Method Article
2D-HELS MS Seq: Um método geral baseado em LC-MS para sequenciamento direto e de novo de misturas de RNA com diferentes modificações de nucleotídeos
Neste Artigo
Resumo
Aqui, descrevemos um protocolo detalhado para um método de sequenciamento baseado em LC-MS que pode ser usado como um método direto para sequenciar RNA curto (<35 nt por execução) sem um intermediário de cDNA e como um método geral para sequenciar diferentes modificações de nucleotídeos em um único estudo com precisão de base única.
Resumo
As abordagens de sequenciamento baseadas em espectrometria de massa (MS) têm se mostrado úteis no sequenciamento direto de RNA sem a necessidade de um intermediário de DNA complementar (cDNA). No entanto, tais abordagens raramente são aplicadas como um método de sequenciamento de RNA de novo , mas usadas principalmente como uma ferramenta que pode auxiliar na garantia de qualidade para confirmar sequências conhecidas de amostras de RNA de fita simples purificadas. Recentemente, desenvolvemos um método de sequenciamento direto de RNA integrando uma estratégia de marcação final hidrofóbica de tempo de retenção de massa bidimensional no sequenciamento baseado em MS (2D-HELS MS Seq). Este método é capaz de sequenciar com precisão sequências de RNA únicas, bem como misturas contendo até 12 sequências de RNA distintas. Além dos quatro ribonucleotídeos canônicos (A, C, G e U), o método tem a capacidade de sequenciar oligonucleotídeos de RNA contendo nucleotídeos modificados. Isso é possível porque a nucleobase modificada possui uma massa intrinsecamente única que pode ajudar em sua identificação e sua localização na sequência de RNA, ou pode ser convertida em um produto com uma massa única. Neste estudo, utilizamos RNA, incorporando dois nucleotídeos modificados representativos (pseudouridina (Ψ) e 5-metilcitosina (m5C)), para ilustrar a aplicação do método para o sequenciamento de novo de um único oligonucleotídeo de RNA, bem como uma mistura de oligonucleotídeos de RNA, cada um com uma sequência diferente e/ou nucleotídeos modificados. Os procedimentos e protocolos descritos aqui para sequenciar esses RNAs modelo serão aplicáveis a outras amostras curtas de RNA (<35 nt) ao usar um sistema LC-MS padrão de alta resolução e também podem ser usados para verificação de sequência de oligonucleotídeos de RNA terapêuticos modificados. No futuro, com o desenvolvimento de algoritmos mais robustos e com melhores instrumentos, esse método poderá permitir o sequenciamento de amostras biológicas mais complexas.
Introdução
Métodos de sequenciamento baseados em espectrometria de massa (MS), incluindo MS de cima para baixo e MS em tandem 1,2,3,4, foram desenvolvidos para sequenciamento direto de RNA. No entanto, técnicas de fragmentação in situ para gerar efetivamente escadas de RNA de alta qualidade em espectrômetros de massa atualmente não podem ser aplicadas ao sequenciamento de novo 5,6. Além disso, não é muito trivial analisar os dados tradicionais de MS unidimensional (1D) para sequenciamento de novo de até mesmo uma sequência de RNA purificada, e seria ainda mais desafiador para o sequenciamento de MS de amostras mistas de RNA 7,8. Portanto, um método de sequenciamento de RNA baseado em cromatografia líquida (LC)-MS bidimensional (2D) foi desenvolvido, incorporando a produção de escadas de tempo de retenção de massa 2D (tR) para substituir as escadas de massa 1D, tornando muito mais fácil identificar os componentes da escada necessários para o sequenciamento de novo de RNAs8. No entanto, o método de sequenciamento de RNA baseado em LC-MS 2D é limitado principalmente ao RNA curto sintético purificado, pois não pode ler uma sequência completa baseada apenas em uma única escada, mas deve contar com duas escadas adjacentes coexistentes (escadas de 5' e 3')8. Mais especificamente, essa abordagem requer leituras bidirecionais de extremidade emparelhada para leitura de nucleobases terminais na região de baixa massa8. A complexidade adicional da leitura de extremidade emparelhada resulta neste método sendo insustentável para o sequenciamento de misturas de RNA porque a confusão é levantada sobre qual fragmento de escada pertence a qual escada para as amostras desconhecidas.
Para superar as barreiras acima mencionadas nas abordagens de sequenciamento de RNA baseadas em MS e ampliar essas aplicações no sequenciamento direto de RNA, duas questões devem ser abordadas: 1) como gerar uma escada de massa de alta qualidade que possa ser usada para ler uma sequência completa, do primeiro nucleotídeo ao último em uma fita de RNA, e 2) como identificar efetivamente cada escada de RNA/massa em um conjunto de dados complexo de MS. Juntamente com a degradação ácida bem controlada, desenvolvemos um novo método de sequenciamento introduzindo uma estratégia de marcação final hidrofóbica (HELS) na técnica de sequenciamento baseada em MS e abordamos com sucesso esses dois problemas adicionando uma etiqueta hidrofóbica nas extremidades 5 '- e / ou 3' dos RNAs a serem sequenciados9. Este método cria uma escada de sequência "ideal" a partir do RNA - cada fragmento de escada deriva da clivagem de RNA específica do local exclusivamente em cada ligação fosfodiéster, e a diferença de massa entre dois fragmentos de escada adjacentes é a massa exata do nucleotídeo ou modificação de nucleotídeo nessa posição 8,9,10. Isso é possível porque incluímos uma etapa de hidrólise ácida altamente controlada, que fragmenta o RNA, em média, uma vez por molécula, antes de ser injetado no instrumento. Como resultado, cada produto de fragmento de degradação é detectado no espectrômetro de massa e todos os fragmentos juntos formam uma escada de sequenciamento 8,9,10. Essa nova estratégia permite a leitura completa de uma sequência de RNA a partir de uma única escada de uma fita de RNA sem leitura de extremidade emparelhada da outra escada do RNA e, adicionalmente, permite o sequenciamento MS de misturas de RNA com várias fitas diferentes que contêm modificações combinatórias de nucleotídeos9. Ao adicionar uma etiqueta na extremidade 5 '- e / ou 3' do RNA, os fragmentos de escada marcados exibem um atraso significativo de tR, o que pode ajudar a distinguir as duas escadas de massa uma da outra e também da região ruidosa de baixa massa. O deslocamento de massat R causado pela adição da etiqueta hidrofóbica facilita a identificação da escada de massa e simplifica a análise de dados para geração de sequências. Além disso, a adição da etiqueta hidrofóbica pode ajudar a identificar a base terminal na fita, evitando que seu fragmento de escada correspondente esteja na regiãoR de baixa massa e ruído devido ao aumento de massa e hidrofobicidade causado pela marcação, permitindo assim a identificação da sequência completa de um RNA a partir de uma única escada; Não são necessárias leituras de extremidade emparelhada. Como resultado, demonstramos anteriormente o sequenciamento bem-sucedido de uma mistura complexa de até 12 fitas distintas de RNA sem o uso de nenhum algoritmo de sequenciamento avançado9, o que abre a porta para o sequenciamento de novo MS de RNA contendo nucleotídeos canônicos e modificados e torna mais viável o sequenciamento de amostras de RNA mistas e mais complexas. De fato, usando 2D-HELS MS Seq, sequenciamos com sucesso uma população mista de amostras de tRNA10 e estamos expandindo ativamente sua aplicação para outras amostras complexas de RNA.
Para facilitar o sequenciamento 2D-HELS MS Seq para sequenciar diretamente uma gama mais ampla de amostras de RNA, aqui nos concentraremos nos aspectos técnicos dessa abordagem de sequenciamento e cobriremos todas as etapas essenciais necessárias ao aplicar a técnica para o sequenciamento direto de amostras de RNA. Exemplos específicos serão usados para ilustrar a técnica de sequenciamento, incluindo sequências sintéticas de RNA único, misturas de várias sequências de RNA distintas e RNAs modificados contendo nucleotídeos canônicos e modificados, como pseudouridina (ψ) e 5-metilcitosina (m5C). Como todos os RNAs contêm ligações fosfodiéster, qualquer tipo de RNA pode ser hidrolisado com ácido para gerar uma escada de sequência ideal para 2D-HELS MS Seq em condições ideais 8,9. No entanto, a detecção de todos os fragmentos de escada de um determinado RNA depende do instrumento. Em um LC-MS padrão de alta resolução (40K), a quantidade mínima de carga para sequenciar uma amostra de RNA curto purificado (<35 nt) é de 100 pmol por corrida. No entanto, mais material é necessário (até 400 pmol por amostra de RNA) quando experimentos adicionais devem ser conduzidos (por exemplo, para distinguir modificações de bases isoméricas que compartilham massas idênticas). O protocolo usado no sequenciamento do modelo de RNAs modificados sintéticos também será aplicável ao sequenciamento de amostras de RNA mais amplas, incluindo amostras de RNA biológico com modificações de base desconhecidas. No entanto, uma quantidade de amostra ainda maior, como 1000 pmol para sequenciamento de tRNA (~ 76 nt) usando um instrumento LC-MS padrão, é necessária para sequenciar o tRNA completo com todas as modificações, e um algoritmo avançado deve ser desenvolvido para seu sequenciamento de novo 10.
Protocolo
1. Oligonucleotídeos de RNA de design
- Projete oligonucleotídeos de RNA sintéticos com diferentes comprimentos (19 nt, 20 nt e 21 nt), incluindo um (RNA # 6) com nucleotídeos canônicos e modificados. ψ é empregado como modelo para modificações que não alteram a massa, o que é um desafio para o sequenciamento de MS porque tem uma massa idêntica à UM5C é escolhido como modelo para modificações que alteram a massa para demonstrar a robustez da abordagem.
RNA # 1: 5'-HO-CGCAUCUGACUGACCAAAA-OH-3'
RNA #2: 5'-HO-AUAGCCCAGUCAGUCUCUACGC-OH-3'
RNA # 3: 5'-HO-AAACCGUUACCAUUACUGAG-OH-3'
RNA # 4: 5'-HO-GCGUACAUCUUCCCCUUUAU-OH-3'
RNA #5: 5'-HO-GCGGAUUUAGCUCAGUUGGGA-OH-3'
RNA # 6: 5'-HO-AAACCGUψACCAUUAm5CUGAG-OH-3 '
- Dissolver cada ARN sintético em água tratada com dietilpirocarbonato (DEPC) livre de nuclease (expresso em H2O tratado com DEPC, salvo indicação em contrário) para obter uma solução reserva de ARN a 100 mM. As soluções de estoque são armazenadas a longo prazo a -20 °C.
- Para evitar possível degradação da amostra de RNA, use suprimentos experimentais sem RNase, incluindo água tratada com DEPC, tubos de microcentrífuga e pontas de pipeta. Limpe frequentemente as superfícies dos suprimentos de laboratório usando lenços de eliminação de RNase.
2. Rotule a extremidade 3' dos RNAs com biotina
- Protocolo de reação em duas etapas (adenilação e ligadura)
- Adicione 1 μL de tampão de reação de adenilação 10x contendo 50 mM de acetato de sódio, pH 6,0, 10 mM MgCl2, 5 mM de diclorodifeniltricloroetano (DTT), 0,1 mM de ácido etilenodiaminotetracético (EDTA), 1 μL de 1 mM de ATP, 1 μL de 100 μM de bifosfato de citidina biotinilado (pCp-biotina), 1 μL de 50 μM de Mth RNA ligase e 6 μL de H2O tratado com DEPC (um volume total de 10 μL) em um tubo de PCR de 0,2 mL de paredes finas sem RNase.
NOTA: Armazene os reagentes a -20 °C antes da reação em duas etapas. Descongele os reagentes à temperatura ambiente e misture bem por vórtice e centrifugação antes de adicionar à reação. - Incubar a reação em uma máquina de PCR a 65 °C por 1 h e inativar a reação a 85 °C por 5 min.
- Conduza a etapa de ligação em um tubo de PCR de 0,2 mL de parede fina e sem RNase contendo 10 μL de solução de reação da etapa anterior, adicionando 3 μL de tampão de reação de RNA ligase 10x T4 contendo 50 mM de tris(hidroximetil)aminometano (Tris)-HCl, pH 7,8, 10 mM MgCl2, 1 mM DTT, 1,5 μL do estoque de amostra de 100 mM do RNA a ser sequenciado, 3 μL de dimetilsulfóxido anidro (DMSO) para atingir 10% (v/v), 1 μL de T4 RNA ligase (10 unidades/μL) e 11,5 μL de H2O tratado com DEPC (para um volume total de 30 mL). Incubar a reação durante a noite a 16 °C numa máquina de PCR.
NOTA: Combine os componentes da reação à temperatura ambiente devido ao alto ponto de congelamento do DMSO (18.45 °C). - Incubar a reação durante a noite a 16 °C.
- Extinguir e purificar a reação por purificação em coluna para remover enzimas e pCp-biotina livre usando Oligo Clean & Concentrator (Zymo Research, Irvine, CA, EUA). Oligo Binding Buffer, DNA Wash Buffer, colunas de rotação e tubos de coleta são fornecidos no kit. Adicione 20 mL de H2O tratado com DEPC à solução de reação para atingir um volume de amostra de 50 mL antes de adicionar o tampão de ligação.
- Adicione 100 mL de tampão de ligação a cada solução de reação. Adicione 400 μL de etanol, misture por pipetagem e transfira a mistura para a coluna. Centrifugue a 10.000 x g por 30 s. Descarte o fluxo.
- Adicione 750 μL de tampão de lavagem de DNA à coluna. Centrifugue a 10.000 x g e velocidade máxima por 30 s e 1 minuto, respectivamente.
- Transfira a coluna para um tubo de microcentrífuga de 1,5 mL. Adicione 15 μL de H2O tratado com DEPC à coluna e centrifugue a 10.000 x g por 30 s para eluir o produto de RNA.
NOTA: As amostras podem ser armazenadas a -20 °C nesta fase até que a próxima etapa seja executada.
- Adicione 1 μL de tampão de reação de adenilação 10x contendo 50 mM de acetato de sódio, pH 6,0, 10 mM MgCl2, 5 mM de diclorodifeniltricloroetano (DTT), 0,1 mM de ácido etilenodiaminotetracético (EDTA), 1 μL de 1 mM de ATP, 1 μL de 100 μM de bifosfato de citidina biotinilado (pCp-biotina), 1 μL de 50 μM de Mth RNA ligase e 6 μL de H2O tratado com DEPC (um volume total de 10 μL) em um tubo de PCR de 0,2 mL de paredes finas sem RNase.
- Protocolo de reação em uma etapa
- Realize uma reação de marcação em uma etapa combinando 2 μL de 150 μM de adenosina-5'-5'-difosfato-{5'-(citidina-2'-O-metil-3'-fosfato-TEG}C-biotina (AppCp-biotina), 3 μL de tampão de reação de ligase 10x, 1,5 μL do estoque de amostra de 100 mM do RNA a ser sequenciado, 3 μL de DMSO anidro para atingir 10% (v/v), 1 μL de RNA ligase T4 (10 unidades/μL), e 19,5 μL de H2O tratado com DEPC (para um volume total de 30 mL) em um tubo de microcentrífuga livre de RNase de 1,5 mL.
- Incubar a reação durante a noite a 16 °C numa máquina de PCR.
- Execute a purificação da coluna conforme descrito acima nas etapas 2.1.5 a 2.1.8.
NOTA: Prepare um tubo de reação separado/exclusivo para cada amostra de RNA (escala de 150 pmol de RNA). A marcação da extremidade 5' do(s) RNA(s) com sulfo-cianina3 (Cy3) ou Cy3 pode ser necessária (por exemplo, para verificação de sequenciamento bidirecional). O método é diferente do da 3'-biotinilação e é descrito em publicação anterior9.
3. Capture amostra de RNA biotinilado em grânulos de estreptavidina
- Ative 200 μL de esferas magnéticas de estreptavidina C1 adicionando 200 μL de 1x tampão P&B (5 mM Tris-HCl, pH 7,5, 0,5 mM EDTA, 1 M NaCl) em um tubo de microcentrífuga livre de RNase de 1,5 mL. Vortex esta solução e coloque-a em um suporte magnético por 2 min. Em seguida, rejeitar o sobrenadante pipetando cuidadosamente a solução.
- Lave os grânulos duas vezes com 200 μL de Solução A (NaOH 0,1 M tratado com DEPC e NaCl 0,05 M tratado com DEPC) e uma vez em 200 μL de Solução B (NaCl 0,1 M tratado com DEPC). Para cada etapa de lavagem, vortex a solução e coloque-a em um suporte magnético por 2 min, seguido de descarte do sobrenadante. Em seguida, adicione 100 μL de 2x tampão P&B (10 mM Tris-HCl, pH 7,5, 1 mM EDTA, 2 M NaCl).
- Adicione 1x tampão P&B à amostra de RNA biotinilado até que o volume seja de 100 μL. Em seguida, adicione esta solução às esferas lavadas armazenadas em 100 μL de 2x tampão P&B. Incubar durante 30 min à temperatura ambiente num agitador de plataforma de balanço a 100 rpm. Coloque o tubo em um suporte magnético por 2 min e descarte o sobrenadante.
- Lave os grânulos revestidos 3 vezes em 1x tampão P&B e meça a concentração final de sobrenadante em cada etapa de lavagem da Nanodrop para análise de recuperação, para confirmar que as moléculas de RNA alvo permanecem nos grânulos.
- Incubar as esferas em EDTA 10 mM, pH 8,2 com 95% de formamida a 65 °C durante 5 min numa máquina de PCR. Mantenha o tubo no suporte magnético por 2 min e colete o sobrenadante (contendo os RNAs biotinilados liberados das esferas de estreptavidina) por pipeta.
NOTA: Esta etapa de separação física antes da degradação ácida é usada apenas para sequenciamento de RNA # 1 na Figura 1c e não é obrigatória para o 2D-HELS MS Seq, uma vez que o marcador de biotina hidrofóbica pode fazer com que os fragmentos de escada marcados com 3 'tenham um tR significativamente atrasado durante a medição de LC-MS, o que pode distinguir claramente os fragmentos de escada 3' marcados dos fragmentos de escada 5' não marcados no gráfico 2D mass-tR .
4. Hidrólise ácida de RNA para gerar escadas de MS para sequenciamento
- Divida cada amostra de RNA em três alíquotas iguais. Por exemplo, divida uma amostra de RNA com um volume de 15 μL de amostra de RNA em três alíquotas de 5 μL.
- Adicionar um volume igual de ácido fórmico para obter 50% (v/v) de ácido fórmico na mistura de reacção 8,9.
- Incubar a reação a 40 °C em uma máquina de PCR, com uma reação por 2 min, uma por 5 min e outra por 15 min, respectivamente.
- Extinguir a degradação ácida congelando imediatamente a amostra em gelo seco após o término de cada reação.
- Use um concentrador centrífugo a vácuo para secar a amostra. A amostra é normalmente completamente seca em 30 minutos e o ácido fórmico é removido junto com o H2O durante o processo de secagem porque o ácido fórmico tem um ponto de ebulição (100,8 ° C) semelhante ao do H2O (100 ° C).
- Suspenda e combine um total de três amostras secas em 20 μL de H2O tratado com DEPC para medição de LC-MS.
NOTA: As amostras podem ser armazenadas a -20 °C nesta fase enquanto aguardam a medição de LC-MS.
5. Converta ψ para aduto CMC-ψ
- Adicione 80 μL de H2O tratado com DEPC em um tubo de microcentrífuga livre de RNase de 1,5 mL contendo 0,0141 g de N-ciclohexil-Nʹ-(2-morfolinoetil)-carbodiimida meto-p-toluenossulfonato (CMC) e 0,07 g de ureia. Adicione 10 μL do estoque de amostra de 100 μM do RNA a ser sequenciado, 8 μL de tampão bicina 1 M (pH 8,3) e 1,28 μL de EDTA 0,5 M. Adicione H2O tratado com DEPC para atingir um volume total de 160 μL. As concentrações finais são 0,17 M CMC, 7 M de ureia e 4 mM de EDTA em 50 mM de bicina (pH 8,3)11.
NOTA: Este protocolo é aplicável a uma única sequência de RNA sintético ou misturas de RNA. - Divida a solução de reação de 160 μL em quatro alíquotas iguais em tubos de PCR de 0,2 mL de paredes finas e sem RNase e incube a 37 °C por 20 min em uma máquina de PCR.
NOTA: 50 μL por tubo é o volume máximo de reação que pode ser usado em uma máquina de PCR. - Extinguir cada reação com 10 μL de acetato de sódio 1,5 M e EDTA 0,5 mM (pH 5,6).
- Execute a purificação da coluna com quatro colunas de rotação paralelas para remover reagentes excessivos de acordo com o procedimento descrito nas etapas 2.1.5-2.1.8. Dissolva o produto purificado em 15 μL de H2O tratado com DEPC em cada tubo de microcentrífuga sem RNase de 1,5 mL.
- Transfira o produto purificado para quatro tubos de PCR de 0,2 mL de paredes finas e sem RNase. Adicione 20 μL de tampão 0,1 M Na2CO3 (pH 10,4) em cada 15 μL de produto purificado e adicione H2O tratado com DEPC para fazer um volume final de 40 μL para cada tubo de reação (no total quatro tubos). Incubar a reação a 37 °C durante 2 h numa máquina de PCR.
- Extinguir e purificar a reação por purificação da coluna com quatro colunas de centrifugação paralelas, conforme descrito na etapa 2.1.5. Eluir o produto convertido em CMC-ψ em um tubo de microcentrífuga livre de RNase de 1,5 mL, cada um com 15 μL de H2O tratado com DEPC.
- Combine a amostra purificada convertida em CMC-ψ de quatro tubos de coleta em um tubo. Realize a degradação do ácido fórmico 50% (v/v) de acordo com os procedimentos descritos nas etapas 4.1-4.6 para gerar escadas de MS para sequenciamento.
6. Medição LC-MS
- Prepare fases móveis para medição LC-MS. A fase móvel A é hexafluoro-2-propanol 25 mM com diisopropilamina 10 mM em água de grau LC-MS; a fase móvel B é o metanol.
- Transfira a amostra para o frasco de amostra LC-MS para análise. Cada volume de injeção de amostra é de 20 μL contendo 100-400 pmol de RNA.
- Use as seguintes condições de LC: temperatura da coluna de 35 °C, vazão de 0,3 mL/min; um gradiente linear de 2–20% da fase móvel B ao longo de 15 minutos, seguido por uma etapa de lavagem de 2 minutos com 90% da fase móvel B.
NOTA: Para rótulos finais mais hidrofóbicos, como Cy3 e sulfo-Cy3, conforme mencionado na Seção 2, uma porcentagem maior de solvente orgânico pode ser necessária para a eluição da amostra (ou seja, um gradiente semelhante pode ser usado, mas com uma faixa percentual aumentada da fase móvel B). Por exemplo, 2–38% da fase móvel B em 30 minutos com uma etapa de lavagem de 2 minutos com 90% da fase móvel B. - Separe e analise amostras em um espectrômetro de massa Agilent Q-TOF (Quadrupole Time-of-Flight) acoplado a um sistema LC equipado com um amostrador automático e um sistema MS HPLC (Cromatografia Líquida de Alta Eficiência). A coluna LC é uma coluna C18 de 50 mm x 2,1 mm com um tamanho de partícula de 1,7 μm. Use as seguintes configurações do MS: modo de íons negativos; alcance, 350 m/z a 3200 m/z; taxa de varredura, 2 espectros/s; fluxo de gás de secagem, 17 L/min; temperatura do gás de secagem, 250 °C; pressão do nebulizador, 30 psig; tensão capilar, 3500 V; e tensão do fragmentor, 365 V. Observe que esses parâmetros são específicos para o tipo ou modelo de espectrômetro de massa que está sendo usado.
- Adquira dados com o software de aquisição Agilent MassHunter. Use o fluxo de trabalho de extração de recursos moleculares (MFE) da Agilent para extrair informações compostas, incluindo massa, tempo de retenção, volume (a abundância de MFE para as respectivas espécies de íons) e índice de qualidade, etc. Use as seguintes configurações de MFE: "formato de dados centróide, pequenas moléculas (cromatográficas), pico com altura ≥ 100, até um máximo de 1000, índice de qualidade ≥ 50".
NOTA: Otimize as configurações de MFE para extrair o maior número possível de compostos potenciais, até um máximo de 1000, com índices de qualidade de ≥ 50.
7. Automatizar a geração de sequências de RNA por um algoritmo computacional
NOTA: Este procedimento é mostrado apenas para o RNA # 1 na Figura 1c.
- Classificar os compostos extraídos MFE por ordem decrescente de volume (intensidade de pico) e tR. Execute a pré-seleção de dados por meio de 1) configuração tR de 4 a 10 min para selecionar os fragmentos de RNA marcados pela biotina, uma vez que os tRs dos componentes da escada de massa marcados com biotina são deslocados para esta janela tR (4 min a 10 min) e 2) usando uma ordem de magnitude maior de compostos de entrada do que o número de fragmentos de escada para computação de algoritmo para reduzir a quantidade de dados com base no volume. Por exemplo, para um RNA de 20 nt, 20 componentes de escadaR de massa t marcados serão necessários para o sequenciamento do RNA de 20 nt e, portanto, 200 compostos do arquivo de dados MFE serão selecionados com base no volume. Observe que a janela tR pode ser diferente quando um tipo ou modelo diferente de espectrômetro de massa é usado.
- Realize o processamento de dados e a geração de sequências de RNA # 1 usando uma versão revisada de um algoritmo publicado8. Os códigos-fonte do algoritmo revisado são descritos anteriormente (https://academic-oup-com.remotexs.ntu.edu.sg/nar/article/47/20/e125/5558343#supplementary-data)9.
- Além de automatizar a geração de sequências usando o algoritmo, calcule manualmente as diferenças de massa entre dois componentes de escada adjacentes para chamadas de base. Todas as bases do RNA podem ser chamadas manualmente e combinadas com as teóricas no banco de dados de nucleotídeos e modificações de RNA8; assim, a sequência completa da fita de RNA pode ser lida com precisão manualmente, o que é usado para confirmar a precisão da sequência relatada pelo algoritmo lida. Mais estruturas de modificações de RNA podem ser encontradas em bancos de dados de modificação de RNA12, e suas massas teóricas correspondentes são obtidas por ChemBioDraw. Nas Tabelas S1–S2, a diferença de massa de ppm (partes por milhão) é mostrada ao comparar a massa observada com sua massa teórica para um componente específico da escada, e um valor inferior a 10 ppm é considerado uma boa correspondência para cada chamada de base.
8. Sequenciamento de misturas de RNA
- Rotule uma mistura de cinco fitas de RNA (RNA # 1 a # 5) em suas extremidades 3 'com A(5')pp(5')Cp-TEG-biotina usando um protocolo de uma etapa descrito na etapa 2.2. Em um volume total de 150 μL de solução de reação, adicione 15 μL de tampão de reação de RNA ligase 10x T4, 1,5 μL de cada fita de RNA (estoque de 100 μM de RNA # 1 a # 5, respectivamente, para um volume total de 7,5 μL), 10 μL de 150 μM A (5 ') pp (5') Cp-TEG-biotina, 15 μL de DMSO anidro, 5 μL de T4 RNA ligase (10 unidades/μL) e 97,5 μL de H2O tratado com DEPC. Distribua igualmente a solução de reação em cinco alíquotas. Cada tubo de microcentrífuga sem RNase contém 30 μL de solução de reação.
- Incubar a reação durante a noite a 16 °C numa máquina de PCR.
- Execute a purificação da coluna de acordo com o procedimento descrito nas etapas 2.1.5-2.1.8 com cinco colunas de centrifugação paralelas. Eluir uma amostra de mistura de fitas de 5 RNA 3'-biotiniladas (mistura de RNA # 1 a # 5) para um tubo de microcentrífuga livre de RNase de 1,5 mL, cada um com 15 μL de H2O tratado com DEPC.
- Combine as amostras de mistura purificada dos cinco tubos de coleta em um tubo. Efectuar a degradação do ácido fórmico de acordo com o procedimento descrito no ponto 4.
- Meça amostras por LC-MS conforme descrito na Seção 6 e analise os dados usando o software de análise de dados com configurações otimizadas de MFE para extrair dados contendo massa, tR e volume, conforme descrito na etapa 6.5. O algoritmo típico de processamento e chamada de base não é aplicado devido ao aumento significativo da complexidade dos dados resultante da mistura. Todas as bases no RNA da amostra mista são chamadas manualmente em um método semelhante à Seção 7.3 e combinam bem com as teóricas no banco de dados de nucleotídeos e modificação de RNA8, portanto, as sequências completas de todas as cinco fitas de RNA na amostra mista são lidas com precisão. Nas Tabelas S7–S11, todas as informações são listadas, incluindo massa observada, tR, volume, índice de qualidade e diferença de massa ppm.
Resultados
Introduzindo uma etiqueta de biotina na extremidade 3' do RNA para produzir escadasR de massa facilmente identificáveis. O fluxo de trabalho da abordagem 2D-HELS MS Seq é demonstrado na Figura 1a. O marcador hidrofóbico de biotina introduzido na extremidade 3' do RNA (ver Seção 2) aumenta as massas e tRs dos componentes da escada marcados com 3' quando comparados aos de suas contrapartes não marcadas. Assim, a curva da escada 3' é deslocada para valores maiores do eixo y (devido ao aumento do tRs) e deslocada para valores maiores do eixo x (devido ao aumento das massas) no gráfico 2D massa-tR . A Figura 1b mostra o protocolo de preparação da amostra, incluindo a introdução de uma etiqueta de biotina na extremidade 3' do RNA para 2D-HELS MS Seq. A Figura 1c demonstra a separação da escada 3' da escada 5' e outros fragmentos indesejados em um gráfico 2D mass-tR com base em mudanças sistemáticas em tRdos fragmentos de escada mass-tR marcados com 3'-biotina do RNA # 1. A curva de escada 3' sozinha fornece uma sequência completa de RNA # 1, e a curva de escada 5' que não mostra um deslocamento tR fornece a sequência reversa, mas requer emparelhamento final para ler a base terminal8. Com esta estratégia de 2D-HELS, o emparelhamento final não é necessário como relatado anteriormente e toda a sequência de RNA pode ser lida completamente a partir de apenas uma curva de escada marcada8. Como tal, é possível sequenciar amostras mistas contendo múltiplos RNAs, por exemplo, duas fitas de RNA de comprimentos diferentes (RNA # 1 e RNA # 2, 19 nt e 20 nt, respectivamente) com um marcador de 5'-biotina em cada RNA (Figura 1d).
Convertendo ψ em seu aduto CMC-ψ para 2D-HELS MS Seq. ψ é uma modificação de nucleotídeo difícil para sequenciamento baseado em MS porque tem a mesma massa que a uridina (U). Para diferenciar essas duas bases uma da outra, tratamos o RNA com CMC, que converte um ψ em um aduto ψ CMC (ver Seção 5). O aduto tem uma massa diferente de U e pode ser diferenciado no 2D-HELS MS Seq. A Figura 2a mostra o perfil de HPLC do produto bruto da reação convertendo ψ em seu aduto CMC no RNA # 6. Ao integrar seus picos de UV, calculamos a conversão percentual e 42% ψ é convertido em seu aduto CMC-ψ após o processo ilustrado na Seção 5. Após a degradação ácida e a medição de LC-MS, adquirimos manualmente a sequência com base em escadas não convertidas em CMC e escadas convertidas em CMC identificadas a partir dos dados processados por algoritmos 8,9. Uma curva vermelha se ramifica da curva cinza a partir de ψ na posição 8 no RNA # 6 (Figura 2b) devido à conversão parcial de ψ para o aduto CMC-ψ. Devido à massa e hidrofobicidade do CMC, essa conversão resulta em um aumento de 252,2076 Dalton na massa e um aumento significativo em tR para cada componente de escada contendo aduto CMC-ψ quando comparado ao seu equivalente não convertido. Assim, uma mudança dramática começando na posição 8 no RNA # 6 pode ser observada no gráfico 2D mass-tR, indicando que a posição 8 é de fato uma ψ no RNA # 6.
Sequenciamento de misturas de RNA. Uma mistura de cinco fitas de RNA diferentes é sequenciada pela abordagem 2D-HELS MS Seq com marcação de extremidade 3' (ver Seção 8). A preocupação com o sequenciamento de RNAs mistos é que várias curvas de escada no gráfico 2D mass-tR podem se sobrepor umas às outras quando todas compartilham os mesmos pontos de partida (a etiqueta hidrofóbica no gráfico 2D mass-tR). No entanto, a chamada de base é feita uma a uma, cada uma com base em uma diferença de massa entre dois fragmentos de escada adjacentes nos dados do MFE. A chamada de base correta pode ser feita desde que cada diferença de massa corresponda bem (uma diferença PPM MS < 10) com uma das massas teóricas de nucleotídeos canônicos ou modificados no conjunto de dados 8,9. Na análise das amostras de RNA multiplexadas, o algoritmo típico de processamento e chamada de base usado nas Figuras 1 e 2 não é usado principalmente devido ao aumento significativo da complexidade dos dados resultante da mistura. Essas sequências são chamadas manualmente por meio do cálculo da diferença de massa entre dois fragmentos de escada de massa adjacentes e comparando-a com a massa teórica do nucleotídeo no conjunto de dados9. Qualquer base correspondente com um PPM em massa <10 é escolhida como a identidade da base nesta posição. Com esse cálculo manual base a base para chamada de base, todas as sequências na mistura são sequenciadas com precisão. O software OriginLab é usado para reconstruir um gráfico 2D mass-tR, no qual o tR inicial para cada sequência é normalizado sistematicamente para melhor visualização de cinco sequências de RNA diferentes (Figura 3). Sem essa normalização, os códigos de letras (ou seja, A, C, G e U) para as sequências de todos os cinco RNAs seriam agrupados no gráfico (Figura S1), resultando em menos facilidade de visualização em comparação com o relatado na Figura 3. Os resultados do sequenciamento demonstram que a abordagem 2D-HELS MS Seq não se limita apenas ao sequenciamento de RNAs purificados de fita simples, mas também, mais importante, misturas de RNA com múltiplas fitas de RNA. Algoritmos estão atualmente em desenvolvimento para automatizar o processo de chamada de base e geração de sequência.
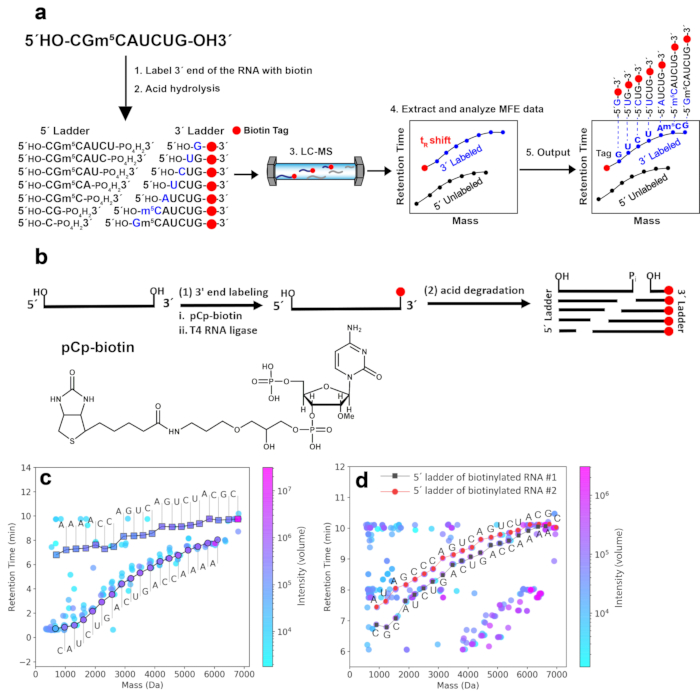
Figura 1. 2D-HELS MS Seq de amostras representativas de RNA. (a) Fluxo de trabalho para 2D-HELS MS Seq. As principais etapas incluem 1) marcação hidrofóbica de RNA a ser sequenciado, 2) hidrólise ácida, 3) medição de LC-MS, 4) extração e análise de dados MFE e 5) geração de sequência por meio de algoritmos ou cálculo manual. (b) Protocolo de preparação de amostras, incluindo a introdução de uma etiqueta de biotina na extremidade 3' do RNA para 2D-HELS MS Seq. (c) Separação da escada 3' da escada 5' e outros fragmentos indesejados em um gráfico de tempo de retenção de massa 2D (tR) com base em mudanças sistemáticas em tRs de fragmentos de escada de massa tR marcados com 3'-biotina do RNA # 1 (19 nt). As sequências são de novo e lidas automaticamente diretamente por um algoritmo de chamada de base9. (d) Sequenciamento simultâneo de RNA # 1 e RNA # 2 marcados com 5'-biotina, 19 nt e 20 nt, respectivamente. Os métodos para introduzir uma marca de biotina na extremidade 5' do RNA são diferentes dos da 3'-biotinilação e podem ser encontrados no protocolo publicado anteriormente9. A extremidade 5' de dois RNAs (RNA # 1 e RNA # 2) é biotinilada e suas escadas 5'-biotiniladas podem ser facilmente identificadas; ambas as escadas 5'-biotiniladas são facilmente separadas de suas escadas 3' não rotuladas no gráfico 2D mass-tR após LC-MS, porque os componentes da escada biotinilada têm os maiores deslocamentos tR devido à hidrofobicidade da biotina, enquanto os componentes da escada não marcados estão na região tR inferior. Embora as escadas 5' e as escadas 3' coexistam, elas não interferem na interpretação da sequência de duas fitas mistas de RNA. Cada sequência desses dois RNAs é adquirida manualmente de escadas 5'-biotiniladas com base nos dados processados por algoritmos computacionais 8,9. Esta figura foi modificada de Zhang et al.9. Clique aqui para ver uma versão maior desta figura.

Figura 2. Convertendo pseudouridina (ψ) em seu aduto para 2D-HELS MS Seq. (a) Perfil de HPLC do produto bruto da reação convertendo ψ em seu aduto CMC em um RNA de 20 nt (RNA # 6) que contém um ψ. (b) Sequenciamento de um RNA contendo ψ # 6. A conversão do ψ para os adutos CMC-ψ (ψ*) resulta em um aumento de massa de 252,2076 Dalton e um aumento significativo em tR devido à sua massa e hidrofobicidade do CMC. Assim, uma mudança dramática começando na posição de 8 pode ser observada no gráficoR de massa-t, indicando que esta é uma ψ na posição de 8 na sequência de RNA. As sequências são adquiridas manualmente com base nos dados processados por algoritmos computacionais 8,9. Esta figura foi modificada de Zhang et al.9. Clique aqui para ver uma versão maior desta figura.

Figura 3. Sequenciamento de misturas de RNA contendo cinco RNAs distintos. Uma biotina é usada para marcar cada RNA em sua extremidade 3' antes do 2D-HELS MS Seq. Para cada sequência, os valores iniciais de tR são normalizados sistematicamente para iniciar em intervalos de 7 minutos para facilitar a visualização. As diferenças absolutas entre o valor tR inicial e os tRs subsequentes permanecem inalteradas para cada um dos cinco RNAs e, portanto, é mais fácil visualizar cada um deles no mesmo gráfico. Todas as bases são identificadas calculando manualmente as diferenças de massa de dois componentes de escada adjacentes e combinando-as com as diferenças teóricas de massa no nucleotídeo de RNA e no banco de dados de modificação8; os gráficos da Figura 3 são reconstruídos usando o OriginLab com base em dados manuais de chamada de base e sequenciamento (consulte a seção de sequenciamento de misturas de RNA em resultados representativos). A figura 2D massa-tR dos cinco RNAs mistos sem normalização tR é mostrada na Figura S1. Clique aqui para ver uma versão maior desta figura.
Figura S1. Clique aqui para baixar este arquivo.
Discussão
Ao contrário da fragmentação de MS baseada em tandem, a hidrólise ácida altamente controlada é usada na abordagem 2D-HELS MS Seq para fragmentar o RNA antes da análise com um espectrômetro de massa 9,10. Como resultado, cada fragmento degradado por ácido pode ser detectado pelo instrumento, formando o equivalente a uma escada de sequenciamento. Em condições ideais, este método cria uma escada de sequência "ideal" do RNA por meio de, em média, uma clivagem de RNA específica do local por molécula exclusivamente em uma ligação fosfodiéster 8,9,10. Depois que cada fragmento degradado é medido pelo espectrômetro de massa em uma única corrida, a diferença de massa entre dois fragmentos de escada adjacentes corresponde à massa exata do nucleotídeo de RNA ou modificação nessa posição. Cada modificação de RNA tem uma massa intrínseca única que pode ajudar a identificá-la e localizá-la no RNA, ou pode ser convertida em uma com uma massa única. Assim, em teoria, este método pode relatar a identidade e localização de nucleotídeos canônicos e modificados para sequenciamento de novo e direto de qualquer RNA. No entanto, diferentes escadas de sequência podem se sobrepor, complicando a análise de dados de EM e dificultando o sequenciamento de RNA por EM na prática.
Um dos benefícios da etiqueta 3'-hidrofóbica é que ela supera um grande desafio em qualquer método de fragmentação, ou seja, que cada molécula de RNA deve ser clivada em exatamente dois fragmentos (e idealmente não mais): um fragmento contendo a extremidade 5' original e o outro contendo a extremidade 3' original do RNA. Portanto, cada evento de clivagem produz dois fragmentos, produzindo duas escadas - uma medida a partir da extremidade 5' e a outra a partir da extremidade 3'. Sempre há ambigüidade na determinação de qual pico de MS pertence a qual escada. Isso se torna mais problemático em uma mistura de vários RNAs diferentes, devido à geração de um grande número de escadas de sequência sobrepostas. No entanto, como todos os fragmentos de escada das extremidades 3' são marcados com uma etiqueta hidrofóbica, eles exibem tRs muito mais longos (Figura 1a). Como resultado, podemos obter escadas claras e inequívocas nos dados 2D mass-tR derivados exclusivamente do RNA marcado com 3'. Notavelmente, estamos otimizando abordagens para marcar seletivamente a extremidade 5 '- ou 3' de qualquer RNA usando diferentes métodos de conjugação química9. Também podemos realizar o sequenciamento bidirecional, que não é usado para determinar a(s) base(s) terminal(is) aqui, mas é usado para fornecer informações de sequência idênticas duas vezes ao ler nas direções 5' e 3' (ou seja, verificação de sequenciamento bidirecional) e, assim, melhorar ainda mais a precisão do sequenciamento.
Para o sequenciamento de novo de amostras de RNA desconhecidas, especialmente para amostras biológicas complexas, é necessário um algoritmo geral e robusto para processar uma grande quantidade de dados de LC-MS para geração de sequência de maneira precisa e eficiente, que recentemente se tornou disponível por meio de outros trabalhos publicados10. Embora esses algoritmos tenham sido usados para sequenciamento de amostras mais complicadas10, neste estudo, realizamos chamadas manuais de base para geração de sequências, salvo indicação em contrário. Nosso objetivo é cobrir todas as etapas-chave do 2D-HELS MS Seq e gostaríamos de ilustrar o processo durante o qual, mesmo sem usar algoritmos de sequenciamento adicionais, ainda podemos ler manualmente as sequências do RNA a ser sequenciado. Para facilitar a visualização e identificar mais rapidamente os fragmentos de escada necessários para o sequenciamento no gráfico 2D mass-tR , os arquivos MFE de cada execução de LC-MS são processados por uma versão revisada de um algoritmo publicado8 antes de ler suas sequências, salvo indicação em contrário. O algoritmo publicado não pode ser usado diretamente para ler as sequências dos dados LC-MS, mas parte de sua função ainda pode ser usada para processar os dados - agrupar hierarquicamente os adutos de massa por meio desse algoritmo aumentará a intensidade de cada componente da escada, o que, por sua vez, reduz a complexidade dos dados, especialmente na região crucial onde as leituras de sequência são geradas8, 9.
Uma das etapas cruciais durante a preparação da amostra para 2D-HELS MS Seq resulta na melhoria da eficiência da marcação de extremidade hidrofóbica de RNA. Uma alta eficiência de marcação pode ajudar a reduzir a quantidade de amostra de RNA necessária para gerar sinais de MS dos quais os dados de sequência dependem. Para aumentar a eficiência da marcação, empregamos novas estratégias de marcação, incluindo o uso de AppCp-biotina ativada para evitar a etapa de adenilação ao marcar a extremidade 3' do RNA. O rendimento da reação para marcar a extremidade 3' de um RNA de 19 nt com biotina (consulte a etapa 2.2) pode ser melhorado de 60% para ~ 95% 9 usando este método de uma etapa. Com a marcação eficiente, somos capazes de sequenciar uma amostra mista contendo até 12 RNAs distintos, conforme descrito anteriormente9. Neste estudo, usamos uma mistura de cinco RNAs como exemplo representativo para ilustrar o processo de sequenciamento. Também detectamos todos os fragmentos de escada necessários para um sequenciamento preciso e lemos as sequências completas de cada uma das cinco sequências de RNA na mistura. Maior eficiência de rotulagem não apenas ajuda a minimizar a quantidade de carga de amostra, mas também auxilia na redução significativa da complexidade dos dados durante a análise de dados downstream para geração de sequência. Novas reações estão atualmente em desenvolvimento para alcançar rendimento quantitativo na marcação de RNAs nas extremidades 5' e 3'.
Ao sequenciar o RNA # 1, conforme mostrado na Figura 1c, as etapas de captura e liberação de estreptavidina são usadas para separar fisicamente o RNA biotinilado # 1 antes da degradação do ácido (ver Seção 3). Isso remove uma pequena porção de RNA não marcado e, subsequentemente, resulta em maior facilidade de identificação visual das escadas de massa marcadas no gráfico 2D mass-tR . No entanto, a etapa de separação física não é obrigatória porque os fragmentos de escada de RNA biotinilado têm tRs atrasados / mais longos devido à hidrofobicidade da etiqueta de biotina quando comparados aos seus equivalentes não marcados. Além disso, a chamada de base não depende da separação física, mas depende das diferenças de massa dos componentes adjacentes da escada de massa, portanto, a chamada de base correta pode ser alcançada desde que as diferenças de massa de dois componentes adjacentes da escada correspondam bem com as massas correspondentes de um nucleotídeo específico ou modificação no nucleotídeo de RNA e na base de data de modificação8. Um algoritmo computacional está atualmente em desenvolvimento para automatizar a chamada de base e a geração de sequências.
As configurações de MFE durante a exportação de dados LC-MS originais (no tipo de arquivo .d) para arquivos de planilha são altamente cruciais para o processamento de dados e a geração de sequência subsequente (consulte a Seção 6.5). Por exemplo, testamos a configuração MFE "pico com altura" em uma faixa de 100 a 1000 e notamos que a configuração de 100 pode nos fornecer 2 vezes mais compostos do que os da configuração 1000. Para evitar a falta de componentes da escada, podemos ajustar a configuração MFE durante o fluxo de trabalho de sequenciamento. Essa configuração provavelmente depende da resolução de massa do instrumento, da quantidade de fragmentos de escada de massa e da complexidade dos dados. Além disso, é importante usar o conjunto de dados do centróide e a configuração do tipo cromatográfico para pequenas moléculas. O índice de qualidade pode variar de 50% a 100% com base na qualidade dos dados.
O instrumento LC-MS que usamos no estudo tem uma resolução de massa superior de ~ 40K, limitando o método a sequenciar apenas RNA com menos de 35 bases de comprimento. No entanto, o comprimento exato de leitura desse método depende do instrumento; Instrumentos mais avançados com maior poder de resolução podem levar a um comprimento de leitura mais longo. Da mesma forma, o rendimento, ou seja, quantas sequências de RNA podem ser sequenciadas simultaneamente em uma única execução de LC-MS, ainda precisa ser explorado, embora tenhamos sequenciado manualmente uma mistura de amostra de RNA de até 12 fitas de RNA distintas, mesmo sem o uso de qualquer algoritmo9. Com o fluxo de trabalho atual, ~ 100 pmol de RNA curto (<35 nt) é necessário para cada execução de LC-MS. A quantidade de carga aumenta quando experimentos adicionais são necessários: para diferenciar modificações de nucleotídeos isoméricos, normalmente são necessários até 400 pmol de RNA. Para sequenciar tRNA específico como tRNAPhe, ~ 1000 pmol de amostra podem ser necessários para sequenciamento e análise de modificação. No entanto, esperamos que as quantidades de carga de amostra necessárias sejam reduzidas em instrumentos LC-MS com maior sensibilidade. Com melhorias na eficiência de marcação de amostras, algoritmo de sequenciamento e sensibilidade e resolução do instrumento, esperamos que nosso método seja aplicável a uma gama mais ampla de amostras de RNA, especialmente aquelas com várias modificações de RNA.
Divulgações
Os autores depositaram uma patente provisória relacionada à tecnologia discutida neste manuscrito.
Agradecimentos
Os autores reconhecem a doação R21 do National Institutes of Health (1R21HG009576) para S. Z. e W. L. e as bolsas de Apoio Institucional para Pesquisa e Criatividade do New York Institute of Technology (NYIT) para S. Z., que apoiaram este trabalho. Os autores gostariam de agradecer ao estudante de doutorado Xuanting Wang (Columbia University) por ajudar na criação de figuras, e agradecer ao Prof. Michael Hadjiargyrou (NYIT), Prof. Jingyue Ju (Columbia University), Drs. James Russo, Shiv Kumar, Xiaoxu Li, Steffen Jockusch e outros membros do Ju lab (Columbia University), Dr. Yongdong Wang (Cerno Bioscience), Meina Aziz (NYIT) e Wenhao Ni (NYIT) por discussões e sugestões úteis para nosso manuscrito.
Materiais
| Name | Company | Catalog Number | Comments |
| 5' DNA Adenylation kit | New England Biolabs | E2610S | 50uM concentration |
| 6550 Q-TOF mass spectrometer | Agilent Technologies | 5991-2116EN | Coupled to a 1290 Infinity LC system |
| A(5´)pp(5´)Cp-TEG-biotin-3´ | ChemGenes | 91718 | HPLC purified |
| ATPγS | Sigma-Aldrich | 11162306001 | Lithium salt |
| Bicine | Sigma-Aldrich | B8660 | BioXtra, ≥99% (titration) |
| Biotin maleimide | Vector Laboratories | SP-1501 | Long arm |
| C18 column | Waters | 186003532 | 50 mm × 2.1 mm Xbridge C18 column with a particle size of 1.7 μm |
| Centrifugal Vacuum Concentrator | Labconco | Refrig 115v/60hz 7310022 | Labconco CentriVap |
| ChemBioDraw | PerkinElmer | ChemDraw Prime | Generate a chemical structure and property data of structures & fragments |
| CMC (N-cyclohexyl-N?-(2-morpholinoethyl)-carbodiimide metho-p-toluenesulfonate) | Sigma-Aldrich | 2491-17-0 | 95% Purifiy |
| Cyanine3 maleimide (Cy3) | Lumiprobe | 11080 | Water insoluble |
| DEPC-treated water | Thermo Fisher Scientific | AM9906 | Autoclaved, certified nuclease-free |
| Diisopropylamine (DIPA) | Thermo Fisher Scientific | 108-18-9 | 99% Alfa Aesar |
| DMSO | Sigma-Aldrich | 276855 | Anhydrous dimethyl sulfoxide, 99.9% |
| EDTA | Sigma-Aldrich | E6758 | Anhydrous, crystalline, BioReagent, suitable for cell culture |
| Formic acid | Merck | 64-18-6 | 98-100%, ACS reag, Ph Eur |
| Hexafluoro-2-propanol (HFIP) | Thermo Fisher Scientific | 920-66-1 | 99% Acros Organics |
| LC-MS sample vials | Thermo Fisher Scientific | C4000-11 | Plastic screw thread vials |
| LC-MS vial caps | Thermo Fisher Scientific | C5000-54A | Autosampler vial screw thread caps |
| Na2CO3 buffer | Sigma-Aldrich | 88975 | BioUltra, >0.1 M Na2CO3, >0.2 M NaHCO3 |
| Oligo Clean & Concentrator | Zymo Research | D4060 | Spin column |
| OriginLab | OriginLab | OriginPro | Data analysis and graphing software |
| pCp-biotin | TriLink BioTechnologies | NU-1706-BIO | 20 ul (1 mM) |
| RNA #1--#6 | Integrated DNA Technologies | Custom RNA oligos | 19nt-21nt single-stranded RNAs, used without further purification |
| Rocking platform shaker | VWR | Orbital Shaker Standard 1000 | Speed Range 40 to 300 rpm |
| Streptavidin magnetic beads | Thermo Fisher Scientific | 88816 | Binding approx. 55ug biotinylated rabbit lgG per mg of beads |
| Sulfonated Cyanine3 maleimide | Lumiprobe | 11380 | Water soluble |
| T4 DNA ligase 1 | New England Biolabs | M0202S | 400 units/uL |
| T4 polynucleotide kinase | Sigma-Aldrich | T4PNK-RO | From phage T4 am N81 pse T1 infected Escherichia coli BB |
| Tris-HCl buffer | Sigma-Aldrich | T6455 | Tris-HCl Buffer, pH 10, 10×, Antigen Retriever |
| Urea | Sigma-Aldrich | 81871 | Urea for synthesis. CAS No. 57-13-6, EC Number 200-315-5. |
Referências
- Addepalli, B., Venus, S., Thakur, P., Limbach, P. A. Novel ribonuclease activity of cusativin from Cucumis sativus for mapping nucleoside modifications in RNA. Analytical and Bioanalytical Chemistry. 409 (24), 5645-5654 (2017).
- Gao, H., Liu, Y., Rumley, M., Yuan, H., Mao, B. Sequence confirmation of chemically modified RNAs using exonuclease digestion and matrix-assisted laser desorption/ionization time-of-flight mass spectrometry. Rapid Communications in Mass Spectrometry. 23 (21), 3423-3430 (2009).
- McLuckey, S. A., Van Berkel, G. J., Glish, G. L. Tandem mass spectrometry of small, multiply charged oligonucleotides. Journal of The American Society for Mass Spectrometry. 3 (1), 60-70 (1992).
- Fountain, K. J., Gilar, M., Gebler, J. C. Analysis of native and chemically modified oligonucleotides by tandem ion-pair reversed-phase high-performance liquid chromatography/electrospray ionization mass spectrometry. Rapid Communications in Mass Spectrometry. 17 (7), 646-653 (2003).
- Taucher, M., Breuker, K. Characterization of modified RNA by top-down mass spectrometry. Angewandte Chemie International Edition in English. 51 (45), 11289-11292 (2012).
- Kellner, S., Burhenne, J., Helm, M. Detection of RNA modifications. RNA Biology. 7 (2), 237-247 (2010).
- Thomas, B., Akoulitchev, A. V. Mass spectrometry of RNA. Trends in Biochemical Sciences. 31 (3), 173-181 (2006).
- Bjorkbom, A., et al. Bidirectional direct sequencing of noncanonical RNA by two-dimensional analysis of mass chromatograms. Journal of the American Chemical Society. 137 (45), 14430-14438 (2015).
- Zhang, N., et al. A general LC-MS-based RNA sequencing method for direct analysis of multiple-base modifications in RNA mixtures. Nucleic Acids Research. 47 (20), 125 (2019).
- Zhang, N., et al. Direct sequencing of tRNA by 2D-HELS-AA MS Seq reveals its different isoforms and dynamic base modifications. ACS Chemical Biology. 15 (6), 1464-1472 (2020).
- Bakin, A., Ofengand, J. Four newly located pseudouridylate residues in Escherichia coli 23S ribosomal RNA are all at the peptidyltransferase center: analysis by the application of a new sequencing technique. Biochemistry. 32 (37), 9754-9762 (1993).
- Cantara, W. A., et al. The RNA Modification Database, RNAMDB: 2011 update. Nucleic Acids Research. 39 (Database issue), D195-D201 (2011).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados