Method Article
2D-HELS MS Seq: Un método general basado en LC-MS para la secuenciación directa y de novo de mezclas de ARN con diferentes modificaciones de nucleótidos
En este artículo
Resumen
Aquí, describimos un protocolo detallado para un método de secuenciación basado en LC-MS que se puede utilizar como método directo para secuenciar ARN corto (<35 nt por ejecución) sin un intermedio de ADNc, y como método general para secuenciar diferentes modificaciones de nucleótidos en un solo estudio con precisión de una sola base.
Resumen
Se ha demostrado que los enfoques de secuenciación basados en espectrometría de masas (MS) son útiles en la secuenciación directa de ARN sin necesidad de un intermediario de ADN complementario (ADNc). Sin embargo, estos enfoques rara vez se aplican como un método de secuenciación de ARN de novo , sino que se utilizan principalmente como una herramienta que puede ayudar en el aseguramiento de la calidad para confirmar secuencias conocidas de muestras de ARN monocatenario purificadas. Recientemente, desarrollamos un método de secuenciación directa de ARN mediante la integración de una estrategia de etiquetado final hidrofóbico en tiempo de retención de masa en 2 dimensiones en secuenciación basada en MS (2D-HELS MS Seq). Este método es capaz de secuenciar con precisión secuencias individuales de ARN, así como mezclas que contienen hasta 12 secuencias de ARN distintas. Además de los cuatro ribonucleótidos canónicos (A, C, G y U), el método tiene la capacidad de secuenciar oligonucleótidos de ARN que contienen nucleótidos modificados. Esto es posible porque la nucleobase modificada tiene una masa intrínsecamente única que puede ayudar en su identificación y su ubicación en la secuencia de ARN, o se puede convertir en un producto con una masa única. En este estudio, hemos utilizado ARN, incorporando dos nucleótidos modificados representativos (pseudouridina (Ψ) y 5-metilcitosina (m5C)), para ilustrar la aplicación del método para la secuenciación de novo de un solo oligonucleótido de ARN, así como una mezcla de oligonucleótidos de ARN, cada uno con una secuencia diferente y/o nucleótidos modificados. Los procedimientos y protocolos descritos aquí para secuenciar estos ARN modelo serán aplicables a otras muestras cortas de ARN (<35 nt) cuando se utilice un sistema LC-MS estándar de alta resolución, y también se pueden utilizar para la verificación de la secuencia de oligonucleótidos de ARN terapéuticos modificados. En el futuro, con el desarrollo de algoritmos más robustos y con mejores instrumentos, este método podría permitir la secuenciación de muestras biológicas más complejas.
Introducción
Se han desarrollado métodos de secuenciación basados en espectrometría de masas (MS), incluida la MS de arriba hacia abajo y la MS en tándem 1,2,3,4, para la secuenciación directa de ARN. Sin embargo, las técnicas de fragmentación in situ para generar eficazmente escaleras de ARN de alta calidad en espectrómetros de masas actualmente no se pueden aplicar a la secuenciación de novo 5,6. Además, no es muy trivial analizar los datos tradicionales de MS unidimensional (1D) para la secuenciación de novo de incluso una secuencia de ARN purificado, y sería aún más difícil para la secuenciación de MS de muestras de ARN mixto 7,8. Por lo tanto, se ha desarrollado un método de secuenciación de ARN basado en cromatografía líquida (LC)-MS bidimensional (2D), que incorpora la producción de escaleras de tiempo de retención de masa (tR) 2D para reemplazar las escaleras de masa 1D, lo que facilita mucho la identificación de los componentes de la escalera necesarios para la secuenciación de novo de ARN8. Sin embargo, el método de secuenciación de ARN 2D LC-MS se limita principalmente a ARN corto sintético purificado, ya que no puede leer una secuencia completa basándose únicamente en una sola escalera, sino que debe basarse en dos escaleras adyacentes coexistentes (escaleras de 5' y 3')8. Más específicamente, este enfoque requiere lecturas bidireccionales de extremos emparejados para leer nucleobases terminales en la región de baja masa8. La complejidad añadida de la lectura de los extremos emparejados hace que este método sea insostenible para la secuenciación de mezclas de ARN porque se genera confusión sobre qué fragmento de escalera pertenece a qué escalera para las muestras desconocidas.
Para superar las barreras mencionadas anteriormente en los enfoques de secuenciación de ARN basados en MS y ampliar dichas aplicaciones en la secuenciación directa de ARN, se deben abordar dos cuestiones: 1) cómo generar una escalera de masa de alta calidad que se pueda utilizar para leer una secuencia completa, desde el primer nucleótido hasta el último en una cadena de ARN, y 2) cómo identificar eficazmente cada escalera de ARN/masa en un conjunto de datos complejos de MS. Junto con la degradación ácida bien controlada, hemos desarrollado un nuevo método de secuenciación mediante la introducción de una estrategia de etiquetado final hidrofóbico (HELS) en la técnica de secuenciación basada en MS, y abordamos con éxito estos dos problemas mediante la adición de una etiqueta hidrofóbica en el extremo 5' y/o 3' de los ARN que se van a secuenciar9. Este método crea una escalera de secuencia "ideal" a partir de ARN: cada fragmento de escalera deriva de la escisión de ARN específica del sitio exclusivamente en cada enlace fosfodiéster, y la diferencia de masa entre dos fragmentos de escalera adyacentes es la masa exacta del nucleótido o la modificación del nucleótido en esa posición 8,9,10. Esto es posible porque incluimos una etapa de hidrólisis ácida altamente controlada, que fragmenta el ARN, en promedio, una vez por molécula, antes de que se inyecte en el instrumento. Como resultado, cada producto de fragmento de degradación se detecta en el espectrómetro de masas y todos los fragmentos juntos forman una escalera de secuenciación 8,9,10. Esta nueva estrategia permite la lectura completa de una secuencia de ARN de una sola escalera de una cadena de ARN sin la lectura de extremos emparejados de la otra escalera del ARN y, además, permite la secuenciación de MS de mezclas de ARN con múltiples hebras diferentes que contienen modificaciones combinatorias de nucleótidos9. Al agregar una etiqueta en el extremo 5' y/o 3' del ARN, los fragmentos de escalera marcados muestran un retraso significativo de tR, lo que puede ayudar a distinguir las dos escaleras de masa entre sí y también de la región ruidosa de baja masa. El desplazamiento de masa-tR causado por la adición de la etiqueta hidrofóbica facilita la identificación de la escalera de masa y simplifica el análisis de datos para la generación de secuencias. Además, la adición de la etiqueta hidrofóbica puede ayudar a identificar la base terminal en la hebra al evitar que su fragmento de escalera correspondiente esté en la regiónR de baja masa y baja ruido debido al aumento de masa e hidrofobicidad causado por la etiqueta, lo que permite la identificación de la secuencia completa de un ARN a partir de una sola escalera; No se requieren lecturas de extremo emparejado. Como resultado, hemos demostrado previamente la secuenciación exitosa de una mezcla compleja de hasta 12 hebras distintas de ARN sin el uso de ningún algoritmo de secuenciación avanzado9, lo que abre la puerta a la secuenciación MS de novo de ARN que contiene nucleótidos canónicos y modificados y lo hace más factible para la secuenciación de muestras de ARN mixtas y más complejas. De hecho, utilizando 2D-HELS MS Seq, incluso hemos secuenciado con éxito una población mixta de muestras de ARNt10 y estamos ampliando activamente su aplicación a otras muestras de ARN complejas.
Para facilitar que 2D-HELS MS Seq secuencie directamente una gama más amplia de muestras de ARN, aquí nos centraremos en los aspectos técnicos de este enfoque de secuenciación y cubriremos todos los pasos esenciales necesarios al aplicar la técnica hacia la secuenciación directa de muestras de ARN. Se utilizarán ejemplos específicos para ilustrar la técnica de secuenciación, incluidas secuencias sintéticas de ARN único, mezclas de múltiples secuencias de ARN distintas y ARN modificados que contienen nucleótidos canónicos y modificados, como pseudouridina (ψ) y 5-metilcitosina (m5C). Dado que todos los ARN contienen enlaces fosfodiéster, cualquier tipo de ARN puede ser hidrolizado con ácido para generar una escalera de secuencia ideal para 2D-HELS MS Seq en condiciones óptimas 8,9. Sin embargo, la detección de todos los fragmentos de escalera de un ARN dado depende del instrumento. En una LC-MS estándar de alta resolución (40K), la cantidad mínima de carga para secuenciar una muestra de ARN corto purificada (<35 nt) es de 100 pmol por ejecución. Sin embargo, se requiere más material (hasta 400 pmol por muestra de ARN) cuando se deben realizar experimentos adicionales (por ejemplo, para distinguir modificaciones de bases isoméricas que comparten masas idénticas). El protocolo utilizado en la secuenciación del modelo de ARN sintético modificado también será aplicable a la secuenciación de muestras de ARN más amplias, incluidas las muestras de ARN biológico con modificaciones de bases desconocidas. Sin embargo, se requiere una cantidad de muestra aún mayor, como 1000 pmol para secuenciar el ARNt (~76 nt) utilizando un instrumento LC-MS estándar, para secuenciar el ARNt completo con todas las modificaciones, y se debe desarrollar un algoritmo avanzado para su secuenciación de novo 10.
Protocolo
1. Diseño de oligonucleótidos de ARN
- Diseñar oligonucleótidos de ARN sintéticos con diferentes longitudes (19 nt, 20 nt y 21 nt), incluyendo uno (ARN #6) con nucleótidos canónicos y modificados. ψ se emplea como modelo para modificaciones que no alteran la masa, lo cual es un desafío para la secuenciación de MS porque tiene una masa idéntica a la de U. m5C se elige como modelo para las modificaciones que alteran la masa para demostrar la solidez del enfoque.
ARN #1: 5'-HO-CGCAUCUGACUGACCAAAA-OH-3'
ARN #2: 5'-HO-AUAGCCCAGUCAGUCUACGC-OH-3'
ARN #3: 5'-HO-AAACCGUUACCAUUACUGAG-OH-3'
ARN #4: 5'-HO-GCGUACAUCUUCCCCUUUAU-OH-3'
ARN #5: 5'-HO-GCGGAUUUAGCUCAGUUGGGA-OH-3'
ARN #6: 5'-HO-AAACCGUψACCAUUAm5CUGAG-OH-3'
- Disuelva cada ARN sintético en agua tratada con pirocarbonato de dietilo (DEPC) libre de nucleasas (expresada comoH2O tratada con DEPC, a menos que se indique lo contrario) para obtener una solución madre de ARN de 100 mM. Las soluciones madre se almacenan a largo plazo a -20 °C.
- Para evitar la posible degradación de la muestra de ARN, utilice suministros experimentales sin ARNasa, como agua tratada con DEPC, tubos de microcentrífuga y puntas de pipeta. Limpie con frecuencia las superficies de los suministros de laboratorio con toallitas de eliminación de RNasa.
2. Etiquete el extremo 3' de los ARN con biotina
- Protocolo de reacción en dos pasos (adenilación y ligadura)
- Agregue 1 μL de tampón de reacción de adenilación 10x que contiene 50 mM de acetato de sodio, pH 6.0, 10 mM MgCl2, 5 mM de diclorodifeniltricloroetano (DTT), 0.1 mM de ácido etilendiaminotetraacético (EDTA), 1 μL de 1 mM de ATP, 1 μL de 100 μM de bisfosfato de citidina biotinilado (pCp-biotina), 1 μL de 50 μM Mth ARN ligasa y 6 μL de H2O tratado con DEPC (un volumen total de 10 μL) en un tubo de PCR de pared delgada de 0,2 mL libre de ARNasa.
NOTA: Almacene los reactivos a -20 °C antes de la reacción en dos pasos. Descongele los reactivos a temperatura ambiente y mezcle bien mediante vórtice y centrifugado antes de agregarlos a la reacción. - Incubar la reacción en una máquina de PCR a 65 °C durante 1 h e inactivar la reacción a 85 °C durante 5 min.
- Llevar a cabo la etapa de ligadura en un tubo de PCR de 0,2 mL de pared delgada libre de ARNasa que contenga 10 μL de solución de reacción del paso anterior añadiendo 3 μL de tampón de reacción de ARN ligasa T4 10x que contenga 50 mM de tris(hidroximetil)aminometano (Tris)-HCl, pH 7,8, 10 mM MgCl2, 1 mM DTT, 1,5 μL del stock de muestra de 100 mM del ARN que se va a secuenciar. 3 μL de dimetilsulfóxido anhidro (DMSO) para alcanzar el 10% (v/v), 1 μL de ARN ligasa T4 (10 unidades/μL) y 11,5 μL deH2O tratado con DEPC (para un volumen total de 30 mL). Incubar la reacción durante la noche a 16 °C en una máquina de PCR.
NOTA: Combine los componentes de la reacción a temperatura ambiente debido al alto punto de congelación del DMSO (18,45 °C). - Incubar la reacción durante la noche a 16 °C.
- Apague y purifique la reacción mediante la purificación en columna para eliminar las enzimas y liberar pCp-biotina utilizando Oligo Clean & Concentrator (Zymo Research, Irvine, CA, EE. UU.). El kit incluye tampón de unión de oligonucleótidos, tampón de lavado de ADN, columnas de centrifugación y tubos de recolección. Agregue 20 mL de H2O tratado con DEPC a la solución de reacción para alcanzar un volumen de muestra de 50 mL antes de agregar el tampón de unión.
- Agregue 100 mL de tampón aglutinante a cada solución de reacción. Agregue 400 μL de etanol, mezcle por pipeteo y transfiera la mezcla a la columna. Centrífuga a 10.000 x g durante 30 s. Deseche el flujo continuo.
- Añada 750 μL de tampón de lavado de ADN a la columna. Centrífuga a 10.000 x g y velocidad máxima durante 30 s y 1 minuto, respectivamente.
- Transfiera la columna a un tubo de microcentrífuga de 1,5 mL. Añadir 15 μL deH2Otratado con DEPC a la columna y centrifugar a 10.000 x g durante 30 s para eluir el producto de ARN.
NOTA: Las muestras se pueden almacenar a -20 °C en esta etapa hasta que se realice el siguiente paso.
- Agregue 1 μL de tampón de reacción de adenilación 10x que contiene 50 mM de acetato de sodio, pH 6.0, 10 mM MgCl2, 5 mM de diclorodifeniltricloroetano (DTT), 0.1 mM de ácido etilendiaminotetraacético (EDTA), 1 μL de 1 mM de ATP, 1 μL de 100 μM de bisfosfato de citidina biotinilado (pCp-biotina), 1 μL de 50 μM Mth ARN ligasa y 6 μL de H2O tratado con DEPC (un volumen total de 10 μL) en un tubo de PCR de pared delgada de 0,2 mL libre de ARNasa.
- Protocolo de reacción en un solo paso
- Realice una reacción de etiquetado en un solo paso combinando 2 μL de 150 μM de adenosina-5'-5'-difosfato-{5'-(citidina-2'-O-metil-3'-fosfato-TEG}C-biotina (AppCp-biotina), 3 μL de tampón de reacción de ligasa 10x, 1,5 μL del stock de muestra de 100 mM del ARN que se va a secuenciar, 3 μL de DMSO anhidro para alcanzar el 10% (v/v), 1 μL de ARN ligasa T4 (10 unidades/μL), y 19,5 μL deH2Otratado con DEPC (para un volumen total de 30 mL) en un tubo de microcentrífuga libre de ARNasa de 1,5 mL.
- Incubar la reacción durante la noche a 16 °C en una máquina de PCR.
- Realice la purificación de la columna como se describe anteriormente en los pasos 2.1.5-2.1.8.
NOTA: Prepare un tubo de reacción separado/exclusivo para cada muestra de ARN (escala de 150 pmol de ARN). Puede ser necesario marcar el extremo 5' del ARN con sulfo-cianina 3 (Cy3) o Cy3 (por ejemplo, para la verificación de la secuenciación bidireccional). El método es diferente al de la biotinilación 3' y se describe en una publicación anterior9.
3. Captura de una muestra de ARN biotinilado en perlas de estreptavidina
- Active 200 μL de perlas magnéticas de estreptavidina C1 añadiendo 200 μL de 1x tampón B&W (5 mM de Tris-HCl, pH 7,5, 0,5 mM EDTA, 1 M NaCl) en un tubo de microcentrífuga sin RNasa de 1,5 mL. Agite esta solución y colóquela en un soporte magnético durante 2 minutos. A continuación, deseche el sobrenadante pipeteando cuidadosamente la solución.
- Lave las perlas dos veces con 200 μL de solución A (0,1 M de NaOH tratado con DEPC y 0,05 M de NaCl con DEPC) y una vez con 200 μL de solución B (0,1 M de NaCl con DEPC). Para cada paso de lavado, agite la solución en vórtice y colóquela en un soporte magnético durante 2 minutos, seguido de desechar el sobrenadante. A continuación, añada 100 μL de tampón 2x B&W (10 mM de Tris-HCl, pH 7,5, 1 mM de EDTA, 2 M de NaCl).
- Agregue 1x tampón en blanco y negro a la muestra de ARN biotinilado hasta que el volumen sea de 100 μL. A continuación, añada esta solución a las perlas lavadas almacenadas en 100 μL de tampón B&W 2x. Incubar durante 30 min a temperatura ambiente en un agitador de plataforma oscilante a 100 rpm. Coloque el tubo en un soporte magnético durante 2 minutos y deseche el sobrenadante.
- Lave las perlas recubiertas 3 veces en 1x tampón B&W y mida la concentración final de sobrenadante en cada paso de lavado con Nanodrop para el análisis de recuperación, para confirmar que las moléculas de ARN objetivo permanecen en las perlas.
- Incubar las perlas en 10 mM de EDTA, pH 8,2 con formamida al 95% a 65 °C durante 5 min en una máquina de PCR. Mantenga el tubo en el soporte magnético durante 2 minutos y recoja el sobrenadante (que contiene los ARN biotinilados liberados por las perlas de estreptavidina) mediante una pipeta.
NOTA: Este paso de separación física antes de la degradación del ácido solo se usa para la secuenciación del ARN # 1 en la Figura 1c, y no es obligatorio para el 2D-HELS MS Seq ya que la etiqueta de biotina hidrofóbica puede hacer que los fragmentos de escalera marcados con 3' tengan un tR significativamente retrasado durante la medición de LC-MS, lo que puede distinguir claramente los fragmentos de escalera de 3' marcados de los fragmentos de escalera de 5' no marcados en el gráficoR de masa-t 2D.
4. Hidrólisis ácida de ARN para generar escaleras de MS para la secuenciación
- Divida cada muestra de ARN en tres alícuotas iguales. Por ejemplo, divida una muestra de ARN con un volumen de 15 μL de muestra de ARN en tres alícuotas de 5 μL.
- Añadir un volumen igual de ácido fórmico para obtener un 50% (v/v) de ácido fórmico en la mezcla de reacción 8,9.
- Incubar la reacción a 40 °C en una máquina de PCR, con una reacción durante 2 minutos, otra durante 5 minutos y otra durante 15 minutos, respectivamente.
- Enfríe la degradación del ácido congelando inmediatamente la muestra en hielo seco después de que finalice cada reacción.
- Utilice un concentrador de vacío centrífugo para secar la muestra. Por lo general, la muestra se seca por completo en 30 minutos, y el ácido fórmico se elimina junto con elH2Odurante el proceso de secado porque el ácido fórmico tiene un punto de ebullición (100,8 °C) similar al delH2O(100 °C).
- Suspender y combinar un total de tres muestras secas en 20 μL de H2O tratado con DEPC para la medición de LC-MS.
NOTA: Las muestras se pueden almacenar a -20 °C en esta etapa mientras se espera la medición de LC-MS.
5. Convertir ψ a aducto CMC-ψ
- Añadir 80 μL de H2O tratado con DEPC en un tubo de microcentrífuga libre de RNasa de 1,5 mL que contenga 0,0141 g de N-ciclohexil-Nʹ-(2-morfolinoetil)-carbodiimida meto-p-toluenosulfonato (CMC) y 0,07 g de urea. Añadir 10 μL de los 100 μM de muestra del ARN a secuenciar, 8 μL de tampón bicina 1 M (pH 8,3) y 1,28 μL de EDTA 0,5 M. Agregue H2O tratado con DEPC para alcanzar un volumen total de 160 μL. Las concentraciones finales son 0,17 M CMC, 7 M urea y 4 mM EDTA en bicina 50 mM (pH 8,3)11.
NOTA: Este protocolo es aplicable a una sola secuencia de ARN sintético o a mezclas de ARN. - Divida la solución de reacción de 160 μL en cuatro alícuotas iguales en tubos de PCR de 0,2 mL de pared delgada y sin RNasa e incube a 37 °C durante 20 min en una máquina de PCR.
NOTA: 50 μL por tubo es el volumen máximo de reacción que se puede utilizar en una máquina de PCR. - Apague cada reacción con 10 μL de acetato de sodio 1,5 M y EDTA 0,5 mM (pH 5,6).
- Realice la purificación de la columna con cuatro columnas de espín paralelas para eliminar el exceso de reactivos de acuerdo con el procedimiento descrito en los pasos 2.1.5-2.1.8. Disuelva el producto purificado en 15 μL de H2O tratado con DEPC en cada tubo de microcentrífuga libre de ARNasa de 1,5 mL.
- Transfiera el producto purificado a cuatro tubos de PCR de 0,2 mL de pared delgada y libres de RNasa. Añadir 20 μL de tampón 0,1 M de Na2CO3 (pH 10,4) en cada 15 μL de producto purificado y añadir H2O tratado con DEPC para obtener un volumen final de 40 μL para cada tubo de reacción (en total cuatro tubos). Incubar la reacción a 37 °C durante 2 h en una máquina de PCR.
- Apagar y purificar la reacción mediante la purificación en columna con cuatro columnas de espín paralelas como se describe en el paso 2.1.5. Eluir el producto convertido en CMC-ψ en un tubo de microcentrífuga libre de ARNasa de 1,5 mL, cada uno con 15 μL de H2O tratado con DEPC.
- Combine la muestra convertida CMC-ψ purificada de cuatro tubos de recolección en un solo tubo. Realice la degradación del ácido fórmico al 50% (v/v) de acuerdo con los procedimientos descritos en los pasos 4.1-4.6 para generar escaleras de MS para la secuenciación.
6. Medición LC-MS
- Prepare las fases móviles para la medición de LC-MS. La fase móvil A es 25 mM de hexafluoro-2-propanol con 10 mM de diisopropilamina en agua de grado LC-MS; La fase móvil B es el metanol.
- Transfiera la muestra al vial de muestra LC-MS para su análisis. El volumen de inyección de cada muestra es de 20 μL y contiene 100-400 pmol de ARN.
- Utilice las siguientes condiciones de LC: temperatura de la columna de 35 °C, caudal de 0,3 mL/min; un gradiente lineal del 2 al 20 % de la fase B móvil durante 15 minutos, seguido de una etapa de lavado de 2 minutos con una fase B móvil del 90 %.
NOTA: En el caso de las etiquetas finales más hidrofóbicas, como Cy3 y sulfo-Cy3, como se menciona en la Sección 2, puede ser necesario un mayor porcentaje de disolvente orgánico para la elución de la muestra (es decir, se puede utilizar un gradiente similar pero con un mayor rango porcentual de fase móvil B). Por ejemplo, 2-38% fase B móvil durante 30 min con un paso de lavado de 2 min con 90% fase B móvil. - Separe y analice muestras en un espectrómetro de masas Agilent Q-TOF (Quadrupole Time-of-Flight) acoplado a un sistema LC equipado con un muestreador automático y un sistema MS HPLC (cromatografía líquida de alto rendimiento). La columna LC es una columna C18 de 50 mm x 2,1 mm con un tamaño de partícula de 1,7 μm. Utilice los siguientes ajustes de MS: modo de iones negativos; rango, de 350 m/z a 3200 m/z; velocidad de exploración, 2 espectros/s; caudal de gas de secado, 17 L/min; temperatura del gas de secado, 250 °C; presión del nebulizador, 30 psig; voltaje capilar, 3500 V; y voltaje del fragmentador, 365 V. Tenga en cuenta que estos parámetros son específicos del tipo o modelo de espectrómetro de masas que se utiliza.
- Adquiera datos con el software de adquisición Agilent MassHunter. Utilice el flujo de trabajo de extracción de características moleculares (MFE) de Agilent para extraer información de compuestos, incluida la masa, el tiempo de retención, el volumen (la abundancia de MFE para las especies de iones respectivas) y la puntuación de calidad, etc. Utilice los siguientes ajustes de MFE: "formato de datos de centroide, moléculas pequeñas (cromatográficas), pico con altura ≥ 100, hasta un máximo de 1000, puntuación de calidad ≥ 50".
NOTA: Optimice la configuración de MFE para extraer tantos compuestos potenciales como sea posible, hasta un máximo de 1000, con puntuaciones de calidad de ≥ 50.
7. Automatizar la generación de secuencias de ARN mediante un algoritmo computacional
NOTA: Este procedimiento se muestra solo para el ARN #1 en la Figura 1c.
- Clasifique los compuestos extraídos con MFE en orden decreciente de volumen (intensidad máxima) y tR. Realice la preselección de datos a través de 1) configurando tR de 4 a 10 min para seleccionar los fragmentos de ARN marcados por la biotina, ya que los tRs de los componentes de la escalera de masa marcados con biotina se desplazan a esta ventana tR (4 min a 10 min), y 2) utilizando un orden de magnitud mayor de compuestos de entrada que el número de fragmentos de escalera para que el cálculo del algoritmo reduzca la cantidad de datos en función del volumen. Por ejemplo, para un ARN de 20 nt, se requerirán 20 componentes de escaleraR de masa-t marcados para la secuenciación del ARN de 20 nt y, por lo tanto, se seleccionarán 200 compuestos del archivo de datos MFE en función del volumen. Tenga en cuenta que la ventana tR puede ser diferente cuando se utiliza un tipo o modelo diferente de espectrómetro de masas.
- Realizar el procesamiento de datos y la generación de secuencias de ARN #1 utilizando una versión revisada de un algoritmo publicado8. Los códigos fuente del algoritmo revisado se describen anteriormente (https://academic-oup-com.remotexs.ntu.edu.sg/nar/article/47/20/e125/5558343#supplementary-data)9.
- Además de automatizar la generación de secuencias mediante el algoritmo, calcule manualmente las diferencias de masa entre dos componentes de escalera adyacentes para la llamada base. Todas las bases del ARN pueden llamarse manualmente y cotejarse con las teóricas de la base de datos de nucleótidos y modificaciones de ARN8; por lo tanto, la secuencia completa de la cadena de ARN se puede leer con precisión manualmente, lo que se utiliza para confirmar la precisión de la lectura de la secuencia informada por el algoritmo. Se pueden encontrar más estructuras de modificaciones de ARN en las bases de datos de modificaciones de ARN12, y sus masas teóricas correspondientes se obtienen mediante ChemBioDraw. En las Tablas S1-S2, la diferencia de masa en ppm (partes por millón) se muestra al comparar la masa observada con su masa teórica para un componente específico de la escalera, y un valor inferior a 10 ppm se considera una buena coincidencia para cada llamada base.
8. Secuenciación de mezclas de ARN
- Etiquete una mezcla de cinco cadenas de ARN (ARN #1 a #5) en sus extremos 3' con A(5')pp(5')Cp-TEG-biotina utilizando un protocolo de un solo paso descrito en el paso 2.2. En un volumen total de 150 μL de solución de reacción, agregue 15 μL de 10x tampón de reacción de ARN ligasa T4, 1,5 μL de cada hebra de ARN (100 μM de ARN #1 a #5, respectivamente, para un volumen total de 7,5 μL), 10 μL de 150 μM A(5')pp(5')Cp-TEG-biotina, 15 μL de DMSO anhidro, 5 μL de ARN ligasa T4 (10 unidades/μL) y 97,5 μL de H2O tratado con DEPC. Distribuya uniformemente la solución de reacción en cinco alícuotas. Cada tubo de microcentrífuga libre de RNasa contiene 30 μL de solución de reacción.
- Incubar la reacción durante la noche a 16 °C en una máquina de PCR.
- Realice la purificación de la columna de acuerdo con el procedimiento descrito en los pasos 2.1.5-2.1.8 con cinco columnas de centrifugación paralelas. Eluir una muestra de mezcla de 5 hebras de ARN 3' biotiniladas (mezcla de ARN #1 a #5) en un tubo de microcentrífuga libre de ARNasa de 1,5 mL, cada uno con 15 μL deH2O, tratado con DEPC.
- Combine las muestras de mezcla purificada de los cinco tubos de recolección en un solo tubo. Realizar la degradación del ácido fórmico de acuerdo con el procedimiento descrito en la sección 4.
- Mida las muestras mediante LC-MS como se describe en la Sección 6 y analice los datos utilizando el software de análisis de datos con configuraciones MFE optimizadas para extraer datos que contengan masa, tR y volumen como se describe en el paso 6.5. El algoritmo típico de procesamiento y llamada a bases no se aplica debido al aumento significativo de la complejidad de los datos resultante de la mezcla. Todas las bases en el ARN de la muestra mezclada se llaman manualmente en un método similar a la Sección 7.3 y coinciden bien con las teóricas en la base de datos de nucleótidos y modificaciones de ARN8, por lo que las secuencias completas de las cinco hebras de ARN en la muestra mezclada se leen con precisión. En las Tablas S7-S11, se enumera toda la información, incluida la masa observada, tR, el volumen, la puntuación de calidad y la diferencia de masa en ppm.
Resultados
Introducción de una etiqueta de biotina en el extremo 3' del ARN para producir escaleras de masa-tR fácilmente identificables. En la Figura 1a se muestra el flujo de trabajo del enfoque MS Seq 2D-HELS. La marca hidrofóbica de biotina introducida en el extremo 3' del ARN (ver Sección 2) aumenta las masas y tRs de los componentes de la escalera marcados con 3' en comparación con los de sus contrapartes no marcadas. Por lo tanto, la curva de la escalera 3' se desplaza a valores mayores del eje Y (debido al aumento de tRs) y se desplaza a valores mayores del eje x (debido al aumento de las masas) en el gráfico 2Dmasa-t R . La Figura 1b muestra el protocolo de preparación de la muestra, incluida la introducción de una etiqueta de biotina en el extremo 3' del ARN para 2D-HELS MS Seq. La Figura 1c muestra la separación de la escalera 3' de la escalera 5' y otros fragmentos no deseados en un gráfico 2D de masa-tR basado en cambios sistemáticos en tRde los fragmentos de escalera de masa-tR marcados con 3' biotina del ARN # 1. La curva de escalera de 3' por sí sola proporciona una secuencia completa de ARN # 1, y la curva de escalera de 5' que no muestra un desplazamiento tR proporciona la secuencia inversa, pero requiere emparejamiento final para leer la baseterminal 8. Con esta estrategia de 2D-HELS, no se requiere el emparejamiento final como se informó anteriormente y la secuencia completa de ARN se puede leer completamente desde una sola curva de escalera marcada8. Como tal, es posible secuenciar muestras mixtas que contienen múltiples ARN, por ejemplo, dos cadenas de ARN de diferentes longitudes (ARN #1 y ARN #2, 19 nt y 20 nt, respectivamente) con una etiqueta de biotina 5' en cada ARN (Figura 1d).
Conversión de ψ a su aductor CMC-ψ para 2D-HELS MS Seq. ψ es una modificación nucleótida difícil para la secuenciación basada en MS porque tiene la misma masa que la uridina (U). Para diferenciar estas dos bases entre sí, tratamos el ARN con CMC, que convierte un ψ en un aducto CMC-ψ (ver sección 5). El aducto tiene una masa diferente a la suya y se puede diferenciar en la secuencia MS de 2D-HELS. La figura 2a muestra el perfil de HPLC del producto bruto de la reacción que convierte ψ a su aducto CMC en el ARN # 6. Al integrar sus picos UV, calculamos el porcentaje de conversión y el 42% ψ se convierte en su aducto CMC-ψ después del proceso ilustrado en la Sección 5. Después de la degradación ácida y la medición de LC-MS, adquirimos manualmente la secuencia basada tanto en las escaleras no convertidas en CMC como en las escaleras convertidas en CMC identificadas a partir de los datos procesados por el algoritmo 8,9. Una curva roja se ramifica desde la curva gris a partir de ψ en la posición 8 en el ARN # 6 (Figura 2b) debido a la conversión parcial de ψ al aducto CMC-ψ. Debido a la masa y la hidrofobicidad de la CMC, esta conversión da como resultado un aumento de 252,2076 Dalton en la masa y un aumento significativo en tR para cada componente de escalera que contiene aductos CMC-ψ en comparación con su contraparte no convertida. Por lo tanto, se puede observar un cambio dramático a partir de la posición 8 en el ARN # 6 en el gráficoR de masa-t 2D, lo que indica que la posición 8 es de hecho un ψ en el ARN # 6.
Secuenciación de mezclas de ARN. Se secuencia una mezcla de cinco cadenas de ARN diferentes mediante el enfoque 2D-HELS MS Seq con marcaje en el extremo 3' (ver sección 8). La preocupación por la secuenciación de ARN mixtos es que múltiples curvas de escalera en el gráfico 2Dmasa-t R pueden superponerse entre sí cuando todas comparten los mismos puntos de partida (la etiqueta hidrofóbica en el gráfico 2Dmasa-t R). Sin embargo, la llamada base se realiza una por una, cada una basada en una diferencia de masa entre dos fragmentos de escalera adyacentes en los datos MFE. La llamada de base correcta se puede realizar siempre que cada diferencia de masa coincida bien (una diferencia de MS PPM < 10) con una de las masas teóricas de nucleótidos canónicos o modificados en el conjunto de datos 8,9. En el análisis de las muestras de ARN multiplexadas, el algoritmo típico de procesamiento y llamada de bases utilizado en las Figuras 1 y 2 no se utiliza principalmente debido al aumento significativo de la complejidad de los datos resultante de la mezcla. Estas secuencias se llaman bases manualmente mediante el cálculo de la diferencia de masa entre dos fragmentos de escalera de masa adyacentes y la comparan con la masa teórica del nucleótido en el conjunto de datos9. Cualquier base coincidente con una masa PPM <10 se elige como identidad base en esta posición. Con este cálculo manual base por base para la llamada de bases, todas las secuencias de la mezcla se secuencian con precisión. El software OriginLab se utiliza para reconstruir un gráfico 2D demasa-t R, en el que el tR inicial para cada secuencia se normaliza sistemáticamente para visualizar mejor cinco secuencias de ARN diferentes (Figura 3). Sin dicha normalización, los códigos de letras (es decir, A, C, G y U) para las secuencias de los cinco ARN se agruparían en el gráfico (Figura S1), lo que resultaría en una menor facilidad de visualización en comparación con lo informado en la Figura 3. Los resultados de la secuenciación demuestran que el enfoque 2D-HELS MS Seq no solo se limita a la secuenciación de ARN monocatenario purificados, sino también, lo que es más importante, a mezclas de ARN con múltiples cadenas de ARN. Actualmente se están desarrollando algoritmos para automatizar el proceso de llamada a bases y generación de secuencias.
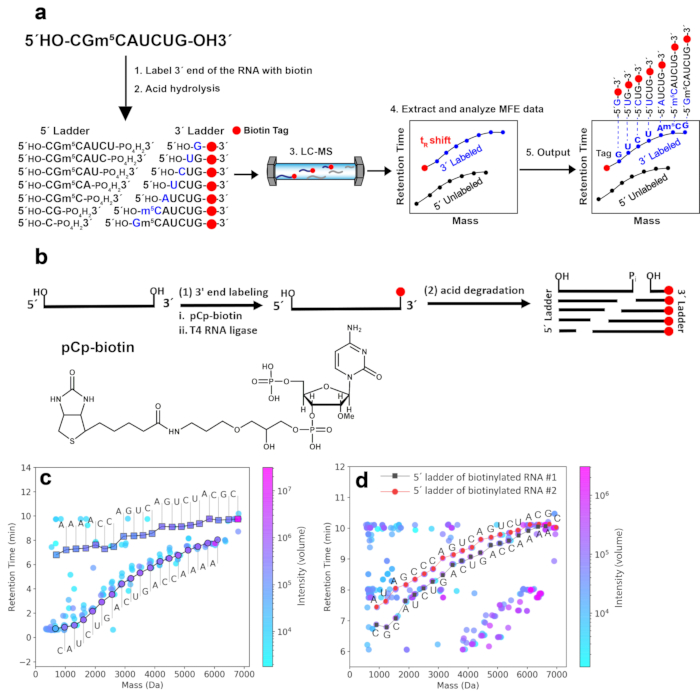
Figura 1: 2D-HELS MS Seq de muestras representativas de ARN. (a) Flujo de trabajo para 2D-HELS MS Seq. Los pasos principales incluyen 1) el etiquetado hidrofóbico del ARN que se va a secuenciar, 2) la hidrólisis ácida, 3) la medición de LC-MS, 4) la extracción y el análisis de datos MFE, y 5) la generación de secuencias mediante algoritmos o cálculo manual. (b) Protocolo de preparación de muestras que incluye la introducción de una etiqueta de biotina en el extremo 3' del ARN para 2D-HELS MS Seq. (c) Separación de la escalera 3' de la escalera 5' y otros fragmentos no deseados en un gráfico de tiempo de retención de masa 2D (tR) basado en cambios sistemáticos en tRs de fragmentos de escalera de masa tR marcados con 3' biotina de ARN # 1 (19 nt). Las secuencias son de novo y se leen automáticamente directamente mediante un algoritmo de llamada a base9. (d) Secuenciación simultánea de ARN #1 y ARN #2, 19 nt y 20 nt marcados con 5'-biotina, respectivamente. Los métodos para introducir una etiqueta de biotina en el extremo 5' del ARN son diferentes a los de la biotinilación 3', y se pueden encontrar en el protocolo publicado anteriormente9. El extremo 5' de dos ARN (ARN #1 y ARN #2) está biotinilado y sus escaleras 5'-biotiniladas se pueden identificar fácilmente; ambas escaleras 5' biotiniladas se separan fácilmente de sus escaleras 3' no marcadas en el gráfico 2Dmasa-t R después de LC-MS, porque los componentes de la escalera biotinilada tienen los mayores desplazamientos tR debido a la hidrofobicidad de la biotina, mientras que los componentes de la escalera no marcados están en la región tR inferior. Aunque las escaleras 5' y 3' coexisten, no interfieren con la interpretación de la secuencia de dos cadenas mixtas de ARN. Cada secuencia de estos dos ARN se adquiere manualmente a partir de escaleras biotiniladas 5' basadas en los datos procesados por el algoritmo computacional 8,9. Esta figura ha sido modificada a partir de Zhang et al.9. Haga clic aquí para ver una versión más grande de esta figura.

Figura 2. Conversión de pseudouridina (ψ) en su aducto para 2D-HELS MS Seq. (a) Perfil de HPLC del producto bruto de la reacción que convierte ψ a su aducto CMC en un ARN de 20 nt (ARN # 6) que contiene un ψ. (b) Secuenciación de un ARN # 6 que contiene ψ. La conversión de la ψ a los aductos CMC-ψ (ψ*) da como resultado un aumento de 252,2076 Dalton en masa y un aumento significativo en tR debido a su masa e hidrofobicidad de la CMC. Por lo tanto, se puede observar un cambio dramático que comienza en la posición de 8 en el gráfico de masa-tR, lo que indica que se trata de un ψ en la posición de 8 en la secuencia de ARN. Las secuencias se adquieren manualmente en función de los datos procesados por el algoritmo computacional 8,9. Esta figura ha sido modificada a partir de Zhang et al.9. Haga clic aquí para ver una versión más grande de esta figura.

Figura 3. Secuenciación de mezclas de ARN que contienen cinco ARN distintos. Se utiliza una biotina para marcar cada ARN en su extremo 3' antes de la secuencia MS de 2D-HELS. Para cada secuencia, los valores tR iniciales se normalizan sistemáticamente para comenzar a intervalos de 7 minutos para facilitar la visualización. Las diferencias absolutas entre el valor tR inicial y el valor tRs subsiguiente permanecen inalteradas para cada uno de los cinco ARN, por lo que es más fácil visualizar cada uno de ellos en el mismo gráfico. Todas las bases se identifican calculando manualmente las diferencias de masa de dos componentes de la escalera adyacentes y comparándolas con las diferencias de masa teóricas en la base de datos de modificación y nucleótidos de ARN8; los gráficos de la Figura 3 se reconstruyen utilizando OriginLab en función de los datos de secuenciación y llamada de bases manuales (consulte la Sección de secuenciación de mezclas de ARN en Resultados representativos). En la Figura S1 se muestra la cifra 2Dmasa-t R de los cinco ARN mixtos sin normalización de tR . Haga clic aquí para ver una versión más grande de esta figura.
Figura S1. Haga clic aquí para descargar este archivo.
Discusión
A diferencia de la fragmentación de MS basada en tándem, la hidrólisis ácida altamente controlada se utiliza en el enfoque 2D-HELS MS Seq para fragmentar el ARN antes del análisis con un espectrómetro de masas 9,10. Como resultado, el instrumento puede detectar cada fragmento degradado en ácido, formando el equivalente a una escalera de secuenciación. En condiciones óptimas, este método crea una escalera de secuencia "ideal" a partir de ARN a través, en promedio, de una escisión de ARN específico por sitio molécula exclusivamente en un enlace fosfodiéster 8,9,10. Después de que el espectrómetro de masas mide cada fragmento degradado en una sola ejecución, la diferencia de masa entre dos fragmentos de escalera adyacentes corresponde a la masa exacta del nucleótido de ARN o modificación en esa posición. Cada modificación de ARN tiene una masa única intrínseca que puede ayudar a identificarla y localizarla en el ARN, o se puede convertir en una con una masa única. Por lo tanto, en teoría, este método puede informar la identidad y la ubicación de nucleótidos canónicos y modificados para la secuenciación directa y de novo de cualquier ARN. Sin embargo, las diferentes escalas de secuencias pueden superponerse entre sí, lo que complica el análisis de los datos de la EM y dificulta la secuenciación del ARN por EM en la práctica.
Uno de los beneficios de la etiqueta hidrofóbica 3' es que supera un desafío importante en cualquier método de fragmentación , es decir, que cada molécula de ARN debe dividirse exactamente en dos fragmentos (e idealmente no más): un fragmento que contiene el extremo 5' original y el otro que contiene el extremo 3' original del ARN. Por lo tanto, cada evento de escisión produce dos fragmentos, produciendo dos escaleras, una medida desde el extremo 5' y la otra desde el extremo 3'. Siempre hay ambigüedad a la hora de determinar qué pico de EM pertenece a cada escalera. Esto se vuelve más problemático en una mezcla de varios ARN diferentes, debido a la generación de un gran número de escaleras de secuencia superpuestas. Sin embargo, dado que todos los fragmentos de escalera de los extremos 3' están etiquetados con una etiqueta hidrofóbica, exhiben tRs mucho más largos (Figura 1a). Como resultado, podemos obtener escaleras claras e inequívocas enlos datos R de masa-t 2D derivados exclusivamente del ARN marcado con 3'. En particular, estamos optimizando enfoques para etiquetar selectivamente el extremo 5' o 3' de cualquier ARN utilizando diferentes métodos de conjugación química. También podemos realizar la secuenciación bidireccional, que no se utiliza para determinar la(s) base(s) terminal(es) aquí, sino que se utiliza para proporcionar información de secuencia idéntica dos veces cuando se lee desde direcciones 5' y 3' (es decir, verificación de secuenciación bidireccional), y por lo tanto mejorar aún más la precisión de la secuenciación.
Para la secuenciación de novo de muestras de ARN desconocidas, especialmente para muestras biológicas complejas, se requiere un algoritmo general y robusto para procesar una gran cantidad de datos de LC-MS para la generación de secuencias de manera precisa y eficiente, que recientemente se ha puesto a disposición a través de otros trabajos publicados10. A pesar de que estos algoritmos se han utilizado para la secuenciación de muestras más complicadas10, en este estudio se realizó la llamada manual de bases para la generación de secuencias, a menos que se indique lo contrario. Nuestro objetivo es cubrir todos los pasos clave en el 2D-HELS MS Seq, y nos gustaría ilustrar el proceso durante el cual, incluso sin utilizar algoritmos de secuenciación adicionales, podemos leer manualmente las secuencias del ARN que se va a secuenciar. Para facilitar la visualización e identificar más rápidamente los fragmentos de escalera necesarios para la secuenciación en el gráfico 2D mass-tR, los archivos MFE de cada ejecución de LC-MS son procesados por una versión revisada de un algoritmo publicado8 antes de leer sus secuencias, a menos que se indique lo contrario. El algoritmo publicado no se puede usar directamente para leer las secuencias de los datos de LC-MS, pero parte de su función aún se puede usar para procesar los datos: agrupar jerárquicamente aductos de masa a través de este algoritmo aumentará la intensidad de cada componente de la escalera, lo que a su vez reduce la complejidad de los datos, especialmente en la región crucial dondese generan las lecturas de secuencia. 9.
Uno de los pasos cruciales durante la preparación de muestras para 2D-HELS MS Seq da como resultado la mejora de la eficiencia del etiquetado final de la etiqueta hidrofóbica de ARN. Una alta eficiencia de etiquetado puede ayudar a reducir la cantidad de muestra de ARN necesaria para generar señales de MS en las que se basan los datos de secuencia. Con el fin de aumentar la eficiencia del etiquetado, empleamos nuevas estrategias de etiquetado, incluido el uso de AppCp-biotina activada para evitar el paso de adenilación al etiquetar el extremo 3' del ARN. El rendimiento de la reacción para marcar el extremo 3' de un ARN de 19 nt con biotina (ver paso 2.2) se puede mejorar del 60% a ~95%9 utilizando este método de un solo paso. Con el etiquetado eficiente, podemos secuenciar una muestra mixta que contiene hasta 12 ARN distintos como se describió anteriormente9. En este estudio, utilizamos una mezcla de cinco ARN como ejemplo representativo para ilustrar el proceso de secuenciación. También detectamos todos los fragmentos de escalera necesarios para una secuenciación precisa y leemos las secuencias completas de cada una de las cinco secuencias de ARN de la mezcla. Una mayor eficiencia de etiquetado no solo ayuda a minimizar la cantidad de carga de la muestra, sino que también ayuda a reducir significativamente la complejidad de los datos durante el análisis de datos posterior para la generación de secuencias. Actualmente se están desarrollando nuevas reacciones para lograr un rendimiento cuantitativo en el marcaje de ARN en los extremos 5' y 3'.
Al secuenciar el ARN #1 como se muestra en la Figura 1c, se utilizan los pasos de captura y liberación de estreptavidina para separar físicamente el ARN #1 biotinilado antes de la degradación del ácido (ver Sección 3). Esto elimina una pequeña porción de ARN no marcado y, posteriormente, da como resultado una mayor facilidad de identificación visual de las escaleras de masa marcadas en el gráficoR de masa-t 2D. Sin embargo, el paso de separación física no es obligatorio porque los fragmentos de la escalera de ARN biotinilado tienen tRs retrasados/más largos debido a la hidrofobicidad de la etiqueta de biotina en comparación con sus contrapartes no marcadas. Además, la llamada de base no se basa en la separación física, sino que se basa en las diferencias de masa de los componentes de la escalera de masa adyacentes, por lo tanto, se puede lograr la llamada de base correcta siempre que las diferencias de masa de dos componentes de la escalera adyacentes coincidan bien con las masas correspondientes de un nucleótido en particular o modificación en el nucleótido de ARN y la base de fecha de modificación8. Actualmente se está desarrollando un algoritmo computacional para automatizar la llamada de bases y la generación de secuencias.
Los ajustes de MFE durante la exportación de datos LC-MS originales (en el tipo de archivo .d) a archivos de hoja de cálculo son muy cruciales para el procesamiento de datos y la posterior generación de secuencias (consulte la Sección 6.5). Por ejemplo, probamos el ajuste MFE "pico con altura" en un rango de 100 a 1000 y nos dimos cuenta de que el ajuste de 100 puede proporcionarnos 2 veces más compuestos que los del ajuste 1000. Para evitar que se pierda ningún componente de la escalera, podemos ajustar la configuración de MFE durante el flujo de trabajo de secuenciación. Es probable que esta configuración dependa de la resolución de masa del instrumento, la cantidad de fragmentos de escalera de masa y la complejidad de los datos. Además, es importante utilizar el conjunto de datos de centroide y la configuración de tipo cromatográfico para moléculas pequeñas. La puntuación de calidad puede variar del 50 % al 100 % en función de la calidad de los datos.
El instrumento LC-MS que utilizamos en el estudio tiene una resolución de masa superior de ~40K, lo que limita el método a secuenciar solo ARN de menos de 35 bases de largo. Sin embargo, la longitud de lectura exacta de este método depende del instrumento; Los instrumentos más avanzados con mayor poder de resolución pueden conducir a una mayor longitud de lectura. Del mismo modo, el rendimiento, es decir, cuántas secuencias de ARN se pueden secuenciar simultáneamente en una sola ejecución de LC-MS, aún no se ha explorado, aunque secuenciamos manualmente una mezcla de muestras de ARN de hasta 12 hebras de ARN distintas, incluso sin el uso de ningún algoritmo9. Con el flujo de trabajo actual, se requieren ~100 pmol de ARN corto (<35 nt) para cada ejecución de LC-MS. La cantidad de carga aumenta cuando se necesitan experimentos adicionales: para diferenciar las modificaciones de los nucleótidos isoméricos, normalmente se requieren hasta 400 pmol de ARN. Para la secuenciación de ARNt específicos como el ARNtPhe, es posible que se necesiten ~ 1000 pmol de muestra para el análisis de secuenciación y modificación. Sin embargo, esperamos que las cantidades requeridas de carga de muestras disminuyan en los instrumentos LC-MS con mayor sensibilidad. Con las mejoras en la eficiencia del etiquetado de muestras, el algoritmo de secuenciación y la sensibilidad y resolución del instrumento, esperamos que nuestro método sea aplicable a una gama más amplia de muestras de ARN, especialmente aquellas con diversas modificaciones de ARN.
Divulgaciones
Los autores han presentado una patente provisional relacionada con la tecnología discutida en este manuscrito.
Agradecimientos
Los autores agradecen la subvención R21 de los Institutos Nacionales de Salud (1R21HG009576) a S. Z. y W. L. y las subvenciones de Apoyo Institucional para la Investigación y la Creatividad del Instituto de Tecnología de Nueva York (NYIT) a S. Z., que apoyaron este trabajo. Los autores desean agradecer al estudiante de doctorado Xuanting Wang (Universidad de Columbia) por ayudar en la creación de figuras, y agradecer al Prof. Michael Hadjiargyrou (NYIT), al Prof. Jingyue Ju (Universidad de Columbia), a los Dres. James Russo, Shiv Kumar, Xiaoxu Li, Steffen Jockusch y a otros miembros del laboratorio Ju (Universidad de Columbia), al Dr. Yongdong Wang (Cerno Bioscience), Meina Aziz (NYIT) y Wenhao Ni (NYIT) por sus útiles discusiones y sugerencias para nuestro manuscrito.
Materiales
| Name | Company | Catalog Number | Comments |
| 5' DNA Adenylation kit | New England Biolabs | E2610S | 50uM concentration |
| 6550 Q-TOF mass spectrometer | Agilent Technologies | 5991-2116EN | Coupled to a 1290 Infinity LC system |
| A(5´)pp(5´)Cp-TEG-biotin-3´ | ChemGenes | 91718 | HPLC purified |
| ATPγS | Sigma-Aldrich | 11162306001 | Lithium salt |
| Bicine | Sigma-Aldrich | B8660 | BioXtra, ≥99% (titration) |
| Biotin maleimide | Vector Laboratories | SP-1501 | Long arm |
| C18 column | Waters | 186003532 | 50 mm × 2.1 mm Xbridge C18 column with a particle size of 1.7 μm |
| Centrifugal Vacuum Concentrator | Labconco | Refrig 115v/60hz 7310022 | Labconco CentriVap |
| ChemBioDraw | PerkinElmer | ChemDraw Prime | Generate a chemical structure and property data of structures & fragments |
| CMC (N-cyclohexyl-N?-(2-morpholinoethyl)-carbodiimide metho-p-toluenesulfonate) | Sigma-Aldrich | 2491-17-0 | 95% Purifiy |
| Cyanine3 maleimide (Cy3) | Lumiprobe | 11080 | Water insoluble |
| DEPC-treated water | Thermo Fisher Scientific | AM9906 | Autoclaved, certified nuclease-free |
| Diisopropylamine (DIPA) | Thermo Fisher Scientific | 108-18-9 | 99% Alfa Aesar |
| DMSO | Sigma-Aldrich | 276855 | Anhydrous dimethyl sulfoxide, 99.9% |
| EDTA | Sigma-Aldrich | E6758 | Anhydrous, crystalline, BioReagent, suitable for cell culture |
| Formic acid | Merck | 64-18-6 | 98-100%, ACS reag, Ph Eur |
| Hexafluoro-2-propanol (HFIP) | Thermo Fisher Scientific | 920-66-1 | 99% Acros Organics |
| LC-MS sample vials | Thermo Fisher Scientific | C4000-11 | Plastic screw thread vials |
| LC-MS vial caps | Thermo Fisher Scientific | C5000-54A | Autosampler vial screw thread caps |
| Na2CO3 buffer | Sigma-Aldrich | 88975 | BioUltra, >0.1 M Na2CO3, >0.2 M NaHCO3 |
| Oligo Clean & Concentrator | Zymo Research | D4060 | Spin column |
| OriginLab | OriginLab | OriginPro | Data analysis and graphing software |
| pCp-biotin | TriLink BioTechnologies | NU-1706-BIO | 20 ul (1 mM) |
| RNA #1--#6 | Integrated DNA Technologies | Custom RNA oligos | 19nt-21nt single-stranded RNAs, used without further purification |
| Rocking platform shaker | VWR | Orbital Shaker Standard 1000 | Speed Range 40 to 300 rpm |
| Streptavidin magnetic beads | Thermo Fisher Scientific | 88816 | Binding approx. 55ug biotinylated rabbit lgG per mg of beads |
| Sulfonated Cyanine3 maleimide | Lumiprobe | 11380 | Water soluble |
| T4 DNA ligase 1 | New England Biolabs | M0202S | 400 units/uL |
| T4 polynucleotide kinase | Sigma-Aldrich | T4PNK-RO | From phage T4 am N81 pse T1 infected Escherichia coli BB |
| Tris-HCl buffer | Sigma-Aldrich | T6455 | Tris-HCl Buffer, pH 10, 10×, Antigen Retriever |
| Urea | Sigma-Aldrich | 81871 | Urea for synthesis. CAS No. 57-13-6, EC Number 200-315-5. |
Referencias
- Addepalli, B., Venus, S., Thakur, P., Limbach, P. A. Novel ribonuclease activity of cusativin from Cucumis sativus for mapping nucleoside modifications in RNA. Analytical and Bioanalytical Chemistry. 409 (24), 5645-5654 (2017).
- Gao, H., Liu, Y., Rumley, M., Yuan, H., Mao, B. Sequence confirmation of chemically modified RNAs using exonuclease digestion and matrix-assisted laser desorption/ionization time-of-flight mass spectrometry. Rapid Communications in Mass Spectrometry. 23 (21), 3423-3430 (2009).
- McLuckey, S. A., Van Berkel, G. J., Glish, G. L. Tandem mass spectrometry of small, multiply charged oligonucleotides. Journal of The American Society for Mass Spectrometry. 3 (1), 60-70 (1992).
- Fountain, K. J., Gilar, M., Gebler, J. C. Analysis of native and chemically modified oligonucleotides by tandem ion-pair reversed-phase high-performance liquid chromatography/electrospray ionization mass spectrometry. Rapid Communications in Mass Spectrometry. 17 (7), 646-653 (2003).
- Taucher, M., Breuker, K. Characterization of modified RNA by top-down mass spectrometry. Angewandte Chemie International Edition in English. 51 (45), 11289-11292 (2012).
- Kellner, S., Burhenne, J., Helm, M. Detection of RNA modifications. RNA Biology. 7 (2), 237-247 (2010).
- Thomas, B., Akoulitchev, A. V. Mass spectrometry of RNA. Trends in Biochemical Sciences. 31 (3), 173-181 (2006).
- Bjorkbom, A., et al. Bidirectional direct sequencing of noncanonical RNA by two-dimensional analysis of mass chromatograms. Journal of the American Chemical Society. 137 (45), 14430-14438 (2015).
- Zhang, N., et al. A general LC-MS-based RNA sequencing method for direct analysis of multiple-base modifications in RNA mixtures. Nucleic Acids Research. 47 (20), 125 (2019).
- Zhang, N., et al. Direct sequencing of tRNA by 2D-HELS-AA MS Seq reveals its different isoforms and dynamic base modifications. ACS Chemical Biology. 15 (6), 1464-1472 (2020).
- Bakin, A., Ofengand, J. Four newly located pseudouridylate residues in Escherichia coli 23S ribosomal RNA are all at the peptidyltransferase center: analysis by the application of a new sequencing technique. Biochemistry. 32 (37), 9754-9762 (1993).
- Cantara, W. A., et al. The RNA Modification Database, RNAMDB: 2011 update. Nucleic Acids Research. 39 (Database issue), D195-D201 (2011).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ISSN 2578-6326
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados
Utilizamos cookies para mejorar su experiencia en nuestra página web.
Al continuar usando nuestro sitio web o al hacer clic en 'Continuar', está aceptando nuestras cookies.