Method Article
2D-HELS MS SEQ: Общий метод на основе LC-MS для прямого и de novo секвенирования смесей РНК с различными модификациями нуклеотидов
В этой статье
Резюме
В данной статье мы описываем подробный протокол метода секвенирования на основе LC-MS, который может быть использован как прямой метод для секвенирования короткой РНК (<35 nt на прогон) без промежуточного продукта кДНК, так и в качестве общего метода для секвенирования различных модификаций нуклеотидов в одном исследовании с точностью до одного основания.
Аннотация
Было показано, что подходы к секвенированию, основанные на масс-спектрометрии (МС), полезны для прямого секвенирования РНК без необходимости использования промежуточного продукта комплементарной ДНК (кДНК). Тем не менее, такие подходы редко применяются в качестве метода секвенирования РНК de novo , а используются в основном в качестве инструмента, который может помочь в обеспечении качества для подтверждения известных последовательностей очищенных образцов одноцепочечной РНК. Недавно мы разработали метод прямого секвенирования РНК, интегрировав 2-мерную стратегию гидрофобного мечения концов по времени удержания массы в секвенирование на основе РС (2D-HELS MS Seq). Этот метод способен точно секвенировать как одиночные последовательности РНК, так и смеси, содержащие до 12 различных последовательностей РНК. В дополнение к четырем каноническим рибонуклеотидам (A, C, G и U), метод обладает способностью секвенировать РНК-олигонуклеотиды, содержащие модифицированные нуклеотиды. Это возможно потому, что модифицированное нуклеиновое основание либо обладает уникальной по своей природе массой, которая может помочь в его идентификации и размещении в последовательности РНК, либо может быть преобразовано в продукт с уникальной массой. В этом исследовании мы использовали РНК, включающую два репрезентативных модифицированных нуклеотида (псевдоуридин (Ψ) и 5-метилцитозин (m5C)), чтобы проиллюстрировать применение метода для секвенирования de novo одного олигонуклеотида РНК, а также смеси олигонуклеотидов РНК, каждый из которых имеет свою последовательность и/или модифицированные нуклеотиды. Описанные здесь процедуры и протоколы для секвенирования этих модельных РНК будут применимы к другим образцам коротких РНК (<35 нт) при использовании стандартной системы LC-MS высокого разрешения, а также могут быть использованы для верификации последовательности модифицированных терапевтических РНК-олигонуклеотидов. В будущем, с развитием более надежных алгоритмов и более совершенных инструментов, этот метод может позволить секвенировать более сложные биологические образцы.
Введение
Для прямого секвенирования РНК разработаны методы секвенирования, основанные на масс-спектрометрии (МС), в том числе нисходящая МСи тандемная МС 1,2,3,4. Тем не менее, методы фрагментации in situ для эффективного получения высококачественных РНК-лестниц в масс-спектрометрах в настоящее время не могут быть применены для секвенирования de novo 5,6. Кроме того, не очень тривиально проанализировать традиционные одномерные (1D) данные МС для de novo секвенирования даже одной очищенной последовательности РНК, а для МС секвенирования образцов смешанных РНК 7,8 это было бы еще сложнее. В связи с этим был разработан метод секвенирования РНК на основе двумерной (2D) жидкостной хроматографии (LC)-MS, включающий в себя производство двумерных лестниц времени удержания массы (tR) для замены 1D-лестниц массы, что значительно упрощает идентификацию компонентов лестницы, необходимых для de novo секвенирования РНК8. Тем не менее, метод секвенирования РНК на основе 2D LC-MS в основном ограничен очищенной синтетической короткой РНК, поскольку он не может считывать полную последовательность исключительно на основе одной единственной лестницы, но должен полагаться на две сосуществующие соседние лестницы (5'- и 3'-лестницы)8. В частности, этот подход требует двунаправленного чтения парных концов для чтения терминальных нуклеиновых оснований в области с малой массой8. Дополнительная сложность чтения парных концов приводит к тому, что этот метод несостоятелен для секвенирования смесей РНК, поскольку возникает путаница в вопросе о том, какой фрагмент лестницы к какой лестнице принадлежит для неизвестных образцов.
Чтобы преодолеть вышеупомянутые барьеры в подходах к секвенированию РНК на основе РС и расширить их применение в прямом секвенировании РНК, необходимо решить две проблемы: 1) как создать высококачественную лестницу масс, которую можно использовать для чтения полной последовательности, от первого нуклеотида до последнего в цепи РНК, и 2) как эффективно идентифицировать каждую лестницу РНК/массы в сложном наборе данных РС. Вместе с хорошо контролируемой кислотной деградацией мы разработали новый метод секвенирования, введя стратегию гидрофобного мечения концов (HELS) в технику секвенирования на основе МС, и успешно решили эти две проблемы, добавив гидрофобную метку на 5'- и/или 3'-конце РНК, подлежащих секвенированию9. Этот метод создает «идеальную» лестницу последовательностей из РНК — каждый фрагмент лестницы происходит из сайт-специфичного расщепления РНК исключительно в каждой фосфодиэфирной связи, а разница масс между двумя соседними фрагментами лестницы равна точной массе либо нуклеотида, либо нуклеотидной модификации в этой позиции 8,9,10. Это возможно, потому что мы включаем строго контролируемую стадию кислотного гидролиза, которая фрагментирует РНК в среднем один раз на молекулу, прежде чем она будет введена в инструмент. В результате каждый продукт деградации детектируется на масс-спектрометре и все фрагменты вместе образуют лестницу секвенирования 8,9,10. Эта новая стратегия позволяет полностью считывать последовательность РНК с одной единственной лестницы цепи РНК без чтения парных концов с другой лестницы РНК, а также позволяет проводить МС-секвенирование смесей РНК с несколькими различными цепями, которые содержат комбинаторные нуклеотидные модификации9. При добавлении метки на 5'- и/или 3'-конце РНК, меченые фрагменты лестницы демонстрируют значительную задержку tR, что может помочь отличить две массовые лестницы друг от друга, а также от шумной области с низкой массой. СдвигR массы-t, вызванный добавлением гидрофобной метки, облегчает идентификацию лестницы массы и упрощает анализ данных для генерации последовательностей. Кроме того, добавление гидрофобной метки может помочь идентифицировать концевое основание в цепи, предотвращая нахождение соответствующего фрагмента лестницы в зашумленной областиR с низкой массой из-за увеличения массы и гидрофобности, вызванного меткой, что позволяет идентифицировать полную последовательность РНК из одного лесенка; Чтение с парными концами не требуется. В результате, ранее мы продемонстрировали успешное секвенирование сложной смеси до 12 различных нитей РНК без использования какого-либо усовершенствованного алгоритма секвенирования9, что открывает двери для de novo MS-секвенирования РНК, содержащей как канонические, так и модифицированные нуклеотиды, и делает его более пригодным для секвенирования смешанных и более сложных образцов РНК. На самом деле, используя 2D-HELS MS Seq, мы даже успешно секвенировали смешанную популяцию образцов тРНК10 и активно расширяем его применение на другие сложные образцы РНК.
Чтобы облегчить 2D-HELS MS Seq для прямого секвенирования более широкого диапазона образцов РНК, здесь мы сосредоточимся на технических аспектах этого подхода к секвенированию и рассмотрим все основные шаги, необходимые при применении метода прямого секвенирования образцов РНК. Для иллюстрации метода секвенирования будут использованы конкретные примеры, включая синтетические одиночные последовательности РНК, смеси нескольких различных последовательностей РНК и модифицированные РНК, содержащие как канонические, так и модифицированные нуклеотиды, такие как псевдоуридин (ψ) и 5-метилцитозин (m5C). Поскольку все РНК содержат фосфодиэфирные связи, любой тип РНК может быть гидролизован кислотой для создания идеальной лестницы последовательностей для 2D-HELS MS Seq при оптимальных условиях 8,9. Однако обнаружение всех лестничных фрагментов данной РНК зависит от прибора. На стандартном LC-MS с высоким разрешением (40K) минимальная нагрузка для секвенирования очищенного образца короткой РНК (<35 нт) составляет 100 пмоль за прогон. Тем не менее, требуется больше материала (до 400 пмоль на образец РНК), когда необходимо провести дополнительные эксперименты (например, чтобы различить изомерные модификации оснований, которые имеют одинаковую массу). Протокол, используемый при секвенировании модельных синтетических модифицированных РНК, будет также применим для секвенирования более широких образцов РНК, включая образцы биологических РНК с неизвестными модификациями оснований. Тем не менее, для секвенирования полной тРНК со всеми модификациями требуется еще большее количество образца, например, 1000 пмоль для секвенирования тРНК (~76 нт) с использованием стандартного инструмента LC-MS, и для ее секвенирования de novo 10 должен быть разработан усовершенствованный алгоритм.
протокол
1. Дизайн РНК-олигонуклеотидов
- Конструируйте синтетические олигонуклеотиды РНК разной длины (19 нт, 20 нн и 21 нт), в том числе один (РНК #6) как с каноническими, так и с модифицированными нуклеотидами. ψ используется в качестве модели для модификаций, не изменяющих массу, что является сложной задачей для секвенирования MS, поскольку он имеет массу, идентичную U. m5C выбран в качестве модели для модификаций, изменяющих массу, чтобы продемонстрировать надежность подхода.
РНК #1: 5'-HO-CGCAUCUGACACCAAAA-OH-3'
РНК #2: 5'-HO-AUAGCCCAGUCAGUCUACGC-OH-3'
РНК #3: 5'-HO-AAACCGUUACCAUUACUGAG-OH-3'
РНК #4: 5'-HO-GCGUACAUUCCCCUUUUAU-OH-3'
РНК #5: 5'-HO-GCGGAUUUAGCUCAGUUGGA-OH-3'
РНК #6: 5'-HO-AAACCGUψACCAUUAm5CUGAG-OH-3'
- Растворите каждую синтетическую РНК в воде, обработанной диэтилпирокарбонатом (DEPC) без нуклеаз (выраженной как обработанный DEPC H2O, если не указано иное) для получения 100 мМ раствора РНК. Стоковые растворы хранятся длительное время при температуре -20 °С.
- Чтобы избежать возможной деградации образца РНК, используйте экспериментальные расходные материалы, не содержащие РНКазы, включая воду, обработанную DEPC, микроцентрифужные пробирки и наконечники для пипеток. Часто протирайте поверхности лабораторных принадлежностей салфетками для удаления РНКазы.
2. Пометьте 3'-конец РНК биотином
- Двухэтапный протокол реакции (аденилирование и лигирование)
- Добавьте 1 мкл 10-кратного реакционного буфера аденилирования, содержащего 50 мМ ацетата натрия, pH 6,0, 10 мМ MgCl2, 5 мМ дихлордифенилтрихлорэтана (DTT), 0,1 мМ этилендиаминтетрауксусной кислоты (ЭДТА), 1 мкл 1 мМ АТФ, 1 мкл 100 мкМ биотинилированного цитидина бисфосфата (pCp-биотин), 1 мкл 50 мкМ Mth РНК-лигазы и 6 мкл обработанного DEPC H2O (общий объем 10 мкл) в тонкостенную ПЦР-пробирку объемом 0,2 мл, не содержащую РНКазы.
ПРИМЕЧАНИЕ: Храните реагенты при температуре -20 °C перед двухступенчатой реакцией. Разморозьте реагенты при комнатной температуре и хорошо перемешайте путем вортексирования и центрифугирования перед добавлением в реакцию. - Инкубировать реакцию в ПЦР-аппарате при 65 °C в течение 1 ч и инактивировать реакцию при 85 °C в течение 5 мин.
- Провести стадию лигирования в тонкостенной ПЦР-пробирке объемом 0,2 мл без РНКазы, содержащей 10 мкл реакционного раствора предыдущей стадии, путем добавления 3 мкл 10x T4 реакционного буфера РНК-лигазы, содержащего 50 мМ трис(гидроксиметил)аминометана (Tris)-HCl, pH 7,8, 10 мМ MgCl2, 1 мМ DTT, 1,5 мкл из 100 мМ запаса образцов РНК, подлежащей секвенированию, 3 мкл безводного диметилсульфоксида (ДМСО) до 10% (v/v), 1 мкл T4 РНК-лигазы (10 ед/мкл) и 11,5 мкл обработанного DEPC H2O (общий объем 30 мл). Инкубируйте реакцию в течение ночи при 16 °C в ПЦР-машине.
ПРИМЕЧАНИЕ: Соедините компоненты реакции при комнатной температуре из-за высокой температуры замерзания ДМСО (18,45 °C). - Инкубируйте реакцию в течение ночи при 16 °C.
- Погасите и очистите реакцию путем очистки колонки для удаления ферментов и свободного pCp-биотина с помощью Oligo Clean & Concentrator (Zymo Research, Ирвайн, Калифорния, США). В комплект входят олигосвязывающий буфер, буфер для промывки ДНК, спин-колонки и пробирки для сбора. Добавьте 20 мл обработанного DEPC H2O в реакционный раствор до объема образца 50 мл перед добавлением связывающего буфера.
- Добавьте 100 мл связующего буфера в каждый реакционный раствор. Добавьте 400 μл этанола, перемешайте с помощью пипетирования и переложите смесь в колонку. Центрифуга при 10 000 x g в течение 30 с. Откажитесь от проточного потока.
- Добавьте в колонку 750 мкл буфера для промывки ДНК. Центрифуга при 10 000 x g и максимальной скорости в течение 30 с и 1 минуты соответственно.
- Перенесите колонку в микроцентрифужную пробирку объемом 1,5 мл. Добавьте в колонку 15 мкл обработанного DEPC H2O и центрифугируйте при давлении 10 000 x g в течение 30 с для элюирования РНК-продукта.
ПРИМЕЧАНИЕ: На этом этапе образцы могут храниться при температуре -20 °C до тех пор, пока не будет выполнен следующий этап.
- Добавьте 1 мкл 10-кратного реакционного буфера аденилирования, содержащего 50 мМ ацетата натрия, pH 6,0, 10 мМ MgCl2, 5 мМ дихлордифенилтрихлорэтана (DTT), 0,1 мМ этилендиаминтетрауксусной кислоты (ЭДТА), 1 мкл 1 мМ АТФ, 1 мкл 100 мкМ биотинилированного цитидина бисфосфата (pCp-биотин), 1 мкл 50 мкМ Mth РНК-лигазы и 6 мкл обработанного DEPC H2O (общий объем 10 мкл) в тонкостенную ПЦР-пробирку объемом 0,2 мл, не содержащую РНКазы.
- Протокол одноэтапной реакции
- Провести одностадийную реакцию мечения путем объединения 2 мкл 150 мкМ аденозин-5'-5'-дифосфат-{5'-(цитидин-2'-O-метил-3'-фосфат-TEG}C-биотин (AppCp-биотин), 3 мкл 10x лигазного реакционного буфера, 1,5 мкл из 100 мМ образца РНК, подлежащей секвенированию, 3 мкл безводного ДМСО для получения 10% (v/v), 1 мкл Т4 РНК-лигазы (10 единиц/мкл), и 19,5 мкл обработанного DEPC H2O (общий объем 30 мл) в микроцентрифужной пробирке объемом 1,5 мл без РНКазы.
- Инкубируйте реакцию в течение ночи при 16 °C в ПЦР-машине.
- Проведите очистку колонок, как описано выше в пунктах 2.1.5-2.1.8.
ПРИМЕЧАНИЕ: Подготовьте отдельную/эксклюзивную реакционную пробирку для каждого образца РНК (масштаб РНК 150 пмоль). Может потребоваться мечение 5'-конца РНК сульфо-цианином3 (Cy3) или Cy3 (например, для верификации двунаправленного секвенирования). Этот метод отличается от метода 3'-биотинилирования и описан в предыдущей публикации9.
3. Захват биотинилированной РНК на шариках стрептавидина
- Активируйте 200 мкл магнитных шариков стрептавидина C1, добавив 200 мкл 1x B&W буфера (5 мМ Tris-HCl, pH 7,5, 0,5 мМ ЭДТА, 1 М NaCl) в микроцентрифужную пробирку объемом 1,5 мл без РНКазы. Сделайте этот раствор вихрем и поместите его на магнитную подставку на 2 минуты. Затем выбросьте надосадочную жидкость, тщательно отпиливая раствор.
- Дважды промойте шарики 200 μL раствора A (0,1 M NaCl, обработанного DEPC и 0,05 M NaCl, обработанного DEPC) и один раз 200 μл раствора B (0,1 M NaCl, обработанного DEPC). На каждом этапе стирки раствор перебивается вихрем и помещается на магнитную подставку на 2 минуты, после чего удаляется надосадочная жидкость. Затем добавьте 100 μL 2x B&W буфера (10 мМ Tris-HCl, pH 7,5, 1 mM EDTA, 2 M NaCl).
- Добавьте 1x черно-белый буфер к биотинилированному образцу РНК до тех пор, пока объем не достигнет 100 мкл. Затем добавьте этот раствор к промытым шарикам, хранящимся в 100 мкл 2x черно-белого буфера. Инкубировать в течение 30 минут при комнатной температуре на качающейся платформе при 100 оборотах в минуту. Поместите трубку на магнитную подставку на 2 минуты и выбросьте надосадочную жидкость.
- Промойте покрытые бусины 3 раза в 1x черно-белом буфере и измерьте конечную концентрацию надосадочной жидкости на каждом этапе промывки с помощью Nanodrop для анализа восстановления, чтобы подтвердить, что целевые молекулы РНК остаются на бусинах.
- Инкубировать шарики в 10 мМ ЭДТА, pH 8,2 с 95% формамидом при 65 °C в течение 5 минут в ПЦР-машине. Подержите трубку на магнитной подставке в течение 2 минут и соберите надосадочную жидкость (содержащую биотинилированные РНК, высвобождаемые из шариков стрептавидина) с помощью пипета.
Примечание: Эта стадия физического разделения перед кислотной деградацией используется только для секвенирования РНК#1 на рисунке 1c и не является обязательной для 2D-HELS MS Seq, поскольку гидрофобная метка биотина может привести к тому, что 3'-меченые фрагменты лестницы будут иметь значительную задержку tR во время измерения LC-MS, что может четко отличать меченые 3'-лестничные фрагменты от немеченых 5'-лестничных фрагментов на графике 2D mass-tR .
4. Кислотный гидролиз РНК для получения МС-лестниц для секвенирования
- Разделите каждый образец РНК на три равные аликвоты. Например, разделить образец РНК объемом 15 мкл на три аликвоты по 5 мкл.
- Добавьте равный объем муравьиной кислоты для получения 50% (v/v) муравьиной кислоты в реакционной смеси 8,9.
- Инкубируйте реакцию при 40 °C в ПЦР-машине, причем одна реакция длится 2 мин, одна — 5 мин и одна — 15 мин соответственно.
- Угасите кислотное разложение, немедленно замораживая образец на сухом льду после завершения каждой реакции.
- Используйте центробежный вакуумный концентратор для сушки образца. Образец обычно полностью высушивают в течение 30 минут, и муравьиную кислоту удаляют вместе с H2O в процессе сушки, поскольку муравьиная кислота имеет температуру кипения (100,8 °C), аналогичную температуре кипения H2O (100 °C).
- Суспендируйте и объедините в общей сложности три высушенных образца в 20 мкл обработанного DEPC H2O для измерения ЖХ-МС.
ПРИМЕЧАНИЕ: На этом этапе образцы могут храниться при температуре -20 °C в ожидании измерения LC-MS.
5. Преобразование ψ в аддукт CMC-ψ
- Добавьте 80 мкл обработанного DEPC H2O в микроцентрифужную пробирку объемом 1,5 мл без РНКазы, содержащую 0,0141 г N-циклогексил-Nʹ-(2-морфолиноэтил)-карбодиимида мето--толуолсульфоната (КМЦ) и 0,07 г мочевины. Добавьте 10 мкл из 100 мкМ образца секвенируемой РНК, 8 мкл 1 М бицинового буфера (pH 8,3) и 1,28 мкл 0,5 М ЭДТА. Добавьте обработанный DEPC H2O, чтобы получить общий объем 160 μл. Конечные концентрации составляют 0,17 М КМЦ, 7 М мочевины и 4 мМ ЭДТА в 50 мМ бицине (pH 8,3)11.
Примечание: Этот протокол применим как к одной последовательности синтетической РНК, так и к смесям РНК. - Разделите реакционный раствор объемом 160 мкл на четыре равные аликвоты в тонкостенных ПЦР-пробирках объемом 0,2 мл без РНКазы и инкубируйте при температуре 37 °C в течение 20 мин в ПЦР-машине.
ПРИМЕЧАНИЕ: 50 μL на пробирку - это максимальный реакционный объем, который может быть использован в ПЦР-машине. - Гасите каждую реакцию 10 мкл 1,5 М ацетата натрия и 0,5 мМ ЭДТА (pH 5,6).
- Проведите очистку колонок с помощью четырех параллельных спиновых колонок для удаления избыточных реагентов в соответствии с процедурой, описанной в шагах 2.1.5-2.1.8. Растворите очищенный продукт в 15 мкл обработанного DEPC H2O в каждой микроцентрифужной пробирке объемом 1,5 мл без РНКазы.
- Переложите очищенный продукт в четыре тонкостенные ПЦР-пробирки объемом 0,2 мл без РНКазы. Добавьте 20 μL 0,1 M Na2CO3 буфера (pH 10,4) в каждые 15 μL очищенного продукта и добавьте обработанный DEPC H2O, чтобы получить окончательный объем 40 μL для каждой реакционной пробирки (всего четыре пробирки). Инкубируйте реакцию при 37 °C в течение 2 ч в ПЦР-аппарате.
- Утолите и очистите реакцию путем очистки колонки с помощью четырех параллельных спиновых колонок, как описано в шаге 2.1.5. Элюируйте преобразованный продукт CMC-ψ в микроцентрифужную пробирку объемом 1,5 мл без РНКазы каждая, добавив 15 мкл обработанного DEPC H2O.
- Объедините очищенный CMC-ψ преобразованный образец из четырех пробирок для сбора в одну пробирку. Выполните разложение муравьиной кислоты на 50% (v/v) в соответствии с процедурами, описанными в шагах 4.1-4.6, для создания лестниц MS для секвенирования.
6. Измерение ЖК-МС
- Подготовка подвижных фаз для измерения ЖХ-МС. Подвижная фаза А представляет собой 25 мМ гексафтор-2-пропанол с 10 мМ диизопропиламином в воде класса LC-MS; подвижная фаза В – метанол.
- Перенесите образец во флакон с образцом LC-MS для анализа. Объем каждого образца для инъекции составляет 20 μл, содержащий 100-400 пмоль РНК.
- Используйте следующие условия LC: температура столба 35 °C, расход 0,3 мл/мин; линейный градиент от 2–20% подвижной фазы B в течение 15 мин с последующим этапом промывки 2 мин с 90% подвижной фазой B.
ПРИМЕЧАНИЕ: Для более гидрофобных концевых этикеток, таких как Cy3 и sulfo-Cy3, как указано в разделе 2, для элюирования образца может потребоваться более высокий процент органического растворителя (т.е. можно использовать аналогичный градиент, но с увеличенным процентным диапазоном подвижной фазы В). Например, 2–38% подвижной фазы B в течение 30 мин с шагом стирки 2 мин с 90% подвижной фазы B. - Разделяйте и анализируйте образцы на масс-спектрометре Agilent Q-TOF (Quadrupole Time-of-Flight), подключенном к системе LC, оснащенной автосамплером и системой MS HPLC (High Performance Liquid Chromatography). Колонка LC представляет собой колонку C18 размером 50 мм x 2,1 мм с размером частиц 1,7 мкм. Используйте следующие настройки MS: режим отрицательных ионов; дальность, от 350 м/з до 3200 м/з; скорость сканирования, 2 спектра/с; расход сушильного газа, 17 л/мин; температура сушильного газа, 250 °C; давление в небулайзере, 30 фунтов на кв. дюйм изб; капиллярное напряжение, 3500 В; и напряжение фрагментора, 365 В. Обратите внимание, что эти параметры специфичны для типа или модели используемого масс-спектрометра.
- Собирайте данные с помощью программного обеспечения Agilent MassHunter для сбора данных. Используйте рабочий процесс молекулярного извлечения признаков (MFE) Agilent для извлечения информации о соединениях, включая массу, время удержания, объем (распространенность MFE для соответствующих ионов), показатель качества и т. д. Используйте следующие настройки MFE: "формат данных центроида, малые молекулы (хроматографические), пик с высотой ≥ 100, максимум до 1000, показатель качества ≥ 50".
ПРИМЕЧАНИЕ: Оптимизируйте настройки MFE, чтобы извлечь как можно больше потенциальных соединений, максимум до 1000, с показателями качества ≥ 50.
7. Автоматизировать генерацию последовательностей РНК с помощью вычислительного алгоритма
ПРИМЕЧАНИЕ: Эта процедура показана только для РНК #1 на рисунке 1c.
- Отсортируйте экстрагированные соединения MFE в порядке убывания объема (пиковой интенсивности) и tR. Выполните предварительный отбор данных следующим образом : 1) установив tR от 4 до 10 мин для выбора фрагментов РНК, меченных биотином, поскольку tRкомпонентов лестницы, меченных биотином, смещаются в это окно tR (от 4 мин до 10 мин), и 2) используя на порядок большее количество входных соединений, чем количество фрагментов лестницы, для вычисления алгоритма с целью уменьшения объема данных в зависимости от объема. Например, для 20 nt РНК для секвенирования 20nt РНК потребуется 20 меченых компонент лестницы mass-t R, и, таким образом, 200 соединений из файла данных MFE будут выбраны на основе объема. Обратите внимание, что окно tR может отличаться при использовании другого типа или модели масс-спектрометра.
- Выполнение обработки данных и генерации последовательностей РНК #1 с использованием переработанной версии опубликованного алгоритма8. Исходные коды пересмотренного алгоритма описаны ранее (https://academic-oup-com.remotexs.ntu.edu.sg/nar/article/47/20/e125/5558343#supplementary-data)9.
- В дополнение к автоматизации генерации последовательностей с помощью алгоритма, вручную рассчитайте разность масс между двумя соседними компонентами лестницы для вызова базы. Все основания в РНК могут быть вызваны вручную и сопоставлены с теоретическими в базе данных нуклеотидов и модификаций РНК8; таким образом, полная последовательность цепи РНК может быть точно прочитана вручную, что используется для подтверждения точности считывания последовательности, сообщенной алгоритмом. Больше структур модификаций РНК можно найти в базах данных модификаций РНК12, а их соответствующие теоретические массы получены с помощью ChemBioDraw. В таблицах S1–S2 разница в массе ppm (частей на миллион) показана при сравнении наблюдаемой массы с ее теоретической массой для конкретного компонента лестницы, и значение менее 10 ppm считается хорошим совпадением для каждого вызова основания.
8. Секвенирование смесей РНК
- Пометьте смесь из пяти цепей РНК (РНК от #1 до #5) на их 3'-концах биотином A(5')pp(5')Cp-TEG-биотином, используя одноэтапный протокол, описанный в шаге 2.2. В общий объем 150 мкл реакционного раствора добавить 15 мкл 10x T4 реакционного буфера РНК-лигазы, по 1,5 мкл каждой нити РНК (по 100 мкМ запас РНК от #1 до #5 соответственно, для общего объема 7,5 мкл), 10 мкл 150 мкМ A(5')pp(5')Cp-TEG-биотина, 15 мкл безводного ДМСО, 5 мкл Т4 РНК-лигазы (10 ед/мкл) и 97,5 мкл обработанного DEPC H2O. Равномерно распределите реакционный раствор на пять аликвот. Каждая микроцентрифужная пробирка, не содержащая РНКазы, содержит 30 мкл реакционного раствора.
- Инкубируйте реакцию в течение ночи при 16 °C в ПЦР-машине.
- Проведите очистку колонны в соответствии с процедурой, описанной в шагах 2.1.5-2.1.8, с пятью параллельными спиновыми колоннами. Разбавьте образец смеси 3'-биотинилированных 5 нитей РНК (смесь РНК от #1 до #5) в микроцентрифужную пробирку объемом 1,5 мл без РНКазы с 15 мкл обработанного DEPC H2O.
- Соедините очищенные образцы смеси из пяти пробирок для сбора в одну пробирку. Выполните разложение муравьиной кислоты в соответствии с процедурой, описанной в разделе 4.
- Измерьте образцы с помощью LC-MS, как описано в разделе 6, и проанализируйте данные с помощью программного обеспечения для анализа данных с оптимизированными настройками MFE для извлечения данных, содержащих массу, tR и объем, как описано в шаге 6.5. Типичный алгоритм обработки и вызова базы не применяется из-за значительно возросшей сложности данных, возникающей в результате смешивания. Все основания в РНК смешанного образца вызываются вручную в методе, аналогичном разделу 7.3, и хорошо согласуются с теоретическими основаниями в базе данных нуклеотидов и модификаций РНК8, таким образом, полные последовательности всех пяти цепей РНК в смешанном образце точно считываются. В таблицах S7–S11 перечислена вся информация, включая наблюдаемую массу,t R, объем, показатель качества и разницу в массе ppm.
Результаты
Введение метки биотина на 3'-конец РНК для получения легко идентифицируемых лестниц с массой tR . Рабочий процесс подхода 2D-HELS MS Seq показан на рисунке 1a. Гидрофобная метка биотина, вводимая на 3'-конец РНК (см. раздел 2), увеличивает массу и tR3'-меченых компонентов лестницы по сравнению с их немечеными аналогами. Таким образом, 3'-лестничная кривая смещается к большим значениям по оси y (из-за увеличения tRs) и смещается к большим значениям по оси x (из-за увеличения масс) на графике 2D mass-tR . На рисунке 1b показан протокол подготовки образца, включающий введение метки биотина на 3'-конец РНК для 2D-HELS MS Seq. На рисунке 1c показано отделение 3'-лестницы от 5'-лестницы и других нежелательных фрагментов на 2D-графике mass-tR на основе систематических изменений tRфрагментов 3'-биотин-меченных массой tR РНК #1. Одна только 3'-лестничная кривая дает полную последовательность РНК #1, а 5'-лестничная кривая, которая не показывает сдвиг tR , обеспечивает обратную последовательность, но требует конечного спаривания для чтения терминального основания8. При использовании этой стратегии 2D-HELS не требуется спаривание концов, как сообщалось ранее, и вся последовательность РНК может быть полностью прочитана только из одной меченой лестничной кривой8. Таким образом, можно секвенировать смешанные образцы, содержащие несколько РНК, например, две нити РНК разной длины (РНК #1 и РНК #2, 19 нт и 20 нт соответственно) с меткой 5'-биотина на каждой РНК (рис. 1d).
Преобразование ψ в аддукт CMC-ψ для 2D-HELS MS Seq. ψ является сложной модификацией нуклеотидов для секвенирования на основе РС, поскольку он имеет ту же массу, что и уридин (U). Чтобы отличить эти два основания друг от друга, мы обрабатываем РНК с помощью CMC, который превращает ψ в аддукт CMC-ψ (см. раздел 5). Аддукт имеет массу, отличную от U и может быть дифференцирован в 2D-HELS MS Seq. На рисунке 2a показан профиль ВЭЖХ сырого продукта реакции, превращающего ψ в его CMC-аддукт в РНК #6. Интегрируя их УФ-пики, мы рассчитали процент конверсии, и 42% ψ преобразуется в его аддукт CMC-ψ после процесса, проиллюстрированного в разделе 5. После измерения кислотной деградации и ЖХ-МС мы вручную получили последовательность на основе как не-CMC-преобразованных лестниц, так и CMC-преобразованных лестниц, идентифицированных по данным, обработанным алгоритмом 8,9. Красная кривая ответвляется вверх от серой кривой, начиная с ψ в положении 8 в РНК #6 (рис. 2b) из-за частичной конверсии ψ в аддукт CMC-ψ. Из-за массы и гидрофобности CMC это преобразование приводит к увеличению массы по Дальтону на 252,2076 и значительному увеличению tR для каждого компонента лестницы, содержащего аддукты CMC-ψ, по сравнению с его непреобразованным аналогом. Таким образом, резкий сдвиг, начинающийся с позиции 8 в РНК #6, можно наблюдать на графике 2D mass-tR, что указывает на то, что позиция 8 действительно является ψ в РНК #6.
Секвенирование смесей РНК. Смесь из пяти различных цепей РНК секвенируется с помощью подхода 2D-HELS MS Seq с 3'-концевой мечкой (см. раздел 8). Проблема секвенирования смешанных РНК заключается в том, что множественные лестничные кривые на 2D графике mass-tR могут перекрываться друг с другом, когда все они имеют одни и те же начальные точки (гидрофобная метка на графике 2D mass-tR). Тем не менее, базовые вызовы выполняются один за другим, каждый из которых основан на разнице в массе между двумя соседними фрагментами лестницы в данных MFE. Правильный вызов основания может быть сделан при условии, что каждая разница масс хорошо согласуется (разница PPM MS < 10) с одной из теоретических масс канонических или модифицированных нуклеотидов в пуле данных 8,9. При анализе образцов мультиплексированной РНК типичный алгоритм обработки и вызова оснований, использованный на рисунках 1 и 2, не используется в основном из-за значительно возросшей сложности данных, возникающей в результате смешивания. Эти последовательности вызываются вручную путем вычисления разницы масс между двумя соседними фрагментами лестницы масс и сравнения ее с теоретической массой нуклеотида в пуле данных9. Любое сопоставленное основание с массой PPM <10 выбирается в качестве тождества основания в этой позиции. С помощью этого ручного расчета для вызова базы все последовательности в смеси точно секвенированы. Программное обеспечение OriginLab используется для реконструкции 2D-графика массы-tR, в котором начальное tR для каждой последовательности систематически нормализуется для лучшей визуализации пяти различных последовательностей РНК (рис. 3). Без такой нормализации буквенные коды (т.е. A, C, G и U) для последовательностей всех пяти РНК были бы скучены друг к другу на графике (рисунок S1), что привело бы к меньшей простоте визуализации по сравнению с тем, что показано на рисунке 3. Результаты секвенирования показывают, что подход 2D-HELS MS Seq не ограничивается только секвенированием очищенных одноцепочечных РНК, но и, что более важно, смесей РНК с несколькими цепями РНК. В настоящее время разрабатываются алгоритмы для автоматизации процесса вызова базы и генерации последовательности.
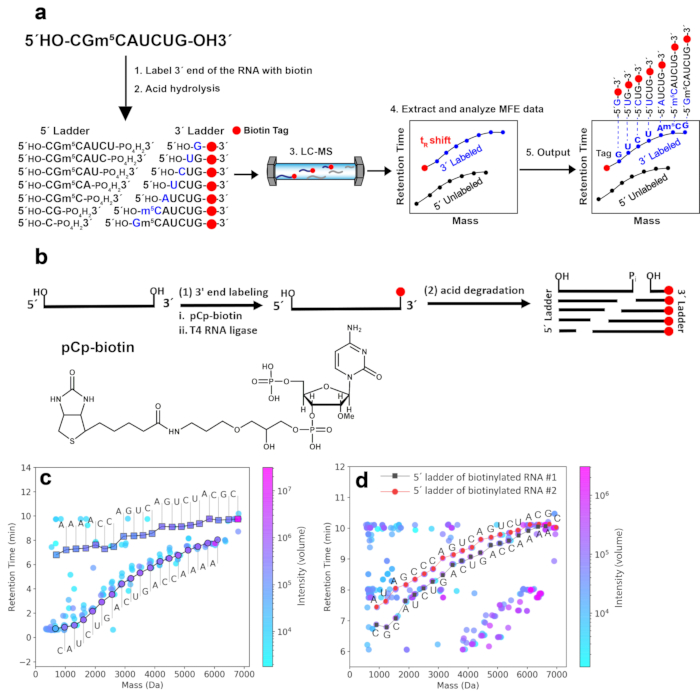
Рисунок 1. 2D-HELS MS Seq репрезентативных образцов РНК. (a) Рабочий процесс для 2D-HELS MS Seq. Основные этапы включают в себя: 1) гидрофобное мечение РНК, подлежащую секвенированию, 2) кислотный гидролиз, 3) измерение LC-MS, 4) экстракцию и анализ данных MFE и 5) генерацию последовательностей с помощью алгоритмов или ручных вычислений. (b) Протокол пробоподготовки, включающий введение метки биотина на 3'-конец РНК для 2D-HELS MS Seq. (c) Отделение 3'-лестницы от 5'-лестницы и других нежелательных фрагментов на 2D-графике времени удержания массы (tR) на основе систематических изменений tRс 3'-биотин-меченными массой tR фрагментов лестницы РНК #1 (19 nt). Последовательности являются de novo и автоматически считываются непосредственно алгоритмом вызова базы9. (d) Одновременное секвенирование меченых 5'-биотином РНК #1 и РНК #2, 19 нт и 20 нт соответственно. Методы введения метки биотина на 5'-конец РНК отличаются от методов 3'-биотинилирования, и их можно найти в предыдущем опубликованном протоколе9. 5'-конец двух РНК (РНК #1 и РНК #2) биотинилированы, и их 5'-биотинилированные лестницы могут быть легко идентифицированы; обе 5'-биотинилированные лестницы легко отделяются от их немеченых 3'-лестниц на 2D-графике массы-tR после LC-MS, потому что биотинилированные компоненты лестницы имеют большие сдвиги tR из-за гидрофобности биотина, в то время как немеченные компоненты лестницы находятся в нижней области tR. Несмотря на то, что 5'-лестницы и 3'-лестницы сосуществуют, они не препятствуют интерпретации последовательности двух смешанных нитей РНК. Каждая последовательность этих двух РНК вручную получена из 5'-биотинилированных лестниц на основе данных, обработанных вычислительным алгоритмом 8,9. Эта цифра была изменена по сравнению с Zhang et al.9. Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этой цифры.

Рисунок 2. Преобразование псевдоуридина (ψ) в его аддукт для 2D-HELS MS Seq. (a) Профиль ВЭЖХ сырого продукта реакции, превращающего ψ в его аддукт CMC в 20 nt РНК (РНК #6), содержащей одну ψ. (b) Секвенирование ψ-содержащей РНК #6. Преобразование ψ в аддукты CMC-ψ (ψ*) приводит к увеличению массы Дальтона на 252,2076 и значительному увеличению tR из-за его массы и гидрофобности CMC. Таким образом, на графике массы-tR можно наблюдать резкий сдвиг, начинающийся в положении 8, что указывает на то, что это ψ в положении 8 в последовательности РНК. Последовательности получены вручную на основе данных, обработанных вычислительным алгоритмом 8,9. Эта цифра была изменена по сравнению с Zhang et al.9. Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этой цифры.

Рисунок 3. Секвенирование смесей РНК, содержащих пять различных РНК. Биотин используется для маркировки каждой РНК на ее 3'-конце перед 2D-HELS MS Seq. Для каждой последовательности начальные значения tR систематически нормализуются с интервалом в 7 минут для удобства визуализации. Абсолютные различия между начальным значением tR и последующими tRs остаются неизменными для каждой из пяти РНК, и, таким образом, легче визуализировать каждую из них на одном графике. Все основания идентифицируются путем ручного вычисления разности масс двух смежных компонентов лестницы и сопоставления их с теоретическими разницами масс в базе данных нуклеотидов и модификаций РНК8; графики для рисунка 3 реконструированы с помощью OriginLab на основе данных ручного вызова оснований и секвенирования (см. раздел «Секвенирование смесей РНК» в разделе «Репрезентативные результаты»). Двумерная масса-tR фигуры пяти смешанных РНК без нормализации tR показана на рисунке S1. Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этой цифры.
Рисунок S1. Пожалуйста, нажмите здесь, чтобы скачать этот файл.
Обсуждение
В отличие от фрагментации МС на основе тандема, в подходе 2D-HELS MS Seq используется высококонтролируемый кислотный гидролиз для фрагментации РНК перед анализом с помощью масс-спектрометра 9,10. В результате каждый фрагмент, разложившийся кислотой, может быть обнаружен прибором, образуя эквивалент лестницы секвенирования. При оптимальных условиях этот метод создает «идеальную» лестницу последовательностей из РНК посредством, в среднем, расщепления сайт-специфичной РНК по одному на молекулу исключительно при фосфодиэфирной связи 8,9,10. После того, как каждый деградировавший фрагмент измеряется масс-спектрометром за один прогон, разница масс между двумя соседними лестничными фрагментами соответствует точной массе нуклеотида РНК или модификации в этом положении. Каждая модификация РНК либо имеет внутреннюю уникальную массу, которая может помочь идентифицировать и локализовать ее в РНК, либо может быть преобразована в РНК с уникальной массой. Таким образом, теоретически этот метод может сообщать об идентичности и расположении как канонических, так и модифицированных нуклеотидов для de novo и прямого секвенирования любой РНК. Тем не менее, различные лестницы последовательностей могут накладываться друг на друга, что усложняет анализ данных РС и затрудняет секвенирование РНК с помощью РС на практике.
Одно из преимуществ 3'-гидрофобной метки заключается в том, что она преодолевает главную проблему в любом методе фрагментации, а именно, что каждая молекула РНК должна быть расщеплена ровно на два фрагмента (и в идеале не более): один фрагмент содержит исходный 5'-конец, а другой содержит исходный 3'-конец РНК. Таким образом, при каждом событии расщепления образуются два фрагмента, образующие две лестницы — одну, измеренную от 5'-конца, а другую от 3'-конца. Всегда существует неопределенность в определении того, какой пик МС к какой лестнице принадлежит. Это становится более проблематичным в смеси нескольких различных РНК из-за генерации большого количества перекрывающихся лестниц последовательностей. Однако, поскольку все фрагменты лестницы с 3'-концов помечены гидрофобной меткой, они демонстрируют гораздо более длинную tRs (рис. 1a). В результате мы можем получить четкие и однозначные лестницы в 2D-данных mass-tR , полученных исключительно из 3'-меченой РНК. В частности, мы оптимизируем подходы к селективному мечению 5'- или 3'-конца любой РНК с использованием различных методов химической конъюгации9. Мы также можем выполнить двунаправленное секвенирование, которое здесь не используется для определения концевой базы (баз) терминала, но используется для предоставления идентичной информации о последовательности дважды при чтении как с 5'-, так и с 3'-направлением (т.е. двунаправленная проверка секвенирования), что еще больше повышает точность секвенирования.
Для de novo секвенирования неизвестных образцов РНК, особенно для сложных биологических образцов, требуется общий и надежный алгоритм для точной и эффективной обработки большого объема данных LC-MS для генерации последовательностей, который недавно стал доступен в других опубликованных работах. Несмотря на то, что эти алгоритмы использовались для секвенирования более сложных образцов10, в данном исследовании мы выполнили ручной вызов базы для генерации последовательности, если не указано иное. Мы стремимся охватить все ключевые этапы 2D-HELS MS Seq и хотели бы проиллюстрировать процесс, в ходе которого даже без использования дополнительных алгоритмов секвенирования мы все еще можем вручную считывать последовательности РНК, подлежащей секвенированию. Для упрощения визуализации и более быстрой идентификации фрагментов лестницы, необходимых для секвенирования на 2D-графике mass-tR , файлы MFE каждого прогона LC-MS обрабатываются пересмотренной версией опубликованного алгоритма8 перед чтением их последовательностей, если не указано иное. Опубликованный алгоритм не может быть использован непосредственно для считывания последовательностей из данных LC-MS, но часть его функции все же может быть использована для обработки данных — иерархическая кластеризация массовых аддуктов с помощью этого алгоритма увеличит интенсивность каждого компонента лестницы, что, в свою очередь, уменьшит сложность данных, особенно в критической области, где генерируются чтенияпоследовательностей. 9.
Одним из важнейших этапов подготовки образцов для 2D-HELS MS Seq является повышение эффективности маркировки концов РНК-гидрофобных меток. Высокая эффективность мечения может помочь уменьшить количество образца РНК, необходимого для генерации сигналов МС, на которые опираются данные секвенирования. Для повышения эффективности мечения мы применяем новые стратегии мечения, в том числе с использованием активированного AppCp-биотина, чтобы избежать этапа аденилирования при мечении 3'-конца РНК. Выход реакции для мечения 3'-конца 19-нт РНК биотином (см. шаг 2.2) может быть увеличен с 60% до ~95%9 с использованием этого одноэтапного метода. Благодаря эффективному мечению мы можем секвенировать смешанный образец, содержащий до 12 различных РНК, как описано ранее9. В этом исследовании мы используем смесь из пяти РНК в качестве репрезентативного примера, чтобы проиллюстрировать процесс секвенирования. Мы также обнаруживаем все фрагменты лестницы, необходимые для точного секвенирования, и считываем полные последовательности каждой из пяти последовательностей РНК в смеси. Более высокая эффективность маркировки не только помогает свести к минимуму объем загрузки образца, но и способствует значительному снижению сложности данных при последующем анализе данных для генерации последовательностей. В настоящее время разрабатываются новые реакции для достижения количественного выхода в мечении РНК как на 5', так и на 3'-концах.
При секвенировании РНК #1, как показано на рисунке 1c, этапы захвата и высвобождения стрептавидина используются для физического разделения биотинилированной РНК #1 перед кислотной деградацией (см. раздел 3). Это удаляет небольшую часть немеченой РНК и впоследствии приводит к большей простоте визуальной идентификации меченых массовых лестниц на 2D-графике массы-tR . Тем не менее, стадия физического разделения не является обязательной, поскольку биотинилированные фрагменты РНК-лестницы имеют задержку/увеличениеtRs из-за гидрофобности биотиновой метки по сравнению с их немечеными аналогами. Кроме того, вызов оснований не основан на физическом разделении, а опирается на разность масс соседних компонентов лестницы масс, таким образом, правильный вызов основания может быть достигнут при условии, что различия в массах двух соседних компонентов лестницы хорошо согласуются с соответствующими массами конкретного нуклеотида или модификации в нуклеотиде РНК и дате модификации8. В настоящее время разрабатывается вычислительный алгоритм для автоматизации вызова базы и генерации последовательностей.
Настройки MFE при экспорте исходных данных LC-MS (в файле типа .d) в файлы электронных таблиц имеют решающее значение для обработки данных и последующей генерации последовательностей (см. раздел 6.5). Например, мы протестировали настройку MFE «пик с высотой» в диапазоне от 100 до 1000 и заметили, что установка 100 может дать нам в 2 раза больше соединений, чем установка 1000. Чтобы не пропустить какие-либо компоненты лестницы, мы можем настроить настройку MFE во время рабочего процесса секвенирования. Эта настройка, вероятно, зависит от разрешения по массе прибора, количества фрагментов лестницы масс и сложности данных. Кроме того, важно использовать набор данных центроида и настройку хроматографического типа для малых молекул. Показатель качества может варьироваться от 50 % до 100 % в зависимости от качества данных.
Прибор LC-MS, который мы используем в исследовании, имеет верхнее массовое разрешение ~40K, что ограничивает метод только секвенированием РНК длиной менее 35 оснований. Однако точная длина чтения этого метода зависит от прибора; Более совершенные приборы с более высокой разрешающей способностью могут привести к увеличению длины считывания. Аналогичным образом, пропускная способность, т.е. количество последовательностей РНК может быть одновременно секвенировано в одном прогоне LC-MS, остается неизученным, хотя мы вручную секвенировали смесь образца РНК до 12 различных нитей РНК даже без использования какого-либо алгоритма. При текущем рабочем процессе требуется ~100 пмоль короткой РНК (<35 нт) для каждого прогона LC-MS. Количество нагрузки увеличивается, когда требуются дополнительные эксперименты: для дифференцировки изомерных нуклеотидных модификаций обычно требуется до 400 пмоль РНК. Для секвенирования специфических тРНК, таких как тРНКPhe, может потребоваться ~1000 пмоль образца для секвенирования и анализа модификаций. Тем не менее, мы ожидаем, что требуемое количество загружаемых образцов будет уменьшено на приборах LC-MS с более высокой чувствительностью. С улучшением эффективности маркировки образцов, алгоритма секвенирования, а также чувствительности и разрешения прибора мы ожидаем, что наш метод будет применим к более широкому спектру образцов РНК, особенно с различными модификациями РНК.
Раскрытие информации
Авторы подали заявку на предварительный патент, относящийся к технологии, обсуждаемой в данной рукописи.
Благодарности
Авторы выражают признательность за грант R21 от Национальных институтов здравоохранения (1R21HG009576) для S. Z. и W. L. и гранты Нью-Йоркского технологического института (NYIT) на институциональную поддержку исследований и творчества для S. Z., которая поддержала эту работу. Авторы хотели бы поблагодарить аспиранта Сюаньтин Ван (Колумбийский университет) за помощь в создании фигур, а также профессора Майкла Хаджиаргиру (NYIT), профессора Цзинъюэ Джу (Колумбийский университет), докторов Джеймса Руссо, Шива Кумара, Сяосюй Ли, Стеффена Джокуша и других членов лаборатории Цзюй (Колумбийский университет), доктора Юндонга Вана (Cerno Bioscience), Мейну Азиз (NYIT) и Вэньхао Ни (NYIT) за полезные обсуждения и предложения по нашей рукописи.
Материалы
| Name | Company | Catalog Number | Comments |
| 5' DNA Adenylation kit | New England Biolabs | E2610S | 50uM concentration |
| 6550 Q-TOF mass spectrometer | Agilent Technologies | 5991-2116EN | Coupled to a 1290 Infinity LC system |
| A(5´)pp(5´)Cp-TEG-biotin-3´ | ChemGenes | 91718 | HPLC purified |
| ATPγS | Sigma-Aldrich | 11162306001 | Lithium salt |
| Bicine | Sigma-Aldrich | B8660 | BioXtra, ≥99% (titration) |
| Biotin maleimide | Vector Laboratories | SP-1501 | Long arm |
| C18 column | Waters | 186003532 | 50 mm × 2.1 mm Xbridge C18 column with a particle size of 1.7 μm |
| Centrifugal Vacuum Concentrator | Labconco | Refrig 115v/60hz 7310022 | Labconco CentriVap |
| ChemBioDraw | PerkinElmer | ChemDraw Prime | Generate a chemical structure and property data of structures & fragments |
| CMC (N-cyclohexyl-N?-(2-morpholinoethyl)-carbodiimide metho-p-toluenesulfonate) | Sigma-Aldrich | 2491-17-0 | 95% Purifiy |
| Cyanine3 maleimide (Cy3) | Lumiprobe | 11080 | Water insoluble |
| DEPC-treated water | Thermo Fisher Scientific | AM9906 | Autoclaved, certified nuclease-free |
| Diisopropylamine (DIPA) | Thermo Fisher Scientific | 108-18-9 | 99% Alfa Aesar |
| DMSO | Sigma-Aldrich | 276855 | Anhydrous dimethyl sulfoxide, 99.9% |
| EDTA | Sigma-Aldrich | E6758 | Anhydrous, crystalline, BioReagent, suitable for cell culture |
| Formic acid | Merck | 64-18-6 | 98-100%, ACS reag, Ph Eur |
| Hexafluoro-2-propanol (HFIP) | Thermo Fisher Scientific | 920-66-1 | 99% Acros Organics |
| LC-MS sample vials | Thermo Fisher Scientific | C4000-11 | Plastic screw thread vials |
| LC-MS vial caps | Thermo Fisher Scientific | C5000-54A | Autosampler vial screw thread caps |
| Na2CO3 buffer | Sigma-Aldrich | 88975 | BioUltra, >0.1 M Na2CO3, >0.2 M NaHCO3 |
| Oligo Clean & Concentrator | Zymo Research | D4060 | Spin column |
| OriginLab | OriginLab | OriginPro | Data analysis and graphing software |
| pCp-biotin | TriLink BioTechnologies | NU-1706-BIO | 20 ul (1 mM) |
| RNA #1--#6 | Integrated DNA Technologies | Custom RNA oligos | 19nt-21nt single-stranded RNAs, used without further purification |
| Rocking platform shaker | VWR | Orbital Shaker Standard 1000 | Speed Range 40 to 300 rpm |
| Streptavidin magnetic beads | Thermo Fisher Scientific | 88816 | Binding approx. 55ug biotinylated rabbit lgG per mg of beads |
| Sulfonated Cyanine3 maleimide | Lumiprobe | 11380 | Water soluble |
| T4 DNA ligase 1 | New England Biolabs | M0202S | 400 units/uL |
| T4 polynucleotide kinase | Sigma-Aldrich | T4PNK-RO | From phage T4 am N81 pse T1 infected Escherichia coli BB |
| Tris-HCl buffer | Sigma-Aldrich | T6455 | Tris-HCl Buffer, pH 10, 10×, Antigen Retriever |
| Urea | Sigma-Aldrich | 81871 | Urea for synthesis. CAS No. 57-13-6, EC Number 200-315-5. |
Ссылки
- Addepalli, B., Venus, S., Thakur, P., Limbach, P. A. Novel ribonuclease activity of cusativin from Cucumis sativus for mapping nucleoside modifications in RNA. Analytical and Bioanalytical Chemistry. 409 (24), 5645-5654 (2017).
- Gao, H., Liu, Y., Rumley, M., Yuan, H., Mao, B. Sequence confirmation of chemically modified RNAs using exonuclease digestion and matrix-assisted laser desorption/ionization time-of-flight mass spectrometry. Rapid Communications in Mass Spectrometry. 23 (21), 3423-3430 (2009).
- McLuckey, S. A., Van Berkel, G. J., Glish, G. L. Tandem mass spectrometry of small, multiply charged oligonucleotides. Journal of The American Society for Mass Spectrometry. 3 (1), 60-70 (1992).
- Fountain, K. J., Gilar, M., Gebler, J. C. Analysis of native and chemically modified oligonucleotides by tandem ion-pair reversed-phase high-performance liquid chromatography/electrospray ionization mass spectrometry. Rapid Communications in Mass Spectrometry. 17 (7), 646-653 (2003).
- Taucher, M., Breuker, K. Characterization of modified RNA by top-down mass spectrometry. Angewandte Chemie International Edition in English. 51 (45), 11289-11292 (2012).
- Kellner, S., Burhenne, J., Helm, M. Detection of RNA modifications. RNA Biology. 7 (2), 237-247 (2010).
- Thomas, B., Akoulitchev, A. V. Mass spectrometry of RNA. Trends in Biochemical Sciences. 31 (3), 173-181 (2006).
- Bjorkbom, A., et al. Bidirectional direct sequencing of noncanonical RNA by two-dimensional analysis of mass chromatograms. Journal of the American Chemical Society. 137 (45), 14430-14438 (2015).
- Zhang, N., et al. A general LC-MS-based RNA sequencing method for direct analysis of multiple-base modifications in RNA mixtures. Nucleic Acids Research. 47 (20), 125(2019).
- Zhang, N., et al. Direct sequencing of tRNA by 2D-HELS-AA MS Seq reveals its different isoforms and dynamic base modifications. ACS Chemical Biology. 15 (6), 1464-1472 (2020).
- Bakin, A., Ofengand, J. Four newly located pseudouridylate residues in Escherichia coli 23S ribosomal RNA are all at the peptidyltransferase center: analysis by the application of a new sequencing technique. Biochemistry. 32 (37), 9754-9762 (1993).
- Cantara, W. A., et al. The RNA Modification Database, RNAMDB: 2011 update. Nucleic Acids Research. 39 (Database issue), D195-D201 (2011).
Перепечатки и разрешения
Запросить разрешение на использование текста или рисунков этого JoVE статьи
Запросить разрешениеСмотреть дополнительные статьи
This article has been published
Video Coming Soon
Авторские права © 2025 MyJoVE Corporation. Все права защищены