Method Article
2D-HELS MS Seq:異なるヌクレオチド修飾を持つRNA混合物の直接およびde novoシーケンシングのための一般的なLC-MSベースの方法
要約
ここでは、cDNA中間体を使用せずに短いRNA(ランあたり<35 nt)をシーケンシングする直接的な方法として、また、単一の研究で異なるヌクレオチド修飾を単一塩基の精度でシーケンシングする一般的な方法として使用できるLC-MSベースのシーケンシング法の詳細なプロトコールについて説明します。
要約
質量分析(MS)ベースのシーケンシングアプローチは、相補的DNA(cDNA)中間体を必要とせずにRNAを直接シーケンシングするのに有用であることが示されています。しかし、このようなアプローチは de novo RNAシーケンシング法として適用されることはほとんどなく、主に精製された一本鎖RNAサンプルの既知の配列を確認するための品質保証を支援するツールとして使用されています。最近、私たちは、2次元質量保持時間疎水性末端標識戦略をMSベースのシーケンシング(2D-HELS MS Seq)に統合することにより、ダイレクトRNAシーケンシング法を開発しました。この方法は、単一のRNA配列だけでなく、最大12の異なるRNA配列を含む混合物も正確にシーケンシングすることができます。この分析法には、4つの標準的なリボヌクレオチド(A、C、G、U)に加えて、修飾ヌクレオチドを含むRNAオリゴヌクレオチドのシーケンシング能力があります。これが可能なのは、修飾された核酸塩基が、その同定とRNA配列内の位置に役立つ固有の固有の質量を持っているか、または固有の質量を持つ製品に変換できるためです。本研究では、代表的な修飾ヌクレオチド2種(シュードウリジン(Ψ)と5-メチルシトシン(m5C))を組み込んだRNAを用いて、単一のRNAオリゴヌクレオチドと、それぞれ異なる配列および/または修飾ヌクレオチドを持つRNAオリゴヌクレオチドの混合物の de novo シーケンシング法の適用例を示しました。これらのモデル RNA のシーケンシングに関する本書に記載されている手順とプロトコールは、標準的な高分解能 LC-MS システムを使用する場合、他のショート RNA サンプル(<35 nt)に適用でき、修飾された治療用 RNA オリゴヌクレオチドのシーケンシング検証にも使用できます。将来的には、より堅牢なアルゴリズムとより優れた機器の開発により、この方法はより複雑な生物学的サンプルのシーケンシングを可能にする可能性があります。
概要
トップダウンMSやタンデムMS 1,2,3,4などの質量分析(MS)ベースのシーケンシング法は、RNAの直接シーケンシングのために開発されました。しかし、質量分析計で高品質のRNAラダーを効果的に生成するためのin situフラグメンテーション技術は、現在、de novoシーケンシングには適用できません5,6。さらに、精製されたRNA配列を1つだけde novoシーケンシングするために従来の1次元(1D)MSデータを分析することは非常に簡単ではなく、混合RNAサンプルのMSシーケンシングはさらに困難になります7,8。したがって、2次元(2D)液体クロマトグラフィー(LC)-MSベースのRNAシーケンシング法が開発され、1D質量ラダーに代わる2D質量保持時間(tR)ラダーの生成が組み込まれ、RNA8のde novoシーケンシングに必要なラダー成分の同定がはるかに容易になりました。しかし、2D LC-MSベースのRNAシーケンシング法は、1つのラダーのみに基づいて完全な配列を読み取ることはできず、2つの共存する隣接するラダー(5'-および3'-ラダー)8に依存する必要があるため、主に精製された合成短RNAに限定されます。より具体的には、このアプローチでは、低質量領域8の末端核酸塩基を読み取るために、双方向のペアエンドリードが必要です。ペアエンド読み取りの複雑さが増すと、未知のサンプルのどのラダーフラグメントがどのラダーに属するかについて混乱が生じるため、この方法はRNA混合物のシーケンシングには適しなくなります。
MSベースのRNAシーケンシングアプローチにおける上記の障壁を克服し、ダイレクトRNAシーケンシングにおけるそのようなアプリケーションを拡大するには、2つの問題に対処する必要があります:1)RNA鎖の最初のヌクレオチドから最後のヌクレオチドまでの完全な配列を読み取るために使用できる高品質のマスラダーをどのように作製するか、2)複雑なMSデータセットで各RNA/マスラダーを効果的に同定する方法。我々は、酸分解の制御を十分に制御するとともに、MSベースのシーケンシング技術に疎水性末端標識戦略(HELS)を導入することで新しいシーケンシング法を開発し、シーケンシングするRNAの5'末端および/または3'末端に疎水性タグを追加することで、これら2つの問題にうまく対処した9。この方法は、RNAから「理想的な」配列ラダーを作成します—各ラダーフラグメントは、各ホスホジエステル結合のみでの部位特異的なRNA切断に由来し、2つの隣接するラダーフラグメント間の質量差は、その位置でのヌクレオチドまたはヌクレオチド修飾のいずれかの正確な質量です8,9,10.これが可能なのは、高度に制御された酸性加水分解ステップが含まれており、RNAを装置に注入する前に、分子ごとに平均して1回断片化するためです。その結果、各分解フラグメント生成物は質量分析計で検出され、すべてのフラグメントが一緒になってシーケンシングラダー8,9,10を形成する。この新しい戦略により、RNAの一方のラダーからRNA配列を完全に読み取ることができ、もう一方のRNAラダーからペアエンドを読み取ることなく、さらに、組み合わせヌクレオチド修飾を含む複数の異なる鎖を持つRNA混合物のMSシーケンシングが可能になります9。RNAの5'末端および/または3'末端にタグを追加することで、標識されたラダーフラグメントはtRの有意な遅延を示し、2つの質量ラダーを互いに区別したり、ノイズの多い低質量領域から区別したりするのに役立ちます。疎水性タグの追加によるmass-tRシフトにより、質量ラダーの同定が容易になり、シーケンス生成のためのデータ解析が簡素化されます。さらに、疎水性タグの添加は、タグによって引き起こされる質量および疎水性の増加により、対応するラダーフラグメントがノイズの多い低質量-t R領域に存在するのを防ぐことにより、ストランドの末端基部を同定するのに役立ち、したがって、単一のラダーからRNAの完全な配列を同定することを可能にする。ペアエンドリードは必要ありません。その結果、高度なシーケンシングアルゴリズムを使用せずに、最大12の異なるRNA鎖の複雑な混合物のシーケンシングに成功したこと9をこれまでに実証しており、これにより、標準ヌクレオチドと修飾ヌクレオチドの両方を含むRNAのde novo MSシーケンシングへの扉が開かれ、混合されたより複雑なRNAサンプルのシーケンシングがより実現可能になります。実際、2D-HELS MS Seqを用いて、tRNAサンプル10の混合集団のシーケンシングに成功し、その応用を他の複雑なRNAサンプルにも積極的に拡大しています。
2D-HELS MS Seqが広範囲のRNAサンプルを直接シーケンシングできるように、ここではこのシーケンシングアプローチの技術的側面に焦点を当て、RNAサンプルの直接シーケンシングに技術を適用する際に必要なすべての重要なステップについて説明します。合成単一RNA配列、複数の異なるRNA配列の混合物、およびシュードウリジン(ψ)および5-メチルシトシン(m5C)などの標準ヌクレオチドおよび修飾ヌクレオチドの両方を含む修飾RNAを含む、シーケンシング技術を説明するために具体例を使用する。すべてのRNAはホスホジエステル結合を含んでいるため、あらゆるタイプのRNAを酸加水分解して、最適な条件下で2D-HELS MS Seqの理想的な配列ラダーを生成することができます8,9。ただし、特定のRNAのすべてのラダーフラグメントの検出は、機器によって異なります。標準的な高分解能 LC-MS(40K)では、精製された短い RNA サンプル(<35 nt)のシーケンシングの最小ローディング量は、1 回あたり 100 pmol です。ただし、追加の実験を行う必要がある場合(たとえば、同一の質量を共有する異性体塩基修飾を区別するため)には、より多くの材料が必要です(RNAサンプルあたり最大400 pmol)。モデル合成修飾RNAのシーケンシングに使用されるプロトコルは、未知の塩基修飾を持つ生物学的RNAサンプルを含む、より広範なRNAサンプルのシーケンシングにも適用できます。ただし、すべての修飾を含む完全なtRNAをシーケンシングするには、標準的なLC-MS機器を使用してtRNAをシーケンシングするための1000 pmol(~76 nt)など、さらに大量のサンプルが必要であり、そのde novoシーケンシング10には高度なアルゴリズムを開発する必要があります。
プロトコル
1. RNAオリゴヌクレオチドの設計
- 異なる長さ(19 nt、20 nt、21 nt)の合成RNAオリゴヌクレオチドを設計し、そのうちの1つ(RNA #6)は標準ヌクレオチドと修飾ヌクレオチドの両方を含みます。ψは、質量を変化させない修飾のモデルとして採用されていますが、これは U.m と同一の質量を持つため、MS シーケンシングでは困難です。このアプローチの堅牢性を実証するために、質量を変化させる修飾のモデルとして 5 C が選択されています。
RNA #1: 5'-HO-CGCAUCUGACUGACCAAAAA-OH-3'
RNA #2: 5'-HO-AUAGCCCAGUCAGUACGC-OH-3'
RNA #3: 5'-HO-AAACCGUUACCAUUACUGAG-OH-3'
RNA #4: 5'-HO-GCGUACAUCUUCCCCUUUAU-OH-3'
RNA #5: 5'-HO-GCGGAUUUAGCUCAGUUGGA-OH-3'
RNA #6: 5'-HO-AAACCGUψACCAUUAm5CUGAG-OH-3'
- 各合成RNAをヌクレアーゼフリーのジエチルピロカーボネート(DEPC)処理水(特に指定がない限りDEPC処理H2Oと表される)に溶解して、100 mM RNAストック溶液を得ます。ストック溶液は-20°Cで長期保存します。
- RNAサンプルの分解を避けるために、DEPC処理水、微量遠心チューブ、ピペットチップなど、RNaseフリーの実験用消耗品を使用してください。RNase除去ワイプを使用して、ラボ用品の表面を頻繁に拭きます。
2. RNAの3'末端をビオチンで標識します。
- 2段階反応プロトコール(アデニル化とライゲーション)
- 50 mM 酢酸ナトリウム、pH 6.0、10 mM MgCl2、5 mM ジクロロジフェニルトリクロロエタン (DTT)、0.1 mM エチレンジアミン四酢酸 (EDTA)、1 μL 1 μL 1 mM ATP、100 μM ビオチン化シチジンビスリン酸 (pCp-ビオチン) 1 μL 1 μL、50 μM Mth RNA リガーゼ 1 μL および 6 μL の DEPC 処理 H2O (総容量 10 μL) を含む 10x アデニル化反応バッファーを RNase フリーの薄肉 0.2 mL PCR チューブに加えます。
注:試薬は、2ステップ反応の前に-20°Cで保存してください。試薬を室温で解凍し、ボルテックスと遠心分離でよく混合してから反応に加えます。 - PCR装置で反応液を65°Cで1時間インキュベートし、85°Cで5分間反応液を不活性化します。
- 前のステップの反応液10 μLを含むRNaseフリーの薄肉0.2 mL PCRチューブで、50 mM tris(hydroxymethyl)aminomethane (Tris)-HCl、pH 7.8、10 mM MgCl2、1 mM DTT、シーケンシングするRNAの100 mMサンプルストックの1.5 μLを含む10x T4 RNAリガーゼ反応バッファーを3 μL添加してライゲーションステップを行います。 10%(v/v)に達するには3 μLの無水ジメチルスルホキシド(DMSO)、1 μLのT4 RNAリガーゼ(10 units/μL)、および11.5 μLのDEPC処理H2O(総容量30 mL)が必要です。反応液をPCR装置で16°Cで一晩インキュベートします。
注:DMSOは凝固点が高い(18.45°C)ため、反応成分を室温で組み合わせてください。 - 反応液を16°Cで一晩インキュベートします。
- Oligo Clean & Concentrator(Zymo Research、カリフォルニア州アーバイン、米国)を使用して、カラム精製により反応をクエンチおよび精製し、酵素と遊離pCp-ビオチンを除去します。オリゴ結合バッファー、DNA洗浄バッファー、スピンカラム、コレクションチューブがキットに含まれています。Binding Bufferを添加する前に、反応溶液に20 mLのDEPC処理H2Oを加えてサンプル容量を50 mLにします。
- 各反応溶液に100 mLの結合バッファーを加えます。エタノール400μLを加え、ピペッティングで混合し、カラムに移します。10,000 x g で30秒間遠心分離します。フロースルーを破棄します。
- 750 μL の DNA Wash Buffer をカラムに加えます。10,000 x g で遠心分離し、最大速度をそれぞれ30秒と1分間遠心分離します。
- カラムを1.5 mLの微量遠心チューブに移します。DEPC処理したH2O15 μLをカラムに加え、10,000 x g で30秒間遠心分離してRNA産物を溶出します。
注:この段階では、次のステップが実行されるまで、サンプルを-20°Cで保存できます。
- 50 mM 酢酸ナトリウム、pH 6.0、10 mM MgCl2、5 mM ジクロロジフェニルトリクロロエタン (DTT)、0.1 mM エチレンジアミン四酢酸 (EDTA)、1 μL 1 μL 1 mM ATP、100 μM ビオチン化シチジンビスリン酸 (pCp-ビオチン) 1 μL 1 μL、50 μM Mth RNA リガーゼ 1 μL および 6 μL の DEPC 処理 H2O (総容量 10 μL) を含む 10x アデニル化反応バッファーを RNase フリーの薄肉 0.2 mL PCR チューブに加えます。
- ワンステップ反応プロトコール
- 2 μL の 150 μM アデノシン-5'-5'-ジリン酸-{5'-(シチジン-2'-O-メチル-3'-リン酸-TEG}C-ビオチン (AppCp-ビオチン)、3 μL の 10x リガーゼ反応バッファー、1.5 μL のシーケンシング対象 RNA の 100 mM サンプルストック、3 μL の無水 DMSO を 10% (v/v) に到達、1 μL の T4 RNA リガーゼ (10 units/μL) を組み合わせて 1 ステップ標識反応を行います。 19.5 μL の DEPC 処理 H2O (総容量 30 mL) を 1.5 mL の RNase フリー微量遠心チューブに入れます。
- 反応液をPCR装置で16°Cで一晩インキュベートします。
- 上記のステップ2.1.5-2.1.8で説明したようにカラム精製を行います。
注:各RNAサンプル(RNAの150 pmolスケール)に対して、個別の/排他的な反応チューブを準備します。RNAの5'末端をスルホシアニン3(Cy3)またはCy3で標識することが必要な場合があります(例えば、双方向シーケンシングの検証のため)。この方法は3'-ビオチン化の方法とは異なり、以前の論文9に記載されている。
3. ビオチン化RNAサンプルをストレプトアビジンビーズに捕捉
- 1.5 mL RNaseフリーの微量遠心チューブに 200 μL の 1x B&W バッファー (5 mM Tris-HCl、pH 7.5、0.5 mM EDTA、1 M NaCl) を添加して、200 μL のストレプトアビジン C1 マグネットビーズを活性化します。この溶液をボルテックスし、マグネットスタンドに2分間置きます。次に、溶液を慎重にピペッティングして上清を捨てます。
- ビーズを200 μLの溶液A(DEPC処理0.1 M NaOHおよびDEPC処理0.05 M NaCl)で2回、200 μLの溶液B(DEPC処理0.1 M NaCl)で1回洗浄します。各洗浄ステップで、溶液をボルテックスしてマグネットスタンドに2分間置き、続いて上清を捨てます。次に、2x B&Wバッファー(10 mM Tris-HCl、pH 7.5、1 mM EDTA、2 M NaCl)100 μLを加えます。
- ビオチン化RNAサンプルに1x B&Wバッファーを添加し、容量が100 μLになるまで加えます。次に、この溶液を100 μLの2x B&Wバッファーに保存した洗浄済みビーズに加えます。100 rpmのロッキングプラットフォームシェーカーで室温で30分間インキュベートします。チューブをマグネットスタンドに2分間置き、上清を捨てます。
- コーティングしたビーズを1x B&Wバッファーで3回洗浄し、各洗浄ステップで上清の最終濃度をNanodropで測定して回収分析を行い、目的のRNA分子がビーズに残っていることを確認します。
- ビーズを10 mM EDTA、pH 8.2、95%ホルムアミド、65°C、65°Cで5分間PCRマシンでインキュベートします。チューブをマグネットスタンドに2分間置いておき、上清(ストレプトアビジンビーズから放出されたビオチン化RNAを含む)をピペットで回収します。
注:酸分解前のこの物理的分離ステップは、 図 1c の RNA#1 のシーケンシングにのみ使用され、疎水性ビオチン標識により LC-MS 測定中に 3' 標識ラダーフラグメントの tR が大幅に遅延する可能性があるため、2D-HELS MS Seq では必須ではありません。これにより、2D mass-tR プロットで標識された 3'-ラダーフラグメントと標識されていない 5'-ラダーフラグメントを明確に区別できます。
4. シーケンシング用のMSラダーを生成するためのRNAの酸加水分解
- 各RNAサンプルを3つの等しいアリコートに分割します。例えば、15 μL の RNA サンプルを 5 μL の 3 つのアリコートに分割します。
- 等量のギ酸を添加して、反応混合物8,9で50%(v / v)ギ酸を達成します。
- PCR装置で反応を40°Cでインキュベートし、1つの反応をそれぞれ2分間、1つの反応を5分間、1つの反応を15分間実行します。
- 酸分解を急冷するには、各反応終了後すぐにサンプルをドライアイスで凍結します。
- 遠心真空濃縮器を使用してサンプルを乾燥させます。通常、サンプルは30分以内に完全に乾燥され、ギ酸の沸点(100.8°C)はH2O(100°C)と同程度であるため、乾燥プロセス中にヒ酸はH2Oとともに除去されます。
- LC-MS測定のために、DEPC処理したH2Oの20 μLに合計3つの乾燥サンプルを懸濁して組み合わせます。
注:この段階では、LC-MS測定を待つ間、サンプルを-20°Cで保存できます。
5. ψをCMC-ψ付加物に変換する
- 0.0141 gのN-シクロヘキシル-Nʹ-(2-モルフォリノエチル)-カルボジイミドメト-p-トルエンスルホン酸(CMC)と0.07 gの尿素を含む1.5 mLのRNaseフリー微量遠心チューブに、DEPC処理したH2O80 μLを加えます。シーケンシングするRNAの100 μMサンプルストック10 μL、1 Mビシンバッファー(pH 8.3)8 μL、0.5 M EDTA1.28 μLを添加します。DEPC 処理した H2O を添加して総容量 160 μL にすると、最終濃度は 50 mM ビシン (pH 8.3) 中に 0.17 M CMC、7 M 尿素、4 mM EDTA です11。
注:このプロトコルは、単一の合成RNA配列またはRNA混合物のいずれかに適用できます。 - 160 μLの反応溶液をRNaseフリーの薄肉0.2 mL PCRチューブで4等分し、PCRマシンで37°Cで20分間インキュベートします。
注:チューブあたり50μLは、PCR装置で使用できる最大反応量です。 - 10 μLの1.5 M酢酸ナトリウムと0.5 mM EDTA(pH 5.6)で各反応をクエンチします。
- 4つの平行スピンカラムでカラム精製を行い、ステップ2.1.5-2.1.8で説明されている手順に従って過剰な反応物を除去します。精製した製品を、各1.5 mL RNaseフリー微量遠心チューブ内の15 μLのDEPC処理H2Oに溶解します。
- 精製した製品を4本のRNaseフリーの薄肉0.2 mL PCRチューブに移します。精製された製品15 μLごとに0.1 M Na2CO3バッファー(pH 10.4)を20 μL加え、DEPC処理されたH2Oを添加すると、各反応チューブ(合計4本のチューブ)の最終容量が40 μLになります。反応液を37°Cで2時間PCR装置でインキュベートします。
- ステップ2.1.5で説明したように、4つの平行スピンカラムによるカラム精製により、反応をクエンチおよび精製します。CMC-ψ変換した製品を、それぞれ15 μLのDEPC処理H2Oを含む1.5 mLのRNaseフリー微量遠心チューブに溶出します。
- 精製したCMC-ψ変換されたサンプルを4つのコレクションチューブから1本のチューブに結合します。ステップ 4.1 から 4.6 で説明されている手順に従ってギ酸分解を 50% (v/v) 行い、シーケンシング用の MS ラダーを生成します。
6. LC-MS測定
- LC-MS 測定用の移動相を調製します。移動相 A は、25 mM ヘキサフルオロ-2-プロパノールと 10 mM ジイソプロピルアミンを LC-MS グレードの水に溶かしたものです。移動相Bはメタノールです。
- サンプルをLC-MSサンプルバイアルに移し、分析します。各サンプル注入量は、100〜400 pmolのRNAを含む20 μLです。
- 以下のLC条件を使用してください:カラム温度35°C、流速0.3mL/分;2 〜 20% 移動相 B から 15 分間かけて線形グラジエントを行い、その後 90% 移動相 B で 2 分間の洗浄ステップを行います。
注:セクション 2 で述べた Cy3 や sulfo-Cy3 などの疎水性の末端標識では、サンプル溶出に高い割合の有機溶媒が必要になる場合があります(つまり、同様のグラジエントを使用できますが、移動相 B の割合範囲が広くなります)。例えば、移動相 B を 2 〜 38% で 30 分間、2 分間の洗浄ステップで 90% 移動相 B を 30 分間混合します。 - Agilent Q-TOF(Quadrupole Time-of-Flight)質量分析計と、オートサンプラーおよびMS HPLC(High Performance Liquid Chromatography)システムを搭載したLCシステムを組み合わせて、サンプルを分離して分析します。LCカラムは、粒子径が1.7 μmの50 mm x 2.1 mmC18カラムです。次のMS設定を使用します:マイナスイオンモード。範囲、350 m / z〜3200 m / z;スキャンレート、2スペクトル/秒;乾燥ガス流量、17 L / min;乾燥ガス温度、250°C;ネブライザー圧力、30 psig;キャピラリー電圧、3500 V;フラグメント電圧、365V。これらのパラメータは、使用する質量分析計のタイプまたはモデルに固有のものであることに注意してください。
- Agilent MassHunter 取得ソフトウェアでデータを取得します。Agilent 分子特徴抽出(MFE)ワークフローを使用して、質量、保持時間、容量(各イオン種のMFE存在量)、品質スコアなどの化合物情報を抽出します。 次のMFE設定を使用します:「重心データ形式、低分子(クロマトグラフィー)、高さ≥100のピーク、最大1000、品質スコア≥50」。
注:MFE設定を最適化して、最大1000個まで、品質スコアが50≥、できるだけ多くの潜在的な化合物を抽出します。
7. 計算アルゴリズムによるRNA配列生成の自動化
注:この手順は、 図1cのRNA #1についてのみ示されています。
- MFE抽出化合物を、体積(ピーク強度)とtRの減少順にソートします。1)ビオチン標識された質量ラダー成分のtRsがこのtRウィンドウ(4分から10分)にシフトされるため、ビオチンによって標識されたRNAフラグメントを選択するためにt Rを4分から10分に設定して選択し、2)アルゴリズム計算のためにラダーフラグメントの数よりも桁違いに高いインプット化合物を使用して、ボリュームに基づくデータ量を減らすことで、データの事前選択を行います。例えば、20 nt RNAの場合、20 nt RNAのシーケンシングには20個の標識されたmass-t Rラダー成分が必要になるため、MFEデータファイルから200個の化合物が体積に基づいて選択されます。質量分析計の種類や機種が異なると、t Rウィンドウが異なる場合がありますので、ご注意ください。
- 公開されたアルゴリズム8の改訂版を使用してRNA #1のデータ処理と配列生成を実行します。改訂されたアルゴリズムのソースコードは、前述(https://academic-oup-com.remotexs.ntu.edu.sg/nar/article/47/20/e125/5558343#supplementary-data)9に記載されている。
- アルゴリズムを使用してシーケンス生成を自動化するだけでなく、ベースコールのために2つの隣接するラダーコンポーネント間の質量差を手動で計算します。RNA内のすべての塩基は手動で呼び出すことができ、RNAヌクレオチドおよび修飾データベース8の理論上の塩基と照合できます。したがって、RNA鎖の完全な配列を手動で正確に読み取ることができ、アルゴリズムによって報告された配列の読み取り精度を確認するために使用されます。RNA修飾のさらなる構造は、RNA修飾データベース12に見出すことができ、それらに対応する理論質量はChemBioDrawによって得られる。表 S1 から S2 では、特定のラダー成分について観測された質量とその理論質量と比較すると、ppm (100 万分の 10 万) の質量差が示されており、10 ppm 未満の値は各ベースコールで良好に一致していると見なされます。
8. RNA混合物のシーケンシング
- ステップ2.2で説明したワンステッププロトコールを使用して、3'末端の5本のRNA鎖(RNA #1〜#5)の混合物をA(5')pp(5')Cp-TEG-ビオチンで標識します。総容量150 μLの反応液中に、10x T4 RNAリガーゼ反応バッファー15 μL、各RNA鎖1.5 μL(RNA #1から#5のストック100 μM、総容量7.5 μL)、150 μM A(5')pp(5')Cp-TEG-ビオチン10 μL、無水DMSO15 μL、 5 μL の T4 RNA リガーゼ (10 units/μL) と 97.5 μL の DEPC 処理 H2O. 反応溶液を 5 つのアリコートに均等に分配します。各RNaseフリー微量遠心チューブには、30 μLの反応溶液が含まれています。
- 反応液をPCR装置で16°Cで一晩インキュベートします。
- ステップ2.1.5-2.1.8で説明した手順に従って、5本の平行スピンカラムを使用してカラム精製を行います。3'-ビオチン化5 RNA鎖の混合物サンプル(RNA #1から#5の混合物)を、15 μLのDEPC処理H2Oを含む1.5 mLのRNaseフリー微量遠心チューブに溶出します。
- 5つの収集チューブから精製された混合物サンプルを1つのチューブに結合します。セクション4に記載されている手順に従ってギ酸分解を行います。
- セクション 6 で説明したように LC-MS でサンプルを測定し、ステップ 6.5 で説明したように、最適化された MFE 設定のデータ解析ソフトウェアを使用してデータを分析して、質量、tR、および体積を含むデータを抽出します。一般的な処理アルゴリズムとベースコールアルゴリズムは、混合物によってデータの複雑さが大幅に増加するため、適用されません。混合サンプルのRNA中のすべての塩基は、セクション7.3と同様の方法で手動で呼び出され、RNAヌクレオチドおよび修飾データベース8の理論上の塩基とよく一致するため、混合サンプル中の5本すべてのRNA鎖の完全な配列が正確に読み取られます。 表S7〜S11には、観測質量、t R、体積、品質スコア、ppm質量差など、すべての情報がリストされています。
結果
RNAの3'末端にビオチンタグを導入し、容易に識別できるmass-tRラダーを作製します。2D-HELS MS Seqアプローチのワークフローを図1aに示します。RNAの3'末端に導入された疎水性ビオチン標識(セクション2を参照)は、標識されていないラダー成分と比較して、3'標識ラダー成分の質量とtRsを増加させます。したがって、2D mass-tR プロットでは、3' ラダー曲線は y 軸の値が大きい値にシフトされ (tRs の増加による)、x 軸の値が大きい値にシフトします (質量の増加による)。図 1b は、2D-HELS MS Seq の 3'-末端へのビオチンタグの導入を含むサンプル調製プロトコルを示しています。図 1c は、RNA #1 の 3'-ビオチン標識 mass-tR ラダーフラグメントの tRs の系統的変化に基づく 2D mass-tR プロット上の 5'-ラダーおよびその他の望ましくないフラグメントからの 3'-ラダーの分離を示しています。3'-ラダー曲線だけでRNA #1の完全な配列が得られ、t Rシフトを示さない5'-ラダー曲線は逆の配列を提供するが、ターミナルベース8を読み取るためにはエンドペアリングが必要である。この2D-HELSの戦略では、以前に報告されたような末端対形成は必要なく、1つの標識ラダーカーブ8からのみRNA配列全体を完全に読み取ることができる。そのため、複数のRNAを含む混合サンプル、例えば、長さの異なる2本のRNA鎖(RNA #1とRNA #2、それぞれ19 ntと20 nt)を、各RNAに5'-ビオチン標識でシーケンシングすることができます(図1d)。
ψをCMC-ψ付加体に変換し、2D-HELS MS Seq.ψは、ウリジン(U)と同じ質量を持つため、MSベースのシーケンシングでは難しいヌクレオチド修飾です。これら2つの塩基を互いに区別するために、RNAをCMCで処理し、ψをCMC-ψ付加物に変換します(セクション5を参照)。付加物の質量はU型とは異なり、2D-HELS MS Seq.で区別できます。図2aは、ψをRNA #6のCMC付加物に変換する反応の粗生成物のHPLCプロファイルを示しています。それらの UV ピークを積分することにより、コンバージョン率を計算し、セクション 5 で示したプロセス後に 42% の ψ が CMC-ψ付加物に変換されます。酸分解およびLC-MS測定の後、アルゴリズム処理データ8,9から同定された非CMC変換ラダーおよびCMC変換ラダーの両方に基づいて、手動で配列を取得した。赤い曲線は、ψがCMC-ψ付加体に部分的に変換されるため、RNA #6(図2b)の8位のψから始まる灰色の曲線から分岐します。CMCの質量と疎水性のため、この変換により、各CMC-ψ付加物含有ラダー成分の質量は252.2076ダルトン増加し、非変換のラダーコンポーネントと比較すると、tRが大幅に増加します。したがって、RNA #6 の位置 8 から始まる劇的なシフトは、2D mass-tR プロットで観察でき、位置 8 が実際に RNA #6 のψであることを示しています。
RNA混合物のシーケンシング。5つの異なるRNA鎖の混合物を、3'末端標識による2D-HELS MS Seqアプローチによりシーケンシングします(セクション8を参照)。混合RNAのシーケンシングに関する懸念は、2D mass-tRプロットの複数のラダー曲線が同じ開始点(2D mass-tRプロットの疎水性タグ)を共有する場合に、互いに重なる可能性があることです。ただし、ベースコールは 1 つずつ行われ、それぞれが MFE データ内の 2 つの隣接するラダーフラグメント間の質量差に基づいて行われます。各質量差がデータプール8,9内の標準ヌクレオチドまたは修飾ヌクレオチドの理論質量の1つとよく一致する(PPM MS差<10)限り、正しいベースコールを行うことができます。マルチプレックス化されたRNAサンプルの分析では、主に混合物に起因するデータの複雑さが大幅に増加するため、図1および図2で使用されている一般的な処理およびベースコールアルゴリズムは使用されません。これらの配列は、2つの隣接する質量ラダーフラグメント間の質量差を計算し、それをデータプール9内のヌクレオチドの理論質量と比較することにより、手動でベースコールされる。質量 PPM <10 の一致したベースは、この位置のベース ID として選択されます。このベースコールのベースごとの手動計算により、混合物内のすべてのシーケンスが正確にシーケンスされます。OriginLabソフトウェアを使用して、2D mass-tRプロットを再構築し、各配列の開始tRを体系的に正規化して、5つの異なるRNA配列をよりよく視覚化します(図3)。このような正規化を行わないと、5つのRNAすべての配列の文字コード(A、C、G、U)がプロット上に密集し(図S1)、図3で報告したものに比べて視覚化のしやすさが低くなります。シーケンシングの結果は、2D-HELS MS Seqアプローチが精製された一本鎖RNAのシーケンシングに限定されるだけでなく、さらに重要なことに、複数のRNA鎖を持つRNA混合物にも限定されることを示しています。現在、ベースコールとシーケンス生成のプロセスを自動化するためのアルゴリズムが開発されています。
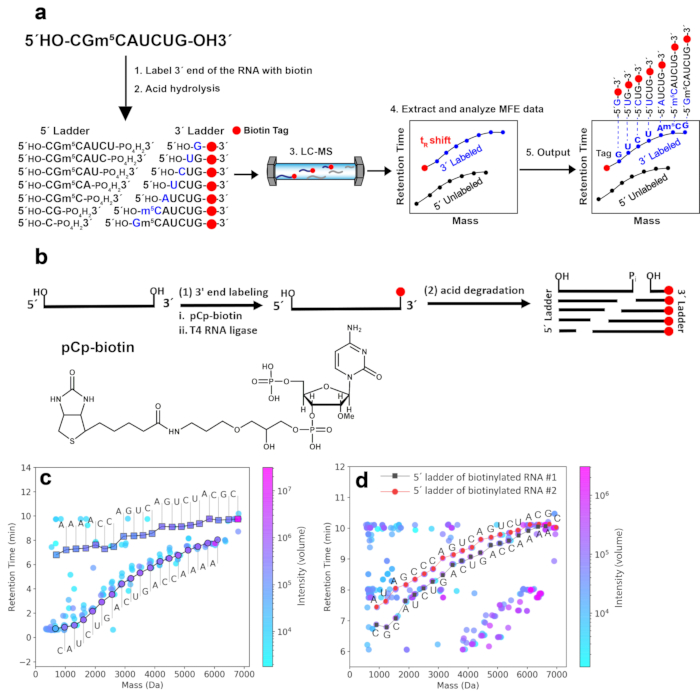
図1. 代表的なRNAサンプルの2D-HELS MS Seq。 (a) 2D-HELS MS Seq.主なステップには、1)配列決定するRNAの疎水性タグ標識、2)酸加水分解、3)LC-MS測定、4)MFEデータの抽出と分析、5)アルゴリズムまたは手動計算による配列生成が含まれます。(b) 2D-HELS MS Seq. (c) 3'-biotin-labeledmass-t R ladder fragments of RNA #1 (19 nt) の tRs の系統的変化に基づく 2D 質量保持時間 (tR) プロットにおける 5'-ラダーからの 3'-ラダーおよびその他の望ましくないフラグメントの分離を含むサンプル調製プロトコル。配列はde novoであり、ベースコールアルゴリズム9によって直接自動的に読み出されます。(d)5'-ビオチン標識RNA #1およびRNA #2の同時シーケンシング、それぞれ19 ntおよび20 nt。ビオチンタグをRNAの5'末端に導入する方法は、3'-ビオチン化の方法とは異なり、以前に公開されたプロトコル9で見つけることができます。2つのRNA(RNA #1およびRNA #2)の5'末端はビオチン化されており、それらの5'-ビオチン化ラダーは容易に同定できます。LC-MS後の2D mass-t Rプロットでは、ビオチン化ラダー成分はビオチンの疎水性によりtRシフトが大きいのに対し、非標識ラダー成分は低tR領域にあるため、LC-MS後の2D mass-tRプロットでは、両方の5'-ビオチン化ラダーを非標識3'-ラダーから容易に分離できます。5'-ラダーと3'-ラダーは共存しますが、2つの混合RNA鎖の配列解釈を妨げません。これら2つのRNAの各配列は、計算アルゴリズムで処理されたデータ8,9に基づいて、5'-ビオチン化ラダーから手動で取得される。この図はZhang et al.9から修正されています。この図の拡大版を表示するには、ここをクリックしてください。

図 2.ψ 2D-HELS MS Seq. (a)1つのψを含む20 nt RNA(RNA #6)のCMC付加体に変換する反応ψ粗生成物のHPLCプロファイル(b)ψ含有RNA#6のシーケンシング。ψをCMC-ψ付加体(ψ*)に変換すると、CMCの質量と疎水性により、質量は252.2076ダルトン増加し、tR は大幅に増加します。したがって、mass-tR プロットでは8の位置から始まる劇的なシフトが観察でき、これがRNA配列の8の位置でのψであることを示しています。配列は、計算アルゴリズムで処理されたデータ8、9に基づいて手動で取得される。この図はZhang et al.9から修正されています。 この図の拡大版を表示するには、ここをクリックしてください。

図 3.5つの異なるRNAを含むRNA混合物のシーケンシング。ビオチンは、2D-HELS MS Seqの前に各RNAの3'末端を標識するために使用されます。各シーケンスについて、開始t R 値は、視覚化を容易にするために 7 分間隔で開始するように体系的に正規化されます。開始 tR 値と後続のt Rs の絶対差は、5 つの RNA のそれぞれで変わらないため、同じプロットでそれぞれを視覚化しやすくなります。すべての塩基は、隣接する2つのラダー成分の質量差を手動で計算し、それらをRNAヌクレオチドおよび修飾データベース8の理論質量差と一致させることによって同定されます。図3のプロットは、手動のベースコールとシーケンシングデータに基づいてOriginLabを使用して再構築されます(代表的な結果のRNA混合物のシーケンシングのセクションを参照)。tRの標準化を行わない5つの混合RNAの2Dmass-t R図を図S1に示します。この図の拡大版を表示するには、ここをクリックしてください。
ディスカッション
タンデムベースのMSフラグメンテーションとは異なり、高度に制御された酸性加水分解は、質量分析計9,10での分析前にRNAをフラグメンテーションするために、2D-HELS MS Seqアプローチで使用されます。その結果、酸分解された各フラグメントを装置で検出でき、シーケンシングラダーと同等のものを形成します。最適な条件下では、この方法は、ホスホジエステル結合8,9,10でのみ、平均して分子あたり1つの部位特異的RNA切断を介して、RNAから「理想的な」配列ラダーを作成する。分解された各フラグメントを質量分析計で 1 回の分析で測定した後、隣接する 2 つのラダーフラグメント間の質量差は、その位置での RNA ヌクレオチドまたは修飾の正確な質量に対応します。各RNA修飾は、RNA内での同定と位置特定に役立つ固有の固有の質量を持つか、または固有の質量を持つものに変換することができます。したがって、理論的には、この方法では、任意のRNAのde novoおよび直接シーケンシングのために、標準ヌクレオチドと修飾ヌクレオチドの両方の同一性と位置を報告できます。しかし、異なる配列ラダーが互いに重なり合う場合があり、MSデータ解析が複雑になり、MSによるRNAシーケンシングが実際に困難になります。
3'-疎水性タグの利点の1つは、あらゆるフラグメンテーション法における大きな課題、 すなわち、すべてのRNA分子を2つのフラグメントに切断する必要がある(理想的にはそれ以上ではない)こと、つまり、1つは元の5'末端を含み、もう1つはRNAの元の3'末端を含むフラグメントであるということです。したがって、各切断イベントでは 2 つのフラグメントが生成され、2 つのはしごが生成されます (1 つは 5' 末端から測定され、もう 1 つは 3' 末端から測定されます)。どのMSピークがどのラダーに属しているかを判断するには、常に曖昧さがあります。これは、いくつかの異なるRNAが混在している場合、重複する配列ラダーが多数生成されるため、より問題になります。しかし、3'末端からのすべてのラダーフラグメントは疎水性タグで標識されているため、はるかに長いtRsを示します(図1a)。その結果、3'標識RNAのみからのみ得られた2D質量-tR データにおいて、明確で明確なラダーを得ることができます。特に、我々は、異なる化学的結合法9を用いて、任意のRNAの5'末端または3'末端のいずれかを選択的に標識するアプローチを最適化している。また、ここでは端子ベースの決定には使用されませんが、5'方向と3'方向の両方から読み取ったときに同一のシーケンス情報を2回提供するために使用され(つまり、双方向シーケンシング検証)、シーケンシングの精度をさらに向上させるために、双方向シーケンシングを実行することもできます。
未知のRNAサンプルの de novo シーケンシング、特に複雑な生物学的サンプルの場合、シーケンス生成のための大量のLC-MSデータを正確かつ効率的な方法で処理するための一般的で堅牢なアルゴリズムが必要であり、これは最近、他の発表された研究10を通じて利用可能になりました。これらのアルゴリズムは、より複雑なサンプル10のシーケンシングに使用されてきましたが、この研究では、特に指示がない限り、シーケンス生成のために手動ベースコールを実行しました。私たちは、2D-HELS MS Seqのすべての主要なステップをカバーすることを目指しており、追加のシーケンシングアルゴリズムを使用しなくても、シーケンシングするRNAの配列を手動で読み出すことができるプロセスを説明したいと思います。可視化を容易にし、2D mass-tR プロットのシーケンシングに必要なラダーフラグメントをより迅速に同定するために、各LC-MSランのMFEファイルは、特に明記されていない限り、公開されたアルゴリズム8 の改訂版で処理されてから、その配列を読み取ることができます。公開されたアルゴリズムは、LC-MSデータから配列を読み取るために直接使用することはできませんが、その機能の一部はデータの処理に引き続き使用できます—このアルゴリズムを通じて質量付加物を階層的にクラスタリングすると、各ラダー成分の強度が増大し、特にシーケンスリードが生成される重要な領域でデータの複雑さが軽減されます8。9.
2D-HELS MS Seqのサンプル調製における重要なステップの1つは、RNA疎水性タグの末端標識効率の改善です。高い標識効率は、配列データが依存するMSシグナルの生成に必要なRNAサンプルの量を減らすのに役立ちます。標識効率を高めるために、RNAの3'末端を標識する際にアデニル化ステップを回避するために活性化AppCp-ビオチンを使用するなど、新しい標識戦略を採用しています。19 nt RNAの3'末端をビオチンで標識する反応(ステップ2.2を参照)の収率は、このワンステップ法を使用して60%から~95%9 に改善できます。効率的な標識により、前述の9と同様に、最大12の異なるRNAを含む混合サンプルのシーケンシングを行うことができます9。この研究では、5つのRNAの混合物を代表例として使用して、シーケンシングプロセスを説明します。また、正確なシーケンシングに必要なすべてのラダーフラグメントを検出し、混合物中の5つのRNA配列のそれぞれの完全な配列を読み取ります。ラベリング効率の向上は、サンプルのロード量を最小限に抑えるだけでなく、シーケンス生成のためのダウンストリームデータ解析中のデータの複雑さを大幅に軽減するのにも役立ちます。現在、5'末端と3'末端の両方でRNAを標識する際に定量的な収率を達成するための新しい反応が開発されています。
図1cに示すようにRNA #1のシーケンシングを行う場合、ストレプトアビジンの捕捉および放出ステップを使用して、酸分解の前にビオチン化RNA #1を物理的に分離します(セクション3を参照)。これにより、非標識RNAのごく一部が除去され、その結果、2D mass-tRプロットで標識された質量ラダーを視覚的に同定しやすくなります。ただし、ビオチン化RNAラダーフラグメントは、非標識の対応物と比較して、ビオチンタグからの疎水性によりtRsが遅延/長くなるため、物理的分離ステップは必須ではありません。さらに、ベースコールは物理的な分離に依存しないが、隣接する質量ラダー成分の質量差に依存するため、2つの隣接するラダー成分の質量差が特定のヌクレオチドまたはRNAヌクレオチドおよび修飾日付ベース8の対応する質量とよく一致する限り、正しいベースコールを達成できる。現在、ベースコールとシーケンス生成を自動化するための計算アルゴリズムが開発中です。
元の LC-MS データ(ファイルタイプ .d)をスプレッドシートファイルにエクスポートする際の MFE 設定は、データ処理とその後のシーケンス生成(セクション 6.5 を参照)にとって非常に重要です。たとえば、MFE 設定の「高さのあるピーク」を 100 から 1000 の範囲でテストしたところ、100 に設定すると、1000 に設定すると 1000 の場合よりも 2 倍多くの化合物が得られることがわかりました。ラダーコンポーネントの欠落を避けるために、シーケンシングワークフロー中にMFE設定を調整できます。この設定は、装置の質量分解能、質量ラダーフラグメントの量、およびデータの複雑さに依存すると考えられます。さらに、低分子の重心データセットとクロマトグラフィータイプの設定を使用することが重要です。品質スコアは、データ品質に基づいて 50% から 100% まで変更できます。
この研究で使用している LC-MS 装置の質量分解能は ~40K を超えており、この分析法は 35 塩基未満の RNA のシーケンシングのみに制限されています。ただし、このメソッドの正確な読み取り長は機器によって異なります。より高い分解能を持つより高度な機器は、読み取り長が長くなる可能性があります。同様に、1回のLC-MSランで同時にシーケンシングできるRNA配列の数であるスループットについては、まだ調査されていませんが、アルゴリズムを使用せずに最大12の異なるRNA鎖のRNAサンプルの混合物を手動でシーケンシングしました9。 現在のワークフローでは、LC-MS の各分析に ~100 pmol の短 RNA(<35 nt)が必要です。追加の実験が必要になると、ローディング量が増加します:異性体のヌクレオチド修飾を区別するためには、通常、最大400 pmolのRNAが必要です。tRNAPheのような特異的なtRNAのシーケンシングには、シーケンシングと修飾解析に~1000 pmolのサンプルが必要になる場合があります。ただし、LC-MS装置では、感度が高いほど必要なサンプル負荷量が減少することが予想されます。サンプルの標識効率、シーケンシングアルゴリズム、装置の感度と分解能の向上により、私たちの分析法は、より幅広いRNAサンプル、特にさまざまなRNA修飾を持つサンプルに適用できると期待しています。
開示事項
著者らは、この原稿で議論されている技術に関連する仮特許を出願しました。
謝辞
著者らは、この研究を支援した国立衛生研究所(National Institutes of Health)(1R21HG009576)からS.Z.およびW.L.へのR21助成金、およびニューヨーク工科大学(NYIT)のS.Z.への研究と創造性のための機関支援助成金に感謝します。著者らは、図作成を支援してくださった博士課程の学生Xuanting Wang氏(コロンビア大学)に感謝するとともに、Michael Hadjiargyrou教授(NYIT)、Jingyue Ju教授(コロンビア大学)、James Russo博士、Shiv Kumar博士、Xiaoxu Li博士、Steffen Jockusch博士、Ju研究室の他のメンバー(コロンビア大学)、Yongdong Wang博士(Cerno Bioscience)、Meina Aziz氏(NYIT)、Wenhao Ni氏(NYIT)に感謝します。
資料
| Name | Company | Catalog Number | Comments |
| 5' DNA Adenylation kit | New England Biolabs | E2610S | 50uM concentration |
| 6550 Q-TOF mass spectrometer | Agilent Technologies | 5991-2116EN | Coupled to a 1290 Infinity LC system |
| A(5´)pp(5´)Cp-TEG-biotin-3´ | ChemGenes | 91718 | HPLC purified |
| ATPγS | Sigma-Aldrich | 11162306001 | Lithium salt |
| Bicine | Sigma-Aldrich | B8660 | BioXtra, ≥99% (titration) |
| Biotin maleimide | Vector Laboratories | SP-1501 | Long arm |
| C18 column | Waters | 186003532 | 50 mm × 2.1 mm Xbridge C18 column with a particle size of 1.7 μm |
| Centrifugal Vacuum Concentrator | Labconco | Refrig 115v/60hz 7310022 | Labconco CentriVap |
| ChemBioDraw | PerkinElmer | ChemDraw Prime | Generate a chemical structure and property data of structures & fragments |
| CMC (N-cyclohexyl-N?-(2-morpholinoethyl)-carbodiimide metho-p-toluenesulfonate) | Sigma-Aldrich | 2491-17-0 | 95% Purifiy |
| Cyanine3 maleimide (Cy3) | Lumiprobe | 11080 | Water insoluble |
| DEPC-treated water | Thermo Fisher Scientific | AM9906 | Autoclaved, certified nuclease-free |
| Diisopropylamine (DIPA) | Thermo Fisher Scientific | 108-18-9 | 99% Alfa Aesar |
| DMSO | Sigma-Aldrich | 276855 | Anhydrous dimethyl sulfoxide, 99.9% |
| EDTA | Sigma-Aldrich | E6758 | Anhydrous, crystalline, BioReagent, suitable for cell culture |
| Formic acid | Merck | 64-18-6 | 98-100%, ACS reag, Ph Eur |
| Hexafluoro-2-propanol (HFIP) | Thermo Fisher Scientific | 920-66-1 | 99% Acros Organics |
| LC-MS sample vials | Thermo Fisher Scientific | C4000-11 | Plastic screw thread vials |
| LC-MS vial caps | Thermo Fisher Scientific | C5000-54A | Autosampler vial screw thread caps |
| Na2CO3 buffer | Sigma-Aldrich | 88975 | BioUltra, >0.1 M Na2CO3, >0.2 M NaHCO3 |
| Oligo Clean & Concentrator | Zymo Research | D4060 | Spin column |
| OriginLab | OriginLab | OriginPro | Data analysis and graphing software |
| pCp-biotin | TriLink BioTechnologies | NU-1706-BIO | 20 ul (1 mM) |
| RNA #1--#6 | Integrated DNA Technologies | Custom RNA oligos | 19nt-21nt single-stranded RNAs, used without further purification |
| Rocking platform shaker | VWR | Orbital Shaker Standard 1000 | Speed Range 40 to 300 rpm |
| Streptavidin magnetic beads | Thermo Fisher Scientific | 88816 | Binding approx. 55ug biotinylated rabbit lgG per mg of beads |
| Sulfonated Cyanine3 maleimide | Lumiprobe | 11380 | Water soluble |
| T4 DNA ligase 1 | New England Biolabs | M0202S | 400 units/uL |
| T4 polynucleotide kinase | Sigma-Aldrich | T4PNK-RO | From phage T4 am N81 pse T1 infected Escherichia coli BB |
| Tris-HCl buffer | Sigma-Aldrich | T6455 | Tris-HCl Buffer, pH 10, 10×, Antigen Retriever |
| Urea | Sigma-Aldrich | 81871 | Urea for synthesis. CAS No. 57-13-6, EC Number 200-315-5. |
参考文献
- Addepalli, B., Venus, S., Thakur, P., Limbach, P. A. Novel ribonuclease activity of cusativin from Cucumis sativus for mapping nucleoside modifications in RNA. Analytical and Bioanalytical Chemistry. 409 (24), 5645-5654 (2017).
- Gao, H., Liu, Y., Rumley, M., Yuan, H., Mao, B. Sequence confirmation of chemically modified RNAs using exonuclease digestion and matrix-assisted laser desorption/ionization time-of-flight mass spectrometry. Rapid Communications in Mass Spectrometry. 23 (21), 3423-3430 (2009).
- McLuckey, S. A., Van Berkel, G. J., Glish, G. L. Tandem mass spectrometry of small, multiply charged oligonucleotides. Journal of The American Society for Mass Spectrometry. 3 (1), 60-70 (1992).
- Fountain, K. J., Gilar, M., Gebler, J. C. Analysis of native and chemically modified oligonucleotides by tandem ion-pair reversed-phase high-performance liquid chromatography/electrospray ionization mass spectrometry. Rapid Communications in Mass Spectrometry. 17 (7), 646-653 (2003).
- Taucher, M., Breuker, K. Characterization of modified RNA by top-down mass spectrometry. Angewandte Chemie International Edition in English. 51 (45), 11289-11292 (2012).
- Kellner, S., Burhenne, J., Helm, M. Detection of RNA modifications. RNA Biology. 7 (2), 237-247 (2010).
- Thomas, B., Akoulitchev, A. V. Mass spectrometry of RNA. Trends in Biochemical Sciences. 31 (3), 173-181 (2006).
- Bjorkbom, A., et al. Bidirectional direct sequencing of noncanonical RNA by two-dimensional analysis of mass chromatograms. Journal of the American Chemical Society. 137 (45), 14430-14438 (2015).
- Zhang, N., et al. A general LC-MS-based RNA sequencing method for direct analysis of multiple-base modifications in RNA mixtures. Nucleic Acids Research. 47 (20), 125 (2019).
- Zhang, N., et al. Direct sequencing of tRNA by 2D-HELS-AA MS Seq reveals its different isoforms and dynamic base modifications. ACS Chemical Biology. 15 (6), 1464-1472 (2020).
- Bakin, A., Ofengand, J. Four newly located pseudouridylate residues in Escherichia coli 23S ribosomal RNA are all at the peptidyltransferase center: analysis by the application of a new sequencing technique. Biochemistry. 32 (37), 9754-9762 (1993).
- Cantara, W. A., et al. The RNA Modification Database, RNAMDB: 2011 update. Nucleic Acids Research. 39 (Database issue), D195-D201 (2011).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved