Method Article
Kanser hücresel Phenotypes üzerinde Mikroçevresel etki Interrogate için Microdiziler kullanma
Bu Makalede
Özet
Burada sunulan yöntemin amacı, mikroortam mikrodiziler (Mema) nasıl imal edilebilir ve kültürlü hücrelerin fenotürü üzerinde basit Kombinatoryal mikroortamlar binlerce etkisini sorgulamak için kullanılan göstermek için.
Özet
Mikroortamın hücrelerin phenoype üzerindeki etkisini anlamak, hem çözünür büyüme faktörleri hem de mikro ortamda matris ilişkili proteinlerin kompleks karışımı nedeniyle zor bir sorundur. Ayrıca, mikroortamlar içinde vitro modelleme için kolayca kullanılabilir reaktifler genellikle tamamen tanımlanmış ve toplu değişkenlik için toplu muzdarip proteinlerin karmaşık karışımları kullanın. Mikro Mikroarray (Mema) platformu tek bir tahlil içinde hücresel fenotipleri üzerindeki etkileri için mikro çevre proteinleri binlerce basit kombinasyonları değerlendirilmesi için izin verir. Memalar, bireysel liglerin dizilmiş ekstrenüler matris (ECM) proteinlerini içeren ayrı kuyulara eklenmesini sağlayan iyi plakalar halinde hazırlanmaktadır. Her basılı ECM ile çözünür ligand kombinasyonu benzersiz bir kombinasyon oluşturur. Tipik bir Mema tahlil hücre tek bir tahlil maruz kalan 2.500 benzersiz Kombinatoryal mikroortamlar büyük içerir. Bir test durumu olarak, meme kanseri hücre hattı MCF7 MEMA platformunda kaplama oldu. Bu tahlil Analizi hem geliştirmek ve bu hücrelerin büyümesi ve proliferasyonu inhibe faktörler tespit. MEMA platformu son derece esnektir ve kanser araştırmalarının ötesinde diğer biyolojik sorularla kullanılmak üzere uzatılabilir.
Giriş
İki boyutlu (2D) monolayers plastik üzerinde kanser hücre hatları kültür kanser araştırmacıları için büyük workhorses vardır biri kalır. Ancak, mikroortam giderek hücresel fenotürleri etkisi yeteneği için tanınıyor. Kanserde, tümör mikroçevre, büyüme, hayatta kalma, invazyon ve tedaviye yanıt dahil olmak üzere birden fazla hücresel davranışları etkileyen bilinmektedir1,2. Geleneksel Tek tabakalı hücre kültürler genellikle mikroçevre etkileri eksikliği, hangi daha karmaşık üç boyutlu gelişimine yol açmıştır (3D) hücreleri büyümek için tespit, ticari olarak kullanılabilir arıtılmış Bodrum membran özler de dahil olmak üzere. Ancak, bu saflaştırılmış matrisler genellikle kullanmak ve toplu değişkenlik3 ve karmaşık kompozisyonlar3gibi teknik sorunlardan muzdarip karmaşıklaşır. Sonuç olarak, hücresel fenotürleri etkileyen olabilir belirli proteinler için fonksiyon atamak zor olabilir3.
Bu sınırlamaları gidermek için, mikroortam Mikroarray (Mema) teknolojisini geliştirdik, bu da mikro 'ı basit bileşimsiz matris (ECM) ve çözünebilir büyüme faktörü proteinlerinin kolay kombinasyonuna düşüren4,5 . MEMA platformu, hücrelerin davranışını etkileyen baskın mikroçevresel faktörlerin tanımlanması sağlar. Bir dizi biçimi kullanarak, mikro ortam faktörlerinin kombinasyonları binlerce tek bir denemede farklı teknik kullanılarak olabilir. MEMA burada açıklanan interrogates ~ 2.500 farklı benzersiz mikroçevre koşulları. İyi plakalara yazdırılan ECM proteinleri, hücrelerin kültürlü olması üzerine büyüme pedleri oluşturur. Çözünür ligler bireysel kuyulara eklenir, hücrelerin maruz kaldığı her farklı noktada benzersiz Kombinatoryal mikroortamlar (ECM + ligand) oluşturma. Hücreler birkaç gün boyunca kültürlü, sonra sabit, lekeli, ve bu özel mikroçevre kombinasyonları maruz kalma sonucu hücresel fenotipleri değerlendirmek için görüntülenmiştir. Mikroortamlar basit kombinasyonları olduğundan, hücrelerde büyük fenotipi değişiklikler yapan proteinlerin tanımlanması basittir. Memalar, hücre kaderi kararları ve terapi4,5,6,7tepki sürücü olanlar da dahil olmak üzere, birden fazla hücresel fenotürleri etkileyen faktörleri belirlemek için başarıyla kullanılmıştır. Bu tepkiler basit 2D deneylerde doğrulanabilir ve daha sonra tümör mikroortamının karmaşıklığını daha tam olarak tekrar eden koşullar altında değerlendirilebilir. MEMA platformu, iyi fenotip biyomarkerlerin mevcut olması koşuluyla, çeşitli hücre türleri ve uç noktalarıyla oldukça uyarlanabilir.
Protokol
Not: Tahmini saat de dahil olmak üzere tüm MEMA işleminin genel bakışı Şekil 1' de gösterilen akış diyagramında özetlenmiştir. Bu protokol 8-kuyu plakaları içinde MEMAs imalatı ayrıntıları. Protokol diğer plakalar veya slaytlar için uyarlanabilir.
1. protein, Seyreltilme ve boyama tamponları hazırlanması
- ECMs, ligler ve sitokinlerin oda sıcaklığına (RT) ve kısaca santrifüjlerin dengelenme şişeleri. Uygun RT arabelleğinin uygun hacmini ürün veri sayfasında belirtildiği şekilde ekleyin. Stok konsantrasyonları için üreticinin tavsiyesini takip edin.
Not: Tablo 1 ve Tablo 2' de hisse senetleri ve nihai konsantrasyonları Ile ligler ve ECMS tam listesi sağlanmıştır. Her iki ligler ve ECMs genellikle standart 2 günlük kültür assays bir biyolojik etkisi ortaya çıkarır üretici tarafından önerilen aralığın en yüksek konsantrasyonda kullanılır. Kontaminasyonu önlemek için laminar akımı altında yavaşça ve biyogüvenlik dolapları proteinleri ele. - 1 h için RT 'de hafif sallanan şişeleri kulkaylar. Bu onları denature neden olabilir gibi protein Vortex etmeyin.
- Uzun süreli depolama için aliquot proteinleri böylece tüm plakaya sadece tekrarlanan dondurma/çözme döngüleri ile bozulma önlemek için tek kullanımlık. Likofilize proteinlerin-80 °C ' de (aksi belirtilmediği sürece) gerekli olana kadar saklayın. : (İ) protein adı, (ii) hazırlanan Tarih, (iii) lot/toplu iş numarası, (iv) Tedarikçi, (v) katalog numarası, (vi) konsantrasyon, (vii) hacim ve (VIII) preparer gibi gelecekteki referanslar için tüm meta verileri toplamak için özen yapın.
- % 20 (v/v) gliserol, 10 mM EDTA, 200 mM Tris-HCl, pH 7,2 ve filtre sterilizasyonu içeren seyreltilici tampon hazırlayın. Bu tamponu steril tutun ve RT 'de saklayın.
- % 2 (w/v) BSA, 1 mM MgCl2ve 0,02% Nan3 ' ü fosfat-TAMPONLU tuz (PBS) içeren boyama tamponunu hazırlayın. 4 °C ' de filtreleyin ve saklayın.
2. ECM kaynak plakasının hazırlanması
- ECM proteinleri, yazılı ve buzda çözülecek olan seyreltilmemeli stokları çıkarın. Meta veri izleme için tüm lot numaralarını kaydedin.
- Çözülür proteinleri yavaşça doğru Resuspension sağlamak ve bir santrifüjte aşağı döndürmek için fiske tüpler.
-
ECM baskı karışımları (EPMs) ve randomize 384-Well kaynak plakaları yaratacak bir sıvı taşıma robotu tarafından kullanılacak bir floresan fiducial olun.
Not: 384-Well kaynak plakaları, 8 kuyu plakalarına yazdırılan dizileri oluşturmak için bir dokunma pin dizisi yazıcısı tarafından kullanılacaktır.- Etiket 1,5 her EPM ve fiducial için mL mikrosantrifüjler tüpleri.
- Her bir EPM 'ye, 125 μL seyreltilme tamponunu birleştirerek (bkz. Adım 1,4) ECM stokunun uygun hacmine göre hazırlayın ve karışımı PBS ile toplam 250 μL hacmine getirin. Her EPM tüpünün son konsantrasyonları 1x ECM protein, 5 mM EDTA,% 10 gliserol ve 100 mM Tris olacaktır.
- Üretici tarafından belirtilen uygun tamponda çözülerek floresan fiducial hazırlayın ve 250 μL 'yi etiketli bir fiducial tüpe aktarın.
3. sıvı Işleyici kullanarak kaynak plakasının oluşturulması
- ECMs konumlarını randomizes ve kullanılan dizi yazıcı pin kafası için optimize edilmiş bir 384-kuyu plaka düzeni tasarlayın. Dizi oryantasyonunda yardımcı olmak için her iyi satır 1, sütun 1 pozisyon yazdırılır böylece fiducial yerleşimi tasarlayın.
Not: Her ECM 'nin toplam 14 − 15 çoğaltır güçlü veri sağlamak için kullanılır. Bağlayıcı tekdüzelik değerlendirmesi için sağlam bir ek veren kollajen veya başka bir ECM ek çoğaltır dahil. Düzen ilgi ECMs sayısına bağlı olarak birden fazla 384-kuyu plakaları kullanmak gerekebilir. - EPM tüpleri soğutulmuş bir tüp raf ile veya soğuk bir odada bulunan bir sıvı taşıma robotu kullanarak, 4 °C ' de tüpler tutarak, bir sıvı işleyici aktarın.
- Sıvı iþleyicinin yazılımını kullanarak, her EPM 'nin 15 μL 'i ve 384-Well kaynak plaka (lar) içinde önceden belirlenmiş kuyulara fiducial aktarmak için bir program çalıştırın.
- Pipet, baskı işlemi sırasında nem ve kurutmaya karşı koruma artırmak için kullanılmayan kuyuların içine PBS.
Not: 4 x 7 pin kafası için optimize edilmiş ve ı bloğu ve PBS kollajen içeren bir 384-iyi kaynak plaka seti örneği için Şekil 2 bakın. - Plakayı (s) mühürleyin ve yazdırmaya hazır olana kadar 4 °C ' de tutun.
4. bir dizi baskı robotu kullanarak MEMAs yazdırma
Not: Protokolün aşağıdaki bölümü, MCF7 hücrelerinin büyümesi ve proliferasyonu üzerinde farklı mikroçevre proteinlerinin etkisini araştırmak için MEMA 'nın hazırlanması ve kullanımını özellikle açıklar. Ancak, protokol kolayca farklı ligler, ECMs ve hücre diğer hücre hatları ve ilgi uç noktalarını incelemek için kullanılmak üzere adapte edilebilir.
-
Bir dokunmatik PIN yazıcısı kullanarak, EPMs ve fiducial lekeler 8 kuyu plakaları içine yazdırın. Tekrarlanabilirlik sağlamak için her ECM koşulunun birden çok çoğaltır yazdırın.
Not: diğer plaka formatları veya slaytlar yazdırmak için kullanılabilir, ancak optimum spot oluşumu elde etmek için tampon optimizasyonu gerekebilir.- 4 x 7 baskı kafası konfigürasyonda düzenlenmiş 350 μm çap pimleri kullanarak MEMA için ECMs 'i yazdırın. 8-Well plakalardaki dizileri, toplam ~ 700 Spotlar için 35 satır olarak 20 sütun olarak yazdırın. Bu plaklarda daha büyük diziler mümkündür ancak hem hücre bağlaması hem de boyama sırasında artan kenar efektlerinin takas edilmesi ile birlikte gelir.
- Yazdırdıktan sonra, kullanım öncesinde en az 3 gün boyunca bir kurutucu içinde depo plakaları.
5. ligand tedavi plakalarının oluşturulması
- İlgi liglerini de içeren bir 96-Well plaka düzeni Tasarla. Aynı anda birçok MEMA plakasının tedavisinde kolaylaştırmak için, 8-Well MEMAs ve 96-Well plaka kuyuları arasında sıvı aktarmak için 4 aralıklı ipuçları ile çok kanallı pipet kullanımı için izin aralığı ile bu plaka tasarımı.
Not: Bu protokolde, Tablo 2 ' de listelenen ligin tam kümesi kullanılır. - Buz üzerinde ligin çözülmesi. Kısaca Flick ve her tüp aşağı spin.
- Üreticinin önerilen arabelleğini (genellikle PBS) kullanarak 200x çalışma stokunda seyreltmeli ligeler.
- Her 200x ligand stoktaki pipet 10 μL, 96-kuyu plakasının içinde karşılık gelen kuyu içine girer.
- -20 °C ' de mühür ve saklama plakaları.
Not: Akış Analizi için tüm meta verileri yakalama, gruplar halinde ligand tedavi plakaları olun.
6. MEMAs üzerindeki hücreler kültür
- Çift distile su (ddH2O) içinde 1% kirlenme engelleme maddesi (malzeme tablosu) içeren, kirlenme yapmayan engelleme tamponunun başına 2 ml Ile 20 dakika boyunca Memas engelleyin.
- Aspirate engelleme tampon ve üçlü durulama kuyuları PBS ile. Kurutma önlemek için, hücre kaplama için hazır olana kadar kuyularda PBS son hacmi bırakın.
Not: MEMAs üzerinde hücre kültürü adımları için iki tezgah çalışanı olması son derece yararlıdır. Bir tezgah çalışanı aspirasyon adımlarını gerçekleştirebilir, ikincisi ise ek adımlar gerçekleştirir. 1 mL çok kanallı pipet ile pipetleme için 8-kuyu plakasını ve iki Pasteur pipeti ile Y-Splitter ile aynı anda birden fazla kuyu aspirine uyacak şekilde aralıklı ipuçları kullanmak önerilir. - Tohum 2 x 105 MCF7 hücrelerde her Iyi 2 ml Dulbecco 'Nun modifiye kartal Orta (dmem) orta içeren 10% fetal sığır serumu (FBS).
Not: Tam MEMA deneyinden önce, MEMA noktalarının yüksek hücre numaralarıyla (ancak confluent olmayan) istenen deneysel sürenin sonunda hücre numaralarını en iyi duruma getirmek için bir hücre titrasyon deneyi gerçekleştirin. - 2 − 18 h yapışma, aspirat orta ve 2 mL azaltılmış büyüme orta (DMEM ile 0,1% FBS) ile değiştirin.
Not: Azaltılmış serum (örn.% 0,1 FBS) veya büyüme faktörü-tükenmiş koşullar Şu anda belirli liglerin uyarıcı etkisini izole etmek için kullanılabilir. - Buz üzerinde bir ligand tedavi plakasını çözün. Santrifüjlü çözünen plaka 200 x g 'de 1 dk.
- Transfer 200 μL, kültür plakasının her bir kuyunda, tedavi plakasında uygun bir şekilde orta olan. Pipet yukarı ve aşağı orta ile ligand hacmi karıştırın ve MEMA plaka uygun iyi geri bu karışımı aktarmak için.
- Elle hafifçe kaya ve MEMA plakaları inküyaya dönün. 37 °C ve 5% CO2' de ligand/ECM kombinasyonunun varlığını deneme süresi için kültür.
Not: Tipik bir MEMA deneyi 72 h için çalışır; uzun süreli deneyler orta ve yeniden tedavi ligand ile değiştirilmesi gerekebilir. - 71 h 'de nabız MEMA kuyuları 10 μM son konsantrasyon için 100x 5-etynyl-2'-deoxyuridine (EdU) ile. EdU ile deneysel koşullarda, 37 °C ' de 1 h ve% 5 CO2' de inkük edilir.
Not: Diğer canlı hücre uygulamaları da şu anda kullanılabilir.
7. sabitleme ve boyama MEMAs
- Sonra 72 h ve herhangi bir canlı hücre tedavileri, Aspire kuyuları. Min 'de 15 dakika boyunca 2% civarında formaldehite (PFA) başına 2 ml 'de Memas 'ı düzeltin.
- Aspirate PFA. 15 dk için 0,1% noniyonik yüzey başına 2 mL ile geçirgen.
- Noniyonik yüzey kaynağı aspirate ve PBS iyi başına 2 mL ile yıkayın. Aspirate PBS. 0,05% polisorbat 20 (PBS-T) ile 2 ml PBS ile yıkayın.
Not: MEMA yüzeyi hidrofobiktir ve leke ve antikor inkübasyon öncesinde PBS-T ile yıkanmaması, kuluçk basamakları sırasında kuyularda boşlukların oluşması ve boyama yapılarına yol açacaktır. - Aspirate PBS-T. EdU algılama reaksiyon reaktifleri ekleyin. RT 'de 1 saat boyunca kulbirer yapın, sallanan ve ışıkta korunur. 1 h inkübasyon sonra, sağlanan ticari gidermek tampon ile reaksiyonu azaltma.
Not: EdU algılama ve boyama/antikor basamakları maliyeti azaltmak için iyi başına 1,5 mL 'de gerçekleştirilebilir. - Su giderici tamponu aspirate ve lekeleri veya antikorlar ile inküp önce PBS-T ile yıkayın.
- MEMA kuyularını% 2 (w/v) Sığır serum albümin (BSA), 1 mM MgCl2 ve 0,02% Nan3 gece 4 °c ' de içeren boyama tamponunda histon H3K9me3 (1:1000) ve fibrillarin (1:400) karşı antikorlar ile kulbin.
Not: Tam MEMA kümesinde kullanmadan önce optimum konsantrasyonları belirlemek için antikor titrasyonları gerçekleştirin. - Primer antikor veya leke İnkübasyon sonrasında, 2x PBS ile ve bir kez PBS-T ile yıkayın kuyuları.
- İkincil antikorlar ekleyin (eşek Anti-fare IgG ve eşek Anti-tavşan IgG, hem 1:300) ve 0,5 μg/mL 4 ′ 6 ‐ diamidino ‐ 2 ‐ phenylindole (DAPI). Karanlıkta RT 'de 1 saat boyunca inküye yapın.
- 2 mL 'Lik PBS 'ye kadar, iki adet x, 2 ml ile yıkayın.
- Işıktan korunan 4 °C ' de PBS 'de daha sonra görüntüleme için görüntülenmiş MEMAs 'a devam edin.
8. MEMAs 'ın görüntülenmesi
- Uygun floresan algılama kanalları ile otomatik görüntüleme sistemi üzerinde görüntü MEMA.
- Bir görüntü yönetim sistemine elde edilen görüntü verilerini çıktı. Hücreleri segment ve Cell Profiler8kullanarak yoğunluk düzeylerini hesaplayın.
9. veri analizi
Not: Veri Analizi normalleştirme, varyasyon düzeltme ve ham CellProfiler türetilmiş veri özetleme oluşur. Bu örnekte, özel kod ile R-ortamı tüm adımları gerçekleştirmek için kullanılır. Ancak, herhangi bir istatistiksel ortam veya yazılım programı eşdeğer eylemleri gerçekleştirmek için kullanılabilir. Çözümleme için R ortamı için açık kaynak özel kod örneği kullanılabilir: https://www.SYNAPSE.org/#!Synapse:syn2862345/wiki/72486.
- Bölümlenmiş görüntü verilerini preprocess ve Normalleştir.
- DAPı lekeli çekirdekleri kullanarak spot hücre sayısını belirleyin.
- Hücreleri EdU+olarak etiketlemek için edu yoğunluğu otomatik kapı. Her noktada EdU+ hücre oranını kullanarak proliferasyonu ölçün.
- Sitoplazmik lekeleri ve nükleer morfoloji ölçümlerini spot düzeyde özetlemektedir.
- Veri kalitesini artırmak için veriler üzerinde istenmeyen varyasyon (RUV) normalleştirme kaldırılması gerçekleştirin9.
Not: Bu yaklaşım, her yoğunluğa ve morfoloji sinyaline, daha önce9olarak açıklandığı gibi satırlar ve lekeler kullanarak diziler içeren bir matris olarak bağımsız olarak uygulanır. - Uzamsal veya yoğunlukla ilgili efektler için düzeltmek için bağımsız değişkenler olarak dizi satırını ve dizi sütununu kullanarak ruv normalleştirilmiş kalıntılar için iki değişkenli Loess normalleştirme uygulayın.
- Normalleştirme tamamlandıktan sonra medyan, raporlama ve daha fazla çözümleme için her mikro ortam koşulu için çoğaltır özetler.
Sonuçlar
Hücre büyümesi ve proliferasyonu üzerinde mikroçevresel etkileri kolaylaştırmak ve hücre büyümesini ve proliferasyonu teşvik eden veya engellenmiş olan koşulları belirlemek için, meme kanseri hücre hattı MCF7 protokolde açıklandığı gibi sekiz 8-iyi Memas bir dizi üzerinde seribaşı oldu. Bu tahlil 48 farklı ECMs ve 57 farklı ligler, toplam 2736 Kombinatoryal mikroçevre koşulları için hücreleri maruz. Kültüre 71 h sonra, hücreler EdU ile darbeli, sabit, geçirgen, ve DAPı ile lekelenmiş, EdU algılama için reaksiyon, bir anti-fiilin antikor, ve bir anti-H3K9me3 antikor. Hücreler yüksek içerik mikroskobu üzerinde görüntülenmiş. Görüntüler bir Omero sunucusuna yüklendi10, cellprofiler8kullanılarak bölümlenmiş ve normalleştirilmiş ve R9analiz. Aşağıda açıklanan sonuçlar DAPı ve EdU sinyallerine odaklanmaktadır.
MEMAs 'ın görüntü analizi platformu, belirli bir ligand (Şekil 3a) ile tedavi edilen hücreler Için 2n ve 4N fraksiyonları gösteren DNA içerik grafikleri gibi akış sitometri yaklaşımlarından mevcut olanlara benzer bazı sonuçlar verir. yoğunluğu ve alanı. Bu araziler, G1 veya G2 aşamalarında hücrelere karşılık gelen açık bimodal zirveler ile gösterilen karşı aktif hücre bisiklet teşvik koşulları için kanıt sağlamak vs büyüme tutuklandı hücreler, kontrol koşullarına göre doruklarında değişiklikler gösterecektir. Hücre numarasını ve boyama yoğunluğu verilerini, mikroortamın (bir eksendeki ligin, ikinci eksendeki ECM 'nin) her iki hücre numarasına (Şekil 3B) ve edu 'nın (Şekil 3C) üzerinde etkisi olan verileri özetlemek için kullanırız. daha kolay heatmap renk ve yoğunluğu değişiklikler olarak görülebilir. Bu arazilerin görüldüğü gibi, ECM durumu güçlü bir şekilde hücre numarasını veya EdU pozitifliği etkilmediği için etkilerinin çoğu ligand odaklı. Nidogen-1, bu ECM molekülünün varlığı MCF7 ' in hücre bağlamasını ve büyümesini inhibe ettiği için net bir istisna oluşturmaktadır. FGF6 ve NRG1α (grafiklerde NRG 1.1) gibi ligler hücre numarasını geliştirir ve EdU 'nın yüksek oranlarına sahiptir, ancak AREG ve NRG1-SMDF (grafiklerde NRG 1.10) gibi ligler hücre bağlamasını ve/veya hücrelerin büyümesini inhibe eder. Bu bulgular, hücre numarası ve EdU pozitifliğinde net bir farkın belirgin olduğu noktalar üzerinde büyüyen hücrelerin görüntüleri tarafından desteklenir (örnek Şekil 3D'de bakın).
MEMA platformu daha yeni bir teknoloji olduğundan, sonuçlar ayrı olarak doğrulandı. MCF7 hücreler,% 10 FBS ile DMEM Orta içinde kollajen ile kaplanmış 24-kuyu plakaları içine tohumlandı. 18 saat sonra, medya azaltılmış büyüme ortamı için takas edildi (DMEM ile 0,1% FBS) ve hücreler NRG1α ile tedavi edildi, FGF6, veya AREG ve kültür 72 h. EdU eklendi 1 h önce fiks. Hücreler DAPı ile lekelenmiş ve EdU 'nın birleştirilme, görüntülenmiş, segmentli ve analiz edildi. MEMA platformundan elde edilen sonuçlara benzer şekilde, FGF6 ve NRG1α her ikisi de daha yüksek hücre numaralarına (Şekil 4A) ve edu Kuruluş oranlarına (ŞEKIL 4B) göre Areg işlenmiş hücrelere kıyasla, gözlemlerimizi doğrulama Orijinal MEMA deneyleri.

Şekil 1 : Tipik BIR Mema deneyinin farklı aşamaları için iş akışını ve zaman çizelgesini gösteren akış şeması. MEMAs yazdırıldığında, kullanım öncesi birkaç ay boyunca kurutulmuş oda sıcaklığında saklanabilirler. Tipik olarak, deneysel faz 3 − 4 gün sürer, ancak bazı yavaş büyüyen Primer hücreler MEMAs üzerinde 2 hafta boyunca kültürlüdür. Bu figürün daha büyük bir versiyonunu görmek Için lütfen tıklayınız.

Şekil 2 : Dizi baskı Için ECM kaynak plakası düzeni. Kollajen bloğu MEMA üzerine bir kılavuz olarak yazdırılır, bu da kuyular arasında daha sağlam normalleşme için izin veren son derece tekrarlayan bir koşul kümesi sağlar. PBS dolu kuyular, baskı işlemi sırasında buharlaşma önlenmesinde yardımcı olmak için nem sağlar. Bu figürün daha büyük bir versiyonunu görmek Için lütfen tıklayınız.

Şekil 3 : Tipik BIR Mema deneyinden oluşturulan veri örnekleri. (A) farklı ligler ile tedavi edilen bir 8-kuyu plakasından hücre sayar karşı BINNED DAPI yoğunluğu değerlerinin hücre döngüsü profilleri, iki taraflı DAPI yoğunluğu boya gösteren G1 ile G2 hücre döngüsü aşamasında hücreleri gösterir. (B) heatmap hiyerarşik kümeleme kullanarak benzerlik tarafından kümelenmiş normalleştirilmiş spot hücre sayar gösteriliyor. Kırmızı daha yüksek hücre numarasını gösterir ve mavi daha düşük hücre numarasıdır. Ligler x ekseni üzerinde, ECMs y ekseni üzerindedir. (C) heatmap normalleştirilmiş edu eklenmesi gösteren, kırmızı daha yüksek ve mavi gösteren daha düşük edu dahil. Ligler x ekseni üzerinde, ECMs y ekseni üzerindedir. (D) NRG1-α ile tedavi edilen, yüksek edu birleştirmesi (pembe çekirdekler) gösteren bir Mema NOKTASıNDA büyüyen MCF7 hücre örneği. Yeşil leke hücre maskesi ve mavi DAPı olduğunu. Bu figürün daha büyük bir versiyonunu görmek Için lütfen tıklayınız.
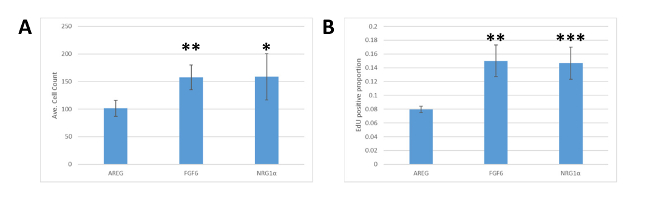
Şekil 4 : MEMA doğrulama hücre kültüründe sonuçlanır. (A) farklı LIGLERLE MCF7 tedavisinde kaynaklanan hücre numarasının ölçülmesini. MCF7 hücrelerin eşdeğer numaraları, daha sonra AREG, FGF6 veya NRG1α ile tedavi edilen Multiwall plakalara kaplamalı. AREG ile tedavi Wells FGF6 ile tedavi olandan önemli ölçüde daha az hücre vardı (* * öğrenci t-testi p-değerleri daha az 0,01) veya NRG1α (* gösterir 0,05 bir p-değeri) 72 h sonrası ligand tedavisi. (B) Apanelinde olduğu gibi, farklı LIGLERLE tedavi nedeniyle MCF7 yılında edu 'nın eklenmesi seviyesinin ölçülmesi. AREG tedavisi, EdU içeren hücrelerin FGF6 (* *, p < 0,01) veya NRG1α (** *, p = 0,01) ile tedavi edilen hücrelerin önemli ölçüde daha düşük bir oranıyla sonuçlanır. Hata çubukları standart sapma temsil eder. Bu figürün daha büyük bir versiyonunu görmek Için lütfen tıklayınız.
| Protein adı | Uniprot KIMLIĞI | Stok Konsantrasyon (μg/mL) | Son Konsantrasyon (μg/mL) |
| ANGPT1 | 1 | Q15389 | 1 | 100 | 0,04 |
| ANGPT2 | 1 | O15123 | 1 | 100 | 0,2 |
| Areg | P15514 | 100 | 0,02 |
| BMP2 | P12643 | 100 | 0,1 |
| BMP3 | P12645 | 1000 | 0,1 |
| BMP4 | P12644 | 100 | 0,1 |
| BMP5 | 1 | P22003 | 1 | 100 | 0,1 |
| BMP6 | P22004 | 100 | 0,1 |
| BMP7 | P18075 | 100 | 0,1 |
| CSF2 | P04141 | 100 | 0,02 |
| CTGF | 1 | P29279 | 1 | 100 | 0,05 |
| CXCL12 | Alfa | P48061 | 2 | 100 | 0,01 |
| CXCL12 | Beta | P48061 | 1 | 100 | 0,03 |
| CXCL1 | P09341 | 100 | 0,004 |
| CXCL8 | 1 | P10145 | 1 | 100 | 0,3 |
| DLL1 | 1 | O00548 | 1 | 500 | 0,5 |
| DLL4 | Q9NR61 | 200 | 0,6 |
| EGF | 1 | P01133 | 1 | 500 | 0,01 |
| FASLG | 1 | P48023 | 1 | 10 | 0,02 |
| FGF2 | 3 | P09038 | 2 | 100 | 0,01 |
| FGF6 | P10767 | 100 | 0,01 |
| FLT3LG | 1 | P49771 | 1 | 50 | 0,001 |
| GPNMB | 1 | Q14956 | 1 | 100 | 0,5 |
| HGF | 1 | P14210 | 1 | 50 | 0,04 |
| IGF1 | 1 | P05019 | 1 | 200 | 0,01 |
| IGFBP2 | P18065 | 100 | 0,05 |
| IGFBP3 | 1 | P17936 | 1 | 100 | 0,1 |
| IL13 | P35225 | 100 | 0,01 |
| IL15 | IL15S48AA | P40933 | 1 | 50 | 0,01 |
| IL1B | P01584 | 25 | 0,001 |
| IL6 | P05231 | 100 | 0,01 |
| IL7 | 1 | P13232 | 1 | 100 | 0,01 |
| JAG1 | 1 | P78504 | 1 | 200 | 0,5 |
| JAG2 | Uzun | Q9Y219 | 1 | 100 | 0,5 |
| KITLG | 1 | P21583 | 1 | 100 | 0,005 |
| KNG1 | HMW | P01042 | 1 | 100 | 0,2 |
| LEP | P41159 | 1000 | 0,002 |
| LYVE1 | Q9Y5Y7 | 100 | 0,05 |
| NRG1 | 10 | Q02297 | 10 | 100 | 0,01 |
| NRG1 | 1 | Q02297 | 1 | 100 | 0,05 |
| NRG1 | 6 | Q02297 | 6 | 100 | 0,01 |
| GFAB | go1990265 | 100 | 0,05 |
| PDGFB | 1 | P01127 | 1 | 100 | 0,05 |
| PTN | P21246 | 100 | 0,5 |
| SHH | Q15465 | 100 | 0,5 |
| TGFB1 | | Cterminus | P01137 | Cterminus | 20 | 0,01 |
| TGFB1 | | Tur | P01137 | Tur | 100 | 0,15 |
| TGFB2 | A | P61812 | 1 | 20 | 0,01 |
| THPO | 1 | P40225 | 1 | 50 | 0,002 |
| TNFRSF11B | O00300 | 100 | 0,02 |
| TNFSF11 | 1 | O14788 | 1 | 100 | 0,01 |
| Tnf | P01375 | 100 | 0,01 |
| VEGFA | VEGF206 | P15692 | 1 | 100 | 0,01 |
| WNT10A | Q9GZT5 | 100 | 0,1 |
| WNT3A | 1 | P56704 | 1 | 200 | 0,1 |
| Wnt5a | 1 | P22725 | 1 | 100 | 0,1 |
Tablo 1: MEMA denemeleri için kullanılan ligin tam listesi. Uniprot KIMLIĞI, stok konsantrasyonları ve son çalışma konsantrasyonları sağlanır.
| ECM protein | Tek Protid | Stok konsantrasyonu (μg/mL) | Son Konsantrasyon (μg/mL) | Notlar |
| ALCAM | 1 | Q13740 | 1 | 100 | 30 | |
| CDH20 | Q9HBT6 | 300 | 80 | |
| CDH6 | 1 | P55285 | 1 | 100 | 40 | |
| CDH8 | P55286 | 100 | 20 | |
| CD44 | 1 | P16070 | 1 | 100 | 30 | |
| CEACAM6 | P40199 | 100 | 30 | |
| COL1A1 | P02453 | 5000 | 200 | birden fazla uniprot kimlikleri ile birden fazla alt paketleri |
| COL2A1 | 2 | P02458 | 2 | 1000 | 200 | |
| COL3A1 | 1 | P02461 | 1 | 1000 | 200 | |
| COL4A1 | 1 | P02462 | 1 | 1000 | 200 | birden fazla uniprot kimlikleri ile birden fazla alt paketleri |
| COL5A1 | P20908 | 1000 | 200 | |
| COL23A1 | 1 | Q86Y22 | 1 | 200 | 80 | |
| DSG2 | Q14126 | 100 | 30 | |
| CDH1 | 1 | P12830 | 1 | 100 | 40 | |
| ECM1 | 1 | Q16610 | 1 | 100 | 40 | |
| FN1 | 1 | P02751 | 1 | 1000 | 200 | |
| GAP43 | 1 | P17677 | 1 | 158 | 40 | |
| HyA-500K | 1000 | 200 | LOR-0005 | |
| HyA-50K | 1000 | 200 | LOR-0007 | |
| ICAM1 | P05362 | 400 | 80 | |
| ALCAM | 1 | Q13740 | 1 | 100 | 30 | |
| CDH20 | Q9HBT6 | 300 | 80 | |
| CDH8 | P55286 | 100 | 20 | |
| CD44 | 1 | P16070 | 1 | 100 | 30 | |
| CEACAM6 | P40199 | 100 | 30 | |
| DSG2 | Q14126 | 100 | 30 | |
| CDH15 | P55291 | 100 | 20 | |
| VCAM1 | 1 | P19320 | 1 | 1000 | 200 | |
| LAMA1 | P25391 | 500 | 200 | birden fazla uniprot kimlikleri ile birden fazla alt paketleri |
| LAMA3 | 2 | Q16787 | 2 | 130 | 40 | |
| Lum | P51884 | 200 | 80 | |
| CDH15 | P55291 | 100 | 20 | |
| NID1 | 1 | P14543 | 1 | 100 | 9,3 μg/mL Nid, 130 μg/mL Lam, 46,5 μg/mL COL4 | + COL4 ve laminin |
| Omd | Q99983 | 100 | 40 | |
| SPP1 | A | P10451 | 1 | 100 | 40 | |
| CDH3 | 1 | P22223 | 1 | 100 | 40 | |
| PECAM1 | Uzun | P16284 | 1 | 150 | 40 | |
| TNC | 1 | P24821 | 1 | 500 | 200 | |
| VCAM1 | 1 | P19320 | 1 | 1000 | 200 | |
| Vtn | P04004 | 100 | 40 | |
| Bgn | P21810 | 100 | 40 | |
| DCN | A | P07585 | 1 | 300 | 80 | |
| POSTN | 1 | Q15063 | 1 | 100 | 40 | |
| Sparc | P09486 | 100 | 40 | |
| THBS1 | 1 | P07996 | 1 | 100 | 40 | |
| BCAN | 1 | Q96GW7 | 1 | 100 | 40 | |
| ELN | 3 | P15502 | 3 | 1000 | 200 | |
| FBN1 | P35555 | 254 | 80 |
Tablo 2: MEMA deneylerinde kullanılan ECM proteinleri ve koşullarının tam listesi. Uniprot KIMLIĞI, stok konsantrasyonları ve son çalışma konsantrasyonları sağlanır. Bazı durumlarda, yazdırılan koşul, notlar sütununda belirtilen bir protein kompleksini veya birden fazla proteinin birleşimini temsil eder.
Tartışmalar
"Dimensionality" ve içeriğin önemi, mikroçevre11ile etkileşimi ile kanser hücrelerinin karakterizasyonu araçları olarak in vitro kültür sistemlerinin geliştirilmesi konusunda motive edici bir faktör olmuştur ve in vitro yeteneği Kültür sistemleri in vivo çevre taklit etmek için bu kültür sistemleri geliştirmek için arayışı arkasında bir itici kuvvettir. In vitro sistemlerde, ancak, oldukça basitleştirilmiş bir model12aşağı kompleks in vivo durumda damıtmak yeteneğini nedeniyle kanser araştırması önemli araçlar kalır.
2D sistemleri ECMS ve ligler içerebilir rağmen, onlar geleneksel Kombinatoryal pertubagens geniş bir panel sorgulamak için verim kapasiteleri yoksun. Popüler ticari Bodrum membran özler 3D kültür için izin, ancak proteinleri dikkatle tanımlanmış bir panel yerine eksikliği. Ticari özler genellikle tamamen tanımlanan kompozisyon muzdarip, hangi analiz ve sonuç önemli toplu-toplu varyasyon3,13neden olabilir. MEMA platformu bu engelleri aşmak, hücresel fenotiplerde değişiklik çalışması için izin, metabolik aktivite, farklılaşma durumu, ve hücre büyümesi ve proliferasyon varyasyonları belirli ve tanımlanmış endojen tarafından modüler olarak Faktörler.
MEMA platformu, mikroortamın (ECM ve çözünür faktörlerin) hücrelerin fenotipi üzerinde etkisini değerlendirmek için güçlü, orta-yüksek verimlilik yaklaşımıdır. Platform, kullanılabilen asder ve hücreler için büyük bir esneklik gösterir. Hem çözünür liglerin hem de hücrelerin maruz kaldığı ECM proteinlerinin etkilerini gözlemleyebiliriz. Nitekim, son zamanlarda bu liglerin HER2 hedefli İnhibitörlere karşı büyük bir direnç sürücüsü olduğunu bulduk, ancak bu etkiler ECM5tarafından modüle edilebilir. Akciğer, mesane, prostat, meme ve pankreas yanı sıra indüklenen pluripotent kök (IPS) hücreleri de dahil olmak üzere farklı hücre türlerinden türetilen Primer hücreler ve hücre çizgileri de dahil olmak üzere çeşitli hücreler, başarıyla MEMA platformunda kültürlü olmuştur (bkz. başvurular5,7,14) örnekleri. Farklı lekelerin kullanımı, hücre büyümesi, farklılaşma ve metabolizma dahil olmak üzere birden fazla hücresel uç noktasının okunmasını sağlar. Diğer araştırmacılar, MEMA platforma ek bir boyut ekleyerek, sertlik veya elastik modülün etkisini sorgulamak için platformu uzattı15. Son olarak, Platform, ilaç etkinliğini artıran veya inhibe eden mikroçevre koşullarının tanımlanması için ilaç ekranlarının gerçekleştirilmesini sağlar, biz ve diğerleri son zamanlarda5,14,15 ' e bildirmişlerdir. .
Belki de bir MEMA deneyinin başarısını en önemli adım hücre kaplama yoğunluğu optimize etmektir. Hücrelerin yoğunluğunu optimize etmek, sağlam veri sağlamak için yeterli hücrelerin mevcut olmasını sağlar, ancak o kadar çok nokta aşırı konfluent olur. Özellikle proliferasyon bir uç nokta olarak kullanıldığında, düşük proliferasyon oranlarının mikroçevresel faktörlerle etkileşimlerin bir sonucu olup olmadığını belirlemek imkansız hale getirmek veya temas inhibisyonu nedeniyle konfluent lekeler önemli ölçüde sonuçları konabilir yüksek hücresel yoğunluk. Hücre titrasyon denemeleri bu sorunları ortaya çıkarabilir, spot başına ortalama hücre numaraları kaplama hücrelerin sayısı artan doğrusal bir artış gösterecektir, ancak sonunda Plato olacaktır. En iyi hücre numarası eğrinin doğrusal aralığında seçilmelidir.
Yukarıda belirtildiği gibi, MEMA platformu esnektir ve farklı yüzeylere sahip çeşitli substratlar üzerinde hazırlanabilir. Bunlar arasında cam slaytlar ve Multiwall Plate formatları yer aldı. Bizim deneyimimizde, tüm yüzey kimyaları Mema baskı için, biz kötü yapışma özellikleri ve diğer yüksek yapışma yüzeylerde hücre yapışmasını engellemek için yetersizlik nedeniyle bazı yüzeylerde spot dekolmanı gözlemledik için izin verilmez. Ayrıca, farklı substratlar arasında değişen tampon koşullarının optimizasyonu gerektirir, aynı baskı tampon ile baskı performansı yüzey kimyası bağlı olarak değişebilir gibi.
Yazdırılan ECM noktaların çapı veri kalitesinde önemli bir rol oynamaktadır. Genel olarak, kullanımda dizici için kullanılabilen en büyük çap baskı pimlerini kullanmanızı öneririz (Şu anda 350 μm çap iğne kullanıyoruz). Daha büyük çaplı lekeler, daha küçük çaplı pimler ile üretilenden daha sağlam verilere neden olan bir noktayı daha fazla sayıda hücreye kaplayacak şekilde sağlar. Hücrelerin bağlanması Stokastik bir süreçtir olduğundan, orijinal her noktaya bağlı hücrelerin sayısı ile ilgili veri değişkenlik yüksek derecede olma eğilimindedir. Böylece, her ECM koşulu için çok sayıda çoğaltır yazdırmanızı öneririz. Güçlü istatistiklerin sağlanması için mevcut baskı Koşullarımız ile her bir kuyunda 10 − 15 ECM çoğaltır.
Biz geçmiş deneylerde, en çok, ligand etkileri ECM etkileri üzerinde hakim eğilimindedir kaydetti. Bu, güçlü hücre bağlayıcılığı sağlayan tüm ECM lekeleri için kollajen ı ekleme kararımız nedeniyle kısmen olabilir. Ancak, bu da ECM etkileri homojenize olduğuna inanıyoruz, çoğu noktalar bir şekilde kolajen benzer bir şekilde davranmaya eğilimindedir ı. kollajen dışlamak için spot kompozisyon değiştirme ile etkileşiminin bir sonucu olarak diferansiyel hücre davranışına neden olabilir ECM, ama aynı zamanda önemli ölçüde etkiler hücre bağlayıcı, daha birçok işgal olmayan noktalar sonuçlanan. Kullanıcılar ECM bileşiminin bu farklılıkları akılda tutmaları, özellikle kök ve progenitör hücreler ve farklılaşma ile ilgilenenler, matrisin önemli bir etki yaratabileceği16.
Biz genellikle nispeten kısa bir süre için Mema deneyleri gerçekleştirmek (örn., 72 h maksimum). Hücrelerin noktalar (engelleme arabellek bizim deneyimimizde noktalar dışında büyüme için izin vermez) kısıtlı olmasıdır. Hızla bölünmüş hücreler ile, büyüme daha uzun 72 h nokta, hangi sırayla hücreler kalabalık ve birbirlerine kazık gibi görüntü segmentasyon karmaşıklaşır, ve aynı zamanda büyüme tutuklama temas inhibisyonu ile oluşabilir olarak verileri etkileyebilir büyümeye yol açacaktır. Çok yavaş büyüyen primer hücrelerle (10 − 14 gün) daha uzun tedaviler gerçekleştirdik, ancak medyayı değiştirmek ve her 3 − 4 günde bir Ligat doldurmak için bu konuda bakım yapılmalıdır.
MEMA platformunu geliştirmek için devam eden çabalar, iki ilgi alanına odaklanmıştır, daha küçük kültür gemileri içinde görüntüleme ve optimizasyon için optik kalitesinin maksimizasyonu. Optik kalite, araştırmacılar, ilgi kendi işaretçileri alt hücre lokalizasyonu belirlemek için yüksek çözünürlüklü mikroskobik gerektirdiğinde önemli bir faktör haline gelir. İlk ekranlar yüksek verim mikroskoplar daha düşük çözünürlükte daha yüksek çözünürlüklü araçlarla ilgi belirli noktalar görüntüleme takip yapılabilir, ancak substrat optik özellikleri zayıf ise görüntü kalitesi tehlikeye olabilir. Substratın optik özelliklerinin geliştirilmesi, araştırmacılar yüksek çözünürlükte Seçilen görüntüleri yeniden elde etmek için gerek kalmadan üst çözünürlüklü görüntüleme sistemlerinde ilk ekranları gerçekleştirmek için izin verir. Son olarak, 96-Well plakaları gibi küçük kültür gemilerin MEMAs gerçekleştirmek için yeteneği, tedavi hacmi bir azalma ve sorguya ligler ve çoğaltır bir genişleme izin verir. Bu geçiş yeni kültür gemileri içinde substrat-tampon-protein etkileşimleri ve dizi baskı optimizasyonu gerektirir. Böyle devam eden çabaları MEMA platformu geliştirmek ve daha sonra daha sonra incelenebilir hücre türleri, çeşitli hücresel fenotürleri değiştirmek ilgili mikroçevresel proteinleri belirlemek için güçlü yetenekleri üzerine genişletmek olacak teyit edilir.
Açıklamalar
Yazarların ifşa etmesi gereken hiçbir şey yok.
Teşekkürler
Bu çalışma, ağ hücresel Imzaları (LıNCS) Grant HG008100 (J.W.G., L.M.H. ve J. E. K) NıH ortak fon Kütüphanesi tarafından destekleniyordu.
Malzemeler
| Name | Company | Catalog Number | Comments |
| Aushon 2470 | Aushon BioSystems | Arrayer robot system used in the protocol | |
| Nikon HCA | Nikon | High Content Imaging system designed around Nikon Eclipse Ti Inverted Microscope | |
| BioTek Precision XS liquid Handler | BioTek | liquid handling robot used in the protocol | |
| Trizma hydrochloride buffer solution | Sigma | T2069 | |
| EDTA | Invitrogen | 15575-038 | |
| Glycerol | Sigma | G5516 | |
| Triton X100 | Sigma | T9284 | |
| Tween 20 | Sigma | P7949 | |
| Kolliphor P338 | BASF | 50424591 | |
| 384-well microarray plate, cylindrical well | Thermo Fisher | ab1055 | |
| Nunc 8 well dish | Thermo Fisher | 267062 | |
| Paraformaldehyde 16% solution | Electron Microscopy Science | 15710 | |
| BSA | Fisher | BP-1600 | |
| Sodium Azide | Sigma | S2002 | |
| Cell Mask | Molecular Probes | H32713 | |
| Click-iTEdU Alexa Fluor | Molecular Probes | C10357 | |
| DAPI | Promo Kine | PK-CA70740043 | |
| ALCAM | R & D Systems | 656-AL | ECM |
| Cadherin-20 (CDH20) | R & D Systems | 5604-CA | ECM |
| Cadherin-6 (CDH6) | R & D Systems | 2715-CA | ECM |
| Cadherin-8 (CDH8) | R & D Systems | 188-C8 | ECM |
| CD44 | R & D Systems | 3660-CD | ECM |
| CEACAM6 | R & D Systems | 3934-CM | ECM |
| Collagen I | Cultrex | 3442-050-01 | ECM |
| Collagen Type II | Millipore | CC052 | ECM |
| Collagen Type III | Millipore | CC054 | ECM |
| Collagen Type IV | Sigma | C5533 | ECM |
| Collagen Type V | Millipore | CC077 | ECM |
| COL23A1 | R & D Systems | 4165-CL | ECM |
| Desmoglein 2 | R & D Systems | 947-DM | ECM |
| E-cadherin (CDH1) | R & D Systems | 648-EC | ECM |
| ECM1 | R & D Systems | 3937-EC | ECM |
| Fibronectin | R & D Systems | 1918-FN | ECM |
| GAP43 | Abcam | ab114188 | ECM |
| HyA-500K | R & D Systems | GLR002 | ECM |
| HyA-50K | R & D Systems | GLR001 | ECM |
| ICAM-1 | R & D Systems | 720-IC | ECM |
| Laminin | Sigma | L6274 | ECM |
| Laminin-5 | Abcam | ab42326 | ECM |
| Lumican | R & D Systems | 2846-LU | ECM |
| M-Cad (CDH15) | R & D Systems | 4096-MC | ECM |
| Nidogen-1 | R & D Systems | 2570-ND | ECM |
| Osteoadherin/OSAD | R & D Systems | 2884-AD | ECM |
| Osteopontin (SPP) | R & D Systems | 1433-OP | ECM |
| P-Cadherin (CDH3) | R & D Systems | 861-PC | ECM |
| PECAM1 | R & D Systems | ADP6 | ECM |
| Tenascin C | R & D Systems | 3358-TC | ECM |
| VCAM1 | R & D Systems | ADP5 | ECM |
| Vitronectin | R & D Systems | 2308-VN | ECM |
| Biglycan | R & D Systems | 2667-CM | ECM |
| Decorin | R & D Systems | 143-DE | ECM |
| Periostin | R & D Systems | 3548-F2 | ECM |
| SPARC/osteonectin | R & D Systems | 941-SP | ECM |
| Thrombospondin-1/2 | R & D Systems | 3074-TH | ECM |
| Brevican | R & D Systems | 4009-BC | ECM |
| Elastin | BioMatrix | 5052 | ECM |
| Fibrillin | Lynn Sakai Lab OHSU | N/A | ECM |
| ANGPT2 | RnD_Systems_Own | 623-AN-025 | Ligand |
| IL1B | RnD_Systems_Own | 201-LB-005 | Ligand |
| CXCL8 | RnD_Systems_Own | 208-IL-010 | Ligand |
| IGF1 | RnD_Systems_Own | 291-G1-200 | Ligand |
| TNFRSF11B | RnD_Systems_Own | 185-OS | Ligand |
| BMP6 | RnD_Systems_Own | 507-BP-020 | Ligand |
| FLT3LG | RnD_Systems_Own | 308-FK-005 | Ligand |
| CXCL1 | RnD_Systems_Own | 275-GR-010 | Ligand |
| DLL4 | RnD_Systems_Own | 1506-D4-050 | Ligand |
| HGF | RnD_Systems_Own | 294-HGN-005 | Ligand |
| Wnt5a | RnD_Systems_Own | 645-WN-010 | Ligand |
| CTGF | Life_Technologies_Own | PHG0286 | Ligand |
| LEP | RnD_Systems_Own | 398-LP-01M | Ligand |
| FGF2 | Sigma_Aldrich_Own | SRP4037-50UG | Ligand |
| FGF6 | RnD_Systems_Own | 238-F6 | Ligand |
| IL7 | RnD_Systems_Own | 207-IL-005 | Ligand |
| TGFB1 | RnD_Systems_Own | 246-LP-025 | Ligand |
| PDGFB | RnD_Systems_Own | 220-BB-010 | Ligand |
| WNT10A | Genemed_Own | 90009 | Ligand |
| PTN | RnD_Systems_Own | 252-PL-050 | Ligand |
| BMP3 | RnD_Systems_Own | 113-BP-100 | Ligand |
| BMP4 | RnD_Systems_Own | 314-BP-010 | Ligand |
| TNFSF11 | RnD_Systems_Own | 390-TN-010 | Ligand |
| CSF2 | RnD_Systems_Own | 215-GM-010 | Ligand |
| BMP5 | RnD_Systems_Own | 615-BMC-020 | Ligand |
| DLL1 | RnD_Systems_Own | 1818-DL-050 | Ligand |
| NRG1 | RnD_Systems_Own | 296-HR-050 | Ligand |
| KNG1 | RnD_Systems_Own | 1569-PI-010 | Ligand |
| GPNMB | RnD_Systems_Own | 2550-AC-050 | Ligand |
| CXCL12 | RnD_Systems_Own | 350-NS-010 | Ligand |
| IL15 | RnD_Systems_Own | 247-ILB-005 | Ligand |
| TNF | RnD_Systems_Own | 210-TA-020 | Ligand |
| IGFBP3 | RnD_Systems_Own | 675-B3-025 | Ligand |
| WNT3A | RnD_Systems_Own | 5036-WNP-010 | Ligand |
| PDGFAB | RnD_Systems_Own | 222-AB | Ligand |
| AREG | RnD_Systems_Own | 262-AR-100 | Ligand |
| JAG1 | RnD_Systems_Own | 1277-JG-050 | Ligand |
| BMP7 | RnD_Systems_Own | 354-BP-010 | Ligand |
| TGFB2 | RnD_Systems_Own | 302-B2-010 | Ligand |
| VEGFA | RnD_Systems_Own | 293-VE-010 | Ligand |
| IL6 | RnD_Systems_Own | 206-IL-010 | Ligand |
| CXCL12 | RnD_Systems_Own | 351-FS-010 | Ligand |
| NRG1 | RnD_Systems_Own | 378-SM | Ligand |
| IGFBP2 | RnD_Systems_Own | 674-B2-025 | Ligand |
| SHH | RnD_Systems_Own | 1314-SH-025 | Ligand |
| FASLG | RnD_Systems_Own | 126-FL-010 | Ligand |
Referanslar
- Hanahan, D., Coussens, L. M. Accessories to the crime: functions of cells recruited to the tumor microenvironment. Cancer Cell. 21 (3), 309-322 (2012).
- Quail, D. F., Joyce, J. A. Microenvironmental regulation of tumor progression and metastasis. Nature Medicine. 19 (11), 1423-1437 (2013).
- Hughes, C. S., Postovit, L. M., Lajoie, G. A. Matrigel: a complex protein mixture required for optimal growth of cell culture. Proteomics. 10 (9), 1886-1890 (2010).
- LaBarge, M. A., et al. Human mammary progenitor cell fate decisions are products of interactions with combinatorial microenvironments. Integrative Biology (Cambridge). 1 (1), 70-79 (2009).
- Watson, S. S., et al. Microenvironment-Mediated Mechanisms of Resistance to HER2 Inhibitors Differ between HER2+ Breast Cancer Subtypes. Cell Systems. 6 (3), 329-342 (2018).
- Ranga, A., et al. 3D niche microarrays for systems-level analyses of cell fate. Nature Communications. 5, 4324 (2014).
- Malta, D. F. B., et al. Extracellular matrix microarrays to study inductive signaling for endoderm specification. Acta Biomater. 34, 30-40 (2016).
- Kamentsky, L., et al. Improved structure, function and compatibility for CellProfiler: modular high-throughput image analysis software. Bioinformatics. 27 (8), 1179-1180 (2011).
- Gagnon-Bartsch, J. A., Jacob, L., Speed, T. P. Removing Unwanted Variation from High Dimensional Data with Negative Controls. University of California, Berkeley, Department of Statistics, University of California, Berkeley. , (2013).
- Allan, C., et al. OMERO: flexible, model-driven data management for experimental biology. Nature Methods. 9 (3), 245-253 (2012).
- Simian, M., Bissell, M. J. Organoids: A historical perspective of thinking in three dimensions. Journal of Cell Biology. 216 (1), 31-40 (2017).
- Bissell, M. J. The differentiated state of normal and malignant cells or how to define a "normal" cell in culture. International Review of Cytology. 70, 27-100 (1981).
- Serban, M. A., Prestwich, G. D. Modular extracellular matrices: solutions for the puzzle. Methods. 45 (1), 93-98 (2008).
- Kaylan, K. B., et al. Mapping lung tumor cell drug responses as a function of matrix context and genotype using cell microarrays. Integrative Biology (Cambridge). 8 (12), 1221-1231 (2016).
- Lin, C. H., Jokela, T., Gray, J., LaBarge, M. A. Combinatorial Microenvironments Impose a Continuum of Cellular Responses to a Single Pathway-Targeted Anti-cancer Compound. Cell Reports. 21 (2), 533-545 (2017).
- Gjorevski, N., et al. Designer matrices for intestinal stem cell and organoid culture. Nature. 539 (7630), 560-564 (2016).
Yeniden Basımlar ve İzinler
Bu JoVE makalesinin metnini veya resimlerini yeniden kullanma izni talebi
Izin talebiDaha Fazla Makale Keşfet
This article has been published
Video Coming Soon
JoVE Hakkında
Telif Hakkı © 2020 MyJove Corporation. Tüm hakları saklıdır