Method Article
Использование Microarrays для допроса микроenvironmentale влияние на клеточные фенотипы при раке
В этой статье
Резюме
Цель метода, представленного здесь, состоит в том, чтобы показать, как микроокружение microarrays (MEMA) могут быть изготовлены и использованы для допроса воздействия тысяч простых комбинаторных микросред на фенотип культивированных клеток.
Аннотация
Понимание влияния микроокружения на фенотип клеток является сложной проблемой из-за сложной смеси как растворимых факторов роста, так и матричных белков в микроокружении in vivo. Кроме того, легкодоступные реагенты для моделирования микросред в пробирке обычно используют сложные смеси белков, которые не полностью определены и страдают от пакетной к пакетной изменчивости. Платформа микроокружения microarray (MEMA) позволяет оценить тысячи простых комбинаций белков микросреды для их воздействия на клеточные фенотипы в одном анализе. MEMAs готовятся в колодцах, что позволяет добавление отдельных лигандов отделять скважины, содержащие массивные внеклеточные матричные белки (ECM). Сочетание растворимого лиганда с каждым печатным ECM образует уникальное сочетание. Типичный анализ MEMA содержит более 2500 уникальных комбинаторных микросред, что клетки подвергаются в одном анализе. В качестве тестового случая, линия клеток рака молочной железы MCF7 была покрыта на платформе MEMA. Анализ этого анализа выявил факторы, которые как усиливают и препятствуют росту и распространению этих клеток. Платформа MEMA является очень гибкой и может быть расширена для использования с другими биологическими вопросами, помимо исследований рака.
Введение
Культирование раковых клеток линий на пластике в двумерных (2D) монослой остается одним из основных рабочих лошадок для исследователей рака. Тем не менее, микросреда все больше признается за его способность влиять на клеточные фенотипы. При раке, микроокружение опухоли, как известно, влияет на несколько клеточного поведения, в том числе рост, выживание, вторжение, и ответ на терапию1,2. Традиционные монослойные клеточные культуры, как правило, не имеют влияния микроэкологии, что привело к разработке более сложных трехмерных (3D) анализов для выращивания клеток, в ключая коммерчески доступные очищенные экстракты мембраны подвала. Тем не менее, эти очищенные матрицы, как правило, сложны в использовании и страдают от технических проблем, таких как вариабельность пакетов3 и сложные композиции3. В результате, это может быть трудно назначить функцию конкретных белков, которые могут влиять на клеточные фенотипы3.
Для устранения этих ограничений мы разработали технологию микроокружения microarray (MEMA), которая сводит микроокружение к простым сочетаниям внеклеточной матрицы (ECM) и растворимых белков фактора роста4,5 . Платформа MEMA позволяет выявлять доминирующие микроэкологические факторы, влияющие на поведение клеток. Используя формат массива, тысячи комбинаций факторов микросреды могут быть рассмотрены в одном эксперименте. MEMA описал здесь допрашивает 2500 различных уникальных условий микросреды. Белки ECM, напечатанные в хорошо пластины образуют рост колодки, на которых клетки могут быть культивированы. Растворимые лиганды добавляются в отдельные скважины, создавая уникальные комбинаторные микросреды (ECM и ligand) на каждом различном месте, к которому подвергаются клетки. Клетки культивируются в течение нескольких дней, затем фиксируются, окрашиваются и образуются для оценки клеточных фенотипов в результате воздействия этих специфических комбинаций микросреды. Поскольку микросреда представляет собой простые комбинации, легко определить белки, которые управляют основными фенотипическими изменениями в клетках. MEMAs были успешно использованы для выявления факторов, которые влияют на несколько клеточных фенотипов, в том числе те, которые управляют решениями судьбы клеток и ответ на терапию4,5,6,7. Эти ответы могут быть проверены в простых 2D-экспериментов, а затем могут быть оценены в условиях, которые более полно резюмировать сложность микросреды опухоли. Платформа MEMA хорошо адаптируется к различным типам клеток и конечным точкам, при условии, что имеются хорошие фенотипические биомаркеры.
протокол
ПРИМЕЧАНИЕ: Обзор всего процесса MEMA, включая расчетное время, изложен на диаграмме потока, показанной на рисунке 1. В этом протоколе подробно описано изготовление MEMA в 8-колодцах. Протокол может быть адаптирован для других пластин или слайдов.
1. Подготовка белков, разбавитель, и окрашивания буферов
- Равновесие флаконов ЭКМ, лигандов и цитокинов к комнатной температуре (RT) и краткоцентрике. Добавьте соответствующий объем соответствующего буфера RT, указанного в листе данных о продукте. Следуйте рекомендациям производителя в расчете на концентрацию запасов.
ПРИМЕЧАНИЕ: Полный список лигандов и ЭКМ с их запасами и конечными концентрациями приведен в таблице 1 и таблице2. Оба лиганда и ЭКМ, как правило, используются при самой высокой концентрации диапазона, рекомендованных производителем, что вызывает биологический эффект в стандартных 2-дневный анализ культуры. Ручка белки мягко и в биобезопасности шкафы под ламинарный поток, чтобы избежать загрязнения. - Инкубировать флаконы с нежным раскачиванием на RT за 1 ч. Не вихревые белки, так как это может привести к их денатурации.
- Белки Aliquot для долгосрочного хранения так, что все aliquots одноразового использования только для того чтобы во избежание деградация с повторными циклами замораживания/оттепели. Храните лиофилизированные белки при -80 градусах по Цельсию (если не указано иное) до необходимости. Позаботьтесь о сборе всех метаданных для будущей ссылки, таких как: (i) название белка, (ii) дата подготовлена, (iii) лот/номер партии, (iv) поставщик, (v) номер каталога, (vi) концентрация, (vii) объем, и (viii) подготовителя.
- Приготовьте разбавительный буфер, содержащий 20% (v/v) глицерол, 10 мМ EDTA, 200 мМ Tris-HCl, pH 7.2, и фильтр стерилизовать. Держите этот буфер стерильным и храните на RT.
- Подготовка окрашивания буфера, содержащего 2% (w/v) BSA, 1 мМ MgCl2, и 0.02% NaN3 в фосфат-буферных солив (PBS). Фильтр и хранить при 4 градусах По Цельсию.
2. Подготовка ECM Исходная плита
- Удалите алицитные запасы белков ECM для печати и оттаивания на льду. Запись всех номеров лотов для отслеживания метаданных.
- Флик трубки оттаяли белков осторожно, чтобы обеспечить надлежащее повторное приостановление и спина вниз в центрифуге.
-
Сделать ECM печати смесей (EPMs) и флуоресцентные фидуциальное для использования жидким роботом обработки, который создаст рандомизированных 384-хорошо источник пластин.
ПРИМЕЧАНИЕ: 384-хорошо источник пластин ы будет использоваться сенсорным контактным массивом принтера для создания печатных массивов в 8 хорошо пластин.- Этикетка 1,5 мл микроцентрифуговых труб для каждого EPM и фидуциальной.
- Подготовьте каждый EPM, объединив 125 л разбавительного буфера (см. шаг 1.4) с соответствующим объемом eCM запаса и довести смесь до общего объема 250 Зл с PBS. Окончательные концентрации в каждой трубке EPM будут 1x eCM протеин, 5 mM EDTA, 10% glycerol, и 100 mM Tris.
- Подготовьте флуоресцентный фидуциал, растворив его в соответствующем буфере, указанном производителем, и перенесите 250 л на маркированную фидуциальную трубку.
3. Создание исходной плиты с использованием жидкого обработчика
- Дизайн 384-луп овойте, который рандомизирует позиции ECM и оптимизируется для используемой головки контактного кода принтера массива. Дизайн размещения фидуциальных так, что он будет напечатан в строке 1, столбец 1 положение каждого хорошо, чтобы помочь в ориентации массива.
ПРИМЕЧАНИЕ: Для обеспечения надежных данных используется в общей сложности 14–15 репликаций каждого ECM. Включите дополнительные репликации коллагена или другого ECM, который дает надежное вложение для оценки единообразия связывания. Макет, возможно, потребуется использовать несколько 384-колодец пластин в зависимости от количества ECMs интерес. - Передача EPM трубки жидких обработчик, сохраняя трубки на 4 кВ либо с охлажденный трубки стойки или с помощью жидкой обработки робота, расположенного в холодной комнате.
- Используя программное обеспечение обработчика жидкости, запустите программу для переноса 15 qL каждого EPM и фидуциальных к заранее назначенным скважинам в пределах 384-хорошей пластины источника (ы).
- Pipet PBS в любые неиспользованные скважины для повышения влажности и защиты от высыхания во время процесса печати.
ПРИМЕЧАНИЕ: См Рисунок 2 на примере 384-хорошо набор пластины источника, который оптимизирован для 4 х 7 контактный головка и включает в себя коллаген i блок и PBS. - Печать пластины (ы) и держать при 4 градусах По Цельсию до готовности к печати.
4. Печать MEMAs с использованием робота-печати array
ПРИМЕЧАНИЕ: Следующая часть протокола конкретно описывает подготовку и использование MEMA для исследования влияния различных белков микросреды на рост и распространение клеток MCF7. Тем не менее, протокол может быть легко адаптирован для использования различных лигандов, ECMs и клеток для изучения других клеточных линий и конечных точек интереса.
-
Использование сенсорного принтера, печать EPMs и фидуциальных пятен в 8 хорошо пластин. Печать нескольких репликаций каждого состояния ECM для обеспечения воспроизводимости.
ПРИМЕЧАНИЕ: Другие форматы пластин или слайды могут быть использованы для печати, но оптимизация буфера может потребоваться для достижения оптимального образования точек.- Печать ECMs для MEMA с использованием 350 мкм диаметром булавки расположены в 4 х 7 печати головы конфигурации. Печать массивов в 8-ну колодцев, как 20 столбцов на 35 строк, в общей сложности 700 пятен. Большие массивы возможны в этих пластинах, но поставляются с компромиссом увеличенных эффектов края как в связывании клеток и окрашивания.
- После печати храните тарелки в обезвраживаемом месте в течение как минимум 3 дней до использования.
5. Создание лигандских лечебных пластин
- Дизайн 96-ну хорошо планировки пластины в том числе лиганды интерес. Чтобы облегчить обработку многих пластин MEMA сразу, спроектируйте эту пластину с интервалом, что позволяет использовать многоканальный трубчатый с 4 расставленными наконечниками для передачи жидкостей между скважинами 8-хорошо MEMAs и 96-хорошей пластиной.
ПРИМЕЧАНИЕ: В этом протоколе используется полный набор лигандов, перечисленных в таблице 2. - Оттепель лиганды на льду. Кратко Флик и спина вниз каждой трубки.
- Разбавлять лиганды до 200-х рабочих запасов с помощью рекомендованный буфер производителя (обычно PBS).
- Пипер 10 л каждого 200-х лиганда в соответствующую скважину в пределах 96-колодца пластины.
- Печать и хранить тарелки при -20 градусах Цельсия.
ПРИМЕЧАНИЕ: Сделать лиганд лечения пластин в партиях, захватив все метаданные для анализа вниз по течению.
6. Культивирование ячеек на МЕМА
- Блок MEMAs в течение 20 минут с 2 мл на скважину не-загрязнения блокирующий буфер, содержащий 1% незагрязняющий блокирующий агент(Таблица материалов) в двойной дистиллированной воде (ddH2O).
- Аспир блокирующий буфер и тройной промывки скважин с помощью PBS. Для предотвращения высыхания, оставьте окончательный объем PBS в скважинах до готовности к клеточному покрытию.
ПРИМЕЧАНИЕ: Это чрезвычайно полезно иметь два скамейки работников для клеточной культуры шаги на MEMAs. Один работник скамейки может выполнять шаги аспирации, в то время как второй выполняет дополнительные шаги. Рекомендуется использовать 1 мл многоканального пайпета с наконечниками, расположенными в соответствии с 8-хорошо пластины для pipetting и Y-сплиттер с двумя пастерпицы, чтобы аспирировать несколько скважин одновременно. - Семена 2 х 105 клеток MCF7 на скважину в 2 мл модифицированной среды Орла Dulbecco (DMEM), содержащей 10% сыворотки крупного рогатого скота плода (FBS).
ПРИМЕЧАНИЕ: Перед полным экспериментом MEMA, выполнить эксперимент титрования клеток для оптимизации числа клеток, таким образом, что пятна MEMA имеют высокие номера клеток (но не конфлеятор) в конце желаемой экспериментальной продолжительности. - После 2–18 ч слякоть, аспирируйте среднюю и заменяйте 2 мл среды с пониженным ростом (DMEM с 0,1% FBS).
ПРИМЕЧАНИЕ: Снижение сыворотки (например, 0,1% FBS) или фактор роста истощенных условиях могут быть использованы в это время для изоляции стимулирующего воздействия конкретных лигандов. - Оттепель лигандовой лечебной пластины на льду. Центрифуга разморожена по 200 х г в течение 1 мин.
- Перенесите 200 л среды от каждого колодца в культурной тарелке к соответствующему колодцу в лечебной пластине. Pipet вверх и вниз, чтобы смешать объем лиганда со средним и передать эту смесь обратно в соответствующий хорошо в пластине MEMA.
- Слегка раскачивайтесь вручную и возвращайте пластины MEMA в инкубатор. Культура на протяжении всего эксперимента при наличии комбинации лиганд/Экм при 37 градусах Цельсия и 5% CO2.
ПРИМЕЧАНИЕ: Типичный эксперимент MEMA длится 72 ч; более длительные эксперименты могут потребовать замены средней и повторной обработки лигандой. - Пульс овсяные скважины MEMA при 71 ч со 100х 5-этинил-2'-деоксюридин (EdU) для окончательной концентрации 10 мкм. Инкубируйтев экспериментальных условиях с EdU для 1 ч при 37 C и 5% CO 2.
ПРИМЕЧАНИЕ: Другие живые клетки лечения также могут быть использованы в это время.
7. Фиксация и окрашивание МЕМА
- После 72 ч и любых живых клеточных процедур, аспирных колодцев. Исправить MEMAs в 2 мл на скважину 2% параформальдегида (PFA) в течение 15 минут на RT.
- Аспир ПФА. Пермяки с 2 мл на скважину 0,1% неионического сурфактанта в течение 15 мин.
- Аспирируй неионический сурфактант и мыть с 2 мл на скважину PBS. Аспирируй PBS. Мытье с 2 мл PBS с 0,05% полисорбат 20 (PBS-T).
ПРИМЕЧАНИЕ: Поверхность MEMA гидрофобна, и отказ мыть с PBS-T до инкубации пятна и антитела приведет к образованию пустот в скважинах во время инкубационных шагов и приведет к окрашиванию артефактов. - Аспират PBS-T. Добавьте реагенты реакции обнаружения EdU. Инкубировать в течение 1 ч на RT, качаясь и защищены от света. После 1 ч инкубации, утолить реакцию с предусмотренным коммерческим буфером утоления.
ПРИМЕЧАНИЕ: EdU обнаружения и окрашивания / антитела шаги могут быть выполнены в 1,5 мл на хорошо, чтобы уменьшить стоимость. - Призадыхать утолить буфер и промыть PBS-T до инкубации с пятнами или антителами.
- Инкубировать MEMA скважины с антителами против гистона H3K9me3 (1:1,000) и фибрилларин (1:400) в окрашивание буфера, содержащего 2% (w/v) бычьей сыворотки альбумина (BSA), 1 мМ MgCl2 и 0.02% NaN3 ночь на 4 кв.
ПРИМЕЧАНИЕ: Выполните титрования антител, чтобы определить оптимальные концентрации до их использования на полном наборе MEMA. - После первичного антитела или пятно инкубации, мыть колодцы 2x с PBS и один раз с PBS-T.
- Добавьте вторичные антитела (осел анти-мышь IgG и осел анти-кролик IgG, как 1:300) и 0,5 мкг /мл 4 "6-диамидино-2-фенилинзол (DAPI). Инкубировать 1 ч на RT в темноте.
- Вымойте скважины 2x с 2 мл на скважину PBS, оставляя их в финале 2 мл PBS.
- Приступить к визуализации или хранить окрашенные MEMAs для последующего изображения в PBS при 4 градусах по Цельсию, защищенных от света.
8. Изображение МЕМА
- Изображение MEMA на автоматизированной системе визуализации с соответствующими каналами флуоресцентного обнаружения.
- Вывод данных изображений в систему управления изображениями. Сегментные клетки и вычислить уровни интенсивности с помощью CellProfiler8.
9. Анализ данных
ПРИМЕЧАНИЕ: Анализ данных состоит из нормализации, коррекции изменений и обобщения необработанных данных CellProfiler. В этом случае R-среда с пользовательским кодом используется для выполнения всех шагов. Тем не менее, любая статистическая среда или программа программного обеспечения могут быть использованы для выполнения эквивалентных действий. Пример пользовательского кода с открытым исходным кодом для среды R для анализа доступен по адресу: https://www.synapse.org/#!Synapse:syn2862345/wiki/72486.
- Предварительное обработка и нормализация сегментированных данных изображений.
- Определите количество точечных клеток с помощью окрашенных ядер DAPI.
- Авто-ворота EdU интенсивность для обозначения клеток, как EdU. Измерьте пролиферацию, используя пропорцию клеток EdUв каждом месте.
- Медианные суммируют цитоплазматические пятна и измерения ядерной морфологии на спот-уровне.
- Выполните удаление нежелательных изменений (RUV) нормализации на данных для улучшения качества данных9.
ПРИМЕЧАНИЕ: Этот подход применяется к каждой интенсивности и морфологии сигнал атебов независимо как матрица с массивами, использующими строки и пятна, как столбцы, как описано ранее9. - Применить нормализацию двухвариатной лэсск к нормализованным остаткам RUV, используя строку массива и столбец массива в качестве независимых переменных для коррекции пространственных или связанных с интенсивностью эффектов.
- Как только нормализация завершена, медиана суммирует репликации для каждого состояния микросреды для отчетности и дальнейшего анализа.
Результаты
Для упрощения воздействия микроenvironmentalов на рост и пролиферацию клеток и выявления условий, способствующих или препятствуя росту и пролиферации клеток, линия клеток рака молочной железы MCF7 была посеяна на комплекте из восьми 8-ну хорошо MEMAs, как описано в протоколе. Этот анализ подвергал клетки 48 различным ЭКМ и 57 различным лигандам, в общей сложности 2736 комбинаторных микроэкологических условий. После 71 ч в культуре, клетки были импульсивные с EdU, фиксированные, permeablized, и окрашенные DAPI, реакция на обнаружение EdU, анти-фибриллин антитела, и анти-H3K9me3 антитела. Клетки были изображены на микроскопе с высоким содержанием. Изображения были загружены на сервер Omero10, сегментированные с помощью CellProfiler8, и нормализованы и проанализированы в R9. Результаты, описанные ниже, сосредоточены на сигналах DAPI и EdU.
Платформа анализа изображений MEMAs дает некоторые результаты, аналогичные тем, которые доступны из подходов цитометрии потока, таких как участки содержания ДНК, показывающие 2N и 4N фракции для клеток, обработанных данным лигандой (Рисунок 3A), на основе DAPI интенсивности и площади. Эти участки предоставляют доказательства условий, которые способствуют активному циклу клеток по сравнению с обозначенными четкими бимодальными пиками, соответствующими ячейкам в фазах G1 или G2 против арестованных клеток роста, которые показали бы изменения в пиках по сравнению с условиями контроля. Мы используем номер ячейки и данные интенсивности окрашивания для обобщения данных, где влияние микроокружения (лиганды на одну ось, ECM на второй оси) на обоих сотовых номера(рисунок 3B) и EdU включения (Рисунок 3C) может быть более легко рассматривать как изменения в цвете тепловой карты и интенсивности. Как видно из этих участков, многие эффекты лиганд-управляемых, как условие ECM не сильно влияние номер ячейки или EdU положительности. Нидоген-1 является явным исключением, так как наличие этой молекулы ECM подавляет связывание клеток и рост MCF7. Лиганды, такие как FGF6 и NRG1 (NRG1.1 на участках) повышают число клеток и имеют высокие показатели регистрации EdU, в то время как лиганды, такие как AREG и NRG1-smdf (NRG1.10 на участках), препятствуют связыванию клеток и/или росту клеток. Эти выводы подтверждаются изображениями клеток, растущих на пятнах, где очевидна разница в количестве клеток и положительности EdU (см. пример на рисунке 3D).
Поскольку платформа MEMA является новой технологией, результаты были проверены в отдельных анализах. Клетки MCF7 были посеяны в 24-колодские пластины, покрытые коллагеном I в среде DMEM с 10% FBS. После 18 ч, средства массовой информации были обменяны на снижение среды роста (DMEM с 0,1% FBS) и клетки были обработаны NRG1 ", FGF6, или AREG и культивируется для 72 ч. EdU был добавлен 1 ч до фиксации. Клетки были окрашены DAPI и для edU включения, изображения, сегментированы и проанализированы. Подобно результатам, полученным с платформы MEMA, FGF6 и NRG1' оба привели к более высоким числам клеток(Рисунок 4A) и EdU инкорпорации ставки (Рисунок 4B) по сравнению с AREG обработанных клеток, проверки наших наблюдений в оригинальные эксперименты MEMA.

Рисунок 1 диаграмма потока, показывающая рабочий процесс и сроки для различных фаз типичного эксперимента MEMA. После того, как MEMAs печатаются, они могут храниться при комнатной температуре высохли в течение нескольких месяцев до использования. Как правило, экспериментальная фаза длится от 3 до 4 дней, но некоторые медленно растущие первичные клетки культивируются на MEMA в течение 2 недель. Пожалуйста, нажмите здесь, чтобы просмотреть большую версию этой цифры.

Рисунок 2 : макет исходной пластины ECM для печати массива. Блок коллагена печатается на MEMA как сетка, которая обеспечивает весьма повторяющийся набор условий, которые позволяют более надежную нормализацию между скважинами. Заполненные PBS скважины обеспечивают влажность, чтобы помочь в предотвращении испарения во время процесса печати. Пожалуйста, нажмите здесь, чтобы просмотреть большую версию этой цифры.

Рисунок 3 : Примеры данных, полученных в результате типичного эксперимента MEMA. (A) Профили цикла клетки binned значений интенсивности DAPI против отсчетов клетки от одного 8-наилучшим образом плиты обработанных с по-разному ligands, показывая biphasic интенсивность DAPI окрашивая показывая клетки в участке цикла G1 против участка цикла клетки G2. (B) Тепловая карта, показывающая нормализованные количество точечных ячеек, сгруппированных по сходству с помощью иерархической кластеризации. Красный цвет указывает на более высокое число ячеек, а синий — на более низкое число ячеек. Лиганды находятся на оси x, ЭКМ - на оси y. (C) Heatmap, показывающий нормализованную инкорпорацию EdU, с красным цветом, указывающим более высокий и синий указывающий более низкий incorporation EdU. Лиганды находятся на оси x, ЭКМ - на оси y. (D) Пример MCF7 клеток, растущих на месте MEMA, обработанных NRG1-я, показывающие высокие показатели регистрации EdU (розовые ядра). Зеленое пятно - это клеточная маска, а синий - DAPI. Пожалуйста, нажмите здесь, чтобы просмотреть большую версию этой цифры.
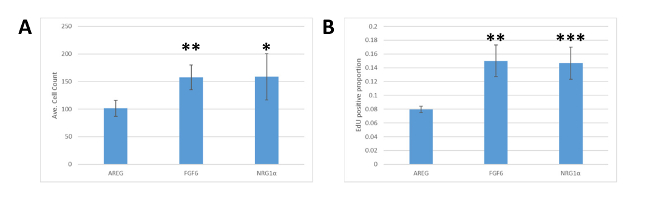
Рисунок 4 : Проверка MEMA приводит к культуре клеток. (A) Количественная оценка номера клеток в результате лечения MCF7 с различными лигандами. Эквивалентные числа клеток MCF7 были покрыты мультистенными пластинами, а затем обработаны либо AREG, FGF6, или NRG1. Скважины, обработанные AREG, имели значительно меньше клеток, чем те, которые лечились FGF6(с указанием значения t-test p-значения студента менее 0,01) или NRG1 » (-указывает на p-значение 0.05) при 72 h после лиганд лечения. (B) Количественная оценка уровня включения EdU в MCF7 из-за лечения различными лигандами, как в панели A. Лечение AREG приводит к значительно меньшей доле клеток, включающих EdU, чем клетки, обработанные FGF6(яп. Бары ошибок представляют собой стандартное отклонение. Пожалуйста, нажмите здесь, чтобы просмотреть большую версию этой цифры.
| Название белка | Uniprot ID | Фондовая Концентрации (мкг/мл) | Окончательный Концентрации (мкг/мл) |
| ANGPT1-1 | No15389-1 | 100 | 0,04 |
| ANGPT2-1 | O15123-1 | 100 | 0,2 |
| Арег | P15514 | 100 | 0,02 |
| BMP2 | P12643 | 100 | 0,1 |
| BMP3 | P12645 | 1000 г. | 0,1 |
| BMP4 | P12644 | 100 | 0,1 |
| БМП5-1 | P22003-1 | 100 | 0,1 |
| BMP6 | P22004 | 100 | 0,1 |
| БМП7 | P18075 | 100 | 0,1 |
| CSF2 | P04141 | 100 | 0,02 |
| CTGF-1 | P29279-1 | 100 | 0,05 |
| CXCL12 Альфа | P48061-2 | 100 | 0,01 |
| CXCL12 Бета-версия | P48061-1 | 100 | 0,03 |
| CXCL1 | P09341 | 100 | 0,004 |
| CXCL8'1 | P10145-1 | 100 | 0,3 |
| DLL1'1 | O00548'1 | 500 | 0,5 |
| DLL4 | No9NR61 | 200 г. | 0,6 |
| EGF-1 | P01133-1 | 500 | 0,01 |
| ФАСЛГ-1 | P48023-1 | 10 Лет | 0,02 |
| FGF2-3 | P09038-2 | 100 | 0,01 |
| FGF6 | P10767 | 100 | 0,01 |
| FLT3LG-1 | P49771-1 | 50 лет | 0,001 |
| ГПНМБ-1 | No14956-1 | 100 | 0,5 |
| HGF-1 | P14210-1 | 50 лет | 0,04 |
| IGF1-1 | P05019-1 | 200 г. | 0,01 |
| IGFBP2 | P18065 | 100 | 0,05 |
| IGFBP3'1 | P17936-1 | 100 | 0,1 |
| IL13 | P35225 | 100 | 0,01 |
| IL15 IL15S48AA | P40933-1 | 50 лет | 0,01 |
| IL1B | P01584 | 25 | 0,001 |
| IL6 | P05231 | 100 | 0,01 |
| IL7-1 | P13232-1 | 100 | 0,01 |
| JAG1'1 | P78504-1 | 200 г. | 0,5 |
| JAG2 Длинные | No9Y219-1 | 100 | 0,5 |
| КИТЛГ-1 | P21583-1 | 100 | 0,005 |
| КНГ1З HMW | P01042-1 | 100 | 0,2 |
| Lep | P41159 | 1000 г. | 0,002 |
| LYVE1 | 9Y5Y7 | 100 | 0,05 |
| NRG1-10 | 02297-10 | 100 | 0,01 |
| NRG1-1 | No02297-1 | 100 | 0,05 |
| NRG1-6 | No02297-6 | 100 | 0,01 |
| PDGFAB | go1990265 | 100 | 0,05 |
| PDGFB-1 | P01127-1 | 100 | 0,05 |
| Ptn | P21246 | 100 | 0,5 |
| Тсс | No15465 | 100 | 0,5 |
| TGFB1» Ктермининус | P01137 Ктермининус | 20 | 0,01 |
| TGFB1» Коленях | P01137 Коленях | 100 | 0,15 |
| TGFB2 A | P61812-1 | 20 | 0,01 |
| ТПО1 | P40225-1 | 50 лет | 0,002 |
| TNFRSF11B | O00300 | 100 | 0,02 |
| TNFSF11 | O14788-1 | 100 | 0,01 |
| Фно | P01375 | 100 | 0,01 |
| VEGFA VEGF206 | P15692-1 | 100 | 0,01 |
| WNT10A | No9ГЗТ5 | 100 | 0,1 |
| WNT3A-1 | P56704-1 | 200 г. | 0,1 |
| Wnt5a'1 | P22725-1 | 100 | 0,1 |
Таблица 1: Полный список лигандов, используемых для экспериментов MEMA. Предоставляется uniprot ID, концентрации запасов и конечная рабочая концентрация.
| ECM Белок | Единороссы | Концентрация запасов (мкг/мл) | Окончательный Концентрации (мкг/мл) | Заметки |
| АЛКАМ-1 | No13740-1 | 100 | 30 год | |
| CDH20 | No9HBT6 | 300 г. | 80 | |
| CDH6'1 | P55285-1 | 100 | 40 г. | |
| CDH8 | P55286 | 100 | 20 | |
| CD44-1 | P16070-1 | 100 | 30 год | |
| CEACAM6 | P40199 | 100 | 30 год | |
| COL1A1 | P02453 | 5000 | 200 г. | несколько подразделений с несколькими идентификаторами uniprot |
| COL2A1-2 | P02458-2 | 1000 г. | 200 г. | |
| COL3A1-1 | P02461-1 | 1000 г. | 200 г. | |
| COL4A1-1 | P02462-1 | 1000 г. | 200 г. | несколько подразделений с несколькими идентификаторами uniprot |
| COL5A1 | P20908 | 1000 г. | 200 г. | |
| COL23A1-1 | No86Y22-1 | 200 г. | 80 | |
| DSG2 | No14126 | 100 | 30 год | |
| CDH1'1 | P12830-1 | 100 | 40 г. | |
| ECM1'1 | No16610-1 | 100 | 40 г. | |
| FN1-1 | P02751-1 | 1000 г. | 200 г. | |
| GAP43'1 | P17677-1 | 158 г. | 40 г. | |
| HyA-500K | 1000 г. | 200 г. | ЛОР-0005 | |
| HyA-50K | 1000 г. | 200 г. | ЛОР-0007 | |
| ICAM1 | P05362 | 400 | 80 | |
| АЛКАМ-1 | No13740-1 | 100 | 30 год | |
| CDH20 | No9HBT6 | 300 г. | 80 | |
| CDH8 | P55286 | 100 | 20 | |
| CD44-1 | P16070-1 | 100 | 30 год | |
| CEACAM6 | P40199 | 100 | 30 год | |
| DSG2 | No14126 | 100 | 30 год | |
| CDH15 | P55291 | 100 | 20 | |
| VCAM1'1 | P19320-1 | 1000 г. | 200 г. | |
| LAMA1 | P25391 | 500 | 200 г. | несколько подразделений с несколькими идентификаторами uniprot |
| ЛАМА3-2 | No16787-2 | 130 г. | 40 г. | |
| Lum | P51884 | 200 г. | 80 | |
| CDH15 | P55291 | 100 | 20 | |
| NID1'1 | P14543-1 | 100 | 9,3 мкг/мл Нид, 130 мкг/мл Лам, 46,5 мкг/мл COL4 | ЗКОЛ4 и ламинин |
| Omd | No99983 | 100 | 40 г. | |
| SPP1 A | P10451-1 | 100 | 40 г. | |
| CDH3'1 | P22223-1 | 100 | 40 г. | |
| ПЕКАМ1З Длинные | P16284-1 | 150 г. | 40 г. | |
| ТНК-1 | P24821-1 | 500 | 200 г. | |
| VCAM1'1 | P19320-1 | 1000 г. | 200 г. | |
| Vtn | P04004 | 100 | 40 г. | |
| Bgn | P21810 | 100 | 40 г. | |
| ДКНЗ A | P07585-1 | 300 г. | 80 | |
| ПОСТН-1 | No15063-1 | 100 | 40 г. | |
| Sparc | P09486 | 100 | 40 г. | |
| THBS1'1 | P07996-1 | 100 | 40 г. | |
| BCAN-1 | No96GW7-1 | 100 | 40 г. | |
| АНО-3 | P15502-3 | 1000 г. | 200 г. | |
| FBN1 | P35555 | 254 г. | 80 |
Таблица 2: Полный список белков ECM и условий, которые используются в экспериментах MEMA. Предоставляется uniprot ID, концентрации запасов и конечная рабочая концентрация. В некоторых случаях печатное состояние представляет собой белковый комплекс или комбинацию нескольких белков, что указано в колонке Notes.
Обсуждение
Важность "мерности" и контекста был мотивирующее фактор в развитии систем культуры in vitro в качестве инструментов в характеристике раковых клеток через их взаимодействие с микросредой11, и способность in vitro культурные системы для имитации среды in vivo является движущей силой стремления улучшить эти культурные системы. In vitro системы, однако, остаются важными инструментами исследования рака именно из-за их способности перегонять сложные in vivo ситуации вплоть до упрощенной модели12.
Хотя 2D-системы могут включать ЭКМ и лиганды, они традиционно не имеют пропускной способности для допроса широкой панели комбинаторных пертубагенов. Популярные коммерческие экстракты мембраны подвала позволяют культивировать в 3D, но не хватает происхождения тщательно определенной панели белков. Коммерческие экстракты обычно страдают от неполно определенного состава, что может заставить засомниться анализ и привести к значительному варианту партии к партии3,13. Платформа MEMA преодолевает эти барьеры, что позволяет изучать изменения в клеточных фенотипах, метаболической активности, статусе дифференциации и вариациях роста и пролиферации клеток, поскольку они модулируются конкретными и определенными эндогенными Факторов.
Платформа MEMA представляет собой мощный, средне- и высокопроизводительный подход для оценки влияния микросреды (как ECM, так и растворимых факторов) на фенотип клеток. Платформа показывает большую гибкость для типов анализов и ячеек, для которых он может быть использован. Мы можем наблюдать эффекты как растворимых лигандов, так и белков ECM, которым подвергаются клетки. Действительно, мы недавно обнаружили, что лиганды были основным фактором сопротивления HER2-целевых ингибиторов, но что эти эффекты могут быть модулированы ECM5. Разнообразие клеток, включая первичные клетки и клеточные линии, полученные из различных типов клеток, включая легкие, мочевой пузырь, простату, грудь и поджелудочную железу, а также индуцированные плюрипотентные стволовые клетки (iPS), были успешно культивированы на платформе MEMA (см. примеры вссылках 5,7,14). Использование различных пятен позволяет считывать несколько клеточных конечных точек, включая рост клеток, дифференциацию и обмен веществ. Другие исследователи расширили платформу, чтобы допросить влияние жесткости или упругого модуля, добавив дополнительное измерение к платформе MEMA15. Наконец, платформа поддается выполнению наркотиков экраны для идентификации микроэкологических условий, которые либо повышения или ингибировать эффективность препарата, как мы и другие недавно сообщили5,14,15 .
Возможно, наиболее важным шагом к успеху эксперимента MEMA является оптимизация плотности клеточного покрытия. Оптимизация плотности ячеек гарантирует наличие достаточного количества ячеек для предоставления надежных данных, но не настолько многих, что пятно становится слишком сливовым. Слияние пятен может значительно затруднить результаты, особенно если распространение используется в качестве конечной точки, что делает невозможным определить, являются ли низкие показатели распространения результатом взаимодействия с микрофакторами или из-за контакта высокой плотностью клеток. Эксперименты по титроции клеток могут выявить эти проблемы, так как средние цифры клеток на место продемонстрируют линейное увеличение с увеличением числа клеток, покрытых, но в конечном итоге плато. Оптимальный номер ячейки должен быть выбран в линейном диапазоне кривой.
Как уже упоминалось выше, платформа MEMA является гибкой и может быть подготовлена на различных субстратов с различными поверхностями. К ним относятся стеклянные слайды и многостенные форматы пластин. По нашему опыту, не все поверхностные химии поддаются печати MEMA, так как мы наблюдали точечный отряд на некоторых поверхностях из-за плохих свойств стыковки и невозможности блокировать сливки клеток на других поверхностях высокой адгезии. Кроме того, изменение между различными субстратами требует оптимизации условий буфера, так как производительность печати с одним и тем же буфером печати может варьироваться в зависимости от химии поверхности.
Диаметр печатных eCM пятна играет важную роль в качестве данных. В целом, мы рекомендуем использовать самые большие штыри печати диаметра, доступные для массивителя в использовании (в настоящее время мы используем 350 мкм диаметр контактов). Пятна большего диаметра позволяют большему числу клеток занимать пятно, что приводит к более надежным данным, чем генерируются с прыгним меньшего диаметра. Поскольку связывание клеток является стохастический процесс, там, как правило, высокая степень изменчивости в данных, что связано с числом клеток, первоначально прикрепленных к каждому месту. Таким образом, мы рекомендуем печатать большое количество репликантов для каждого состояния ECM. Мы печатаем репликации ECM в каждой скважине с нашими текущими условиями печати, чтобы обеспечить надежную статистику.
Мы отмечали в наших прошлых экспериментах, что по большей части, лиганд эффекты, как правило, доминируют над ECM эффекты. Это может быть отчасти связано с нашим решением добавить коллаген I ко всем пятнам ECM, что обеспечивает надежную связывание клеток. Тем не менее, мы считаем, что это может также гомогенизовать eCM эффекты, так как большинство пятен, как правило, ведут себя таким образом, очень похож на коллаген I. Изменение точечного состава, чтобы исключить коллагена я могу привести к дифференциальной клеточного поведения в результате взаимодействия с ECM, но и значительно влияет на связывание клеток, в результате чего гораздо больше незанятых мест. Пользователи должны адаптировать их ECM состав с учетом этих различий, особенно тех, кто заинтересован в стволовых и прародителей клеток и дифференциации, где матрица может иметь значительное влияние16.
Обычно мы выполняем анализы MEMA в течение относительно коротких периодов времени (например, максимум 72 ч). Это потому, что клетки ограничены пятнами (блокирующий буфер не позволяет роста за пределами пятен в нашем опыте). С быстро делением клеток, рост дольше, чем 72 ч приведет к разрастанию пятна, что, в свою очередь, усложняет сегментации изображения, как клетки становятся переполненными и накапливаются друг на друга, а также может повлиять на данные, как рост арест может произойти с торможением контакта. Мы проводили более длительные процедуры с очень медленно растущими первичными клетками (10–14 дней), но в этих анализах необходимо проявлять осторожность, чтобы изменить медиа- и пополнить лиганды каждые 3–4 дня.
Продолжающиеся усилия по разработке платформы MEMA сосредоточены на двух областях, представляющих интерес, максимизации оптического качества для визуализации и оптимизации в небольших культурных сосудах. Оптическое качество становится решающим фактором, когда исследователям требуется микроскопия с более высоким разрешением для определения субклеточной локализации их маркеров интереса. Первоначальные экраны могут быть выполнены с более низким разрешением на высокопроизводительных микроскопах с последующим изображением конкретных пятен, представляющих интерес на приборах с более высоким разрешением, но качество изображения может быть нарушено, если оптические свойства субстрата являются плохими. Улучшение оптических свойств субстрата позволит исследователям выполнять первоначальные экраны на системах визуализации с высоким разрешением без необходимости повторного получения выбранных изображений с более высоким разрешением. Наконец, способность выполнять МЕМА в небольших сосудах культуры, таких как 96-колодцы, позволит сократить объем обработки и расширение допрошенных лигандов и репликаций. Этот переход требует оптимизации взаимодействий субстрата-буфера-белка и печати массивов в новых сосудах культуры. Такие постоянные усилия улучшат платформу MEMA и расширят ее мощные возможности для выявления соответствующих микроэкологических белков, которые изменяют клеточные фенотипы для различных типов клеток, которые затем могут быть впоследствии исследованы в подтверждающие анализы.
Раскрытие информации
Авторам нечего раскрывать.
Благодарности
Эта работа была поддержана NIH Общий фонд Библиотека сетевых сотовых подписей (LINCS) грант HG008100 (J.W.G., L.M.H., и J.E.K.).
Материалы
| Name | Company | Catalog Number | Comments |
| Aushon 2470 | Aushon BioSystems | Arrayer robot system used in the protocol | |
| Nikon HCA | Nikon | High Content Imaging system designed around Nikon Eclipse Ti Inverted Microscope | |
| BioTek Precision XS liquid Handler | BioTek | liquid handling robot used in the protocol | |
| Trizma hydrochloride buffer solution | Sigma | T2069 | |
| EDTA | Invitrogen | 15575-038 | |
| Glycerol | Sigma | G5516 | |
| Triton X100 | Sigma | T9284 | |
| Tween 20 | Sigma | P7949 | |
| Kolliphor P338 | BASF | 50424591 | |
| 384-well microarray plate, cylindrical well | Thermo Fisher | ab1055 | |
| Nunc 8 well dish | Thermo Fisher | 267062 | |
| Paraformaldehyde 16% solution | Electron Microscopy Science | 15710 | |
| BSA | Fisher | BP-1600 | |
| Sodium Azide | Sigma | S2002 | |
| Cell Mask | Molecular Probes | H32713 | |
| Click-iTEdU Alexa Fluor | Molecular Probes | C10357 | |
| DAPI | Promo Kine | PK-CA70740043 | |
| ALCAM | R & D Systems | 656-AL | ECM |
| Cadherin-20 (CDH20) | R & D Systems | 5604-CA | ECM |
| Cadherin-6 (CDH6) | R & D Systems | 2715-CA | ECM |
| Cadherin-8 (CDH8) | R & D Systems | 188-C8 | ECM |
| CD44 | R & D Systems | 3660-CD | ECM |
| CEACAM6 | R & D Systems | 3934-CM | ECM |
| Collagen I | Cultrex | 3442-050-01 | ECM |
| Collagen Type II | Millipore | CC052 | ECM |
| Collagen Type III | Millipore | CC054 | ECM |
| Collagen Type IV | Sigma | C5533 | ECM |
| Collagen Type V | Millipore | CC077 | ECM |
| COL23A1 | R & D Systems | 4165-CL | ECM |
| Desmoglein 2 | R & D Systems | 947-DM | ECM |
| E-cadherin (CDH1) | R & D Systems | 648-EC | ECM |
| ECM1 | R & D Systems | 3937-EC | ECM |
| Fibronectin | R & D Systems | 1918-FN | ECM |
| GAP43 | Abcam | ab114188 | ECM |
| HyA-500K | R & D Systems | GLR002 | ECM |
| HyA-50K | R & D Systems | GLR001 | ECM |
| ICAM-1 | R & D Systems | 720-IC | ECM |
| Laminin | Sigma | L6274 | ECM |
| Laminin-5 | Abcam | ab42326 | ECM |
| Lumican | R & D Systems | 2846-LU | ECM |
| M-Cad (CDH15) | R & D Systems | 4096-MC | ECM |
| Nidogen-1 | R & D Systems | 2570-ND | ECM |
| Osteoadherin/OSAD | R & D Systems | 2884-AD | ECM |
| Osteopontin (SPP) | R & D Systems | 1433-OP | ECM |
| P-Cadherin (CDH3) | R & D Systems | 861-PC | ECM |
| PECAM1 | R & D Systems | ADP6 | ECM |
| Tenascin C | R & D Systems | 3358-TC | ECM |
| VCAM1 | R & D Systems | ADP5 | ECM |
| Vitronectin | R & D Systems | 2308-VN | ECM |
| Biglycan | R & D Systems | 2667-CM | ECM |
| Decorin | R & D Systems | 143-DE | ECM |
| Periostin | R & D Systems | 3548-F2 | ECM |
| SPARC/osteonectin | R & D Systems | 941-SP | ECM |
| Thrombospondin-1/2 | R & D Systems | 3074-TH | ECM |
| Brevican | R & D Systems | 4009-BC | ECM |
| Elastin | BioMatrix | 5052 | ECM |
| Fibrillin | Lynn Sakai Lab OHSU | N/A | ECM |
| ANGPT2 | RnD_Systems_Own | 623-AN-025 | Ligand |
| IL1B | RnD_Systems_Own | 201-LB-005 | Ligand |
| CXCL8 | RnD_Systems_Own | 208-IL-010 | Ligand |
| IGF1 | RnD_Systems_Own | 291-G1-200 | Ligand |
| TNFRSF11B | RnD_Systems_Own | 185-OS | Ligand |
| BMP6 | RnD_Systems_Own | 507-BP-020 | Ligand |
| FLT3LG | RnD_Systems_Own | 308-FK-005 | Ligand |
| CXCL1 | RnD_Systems_Own | 275-GR-010 | Ligand |
| DLL4 | RnD_Systems_Own | 1506-D4-050 | Ligand |
| HGF | RnD_Systems_Own | 294-HGN-005 | Ligand |
| Wnt5a | RnD_Systems_Own | 645-WN-010 | Ligand |
| CTGF | Life_Technologies_Own | PHG0286 | Ligand |
| LEP | RnD_Systems_Own | 398-LP-01M | Ligand |
| FGF2 | Sigma_Aldrich_Own | SRP4037-50UG | Ligand |
| FGF6 | RnD_Systems_Own | 238-F6 | Ligand |
| IL7 | RnD_Systems_Own | 207-IL-005 | Ligand |
| TGFB1 | RnD_Systems_Own | 246-LP-025 | Ligand |
| PDGFB | RnD_Systems_Own | 220-BB-010 | Ligand |
| WNT10A | Genemed_Own | 90009 | Ligand |
| PTN | RnD_Systems_Own | 252-PL-050 | Ligand |
| BMP3 | RnD_Systems_Own | 113-BP-100 | Ligand |
| BMP4 | RnD_Systems_Own | 314-BP-010 | Ligand |
| TNFSF11 | RnD_Systems_Own | 390-TN-010 | Ligand |
| CSF2 | RnD_Systems_Own | 215-GM-010 | Ligand |
| BMP5 | RnD_Systems_Own | 615-BMC-020 | Ligand |
| DLL1 | RnD_Systems_Own | 1818-DL-050 | Ligand |
| NRG1 | RnD_Systems_Own | 296-HR-050 | Ligand |
| KNG1 | RnD_Systems_Own | 1569-PI-010 | Ligand |
| GPNMB | RnD_Systems_Own | 2550-AC-050 | Ligand |
| CXCL12 | RnD_Systems_Own | 350-NS-010 | Ligand |
| IL15 | RnD_Systems_Own | 247-ILB-005 | Ligand |
| TNF | RnD_Systems_Own | 210-TA-020 | Ligand |
| IGFBP3 | RnD_Systems_Own | 675-B3-025 | Ligand |
| WNT3A | RnD_Systems_Own | 5036-WNP-010 | Ligand |
| PDGFAB | RnD_Systems_Own | 222-AB | Ligand |
| AREG | RnD_Systems_Own | 262-AR-100 | Ligand |
| JAG1 | RnD_Systems_Own | 1277-JG-050 | Ligand |
| BMP7 | RnD_Systems_Own | 354-BP-010 | Ligand |
| TGFB2 | RnD_Systems_Own | 302-B2-010 | Ligand |
| VEGFA | RnD_Systems_Own | 293-VE-010 | Ligand |
| IL6 | RnD_Systems_Own | 206-IL-010 | Ligand |
| CXCL12 | RnD_Systems_Own | 351-FS-010 | Ligand |
| NRG1 | RnD_Systems_Own | 378-SM | Ligand |
| IGFBP2 | RnD_Systems_Own | 674-B2-025 | Ligand |
| SHH | RnD_Systems_Own | 1314-SH-025 | Ligand |
| FASLG | RnD_Systems_Own | 126-FL-010 | Ligand |
Ссылки
- Hanahan, D., Coussens, L. M. Accessories to the crime: functions of cells recruited to the tumor microenvironment. Cancer Cell. 21 (3), 309-322 (2012).
- Quail, D. F., Joyce, J. A. Microenvironmental regulation of tumor progression and metastasis. Nature Medicine. 19 (11), 1423-1437 (2013).
- Hughes, C. S., Postovit, L. M., Lajoie, G. A. Matrigel: a complex protein mixture required for optimal growth of cell culture. Proteomics. 10 (9), 1886-1890 (2010).
- LaBarge, M. A., et al. Human mammary progenitor cell fate decisions are products of interactions with combinatorial microenvironments. Integrative Biology (Cambridge). 1 (1), 70-79 (2009).
- Watson, S. S., et al. Microenvironment-Mediated Mechanisms of Resistance to HER2 Inhibitors Differ between HER2+ Breast Cancer Subtypes. Cell Systems. 6 (3), 329-342 (2018).
- Ranga, A., et al. 3D niche microarrays for systems-level analyses of cell fate. Nature Communications. 5, 4324 (2014).
- Malta, D. F. B., et al. Extracellular matrix microarrays to study inductive signaling for endoderm specification. Acta Biomater. 34, 30-40 (2016).
- Kamentsky, L., et al. Improved structure, function and compatibility for CellProfiler: modular high-throughput image analysis software. Bioinformatics. 27 (8), 1179-1180 (2011).
- Gagnon-Bartsch, J. A., Jacob, L., Speed, T. P. Removing Unwanted Variation from High Dimensional Data with Negative Controls. University of California, Berkeley, Department of Statistics, University of California, Berkeley. , (2013).
- Allan, C., et al. OMERO: flexible, model-driven data management for experimental biology. Nature Methods. 9 (3), 245-253 (2012).
- Simian, M., Bissell, M. J. Organoids: A historical perspective of thinking in three dimensions. Journal of Cell Biology. 216 (1), 31-40 (2017).
- Bissell, M. J. The differentiated state of normal and malignant cells or how to define a "normal" cell in culture. International Review of Cytology. 70, 27-100 (1981).
- Serban, M. A., Prestwich, G. D. Modular extracellular matrices: solutions for the puzzle. Methods. 45 (1), 93-98 (2008).
- Kaylan, K. B., et al. Mapping lung tumor cell drug responses as a function of matrix context and genotype using cell microarrays. Integrative Biology (Cambridge). 8 (12), 1221-1231 (2016).
- Lin, C. H., Jokela, T., Gray, J., LaBarge, M. A. Combinatorial Microenvironments Impose a Continuum of Cellular Responses to a Single Pathway-Targeted Anti-cancer Compound. Cell Reports. 21 (2), 533-545 (2017).
- Gjorevski, N., et al. Designer matrices for intestinal stem cell and organoid culture. Nature. 539 (7630), 560-564 (2016).
Перепечатки и разрешения
Запросить разрешение на использование текста или рисунков этого JoVE статьи
Запросить разрешениеThis article has been published
Video Coming Soon
Авторские права © 2025 MyJoVE Corporation. Все права защищены