Method Article
Uso de microarrays para interrogar el impacto microambiental en fenotipos celulares en cáncer
En este artículo
Resumen
El propósito del método presentado aquí es mostrar cómo los microarrays de microambiente (MEMA) pueden ser fabricados y utilizados para interrogar el impacto de miles de microambientes combinatorios simples en el fenotipo de las células cultivadas.
Resumen
Comprender el impacto del microambiente en el fenotipo de las células es un problema difícil debido a la compleja mezcla de factores de crecimiento solubles y proteínas asociadas a la matriz en el microambiente in vivo. Además, los reactivos fácilmente disponibles para el modelado de microambientes in vitro suelen utilizar mezclas complejas de proteínas que están incompletamente definidas y sufren de variabilidad lote a lote. La plataforma de microarray (MEMA) permite evaluar miles de combinaciones simples de proteínas de microambiente por su impacto en los fenotipos celulares en un solo ensayo. Los MEMA se preparan en placas de pozos, lo que permite la adición de ligandos individuales para separar los pocillos que contienen proteínas de matriz de matriz extracelular (ECM). La combinación del ligando soluble con cada ECM impreso forma una combinación única. Un ensayo MEMA típico contiene más de 2.500 microambientes combinatoriales únicos a los que las células están expuestas en un solo ensayo. Como caso de prueba, la línea celular de cáncer de mama MCF7 estaba chapada en la plataforma MEMA. El análisis de este ensayo identificó factores que mejoran e inhiben el crecimiento y la proliferación de estas células. La plataforma MEMA es altamente flexible y se puede ampliar para su uso con otras cuestiones biológicas más allá de la investigación del cáncer.
Introducción
El cultivo de líneas celulares cancerosas en plástico en monocapas bidimensionales (2D) sigue siendo uno de los principales caballos de batalla para los investigadores del cáncer. Sin embargo, el microambiente está siendo cada vez más reconocido por su capacidad para afectar a los fenotipos celulares. En el cáncer, se sabe que el microambiente tumoral influye en múltiples comportamientos celulares, incluyendo el crecimiento, la supervivencia, la invasión y la respuesta a la terapia1,2. Los cultivos celulares monocapa tradicionales suelen carecer de influencias en el microambiente, lo que ha llevado al desarrollo de ensayos tridimensionales (3D) más complejos para cultivar células, incluidos los extractos de membrana de sótano purificados disponibles comercialmente. Sin embargo, estas matrices purificadas suelen ser complicadas de usar y sufren de problemas técnicos como la variabilidad de lotes3 y composiciones complejas3. Como resultado, puede ser difícil asignar la función a proteínas específicas que pueden estar afectando a los fenotipos celulares3.
Para hacer frente a estas limitaciones, hemos desarrollado la tecnología de microarray (MEMA), que reduce el microambiente a combinaciones simples de matriz extracelular (ECM) y proteínas de factor de crecimiento soluble4,5 . La plataforma MEMA permite identificar factores microambientales dominantes que afectan el comportamiento de las células. Mediante el uso de un formato de matriz, se pueden analizar miles de combinaciones de factores de microambiente en un solo experimento. El MEMA descrito aquí interroga 2.500 condiciones únicas de microambiente diferentes. Las proteínas ECM impresas en placas de pozos forman almohadillas de crecimiento sobre las cuales se pueden cultivar células. Los ligandos solubles se añaden a los pozos individuales, creando microambientes combinatorias únicos (ECM + ligando) en cada punto diferente al que están expuestas las células. Las células se cultivan durante varios días, luego se fijan, manchan e imágenes para evaluar los fenotipos celulares como resultado de la exposición a estas combinaciones específicas de microambiente. Dado que los microambientes son combinaciones simples, es sencillo identificar proteínas que impulsan cambios fenotípicos importantes en las células. Los MEMA se han utilizado con éxito para identificar factores que influyen en múltiples fenotipos celulares, incluyendo aquellos que impulsan las decisiones del destino celular y la respuesta alaterapia4,5,6,7. Estas respuestas se pueden validar en experimentos 2D simples y luego se pueden evaluar en condiciones que recapitulan más plenamente la complejidad del microambiente tumoral. La plataforma MEMA es altamente adaptable a una variedad de tipos de células y puntos finales, siempre que estén disponibles buenos biomarcadores fenotípicos.
Protocolo
NOTA: En el diagrama de flujo que se muestra en la Figura 1se describe una descripción general de todo el proceso MEMA, incluido el tiempo estimado. Este protocolo detalla la fabricación de MEMAs en placas de 8 pocillos. El protocolo puede adaptarse a otras placas o portaobjetos.
1. Preparación de tampones de proteínas, diluyentes y tinción
- Equilibrar viales de EME, ligandos y citoquinas a temperatura ambiente (RT) y centrífuga brevemente. Agregue el volumen adecuado del búfer RT adecuado como se indica en la hoja de datos del producto. Siga las recomendaciones del fabricante para las concentraciones de stock.
NOTA: En el Cuadro 1 y en el Cuadro 2se ofrece una lista completa de los ligandos y eFEC con sus existencias y concentraciones finales. Tanto los ligandos como los EME se utilizan típicamente en la concentración más alta de la gama recomendada por el fabricante que provoca un efecto biológico en los ensayos de cultivo estándar de 2 días. Manipule las proteínas suavemente y en los gabinetes de bioseguridad bajo flujo laminar para evitar la contaminación. - Incubar viales con balanceo suave a RT durante 1 h. No vórtice proteínas, ya que esto puede hacer que desnaturalicen.
- Proteínas aliquot para almacenamiento a largo plazo de modo que todas las alícuotas sean de un solo uso para evitar la degradación con ciclos repetidos de congelación/descongelación. Almacene las proteínas liofilizadas a -80 oC (a menos que se especifique lo contrario) hasta que sea necesario. Tenga cuidado de recopilar todos los metadatos para futuras referencias, tales como: (i) nombre de proteína, (ii) fecha preparada, (iii) número de lote/lote, (iv) proveedor, (v) número de catálogo, (vi) concentración, (vii) volumen y (viii) preparador.
- Prepare el tampón diluente que contenga 20% (v/v) glicerol, 10 mM EDTA, 200 mM Tris-HCl, pH 7.2 y esterilizar filtro. Mantenga este búfer estéril y guárdelo en RT.
- Prepare el tampón de tinción que contenga 2% (p/v) de BSA, 1 mM MgCl2y 0,02% NaN3 en solución salina tamponada de fosfato (PBS). Filtrar y almacenar a 4oC.
2. Preparación de una placa de origen ECM
- Retire las existencias alícuotas de proteínas ECM que se imprimirán y descongelarán en hielo. Registre todos los números de lote para el seguimiento de metadatos.
- Flick tubos de proteínas descongeladas suavemente para asegurar la resuspensión adecuada y girar hacia abajo en una centrífuga.
-
Haga mezclas de impresión ECM (ECM) y un fiduciario fluorescente para ser utilizado por un robot de manipulación de líquidos que creará las placas de fuente aleatorias de 384 pocillos.
NOTA: Las placas de origen de 384 pocillos serán utilizadas por una impresora de matriz de pines táctiles para crear las matrices impresas en 8 placas de pozo.- Etiquetar tubos de microcentrífuga de 1,5 ml para cada EPM y el fiduciario.
- Preparar cada EPM combinando 125 l de tampón diluyente (ver paso 1.4) con el volumen adecuado de stock de ECM y llevar la mezcla hasta un volumen total de 250 l con PBS. Las concentraciones finales en cada tubo EPM serán 1x proteína ECM, 5 mM EDTA, 10% glicerol y 100 mM Tris.
- Preparar un fiducial fluorescente disolviéndolo en el tampón apropiado especificado por el fabricante y transferir 250 l a un tubo fiduciario etiquetado.
3. Creación de la placa de origen mediante un manipulador de líquidos
- Diseñe un diseño de placa de 384 pocillos que aleatore las posiciones de los EME y esté optimizado para el cabezal de pasador de impresora de matriz que se está utilizando. Diseñe la colocación del fiducial para que se imprima en la fila 1, columna 1 posición de cada pozo para ayudar en la orientación de la matriz.
NOTA: Se utilizan un total de 14 a 15 réplicas de cada ECM para garantizar datos robustos. Incluya réplicas adicionales de colágeno u otro ECM que produzca un accesorio robusto para evaluar la uniformidad de la unión. El diseño puede necesitar utilizar varias placas de 384 pocillos dependiendo del número de ECM de interés. - Transfiera los tubos EPM a un manipulador de líquidos, manteniendo los tubos a 4 oC, ya sea con un bastidor de tubo refrigerado o utilizando un robot de manipulación de líquidos ubicado en una cámara fría.
- Usando el software del controlador de líquidos, ejecute un programa para transferir 15 l de cada EPM y el fiduciario a los pozos predesignados dentro de la(s) placa(s) fuente(es) de 384 pocillos.
- Pipet PBS en cualquier pozo no utilizado para aumentar la humedad y proteger contra la desecación durante el proceso de impresión.
NOTA: Vea la Figura 2 para ver un ejemplo de un conjunto de placas de origen de 384 pocillos que está optimizado para una cabeza de 4 x 7 pines e incluye un bloque de colágeno I y PBS. - Sellar la(s) placa(s) y mantenerla a 4oC hasta que esté lista para imprimir.
4. Impresión de MEMA utilizando un robot de impresión de matriz
NOTA: La siguiente parte del protocolo describe específicamente la preparación y el uso de MEMA para investigar el impacto de diferentes proteínas de microambiente en el crecimiento y proliferación de células MCF7. Sin embargo, el protocolo se puede adaptar fácilmente para utilizar diferentes ligandos, eCM y celdas para estudiar otras líneas de celda y puntos finales de interés.
-
Con una impresora de pasador táctil, imprima ECM y puntos fiduciarios en 8 placas de pozo. Imprima múltiples réplicas de cada condición de ECM para garantizar la reproducibilidad.
NOTA: Otros formatos de placas o diapositivas se pueden utilizar para la impresión, pero puede ser necesaria la optimización del búfer para lograr una formación de puntos óptima.- Imprima los ECM para el MEMA utilizando pasadores de 350 m de diámetro dispuestos en una configuración de cabezal de impresión de 4 x 7. Imprima las matrices en las placas de 8 pocillos como 20 columnas por 35 filas, para un total de 700 puntos. Las matrices más grandes son posibles en estas placas, pero vienen con un equilibrio de efectos de borde aumentados en la unión celular y la tinción.
- Después de imprimir, guarde las placas en un desecador durante un mínimo de 3 días antes de su uso.
5. Creación de placas de tratamiento de ligadas
- Diseñe un diseño de placa de 96 pocillos que incluya ligandos de interés. Para facilitar el tratamiento de muchas placas MEMA a la vez, diseñe esta placa con espaciado que permita el uso de un pipeta multicanal con 4 puntas espaciadas para transferir líquidos entre los pocillos de los MEMA de 8 pocillos y una placa de 96 pocillos.
NOTA: En este protocolo, se utiliza el conjunto completo de ligandos enumerados en el Cuadro 2. - Descongelar los ligandos en el hielo. Pase brevemente y gire por cada tubo.
- Diluir los ligandos a un stock de trabajo 200x utilizando el búfer recomendado por el fabricante (normalmente PBS).
- Pipet 10 l de cada lintíqueta de 200x en el pozo correspondiente dentro de la placa de 96 pocillos.
- Sellar y almacenar las placas a -20 oC.
NOTA: Haga placas de tratamiento de ligandos en lotes, capturando todos los metadatos para el análisis posterior.
6. Células de cultivo en MEMa
- Bloquee los MEMA durante 20 min con 2 ml por pozo de tampón de bloqueo sin inserción que contenga un 1% de agente de bloqueo sin incrustantes (Tablade materiales)en agua destilada doble (ddH2O).
- Búfer de bloqueo de aspiración y pozos de enjuague triple con PBS. Para evitar la desecación, deje el volumen final de PBS en pozos hasta que esté listo para el revestimiento celular.
NOTA: Es extremadamente útil tener dos trabajadores de banco para los pasos de cultivo celular en MEMa. Un trabajador de banco puede realizar pasos de aspiración, mientras que el segundo realiza pasos de adición. Se recomienda utilizar una tubería multicanal de 1 ml con puntas espaciadas para que coincida con la placa de 8 pocillos para pipeteo y un divisor Y con dos pipetas Pasteur para aspirar múltiples pozos a la vez. - Semilla 2 x 105 Células MCF7 por pozo en 2 ml del medio medio de águila modificado (DMEM) de Dulbecco que contiene 10% suero bovino fetal (FBS).
NOTA: Antes de un experimento COMPLETO de MEMA, realice un experimento de valoración celular para optimizar los números de celda de modo que los puntos MEMA tengan números de celda altos (pero no confluentes) al final de la duración experimental deseada. - Después de 2 a 18 h de adherencia, aspirar medio y reemplazar con 2 mL de medio de crecimiento reducido (DMEM con 0,1% FBS).
NOTA: En este momento se puede utilizar en este momento un suero reducido (p. ej., 0,1% FBS) o condiciones agotadas por factores de crecimiento para aislar el impacto estimulante de ligandos específicos. - Descongelar una placa de tratamiento de ligando en hielo. Placa descongelación centrífuga a 200 x g durante 1 min.
- Transfiera 200 ml de medio de cada pocal de la placa de cultivo al pozo adecuado de la placa de tratamiento. Pipet hacia arriba y hacia abajo para mezclar el volumen de ligando con medio y transferir esta mezcla de nuevo al pozo apropiado en la placa MEMA.
- Ligeramente roca a mano y devolver las placas MEMA a la incubadora. Cultivo durante la duración del experimento en presencia de la combinación de ligando/ECM a 37oC y 5% de CO2.
NOTA: Un experimento MEMA típico se ejecuta durante 72 h; experimentos de mayor duración pueden requerir la sustitución de medio y re-tratamiento con ligando. - Pulse pozos MEMA a 71 h con 100x 5-etil-2'-desoxiuridina (EdU) para una concentración final de 10 m. Incubar en condiciones experimentales con EdU durante 1 h a 37oC y 5% CO2.
NOTA: Otros tratamientos de células vivas también se pueden utilizar en este momento.
7. Fijación y tinción de MEMa
- Después de 72 h y cualquier tratamiento celular vivo, aspirar pozos. Fijar MEMA en 2 mL por pozo de 2% paraformaldehído (PFA) durante 15 min en RT.
- Aspirar PFA. Permeabilizar con 2 ml por pozo de tensioactivo no iónico 0,1% durante 15 min.
- Aspirar el tensioactivo no iónico y lavar con 2 mL por pozo de PBS. Aspirar PBS. Lavar con 2 ml de PBS con 0,05% de polisorbato 20 (PBS-T).
NOTA: La superficie MEMA es hidrófoba, y el no lavarse con PBS-T antes de la incubación de manchas y anticuerpos dará lugar a la formación de vacíos en pozos durante las etapas de incubación y dará lugar a artefactos de tinción. - Aspirar PBS-T. Agregue reactivos de reacción de detección de EdU. Incubar durante 1 h a RT, balanceándose y protegido de la luz. Después de 1 h de incubación, atemple la reacción con el tampón de temple comercial proporcionado.
NOTA: Los pasos de detección y tinción/anticuerpos de EdU se pueden realizar en 1,5 ml por pozo para reducir el coste. - Aspirar el tampón de temple y lavar con PBS-T antes de incubar con manchas o anticuerpos.
- Incubar pozos MEMA con anticuerpos contra la histona H3K9me3 (1:1.000) y la fibrilarina (1:400) en tampón de tinción que contenga un 2% (p/v) de albúmina sérica bovina (BSA), 1 mM MgCl2 y 0,02% NaN3 durante la noche a 4oC.
NOTA: Realice valoraciones de anticuerpos para determinar las concentraciones óptimas antes de usarlas en un conjunto completo de MEMA. - Después de la incubación primaria de anticuerpos o manchas, lave los pozos 2 veces con PBS y una vez con PBS-T.
- Añadir anticuerpos secundarios (nelvón anti-ratón IgG y burro anti-conejo IgG, ambos 1:300) y 0.5 g/mL 4'6'diamidino-2-phenylindole (DAPI). Incubar durante 1 h a RT en la oscuridad.
- Lavar los pozos 2veces con 2 mL por pozo de PBS, dejándolos en los últimos 2 ml de PBS.
- Proceda a la toma de imágenes o almacene MEMA manchados para obtener imágenes posteriores en PBS a 4 oC protegidos de la luz.
8. Imágenes de MEMAs
- Imagen MEMA en un sistema de imágenes automatizado con canales de detección fluorescentes adecuados.
- Salida de datos de imagen resultantes a un sistema de gestión de imágenes. Segmente las celdas y calcule los niveles de intensidad utilizando CellProfiler8.
9. Análisis de datos
NOTA: El análisis de datos consiste en la normalización, la corrección de variaciones y la integración de los datos derivados de CellProfiler sin procesar. En este caso, el entorno R con código personalizado se utiliza para realizar todos los pasos. Sin embargo, cualquier entorno estadístico o programa de software se puede utilizar para realizar las acciones equivalentes. Un ejemplo del código personalizado de código abierto para el entorno de R para el análisis está disponible en: https://www.synapse.org/#!Synapse:syn2862345/wiki/72486.
- Preprocesar y normalizar los datos de imagen segmentados.
- Determine el recuento de células planas utilizando los núcleos manchados DAPI.
- Auto-gate Intensidad EdU para etiquetar celdas como EdU+. Mida la proliferación usando la proporción de Células EDU+ en cada punto.
- Mediana resumen las manchas citoplasmáticas y las mediciones de morfología nuclear a nivel de punto.
- Realizar la eliminación de la normalización de variación no deseada (RUV) en los datos para mejorar la calidad de los datos9.
NOTA: Este enfoque se aplica a cada señal de intensidad y morfología de forma independiente comouna matriz con matrices utilizando las filas y puntos como las columnas como se describió anteriormente 9. - Aplique la normalización de loess bivariados a los residuos normalizados del RUV utilizando la columna de fila y matriz de matriz como variables independientes para corregir los efectos espaciales o relacionados con la intensidad.
- Una vez completada la normalización, la mediana resume las réplicas de cada condición de microambiente para la presentación de informes y análisis posteriores.
Resultados
Para simplificar los impactos microambientales en el crecimiento y la proliferación celular e identificar las condiciones que promovieron o inhibió el crecimiento celular y la proliferación, la línea celular de cáncer de mama MCF7 se sembraba en un conjunto de ocho MEMA de 8 pozos como se describe en el protocolo. Este ensayo expuso las células a 48 EME diferentes y 57 ligandos diferentes, para un total de 2736 condiciones microambientales combinatorias. Después de 71 h en cultivo, las células fueron pulsadas con EdU, fijas, permeablizadas y manchadas con DAPI, la reacción para la detección de EdU, un anticuerpo anti-fibrillina y un anticuerpo anti-H3K9me3. Las células fueron imágenes en un microscopio de alto contenido. Las imágenes se cargaron en un servidor Omero10,segmentadas utilizando CellProfiler8,y normalizadas y analizadas en R9. Los resultados descritos a continuación se centran en las señales DAPI y EdU.
La plataforma de análisis de imágenes de MEMAs produce algunos resultados similares a los disponibles a partir de enfoques de citometría de flujo, como las gráficas de contenido de ADN que muestran fracciones 2N y 4N para células tratadas con un ligando determinado (Figura3A),basadas en la DAPI intensidad y área. Estas gráficas proporcionan evidencia de condiciones que promueven el ciclo celular activo frente a lo indicado por picos bimodales claros correspondientes a células en fases G1 o G2 frente a células arrestadas de crecimiento, que mostrarían cambios en los picos en comparación con las condiciones de control. Utilizamos el número de celda y los datos de intensidad de tinción para resumir los datos, donde el impacto del microambiente (ligands en un eje, ECM en el segundo eje) en ambos números de celda (Figura3B) y incorporación de EdU (Figura3C) se puede ver más fácilmente como cambios en el color y la intensidad del mapa de calor. Como se ve en estas parcelas, muchos de los efectos son impulsados por ligando, ya que la condición de ECM no impactó fuertemente el número de celda o la positividad edU. Nidogen-1 es una clara excepción, ya que la presencia de esta molécula ECM inhibe la unión celular y el crecimiento de MCF7. Los ligandos como FGF6 y NRG1 (NRG1.1 en parcelas) mejoran el número de celdas y tienen altas tasas de incorporación de EdU, mientras que los ligandos como AREG y NRG1-smdf (NRG1.10 en parcelas) inhiben la unión celular y/o el crecimiento de células. Estos hallazgos están respaldados por las imágenes de las células que crecen en las manchas, donde una clara diferencia en el número de celda y la positividad de EdU es evidente (véase el ejemplo en la Figura 3D).
Dado que la plataforma MEMA es una tecnología más reciente, los resultados se validaron en ensayos separados. Las células MCF7 fueron sembradas en placas de 24 pocillos recubiertas con colágeno I en medio DMEM con 10% FBS. Después de 18 h, los medios de comunicación se intercambiaron por medio de crecimiento reducido (DMEM con 0,1% FBS) y las células fueron tratadas con NRG1, FGF6 o AREG y cultivadas por 72 h. EdU se añadió 1 h antes de la fijación. Las celdas se tiñieron con DAPI y para la incorporación de EdU, se imágena, segmentaron y analizaron. Al igual que los resultados obtenidos de la plataforma MEMA, el FGF6 y el NRG1 dieron lugar a números de celda más altos (Figura4A) y tasas de incorporación de EdU (Figura4B) en comparación con las células tratadas con AREG, validando nuestras observaciones en los experimentos originales de MEMA.

Figura 1 : gráfico de flujo que muestra el flujo de trabajo y la línea de tiempo para las diferentes fases de un experimento MEMA típico. Una vez que se imprimen los MEMA, se pueden almacenar a temperatura ambiente desecado durante varios meses antes de su uso. Típicamente, la fase experimental dura 3 a 4 días, pero algunas células primarias de crecimiento lento se han cultivado en MEMAs por hasta 2 semanas. Haga clic aquí para ver una versión más grande de esta figura.

Figura 2 : Diseño de placa fuente ECM para la impresión de matrices. El bloque de colágeno se imprime en MEMA como una cuadrícula, lo que proporciona un conjunto altamente repetitivo de condiciones que permiten una normalización más robusta entre pozos. Los pozos rellenos de PBS proporcionan humedad para ayudar en la prevención de la evaporación durante el proceso de impresión. Haga clic aquí para ver una versión más grande de esta figura.

Figura 3 : Ejemplos de datos generados a partir de un experimento MEMA típico. (A) Perfiles de ciclo celular de valores de intensidad DAPI en la penada frente a recuentos celulares de una placa de 8 pocillos tratada con diferentes ligandos, mostrando la intensidad de la DAPI bifásica que indica las células en la fase del ciclo celular G1 frente a la Fase del ciclo celular G2. (B) Mapa de calor que muestra los recuentos de celdas de punto normalizados agrupados por similitud mediante agrupación jerárquica. El rojo indica un número de celda más alto, y el azul es el número de celda más bajo. Los ligandos están en el eje X, los EM están en el eje Y. (C) Mapa de calor que muestra la incorporación normalizada de EdU, con rojo indicando mayor y azul indicando menor incorporación de EdU. Los ligandos están en el eje X, los EM están en el eje Y. (D) Ejemplo de células MCF7 que crecen en un punto MEMA tratado con NRG1-- que muestra altas tasas de incorporación de EdU (núcleos rosados). La mancha verde es máscara de celda y azul es DAPI. Haga clic aquí para ver una versión más grande de esta figura.
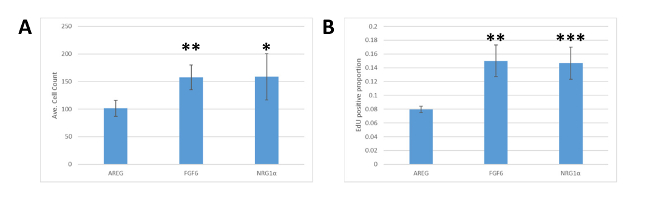
Figura 4 : La validación de los resultados de MEMA en el cultivo celular. (A) Cuantificación del número celular resultante del tratamiento de MCF7 con diferentes ligandos. Los números equivalentes de células MCF7 se clasificaron en placas multipared y luego se trataron con AREG, FGF6 o NRG1. Los pozos tratados con AREG tenían significativamente menos células que las tratadas con FGF6 (** que indica los valores p de la prueba t del estudiante inferiores a 0,01) o NRG1 (* indica un valor p de 0,05) a 72 h después del tratamiento con ligando. (B) Cuantificación del nivel de incorporación de EdU en MCF7 debido al tratamiento con diferentes ligandos, como en el panel A. El tratamiento con AREG da como resultado una proporción significativamente menor de células que incorporan EdU que las células tratadas con FGF6 (**, p < 0.01) o NRG1 (***, p a 0,01). Las barras de error representan la desviación estándar. Haga clic aquí para ver una versión más grande de esta figura.
| Nombre de la proteína | Uniprot ID | Acción Concentración (g/mL) | Final Concentración (g/mL) |
| ANGPT1-1 | Q15389-1 | 100 | 0.04 |
| ANGPT2-1 | O15123-1 | 100 | 0.2 |
| AREG | P15514 | 100 | 0.02 |
| BMP2 | P12643 | 100 | 0.1 |
| BMP3 | P12645 | 1000 | 0.1 |
| BMP4 | P12644 | 100 | 0.1 |
| BMP5-1 | P22003-1 | 100 | 0.1 |
| BMP6 | P22004 | 100 | 0.1 |
| BMP7 | P18075 | 100 | 0.1 |
| CSF2 | P04141 | 100 | 0.02 |
| CTGF-1 | P29279-1 | 100 | 0.05 |
| CXCL12 Alfa | P48061-2 | 100 | 0.01 |
| CXCL12 Beta | P48061-1 | 100 | 0.03 |
| CXCL1 | P09341 | 100 | 0.004 |
| CXCL8-1 | P10145-1 | 100 | 0.3 |
| DLL1-1 | O00548-1 | 500 | 0.5 |
| DLL4 | Q9NR61 | 200 | 0,6 |
| EGF-1 | P01133-1 | 500 | 0.01 |
| FASLG-1 | P48023-1 | 10 | 0.02 |
| FGF2-3 | P09038-2 | 100 | 0.01 |
| FGF6 | P10767 | 100 | 0.01 |
| FLT3LG-1 | P49771-1 | 50 | 0.001 |
| GPNMB-1 | Q14956-1 | 100 | 0.5 |
| HGF-1 | P14210-1 | 50 | 0.04 |
| IGF1-1 | P05019-1 | 200 | 0.01 |
| IGFBP2 | P18065 | 100 | 0.05 |
| IGFBP3-1 | P17936-1 | 100 | 0.1 |
| IL13 | P35225 | 100 | 0.01 |
| IL15 IL15S48AA | P40933-1 | 50 | 0.01 |
| IL1B | P01584 | 25 | 0.001 |
| IL6 | P05231 | 100 | 0.01 |
| IL7-1 | P13232-1 | 100 | 0.01 |
| JAG1-1 | P78504-1 | 200 | 0.5 |
| JAG2 Largo | Q9Y219-1 | 100 | 0.5 |
| KITLG-1 | P21583-1 | 100 | 0.005 |
| KNG1 Hmw | P01042-1 | 100 | 0.2 |
| Lep | P41159 | 1000 | 0.002 |
| LYVE1 | Q9Y5Y7 | 100 | 0.05 |
| NRG1-10 | Q02297-10 | 100 | 0.01 |
| NRG1-1 | Q02297-1 | 100 | 0.05 |
| NRG1-6 | Q02297-6 | 100 | 0.01 |
| PDGFAB | go1990265 | 100 | 0.05 |
| PDGFB-1 | P01127-1 | 100 | 0.05 |
| Ptn | P21246 | 100 | 0.5 |
| Shh | Q15465 | 100 | 0.5 |
| TGFB1 Cterminus | P01137 Cterminus | 20 | 0.01 |
| TGFB1 MA1 | P01137 MA1 | 100 | 0.15 |
| TGFB2 Un | P61812-1 | 20 | 0.01 |
| THPO-1 | P40225-1 | 50 | 0.002 |
| TNFRSF11B | O00300 | 100 | 0.02 |
| TNFSF11-1 | O14788-1 | 100 | 0.01 |
| Tnf | P01375 | 100 | 0.01 |
| VEGFA VEGF206 | P15692-1 | 100 | 0.01 |
| WNT10A | Q9GZT5 | 100 | 0.1 |
| WNT3A-1 | P56704-1 | 200 | 0.1 |
| Wnt5a-1 | P22725-1 | 100 | 0.1 |
Tabla 1: La lista completa de ligandos utilizados para los experimentos MEMA. Se proporcionan el ID de uniprot, las concentraciones de stock y las concentraciones finales de trabajo.
| Proteína ECM | UniprotID | Concentración de stock (g/mL) | Final Concentración (g/mL) | Notas |
| ALCAM 1 | Q13740-1 | 100 | 30 | |
| CDH20 | Q9HBT6 | 300 | 80 | |
| CDH6-1 | P55285-1 | 100 | 40 | |
| CDH8 | P55286 | 100 | 20 | |
| CD44-1 | P16070-1 | 100 | 30 | |
| CEACAM6 | P40199 | 100 | 30 | |
| COL1A1 | P02453 | 5000 | 200 | múltiples subunidades con múltiples identificadores uniprot |
| COL2A1-2 | P02458-2 | 1000 | 200 | |
| COL3A1-1 | P02461-1 | 1000 | 200 | |
| COL4A1-1 | P02462-1 | 1000 | 200 | múltiples subunidades con múltiples identificadores uniprot |
| COL5A1 | P20908 | 1000 | 200 | |
| COL23A1-1 | Q86Y22-1 | 200 | 80 | |
| DSG2 | Q14126 | 100 | 30 | |
| CDH1-1 | P12830-1 | 100 | 40 | |
| ECM1-1 | Q16610-1 | 100 | 40 | |
| FN1-1 | P02751-1 | 1000 | 200 | |
| GAP43-1 | P17677-1 | 158 | 40 | |
| HyA-500K | 1000 | 200 | LOR-0005 | |
| HyA-50K | 1000 | 200 | LOR-0007 | |
| ICAM1 | P05362 | 400 | 80 | |
| ALCAM 1 | Q13740-1 | 100 | 30 | |
| CDH20 | Q9HBT6 | 300 | 80 | |
| CDH8 | P55286 | 100 | 20 | |
| CD44-1 | P16070-1 | 100 | 30 | |
| CEACAM6 | P40199 | 100 | 30 | |
| DSG2 | Q14126 | 100 | 30 | |
| CDH15 | P55291 | 100 | 20 | |
| VCAM1-1 | P19320-1 | 1000 | 200 | |
| LAMA1 | P25391 | 500 | 200 | múltiples subunidades con múltiples identificadores uniprot |
| LAMA3-2 | Q16787-2 | 130 | 40 | |
| Lum | P51884 | 200 | 80 | |
| CDH15 | P55291 | 100 | 20 | |
| NID1-1 | P14543-1 | 100 | 9,3 g/mL Nid, 130 g/mL Lam, 46,5 g/mL COL4 | +COL4 y laminina |
| Omd | Q99983 | 100 | 40 | |
| SPP1 Un | P10451-1 | 100 | 40 | |
| CDH3-1 | P22223-1 | 100 | 40 | |
| PECAM1 Largo | P16284-1 | 150 | 40 | |
| Cnc 1 | P24821-1 | 500 | 200 | |
| VCAM1-1 | P19320-1 | 1000 | 200 | |
| VTN | P04004 | 100 | 40 | |
| Bgn | P21810 | 100 | 40 | |
| DCN Un | P07585-1 | 300 | 80 | |
| POSTN-1 | Q15063-1 | 100 | 40 | |
| Sparc | P09486 | 100 | 40 | |
| THBS1-1 | P07996-1 | 100 | 40 | |
| BCAN-1 | Q96GW7-1 | 100 | 40 | |
| ELN-3 | P15502-3 | 1000 | 200 | |
| FBN1 | P35555 | 254 | 80 |
Tabla 2: La lista completa de proteínas Y condiciones de ECM que se utilizan en los experimentos MEMA. Se proporcionan el ID de uniprot, las concentraciones de stock y las concentraciones finales de trabajo. En algunos casos, la condición impresa representa un complejo proteico o una combinación de múltiples proteínas, que se indica en la columna Notas.
Discusión
La importancia de la "dimensionalidad" y el contexto ha sido un factor motivador en el desarrollo de sistemas de cultivo in vitro como herramientas en la caracterización de las células cancerosas a través de su interacción con el microambiente11,y la capacidad de in vitro sistemas de cultura para imitar el entorno in vivo es una fuerza impulsora detrás de la búsqueda de mejorar esos sistemas de cultivo. Los sistemas in vitro, sin embargo, siguen siendo herramientas significativas de la investigación del cáncer precisamente debido a su capacidad para destilar la compleja situación in vivo hasta un modelo simplificado12.
Aunque los sistemas 2D pueden incluir EME y ligandos, tradicionalmente han carecido de las capacidades de producción para interrogar a un amplio panel de pertubagens combinatoriales. Los extractos de membrana de sótano comercial populares permiten el cultivo en 3D, pero carecen de la procedencia de un panel cuidadosamente definido de proteínas. Los extractos comerciales suelen sufrir de composición incompletamente definida, que puede confundir el análisis y dar lugar a una variación significativa de lote a lote3,13. La plataforma MEMA supera estas barreras, permitiendo el estudio de alteraciones en los fenotipos celulares, actividad metabólica, estado de diferenciación y variaciones en el crecimiento y proliferación celular, ya que son moduladas por endógenos específicos y definidos Factores.
La plataforma MEMA es un enfoque potente, de rendimiento medio a alto para evaluar el impacto del microambiente (tanto eCM como factores solubles) en el fenotipo de las células. La plataforma muestra una gran flexibilidad para los tipos de ensayos y celdas para los que se puede utilizar. Podemos observar los efectos tanto de los ligandos solubles como de las proteínas ECM a las que están expuestas las células. De hecho, recientemente encontramos que los ligandos eran un importante impulsor de la resistencia a los inhibidores dirigidos a HER2, pero que estos efectos podían ser modulados por el ECM5. Una variedad de células, incluyendo células primarias y líneas celulares derivadas de diferentes tipos de células incluyendo pulmón, vejiga, próstata, mama y páncreas, así como células madre pluripotentes inducidas (iPS), se han cultivado con éxito en la plataforma MEMA (ver ejemplos en las referencias5,7,14). El uso de diferentes manchas permite la lectura de múltiples puntos finales celulares, incluyendo el crecimiento celular, diferenciación, y el metabolismo. Otros investigadores han ampliado la plataforma para interrogar el impacto de la rigidez o el módulo elástico, añadiendo una dimensión adicional a la plataforma MEMA15. Por último, la plataforma es susceptible de realizar pantallas de fármacos para la identificación de condiciones de microambiente que mejoran o inhiben la eficacia de los fármacos, como nosotros y otros hemos informado recientemente5,14,15 .
Tal vez el paso más crítico para el éxito de un experimento MEMA es optimizar la densidad de chapado celular. La optimización de la densidad de las células garantiza que haya suficientes celdas para proporcionar datos robustos, pero no tantos que el punto se vuelva demasiado confluente. Las manchas confluentes pueden confundir significativamente los resultados, especialmente si la proliferación se utiliza como un punto final, por lo que es imposible determinar si las bajas tasas de proliferación son el resultado de interacciones con factores microambientales o debido a la inhibición del contacto alta densidad celular. Los experimentos de valoración celular pueden revelar estos problemas, ya que el número promedio de celdas por punto demostrará un aumento lineal con un número creciente de celdas chapadas, pero eventualmente se meseta. El número de celda óptimo debe elegirse en el rango lineal de la curva.
Como se mencionó anteriormente, la plataforma MEMA es flexible y se puede preparar en una variedad de sustratos con diferentes superficies. Estos incluyen portaobjetos de vidrio y formatos de placas multipared. En nuestra experiencia, no todas las químicas de superficie son susceptibles a la impresión MEMA, ya que hemos observado desprendimiento de manchas en algunas superficies debido a las malas propiedades de adhesión y la incapacidad de bloquear la adhesión celular en otras superficies de alta adhesión. Además, el cambio entre diferentes sustratos requiere la optimización de las condiciones de amortiguación, ya que el rendimiento de la impresión con el mismo búfer de impresión puede variar dependiendo de la química de la superficie.
El diámetro de las manchas ECM impresas desempeña un papel importante en la calidad de los datos. En general, se recomienda utilizar los pasadores de impresión de mayor diámetro disponibles para el arrayer en uso (actualmente utilizamos pasadores de diámetro de 350 m). Las manchas de mayor diámetro permiten que un mayor número de celdas ocupen un punto, lo que tiende a dar lugar a datos más robustos de los que se generan con pasadores de diámetro más pequeño. Dado que la unión de las celdas es un proceso estocástico, tiende a haber un alto grado de variabilidad en los datos que está relacionado con el número de celdas originalmente unidas a cada punto. Por lo tanto, se recomienda imprimir un gran número de réplicas para cada condición de ECM. Imprimimos réplicas de ECM de 10 a 15 en cada pozo con nuestras condiciones de impresión actuales para garantizar estadísticas sólidas.
Hemos observado en nuestros experimentos anteriores que en su mayor parte, los efectos de ligando tienden a dominar sobre los efectos ECM. Esto puede deberse en parte a nuestra decisión de agregar colágeno I a todos los puntos ECM, lo que garantiza una unión celular robusta. Sin embargo, creemos que esto también puede homogeneizar los efectos ECM, ya que la mayoría de las manchas tienden a comportarse de una manera muy similar al colágeno I. Alterar la composición del punto para excluir el colágeno puede resultar en el comportamiento celular diferencial como resultado de la interacción con el ECM, pero también afecta significativamente la unión celular, lo que resulta en muchos más puntos desocupados. Los usuarios deben adaptar su composición ECM teniendo en cuenta estas diferencias, particularmente aquellos interesados en células madre y progenitoras y diferenciación, donde la matriz puede tener un impacto significativo16.
Por lo general, realizamos los ensayos MEMA durante períodos de tiempo relativamente cortos (por ejemplo, 72 h máximo). Esto se debe a que las celdas están restringidas a los puntos (el búfer de bloqueo no permite el crecimiento fuera de los puntos de nuestra experiencia). Con las células que se dividen rápidamente, el crecimiento de más de 72 h conducirá a un crecimiento excesivo del punto, lo que a su vez complica la segmentación de la imagen a medida que las células se llenan y se acumulan entre sí, y también puede afectar a los datos, ya que el detención del crecimiento puede ocurrir con la inhibición del contacto. Hemos realizado tratamientos más largos con células primarias de crecimiento muy lento (10-14 días), pero se debe tener cuidado en estos ensayos para cambiar los medios y reponer los ligandos cada 3 a 4 días.
Los esfuerzos continuos para desarrollar la plataforma MEMA se centran en dos áreas de interés, la maximización de la calidad óptica para la toma de imágenes y la optimización dentro de recipientes de cultivo más pequeños. La calidad óptica se convierte en un factor crucial cuando los investigadores requieren una microscopía de mayor resolución para identificar la localización subcelular de sus marcadores de interés. Las pantallas iniciales se pueden realizar a menor resolución en microscopios de alto rendimiento seguidos de imágenes de puntos de interés específicos en instrumentos de mayor resolución, pero la calidad de imagen puede verse comprometida si las propiedades ópticas del sustrato son deficientes. La mejora de las propiedades ópticas del sustrato permitiría a los investigadores realizar las pantallas iniciales en sistemas de imágenes de alta resolución sin necesidad de volver a adquirir imágenes seleccionadas a mayor resolución. Por último, la capacidad de realizar MEMa en recipientes de cultivo más pequeños, como placas de 96 pocillos, permitiría una reducción en el volumen de tratamiento y una expansión de ligandos y réplicas interrogados. Esta transición requiere la optimización de las interacciones entre sustrato-buffer-proteína e impresión de matrices dentro de nuevos recipientes de cultivo. Tales esfuerzos en curso mejorarán la plataforma MEMA y ampliarán sus poderosas capacidades para identificar proteínas microambientales relevantes que alteran los fenotipos celulares para una variedad de tipos celulares, que luego pueden ser investigados en ensayos confirmatorios.
Divulgaciones
Los autores no tienen nada que revelar.
Agradecimientos
Este trabajo fue apoyado por la subvención HG008100 (J.W.G., L.M.H. y J.E.K. de la Biblioteca del Fondo Común de NIH).
Materiales
| Name | Company | Catalog Number | Comments |
| Aushon 2470 | Aushon BioSystems | Arrayer robot system used in the protocol | |
| Nikon HCA | Nikon | High Content Imaging system designed around Nikon Eclipse Ti Inverted Microscope | |
| BioTek Precision XS liquid Handler | BioTek | liquid handling robot used in the protocol | |
| Trizma hydrochloride buffer solution | Sigma | T2069 | |
| EDTA | Invitrogen | 15575-038 | |
| Glycerol | Sigma | G5516 | |
| Triton X100 | Sigma | T9284 | |
| Tween 20 | Sigma | P7949 | |
| Kolliphor P338 | BASF | 50424591 | |
| 384-well microarray plate, cylindrical well | Thermo Fisher | ab1055 | |
| Nunc 8 well dish | Thermo Fisher | 267062 | |
| Paraformaldehyde 16% solution | Electron Microscopy Science | 15710 | |
| BSA | Fisher | BP-1600 | |
| Sodium Azide | Sigma | S2002 | |
| Cell Mask | Molecular Probes | H32713 | |
| Click-iTEdU Alexa Fluor | Molecular Probes | C10357 | |
| DAPI | Promo Kine | PK-CA70740043 | |
| ALCAM | R & D Systems | 656-AL | ECM |
| Cadherin-20 (CDH20) | R & D Systems | 5604-CA | ECM |
| Cadherin-6 (CDH6) | R & D Systems | 2715-CA | ECM |
| Cadherin-8 (CDH8) | R & D Systems | 188-C8 | ECM |
| CD44 | R & D Systems | 3660-CD | ECM |
| CEACAM6 | R & D Systems | 3934-CM | ECM |
| Collagen I | Cultrex | 3442-050-01 | ECM |
| Collagen Type II | Millipore | CC052 | ECM |
| Collagen Type III | Millipore | CC054 | ECM |
| Collagen Type IV | Sigma | C5533 | ECM |
| Collagen Type V | Millipore | CC077 | ECM |
| COL23A1 | R & D Systems | 4165-CL | ECM |
| Desmoglein 2 | R & D Systems | 947-DM | ECM |
| E-cadherin (CDH1) | R & D Systems | 648-EC | ECM |
| ECM1 | R & D Systems | 3937-EC | ECM |
| Fibronectin | R & D Systems | 1918-FN | ECM |
| GAP43 | Abcam | ab114188 | ECM |
| HyA-500K | R & D Systems | GLR002 | ECM |
| HyA-50K | R & D Systems | GLR001 | ECM |
| ICAM-1 | R & D Systems | 720-IC | ECM |
| Laminin | Sigma | L6274 | ECM |
| Laminin-5 | Abcam | ab42326 | ECM |
| Lumican | R & D Systems | 2846-LU | ECM |
| M-Cad (CDH15) | R & D Systems | 4096-MC | ECM |
| Nidogen-1 | R & D Systems | 2570-ND | ECM |
| Osteoadherin/OSAD | R & D Systems | 2884-AD | ECM |
| Osteopontin (SPP) | R & D Systems | 1433-OP | ECM |
| P-Cadherin (CDH3) | R & D Systems | 861-PC | ECM |
| PECAM1 | R & D Systems | ADP6 | ECM |
| Tenascin C | R & D Systems | 3358-TC | ECM |
| VCAM1 | R & D Systems | ADP5 | ECM |
| Vitronectin | R & D Systems | 2308-VN | ECM |
| Biglycan | R & D Systems | 2667-CM | ECM |
| Decorin | R & D Systems | 143-DE | ECM |
| Periostin | R & D Systems | 3548-F2 | ECM |
| SPARC/osteonectin | R & D Systems | 941-SP | ECM |
| Thrombospondin-1/2 | R & D Systems | 3074-TH | ECM |
| Brevican | R & D Systems | 4009-BC | ECM |
| Elastin | BioMatrix | 5052 | ECM |
| Fibrillin | Lynn Sakai Lab OHSU | N/A | ECM |
| ANGPT2 | RnD_Systems_Own | 623-AN-025 | Ligand |
| IL1B | RnD_Systems_Own | 201-LB-005 | Ligand |
| CXCL8 | RnD_Systems_Own | 208-IL-010 | Ligand |
| IGF1 | RnD_Systems_Own | 291-G1-200 | Ligand |
| TNFRSF11B | RnD_Systems_Own | 185-OS | Ligand |
| BMP6 | RnD_Systems_Own | 507-BP-020 | Ligand |
| FLT3LG | RnD_Systems_Own | 308-FK-005 | Ligand |
| CXCL1 | RnD_Systems_Own | 275-GR-010 | Ligand |
| DLL4 | RnD_Systems_Own | 1506-D4-050 | Ligand |
| HGF | RnD_Systems_Own | 294-HGN-005 | Ligand |
| Wnt5a | RnD_Systems_Own | 645-WN-010 | Ligand |
| CTGF | Life_Technologies_Own | PHG0286 | Ligand |
| LEP | RnD_Systems_Own | 398-LP-01M | Ligand |
| FGF2 | Sigma_Aldrich_Own | SRP4037-50UG | Ligand |
| FGF6 | RnD_Systems_Own | 238-F6 | Ligand |
| IL7 | RnD_Systems_Own | 207-IL-005 | Ligand |
| TGFB1 | RnD_Systems_Own | 246-LP-025 | Ligand |
| PDGFB | RnD_Systems_Own | 220-BB-010 | Ligand |
| WNT10A | Genemed_Own | 90009 | Ligand |
| PTN | RnD_Systems_Own | 252-PL-050 | Ligand |
| BMP3 | RnD_Systems_Own | 113-BP-100 | Ligand |
| BMP4 | RnD_Systems_Own | 314-BP-010 | Ligand |
| TNFSF11 | RnD_Systems_Own | 390-TN-010 | Ligand |
| CSF2 | RnD_Systems_Own | 215-GM-010 | Ligand |
| BMP5 | RnD_Systems_Own | 615-BMC-020 | Ligand |
| DLL1 | RnD_Systems_Own | 1818-DL-050 | Ligand |
| NRG1 | RnD_Systems_Own | 296-HR-050 | Ligand |
| KNG1 | RnD_Systems_Own | 1569-PI-010 | Ligand |
| GPNMB | RnD_Systems_Own | 2550-AC-050 | Ligand |
| CXCL12 | RnD_Systems_Own | 350-NS-010 | Ligand |
| IL15 | RnD_Systems_Own | 247-ILB-005 | Ligand |
| TNF | RnD_Systems_Own | 210-TA-020 | Ligand |
| IGFBP3 | RnD_Systems_Own | 675-B3-025 | Ligand |
| WNT3A | RnD_Systems_Own | 5036-WNP-010 | Ligand |
| PDGFAB | RnD_Systems_Own | 222-AB | Ligand |
| AREG | RnD_Systems_Own | 262-AR-100 | Ligand |
| JAG1 | RnD_Systems_Own | 1277-JG-050 | Ligand |
| BMP7 | RnD_Systems_Own | 354-BP-010 | Ligand |
| TGFB2 | RnD_Systems_Own | 302-B2-010 | Ligand |
| VEGFA | RnD_Systems_Own | 293-VE-010 | Ligand |
| IL6 | RnD_Systems_Own | 206-IL-010 | Ligand |
| CXCL12 | RnD_Systems_Own | 351-FS-010 | Ligand |
| NRG1 | RnD_Systems_Own | 378-SM | Ligand |
| IGFBP2 | RnD_Systems_Own | 674-B2-025 | Ligand |
| SHH | RnD_Systems_Own | 1314-SH-025 | Ligand |
| FASLG | RnD_Systems_Own | 126-FL-010 | Ligand |
Referencias
- Hanahan, D., Coussens, L. M. Accessories to the crime: functions of cells recruited to the tumor microenvironment. Cancer Cell. 21 (3), 309-322 (2012).
- Quail, D. F., Joyce, J. A. Microenvironmental regulation of tumor progression and metastasis. Nature Medicine. 19 (11), 1423-1437 (2013).
- Hughes, C. S., Postovit, L. M., Lajoie, G. A. Matrigel: a complex protein mixture required for optimal growth of cell culture. Proteomics. 10 (9), 1886-1890 (2010).
- LaBarge, M. A., et al. Human mammary progenitor cell fate decisions are products of interactions with combinatorial microenvironments. Integrative Biology (Cambridge). 1 (1), 70-79 (2009).
- Watson, S. S., et al. Microenvironment-Mediated Mechanisms of Resistance to HER2 Inhibitors Differ between HER2+ Breast Cancer Subtypes. Cell Systems. 6 (3), 329-342 (2018).
- Ranga, A., et al. 3D niche microarrays for systems-level analyses of cell fate. Nature Communications. 5, 4324 (2014).
- Malta, D. F. B., et al. Extracellular matrix microarrays to study inductive signaling for endoderm specification. Acta Biomater. 34, 30-40 (2016).
- Kamentsky, L., et al. Improved structure, function and compatibility for CellProfiler: modular high-throughput image analysis software. Bioinformatics. 27 (8), 1179-1180 (2011).
- Gagnon-Bartsch, J. A., Jacob, L., Speed, T. P. Removing Unwanted Variation from High Dimensional Data with Negative Controls. University of California, Berkeley, Department of Statistics, University of California, Berkeley. , (2013).
- Allan, C., et al. OMERO: flexible, model-driven data management for experimental biology. Nature Methods. 9 (3), 245-253 (2012).
- Simian, M., Bissell, M. J. Organoids: A historical perspective of thinking in three dimensions. Journal of Cell Biology. 216 (1), 31-40 (2017).
- Bissell, M. J. The differentiated state of normal and malignant cells or how to define a "normal" cell in culture. International Review of Cytology. 70, 27-100 (1981).
- Serban, M. A., Prestwich, G. D. Modular extracellular matrices: solutions for the puzzle. Methods. 45 (1), 93-98 (2008).
- Kaylan, K. B., et al. Mapping lung tumor cell drug responses as a function of matrix context and genotype using cell microarrays. Integrative Biology (Cambridge). 8 (12), 1221-1231 (2016).
- Lin, C. H., Jokela, T., Gray, J., LaBarge, M. A. Combinatorial Microenvironments Impose a Continuum of Cellular Responses to a Single Pathway-Targeted Anti-cancer Compound. Cell Reports. 21 (2), 533-545 (2017).
- Gjorevski, N., et al. Designer matrices for intestinal stem cell and organoid culture. Nature. 539 (7630), 560-564 (2016).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados