Method Article
マイクロアレイを用いてがん細胞型に及ぼす微小環境への影響を調知る
要約
ここで提示される方法の目的は、マイクロ環境マイクロアレイ(MEMA)を作製し、培養細胞の表現型に対する何千もの単純な組み合わせ微小環境の影響を調べるために使用する方法を示すことです。
要約
細胞の表現型に対する微小環境の影響を理解することは、生体内の微小環境における可溶性成長因子とマトリックス関連タンパク質の両方の複雑な混合物による困難な問題である。さらに、インビトロでの微小環境のモデリングに容易に利用可能な試薬は、通常、不完全に定義され、バッチからバッチの変動に苦しむタンパク質の複雑な混合物を利用します。マイクロ環境マイクロアレイ(MEMA)プラットフォームは、単一のアッセイにおける細胞表現型への影響について、マイクロ環境タンパク質の数千もの単純な組み合わせの評価を可能にします。MEMAはウェルプレートで調製され、個々のリガンドをアレイされた細胞外マトリックス(ECM)タンパク質を含むウェルを分離することができます。各印刷されたECMとの可溶性リガンドの組み合わせは、ユニークな組み合わせを形成します。典型的なMEMAアッセイには、細胞が単一のアッセイでさらされている2,500を超えるユニークな組み合わせ微小環境が含まれています。試験ケースとして、乳癌細胞株MCF7をMEMAプラットフォーム上でメッキした。このアッセイの分析は、これらの細胞の増殖および増殖を増強および阻害する因子を同定した。MEMAのプラットホームは非常に適用範囲が広く、癌の研究を超えて他の生物学的質問と使用するために拡張することができる。
概要
2次元(2D)単層におけるプラスチック上の癌細胞株の培養は、がん研究者にとって依然として主要な主力の1つである。しかし、微小環境は、細胞の型示しに影響を与える能力のためにますます認識されています。癌において、腫瘍微小環境は、成長、生存、侵侵入、および治療1、2への応答を含む複数の細胞行動に影響を与える知られている。従来の単層細胞培養は、通常、微小環境の影響を欠き、市販の精製基膜抽出物を含む細胞を成長させるより複雑な3次元(3D)アッセイの開発につながっている。しかし、これらの精製行列は、通常、使用が複雑であり、バッチ変動3や複雑な組成3などの技術的な問題に苦しむ。その結果、細胞表現型3に影響を与える可能性のある特定のタンパク質に機能を割り当てるのは困難な場合があります。
これらの限界に対処するため、微小環境マイクロアレイ(MEMA)技術を開発し、微小環境を細胞外マトリックス(ECM)と可溶性成長因子タンパク質の単純な組み合わせにまで削減する4,5.MEMAプラットフォームは、細胞の挙動に影響を与える主要な微小環境因子の同定を可能にします。配列形式を使用することで、何千もの微小環境因子の組み合わせを1回の実験でアッセイすることができます。ここで説明するMEMAは、約2,500の異なるユニークな微小環境条件を尋問する。ウェルプレートに印刷されたECMタンパク質は、細胞を培養できる成長パッドを形成します。可溶性リガンドは個々のウェルに添加され、細胞が露出する各異なるスポットにユニークな組み合わせ微小環境(ECM +リガンド)を作成します。細胞は数日間培養され、次いで固定され、染色され、これらの特定の微小環境の組み合わせへの曝露の結果として細胞表現型を評価するために画像化される。微小環境は単純な組み合わせであるため、細胞の主要な現象的変化を駆動するタンパク質を特定するのは簡単です。MEMAは、細胞の運命決定と治療4、5、6、7への応答を駆動するものを含む、複数の細胞表現型に影響を与える因子を同定するために正常に使用されている。これらの応答は、単純な2D実験で検証することができ、腫瘍微小環境の複雑さをより完全に要約する条件下で評価することができます。MEMAプラットフォームは、良好なプノチピックバイオマーカーが利用可能である場合、様々な細胞タイプおよびエンドポイントに非常に適応可能です。
プロトコル
注:MEMA プロセス全体の概要 (推定時間を含む) は、図 1に示すフロー図に概説されています。このプロトコルは、8ウェルプレートにおけるMEMAの製造について詳しく述べています。プロトコルは、他のプレートまたはスライドに適合してもよい。
1. タンパク質、希釈剤、染色バッファーの調製
- EPM、リガンド、サイトカインのバイアルを室温(RT)と短時間遠心分離機に平衡化します。製品データシートに示されているように、適切なRTバッファの適切なボリュームを追加します。在庫濃度に関するメーカーの推奨事項に従ってください。
注:リガンドとEPMの在庫と最終濃度の完全なリストは、表1および表2に記載されています。リガンドとEPMの両方は、通常、標準的な2日間培養アッセイで生物学的効果を引き出す製造業者が推奨する範囲の最高濃度で使用されます。汚染を避けるために層流の下で穏やかに、バイオセーフティキャビネットでタンパク質を扱う。 - RTで1時間の穏やかな揺れでバイアルをインキュベートします。これは、変性を引き起こす可能性がありますので、渦タンパク質をしないでください。
- アリコートタンパク質は長期保存のため、すべてのアリコートは、繰り返し凍結/解凍サイクルで分解を避けるためにのみ単一の使用です。凍結乾燥タンパク質は、必要になるまで-80 °C(特に指定がない限り)に保管してください。(i) タンパク質名、(ii) 準備日、(iii)ロット/バッチ番号、(iv)サプライヤー、(v)カタログ番号、(vi)濃度、(vii)容積、(viii)調製機など、将来の参照のためにすべてのメタデータを収集するように注意してください。
- 20%(v/v)グリセロール、10mM EDTA、200mMトリス-HCl、pH 7.2を含む希釈剤バッファーを調製し、濾過殺します。このバッファを無菌に保ち、RTに保存してください。
- リン酸緩衝生理食液(PBS)に2%(w/v)BSA、1mM MgCl 2、および0.02%NaN3を含む染色バッファーを調製する。フィルターを取り、4 °Cで保存します。
2. ECMソースプレートの調製
- 印刷するECMタンパク質のアリクォートストックを取り除き、氷上で解凍します。メタデータ追跡用のすべてのロット番号を記録します。
- 解凍されたタンパク質のチューブを穏やかにフリックして、適切なリサスペンションを確保し、遠心分離機でスピンダウンします。
-
無作為化された384ウェルソースプレートを作成する液体処理ロボットで使用するECMプリント混合物(EPM)と蛍光線を作ります。
注:384ウェルソースプレートは、タッチピンアレイプリンタで使用され、8ウェルプレートで印刷されたアレイを作成します。- 各EPMおよび受託者のためのラベル1.5 mLマイクロセントリフュージチューブ。
- 125 μL の希釈バッファー (ステップ 1.4 を参照) を適切な体積の ECM ストックと組み合わせて各 EPM を準備し、PBS で合計 250 μL の総体積まで混合物を持ち込みます。各EPMチューブの最終濃度は、1x ECMタンパク質、5 mM EDTA、10%グリセロール、および100mMトリスになります。
- 蛍光繊維を製造元が指定した適切なバッファーに溶解して調作し、250 μLを標識された受託チューブに移します。
3. 液体ハンドラを用したソースプレートの作成
- EPM の位置をランダム化し、使用するアレイ プリンタ ピン ヘッド用に最適化された 384 ウェル プレート レイアウトを設計します。各ウェルの行 1、列 1 の位置に印刷するように、fiducial の配置を設計し、配列の向きを支援します。
注:堅牢なデータを確保するために、各 ECM の合計 14-15 レプリケートが使用されます。結合の均一性の評価のための堅牢な添付ファイルをもたらすコラーゲンまたは別のECMの追加の複製を含めます。対象の EEC の数に応じて、レイアウトで複数の 384 ウェル プレートを使用する必要がある場合があります。 - EPMチューブを液体ハンドラに移し、冷却されたチューブラックでチューブを4°Cに保つか、コールドルームにある液体取り扱いロボットを使用します。
- 液体ハンドラのソフトウェアを使用して、各EPMの15 μLを転送するプログラムを実行し、384ウェルソースプレート内の事前指定ウェルにフィデューシャルを転送します。
- ピペットPBSは、印刷プロセス中に湿度を高め、乾燥から保護するために、任意の未使用の井戸にピペットPBS。
注:4 x 7 ピン ヘッド用に最適化され、コラーゲン I ブロックと PBS を含む 384 ウェル ソース プレート セットの例については、図 2を参照してください。 - シールプレートを、印刷する準備ができるまで4°Cに保ちます。
4. アレイ印刷ロボットを用いたMEMの印刷
注:プロトコルの次の部分は、MCF7細胞の増殖および増殖に対する異なる微小環境タンパク質の影響を調査するためのMEMAの調製および使用について具体的に説明する。しかし、プロトコルは、異なるリガンド、EEC、および細胞を使用して、他の細胞株および目的のエンドポイントを研究するように容易に適応させることができる。
-
タッチピンプリンタを使用して、EPMとフィデューシャルスポットを8つのウェルプレートに印刷します。再現性を確保するために、各 ECM 条件の複数の反復を印刷します。
注:他のプレートフォーマットやスライドは印刷に使用できますが、最適なスポット形成を達成するためにバッファの最適化が必要になる場合があります。- 4 x 7 プリント ヘッド構成に配置された 350 μm の直径ピンを使用して、MEMA の ECM を印刷します。8ウェルプレートの配列を20列35列で印刷し、合計で約700個のスポットを印刷します。より大きい配列はこれらの版で可能であるが、細胞結合および染色の両方の増加した端効果のトレードオフと来る。
- 印刷後は、使用の3日前までにデシケータにプレートを保管してください。
5. リガンド処理プレートの作成
- 関心のあるリガンドを含む96ウェルプレートレイアウトを設計します。一度に多くのMEMAプレートの処理を容易にするために、8ウェルMEMAと96ウェルプレートの間で液体を転送するために4つの間隔の先端を持つマルチチャンネルピペを使用することを可能にする間隔でこのプレートを設計します。
注:このプロトコルでは、表2に記載されているリガンドの完全なセットが利用される。 - 氷の上でリガンドを解凍します。簡単にフリックし、各チューブをスピンダウン。
- 製造業者の推奨バッファー(通常はPBS)を使用して、200倍の作業ストックにリガンドを希釈します。
- 各200xリガンドストックのピペ10μLを96ウェルプレート内の対応ウェルに入れます。
- -20 °Cでプレートをシールして保管します。
注:リガンド処理プレートをバッチで作成し、ダウンストリーム解析用のすべてのメタデータをキャプチャします。
6. MEMA上の細胞の培養
- 二重蒸留水(ddH2 O)で1%の非ファウリング遮断剤(材料の表)を含む非ファウリング遮断バッファーのウェルあたり2mLで20分間MEMをブロックします。
- PBSを使用してブロッキングバッファとトリプルリンスウェルを吸引します。乾燥を防ぐために、細胞めっきの準備ができるまで、PBSの最終体積を井戸に残します。
注:MEMの細胞培養ステップのために2人のベンチワーカーを持つことは非常に有用です。1人のベンチワーカーは吸引ステップを実行でき、2人目は加算ステップを実行します。ピペット用の8ウェルプレートと一度に複数のウェルを吸引する2つのパスツールピペットを持つYスプリッターに一致するように間隔をあけた先端を持つ1 mLマルチチャンネルピペットを使用することをお勧めします。 - 10%の胎児ウシ血清(FBS)を含有するダルベッコの改変イーグル培地(DMEM)培地の2mL中の種子2 x 105 MCF7細胞。
注:完全なMEMA実験の前に、MEMAスポットが目的の実験期間の終わりに高い細胞数を持つ(しかし、コンフルエントではない)ので、細胞数を最適化するために細胞滴定実験を行います。 - 2−18hの接着後、吸引培地を2mLの低成長培地(DMEMを0.1%FBSで置き換える)。
注:減少した血清(例えば、0.1%FBS)または成長因子枯渇条件は、この時点で特定のリガンドの刺激的影響を分離するために使用することができる。 - 氷の上にリガンド処理プレートを解凍します。遠心分離機は1分間200 x gでプレートを解凍しました。
- 培養板の各ウェルから200μLの培地を処理プレート内の適切なウェルに移す。リガンドの体積を媒体と混ぜ合わせ、この混合物をMEMAプレートの適切な井戸に戻すために上下にピペを行います。
- 手で軽く揺れ、MEMAプレートをインキュベーターに戻します。37°Cおよび5%CO2でのリガンド/ECM組み合わせの存在下での実験の持続期間のための培養。
注:典型的なMEMA実験は72時間実行されます。より長い期間の実験は、培地の置換とリガンドとの再処理を必要とする場合があります。 - パルスMEMAウェルは、100x 5-エチニル-2'-デオキシュリジン(EdU)で71時間で、最終的な濃度は10μMのインキュベートで、EdUを1時間37°Cおよび5%CO2で試験条件でインキュベートする。
注:他の生細胞治療もこの時点で使用されてもよい。
7. MEMの固定と染色
- 72時間と任意の生細胞治療の後、井戸を吸引する。RTで2%パラホルムアルデヒド(PFA)のウェルあたり2 mLでMEMAを15分間固定します。
- PFAを吸引する。0.1%のニオニオニック界面活性剤のウェルあたり2 mLで15分間透過性を測定する。
- ニオニオニック界面活性剤を吸引し、PBSのウェルあたり2 mLで洗浄します。吸引PBS。0.05%ポリソルベート20(PBS-T)で2mLのPBSで洗浄します。
注:MEMA表面は疎水性であり、染色および抗体のインキュベーションの前にPBS-Tで洗浄しないと、インキュベーションステップ中にウェルに空隙が形成され、染色アーティファクトが生じます。 - 吸引PBS-T.EdU検出反応試薬を添加する。RTで1時間インキュベートし、揺れ、光から保護します。1時間インキュベーション後、提供された市販のクエンチバッファーとのクエンチ反応。
注:EdU検出および染色/抗体ステップは、コストを削減するためにウェルあたり1.5mLで行うことができます。 - クエンチバッファーを吸引し、汚れまたは抗体でインキュベートする前にPBS-Tで洗浄します。
- ヒストンH3K9me3(1:1,000)およびフィブリラリン(1:400)に対する抗体を使用してMEMAウェルをインキュベートし、2%(w/v)ウシ血清アルブミン(BSA)を含む染色バッファーで、1mM MgCl2および0.02%NaN3を4°Cで一晩使用する。
注:完全なMEMAセットでそれらを使用する前に最適な濃度を決定するために抗体滴定を行います。 - 一次抗体または染色インキュベーションに続いて、PBSでウェル2xを洗浄し、PBS-Tで1回洗浄します。
- 二次抗体(ロバ抗マウスIgGおよびロバ抗ウサギIgG、両方とも1:300)および0.5 μg/mL 4'6‐diamidino‐2‐フェニリンドール(DAPI)を添加する。暗闇の中でRTで1時間インキュベートします。
- PBSのウェルあたり2 mLでウェル2xを洗浄し、最終的な2 mL PBSにそれらを残します。
- 光から保護された4°CでPBSで後のイメージングのために染色されたMEMをイメージングまたは保存してください。
8. MEMAのイメージング
- 適切な蛍光検出チャネルを備えた自動イメージングシステム上の画像MEMA。
- 結果の画像データをイメージ管理システムに出力します。セルをセグメント化し、CellProfiler8を使用して強度レベルを計算します。
9. データ分析
注:データ分析は、Raw CellProfiler 派生データの正規化、変動補正、および要約で構成されます。この例では、カスタム コードを使用する R 環境を使用して、すべての手順を実行します。ただし、任意の統計環境またはソフトウェア プログラムを使用して、同等のアクションを実行できます。分析用の R 環境のオープン ソース カスタム コードの例は、https://www.synapse.org/#!Synapse:syn2862345/wiki/72486 にあります。
- セグメント化されたイメージ データを前処理して正規化します。
- DAPI染色された核を使用してスポットセル数を決定します。
- セルを EdU+としてラベル付けする自動ゲート EdU 強度 。各スポットにおけるEdU+細胞の割合を用いて増殖を測定する。
- 中央値は、サイトレベルで細胞質汚れと核形態の測定値を要約します。
- データ品質を向上させるために、データに対する不要なバリエーション (RUV) 正規化の除去を実行します 9.
注:このアプローチは、前述の9のように列として行とスポットを使用する配列を持つ行列として、各強度信号と形態信号に個別に適用されます。 - 空間的または強度に関連する効果を補正するために、配列行と配列列を独立変数として使用して RUV 正規化された残差に二変量ロース正規化を適用します。
- 正規化が完了したら、中央値は、レポート作成およびさらなる分析のために、各マイクロ環境条件の反復を要約します。
結果
細胞増殖および増殖に対する微小環境影響を簡素化し、細胞増殖および増殖を促進または阻害する条件を同定するために、乳癌細胞株MCF7は、プロトコルに記載されている8ウェルMEMAのセットに播種した。このアッセイは、合計2736の組み合わせ微小環境条件のために、48の異なるEPMと57の異なるリガンドに細胞を曝露しました。培養71時間後、細胞をEdUでパルスし、固定、透過化、DAPI、EdU検出に対する反応、抗フィブリリン抗体、および抗H3K9me3抗体で染色した。細胞を高含有量の顕微鏡で画像化した。画像はOmeroサーバー10にアップロードされ、CellProfiler8を使用してセグメント化され、R9で正規化および分析されました。以下に説明する結果は、DAPI シグナルと EdU シグナルに焦点を当てています。
MEMAの画像解析プラットフォームは、DAPIに基づいて所定のリガンドで処理された細胞に対して2Nおよび4N画分を示すDNA含有量プロットなど、フローサイトメトリーアプローチから得られるものと同様の結果をもたらす(図3A)。強度と面積。これらのプロットは、G1またはG2相の細胞に対応する明確なバイモーダルピークと、制御条件と比較してピークの変化を示す成長止血細胞によって示される、活性細胞循環を促進する条件の証拠を提供する。細胞数と染色強度データを使用してデータを要約し、微小環境(1軸のリガンド、2番目の軸上のECM)の両方の細胞数(図3B)とEdU組み込み(図3C)の影響を要約します。ヒートマップの色と強度の変化として、より簡単に見ることができます。これらのプロットから分かるように、ECM条件は細胞数またはEdU陽性に強く影響を与えなかったため、効果の多くはリガンド駆動である。ニドゲン-1は、このECM分子の存在がMCF7の細胞結合および増殖を阻害するように、明らかな例外である。FGF6やNRG1α(プロット上のNRG1.1)などのリガンドは細胞数を増強し、EdUの取り込み率が高い一方で、AREGやNRG1-smdf(プロット上のNRG1.10)などのリガンドは細胞結合および/または細胞の増殖を阻害する。これらの知見は、細胞数とEdU陽性の明確な違いが明らかなスポット上で成長する細胞の画像によって支持される(図3Dの例を参照)。
MEMAプラットフォームは新しい技術であるため、結果は別々のアッセイで検証されました。MCF7細胞を10%FBSでDMEM培地中にコラーゲンIで被覆した24ウェルプレートに播種した。18時間後、培地を還元増殖培地(0.1%FBSのDMEM)と交換し、細胞をNRG1α、FGF6、またはAREGで処理し、72時間のEdUを固定前に1時間添加した。細胞をDAPIおよびEdU組み込み用に染色し、画像化、セグメント化、および分析した。MEMAプラットフォームから得られた結果と同様に、FGF6とNRG1αはいずれもAREG処理細胞と比較して、より高い細胞数(図4A)とEdU組み込み率(図4B)を生み出し、我々の観察を検証した。元のMEMA実験。

図 1: 典型的な MEMA 実験のさまざまなフェーズのワークフローとタイムラインを示すフロー チャート。MEMを印刷すると、使用前に数ヶ月間室温で保存できます。通常、実験段階は3~4日間続くが、一部の遅い成長原発細胞はMEMA上で最大2週間培養されている。この図のより大きなバージョンを表示するには、ここをクリックしてください。

図 2: 配列印刷用の ECM ソースプレートレイアウト。コラーゲンブロックはグリッドとしてMEMAに印刷され、ウェル間のより堅牢な正規化を可能にする非常に反復的な条件のセットを提供します。PBS充填井戸は、印刷プロセス中の蒸発防止に役立つ湿度を提供します。この図のより大きなバージョンを表示するには、ここをクリックしてください。

図 3:典型的なMEMA実験から生成されたデータの例。(A) ビンニングされたDAPI強度値の細胞周期プロファイルと、異なるリガンドで処理された1つの8ウェルプレートからの細胞数に対する細胞数は、G1対G2細胞周期相における細胞を示す二相性DAPI強度染色を示す。(B) 階層クラスタリングを用いて類似性によってクラスタ化された正規化されたスポットセル数を示すヒートマップ。赤はセル番号が大きいことを示し、青はセル番号が小さいです。リガンドは X 軸上にあり、EPM は Y 軸上にあります。(C) 正規化された EdU の組み込みを示すヒートマップで、赤は高く、青は EdU の組み込みを低く示します。リガンドは X 軸上にあり、EPM は Y 軸上にあります。(D) EDU取り込み(ピンク核)の高い率を示すNRG1-αで処理したMEMAスポットで増殖するMCF7細胞の例。緑色の染色はセルマスク、青はDAPIです。この図のより大きなバージョンを表示するには、ここをクリックしてください。
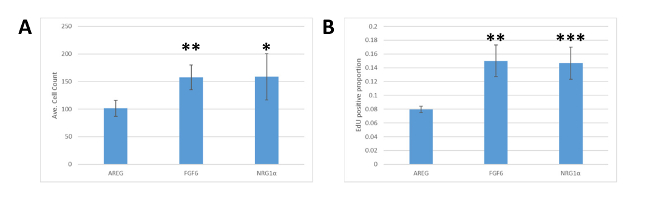
図 4: MEMAの検証は細胞培養をもたらす。(A) 異なるリガンドによるMCF7の治療に起因する細胞数の定量化。同等の数のMCF7細胞をマルチウォールプレートにめっきし、AREG、FGF6、またはNRG1αのいずれかで処理した。AREGで処理したウェルは、72h後リガンド処理でFGF6(0.01未満の学生のt検定p値を示す**)またはNRG1α(*は0.05のp値を示す)で処理された細胞よりも有意に少なかった。 (B) パネルAのように、異なるリガンドによる治療によるMCF7におけるEdUの定量の定量化。 AREG治療は、FGF6(**、p<0.01)またはNRG1α(***、p=0.01)で処理した細胞よりもEdUを組み込んだ細胞の有意に低い割合をもたらす。誤差符は標準偏差を表します。この図のより大きなバージョンを表示するには、ここをクリックしてください。
| タンパク質名 | ユニプロト ID | 株式 濃度 (μg/mL) | 最終的な 濃度 (μg/mL) |
| ANGPT1|1 | Q15389|1 | 100人 | 0.04 |
| ANGPT2|1 | O15123|1 | 100人 | 0.2年 |
| アレグ | P15514 | 100人 | 0.02 |
| BMP2 | P12643 | 100人 | 0.1年 |
| BMP3 | P12645 | 1000年 | 0.1年 |
| BMP4 | P12644 | 100人 | 0.1年 |
| BMP5|1 | P22003|1 | 100人 | 0.1年 |
| BMP6 | P22004 | 100人 | 0.1年 |
| BMP7 | P18075 | 100人 | 0.1年 |
| CSF2 | P04141 | 100人 | 0.02 |
| CTGF|1 | P29279|1 | 100人 | 0.05 |
| CXCL12 |アルファ | P48061|2 | 100人 | 0.01 |
| CXCL12 |Beta | P48061|1 | 100人 | 0.03 |
| CXCL1 | P09341 | 100人 | 0.004 |
| CXCL8|1 | P10145|1 | 100人 | 0.3年 |
| DLL1|1 | O00548|1 | 500名 | 0.5年 |
| DLL4 | Q9NR61 | 200年 | 0.6年 |
| EGF|1 | P01133|1 | 500名 | 0.01 |
| ファスルグ|1 | P48023|1 | 10歳 | 0.02 |
| FGF2|3 | P09038|2 | 100人 | 0.01 |
| FGF6 | P10767 | 100人 | 0.01 |
| FLT3LG|1 | P49771|1 | 50歳 | 0.001 |
| GPNMB|1 | Q14956|1 | 100人 | 0.5年 |
| HGF|1 | P14210|1 | 50歳 | 0.04 |
| IGF1|1 | P05019|1 | 200年 | 0.01 |
| IGFBP2 | P18065 | 100人 | 0.05 |
| IGFBP3|1 | P17936|1 | 100人 | 0.1年 |
| IL13 | P35225 | 100人 | 0.01 |
| IL15 |IL15S48AA | P40933|1 | 50歳 | 0.01 |
| IL1B | P01584 | 25名 | 0.001 |
| IL6 | P05231 | 100人 | 0.01 |
| IL7|1 | P13232|1 | 100人 | 0.01 |
| JAG1|1 | P78504|1 | 200年 | 0.5年 |
| JAG2 |長い | Q9Y219|1 | 100人 | 0.5年 |
| キットルグ|1 | P21583|1 | 100人 | 0.005 |
| KNG1 |HMW | P01042|1 | 100人 | 0.2年 |
| Lep | P41159 | 1000年 | 0.002 |
| ライブ1 | Q9Y5Y7 | 100人 | 0.05 |
| NRG1|10 | Q02297|10 | 100人 | 0.01 |
| NRG1|1 | Q02297|1 | 100人 | 0.05 |
| NRG1|6 | Q02297|6 | 100人 | 0.01 |
| PDGFAB | ゴー1990265 | 100人 | 0.05 |
| PDGFB|1 | P01127|1 | 100人 | 0.05 |
| Ptn | P21246 | 100人 | 0.5年 |
| しゃべる | Q15465 | 100人 | 0.5年 |
| TGFB1 ||クテルミナス | P01137 |クテルミナス | 20歳 | 0.01 |
| TGFB1 ||ラップ | P01137 |ラップ | 100人 | 0.15 |
| TGFB2 |A | P61812|1 | 20歳 | 0.01 |
| THPO|1 | P40225|1 | 50歳 | 0.002 |
| TNFRSF11B | O00300 | 100人 | 0.02 |
| TNFSF11|1 | O14788|1 | 100人 | 0.01 |
| Tnf | P01375 | 100人 | 0.01 |
| VEGFA |VEGF206 | P15692|1 | 100人 | 0.01 |
| WNT10A | Q9GZT5 | 100人 | 0.1年 |
| WNT3A|1 | P56704|1 | 200年 | 0.1年 |
| Wnt5a|1 | P22725|1 | 100人 | 0.1年 |
表1:MEMA実験に使用されるリガンドの全リスト。ユニプロトID、ストック濃度、および最終作業濃度が提供されます。
| ECMタンパク質 | ユニプロティッド | 在庫集中 (μg/mL) | 最終的な 濃度 (μg/mL) | ノート |
| アルカム|1 | Q13740|1 | 100人 | 30歳 | |
| CDH20 | Q9HBT6 | 300人 | 80歳 | |
| CDH6|1 | P55285|1 | 100人 | 40歳 | |
| CDH8 | P55286 | 100人 | 20歳 | |
| CD44|1 | P16070|1 | 100人 | 30歳 | |
| セカム6 | P40199 | 100人 | 30歳 | |
| コル1A1 | P02453 | 5000年 | 200年 | 複数の単一プロト ID を持つ複数のサブユニット |
| コル2A1|2 | P02458|2 | 1000年 | 200年 | |
| コル3A1|1 | P02461|1 | 1000年 | 200年 | |
| コル4A1|1 | P02462|1 | 1000年 | 200年 | 複数の単一プロト ID を持つ複数のサブユニット |
| コル5A1 | P20908 | 1000年 | 200年 | |
| COL23A1|1 | Q86Y22|1 | 200年 | 80歳 | |
| DSG2 | Q14126 | 100人 | 30歳 | |
| CDH1|1 | P12830|1 | 100人 | 40歳 | |
| ECM1|1 | Q16610|1 | 100人 | 40歳 | |
| FN1|1 | P02751|1 | 1000年 | 200年 | |
| GAP43|1 | P17677|1 | 158名 | 40歳 | |
| ハイア-500K | 1000年 | 200年 | LOR-0005 | |
| ハイア-50K | 1000年 | 200年 | LOR-0007 | |
| ICAM1 | P05362 | 400人 | 80歳 | |
| アルカム|1 | Q13740|1 | 100人 | 30歳 | |
| CDH20 | Q9HBT6 | 300人 | 80歳 | |
| CDH8 | P55286 | 100人 | 20歳 | |
| CD44|1 | P16070|1 | 100人 | 30歳 | |
| セカム6 | P40199 | 100人 | 30歳 | |
| DSG2 | Q14126 | 100人 | 30歳 | |
| CDH15 | P55291 | 100人 | 20歳 | |
| VCAM1|1 | P19320|1 | 1000年 | 200年 | |
| ラマ1 | P25391 | 500名 | 200年 | 複数の単一プロト ID を持つ複数のサブユニット |
| ラマ3|2 | Q16787|2 | 130の | 40歳 | |
| ラム | P51884 | 200年 | 80歳 | |
| CDH15 | P55291 | 100人 | 20歳 | |
| NID1|1 | P14543|1 | 100人 | 9.3 μg/mLニド、130 μg/mLラム、46.5 μg/mL COL4 | +COL4とラミニン |
| Omd | Q99983 | 100人 | 40歳 | |
| SPP1 |A | P10451|1 | 100人 | 40歳 | |
| CDH3|1 | P22223|1 | 100人 | 40歳 | |
| ペクマーム1 |長い | P16284|1 | 150名 | 40歳 | |
| TNC|1 | P24821|1 | 500名 | 200年 | |
| VCAM1|1 | P19320|1 | 1000年 | 200年 | |
| VTN | P04004 | 100人 | 40歳 | |
| レフ | P21810 | 100人 | 40歳 | |
| DCN |A | P07585|1 | 300人 | 80歳 | |
| ポスト|1 | Q15063|1 | 100人 | 40歳 | |
| Sparc | P09486 | 100人 | 40歳 | |
| THBS1|1 | P07996|1 | 100人 | 40歳 | |
| BCAN|1 | Q96GW7|1 | 100人 | 40歳 | |
| エルン|3 | P15502|3 | 1000年 | 200年 | |
| FBN1 | P35555 | 254名 | 80歳 |
表2:MEMA実験で使用されるECMタンパク質および条件の完全なリスト。ユニプロトID、ストック濃度、および最終作業濃度が提供されます。場合によっては、印刷条件は、タンパク質複合体または複数のタンパク質の組み合わせを表し、Notes列に示されます。
ディスカッション
「次元性」と文脈の重要性は、微小環境11との相互作用を通じて癌細胞の特性評価のツールとしてのインビトロ培養システムの開発の動機付け要因となっている11、およびインビトロの能力インビボ環境を模倣する文化システムは、これらの文化システムを改善するための探求の原動力です。しかし、インビトロシステムは、単純化されたモデル12にインビボ状況の複合体を蒸留する能力のために、正確に癌研究の重要なツールを残しています。
2D システムには EPM とリガンドを含めることができますが、従来は、組み合わせのパーチュバゲンの広いパネルを調べるためのスループット能力が欠けていました。一般的な市販の地下膜抽出物は、3Dで培養することができますが、タンパク質の慎重に定義されたパネルの証明を欠いています。市販の抽出物は、通常、不完全に定義された組成物に苦しんでおり、分析を混乱させ、有意なバッチ間変動3、13をもたらす可能性がある。MEMAプラットフォームは、これらの障壁を克服し、細胞表現型の変化、代謝活性、分化状態、および細胞の増殖および増殖の変化を特定および定義された内因性によって調節することを可能にする。要因。
MEMAプラットフォームは、細胞の表現型に対する微小環境(ECMおよび可溶性因子の両方)の影響を評価するための強力な中~ハイスループットアプローチです。プラットホームはそれを利用することができるアッセイおよび細胞のタイプのための大きい柔軟性を示す。可溶性リガンドと細胞が露出するECMタンパク質の両方からの効果を観察することができます。実際、我々は最近、リガンドがHER2標的阻害剤に対する耐性の主要な要因であったが、これらの効果はECM5によって変調することができることを発見した。肺、膀胱、前立腺、乳房、膵臓などの異なる細胞型に由来する原発細胞および細胞株、および誘導多能性幹細胞(iPS)細胞を含む様々な細胞がMEMAプラットフォーム上で正常に培養されている(参照)参照5,7,14) の例。異なる染色を使用すると、細胞の増殖、分化、代謝を含む複数の細胞エンドポイントの読み出しが可能になります。他の研究者は、剛性または弾性率の影響を尋問するためにプラットフォームを拡張し、MEMAプラットフォーム15に追加の次元を追加しました。最後に、プラットフォームは、我々と他の人が最近報告したように、薬物の有効性を増強または阻害する微小環境条件の同定のための薬物スクリーンを実行するのに適しています 5,14,15.
おそらく、MEMA実験の成功に最も重要なステップは、細胞めっき密度を最適化することです。細胞の密度を最適化することで、堅牢なデータを提供するのに十分なセルが存在しますが、スポットが過度にコンフルエントになるほど多くはありません。コンフルエントスポットは、特に増殖がエンドポイントとして使用される場合、結果を著しく混乱させる可能性があり、低増殖率が微小環境因子との相互作用の結果であるか、またはからの接触阻害による影響によるものであるかを判断することが不可能です。高い細胞密度。細胞滴定実験は、スポットあたりの平均細胞数がめっきされた細胞の数の増加に伴って線形増加を示すが、最終的には高原となるので、これらの問題を明らかにすることができる。最適なセル番号は、曲線の線形範囲で選択する必要があります。
前述のように、MEMAプラットフォームは柔軟で、異なる表面を持つ様々な基板上に準備することができます。これには、ガラススライドとマルチウォールプレートフォーマットが含まれます。私たちの経験では、すべての表面化学物質がMEMA印刷に適しているわけではありませんが、接着性が低く、他の高い接着面で細胞の接着を遮断できないため、一部の表面のスポット剥離が見られます。さらに、異なる基板間で変化すると、同じ印刷バッファーを持つ印刷の性能は表面化学によって異なるため、バッファ条件の最適化が必要になります。
印刷された ECM スポットの直径は、データの品質において重要な役割を果たします。一般に、アレイアに使用できる最大径プリントピンを使用することをお勧めします(現在は350μm径ピンを使用しています)。直径のスポットが大きいほど、スポットを占有するセルの数が多くなり、直径の小さいピンで生成されるよりも堅牢なデータが生成される傾向があります。細胞の結合は確率的なプロセスであるため、各スポットに最初に付着した細胞の数に関連するデータには高いばらつきがある傾向がある。したがって、ECM 条件ごとに多数の反復を印刷することをお勧めします。現在の印刷条件で各ウェルに10−15 ECMレプリケートを印刷し、堅牢な統計を確保します。
我々は、過去の実験で、ほとんどの場合、リガンド効果がECM効果よりも支配する傾向があることを指摘しました。これは、すべてのECMスポットにコラーゲンIを追加するという当社の決定によるもので、堅牢な細胞結合を保証します。しかし、これはECM効果も均質化する可能性があると考え、ほとんどのスポットはコラーゲンIと非常によく似た様式で振る舞う傾向がある。ECMは、細胞結合にも大きな影響を与えるが、より多くの非占有スポットをもたらす。ユーザーは、これらの違いを念頭に置いてECM組成物を調整する必要があり、特に幹および前駆細胞および分化に興味がある人は、マトリックスが有意な影響を及ぼす可能性がある16。
我々は通常、比較的短い期間(例えば、最大72時間)のMEMAアッセイを行います。これは、セルがスポットに拘束されているためです(ブロッキングバッファは、私たちの経験のスポットの外で成長を許可しません)。細胞を急速に分裂させると、72時間を超える増殖がスポットの過剰増殖を引き起こし、細胞が混雑して互いに積み重なるにつれて画像のセグメンテーションが複雑になり、接触阻害で増殖停止が起こりうるデータにも影響を与える可能性があります。我々は非常に遅い成長原発細胞(10−14日)でより長い治療を行ったが、メディアを変更し、3−4日ごとにリガンドを補充するために、これらのアッセイで注意が必要である。
MEMAプラットフォームの開発に継続的な取り組みは、小さな培養容器内でのイメージングと最適化のための光学品質の最大化という2つの関心分野に焦点を当てています。光学品質は、研究者が目的のマーカーの細胞内局在度を識別するために、より高い分解能顕微鏡検査を必要とする場合に重要な要素となります。初期画面は、高解像度の顕微鏡で低解像度で実行し、その後、高解像度の計測器で特定のスポットをイメージングすることができますが、基板の光学特性が悪い場合は画質が損なわれる可能性があります。基板の光学特性の向上により、研究者は、高解像度の画像を高解像度で再取得することなく、高解像度イメージングシステム上で初期スクリーンを実行することができます。最後に、96ウェルプレートなどの小さな培養容器でMEMAを実行する能力は、治療量の減少と尋問されたリガンドおよび複製の拡大を可能にするであろう。この移行には、基板バッファータンパク質相互作用の最適化と、新しい培養容器内での配列印刷が必要です。このような継続的な取り組みは、MEMAプラットフォームを改善し、その強力な能力を拡張して、様々な細胞タイプの細胞現象を変化させる関連する微小環境タンパク質を同定し、その後に調査することができます。確認アッセイ。
開示事項
著者は何も開示していない。
謝辞
この研究は、NIHコモン・ファンド・ライブラリー・オブ・ネットワーク・セルラー・シグネチャー(LINCS)助成金HG008100(J.W.G.、L.M.H.、J.E.K)によって支援されました。
資料
| Name | Company | Catalog Number | Comments |
| Aushon 2470 | Aushon BioSystems | Arrayer robot system used in the protocol | |
| Nikon HCA | Nikon | High Content Imaging system designed around Nikon Eclipse Ti Inverted Microscope | |
| BioTek Precision XS liquid Handler | BioTek | liquid handling robot used in the protocol | |
| Trizma hydrochloride buffer solution | Sigma | T2069 | |
| EDTA | Invitrogen | 15575-038 | |
| Glycerol | Sigma | G5516 | |
| Triton X100 | Sigma | T9284 | |
| Tween 20 | Sigma | P7949 | |
| Kolliphor P338 | BASF | 50424591 | |
| 384-well microarray plate, cylindrical well | Thermo Fisher | ab1055 | |
| Nunc 8 well dish | Thermo Fisher | 267062 | |
| Paraformaldehyde 16% solution | Electron Microscopy Science | 15710 | |
| BSA | Fisher | BP-1600 | |
| Sodium Azide | Sigma | S2002 | |
| Cell Mask | Molecular Probes | H32713 | |
| Click-iTEdU Alexa Fluor | Molecular Probes | C10357 | |
| DAPI | Promo Kine | PK-CA70740043 | |
| ALCAM | R & D Systems | 656-AL | ECM |
| Cadherin-20 (CDH20) | R & D Systems | 5604-CA | ECM |
| Cadherin-6 (CDH6) | R & D Systems | 2715-CA | ECM |
| Cadherin-8 (CDH8) | R & D Systems | 188-C8 | ECM |
| CD44 | R & D Systems | 3660-CD | ECM |
| CEACAM6 | R & D Systems | 3934-CM | ECM |
| Collagen I | Cultrex | 3442-050-01 | ECM |
| Collagen Type II | Millipore | CC052 | ECM |
| Collagen Type III | Millipore | CC054 | ECM |
| Collagen Type IV | Sigma | C5533 | ECM |
| Collagen Type V | Millipore | CC077 | ECM |
| COL23A1 | R & D Systems | 4165-CL | ECM |
| Desmoglein 2 | R & D Systems | 947-DM | ECM |
| E-cadherin (CDH1) | R & D Systems | 648-EC | ECM |
| ECM1 | R & D Systems | 3937-EC | ECM |
| Fibronectin | R & D Systems | 1918-FN | ECM |
| GAP43 | Abcam | ab114188 | ECM |
| HyA-500K | R & D Systems | GLR002 | ECM |
| HyA-50K | R & D Systems | GLR001 | ECM |
| ICAM-1 | R & D Systems | 720-IC | ECM |
| Laminin | Sigma | L6274 | ECM |
| Laminin-5 | Abcam | ab42326 | ECM |
| Lumican | R & D Systems | 2846-LU | ECM |
| M-Cad (CDH15) | R & D Systems | 4096-MC | ECM |
| Nidogen-1 | R & D Systems | 2570-ND | ECM |
| Osteoadherin/OSAD | R & D Systems | 2884-AD | ECM |
| Osteopontin (SPP) | R & D Systems | 1433-OP | ECM |
| P-Cadherin (CDH3) | R & D Systems | 861-PC | ECM |
| PECAM1 | R & D Systems | ADP6 | ECM |
| Tenascin C | R & D Systems | 3358-TC | ECM |
| VCAM1 | R & D Systems | ADP5 | ECM |
| Vitronectin | R & D Systems | 2308-VN | ECM |
| Biglycan | R & D Systems | 2667-CM | ECM |
| Decorin | R & D Systems | 143-DE | ECM |
| Periostin | R & D Systems | 3548-F2 | ECM |
| SPARC/osteonectin | R & D Systems | 941-SP | ECM |
| Thrombospondin-1/2 | R & D Systems | 3074-TH | ECM |
| Brevican | R & D Systems | 4009-BC | ECM |
| Elastin | BioMatrix | 5052 | ECM |
| Fibrillin | Lynn Sakai Lab OHSU | N/A | ECM |
| ANGPT2 | RnD_Systems_Own | 623-AN-025 | Ligand |
| IL1B | RnD_Systems_Own | 201-LB-005 | Ligand |
| CXCL8 | RnD_Systems_Own | 208-IL-010 | Ligand |
| IGF1 | RnD_Systems_Own | 291-G1-200 | Ligand |
| TNFRSF11B | RnD_Systems_Own | 185-OS | Ligand |
| BMP6 | RnD_Systems_Own | 507-BP-020 | Ligand |
| FLT3LG | RnD_Systems_Own | 308-FK-005 | Ligand |
| CXCL1 | RnD_Systems_Own | 275-GR-010 | Ligand |
| DLL4 | RnD_Systems_Own | 1506-D4-050 | Ligand |
| HGF | RnD_Systems_Own | 294-HGN-005 | Ligand |
| Wnt5a | RnD_Systems_Own | 645-WN-010 | Ligand |
| CTGF | Life_Technologies_Own | PHG0286 | Ligand |
| LEP | RnD_Systems_Own | 398-LP-01M | Ligand |
| FGF2 | Sigma_Aldrich_Own | SRP4037-50UG | Ligand |
| FGF6 | RnD_Systems_Own | 238-F6 | Ligand |
| IL7 | RnD_Systems_Own | 207-IL-005 | Ligand |
| TGFB1 | RnD_Systems_Own | 246-LP-025 | Ligand |
| PDGFB | RnD_Systems_Own | 220-BB-010 | Ligand |
| WNT10A | Genemed_Own | 90009 | Ligand |
| PTN | RnD_Systems_Own | 252-PL-050 | Ligand |
| BMP3 | RnD_Systems_Own | 113-BP-100 | Ligand |
| BMP4 | RnD_Systems_Own | 314-BP-010 | Ligand |
| TNFSF11 | RnD_Systems_Own | 390-TN-010 | Ligand |
| CSF2 | RnD_Systems_Own | 215-GM-010 | Ligand |
| BMP5 | RnD_Systems_Own | 615-BMC-020 | Ligand |
| DLL1 | RnD_Systems_Own | 1818-DL-050 | Ligand |
| NRG1 | RnD_Systems_Own | 296-HR-050 | Ligand |
| KNG1 | RnD_Systems_Own | 1569-PI-010 | Ligand |
| GPNMB | RnD_Systems_Own | 2550-AC-050 | Ligand |
| CXCL12 | RnD_Systems_Own | 350-NS-010 | Ligand |
| IL15 | RnD_Systems_Own | 247-ILB-005 | Ligand |
| TNF | RnD_Systems_Own | 210-TA-020 | Ligand |
| IGFBP3 | RnD_Systems_Own | 675-B3-025 | Ligand |
| WNT3A | RnD_Systems_Own | 5036-WNP-010 | Ligand |
| PDGFAB | RnD_Systems_Own | 222-AB | Ligand |
| AREG | RnD_Systems_Own | 262-AR-100 | Ligand |
| JAG1 | RnD_Systems_Own | 1277-JG-050 | Ligand |
| BMP7 | RnD_Systems_Own | 354-BP-010 | Ligand |
| TGFB2 | RnD_Systems_Own | 302-B2-010 | Ligand |
| VEGFA | RnD_Systems_Own | 293-VE-010 | Ligand |
| IL6 | RnD_Systems_Own | 206-IL-010 | Ligand |
| CXCL12 | RnD_Systems_Own | 351-FS-010 | Ligand |
| NRG1 | RnD_Systems_Own | 378-SM | Ligand |
| IGFBP2 | RnD_Systems_Own | 674-B2-025 | Ligand |
| SHH | RnD_Systems_Own | 1314-SH-025 | Ligand |
| FASLG | RnD_Systems_Own | 126-FL-010 | Ligand |
参考文献
- Hanahan, D., Coussens, L. M. Accessories to the crime: functions of cells recruited to the tumor microenvironment. Cancer Cell. 21 (3), 309-322 (2012).
- Quail, D. F., Joyce, J. A. Microenvironmental regulation of tumor progression and metastasis. Nature Medicine. 19 (11), 1423-1437 (2013).
- Hughes, C. S., Postovit, L. M., Lajoie, G. A. Matrigel: a complex protein mixture required for optimal growth of cell culture. Proteomics. 10 (9), 1886-1890 (2010).
- LaBarge, M. A., et al. Human mammary progenitor cell fate decisions are products of interactions with combinatorial microenvironments. Integrative Biology (Cambridge). 1 (1), 70-79 (2009).
- Watson, S. S., et al. Microenvironment-Mediated Mechanisms of Resistance to HER2 Inhibitors Differ between HER2+ Breast Cancer Subtypes. Cell Systems. 6 (3), 329-342 (2018).
- Ranga, A., et al. 3D niche microarrays for systems-level analyses of cell fate. Nature Communications. 5, 4324 (2014).
- Malta, D. F. B., et al. Extracellular matrix microarrays to study inductive signaling for endoderm specification. Acta Biomater. 34, 30-40 (2016).
- Kamentsky, L., et al. Improved structure, function and compatibility for CellProfiler: modular high-throughput image analysis software. Bioinformatics. 27 (8), 1179-1180 (2011).
- Gagnon-Bartsch, J. A., Jacob, L., Speed, T. P. Removing Unwanted Variation from High Dimensional Data with Negative Controls. University of California, Berkeley, Department of Statistics, University of California, Berkeley. , (2013).
- Allan, C., et al. OMERO: flexible, model-driven data management for experimental biology. Nature Methods. 9 (3), 245-253 (2012).
- Simian, M., Bissell, M. J. Organoids: A historical perspective of thinking in three dimensions. Journal of Cell Biology. 216 (1), 31-40 (2017).
- Bissell, M. J. The differentiated state of normal and malignant cells or how to define a "normal" cell in culture. International Review of Cytology. 70, 27-100 (1981).
- Serban, M. A., Prestwich, G. D. Modular extracellular matrices: solutions for the puzzle. Methods. 45 (1), 93-98 (2008).
- Kaylan, K. B., et al. Mapping lung tumor cell drug responses as a function of matrix context and genotype using cell microarrays. Integrative Biology (Cambridge). 8 (12), 1221-1231 (2016).
- Lin, C. H., Jokela, T., Gray, J., LaBarge, M. A. Combinatorial Microenvironments Impose a Continuum of Cellular Responses to a Single Pathway-Targeted Anti-cancer Compound. Cell Reports. 21 (2), 533-545 (2017).
- Gjorevski, N., et al. Designer matrices for intestinal stem cell and organoid culture. Nature. 539 (7630), 560-564 (2016).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved