Method Article
利用微阵列研究微环境对癌症细胞表型的影响
摘要
这里介绍的方法的目的是展示如何制造微环境微阵列 (MEMA) 并用于询问数千种简单组合微环境对培养细胞表型的影响。
摘要
由于可溶性生长因子和基质相关蛋白在体内微环境中的混合混合,理解微环境对细胞表型的影响是一个难题。此外,用于体外微环境建模的现成试剂通常利用未完全定义的蛋白质的复杂混合物,并遭受批次到批次的变异。微环境微阵列 (MEMA) 平台允许评估数千种微环境蛋白的简单组合,以在单个测定中评估其对细胞表型的影响。MEMEA在井板中制备,允许添加单独的配体来分离含有阵列细胞外基质(ECM)蛋白质的孔。可溶性配体与每个印刷的 ECM 的组合形成了独特的组合。典型的 MEMA 测定包含超过 2,500 个独特的组合微环境,细胞在单个检测中暴露。作为测试案例,乳腺癌细胞系MCF7被镀在MEMA平台上。分析这种测定确定了增强和抑制这些细胞的生长和增殖的因素。MEMA 平台非常灵活,可扩展用于癌症研究以外的其他生物学问题。
引言
在二维(2D)单层塑料上培养癌细胞系仍然是癌症研究人员的主要工作之一。然而,微环境因其影响细胞表型的能力而日益得到认可。在癌症中,肿瘤微环境已知影响多种细胞行为,包括生长、存活、入侵和对治疗1、2的反应。传统的单层细胞培养通常缺乏微环境影响,这导致开发更复杂的三维(3D)检测来生长细胞,包括市售的纯化基底膜提取物。然而,这些纯化的基质通常使用复杂,并遭受技术问题,如批次变异性3和复杂组合物3。因此,很难将功能分配给可能影响细胞表型3的特定蛋白质。
为了弥补这些限制,我们开发了微环境微阵列 (MEMA) 技术,该技术将微环境简化为细胞外基质 (ECM) 和可溶性生长因子蛋白4、5 的简单组合.MEMA 平台能够识别影响细胞行为的显性微环境因素。通过使用数组格式,可以在单个实验中测定数千个微环境因子的组合。此处描述的 MEMA 询问了 2,500 种不同的独特微环境条件。印在井板的ECM蛋白形成生长垫,细胞可以赖以培养。可溶性配体被添加到单个孔中,在细胞暴露的每个不同点上创建独特的组合微环境(ECM + 配体)。细胞被培养数天,然后固定,染色和图像,以评估细胞表型作为暴露于这些特定的微环境组合的结果。由于微环境是简单的组合,因此很容易识别驱动细胞中主要型型变化的蛋白质。MEMEA已经成功地用于识别影响多细胞表型的因素,包括那些驱动细胞命运决定和对治疗4、5、6、7的反应的因素。这些反应可以在简单的2D实验中验证,然后可以在更全面地概括肿瘤微环境复杂性的条件下进行评估。MEMA 平台高度适应各种细胞类型和端点,前提是提供良好的型型生物标志物。
研究方案
注:图 1所示的流程图概述了整个 MEMA 流程(包括估计时间) 。该协议详细介绍了 8 孔板中 MEMEA 的制造。该协议可适用于其他板或幻灯片。
1. 蛋白质、稀释剂和染色缓冲液的制备
- 将 EMS、配体和细胞因子小瓶与室温 (RT) 和短暂离心器进行平衡。如产品数据表所示,添加适当的 RT 缓冲区的适当体积。按照制造商关于库存集中的建议进行操作。
注:表1及表2列有配体及EECM及其储存及最终浓度的完整清单。配体和 EEC 通常以制造商建议的最高浓度使用,在标准 2 天培养性检测中产生生物效应。在层流下轻轻处理蛋白质,在生物安全柜中处理,以避免污染。 - 在RT下用柔和的摇动孵育小瓶1小时。不要涡旋蛋白质,因为这可能导致它们变性。
- 用于长期存储的等分蛋白,以便所有等分只用于避免重复的冻结/解冻循环降解。将冻干蛋白储存在-80°C(除非另有说明),直到需要为止。注意收集所有元数据以供将来参考,例如:(一) 蛋白质名称、(ii) 准备日期、(iii) 批号/批号、(iv) 供应商、(v) 目录号、(vi) 浓度、(vii) 体积和(八) 准备者。
- 准备含有20%(v/v)甘油、10mM EDTA、200 mM Tris-HCl、pH 7.2和过滤器消毒的稀释液缓冲液。保持此缓冲液无菌并储存在 RT 处。
- 在磷酸盐缓冲盐水(PBS)中制备含有2%(w/v)BSA、1 mM MgCl2和0.02%NaN3的染色缓冲液。过滤并储存在4°C。
2. 准备 ECM 源板
- 去除要打印的ECM蛋白的并键库存,并在冰上解冻。记录所有批号以进行元数据跟踪。
- 轻轻轻拂解冻蛋白质的管,以确保在离心机中正确重新悬浮和旋转。
-
使 ECM 打印混合物 (EEP) 和荧光基准由液体处理机器人使用,以创建随机 384 孔源板。
注:触摸引脚阵列打印机将使用 384 孔源板创建 8 孔板中的打印阵列。- 为每个 EPM 和基准标出 1.5 mL 微离心管。
- 将 125 μL 稀释剂缓冲液(参见步骤 1.4)与适当的 ECM 库存量相结合,将混合液的总体积与 PBS 结合至 250 μL,从而制备每个 EPM。每个 EPM 管的最终浓度为 1x ECM 蛋白、5 mM EDTA、10% 甘油和 100 mM Tris。
- 在制造商指定的适当缓冲液中溶解荧光基准,并将其转移到标有基准管的 250 μL。
3. 使用液体处理程序创建源板
- 设计 384 孔板布局,随机化 EVM 的位置,并针对所使用的阵列打印机销头进行了优化。设计基准的位置,使其打印在每口井的第 1 行第 1 列位置中,以帮助阵列方向。
注:每个 ECM 总共使用 14-15 个复制来确保可靠的数据。包括胶原蛋白或其他ECM的附加复制,产生强大的附件,以评估结合的均匀性。布局可能需要使用多个 384 孔板,具体取决于感兴趣的 EVM 数量。 - 将 EPM 管转移到液体处理器上,使用冷却管架或使用位于冷室的液体处理机器人将管保持在 4°C。
- 使用液体处理程序的软件,运行一个程序,将每个 EPM 的 15 μL 和基准转移到 384 孔源板内的预先指定的井。
- 将 PBS 移入任何未使用的井中,以增加湿度,防止在打印过程中干燥。
注:参见图2,了解针对 4 x 7 引脚头进行优化且包括胶原蛋白 I 块和 PBS 的 384 孔源板集的示例。 - 密封板,并保持在 4°C,直到准备打印。
4. 使用阵列打印机器人打印 MEMA
注:协议的第以下部分特别描述了MEMA的制备和使用,以研究不同微环境蛋白对MCF7细胞生长和增殖的影响。然而,该协议可以很容易地适应使用不同的配体、EVM和细胞来研究其他细胞系和感兴趣的端点。
-
使用触摸销打印机将 EVM 和基准点打印到 8 个孔板中。打印每个 ECM 条件的多个副本,以确保可重复性。
注:其他印版格式或幻灯片可用于打印,但可能需要优化缓冲器以实现最佳的点形成。- 使用 4 x 7 打印头配置中排列的 350 μm 直径引脚打印 MEMA 的 EVM。将 8 孔板中的数组打印为 20 列,由 35 行打印,总计约 700 个点。这些板中可以有较大的阵列,但附带了在细胞结合和染色中增加边缘效应的权衡。
- 打印后,在干燥器中存放印版至少 3 天,否则使用时间将提前。
5. 制作木质处理板
- 设计 96 孔板布局,包括感兴趣的配体。为了便于同时处理许多MEMA板,设计这个带间距的板,允许使用带有4个间隔尖端的多通道移液器,在8孔MEMA和96孔板之间的液体转移。
注:在此协议中,使用了表 2中列出的全套配体。 - 在冰上解冻配体。短暂轻拂并旋转每个管。
- 使用制造商推荐的缓冲液(通常为 PBS)将配体稀释到 200x 工作库存中。
- 将每个 200x 配体股票的移液 10 μL 放入 96 孔板内的相应井中。
- 密封并储存板在-20°C。
注:分批制作配体处理板,捕获所有元数据进行下游分析。
6. 在MEMEA上培育细胞
- 在双蒸馏水中(ddH2O)中,用每口2 mL的非结垢阻隔缓冲液(含1%的非污物阻隔剂(材料表))块住MEM2分钟。
- 吸气阻隔缓冲液和三重冲洗井与PBS。为了防止干燥,将PBS的最终体积留在井中,直到准备好进行细胞电镀。
注:在 MEMEA 上为细胞培养步骤提供两个工作台工作人员是非常有帮助的。一个工作台工作人员可以执行吸入步骤,而第二个工作台执行加步。建议使用带间距的 1 mL 多通道移液器,以匹配用于移液的 8 孔板,以及带有两个巴斯德移液器的 Y 分孔器,以便同时吸出多个孔。 - 种子 2 x 105 MCF7 细胞每井 2 mL 的 Dulbecco 的改性 Eagle 培养基 (DMEM) 培养基含有 10% 的胎儿牛血清 (FBS)。
注:在进行完整的MEMA实验之前,执行细胞滴定实验以优化细胞数量,使MEMA点在所需的实验持续时间结束时具有高细胞数(但不汇合)。 - 粘附2~18小时后,吸气介质,用2mL的减生长介质(DMEM,0.1%FBS)代替。
注:减少的血清(例如0.1%FBS)或生长因子耗尽条件可用于此时分离特定配体的刺激作用。 - 在冰上解冻配体处理板。离心解冻板在200 x g 1分钟。
- 将200μL的介质从培养板中的每个孔转移到处理板中的相应孔。上下移管,将配体体积与介质混合,并将混合物移回MEMA板中的相应孔。
- 用手轻轻摇动,将MEMA板返回到孵化器。在37°C和5%CO2下,在配体/ECM组合存在的情况下,在实验期间培养。
注:典型的MEMA实验运行72小时;较长的实验可能需要更换介质和重新处理配体。 - 脉冲MEMA井在71小时与100x 5-ethynyl-2'-脱氧尿氨酸(EdU)的最终浓度为10μM。在实验条件下孵育与EdU在37°C和5%CO2的1小时。
注:此时也可使用其他活细胞治疗。
7. 修复和染色 MEMA
- 经过72小时和任何活细胞治疗,吸气井。在RT处将MEMA固定在2 mL中,每口2%的甲醛(PFA)为15分钟。
- 吸气PFA。每口2mL的渗透剂为0.1%非离子表面活性剂,15分钟。
- 吸气非离子表面活性剂,每井PBS用2 mL洗涤。吸气PBS。用 0.05% 聚索巴20 (PBS-T) 用 2 mL 的 PBS 清洗。
注:MEMA 表面是疏水性的,在染色和抗体孵育之前不用 PBS-T 清洗将导致在孵育步骤期间在井中形成空隙,并产生染色伪影。 - 吸气 PBS-T.添加 EdU 检测反应试剂。在RT孵育1小时,摇动,免受光线照射。1小时孵育后,用提供的商业淬火缓冲液淬火反应。
注:EdU 检测和染色/抗体步骤可在每口井 1.5 mL 下执行,以降低成本。 - 吸气缓冲液,在用污渍或抗体孵育之前用PBS-T清洗。
- 在含有2%(w/v)牛血清白蛋白(BSA)、1 mM MgCl2和0.02%NaN3在4°C过夜的染色缓冲液中孵育MEMA井,以抑制组蛋白H3K9me3(1:1,000)和纤维素(1:400)。
注:在一个完整的MEMA集上使用抗体点位之前,先确定最佳浓度。 - 原发抗体或染色孵育后,用PBS清洗井2倍,用PBS-T洗净。
- 添加二级抗体(驴抗小鼠IgG和驴抗兔子IgG,均为1:300)和0.5微克/mL 4°6°二酰胺2_phenyinole(DAPI)。在黑暗中在RT孵育1小时。
- 洗井 2x 每井 PBS 2 mL,将其留在最后 2 mL PBS 中。
- 继续成像或存储染色MEMA,以便以后在PBS中以4°C进行成像,防止光线照射。
8. MEMEA成像
- 在具有适当荧光检测通道的自动成像系统上成像 MEMA。
- 将生成的图像数据输出到图像管理系统。使用细胞探查器8对单元格进行分割并计算强度水平。
9. 数据分析
注:数据分析包括原始CellProfiler派生数据的规范化、变异校正和汇总。在这种情况下,具有自定义代码的 R 环境用于执行所有步骤。但是,可以使用任何统计环境或软件程序来执行等效操作。用于分析的 R 环境的开源自定义代码的示例位于:https://www.synapse.org/#!Synapse:syn2862345/wiki/72486 。
- 预处理和规范化分段图像数据。
- 使用 DAPI 染色核确定点细胞计数。
- 自动栅极 EdU 强度将单元格标记为 EdU=。使用每个点中的 EdU+细胞比例测量增殖。
- 中位数汇总了细胞质污渍和现场水平的核形态测量。
- 对数据执行删除不需要的变体 (RUV) 规范化,以提高数据质量9。
注:此方法作为矩阵独立应用于每个强度和形态信号,数组使用行和点作为前面描述的列。 - 使用数组行和数组列作为独立变量对 RUV 规范化残差应用双变量黄土规范化,以校正空间或强度相关效果。
- 完成规范化后,中位数将汇总每个微环境条件的复制,以便进行报告和进一步分析。
结果
为了简化微环境对细胞生长和增殖的影响,并确定促进或抑制细胞生长和增殖的条件,乳腺癌细胞系MCF7被播种在协议中描述的一组8 8孔MEMA上。这种测定使细胞接触48种不同的EMS和57种不同的配体,共2736个组合微环境条件。在培养71小时后,用EdU脉冲细胞,固定,渗透,并沾染DAPI,EdU检测反应,抗纤维蛋白抗体,抗H3K9me3抗体。细胞在高含量显微镜上成像。图像被上传到一个Omero服务器10,分段使用CellProfiler8,并在R9规范化和分析。下面描述的结果侧重于 DAPI 和 EdU 信号。
MEMA的图像分析平台产生一些类似于流式细胞学方法的结果,例如,基于DAPI,DNA含量图显示使用给定配体治疗的细胞的2N和4N分数(图3A)。强度和面积。这些图提供了促进活性细胞循环的条件的证据,而与G1或G2相中与生长促动细胞对应的透明双模态峰与生长控制细胞相比,这些条件将显示与控制条件相比的峰值变化。我们使用细胞数和染色强度数据来汇总数据,其中微环境(一个轴上的配体,第二个轴上的 ECM)对两个单元数(图 3B) 和 EdU 合并的影响(图 3C)可以更容易地看到热图颜色和强度的变化。从这些图中可以看出,许多影响是由配体驱动的,因为ECM条件没有强烈地影响细胞数或EdU的积极性。尼多根-1是一个明显的例外,因为这种ECM分子的存在抑制细胞结合和MCF7的生长。配体(如 FGF6 和 NRG1+(图上的 NRG1.1)可增强细胞数量,并具有高 EDU 结合率,而配体(如 AREG 和 NRG1-smdf(图上的 NRG1.10)抑制细胞结合和/或细胞生长。这些发现得到了在斑点上生长的细胞图像的支持,其中细胞数和EdU正性有明显差异(参见图3D中的示例)。
由于MEMA平台是一种较新的技术,结果在单独的检测中得到了验证。MCF7细胞被播种到24孔板,在DMEM介质中涂有胶原蛋白I,具有10%的FBS。18小时后,用培养基交换培养基(DMEM,FBS为0.1%),用NRG1+、FGF6或AREG处理细胞,并培养72小时。在固定前1小时加入EdU。细胞被DAPI和EdU合并、成像、分割和分析所染色。与从MEMA平台获得的结果相似,FGF6和NRG1®都产生了更高的细胞数(图4A)和EDU加入率(图4B)与AREG处理的细胞相比,验证了我们的观察结果。原始的MEMA实验。

图 1:显示典型 MEMA 实验不同阶段的工作流和时间线的流程图。一旦MEMEA被打印出来,它们就可以在室温干燥前几个月储存。通常,实验阶段持续3⁄4天,但一些生长缓慢的原细胞已经在MEMA上培养了长达2周。请点击此处查看此图的较大版本。

图 2:用于阵列打印的 ECM 源板布局。胶原蛋白块作为网格打印到 MEMA 上,它提供了一组高度重复的条件,允许在井间实现更稳健的规范化。充满 PBS 的井提供湿度,有助于防止印刷过程中的蒸发。请点击此处查看此图的较大版本。

图 3:从典型的MEMA实验生成的数据示例。(A) 用不同配体处理的8孔板的细胞周期曲线与细胞计数的细胞周期曲线,显示G1与G2细胞周期阶段的双相DAPI强度染色指示细胞。(B) 热图显示使用分层聚类按相似性聚类的规范化点单元计数。红色表示较高的单元格数,蓝色表示单元格编号较低。配体位于 x 轴上,EVM 位于 y 轴上。(C) 热图显示标准化 EDU 合并,红色表示较高,蓝色表示较低的 EDU 合并。配体位于 x 轴上,EVM 位于 y 轴上。(D) MCF7 细胞生长在使用 NRG1-α 处理的 MEMA 点上生长的示例,显示 EDU 结合率高(粉红色核)。绿色污渍是细胞面罩,蓝色是 DAPI。请点击此处查看此图的较大版本。
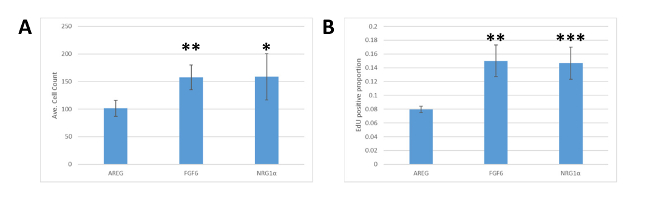
图 4:MEMA的验证导致细胞培养。(A) 用不同配体治疗MCF7后产生的细胞数的定量。将等效数量的MCF7细胞镀入多壁板,然后用AREG、FGF6或NRG1+进行处理。使用AREG治疗的井的细胞比使用FGF6(表示学生的t-test p值小于0.01)或NRG1+(=表示p值0.05)在配体治疗后,其细胞要少得多。(B) 由于处理不同的配体,在 MCF7 中纳入的 EDU 所加入水平的量化,如小组A中。AREG治疗导致与使用FGF6(α,p <0.01)或NRG1+(+,p = 0.01)治疗的细胞相比,纳入EdU的细胞比例明显较低。误差条表示标准偏差。请点击此处查看此图的较大版本。
| 蛋白质名称 | 统一 ID | 股票 浓度 (μg/mL) | 最后 浓度 (μg/mL) |
| ANGPT1=1 | 问题15389+1 | 100 | 0.04 |
| ANGPT2=1 | O15123+1 | 100 | 0.2 |
| AREG | P15514 | 100 | 0.02 |
| BMP2 | P12643 | 100 | 0.1 |
| BMP3 | P12645 | 1000 | 0.1 |
| BMP4 | P12644 | 100 | 0.1 |
| BMP5+1 | P22003_1 | 100 | 0.1 |
| BMP6 | P22004 | 100 | 0.1 |
| BMP7 | P18075 | 100 | 0.1 |
| CSF2 | P04141 | 100 | 0.02 |
| CTGF=1 | P29279_1 | 100 | 0.05 |
| CXCL12*阿 尔 法 | P48061_2 | 100 | 0.01 |
| CXCL12*试用版 | P48061+1 | 100 | 0.03 |
| CXCL1 | P09341 | 100 | 0.004 |
| CXCL8+1 | P10145_1 | 100 | 0.3 |
| DLL1=1 | O00548+1 | 500 | 0.5 |
| DLL4 | Q9NR61 | 200 | 0.6 |
| EGF=1 | P01133+1 | 500 | 0.01 |
| FASLG+1 | P48023+1 | 10 | 0.02 |
| FGF2×3 | P09038_2 | 100 | 0.01 |
| FGF6 | P10767 | 100 | 0.01 |
| FLT3LG+1 | P49771+1 | 50 | 0.001 |
| GPNMB=1 | 问题14956+1 | 100 | 0.5 |
| HGF=1 | P14210+1 | 50 | 0.04 |
| IGF1+1 | P05019+1 | 200 | 0.01 |
| IGFBP2 | P18065 | 100 | 0.05 |
| IGFBP3+1 | P17936+1 | 100 | 0.1 |
| IL13 | P35225 | 100 | 0.01 |
| IL15+IL15S48AA | P40933_1 | 50 | 0.01 |
| IL1B | P01584 | 25 | 0.001 |
| IL6 | P05231 | 100 | 0.01 |
| IL7+1 | P13232+1 | 100 | 0.01 |
| JAG1+1 | P78504+1 | 200 | 0.5 |
| JAG2*长 | Q9Y219+1 | 100 | 0.5 |
| 基特LG+1 | P21583+1 | 100 | 0.005 |
| KNG1+HMW | P01042+1 | 100 | 0.2 |
| LEP | P41159 | 1000 | 0.002 |
| LYVE1 | Q9Y5Y7 | 100 | 0.05 |
| NRG1=10 | Q02297_10 | 100 | 0.01 |
| NRG1=1 | Q02297+1 | 100 | 0.05 |
| NRG1_6 | Q02297_6 | 100 | 0.01 |
| PDGFAB | go1990265 | 100 | 0.05 |
| PDGFB=1 | P01127+1 | 100 | 0.05 |
| PTN | P21246 | 100 | 0.5 |
| 嘘 | Q15465 | 100 | 0.5 |
| TGFB1|特梅努斯 | P01137*特梅努斯 | 20 | 0.01 |
| TGFB1|圈 | P01137*圈 | 100 | 0.15 |
| TGFB2*A | P61812+1 | 20 | 0.01 |
| THPO=1 | P40225+1 | 50 | 0.002 |
| TNFRSF11B | O00300 | 100 | 0.02 |
| TNFSF11+1 | O14788+1 | 100 | 0.01 |
| Tnf | P01375 | 100 | 0.01 |
| VEGFA*VEGF206 | P15692+1 | 100 | 0.01 |
| WNT10A | Q9GZT5 | 100 | 0.1 |
| WNT3A+1 | P56704_1 | 200 | 0.1 |
| Wnt5a_1 | P22725+1 | 100 | 0.1 |
表1:用于MEMA实验的配体的完整列表。提供单prot ID、库存浓度和最终工作浓度。
| ECM 蛋白 | 乌尼普罗特德 | 库存集中 (μg/mL) | 最后 浓度 (μg/mL) | 笔记 |
| 阿尔卡姆+1 | Q13740+1 | 100 | 30 | |
| CDH20 | Q9HBT6 | 300 | 80 | |
| CDH6+1 | P55285+1 | 100 | 40 | |
| CDH8 | P55286 | 100 | 20 | |
| CD44+1 | P16070×1 | 100 | 30 | |
| CEACAM6 | P40199 | 100 | 30 | |
| COL1A1 | P02453 | 5000 | 200 | 具有多个单prot ID 的多个子单元 |
| COL2A1_2 | P02458_2 | 1000 | 200 | |
| COL3A1+1 | P02461+1 | 1000 | 200 | |
| COL4A1+1 | P02462+1 | 1000 | 200 | 具有多个单prot ID 的多个子单元 |
| COL5A1 | P20908 | 1000 | 200 | |
| COL23A1+1 | Q86Y22+1 | 200 | 80 | |
| DSG2 | Q14126 | 100 | 30 | |
| CDH1+1 | P12830×1 | 100 | 40 | |
| ECM1+1 | Q16610+1 | 100 | 40 | |
| FN1+1 | P02751+1 | 1000 | 200 | |
| GAP43_1 | P17677_1 | 158 | 40 | |
| HyA-500K | 1000 | 200 | LOR-0005 | |
| HyA-50K | 1000 | 200 | LOR-0007 | |
| ICAM1 | P05362 | 400 | 80 | |
| 阿尔卡姆+1 | Q13740+1 | 100 | 30 | |
| CDH20 | Q9HBT6 | 300 | 80 | |
| CDH8 | P55286 | 100 | 20 | |
| CD44+1 | P16070×1 | 100 | 30 | |
| CEACAM6 | P40199 | 100 | 30 | |
| DSG2 | Q14126 | 100 | 30 | |
| CDH15 | P55291 | 100 | 20 | |
| VCAM1_1 | P19320+1 | 1000 | 200 | |
| 拉马1 | P25391 | 500 | 200 | 具有多个单prot ID 的多个子单元 |
| 喇嘛3+2 | Q16787_2 | 130 | 40 | |
| 卢姆 | P51884 | 200 | 80 | |
| CDH15 | P55291 | 100 | 20 | |
| NID1+1 | P14543+1 | 100 | 9.3 μg/mL Nid, 130 μg/mL 林, 46.5 μg/mL COL4 | #COL4 和拉米宁 |
| OMD | Q99983 | 100 | 40 | |
| SPP1*A | P10451+1 | 100 | 40 | |
| CDH3+1 | P22223+1 | 100 | 40 | |
| 佩塔姆1+长 | P16284_1 | 150 | 40 | |
| 跨国公司+1 | P24821+1 | 500 | 200 | |
| VCAM1_1 | P19320+1 | 1000 | 200 | |
| VTN | P04004 | 100 | 40 | |
| BGN | P21810 | 100 | 40 | |
| DCN_A | P07585+1 | 300 | 80 | |
| 已分站号=1 | Q15063+1 | 100 | 40 | |
| Sparc | P09486 | 100 | 40 | |
| THBS1=1 | P07996_1 | 100 | 40 | |
| BCAN=1 | Q96GW7+1 | 100 | 40 | |
| 民族解放军+3 | P15502×3 | 1000 | 200 | |
| FBN1 | P35555 | 254 | 80 |
表2:MEMA实验中使用的ECM蛋白和条件的完整列表。提供单prot ID、库存浓度和最终工作浓度。在某些情况下,打印条件表示蛋白质复合体或多种蛋白质的组合,在"注释"列中指示。
讨论
"维数"和语境的重要性一直是体外培养系统发展的动力,通过它们与微环境的相互作用,以及体外细胞的能力,作为癌细胞表征的工具。模仿体内环境的文化系统是寻求改善这些文化系统的动力。然而,体外系统仍然是癌症研究的重要工具,正是因为它们能够将复杂的体内情况提炼为简化的12型。
虽然 2D 系统可以包括 EVM 和配体,但它们传统上缺乏对宽范围组合式 pertubagen 进行质询的吞吐量能力。流行的商业基底膜提取物允许在3D中培养,但缺乏精心定义的蛋白质面板的来源。商业提取物通常受到不完全定义的成分的影响,这可能混淆分析,并导致大量的批次到批次的变化3,13。MEMA平台克服了这些障碍,允许研究细胞表型的变化,代谢活性,分化状态,以及细胞生长和增殖的变化,因为它们是由特定和定义的内源性调节的因素。
MEMA 平台是一种强大的中高通量方法,用于评估微环境(ECM 和可溶性因素)对细胞表型的影响。该平台在可利用的测定和细胞类型方面表现出极大的灵活性。我们可以观察细胞暴露的可溶性配体和ECM蛋白的效果。事实上,我们最近发现配体是抗HER2靶向抑制剂的主要驱动因素,但这些影响可以通过ECM5来调节。各种细胞,包括从不同细胞类型(包括肺、膀胱、前列腺、乳房和胰腺)衍生的初级细胞和细胞系,以及诱导多能干细胞(iPS)细胞,已在MEMA平台上成功培养(参见参考文献5、7、14中的示例。使用不同的污渍允许读取多个细胞端点,包括细胞生长、分化和代谢。其他研究人员已经扩展了平台来询问刚度或弹性模量的影响,为MEMA平台15增加了一个维度。最后,该平台适合执行药物筛选,以识别微环境条件,提高或抑制药物疗效,正如我们和其他人最近报告5,14,15.
也许MEMA实验成功的关键步骤是优化细胞电镀密度。优化细胞的密度可确保有足够的细胞来提供可靠的数据,但不超过这么多,使点变得过度汇入。康康点会显著混淆结果,特别是如果将扩散用作终点,因此无法确定低增殖率是微环境因素相互作用的结果,还是由于接触抑制。高细胞密度。细胞滴定实验可以揭示这些问题,因为每个点的平均细胞数将证明随着镀层细胞数量的增加而呈线性增长,但最终会趋于稳定。应在曲线的线性范围内选择最佳像元数。
如上所述,MEMA 平台非常灵活,可在具有不同表面的各种基板上制备。其中包括玻璃玻片和多壁板格式。根据我们的经验,并非所有表面化学品都适合 MEMA 打印,因为我们观察到某些表面的点分离,因为粘附性能差,并且无法阻止其他高粘附表面上的细胞粘附。此外,不同基板之间的变化需要优化缓冲条件,因为使用同一打印缓冲液的打印性能可能因表面化学性质而异。
打印 ECM 斑点的直径在数据质量中起着重要作用。通常,我们建议使用用于使用的阵列器的最大直径打印引脚(我们目前使用直径为 350 μm 的引脚)。直径较大的点允许更多的细胞占据一个点,这往往产生比用较小直径引脚生成的数据更健壮的数据。由于细胞的绑定是一个随机过程,因此数据中确实有高度的变异性,这与最初附加到每个点的单元格数有关。因此,我们建议为每个 ECM 条件打印大量复制。我们根据当前的打印条件在每个井中打印 10×15 ECM,以确保可靠的统计数据。
我们在过去的实验中注意到,在大多数方面,配体效应往往主导ECM效应。这可能是由于我们决定在所有ECM点添加胶原蛋白I,这确保了强健的细胞结合。然而,我们认为,这也可能同质化ECM效应,因为大多数斑点的行为方式往往与胶原蛋白I非常相似。 改变点组成以排除胶原蛋白I可能导致与ECM,但也显著影响细胞结合,导致更多的空置点。用户应调整他们的ECM组合,同时牢记这些差异,特别是那些对干细胞和祖细胞和分化感兴趣的用户,其中基质可以产生显著的影响16。
我们通常执行 MEMA 测定相对较短的时间(例如,最大 72 小时)。这是因为细胞被限制在斑点(阻塞缓冲器不允许在我们的经验的斑点之外生长)。随着细胞的快速分裂,生长超过72小时将导致斑点的过度生长,这反过来又使图像分割复杂化,因为细胞变得拥挤并相互堆积,并且还会影响数据,因为生长抑制可以发生接触抑制。我们已经对生长非常缓慢的原发性细胞(10-14天)进行了更长的治疗,但必须小心这些检测,以改变培养剂,每3⁄4天补充配体。
持续开发MEMA平台的努力集中在两个感兴趣的领域,即最大化光学质量,在较小的培养容器内进行成像和优化。当研究人员需要更高分辨率的显微镜来识别其感兴趣的标记的亚细胞定位时,光学质量就成为一个关键因素。在高通量显微镜上,可以以较低的分辨率执行初始屏幕,然后对高分辨率仪器上的特定兴趣点进行成像,但如果基板的光学性能较差,图像质量可能会受到影响。改进基板的光学性能将使研究人员能够在高分辨率成像系统上执行初始屏幕,而无需以更高的分辨率重新获取选定的图像。最后,在较小的培养容器(如96孔板)中执行MEMEA的能力,将允许减少治疗量,并扩大被询问的配体和复制。这种过渡需要优化新培养容器内的基质-缓冲蛋白相互作用和阵列打印。这种持续的努力将改进MEMA平台,并扩展其强大的能力,以识别相关的微环境蛋白,改变细胞表型的各种细胞类型,然后可以随后研究确认性测定。
披露声明
作者没有什么可透露的。
致谢
这项工作得到了NIH网络蜂窝签名共同基金图书馆(LINCS)赠款HG008100(J.W.G.、L.M.H.和J.E.K)的支持。
材料
| Name | Company | Catalog Number | Comments |
| Aushon 2470 | Aushon BioSystems | Arrayer robot system used in the protocol | |
| Nikon HCA | Nikon | High Content Imaging system designed around Nikon Eclipse Ti Inverted Microscope | |
| BioTek Precision XS liquid Handler | BioTek | liquid handling robot used in the protocol | |
| Trizma hydrochloride buffer solution | Sigma | T2069 | |
| EDTA | Invitrogen | 15575-038 | |
| Glycerol | Sigma | G5516 | |
| Triton X100 | Sigma | T9284 | |
| Tween 20 | Sigma | P7949 | |
| Kolliphor P338 | BASF | 50424591 | |
| 384-well microarray plate, cylindrical well | Thermo Fisher | ab1055 | |
| Nunc 8 well dish | Thermo Fisher | 267062 | |
| Paraformaldehyde 16% solution | Electron Microscopy Science | 15710 | |
| BSA | Fisher | BP-1600 | |
| Sodium Azide | Sigma | S2002 | |
| Cell Mask | Molecular Probes | H32713 | |
| Click-iTEdU Alexa Fluor | Molecular Probes | C10357 | |
| DAPI | Promo Kine | PK-CA70740043 | |
| ALCAM | R & D Systems | 656-AL | ECM |
| Cadherin-20 (CDH20) | R & D Systems | 5604-CA | ECM |
| Cadherin-6 (CDH6) | R & D Systems | 2715-CA | ECM |
| Cadherin-8 (CDH8) | R & D Systems | 188-C8 | ECM |
| CD44 | R & D Systems | 3660-CD | ECM |
| CEACAM6 | R & D Systems | 3934-CM | ECM |
| Collagen I | Cultrex | 3442-050-01 | ECM |
| Collagen Type II | Millipore | CC052 | ECM |
| Collagen Type III | Millipore | CC054 | ECM |
| Collagen Type IV | Sigma | C5533 | ECM |
| Collagen Type V | Millipore | CC077 | ECM |
| COL23A1 | R & D Systems | 4165-CL | ECM |
| Desmoglein 2 | R & D Systems | 947-DM | ECM |
| E-cadherin (CDH1) | R & D Systems | 648-EC | ECM |
| ECM1 | R & D Systems | 3937-EC | ECM |
| Fibronectin | R & D Systems | 1918-FN | ECM |
| GAP43 | Abcam | ab114188 | ECM |
| HyA-500K | R & D Systems | GLR002 | ECM |
| HyA-50K | R & D Systems | GLR001 | ECM |
| ICAM-1 | R & D Systems | 720-IC | ECM |
| Laminin | Sigma | L6274 | ECM |
| Laminin-5 | Abcam | ab42326 | ECM |
| Lumican | R & D Systems | 2846-LU | ECM |
| M-Cad (CDH15) | R & D Systems | 4096-MC | ECM |
| Nidogen-1 | R & D Systems | 2570-ND | ECM |
| Osteoadherin/OSAD | R & D Systems | 2884-AD | ECM |
| Osteopontin (SPP) | R & D Systems | 1433-OP | ECM |
| P-Cadherin (CDH3) | R & D Systems | 861-PC | ECM |
| PECAM1 | R & D Systems | ADP6 | ECM |
| Tenascin C | R & D Systems | 3358-TC | ECM |
| VCAM1 | R & D Systems | ADP5 | ECM |
| Vitronectin | R & D Systems | 2308-VN | ECM |
| Biglycan | R & D Systems | 2667-CM | ECM |
| Decorin | R & D Systems | 143-DE | ECM |
| Periostin | R & D Systems | 3548-F2 | ECM |
| SPARC/osteonectin | R & D Systems | 941-SP | ECM |
| Thrombospondin-1/2 | R & D Systems | 3074-TH | ECM |
| Brevican | R & D Systems | 4009-BC | ECM |
| Elastin | BioMatrix | 5052 | ECM |
| Fibrillin | Lynn Sakai Lab OHSU | N/A | ECM |
| ANGPT2 | RnD_Systems_Own | 623-AN-025 | Ligand |
| IL1B | RnD_Systems_Own | 201-LB-005 | Ligand |
| CXCL8 | RnD_Systems_Own | 208-IL-010 | Ligand |
| IGF1 | RnD_Systems_Own | 291-G1-200 | Ligand |
| TNFRSF11B | RnD_Systems_Own | 185-OS | Ligand |
| BMP6 | RnD_Systems_Own | 507-BP-020 | Ligand |
| FLT3LG | RnD_Systems_Own | 308-FK-005 | Ligand |
| CXCL1 | RnD_Systems_Own | 275-GR-010 | Ligand |
| DLL4 | RnD_Systems_Own | 1506-D4-050 | Ligand |
| HGF | RnD_Systems_Own | 294-HGN-005 | Ligand |
| Wnt5a | RnD_Systems_Own | 645-WN-010 | Ligand |
| CTGF | Life_Technologies_Own | PHG0286 | Ligand |
| LEP | RnD_Systems_Own | 398-LP-01M | Ligand |
| FGF2 | Sigma_Aldrich_Own | SRP4037-50UG | Ligand |
| FGF6 | RnD_Systems_Own | 238-F6 | Ligand |
| IL7 | RnD_Systems_Own | 207-IL-005 | Ligand |
| TGFB1 | RnD_Systems_Own | 246-LP-025 | Ligand |
| PDGFB | RnD_Systems_Own | 220-BB-010 | Ligand |
| WNT10A | Genemed_Own | 90009 | Ligand |
| PTN | RnD_Systems_Own | 252-PL-050 | Ligand |
| BMP3 | RnD_Systems_Own | 113-BP-100 | Ligand |
| BMP4 | RnD_Systems_Own | 314-BP-010 | Ligand |
| TNFSF11 | RnD_Systems_Own | 390-TN-010 | Ligand |
| CSF2 | RnD_Systems_Own | 215-GM-010 | Ligand |
| BMP5 | RnD_Systems_Own | 615-BMC-020 | Ligand |
| DLL1 | RnD_Systems_Own | 1818-DL-050 | Ligand |
| NRG1 | RnD_Systems_Own | 296-HR-050 | Ligand |
| KNG1 | RnD_Systems_Own | 1569-PI-010 | Ligand |
| GPNMB | RnD_Systems_Own | 2550-AC-050 | Ligand |
| CXCL12 | RnD_Systems_Own | 350-NS-010 | Ligand |
| IL15 | RnD_Systems_Own | 247-ILB-005 | Ligand |
| TNF | RnD_Systems_Own | 210-TA-020 | Ligand |
| IGFBP3 | RnD_Systems_Own | 675-B3-025 | Ligand |
| WNT3A | RnD_Systems_Own | 5036-WNP-010 | Ligand |
| PDGFAB | RnD_Systems_Own | 222-AB | Ligand |
| AREG | RnD_Systems_Own | 262-AR-100 | Ligand |
| JAG1 | RnD_Systems_Own | 1277-JG-050 | Ligand |
| BMP7 | RnD_Systems_Own | 354-BP-010 | Ligand |
| TGFB2 | RnD_Systems_Own | 302-B2-010 | Ligand |
| VEGFA | RnD_Systems_Own | 293-VE-010 | Ligand |
| IL6 | RnD_Systems_Own | 206-IL-010 | Ligand |
| CXCL12 | RnD_Systems_Own | 351-FS-010 | Ligand |
| NRG1 | RnD_Systems_Own | 378-SM | Ligand |
| IGFBP2 | RnD_Systems_Own | 674-B2-025 | Ligand |
| SHH | RnD_Systems_Own | 1314-SH-025 | Ligand |
| FASLG | RnD_Systems_Own | 126-FL-010 | Ligand |
参考文献
- Hanahan, D., Coussens, L. M. Accessories to the crime: functions of cells recruited to the tumor microenvironment. Cancer Cell. 21 (3), 309-322 (2012).
- Quail, D. F., Joyce, J. A. Microenvironmental regulation of tumor progression and metastasis. Nature Medicine. 19 (11), 1423-1437 (2013).
- Hughes, C. S., Postovit, L. M., Lajoie, G. A. Matrigel: a complex protein mixture required for optimal growth of cell culture. Proteomics. 10 (9), 1886-1890 (2010).
- LaBarge, M. A., et al. Human mammary progenitor cell fate decisions are products of interactions with combinatorial microenvironments. Integrative Biology (Cambridge). 1 (1), 70-79 (2009).
- Watson, S. S., et al. Microenvironment-Mediated Mechanisms of Resistance to HER2 Inhibitors Differ between HER2+ Breast Cancer Subtypes. Cell Systems. 6 (3), 329-342 (2018).
- Ranga, A., et al. 3D niche microarrays for systems-level analyses of cell fate. Nature Communications. 5, 4324 (2014).
- Malta, D. F. B., et al. Extracellular matrix microarrays to study inductive signaling for endoderm specification. Acta Biomater. 34, 30-40 (2016).
- Kamentsky, L., et al. Improved structure, function and compatibility for CellProfiler: modular high-throughput image analysis software. Bioinformatics. 27 (8), 1179-1180 (2011).
- Gagnon-Bartsch, J. A., Jacob, L., Speed, T. P. Removing Unwanted Variation from High Dimensional Data with Negative Controls. University of California, Berkeley, Department of Statistics, University of California, Berkeley. , (2013).
- Allan, C., et al. OMERO: flexible, model-driven data management for experimental biology. Nature Methods. 9 (3), 245-253 (2012).
- Simian, M., Bissell, M. J. Organoids: A historical perspective of thinking in three dimensions. Journal of Cell Biology. 216 (1), 31-40 (2017).
- Bissell, M. J. The differentiated state of normal and malignant cells or how to define a "normal" cell in culture. International Review of Cytology. 70, 27-100 (1981).
- Serban, M. A., Prestwich, G. D. Modular extracellular matrices: solutions for the puzzle. Methods. 45 (1), 93-98 (2008).
- Kaylan, K. B., et al. Mapping lung tumor cell drug responses as a function of matrix context and genotype using cell microarrays. Integrative Biology (Cambridge). 8 (12), 1221-1231 (2016).
- Lin, C. H., Jokela, T., Gray, J., LaBarge, M. A. Combinatorial Microenvironments Impose a Continuum of Cellular Responses to a Single Pathway-Targeted Anti-cancer Compound. Cell Reports. 21 (2), 533-545 (2017).
- Gjorevski, N., et al. Designer matrices for intestinal stem cell and organoid culture. Nature. 539 (7630), 560-564 (2016).
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。