Method Article
Verwendung von Mikroarrays zur Vernehmung von Mikroumweltauswirkungen auf zelluläre Phänotypen bei Krebs
In diesem Artikel
Zusammenfassung
Der Zweck der hier vorgestellten Methode ist es, zu zeigen, wie Mikroumgebungsmikroarrays (MEMA) hergestellt und verwendet werden können, um die Auswirkungen von Tausenden von einfachen kombinatorischen Mikroumgebungen auf den Phänotyp kultivierter Zellen abzufragen.
Zusammenfassung
Das Verständnis der Auswirkungen der Mikroumgebung auf den Phänotyp von Zellen ist aufgrund der komplexen Mischung sowohl aus löslichen Wachstumsfaktoren als auch matrixassoziierten Proteinen in der Mikroumgebung in vivo ein schwieriges Problem. Darüber hinaus verwenden leicht verfügbare Reagenzien für die Modellierung von Mikroumgebungen in vitro in der Regel komplexe Mischungen von Proteinen, die unvollständig definiert sind und unter der Variabilität von Charge zu Charge leiden. Die Microenvironment Microarray (MEMA)-Plattform ermöglicht die Bewertung tausender einfacher Kombinationen von Mikroumweltproteinen für ihre Auswirkungen auf zelluläre Phänotypen in einem einzigen Test. Die MEMAs werden in Brunnenplatten hergestellt, was die Zugabe einzelner Liganden ermöglicht, um Brunnen zu trennen, die arrayierte extrazelluläre Matrixproteine (ECM) enthalten. Die Kombination des löslichen Liganden mit jedem gedruckten ECM bildet eine einzigartige Kombination. Ein typischer MEMA-Assay enthält mehr als 2.500 einzigartige kombinatorische Mikroumgebungen, denen Zellen in einem einzigen Assay ausgesetzt sind. Als Testfall wurde die Brustkrebszelllinie MCF7 auf der MEMA-Plattform plattiert. Die Analyse dieses Assays identifizierte Faktoren, die das Wachstum und die Proliferation dieser Zellen sowohl verbessern als auch hemmen. Die MEMA-Plattform ist hochflexibel und kann für den Einsatz mit anderen biologischen Fragen über die Krebsforschung hinaus erweitert werden.
Einleitung
Die Kultivierung von Krebszelllinien auf Kunststoff in zweidimensionalen (2D) Monolayern bleibt eines der Wichtigsten Arbeitspferde für Krebsforscher. Jedoch, die Mikroumgebung wird zunehmend für seine Fähigkeit, zelluläre Phänotypen zu beeinflussen erkannt. Bei Krebs, die Tumor-Mikroumgebung ist bekannt, mehrere zelluläre Verhaltensweisen beeinflussen, einschließlich Wachstum, Überleben, Invasion, und Reaktion auf Therapie1,2. Herkömmliche monolayer Zellkulturen haben in der Regel keine Mikroumgebungseinflüsse, was zur Entwicklung komplexerer dreidimensionaler (3D) Assays zum Wachsen von Zellen geführt hat, einschließlich kommerziell erhältlicher gereinigter Kellermembranextrakte. Diese gereinigten Matrizen sind jedoch in der Regel kompliziert zu verwenden und leiden unter technischen Problemen wie Chargenvariabilität3 und komplexen Zusammensetzungen3. Infolgedessen kann es schwierig sein, bestimmten Proteinen, die sich aufzelluläre Phänotypen auswirken können, eine Funktion zuzuweisen 3 .
Um diese Einschränkungen zu beheben, haben wir die Microenvironment Microarray (MEMA) Technologie entwickelt, die die Mikroumgebung auf einfache Kombinationen von extrazellulärer Matrix (ECM) und löslichen Wachstumsfaktorproteinenreduziert 4,5 . Die MEMA-Plattform ermöglicht die Identifizierung dominanter Mikroumweltfaktoren, die das Verhalten von Zellen beeinflussen. Mithilfe eines Arrayformats können Tausende von Kombinationen von Mikroumgebungsfaktoren in einem einzigen Experiment ermittelt werden. Das hier beschriebene MEMA hinterfragt 2.500 verschiedene einzigartige Mikroumgebungsbedingungen. ECM-Proteine, die in Brunnenplatten gedruckt werden, bilden Wachstumspads, auf denen Zellen kultiviert werden können. Lösliche Liganden werden einzelnen Brunnen zugesetzt, wodurch an jedem einzelnen Ort, dem die Zellen ausgesetzt sind, einzigartige kombinatorische Mikroumgebungen (ECM + Ligand) entstehen. Zellen werden für mehrere Tage kultiviert, dann fixiert, gefärbt und abgebildet, um zelluläre Phänotypen als Ergebnis der Exposition gegenüber diesen spezifischen Mikroumgebungskombinationen zu bewerten. Da es sich bei den Mikroumgebungen um einfache Kombinationen handelt, ist es einfach, Proteine zu identifizieren, die große phänotypische Veränderungen in den Zellen antreiben. MEMAs wurden erfolgreich verwendet, um Faktoren zu identifizieren, die mehrere zelluläre Phänotypen beeinflussen, einschließlich derjenigen, die Entscheidungen über das Zellschicksal und die Reaktion auf Therapie4,5,6,7vorantreiben. Diese Reaktionen können in einfachen 2D-Experimenten validiert und dann unter Bedingungen beurteilt werden, die die Komplexität der Tumormikroumgebung vollständiger rekapitulieren. Die MEMA-Plattform ist sehr anpassungsfähig an eine Vielzahl von Zelltypen und Endpunkten, vorausgesetzt, dass gute phänotypische Biomarker verfügbar sind.
Protokoll
HINWEIS: Eine Übersicht über den gesamten MEMA-Prozess, einschließlich der geschätzten Zeit, ist im Flussdiagramm in Abbildung 1dargestellt. Dieses Protokoll beschreibt die Herstellung von MEMAs in 8-Well-Platten. Das Protokoll kann für andere Platten oder Dias angepasst werden.
1. Herstellung von Protein-, Verdünnungs- und Färbepuffern
- Ausgleich durch Fläschchen von ECMs, Liganden und Zytokinen auf Raumtemperatur (RT) und kurz Zentrifuge. Fügen Sie das entsprechende Volumen des entsprechenden RT-Puffers hinzu, wie auf dem Produktdatenblatt angegeben. Folgen Sie der Empfehlung des Herstellers für Aktienkonzentrationen.
HINWEIS: Eine vollständige Liste der Liganden und EKOM mit ihren Bestands- und Endkonzentrationen ist Tabelle 1 und Tabelle 2enthalten. Sowohl Liganden als auch ECMs werden in der Regel in der höchsten Konzentration des vom Hersteller empfohlenen Bereichs verwendet, der eine biologische Wirkung in Standard-2-Tages-Kultur-Assays hervorruft. Behandeln Sie Proteine schonend und in Biosicherheitsschränken unter laminarem Fluss, um Kontaminationen zu vermeiden. - Inkubieren Fläschchen mit sanftem Schaukeln bei RT für 1 h. Wirbelproteine nicht, da dies dazu führen kann, dass sie denaturieren.
- Aliquot-Proteine für die Langfristige Lagerung, so dass alle Aliquots nur zur Vermeidung von Abbau mit wiederholten Frost-/Tauzyklen verwendet werden. Lyophilisierte Proteine bei -80 °C (sofern nicht anders angegeben) lagern, bis dies erforderlich ist. Achten Sie darauf, alle Metadaten für zukünftige Referenzen zu sammeln, wie z. B.: (i) Proteinname, (ii) vorbereitetes Datum, (iii) Chargen-/Chargennummer, (iv) Lieferant, (v) Katalognummer, (vi) Konzentration, (vii) Volumen und (viii) Vorbereiter.
- Bereiten Sie Verdünnungspuffer mit 20% (v/v) Glycerin, 10 mM EDTA, 200 mM Tris-HCl, pH 7.2 und Filter sterilisieren. Bewahren Sie diesen Puffer steril auf und lagern Sie ihn bei RT.
- Bereiten Sie einen Färbepuffer vor, der 2% (w/v) BSA, 1 mM MgCl2und 0,02% NaN3 in phosphatgepufferter Kochsaline (PBS) enthält. Filtern und lagern bei 4 °C.
2. Vorbereitung einer ECM-Quellenplatte
- Entfernen Sie aliquoted Bestände an ECM-Proteinen, die gedruckt werden sollen, und tauen Sie auf Eis. Zeichnen Sie alle Chargennummern für die Metadatenverfolgung auf.
- Flick Tuben von aufgetauten Proteinen sanft, um eine ordnungsgemäße Resuspension zu gewährleisten und in einer Zentrifuge nach unten zu drehen.
-
Machen Sie ECM-Druckmischungen (EPMs) und einen fluoreszierenden Fiducial, der von einem Flüssigkeitshandhabungsroboter verwendet werden kann, der die randomisierten 384-Well-Quellenplatten erstellt.
HINWEIS: Die 384-Well-Quellplatten werden von einem Touch-Pin-Array-Drucker verwendet, um die gedruckten Arrays in 8 Well-Platten zu erstellen.- Etikettieren Sie 1,5 ml Mikrozentrifugenrohre für jedes EPM und das Treuhandland.
- Bereiten Sie jedes EPM vor, indem Sie 125 L Verdünnungspuffer (siehe Schritt 1.4) mit dem entsprechenden Volumen des ECM-Bestands kombinieren und das Gemisch mit PBS auf ein Gesamtvolumen von 250 l bringen. Die Endkonzentrationen in jedem EPM-Röhrchen sind 1x ECM-Protein, 5 mM EDTA, 10% Glycerin und 100 mM Tris.
- Bereiten Sie ein fluoreszierendes Fiducial vor, indem Sie es in dem vom Hersteller angegebenen Puffer auflösen und 250 l in ein gekennzeichnetes Fiducial-Rohr übertragen.
3. Erstellung der Quellplatte mit einem Flüssigkeitshandler
- Entwerfen Sie ein 384-Well-Plattenlayout, das die Positionen der ECMs randomisiert und für den verwendeten Arraydrucker-Pinkopf optimiert ist. Entwerfen Sie die Platzierung des Treuhandsplatzes so, dass er in der Zeile 1, Spalte 1 Position jedes Brunnens gedruckt wird, um die Arrayausrichtung zu unterstützen.
HINWEIS: Zur Sicherstellung robuster Daten werden insgesamt 14 bis 15 Replikationen jedes ECM verwendet. Fügen Sie zusätzliche Replikationen von Kollagen oder einem anderen ECM ein, das eine robuste Befestigung für die Beurteilung der Homogenität der Bindung liefert. Das Layout muss je nach Anzahl der ecMs möglicherweise mehrere 384-Well-Platten verwenden. - Übertragen Sie EPM-Rohre auf einen Flüssigkeitshandler und halten Sie Rohre bei 4 °C, entweder mit einem gekühlten Rohrträger oder mit einem Flüssigkeitshandhabungsroboter in einem Kühlraum.
- Führen Sie mithilfe der Software des Flüssigkeitshandlers ein Programm aus, um 15 L jedes EPM und das Treuhandsystem in die vordefinierten Brunnen innerhalb der 384-Well-Quellplatte(n) zu übertragen.
- Pipetten PBS in alle ungenutzten Brunnen, um die Luftfeuchtigkeit zu erhöhen und vor Austrocknung während des Druckprozesses zu schützen.
HINWEIS: Siehe Abbildung 2 für ein Beispiel für ein 384-Well-Quellplattenset, das für einen 4 x 7-poligen Kopf optimiert ist und einen Kollagen-I-Block und PBS enthält. - Platten versiegeln und bei 4 °C halten, bis sie druckfertig sind.
4. Drucken von MEMAs mit einem Array-Druckroboter
HINWEIS: Im folgenden Teil des Protokolls wird speziell die Herstellung und Verwendung von MEMA beschrieben, um die Auswirkungen verschiedener Mikroumweltproteine auf das Wachstum und die Proliferation von MCF7-Zellen zu untersuchen. Das Protokoll kann jedoch leicht angepasst werden, um verschiedene Liganden, ECMs und Zellen zu verwenden, um andere Zelllinien und Endpunkte von Interesse zu untersuchen.
-
Drucken Sie mit einem Touch-Pin-Drucker EPMs und Fiducial-Spots in 8 Brunnenplatten. Drucken Sie mehrere Replikationen jeder ECM-Bedingung, um die Reproduzierbarkeit sicherzustellen.
HINWEIS: Andere Plattenformate oder Dias können für den Druck verwendet werden, aber Pufferoptimierung kann erforderlich sein, um eine optimale Punktbildung zu erreichen.- Drucken Sie die ECMs für den MEMA mit Pins mit einem Durchmesser von 350 m, die in einer 4 x 7 Druckkopfkonfiguration angeordnet sind. Drucken Sie die Arrays in den 8-Well-Platten als 20 Spalten in 35 Zeilen, für insgesamt 700 Punkte. Größere Arrays sind in diesen Platten möglich, haben aber einen Kompromiss von erhöhten Kanteneffekten sowohl in der Zellbindung als auch bei der Färbung.
- Nach dem Drucken platten mindestens 3 Tage vor gebrauchen in einem Trockenschrank lagern.
5. Erstellung von Ligand-Behandlungsplatten
- Entwerfen Sie ein 96-Well-Platten-Layout mit Liganden von Interesse. Um die Behandlung vieler MEMA-Platten auf einmal zu erleichtern, entwerfen Sie diese Platte mit einem Abstand, der die Verwendung einer Mehrkanalpipette mit 4 Abstandsspitzen ermöglicht, um Flüssigkeiten zwischen den Brunnen von 8-Well-MEMAs und einer 96-Well-Platte zu übertragen.
HINWEIS: In diesem Protokoll wird der vollständige Satz von Liganden verwendet, die in Tabelle 2 aufgeführt sind. - Liganden auf Eis auftauen. Kurz streichen und drehen Sie jede Röhre nach unten.
- Verdünnen Sie Liganden mit dem vom Hersteller empfohlenen Puffer (in der Regel PBS) auf einen 200-fachen Arbeitsbestand.
- Pipetten Sie 10 l von jedem 200x Ligand-Lager in den entsprechenden Brunnen innerhalb der 96-Well-Platte.
- Dichtungs- und Lagerplatten bei -20 °C.
HINWEIS: Erstellen Sie Liganden-Behandlungsplatten in Chargen und erfassen Sie alle Metadaten für die nachgelagerte Analyse.
6. Culturing Cells auf MEMAs
- Block MEMAs für 20 min mit 2 ml pro Brunnen des nicht-fouling Blockierpuffers mit 1% nicht-fouling Blockierungsmittel (Tabelle der Materialien) in doppeldestilliertem Wasser (ddH2O).
- Aspirieren Sie Blockierpuffer und dreifache Spülbrunnen mit PBS. Um die Austrocknung zu verhindern, lassen Sie das endgültige Volumen der PBS in Brunnen, bis sie für die Zellbeschichtung bereit sind.
HINWEIS: Es ist äußerst hilfreich, zwei Bankarbeiter für Zellkulturschritte auf MEMAs zu haben. Ein Bankarbeiter kann Aspirationsschritte ausführen, während der zweite Additionsschritte durchführt. Es wird empfohlen, eine 1 ml Mehrkanalpipette mit Spitzen zu verwenden, die der 8-Well-Platte für das Pipettieren entsprechen, und einen Y-Splitter mit zwei Pasteur Pipetten, um mehrere Brunnen gleichzeitig zu aspirieren. - Samen 2 x 105 MCF7-Zellen pro Brunnen in 2 ml des modifizierten Eagle-Mediums (DMEM) von Dulbecco, das 10% fetales Rinderserum (FBS) enthält.
HINWEIS: Führen Sie vor einem vollständigen MEMA-Experiment ein Zelltitrationsexperiment durch, um die Zellzahlen so zu optimieren, dass MEMA-Spots am Ende der gewünschten experimentellen Dauer hohe Zellzahlen (aber nicht konfluent) haben. - Nach 2-18 h Haftung, Aspiratmedium und ersetzen durch 2 ml wachstumsreduziertes Medium (DMEM mit 0,1% FBS).
HINWEIS: Reduziertes Serum (z. B. 0,1% FBS) oder wachstumsfaktorreduzierte Bedingungen können zu diesem Zeitpunkt verwendet werden, um die stimulierende Wirkung bestimmter Liganden zu isolieren. - Eine Ligandenbehandlungsplatte auf Eis auftauen. Zentrifuge aufgetautPlatte bei 200 x g für 1 min.
- 200 L Medium von jedem Brunnen in der Kulturplatte auf den entsprechenden Brunnen in der Behandlungsplatte übertragen. Nach oben und unten, um Ligandenvolumen mit Medium zu mischen und diese Mischung wieder auf den entsprechenden Brunnen in der MEMA-Platte zu übertragen.
- Leicht von Hand schaukeln und MEMA-Platten zum Inkubator zurückgeben. Kultur für die Dauer des Experiments in Gegenwart der Liganden/ECM-Kombinationbei 37 °C und 5% CO2 .
HINWEIS: Ein typisches MEMA-Experiment läuft 72 h; längerfristige Experimente können den Austausch von Medium und eine erneute Behandlung mit Ligand erfordern. - Pulsieren Sie MEMA-Bohrungen bei 71 h mit 100x 5-Ethynyl-2'-Deoxyuridin (EdU) für eine Endkonzentration von 10 m. Inkubieren unter experimentellen Bedingungen mit EdU für 1 h bei 37 °C und 5%CO2.
HINWEIS: Zu diesem Zeitpunkt können auch andere Live-Zell-Behandlungen angewendet werden.
7. Fixierung und Färbung von MEMAs
- Nach 72 h und alle Live-Zell-Behandlungen, Aspirieren Brunnen. Fix MEMAs in 2 ml pro Bohrung von 2% Paraformaldehyd (PFA) für 15 min bei RT.
- Aspirieren PFA. Permeabilisieren Sie mit 2 ml pro Brunnen von 0,1% nichtionischem Tensid für 15 min.
- Das nichtionische Tensid ansaugen und mit 2 ml pro PBS-Brunnen waschen. Aspirieren PBS. Mit 2 ml PBS mit 0,05% Polysorbat 20 (PBS-T) waschen.
HINWEIS: Die MEMA-Oberfläche ist hydrophob, und das Nichtwaschen mit PBS-T vor der Flecken- und Antikörperinkubation führt zur Bildung von Hohlräumen in Brunnen während der Inkubationsschritte und führt zu Färbungsartefakten. - Aspirate PBS-T. Fügen Sie EdU-Erkennungsreaktionsreagenzien hinzu. Inkubieren für 1 h bei RT, schaukeln und vor Licht geschützt. Nach 1 h Inkubation, Löschreaktion mit dem mitgelieferten kommerziellen Quench-Puffer.
HINWEIS: EdU-Erkennungs- und Färbe-/Antikörperschritte können in 1,5 ml pro Brunnen durchgeführt werden, um die Kosten zu senken. - Den Abschreckpuffer ansaugen und vor der Inkubation mit Flecken oder Antikörpern mit PBS-T waschen.
- MEMA-Brunnen mit Antikörpern gegen Histon H3K9me3 (1:1.000) und Fibrillarin (1:400) im Färbepuffer mit 2% (w/v) Rinderserumalbumin (BSA), 1 mM MgCl2 und 0,02% NaN3 über Nacht bei 4 °C inkubieren.
HINWEIS: Führen Sie Antikörpertitrationen durch, um optimale Konzentrationen zu bestimmen, bevor Sie sie auf einem vollständigen MEMA-Set verwenden. - Nach Primärantikörper oder Fleckeninkubation, waschen Sie Brunnen 2x mit PBS und einmal mit PBS-T.
- Sekundäre Antikörper (Esel-Anti-Maus-IgG und Esel-Anti-Kaninchen-IgG, beide 1:300) und 0,5 g/ml 4 x 6-Diamidino-2-Phenylindole (DAPI) hinzufügen. 1 h bei RT im Dunkeln inkubieren.
- Waschen Sie Brunnen 2x mit 2 ml pro Brunnen von PBS, so dass sie in den letzten 2 ml PBS.
- Gehen Sie zur Abbildung oder Lagerung von gebeizten MEMAs für die spätere Bildgebung in PBS bei 4 °C, die vor Licht geschützt sind.
8. Bildgebung von MEMAs
- Bild MEMA auf einem automatisierten Bildgebungssystem mit geeigneten Fluoreszenzdetektionskanälen.
- Ausgabe resultierender Bilddaten an ein Bildverwaltungssystem. Segmentieren von Zellen und berechnen Sie Intensitätsstufen mit CellProfiler8.
9. Datenanalyse
HINWEIS: Die Datenanalyse besteht aus Normalisierung, Variationskorrektur und Zusammenfassung der von CellProfiler abgeleiteten Rohdaten. In diesem Fall wird die R-Umgebung mit benutzerdefiniertem Code verwendet, um alle Schritte auszuführen. Jede statistische Umgebung oder jedes Softwareprogramm kann jedoch verwendet werden, um die entsprechenden Aktionen auszuführen. Ein Beispiel für den benutzerdefinierten Open-Source-Code für die R-Umgebung zur Analyse finden Sie unter: https://www.synapse.org/#!Synapse:syn2862345/wiki/72486.
- Vorverarbeiten und Normalisieren der segmentierten Bilddaten.
- Bestimmen Sie die Anzahl der Punktzellen mithilfe der DAPI-gebeizten Kerne.
- Auto-Gate EdU-Intensität, um Zellen als EdU+zu kennzeichnen. Messen Sie die Proliferation anhand des Anteils der EdU+ Zellen an jedem Punkt.
- Median fassen zytoplasmatische Flecken und Kernmorphologiemessungen auf Spot-Ebene zusammen.
- Entfernen der RUV-Normalisierung unerwünschter Variationen (RUV), um die Datenqualität zu verbessern9.
HINWEIS: Dieser Ansatz wird unabhängig voneinander auf jedes Intensitäts- und Morphologiesignal angewendet, als Matrix mit Arrays, die die Zeilen und Flecken als Spalten verwenden, wie zuvorbeschrieben 9. - Wenden Sie die bivariate Lössnormalisierung auf die normalisierten RUV-Residuen an, indem Sie die Arrayzeile und die Arrayspalte als unabhängige Variablen verwenden, um räumliche oder intensitätsbezogene Effekte zu korrigieren.
- Nach Abschluss der Normalisierung fassen Die Medianreplikationen für jede Mikroumgebungsbedingung für die Berichterstattung und weitere Analysen zusammen.
Ergebnisse
Um die Auswirkungen der Mikroumwelt auf das Zellwachstum und die Zellproliferation zu vereinfachen und Bedingungen zu identifizieren, die das Zellwachstum und die Zellproliferation förderten oder hemmten, wurde die Brustkrebszelllinie MCF7 auf einer Reihe von acht 8-Well-MEMAs gesät, wie im Protokoll beschrieben. Dieser Assay setzte die Zellen 48 verschiedenen EKMs und 57 verschiedenen Liganden für insgesamt 2736 kombinatorische mikroökologische Bedingungen aus. Nach 71 h in der Kultur wurden Zellen mit EdU gepulst, fixiert, permeablisiert und mit DAPI, der Reaktion zur EdU-Erkennung, einem Antifibrillin-Antikörper und einem Anti-H3K9me3-Antikörper gefärbt. Die Zellen wurden auf einem Mikroskop mit hohem Gehalt abgebildet. Die Bilder wurden auf einen Omero-Server10hochgeladen, mit CellProfiler8segmentiert und in R9normalisiert und analysiert. Die unten beschriebenen Ergebnisse konzentrieren sich auf die DAPI- und EdU-Signale.
Die Bildanalyseplattform von MEMAs liefert einige Ergebnisse, die denen von Flow-Zytometrie-Ansätzen ähneln, wie z. B. DNA-Inhaltsdiagramme, die 2N- und 4N-Fraktionen für Zellen zeigen, die mit einem gegebenen Liganden behandelt werden (Abbildung 3A), basierend auf der DAPI Intensität und Fläche. Diese Diagramme liefern Beweise für Bedingungen, die aktivezellradfahren im Vergleich zu fördern, wie durch klare bimodale Spitzen entsprechend Zellen in G1- oder G2-Phasen vs. Wachstums-verhafteten Zellen angezeigt, die Veränderungen in den Spitzen im Vergleich zu Kontrollbedingungen zeigen würden. Wir verwenden die Daten zur Zellzahl und Färbeintensität, um die Daten zusammenzufassen, wobei die Auswirkungen der Mikroumgebung (Liganden auf einer Achse, ECM auf der zweiten Achse) auf die Zellennummer (Abbildung3B) und die EdU-Integration (Abbildung 3C) kann leichter als Änderungen in der Wärmemapfarbe und -intensität gesehen werden. Aus diesen Diagrammen gesehen, sind viele der Effekte Liganden-gesteuert, da die ECM-Bedingung die Zellzahl oder EdU-Positivität nicht stark beeinflusste. Nidogen-1 ist eine klare Ausnahme, da das Vorhandensein dieses ECM-Moleküls die Zellbindung und das Wachstum von MCF7 hemmt. Liganden wie FGF6 und NRG1 (NRG1.1 auf Parzellen) erhöhen die Zellzahl und weisen hohe EdU-Einbauraten auf, während Liganden wie AREG und NRG1-smdf (NRG1.10 auf Parzellen) die Zellbindung und/oder das Wachstum von Zellen hemmen. Diese Ergebnisse werden durch die Bilder der Zellen unterstützt, die an den Stellen wachsen, wo ein deutlicher Unterschied in der Zellzahl und der EdU-Positivität erkennbar ist (siehe Beispiel in Abbildung 3D).
Da es sich bei der MEMA-Plattform um eine neuere Technologie handelt, wurden die Ergebnisse in separaten Assays validiert. MCF7-Zellen wurden in 24-Well-Platten mit Kollagen I in DMEM Medium mit 10% FBS beschichtet. Nach 18 h wurden Medien gegen ein reduziertes Wachstumsmedium (DMEM mit 0,1% FBS) ausgetauscht und Zellen mit NRG1, FGF6 oder AREG behandelt und für 72 h kultiviert. EdU wurde 1 h vor der Fixierung hinzugefügt. Zellen wurden mit DAPI und zur EdU-Einarbeitung befleckt, abgebildet, segmentiert und analysiert. Ähnlich wie die Ergebnisse der MEMA-Plattform führten sowohl FGF6 als auch NRG1 a zu höheren Zellzahlen (Abbildung 4A) und EdU-Gründungsraten (Abbildung 4B) im Vergleich zu AREG-behandelten Zellen, die unsere Beobachtungen in die ursprünglichen MEMA-Experimente.

Abbildung 1 : Flussdiagramm mit dem Workflow und der Zeitachse für die verschiedenen Phasen eines typischen MEMA-Experiments. Sobald die MEMAs gedruckt sind, können sie vor der Verwendung mehrere Monate bei Raumtemperatur gelagert werden. In der Regel dauert die experimentelle Phase 3 bis 4 Tage, aber einige langsam wachsende Primärzellen wurden auf MEMAs für bis zu 2 Wochen kultiviert. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.

Abbildung 2 : ECM-Quellplattenlayout für den Arraydruck. Der Kollagenblock wird als Gitter auf MEMA gedruckt, was eine sehr sich wiederholende Reihe von Bedingungen bietet, die eine robustere Normalisierung zwischen Brunnen ermöglichen. Die PBS-gefüllten Brunnen sorgen für Feuchtigkeit, um die Verdunstung während des Druckprozesses zu verhindern. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.

Abbildung 3 : Beispiele für Daten, die aus einem typischen MEMA-Experiment generiert wurden. (A) Zellzyklusprofile von binned DAPI-Intensitätswerten im Vergleich zu Zellzahlen aus einer 8-Well-Platte, die mit verschiedenen Liganden behandelt wird, was eine biphasische DAPI-Intensitätsfärbung zeigt, die Zellen in der G1-Zellenzyklusphase anzeigt. (B) Heatmap mit normalisierten Punktzellenzahlen, gruppiert nach Ähnlichkeit mithilfe hierarchischer Clustering. Rot gibt eine höhere Zellenzahl an, und blau ist die niedrigere Zellennummer. Liganden befinden sich auf der x-Achse, ECMs auf der y-Achse. (C) Heatmap mit normalisierter EdU-Eingemeindung, wobei rot auf höhere und blaue Angabe einer niedrigeren EdU-Einbauung hinweist. Liganden befinden sich auf der x-Achse, ECMs auf der y-Achse. (D) Beispiel für MCF7-Zellen, die an einem MEMA-Spot wachsen, der mit NRG1-- mit hohen EdU-Indukationsraten (rosa Kerne) behandelt wird. Grüner Fleck ist Zellmaske und blau ist DAPI. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
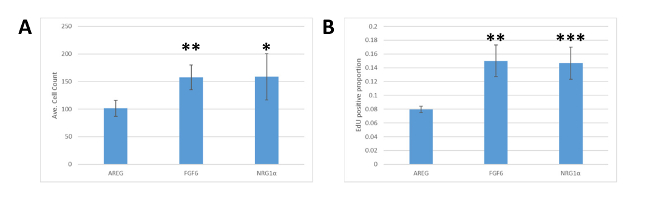
Abbildung 4 : Validierung von MEMA führt zu Zellkultur. (A) Quantifizierung der Zellzahl, die sich aus der Behandlung von MCF7 mit verschiedenen Liganden ergibt. Äquivalente Anzahl von MCF7-Zellen wurden in Mehrwandplatten plattiert und dann entweder mit AREG, FGF6 oder NRG1 behandelt. Brunnen, die mit AREG behandelt wurden, hatten deutlich weniger Zellen als mit FGF6 (** mit Angabe der t-Test-p-Werte des Schülers unter 0,01) oder NRG1 (* gibt einen p-Wert von 0,05) bei 72 h nach Ligandenbehandlung an. (B) Quantifizierung des Niveaus der EdU-Einbindung in MCF7 aufgrund der Behandlung mit verschiedenen Liganden, wie in Panel A. Die AREG-Behandlung führt zu einem deutlich geringeren Anteil von Zellen, die EdU enthalten, als Zellen, die mit FGF6 (**, p < 0,01) oder NRG1 (***, p = 0,01) behandelt wurden. Fehlerbalken stellen eine Standardabweichung dar. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
| Proteinname | Uniprot ID | vorrat konzentration (g/ml) | Finale konzentration (g/ml) |
| ANGPT1|1 | Q15389|1 | 100 | 0,04 |
| ANGPT2|1 | O15123|1 | 100 | 0,2 |
| AREG | P15514 | 100 | 0,02 |
| BMP2 | P12643 | 100 | 0,1 |
| BMP3 | P12645 | 1000 | 0,1 |
| BMP4 | P12644 | 100 | 0,1 |
| BMP5|1 | P22003|1 | 100 | 0,1 |
| BMP6 | P22004 | 100 | 0,1 |
| BMP7 | P18075 | 100 | 0,1 |
| CSF2 | P04141 | 100 | 0,02 |
| CTGF|1 | P29279|1 | 100 | 0,05 |
| CXCL12| alpha | P48061|2 | 100 | 0,01 |
| CXCL12| Beta | P48061|1 | 100 | 0,03 |
| CXCL1 | P09341 | 100 | 0,004 |
| CXCL8|1 | P10145|1 | 100 | 0,3 |
| DLL1|1 | O00548|1 | 500 | 0,5 |
| DLL4 | Q9NR61 | 200 | 0,6 |
| EGF|1 | P01133|1 | 500 | 0,01 |
| FASLG|1 | P48023|1 | 10 | 0,02 |
| FGF2|3 | P09038|2 | 100 | 0,01 |
| FGF6 | P10767 | 100 | 0,01 |
| FLT3LG|1 | P49771|1 | 50 | 0,001 |
| GPNMB|1 | Q14956|1 | 100 | 0,5 |
| HGF|1 | P14210|1 | 50 | 0,04 |
| IGF1|1 | P05019|1 | 200 | 0,01 |
| IGFBP2 | P18065 | 100 | 0,05 |
| IGFBP3|1 | P17936|1 | 100 | 0,1 |
| IL13 | P35225 | 100 | 0,01 |
| IL15| IL15S48AA | P40933|1 | 50 | 0,01 |
| IL1B | P01584 | 25 | 0,001 |
| IL6 | P05231 | 100 | 0,01 |
| IL7|1 | P13232|1 | 100 | 0,01 |
| JAG1|1 | P78504|1 | 200 | 0,5 |
| JAG2| lang | Q9Y219|1 | 100 | 0,5 |
| KITLG|1 | P21583|1 | 100 | 0,005 |
| KNG1 | HMW | P01042|1 | 100 | 0,2 |
| Lep | P41159 | 1000 | 0,002 |
| LYVE1 | Q9Y5Y7 | 100 | 0,05 |
| NRG1|10 | Q02297|10 | 100 | 0,01 |
| NRG1|1 | Q02297|1 | 100 | 0,05 |
| NRG1|6 | Q02297|6 | 100 | 0,01 |
| PDGFAB | go1990265 | 100 | 0,05 |
| PDGFB|1 | P01127|1 | 100 | 0,05 |
| Ptn | P21246 | 100 | 0,5 |
| Shh | Q15465 | 100 | 0,5 |
| TGFB1|| Cterminus | P01137| Cterminus | 20 | 0,01 |
| TGFB1|| auflecken | P01137| auflecken | 100 | 0,15 |
| TGFB2| pro | P61812|1 | 20 | 0,01 |
| THPO|1 | P40225|1 | 50 | 0,002 |
| TNFRSF11B | O00300 | 100 | 0,02 |
| TNFSF11|1 | O14788|1 | 100 | 0,01 |
| Tnf | P01375 | 100 | 0,01 |
| VEGFA| VEGF206 | P15692|1 | 100 | 0,01 |
| WNT10A | Q9GZT5 | 100 | 0,1 |
| WNT3A|1 | P56704|1 | 200 | 0,1 |
| Wnt5a|1 | P22725|1 | 100 | 0,1 |
Tabelle 1: Die vollständige Liste der Liganden, die für die MEMA-Experimente verwendet werden. Die Uniprot-ID, die Lagerkonzentrationen und die endlichen Arbeitskonzentrationen werden bereitgestellt.
| ECM Protein | UniprotID | Lagerkonzentration (g/ml) | Finale konzentration (g/ml) | Notizen |
| ALCAM|1 | Q13740|1 | 100 | 30 | |
| CDH20 | Q9HBT6 | 300 | 80 | |
| CDH6|1 | P55285|1 | 100 | 40 | |
| CDH8 | P55286 | 100 | 20 | |
| CD44|1 | P16070|1 | 100 | 30 | |
| CEACAM6 | P40199 | 100 | 30 | |
| COL1A1 | P02453 | 5000 | 200 | mehrere Untereinheiten mit mehreren Uniprot-IDs |
| COL2A1|2 | P02458|2 | 1000 | 200 | |
| COL3A1|1 | P02461|1 | 1000 | 200 | |
| COL4A1|1 | P02462|1 | 1000 | 200 | mehrere Untereinheiten mit mehreren Uniprot-IDs |
| COL5A1 | P20908 | 1000 | 200 | |
| COL23A1|1 | Q86Y22|1 | 200 | 80 | |
| DSG2 | Q14126 | 100 | 30 | |
| CDH1|1 | P12830|1 | 100 | 40 | |
| ECM1|1 | Q16610|1 | 100 | 40 | |
| FN1|1 | P02751|1 | 1000 | 200 | |
| GAP43|1 | P17677|1 | 158 | 40 | |
| Hya-500K | 1000 | 200 | LOR-0005 | |
| Hya-50K | 1000 | 200 | LOR-0007 | |
| ICAM1 | P05362 | 400 | 80 | |
| ALCAM|1 | Q13740|1 | 100 | 30 | |
| CDH20 | Q9HBT6 | 300 | 80 | |
| CDH8 | P55286 | 100 | 20 | |
| CD44|1 | P16070|1 | 100 | 30 | |
| CEACAM6 | P40199 | 100 | 30 | |
| DSG2 | Q14126 | 100 | 30 | |
| CDH15 | P55291 | 100 | 20 | |
| VCAM1|1 | P19320|1 | 1000 | 200 | |
| LAMA1 | P25391 | 500 | 200 | mehrere Untereinheiten mit mehreren Uniprot-IDs |
| LAMA3|2 | Q16787|2 | 130 | 40 | |
| Lum | P51884 | 200 | 80 | |
| CDH15 | P55291 | 100 | 20 | |
| NID1|1 | P14543|1 | 100 | 9,3 g/ml Nid, 130 g/ml Lam, 46,5 g/ml COL4 | +COL4 und Laminin |
| Omd | Q99983 | 100 | 40 | |
| SPP1| pro | P10451|1 | 100 | 40 | |
| CDH3|1 | P22223|1 | 100 | 40 | |
| PECAM1| lang | P16284|1 | 150 | 40 | |
| TNC|1 | P24821|1 | 500 | 200 | |
| VCAM1|1 | P19320|1 | 1000 | 200 | |
| Vtn | P04004 | 100 | 40 | |
| Bgn | P21810 | 100 | 40 | |
| DCN| pro | P07585|1 | 300 | 80 | |
| POSTN|1 | Q15063|1 | 100 | 40 | |
| Sparc | P09486 | 100 | 40 | |
| THBS1|1 | P07996|1 | 100 | 40 | |
| BCAN|1 | Q96GW7|1 | 100 | 40 | |
| ELN|3 | P15502|3 | 1000 | 200 | |
| FBN1 | P35555 | 254 | 80 |
Tabelle 2: Die vollständige Liste der ECM-Proteine und -Bedingungen, die in den MEMA-Experimenten verwendet werden. Die Uniprot-ID, die Lagerkonzentrationen und die endlichen Arbeitskonzentrationen werden bereitgestellt. In einigen Fällen stellt der gedruckte Zustand einen Proteinkomplex oder eine Kombination mehrerer Proteine dar, die in der Spalte Notizen angegeben ist.
Diskussion
Die Bedeutung von "Dimensionalität" und Kontext war ein motivierender Faktor bei der Entwicklung von In-vitro-Kultursystemen als Werkzeuge für die Charakterisierung von Krebszellen durch ihre Interaktion mit der Mikroumgebung11und die Fähigkeit von In-vitro-Kultursystemen Kultursysteme, um die in vivo-Umgebung nachzuahmen, sind eine treibende Kraft hinter dem Streben, diese Kultursysteme zu verbessern. In-vitro-Systeme bleiben jedoch wichtige Instrumente der Krebsforschung gerade wegen ihrer Fähigkeit, die komplexe in vivo-Situation bis auf ein vereinfachtes Modell zu destillieren12.
Obwohl 2D-Systeme ECMs und Liganden enthalten können, fehlten ihnen traditionell die Durchsatzkapazitäten, um eine breite Palette kombinatorischer Pertubagen abzuhören. Beliebte kommerzielle Kellermembran-Extrakte ermöglichen die Kultivierung in 3D, aber es fehlt die Provenienz eines sorgfältig definierten Panels von Proteinen. Die kommerziellen Extrakte leiden in der Regel unter unvollständig definierter Zusammensetzung, die die Analyse verwirren und zu einer signifikanten Charge-zu-Charge-Variation3,13führen kann. Die MEMA-Plattform überwindet diese Barrieren und ermöglicht die Untersuchung von Veränderungen bei zellulären Phänotypen, metabolischer Aktivität, Differenzierungsstatus und Variationen des Zellwachstums und der Zellproliferation, da sie durch spezifische und definierte endogene Faktoren.
Die MEMA-Plattform ist ein leistungsstarker Ansatz mit mittlerem bis hohem Durchsatz, um die Auswirkungen der Mikroumgebung (sowohl ECM als auch lösliche Faktoren) auf den Phänotyp von Zellen zu bewerten. Die Plattform zeigt große Flexibilität für die Arten von Assays und Zellen, für die sie genutzt werden kann. Wir können Effekte sowohl von löslichen Liganden als auch von den ECM-Proteinen beobachten, denen die Zellen ausgesetzt sind. In der Tat haben wir vor kurzem festgestellt, dass Liganden ein wichtiger Treiber für die Resistenz gegen HER2-gezielte Inhibitoren waren, aber dass diese Effekte durch das ECM5moduliert werden könnten. Eine Vielzahl von Zellen, einschließlich Primärzellen und Zelllinien, die aus verschiedenen Zelltypen wie Lunge, Blase, Prostata, Brust und Bauchspeicheldrüse sowie induzierten pluripotenten Stammzellen (iPS) stammen, wurden erfolgreich auf der MEMA-Plattform kultiviert (siehe Beispiele in Referenzen5,7,14). Die Verwendung verschiedener Flecken ermöglicht das Auslesen mehrerer zellulärer Endpunkte, einschließlich Zellwachstum, Differenzierung und Stoffwechsel. Andere Forscher haben die Plattform erweitert, um die Auswirkungen von Steifigkeit oder elastischem Modul zu behören, was der MEMA-Plattform15eine zusätzliche Dimension hinzufügt. Schließlich ist die Plattform für die Durchführung von Medikamenten-Screens zur Identifizierung von Mikroumgebungsbedingungen, die entweder verbessern oder hemmen Die Wirksamkeit von Medikamenten, wie wir und andere vor kurzem berichtet haben5,14,15 .
Der vielleicht wichtigste Schritt zum Erfolg eines MEMA-Experiments ist die Optimierung der Zellbeschichtungsdichte. Die Optimierung der Dichte der Zellen stellt sicher, dass genügend Zellen vorhanden sind, um robuste Daten bereitzustellen, aber nicht so viele, dass der Spot übermäßig konfluent wird. Konfluente Flecken können die Ergebnisse erheblich verwirren, insbesondere wenn die Proliferation als Endpunkt verwendet wird, was es unmöglich macht, festzustellen, ob niedrige Proliferationsraten auf Wechselwirkungen mit Mikroumweltfaktoren oder auf Kontakthemmungen durch hohe Zelldichte. Zelltitrationsexperimente können diese Probleme aufdecken, da die durchschnittlichen Zellzahlen pro Punkt eine lineare Zunahme mit einer zunehmenden Anzahl von zellenbeschichteten zeigen, aber schließlich Plateau. Die optimale Zellenzahl sollte im linearen Bereich der Kurve gewählt werden.
Wie bereits erwähnt, ist die MEMA-Plattform flexibel und kann auf einer Vielzahl von Substraten mit unterschiedlichen Oberflächen hergestellt werden. Dazu gehören Glasschlitten und Multiwall-Plattenformate. Nach unserer Erfahrung sind nicht alle Oberflächenchemikalien für den MEMA-Druck zugänglich, da wir auf einigen Oberflächen aufgrund schlechter Haftungseigenschaften und der Unfähigkeit, die Zellhaftung auf anderen hohen Haftungsflächen zu blockieren, eine Punktablösung beobachtet haben. Darüber hinaus erfordert der Wechsel zwischen verschiedenen Substraten die Optimierung der Pufferbedingungen, da die Leistung des Drucks mit demselben Druckpuffer je nach Oberflächenchemie variieren kann.
Der Durchmesser der gedruckten ECM-Spots spielt eine wichtige Rolle bei der Qualität der Daten. Im Allgemeinen empfehlen wir die Verwendung der druckbaren Stifte mit dem größten Durchmesser, die für den im Einsatz befindlichen Arrayer verfügbar sind (wir verwenden derzeit Pins mit einem Durchmesser von 350 m). Größere Durchmesser-Spots ermöglichen es einer größeren Anzahl von Zellen, einen Punkt zu besetzen, was tendenziell zu robusteren Daten führt, als mit Pins mit kleinerem Durchmesser erzeugt werden. Da die Bindung der Zellen ein stochastischer Prozess ist, gibt es in der Regel einen hohen Grad an Variabilität in den Daten, die mit der Anzahl der Zellen zusammenhängen, die ursprünglich an jeden Punkt angehängt wurden. Daher empfehlen wir, eine große Anzahl von Replikationen für jede ECM-Bedingung zu drucken. Wir drucken 10-15 ECM-Replikationen in jedem Brunnen mit unseren aktuellen Druckbedingungen, um robuste Statistiken zu gewährleisten.
Wir haben in unseren früheren Experimenten festgestellt, dass ligand-Effekte zum größten Teil über ECM-Effekte dominieren. Dies kann zum Teil auf unsere Entscheidung zurückzuführen sein, Kollagen I zu allen ECM-Spots hinzuzufügen, was eine robuste Zellbindung gewährleistet. Wir glauben jedoch, dass dies auch die ECM-Effekte homogenisieren kann, da die meisten Flecken dazu neigen, sich in einer Weise zu verhalten, die kollagen I sehr ähnlich ist. Die Änderung der Spotzusammensetzung, um Kollagen auszuschließen, kann zu einem differentialen Zellverhalten als Folge der Interaktion mit dem ECM, wirkt sich aber auch signifikant auf die Zellbindung aus, was zu viel mehr unbesetzten Stellen führt. Benutzer sollten ihre ECM-Zusammensetzung unter Berücksichtigung dieser Unterschiede anpassen, insbesondere diejenigen, die an Stamm- und Vorläuferzellen und Differenzierung interessiert sind, wobei die Matrix eine signifikante Wirkung haben kann16.
Wir führen die MEMA-Assays in der Regel für relativ kurze Zeiträume (z. B. maximal 72 h) durch. Dies liegt daran, dass die Zellen auf die Flecken beschränkt sind (der Blockierpuffer lässt kein Wachstum außerhalb der Punkte unserer Erfahrung zu). Bei schnell teilenden Zellen führt ein Wachstum von mehr als 72 h zu einem Überwuchern des Spots, was wiederum die Bildsegmentierung erschwert, wenn zellen überfüllt werden und sich aneinander anhäufen, und kann sich auch auf Daten auswirken, da Wachstumsstillstand mit Kontakthemmung auftreten kann. Wir haben längere Behandlungen mit sehr langsam wachsenden Primärzellen durchgeführt (10 bis 14 Tage), aber in diesen Assays muss darauf geachtet werden, die Medien zu verändern und Liganden alle 3 bis 4 Tage aufzufüllen.
Die kontinuierlichen Bemühungen um die Entwicklung der MEMA-Plattform konzentrieren sich auf zwei Interessensbereiche, die Maximierung der optischen Qualität für die Bildgebung und Optimierung in kleineren Kulturgefäßen. Optische Qualität wird ein entscheidender Faktor, wenn Forscher eine Mikroskopie mit höherer Auflösung benötigen, um die subzelluläre Lokalisierung ihrer Marker von Interesse zu identifizieren. Erste Bildschirme können mit niedrigerer Auflösung an Hochdurchsatzmikroskopen durchgeführt werden, gefolgt von der Abbildung bestimmter Sehenswürdigkeiten auf Instrumenten mit höherer Auflösung, aber die Bildqualität kann beeinträchtigt werden, wenn die optischen Eigenschaften des Substrats schlecht sind. Eine Verbesserung der optischen Eigenschaften des Substrats würde es den Forschern ermöglichen, die ersten Bildschirme auf hochauflösenden Bildgebungssystemen durchzuführen, ohne ausgewählte Bilder mit höherer Auflösung wieder erfassen zu müssen. Schließlich würde die Fähigkeit, MEMAs in kleineren Kulturgefäßen wie 96-Well-Platten durchzuführen, eine Verringerung des Behandlungsvolumens und eine Erweiterung der verhörten Liganden und Replikationen ermöglichen. Dieser Übergang erfordert die Optimierung von Substrat-Puffer-Protein-Wechselwirkungen und Arraydruck in neuen Kulturgefäßen. Solche laufenden Bemühungen werden die MEMA-Plattform verbessern und ihre leistungsstarken Fähigkeiten erweitern, um relevante mikroökologische Proteine zu identifizieren, die zelluläre Phänotypen für eine Vielzahl von Zelltypen verändern, die dann anschließend in Bestätigende Assays.
Offenlegungen
Die Autoren haben nichts zu verraten.
Danksagungen
Diese Arbeit wurde von der NIH Common Fund Library of Network Cellular Signatures (LINCS) gefördert, die HG008100 (J.W.G., L.M.H. und J.E.K.) gewährte.
Materialien
| Name | Company | Catalog Number | Comments |
| Aushon 2470 | Aushon BioSystems | Arrayer robot system used in the protocol | |
| Nikon HCA | Nikon | High Content Imaging system designed around Nikon Eclipse Ti Inverted Microscope | |
| BioTek Precision XS liquid Handler | BioTek | liquid handling robot used in the protocol | |
| Trizma hydrochloride buffer solution | Sigma | T2069 | |
| EDTA | Invitrogen | 15575-038 | |
| Glycerol | Sigma | G5516 | |
| Triton X100 | Sigma | T9284 | |
| Tween 20 | Sigma | P7949 | |
| Kolliphor P338 | BASF | 50424591 | |
| 384-well microarray plate, cylindrical well | Thermo Fisher | ab1055 | |
| Nunc 8 well dish | Thermo Fisher | 267062 | |
| Paraformaldehyde 16% solution | Electron Microscopy Science | 15710 | |
| BSA | Fisher | BP-1600 | |
| Sodium Azide | Sigma | S2002 | |
| Cell Mask | Molecular Probes | H32713 | |
| Click-iTEdU Alexa Fluor | Molecular Probes | C10357 | |
| DAPI | Promo Kine | PK-CA70740043 | |
| ALCAM | R & D Systems | 656-AL | ECM |
| Cadherin-20 (CDH20) | R & D Systems | 5604-CA | ECM |
| Cadherin-6 (CDH6) | R & D Systems | 2715-CA | ECM |
| Cadherin-8 (CDH8) | R & D Systems | 188-C8 | ECM |
| CD44 | R & D Systems | 3660-CD | ECM |
| CEACAM6 | R & D Systems | 3934-CM | ECM |
| Collagen I | Cultrex | 3442-050-01 | ECM |
| Collagen Type II | Millipore | CC052 | ECM |
| Collagen Type III | Millipore | CC054 | ECM |
| Collagen Type IV | Sigma | C5533 | ECM |
| Collagen Type V | Millipore | CC077 | ECM |
| COL23A1 | R & D Systems | 4165-CL | ECM |
| Desmoglein 2 | R & D Systems | 947-DM | ECM |
| E-cadherin (CDH1) | R & D Systems | 648-EC | ECM |
| ECM1 | R & D Systems | 3937-EC | ECM |
| Fibronectin | R & D Systems | 1918-FN | ECM |
| GAP43 | Abcam | ab114188 | ECM |
| HyA-500K | R & D Systems | GLR002 | ECM |
| HyA-50K | R & D Systems | GLR001 | ECM |
| ICAM-1 | R & D Systems | 720-IC | ECM |
| Laminin | Sigma | L6274 | ECM |
| Laminin-5 | Abcam | ab42326 | ECM |
| Lumican | R & D Systems | 2846-LU | ECM |
| M-Cad (CDH15) | R & D Systems | 4096-MC | ECM |
| Nidogen-1 | R & D Systems | 2570-ND | ECM |
| Osteoadherin/OSAD | R & D Systems | 2884-AD | ECM |
| Osteopontin (SPP) | R & D Systems | 1433-OP | ECM |
| P-Cadherin (CDH3) | R & D Systems | 861-PC | ECM |
| PECAM1 | R & D Systems | ADP6 | ECM |
| Tenascin C | R & D Systems | 3358-TC | ECM |
| VCAM1 | R & D Systems | ADP5 | ECM |
| Vitronectin | R & D Systems | 2308-VN | ECM |
| Biglycan | R & D Systems | 2667-CM | ECM |
| Decorin | R & D Systems | 143-DE | ECM |
| Periostin | R & D Systems | 3548-F2 | ECM |
| SPARC/osteonectin | R & D Systems | 941-SP | ECM |
| Thrombospondin-1/2 | R & D Systems | 3074-TH | ECM |
| Brevican | R & D Systems | 4009-BC | ECM |
| Elastin | BioMatrix | 5052 | ECM |
| Fibrillin | Lynn Sakai Lab OHSU | N/A | ECM |
| ANGPT2 | RnD_Systems_Own | 623-AN-025 | Ligand |
| IL1B | RnD_Systems_Own | 201-LB-005 | Ligand |
| CXCL8 | RnD_Systems_Own | 208-IL-010 | Ligand |
| IGF1 | RnD_Systems_Own | 291-G1-200 | Ligand |
| TNFRSF11B | RnD_Systems_Own | 185-OS | Ligand |
| BMP6 | RnD_Systems_Own | 507-BP-020 | Ligand |
| FLT3LG | RnD_Systems_Own | 308-FK-005 | Ligand |
| CXCL1 | RnD_Systems_Own | 275-GR-010 | Ligand |
| DLL4 | RnD_Systems_Own | 1506-D4-050 | Ligand |
| HGF | RnD_Systems_Own | 294-HGN-005 | Ligand |
| Wnt5a | RnD_Systems_Own | 645-WN-010 | Ligand |
| CTGF | Life_Technologies_Own | PHG0286 | Ligand |
| LEP | RnD_Systems_Own | 398-LP-01M | Ligand |
| FGF2 | Sigma_Aldrich_Own | SRP4037-50UG | Ligand |
| FGF6 | RnD_Systems_Own | 238-F6 | Ligand |
| IL7 | RnD_Systems_Own | 207-IL-005 | Ligand |
| TGFB1 | RnD_Systems_Own | 246-LP-025 | Ligand |
| PDGFB | RnD_Systems_Own | 220-BB-010 | Ligand |
| WNT10A | Genemed_Own | 90009 | Ligand |
| PTN | RnD_Systems_Own | 252-PL-050 | Ligand |
| BMP3 | RnD_Systems_Own | 113-BP-100 | Ligand |
| BMP4 | RnD_Systems_Own | 314-BP-010 | Ligand |
| TNFSF11 | RnD_Systems_Own | 390-TN-010 | Ligand |
| CSF2 | RnD_Systems_Own | 215-GM-010 | Ligand |
| BMP5 | RnD_Systems_Own | 615-BMC-020 | Ligand |
| DLL1 | RnD_Systems_Own | 1818-DL-050 | Ligand |
| NRG1 | RnD_Systems_Own | 296-HR-050 | Ligand |
| KNG1 | RnD_Systems_Own | 1569-PI-010 | Ligand |
| GPNMB | RnD_Systems_Own | 2550-AC-050 | Ligand |
| CXCL12 | RnD_Systems_Own | 350-NS-010 | Ligand |
| IL15 | RnD_Systems_Own | 247-ILB-005 | Ligand |
| TNF | RnD_Systems_Own | 210-TA-020 | Ligand |
| IGFBP3 | RnD_Systems_Own | 675-B3-025 | Ligand |
| WNT3A | RnD_Systems_Own | 5036-WNP-010 | Ligand |
| PDGFAB | RnD_Systems_Own | 222-AB | Ligand |
| AREG | RnD_Systems_Own | 262-AR-100 | Ligand |
| JAG1 | RnD_Systems_Own | 1277-JG-050 | Ligand |
| BMP7 | RnD_Systems_Own | 354-BP-010 | Ligand |
| TGFB2 | RnD_Systems_Own | 302-B2-010 | Ligand |
| VEGFA | RnD_Systems_Own | 293-VE-010 | Ligand |
| IL6 | RnD_Systems_Own | 206-IL-010 | Ligand |
| CXCL12 | RnD_Systems_Own | 351-FS-010 | Ligand |
| NRG1 | RnD_Systems_Own | 378-SM | Ligand |
| IGFBP2 | RnD_Systems_Own | 674-B2-025 | Ligand |
| SHH | RnD_Systems_Own | 1314-SH-025 | Ligand |
| FASLG | RnD_Systems_Own | 126-FL-010 | Ligand |
Referenzen
- Hanahan, D., Coussens, L. M. Accessories to the crime: functions of cells recruited to the tumor microenvironment. Cancer Cell. 21 (3), 309-322 (2012).
- Quail, D. F., Joyce, J. A. Microenvironmental regulation of tumor progression and metastasis. Nature Medicine. 19 (11), 1423-1437 (2013).
- Hughes, C. S., Postovit, L. M., Lajoie, G. A. Matrigel: a complex protein mixture required for optimal growth of cell culture. Proteomics. 10 (9), 1886-1890 (2010).
- LaBarge, M. A., et al. Human mammary progenitor cell fate decisions are products of interactions with combinatorial microenvironments. Integrative Biology (Cambridge). 1 (1), 70-79 (2009).
- Watson, S. S., et al. Microenvironment-Mediated Mechanisms of Resistance to HER2 Inhibitors Differ between HER2+ Breast Cancer Subtypes. Cell Systems. 6 (3), 329-342 (2018).
- Ranga, A., et al. 3D niche microarrays for systems-level analyses of cell fate. Nature Communications. 5, 4324 (2014).
- Malta, D. F. B., et al. Extracellular matrix microarrays to study inductive signaling for endoderm specification. Acta Biomater. 34, 30-40 (2016).
- Kamentsky, L., et al. Improved structure, function and compatibility for CellProfiler: modular high-throughput image analysis software. Bioinformatics. 27 (8), 1179-1180 (2011).
- Gagnon-Bartsch, J. A., Jacob, L., Speed, T. P. Removing Unwanted Variation from High Dimensional Data with Negative Controls. University of California, Berkeley, Department of Statistics, University of California, Berkeley. , (2013).
- Allan, C., et al. OMERO: flexible, model-driven data management for experimental biology. Nature Methods. 9 (3), 245-253 (2012).
- Simian, M., Bissell, M. J. Organoids: A historical perspective of thinking in three dimensions. Journal of Cell Biology. 216 (1), 31-40 (2017).
- Bissell, M. J. The differentiated state of normal and malignant cells or how to define a "normal" cell in culture. International Review of Cytology. 70, 27-100 (1981).
- Serban, M. A., Prestwich, G. D. Modular extracellular matrices: solutions for the puzzle. Methods. 45 (1), 93-98 (2008).
- Kaylan, K. B., et al. Mapping lung tumor cell drug responses as a function of matrix context and genotype using cell microarrays. Integrative Biology (Cambridge). 8 (12), 1221-1231 (2016).
- Lin, C. H., Jokela, T., Gray, J., LaBarge, M. A. Combinatorial Microenvironments Impose a Continuum of Cellular Responses to a Single Pathway-Targeted Anti-cancer Compound. Cell Reports. 21 (2), 533-545 (2017).
- Gjorevski, N., et al. Designer matrices for intestinal stem cell and organoid culture. Nature. 539 (7630), 560-564 (2016).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenWeitere Artikel entdecken
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten