Method Article
Utilisation de microréseaux pour interroger l'impact microenvironnemental sur les phénotypes cellulaires dans le cancer
Dans cet article
Résumé
Le but de la méthode présentée ici est de montrer comment les microréseaux microenvironnements (MEMA) peuvent être fabriqués et utilisés pour interroger l'impact de milliers de microenvironnements combinatoires simples sur le phénotype des cellules cultivées.
Résumé
Comprendre l'impact du microenvironnement sur le phénotype des cellules est un problème difficile en raison du mélange complexe de facteurs de croissance solubles et de protéines associées à la matrice dans le microenvironnement in vivo. En outre, les réactifs facilement disponibles pour la modélisation des microenvironnements in vitro utilisent généralement des mélanges complexes de protéines qui sont incomplètement définis et souffrent de la variabilité de lot à lot. La plate-forme microenvironnement microréseau (MEMA) permet d'évaluer des milliers de combinaisons simples de protéines microenvironnement pour leur impact sur les phénotypes cellulaires en un seul test. Les MEMA sont préparés dans des plaques de puits, ce qui permet l'ajout de ligands individuels pour séparer les puits contenant des protéines de matrice extracellulaire (ECM). La combinaison du ligand soluble avec chaque ECM imprimé forme une combinaison unique. Un analyse meMA typique contient plus de 2 500 microenvironnements combinatoires uniques auxquels les cellules sont exposées en un seul analyse. Comme cas d'essai, la ligne de cellules de cancer du sein MCF7 a été plaquée sur la plate-forme de MEMA. L'analyse de cet analyse a identifié des facteurs qui améliorent et inhibent la croissance et la prolifération de ces cellules. La plate-forme MEMA est très flexible et peut être étendue pour une utilisation avec d'autres questions biologiques au-delà de la recherche sur le cancer.
Introduction
La culture des lignées de cellules cancéreuses sur le plastique dans les monocouches bidimensionnelles (2D) demeure l'un des principaux chevaux de bataille pour les chercheurs sur le cancer. Cependant, le microenvironnement est de plus en plus reconnu pour sa capacité à avoir un impact sur les phénotypes cellulaires. Dans le cancer, le microenvironnement de tumeur est connu pour influencer les comportements cellulaires multiples, y compris la croissance, la survie, l'invasion, et la réponse à la thérapie1,2. Les cultures traditionnelles des cellules monocouches n'ont généralement pas d'influences microenvironnement, ce qui a conduit au développement d'essais tridimensionnels (3D) plus complexes pour faire pousser des cellules, y compris des extraits de membrane de sous-sol purifiés disponibles dans le commerce. Cependant, ces matrices purifiées sont généralement compliquées à utiliser et souffrent de problèmes techniques tels que la variabilité des lots3 et les compositions complexes3. En conséquence, il peut être difficile d'attribuer la fonction à des protéines spécifiques qui peuvent avoir un impact sur les phénotypes cellulaires3.
Pour remédier à ces limitations, nous avons développé la technologie microenvironnement micro-réseau (MEMA), qui réduit le microenvironnement à de simples combinaisons de matrice extracellulaire (ECM) et de protéines solubles de facteur de croissance4,5 . La plate-forme MEMA permet d'identifier les facteurs microenvironnementaux dominants qui ont un impact sur le comportement des cellules. En utilisant un format de tableau, des milliers de combinaisons de facteurs microenvironnement peuvent être astoyées en une seule expérience. Le MEMA décrit ici interroge 2 500 conditions microenvironnement uniques différentes. Les protéines ECM imprimées dans des plaques de puits forment des plaquettes de croissance sur lesquelles les cellules peuvent être cultivées. Des ligands solubles sont ajoutés à des puits individuels, créant des microenvironnements combinatoires uniques (ECM et ligand) à chaque endroit différent auquel les cellules sont exposées. Les cellules sont cultivées pendant plusieurs jours, puis fixes, tachées et représentées pour évaluer les phénotypes cellulaires à la suite de l'exposition à ces combinaisons microenvironnement spécifiques. Étant donné que les microenvironnements sont des combinaisons simples, il est facile d'identifier les protéines qui entraînent des changements phénotypiques majeurs dans les cellules. Les MEMA ont été utilisés avec succès pour identifier les facteurs qui influencent plusieurs phénotypes cellulaires, y compris ceux qui conduisent les décisions de destin cellulaire et la réponse à la thérapie4,5,6,7. Ces réponses peuvent être validées dans des expériences 2D simples et peuvent ensuite être évaluées dans des conditions qui récapitulent plus complètement la complexité du microenvironnement tumoral. La plate-forme MEMA est très adaptable à une variété de types de cellules et de points de terminaison, à condition que de bons biomarqueurs phénotypiques soient disponibles.
Protocole
REMARQUE: Un aperçu de l'ensemble du processus MEMA, y compris le temps estimé, est décrit dans le diagramme de débit indiqué à la figure 1. Ce protocole détaille la fabrication des MEMA dans des plaques de 8 puits. Le protocole peut être adapté pour d'autres plaques ou diapositives.
1. Préparation de tampons de protéines, de diluants et de taches
- Équilibrez les flacons d'ECM, de ligands et de cytokines à température ambiante (RT) et brièvement centrifugeuse. Ajouter le volume approprié de la mémoire tampon RT appropriée indiquée sur la feuille de données du produit. Suivez la recommandation du fabricant concernant les concentrations de stocks.
REMARQUE: Une liste complète des ligands et des ECM avec leur stock et leurs concentrations finales sont fournies dans le tableau 1 et le tableau 2. Les ligands et les ECM sont généralement utilisés à la plus forte concentration de la gamme recommandée par le fabricant qui provoque un effet biologique dans les essais de culture standard de 2 jours. Manipulez les protéines en douceur et dans des armoires de biosécurité sous un débit laminaire afin d'éviter la contamination. - Incuber les flacons avec un basculement doux à RT pendant 1 h. Ne pas vortex protéines car cela peut les amener à dénaturer.
- Protéines Aliquot pour le stockage à long terme de sorte que tous les aliquots sont une seule utilisation seulement pour éviter la dégradation avec des cycles répétés de gel / dégel. Conserver les protéines lyophilisées à -80 oC (sauf indication contraire) jusqu'à ce qu'elles soient nécessaires. Veillez à recueillir toutes les métadonnées pour référence future, telles que : (i) nom de protéine, (ii) date préparée, (iii) numéro de lot/lot, (iv) fournisseur, (v) numéro de catalogue, (vi) concentration, (vii) volume, et (viii) préparateur.
- Préparer un tampon diluant contenant 20 % (v/v) de glycérol, 10 mM EDTA, 200 mM Tris-HCl, pH 7,2 et filtrer la stérilisation. Gardez ce tampon stérile et entreposez-le à RT.
- Préparer un tampon de coloration contenant 2 % (w/v) BSA, 1 mM MgCl2et 0,02 % NaN3 en salin tamponné par phosphate (PBS). Filtrer et conserver à 4 oC.
2. Préparation d'une plaque source ECM
- Enlever les stocks de protéines ECM aliquoted à imprimer et décongeler sur la glace. Enregistrez tous les numéros de lot pour le suivi des métadonnées.
- Flick tubes de protéines décongelées doucement pour assurer une résuspension appropriée et spin vers le bas dans une centrifugeuse.
-
Faire des mélanges d'impression ECM (EPM) et un fiducial fluorescent qui sera utilisé par un robot de manipulation liquide qui créera les plaques aléatoires de 384 puits.
REMARQUE : Les plaques de source de 384 puits seront utilisées par une imprimante à broches tactiles pour créer les tableaux imprimés dans 8 plaques de puits.- Étiqueter les tubes microcentrifuges de 1,5 mL pour chaque EPM et le fiducial.
- Préparer chaque EPM en combinant 125 l de tampon diluant (voir l'étape 1.4) avec le volume approprié de stock ECM et porter le mélange jusqu'à un volume total de 250 'L avec PBS. Les concentrations finales dans chaque tube EPM seront 1x protéine ECM, 5 mM EDTA, 10% de glycérol et 100 mM Tris.
- Préparer un fiducial fluorescent en le dissolvant dans le tampon approprié spécifié par le fabricant et transférer 250 L sur un tube fiducial étiqueté.
3. Création de la plaque source à l'aide d'un manutentionnaire liquide
- Concevoir une disposition de plaque de 384 puits qui randomise les positions des ECM et est optimisée pour la tête d'épingle d'imprimante de tableau utilisée. Concevoir le placement du fiducial de sorte qu'il sera imprimé dans la rangée 1, colonne 1 position de chaque puits pour aider à l'orientation du tableau.
REMARQUE: Au total, 14 à 15 répliques de chaque ECM sont utilisées pour garantir des données robustes. Inclure des répliques supplémentaires de collagène ou d'un autre ECM qui donne une fixation robuste pour l'évaluation de l'uniformité de la liaison. La disposition peut avoir besoin d'utiliser plusieurs plaques de 384 puits en fonction du nombre d'ECMd d'intérêt. - Transférer les tubes EPM à un gestionnaire liquide, en maintenant les tubes à 4 oC soit avec un support à tube refroidi ou à l'aide d'un robot de manutention liquide situé dans une chambre froide.
- À l'aide du logiciel du gestionnaire de liquide, exécutez un programme pour transférer 15 L de chaque EPM et le fiducial aux puits prédésignés dans la plaque source de 384 puits.
- Pipet PBS dans tous les puits inutilisés pour augmenter l'humidité et se prémunir contre la dessiccation pendant le processus d'impression.
REMARQUE: Voir Figure 2 pour un exemple d'un ensemble de plaques de source de 384 puits qui est optimisé pour une tête de 4 x 7 broches et comprend un bloc de collagène I et PBS. - Sceller la plaque et conserver à 4 oC jusqu'au moment de l'impression.
4. Impression meMA à l'aide d'un robot d'impression Array
REMARQUE: La partie suivante du protocole décrit spécifiquement la préparation et l'utilisation de MEMA pour étudier l'impact de différentes protéines microenvironnement sur la croissance et la prolifération des cellules MCF7. Cependant, le protocole peut facilement être adapté pour utiliser différents ligands, ECMs, et les cellules pour étudier d'autres lignées cellulaires et les points de terminaison d'intérêt.
-
À l'aide d'une imprimante à broches tactiles, imprimez des EPM et des taches fiduciales dans 8 plaques de puits. Imprimez plusieurs répliques de chaque condition ECM pour assurer la reproductibilité.
REMARQUE : D'autres formats de plaques ou diapositives peuvent être utilisés pour l'impression, mais l'optimisation tampon peut être nécessaire pour atteindre une formation optimale de taches.- Imprimez les EMC pour le MEMA à l'aide de broches de 350 m de diamètre disposées en configuration de tête d'impression 4 x 7. Imprimez les tableaux dans les plaques de 8 puits en 20 colonnes par 35 rangées, pour un total de 700 places. De plus grands tableaux sont possibles dans ces plaques, mais viennent avec un compromis des effets de bord accrus dans la liaison cellulaire et la coloration.
- Après l'impression, conserver les assiettes dans un dessiccateur pendant au moins 3 jours avant l'utilisation.
5. Création de plaques de traitement Ligand
- Concevoir une disposition de la plaque de 96 puits, y compris les ligands d'intérêt. Pour faciliter le traitement de nombreuses plaques MEMA à la fois, concevoir cette plaque avec l'espacement qui permet l'utilisation d'une tuyauterie multicanal avec 4 pointes espacées pour transférer des liquides entre les puits de 8-bien MEMaAs et une plaque de 96 puits.
REMARQUE: Dans ce protocole, l'ensemble complet des ligands énumérés dans le tableau 2 sont utilisés. - Décongeler les ligands sur la glace. Faites glisser brièvement et faites tourner chaque tube.
- Diluer les ligands à un stock de travail 200x en utilisant le tampon recommandé par le fabricant (généralement PBS).
- Pipet 10 'L de chaque stock de ligand 200x dans le puits correspondant dans la plaque de 96 puits.
- Sceller et ranger les assiettes à -20 oC.
REMARQUE: Faire des plaques de traitement ligand par lots, en capturant toutes les métadonnées pour l'analyse en aval.
6. Cellules de culture sur meMAs
- Bloquer les MEMA pendant 20 min avec 2 ml par puits de tampon de blocage non encrassé contenant 1 % d'agent de blocage non encrassé (Tableau des matériaux) dans de l'eau double distillée (ddH2O).
- Aspirer le tampon de blocage et triple rinçage avec PBS. Pour éviter la dessiccation, laisser le volume final de PBS dans les puits jusqu'à ce qu'il soit prêt pour le placage cellulaire.
REMARQUE: Il est extrêmement utile d'avoir deux employés de banc pour les étapes de culture cellulaire sur les MEMA. Un employé de banc peut effectuer des étapes d'aspiration, tandis que le deuxième effectue des étapes d'addition. Il est recommandé d'utiliser une tuyauterie multicanal de 1 ml avec des pointes espacées pour correspondre à la plaque de 8 puits pour la pipetière et un Y-splitter avec deux pipettes Pasteur pour aspirer plusieurs puits à la fois. - Seed 2 x 105 cellules MCF7 par puits dans 2 ml du milieu moyen modifié de Dulbecco Eagle (DMEM) contenant 10 % de sérum bovin fœtal (SGF).
REMARQUE: Avant une expérience MEMA complète, effectuez une expérience de titration cellulaire pour optimiser le nombre de cellules de telle sorte que les taches MEMA ont un nombre élevé de cellules (mais ne sont pas confluents) à la fin de la durée expérimentale désirée. - Après 2 à 18 h d'adhérence, aspirez à moyen et remplacez-le par 2 ml de milieu à croissance réduite (DMEM avec 0,1 % de FBS).
REMARQUE: On peut utiliser à ce moment-là des conditions réduites de sérum (p. ex., 0,1 % de SGF) ou des conditions appauvries par les facteurs de croissance pour isoler l'impact stimulant de certains ligands. - Décongeler une plaque de traitement ligand sur la glace. Plaque décongelée centrifugeuse à 200 x g pendant 1 min.
- Transférer 200 l de milieu de chaque puits dans la plaque de culture au puits approprié dans la plaque de traitement. Pipet de haut en bas pour mélanger le volume ligand avec le milieu et transférer ce mélange vers le puits approprié dans la plaque MEMA.
- Légèrement bercer à la main et remettre les plaques MEMA à l'incubateur. Culture pour la durée de l'expérience en présence de la combinaison ligand/ECM à 37 oC et 5 % CO2.
REMARQUE: Une expérience MEMA typique dure 72 h; les expériences de plus longue durée peuvent nécessiter le remplacement du moyen et le retraitement avec du ligand. - Pulsez les puits MEMA à 71 h avec 100x 5-ethynyl-2'-deoxyuridine (EdU) pour une concentration finale de 10 M. Incubate dans des conditions expérimentales avec EdU pour 1 h à 37 oC et 5% CO2.
REMARQUE: D'autres traitements de cellules vivantes peuvent également être utilisés à ce moment.
7. Fixation et souillage des MEMA
- Après 72 h et tous les traitements cellulaires vivants, aspirater les puits. Fixer les MEMA en 2 ml par puits de 2 % de paraformaldéhyde (PFA) pendant 15 min à RT.
- Aspirez PFA. Perméabilize avec 2 ml par puits de 0,1% de surfactant nonionic pendant 15 min.
- Aspirer le surfactant nonionic et laver avec 2 ml par puits de PBS. Aspirez PBS. Laver avec 2 ml de PBS avec 0,05% polysorbate 20 (PBS-T).
REMARQUE: La surface du MEMA est hydrophobe, et le fait de ne pas se laver avec du PBS-T avant que la tache et l'incubation des anticorps n'entraînent la formation de vides dans les puits pendant les étapes d'incubation et donnent lieu à des artefacts de coloration. - Aspirez PBS-T. Ajouter des réactifs de réaction de détection EdU. Incuber pendant 1 h à RT, basculer et protéger de la lumière. Après 1 h d'incubation, étancher la réaction avec le tampon d'étancher commercial fourni.
REMARQUE: La détection de l'EdU et les étapes de coloration/anticorps peuvent être effectuées en 1,5 ml par puits afin de réduire les coûts. - Aspirez le tampon d'étance et lavez-le avec du PBS-T avant d'incuber avec des taches ou des anticorps.
- Incuber des puits MEMA avec des anticorps contre l'histone H3K9me3 (1:1 000) et de la fibrilline (1:400) dans un tampon de coloration contenant 2 % (w/v) d'albumine de sérum bovin (BSA), 1 mM MgCl2 et 0,02 % NaN3 pendant la nuit à 4 oC.
REMARQUE: Effectuez des titrations d'anticorps pour déterminer les concentrations optimales avant de les utiliser sur un ensemble COMPLET de MEMA. - Après l'incubation primaire des anticorps ou des taches, lavez les puits 2x avec du PBS et une fois avec le PBS-T.
- Ajoutez des anticorps secondaires (igG anti-souris à ânes et IgG anti-lapin d'âne, tous deux 1:300) et 0,5 g/mL 4' 6-diamidino-2-phenylindole (DAPI). Incuber pour 1 h à RT dans l'obscurité.
- Laver les puits 2x avec 2 ml par puits de PBS, en les laissant dans les 2 derniers mL PBS.
- Passez à l'imagerie ou entreposez les MEMA tachés pour une imagerie ultérieure en PBS à 4 oC, à l'abri de la lumière.
8. Imagerie des MEMA
- Image MEMA sur un système d'imagerie automatisé avec des canaux de détection fluorescents appropriés.
- Sortie des données d'image résultantes vers un système de gestion d'image. Segmenter les cellules et calculer les niveaux d'intensité à l'aide de CellProfiler8.
9. Analyse des données
REMARQUE: L'analyse des données consiste en la normalisation, la correction des variations et la synthèse des données dérivées de CellProfiler brutes. Dans ce cas, l'environnement R avec code personnalisé est utilisé pour effectuer toutes les étapes. Cependant, tout environnement statistique ou logiciel peut être utilisé pour effectuer les actions équivalentes. Un exemple du code personnalisé open source pour l'environnement R pour analyse est disponible à: https://www.synapse.org/#!Synapse:syn2862345/wiki/72486.
- Prétraiter et normaliser les données d'image segmentées.
- Déterminer le nombre de cellules ponctuelles à l'aide des noyaux tachés DAPI.
- Auto-porte EdU intensité pour étiqueter les cellules comme EdU. Mesurer la prolifération à l'aide de la proportion de cellules EdUà chaque endroit.
- Résumé médian des taches cytoplasmiques et des mesures de morphologie nucléaire au niveau de la tache.
- Effectuer la suppression de la normalisation des variations indésirables (RUV) sur les données afin d'améliorer la qualité des données9.
REMARQUE: Cette approche est appliquée à chaque signal d'intensité et de morphologie indépendamment comme une matrice avec des tableaux utilisant les lignes et les taches comme les colonnes comme décrit précédemment9. - Appliquer la normalisation bivariée de loess aux résidus normalisés de RUV en utilisant la ligne de réseau et la colonne de réseau comme variables indépendantes pour corriger les effets spatiaux ou liés à l'intensité.
- Une fois la normalisation terminée, la médiane résume les répliques pour chaque état microenvironnement pour les rapports et les analyses plus poussées.
Résultats
Afin de simplifier les impacts microenvironnementaux sur la croissance et la prolifération des cellules et d'identifier les conditions qui ont favorisé ou inhibé la croissance et la prolifération cellulaires, la lignée cellulaire du cancer du sein MCF7 a été enseve sur un ensemble de huit MEMA de huit puits tels que décrits dans le protocole. Cet analyse a exposé les cellules à 48 ECM différents et 57 ligands différents, pour un total de 2736 conditions micro-environnementales combinatoires. Après 71 h dans la culture, les cellules ont été pulsées avec EdU, fixe, perméablisée, et souillée avec DAPI, la réaction pour la détection d'EdU, un anticorps anti-fibrillin, et un anticorps anti-H3K9me3. Les cellules ont été imaged sur un microscope à haute teneur. Les images ont été téléchargées sur un serveur Omero10, segmenté à l'aide de CellProfiler8, et normalisé et analysé dans R9. Les résultats décrits ci-dessous se concentrent sur les signaux DAPI et EdU.
La plate-forme d'analyse d'image des MEMA donne des résultats similaires à ceux disponibles à partir des approches de cytométrie de flux, telles que des parcelles de contenu d'ADN montrant des fractions 2N et 4N pour les cellules traitées avec un ligand donné (Figure 3A), basé sur le DAPI l'intensité et la zone. Ces parcelles fournissent des preuves des conditions qui favorisent le cycle cellulaire actif par rapport à ce qui est indiqué par des pics bimodaux clairs correspondant aux cellules en phase G1 ou G2 par rapport aux cellules arrêtées par rapport à la croissance, ce qui montrerait des changements dans les pics par rapport aux conditions de contrôle. Nous utilisons le nombre de cellules et les données d'intensité de coloration pour résumer les données, où l'impact du microenvironnement (ligands sur un axe, ECM sur le deuxième axe) sur les deux numéros de cellule (figure 3B) et EdU incorporation (Figure 3C) peut être plus facilement vu comme des changements dans la couleur et l'intensité de la carte thermique. Comme on le voit dans ces parcelles, beaucoup d'effets sont pilotés par ligand, car l'état d'ECM n'a pas fortement eu d'impact sur le nombre de cellules ou la positivité EdU. Nidogen-1 est une exception claire, car la présence de cette molécule ECM inhibe la liaison cellulaire et la croissance de MCF7. Les ligands tels que FGF6 et NRG1 (NRG1.1 sur les parcelles) augmentent le nombre de cellules et ont des taux élevés d'incorporation d'EdU, tandis que les ligands tels que AREG et NRG1-smdf (NRG1.10 sur les parcelles) inhibent la liaison cellulaire et/ou la croissance des cellules. Ces résultats sont étayés par les images des cellules qui poussent sur les taches, où une nette différence dans le nombre de cellules et la positivité EdU est évidente (voir exemple dans la figure 3D).
Étant donné que la plate-forme MEMA est une technologie plus récente, les résultats ont été validés dans des analyses distinctes. Les cellules MCF7 ont été ensepépinées dans des plaques de 24 puits recouvertes de collagène I dans le milieu DMEM avec 10% de FBS. Après 18 h, les médias ont été échangés contre un milieu de croissance réduit (DMEM avec 0,1 % de FBS) et les cellules ont été traitées avec NRG1, FGF6, ou AREG et cultivées pendant 72 h. EdU a été ajouté 1 h avant la fixation. Les cellules ont été tachées avec DAPI et pour l'incorporation d'EdU, imaged, segmenté, et analysé. À l'instar des résultats obtenus à partir de la plate-forme MEMA, FGF6 et NRG1MD ont tous deux donné lieu à des taux d'incorporation plus élevés (figure 4A) et edU (figure 4B) par rapport aux cellules traitées AREG, validant nos observations dans les expériences MEMA originales.

Figure 1 : Graphique de flux indiquant le flux de travail et la chronologie des différentes phases d'une expérience MEMA typique. Une fois les EMMA imprimés, ils peuvent être conservés à température ambiante desséchées pendant plusieurs mois avant leur utilisation. Typiquement, la phase expérimentale dure 3 à 4 jours, mais certaines cellules primaires à croissance lente ont été cultivées sur les MEMA pendant une période pouvant aller jusqu'à 2 semaines. Veuillez cliquer ici pour voir une version plus grande de ce chiffre.

Figure 2 : Mise en page de la plaque source ECM pour l'impression du tableau. Le bloc de collagène est imprimé sur MEMA comme une grille, ce qui fournit un ensemble très répétitif de conditions qui permettent une normalisation plus robuste entre les puits. Les puits remplis de PBS fournissent de l'humidité pour aider à prévenir l'évaporation pendant le processus d'impression. Veuillez cliquer ici pour voir une version plus grande de ce chiffre.

Figure 3 : Exemples de données générées à partir d'une expérience MEMA typique. (A) Profils de cycle cellulaire des valeurs d'intensité Binned DAPI par rapport aux comptes cellulaires d'une plaque de 8 puits traitée avec différents ligands, montrant la coloration biphasique d'intensité de DAPI indiquant des cellules dans la phase de cycle cellulaire de G1 contre G2. (B) Heatmap montrant des comptes de cellules ponctuelles normalisées regroupés par similitude à l'aide d'amas hiérarchiques. Le rouge indique un nombre de cellules plus élevé, et le bleu est un nombre de cellules inférieur. Les Ligands sont sur l'axe X, les ECM sont sur l'axe y. (C) Heatmap montrant l'incorporation normalisée d'EdU, avec le rouge indiquant plus haut et le bleu indiquant l'incorporation inférieure d'EdU. Les Ligands sont sur l'axe X, les ECM sont sur l'axe y. (D) Exemple de cellules MCF7 poussant sur une tache MEMA traitée avec NRG1-MD montrant des taux élevés d'incorporation d'EdU (noyaux roses). La tache verte est masque cellulaire et le bleu est DAPI. Veuillez cliquer ici pour voir une version plus grande de ce chiffre.
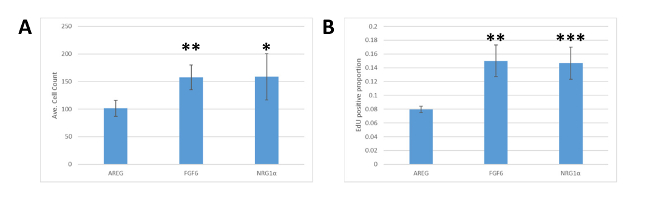
Figure 4 : La validation de LA MEMA entraîne la culture cellulaire. (A) Quantification du nombre cellulaire résultant du traitement de MCF7 avec différents ligands. Des nombres équivalents de cellules MCF7 ont été plaqués dans des plaques multi-murs puis traités avec AREG, FGF6, ou NRG1. Les puits traités par AREG avaient beaucoup moins de cellules que celles traitées par FGF6 (- indiquant les p-values de l'étudiant de moins de 0,01) ou NRG1 (indique une valeur p de 0,05) à 72 h après le traitement de ligand. (B) Quantification du niveau d'incorporation d'EdU dans MCF7 en raison d'un traitement avec différents ligands, comme dans le panneau A. Le traitement AREG se traduit par une proportion significativement plus faiblede cellules incorporant de l'EdU queles cellules traitées par FGF6 ( , p 'lt; 0.01) ou NRG1 ' ( , p - 0,01). Les barres d'erreur représentent l'écart type. Veuillez cliquer ici pour voir une version plus grande de ce chiffre.
| Nom des protéines | ID Uniprot | stock concentration (g/mL) | finale concentration (g/mL) |
| ANGPT1 1 | Q15389-1 | 100 ans | 0,04 |
| ANGPT2 1 | O15123-1 | 100 ans | 0,2 (en) |
| Areg | P15514 P15514 | 100 ans | 0,02 |
| BMP2 BMP2 BMP2 | P12643 (en) | 100 ans | 0,1 |
| BMP3 (en) | P12645 | 1000 | 0,1 |
| BMP4 (en) | P12644 (en) | 100 ans | 0,1 |
| BMP5 1 | P22003-1 | 100 ans | 0,1 |
| BMP6 (en) | P22004 P22004 | 100 ans | 0,1 |
| BMP7 (en) | P18075 P18075 | 100 ans | 0,1 |
| FSC2 | P04141 P041 | 100 ans | 0,02 |
| CTGF1 | P29279 1 | 100 ans | 0,05 |
| CXCL12 Alpha | P48061 | 100 ans | 0,01 |
| CXCL12 Bêta | P48061 | 100 ans | 0,03 |
| CXCL1 (en) | P09341 P09341 | 100 ans | 0,004 |
| CXCL8 1 | P10145 1 | 100 ans | 0,3 |
| DLL11 | O00548 1 | 500 Annonces | 0,5 |
| DLL4 (En) | Q9NR61 (en) | 200 Ans et plus | 0,6 |
| EGF 1 | P01133 1 | 500 Annonces | 0,01 |
| FASLG1 | P48023-1 | 10 Ans et plus | 0,02 |
| FGF2-3 | P09038 -2 | 100 ans | 0,01 |
| FGF6 (en) | P10767 (en) | 100 ans | 0,01 |
| FLT3LG1 | P49771 | 50 Annonces | 0,001 |
| GPNMB1 | Q14956 1 | 100 ans | 0,5 |
| HGF 1 | P14210 1 | 50 Annonces | 0,04 |
| IGF1 1 | P05019 1 | 200 Ans et plus | 0,01 |
| IGFBP2 (en anglais seulement) | P18065 P18065 | 100 ans | 0,05 |
| IGFBP3 | P17936 1 | 100 ans | 0,1 |
| IL13 (EN) | P35225 P35225 | 100 ans | 0,01 |
| IL15 IL15S48AA | P40933 1 | 50 Annonces | 0,01 |
| IL1B (en) | P01584 P01584 | 25 Annonces | 0,001 |
| IL6 (EN) | P05231 P05231 | 100 ans | 0,01 |
| IL7 1 | P13232 1 | 100 ans | 0,01 |
| JAG1 1 | P78504-1 | 200 Ans et plus | 0,5 |
| JAG2 longtemps | Q9Y219 1 | 100 ans | 0,5 |
| KITLG1 | P21583-1 | 100 ans | 0,005 Annonces |
| KNG1 Hmw | P01042 1 | 100 ans | 0,2 (en) |
| LEP (LEP) | P41159 (en) | 1000 | 0,002 |
| LYVE1 (EN) | Q9Y5Y7 | 100 ans | 0,05 |
| NRG1-10 | Q02297-10 | 100 ans | 0,01 |
| NRG1 1 | Q02297-1 | 100 ans | 0,05 |
| NRG1-6 | Q02297 6 | 100 ans | 0,01 |
| PDGFAB PDGFAB | aller1990265 | 100 ans | 0,05 |
| PDGFB1 | P01127 1 | 100 ans | 0,05 |
| Ptn | P21246 (en) | 100 ans | 0,5 |
| SHH (En) | Le T15465 | 100 ans | 0,5 |
| TGFB1MD Cterminus Cterminus | P01137 Cterminus Cterminus | 20 Ans, états-unis | 0,01 |
| TGFB1MD genoux | P01137 genoux | 100 ans | 0,15 |
| TGFB2MD un | P61812-1 | 20 Ans, états-unis | 0,01 |
| THPO 1 | P40225 1 | 50 Annonces | 0,002 |
| TNFRSF11B TNFRSF11B | O00300 O00300 | 100 ans | 0,02 |
| TNFSF11 | O14788 1 | 100 ans | 0,01 |
| Tnf | P01375 P01375 | 100 ans | 0,01 |
| VEGFA VEGF206 (en) | P15692 1 | 100 ans | 0,01 |
| WNT10A (en) | Q9GZT5 | 100 ans | 0,1 |
| WNT3A 1 | P56704-1 | 200 Ans et plus | 0,1 |
| Wnt5a 1 | P22725 1 | 100 ans | 0,1 |
Tableau 1 : La liste complète des ligands utilisés pour les expériences MEMA. L'ID uniprot, les concentrations de stock et les concentrations de travail finales sont fournis.
| Protéines ECM | UniprotID UniprotID | Concentration des stocks (g/mL) | finale concentration (g/mL) | Notes |
| ALCAM1 | Q13740-1 | 100 ans | 30 Ans, états-unis ( | |
| CDH20 | Q9HBT6 (en anglais) | 300 Ans et plus | 80 Ans, états-unis ( | |
| CDH6 1 | P55285 1 | 100 ans | 40 ans, états-unis ( | |
| CDH8 | P55286 P55286 | 100 ans | 20 Ans, états-unis | |
| CD44-1 | P16070 1 | 100 ans | 30 Ans, états-unis ( | |
| CEACAM6 | P40199 (en) | 100 ans | 30 Ans, états-unis ( | |
| COL1A1 (en) | P02453 P02453 | 5000 Annonces | 200 Ans et plus | sous-unités multiples avec plusieurs ids uniprot |
| COL2A1-2 | P02458-2 | 1000 | 200 Ans et plus | |
| COL3A11 | P02461 | 1000 | 200 Ans et plus | |
| COL4A11 | P02462 1 | 1000 | 200 Ans et plus | sous-unités multiples avec plusieurs ids uniprot |
| COL5A1 (en) | P20908 P20908 | 1000 | 200 Ans et plus | |
| COL23A11 | Q86Y22 | 200 Ans et plus | 80 Ans, états-unis ( | |
| DSG2 (en) | Le T14126 | 100 ans | 30 Ans, états-unis ( | |
| CDH1 | P12830 1 | 100 ans | 40 ans, états-unis ( | |
| ECM1 1 | Q16610 1 | 100 ans | 40 ans, états-unis ( | |
| FN1 1 | P02751 | 1000 | 200 Ans et plus | |
| GAP43-1 | P17677 1 | 158 Annonces | 40 ans, états-unis ( | |
| HyA-500K | 1000 | 200 Ans et plus | LOR-0005 | |
| HyA-50K | 1000 | 200 Ans et plus | LOR-0007 | |
| ICAM1 (en) | P05362 P05362 | 400 Ans et plus | 80 Ans, états-unis ( | |
| ALCAM1 | Q13740-1 | 100 ans | 30 Ans, états-unis ( | |
| CDH20 | Q9HBT6 (en anglais) | 300 Ans et plus | 80 Ans, états-unis ( | |
| CDH8 | P55286 P55286 | 100 ans | 20 Ans, états-unis | |
| CD44-1 | P16070 1 | 100 ans | 30 Ans, états-unis ( | |
| CEACAM6 | P40199 (en) | 100 ans | 30 Ans, états-unis ( | |
| DSG2 (en) | Le T14126 | 100 ans | 30 Ans, états-unis ( | |
| CDH15 | P55291 (en) | 100 ans | 20 Ans, états-unis | |
| VCAM1 1 | P19320 1 | 1000 | 200 Ans et plus | |
| LAMA1 (LaMA1) | P25391 (en) | 500 Annonces | 200 Ans et plus | sous-unités multiples avec plusieurs ids uniprot |
| LAMA3-2 | Q16787-2 | 130 Ans et plus | 40 ans, états-unis ( | |
| Lum | P51884 P51884 | 200 Ans et plus | 80 Ans, états-unis ( | |
| CDH15 | P55291 (en) | 100 ans | 20 Ans, états-unis | |
| NID1 1 | P14543 1 | 100 ans | 9,3 g/mL Nid, 130 g/mL Lam, 46,5 g/mL COL4 | COL4 et laminin |
| OMD (OMD) | Q99983 (en) | 100 ans | 40 ans, états-unis ( | |
| SPP1 un | P10451 1 | 100 ans | 40 ans, états-unis ( | |
| CDH3 1 | P22223-1 | 100 ans | 40 ans, états-unis ( | |
| PECAM1 longtemps | P16284-1 | 150 Annonces | 40 ans, états-unis ( | |
| Le TNC1 | P24821 | 500 Annonces | 200 Ans et plus | |
| VCAM1 1 | P19320 1 | 1000 | 200 Ans et plus | |
| VTN (en) | P04004 P0404 | 100 ans | 40 ans, états-unis ( | |
| Bgn | P21810 P21810 | 100 ans | 40 ans, états-unis ( | |
| DCN un | P07585 1 | 300 Ans et plus | 80 Ans, états-unis ( | |
| POSTN 1 | Q15063-1 | 100 ans | 40 ans, états-unis ( | |
| Sparc | P09486 P09486 | 100 ans | 40 ans, états-unis ( | |
| THBS1 1 | P07996 1 | 100 ans | 40 ans, états-unis ( | |
| BCAN1 | Q96GW7-1 | 100 ans | 40 ans, états-unis ( | |
| ELN 3 | P15502 3 | 1000 | 200 Ans et plus | |
| FBN1 (en) | P35555 | 254 Annonces | 80 Ans, états-unis ( |
Tableau 2 : La liste complète des protéines et des conditions d'ECM utilisées dans les expériences MEMA. L'ID uniprot, les concentrations de stock et les concentrations de travail finales sont fournis. Dans certains cas, l'état imprimé représente un complexe protéique ou une combinaison de protéines multiples, qui est indiquée dans la colonne Notes.
Discussion
L'importance de la « dimensionnalité » et du contexte a été un facteur motivant dans le développement de systèmes de culture in vitro comme outils dans la caractérisation des cellules cancéreuses par leur interaction avec le microenvironnement11, et la capacité de l'in vitro les systèmes de culture pour imiter l'environnement in vivo est une force motrice derrière la quête pour améliorer ces systèmes de culture. Les systèmes in vitro, cependant, restent des outils importants de la recherche sur le cancer précisément en raison de leur capacité à distiller la situation complexe in vivo jusqu'à un modèle simplifié12.
Bien que les systèmes 2D puissent inclure des ECM et des ligands, ils n'ont traditionnellement pas les capacités de débit nécessaires pour interroger un large panel de pertubagens combinatoires. Les extraits commerciaux populaires de membrane de sous-sol permettent la culture en 3D, mais manquent de la provenance d'un panneau soigneusement défini de protéines. Les extraits commerciaux souffrent généralement d'une composition incomplètement définie, ce qui peut confondre l'analyse et entraîner une variation importante de lot à lot3,13. La plate-forme MEMA surmonte ces obstacles, permettant l'étude des altérations des phénotypes cellulaires, de l'activité métabolique, du statut de différenciation et des variations de la croissance et de la prolifération cellulaires telles qu'elles sont modulées par des endogènes spécifiques et définis. Facteurs.
La plate-forme MEMA est une approche puissante, moyenne à haute quantité pour évaluer l'impact du microenvironnement (à la fois ECM et facteurs solubles) sur le phénotype des cellules. La plate-forme montre une grande flexibilité pour les types d'essais et de cellules pour lesquelles il peut être utilisé. Nous pouvons observer les effets des ligands solubles et des protéines ECM auxquelles les cellules sont exposées. En effet, nous avons récemment constaté que les ligands étaient un moteur majeur de résistance aux inhibiteurs ciblés par HER2, mais que ces effets pouvaient être modulés par l'ECM5. Une variété de cellules, y compris les cellules primaires et les lignées cellulaires dérivées de différents types de cellules, y compris les poumons, la vessie, la prostate, le sein et le pancréas, ainsi que les cellules souches pluripotentes induites (iPS), ont été cultivées avec succès sur la plate-forme MEMA (voir exemples dans les références5,7,14). L'utilisation de différentes taches permet la lecture de plusieurs paramètres cellulaires, y compris la croissance cellulaire, la différenciation et le métabolisme. D'autres chercheurs ont étendu la plate-forme pour interroger l'impact de la rigidité ou du modulus élastique, ajoutant une dimension supplémentaire à la plate-forme MEMA15. Enfin, la plate-forme est prête à effectuer des écrans de médicaments pour l'identification des conditions microenvironnement qui soit améliorer ou inhiber l'efficacité des médicaments, comme nous et d'autres ont récemment signalé5,14,15 .
Peut-être l'étape la plus critique au succès d'une expérience de MEMA est d'optimiser la densité de placage cellulaire. L'optimisation de la densité des cellules garantit la présence de suffisamment de cellules pour fournir des données robustes, mais pas tant que la tache devient trop confluente. Les taches de confluent peuvent confondre considérablement les résultats, en particulier si la prolifération est utilisée comme point de terminaison, ce qui rend impossible de déterminer si de faibles taux de prolifération sont le résultat d'interactions avec des facteurs microenvironnementaux ou en raison de l'inhibition des contacts densité cellulaire élevée. Les expériences de titration cellulaire peuvent révéler ces problèmes, car le nombre moyen de cellules par tache démontrera une augmentation linéaire avec un nombre croissant de cellules plaquées, mais finira par plafonner. Le numéro de cellule optimal doit être choisi dans la plage linéaire de la courbe.
Comme mentionné ci-dessus, la plate-forme MEMA est flexible et peut être préparée sur une variété de substrats avec différentes surfaces. Il s'agit notamment de diapositives en verre et de formats de plaques multimurs. D'après notre expérience, toutes les chimies de surface ne sont pas propices à l'impression MEMA, car nous avons observé un détachement ponctuel sur certaines surfaces en raison de mauvaises propriétés d'adhérence et de l'incapacité de bloquer l'adhérence cellulaire sur d'autres surfaces d'adhérence élevée. En outre, le changement entre différents substrats nécessite l'optimisation des conditions tampons, car les performances de l'impression avec le même tampon d'impression peuvent varier en fonction de la chimie de surface.
Le diamètre des taches ECM imprimées joue un rôle important dans la qualité des données. En général, nous recommandons d'utiliser les broches d'impression de plus grand diamètre disponibles pour l'entremise en usage (nous utilisons actuellement des broches de 350 m de diamètre). Les taches de diamètre plus grandes permettent à un plus grand nombre de cellules d'occuper une tache, ce qui tend à se traduire par des données plus robustes que celles générées par des broches de plus petit diamètre. Puisque la liaison des cellules est un processus stochastique, il a tendance à y avoir un degré élevé de variabilité dans les données qui est liée au nombre de cellules attachées à l'origine à chaque endroit. Ainsi, nous recommandons d'imprimer un grand nombre de répliques pour chaque condition ECM. Nous imprimons des répliques ECM de 10 à 15 dans chaque puits avec nos conditions d'impression actuelles afin d'assurer des statistiques fiables.
Nous avons noté dans nos expériences passées que, pour la plupart, les effets ligandont ont tendance à dominer les effets ECM. Cela peut être en partie dû à notre décision d'ajouter du collagène I à toutes les taches ECM, ce qui assure une liaison cellulaire robuste. Cependant, nous croyons que cela peut également homogénéiser les effets ECM, comme la plupart des taches ont tendance à se comporter d'une manière très similaire au collagène I. Modifier la composition de tache pour exclure le collagène I peut entraîner un comportement cellulaire différentiel à la suite de l'interaction avec le ECM, mais aussi des impacts significatifs de liaison cellulaire, résultant en beaucoup plus de taches inoccupées. Les utilisateurs doivent adapter leur composition ECM en gardant ces différences à l'esprit, en particulier ceux qui s'intéressent aux cellules souches et progénitrices et la différenciation, où la matrice peut avoir un impact significatif16.
Nous effectuons généralement les tests MEMA pour des périodes relativement courtes (p. ex., 72 h maximum). C'est parce que les cellules sont limitées aux taches (le tampon de blocage ne permet pas la croissance en dehors des taches dans notre expérience). Avec des cellules qui se divisent rapidement, la croissance de plus de 72 h entraînera une surcroissance de la tache, ce qui complique la segmentation de l'image à mesure que les cellules s'entassent les unes sur les autres, et peuvent également avoir un impact sur les données, car l'arrêt de croissance peut se produire avec l'inhibition du contact. Nous avons effectué des traitements plus longs avec des cellules primaires à croissance très lente (10 à 14 jours), mais il faut prendre soin de ces tests pour changer les médias et reconstituer les ligands tous les 3 à 4 jours.
Les efforts continus pour développer la plate-forme MEMA sont axés sur deux domaines d'intérêt, la maximisation de la qualité optique pour l'imagerie et l'optimisation au sein de petits navires de culture. La qualité optique devient un facteur crucial lorsque les chercheurs ont besoin d'une microscopie à plus haute résolution pour identifier la localisation subcellulaire de leurs marqueurs d'intérêt. Les écrans initiaux peuvent être effectués à faible résolution sur des microscopes à haut débit suivis de l'imagerie de points d'intérêt spécifiques sur des instruments à résolution plus élevée, mais la qualité de l'image peut être compromise si les propriétés optiques du substrat sont médiocres. L'amélioration des propriétés optiques du substrat permettrait aux chercheurs d'effectuer les premiers écrans sur des systèmes d'imagerie à haute résolution sans avoir besoin de réacquérir des images sélectionnées à une résolution plus élevée. Enfin, la capacité d'effectuer des MEMA dans de plus petits navires de culture, comme des plaques de 96 puits, permettrait de réduire le volume de traitement et d'élargir les ligands et les répliques interrogés. Cette transition nécessite l'optimisation des interactions substrat-tampon-protéine et l'impression de tableaux à l'intérieur de nouveaux vaisseaux de culture. Ces efforts continus amélioreront la plate-forme MEMA et élargiront ses puissantes capacités à identifier les protéines microenvironnementales pertinentes qui modifient les phénotypes cellulaires pour une variété de types de cellules, qui peuvent ensuite être étudiées dans tests de confirmation.
Déclarations de divulgation
Les auteurs n'ont rien à révéler.
Remerciements
Ce travail a été appuyé par la subvention HG008100 (J.W.G., L.M.H.et J.E.K) de la Bibliothèque des signatures cellulaires de réseau (LINCS) du Fonds commun des NIH.
matériels
| Name | Company | Catalog Number | Comments |
| Aushon 2470 | Aushon BioSystems | Arrayer robot system used in the protocol | |
| Nikon HCA | Nikon | High Content Imaging system designed around Nikon Eclipse Ti Inverted Microscope | |
| BioTek Precision XS liquid Handler | BioTek | liquid handling robot used in the protocol | |
| Trizma hydrochloride buffer solution | Sigma | T2069 | |
| EDTA | Invitrogen | 15575-038 | |
| Glycerol | Sigma | G5516 | |
| Triton X100 | Sigma | T9284 | |
| Tween 20 | Sigma | P7949 | |
| Kolliphor P338 | BASF | 50424591 | |
| 384-well microarray plate, cylindrical well | Thermo Fisher | ab1055 | |
| Nunc 8 well dish | Thermo Fisher | 267062 | |
| Paraformaldehyde 16% solution | Electron Microscopy Science | 15710 | |
| BSA | Fisher | BP-1600 | |
| Sodium Azide | Sigma | S2002 | |
| Cell Mask | Molecular Probes | H32713 | |
| Click-iTEdU Alexa Fluor | Molecular Probes | C10357 | |
| DAPI | Promo Kine | PK-CA70740043 | |
| ALCAM | R & D Systems | 656-AL | ECM |
| Cadherin-20 (CDH20) | R & D Systems | 5604-CA | ECM |
| Cadherin-6 (CDH6) | R & D Systems | 2715-CA | ECM |
| Cadherin-8 (CDH8) | R & D Systems | 188-C8 | ECM |
| CD44 | R & D Systems | 3660-CD | ECM |
| CEACAM6 | R & D Systems | 3934-CM | ECM |
| Collagen I | Cultrex | 3442-050-01 | ECM |
| Collagen Type II | Millipore | CC052 | ECM |
| Collagen Type III | Millipore | CC054 | ECM |
| Collagen Type IV | Sigma | C5533 | ECM |
| Collagen Type V | Millipore | CC077 | ECM |
| COL23A1 | R & D Systems | 4165-CL | ECM |
| Desmoglein 2 | R & D Systems | 947-DM | ECM |
| E-cadherin (CDH1) | R & D Systems | 648-EC | ECM |
| ECM1 | R & D Systems | 3937-EC | ECM |
| Fibronectin | R & D Systems | 1918-FN | ECM |
| GAP43 | Abcam | ab114188 | ECM |
| HyA-500K | R & D Systems | GLR002 | ECM |
| HyA-50K | R & D Systems | GLR001 | ECM |
| ICAM-1 | R & D Systems | 720-IC | ECM |
| Laminin | Sigma | L6274 | ECM |
| Laminin-5 | Abcam | ab42326 | ECM |
| Lumican | R & D Systems | 2846-LU | ECM |
| M-Cad (CDH15) | R & D Systems | 4096-MC | ECM |
| Nidogen-1 | R & D Systems | 2570-ND | ECM |
| Osteoadherin/OSAD | R & D Systems | 2884-AD | ECM |
| Osteopontin (SPP) | R & D Systems | 1433-OP | ECM |
| P-Cadherin (CDH3) | R & D Systems | 861-PC | ECM |
| PECAM1 | R & D Systems | ADP6 | ECM |
| Tenascin C | R & D Systems | 3358-TC | ECM |
| VCAM1 | R & D Systems | ADP5 | ECM |
| Vitronectin | R & D Systems | 2308-VN | ECM |
| Biglycan | R & D Systems | 2667-CM | ECM |
| Decorin | R & D Systems | 143-DE | ECM |
| Periostin | R & D Systems | 3548-F2 | ECM |
| SPARC/osteonectin | R & D Systems | 941-SP | ECM |
| Thrombospondin-1/2 | R & D Systems | 3074-TH | ECM |
| Brevican | R & D Systems | 4009-BC | ECM |
| Elastin | BioMatrix | 5052 | ECM |
| Fibrillin | Lynn Sakai Lab OHSU | N/A | ECM |
| ANGPT2 | RnD_Systems_Own | 623-AN-025 | Ligand |
| IL1B | RnD_Systems_Own | 201-LB-005 | Ligand |
| CXCL8 | RnD_Systems_Own | 208-IL-010 | Ligand |
| IGF1 | RnD_Systems_Own | 291-G1-200 | Ligand |
| TNFRSF11B | RnD_Systems_Own | 185-OS | Ligand |
| BMP6 | RnD_Systems_Own | 507-BP-020 | Ligand |
| FLT3LG | RnD_Systems_Own | 308-FK-005 | Ligand |
| CXCL1 | RnD_Systems_Own | 275-GR-010 | Ligand |
| DLL4 | RnD_Systems_Own | 1506-D4-050 | Ligand |
| HGF | RnD_Systems_Own | 294-HGN-005 | Ligand |
| Wnt5a | RnD_Systems_Own | 645-WN-010 | Ligand |
| CTGF | Life_Technologies_Own | PHG0286 | Ligand |
| LEP | RnD_Systems_Own | 398-LP-01M | Ligand |
| FGF2 | Sigma_Aldrich_Own | SRP4037-50UG | Ligand |
| FGF6 | RnD_Systems_Own | 238-F6 | Ligand |
| IL7 | RnD_Systems_Own | 207-IL-005 | Ligand |
| TGFB1 | RnD_Systems_Own | 246-LP-025 | Ligand |
| PDGFB | RnD_Systems_Own | 220-BB-010 | Ligand |
| WNT10A | Genemed_Own | 90009 | Ligand |
| PTN | RnD_Systems_Own | 252-PL-050 | Ligand |
| BMP3 | RnD_Systems_Own | 113-BP-100 | Ligand |
| BMP4 | RnD_Systems_Own | 314-BP-010 | Ligand |
| TNFSF11 | RnD_Systems_Own | 390-TN-010 | Ligand |
| CSF2 | RnD_Systems_Own | 215-GM-010 | Ligand |
| BMP5 | RnD_Systems_Own | 615-BMC-020 | Ligand |
| DLL1 | RnD_Systems_Own | 1818-DL-050 | Ligand |
| NRG1 | RnD_Systems_Own | 296-HR-050 | Ligand |
| KNG1 | RnD_Systems_Own | 1569-PI-010 | Ligand |
| GPNMB | RnD_Systems_Own | 2550-AC-050 | Ligand |
| CXCL12 | RnD_Systems_Own | 350-NS-010 | Ligand |
| IL15 | RnD_Systems_Own | 247-ILB-005 | Ligand |
| TNF | RnD_Systems_Own | 210-TA-020 | Ligand |
| IGFBP3 | RnD_Systems_Own | 675-B3-025 | Ligand |
| WNT3A | RnD_Systems_Own | 5036-WNP-010 | Ligand |
| PDGFAB | RnD_Systems_Own | 222-AB | Ligand |
| AREG | RnD_Systems_Own | 262-AR-100 | Ligand |
| JAG1 | RnD_Systems_Own | 1277-JG-050 | Ligand |
| BMP7 | RnD_Systems_Own | 354-BP-010 | Ligand |
| TGFB2 | RnD_Systems_Own | 302-B2-010 | Ligand |
| VEGFA | RnD_Systems_Own | 293-VE-010 | Ligand |
| IL6 | RnD_Systems_Own | 206-IL-010 | Ligand |
| CXCL12 | RnD_Systems_Own | 351-FS-010 | Ligand |
| NRG1 | RnD_Systems_Own | 378-SM | Ligand |
| IGFBP2 | RnD_Systems_Own | 674-B2-025 | Ligand |
| SHH | RnD_Systems_Own | 1314-SH-025 | Ligand |
| FASLG | RnD_Systems_Own | 126-FL-010 | Ligand |
Références
- Hanahan, D., Coussens, L. M. Accessories to the crime: functions of cells recruited to the tumor microenvironment. Cancer Cell. 21 (3), 309-322 (2012).
- Quail, D. F., Joyce, J. A. Microenvironmental regulation of tumor progression and metastasis. Nature Medicine. 19 (11), 1423-1437 (2013).
- Hughes, C. S., Postovit, L. M., Lajoie, G. A. Matrigel: a complex protein mixture required for optimal growth of cell culture. Proteomics. 10 (9), 1886-1890 (2010).
- LaBarge, M. A., et al. Human mammary progenitor cell fate decisions are products of interactions with combinatorial microenvironments. Integrative Biology (Cambridge). 1 (1), 70-79 (2009).
- Watson, S. S., et al. Microenvironment-Mediated Mechanisms of Resistance to HER2 Inhibitors Differ between HER2+ Breast Cancer Subtypes. Cell Systems. 6 (3), 329-342 (2018).
- Ranga, A., et al. 3D niche microarrays for systems-level analyses of cell fate. Nature Communications. 5, 4324 (2014).
- Malta, D. F. B., et al. Extracellular matrix microarrays to study inductive signaling for endoderm specification. Acta Biomater. 34, 30-40 (2016).
- Kamentsky, L., et al. Improved structure, function and compatibility for CellProfiler: modular high-throughput image analysis software. Bioinformatics. 27 (8), 1179-1180 (2011).
- Gagnon-Bartsch, J. A., Jacob, L., Speed, T. P. Removing Unwanted Variation from High Dimensional Data with Negative Controls. University of California, Berkeley, Department of Statistics, University of California, Berkeley. , (2013).
- Allan, C., et al. OMERO: flexible, model-driven data management for experimental biology. Nature Methods. 9 (3), 245-253 (2012).
- Simian, M., Bissell, M. J. Organoids: A historical perspective of thinking in three dimensions. Journal of Cell Biology. 216 (1), 31-40 (2017).
- Bissell, M. J. The differentiated state of normal and malignant cells or how to define a "normal" cell in culture. International Review of Cytology. 70, 27-100 (1981).
- Serban, M. A., Prestwich, G. D. Modular extracellular matrices: solutions for the puzzle. Methods. 45 (1), 93-98 (2008).
- Kaylan, K. B., et al. Mapping lung tumor cell drug responses as a function of matrix context and genotype using cell microarrays. Integrative Biology (Cambridge). 8 (12), 1221-1231 (2016).
- Lin, C. H., Jokela, T., Gray, J., LaBarge, M. A. Combinatorial Microenvironments Impose a Continuum of Cellular Responses to a Single Pathway-Targeted Anti-cancer Compound. Cell Reports. 21 (2), 533-545 (2017).
- Gjorevski, N., et al. Designer matrices for intestinal stem cell and organoid culture. Nature. 539 (7630), 560-564 (2016).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon