Method Article
שימוש במיקרו מערכים לחקור השפעה מיקרוסביבתית על פנוטיפים סלולריים בסרטן
In This Article
Summary
מטרת השיטה המוצגת כאן היא להראות כיצד מיקרומערכים מיקרואקולוגיה (MEMA) ניתן לזייף ולהשתמש כדי לחקור את ההשפעה של אלפי קומבינטורית מיקרו סביבות פשוטות על הפנוטיפ של תאים מתורבתים.
Abstract
הבנת ההשפעה של המיקרוסביבה על הפנוטיפ של התאים היא בעיה קשה בשל התערובת המורכבת של שני גורמי גדילה מסיסים וחלבונים הקשורים מטריקס ב מיקרוסביבה ב vivo. יתר על כן, ריאגנטים זמין עבור מידול של מיקרו סביבות בתוך החוץ בדרך כלל לנצל תערובות מורכבות של חלבונים כי הם מוגדרים לחלוטין וסובלים אצווה להשתנות אצווה. פלטפורמת המיקרו-מיקרו של הסביבה (MEMA) מאפשרת הערכה של אלפי שילובים פשוטים של חלבונים מיקרוסביבתיים להשפעה שלהם על הפנוטיפים הסלולריים בתוך שיטת יחיד. MEMAs מוכנים צלחות היטב, אשר מאפשר תוספת של ליגונים בודדים להפריד בארות המכילות מטריקס מוליכי הרכב (ECM) חלבונים. השילוב של הליטרים מסיסים עם כל ECM מודפס יוצר שילוב ייחודי. שיטת MEMA טיפוסית מכילה יותר מ-2,500 מיקרוסביבות קומבינטורית ייחודיות שתאים חשופים אליהם בתוך שיטת יחיד. כמקרה מבחן, סרטן השד התאים קו MCF7 היה מצופה על פלטפורמת MEMA. ניתוח זה מזוהה גורמים שניהם לשפר ולעכב את הצמיחה והתפשטות של תאים אלה. פלטפורמת MEMA היא גמישה מאוד והוא יכול להיות מורחב לשימוש עם שאלות ביולוגיות אחרות מעבר למחקר הסרטן.
Introduction
Culturing של שורות תאים סרטניים על פלסטיק דו מימדי (2D) monolayers נשאר אחד הסוסים העיקריים עבודה עבור חוקרי סרטן. עם זאת, המיקרו-סביבה מוכרת יותר ויותר בשל יכולתה להשפיע על פנוטיפים סלולריים. בסרטן, מיקרוסביבה הגידול ידוע להשפיע על התנהגויות סלולר מרובות, כולל צמיחה, הישרדות, פלישה, ותגובה לטיפול1,2. תרביות תאים מסורתיים בדרך כלל חוסר השפעות מיקרו הסביבה, אשר הובילה לפיתוח של מורכב יותר תלת-מימדי (3d) בחני לגדול תאים, כולל מסחרית זמין מרתף מטוהרים מתחת תמציות קרום. עם זאת, אלה מטריצות מטוהרים הם בדרך כלל מורכבים להשתמש וסובלים מבעיות טכניות כגון השתנות אצווה3 וקומפוזיציות מורכבות3. כתוצאה מכך, זה יכול להיות קשה להקצות פונקציה חלבונים ספציפיים שעשויים להשפיע על פנוטיפים סלולריים3.
כדי לטפל במגבלות אלה, פיתחנו את הטכנולוגיה microenvironment מיקרו-הסביבה (mema), אשר מפחית את המיקרו-סביבה עד שילובים פשוטים של מטריקס מטריצה (ecm) ומסיס גורם צמיחה חלבונים4,5 . פלטפורמת MEMA מאפשרת זיהוי של גורמים מיקרוסביבתיים דומיננטי המשפיעים על התנהגות של תאים. באמצעות תבנית מערך, ניתן לקבל בניסוי אחד אלפי שילובים של גורמי מיקרו-סביבה. MEMA תיאר כאן חוקר שערים ~ 2,500 שונים בתנאים מיקרו סביבה ייחודית. חלבונים מסוג ECM המודפסים בלוחות הגידול של הטופס שעליהם יכולים להיות מתורבתים. ליגפות מסיסים מתווספים לבארות בודדות, ויוצרות קומבינטורית מיקרו-סביבות ייחודיות (ECM + ligands) בכל נקודה שונה אליה נחשפים התאים. תאים הם מתורבתים במשך מספר ימים, לאחר מכן קבוע, מוכתם, והתמונה כדי להעריך פנוטיפים הסלולר כתוצאה של חשיפה אלה שילובים ספציפיים מיקרו הסביבה. מאז המיקרוסביבות הם שילובים פשוטים, זה פשוט לזהות חלבונים כי כונן שינויים פנוטימית עיקריים בתאים. Memas השתמשו בהצלחה כדי לזהות גורמים המשפיעים על מספר פנוטיפים סלולריים, כולל אלה כי כונן החלטות הגורל תא תגובה לטיפול4,5,6,7. תגובות אלה ניתן לאמת בניסויים 2D פשוטים ולאחר מכן ניתן להעריך בתנאים כי יותר לכידה מלאה של המורכבות של מיקרוסביבה הגידול. פלטפורמת MEMA היא להתאים מאוד למגוון של סוגי תאים ונקודות קצה, בתנאי כי בסמנים פנוטיפים טובים זמינים.
Protocol
הערה: מבט כולל על תהליך MEMA כולו, כולל זמן משוער, מתואר בדיאגרמת הזרימה המוצגת באיור 1. פרוטוקול זה מפרט את הייצור של MEMAs בצלחות בנות 8. הפרוטוקול עשוי להיות מותאם עבור לוחות אחרים או שקופיות.
1. הכנת מאגרי חלבונים, מדלל וכתמים
- מבחנות של ECMs, ליגניות, ו ציטוקינים לטמפרטורת החדר (RT) ובקצרה צנטריפוגה. הוסף את הנפח המתאים של מאגר ה-RT המתאים כפי שמצוין בגליון הנתונים של המוצר. בצע את המלצת היצרן עבור ריכוזי מניות.
הערה: רשימה מלאה של ליגנדס ו ECMs עם המניות שלהם וריכוזי הסופי ניתנים בטבלה 1 ושולחן 2. הן ליגנדס והן ECMs משמשות בדרך כלל בריכוז הגבוה ביותר של הטווח המומלץ על ידי היצרן, אשר מעורר את ההשפעה הביולוגית שלו בתקן 2 יום התרבות. מטפלים בחלבונים בעדינות ובארונות ביו-בטיחות בזרימה שכבתית כדי למנוע זיהום. - דגירה מבחנות עם נדנדה עדין ב-RT עבור 1 h. לא מערבולת חלבונים כמו זה יכול לגרום להם להספרות.
- מחלקים חלבונים עבור אחסון לטווח ארוך כך כל aliquot הם שימוש יחיד רק כדי למנוע השפלה עם חוזר הקפאה/הפשרה מחזורי. מאחסנים חלבונים ב-80 ° צ' (אלא אם צוין אחרת) עד לצורך. דאג לאסוף את כל המטא-נתונים לעיון עתידי, כגון: (i) שם חלבון, (ii) תאריך מוכן, (iii) הרבה/אצווה מספר, (iv) הספק, (v) מספר קטלוג, (vi) ריכוז, (vii) נפח, ו (viii) הכנה.
- הכנת מאגר דילול המכיל 20% (v/v) גליצרול, 10 מ"מ EDTA, 200 mM טריס-HCl, pH 7.2, ומסנן לעקר. שמור על מאגר זה סטרילי ואחסן ב-RT.
- הכנת מאגר כתמים המכיל 2% (w/v) BSA, 1 מ"מ MgCl2, ו 0.02% נאן3 בתמיסת מלח פוספט באגירה (PBS). לסנן ולאחסן ב-4 ° c.
2. הכנת לוח מקור ECM
- הסר מניות מצוטט של חלבונים ECM להיות מודפס ולהפשיר על הקרח. הקלט את כל המספרים הרבים עבור מעקב אחר מטא-נתונים.
- קפיצי צינורות של חלבונים המופשטים בעדינות כדי להבטיח resuspension נכונה ספין למטה בצנטריפוגה.
-
הפוך תערובות ECM הדפסה (EPMs) ו פלורסנט fiducial לשמש על ידי רובוט טיפול נוזלי שתיצור את הצלחות אקראי 384-ובכן מקור.
הערה: לוחות המקור של 384-והטוב ישמשו מדפסת מערך של סיכת מגע כדי ליצור את המערכים המודפסים ב-8 לוחות הבאר.- תווית 1.5 mL לצינורות מיקרוצנטריפוגה עבור כל EPM והfiducial.
- הכן כל EPM על ידי שילוב 125 μL של מאגר דילול (ראה שלב 1.4) עם הנפח המתאים של מלאי ECM ולהביא את התערובת עד נפח כולל של 250 μL עם PBS. הריכוזים הסופיים בכל צינור EPM יהיה 1 x ECM חלבון, 5 מ"מ EDTA, 10% גליצרול, ו 100 mM טריס.
- הכן fiducial פלורסנט על ידי המסת אותו במאגר המתאים שצוין על ידי היצרן והעבר 250 μL לשפופרת fiducial מתויג.
3. יצירת לוח המקור באמצעות מטפל נוזלי
- עיצוב לוח 384-ובכן, המתאר את המיקומים של ה-ECMs וממוטב לשימוש בראש סיכת המדפסת המשמש כמערך. עצב את מיקום הfiducial כך שהוא יודפס בשורה 1, עמודה 1 מיקום כל טוב כדי לסייע בכיוון מערך.
הערה: סך של 14/15 שכפול של כל ECM משמשים להבטחת נתונים חזקים. כלול משכפל נוסף של קולגן או ECM אחרת התשואות חזקה להערכת אחידות של הכריכה. ייתכן שהפריסה תצטרך לנצל מספר 384-הצלחות מרובות בהתאם למספר ה-ECMs של הריבית. - העבר צינורות EPM למטפל נוזלי, שמירה על צינורות ב -4 ° c או עם מתלה צינור מקורר או באמצעות רובוט טיפול נוזלי הממוקם בחדר קר.
- באמצעות התוכנה של המטפל נוזל, להפעיל תוכנית להעביר 15 μL של כל EPM ואת fiducial לבארות מראש בתוך לוחית מקור 384-היטב (s).
- Pipet PBS לתוך בארות לא מנוצל כדי להגביר את הלחות והשמירה מפני התייבשות במהלך תהליך ההדפסה.
הערה: ראה איור 2 לדוגמה מערכת לוחית מקור 384-היטב הממוטב עבור 4 x 7 פינים הראש וכולל קולגן אני לחסום ו PBS. - לסגור את הצלחות ולשמור ב 4 ° צ' עד מוכן להדפסה.
4. הדפסת MEMAs באמצעות רובוט מערך הדפסה
הערה: החלק הבא של הפרוטוקול מתאר במפורש את ההכנה והשימוש של MEMA כדי לחקור את ההשפעה של חלבונים מיקרוסביבתיים שונים על הצמיחה והתפשטות של תאים MCF7. עם זאת, ניתן להתאים את הפרוטוקול בקלות לשימוש בליפסים שונים, ECMs ותאים ללימוד קווי תאים אחרים ונקודות קצה של ריבית.
-
באמצעות מדפסת סיכות מגע, הדפס EPMs ו fiducial כתמים לתוך 8 צלחות היטב. הדפס שכפול מרובים של כל תנאי ECM כדי להבטיח את התוכונות.
הערה: ניתן להשתמש בתבניות לוח או שקופיות אחרות להדפסה, אך ייתכן שיהיה צורך במיטוב מאגר כדי להשיג היווצרות ספוט אופטימלי.- הדפס את ה-ecms עבור mema באמצעות פינים בקוטר 350 יקרומטר המסודרים בתצורת ראש הדפסה של 4 x 7. הדפס את המערכים בלוחות של 8-היטב כמו 20 עמודות על-ידי 35 שורות, עבור סך של ~ 700 נקודות. מערכים גדולים יותר אפשריים בצלחות אלה, אך באים עם הסחר-off של השפעות קצה מוגבר הן כריכת תאים וכתמים.
- לאחר הדפסה, לאחסן צלחות desiccator לפחות 3 ימים לפני השימוש.
5. יצירת צלחות ליגוטיפול
- עיצוב לוח 96-ובכן, כולל ליגניות של עניין. כדי להקל על הטיפול של צלחות MEMA רבים בבת אחת, לעצב את הצלחת הזאת עם מרווח המאפשר שימוש של מרובת ערוצים pipet עם 4 טיפים במרווחים כדי להעביר נוזלים בין הבארות של 8-טוב Mema וצלחת 96-באר.
הערה: בפרוטוקול זה, משתמשים בערכה המלאה של ליגניות המפורטות בטבלה 2 . - . להפשיר ליגנדס על קרח קפיצי בקצרה וסובב כל צינור.
- השתמש במאגר המומלץ של היצרן (בדרך כלל PBS) כדי לדלל מלאי של 200x.
- Pipet 10 μL של כל 200x ליטר ומלאי לתוך הטוב המתאים בתוך הצלחת 96-באר.
- הרכב לוחות החותם והחנות ב-20 ° c.
הערה: להפוך לוחיות טיפול לחות בקבוצות, לכידת כל המטא-נתונים עבור ניתוח במורד הזרם.
6. תאים culturing על MEMAs
- לחסום MEMAs עבור 20 דקות עם 2 מ ל לכל היותר של מאגר חסימה ללא התפסות המכיל 1% שאינם מפוסות סוכן חסימה (טבלת חומרים) במים כפולים מזוקקים (ddh2O).
- ומשלש בארות שוטפים. עם הערוץ הPBS כדי למנוע התייבשות, השאר את הנפח הסופי של PBS בבארות עד שמוכן לציפוי תאים.
הערה: זה מאוד מועיל יש שני עובדי ספסל עבור שלבי תרבות התא על MEMAs. אחד מעובדי הספסל יכול לבצע שלבי השאיפה, ואילו השני מבצע צעדים בתוספת. מומלץ להשתמש 1 מ"ל pipet רב-ערוצי עם טיפים במרווחים כדי להתאים את הצלחת 8-הבאר עבור ליטוף ו-מפצל Y עם שני פיפטות פסטר לפתי בארות מרובות בבת אחת. - זרע 2 x 105 MCF7 תאים לכל טוב ב 2 מ ל של דולבקה שונה של הנשר הבינוני (dmem דיום) בינונית המכילה 10% סרום העוברי (fbs).
הערה: לפני ניסוי MEMA מלא, לבצע ניסוי טיטור תא כדי למטב את מספרי התאים כגון מקומות MEMA יש מספרי תאים גבוהים (אבל הם לא שוטפת) בסוף המשך הניסיוני הרצוי. - לאחר 2-18 שעות של הדבקה, מפחית בינונית ולהחליף עם 2 מ ל של בינונית צמיחה מופחתת (DMEM עם 0.1% FBS).
הערה: סרום מופחת (למשל, 0.1% FBS) או גורם גדילה-תנאים מופחתים ניתן להשתמש בשלב זה כדי לבודד את ההשפעה מגירוי של ליגניות ספציפיות. - . להפשיר צלחת ליגוטיפול בקרח צנטריפוגה הופלה את הצלחת ב 200 x g עבור 1 דקות.
- העברת 200 μL של בינוני מכל באר בלוח התרבות לבאר המתאים בצלחת הטיפול. Pipet למעלה ולמטה כדי לערבב נפח עם בינוני ולהעביר את התערובת בחזרה היטב המתאים בצלחת MEMA.
- להקל על הרוק ביד ולהחזיר את לוחיות MEMA לחממה. תרבות למשך הניסוי בנוכחות שילוב ligand/ECM ב 37 ° c ו-5% CO2.
הערה: ניסוי MEMA טיפוסי פועל עבור 72 h; ניסויים משך זמן ארוך עשויים לדרוש החלפת בינונית וטיפול מחדש עם ligand. - דופק MEMA וולס ב 71 h עם 100x 5-ethynyl-2'-deoxyuridine (EdU) לריכוז הסופי של 10 μM. דגירה בתנאים ניסיוניים עם EdU עבור 1 h ב 37 ° צ' ו 5% CO2.
הערה: בשלב זה ניתן להשתמש גם בטיפולי תאים חיים אחרים.
7. תיקון וצביעת MEMAs
- לאחר 72 h וכל טיפולי תאים חיים, מנושמים בארות. תקן MEMAs ב 2 מ ל לכל טוב של 2% פאראמפורמלדהיד (בתחתית) עבור 15 דקות at RT.
- . מתיף את הכדורגלן החדירות עם 2 מ ל לבאר של 0.1% nonionic החומרים עבור 15 דקות.
- ולשטוף עם 2 מ ל לכל טוב של PBS. . שמתי את הערוץ לשטוף עם 2 מ ל של PBS עם 0.05% polysorbate 20 (PBS-T).
הערה: המשטח MEMA הוא הידרופובי, וכישלון לשטוף עם PBS-T לפני דגירה הכתם נוגדן יגרום להיווצרות חללים בבארות במהלך שלבי הדגירה ולתת היווצרות חפצי הצביעת. - . מנושף את הPBS-טי הוסף את התגובה של EdU לגילוי ריאגנטים. הגנה מפני 1 h ב RT, נדנדה ומוגן מפני אור. אחרי 1 h הדגירה, תגובת כיבוי עם מאגר כיבוי מסחרי סיפק.
הערה: EdU שלבים לזיהוי וכתמים/נוגדנים עשויים להתבצע ב-1.5 mL לכל טוב כדי להפחית את העלות. - ולשטוף עם PBS-T לפני הדגירה עם כתמים או נוגדנים.
- מודדת mema בארות עם נוגדנים נגד היסטון H3K9me3 (1:1000) ו fibrillarin (1:400) במאגר מכתים המכיל 2% (w/v) סרום פרה אלבומין (bsa), 1 מ"מ mgcl2 ו 0.02% נאן3 לילה ב 4 ° c.
הערה: ביצוע החדירות נוגדן כדי לקבוע ריכוזים אופטימליים לפני השימוש בהם על ערכת MEMA מלא. - לאחר הנוגדן העיקרי או הדגירה כתם, לשטוף את הבארות 2x עם PBS ופעם עם PBS-T.
- הוסף נוגדנים משניים (חמור אנטי עכבר IgG וחמור אנטי ארנב IgG, שניהם 1:300) ו 0.5 μg/mL 4 ′ 6 ‐ diamidino ‐ 2 ‐ פניינילידול (DAPI). . בסדר.
- לשטוף את הבארות 2x עם 2 מ ל עבור כל טוב של PBS, להשאיר אותם בגמר 2 mL PBS.
- להמשיך להדמיה או לאחסן MEMAs מוכתם להדמיה מאוחר יותר ב-PBS ב-4 ° צ' מוגן מפני אור.
8. הדמיה של MEMAs
- תמונה MEMA במערכת הדמיה אוטומטית עם ערוצים מתאימים לזיהוי פלורסנט.
- פלט נתוני תמונה שהתקבלו למערכת ניהול תמונות. פלח תאים וחשב רמות אינטנסיביות באמצעות CellProfiler8.
9. ניתוח נתונים
הערה: ניתוח נתונים מורכב מנורמליזציה, תיקון וריאציה וסיכום של הנתונים הנגזרים מחברת CellProfiler הגולמית. במקרה זה, הסביבה R עם קוד מותאם אישית משמשת לביצוע כל השלבים. עם זאת, כל סביבה או תוכנית תוכנה סטטיסטית יכולים להיות מנוצלים כדי לבצע את הפעולות המקבילות. דוגמה לקוד המותאם אישית של המקור הפתוח עבור הסביבה R לניתוח זמינה ב: https://www.synapse.org/#!Synapse:syn2862345/wiki/72486.
- לעבד מראש ולנרמל את נתוני התמונה מקוטע.
- קבע ספירת תאי ספוט תוך שימוש בגרעינים המוכתמים של DAPI.
- עוצמת שער אוטומטי EdU כדי לתייג תאים כ-EdU+. מדידת התפשטות באמצעות הפרופורציה של EdU+ תאים בכל נקודה.
- חציון מסכם כתמים cytoplasmic ומדידות המבנה הגרעיני ברמת ספוט.
- בצע הסרה של נורמליזציה לא רצויה (RUV) בנתונים כדי לשפר את איכות הנתונים9.
הערה: גישה זו מוחלת על כל עוצמה ואות מורפולוגיה באופן עצמאי כמטריצה עם מערכים תוך שימוש בשורות ובנקודות כמו העמודות כמתואר בעבר9. - החל נרמול מחלות bivariate ל-RUV מנורמלים מנורמל באמצעות שורת המערך ועמודת המערך כמשתנים עצמאיים כדי לתקן את ההשפעות הקשורות למרחב או לאינטנסיביות.
- לאחר השלמת הנורמליזציה, החציון מסכם את המשכפלת עבור כל תנאי של סביבת מיקרו עבור דיווח וניתוח נוסף.
תוצאות
כדי לפשט השפעות מיקרוסביבתיות על צמיחת תאים והתפשטות ועל מנת לזהות תנאים שקידמו או מעכבות את צמיחת התאים וההפצה, התאים הסרטניים בסרטן השד MCF7 הופרה על סט של שמונה 8-טוב MEMAs כפי שמתואר בפרוטוקול. שיטת הפעולה חשפה את התאים ל-48 ECMs שונים ו57 ליגפות שונות, עבור סך של 2736 קומבינטורית מיקרו תנאים סביבתיים. לאחר 71 h בתרבות, תאים היו פעמו עם EdU, קבוע, חדירות, והכתים עם DAPI, התגובה של EdU זיהוי, נוגדן אנטי fibrillin, ו נוגדן anti-H3K9me3. התאים התהיו בתמונה במיקרוסקופ תוכן גבוה. התמונות הועלו לשרת Omero10, מחולק באמצעות cellprofiler8, מנורמל וניתח ב-R9. התוצאות המתוארות להלן מתמקדות באותות DAPI ו-EdU.
פלטפורמת ניתוח תמונה של memas תשואות כמה תוצאות דומות לאלה הזמינות של הגישות cy, כגון מגרשים תוכן DNA מראה 2n ו-4n שברים עבור תאים שטופלו עם ליגנד (איור 3a), מבוסס על dapi עוצמה ואזור. מגרשים אלה מספקים ראיות לתנאים המקדמים רכיבה על אופניים פעילים לעומת המצוין על ידי פסגות דו-אופנית ברור המתאימים לתאים ב-G1 או מG2 שלבים לעומת הצמיחה בתאים שנעצרו, אשר מראים שינויים בפסגות לעומת תנאי שליטה. אנו משתמשים במספר התאים ומכתים נתונים בעוצמה כדי לסכם את הנתונים, כאשר ההשפעה של המיקרו-סביבה (ליגטרים על ציר אחד, ECM בציר השני) בשני מספר התאים (איור 3ב) ו-EdU התאגדות (איור 3ג) ניתן לראות בקלות רבה יותר שינויים בצבע ובאינטנסיביות של מפת החום. כפי שנראה מתוך מגרשים אלה, רבים מהאפקטים הם ligand-מונחה, כמו מצבו של ECM לא השפיע מאוד על מספר התאים או EdU חיוביות. Nidogen -1 הוא חריג ברור, כנוכחות של מולקולת ECM זו מעכב את כריכת התא ואת הצמיחה של MCF7. ליגנדס כגון FGF6 ו-NRG1α (NRG 1.1 על מגרשים) לשפר את מספר התא ויש להם שיעור גבוה של התאגדות EdU, בעוד ליגנדס כגון AREG ו NRG1-smdf (NRG 1.10 על מגרשים) לעכב את כריכת התא ו/או צמיחה של תאים. ממצאים אלה נתמכים על ידי תמונות של התאים הגדלים על כתמים, שם הבדל ברור מספר התא ו-EdU חיוביות הוא ניכר (ראה למשל באיור 3ד).
מאחר שפלטפורמת MEMA היא טכנולוגיה חדשה יותר, התוצאות אומתו בהסכמה נפרדת. MCF7 תאים שופרה לתוך 24-היטב צלחות מצופות עם קולגן אני במדיום Dמאמ עם 10% FBS. לאחר 18 h, התקשורת הוחלפו עבור בינונית צמיחה מופחתת (DMEM עם 0.1% FBS) ותאים טופלו עם NRG1α, FGF6, או AREG ותרבותי עבור 72 h. EdU התווסף 1 h לפני קיבעון. תאים היו מוכתמים ב-DAPI ועבור EdU התאגדות, התמונה, מקוטע, וניתח. בדומה לתוצאות שהתקבלו מפלטפורמת mema, FGF6 ו-NRG1α שניהם העניקו מספרי תאים גבוהים יותר (איור 4A) ו-EdU שיעורי התאגדות (איור 4B) לעומת התאים התייחסו areg, אימות התצפיות שלנו ב ניסויי MEMA המקורי.

איור 1 : תרשים זרימה המציג את זרימת העבודה ואת ציר הזמן עבור השלבים השונים של ניסוי MEMA טיפוסי. לאחר הדפסת MEMAs, הם יכולים להיות מאוחסנים בטמפרטורת החדר מיובש במשך מספר חודשים לפני השימוש. בדרך כלל, השלב הנסיוני נמשך 3 עד 4 ימים, אבל כמה תאים העיקריים הגדלים איטי כבר מתורבתים על MEMAs עד 2 שבועות. אנא לחץ כאן כדי להציג גירסה גדולה יותר של איור זה.

איור 2 : פריסת מקור Ecm להדפסת מערך. בלוק הקולגן מודפס על MEMA כרשת, אשר מספק מערכת מאוד חוזרים ונשנים המאפשרים נורמליזציה חזקה יותר בין בארות. הבארות המלאות מספקות לחות לסיוע במניעת התאיידות במהלך תהליך ההדפסה. אנא לחץ כאן כדי להציג גירסה גדולה יותר של איור זה.

איור 3 : דוגמאות של נתונים שנוצרו מניסוי MEMA טיפוסי. (א) מחזור התא פרופילים של ערכי האינטנסיביות dapi בעוצמה לעומת ספירת תאים מאחת 8-הבאר מטופלים עם ליגניות שונות, מראה biphasic dapi מכתים את התאים המצביעים בתאי G1 לעומת המחזור מחזור התא G2. (ב) מפת חום הצגת ספירות של תאי ספוט מנורמל באשכולות לפי דמיון באמצעות קיבוץ באשכולות הירארכי. אדום מציין מספר תאים גבוה יותר וכחול הוא מספר תא נמוך יותר. ליגנדס על ציר ה-x, ECMs נמצאים על ציר y. (ג) מיפוי החום מציג שילוב של מנורמל edu, כאשר אדום המציין שילוב גבוה יותר וכחול המעיד על התאגדות של edu נמוכה יותר. ליגנדס על ציר ה-x, ECMs נמצאים על ציר y. (ד) דוגמה לתאים MCF7 הגדלים בספוט mema שטופלו ב-NRG1-α המציגה שיעור גבוה של שילוב של EdU (גרעינים ורודים). כתם ירוק הוא מסכת תאים וכחול הוא DAPI. אנא לחץ כאן כדי להציג גירסה גדולה יותר של איור זה.
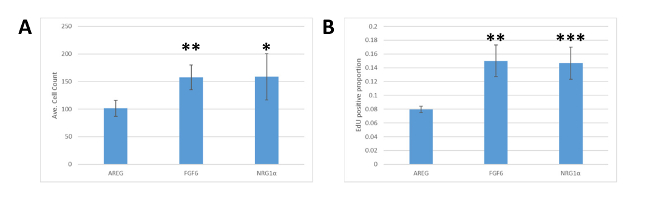
איור 4 : ואלידציה של תוצאות MEMA בתרבית תאים. (א) כימות של מספר תא כתוצאה מטיפול בMCF7 עם ליגפות שונות. מספרים שווי ערך של תאים MCF7 היו מצופים לתוך לוחות multiwall ולאחר מכן מטופלים עם AREG, FGF6, או NRG1α. ולס מטופלים עם AREG היו באופן משמעותי פחות תאים מאשר אלה שטופלו עם FGF6 (* * המציין t-הבדיקה של התלמיד p-ערכים פחות מ 0.01) או NRG1α (* מציין ערך p של 0.05) ב 72 h post ו טיפול. (ב) קוונפיקציה של רמת התאגדות של EDU ב MCF7 עקב טיפול עם ליגפות שונות, כמו פאנל A. AREG תוצאות הטיפול בחלק נמוך באופן משמעותי של תאים המשלבים EdU מאשר תאים שטופלו עם FGF6 (* *, p < 0.01) או NRG1α (* * *, p = 0.01). קווי שגיאה מייצגים סטיית תקן. אנא לחץ כאן כדי להציג גירסה גדולה יותר של איור זה.
| שם החלבון | מזהה יוניפרוט | לאי ריכוז (μg/mL) | סופי ריכוז (μg/mL) |
| ANGPT1 | 1 | Q15389 | 1 | 100 | 0.04 |
| ANGPT2 | 1 | O15123 | 1 | 100 | 0.2 |
| ארג'י | P15514 | 100 | 0.02 |
| BMP2 | P12643 | 100 | 0.1 |
| BMP3 | P12645 | 1000 | 0.1 |
| BMP4 | P12644 | 100 | 0.1 |
| BMP5 | 1 | P22003 | 1 | 100 | 0.1 |
| BMP6 | P22004 | 100 | 0.1 |
| BMP7 | P18075 | 100 | 0.1 |
| CSF2 | P04141 | 100 | 0.02 |
| CTGF | 1 | P29279 | 1 | 100 | 0.05 |
| CXCL12 | אלפא | P48061 | 2 | 100 | 0.01 |
| CXCL12 | בטא | P48061 | 1 | 100 | 0.03 |
| CXCL1 | P09341 | 100 | 0.004 |
| CXCL8 | 1 | P10145 | 1 | 100 | 0.3 |
| DLL1 | 1 | O00548 | 1 | 500 | 0.5 |
| DLL4 | Q9NR61 | 200 | 0.6 |
| EGF | 1 | P01133 | 1 | 500 | 0.01 |
| FASLG | 1 | P48023 | 1 | 10 | 0.02 |
| FGF2 | 3 | P09038 | 2 | 100 | 0.01 |
| FGF6 | P10767 | 100 | 0.01 |
| FLT3LG | 1 | P49771 | 1 | 50 | 0.001 |
| GPNMB | 1 | Q14956 | 1 | 100 | 0.5 |
| HGF | 1 | P14210 | 1 | 50 | 0.04 |
| IGF1 | 1 | P05019 | 1 | 200 | 0.01 |
| IGFBP2 | P18065 | 100 | 0.05 |
| IGFBP3 | 1 | P17936 | 1 | 100 | 0.1 |
| IL13 | P35225 | 100 | 0.01 |
| IL15 | IL15S48AA | P40933 | 1 | 50 | 0.01 |
| IL1B | P01584 | 25 | 0.001 |
| IL6 | P05231 | 100 | 0.01 |
| IL7 | 1 | P13232 | 1 | 100 | 0.01 |
| JAG1 | 1 | P78504 | 1 | 200 | 0.5 |
| JAG2 | זמן | Q9Y219 | 1 | 100 | 0.5 |
| KITLG | 1 | P21583 | 1 | 100 | 0.005 |
| KNG1 | היחידה ל, HMW | P01042 | 1 | 100 | 0.2 |
| LEP | P41159 | 1000 | 0.002 |
| LYVE1 | Q9Y5Y7 | 100 | 0.05 |
| NRG1 | 10 | Q02297 | 10 | 100 | 0.01 |
| NRG1 | 1 | Q02297 | 1 | 100 | 0.05 |
| NRG1 | 6 | Q02297 | 6 | 100 | 0.01 |
| היחידה ל, האוקיינוס האטלנטי | go1990265 | 100 | 0.05 |
| PDGFB | 1 | P01127 | 1 | 100 | 0.05 |
| PTN | P21246 | 100 | 0.5 |
| ששש | Q15465 | 100 | 0.5 |
| TGFB1 | | קטרמינוס | P01137 | קטרמינוס | 20 | 0.01 |
| TGFB1 | | ברכיים | P01137 | ברכיים | 100 | 0.15 |
| TGFB2 | קצת | P61812 | 1 | 20 | 0.01 |
| THPO | 1 | P40225 | 1 | 50 | 0.002 |
| TNFRSF11B | O00300 | 100 | 0.02 |
| TNFSF11 | 1 | O14788 | 1 | 100 | 0.01 |
| TNF | P01375 | 100 | 0.01 |
| ובכל היותר | VEGF206 | P15692 | 1 | 100 | 0.01 |
| WNT10A | Q9GZT5 | 100 | 0.1 |
| WNT3A | 1 | P56704 | 1 | 200 | 0.1 |
| Wnt5a | 1 | P22725 | 1 | 100 | 0.1 |
טבלה 1: הרשימה המלאה של ליגניות המשמשות לניסויי MEMA. המזהה היחיד, ריכוזי המניות וריכוזי העבודה הסופיים מסופקים.
| חלבון ECM | UniprotID | מניות ריכוז (μg/mL) | סופי ריכוז (μg/mL) | ערות |
| ALCAM | 1 | Q13740 | 1 | 100 | 30 | |
| CDH20 | Q9HBT6 | 300 | 80 | |
| CDH6 | 1 | P55285 | 1 | 100 | 40 | |
| CDH8 | P55286 | 100 | 20 | |
| CD44 | 1 | P16070 | 1 | 100 | 30 | |
| CEACAM6 | P40199 | 100 | 30 | |
| COL1A1 | P02453 | 5000 | 200 | יחידות משנה מרובות עם מספר מזהי ייחודיות |
| COL2A1 | 2 | P02458 | 2 | 1000 | 200 | |
| COL3A1 | 1 | P02461 | 1 | 1000 | 200 | |
| COL4A1 | 1 | P02462 | 1 | 1000 | 200 | יחידות משנה מרובות עם מספר מזהי ייחודיות |
| COL5A1 | P20908 | 1000 | 200 | |
| COL23A1 | 1 | Q86Y22 | 1 | 200 | 80 | |
| DSG2 | Q14126 | 100 | 30 | |
| CDH1 | 1 | P12830 | 1 | 100 | 40 | |
| ECM1 | 1 | Q16610 | 1 | 100 | 40 | |
| FN1 | 1 | P02751 | 1 | 1000 | 200 | |
| GAP43 | 1 | P17677 | 1 | 158 | 40 | |
| מיכל מק | 1000 | 200 | 0005 לור | |
| מיכל שלמה | 1000 | 200 | 0007 לור | |
| ICAM1 | P05362 | 400 | 80 | |
| ALCAM | 1 | Q13740 | 1 | 100 | 30 | |
| CDH20 | Q9HBT6 | 300 | 80 | |
| CDH8 | P55286 | 100 | 20 | |
| CD44 | 1 | P16070 | 1 | 100 | 30 | |
| CEACAM6 | P40199 | 100 | 30 | |
| DSG2 | Q14126 | 100 | 30 | |
| CDH15 | P55291 | 100 | 20 | |
| VCAM1 | 1 | P19320 | 1 | 1000 | 200 | |
| LAMA1 | P25391 | 500 | 200 | יחידות משנה מרובות עם מספר מזהי ייחודיות |
| LAMA3 | 2 | Q16787 | 2 | 130 | 40 | |
| לאם | P51884 | 200 | 80 | |
| CDH15 | P55291 | 100 | 20 | |
| NID1 | 1 | P14543 | 1 | 100 | 9.3 μg/mL ניד, 130 μg/mL לאם, 46.5 μg/mL COL4 | + COL4 ולמינציה |
| OMD | Q99983 | 100 | 40 | |
| SPP1 | קצת | P10451 | 1 | 100 | 40 | |
| CDH3 | 1 | P22223 | 1 | 100 | 40 | |
| PECAM1 | זמן | P16284 | 1 | 150 | 40 | |
| TNC | 1 | P24821 | 1 | 500 | 200 | |
| VCAM1 | 1 | P19320 | 1 | 1000 | 200 | |
| היכל ה, VTN | P04004 | 100 | 40 | |
| BGN | P21810 | 100 | 40 | |
| DCN | קצת | P07585 | 1 | 300 | 80 | |
| POSTN | 1 | Q15063 | 1 | 100 | 40 | |
| ב SPARC | P09486 | 100 | 40 | |
| THBS1 | 1 | P07996 | 1 | 100 | 40 | |
| B האם | 1 | Q96GW7 | 1 | 100 | 40 | |
| ELN | 3 | P15502 | 3 | 1000 | 200 | |
| FBN1 | P35555 | 254 | 80 |
טבלה 2: הרשימה המלאה של חלבוני ECM והתנאים המשמשים בניסויי MEMA. המזהה היחיד, ריכוזי המניות וריכוזי העבודה הסופיים מסופקים. במקרים מסוימים, התנאי המודפס מייצג מתחם חלבונים או שילוב של מספר חלבונים, המצוין בעמודה Notes.
Discussion
חשיבותה של "ממדי" והקשר היה גורם מניע בפיתוח במערכות תרבות מבחנה ככלים לאפיון תאים סרטניים באמצעות האינטראקציה שלהם עם המיקרו-סביבה11, והיכולת של מבחנה מערכות תרבות לחקות את הסביבה vivo הוא כוח המניע מאחורי החיפוש כדי לשפר את מערכות התרבות. אולם, במערכות חוץ גופית, נותרו כלים משמעותיים של מחקר הסרטן בדיוק בשל יכולתם לזקק את הקומפלקס במצב vivo למטה לדגם מפושט12.
למרות שמערכות דו-ממדיות יכולות לכלול ECMs וליגנדס, הן היו חסרה באופן מסורתי בקיבולות התפוקה כדי לחקור פאנל רחב של קומבינטורית pertubagens. פופולרי מסחרי המרתף תמציות הקרום לאפשר culturing ב-3D, אבל חוסר מקורו של פאנל מוגדר בקפידה של חלבונים. תמציות מסחריות סובלים בדרך כלל מהרכב מוגדר לחלוטין, אשר יכול לבלבל את הניתוח ולגרום משמעותי אצווה-to-אצווה וריאציה3,13. פלטפורמת MEMA גוברת על המחסומים האלה, המאפשר לחקר שינויים בפנוטיפים סלולריים, פעילות מטבולית, בידול סטטוס, וריאציות של צמיחת תאים והתפשטות כפי שהם מאופנן על ידי אנדודוגני ספציפיים ומוגדרים גורמים.
פלטפורמת MEMA היא גישה רבת עוצמה, בינונית לתפוקה גבוהה כדי להעריך את ההשפעה של המיקרו-סביבה (הן הגורמים ECM ו מסיסים) על הפנוטיפ של תאים. הפלטפורמה מראה גמישות רבה עבור סוגי בחני ותאים שעבורם ניתן לעשות שימוש. אנו יכולים להתבונן בהשפעות משני הליטרים מסיסים ומחלבוני ECM שאליהם נחשפים התאים. אכן, לאחרונה מצאנו כי ליגנדס היה הנהג העיקרי של התנגדות HER2 ממוקדות מעכבי, אבל השפעות אלה יכול להיות מאופנן על ידי ECM5. מגוון של תאים, כולל תאים ראשוניים וקווי התא נגזר סוגי תאים שונים כולל ריאות, שלפוחית השתן, ערמונית, שד, ולבלב, כמו גם המושרה (שב ס) תאים גזע (iPS), היו מתורבתים בהצלחה על פלטפורמת MEMA (לראות דוגמאות בהפניות5,7,14). השימוש בכתמים שונים מאפשר את הבדיקה של נקודות קצה סלולריות מרובות, כולל צמיחת תאים, בידול וחילוף חומרים. חוקרים אחרים הרחיבו את הפלטפורמה כדי לחקור את ההשפעה של נוקשות או מודול אלסטי, הוספת ממד נוסף לפלטפורמת MEMA15. לבסוף, הפלטפורמה היא קלה לביצוע מסכי סמים לזיהוי מצבי סביבה מיקרו המשפרים או מעכבים את יעילות הסמים, כפי שאנו ואחרים דיווחו לאחרונה5,14,15 .
ייתכן שהצעד הקריטי ביותר להצלחת ניסוי MEMA ממטב את צפיפות הציפוי בתאים. אופטימיזציה של צפיפות התאים מבטיחה כי מספיק תאים נוכחים כדי לספק נתונים חזקים, אבל לא כל כך הרבה כי המקום הופך להיות מיותר מדי. מקומות confluent יכולים באופן משמעותי לבלבל את התוצאות, במיוחד אם התפשטות משמש כנקודת קצה, מה שהופך את זה בלתי אפשרי לקבוע אם שיעורי התפשטות נמוכה הם תוצאה של אינטראקציות עם גורמים מיקרוסביבתיים או עקב העיכוב המגע מ צפיפות תאית גבוהה. הניסויים תא טיטור יכול לחשוף את הבעיות האלה, כמו מספרי התאים הממוצעים בנקודה יהיה להפגין עלייה ליניארית עם הגדלת מספר התאים מצופה, אבל בסופו של דבר מישור. יש לבחור את מספר התא האופטימלי בטווח הליניארי של העקומה.
כפי שהוזכר לעיל, פלטפורמת MEMA היא גמישה והוא יכול להיות מוכן על מגוון של מצעים עם משטחים שונים. אלה כוללים שקופיות זכוכית ותבניות לוחית מרובת קירות. בניסיון שלנו, לא כל פני השטח chemistries הם להדפסה MEMA, כפי שראינו התנתקות ספוט על משטחים מסוימים בשל תכונות הדבקה עניים וחוסר יכולת לחסום הדבקה תא על משטחים אחרים הדבקה גבוהה. יתר על כן, שינוי בין שונות שונים מחייבת אופטימיזציה של תנאי מאגר, כמו ביצועי ההדפסה עם מאגר ההדפסה זהה יכול להשתנות בהתאם לכימיה פני השטח.
קוטרו של מקומות ECM המודפסים ממלא תפקיד חשוב באיכות הנתונים. באופן כללי, אנו ממליצים באמצעות סיכות הדפסה בקוטר הגדול ביותר הזמינים עבור הארייר בשימוש (אנחנו כרגע משתמשים בפינים בקוטר יקרומטר 350). נקודות בקוטר גדולות יותר מאפשרות למספר גדול יותר של תאים לכבוש נקודה, שנוטה לגרום לנתונים חזקים יותר מאשר שנוצרו באמצעות פינים בקוטר קטן יותר. מאחר שאיגוד התאים הוא תהליך סטוכסטי, יש נטייה להיות מידה גבוהה של שינויים בנתונים הקשורים למספר התאים המחוברים במקור לכל נקודה. לכן, אנו ממליצים להדפיס מספר רב של משכפל עבור כל תנאי ECM. אנו מדפיסים 10/15 לשכפל ECM בכל הקשור לתנאי ההדפסה הנוכחיים שלנו כדי להבטיח סטטיסטיקות חזקות.
אנו ציינו בניסויים הקודמים שלנו שרוב החלקים, הליגוהאפקטים נוטים להשתלט על אפקטי ECM. זה עשוי להיות חלק בגלל ההחלטה שלנו להוסיף קולגן אני לכל כתמים ECM, אשר מבטיח כריכת תאים חזקים. עם זאת, אנו מאמינים כי זה עשוי גם המגון את ההשפעות ECM, כמו רוב המקומות נוטים להתנהג בצורה דומה מאוד קולגן אני. שינוי הרכב ספוט להוציא קולגן אני עלול לגרום להתנהגות תא דיפרנציאלי כתוצאה של אינטראקציה עם ECM, אבל גם משפיעה באופן משמעותי על כריכת התא, וכתוצאה מכך מקומות רבים יותר פנויים. משתמשים צריכים להתאים את הרכב ECM שלהם שמירה על הבדלים אלה בראש, במיוחד אלה המעוניינים בתאי גזע ומחולל קדמון ובידול, שם המטריצה יכולה להיות השפעה משמעותית16.
אנחנו בדרך כלל לבצע את mema בחני במשך תקופה קצרה יחסית של זמן (למשל, 72 h מקסימום). הסיבה לכך היא שהתאים מוגבל לנקודות (המאגר החוסם אינו מאפשר צמיחה מחוץ לנקודות הניסיון שלנו). עם חלוקת תאים במהירות, צמיחה יותר מ 72 h יוביל לצמיחת יתר של המקום, אשר בתורו מסבך את פילוח התמונה כמו תאים להיות צפוף להצטבר אחד על השני, והוא יכול גם להשפיע על הנתונים כמו מעצר גדילה יכול להתרחש עם עיכוב מגע. ביצעו טיפולים ארוכים יותר עם תאים ראשוניים הגדלים לאט מאוד (10-14 ימים), אבל יש לנקוט באמצעים אלה כדי לשנות את התקשורת ולחדש לחדש כל 3-4 ימים.
מאמצים ממשיכים לפתח את פלטפורמת MEMA מתמקדים שני תחומי עניין, מקסימיזציה של האיכות האופטית לדימות ואופטימיזציה בתוך כלי התרבות הקטנים. איכות אופטית הופכת גורם מכריע כאשר החוקרים דורשים מיקרוסקופ ברזולוציה גבוהה יותר כדי לזהות לוקליזציה subcellular של סמני העניין שלהם. מסכים ראשוניים ניתן לבצע ברזולוציה נמוכה יותר על מיקרוסקופים תפוקה גבוהה ואחריו הדמיה של נקודות עניין ספציפיות על מכשירים ברזולוציה גבוהה יותר, אבל איכות התמונה יכול להיות בסכנה אם המאפיינים האופטיים של המצע הם עניים. שיפור התכונות האופטיות של המצע יאפשר לחוקרים לבצע את המסכים הראשוניים על מערכות דימות ברזולוציה גבוהה ללא צורך לרכוש מחדש תמונות נבחרות ברזולוציה גבוהה יותר. לבסוף, היכולת לבצע MEMAs בכלי תרבות קטנים יותר, כגון 96-צלחות, תאפשר הפחתה בנפח הטיפול והתרחבות של ליגפות ושכפול נחקרים. מעבר זה מחייב אופטימיזציה של אינטראקציות המצע-חלבון-מאגר-חלבונים והדפסת מערך בתוך כלי תרבות חדשים. מאמצים מתמשכים כאלה ישפרו את פלטפורמת mema ותרחיב על יכולותיה החזקות כדי לזהות חלבונים מיקרוסביבתיים רלוונטיים המשנים את הפנוטיפים הסלולריים למגוון סוגי תאים, שניתן לאחר מכן לחקור לאחר מכן ב confirmatory אמר.
Disclosures
. למחברים אין מה לגלות
Acknowledgements
עבודה זו נתמכת על ידי ספריית הקרן המשותפת של NIH של הרשת חתימות סלולריות (LINCS) גרנט HG008100 (J.W.G., L.M.H., ו-J. E. K).
Materials
| Name | Company | Catalog Number | Comments |
| Aushon 2470 | Aushon BioSystems | Arrayer robot system used in the protocol | |
| Nikon HCA | Nikon | High Content Imaging system designed around Nikon Eclipse Ti Inverted Microscope | |
| BioTek Precision XS liquid Handler | BioTek | liquid handling robot used in the protocol | |
| Trizma hydrochloride buffer solution | Sigma | T2069 | |
| EDTA | Invitrogen | 15575-038 | |
| Glycerol | Sigma | G5516 | |
| Triton X100 | Sigma | T9284 | |
| Tween 20 | Sigma | P7949 | |
| Kolliphor P338 | BASF | 50424591 | |
| 384-well microarray plate, cylindrical well | Thermo Fisher | ab1055 | |
| Nunc 8 well dish | Thermo Fisher | 267062 | |
| Paraformaldehyde 16% solution | Electron Microscopy Science | 15710 | |
| BSA | Fisher | BP-1600 | |
| Sodium Azide | Sigma | S2002 | |
| Cell Mask | Molecular Probes | H32713 | |
| Click-iTEdU Alexa Fluor | Molecular Probes | C10357 | |
| DAPI | Promo Kine | PK-CA70740043 | |
| ALCAM | R & D Systems | 656-AL | ECM |
| Cadherin-20 (CDH20) | R & D Systems | 5604-CA | ECM |
| Cadherin-6 (CDH6) | R & D Systems | 2715-CA | ECM |
| Cadherin-8 (CDH8) | R & D Systems | 188-C8 | ECM |
| CD44 | R & D Systems | 3660-CD | ECM |
| CEACAM6 | R & D Systems | 3934-CM | ECM |
| Collagen I | Cultrex | 3442-050-01 | ECM |
| Collagen Type II | Millipore | CC052 | ECM |
| Collagen Type III | Millipore | CC054 | ECM |
| Collagen Type IV | Sigma | C5533 | ECM |
| Collagen Type V | Millipore | CC077 | ECM |
| COL23A1 | R & D Systems | 4165-CL | ECM |
| Desmoglein 2 | R & D Systems | 947-DM | ECM |
| E-cadherin (CDH1) | R & D Systems | 648-EC | ECM |
| ECM1 | R & D Systems | 3937-EC | ECM |
| Fibronectin | R & D Systems | 1918-FN | ECM |
| GAP43 | Abcam | ab114188 | ECM |
| HyA-500K | R & D Systems | GLR002 | ECM |
| HyA-50K | R & D Systems | GLR001 | ECM |
| ICAM-1 | R & D Systems | 720-IC | ECM |
| Laminin | Sigma | L6274 | ECM |
| Laminin-5 | Abcam | ab42326 | ECM |
| Lumican | R & D Systems | 2846-LU | ECM |
| M-Cad (CDH15) | R & D Systems | 4096-MC | ECM |
| Nidogen-1 | R & D Systems | 2570-ND | ECM |
| Osteoadherin/OSAD | R & D Systems | 2884-AD | ECM |
| Osteopontin (SPP) | R & D Systems | 1433-OP | ECM |
| P-Cadherin (CDH3) | R & D Systems | 861-PC | ECM |
| PECAM1 | R & D Systems | ADP6 | ECM |
| Tenascin C | R & D Systems | 3358-TC | ECM |
| VCAM1 | R & D Systems | ADP5 | ECM |
| Vitronectin | R & D Systems | 2308-VN | ECM |
| Biglycan | R & D Systems | 2667-CM | ECM |
| Decorin | R & D Systems | 143-DE | ECM |
| Periostin | R & D Systems | 3548-F2 | ECM |
| SPARC/osteonectin | R & D Systems | 941-SP | ECM |
| Thrombospondin-1/2 | R & D Systems | 3074-TH | ECM |
| Brevican | R & D Systems | 4009-BC | ECM |
| Elastin | BioMatrix | 5052 | ECM |
| Fibrillin | Lynn Sakai Lab OHSU | N/A | ECM |
| ANGPT2 | RnD_Systems_Own | 623-AN-025 | Ligand |
| IL1B | RnD_Systems_Own | 201-LB-005 | Ligand |
| CXCL8 | RnD_Systems_Own | 208-IL-010 | Ligand |
| IGF1 | RnD_Systems_Own | 291-G1-200 | Ligand |
| TNFRSF11B | RnD_Systems_Own | 185-OS | Ligand |
| BMP6 | RnD_Systems_Own | 507-BP-020 | Ligand |
| FLT3LG | RnD_Systems_Own | 308-FK-005 | Ligand |
| CXCL1 | RnD_Systems_Own | 275-GR-010 | Ligand |
| DLL4 | RnD_Systems_Own | 1506-D4-050 | Ligand |
| HGF | RnD_Systems_Own | 294-HGN-005 | Ligand |
| Wnt5a | RnD_Systems_Own | 645-WN-010 | Ligand |
| CTGF | Life_Technologies_Own | PHG0286 | Ligand |
| LEP | RnD_Systems_Own | 398-LP-01M | Ligand |
| FGF2 | Sigma_Aldrich_Own | SRP4037-50UG | Ligand |
| FGF6 | RnD_Systems_Own | 238-F6 | Ligand |
| IL7 | RnD_Systems_Own | 207-IL-005 | Ligand |
| TGFB1 | RnD_Systems_Own | 246-LP-025 | Ligand |
| PDGFB | RnD_Systems_Own | 220-BB-010 | Ligand |
| WNT10A | Genemed_Own | 90009 | Ligand |
| PTN | RnD_Systems_Own | 252-PL-050 | Ligand |
| BMP3 | RnD_Systems_Own | 113-BP-100 | Ligand |
| BMP4 | RnD_Systems_Own | 314-BP-010 | Ligand |
| TNFSF11 | RnD_Systems_Own | 390-TN-010 | Ligand |
| CSF2 | RnD_Systems_Own | 215-GM-010 | Ligand |
| BMP5 | RnD_Systems_Own | 615-BMC-020 | Ligand |
| DLL1 | RnD_Systems_Own | 1818-DL-050 | Ligand |
| NRG1 | RnD_Systems_Own | 296-HR-050 | Ligand |
| KNG1 | RnD_Systems_Own | 1569-PI-010 | Ligand |
| GPNMB | RnD_Systems_Own | 2550-AC-050 | Ligand |
| CXCL12 | RnD_Systems_Own | 350-NS-010 | Ligand |
| IL15 | RnD_Systems_Own | 247-ILB-005 | Ligand |
| TNF | RnD_Systems_Own | 210-TA-020 | Ligand |
| IGFBP3 | RnD_Systems_Own | 675-B3-025 | Ligand |
| WNT3A | RnD_Systems_Own | 5036-WNP-010 | Ligand |
| PDGFAB | RnD_Systems_Own | 222-AB | Ligand |
| AREG | RnD_Systems_Own | 262-AR-100 | Ligand |
| JAG1 | RnD_Systems_Own | 1277-JG-050 | Ligand |
| BMP7 | RnD_Systems_Own | 354-BP-010 | Ligand |
| TGFB2 | RnD_Systems_Own | 302-B2-010 | Ligand |
| VEGFA | RnD_Systems_Own | 293-VE-010 | Ligand |
| IL6 | RnD_Systems_Own | 206-IL-010 | Ligand |
| CXCL12 | RnD_Systems_Own | 351-FS-010 | Ligand |
| NRG1 | RnD_Systems_Own | 378-SM | Ligand |
| IGFBP2 | RnD_Systems_Own | 674-B2-025 | Ligand |
| SHH | RnD_Systems_Own | 1314-SH-025 | Ligand |
| FASLG | RnD_Systems_Own | 126-FL-010 | Ligand |
References
- Hanahan, D., Coussens, L. M. Accessories to the crime: functions of cells recruited to the tumor microenvironment. Cancer Cell. 21 (3), 309-322 (2012).
- Quail, D. F., Joyce, J. A. Microenvironmental regulation of tumor progression and metastasis. Nature Medicine. 19 (11), 1423-1437 (2013).
- Hughes, C. S., Postovit, L. M., Lajoie, G. A. Matrigel: a complex protein mixture required for optimal growth of cell culture. Proteomics. 10 (9), 1886-1890 (2010).
- LaBarge, M. A., et al. Human mammary progenitor cell fate decisions are products of interactions with combinatorial microenvironments. Integrative Biology (Cambridge). 1 (1), 70-79 (2009).
- Watson, S. S., et al. Microenvironment-Mediated Mechanisms of Resistance to HER2 Inhibitors Differ between HER2+ Breast Cancer Subtypes. Cell Systems. 6 (3), 329-342 (2018).
- Ranga, A., et al. 3D niche microarrays for systems-level analyses of cell fate. Nature Communications. 5, 4324 (2014).
- Malta, D. F. B., et al. Extracellular matrix microarrays to study inductive signaling for endoderm specification. Acta Biomater. 34, 30-40 (2016).
- Kamentsky, L., et al. Improved structure, function and compatibility for CellProfiler: modular high-throughput image analysis software. Bioinformatics. 27 (8), 1179-1180 (2011).
- Gagnon-Bartsch, J. A., Jacob, L., Speed, T. P. Removing Unwanted Variation from High Dimensional Data with Negative Controls. University of California, Berkeley, Department of Statistics, University of California, Berkeley. , (2013).
- Allan, C., et al. OMERO: flexible, model-driven data management for experimental biology. Nature Methods. 9 (3), 245-253 (2012).
- Simian, M., Bissell, M. J. Organoids: A historical perspective of thinking in three dimensions. Journal of Cell Biology. 216 (1), 31-40 (2017).
- Bissell, M. J. The differentiated state of normal and malignant cells or how to define a "normal" cell in culture. International Review of Cytology. 70, 27-100 (1981).
- Serban, M. A., Prestwich, G. D. Modular extracellular matrices: solutions for the puzzle. Methods. 45 (1), 93-98 (2008).
- Kaylan, K. B., et al. Mapping lung tumor cell drug responses as a function of matrix context and genotype using cell microarrays. Integrative Biology (Cambridge). 8 (12), 1221-1231 (2016).
- Lin, C. H., Jokela, T., Gray, J., LaBarge, M. A. Combinatorial Microenvironments Impose a Continuum of Cellular Responses to a Single Pathway-Targeted Anti-cancer Compound. Cell Reports. 21 (2), 533-545 (2017).
- Gjorevski, N., et al. Designer matrices for intestinal stem cell and organoid culture. Nature. 539 (7630), 560-564 (2016).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved