Method Article
استخدام المصفوفات الدقيقة لاستجواب الأثر البيئي الدقيق على الأنماط الظاهرية الخلوية في السرطان
In This Article
Summary
الغرض من الطريقة المعروضة هنا هو إظهار كيف يمكن تصنيع النُفَج ّة الدقيقة للبيئة الدقيقة (MEMA) واستخدامها لاستجواب تأثير آلاف البيئات الدقيقة المؤتلفة البسيطة على النمط الظاهري للخلايا المستزرعة.
Abstract
فهم تأثير البيئة الدقيقة على النمط الظاهري للخلايا مشكلة صعبة بسبب الخليط المعقد من كل من عوامل النمو القابلة للذوبان والبروتينات المرتبطة بالمصفوفة في البيئة الدقيقة في الجسم الحي. وعلاوة على ذلك، فإن الكواشف المتاحة بسهولة لنمذجة البيئات الدقيقة في المختبر تستخدم عادة خليطا معقدا من البروتينات التي يتم تعريفها بشكل غير كامل وتعاني من تقلب الدفعة إلى الدفعية. تسمح منصة microarray microenvironment (MEMA) بتقييم الآلاف من التركيبات البسيطة من بروتينات البيئة الدقيقة لتأثيرها على الأنماط الظاهرية الخلوية في اختبار واحد. يتم إعداد MEMAs في لوحات جيدا، والذي يسمح بإضافة الأربطة الفردية لفصل الآبار التي تحتوي على مصفوفة مصفوفة مصفوفة مصفوفة مصفوفة (ECM) البروتينات. مزيج من الرباط القابلة للذوبان مع كل ECM المطبوعة تشكل مزيجا فريدا من نوعه. يحتوي اختبار MEMA النموذجي على أكثر من 2500 بيئة صغيرة مؤتلفة فريدة من نوعها تتعرض لها الخلايا في اختبار واحد. كحالة اختبار، تم طلاء خط خلايا سرطان الثدي MCF7 على منصة MEMA. وقد حدد تحليل هذا التحليل العوامل التي تعزز وتعوق نمو وانتشار هذه الخلايا. منصة MEMA مرنة للغاية ويمكن تمديدها للاستخدام مع المسائل البيولوجية الأخرى خارج أبحاث السرطان.
Introduction
يبقى زراعة خطوط الخلايا السرطانية على البلاستيك في الطبقات الأحادية ثنائية الأبعاد (ثنائية الأبعاد) واحدة من خيول العمل الرئيسية للباحثين عن السرطان. ومع ذلك، يتم الاعتراف بشكل متزايد البيئة الدقيقة لقدرتها على التأثير على الأنماط الظاهرية الخلوية. في السرطان، ومن المعروف أن البيئة الدقيقة الورم تؤثر على السلوكيات الخلوية متعددة، بما في ذلك النمو، والبقاء على قيد الحياة، والغزو، والاستجابة للعلاج1،2. عادة ما تفتقر ثقافات الخلايا أحادية الطبقة التقليدية إلى تأثيرات البيئة الدقيقة، مما أدى إلى تطوير تجارب ثلاثية الأبعاد (ثلاثية الأبعاد) أكثر تعقيداً لزراعة الخلايا، بما في ذلك مستخلصات غشاء الطابق السفلي النقي المتاحة تجارياً. ومع ذلك، فإن هذه المصفوفات النقية عادة ما تكون معقدة للاستخدام وتعاني من مشاكل تقنية مثل تقلب الدفعة3 والتركيبات المعقدة3. ونتيجة لذلك، قد يكون من الصعب تعيين وظيفة لبروتينات محددة قد تؤثر على الأنماط الظاهرية الخلوية3.
لمعالجة هذه القيود، قمنا بتطوير تقنية microarray microenvironment (MEMA)، مما يقلل من البيئة الدقيقة وصولا ً إلى تركيبات بسيطة من المصفوفة خارج الخلية (ECM) وبروتينات عامل النمو القابلة للذوبان4،5 . منصة MEMA تمكن من تحديد العوامل البيئية الصغرية المهيمنة التي تؤثر على سلوك الخلايا. باستخدام تنسيق صفيف، يمكن وضع حد لآلاف المجموعات من عوامل البيئة الدقيقة في تجربة واحدة. ال [مّا] يوصف هنا يستجوب ~2,500 مختلفة فريدة [ميكروفيول] شروط. البروتينات ECM المطبوعة في لوحات جيدا تشكل منصات النمو التي يمكن أن تكون المستزرعة الخلايا. يتم إضافة الأربطة القابلة للذوبان إلى الآبار الفردية، مما يخلق بيئات صغيرة مؤتلفة فريدة من نوعها (ECM + ligand) على كل بقعة مختلفة تتعرض لها الخلايا. يتم زراعة الخلايا لعدة أيام، ثم ثابتة، ملطخة، وصورة لتقييم الأنماط الظاهرية الخلوية نتيجة للتعرض لهذه تركيبات البيئة الدقيقة المحددة. وبما أن البيئات الدقيقة هي تركيبات بسيطة، فمن السهل تحديد البروتينات التي تدفع التغيرات الفينوتية الرئيسية في الخلايا. وقد استخدمت MEMAs بنجاح لتحديد العوامل التي تؤثر على الأنماط الظاهرية الخلويةالمتعددة، بما في ذلك تلك التي تدفع قرارات مصير الخلية والاستجابة للعلاج 4،5،6،7. ويمكن التحقق من صحة هذه الاستجابات في تجارب بسيطة 2D ومن ثم يمكن تقييمها في ظل ظروف أن يلخص بشكل أكمل تعقيد البيئة الدقيقة الورم. منصة MEMA قابلة للتكيف إلى حد كبير مع مجموعة متنوعة من أنواع الخلايا ونقاط النهاية، شريطة أن تتوفر علامات حيوية فينوتيبيك جيدة.
Protocol
ملاحظة: ويرد في الرسم التخطيطي للتدفق المبين في الشكل 1عرض عام للعملية MEMA بأكملها، بما في ذلك الوقت المقدر. هذا البروتوكول تفاصيل تصنيع MEMAs في لوحات 8-جيدا. قد يتم تكييف البروتوكول للوحات أو شرائح أخرى.
1. إعداد البروتين، ومخفف، وتلطيخ المخازن المؤقتة
- قارورة متساوية من ECMs، الأربطة، والسيتوكينات إلى درجة حرارة الغرفة (RT) ولفترة وجيزة الطرد المركزي. إضافة وحدة التخزين المناسبة من المخزن المؤقت RT المناسبة كما هو موضح في ورقة بيانات المنتج. اتبع توصية الشركة المصنعة لتركيزات المخزون.
ملاحظة: وترد في الجدول 1 والجدول 2قائمة كاملة بالأنابيب والآليات المالية الأوروبية بمخزونها وتركيزاتها النهائية. وعادة ما تستخدم كل من الأربطة وECMs في أعلى تركيز من النطاق الموصى به من قبل الشركة المصنعة التي تثير تأثير بيولوجي في معيار 2 يوم اختبارات الثقافة. التعامل مع البروتينات بلطف وفي خزانات السلامة البيولوجية تحت تدفق laminar لتجنب التلوث. - احتضان قارورة مع هزاز لطيف في RT لمدة 1 ساعة. لا دوامة البروتينات لأن هذا يمكن أن يسبب لهم تشويه.
- البروتينات Aliquot للتخزين على المدى الطويل بحيث جميع aliquots هي استخدام واحد فقط لتجنب التدهور مع دورات التجميد / ذوبان المتكررة. تخزين البروتينات الليوفيلة في -80 درجة مئوية (ما لم ينص على خلاف ذلك) حتى الحاجة. الحرص على جمع جميع البيانات الوصفية للرجوع إليها في المستقبل، مثل: '1' اسم البروتين، '2' التاريخ المعد، '3' رقم اللوت/الدفعة، '4' المورد، '5' رقم الفهرس، '6' التركيز، '7' الحجم، '8' معد.
- إعداد العازلة مخفف تحتوي على 20٪ (V / V) الجلسرين، 10 MM EDTA، 200 mM تريس-حمض الهيدروكلوريك، درجة الحموضة 7.2، وتعقيم فلتر. الاحتفاظ بهذا المخزن المؤقت معقم ًا وتخزينه في RT.
- إعداد العازلة تلطيخ تحتوي على 2٪ (ث / الخامس)BSA، 1 M M MgCl 2، و 0.02٪ NaN3 في محلول ملحي الفوسفات المخزنة (PBS). تصفية وتخزين في 4 درجة مئوية.
2. إعداد لوحة مصدر ECM
- إزالة المخزونات من البروتينات ECM لطباعتها وذوبانها على الجليد. تسجيل جميع أرقام الكثير لتتبع البيانات الوصفية.
- نفض الغبار أنابيب من البروتينات المذابة بلطف لضمان إعادة تعليق السليم وتدور إلى أسفل في جهاز طرد مركزي.
-
جعل خليط الطباعة ECM (EPMs) وfiducial الفلورسنت لاستخدامها من قبل الروبوت التعامل مع السائل من شأنها أن تخلق لوحات مصدر 384 جيدا معشاة.
ملاحظة: سيتم استخدام لوحات المصدر 384-well بواسطة طابعة صفيف دبوس تعمل باللمس لإنشاء صفائف مطبوعة في 8 لوحات جيدا.- تسمية أنابيب الطرد المركزي الدقيقة 1.5 مل لكل EPM وfiducial.
- إعداد كل EPM عن طريق الجمع بين 125 ميكرولتر من المخزن المؤقت مخفف (انظر الخطوة 1.4) مع الحجم المناسب من مخزون ECM وجعل الخليط يصل إلى حجم إجمالي 250 درجة مئوية مع PBS. التركيزات النهائية في كل أنبوب EPM سيكون 1X بروتين ECM, 5 MM EDTA, 10% الجلسرين, و 100 mM Tris.
- إعداد fiducial الفلورسنت عن طريق حلها في المخزن المؤقت المناسب المحدد من قبل الشركة المصنعة ونقل 250 € L إلى أنبوب fiducial المسمى.
3. إنشاء لوحة المصدر باستخدام معالج سائل
- تصميم تخطيط لوحة 384 جيدا أن العشوائية مواقف ECMs والأمثل لمحور دبوس طابعة صفيف المستخدمة. تصميم موضع fiducial بحيث سيتم طباعتها في الصف 1، العمود 1 موقف كل بئر للمساعدة في اتجاه الصفيف.
ملاحظة: يتم استخدام ما مجموعه 14-15 نسخة متماثلة من كل إدارة المحتوى في المؤسسة لضمان بيانات قوية. وتشمل تكرار إضافية من الكولاجين أو آخر ECM التي تسفر عن مرفق قوي لتقييم توحيد الربط. قد يحتاج التخطيط إلى استخدام لوحات متعددة 384-جيدا اعتمادا على عدد ECMs ذات الفائدة. - نقل أنابيب EPM إلى معالج السائل، والحفاظ على أنابيب في 4 درجة مئوية إما مع رف أنبوب تبريد أو باستخدام الروبوت التعامل مع السائل الموجود في غرفة باردة.
- باستخدام برنامج المعالج السائل، قم بتشغيل برنامج لنقل 15 ميكرولتر من كل EPM والفيدوسيال إلى الآبار المحددة مسبقاً داخل لوحة (لوحات) مصدر 384 بئر.
- Pipet PBS في أي آبار غير المستخدمة لزيادة الرطوبة والحذر من الجفاف أثناء عملية الطباعة.
ملاحظة: انظر الشكل 2 للحصول على مثال على مجموعة لوحة مصدر 384 جيدا التي تم تحسينها ل4 × 7 دبوس الرأس ويشمل الكولاجين أنا كتلة وPBS. - ختم لوحة (ق) والحفاظ على 4 درجة مئوية حتى تكون جاهزة للطباعة.
4. طباعة MEMAs باستخدام مجموعة الطباعة الروبوت
ملاحظة: ويصف الجزء التالي من البروتوكول على وجه التحديد إعداد واستخدام وزارة البيئة والبيئة للتحقيق في تأثير مختلف بروتينات البيئة الدقيقة على نمو وانتشار خلايا MCF7. ومع ذلك، يمكن بسهولة تكييف البروتوكول لاستخدام الأربطة المختلفة، ECMs، والخلايا لدراسة خطوط الخلايا الأخرى ونقاط النهاية ذات الأهمية.
-
باستخدام طابعة دبوس تعمل باللمس، طباعة EPMs والبقع fiducial في 8 لوحات جيدا. طباعة نسخ متماثلة متعددة من كل شرط ECM لضمان التكرار.
ملاحظة: يمكن استخدام تنسيقات لوحة أخرى أو الشرائح للطباعة ولكن قد تكون هناك حاجة إلى تحسين المخزن المؤقت لتحقيق تكوين بقعة الأمثل.- اطبع ECMs لـ MEMA باستخدام دبابيس قطرها 350 ميكرومتر مرتبة في تكوين رأس الطباعة 4 × 7. اطبع الصفائف في لوحات 8-well كـ 20 عمودًا بـ 35 صفًا، لما مجموعه 700 نقطة تقريبًا. صفائف أكبر ممكنة في هذه اللوحات ولكن تأتي مع مقايضة من زيادة آثار الحافة في كل من ربط الخلية وتلطيخ.
- بعد الطباعة، قم بتخزين لوحات في المجفف لمدة لا تقل عن 3 أيام قبل الاستخدام.
5. إنشاء لوحات العلاج Ligand
- تصميم تخطيط لوحة 96 جيدا بما في ذلك ligands من الفائدة. لتسهيل علاج العديد من لوحات MEMA في وقت واحد، وتصميم هذه اللوحة مع تباعد التي تسمح لاستخدام الأنابيب متعددة القنوات مع 4 نصائح متباعدة لنقل السوائل بين آبار MEMAs 8-جيدا ولوحة 96 جيدا.
ملاحظة: وفي هذا البروتوكول، تُستخدم المجموعة الكاملة من الأربطة المدرجة في الجدول 2. - ذوبان الأربطة على الجليد. نفض الغبار لفترة وجيزة وتدور أسفل كل أنبوب.
- تخفيف الأربطة إلى مخزون عمل 200x باستخدام المخزن المؤقت الموصى بها من قبل الشركة المصنعة (عادة PBS).
- Pipet 10 درجة مئوية من كل 200x ligand الأسهم في البئر المقابلة داخل لوحة 96 جيدا.
- ختم وتخزين لوحات في -20 درجة مئوية.
ملاحظة: جعل لوحات العلاج ligand على دفعات، والتقاط جميع البيانات الوصفية لتحليل المصب.
6. زراعة الخلايا على MEMAs
- كتلة MEMAs لمدة 20 دقيقة مع 2 مل لكل بئر من العازلة حظر غيرقاذورات تحتوي على 1٪ عامل حظر غير قاذورات (جدول المواد) في الماء المقطر المزدوج (ddH2O).
- يستنشق حجب العازلة وآبار شطف الثلاثي مع PBS. لمنع الجفاف، وترك الحجم النهائي من PBS في الآبار حتى جاهزة لطلاء الخلايا.
ملاحظة: من المفيد للغاية أن يكون هناك عاملان في مقاعد البدلاء لخطوات ثقافة الخلايا على الوزارة البحرية والزراعة. يمكن لأحد العاملين في مقاعد البدلاء تنفيذ خطوات الطموح، بينما يقوم الثاني بتنفيذ خطوات إضافية. فمن المستحسن استخدام الأنابيب متعددة القنوات 1 مل مع نصائح متباعدة لتتناسب مع لوحة 8-جيدا لpipetting وY-الخائن مع اثنين من ماصات باستور لستنشق آبار متعددة في وقت واحد. - البذور 2 × 105 خلايا MCF7 لكل بئر في 2 مل من دولبكو تعديل النسر المتوسط (DMEM) المتوسطة التي تحتوي على 10٪ مصل البقر الجنيني (FBS).
ملاحظة: قبل إجراء تجربة كاملة لـ MEMA، قم بإجراء تجربة معايرة الخلايا لتحسين أرقام الخلايا بحيث تحتوي بقع MEMA على أرقام خلايا عالية (ولكنها لا تتلاءم) في نهاية المدة التجريبية المطلوبة. - بعد 2-18 ساعة من التصاق، يستنشق المتوسطة واستبدالها ب2 مل من متوسط النمو المنخفض (DMEM مع 0.1٪ FBS).
ملاحظة: يمكن استخدام المصل المنخفض (على سبيل المثال، 0.1٪ FBS) أو ظروف النمو المنضب ة عامل في هذا الوقت لعزل التأثير التحفيزي للأربطة محددة. - إذابة لوحة علاج الرباط على الجليد. لوحة مذوبة بالطرد المركزي في 200 × غرام لمدة دقيقة واحدة.
- نقل 200 درجة مئوية من المتوسطة من كل بئر في لوحة الثقافة إلى البئر المناسب في لوحة العلاج. Pipet صعودا وهبوطا لخلط حجم ligand مع المتوسطة ونقل هذا الخليط مرة أخرى إلى البئر المناسبة في لوحة MEMA.
- صخرة طفيفة باليد وإعادة لوحات MEMA إلى الحاضنة. الثقافة لمدة التجربة في وجود مزيج ligand / ECM في 37 درجة مئوية و 5٪ CO2.
ملاحظة: تجربة MEMA نموذجية تعمل لمدة 72 ساعة; قد تتطلب التجارب لمدة أطول استبدال المتوسطة وإعادة المعالجة مع ligand. - نبض الآبار MEMA في 71 ساعة مع 100X 5-ethynyl-2'-deoxyuridine (EdU) لتركيز نهائي من 10 μM. الحضانة في الظروفالتجريبية مع EdU لمدة 1 ساعة في 37 درجة مئوية و 5٪ CO 2.
ملاحظة: ويمكن أيضا استخدام علاجات الخلايا الحية الأخرى في هذا الوقت.
7. تحديد وتلطيخ المثليون أو الشرق الأوسط والأوسط
- بعد 72 ساعة وأي علاجات الخلايا الحية، يستنشق الآبار. إصلاح MEMAs في 2 مل لكل بئر من 2٪ بارافورماليدهايد (PFA) لمدة 15 دقيقة في RT.
- إستنشق PFA. نفاذية مع 2 مل لكل بئر من 0.1٪ السطحي غير الأيونية لمدة 15 دقيقة.
- يستنشق السطحي غير الأيونية وغسل مع 2 مل لكل بئر من PBS. إستنشق الـ(بي بي إس) غسل مع 2 مل من PBS مع 0.05٪ بوليسوربات 20 (PBS-T).
ملاحظة: سطح MEMA هو الكاره للماء، وعدم غسل مع PBS-T قبل وصمة عار وحضانة الأجسام المضادة سيؤدي إلى تشكيل الفراغات في الآبار خلال خطوات الحضانة وتؤدي إلى تلطيخ القطع الأثرية. - إستنشق PBS-T. إضافة الكواشف رد فعل الكشف EdU. حضانة لمدة 1 ساعة في RT، هزاز ومحمية من الضوء. بعد 1 ساعة الحضانة، وتبريد رد فعل مع المخزن المؤقت التبريد التجارية المقدمة.
ملاحظة: يمكن تنفيذ EdU الكشف وتلطيخ / خطوات الأجسام المضادة في 1.5 مل لكل بئر للحد من التكلفة. - قم بإستقان العازلة للتبريد واغسلها بـ PBS-T قبل الحضانة بالبقع أو الأجسام المضادة.
- احتضان آبار MEMA مع الأجسام المضادة ضد الهيستون H3K9me3 (1:1,000) والفيبريلرين (1:400) في وصمة عار العازلة التي تحتوي على 2٪ (ث / الخامس) ألبومالمصل البقري (BSA)، 1 MM MgCl2 و 0.02٪ NaN3 بين عشية وضحاها في 4 درجة مئوية.
ملاحظة: إجراء التحيّات الأجسام المضادة لتحديد التركيزات المثلى قبل استخدامها على مجموعة MEMA كاملة. - بعد الحمل الأساسي أو وصمة عار، وغسل الآبار 2X مع PBS ومرة واحدة مع PBS-T.
- إضافة الأجسام المضادة الثانوية (حمار المضادة للماوس IgG والحمار المضادة للأرنب IgG، على حد سواء 1:300) و 0.5 ميكروغرام / مل 4′ 6-diamidino-2-phenylindole (DAPI). حضانة لمدة 1 ساعة في RT في الظلام.
- غسل الآبار 2X مع 2 مل لكل بئر من PBS، وتركها في النهائي 2 مل PBS.
- انتقل إلى التصوير أو تخزين MEMAs الملون للتصوير في وقت لاحق في PBS في 4 درجة مئوية محمية من الضوء.
8. تصوير التقييمات المتوسطة والصغيرة والمتوسطة
- صورة MEMA على نظام التصوير الآلي مع قنوات الكشف الفلورسنت المناسبة.
- إخراج بيانات الصورة الناتجة إلى نظام إدارة الصور. تقسيم الخلايا وحساب مستويات الكثافة باستخدام CellProfiler8.
9- تحليل البيانات
ملاحظة: يتكون تحليل البيانات من التطبيع، وتصحيح التباين، وتلخيص البيانات المشتقة من CellProfiler الخام. في هذه الحالة، يتم استخدام بيئة R مع تعليمات برمجية مخصصة لتنفيذ كافة الخطوات. ومع ذلك، يمكن استخدام أي بيئة إحصائية أو برنامج لتنفيذ الإجراءات المكافئة. يتوفر مثال على التعليمات البرمجية المخصصة مفتوحة المصدر لبيئة R للتحليل في: https://www.synapse.org/#!Synapse:syn2862345/wiki/72486.
- قبل المعالجة وتطبيع بيانات الصورة المجزأة.
- تحديد عدد الخلايا الموضعية باستخدام النوى الملطخة DAPI.
- لصناعة السيارات في بوابة كثافة EdU لتسمية الخلايا كما EdU+. قياس الانتشار باستخدام نسبة EdU+ الخلايا في كل بقعة.
- متوسط تلخيص البقع السيتوبلازمية وقياسات مورفولوجيا النووية على المستوى الفوري.
- إجراء إزالة التباين غير المرغوب فيه (RUV) التطبيع على البيانات لتحسين جودة البيانات9.
ملاحظة: يتم تطبيق هذا النهج على كل إشارة كثافة ومورفولوجيا بشكل مستقل كمصفوفة مع صفائف باستخدام الصفوف والبقع كما الأعمدة كما هو موضح سابقا9. - تطبيق التسوية loess ثنائي المتغيرات إلى المخلفات تطبيع RUV باستخدام صف الصفيف وعمود الصفيف كمتغيرات مستقلة لتصحيح للتأثيرات المكانية أو كثافة ذات الصلة.
- وبمجرد الانتهاء من التطبيع، يلخص الوسيط التكرار لكل حالة من شروط البيئة الدقيقة للإبلاغ والمزيد من التحليل.
النتائج
ولتبسيط الآثار البيئية الدقيقة على نمو الخلايا وانتشارها وتحديد الظروف التي تعزز أو تمنع نمو الخلايا وانتشارها، تم زرع خط خلايا سرطان الثدي MCF7 على مجموعة من ثمانية من الاتفاقيات البيئية المتوسطة والمتوسطة 8 الآبار على النحو المبين في البروتوكول. وقد عرّض هذا التقييم الخلايا إلى 48 جهازاً مختلفاً من الـ ECMs و57 ربطاً مختلفاً، لما مجموعه 2736 حالة بيئية مؤتلفة. بعد 71 ساعة في الثقافة، تم نبض الخلايا مع EdU، ثابتة، نفاذيبليد، وملطخة DAPI، رد فعل للكشف عن EdU، والأجسام المضادة للفيبريلين، والأجسام المضادة للH3K9me3. تم تصوير الخلايا على مجهر محتوى عالي. تم تحميل الصور إلى خادم Omero10، مقسمة باستخدام CellProfiler8، وتطبيعها وتحليلها في R9. وتركز النتائج الموضحة أدناه على إشارات DAPI وEdU.
منصة تحليل الصور من MEMAs تسفر عن بعض النتائج مماثلة لتلك المتاحة من طرق قياس التدفق، مثل قطع محتوى الحمض النووي تظهر 2N و 4N الكسور للخلايا المعالجة مع الرباط معين (الشكل3A)،استنادا ً إلى DAPI كثافة والمنطقة. وتوفر هذه المؤامرات أدلة على الظروف التي تعزز ركوب الدراجات النشطة في الخلايا مقابل ما يتبين من القمم المزدوجة الواضحة المقابلة للخلايا في مراحل G1 أو G2 مقابل الخلايا الموقوفة للنمو، مما يظهر تغيرات في القمم مقارنة بظروف التحكم. نحن نستخدم رقم الخلية وتلطيخ البيانات كثافة لتلخيص البيانات، حيث تأثير البيئة الدقيقة (ligands على محور واحد، ECM على المحور الثاني) على كل من رقم الخلية (الشكل3B) وإدو التأسيس (الشكل3C) يمكن أن ينظر إليها بسهولة أكبر كتغيرات في لون الخريطة الحرارية وكثافتها. كما رأينا من هذه المؤامرات، وكثير من الآثار هي ligand يحركها، كما أن حالة ECM لم تؤثر بقوة على رقم الخلية أو الإيجابية EdU. Nidogen-1 هو استثناء واضح، كما أن وجود هذا الجزيء ECM يمنع ربط الخلايا ونمو MCF7. الأربطة مثل FGF6 و NRG1α (NRG1.1 على المؤامرات) تعزيز عدد الخلايا ولها معدلات عالية من التأسيس EDU، في حين أن الأربطة مثل AREG و NRG1-smdf (NRG1.10 على المؤامرات) تمنع ربط الخلايا و / أو نمو الخلايا. وتدعم هذه النتائج من قبل صور الخلايا المتنامية على البقع، حيث الفرق الواضح في عدد الخلايا والإيجابية EdU واضح (انظر المثال في الشكل 3D).
وبما أن منصة MEMA هي تقنية أحدث، فقد تم التحقق من صحة النتائج في تجارب منفصلة. تم زرع خلايا MCF7 في لوحات 24-جيدا المغلفة بالكولاجين الأول في وسط DMEM مع FBS 10٪. بعد 18 ساعة، تم تبادل وسائل الإعلام لمتوسط النمو المنخفض (DMEM مع 0.1٪ FBS) وعولجت الخلايا مع NRG1α، FGF6، أو AREG ومثقف لمدة 72 ساعة. كانت الخلايا ملطخة DAPI ولتأسيس EdU، وصورة، مجزأة، وتحليلها. على غرار النتائج التي تم الحصول عليها من منصة MEMA، FGF6 و NRG1α على حد سواء أدى إلى ارتفاع أعداد الخلايا (الشكل4A) ومعدلات تأسيس EDU (الشكل4B) مقارنة مع الخلايا المعالجة AREG، والتحقق من ملاحظاتنا في تجارب MEMA الأصلية.

الشكل 1 مخطط انسيابي يوضح سير العمل والجدول الزمني للمراحل المختلفة لتجربة MEMA النموذجية. بمجرد طباعة الـ MEMAs، يمكن تخزينها في درجة حرارة الغرفة المجففة لعدة أشهر قبل استخدامها. عادة، تستمر المرحلة التجريبية 3-4 أيام، ولكن تم زراعة بعض الخلايا الأولية البطيئة النمو على الآليات البيئية والمتوسطة لمدة تصل إلى أسبوعين. الرجاء النقر هنا لعرض نسخة أكبر من هذا الرقم.

الشكل 2 تخطيط لوحة مصدر ECM لطباعة الصفيف. تتم طباعة كتلة الكولاجين على MEMA كشبكة، والتي توفر مجموعة متكررة للغاية من الشروط التي تسمح لتطبيع أكثر قوة بين الآبار. توفر الآبار المملوءة بـ PBS الرطوبة للمساعدة في منع التبخر أثناء عملية الطباعة. الرجاء النقر هنا لعرض نسخة أكبر من هذا الرقم.

الشكل 3 أمثلة على البيانات التي تم إنشاؤها من تجربة MEMA نموذجية. (أ) ملامح دورة الخلية من قيم كثافة DAPI معلب مقابل عدد الخلايا من لوحة واحدة 8 جيدا تعامل مع الأربطة المختلفة، مما يدل على كثافة DAPI biphasic تلطيخ تشير إلى الخلايا في G1 مقابل G2 مرحلة دورة الخلية. (B) خريطة الحرارة التي تعرض عدد الخلايا الموضعية التي تم تجميعها بواسطة التشابه باستخدام التجميع الهرمي. يشير اللون الأحمر إلى رقم خلية أعلى، والأزرق هو رقم الخلية السفلي. الأربطة على محور س، ECMs على محور ص. (C) خريطة الحرارة تظهر التأسيس EdU تطبيع، مع الأحمر يشير إلى أعلى والأزرق مما يدل على انخفاض إدو التأسيس. الأربطة على محور س، ECMs على محور ص. (د) مثال على خلايا MCF7 التي تنمو على بقعة MEMA تعامل مع NRG1-α تظهر معدلات عالية من التأسيس EdU (النوى الوردية). وصمة عار خضراء هو قناع الخلية والأزرق هو DAPI. الرجاء النقر هنا لعرض نسخة أكبر من هذا الرقم.
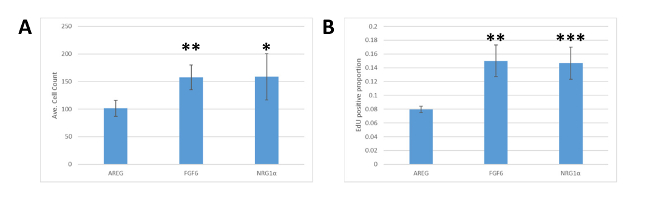
الشكل 4 نتائج التحقق من صحة MEMA في ثقافة الخلية. (أ) التحديد الكمي لعدد الخلايا الناتج عن علاج MCF7 مع أربطة مختلفة. تم طلاء أعداد مكافئة من خلايا MCF7 في لوحات متعددة الجدران ثم تعامل مع AREG أو FGF6 أو NRG1α. الآبار التي عولجت مع AREG كان خلايا أقل بكثير من تلك التي عولجت مع FGF6 (** تشير إلى قيم الطالب t-test p أقل من 0.01) أو NRG1α (* يشير إلى قيمة p من 0.05) في 72 ح بعد العلاج ligand. (ب) التحديد الكمي لمستوى تأسيس EDU في MCF7 بسبب العلاج مع الأربطة المختلفة، كما هو الحال في اللوحة A. ينتج عن علاج AREG نسبة أقل بكثير من الخلايا التي تحتوي على EdU من الخلايا المعالجة بFGF6 (**، p < 0.01) أو NRG1α (***، p = 0.01). تمثل أشرطة الخطأ الانحراف المعياري. الرجاء النقر هنا لعرض نسخة أكبر من هذا الرقم.
| اسم البروتين | معرف يونيبروت | الاسهم تركيز (ميكروغرام/ مل) | النهائي تركيز (ميكروغرام/ مل) |
| ANGPT1|1 | Q15389|1 | 100 | 0.04 |
| ANGPT2|1 | O15123|1 | 100 | 0.2 |
| فى الانمازى | P15514 | 100 | 0.02 |
| BMP2 | P12643 | 100 | 0.1 |
| BMP3 | P12645 | ألف شخص | 0.1 |
| BMP4 | P12644 | 100 | 0.1 |
| BMP5|1 | P22003|1 | 100 | 0.1 |
| BMP6 | P22004 | 100 | 0.1 |
| BMP7 | P18075 | 100 | 0.1 |
| CSF2 | P04141 | 100 | 0.02 |
| CTGF|1 | P29279|1 | 100 | 0.05 |
| CXCL12| الفا | P48061|2 | 100 | 0.01 |
| CXCL12| الإصدار بيتا | P48061|1 | 100 | 0.03 |
| CXCL1 | P09341 | 100 | 0.004 |
| CXCL8|1 | P10145|1 | 100 | 0.3 |
| DLL1|1 | O00548|1 | 500 | 0.5 0.5 |
| DLL4 | Q9NR61 | 200 | 0.6 |
| EGF|1 | P01133|1 | 500 | 0.01 |
| فاسالج|1 | P48023|1 | 10 سنوات | 0.02 |
| FGF2|3 | P09038|2 | 100 | 0.01 |
| FGF6 | P10767 | 100 | 0.01 |
| FLT3LG|1 | P49771|1 | 50 | 0.001 |
| GPNMB|1 | Q14956|1 | 100 | 0.5 0.5 |
| HGF|1 | P14210|1 | 50 | 0.04 |
| منتدى إدارة الإنترنت1|1 | P05019|1 | 200 | 0.01 |
| منتدى إدارة الإنترنتBP2 | P18065 | 100 | 0.05 |
| منتدى إدارة الإنترنتGFBP3|1 | P17936|1 | 100 | 0.1 |
| IL13 | P35225 | 100 | 0.01 |
| IL15 | IL15S48AA | P40933|1 | 50 | 0.01 |
| IL1B | P01584 | 25 | 0.001 |
| IL6 | P05231 | 100 | 0.01 |
| IL7|1 | P13232|1 | 100 | 0.01 |
| JAG1|1 | P78504|1 | 200 | 0.5 0.5 |
| JAG2| طويله | Q9Y219|1 | 100 | 0.5 0.5 |
| كيتإل ج|1 | P21583|1 | 100 | 0.005 |
| KNG1| HMW | P01042|1 | 100 | 0.2 |
| LEP | P41159 | ألف شخص | 0.002 |
| LYVE1 | Q9Y5Y7 | 100 | 0.05 |
| NRG1|10 | Q02297|10 | 100 | 0.01 |
| NRG1|1 | Q02297|1 | 100 | 0.05 |
| NRG1|6 | Q02297|6 | 100 | 0.01 |
| PDGFAB | محمد الدوسري | 100 | 0.05 |
| PDGFB|1 | P01127|1 | 100 | 0.05 |
| Ptn | P21246 | 100 | 0.5 0.5 |
| صه | Q15465 | 100 | 0.5 0.5 |
| TGFB1|| كتيرمينوس | P01137| كتيرمينوس | 20 | 0.01 |
| TGFB1|| اللفه | P01137| اللفه | 100 | 0.15 |
| TGFB2| ألف | P61812|1 | 20 | 0.01 |
| THPO|1 | P40225|1 | 50 | 0.002 |
| في الفرنسيّة | O00300 | 100 | 0.02 |
| TNFSF11|1 | O14788|1 | 100 | 0.01 |
| TNF | P01375 | 100 | 0.01 |
| فيغفا | في GF206 | P15692|1 | 100 | 0.01 |
| WNT10A | Q9GZT5 | 100 | 0.1 |
| WNT3A|1 | P56704|1 | 200 | 0.1 |
| فينت5a|1 | P22725|1 | 100 | 0.1 |
الجدول 1: القائمة الكاملة للأربطة المستخدمة في تجارب وزارة الشؤون الدولية. يتم توفير معرف uniprot، تركيزات المخزون، وتركيزات العمل النهائية.
| بروتين ECM | يونيبروتيد | تركيز المخزون (ميكروغرام/ مل) | النهائي تركيز (ميكروغرام/ مل) | تلاحظ |
| الكاميرا|1 | Q13740|1 | 100 | 30 | |
| CDH20 | Q9HBT6 | 300 | 80 | |
| CDH6|1 | P55285|1 | 100 | 40 | |
| CDH8 | P55286 | 100 | 20 | |
| CD44|1 | P16070|1 | 100 | 30 | |
| CEACAM6 | P40199 | 100 | 30 | |
| COL1A1 | P02453 | 5000 | 200 | وحدات فرعية متعددة مع هويات uniprot متعددة |
| COL2A1|2 | P02458|2 | ألف شخص | 200 | |
| COL3A1|1 | P02461|1 | ألف شخص | 200 | |
| COL4A1|1 | P02462|1 | ألف شخص | 200 | وحدات فرعية متعددة مع هويات uniprot متعددة |
| COL5A1 | P20908 | ألف شخص | 200 | |
| COL23A1|1 | Q86Y22|1 | 200 | 80 | |
| DSG2 | Q14126 | 100 | 30 | |
| CDH1|1 | P12830|1 | 100 | 40 | |
| ECM1|1 | Q16610|1 | 100 | 40 | |
| FN1|1 | P02751|1 | ألف شخص | 200 | |
| GAP43|1 | P17677|1 | 158 | 40 | |
| HyA-500K | ألف شخص | 200 | LOR-0005 | |
| HyA-50K | ألف شخص | 200 | LOR-0007 | |
| ICAM1 | P05362 | 400 | 80 | |
| الكاميرا|1 | Q13740|1 | 100 | 30 | |
| CDH20 | Q9HBT6 | 300 | 80 | |
| CDH8 | P55286 | 100 | 20 | |
| CD44|1 | P16070|1 | 100 | 30 | |
| CEACAM6 | P40199 | 100 | 30 | |
| DSG2 | Q14126 | 100 | 30 | |
| CDH15 | P55291 | 100 | 20 | |
| VCAM1|1 | P19320|1 | ألف شخص | 200 | |
| 1 | P25391 | 500 | 200 | وحدات فرعية متعددة مع هويات uniprot متعددة |
| LAMA3|2 | Q16787|2 | 130 | 40 | |
| لوم | P51884 | 200 | 80 | |
| CDH15 | P55291 | 100 | 20 | |
| NID1|1 | P14543|1 | 100 | 9.3 ميكروغرام/مل نيد، 130 ميكروغرام/مل لام، 46.5 ميكروغرام/مل COL4 | +COL4 واللامينين |
| Omd | Q99983 | 100 | 40 | |
| SPP1| ألف | P10451|1 | 100 | 40 | |
| CDH3|1 | P22223|1 | 100 | 40 | |
| PECAM1| طويله | P16284|1 | 150 | 40 | |
| الشركات عبر الوطنية|1 | P24821|1 | 500 | 200 | |
| VCAM1|1 | P19320|1 | ألف شخص | 200 | |
| Vtn | P04004 | 100 | 40 | |
| Bgn | P21810 | 100 | 40 | |
| DCN| ألف | P07585|1 | 300 | 80 | |
| POSTN|1 | Q15063|1 | 100 | 40 | |
| Sparc | P09486 | 100 | 40 | |
| THBS1|1 | P07996|1 | 100 | 40 | |
| BCAN|1 | Q96GW7|1 | 100 | 40 | |
| جيش التحرير الوطني|3 | P15502|3 | ألف شخص | 200 | |
| FBN1 | P35555 | 254 | 80 |
الجدول 2: القائمة الكاملة لبروتينات والشروط التي تستخدم في تجارب الإدارة البحرية والإدارة. يتم توفير معرف uniprot، تركيزات المخزون، وتركيزات العمل النهائية. في بعض الحالات، تمثل الحالة المطبوعة مركب بروتين أو مزيج من البروتينات المتعددة، والتي يشار إليها في عمود Notes.
Discussion
كانت أهمية "الأبعاد" والسياق عاملاً محفزاً في تطوير أنظمة الثقافة المختبرية كأدوات في توصيف الخلايا السرطانية من خلال تفاعلها مع البيئة الدقيقة11،وقدرة المختبر نظم الثقافة لتقليد البيئة في الجسم الحي هو القوة الدافعة وراء السعي لتحسين تلك النظم الثقافية. في النظم المختبرية، ومع ذلك، لا تزال أدوات هامة من أبحاث السرطان على وجه التحديد بسبب قدرتها على تقطير المجمع في حالة الجسم الحي وصولا الى نموذج مبسط12.
على الرغم من أن أنظمة 2D يمكن أن تشمل ECMs وligands، إلا أنها تفتقر تقليديا إلى قدرات الإنتاجية لاستجواب مجموعة واسعة من pertubagens المؤتلف. مقتطفات غشاء الطابق السفلي التجارية شعبية تسمح للزراعة في 3D، ولكن تفتقر إلى مصدر لوحة محددة بعناية من البروتينات. عادة ما تعاني المقتطفات التجارية من تكوين محدد بشكل غير كامل، والتييمكن أن تشوش التحليل وتؤدي إلى اختلاف كبير من دفعة إلى دفعة 3،13. منصة MEMA يتغلب على هذه الحواجز، مما يسمح لدراسة التعديلات في الأنماط الظاهرية الخلوية، والنشاط الأيضي، وحالة التمايز، والاختلافات في نمو الخلايا وانتشارها كما يتم تعديلها من قبل محلية محددة ومحددة العوامل.
منصة MEMA هو نهج قوي، متوسطة إلى عالية الإنتاجية لتقييم تأثير البيئة الدقيقة (كل من ECM والعوامل القابلة للذوبان) على النمط الظاهري للخلايا. منصة يظهر مرونة كبيرة لأنواع الاختبارات والخلايا التي يمكن استخدامها. يمكننا مراقبة الآثار من كل من الأربطة القابلة للذوبان والبروتينات ECM التي تتعرض الخلايا. في الواقع، وجدنا مؤخرا أن الأربطة كانت المحرك الرئيسي للمقاومة لمثبطات HER2 المستهدفة، ولكن يمكن تعديل هذه الآثار من قبل ECM5. تم بنجاح زراعة مجموعة متنوعة من الخلايا، بما في ذلك الخلايا الأولية وخطوط الخلايا المشتقة من أنواع الخلايا المختلفة بما في ذلك الرئة والمثانة والبروستاتا والثدي والبنكرياس، وكذلك الخلايا الجذعية متعددة القوى المستحثة (iPS)، على منصة MEMA (انظر أمثلة في المراجع5و7و14). استخدام بقع مختلفة يسمح لقراءة نقاط النهاية الخلوية متعددة، بما في ذلك نمو الخلايا، والتمايز، والتمثيل الغذائي. وقد مدد باحثون آخرون منصة لاستجواب تأثير تصلب أو معامل مرن، مضيفا بعدا إضافيا إلى منصة MEMA15. وأخيرا، منصة قابلة لأداء شاشات المخدرات لتحديد ظروف البيئة الدقيقة التي تعزز أو تمنع فعاليةالمخدرات، كما أبلغنا نحن وآخرون مؤخرا 5،14،15 .
ربما الخطوة الأكثر أهمية لنجاح تجربة MEMA هو تحسين كثافة طلاء الخلايا. تحسين كثافة الخلايا يضمن وجود خلايا كافية لتوفير بيانات قوية، ولكن ليس الكثير بحيث تصبح البقعة مُفرطة بشكل مفرط. يمكن للبقع المتجانسة أن تشوش النتائج بشكل كبير، لا سيما إذا تم استخدام الانتشار كنقطة نهاية، مما يجعل من المستحيل تحديد ما إذا كانت معدلات الانتشار المنخفضة نتيجة للتفاعلات مع العوامل البيئية الدقيقة أو بسبب تثبيط الاتصال من عالية الكثافة الخلوية. يمكن أن تكشف تجارب معايرة الخلايا عن هذه المشاكل، حيث أن متوسط أعداد الخلايا لكل بقعة سوف يظهر زيادة خطية مع زيادة أعداد الخلايا المطلية، ولكن في نهاية المطاف سوف هضبة. يجب اختيار رقم الخلية الأمثل في النطاق الخطي للمنحنى.
وكما ذُكر أعلاه، فإن منصة MEMA مرنة ويمكن إعدادها على مجموعة متنوعة من الركائز ذات الأسطح المختلفة. وتشمل هذه الشرائح الزجاجية وصيغ لوحة متعددة الجدران. في تجربتنا، ليست كل الكيمياء السطحية قابلة للطباعة MEMA، كما لاحظنا مفرزة بقعة على بعض الأسطح بسبب خصائص التصاق الفقراء وعدم القدرة على منع التصاق الخلية على أسطح التصاق عالية أخرى. وعلاوة على ذلك، يتطلب التغيير بين ركائز مختلفة تحسين شروط المخزن المؤقت، حيث يمكن أن يختلف أداء الطباعة مع نفس المخزن المؤقت للطباعة استناداً إلى كيمياء السطح.
يلعب قطر بقع ECM المطبوعة دورًا مهمًا في جودة البيانات. بشكل عام، نوصي باستخدام أكبر دبابيس الطباعة قطرها المتاحة للصفر في الاستخدام (نستخدم حاليا دبابيس قطرها 350 م). تسمح بقع قطرأكبر لعدد أكبر من الخلايا باحتلال بقعة، مما يؤدي إلى بيانات أكثر قوة مما يتم إنشاؤه مع دبابيس قطرها أصغر. منذ ربط الخلايا هو عملية العشوائية، وهناك تميل إلى أن تكون درجة عالية من التباين في البيانات التي ترتبط بعدد الخلايا المرفقة أصلا إلى كل بقعة. وبالتالي، نوصي بطباعة عدد كبير من النسخ المتماثللكل حالة ECM. نقوم بطباعة 10-15 ECM تكرار في كل بئر مع ظروف الطباعة الحالية لدينا لضمان إحصاءات قوية.
لقد لاحظنا في تجاربنا الماضية أن معظم هاذا التأثيرات الليكاند تميل إلى الهيمنة على آثار ECM. قد يكون هذا في جزء منه بسبب قرارنا بإضافة الكولاجين الأول إلى جميع بقع ECM، مما يضمن ربط الخلايا قوية. ومع ذلك، نعتقد أن هذا قد أيضا تجانس آثار ECM، كما معظم البقع تميل إلى التصرف بطريقة مشابهة للغاية للكولاجين I. تغيير تكوين بقعة لاستبعاد الكولاجين أنا قد يؤدي إلى سلوك الخلايا التفاضلية نتيجة للتفاعل مع ECM، ولكن أيضا يؤثر بشكل كبير على ربط الخلايا، مما أدى إلى العديد من البقع غير المشغولة. يجب على المستخدمين تكييف تكوين ECM مع الأخذ في الاعتبار هذه الاختلافات، ولا سيما أولئك المهتمين في الخلايا الجذعية والسلف والتمايز، حيث المصفوفة يمكن أن يكون لها تأثير كبير16.
عادة ما نقوم بإجراء الاختبارات MEMA لفترات زمنية قصيرة نسبيا (على سبيل المثال، 72 ساعة كحد أقصى). وذلك لأن الخلايا مقيدة إلى البقع (المخزن المؤقت حظر لا يسمح بالنمو خارج البقع في تجربتنا). مع الخلايا تقسيم بسرعة، والنمو لفترة أطول من 72 ح سوف يؤدي إلى فرط نمو البقعة، مما يعقد بدوره تجزئة الصورة كما تصبح الخلايا مزدحمة وتتراكم على بعضها البعض، ويمكن أيضا أن تؤثر على البيانات كما يمكن أن يحدث اعتقال النمو مع تثبيط الاتصال. لقد قمنا بعلاجات أطول مع خلايا أولية بطيئة النمو (10-14 يومًا)، ولكن يجب توخي الحذر في هذه الاختبارات لتغيير وسائل الإعلام وتجديد الأربطة كل 3-4 أيام.
وتركز الجهود المتواصلة لتطوير منصة MEMA على مجالين من مجالات الاهتمام، هما تعظيم الجودة البصرية للتصوير والتحسين داخل سفن الثقافة الأصغر حجماً. تصبح الجودة البصرية عاملاً حاسماً عندما يحتاج الباحثون إلى فحص مجهري ذي دقة أعلى لتحديد التوطين دون الخلوي لعلامات اهتمامهم. يمكن إجراء الشاشات الأولية بدقة أقل على المجاهر عالية الإنتاجية متبوعة بتصوير بقع محددة ذات أهمية على أدوات ذات دقة أعلى، ولكن يمكن اختراق جودة الصورة إذا كانت الخصائص البصرية للمعيار ضعيفة. ومن شأن تحسين الخصائص البصرية للحصة أن يسمح للباحثين بإجراء الشاشات الأولية على نظم التصوير عالية الاستبانة دون الحاجة إلى استعادة الصور المختارة بدقة أعلى. وأخيراً، فإن القدرة على أداء الأسواق المتوسطة الأجل في السفن الثقافية الأصغر حجماً، مثل لوحات ذات 96 بئراً، من شأنها أن تسمح بتخفيض حجم العلاج وتوسيع الأربطة والكميات المكررة. ويتطلب هذا الانتقال تحسين تفاعلات البروتين العازلة الركيزة وطباعة الصفيف داخل أوعية الثقافة الجديدة. وهذه الجهود الجارية سوف تحسن منصة MEMA وتوسيع قدراتها القوية لتحديد البروتينات البيئية الدقيقة ذات الصلة التي تغير الأنماط الظاهرية الخلوية لمجموعة متنوعة من أنواع الخلايا، والتي يمكن بعد ذلك التحقيق فيها في وقت لاحق في الاختبارات المؤكدة.
Disclosures
وليس لدى أصحاب البلاغ ما يكشفون عنه.
Acknowledgements
وقد تم دعم هذا العمل من قبل مكتبة الصندوق المشترك للشبكات (LINCS) منحة HG008100 (J.W.G., L.M.H., وJ.E.K).
Materials
| Name | Company | Catalog Number | Comments |
| Aushon 2470 | Aushon BioSystems | Arrayer robot system used in the protocol | |
| Nikon HCA | Nikon | High Content Imaging system designed around Nikon Eclipse Ti Inverted Microscope | |
| BioTek Precision XS liquid Handler | BioTek | liquid handling robot used in the protocol | |
| Trizma hydrochloride buffer solution | Sigma | T2069 | |
| EDTA | Invitrogen | 15575-038 | |
| Glycerol | Sigma | G5516 | |
| Triton X100 | Sigma | T9284 | |
| Tween 20 | Sigma | P7949 | |
| Kolliphor P338 | BASF | 50424591 | |
| 384-well microarray plate, cylindrical well | Thermo Fisher | ab1055 | |
| Nunc 8 well dish | Thermo Fisher | 267062 | |
| Paraformaldehyde 16% solution | Electron Microscopy Science | 15710 | |
| BSA | Fisher | BP-1600 | |
| Sodium Azide | Sigma | S2002 | |
| Cell Mask | Molecular Probes | H32713 | |
| Click-iTEdU Alexa Fluor | Molecular Probes | C10357 | |
| DAPI | Promo Kine | PK-CA70740043 | |
| ALCAM | R & D Systems | 656-AL | ECM |
| Cadherin-20 (CDH20) | R & D Systems | 5604-CA | ECM |
| Cadherin-6 (CDH6) | R & D Systems | 2715-CA | ECM |
| Cadherin-8 (CDH8) | R & D Systems | 188-C8 | ECM |
| CD44 | R & D Systems | 3660-CD | ECM |
| CEACAM6 | R & D Systems | 3934-CM | ECM |
| Collagen I | Cultrex | 3442-050-01 | ECM |
| Collagen Type II | Millipore | CC052 | ECM |
| Collagen Type III | Millipore | CC054 | ECM |
| Collagen Type IV | Sigma | C5533 | ECM |
| Collagen Type V | Millipore | CC077 | ECM |
| COL23A1 | R & D Systems | 4165-CL | ECM |
| Desmoglein 2 | R & D Systems | 947-DM | ECM |
| E-cadherin (CDH1) | R & D Systems | 648-EC | ECM |
| ECM1 | R & D Systems | 3937-EC | ECM |
| Fibronectin | R & D Systems | 1918-FN | ECM |
| GAP43 | Abcam | ab114188 | ECM |
| HyA-500K | R & D Systems | GLR002 | ECM |
| HyA-50K | R & D Systems | GLR001 | ECM |
| ICAM-1 | R & D Systems | 720-IC | ECM |
| Laminin | Sigma | L6274 | ECM |
| Laminin-5 | Abcam | ab42326 | ECM |
| Lumican | R & D Systems | 2846-LU | ECM |
| M-Cad (CDH15) | R & D Systems | 4096-MC | ECM |
| Nidogen-1 | R & D Systems | 2570-ND | ECM |
| Osteoadherin/OSAD | R & D Systems | 2884-AD | ECM |
| Osteopontin (SPP) | R & D Systems | 1433-OP | ECM |
| P-Cadherin (CDH3) | R & D Systems | 861-PC | ECM |
| PECAM1 | R & D Systems | ADP6 | ECM |
| Tenascin C | R & D Systems | 3358-TC | ECM |
| VCAM1 | R & D Systems | ADP5 | ECM |
| Vitronectin | R & D Systems | 2308-VN | ECM |
| Biglycan | R & D Systems | 2667-CM | ECM |
| Decorin | R & D Systems | 143-DE | ECM |
| Periostin | R & D Systems | 3548-F2 | ECM |
| SPARC/osteonectin | R & D Systems | 941-SP | ECM |
| Thrombospondin-1/2 | R & D Systems | 3074-TH | ECM |
| Brevican | R & D Systems | 4009-BC | ECM |
| Elastin | BioMatrix | 5052 | ECM |
| Fibrillin | Lynn Sakai Lab OHSU | N/A | ECM |
| ANGPT2 | RnD_Systems_Own | 623-AN-025 | Ligand |
| IL1B | RnD_Systems_Own | 201-LB-005 | Ligand |
| CXCL8 | RnD_Systems_Own | 208-IL-010 | Ligand |
| IGF1 | RnD_Systems_Own | 291-G1-200 | Ligand |
| TNFRSF11B | RnD_Systems_Own | 185-OS | Ligand |
| BMP6 | RnD_Systems_Own | 507-BP-020 | Ligand |
| FLT3LG | RnD_Systems_Own | 308-FK-005 | Ligand |
| CXCL1 | RnD_Systems_Own | 275-GR-010 | Ligand |
| DLL4 | RnD_Systems_Own | 1506-D4-050 | Ligand |
| HGF | RnD_Systems_Own | 294-HGN-005 | Ligand |
| Wnt5a | RnD_Systems_Own | 645-WN-010 | Ligand |
| CTGF | Life_Technologies_Own | PHG0286 | Ligand |
| LEP | RnD_Systems_Own | 398-LP-01M | Ligand |
| FGF2 | Sigma_Aldrich_Own | SRP4037-50UG | Ligand |
| FGF6 | RnD_Systems_Own | 238-F6 | Ligand |
| IL7 | RnD_Systems_Own | 207-IL-005 | Ligand |
| TGFB1 | RnD_Systems_Own | 246-LP-025 | Ligand |
| PDGFB | RnD_Systems_Own | 220-BB-010 | Ligand |
| WNT10A | Genemed_Own | 90009 | Ligand |
| PTN | RnD_Systems_Own | 252-PL-050 | Ligand |
| BMP3 | RnD_Systems_Own | 113-BP-100 | Ligand |
| BMP4 | RnD_Systems_Own | 314-BP-010 | Ligand |
| TNFSF11 | RnD_Systems_Own | 390-TN-010 | Ligand |
| CSF2 | RnD_Systems_Own | 215-GM-010 | Ligand |
| BMP5 | RnD_Systems_Own | 615-BMC-020 | Ligand |
| DLL1 | RnD_Systems_Own | 1818-DL-050 | Ligand |
| NRG1 | RnD_Systems_Own | 296-HR-050 | Ligand |
| KNG1 | RnD_Systems_Own | 1569-PI-010 | Ligand |
| GPNMB | RnD_Systems_Own | 2550-AC-050 | Ligand |
| CXCL12 | RnD_Systems_Own | 350-NS-010 | Ligand |
| IL15 | RnD_Systems_Own | 247-ILB-005 | Ligand |
| TNF | RnD_Systems_Own | 210-TA-020 | Ligand |
| IGFBP3 | RnD_Systems_Own | 675-B3-025 | Ligand |
| WNT3A | RnD_Systems_Own | 5036-WNP-010 | Ligand |
| PDGFAB | RnD_Systems_Own | 222-AB | Ligand |
| AREG | RnD_Systems_Own | 262-AR-100 | Ligand |
| JAG1 | RnD_Systems_Own | 1277-JG-050 | Ligand |
| BMP7 | RnD_Systems_Own | 354-BP-010 | Ligand |
| TGFB2 | RnD_Systems_Own | 302-B2-010 | Ligand |
| VEGFA | RnD_Systems_Own | 293-VE-010 | Ligand |
| IL6 | RnD_Systems_Own | 206-IL-010 | Ligand |
| CXCL12 | RnD_Systems_Own | 351-FS-010 | Ligand |
| NRG1 | RnD_Systems_Own | 378-SM | Ligand |
| IGFBP2 | RnD_Systems_Own | 674-B2-025 | Ligand |
| SHH | RnD_Systems_Own | 1314-SH-025 | Ligand |
| FASLG | RnD_Systems_Own | 126-FL-010 | Ligand |
References
- Hanahan, D., Coussens, L. M. Accessories to the crime: functions of cells recruited to the tumor microenvironment. Cancer Cell. 21 (3), 309-322 (2012).
- Quail, D. F., Joyce, J. A. Microenvironmental regulation of tumor progression and metastasis. Nature Medicine. 19 (11), 1423-1437 (2013).
- Hughes, C. S., Postovit, L. M., Lajoie, G. A. Matrigel: a complex protein mixture required for optimal growth of cell culture. Proteomics. 10 (9), 1886-1890 (2010).
- LaBarge, M. A., et al. Human mammary progenitor cell fate decisions are products of interactions with combinatorial microenvironments. Integrative Biology (Cambridge). 1 (1), 70-79 (2009).
- Watson, S. S., et al. Microenvironment-Mediated Mechanisms of Resistance to HER2 Inhibitors Differ between HER2+ Breast Cancer Subtypes. Cell Systems. 6 (3), 329-342 (2018).
- Ranga, A., et al. 3D niche microarrays for systems-level analyses of cell fate. Nature Communications. 5, 4324 (2014).
- Malta, D. F. B., et al. Extracellular matrix microarrays to study inductive signaling for endoderm specification. Acta Biomater. 34, 30-40 (2016).
- Kamentsky, L., et al. Improved structure, function and compatibility for CellProfiler: modular high-throughput image analysis software. Bioinformatics. 27 (8), 1179-1180 (2011).
- Gagnon-Bartsch, J. A., Jacob, L., Speed, T. P. Removing Unwanted Variation from High Dimensional Data with Negative Controls. University of California, Berkeley, Department of Statistics, University of California, Berkeley. , (2013).
- Allan, C., et al. OMERO: flexible, model-driven data management for experimental biology. Nature Methods. 9 (3), 245-253 (2012).
- Simian, M., Bissell, M. J. Organoids: A historical perspective of thinking in three dimensions. Journal of Cell Biology. 216 (1), 31-40 (2017).
- Bissell, M. J. The differentiated state of normal and malignant cells or how to define a "normal" cell in culture. International Review of Cytology. 70, 27-100 (1981).
- Serban, M. A., Prestwich, G. D. Modular extracellular matrices: solutions for the puzzle. Methods. 45 (1), 93-98 (2008).
- Kaylan, K. B., et al. Mapping lung tumor cell drug responses as a function of matrix context and genotype using cell microarrays. Integrative Biology (Cambridge). 8 (12), 1221-1231 (2016).
- Lin, C. H., Jokela, T., Gray, J., LaBarge, M. A. Combinatorial Microenvironments Impose a Continuum of Cellular Responses to a Single Pathway-Targeted Anti-cancer Compound. Cell Reports. 21 (2), 533-545 (2017).
- Gjorevski, N., et al. Designer matrices for intestinal stem cell and organoid culture. Nature. 539 (7630), 560-564 (2016).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved