Method Article
Биолюминесцентный резонансный перенос энергии (BRET) для измерения взаимодействий CRAF с белками 14-3-3 в живых клетках
В этой статье
Резюме
Этот протокол описывает анализ на основе БРЭТ для измерения взаимодействий киназы CRAF с белками 14-3-3 в живых клетках. В протоколе изложены шаги по подготовке ячеек, считыванию излучений BRET и анализу данных. Также представлен пример результата с идентификацией соответствующих элементов управления и устранением неполадок для оптимизации анализа.
Аннотация
CRAF является первичным эффектором ГТФаз RAS и играет решающую роль в онкогенезе нескольких видов рака, вызванных KRAS. Кроме того, CRAF является горячей точкой для мутаций зародышевой линии, которые, как показано, вызывают развитие RASopathy, синдром Нунан. Все RAF-киназы содержат множественные фосфорилирование-зависимые сайты связывания для 14-3-3 регуляторных белков. Дифференциальное связывание 14-3-3 с этими сайтами играет важную роль в образовании активных димеров RAF на плазматической мембране в условиях передачи сигналов и в поддержании аутоингибирования RAF в условиях покоя. Понимание того, как регулируются эти взаимодействия и как они могут быть модулированы, имеет решающее значение для определения новых терапевтических подходов, нацеленных на функцию RAF. В этой статье я опишу анализ на основе биолюминесцентного резонансного переноса энергии (BRET) для измерения взаимодействий CRAF с белками 14-3-3 в живых клетках. В частности, этот анализ измеряет взаимодействия CRAF, слитых с донором нанолюциферазы, и 14-3-3, слитых с акцептором метки Halo, где взаимодействие RAF и 14-3-3 приводит к передаче энергии от донора к акцептору и генерации сигнала BRET. Протокол также показывает, что этот сигнал может быть нарушен мутациями, которые, как было показано, предотвращают связывание 14-3-3 с каждым из его высокоаффинных стыковочных сайтов RAF. В этом протоколе описаны процедуры посева, трансфекции и повторного посева клеток, а также подробные инструкции по считыванию эмиссии BRET, выполнению анализа данных и подтверждению уровней экспрессии белка. Кроме того, приводятся примеры результатов анализа, а также шаги по оптимизации и устранению неполадок.
Введение
RAF-киназы (ARAF, BRAF и CRAF) являются прямыми эффекторами ГТФаз RAS и инициирующими членами каскада киназ RAF-MEK-ERK, способствующего пролиферации/выживанию. Недавние исследования показали, что экспрессия CRAF играет ключевую роль в онкогенезе нескольких видов рака, вызванных KRAS, включая немелкоклеточный рак легкого и протоковую аденокарциному поджелудочной железы 1,2,3,4,5. Кроме того, мутации зародышевой линии CRAF вызывают особенно тяжелую форму РАСопатии – синдромНунан 6,7. Понимание регуляции CRAF имеет решающее значение для разработки успешных терапевтических подходов, направленных на его функцию в клетках.
Все RAF-киназы можно разделить на два функциональных домена: С-концевой каталитический (CAT) домен и N-концевой регуляторный домен (REG), который контролирует его активность (рис. 1A). Домен REG включает в себя RAS связывающий домен (RBD), цистеин-богатый домен (CRD) и серин/треонин-богатую область (S/T-rich). Примечательно, что S/T-богатая область содержит N'-сайт, который связывается с 14-3-3 фосфорилированием-зависимым образом (S259 в CRAF; Рисунок 1А) 8. Домен CAT включает в себя домен киназы вместе со вторым высокоаффинным сайтом стыковки 14-3-3, называемым сайтом C' (S621 в CRAF; Рисунок 1А) 8. Дифференциальное связывание димерных белков 14-3-3 с сайтами N' и C', наряду с CRD, играет решающую роль как в активации, так и в ингибировании RAF 9,10,11,12,13. В нормальных сигнальных условиях активация RAF инициируется его привлечением к плазматической мембране RAS, что позволяет ему образовывать активные димеры, из которых гетеродимер BRAF-CRAF является преобладающей активной формой 14,15. Биохимические анализы с использованием BRAF и CRAF, наряду с криогенной электронной микроскопией (Cryo-EM) структур димерных BRAF, показывают, что димер 14-3-3 стабилизирует активные димеры RAF, связываясь одновременно с C'-сайтом обоих протомеров RAF (рис. 1B)9,13,16,17. И наоборот, исследования показали, что в условиях покоя RAF принимает цитозольное, аутоингибированное подтверждение, когда домен REG связывается с доменом CAT и ингибирует его активность 12,18,19,20. Это закрытое состояние стабилизируется димером 14-3-3, связанным с CRD и N' сайтом в домене REG и с сайтом C' в домене CAT (рис. 1B)10,13,21. В BRAF эта модель подтверждается недавними крио-ЭМ структурами аутоингибированных мономеров BRAF и нашими предыдущими биохимическими исследованиями 10,12,13,21,22. Однако, в то время как показано, что 14-3-3 играет ингибирующую роль в регуляции23 CRAF, аутоингибированное состояние, подобное BRAF, может играть меньшую роль в регуляции12 CRAF; поэтому необходимы дальнейшие исследования для уточнения механизмов, с помощью которых белки 14-3-3 регулируют активность CRAF. 14-3-3-опосредованная регуляция RAF-киназ требует множества событий фосфорилирования и дефосфорилирования RAF, связывания с различными регуляторными белками и взаимодействия с плазматической мембраной. Поэтому крайне важно, чтобы взаимодействия 14-3-3-RAF измерялись в физиологически значимых условиях и в присутствии интактного липидного бислоя.
Для решения этой проблемы была использована технология NanoBRET (далее именуемая N-BRET; подробную информацию о наборе см. в Таблице материалов) для разработки анализа на основе близости для измерения взаимодействий CRAF с белками 14-3-3 в живых клетках (рис. 1C). Эта система на основе БРЭТ измеряет взаимодействия двух представляющих интерес белков (POI), где один белок помечен донором нанолюциферазы (Nano), а другой — меткой Halo, для мечения лигандом22,24 акцептора энергии Halo618. Взаимодействие представляющих интерес белков приводит к передаче энергии от донора к акцептору, который, в свою очередь, генерирует сигнал BRET (рис. 1C). Чрезвычайно яркий донорский белок Nano (излучение (em) 460 нм) и лиганд Halo618 (em 618 нм) обеспечивают большее спектральное разделение и чувствительность по сравнению с обычным BRET, что делает его идеальной платформой для изучения более слабых взаимодействий и обнаружения тонких изменений в связывании24. Действительно, ранее мы разработали анализ на основе N-BRET для измерения аутоингибиторных взаимодействий доменов RAF REG и CAT, что было важно для характеристики панели мутаций RASopathy в BRAF CRD и продемонстрировало критическую важность этого домена для поддержания аутоингибирования и предотвращения конститутивной активации BRAF12.
Описанный здесь анализ измеряет взаимодействия CRAF, слитого с N-концевой Nano-меткой (Nano-CRAF), и дзета-изоформы 14-3-3, слитой с C-концевой Halo меткой (14-3-3ζ-Halo; Рисунок 1С). Мы показываем, что взаимодействие Nano-CRAF с 14-3-3ζ-Halo генерирует устойчивый сигнал BRET, который, в свою очередь, может быть нарушен мутациями, препятствующими связыванию 14-3-3 с сайтом N' (S259A) и/или сайтом C' (S621A). В следующем протоколе подробно описаны инструкции по выполнению, оптимизации и устранению неполадок этого анализа.
протокол
ПРИМЕЧАНИЕ: Этот анализ проводится на клетках размером 293 фута. Хорошо охарактеризованная и легко трансфицируемая эпителиальная линия, полученная из эмбриональных клеток почек человека. Одна сливающаяся 10-сантиметровая чашка для культивирования этих клеток обычно обеспечивает достаточное количество клеток для засеивания 20 лунок 6-луночных тканевых культуральных планшетов. Шаги 1-3 должны быть выполнены с использованием стерильной техники в шкафу биологической безопасности.
1. Посев клеток (день 1)
ПРИМЕЧАНИЕ: На этом этапе клетки отделяют от чашки (чашек) для культуры тканей, подсчитывают и засевают в 6-луночные планшеты для культивирования тканей для трансфекции на этапе 2 (рис. 2).
- Отсасывайте среду из клеток в 10-сантиметровую чашку. Промойте клетки 5 мл фосфатно-солевого буфера (PBS) и аспиратуйте.
- Добавьте 1 мл трипсина-этилендиаминтетрауксусной кислоты (ЭДТА) и инкубируйте в течение 3-5 минут при 37 °C, чтобы отделить клетки от чашки.
- Добавьте 9 мл полной модифицированной орлиной среды Дульбекко (DMEM) в клетки для нейтрализации трипсина и многократно поднимайте и опускайте пипетку вверх и вниз для получения однородной суспензии одиночных клеток.
- Немедленно перенесите 20 мкл клеток в пробирку объемом 1,7 мл и смешайте с 20 мкл трипанового синего красителя. Подсчитывайте клетки с помощью автоматического счетчика клеток (Table of Materials) или гемоцитометра.
- Разбавьте клетки до 2 x 105 клеток/мл в полной среде DMEM и добавьте по 2 мл в каждую лунку 6-луночного планшета для культуры тканей (4 x 105 клеток/лунка). Инкубируйте клетки при 37 °C и 10%CO2 в течение ночи.
ПРИМЕЧАНИЕ: Рекомендуется использовать элементы 293FT, которые были пройдены менее чем в 20 раз. Ранее мы обнаружили, что использование ячеек с большим числом пассажей может привести к снижению коэффициентов BRET и увеличению вариабельности сигнала от скважины к скважине.
2. Клеточная трансфекция (день 2)
Примечание: Здесь клетки трансфицируются с помощью конструкций экспрессии pCMV5-NanoLuc-CRAF и pCMV5-14-3-3ζ-Halo, а также пустого вектора pCDNA3.1 (Рисунок 2).
- Перед трансфекцией разбавляют N-BRET плазмиды до 5 нг/мкл и pCDNA3,1 до 100 нг/мкл, а также нумеруют набор стерильных пробирок объемом 1,7 мл.
- Добавьте 100 мкл трансфекционной среды (подробнее см. Таблицу материалов ) в каждую пробирку, а также 5 нг pCMV5-NanoLuc-CRAF, 10 нг pCMV5-14-3-3ζ и 200 нг PCDNA3.1.
- Добавьте 2 мкл реагента для трансфекции (подробнее см. Таблицу материалов ) и осторожно перемешайте. Импульс кратковременно вращает пробирки в микроцентрифуге, чтобы убедиться, что вся жидкость собрана на дне пробирок, а затем инкубируется при температуре около 25 °C в течение 15 минут.
- Добавляйте трансфекционные комплексы в клетки по каплям и инкубируйте при 37 °C/10%CO2 в течение 16-20 часов, чтобы обеспечить экспрессию гало- и нано-помеченных белков.
Примечание: Добавление ДНК-носителя пустого вектора (pCDNA3.1) имеет важное значение для достижения высокой эффективности трансфекции конструкций экспрессии N-BRET. Отсутствие добавления пустого вектора приводит к снижению уровней экспрессии белков 14-3-3ζ-Halo и Nano-CRAF и, в свою очередь, приводит к слабым и непостоянным соотношениям BRET, как обсуждалось ранее22.
3. Регирование ячеек (День 3)
Примечание: На этом этапе клетки переносят на 384-луночный планшет и либо лиганд Halo 618 (+лиганд; Таблица материалов) или DMSO (+транспортное средство) добавляется для считывания выбросов BRET на шаге 4. Оставшиеся клетки переносят в свежие 6-луночные культуральные планшеты для вестерн-блоттинга на этапе 5 (рис. 2).
- Соберите следующие материалы и подготовьте рабочую зону, как описано ниже.
- Используя водяную баню при температуре 37 °C, предварительно подогрейте трипсин-ЭДТА, а также бессывороточный фильтрующий материал и 10% пробирный фильтрующий материал с добавлением FBS (подробнее см. Таблицу материалов ).
- В зависимости от количества измеряемых образцов предварительно промаркируйте три комплекта стерильных пробирок объемом 1,7 мл (наборы 1-3) и один комплект стерильных пробирок с коническим дном объемом 15 мл. Оснастите центрифугу с поворотным ведром со вставками для пробирок объемом 15 мл и предварительно охладите до 4 °C.
- Поместите в колпак для тканевых культур следующие предметы: резервуары с реагентами, 384-луночные планшеты для тканевых культур, 6-луночные планшеты для тканевых культур, многоканальную пипетку и наконечники, предметные стекла/камеры для подсчета клеток и трипановый синий краситель (Таблица материалов), а также наборы пробирок объемом 1,7 мл 1-3.
- Соберите и подсчитайте ячейки, как описано ниже.
- Отсадите среду из 6-луночных планшетов и добавьте в клетки 250 мкл трипсина-ЭДТА. Инкубируйте 6-луночные планшеты при температуре 37 °C до тех пор, пока клетки не начнут отделяться (3-5 минут).
- Добавьте 1 мл 10% фильтрующего материала с добавлением FBS в каждую лунку, чтобы нейтрализовать трипсин, и энергично двигайте пипеткой вверх и вниз для получения суспензии одиночных клеток.
- Перенесите 1 мл клеточной суспензии в предварительно помеченные пробирки объемом 15 мл. Добавьте еще 1 мл 10% фильтрующего материала с добавлением FBS в каждую из 15 мл пробирки.
- Переверните пробирки 5 раз, чтобы перемешать, и немедленно перенесите 20 μл клеточной суспензии в набор пробирок 1.
- Центрифугируйте в предварительно охлажденной центрифуге с качающимся ведром в течение 5 минут при ~250 x g. На этапе центрифугирования смешайте 20 мкл трипанового окрашивателя для голубых клеток с клетками в наборе пробирок 1, а затем подсчитайте клетки с помощью автоматического счетчика клеток или гемоцитометра. Типичным является выход 6-8 x 105 клеток/мл.
- Извлеките пробирки объемом 15 мл из центрифуги и аспирируйте среду из клеточных гранул. Суспендируйте клеточные гранулы до концентрации 2 x 105 клеток/мл в бессывороточной среде для анализа и энергично пипетке с получением одноклеточной суспензии.
ПРИМЕЧАНИЕ: Использование бессывороточных аналитических сред используется для замораживания нормальных клеточных сигнальных путей.
- Перепланируйте ячейки в 384-луночных и 6-луночных планшетах, как описано ниже.
- Переверните пробирки объемом 15 мл несколько раз, чтобы получить однородную клеточную суспензию, и перенесите пробирки объемом от 500 мкл до 1,7 мл в наборе 2 и наборе 3.
- Добавьте 0,5 мкл ДМСО (+носитель) в набор 2 и 0,5 мкл лиганда Halo 618 (+лиганд) в набор пробирок 3 и перемешайте пипетку.
- Перенесите суспензии ячеек +vehicle и +ligand в отдельные лунки резервуаров с реагентами. С помощью многоканальной пипетки (Таблица материалов) перенесите 40 мкл суспензии +vehicle cell из резервуаров с реагентами в четырехлуночные лунки 384-луночного планшета для культивирования. Повторите этот шаг для суспензий клеток +лиганда.
- Переложите оставшиеся клетки в свежие 6-луночные культуральные планшеты. Инкубируйте 384-луночные и 6-луночные планшеты в течение ночи при 37 °C и 5%CO2.
4. Чтение выбросов BRET (День 4)
Примечание: На этом этапе субстрат нанолюциферазы (подробнее см. Таблицу материалов ) добавляют к клеткам в 384-луночном культуральном планшете и считывают акцептор N-BRET (618 нм) и донорский (460 нм) излучения (рис. 2). Затем рассчитываются скорректированные коэффициенты BRET.
- Предварительно подогрейте бессывороточную среду для анализа на водяной бане при температуре 37 °C и разморозьте нанолюциферазный субстрат при 25 °C. Разведите субстрат нанолюциферазы в соотношении 1:100 в бессывороточной среде для анализа и перенесите в резервуар с реагентами. Приготовьте достаточное количество этой смеси для добавления 10 μL в каждую лунку 384-луночного планшета, плюс 10%-15% дополнительного объема.
- С помощью многоканальной пипетки (Таблица материалов) перенесите по 10 мкл субстратной смеси в каждую из лунок, содержащих клетки в 384-луночном культуральном планшете. Осторожно вращайте тарелку в течение 1 минуты вручную или с помощью орбитального шейкера.
- Вставьте 384-луночный планшет в многомодовый считыватель планшетов и зарегистрируйте выбросы 460 нм и 618 нм для всех лунок, содержащих ячейки.
ПРИМЕЧАНИЕ: Для многорежимного считывателя пластин, используемого на этом этапе (подробнее см. Таблицу материалов ), использовалась высота считывания 6,5 мм с измерением времени считывания 0,1 с, однако при использовании других считывателей пластин может потребоваться дальнейшая оптимизация. - Рассчитайте исходные соотношения BRET для +vehicle и + лиганда в единицах milliBRET (mBU) по отдельности, используя следующее уравнение:
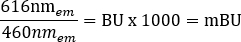
- Рассчитайте скорректированное отношение BRET путем вычитания +vehicle BRET ratio из коэффициента +Ligand (дополнительная таблица 1).
ПРИМЕЧАНИЕ: При сравнении результатов нескольких экспериментов скорректированные соотношения BRET могут быть объединены и нормализованы до среднего значения пары Wildtype (WT) N-BRET, состоящей из Nano-CRAFWT и 14-3-3ζWT-Halo (Дополнительная таблица 1).
5. Подтверждение уровня экспрессии белка (4 и 5 дни)
Примечание: На этом этапе клетки в 6-луночных планшетах лизируют, а уровни экспрессии белков Nano-CRAF и 14-3-3ζ-Halo определяют с помощью вестерн-блоттинга с использованием антител, специфичных к меткам Halo и Nano. (Рисунок 2).
- Добавьте ингибиторы протеазы и фосфатазы в буфер для лизиса NP40 (Таблица материалов), обеспечивая 200 мкл буфера для лизиса на образец.
- Аспиратные среды из 6-луночных планшетов, которые содержат клетки. Промойте клетки один раз 1 мл холодного PBS и аспиратуйте.
- Добавьте 125 мкл буфера для лизиса NP40 в каждую лунку и инкубируйте 6-луночные планшеты при 4 °C на качающейся платформе в течение 15 мин для лизина клеток.
- Перенесите лизаты в пробирки объемом 1,7 мл на льду и центрифугируйте при температуре 20 000 x g в течение 10 минут при 4 °C. Поместите очищенные лизаты обратно на лед и определите концентрацию белка с помощью коммерчески доступных анализов, таких как анализ Брэдфорда или бицинхониновая кислота (BCA).
- Нормализуйте концентрацию белка во всех образцах путем переноса соответствующего объема лизата и буфера для лизиса NP40 в свежие пробирки объемом 1,7 мл до общего объема 100 мкл.
- Вскипятите 5-кратный буфер для образцов белка (Таблица материалов) в течение 1 минуты и добавьте 25 мкл в каждый образец. Прокипятите образцы в течение 5-6 минут, а затем кратковременно центрифугируйте пробирки, чтобы убедиться, что весь образец собирается в основании пробирки.
- Загрузите 25 мкл образца в каждую лунку дубликатов белковых гелей (гель 1 и гель 2), а затем перенесите белки на мембраны из нитроцеллюлозы или PVDF, используя стандартные процедуры вестерн-блоттинга, как описано ранее22.
- Заблокируйте мембраны в 3% BSA-PBS при 25 °C в течение 30 минут, а затем инкубируйте мембраны в течение ночи с антителом Halo (разведение 1:1,000; Гель 1) и либо нанолюциферазу, либо антитела CRAF (разведение 1:500; Гель 2) в трис-буферном физрастворе, с добавлением 0,2% Tween-20 (TBST; Таблица материалов).
- Промойте мембраны 1 раз в течение 5 минут в 10 мл TBST, а затем инкубируйте при комнатной температуре в течение 1 ч с вторичным антителом против мышиного HRP, разведенным в соотношении 1:10 000 в TBST.
- Промойте мембраны 3 раза на качающейся платформе в 10 мл TBST при 25 °C в течение 5 минут каждая. Удалите TBST и визуализируйте белковые полосы с помощью реагентов ECL и либо рентгеновского процессора пленки, либо другой подходящей системы визуализации.
Результаты
При выполнении в соответствии с описанием в данном протоколе (рис. 2) взаимодействие Nano-CRAFWT и 14-3-3ζ-Halo должно привести к скорректированным соотношениям BRET 50-60 мБУ (рис. 3A; Дополнительная таблица 1). CRAF содержит два фосфорилированных 14-3-3 стыковочных сайта, N'-сайт и C'-сайт (рис. 1)8. Таким образом, соответствующие меры контроля для снижения связывания CRAF:14-3-3ζ включают S259A и S621A, которые предотвращают фосфорилирование и, в свою очередь, связывание 14-3-3 с сайтами N' и C'соответственно25. Как показано на рисунке 3A, Nano-CRAFS259A снижает сигнал BRET на ~45%, а Nano-CRAFS621A на ~25%, в то время как двойной мутант S259A/S621A (SS/AA) почти полностью абляирует сигнал CRAF:14-3-3 BRET. Кроме того, результаты нескольких независимых экспериментов можно сравнить, сначала нормализовав результаты каждого эксперимента до среднего скорректированного отношения BRET (дополнительная таблица 1), а затем объединив данные всех трех экспериментов (рисунок 3B). Относительные уровни экспрессии меченых белков также должны быть подтверждены вестерн-блоттингом (рис. 3). Следует отметить, что уровни экспрессии мутантных белков могут отличаться от таковых у WT, что требует соответствующей корректировки трансфицированных количеств мутантных векторов ДНК (Дополнительная таблица 1).
Следует также отметить, что может потребоваться оптимизация этого протокола, особенно при использовании альтернативного считывателя планшетов, клеточных катетеров или реагентов для трансфекции. На рисунке 4А представлены результаты эксперимента по оптимизации, в котором увеличение трансфицированного количества акцепторной плазмиды pCMV5-14-3-3ζ-Halo, сохраняя при этом количество донора pCMV5-NanoLuc-CRAF постоянным, доза зависимо увеличивает сигнал BRET, который затем уменьшается за счет присутствия мутанта CRAFSS/AA с дефицитом 14-3-3-связывания. Однако, если отношение акцептора к донору слишком велико, разница в сигнале между связыванием 14-3-3ζ-Halo с Nano-CRAFWT и отрицательным контролем Nano-CRAFSS/AA будет уменьшена, что, вероятно, отражает насыщение связывания Nano-CRAFWT. Кроме того, после достижения коэффициента BRET, который генерирует воспроизводимые значения с низкой стандартной ошибкой, дальнейшее увеличение отношения акцептора к донору не требуется. Действительно, одним из основных преимуществ этих анализов является возможность измерения взаимодействий белков на физиологических уровнях экспрессии. При разработке новых анализов N-BRET еще одним важным фактором является размещение меток. На рисунке 4B показано влияние переключения метки Halo с C-конца на N-конец 14-3-3ζ на скорректированные соотношения BRET, генерируемые связыванием 14-3-3ζ с Nano-CRAFWT и Nano-CRAFSS/AA. С этой целью должны быть сгенерированы конструкции экспрессии ДНК для каждого из возможных местоположений меток (рис. 4C) и оценено их влияние на соотношения BRET как пары WT, так и известного отрицательного контроля для связывания.

Рисунок 1: Регуляция RAF 14-3-3 белками. (A) Схема структуры домена киназы RAF с указанием консервативных 14-3-3 сайтов стыковки. (B) Модели регуляции RAF белками 14-3-3. (C) Анализ N-BRET для измерения взаимодействий 14-3-3ζ, слитого с C-концевым акцептором Halo tag, и CRAF, слитого с N-концевым донором Nano. Передача энергии от донора к акцептору генерирует сигнал BRET. Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этой цифры.

Рисунок 2: Основные этапы выполнения анализа на основе BRET живых клеток CRAF:14-3-3. Показаны пять основных этапов, описанных в этом протоколе, в том числе: посев, трансфекция и реплатирование клеток, а также методы считывания сигнала BRET и подтверждения уровней экспрессии гало- и нано-меченых белков. Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этой цифры.

Рисунок 3: CRAF:14-3-3 Анализ на основе BRET. Анализ N-BRET сравнивает взаимодействия 14-3-3ζ-Halo либо с Nano-CRAF -WT, либо с мутантными формами -S259A, -S621A и -S259A/S621A (SS/AA), которые ранее показали нарушение связывания CRAF:14-3-325. Уровни экспрессии 14-3-3ζ-Halo (MW: 64 kDa.) и вариантов Nano-CRAF (MW: 93 kDa.) были изучены с помощью вестерн-блоттинга. (A) Скорректированные показания BRET из четырех повторяющихся лунок из одного эксперимента ± стандартным отклонением (SD). (B) Скорректированные показания BRET из трех независимых экспериментов были объединены и нормализованы до Nano-CRAFWT (установлено на 100) ± SD. Статистическая значимость была рассчитана с помощью t-критерия Стьюдента (двуххвостый, предполагающий равную дисперсию). р<0,0001. Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этой цифры.
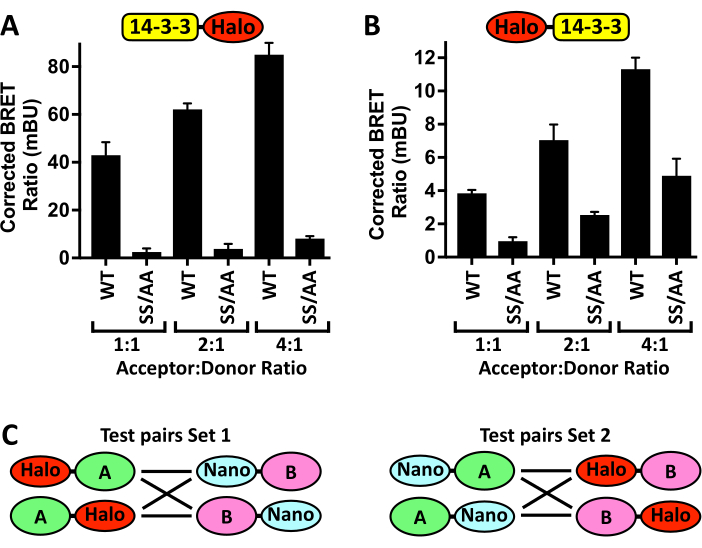
Рисунок 4: Этапы оптимизации анализа. Эффекты котрансфекции возрастающих количеств (A) 14-3-3ζ-Halo или (B) Halo-14-3-3ζ с постоянным количеством Nano-CRAF -WT или -SS/AA (S259A/S621A) на сигнал BRET ± SD. (C) Схема всех возможных пар N-BRET, полученных путем размещения метки Halo или Nano на N-конце или C-конце каждого представляющего интерес белка (A и B). n = 4. Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этой цифры.
Дополнительная таблица 1: Примеры данных анализа на основе CRAF:14-3-3 BRET. Указанные конструкты экспрессии трансфицировали в клетки на 2-й день (верхняя секция). Исходные, скорректированные и нормализованные показания BRET были измерены и рассчитаны на 4-й день (нижняя секция). Пожалуйста, нажмите здесь, чтобы загрузить этот файл.
Обсуждение
Предыдущие исследования показали, что белки 14-3-3 играют решающую роль как в активации, так и в ингибировании киназ RAF. Понимание того, как регулируются эти события связывания и влияние модуляции этих взаимодействий на передачу сигналов RAF и онкогенез, вызванный RAF, может выявить новые терапевтические уязвимости, нацеленные на функцию CRAF. Тем не менее, цикл активации Raf поддерживается множеством ассоциированных белков, посттрансляционных модификаций и изменений в субклеточной локализации8, и поэтому для полного повторения этих условий необходимо измерить взаимодействие RAF с его регуляторами в условиях живых клеток. Этот протокол описывает новый анализ, основанный на близости, который использует превосходное отношение сигнал/шум и спектральное разрешение пары нанолюцифераза-Halo N-BRET для измерения взаимодействий 14-3-3ζ и CRAF в живых клетках.
Несмотря на то, что этот анализ обеспечивает столь необходимую платформу для изучения сложных взаимодействий 14-3-3 и CRAF в физиологически значимых условиях, он имеет ряд ограничений, которые следует учитывать. Во-первых, учитывая, что было показано, что 14-3-3 играет роль как в аутоингибированииRAF, так и в димеризации, могут быть включены дополнительные средства контроля и экспериментальные подходы для различения этих состояний. Например, этот анализ может быть выполнен в сочетании с ранее разработанным анализом аутоингибирования N-BRET, который обеспечивает прямое считывание изменений в аутоингибировании RAF12,22. Кроме того, контрольные группы, такие как мутация CRAF R401H, которая, как было показано, нарушает димеризацию CRAF с другими протомерами RAF15,26, также могут оказаться полезными для анализа этих функций. Наконец, размещение меток Halo и Nano оптимизировано для измерения взаимодействия CRAF:14-3-3, но не может гарантировать, что в результате взаимодействия CRAF или 14-3-3 с другими белками не будут затронуты, а также этот анализ не может определить аффинность связывания CRAF:14-3-3, влияние этих взаимодействий на активность CRAF или место в клетке, где эти взаимодействия происходят. Следовательно, может также потребоваться включение других дополнительных подходов, таких как анализы in vitro, анализы киназы CRAF и исследования субклеточной локализации 12,27,28,29.
При проведении этого анализа, как и любого другого анализа на основе N-BRET, необходимо учитывать ряд факторов. Как показано в этой рукописи и в предыдущей работе22, правильное соотношение трансфекции конструкций pCMV5-Halo (акцептор) и pCMV5-нанолюциферазы (донора), наряду с соответствующим отрицательным контролем, таким как мутант, который, как известно, нарушает связывание POI, имеет решающее значение для оптимизации и тестирования этих анализов. Увеличение экспрессии POI, помеченных Halo, при сохранении постоянных уровней POI, помеченных Nano, приводит к увеличению коэффициентов BRET. Однако, как только привязка будет насыщена, сигнал выровняется, и, в свою очередь, разница между WT и POI с нарушениями привязки уменьшится, что потребует оптимизации акцептор-донор. Еще одним важным фактором в оптимизации анализа является использование пустого векторного носителя ДНК, такого как pCDNA3.1. Включение 200 нг pCDNA3.1. в трансфекционной смеси значительно увеличивает уровни экспрессии гало- и нано-помеченных POI и снижает вариабельность между реплицируемыми лунками и экспериментами22. Однако, учитывая, что эффективность трансфекции может варьироваться в зависимости от реагентов и клеточных линий, количество ДНК-носителя также может нуждаться в титровании для достижения оптимальной экспрессии пары N-BRET, как описано ранее22. Еще одним фактором, который сильно влияет на соотношения BRET, является размещение тегов. Действительно, то, какой POI слит с тегом Halo по сравнению с меткой Nano и размещена ли эта метка на N- или C-конце белка, может повлиять как на амплитуду отношения BRET, так и на способность негативных элементов управления снижать этот сигнал. Поэтому также важно проверить каждую из возможных пар N-BRET при разработке новых анализов.
Повышенная чувствительность и спектральное разрешение, достигнутые N-BRET по сравнению с обычным BRET, позволяют измерять более слабые взаимодействия и тонкие изменения в связывании в живых клетках. Тем не менее, полезность этих анализов для изучения клеточных сигнальных событий не была полностью осознана. Примечательно, что этот анализ, вероятно, может быть адаптирован для изучения взаимодействия 14-3-3 с другими киназами, такими как RAF-киназы ARAF и BRAF, и в сочетании с ранее опубликованными анализами N-BRET для изучения связывания RAS:RAF и аутоингибирования RAF22,30, сформирует основу набора инструментов для измерения регуляции RAF в условиях живых клеток. Кроме того, N-BRET показал себя многообещающим в качестве платформы для скрининга лекарственных препаратовв живых клетках30, и поэтому этот анализ может быть также полезен для идентификации соединений, которые либо стабилизируют ингибитор при взаимодействии 14-3-3 и RAF, либо нарушают действия, участвующие в активации RAF. Учитывая высокую стабильность сигнала нанолюциферазы и способность современных планшетных ридеров одновременно обнаруживать акцепторную и донорскую эмиссию, из этого также следует, что эти анализы могут быть адаптированы для измерения взаимодействий сигнальных белков в режиме реального времени, что будет иметь решающее значение для понимания динамики этих взаимодействий в ответ на предварительную стимуляцию или медикаментозное лечение.
Раскрытие информации
Раскрывать нечего.
Благодарности
Этот проект был частично профинансирован за счет федеральных средств Национального института рака, Национальных институтов здравоохранения, под номером ZIA BC 010329.
Материалы
| Name | Company | Catalog Number | Comments |
| Antibodies | |||
| HaloTag® mouse monoclonal antibody | Promega | G9211 | Antibody for detecting HaloTag tagged proteins by immunoblot |
| NanoLuc® mouse monoclonal antibody | R&D Systems | MAB10026 | Antibody for detecting Nano-tagged proteins by immunoblot |
| CRAF mouse monoclonal antibody (E10) | Santa Crus Biotechnology | sc-7267 | Antibody directly detecting CRAF proteins by immunoblot |
| ECL anti-mouse HRP secondary antibody | Amersham | NA931-1ML | Secondary HRP conjugated mouse antibody (from sheep) |
| Reagents | |||
| X-tremeGENE™ 9 | Roche/Sigma | 6365809001 | |
| NanoBRET™ kit | Promega | N1661 | NanoBRET kit containing Halo 618 ligand and NanoGlo (nanoluciferase) substrate |
| DPBS, without Ca++ and Mg++ | Quality Biologicals | 114-057-101 | |
| Trypsin-EDTA (0.05%), phenol red | Life Technologies | 25300120 | |
| DMEM cell culture media | Life Technologies | 11995073 | High glucose, L-glutamine, phenol red, sodium pyruvate; without HEPES, suppliment media with 10% FBS, 2 mM L-glutamine and 100U penicillin-streptomycin |
| L-Glutamine (200 mM) | Life Technologies | 25030164 | |
| Penicillin-Streptomycin (10,000 U/mL) | Life Technologies | 15140163 | |
| Opti-MEM™ I reduced serum media | Gibco | 31985062 | For cell transfection |
| Opti-MEM reduced serum media, no phenol red | Gibco | 11058021 | For replating cells on Day 3. Supplement with 2 mM L-glutamine and 100U penicillin-streptomycin, along with 10% FBS (where indicated). |
| Invitrogen Trypan Blue Stain | Thermo Scientific | T10282 | |
| NP40 lysis buffer | N/A | N/A | 20 mM Tris (pH 8.0), 137mM NaCl, 10% glycerol, NP40 alternative (Milipore, Cat# 492016). Store at 4 degrees C.. Add the following protease and phosphatase immediately prior to use: 20 µM leupeptin, 0.5 mM sodium orthovanidate, 0.15 U/mL, 1mM PMSF. |
| 5x gel sample buffer | N/A | N/A | 240 mM Tris (pH 8.0), 9.5% SDS, 30% glycerol, 500mM DTT, 3mM bromophenol blue. Store at -20 degrees C. |
| Cell lines | |||
| 293FT cells (human) | Thermo Scientific | R70007 | |
| DNA vectors | |||
| pCMV5-Nano-CRAF WT and mutant | N/A | N/A | |
| pCMV5-14-3-3ζ-Halo | N/A | N/A | |
| Equipment | |||
| EnVision 2104 Multimode Plate Reader | PerkinElmer 2104 | 2104-0010 | 600LP NanoBRET & M460/50 nm NanoBRET emmisions filters, Luminescence 404 mirror, 6.5 mm measurement height and 0.1 s measurement time |
| Invitrogen Countess™ II Automated Cell Counter | Thermo Scientific | AMQAX1000 | |
| ThermoFisher E1-ClipTip™ Multichannel Pipettor | Thermo Scientific | 4672070 | |
| Software | |||
| GraphPad Prism (version 10.0.3) | GraphPad | www.graphpad.com | |
| Other | |||
| ThermoFisher ClipTip Multichannel pipette tips | Thermo Scientific | 94410153 | |
| Reagent Reservoir, 25 mL Divided, Sterile | Thomas Scientific | 1228K16 | |
| Perkin Elmer 384-well CulturPlate™ | PerkinElmer | 6007680 | White, polystyrene, tissue culture treated |
| Countess Cell Counting Chamber Slides | Thermo Scientific | C10228 |
Ссылки
- Blasco, R. B., et al. c-Raf, but not B-Raf, is essential for development of K-Ras oncogene-driven non-small cell lung carcinoma. Cancer Cell. 19, 652-663 (2011).
- Blasco, M. T., et al. Complete regression of advanced pancreatic ductal adenocarcinomas upon combined inhibition of EGFR and C-RAF. Cancer Cell. 35, 573-587 (2019).
- Karreth, F. A., Frese, K. K., DeNicola, G. M., Baccarini, M., Tuveson, D. A. C-Raf is required for the initiation of lung cancer by K-Ras(G12D). Cancer Discov. 1, 128-136 (2011).
- Lito, P., et al. Disruption of CRAF-mediated MEK activation is required for effective MEK inhibition in KRAS mutant tumors. Cancer Cell. 25, 697-710 (2014).
- Sanclemente, M., et al. c-RAF ablation induces regression of advanced Kras/Trp53 mutant lung adenocarcinomas by a mechanism independent of MAPK signaling. Cancer Cell. 33, 217-228 (2018).
- Razzaque, M. A., et al. Germline gain-of-function mutations in RAF1 cause Noonan syndrome. Nat Genet. 39, 1013-1017 (2007).
- Pandit, B., et al. Gain-of-function RAF1 mutations cause Noonan and LEOPARD syndromes with hypertrophic cardiomyopathy. Nat Genet. 39, 1007-1012 (2007).
- Terrell, E. M., Morrison, D. K. Ras-mediated activation of the Raf family kinases. Cold Spring Harb Perspect Med. 9 (1), 033746(2019).
- Kondo, Y., et al. Cryo-EM structure of a dimeric B-Raf:14-3-3 complex reveals asymmetry in the active sites of B-Raf kinases. Science. 366, 109-115 (2019).
- Park, E., et al. Architecture of autoinhibited and active BRAF-MEK1-14-3-3 complexes. Nature. 575 (7783), 545-550 (2019).
- Tzivion, G., Luo, Z., Avruch, J. A dimeric 14-3-3 protein is an essential cofactor for Raf kinase activity. Nature. 394, 88-92 (1998).
- Spencer-Smith, R., et al. RASopathy mutations provide functional insight into the BRAF cysteine-rich domain and reveal the importance of autoinhibition in BRAF regulation. Mol Cell. 82, 4262-4276 (2022).
- Martinez Fiesco, J. A., Durrant, D. E., Morrison, D. K., Zhang, P. Structural insights into the BRAF monomer-to-dimer transition mediated by RAS binding. Nat Commun. 13, 486(2022).
- Freeman, A. K., Ritt, D. A., Morrison, D. K. The importance of Raf dimerization in cell signaling. Small GTPases. 4, 180-185 (2013).
- Freeman, A. K., Ritt, D. A., Morrison, D. K. Effects of Raf dimerization and its inhibition on normal and disease-associated Raf signaling. Mol Cell. 49, 751-758 (2013).
- Rushworth, L. K., Hindley, A. D., O'Neill, E., Kolch, W. Regulation and role of Raf-1/B-Raf heterodimerization. Mol Cell Biol. 26, 2262-2272 (2006).
- Garnett, M. J., Rana, S., Paterson, H., Barford, D., Marais, R. Wild-type and mutant B-RAF activate C-RAF through distinct mechanisms involving heterodimerization. Mol Cell. 20, 963-969 (2005).
- Tran, N. H., Wu, X., Frost, J. A. B-Raf and Raf-1 are regulated by distinct autoregulatory mechanisms. J Biol Chem. 280, 16244-16253 (2005).
- Chong, H., Guan, K. L. Regulation of Raf through phosphorylation and N terminus-C terminus interaction. J Biol Chem. 278, 36269-36276 (2003).
- Cutler, R. E., Stephens, R. M., Saracino, M. R., Morrison, D. K. Autoregulation of the Raf-1 serine/threonine kinase. Proc Natl Acad Sci U S A. 95, 9214-9219 (1998).
- Park, E., et al. Cryo-EM structure of a RAS/RAF recruitment complex. Nat Commun. 14, 4580(2023).
- Spencer-Smith, R., Morrison, D. K. Protocol for measuring BRAF autoinhibition in live cells using a proximity-based NanoBRET assay. STAR Protoc. 4, 102461(2023).
- Clark, G. J., et al. 14-3-3 zeta negatively regulates raf-1 activity by interactions with the Raf-1 cysteine-rich domain. J Biol Chem. 272, 20990-20993 (1997).
- Machleidt, T., et al. NanoBRET--A novel BRET platform for the analysis of protein-protein interactions. ACS Chem Biol. 10, 1797-1804 (2015).
- Hekman, M., et al. Dynamic changes in C-Raf phosphorylation and 14-3-3 protein binding in response to growth factor stimulation: differential roles of 14-3-3 protein binding sites. J Biol Chem. 279, 14074-14086 (2004).
- Hatzivassiliou, G., et al. RAF inhibitors prime wild-type RAF to activate the MAPK pathway and enhance growth. Nature. 464, 431-435 (2010).
- Bondzi, C., Grant, S., Krystal, G. W. A novel assay for the measurement of Raf-1 kinase activity. Oncogene. 19, 5030-5033 (2000).
- Spencer-Smith, R., et al. Inhibition of RAS function through targeting an allosteric regulatory site. Nat Chem Biol. 13 (1), 62-68 (2016).
- Roy, S., et al. 14-3-3 facilitates Ras-dependent Raf-1 activation in vitro and in vivo. Mol Cell Biol. 18, 3947-3955 (1998).
- Durrant, D. E., et al. Development of a high-throughput NanoBRET screening platform to identify modulators of the RAS/RAF interaction. Mol Cancer Ther. 20, 1743-1754 (2021).
Перепечатки и разрешения
Запросить разрешение на использование текста или рисунков этого JoVE статьи
Запросить разрешениеThis article has been published
Video Coming Soon
Авторские права © 2025 MyJoVE Corporation. Все права защищены