Method Article
Dosage basé sur le transfert d’énergie par résonance de bioluminescence (BRET) pour mesurer les interactions de CRAF avec les protéines 14-3-3 dans les cellules vivantes
Dans cet article
Résumé
Ce protocole décrit un test basé sur BRET pour mesurer les interactions de la kinase CRAF avec les protéines 14-3-3 dans des cellules vivantes. Le protocole décrit les étapes de préparation des cellules, de lecture des émissions BRET et d’analyse des données. Un exemple de résultat avec l’identification des contrôles appropriés et le dépannage pour l’optimisation du test est également présenté.
Résumé
CRAF est un effecteur primaire des GTPases RAS et joue un rôle essentiel dans la tumorigenèse de plusieurs cancers induits par KRAS. De plus, CRAF est un point chaud pour les mutations germinales, qui causent la RASopathie développementale, le syndrome de Noonan. Toutes les kinases RAF contiennent de multiples sites de liaison dépendants de la phosphorylation pour les protéines régulatrices 14-3-3. La liaison différentielle de 14-3-3 à ces sites joue un rôle essentiel dans la formation de dimères RAF actifs au niveau de la membrane plasmique dans des conditions de signalisation et dans le maintien de l’auto-inhibition de RAF dans des conditions de repos. Comprendre comment ces interactions sont régulées et comment elles peuvent être modulées est essentiel pour identifier de nouvelles approches thérapeutiques qui ciblent la fonction RAF. Ici, je décris un test basé sur le transfert d’énergie par résonance de bioluminescence (BRET) pour mesurer les interactions de CRAF avec les protéines 14-3-3 dans des cellules vivantes. Plus précisément, ce test mesure les interactions de CRAF fusionné à un donneur de nano luciférase et de 14-3-3 fusionné à un accepteur de marqueurs Halo, où l’interaction de RAF et 14-3-3 entraîne un transfert d’énergie de donneur à accepteur et la génération du signal BRET. Le protocole montre en outre que ce signal peut être perturbé par des mutations qui empêchent la liaison de 14-3-3 à chacun de ses sites d’amarrage RAF de haute affinité. Ce protocole décrit les procédures d’ensemencement, de transfection et de replacage des cellules, ainsi que des instructions détaillées pour lire les émissions BRET, effectuer l’analyse des données et confirmer les niveaux d’expression des protéines. De plus, des exemples de résultats d’analyse, ainsi que des étapes d’optimisation et de dépannage, sont fournis.
Introduction
Les kinases RAF (ARAF, BRAF et CRAF) sont les effecteurs directs des GTPases RAS et les membres initiateurs de la cascade de kinases RAF-MEK-ERK pro-proliférative/pro-survie. Des études récentes ont montré que l’expression de CRAF joue un rôle clé dans la tumorigenèse de plusieurs cancers induits par KRAS, notamment le cancer du poumon non à petites cellules et l’adénocarcinome canalaire pancréatique 1,2,3,4,5. De plus, les mutations germinales de CRAF provoquent une forme particulièrement sévère de la RASopathie, le syndrome de Noonan 6,7. La compréhension de la régulation de la CRAF est essentielle pour développer des approches thérapeutiques efficaces qui ciblent sa fonction dans les cellules.
Toutes les kinases RAF peuvent être divisées en deux domaines fonctionnels, un domaine catalytique C-terminal (CAT) et un domaine régulateur N-terminal (REG), qui contrôle son activité (Figure 1A)8. Le domaine REG englobe le domaine de liaison RAS (RBD), le domaine riche en cystéine (CRD) et une région riche en sérine/thréonine (riche en S/T). Notamment, la région riche en S/T contient le site N', qui se lie à 14-3-3 de manière dépendante de la phosphorylation (S259 dans CRAF ; Graphique 1A) 8. Le domaine CAT englobe le domaine kinase, ainsi qu’un deuxième site d’amarrage 14-3-3 de haute affinité, appelé site C'(S621 en CRAF ; Graphique 1A) 8. La liaison différentielle des protéines dimériques 14-3-3 aux sites N’et C', ainsi que le CRD, jouent un rôle essentiel dans l’activation et l’inhibition de RAF 9,10,11,12,13. Dans des conditions normales de signalisation, l’activation de RAF est initiée par son recrutement dans la membrane plasmique par RAS, ce qui lui permet de former des dimères actifs, dont l’hétérodimère BRAF-CRAF est la forme active prédominante14,15. Les analyses biochimiques avec BRAF et CRAF, ainsi que les structures de microscopie électronique cryogénique (Cryo-EM) des BRAF dimériques, indiquent qu’un dimère 14-3-3 stabilise les dimères RAF actifs en se liant simultanément au site C’des deux protomères RAF (Figure 1B)9,13,16,17. À l’inverse, des études ont montré que dans des conditions de repos, RAF adopte une confirmation cytosolique, auto-inhibée, où le domaine REG se lie au domaine CAT et inhibe son activité 12,18,19,20. Cet état fermé est stabilisé par un dimère 14-3-3 lié au CRD et au site N’dans le domaine REG et au site C’dans le domaine CAT (Figure 1B)10,13,21. Dans BRAF, ce modèle est soutenu par les structures Cryo-EM récentes de monomères BRAF auto-inhibés et par nos études biochimiques précédentes 10,12,13,21,22. Cependant, alors qu’il est démontré que le 14-3-3 joue un rôle inhibiteur dans la régulation23 de la CRA, un état auto-inhibé de type BRAF peut jouer un rôle moindre dans la régulation12 de la CRAC ; par conséquent, d’autres études sont nécessaires pour clarifier les mécanismes par lesquels les protéines 14-3-3 régulent l’activité de CRAF. La régulation des kinases RAF médiée par 14-3-3 nécessite une pléthore d’événements de phosphorylation et de désphosphorylation RAF, la liaison à diverses protéines régulatrices et des interactions avec la membrane plasmique8. Par conséquent, il est essentiel que les interactions 14-3-3-RAF soient mesurées dans des conditions physiologiquement pertinentes et en présence d’une bicouche lipidique intacte.
Pour résoudre ce problème, la technologie NanoBRET (ci-après appelée N-BRET ; voir le tableau des matériaux pour les détails du kit) a été utilisée pour développer un test basé sur la proximité permettant de mesurer les interactions de CRAF avec les protéines 14-3-3 dans les cellules vivantes (Figure 1C). Ce système basé sur BRET mesure les interactions de deux protéines d’intérêt (POI), où une protéine est marquée avec un donneur de nanoluciférase (Nano) et l’autre avec un marqueur Halo, pour le marquage avec le ligand accepteur d’énergie Halo61822,24. L’interaction des protéines d’intérêt entraîne un transfert d’énergie de donneur à accepteur, qui à son tour génère le signal BRET (Figure 1C). La protéine donneuse Nano extrêmement brillante (émission (em) 460 nm) et le ligand Halo618 (em 618 nm) offrent une séparation spectrale et une sensibilité supérieures à celles de BRET conventionnelles, ce qui en fait une plate-forme idéale pour étudier les interactions plus faibles et détecter les changements subtils de liaison24. En effet, nous avons précédemment développé un test basé sur N-BRET pour mesurer les interactions auto-inhibitrices des domaines RAF REG et CAT, ce qui a été essentiel pour la caractérisation d’un panel de mutations RASopathy dans le CRD BRAF et a démontré l’importance critique de ce domaine pour maintenir l’autoinhibition et prévenir l’activation constitutive de BRAF12.
Le test décrit ici mesure les interactions de CRAF, fusionné à une étiquette Nano N-terminale (Nano-CRAF), et de l’isoforme zêta de 14-3-3 fusionnée à l’étiquette Halo C-terminale (14-3-3ζ-Halo ; Figure 1C). Nous montrons que les interactions de Nano-CRAF avec 14-3-3ζ-Halo génèrent un signal BRET robuste, qui peut à son tour être perturbé par des mutations qui empêchent la liaison de 14-3-3 au site N'(S259A) et/ou au site C'(S621A). Le protocole suivant fournit des étapes détaillées pour effectuer, optimiser et dépanner ce test.
Protocole
REMARQUE : Ce test est effectué dans des cellules de 293FT. Une lignée épithéliale bien caractérisée et facilement transfectable dérivée de cellules rénales embryonnaires humaines. Une seule boîte de culture confluente de 10 cm de ces cellules fournit généralement suffisamment de cellules pour ensemencer 20 puits de plaques de culture tissulaire à 6 puits. Les étapes 1 à 3 doivent être effectuées à l’aide d’une technique stérile dans une enceinte de sécurité biologique.
1. Ensemencement cellulaire (jour 1)
REMARQUE : Dans cette étape, les cellules sont détachées de la ou des boîtes de culture tissulaire, comptées et ensemencées dans des plaques de culture tissulaire à 6 puits pour la transfection à l’étape 2 (Figure 2).
- Aspirez le milieu des cellules dans la parabole de 10 cm. Laver les cellules avec 5 mL de solution saline tamponnée au phosphate (PBS) et aspirer.
- Ajouter 1 ml d’acide trypsine-éthylènediaminetétraacétique (EDTA) et incuber pendant 3 à 5 minutes à 37 °C pour détacher les cellules de la boîte.
- Ajoutez 9 ml de milieu complet Dulbecco’s modified eagle (DMEM) dans les cellules pour neutraliser la trypsine et pipetez de haut en bas à plusieurs reprises pour générer une suspension unicellulaire homogène.
- Transférez immédiatement 20 μL de cellules dans un tube de 1,7 ml et mélangez avec 20 μL de colorant bleu trypan. Comptez les cellules à l’aide d’un compteur de cellules automatisé (Table des matériaux) ou d’un hémocytomètre.
- Diluer les cellules à 2 x 105 cellules/mL dans un milieu DMEM complet et ajouter 2 mL dans chaque puits d’une plaque de culture tissulaire à 6 puits (4 x 105 cellules/puits). Incuber les cellules à 37 °C et 10 % de CO2 pendant la nuit.
REMARQUE : Il est recommandé d’utiliser des cellules de 293FT qui ont été passées moins de 20x. Nous avons précédemment constaté que l’utilisation de cellules avec un plus grand nombre de passages peut entraîner une réduction des rapports BRET et une augmentation de la variabilité du signal de puits à puits.
2. Transfection cellulaire (jour 2)
REMARQUE : Ici, les cellules sont transfectées avec les constructions d’expression pCMV5-NanoLuc-CRAF et pCMV5-14-3-3ζ-Halo, ainsi que le vecteur vide pCDNA3.1 (Figure 2).
- Avant la transfection, diluer les plasmides N-BRET à 5 ng/μL et pCDNA3.1 à 100 ng/μL, et numéroter un ensemble de tubes stériles de 1,7 mL.
- Ajoutez 100 μL de milieu de transfection (voir le tableau des matériaux pour plus de détails) dans chaque tube, ainsi que 5 ng de pCMV5-NanoLuc-CRAF, 10 ng de pCMV5-14-3-3ζ et 200 ng de PCDNA3.1.
- Ajouter 2 μL de réactif de transfection (voir le tableau des matériaux pour plus de détails) et agiter doucement pour mélanger. Faites tourner brièvement les tubes dans une microcentrifugeuse pour vous assurer que tout le liquide est recueilli au fond des tubes, puis incubez à environ 25 °C pendant 15 min.
- Ajouter des complexes de transfection goutte à goutte aux cellules et incuber à 37 °C/10 % de CO2 pendant 16 à 20 h pour permettre l’expression des protéines marquées Halo et Nano.
REMARQUE : L’ajout d’un vecteur vide (pCDNA3.1) est essentiel pour obtenir une efficacité de transfection élevée des constructions d’expression de N-BRET. L’absence d’ajout de vecteur vide entraîne une réduction des niveaux d’expression des protéines 14-3-3ζ-Halo et Nano-CRAF et, à son tour, entraîne des rapports BRET faibles et incohérents, comme nous l’avons vu précédemment22.
3. Replacage cellulaire (jour 3)
REMARQUE : Dans cette étape, les cellules sont transférées sur une plaque de 384 puits et soit un ligand Halo 618 (+ligand ; Table des matériaux) ou DMSO (+véhicule) est ajouté pour la lecture des émissions BRET à l’étape 4. Les cellules restantes sont transférées sur des plaques de culture fraîches à 6 puits pour l’analyse par transfert Western à l’étape 5 (figure 2).
- Rassemblez les matériaux suivants et préparez la zone de travail comme décrit ci-dessous.
- À l’aide d’un bain-marie à 37 °C, préchauffer la trypsine-EDTA, avec des milieux de dosage sans sérum et des milieux de dosage supplémentés à 10 % de FBS (voir la table des matériaux pour plus de détails).
- En fonction du nombre d’échantillons à mesurer, pré-étiqueter trois ensembles de tubes stériles de 1,7 mL (ensembles 1 à 3) et un ensemble de tubes stériles à fond conique de 15 mL. Équipez une centrifugeuse à godet oscillant d’inserts de tube de 15 ml et pré-refroidissez à 4 °C.
- Placez les articles suivants dans la hotte de culture tissulaire : réservoirs de réactifs, plaques de culture tissulaire à 384 puits, plaques de culture tissulaire à 6 puits, pipette multicanaux et pointes, lames/chambres de comptage cellulaire et coloration au bleu trypan (table des matériaux), ainsi que les ensembles de tubes 1 à 3 de 1,7 ml.
- Récoltez et comptez les cellules comme décrit ci-dessous.
- Aspirez le milieu des plaques à 6 puits et ajoutez 250 μL de trypsine-EDTA aux cellules. Incuber les plaques à 6 puits à 37 °C jusqu’à ce que les cellules commencent à se détacher (3-5 min).
- Ajouter 1 mL de milieu de dosage complété à 10 % de FBS dans chaque puits pour neutraliser la trypsine et pipeter vigoureusement de haut en bas pour générer une suspension unicellulaire.
- Transférez 1 mL de suspension cellulaire dans les tubes de 15 mL pré-étiquetés. Ajouter 1 mL de milieu de dosage complété à 10 % de FBS dans chacun des tubes de 15 mL.
- Retournez les tubes 5 fois pour mélanger et transférez immédiatement 20 μL de suspension cellulaire dans le tube 1.
- Centrifugeuse dans la centrifugeuse à godet pivotant pré-refroidie pendant 5 min à ~250 x g. Au cours de l’étape de centrifugation, mélangez 20 μL de colorant cellulaire au bleu trypan avec les cellules du tube 1, puis comptez les cellules à l’aide d’un compteur de cellules automatisé ou d’un hémocytomètre. Un rendement de 6-8 x 105 cellules/mL est typique.
- Retirez les tubes de 15 mL de la centrifugeuse et aspirez le média des pastilles de cellule. Remettre les pastilles cellulaires en suspension à 2 x 105 cellules/mL dans un milieu de dosage sans sérum et pipeter vigoureusement pour générer une suspension unicellulaire.
REMARQUE : L’utilisation de milieux de test sans sérum est utilisée pour mettre au repos les voies de signalisation cellulaire normales.
- Replaquez les cellules en plaques à 384 puits et à 6 puits comme décrit ci-dessous.
- Retournez les tubes de 15 mL plusieurs fois pour assurer une suspension cellulaire homogène et transférez les tubes de 500 μL dans les ensembles 2 et 3 de 1,7 mL.
- Ajouter 0,5 μL de DMSO (+véhicule) à la série 2 et 0,5 μL de ligand Halo 618 (+ligand) à la sonde 3 et à la pipette pour mélanger.
- Transférez les suspensions cellulaires +véhicule et +ligand dans des puits séparés des réservoirs de réactifs. À l’aide de la pipette multicanaux (Table des matériaux), transférez 40 μL de la suspension cellulaire +véhicule des réservoirs de réactifs vers les puits quadruplés de la plaque de culture à 384 puits. Répétez cette étape pour les suspensions de cellules +ligand.
- Transférez les cellules restantes dans des plaques de culture fraîches à 6 puits. Incuber les plaques de 384 puits et de 6 puits pendant la nuit à 37 °C et 5 % de CO2.
4. Lecture des émissions de BRET (jour 4)
REMARQUE : Dans cette étape, le substrat de nanoluciférase (voir le tableau des matériaux pour plus de détails) est ajouté aux cellules de la plaque de culture à 384 puits et les émissions de l’accepteur N-BRET (618 nm) et du donneur (460 nm) sont lues (Figure 2). Les rapports BRET corrigés sont ensuite calculés.
- Préchauffer le milieu de dosage sans sérum dans un bain d’eau à 37 °C et décongeler le substrat de nanoluciférase à 25 °C. Diluer le substrat de nanoluciférase 1:100 dans un milieu de dosage sans sérum et transférer dans un réservoir de réactifs. Préparez suffisamment de ce mélange pour ajouter 10 μL dans chaque puits de la plaque de 384 puits, plus 10 à 15 % de volume supplémentaire.
- À l’aide d’une pipette multicanaux (Table of Materials), transférez 10 μL du mélange de substrat dans chacun des puits contenant des cellules dans la plaque de culture à 384 puits. Faites pivoter doucement la plaque pendant 1 min, manuellement ou à l’aide d’un agitateur orbital.
- Insérez la plaque à 384 puits dans le lecteur de plaques multimode et enregistrez les émissions de 460 nm et 618 nm pour tous les puits contenant des cellules.
REMARQUE : Pour le lecteur de plaques multimode utilisé pour cette étape (voir le tableau des matériaux pour plus de détails), une hauteur de lecture de 6,5 mm, avec une mesure de temps de lecture de 0,1 s a été utilisée, mais une optimisation supplémentaire peut être nécessaire si vous utilisez d’autres lecteurs de plaques. - Calculez individuellement les rapports BRET bruts pour +véhicule et +ligand en unités milliBRET (mBU), à l’aide de l’équation suivante :
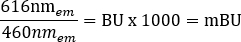
- Calculer le rapport BRET corrigé en soustrayant le rapport BRET +véhicule de celui du +ligand (tableau supplémentaire 1).
REMARQUE : Lorsque l’on compare les résultats de plusieurs expériences, les rapports BRET corrigés peuvent être regroupés et normalisés à la moyenne de la paire N-BRET de type sauvage (WT), composée de Nano-CRAFWT et de 14-3-3ζWT-Halo (tableau supplémentaire 1).
5. Confirmation des niveaux d’expression des protéines (jours 4 et 5)
REMARQUE : Dans cette étape, les cellules des plaques à 6 puits sont lysées et les niveaux d’expression protéique des protéines Nano-CRAF et 14-3-3ζ-Halo sont déterminés par analyse par western blot à l’aide d’anticorps spécifiques aux marqueurs Halo et Nano. (Figure 2).
- Ajouter des inhibiteurs de protéase et de phosphatase au tampon de lyse NP40 (tableau des matériaux), en autorisant 200 μL de tampon de lyse par échantillon.
- Aspirez le milieu à partir de plaques à 6 puits contenant des cellules. Lavez les cellules une fois avec 1 ml de PBS froid et aspirez.
- Ajouter 125 μL de tampon de lyse NP40 dans chaque puits et incuber des plaques de 6 puits à 4 °C sur une plate-forme à bascule pendant 15 min pour lyser les cellules.
- Transférez les lysats dans des tubes de 1,7 mL sur de la glace et centrifugez-les à 20 000 x g pendant 10 min à 4 °C. Remettez les lysats éliminés sur de la glace et déterminez la concentration en protéines à l’aide de dosages disponibles dans le commerce, tels que les dosages de Bradford ou d’acide bicchoninique (BCA).
- Normaliser la concentration en protéines dans tous les échantillons en transférant un volume approprié de lysat et de tampon de lyse NP40 dans des tubes frais de 1,7 mL jusqu’à un volume total de 100 μL.
- Faites bouillir 5 tampons d’échantillon de protéines (table des matériaux) pendant 1 min et ajoutez 25 μL à chaque échantillon. Faites bouillir les échantillons pendant 5 à 6 minutes, puis centrifugez brièvement les tubes par impulsions pour vous assurer que tout l’échantillon s’accumule à la base du tube.
- Chargez 25 μL d’échantillon dans chaque puits de gels de protéines dupliqués (gel 1 et gel 2), puis transférez les protéines sur des membranes de nitrocellulose ou de PVDF à l’aide des procédures standard de transfert Western, comme décrit précédemment22.
- Bloquer les membranes dans 3 % de BSA-PBS à 25 °C pendant 30 minutes, puis incuber les membranes pendant la nuit avec l’anticorps Halo (dilution 1:1 000 ; Gel 1) et soit de la nanoluciférase, soit de l’anticorps CRAF (dilution 1:500 ; Gel 2) dans une solution saline tris-tamponnée, complété par 0,2 % de Tween-20 (TBST ; Table des matériaux).
- Laver les membranes 1x pendant 5 min dans 10 mL de TBST, puis incuber à température ambiante pendant 1 h avec un anticorps secondaire HRP anti-souris, dilué 1:10 000 dans du TBST.
- Laver les membranes 3 fois sur une plate-forme basculante dans 10 mL de TBST à 25 °C pendant 5 min chacune. Retirez le TBST et visualisez les bandes protéiques à l’aide de réactifs ECL et d’un processeur de film à rayons X ou d’un autre système d’imagerie approprié.
Résultats
Lorsqu’elle est effectuée comme décrit dans ce protocole (Figure 2), l’interaction de Nano-CRAFWT et de 14-3-3ζ-Halo devrait produire des rapports BRET corrigés de 50-60 mBU (Figure 3A ; Tableau supplémentaire 1). CRAF contient deux sites d’amarrage 14-3-3 dépendants de la phosphorylation, le site N’et le site C'(Figure 1)8. Par conséquent, les contrôles appropriés pour réduire la liaison CRAF :14-3-3ζ comprennent S259A et S621A, qui empêchent la phosphorylation, et à son tour la liaison 14-3-3, aux sites N’et C', respectivement25. Comme le montre la figure 3A, le Nano-CRAFS259A réduit le signal BRET de ~45% et le Nano-CRAFS621A de ~25%, tandis que le double mutant S259A/S621A (SS/AA) ablate presque entièrement le signal BRET CRAF :14-3-3. De plus, les résultats de plusieurs expériences indépendantes peuvent être comparés en normalisant d’abord les résultats de chaque expérience au rapport BRET corrigé moyen (tableau supplémentaire 1), puis en regroupant les points de données des trois expériences (figure 3B). Les niveaux d’expression relatifs des protéines marquées doivent également être confirmés par l’analyse par transfert Western (figure 3). Notez que les niveaux d’expression des protéines mutantes peuvent différer de ceux de WT, ce qui nécessite que les quantités transfectées de vecteurs d’ADN mutants soient ajustées en conséquence (tableau supplémentaire 1).
Il convient également de noter que l’optimisation de ce protocole peut être nécessaire, en particulier lors de l’utilisation d’un autre lecteur de plaques, de lignées cellulaires ou de réactifs de transfection. La figure 4A montre les résultats d’un exemple d’expérience d’optimisation, où l’augmentation de la quantité transfectée du plasmide accepteur pCMV5-14-3-3ζ-Halo, tout en maintenant constante la quantité de donneur pCMV5-NanoLuc-CRAF, augmente dépendante de la dose le signal BRET, qui est ensuite réduit par la présence du mutant CRAFSS/AA déficient en 14-3-3 liaison. Cependant, si le rapport accepteur/donneur est trop élevé, la différence de signal entre la liaison du 14-3-3ζ-Halo au Nano-CRAFWT et le contrôle négatif du Nano-CRAFSS/AA sera réduite, probablement en raison de la saturation de la liaison du Nano-CRAFWT. De plus, une fois qu’un rapport BRET qui génère des valeurs reproductibles avec une faible erreur-type est atteint, il n’est pas nécessaire d’augmenter davantage le rapport accepteur/donneur. En effet, l’un des avantages majeurs de ces tests est la capacité de mesurer les interactions des protéines aux niveaux d’expression physiologique. Lors de la mise au point de nouveaux tests N-BRET, une autre considération importante est le placement des étiquettes. La figure 4B montre l’effet de la commutation de l’étiquette Halo de l’extrémité C-terminale à l’extrémité N-terminale de 14-3-3ζ sur les rapports BRET corrigés générés par la liaison de 14-3-3ζ à Nano-CRAFWT et Nano-CRAFSS/AA. À cette fin, il faut générer des constructions d’expression de l’ADN pour chacun des emplacements possibles des marqueurs (figure 4C), et évaluer leurs effets sur les rapports BRET de la paire WT et sur celui d’un témoin négatif connu pour la liaison.

Figure 1 : Régulation de la RAF par les protéines 14-3-3. (A) Schéma de la structure du domaine kinase RAF avec les sites d’amarrage 14-3-3 conservés indiqués. (B) Modèles de régulation RAF par les protéines 14-3-3. (C) Test N-BRET mesurant les interactions de 14-3-3ζ fusionné à un accepteur de marqueurs C-terminal Halo et CRAF fusionné à un donneur N-terminal Nano. Le transfert d’énergie du donneur à l’accepteur génère le signal BRET. Veuillez cliquer ici pour voir une version agrandie de cette figure.

Figure 2 : Étapes clés de la réalisation du test CRAF :14-3-3 basé sur le BRET sur cellules vivantes. Les cinq étapes principales décrites dans ce protocole sont illustrées, notamment l’ensemencement, la transfection et le replacage des cellules, ainsi que les méthodes de lecture du signal BRET et de confirmation des niveaux d’expression des protéines marquées Halo et Nano. Veuillez cliquer ici pour voir une version agrandie de cette figure.

Figure 3 : Essai CRAF :14-3-3 basé sur BRET. Test N-BRET comparant les interactions de 14-3-3ζ-Halo avec Nano-CRAF -WT, ou les formes mutantes -S259A, -S621A et -S259A/S621A (SS/AA), dont il a été démontré précédemment qu’elles altèrent la liaison CRAF :14-3-325. Les niveaux d’expression du 14-3-3ζ-Halo (MW : 64 kDa) et des variants Nano-CRAF (MW : 93 kDa) ont été examinés par western blot. (A) Lectures BRET corrigées de quatre puits répétés à partir d’une seule expérience ± écart-type (ET). (B) Les lectures BRET corrigées de trois expériences indépendantes ont été regroupées et normalisées à Nano-CRAFWT (fixé à 100) ± SD. La signification statistique a été calculée par le test t de Student (bilatéral, en supposant une variance égale). p<0.0001. Veuillez cliquer ici pour voir une version agrandie de cette figure.
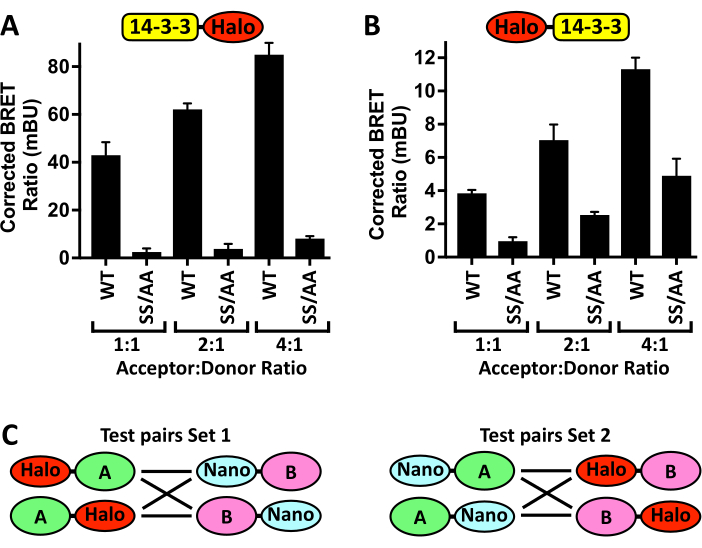
Figure 4 : Étapes d’optimisation du dosage. Les effets de la co-transfection de quantités croissantes de (A) 14-3-3ζ-Halo ou (B) Halo-14-3-3ζ avec une quantité constante de Nano-CRAF -WT ou -SS/AA (S259A/S621A) sur le signal BRET ± SD. (C) Schéma de toutes les paires N-BRET possibles générées en plaçant l’étiquette Halo ou Nano sur l’extrémité N-terminale ou C-terminale de chaque protéine d’intérêt (A et B). n = 4. Veuillez cliquer ici pour voir une version agrandie de cette figure.
Tableau supplémentaire 1 : Exemples de données tirées du test CRAF :14-3-3 basé sur BRET. Les constructions d’expression indiquées ont été transfectées dans les cellules le jour 2 (section supérieure). Les lectures brutes, corrigées et normalisées de BRET ont été mesurées et calculées le jour 4 (section inférieure). Veuillez cliquer ici pour télécharger ce fichier.
Discussion
Des études antérieures ont montré que les protéines 14-3-3 jouent un rôle essentiel dans l’activation et l’inhibition des kinases RAF. Comprendre comment ces événements de liaison sont régulés et les effets de la modulation de ces interactions sur la signalisation RAF et l’oncogenèse induite par RAF pourrait révéler de nouvelles vulnérabilités thérapeutiques qui ciblent la fonction CRAF. Cependant, le cycle d’activation de Raf est soutenu par une pléthore de protéines associées, de modifications post-traductionnelles et de changements dans la localisation subcellulaire8, et donc pour récapituler pleinement ces conditions, les interactions de RAF avec ses régulateurs doivent être mesurées dans des conditions de cellules vivantes. Ce protocole décrit un nouveau test basé sur la proximité qui utilise le rapport signal/bruit supérieur et la résolution spectrale de la paire nanoluciférase-Halo N-BRET pour mesurer les interactions de 14-3-3ζ et CRAF dans les cellules vivantes.
Bien que ce test fournisse une plate-forme indispensable pour étudier les interactions complexes du 14-3-3 et de la CRAF dans des conditions physiologiquement pertinentes, il présente un certain nombre de limites qui doivent être prises en compte. Tout d’abord, étant donné qu’il a été démontré que le 14-3-3 joue un rôle à la fois dans l’auto-inhibition et la dimérisation8 de la RAF, des contrôles supplémentaires et des approches expérimentales peuvent être incorporés pour distinguer ces états. Par exemple, ce test peut être effectué en conjonction avec le test d’auto-inhibition N-BRET précédemment développé, qui fournit une lecture directe des changements dans l’auto-inhibition RAF12,22. De plus, des contrôles tels que la mutation CRAF R401H, dont il a été démontré qu’elle perturbe la dimérisation de CRAF avec d’autres protomères de la RAF15,26, peuvent également s’avérer utiles pour disséquer ces fonctions. Enfin, le placement des marqueurs Halo et Nano est optimisé pour mesurer l’interaction CRAF :14-3-3, mais ne peut pas garantir que les interactions de CRAF ou 14-3-3 avec d’autres protéines ne sont pas affectées en conséquence, ni que ce test ne peut déterminer l’affinité de la liaison CRAF :14-3-3, les effets de ces interactions sur l’activité CRAF ou l’emplacement dans la cellule où ces interactions se produisent. Par conséquent, il peut également être nécessaire d’incorporer d’autres approches complémentaires, telles que les tests de liaison in vitro, les tests de kinase CRAF et les études de localisation subcellulaire 12,27,28,29.
Lors de la réalisation de ce test, ou de tout test basé sur N-BRET, il y a un certain nombre de facteurs à prendre en compte. Comme le montrent ce manuscrit, et dans des travaux antérieurs22, le rapport de transfection correct des constructions pCMV5-Halo (accepteur) et pCMV5-Nanoluciférase (donneur), ainsi qu’un contrôle négatif approprié, tel qu’un mutant connu pour perturber la liaison des POI, est essentiel pour optimiser et tester ces tests. L’augmentation de l’expression du POI marqué Halo, tout en maintenant les niveaux de POI marqués Nano constants, entraîne une augmentation des ratios BRET. Cependant, une fois que la liaison est saturée, le signal se stabilise et, à son tour, le delta entre le WT et les POI altérés par la liaison est réduit, ce qui nécessite une optimisation accepteur-donneur. Un autre facteur critique dans l’optimisation du test est l’utilisation d’un ADN vecteur vide, tel que pCDNA3.1. L’inclusion de 200 ng de pCDNA3.1. dans le mélange de transfection augmente considérablement les niveaux d’expression des POI marqués Halo et Nano et réduit la variabilité entre les puits répliqués et les expériences22. Cependant, étant donné que l’efficacité de la transfection peut varier entre les réactifs et les lignées cellulaires, il peut également être nécessaire de titrer la quantité d’ADN porteur pour obtenir une expression optimale de la paire N-BRET, comme décrit précédemment22. Un autre facteur qui affecte grandement les ratios BRET est le placement des balises. En effet, quel POI est fusionné à l’étiquette Halo par rapport à l’étiquette Nano et si cette étiquette est placée sur l’extrémité N-ou C-terminale de la protéine peut affecter à la fois l’amplitude du rapport BRET et la capacité des contrôles négatifs à réduire ce signal. Par conséquent, il est également important de tester chacune des paires N-BRET possibles lors du développement de nouveaux tests.
L’amélioration de la sensibilité et de la résolution spectrale obtenues par N-BRET par rapport aux BRET conventionnels permet des interactions plus faibles et des changements subtils de liaison à mesurer dans des cellules vivantes. Cependant, l’utilité de ces tests pour l’étude des événements de signalisation cellulaire n’a pas été pleinement réalisée. Notamment, ce test pourrait probablement être adapté pour étudier l’interaction de 14-3-3 avec d’autres kinases, telles que les autres kinases RAF ARAF et BRAF, et lorsqu’il est combiné avec des tests N-BRET précédemment publiés pour étudier la liaison RAS :RAF et l’auto-inhibitionRAF 22,30, il formera la base d’une boîte à outils pour mesurer la régulation RAF dans des conditions de cellules vivantes. De plus, N-BRET s’est avéré prometteur en tant que plateforme de criblage de médicaments à cellulesvivantes 30, et par conséquent, ce test peut également être utile pour identifier des composés qui stabilisent l’inhibiteur avec des interactions de 14-3-3 et RAF ou perturbent ceux impliqués dans l’activation de RAF. Compte tenu de la grande stabilité du signal de la nanoluciférase et de la capacité des lecteurs de plaques modernes à détecter simultanément les émissions accepteurs et donneurs, il s’ensuivrait également que ces tests peuvent être adaptés pour mesurer les interactions des protéines de signalisation en temps réel, ce qui sera essentiel pour comprendre la dynamique de ces interactions en réponse à une stimulation en amont ou à des traitements médicamenteux.
Déclarations de divulgation
Rien à divulguer.
Remerciements
Ce projet a été financé en partie par des fonds fédéraux du National Cancer Institute, National Institutes of Health, sous le numéro de projet ZIA BC 010329.
matériels
| Name | Company | Catalog Number | Comments |
| Antibodies | |||
| HaloTag® mouse monoclonal antibody | Promega | G9211 | Antibody for detecting HaloTag tagged proteins by immunoblot |
| NanoLuc® mouse monoclonal antibody | R&D Systems | MAB10026 | Antibody for detecting Nano-tagged proteins by immunoblot |
| CRAF mouse monoclonal antibody (E10) | Santa Crus Biotechnology | sc-7267 | Antibody directly detecting CRAF proteins by immunoblot |
| ECL anti-mouse HRP secondary antibody | Amersham | NA931-1ML | Secondary HRP conjugated mouse antibody (from sheep) |
| Reagents | |||
| X-tremeGENE™ 9 | Roche/Sigma | 6365809001 | |
| NanoBRET™ kit | Promega | N1661 | NanoBRET kit containing Halo 618 ligand and NanoGlo (nanoluciferase) substrate |
| DPBS, without Ca++ and Mg++ | Quality Biologicals | 114-057-101 | |
| Trypsin-EDTA (0.05%), phenol red | Life Technologies | 25300120 | |
| DMEM cell culture media | Life Technologies | 11995073 | High glucose, L-glutamine, phenol red, sodium pyruvate; without HEPES, suppliment media with 10% FBS, 2 mM L-glutamine and 100U penicillin-streptomycin |
| L-Glutamine (200 mM) | Life Technologies | 25030164 | |
| Penicillin-Streptomycin (10,000 U/mL) | Life Technologies | 15140163 | |
| Opti-MEM™ I reduced serum media | Gibco | 31985062 | For cell transfection |
| Opti-MEM reduced serum media, no phenol red | Gibco | 11058021 | For replating cells on Day 3. Supplement with 2 mM L-glutamine and 100U penicillin-streptomycin, along with 10% FBS (where indicated). |
| Invitrogen Trypan Blue Stain | Thermo Scientific | T10282 | |
| NP40 lysis buffer | N/A | N/A | 20 mM Tris (pH 8.0), 137mM NaCl, 10% glycerol, NP40 alternative (Milipore, Cat# 492016). Store at 4 degrees C.. Add the following protease and phosphatase immediately prior to use: 20 µM leupeptin, 0.5 mM sodium orthovanidate, 0.15 U/mL, 1mM PMSF. |
| 5x gel sample buffer | N/A | N/A | 240 mM Tris (pH 8.0), 9.5% SDS, 30% glycerol, 500mM DTT, 3mM bromophenol blue. Store at -20 degrees C. |
| Cell lines | |||
| 293FT cells (human) | Thermo Scientific | R70007 | |
| DNA vectors | |||
| pCMV5-Nano-CRAF WT and mutant | N/A | N/A | |
| pCMV5-14-3-3ζ-Halo | N/A | N/A | |
| Equipment | |||
| EnVision 2104 Multimode Plate Reader | PerkinElmer 2104 | 2104-0010 | 600LP NanoBRET & M460/50 nm NanoBRET emmisions filters, Luminescence 404 mirror, 6.5 mm measurement height and 0.1 s measurement time |
| Invitrogen Countess™ II Automated Cell Counter | Thermo Scientific | AMQAX1000 | |
| ThermoFisher E1-ClipTip™ Multichannel Pipettor | Thermo Scientific | 4672070 | |
| Software | |||
| GraphPad Prism (version 10.0.3) | GraphPad | www.graphpad.com | |
| Other | |||
| ThermoFisher ClipTip Multichannel pipette tips | Thermo Scientific | 94410153 | |
| Reagent Reservoir, 25 mL Divided, Sterile | Thomas Scientific | 1228K16 | |
| Perkin Elmer 384-well CulturPlate™ | PerkinElmer | 6007680 | White, polystyrene, tissue culture treated |
| Countess Cell Counting Chamber Slides | Thermo Scientific | C10228 |
Références
- Blasco, R. B., et al. c-Raf, but not B-Raf, is essential for development of K-Ras oncogene-driven non-small cell lung carcinoma. Cancer Cell. 19, 652-663 (2011).
- Blasco, M. T., et al. Complete regression of advanced pancreatic ductal adenocarcinomas upon combined inhibition of EGFR and C-RAF. Cancer Cell. 35, 573-587 (2019).
- Karreth, F. A., Frese, K. K., DeNicola, G. M., Baccarini, M., Tuveson, D. A. C-Raf is required for the initiation of lung cancer by K-Ras(G12D). Cancer Discov. 1, 128-136 (2011).
- Lito, P., et al. Disruption of CRAF-mediated MEK activation is required for effective MEK inhibition in KRAS mutant tumors. Cancer Cell. 25, 697-710 (2014).
- Sanclemente, M., et al. c-RAF ablation induces regression of advanced Kras/Trp53 mutant lung adenocarcinomas by a mechanism independent of MAPK signaling. Cancer Cell. 33, 217-228 (2018).
- Razzaque, M. A., et al. Germline gain-of-function mutations in RAF1 cause Noonan syndrome. Nat Genet. 39, 1013-1017 (2007).
- Pandit, B., et al. Gain-of-function RAF1 mutations cause Noonan and LEOPARD syndromes with hypertrophic cardiomyopathy. Nat Genet. 39, 1007-1012 (2007).
- Terrell, E. M., Morrison, D. K. Ras-mediated activation of the Raf family kinases. Cold Spring Harb Perspect Med. 9 (1), 033746(2019).
- Kondo, Y., et al. Cryo-EM structure of a dimeric B-Raf:14-3-3 complex reveals asymmetry in the active sites of B-Raf kinases. Science. 366, 109-115 (2019).
- Park, E., et al. Architecture of autoinhibited and active BRAF-MEK1-14-3-3 complexes. Nature. 575 (7783), 545-550 (2019).
- Tzivion, G., Luo, Z., Avruch, J. A dimeric 14-3-3 protein is an essential cofactor for Raf kinase activity. Nature. 394, 88-92 (1998).
- Spencer-Smith, R., et al. RASopathy mutations provide functional insight into the BRAF cysteine-rich domain and reveal the importance of autoinhibition in BRAF regulation. Mol Cell. 82, 4262-4276 (2022).
- Martinez Fiesco, J. A., Durrant, D. E., Morrison, D. K., Zhang, P. Structural insights into the BRAF monomer-to-dimer transition mediated by RAS binding. Nat Commun. 13, 486(2022).
- Freeman, A. K., Ritt, D. A., Morrison, D. K. The importance of Raf dimerization in cell signaling. Small GTPases. 4, 180-185 (2013).
- Freeman, A. K., Ritt, D. A., Morrison, D. K. Effects of Raf dimerization and its inhibition on normal and disease-associated Raf signaling. Mol Cell. 49, 751-758 (2013).
- Rushworth, L. K., Hindley, A. D., O'Neill, E., Kolch, W. Regulation and role of Raf-1/B-Raf heterodimerization. Mol Cell Biol. 26, 2262-2272 (2006).
- Garnett, M. J., Rana, S., Paterson, H., Barford, D., Marais, R. Wild-type and mutant B-RAF activate C-RAF through distinct mechanisms involving heterodimerization. Mol Cell. 20, 963-969 (2005).
- Tran, N. H., Wu, X., Frost, J. A. B-Raf and Raf-1 are regulated by distinct autoregulatory mechanisms. J Biol Chem. 280, 16244-16253 (2005).
- Chong, H., Guan, K. L. Regulation of Raf through phosphorylation and N terminus-C terminus interaction. J Biol Chem. 278, 36269-36276 (2003).
- Cutler, R. E., Stephens, R. M., Saracino, M. R., Morrison, D. K. Autoregulation of the Raf-1 serine/threonine kinase. Proc Natl Acad Sci U S A. 95, 9214-9219 (1998).
- Park, E., et al. Cryo-EM structure of a RAS/RAF recruitment complex. Nat Commun. 14, 4580(2023).
- Spencer-Smith, R., Morrison, D. K. Protocol for measuring BRAF autoinhibition in live cells using a proximity-based NanoBRET assay. STAR Protoc. 4, 102461(2023).
- Clark, G. J., et al. 14-3-3 zeta negatively regulates raf-1 activity by interactions with the Raf-1 cysteine-rich domain. J Biol Chem. 272, 20990-20993 (1997).
- Machleidt, T., et al. NanoBRET--A novel BRET platform for the analysis of protein-protein interactions. ACS Chem Biol. 10, 1797-1804 (2015).
- Hekman, M., et al. Dynamic changes in C-Raf phosphorylation and 14-3-3 protein binding in response to growth factor stimulation: differential roles of 14-3-3 protein binding sites. J Biol Chem. 279, 14074-14086 (2004).
- Hatzivassiliou, G., et al. RAF inhibitors prime wild-type RAF to activate the MAPK pathway and enhance growth. Nature. 464, 431-435 (2010).
- Bondzi, C., Grant, S., Krystal, G. W. A novel assay for the measurement of Raf-1 kinase activity. Oncogene. 19, 5030-5033 (2000).
- Spencer-Smith, R., et al. Inhibition of RAS function through targeting an allosteric regulatory site. Nat Chem Biol. 13 (1), 62-68 (2016).
- Roy, S., et al. 14-3-3 facilitates Ras-dependent Raf-1 activation in vitro and in vivo. Mol Cell Biol. 18, 3947-3955 (1998).
- Durrant, D. E., et al. Development of a high-throughput NanoBRET screening platform to identify modulators of the RAS/RAF interaction. Mol Cancer Ther. 20, 1743-1754 (2021).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon