Method Article
生細胞におけるCRAFと14-3-3タンパク質との相互作用を測定するための生物発光共鳴エネルギー移動(BRET)ベースのアッセイ
要約
このプロトコルは、生細胞中のCRAFキナーゼと14-3-3タンパク質との相互作用を測定するためのBRETベースのアッセイを説明しています。このプロトコルでは、細胞の調製、BRET排出量の読み取り、およびデータ解析の手順を概説しています。また、アッセイ最適化のための適切なコントロールとトラブルシューティングの特定を含む結果の例も示します。
要約
CRAF は RAS GTPase の主要なエフェクターであり、いくつかの KRAS によるがんの腫瘍形成に重要な役割を果たします。さらに、CRAF は生殖細胞変異のホットスポットであり、発達性 RASopathy、ヌーナン症候群を引き起こすことが示されています。すべてのRAFキナーゼには、14-3-3制御タンパク質のリン酸化依存性結合部位が複数含まれています。これらの部位への14-3-3の差次的結合は、シグナル伝達条件下での原形質膜での活性RAF二量体の形成、および静止条件下でのRAF自己阻害の維持において重要な役割を果たします。これらの相互作用がどのように制御され、どのように調節できるかを理解することは、RAF機能を標的とする新しい治療アプローチを特定するために重要です。ここでは、生細胞におけるCRAFと14-3-3タンパク質との相互作用を測定するための生物発光共鳴エネルギー移動(BRET)ベースのアッセイについて説明します。具体的には、このアッセイは、ナノルシフェラーゼドナーに融合したCRAFとHaloタグアクセプターに融合した14-3-3の相互作用を測定し、RAFと14-3-3の相互作用により、ドナーからアクセプターへのエネルギー移動とBRETシグナルの生成がもたらされます。プロトコルはさらに、このシグナルが、その高親和性RAFドッキング部位のそれぞれへの14-3-3結合を防止することを示す突然変異によって中断され得ることを示している。このプロトコールでは、細胞の播種、トランスフェクション、および再プレーティングの手順と、BRET排出量の読み取り、データ解析の実施、およびタンパク質発現レベルの確認に関する詳細な手順について説明します。さらに、アッセイ結果の例と、最適化およびトラブルシューティングの手順を提供します。
概要
RAFキナーゼ(ARAF、BRAF、CRAF)は、RAS GTPアーゼの直接エフェクターであり、増殖促進型/生存促進型のRAF-MEK-ERKキナーゼカスケードの開始メンバーです。最近の研究では、CRAF の発現が、非小細胞肺がんや膵管腺がん 1,2,3,4,5 など、いくつかの KRAS 駆動型がんの腫瘍形成に重要な役割を果たしていることが示されています。さらに、生殖細胞系CRAF変異は、特に重篤なRASopathyであるヌーナン症候群を引き起こします6,7。CRAF制御を理解することは、細胞内でのCRAF機能を標的とする治療アプローチを成功させるために重要です。
全てのRAFキナーゼは、C末端触媒(CAT)ドメインとN末端調節(REG)ドメインの2つの機能ドメインに分けることができ、これはその活性を制御する(図1A)8。REGドメインには、RAS結合ドメイン(RBD)、システインリッチドメイン(CRD)、およびセリン/スレオニンリッチ領域(S/Tリッチ)が含まれます。特に、S/Tリッチ領域にはN'部位が含まれており、N'部位はリン酸化依存的に14-3-3に結合します(CRAFのS259;図1A)8. CATドメインは、キナーゼドメインを包含し、C'サイトと呼ばれる第2の高親和性14-3-3ドッキングサイト(CRAFではS621;図1A)8.二量体14-3-3タンパク質のN'およびC'部位への差長結合は、CRDとともに、RAFの活性化と阻害の両方に重要な役割を果たします9,10,11,12,13。通常のシグナル伝達条件下では、RAFの活性化はRASによる原形質膜への動員によって開始され、活性二量体を形成することができ、その中でBRAF-CRAFヘテロ二量体が主要な活性型である14,15。BRAFおよびCRAFを用いた生化学的アッセイは、二量体BRAFの極低温電子顕微鏡(Cryo-EM)構造とともに、14-3-3二量体が両方のRAFプロトマーのC'部位に同時に結合することにより活性RAF二量体を安定化させることを示している(図1B)9,13,16,17。逆に、研究は、静止条件下で、RAFが細胞質の自己阻害確認を採用し、REGドメインがCATドメインに結合し、その活性を阻害することを示しました12,18,19,20。この閉状態は、REGドメインのCRDおよびN'サイト、およびCATドメインのC'サイトに結合した14-3-3ダイマーによって安定化される(図1B)10,13,21。BRAFでは、このモデルは、自己阻害BRAFモノマーの最近のクライオ電子顕微鏡構造と、以前の生化学的研究10,12,13,21,22によってサポートされています。しかし、14-3-3はCRAF調節において阻害的な役割を果たすことが示されています23が、BRAFのような自己抑制状態はCRAF調節12においてより少ない役割を果たす可能性があります。したがって、14-3-3タンパク質がCRAF活性を制御するメカニズムを明らかにするためには、さらなる研究が必要です。14-3-3を介したRAFキナーゼの調節には、多数のRAFリン酸化および脱リン酸化イベント、さまざまな調節タンパク質への結合、および原形質膜との相互作用が必要です8。したがって、14-3-3-RAF相互作用は、生理学的に関連性のある条件下で、無傷の脂質二重層の存在下で測定することが重要です。
この問題に対処するために、NanoBRET(以下、N-BRETと表記、キットの詳細については表を参照)技術を利用して、生細胞内のCRAFと14-3-3タンパク質との相互作用を測定するための近接ベースのアッセイを開発しました(図1C)。このBRETベースのシステムは、Halo618エネルギーアクセプターリガンド22,24で標識するために、一方のタンパク質にナノルシフェラーゼ(Nano)ドナー、もう一方にHaloタグが付けられた2つの目的タンパク質(POI)の相互作用を測定する。目的のタンパク質の相互作用により、ドナーからアクセプターへのエネルギー移動が起こり、それが次にBRETシグナルを生成します(図1C)。非常に明るいNanoドナータンパク質(発光(em)460 nm)とHalo618リガンド(em 618 nm)は、従来のBRETよりも優れたスペクトル分離と感度を提供し、弱い相互作用を研究し、結合の微妙な変化を検出するための理想的なプラットフォームとなっています24。実際、我々は以前に、BRAF CRDにおけるRASopathy変異のパネルの特性評価に不可欠であったRAF REGおよびCATドメインの自己抑制性相互作用を測定するためのN-BRETベースのアッセイを開発し、自己抑制を維持し、構成的BRAF活性化を防止するためのこのドメインの決定的な重要性を実証した12。
ここで説明するアッセイは、N末端ナノタグに融合したCRAFと、C末端ハロータグに融合した14-3-3のゼータアイソフォーム(14-3-3ζ-Halo; 図1C)。Nano-CRAFと14-3-3ζ-Haloとの相互作用は、強力なBRETシグナルを生成し、14-3-3がN'部位(S259A)および/またはC'部位(S621A)に結合するのを妨げる突然変異によって破壊される可能性があることを示しています。次のプロトコルは、このアッセイの実行、最適化、およびトラブルシューティングの詳細な手順を示しています。
プロトコル
注:このアッセイは293FT細胞で行われます。ヒト胚性腎臓細胞に由来する、十分に特徴付けられ、容易にトランスフェクト可能な上皮系統。これらの細胞の1つのコンフルエント10 cm培養皿は、通常、6ウェル組織培養プレートの20ウェルを播種するのに十分な細胞を提供します。ステップ1〜3は、生物学的安全キャビネット内で滅菌技術を使用して実行する必要があります。
1. 細胞播種(1日目)
注:このステップでは、細胞を組織培養皿から切り離し、カウントし、ステップ2のトランスフェクションのために6ウェル組織培養プレートに播種します(図2)。
- 10cm皿の細胞から培地を吸引します。5 mLのリン酸緩衝生理食塩水(PBS)で細胞を洗浄し、吸引します。
- 1 mLのトリプシン-エチレンジアミン四酢酸(EDTA)を加え、37°Cで3〜5分間インキュベートして、細胞を皿から分離します。
- 9 mLの完全ダルベッコ改変イーグル培地(DMEM)を細胞に加えてトリプシンを中和し、ピペットを上下に繰り返して均質な単一細胞懸濁液を生成します。
- 直ちに20 μLの細胞を1.7 mLのチューブに移し、20 μLのトリパンブルー染色液と混合します。自動セルカウンター(Table of Materials)または血球計算盤を使用して細胞をカウントします。
- 完全なDMEM培地で細胞を2 x 105 細胞/mLに希釈し、6ウェル組織培養プレート(4 x 105 細胞/ウェル)の各ウェルに2 mLを添加します。細胞を37°Cおよび10%CO2 で一晩インキュベートします。
注:継代が20倍未満の293FTセルを使用することをお勧めします。私たちは以前、継代数の多い細胞を使用すると、BRET比が低下し、ウェル間のシグナルのばらつきが増加する可能性があることを発見しました。
2. 細胞トランスフェクション(2日目)
注:ここでは、細胞にpCMV5-NanoLuc-CRAFおよびpCMV5-14-3-3ζ-Halo発現コンストラクトと、pCDNA3.1エンプティベクターをトランスフェクションします(図2)。
- トランスフェクションの前に、N-BRETプラスミドを5 ng/μLに、pCDNA3.1を100 ng/μLに希釈し、滅菌済みの1.7 mLチューブのセットに番号を付けます。
- 各チューブに100 μLのトランスフェクション培地(詳細は 「材料表 」を参照)を、5 ngのpCMV5-NanoLuc-CRAF、10 ngのpCMV5-14-3-3ζ、および200 ngのPCDNA3.1とともに加えます。
- 2 μLのトランスフェクション試薬(詳細については 、材料表 を参照)を加え、穏やかにボルテックスして混合します。マイクロ遠心分離機でチューブをパルス回転させて、すべての液体がチューブの底に集まることを確認した後、約25°Cで15分間インキュベートします。
- トランスフェクション複合体を細胞に滴下し、37°C/10%CO2 で16〜20時間インキュベートして、Haloタグ付きタンパク質とNanoタグ付きタンパク質を発現させます。
注:エンプティベクター(pCDNA3.1)キャリアDNAの添加は、N-BRET発現コンストラクトの高いトランスフェクション効率を達成するために不可欠です。エンプティベクターを追加しないと、14-3-3ζ-HaloおよびNano-CRAFタンパク質の発現レベルが低下し、前に説明したように、BRET比が弱く一貫性がなくなります22。
3. 細胞再プレーティング(3日目)
注:このステップでは、細胞を384ウェルプレートに移し、Halo 618リガンド(+リガンド; 資料表)または、ステップ4でBRET排出量を読み取るためにDMSO(+vehicle)が追加されます。残りの細胞を新鮮な6ウェル培養プレートに移し、ステップ5でウェスタンブロット分析を行います(図2)。
- 以下の資料を揃え、以下に説明するように作業エリアを準備してください。
- 37°Cのウォーターバス、予温トリプシン-EDTAを使用し、無血清アッセイ培地および10%FBS添加アッセイ培地の両方を使用します(詳細については 、材料の表 を参照)。
- 測定するサンプルの数に基づいて、滅菌1.7 mLチューブ3セット(セット1-3)と滅菌15 mLコニカルボトムチューブ1セットを事前にラベル付けします。スイングバケット遠心分離機に15 mLチューブインサートを装備し、4°Cに予冷します。
- 組織培養フードには、試薬リザーバー、384ウェル組織培養プレート、6ウェル組織培養プレート、マルチチャンネルピペットとチップ、細胞計数スライド/チャンバー、トリパンブルーステイン(材料表)、1.7 mLチューブセット1〜3を入れます。
- 以下で説明するように、セルを採取してカウントします。
- 6ウェルプレートから培地を吸引し、250 μLのトリプシン-EDTAを細胞に加えます。細胞が剥離し始めるまで、6ウェルプレートを37°Cでインキュベートします(3〜5分)。
- 10% FBSを添加したアッセイ培地1 mLを各ウェルに加え、トリプシンを中和し、激しく上下にピペットで動かしてシングルセル懸濁液を生成します。
- 細胞懸濁液1 mLを、あらかじめ標識した15 mLチューブに移します。さらに 10% FBS 添加アッセイ培地 1 mL を各 15 mL チューブに加えます。
- チューブを5倍反転させて混合し、すぐに20μLの細胞懸濁液をチューブセット1に移します。
- 予冷したスイングバケット遠心分離機で、~250 x gで5分間遠心分離します。遠心分離ステップでは、20 μLのトリパンブルー細胞染色液をチューブセット1の細胞と混合し、自動セルカウンターまたは血球計算盤を使用して細胞をカウントします。収量は6-8 x 105 細胞/mLが一般的です。
- 遠心分離機から15 mLチューブを取り出し、細胞ペレットから培地を吸引します。細胞ペレットを無血清アッセイ培地で2 x 105 細胞/mLに再懸濁し、激しくピペットで動かして単一細胞懸濁液を生成します。
注:無血清アッセイ培地の使用は、正常な細胞シグナル伝達経路を静止するために使用されます。
- 以下に説明するように、384ウェルプレートおよび6ウェルプレートでセルを再播種します。
- 15 mLチューブを数回反転させて細胞懸濁液が均一になるようにし、500 μLを1.7 mLチューブのセット2およびセット3に移します。
- 0.5 μL の DMSO (+ビヒクル) をセット 2 に、0.5 μL の Halo 618 リガンド (+リガンド) をチューブセット 3 に加えて、ピペットで混合します。
- +ビヒクルおよび+リガンド細胞懸濁液を試薬リザーバーの別々のウェルに移します。マルチチャンネルピペット(Table of Materials)を使用して、試薬リザーバーから+ビヒクル細胞懸濁液40 μLを384ウェル培養プレートの四重ウェルに移します。+リガンド細胞懸濁液についてこの手順を繰り返します。
- 残りの細胞を新鮮な6ウェル培養プレートに移します。384ウェルプレートと6ウェルプレートを37°Cおよび5%CO2で一晩インキュベートします。
4. BRET排出量の読み方(4日目)
注:このステップでは、ナノルシフェラーゼ基質(詳細は 「材料の表 」を参照)を384ウェル培養プレートの細胞に添加し、N-BRETアクセプター(618 nm)およびドナー(460 nm)の放出を読み取ります(図2)。次に、修正されたBRET比率が計算されます。
- 無血清アッセイ培地を37°Cのウォーターバスで温め、ナノルシフェラーゼ基質を25°Cで解凍します。 ナノルシフェラーゼ基質を無血清アッセイ培地で1:100に希釈し、試薬リザーバーに移します。この混合物を十分な量を調製し、384ウェルプレートの各ウェルに10μLを加え、さらに10%〜15%の追加容量を加えます。
- マルチチャンネルピペット(Table of Materials)を使用して、10 μLの基質混合物を、384ウェル培養プレート内の細胞を含む各ウェルに移します。プレートを手動またはオービタルシェーカーを使用して1分間ゆっくりと回転させます。
- 384ウェルプレートをマルチモードプレートリーダーに挿入し、セルを含むすべてのウェルの460nmおよび618nmの発光を記録します。
注:このステップで使用したマルチモードプレートリーダー( 詳細については、材料の表 を参照)では、読み取り高さ6.5mm、読み取り時間測定0.1秒を使用しましたが、他のプレートリーダーを使用する場合は、さらに最適化が必要になる場合があります。 - 次の式を使用して、+vehicle と +ligand の生の BRET比をミリブレット単位(mBU)で個別に計算します。
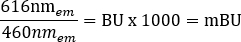
- +Ligand の +Vehicle BRET 比率を差し引いて、補正された BRET比率を計算します(補足表 1)。
注:複数の実験結果を比較する場合、補正されたBRET比をプールし、Nano-CRAFWTと14-3-3ζWT-HaloからなるWildtype(WT)N-BRETペアの平均に正規化することができます(補足表1)。
5. タンパク質発現量の確認(4日目と5日目)
注:このステップでは、6ウェルプレート内の細胞を溶解し、Nano-CRAFおよび14-3-3ζ-Haloタンパク質のタンパク質発現レベルを、HaloおよびNanoタグに特異的な抗体を用いたウェスタンブロット分析により決定します。(図2)。
- NP40溶解バッファー(材料表)にプロテアーゼ阻害剤とホスファターゼ阻害剤を添加すると、サンプルあたり200 μLの溶解バッファーが得られます。
- 細胞を含む6ウェルプレートから培地を吸引します。細胞を1mLの冷たいPBSで一度洗浄し、吸引します。
- 各ウェルに125 μLのNP40溶解バッファーを添加し、6ウェルプレートをロッキングプラットフォーム上で4°Cで15分間インキュベートして細胞を溶解します。
- ライセートを氷上で1.7 mLチューブに移し、20,000 x g で4°Cで10分間遠心分離します。 透明化したライセートを氷上に戻し、BradfordまたはBicinchoninic acid(BCA)アッセイなどの市販のアッセイを使用してタンパク質濃度を測定します。
- 適切な量のライセートとNP40溶解バッファーを新鮮な1.7 mLチューブに総容量100 μLまで移すことにより、すべてのサンプルのタンパク質濃度を標準化します。
- 5倍タンパク質サンプルバッファー(Table of Materials)を1分間煮沸し、各サンプルに25 μLを加えます。サンプルを5〜6分間煮沸してから、チューブを短時間パルス遠心分離して、すべてのサンプルがチューブのベースに集まるようにします。
- 25 μLのサンプルを重複タンパク質ゲル(ゲル1およびゲル2)の各ウェルにロードし、次に、前述の22と同様に、標準的なウェスタンブロッティング手順を使用してタンパク質をニトロセルロースまたはPVDFメンブレンに移します。
- メンブレンを3% BSA-PBSで25°Cで30分間ブロックし、次いでメンブレンをHalo抗体(1:1,000希釈;ゲル 1) およびナノルシフェラーゼまたは CRAF 抗体 (1:500 希釈;トリス緩衝生理食塩水中のゲル 2) に 0.2% Tween-20 (TBST; 資料の表)。
- メンブレンをTBST10 mLで5分間1回洗浄した後、TBSTで1:10,000に希釈した抗マウスHRP二次抗体と室温で1時間インキュベートします。
- メンブレンをロッキングプラットフォーム上で10 mLのTBSTに入れ、25°Cでそれぞれ5分間3回洗浄します。TBSTを除去し、ECL試薬とX線フィルムプロセッサーまたはその他の適切なイメージングシステムを使用してタンパク質バンドを可視化します。
結果
このプロトコル(図2)で説明されているように実行すると、Nano-CRAFWT と14-3-3ζ-Haloの相互作用により、50-60 mBUの補正されたBRET比が生成されます(図3A;補足表1)。CRAFには、リン酸化依存性の2つの14-3-3ドッキングサイト、N'サイトとC'サイトが含まれています(図1)8。したがって、CRAF:14-3-3ζ結合を減少させるための適切な制御には、リン酸化を防止するS259AおよびS621Aが含まれ、さらに14-3-3のN'部位およびC'部位へのそれぞれ25の結合が含まれる。 図3Aに示すように、Nano-CRAFS259A はBRET信号を~45%、Nano-CRAFS621A は~25%減少しますが、S259A/S621A(SS/AA)二重変異体はCRAF:14-3-3 BRET信号をほぼ完全に除去します。さらに、複数の独立した実験の結果は、最初に各実験の結果を平均補正されたBRET比に正規化し(補足表1)、次に3つの実験すべてのデータポイントをプールすることにより比較できます(図3B)。タグ付きタンパク質の相対発現レベルも、ウェスタンブロット分析によって確認する必要があります(図3)。変異タンパク質の発現レベルはWTの発現レベルとは異なる場合があり、変異DNAベクターのトランスフェクト量をそれに応じて調整する必要があることに留意されたい(補足表1)。
また、このプロトコールの最適化は、特に代替のプレートリーダー、細胞株、またはトランスフェクション試薬を使用する場合に必要になる可能性があることにも留意する必要があります。 図4A は、pCMV5-14-3-3ζ-Haloアクセプタープラスミドのトランスフェクト量を増加させ、pCMV5-NanoLuc-CRAFドナーの量を一定に保ちながら、用量依存的にBRETシグナルを増加させ、その後、14-3-3-結合欠損CRAFSS/AA 変異体の存在によってBRETシグナルを減少させる最適化実験の例を示しています。ただし、アクセプターとドナーの比率が高すぎると、Nano-CRAFWT への14-3-3ζ-Halo結合とNano-CRAFSS / AA ネガティブコントロールのシグナルの差が小さくなり、Nano-CRAFWTの結合飽和が反映される可能性があります。また、標準誤差の少ない再現性のある値を生成するBRET比が達成されれば、アクセプター対ドナー比をさらに増やす必要はありません。実際、これらのアッセイの主な利点の1つは、生理学的発現レベルでタンパク質の相互作用を測定できることです。新しいN-BRETアッセイを開発する際には、タグの配置も重要な考慮事項です。 図4B は、14-3-3ζがNano-CRAFWT およびNano-CRAFSS/AAに結合することによって生成される補正BRET比に、HaloタグをC末端からN末端に切り替えることの影響を示しています。この目的のために、可能なタグ位置のそれぞれについてDNA発現コンストラクトを生成する必要があり(図4C)、WTペアと結合に対する既知のネガティブコントロールのBRET比の両方に対するそれらの影響を評価する必要があります。

図1:14-3-3タンパク質によるRAF制御 (A)保存された14-3-3ドッキング部位が示されたRAFキナーゼドメイン構造の概略図。(B)14-3-3タンパク質によるRAF制御のモデル。(C)C末端のHaloタグアクセプターに融合した14-3-3ζとN末端のナノドナーに融合したCRAFの相互作用を測定するN-BRETアッセイ。ドナーからアクセプターへのエネルギー移動により、BRET信号が生成されます。 この図の拡大版を表示するには、ここをクリックしてください。

図2:CRAF:14-3-3生細胞BRETベースのアッセイを実施するための主要なステップ。 このプロトコルで概説されている5つの主要なステップには、細胞の播種、トランスフェクション、および再プレーティング、BRETシグナルの読み取り方法、およびHaloタグ付きタンパク質とNanoタグ付きタンパク質の発現レベルの確認方法が含まれます。 この図の拡大版を表示するには、ここをクリックしてください。

図3:CRAF:14-3-3 BRETベースのアッセイ。 14-3-3ζ-HaloとNano-CRAF -WT、または-S259A、-S621A、および-S259A/S621A(SS/AA)変異体との相互作用を比較するN-BRETアッセイは、CRAF:14-3-3の結合を損なうことを示しました25。14-3-3ζ-Halo(MW:64 kDa.)およびNano-CRAFバリアント(MW:93 kDa.)の発現レベルをウェスタンブロット分析により検討しました。(A)1回の実験で得られた4つの反復ウェルからのBRET測定値±標準偏差(SD)を補正。(B)3つの独立した実験からの補正されたBRET測定値をプールし、Nano-CRAFWT (100に設定)±SDに正規化しました。統計的有意性は、スチューデントのt検定(両側、等分散を仮定)によって計算されました。p<0.0001。 この図の拡大版を表示するには、ここをクリックしてください。
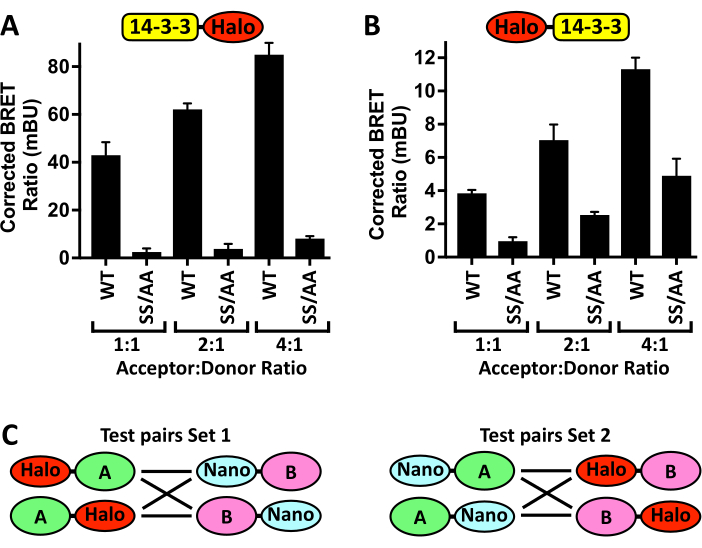
図4:アッセイの最適化ステップ。 (A)14-3-3ζ-Haloまたは(B)Halo-14-3-3ζを一定量のNano-CRAF-WTまたは-SS/AA(S259A/S621A)とBRETシグナル±SDに同時トランスフェクションした場合の影響 (C)HaloまたはNanoタグを目的の各タンパク質(AおよびB)のN末端またはC末端に配置することにより生成されるすべてのN-BRETペアの概略図。n = 4 です。 この図の拡大版を表示するには、ここをクリックしてください。
補足表1:CRAF:14-3-3 BRETベースのアッセイからの例データ。 示された発現コンストラクトを2日目(上部)に細胞にトランスフェクションした。生、補正、および正規化された BRET測定値は、4日目(下部)に測定および計算されました。 このファイルをダウンロードするには、ここをクリックしてください。
ディスカッション
これまでの研究では、14-3-3タンパク質がRAFキナーゼの活性化と阻害の両方に重要な役割を果たすことが示されています。これらの結合イベントがどのように制御され、これらの相互作用を調節することがRAFシグナル伝達とRAF駆動型発癌に及ぼす影響を理解することで、CRAF機能を標的とする新たな治療上の脆弱性が明らかになるかもしれません。しかし、Rafの活性化サイクルは、多数の関連タンパク質、翻訳後修飾、および細胞内局在の変化によって支えられている8ため、これらの条件を完全に再現するためには、RAFとその調節因子との相互作用を生細胞条件下で測定する必要がある。このプロトコルは、生細胞中の14-3-3ζとCRAFの相互作用を測定するためのナノルシフェラーゼ-Halo N-BRETペアの優れた信号対雑音比とスペクトル分解能を利用する新しい近接ベースのアッセイを説明しています。
このアッセイは、生理学的に適切な条件下での14-3-3とCRAFの複雑な相互作用を研究するために切望されていたプラットフォームを提供しますが、考慮すべきいくつかの制限があります。第一に、14-3-3がRAFの自己抑制と二量体化8の両方で役割を果たすことが示されていることを考えると、これらの状態を区別するために追加の制御と実験的アプローチが組み込まれる可能性がある。例えば、このアッセイは、RAF自己阻害12,22の変化を直接読み出すことができる、以前に開発されたN−BRET自己阻害アッセイと組み合わせて実施することができる。さらに、CRAF R401H変異のような制御は、CRAFと他のRAFプロトマーとの二量体化を妨害することが示されている15,26、これらの機能を解剖するのに有用であることが証明されるかもしれない。最後に、HaloタグとNanoタグの配置は、CRAF:14-3-3相互作用の測定に最適化されていますが、CRAFまたは14-3-3と他のタンパク質との相互作用が結果として影響を受けないことを保証するものではなく、このアッセイでは、CRAF:14-3-3結合の親和性、これらの相互作用がCRAF活性に及ぼす影響、またはこれらの相互作用が発生する細胞内の位置を決定することはできません。したがって、in vitro結合アッセイ、CRAFキナーゼアッセイ、および細胞内局在研究12,27,28,29など、他の補完的なアプローチを組み込む必要もあるかもしれない。
このアッセイ、または実際にN-BRETベースのアッセイを実施する際には、考慮すべきいくつかの要因があります。この論文および以前の研究22で示されているように、pCMV5-Halo(アクセプター)およびpCMV5-ナノルシフェラーゼ(ドナー)コンストラクトの正しいトランスフェクション比、およびPOIの結合を破壊することが知られている変異体などの適切なネガティブコントロールは、これらのアッセイの最適化と試験にとって重要です。Halo タグ付き POI の表現を増やし、Nano タグ付き POI のレベルを一定に保つと、BRET比が増加します。ただし、バインディングが飽和すると、信号は横ばいになり、WTとバインディング障害のあるPOIとの間のデルタが減少するため、アクセプタードナーの最適化が必要になります。アッセイの最適化におけるもう一つの重要な要素は、pCDNA3.1のような空のベクターキャリアDNAの使用です。200 ngのpCDNA3.1の包含。トランスフェクションミックスでは、Haloタグ付きPOIおよびNanoタグ付きPOIの発現レベルが大幅に増加し、レプリケートウェルと実験22との間のばらつきが減少する。しかし、トランスフェクション効率が試薬と細胞株の間で異なる可能性があることを考えると、前述のように、N-BRETペアの最適な発現を達成するためには、キャリアDNAの量も滴定する必要があるかもしれない22。BRET比率に大きく影響するもう一つの要因は、タグの配置です。実際、どのPOIがHaloタグとNanoタグに融合しているか、またこのタグがタンパク質のN末端またはC末端に配置されているかどうかは、BRET比の振幅と、このシグナルを減少させるネガティブコントロールの能力の両方に影響を与える可能性があります。したがって、新しいアッセイを開発する際には、可能なN-BRETペアのそれぞれをテストすることも重要です。
N-BRETは従来のBRETよりも感度とスペクトル分解能が向上しているため、生細胞での結合の弱い相互作用や微妙な変化を測定することができます。しかし、これらのアッセイが細胞シグナル伝達事象の研究に有用であることは、まだ十分には認識されていません。特に、このアッセイは、14-3-3と他のキナーゼ、例えば、仲間のRAFキナーゼARAFおよびBRAFとの相互作用を研究するために適応させることができ、RASを研究するために以前に発表されたN-BRETアッセイと組み合わせると、RASを研究するために:RAF結合およびRAF自己阻害22,30は、生細胞条件下でRAF調節を測定するためのツールキットの基礎を形成するであろう。さらに、N−BRETは、生細胞薬物スクリーニングプラットフォーム30として有望であることを示しており、したがって、このアッセイは、14−3−3およびRAFとの相互作用による阻害性を安定化させるか、またはRAF活性化に関与するものを妨害する化合物を同定するためにも有用であり得る。ナノルシフェラーゼシグナルの高い安定性と、アクセプターとドナーの放出を同時に検出する最新のプレートリーダーの能力を考えると、これらのアッセイはシグナル伝達タンパク質の相互作用をリアルタイムで測定するために適応させることができ、これは上流の刺激や薬物治療に応答したこれらの相互作用のダイナミクスを理解するために重要になります。
開示事項
開示するものはありません。
謝辞
このプロジェクトは、プロジェクト番号ZIA BC 010329の下で、国立がん研究所、国立衛生研究所からの連邦資金によって部分的に資金提供されています。
資料
| Name | Company | Catalog Number | Comments |
| Antibodies | |||
| HaloTag® mouse monoclonal antibody | Promega | G9211 | Antibody for detecting HaloTag tagged proteins by immunoblot |
| NanoLuc® mouse monoclonal antibody | R&D Systems | MAB10026 | Antibody for detecting Nano-tagged proteins by immunoblot |
| CRAF mouse monoclonal antibody (E10) | Santa Crus Biotechnology | sc-7267 | Antibody directly detecting CRAF proteins by immunoblot |
| ECL anti-mouse HRP secondary antibody | Amersham | NA931-1ML | Secondary HRP conjugated mouse antibody (from sheep) |
| Reagents | |||
| X-tremeGENE™ 9 | Roche/Sigma | 6365809001 | |
| NanoBRET™ kit | Promega | N1661 | NanoBRET kit containing Halo 618 ligand and NanoGlo (nanoluciferase) substrate |
| DPBS, without Ca++ and Mg++ | Quality Biologicals | 114-057-101 | |
| Trypsin-EDTA (0.05%), phenol red | Life Technologies | 25300120 | |
| DMEM cell culture media | Life Technologies | 11995073 | High glucose, L-glutamine, phenol red, sodium pyruvate; without HEPES, suppliment media with 10% FBS, 2 mM L-glutamine and 100U penicillin-streptomycin |
| L-Glutamine (200 mM) | Life Technologies | 25030164 | |
| Penicillin-Streptomycin (10,000 U/mL) | Life Technologies | 15140163 | |
| Opti-MEM™ I reduced serum media | Gibco | 31985062 | For cell transfection |
| Opti-MEM reduced serum media, no phenol red | Gibco | 11058021 | For replating cells on Day 3. Supplement with 2 mM L-glutamine and 100U penicillin-streptomycin, along with 10% FBS (where indicated). |
| Invitrogen Trypan Blue Stain | Thermo Scientific | T10282 | |
| NP40 lysis buffer | N/A | N/A | 20 mM Tris (pH 8.0), 137mM NaCl, 10% glycerol, NP40 alternative (Milipore, Cat# 492016). Store at 4 degrees C.. Add the following protease and phosphatase immediately prior to use: 20 µM leupeptin, 0.5 mM sodium orthovanidate, 0.15 U/mL, 1mM PMSF. |
| 5x gel sample buffer | N/A | N/A | 240 mM Tris (pH 8.0), 9.5% SDS, 30% glycerol, 500mM DTT, 3mM bromophenol blue. Store at -20 degrees C. |
| Cell lines | |||
| 293FT cells (human) | Thermo Scientific | R70007 | |
| DNA vectors | |||
| pCMV5-Nano-CRAF WT and mutant | N/A | N/A | |
| pCMV5-14-3-3ζ-Halo | N/A | N/A | |
| Equipment | |||
| EnVision 2104 Multimode Plate Reader | PerkinElmer 2104 | 2104-0010 | 600LP NanoBRET & M460/50 nm NanoBRET emmisions filters, Luminescence 404 mirror, 6.5 mm measurement height and 0.1 s measurement time |
| Invitrogen Countess™ II Automated Cell Counter | Thermo Scientific | AMQAX1000 | |
| ThermoFisher E1-ClipTip™ Multichannel Pipettor | Thermo Scientific | 4672070 | |
| Software | |||
| GraphPad Prism (version 10.0.3) | GraphPad | www.graphpad.com | |
| Other | |||
| ThermoFisher ClipTip Multichannel pipette tips | Thermo Scientific | 94410153 | |
| Reagent Reservoir, 25 mL Divided, Sterile | Thomas Scientific | 1228K16 | |
| Perkin Elmer 384-well CulturPlate™ | PerkinElmer | 6007680 | White, polystyrene, tissue culture treated |
| Countess Cell Counting Chamber Slides | Thermo Scientific | C10228 |
参考文献
- Blasco, R. B., et al. c-Raf, but not B-Raf, is essential for development of K-Ras oncogene-driven non-small cell lung carcinoma. Cancer Cell. 19, 652-663 (2011).
- Blasco, M. T., et al. Complete regression of advanced pancreatic ductal adenocarcinomas upon combined inhibition of EGFR and C-RAF. Cancer Cell. 35, 573-587 (2019).
- Karreth, F. A., Frese, K. K., DeNicola, G. M., Baccarini, M., Tuveson, D. A. C-Raf is required for the initiation of lung cancer by K-Ras(G12D). Cancer Discov. 1, 128-136 (2011).
- Lito, P., et al. Disruption of CRAF-mediated MEK activation is required for effective MEK inhibition in KRAS mutant tumors. Cancer Cell. 25, 697-710 (2014).
- Sanclemente, M., et al. c-RAF ablation induces regression of advanced Kras/Trp53 mutant lung adenocarcinomas by a mechanism independent of MAPK signaling. Cancer Cell. 33, 217-228 (2018).
- Razzaque, M. A., et al. Germline gain-of-function mutations in RAF1 cause Noonan syndrome. Nat Genet. 39, 1013-1017 (2007).
- Pandit, B., et al. Gain-of-function RAF1 mutations cause Noonan and LEOPARD syndromes with hypertrophic cardiomyopathy. Nat Genet. 39, 1007-1012 (2007).
- Terrell, E. M., Morrison, D. K. Ras-mediated activation of the Raf family kinases. Cold Spring Harb Perspect Med. 9 (1), 033746(2019).
- Kondo, Y., et al. Cryo-EM structure of a dimeric B-Raf:14-3-3 complex reveals asymmetry in the active sites of B-Raf kinases. Science. 366, 109-115 (2019).
- Park, E., et al. Architecture of autoinhibited and active BRAF-MEK1-14-3-3 complexes. Nature. 575 (7783), 545-550 (2019).
- Tzivion, G., Luo, Z., Avruch, J. A dimeric 14-3-3 protein is an essential cofactor for Raf kinase activity. Nature. 394, 88-92 (1998).
- Spencer-Smith, R., et al. RASopathy mutations provide functional insight into the BRAF cysteine-rich domain and reveal the importance of autoinhibition in BRAF regulation. Mol Cell. 82, 4262-4276 (2022).
- Martinez Fiesco, J. A., Durrant, D. E., Morrison, D. K., Zhang, P. Structural insights into the BRAF monomer-to-dimer transition mediated by RAS binding. Nat Commun. 13, 486(2022).
- Freeman, A. K., Ritt, D. A., Morrison, D. K. The importance of Raf dimerization in cell signaling. Small GTPases. 4, 180-185 (2013).
- Freeman, A. K., Ritt, D. A., Morrison, D. K. Effects of Raf dimerization and its inhibition on normal and disease-associated Raf signaling. Mol Cell. 49, 751-758 (2013).
- Rushworth, L. K., Hindley, A. D., O'Neill, E., Kolch, W. Regulation and role of Raf-1/B-Raf heterodimerization. Mol Cell Biol. 26, 2262-2272 (2006).
- Garnett, M. J., Rana, S., Paterson, H., Barford, D., Marais, R. Wild-type and mutant B-RAF activate C-RAF through distinct mechanisms involving heterodimerization. Mol Cell. 20, 963-969 (2005).
- Tran, N. H., Wu, X., Frost, J. A. B-Raf and Raf-1 are regulated by distinct autoregulatory mechanisms. J Biol Chem. 280, 16244-16253 (2005).
- Chong, H., Guan, K. L. Regulation of Raf through phosphorylation and N terminus-C terminus interaction. J Biol Chem. 278, 36269-36276 (2003).
- Cutler, R. E., Stephens, R. M., Saracino, M. R., Morrison, D. K. Autoregulation of the Raf-1 serine/threonine kinase. Proc Natl Acad Sci U S A. 95, 9214-9219 (1998).
- Park, E., et al. Cryo-EM structure of a RAS/RAF recruitment complex. Nat Commun. 14, 4580(2023).
- Spencer-Smith, R., Morrison, D. K. Protocol for measuring BRAF autoinhibition in live cells using a proximity-based NanoBRET assay. STAR Protoc. 4, 102461(2023).
- Clark, G. J., et al. 14-3-3 zeta negatively regulates raf-1 activity by interactions with the Raf-1 cysteine-rich domain. J Biol Chem. 272, 20990-20993 (1997).
- Machleidt, T., et al. NanoBRET--A novel BRET platform for the analysis of protein-protein interactions. ACS Chem Biol. 10, 1797-1804 (2015).
- Hekman, M., et al. Dynamic changes in C-Raf phosphorylation and 14-3-3 protein binding in response to growth factor stimulation: differential roles of 14-3-3 protein binding sites. J Biol Chem. 279, 14074-14086 (2004).
- Hatzivassiliou, G., et al. RAF inhibitors prime wild-type RAF to activate the MAPK pathway and enhance growth. Nature. 464, 431-435 (2010).
- Bondzi, C., Grant, S., Krystal, G. W. A novel assay for the measurement of Raf-1 kinase activity. Oncogene. 19, 5030-5033 (2000).
- Spencer-Smith, R., et al. Inhibition of RAS function through targeting an allosteric regulatory site. Nat Chem Biol. 13 (1), 62-68 (2016).
- Roy, S., et al. 14-3-3 facilitates Ras-dependent Raf-1 activation in vitro and in vivo. Mol Cell Biol. 18, 3947-3955 (1998).
- Durrant, D. E., et al. Development of a high-throughput NanoBRET screening platform to identify modulators of the RAS/RAF interaction. Mol Cancer Ther. 20, 1743-1754 (2021).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved