Method Article
基于生物发光共振能量转移 (BRET) 的测定法,用于测量 CRAF 与活细胞中 14-3-3 蛋白的相互作用
摘要
该方案描述了一种基于 BRET 的测定法,用于测量 CRAF 激酶与活细胞中 14-3-3 蛋白的相互作用。该协议概述了准备细胞、读取 BRET 发射和数据分析的步骤。还提供了一个示例结果,其中确定了适当的对照和分析优化的故障排除。
摘要
CRAF 是 RAS GTP 酶的主要效应子,在几种 KRAS 驱动的癌症的肿瘤发生中起关键作用。此外,CRAF 是种系突变的热点,种系突变被证明会导致发育性 RASopathy、Noonan 综合征。所有 RAF 激酶都包含 14-3-3 调节蛋白的多个磷酸化依赖性结合位点。14-3-3 与这些位点的差异结合在信号转导条件下质膜上活性 RAF 二聚体的形成和在静止条件下维持 RAF 自抑制中起着重要作用。了解这些相互作用如何被调节以及如何调节它们对于确定靶向 RAF 功能的新治疗方法至关重要。在这里,我描述了一种基于生物发光共振能量转移 (BRET) 的测定法,用于测量 CRAF 与活细胞中 14-3-3 蛋白的相互作用。具体来说,该测定法测量了与纳米荧光素酶供体融合的 CRAF 和与 Halo 标签受体融合的 14-3-3 的相互作用,其中 RAF 和 14-3-3 的相互作用导致供体到受体的能量转移和 BRET 信号的产生。该方案进一步表明,该信号可以被显示阻止 14-3-3 与其每个高亲和力 RAF 停靠位点结合的突变破坏。该实验步骤描述了接种、转染和重新铺板细胞的程序,以及读取 BRET 发射、进行数据分析和确认蛋白质表达水平的详细说明。此外,还提供了示例分析结果以及优化和故障排除步骤。
引言
RAF 激酶 (ARAF、BRAF 和 CRAF) 是 RAS GTP 酶的直接效应子,也是促增殖/促存活 RAF-MEK-ERK 激酶级联反应的起始成员。最近的研究表明,CRAF 表达在几种 KRAS 驱动的癌症的肿瘤发生中起关键作用,包括非小细胞肺癌和胰腺导管腺癌 1,2,3,4,5。此外,种系 CRAF 突变会导致一种特别严重的 RASopathy,即 Noonan 综合征 6,7。了解 CRAF 调节对于开发针对其在细胞中的功能的成功治疗方法至关重要。
所有 RAF 激酶均可分为两个功能结构域,即 C 端催化 (CAT) 结构域和 N 端调节 (REG) 结构域,用于控制其活性(图 1A)8。REG 结构域包括 RAS 结合结构域 (RBD)、富含半胱氨酸的结构域 (CRD) 和富含丝氨酸/苏氨酸的区域 (S/T 富集)。值得注意的是,富含 S/T 的区域包含 N' 位点,该位点以磷酸化依赖性方式与 14-3-3 结合(CRAF 中的 S259;图 1A)8. CAT 结构域包括激酶结构域,以及第二个高亲和力 14-3-3 对接位点,称为 C' 位点(CRAF 中的 S621;图 1A)8. 二聚体 14-3-3 蛋白与 N' 和 C' 位点的差异结合以及 CRD 在 RAF 激活和抑制中起关键作用 9,10,11,12,13。在正常信号转导条件下,RAF 激活是通过 RAS 募集到质膜而启动的,使其能够形成活性二聚体,其中 BRAF-CRAF 异二聚体是主要的活性形式14,15。使用 BRAF 和 CRAF 的生化测定以及二聚体 BRAF 的低温电子显微镜 (Cryo-EM) 结构表明,14-3-3 二聚体通过同时结合两个 RAF 原聚体的 C' 位点来稳定活性 RAF 二聚体(图 1B)9,13,16,17。相反,研究表明,在静止条件下,RAF 采用胞质性、自抑制确认,其中 REG 结构域与 CAT 结构域结合并抑制其活性 12,18,19,20。这种闭合状态通过与 REG 结构域中的 CRD 和 N' 位点以及 CAT 结构域中的 C' 位点结合的 14-3-3 二聚体稳定(图 1B)10,13,21。在 BRAF 中,该模型得到了自体抑制 BRAF 单体的最新冷冻电镜结构以及我们之前的生化研究的支持 10,12,13,21,22。然而,虽然 14-3-3 在 CRAF 调节中起抑制作用23,但类似 BRAF 的自身抑制状态在 CRAF 调节中的作用可能较小12;因此需要进一步的研究来阐明 14-3-3 蛋白调节 CRAF 活性的机制。14-3-3 介导的 RAF 激酶调节需要大量的 RAF 磷酸化和去磷酸化事件、与各种调节蛋白的结合以及与质膜的相互作用8。因此,在生理相关条件下和存在完整脂质双层的情况下测量 14-3-3-RAF 相互作用至关重要。
为了解决这个问题,利用 NanoBRET(以下简称 N-BRET;试剂盒详情见材料表)技术开发了一种基于邻近度的测定法,用于测量 CRAF 与活细胞中 14-3-3 蛋白的相互作用(图 1C)。这种基于 BRET 的系统可测量两种目标蛋白 (POI) 的相互作用,其中一种蛋白质用纳米荧光素酶 (Nano) 供体标记,另一种用 Halo 标签标记,以便用 Halo618 能量受体配体标记22,24。目标蛋白质的相互作用导致供体到受体的能量转移,进而产生 BRET 信号(图 1C)。与传统 BRET 相比,极亮的纳米供体蛋白(发射 (em) 460 nm)和 Halo618 配体 (em 618 nm) 可提供更高的光谱分离和灵敏度,使其成为研究较弱相互作用和检测结合细微变化的理想平台24。事实上,我们之前开发了一种基于 N-BRET 的测定法,用于测量 RAF REG 和 CAT 结构域的自抑制相互作用,这对于表征 BRAF CRD 中的一组 RASopathy 突变至关重要,并证明了该结构域对于维持自身抑制和防止组成型 BRAF 激活的至关重要12。
此处描述的测定法测量与 N 端纳米标签 (Nano-CRAF) 融合的 CRAF 和与 C 端 Halo 标签融合的 14-3-3 的 zeta 亚型(14-3-3ζ-Halo; 图 1C)。我们表明,Nano-CRAF 与 14-3-3ζ-Halo 的相互作用会产生强大的 BRET 信号,该信号反过来又可以被阻止 14-3-3 与 N' 位点 (S259A) 和/或 C' 位点 (S621A) 结合的突变破坏。以下方案提供了执行、优化和排除此分析的详细步骤。
研究方案
注:该测定在 293FT 细胞中进行。来源于人胚胎肾细胞的特征明确且易于转染的上皮细胞系。这些细胞的单个汇合 10 cm 培养皿通常提供足够的细胞来接种 6 孔组织培养板的 20 孔。步骤 1-3 必须在生物安全柜中使用无菌技术进行。
1. 细胞接种(第 1 天)
注:在此步骤中,将细胞从组织培养皿中分离出来,计数并接种在 6 孔组织培养板中,以便在步骤 2 中进行转染(图 2)。
- 从 10 cm 培养皿中的细胞中吸出培养基。用 5 mL 磷酸盐缓冲盐水 (PBS) 洗涤细胞并吸出。
- 加入 1 mL 胰蛋白酶-乙二胺四乙酸 (EDTA),并在 37 °C 下孵育 3-5 分钟,以将细胞从培养皿中分离出来。
- 向细胞中加入 9 mL 完全 Dulbecco 改良的 eagle 培养基 (DMEM) 以中和胰蛋白酶,并反复上下移液以产生均匀的单细胞悬液。
- 立即将 20 μL 细胞转移到 1.7 mL 试管中,并与 20 μL 台盼蓝染色剂混合。使用自动细胞计数仪(材料表)或血细胞计数器对细胞进行计数。
- 在完全 DMEM 培养基中将细胞稀释至 2 x 105 个细胞/mL,并向 6 孔组织培养板的每个孔中加入 2 mL(4 x 105 个细胞/孔)。将细胞在 37 °C 和 10% CO2 下孵育过夜。
注:建议使用传代次数少于 20 次的 293FT 细胞。我们之前发现,使用传代次数更大的细胞会导致 BRET 比率降低和孔间信号变异性增加。
2. 细胞转染(第 2 天)
注意:在这里,用 pCMV5-NanoLuc-CRAF 和 pCMV5-14-3-3ζ-Halo 表达构建体以及 pCDNA3.1 空载体转染细胞(图 2)。
- 转染前,将 N-BRET 质粒稀释至 5 ng/μL,将 pCDNA3.1 稀释至 100 ng/μL,并在一组无菌 1.7 mL 试管中编号。
- 向每个试管中加入 100 μL 转染培养基(详见 材料表 ),以及 5 ng pCMV5-NanoLuc-CRAF、10 ng pCMV5-14-3-3ζ 和 200 ng PCDNA3.1。
- 加入 2 μL 转染试剂(详见 材料表 )并轻轻涡旋混合。在微量离心机中脉冲旋转试管,以确保所有液体都收集在试管底部,然后在 25 °C 左右孵育 15 分钟。
- 向细胞中滴加转染复合物,并在 37 °C/10% CO2 下孵育 16-20 小时,以表达 Halo 和 Nano 标记的蛋白质。
注:添加空载体 (pCDNA3.1) 载体 DNA 对于实现 N-BRET 表达构建体的高转染效率至关重要。如前所述,未能添加空载体会导致 14-3-3ζ-Halo 和 Nano-CRAF 蛋白的表达水平降低,进而导致 BRET 比率较弱且不一致22。
3. 细胞重新铺板(第 3 天)
注:在此步骤中,将细胞转移到 384 孔板和 Halo 618 配体(+配体; 材料表)或添加 DMSO (+vehicle) 以读取步骤 4 中的 BRET 发射。将剩余的细胞转移至新鲜的 6 孔培养板中,以便在步骤 5 中进行蛋白质印迹分析(图 2)。
- 收集以下材料并按照以下说明准备工作区域。
- 使用 37 °C 水浴,预热胰蛋白酶-EDTA,以及无血清检测培养基和 10% FBS 补充的检测培养基(有关详细信息 ,请参阅材料表 )。
- 根据要测量的样品数量,预先标记三组无菌 1.7 mL 试管(套装 1-3)和一组无菌 15 mL 锥形底管。在水平转子离心机上配备 15 mL 试管插件,并预冷至 4 °C。
- 将以下物品放入组织培养罩中:试剂储液槽、384 孔组织培养板、6 孔组织培养板、多通道移液器和吸头、细胞计数玻片/腔室和台盼蓝染色剂(材料表),以及 1.7 mL 试管组 1-3。
- 如下所述收获并计数细胞。
- 从 6 孔板中吸出培养基,并向细胞中加入 250 μL 胰蛋白酶-EDTA。在 37 °C 下孵育 6 孔板,直到细胞开始分离(3-5 分钟)。
- 向每个孔中加入 1 mL 补充 10% FBS 的检测培养基以中和胰蛋白酶,并用力上下移液以产生单细胞悬液。
- 将 1 mL 细胞悬液转移到预先标记的 15 mL 试管中。向每个 15 mL 试管中再添加 1 mL 补充 10% FBS 的检测培养基。
- 将试管倒置 5 次以混合,并立即将 20 μL 细胞悬液转移到试管组 1 中。
- 在预冷的水平吊篮离心机中以 ~250 x g 的速度离心 5 分钟。在离心步骤中,将 20 μL 台盼蓝细胞染色剂与管组 1 中的细胞混合,然后使用自动细胞计数仪或血细胞计数器对细胞进行计数。典型产量为 6-8 x 105 个细胞/mL。
- 从离心机中取出 15 mL 试管,并从细胞沉淀中吸出培养基。在无血清检测培养基中将细胞沉淀重悬至 2 x 105 个细胞/mL,并用力移液以生成单细胞悬液。
注:使用无血清检测培养基用于静止正常细胞信号通路。
- 如下所述,在 384 孔板和 6 孔板中重新铺板。
- 将 15 mL 试管倒置数次以确保细胞悬液均匀,并将 500 μL 转移至 1.7 mL 试管 2 组和组 3 中。
- 将 0.5 μL DMSO(+载体)添加到组 2 中,将 0.5 μL Halo 618 配体(+配体)添加到管组 3 中,然后移液混合。
- 将 +载体和 +配体细胞悬液转移到试剂储液槽的单独孔中。使用多通道移液器(材料表),将 40 μL + 载体细胞悬液从试剂储液槽转移到 384 孔培养板的四份孔中。对 +配体细胞悬液重复此步骤。
- 将剩余细胞转移到新鲜的 6 孔培养板中。将 384 孔板和 6 孔板在 37 °C 和 5% CO2 下孵育过夜。
4. 读取 BRET 排放(第 4 天)
注:在此步骤中,将纳米荧光素酶底物(详见 材料表 )添加到 384 孔培养板中的细胞中,并读取 N-BRET 受体 (618 nm) 和供体 (460 nm) 发射(图 2)。然后计算校正后的 BRET 比率。
- 在 37 °C 水浴中预热无血清检测培养基,并在 25°C 下解冻纳米荧光素酶底物。 在无血清检测培养基中以 1:100 的比例稀释纳米荧光素酶底物,然后转移到试剂储液槽中。准备足够的这种混合物,向 384 孔板的每个孔中加入 10 μL,外加 10%-15% 的额外体积。
- 使用多通道移液器(材料表),将 10 μL 底物混合物转移到 384 孔培养板中含有细胞的每个孔中。轻轻旋转板 1 分钟,手动或使用轨道振荡器。
- 将 384 孔板插入多功能读板器中,并记录所有包含细胞的孔的 460 nm 和 618 nm 发射。
注意:对于用于此步骤的多功能读板器(有关详细信息,请参阅 材料表 ),使用了 6.5 mm 的读取高度和 0.1 s 的读取时间测量,但是如果使用其他读板器,则可能需要进一步优化。 - 使用以下等式分别计算 +载体和 +配体的原始 BRET 比率,以 milliBRET 单位 (mBU) 为单位:
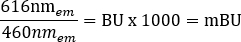
- 通过从 +配体的 BRET 比率中减去 +载体 BRET 比率来计算校正的 BRET 比率(补充表 1)。
注:当比较多个实验的结果时,校正后的 BRET 比率可以合并并标准化为野生型 (WT) N-BRET 对的平均值,该对由 Nano-CRAFWT 和 14-3-3ζWT-Halo 组成(补充表 1)。
5. 确认蛋白表达水平(第 4 天和第 5 天)
注意:在此步骤中,裂解 6 孔板中的细胞,并使用对 Halo 和 Nano 标签具有特异性的抗体通过蛋白质印迹分析确定 Nano-CRAF 和 14-3-3ζ-Halo 蛋白的蛋白质表达水平。(图 2)。
- 向 NP40 裂解缓冲液中加入蛋白酶和磷酸酶抑制剂(材料表),每个样品可使用 200 μL 裂解缓冲液。
- 从含有细胞的 6 孔板中吸出培养基。用 1 mL 冷 PBS 洗涤细胞一次并吸出。
- 向每个孔中加入 125 μL NP40 裂解缓冲液,并在 4 °C 下在摇动平台上孵育 6 孔板 15 分钟以裂解细胞。
- 将裂解物转移至冰上的 1.7 mL 试管中,并在 4 °C 下以 20,000 x g 离心 10 分钟。 将澄清的裂解物放回冰上,并使用市售的测定法测定蛋白质浓度,例如 Bradford 或二辛可宁酸 (BCA) 测定法。
- 通过将适当体积的裂解物和 NP40 裂解缓冲液转移到新鲜的 1.7 mL 试管中,使所有样品中的蛋白质浓度标准化,总体积为 100 μL。
- 将 5x 蛋白质样品缓冲液(材料表)煮沸 1 分钟,并向每个样品中加入 25 μL。将样品煮沸 5-6 分钟,然后短暂脉冲离心试管,以确保所有样品都聚集在试管底部。
- 将 25 μL 样品上样到重复蛋白质凝胶(凝胶 1 和凝胶 2)的每个孔中,然后使用标准蛋白质印迹程序将蛋白质转移到硝酸纤维素或 PVDF 膜上,如前所述22。
- 在 25 °C 下用 3% BSA-PBS 封闭膜 30 分钟,然后用 Halo 抗体(1:1,000 稀释;凝胶 1)和纳米荧光素酶或 CRAF 抗体(1:500 稀释;凝胶 2)溶于 tris 缓冲盐水中,补充有 0.2% Tween-20 (TBST; 材料表)。
- 在 10 mL TBST 中洗涤膜 1 次,每次 5 分钟,然后在室温下与在 TBST 中以 1:10,000 稀释的抗小鼠 HRP 二抗孵育 1 小时。
- 在摇动平台上用 10 mL TBST 在 25 °C 下洗涤膜 3 次,每次 5 分钟。去除 TBST 并使用 ECL 试剂和 X 射线胶片处理器或其他合适的成像系统观察蛋白质条带。
结果
当按照本协议中的描述进行时(图 2),Nano-CRAFWT 和 14-3-3ζ-Halo 的相互作用应产生 50-60 mBU 的校正 BRET 比率(图 3A;补充表 1)。CRAF 包含两个磷酸化依赖性 14-3-3 停靠位点,即 N' 位点和 C' 位点(图 1)8。因此,减少 CRAF:14-3-3ζ 结合的适当对照包括 S259A 和 S621A,它们分别阻止磷酸化,进而阻止 14-3-3 结合到 N' 和 C' 位点25。如图 3A 所示,Nano-CRAFS259A 将 BRET 信号降低 ~45%,Nano-CRAFS621A 降低 ~25%,而 S259A/S621A (SS/AA) 双突变体几乎完全消融 CRAF:14-3-3 BRET 信号。此外,可以通过首先将每个实验的结果归一化为平均校正的 BRET 比率(补充表 1),然后将来自所有三个实验的数据点汇集在一起来比较多个独立实验的结果(图 3B)。标记蛋白的相对表达水平也应通过 western blot 分析来确认(图 3)。请注意,突变蛋白的表达水平可能与 WT 不同,需要相应地调整突变 DNA 载体的转染量(补充表 1)。
还应注意的是,可能需要优化此方案,尤其是在使用替代读板机、细胞系或转染试剂时。 图 4A 显示了一个示例优化实验的结果,其中增加 pCMV5-14-3-3ζ-Halo 受体质粒的转染量,同时保持 pCMV5-NanoLuc-CRAF 供体的量恒定,剂量依赖性地增加 BRET 信号,然后由于 14-3-3 结合缺陷 CRAFSS/AA 突变体的存在而降低。然而,如果受体与供体的比率过高,则 14-3-3ζ-Halo 与 Nano-CRAFWT 与 Nano-CRAFSS/AA 阴性对照的结合差异将减小,这可能反映了 Nano-CRAFWT 的结合饱和度。此外,一旦达到产生低标准误差的可重现值的 BRET 比率,就没有必要进一步增加受体与供体比率。事实上,这些检测的主要优势之一是能够在生理表达水平上测量蛋白质的相互作用。在开发新的 N-BRET 检测时,另一个重要的考虑因素是标签放置。 图 4B 显示了将 Halo 标签从 C 端切换到 14-3-3ζ 的 N 端对 14-3-3ζ 与 Nano-CRAFWT 和 Nano-CRAFSS/AA 结合产生的校正 BRET 比率的影响。为此,应为每个可能的标签位置生成 DNA 表达构建体(图 4C),并应评估它们对 WT 对和已知结合阴性对照的 BRET 比率的影响。

图 1:14-3-3 蛋白对 RAF 的调节。 (A) RAF 激酶结构域结构示意图,显示了保守的 14-3-3 停靠位点。(B) 14-3-3 蛋白的 RAF 调控模型。(C) N-BRET 测定法测量 14-3-3ζ 融合至 C 端 Halo 标签受体和 CRAF 融合至 N 端纳米供体的相互作用。供体到受体的能量转移产生 BRET 信号。 请单击此处查看此图的较大版本。

图 2:执行基于 CRAF:14-3-3 活细胞 BRET 的检测的关键步骤。 显示了该方案中概述的五个主要步骤,包括:接种、转染和重新铺板细胞,以及读取 BRET 信号和确认 Halo 和 Nano 标记蛋白表达水平的方法。 请单击此处查看此图的较大版本。

图 3:CRAF:14-3-3 基于 BRET 的测定。 N-BRET 测定比较了 14-3-3ζ-Halo 与 Nano-CRAF -WT 或 -S259A、-S621A 和 -S259A/S621A (SS/AA) 突变形式的相互作用,先前显示会损害 CRAF:14-3-3 结合25。通过蛋白质印迹分析检测 14-3-3ζ-Halo (MW: 64 kDa.) 和 Nano-CRAF 变体 (MW: 93 kDa.) 的表达水平。(A) 校正了来自单个实验的四个重复孔的 BRET 读数±标准偏差 (SD)。(B) 将来自三个独立实验的校正 BRET 读数合并并归一化为 Nano-CRAFWT (设置为 100)± SD。统计显着性通过学生 t 检验(双尾,假设方差相等)计算。p<0.0001. 请单击此处查看此图的较大版本。
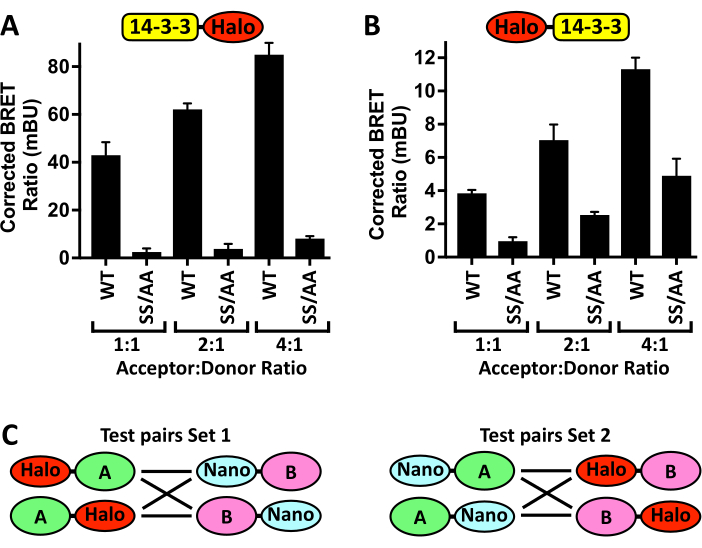
图 4:检测优化步骤。 将增加量的 (A) 14-3-3ζ-Halo 或 (B) Halo-14-3-3ζ 与恒定量的 Nano-CRAF -WT 或 -SS/AA (S259A/S621A) 共转染对 ± SD 的 BRET 信号的影响。(C) 通过将 Halo 或 Nano 标签放置在每种目标蛋白质(A 和 B)的 N 端或 C 端上而生成的所有可能的 N-BRET 对的示意图。n = 4。 请单击此处查看此图的较大版本。
补充表 1:来自 CRAF:14-3-3 基于 BRET 的测定的示例数据。 在第 2 天将指定的表达构建体转染到细胞中(上图)。在第 4 天(下半部分)测量和计算原始、校正和标准化的 BRET 读数。 请点击此处下载此文件。
讨论
先前的研究表明,14-3-3 蛋白在 RAF 激酶的激活和抑制中起关键作用。了解这些结合事件如何被调节以及调节这些相互作用对 RAF 信号转导和 RAF 驱动的肿瘤发生的影响可能会发现针对 CRAF 功能的新治疗脆弱性。然而,Raf 激活周期受到大量相关蛋白、翻译后修饰和亚细胞定位变化的支持8,因此,为了完全概括这些条件,必须在活细胞条件下测量 RAF 与其调节因子的相互作用。该协议描述了一种新颖的基于邻近的测定法,它利用纳米荧光素酶-Halo N-BRET 对的卓越信噪比和光谱分辨率来测量活细胞中 14-3-3ζ 和 CRAF 的相互作用。
虽然该测定为研究 14-3-3 和 CRAF 在生理相关条件下的复杂相互作用提供了一个急需的平台,但它有许多应考虑的局限性。首先,鉴于 14-3-3 已被证明在 RAF 自抑制和二聚化8 中发挥作用,因此可以结合其他对照和实验方法来区分这些状态。例如,该测定可以与先前开发的 N-BRET 自抑制测定法一起进行,该测定法可直接读取 RAF 自抑制变化的读数12,22。此外,CRAF R401H 突变等对照被证明可以破坏 CRAF 与其他 RAF 原聚体的二聚化15,26,也可能被证明可用于剖析这些功能。最后,Halo 和 Nano 标签的放置针对测量 CRAF:14-3-3 相互作用进行了优化,但不能保证 CRAF 或 14-3-3 与其他蛋白质的相互作用不会因此受到影响,该测定也不能确定 CRAF:14-3-3 结合的亲和力、这些相互作用对 CRAF 活性的影响,或这些相互作用在细胞中发生的位置。因此,可能还需要结合其他互补方法,例如体外结合测定、CRAF 激酶测定和亚细胞定位研究 12,27,28,29。
在进行此测定或任何基于 N-BRET 的测定时,需要考虑许多因素。如本手稿和之前的工作22 所示,pCMV5-Halo(受体)和 pCMV5-纳米荧光素酶(供体)构建体的正确转染比率,以及适当的阴性对照,例如已知会破坏 POI 结合的突变体,对于优化和测试这些分析至关重要。增加 Halo 标记的 POI 的表达,同时保持 Nano 标记的 POI 水平恒定,导致 BRET 比率增加。然而,一旦结合饱和,信号将趋于平稳,反过来 WT 和结合受损的 POI 之间的增量将减少,因此需要受体-供体优化。检测优化的另一个关键因素是使用空载体 DNA,例如 pCDNA3.1。包括 200 ng pCDNA3.1。在转染混合物中,可大大增加 Halo 和 Nano 标记的 POI 的表达水平,并降低复制孔和实验之间的变异性22。然而,鉴于试剂和细胞系之间的转染效率可能有所不同,可能还需要滴定载体 DNA 的量以实现 N-BRET 对的最佳表达,如前所述22。另一个对 BRET 比率产生重大影响的因素是标签放置。事实上,哪个 POI 与 Halo 标签融合,哪个 POI 与 Nano 标签融合,以及该标签是否位于蛋白质的 N 端或 C 端,都会影响 BRET 比率的振幅和阴性对照降低该信号的能力。因此,在开发新的检测方法时,测试每个可能的 N-BRET 对也很重要。
与传统 BRET 相比,N-BRET 实现了更高的灵敏度和光谱分辨率,从而可以在活细胞中测量较弱的相互作用和结合的细微变化。然而,这些测定法在研究细胞信号转导事件中的效用尚未完全实现。值得注意的是,该测定可能适用于研究 14-3-3 与其他激酶(如 RAF 激酶 ARAF 和 BRAF)的相互作用,并且当与先前发表的用于研究 RAS:RAF 结合和 RAF 自抑制的 N-BRET 测定相结合时22,30,将构成在活细胞条件下测量 RAF 调节的工具包的基础。此外,N-BRET 已显示出作为活细胞药物筛选平台30 的前景,因此该测定也可用于鉴定通过 14-3-3 和 RAF 相互作用稳定抑制或破坏参与 RAF 激活的化合物。鉴于纳米荧光素酶信号的高稳定性以及现代读板器同时检测受体和供体发射的能力,这些测定也可以适用于实时测量信号蛋白的相互作用,这对于了解这些相互作用响应上游刺激或药物治疗的动力学至关重要。
披露声明
没什么可透露的。
致谢
该项目部分由美国国立卫生研究院国家癌症研究所的联邦资金资助,项目编号为 ZIA BC 010329。
材料
| Name | Company | Catalog Number | Comments |
| Antibodies | |||
| HaloTag® mouse monoclonal antibody | Promega | G9211 | Antibody for detecting HaloTag tagged proteins by immunoblot |
| NanoLuc® mouse monoclonal antibody | R&D Systems | MAB10026 | Antibody for detecting Nano-tagged proteins by immunoblot |
| CRAF mouse monoclonal antibody (E10) | Santa Crus Biotechnology | sc-7267 | Antibody directly detecting CRAF proteins by immunoblot |
| ECL anti-mouse HRP secondary antibody | Amersham | NA931-1ML | Secondary HRP conjugated mouse antibody (from sheep) |
| Reagents | |||
| X-tremeGENE™ 9 | Roche/Sigma | 6365809001 | |
| NanoBRET™ kit | Promega | N1661 | NanoBRET kit containing Halo 618 ligand and NanoGlo (nanoluciferase) substrate |
| DPBS, without Ca++ and Mg++ | Quality Biologicals | 114-057-101 | |
| Trypsin-EDTA (0.05%), phenol red | Life Technologies | 25300120 | |
| DMEM cell culture media | Life Technologies | 11995073 | High glucose, L-glutamine, phenol red, sodium pyruvate; without HEPES, suppliment media with 10% FBS, 2 mM L-glutamine and 100U penicillin-streptomycin |
| L-Glutamine (200 mM) | Life Technologies | 25030164 | |
| Penicillin-Streptomycin (10,000 U/mL) | Life Technologies | 15140163 | |
| Opti-MEM™ I reduced serum media | Gibco | 31985062 | For cell transfection |
| Opti-MEM reduced serum media, no phenol red | Gibco | 11058021 | For replating cells on Day 3. Supplement with 2 mM L-glutamine and 100U penicillin-streptomycin, along with 10% FBS (where indicated). |
| Invitrogen Trypan Blue Stain | Thermo Scientific | T10282 | |
| NP40 lysis buffer | N/A | N/A | 20 mM Tris (pH 8.0), 137mM NaCl, 10% glycerol, NP40 alternative (Milipore, Cat# 492016). Store at 4 degrees C.. Add the following protease and phosphatase immediately prior to use: 20 µM leupeptin, 0.5 mM sodium orthovanidate, 0.15 U/mL, 1mM PMSF. |
| 5x gel sample buffer | N/A | N/A | 240 mM Tris (pH 8.0), 9.5% SDS, 30% glycerol, 500mM DTT, 3mM bromophenol blue. Store at -20 degrees C. |
| Cell lines | |||
| 293FT cells (human) | Thermo Scientific | R70007 | |
| DNA vectors | |||
| pCMV5-Nano-CRAF WT and mutant | N/A | N/A | |
| pCMV5-14-3-3ζ-Halo | N/A | N/A | |
| Equipment | |||
| EnVision 2104 Multimode Plate Reader | PerkinElmer 2104 | 2104-0010 | 600LP NanoBRET & M460/50 nm NanoBRET emmisions filters, Luminescence 404 mirror, 6.5 mm measurement height and 0.1 s measurement time |
| Invitrogen Countess™ II Automated Cell Counter | Thermo Scientific | AMQAX1000 | |
| ThermoFisher E1-ClipTip™ Multichannel Pipettor | Thermo Scientific | 4672070 | |
| Software | |||
| GraphPad Prism (version 10.0.3) | GraphPad | www.graphpad.com | |
| Other | |||
| ThermoFisher ClipTip Multichannel pipette tips | Thermo Scientific | 94410153 | |
| Reagent Reservoir, 25 mL Divided, Sterile | Thomas Scientific | 1228K16 | |
| Perkin Elmer 384-well CulturPlate™ | PerkinElmer | 6007680 | White, polystyrene, tissue culture treated |
| Countess Cell Counting Chamber Slides | Thermo Scientific | C10228 |
参考文献
- Blasco, R. B., et al. c-Raf, but not B-Raf, is essential for development of K-Ras oncogene-driven non-small cell lung carcinoma. Cancer Cell. 19, 652-663 (2011).
- Blasco, M. T., et al. Complete regression of advanced pancreatic ductal adenocarcinomas upon combined inhibition of EGFR and C-RAF. Cancer Cell. 35, 573-587 (2019).
- Karreth, F. A., Frese, K. K., DeNicola, G. M., Baccarini, M., Tuveson, D. A. C-Raf is required for the initiation of lung cancer by K-Ras(G12D). Cancer Discov. 1, 128-136 (2011).
- Lito, P., et al. Disruption of CRAF-mediated MEK activation is required for effective MEK inhibition in KRAS mutant tumors. Cancer Cell. 25, 697-710 (2014).
- Sanclemente, M., et al. c-RAF ablation induces regression of advanced Kras/Trp53 mutant lung adenocarcinomas by a mechanism independent of MAPK signaling. Cancer Cell. 33, 217-228 (2018).
- Razzaque, M. A., et al. Germline gain-of-function mutations in RAF1 cause Noonan syndrome. Nat Genet. 39, 1013-1017 (2007).
- Pandit, B., et al. Gain-of-function RAF1 mutations cause Noonan and LEOPARD syndromes with hypertrophic cardiomyopathy. Nat Genet. 39, 1007-1012 (2007).
- Terrell, E. M., Morrison, D. K. Ras-mediated activation of the Raf family kinases. Cold Spring Harb Perspect Med. 9 (1), 033746(2019).
- Kondo, Y., et al. Cryo-EM structure of a dimeric B-Raf:14-3-3 complex reveals asymmetry in the active sites of B-Raf kinases. Science. 366, 109-115 (2019).
- Park, E., et al. Architecture of autoinhibited and active BRAF-MEK1-14-3-3 complexes. Nature. 575 (7783), 545-550 (2019).
- Tzivion, G., Luo, Z., Avruch, J. A dimeric 14-3-3 protein is an essential cofactor for Raf kinase activity. Nature. 394, 88-92 (1998).
- Spencer-Smith, R., et al. RASopathy mutations provide functional insight into the BRAF cysteine-rich domain and reveal the importance of autoinhibition in BRAF regulation. Mol Cell. 82, 4262-4276 (2022).
- Martinez Fiesco, J. A., Durrant, D. E., Morrison, D. K., Zhang, P. Structural insights into the BRAF monomer-to-dimer transition mediated by RAS binding. Nat Commun. 13, 486(2022).
- Freeman, A. K., Ritt, D. A., Morrison, D. K. The importance of Raf dimerization in cell signaling. Small GTPases. 4, 180-185 (2013).
- Freeman, A. K., Ritt, D. A., Morrison, D. K. Effects of Raf dimerization and its inhibition on normal and disease-associated Raf signaling. Mol Cell. 49, 751-758 (2013).
- Rushworth, L. K., Hindley, A. D., O'Neill, E., Kolch, W. Regulation and role of Raf-1/B-Raf heterodimerization. Mol Cell Biol. 26, 2262-2272 (2006).
- Garnett, M. J., Rana, S., Paterson, H., Barford, D., Marais, R. Wild-type and mutant B-RAF activate C-RAF through distinct mechanisms involving heterodimerization. Mol Cell. 20, 963-969 (2005).
- Tran, N. H., Wu, X., Frost, J. A. B-Raf and Raf-1 are regulated by distinct autoregulatory mechanisms. J Biol Chem. 280, 16244-16253 (2005).
- Chong, H., Guan, K. L. Regulation of Raf through phosphorylation and N terminus-C terminus interaction. J Biol Chem. 278, 36269-36276 (2003).
- Cutler, R. E., Stephens, R. M., Saracino, M. R., Morrison, D. K. Autoregulation of the Raf-1 serine/threonine kinase. Proc Natl Acad Sci U S A. 95, 9214-9219 (1998).
- Park, E., et al. Cryo-EM structure of a RAS/RAF recruitment complex. Nat Commun. 14, 4580(2023).
- Spencer-Smith, R., Morrison, D. K. Protocol for measuring BRAF autoinhibition in live cells using a proximity-based NanoBRET assay. STAR Protoc. 4, 102461(2023).
- Clark, G. J., et al. 14-3-3 zeta negatively regulates raf-1 activity by interactions with the Raf-1 cysteine-rich domain. J Biol Chem. 272, 20990-20993 (1997).
- Machleidt, T., et al. NanoBRET--A novel BRET platform for the analysis of protein-protein interactions. ACS Chem Biol. 10, 1797-1804 (2015).
- Hekman, M., et al. Dynamic changes in C-Raf phosphorylation and 14-3-3 protein binding in response to growth factor stimulation: differential roles of 14-3-3 protein binding sites. J Biol Chem. 279, 14074-14086 (2004).
- Hatzivassiliou, G., et al. RAF inhibitors prime wild-type RAF to activate the MAPK pathway and enhance growth. Nature. 464, 431-435 (2010).
- Bondzi, C., Grant, S., Krystal, G. W. A novel assay for the measurement of Raf-1 kinase activity. Oncogene. 19, 5030-5033 (2000).
- Spencer-Smith, R., et al. Inhibition of RAS function through targeting an allosteric regulatory site. Nat Chem Biol. 13 (1), 62-68 (2016).
- Roy, S., et al. 14-3-3 facilitates Ras-dependent Raf-1 activation in vitro and in vivo. Mol Cell Biol. 18, 3947-3955 (1998).
- Durrant, D. E., et al. Development of a high-throughput NanoBRET screening platform to identify modulators of the RAS/RAF interaction. Mol Cancer Ther. 20, 1743-1754 (2021).
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。