Method Article
Saggio basato sul trasferimento di energia per risonanza di bioluminescenza (BRET) per la misurazione delle interazioni di CRAF con proteine 14-3-3 in cellule vive
In questo articolo
Riepilogo
Questo protocollo descrive un saggio basato su BRET per misurare le interazioni della chinasi CRAF con le proteine 14-3-3 in cellule vive. Il protocollo delinea i passaggi per la preparazione delle celle, la lettura delle emissioni BRET e l'analisi dei dati. Viene inoltre presentato un risultato di esempio con l'identificazione dei controlli appropriati e la risoluzione dei problemi per l'ottimizzazione del saggio.
Abstract
La CRAF è un effettore primario delle GTPasi RAS e svolge un ruolo critico nella tumorigenesi di diversi tumori guidati da KRAS. Inoltre, la CRAF è un hotspot per le mutazioni germinali, che hanno dimostrato di causare la RASopathy evolutiva, la sindrome di Noonan. Tutte le chinasi RAF contengono più siti di legame dipendenti dalla fosforilazione per le proteine regolatorie 14-3-3. Il legame differenziale di 14-3-3 a questi siti gioca un ruolo essenziale nella formazione di dimeri attivi di RAF sulla membrana plasmatica in condizioni di segnalazione e nel mantenimento dell'autoinibizione di RAF in condizioni di quiescenza. Comprendere come queste interazioni sono regolate e come possono essere modulate è fondamentale per identificare nuovi approcci terapeutici che mirano alla funzione dei RAF. Qui, descrivo un saggio basato sul trasferimento di energia per risonanza di bioluminescenza (BRET) per misurare le interazioni di CRAF con le proteine 14-3-3 nelle cellule vive. In particolare, questo test misura le interazioni di CRAF fuso a un donatore di nano luciferasi e 14-3-3 fuso a un accettore di tag Halo, dove l'interazione di RAF e 14-3-3 determina il trasferimento di energia da donatore a accettore e la generazione del segnale BRET. Il protocollo mostra inoltre che questo segnale può essere interrotto da mutazioni che hanno dimostrato di impedire il legame di 14-3-3 a ciascuno dei suoi siti di attracco RAF ad alta affinità. Questo protocollo descrive le procedure per la semina, la trasfetzione e la riplaccatura delle cellule, insieme a istruzioni dettagliate per la lettura delle emissioni di BRET, l'esecuzione dell'analisi dei dati e la conferma dei livelli di espressione proteica. Inoltre, vengono forniti esempi di risultati dei saggi, insieme alle fasi di ottimizzazione e risoluzione dei problemi.
Introduzione
Le chinasi RAF (ARAF, BRAF e CRAF) sono gli effettori diretti delle GTPasi RAS e i membri iniziatori della cascata chinasica RAF-MEK-ERK pro-proliferativa/pro-sopravvivenza. Studi recenti hanno dimostrato che l'espressione di CRAF svolge un ruolo chiave nella tumorigenesi di diversi tumori guidati da KRAS, tra cui il carcinoma polmonare non a piccole cellule e l'adenocarcinoma duttale pancreatico 1,2,3,4,5. Inoltre, le mutazioni germinali della CRAF causano una forma particolarmente grave di RASopathy, la sindrome di Noonan 6,7. Comprendere la regolazione del CRAF è fondamentale per lo sviluppo di approcci terapeutici di successo che mirino alla sua funzione nelle cellule.
Tutte le chinasi RAF possono essere suddivise in due domini funzionali, un dominio catalitico C-terminale (CAT) e un dominio regolatorio N-terminale (REG), che ne controlla l'attività (Figura 1A)8. Il dominio REG comprende il dominio di legame RAS (RBD), il dominio ricco di cisteina (CRD) e una regione ricca di serina/treonina (S/T-rich). In particolare, la regione ricca di S/T contiene il sito N', che si lega a 14-3-3 in modo dipendente dalla fosforilazione (S259 in CRAF; Figura 1A) 8. Il dominio CAT comprende il dominio chinasi, insieme a un secondo sito di docking 14-3-3 ad alta affinità, denominato sito C' (S621 in CRAF; Figura 1A) 8. Il legame differenziale delle proteine dimeriche 14-3-3 ai siti N' e C', insieme alla CRD, svolge un ruolo critico sia nell'attivazione che nell'inibizione di RAF 9,10,11,12,13. In condizioni normali di segnalazione, l'attivazione di RAF è avviata dal suo reclutamento nella membrana plasmatica da parte di RAS, permettendogli di formare dimeri attivi, di cui l'eterodimero BRAF-CRAF è la forma attiva predominante14,15. I saggi biochimici con BRAF e CRAF, insieme alle strutture di microscopia elettronica criogenica (Cryo-EM) di BRAF dimerico, indicano che un dimero 14-3-3 stabilizza i dimeri attivi di RAF legandosi simultaneamente al sito C' di entrambi i protomeri RAF (Figura 1B)9,13,16,17. Al contrario, gli studi hanno dimostrato che in condizioni di quiescenza, RAF adotta una conferma citosolica, autoinibita, in cui il dominio REG si lega al dominio CAT e ne inibisce l'attività 12,18,19,20. Questo stato chiuso è stabilizzato da un dimero 14-3-3 legato al sito CRD e N' nel dominio REG e al sito C' nel dominio CAT (Figura 1B)10,13,21. In BRAF, questo modello è supportato da recenti strutture Cryo-EM di monomeri BRAF autoinibiti e dai nostri precedenti studi biochimici 10,12,13,21,22. Tuttavia, mentre è dimostrato che 14-3-3 svolge un ruolo inibitorio nella regolazione di CRAF23, uno stato autoinibito simile a BRAF può svolgere un ruolo minore nella regolazione di CRAF12; pertanto sono necessari ulteriori studi per chiarire i meccanismi con cui le proteine 14-3-3 regolano l'attività di CRAF. La regolazione mediata da 14-3-3 delle chinasi RAF richiede una pletora di eventi di fosforilazione e de-fosforilazione di RAF, il legame con varie proteine regolatorie e le interazioni con la membrana plasmatica8. Pertanto, è fondamentale che le interazioni 14-3-3-RAF siano misurate in condizioni fisiologicamente rilevanti e in presenza di un doppio strato lipidico intatto.
Per risolvere questo problema, è stata utilizzata la tecnologia NanoBRET (d'ora in poi denominata N-BRET; vedere la Tabella dei materiali per i dettagli del kit) per sviluppare un test basato sulla prossimità per misurare le interazioni di CRAF con le proteine 14-3-3 nelle cellule vive (Figura 1C). Questo sistema basato su BRET misura le interazioni di due proteine di interesse (POI), in cui una proteina è marcata con un donatore di nanoluciferasi (Nano) e l'altra con un tag Halo, per la marcatura con il ligando accettore di energia Halo61822,24. L'interazione delle proteine di interesse si traduce nel trasferimento di energia da donatore a accettore, che a sua volta genera il segnale BRET (Figura 1C). La proteina donatrice Nano estremamente brillante (emissione (em) 460 nm) e il ligando Halo618 (em 618 nm) forniscono una maggiore separazione spettrale e sensibilità rispetto al BRET convenzionale, rendendola una piattaforma ideale per studiare le interazioni più deboli e rilevare sottili cambiamenti nel legame24. Infatti, abbiamo precedentemente sviluppato un test basato su N-BRET per misurare le interazioni autoinibitorie dei domini RAF REG e CAT, che è stato essenziale per la caratterizzazione di un pannello di mutazioni di RASopathy nella CRD di BRAF e ha dimostrato l'importanza critica di questo dominio per mantenere l'autoinibizione e prevenire l'attivazione costitutiva di BRAF12.
Il test qui descritto misura le interazioni tra CRAF, fuso con un tag Nano N-terminale (Nano-CRAF), e l'isoforma zeta di 14-3-3 fusa con il tag Halo C-terminale (14-3-3ζ-Halo; Figura 1C). Dimostriamo che le interazioni di Nano-CRAF con 14-3-3ζ-Halo generano un robusto segnale BRET, che a sua volta può essere interrotto da mutazioni che impediscono il legame di 14-3-3 al sito N' (S259A) e/o al sito C' (S621A). Il seguente protocollo fornisce passaggi dettagliati per l'esecuzione, l'ottimizzazione e la risoluzione dei problemi di questo test.
Protocollo
NOTA: Questo test viene eseguito in celle 293FT. Una linea epiteliale ben caratterizzata e facilmente trasfettabile derivata da cellule renali embrionali umane. Una singola piastra di coltura confluente di 10 cm di queste cellule fornisce in genere cellule sufficienti per la semina di 20 pozzetti di piastre di coltura tissutale a 6 pozzetti. Le fasi 1-3 devono essere eseguite utilizzando una tecnica sterile in una cappa di sicurezza biologica.
1. Semina cellulare (giorno 1)
NOTA: In questa fase, le cellule vengono staccate dalle piastre di coltura tissutale, contate e seminate in piastre di coltura tissutale a 6 pozzetti per la trasfezione nella fase 2 (Figura 2).
- Aspirare il terreno dalle celle nel piatto da 10 cm. Lavare le celle con 5 mL di soluzione salina tamponata con fosfato (PBS) e aspirare.
- Aggiungere 1 mL di acido tripsin-etilendiamminotetraacetico (EDTA) e incubare per 3-5 minuti a 37 °C per staccare le cellule dal piatto.
- Aggiungere 9 mL di terreno eagle modificato (DMEM) completo di Dulbecco alle cellule per neutralizzare la tripsina e pipettare ripetutamente su e giù per generare una sospensione omogenea di singole cellule.
- Trasferire immediatamente 20 μl di cellule in una provetta da 1,7 mL e miscelare con 20 μl di colorante blu di tripano. Contare le cellule utilizzando un contatore automatico di cellule (Tabella dei materiali) o un emocitometro.
- Diluire le cellule a 2 x 105 cellule/mL in terreno DMEM completo e aggiungere 2 mL a ciascun pozzetto di una piastra di coltura tissutale a 6 pozzetti (4 x 105 cellule/pozzetto). Incubare le cellule a 37 °C e 10% di CO2 durante la notte.
NOTA: Si consiglia di utilizzare celle da 293 piedi che sono state passate meno di 20 volte. In precedenza abbiamo scoperto che l'utilizzo di celle con un numero di passaggi maggiore può comportare una riduzione dei rapporti BRET e un aumento della variabilità del segnale da pozzetto a pozzetto.
2. Trasfezione cellulare (Giorno 2)
NOTA: Qui, le cellule vengono trasfettate con i costrutti di espressione pCMV5-NanoLuc-CRAF e pCMV5-14-3-3ζ-Halo, insieme al vettore vuoto pCDNA3.1 (Figura 2).
- Prima della trasfezione, diluire i plasmidi N-BRET a 5 ng/μL e pCDNA3.1 a 100 ng/μL e numerare un set di provette sterili da 1,7 mL.
- Aggiungere 100 μL di terreno di trasfezione (vedere la Tabella dei materiali per i dettagli) a ciascuna provetta, insieme a 5 ng di pCMV5-NanoLuc-CRAF, 10 ng di pCMV5-14-3-3ζ e 200 ng di PCDNA3.1.
- Aggiungere 2 μl di reagente di trasfezione (vedere la Tabella dei materiali per i dettagli) e agitare delicatamente per mescolare. Centrifugare brevemente le provette in una microcentrifuga per assicurarsi che tutto il liquido venga raccolto sul fondo delle provette e quindi incubare a circa 25 °C per 15 minuti.
- Aggiungere complessi di trasfezione goccia a goccia alle cellule e incubare a 37 °C/10% CO2 per 16-20 ore per consentire l'espressione delle proteine marcate con alone e nano.
NOTA: L'aggiunta di un DNA vettore vettore vuoto (pCDNA3.1) è essenziale per ottenere un'elevata efficienza di trasfezione dei costrutti di espressione di N-BRET. La mancata aggiunta di vettore vuoto comporta una riduzione dei livelli di espressione delle proteine 14-3-3ζ-Halo e Nano-CRAF e, a sua volta, si traduce in rapporti BRET deboli e incoerenti, come discusso in precedenza22.
3. Riplaccatura cellulare (Giorno 3)
NOTA: In questa fase, le cellule vengono trasferite su una piastra a 384 pozzetti e il ligando Halo 618 (+ligando; Tabella dei materiali) o DMSO (+veicolo) viene aggiunto per leggere le emissioni BRET nel passaggio 4. Le cellule rimanenti vengono trasferite su piastre di coltura fresche a 6 pozzetti per l'analisi del western blot nella Fase 5 (Figura 2).
- Raccogliere i seguenti materiali e preparare l'area di lavoro come descritto di seguito.
- Utilizzando un bagnomaria a 37 °C, preriscaldare la tripsina-EDTA, insieme a terreni di prova privi di siero e terreni di prova integrati con FBS al 10% (vedere la tabella dei materiali per i dettagli).
- In base al numero di campioni da misurare, pre-etichettare tre set di provette sterili da 1,7 mL (Set 1-3) e un set di provette sterili a fondo conico da 15 mL. Equipaggiare una centrifuga a secchio oscillante con inserti per provette da 15 mL e pre-raffreddare a 4 °C.
- Inserire i seguenti elementi nella cappa di coltura tissutale: serbatoi di reagenti, piastre di coltura tissutale da 384 pozzetti, piastre di coltura tissutale da 6 pozzetti, pipetta multicanale e puntali, vetrini/camere per il conteggio delle cellule e colorazione blu di tripano (Tabella dei materiali), insieme ai set di provette da 1,7 mL 1-3.
- Raccogliere e contare le celle come descritto di seguito.
- Aspirare il terreno dalle piastre a 6 pozzetti e aggiungere 250 μl di tripsina-EDTA alle cellule. Incubare le piastre a 6 pozzetti a 37 °C fino a quando le cellule iniziano a staccarsi (3-5 min).
- Aggiungere 1 mL di terreno di coltura integrato con FBS al 10% in ciascun pozzetto per neutralizzare la tripsina e pipettare energicamente su e giù per generare una sospensione a cellula singola.
- Trasferire 1 mL della sospensione cellulare nelle provette da 15 mL pre-marcate. Aggiungere un altro 1 mL di terreno di prova integrato con FBS al 10% a ciascuna delle provette da 15 mL.
- Capovolgere le provette 5 volte per miscelare e trasferire immediatamente 20 μl di sospensione cellulare nel set di provette 1.
- Centrifugare nella centrifuga a secchiello oscillante pre-raffreddata per 5 minuti a ~250 x g. Durante la fase di centrifugazione, miscelare 20 μL di colorante cellulare blu di tripano con le cellule nel set di provette 1 e quindi contare le cellule utilizzando un contatore di cellule automatizzato o un emocitometro. Una resa di 6-8 x 105 cellule/mL è tipica.
- Rimuovere le provette da 15 mL dalla centrifuga e aspirare il terreno dai pellet cellulari. Risospendere i pellet cellulari a 2 x 105 cellule/mL in terreni di prova privi di siero e pipettare energicamente per generare una sospensione di singola cellula.
NOTA: L'uso di terreni di prova privi di siero viene utilizzato per la quiescenza delle normali vie di segnalazione cellulare.
- Riplaccare le celle in piastre a 384 pozzetti e 6 pozzetti come descritto di seguito.
- Capovolgere più volte le provette da 15 mL per garantire una sospensione cellulare omogenea e trasferire le provette da 500 μL a 1,7 mL dei set 2 e 3 dei set 3.
- Aggiungere 0,5 μl di DMSO (+veicolo) al set 2 e 0,5 μl di ligando Halo 618 (+ligando) al set di provette 3 e pipettare per miscelare.
- Trasferire le sospensioni di cellule +veicolo e +ligando in pozzetti separati di serbatoi di reagenti. Utilizzando la pipetta multicanale (Tabella dei materiali), trasferire 40 μl della sospensione di cellule + veicolo dai serbatoi dei reagenti ai pozzetti quadruplicati della piastra di coltura a 384 pozzetti. Ripetere questo passaggio per le sospensioni di cellule + ligando.
- Trasferire le cellule rimanenti in piastre di coltura fresche a 6 pozzetti. Incubare le piastre da 384 pozzetti e 6 pozzetti per una notte a 37 °C e 5% di CO2.
4. Lettura delle emissioni BRET (Giorno 4)
NOTA: In questa fase il substrato della nanoluciferasi (vedere la Tabella dei materiali per i dettagli) viene aggiunto alle cellule nella piastra di coltura a 384 pozzetti e vengono lette le emissioni dell'accettore N-BRET (618 nm) e del donatore (460 nm) (Figura 2). Vengono quindi calcolati i rapporti BRET corretti.
- Preriscaldare i terreni di prova privi di siero in un bagno d'acqua a 37 °C e scongelare il substrato della nanoluciferasi a 25 °C. Diluire il substrato della nanoluciferasi 1:100 in un terreno di prova privo di siero e trasferirlo in un serbatoio di reagenti. Preparare una quantità sufficiente di questa miscela per aggiungere 10 μl a ciascun pozzetto della piastra a 384 pozzetti, più il 10%-15% di volume extra.
- Utilizzando una pipetta multicanale (Tabella dei materiali), trasferire 10 μl della miscela di substrato in ciascuno dei pozzetti che contengono le cellule nella piastra di coltura a 384 pozzetti. Ruotare delicatamente la piastra per 1 minuto, manualmente o utilizzando uno shaker orbitale.
- Inserire la piastra da 384 pozzetti nel lettore di piastre multimodale e registrare le emissioni di 460 nm e 618 nm per tutti i pozzetti che contengono celle.
NOTA: Per il lettore di piastre multimodale utilizzato per questa fase (vedere la Tabella dei materiali per i dettagli), è stata utilizzata un'altezza di lettura di 6,5 mm, con una misurazione del tempo di lettura di 0,1 s, tuttavia potrebbe essere necessaria un'ulteriore ottimizzazione se si utilizzano altri lettori di piastre. - Calcolare i rapporti BRET grezzi per +veicolo e +ligando in unità milliBRET (mBU) singolarmente, utilizzando la seguente equazione:
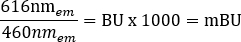
- Calcolare il rapporto BRET corretto sottraendo il rapporto BRET +veicolo da quello del +Ligando (Tabella supplementare 1).
NOTA: Quando si confrontano i risultati di più esperimenti, i rapporti BRET corretti possono essere raggruppati e normalizzati alla media della coppia N-BRET Wildtype (WT), costituita da Nano-CRAFWT e 14-3-3ζWT-Halo (Tabella supplementare 1).
5. Conferma dei livelli di espressione proteica (giorni 4 e 5)
NOTA: In questa fase le cellule nelle piastre a 6 pozzetti vengono lisate e i livelli di espressione proteica delle proteine Nano-CRAF e 14-3-3ζ-Halo vengono determinati mediante analisi western blot utilizzando anticorpi specifici per i tag Halo e Nano. (Figura 2).
- Aggiungere gli inibitori della proteasi e della fosfatasi al tampone di lisi NP40 (Tabella dei materiali), ottenendo 200 μL di tampone di lisi per campione.
- Aspirare i terreni da piastre a 6 pozzetti che contengono cellule. Lavare le cellule una volta con 1 mL di PBS freddo e aspirare.
- Aggiungere 125 μl di tampone di lisi NP40 a ciascun pozzetto e incubare piastre a 6 pozzetti a 4 °C su una piattaforma oscillante per 15 minuti per lisare le cellule.
- Trasferire i lisati in provette da 1,7 mL su ghiaccio e centrifugare a 20.000 x g per 10 minuti a 4 °C. Riposizionare i lisati chiarificati sul ghiaccio e determinare la concentrazione proteica utilizzando saggi disponibili in commercio, come i saggi di Bradford o dell'acido bicinconninico (BCA).
- Normalizzare la concentrazione proteica in tutti i campioni trasferendo un volume appropriato di lisato e tampone di lisi NP40 in provette fresche da 1,7 mL per un volume totale di 100 μL.
- Far bollire 5 tamponi per campioni di proteine (Tabella dei materiali) per 1 minuto e aggiungere 25 μl a ciascun campione. Far bollire i campioni per 5-6 minuti, quindi centrifugare brevemente le provette con impulsi per assicurarsi che tutto il campione si raccolga alla base della provetta.
- Caricare 25 μL di campione in ciascun pozzetto di gel proteici duplicati (Gel 1 e Gel 2) e quindi trasferire le proteine su membrane di nitrocellulosa o PVDF utilizzando le procedure standard di western blotting, come descritto in precedenza22.
- Bloccare le membrane in BSA-PBS al 3% a 25 °C per 30 minuti e quindi incubare le membrane durante la notte con l'anticorpo Halo (diluizione 1:1.000; Gel 1) e anticorpi nanoluciferasi o CRAF (diluizione 1:500; Gel 2) in soluzione salina tris-tamponata, addizionato con Tween-20 allo 0,2% (TBST; Tabella dei materiali).
- Lavare le membrane 1 volta per 5 minuti in 10 mL di TBST e quindi incubare a temperatura ambiente per 1 ora con l'anticorpo secondario anti-topo HRP, diluito 1:10.000 in TBST.
- Lavare le membrane 3 volte su una piattaforma oscillante in 10 mL di TBST a 25 °C per 5 minuti ciascuna. Rimuovere la TBST e visualizzare le bande proteiche utilizzando i reagenti ECL e un processore di pellicole radiografiche o un altro sistema di imaging appropriato.
Risultati
Se eseguita come descritto in questo protocollo (Figura 2), l'interazione di Nano-CRAFWT e 14-3-3ζ-Halo dovrebbe produrre rapporti BRET corretti di 50-60 mBU (Figura 3A; Tabella supplementare 1). CRAF contiene due siti di attracco 14-3-3 dipendenti dalla fosforilazione, il sito N' e il sito C' (Figura 1)8. Pertanto, i controlli appropriati per ridurre il legame CRAF:14-3-3ζ includono S259A e S621A, che impediscono la fosforilazione, e a sua volta il legame 14-3-3, ai siti N' e C', rispettivamente25. Come mostrato nella Figura 3A, il Nano-CRAFS259A riduce il segnale BRET del ~45% e il Nano-CRAFS621A del ~25%, mentre il doppio mutante S259A/S621A (SS/AA) abla quasi completamente il segnale BRET CRAF:14-3-3. Inoltre, i risultati di più esperimenti indipendenti possono essere confrontati normalizzando prima i risultati di ciascun esperimento al rapporto BRET medio corretto (Tabella supplementare 1) e quindi raggruppando i punti dati di tutti e tre gli esperimenti (Figura 3B). I livelli di espressione relativi delle proteine marcate dovrebbero essere confermati anche dall'analisi western blot (Figura 3). Si noti che i livelli di espressione delle proteine mutanti possono variare da quelli di WT, richiedendo che le quantità trasfettate di vettori di DNA mutanti siano regolate di conseguenza (Tabella supplementare 1).
Va inoltre notato che potrebbe essere necessaria l'ottimizzazione di questo protocollo, soprattutto quando si utilizza un lettore di piastre, linee cellulari o reagenti di trasfezione alternativi. La Figura 4A mostra i risultati di un esempio di esperimento di ottimizzazione, in cui aumentando la quantità trasfettata del plasmide accettore pCMV5-14-3-3ζ-Halo, mantenendo costante la quantità di donatore pCMV5-NanoLuc-CRAF, aumenta in modo dose-dipendente il segnale BRET, che viene poi ridotto dalla presenza del mutante CRAFSS/AA deficiente di 14-3-3-binding. Tuttavia, se il rapporto accettore/donatore è troppo alto, la differenza di segnale tra il legame 14-3-3ζ-Halo a Nano-CRAFWT e il controllo negativoNano-CRAF SS/AA sarà ridotta, probabilmente riflettendo la saturazione del legame di Nano-CRAFWT. Inoltre, una volta raggiunto un rapporto BRET che genera valori riproducibili con un basso errore standard, non è necessario un ulteriore aumento del rapporto accettore/donatore. Infatti, uno dei principali vantaggi di questi saggi è la capacità di misurare le interazioni delle proteine a livelli di espressione fisiologica. Quando si sviluppano nuovi saggi N-BRET, un'altra considerazione importante è il posizionamento dei tag. La Figura 4B mostra l'effetto del passaggio del tag Halo dal C-terminale al N-terminale di 14-3-3ζ sui rapporti BRET corretti generati dal legame di 14-3-3ζ a Nano-CRAFWT e Nano-CRAFSS/AA. A tal fine, dovrebbero essere generati costrutti di espressione del DNA per ciascuna delle possibili posizioni di tag (Figura 4C) e dovrebbero essere valutati i loro effetti sui rapporti BRET sia della coppia WT che su quello di un controllo negativo noto per il legame.

Figura 1: Regolazione di RAF da parte di proteine 14-3-3. (A) Schema della struttura del dominio chinasico RAF con indicati i siti di aggancio 14-3-3 conservati. (B) Modelli di regolazione dei RAF da parte di proteine 14-3-3. (C) Saggio N-BRET che misura le interazioni di 14-3-3ζ fuso a un accettore di tag Halo C-terminale e CRAF fuso a un donatore Nano N-terminale. Il trasferimento di energia da donatore a accettore genera il segnale BRET. Clicca qui per visualizzare una versione più grande di questa figura.

Figura 2: Passaggi chiave per l'esecuzione del test CRAF:14-3-3 basato su BRET su cellule vive. Sono mostrati i cinque passaggi principali delineati in questo protocollo, tra cui: semina, trasfetzione e riplaccatura delle cellule, insieme ai metodi per leggere il segnale BRET e confermare i livelli di espressione delle proteine marcate con Halo e Nano. Clicca qui per visualizzare una versione più grande di questa figura.

Figura 3: CRAF:14-3-3 saggio basato su BRET. Saggio N-BRET che confronta le interazioni di 14-3-3ζ-Halo con Nano-CRAF -WT o con le forme mutanti -S259A, -S621A e -S259A/S621A (SS/AA), che hanno dimostrato in precedenza di compromettere il legame CRAF:14-3-325. I livelli di espressione delle varianti 14-3-3ζ-Halo (MW: 64 kDa.) e Nano-CRAF (MW: 93 kDa.) sono stati esaminati mediante analisi western blot. (A) Letture BRET corrette da quattro pozzetti replicati da un singolo esperimento ± deviazione standard (SD). (B) Le letture BRET corrette di tre esperimenti indipendenti sono state raggruppate e normalizzate a Nano-CRAFWT (impostato su 100) ± SD. La significatività statistica è stata calcolata dal test t di Student (due code, assumendo la stessa varianza). pag<0,0001. Clicca qui per visualizzare una versione più grande di questa figura.
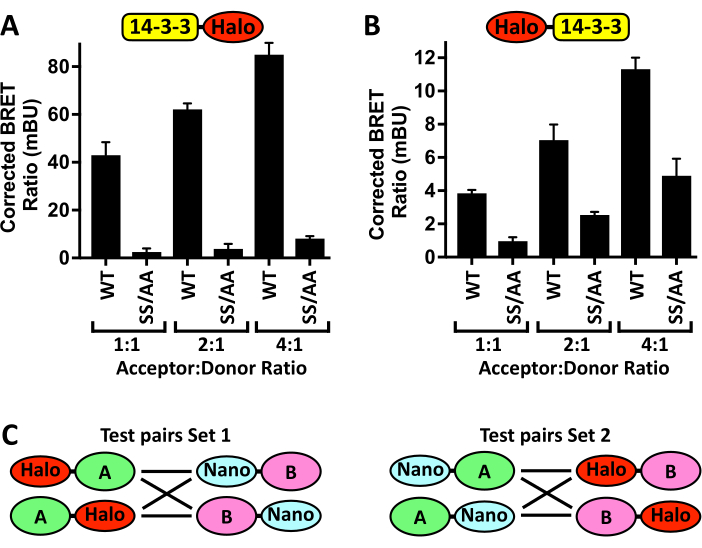
Figura 4: Fasi di ottimizzazione dei saggi. Gli effetti della co-trasfetzione di quantità crescenti di (A) 14-3-3ζ-Halo o (B) Halo-14-3-3ζ con una quantità costante di Nano-CRAF -WT o -SS/AA (S259A/S621A) sul segnale BRET ± SD. (C) Schema di tutte le possibili coppie N-BRET generate posizionando il tag Halo o Nano sull'N-terminale o sul C-terminale di ciascuna proteina di interesse (A e B). n = 4. Clicca qui per visualizzare una versione più grande di questa figura.
Tabella supplementare 1: Dati di esempio dal test basato su BRET CRAF:14-3-3. I costrutti di espressione indicati sono stati trasfettati nelle cellule il giorno 2 (sezione superiore). Le letture BRET grezze, corrette e normalizzate sono state misurate e calcolate il giorno 4 (sezione inferiore). Clicca qui per scaricare questo file.
Discussione
Studi precedenti hanno dimostrato che le proteine 14-3-3 svolgono un ruolo critico sia nell'attivazione che nell'inibizione delle chinasi RAF. Comprendere come questi eventi di legame sono regolati e gli effetti della modulazione di queste interazioni sulla segnalazione RAF e sull'oncogenesi guidata da RAF potrebbe scoprire nuove vulnerabilità terapeutiche che mirano alla funzione di CRAF. Tuttavia, il ciclo di attivazione di Raf è supportato da una pletora di proteine associate, modifiche post-traduzionali e cambiamenti nella localizzazione subcellulare8, e quindi per ricapitolare completamente queste condizioni, le interazioni di RAF con i suoi regolatori devono essere misurate in condizioni di cellule vive. Questo protocollo descrive un nuovo test basato sulla prossimità che utilizza il rapporto segnale/rumore e la risoluzione spettrale superiori della coppia nanoluciferasi-Halo N-BRET per misurare le interazioni di 14-3-3ζ e CRAF in cellule vive.
Sebbene questo test fornisca una piattaforma molto necessaria per studiare le complesse interazioni di 14-3-3 e CRAF in condizioni fisiologicamente rilevanti, presenta una serie di limitazioni che dovrebbero essere considerate. In primo luogo, dato che è stato dimostrato che il 14-3-3 svolge un ruolo sia nell'autoinibizione che nella dimerizzazione della RAF8, possono essere incorporati ulteriori controlli e approcci sperimentali per distinguere tra questi stati. Ad esempio, questo test può essere eseguito in combinazione con il test di autoinibizione N-BRET sviluppato in precedenza, che fornisce una lettura diretta delle variazioni dell'autoinibizione RAF12,22. Inoltre, controlli come la mutazione CRAF R401H, che ha dimostrato di interrompere la dimerizzazione di CRAF con altri protomeri RAF15,26, possono anche rivelarsi utili per sezionare queste funzioni. Infine, il posizionamento dei tag Halo e Nano è ottimizzato per misurare l'interazione CRAF:14-3-3, ma non può garantire che le interazioni di CRAF o 14-3-3 con altre proteine non siano influenzate di conseguenza, né questo test può determinare l'affinità del legame CRAF:14-3-3, gli effetti di queste interazioni sull'attività di CRAF o la posizione nella cellula in cui si verificano queste interazioni. Pertanto, potrebbe essere necessario incorporare anche altri approcci complementari, come i saggi di legame in vitro, i saggi di CRAF chinasi e gli studi di localizzazione subcellulare 12,27,28,29.
Quando si esegue questo test, o qualsiasi test basato su N-BRET, ci sono una serie di fattori da considerare. Come mostrato in questo manoscritto, e nel precedente lavoro22, il corretto rapporto di trasfezione dei costrutti pCMV5-Halo (accettore) e pCMV5-Nanoluciferasi (donatore), insieme a un appropriato controllo negativo, come un mutante noto per interrompere il legame dei POI, è fondamentale per ottimizzare e testare questi saggi. Aumentando l'espressione dei POI marcati Halo, mantenendo costanti i livelli di POI marcati Nano, si ottiene un aumento dei rapporti BRET. Tuttavia, una volta che il legame è saturo, il segnale si stabilizzerà e, a sua volta, il delta tra il WT e i POI con problemi di legame sarà ridotto, rendendo necessaria l'ottimizzazione accettore-donatore. Un altro fattore critico nell'ottimizzazione dei saggi è l'uso di un DNA vettore vettore vuoto, come pCDNA3.1. L'inclusione di 200 ng di pCDNA3.1. nella miscela di trasfezione aumenta notevolmente i livelli di espressione dei POI marcati con Halo- e Nano- e riduce la variabilità tra i pozzetti replicati e gli esperimenti22. Tuttavia, dato che l'efficienza di trasfezione può variare tra reagenti e linee cellulari, potrebbe essere necessario titolare la quantità di DNA vettore per ottenere un'espressione ottimale della coppia N-BRET, come descritto in precedenza22. Un altro fattore che influisce notevolmente sui rapporti BRET è il posizionamento dei tag. Infatti, quale POI è fuso con il tag Halo rispetto al tag Nano e se questo tag è posizionato sull'estremità N o C della proteina può influenzare sia l'ampiezza del rapporto BRET che la capacità dei controlli negativi di ridurre questo segnale. Pertanto, è anche importante testare ciascuna delle possibili coppie N-BRET quando si sviluppano nuovi saggi.
La migliore sensibilità e risoluzione spettrale ottenute da N-BRET rispetto al BRET convenzionale consente di misurare interazioni più deboli e sottili cambiamenti nel legame nelle cellule vive. Tuttavia, l'utilità di questi saggi per lo studio degli eventi di segnalazione cellulare non è stata pienamente compresa. In particolare, questo test potrebbe probabilmente essere adattato per studiare l'interazione di 14-3-3 con altre chinasi, come le altre chinasi RAF ARAF e BRAF, e se combinato con i saggi N-BRET precedentemente pubblicati per lo studio del legame RAS:RAF e dell'autoinibizione RAF22,30, costituirà la base di un kit di strumenti per misurare la regolazione di RAF in condizioni di cellule vive. Inoltre, N-BRET si è dimostrato promettente come piattaforma di screening di farmaci su cellule vive30, e quindi questo test può anche essere utile per identificare composti che stabilizzano l'inibitorio con le interazioni di 14-3-3 e RAF o interrompono quelli coinvolti nell'attivazione di RAF. Data l'elevata stabilità del segnale della nanoluciferasi e la capacità dei moderni lettori di piastre di rilevare simultaneamente le emissioni dell'accettore e del donatore, ne conseguirebbe anche che questi saggi possono essere adattati per misurare le interazioni delle proteine di segnalazione in tempo reale, il che sarà fondamentale per comprendere la dinamica di queste interazioni in risposta alla stimolazione a monte o ai trattamenti farmacologici.
Divulgazioni
Nulla da rivelare.
Riconoscimenti
Questo progetto è stato finanziato in parte con fondi federali del National Cancer Institute, National Institutes of Health, con il numero di progetto ZIA BC 010329.
Materiali
| Name | Company | Catalog Number | Comments |
| Antibodies | |||
| HaloTag® mouse monoclonal antibody | Promega | G9211 | Antibody for detecting HaloTag tagged proteins by immunoblot |
| NanoLuc® mouse monoclonal antibody | R&D Systems | MAB10026 | Antibody for detecting Nano-tagged proteins by immunoblot |
| CRAF mouse monoclonal antibody (E10) | Santa Crus Biotechnology | sc-7267 | Antibody directly detecting CRAF proteins by immunoblot |
| ECL anti-mouse HRP secondary antibody | Amersham | NA931-1ML | Secondary HRP conjugated mouse antibody (from sheep) |
| Reagents | |||
| X-tremeGENE™ 9 | Roche/Sigma | 6365809001 | |
| NanoBRET™ kit | Promega | N1661 | NanoBRET kit containing Halo 618 ligand and NanoGlo (nanoluciferase) substrate |
| DPBS, without Ca++ and Mg++ | Quality Biologicals | 114-057-101 | |
| Trypsin-EDTA (0.05%), phenol red | Life Technologies | 25300120 | |
| DMEM cell culture media | Life Technologies | 11995073 | High glucose, L-glutamine, phenol red, sodium pyruvate; without HEPES, suppliment media with 10% FBS, 2 mM L-glutamine and 100U penicillin-streptomycin |
| L-Glutamine (200 mM) | Life Technologies | 25030164 | |
| Penicillin-Streptomycin (10,000 U/mL) | Life Technologies | 15140163 | |
| Opti-MEM™ I reduced serum media | Gibco | 31985062 | For cell transfection |
| Opti-MEM reduced serum media, no phenol red | Gibco | 11058021 | For replating cells on Day 3. Supplement with 2 mM L-glutamine and 100U penicillin-streptomycin, along with 10% FBS (where indicated). |
| Invitrogen Trypan Blue Stain | Thermo Scientific | T10282 | |
| NP40 lysis buffer | N/A | N/A | 20 mM Tris (pH 8.0), 137mM NaCl, 10% glycerol, NP40 alternative (Milipore, Cat# 492016). Store at 4 degrees C.. Add the following protease and phosphatase immediately prior to use: 20 µM leupeptin, 0.5 mM sodium orthovanidate, 0.15 U/mL, 1mM PMSF. |
| 5x gel sample buffer | N/A | N/A | 240 mM Tris (pH 8.0), 9.5% SDS, 30% glycerol, 500mM DTT, 3mM bromophenol blue. Store at -20 degrees C. |
| Cell lines | |||
| 293FT cells (human) | Thermo Scientific | R70007 | |
| DNA vectors | |||
| pCMV5-Nano-CRAF WT and mutant | N/A | N/A | |
| pCMV5-14-3-3ζ-Halo | N/A | N/A | |
| Equipment | |||
| EnVision 2104 Multimode Plate Reader | PerkinElmer 2104 | 2104-0010 | 600LP NanoBRET & M460/50 nm NanoBRET emmisions filters, Luminescence 404 mirror, 6.5 mm measurement height and 0.1 s measurement time |
| Invitrogen Countess™ II Automated Cell Counter | Thermo Scientific | AMQAX1000 | |
| ThermoFisher E1-ClipTip™ Multichannel Pipettor | Thermo Scientific | 4672070 | |
| Software | |||
| GraphPad Prism (version 10.0.3) | GraphPad | www.graphpad.com | |
| Other | |||
| ThermoFisher ClipTip Multichannel pipette tips | Thermo Scientific | 94410153 | |
| Reagent Reservoir, 25 mL Divided, Sterile | Thomas Scientific | 1228K16 | |
| Perkin Elmer 384-well CulturPlate™ | PerkinElmer | 6007680 | White, polystyrene, tissue culture treated |
| Countess Cell Counting Chamber Slides | Thermo Scientific | C10228 |
Riferimenti
- Blasco, R. B., et al. c-Raf, but not B-Raf, is essential for development of K-Ras oncogene-driven non-small cell lung carcinoma. Cancer Cell. 19, 652-663 (2011).
- Blasco, M. T., et al. Complete regression of advanced pancreatic ductal adenocarcinomas upon combined inhibition of EGFR and C-RAF. Cancer Cell. 35, 573-587 (2019).
- Karreth, F. A., Frese, K. K., DeNicola, G. M., Baccarini, M., Tuveson, D. A. C-Raf is required for the initiation of lung cancer by K-Ras(G12D). Cancer Discov. 1, 128-136 (2011).
- Lito, P., et al. Disruption of CRAF-mediated MEK activation is required for effective MEK inhibition in KRAS mutant tumors. Cancer Cell. 25, 697-710 (2014).
- Sanclemente, M., et al. c-RAF ablation induces regression of advanced Kras/Trp53 mutant lung adenocarcinomas by a mechanism independent of MAPK signaling. Cancer Cell. 33, 217-228 (2018).
- Razzaque, M. A., et al. Germline gain-of-function mutations in RAF1 cause Noonan syndrome. Nat Genet. 39, 1013-1017 (2007).
- Pandit, B., et al. Gain-of-function RAF1 mutations cause Noonan and LEOPARD syndromes with hypertrophic cardiomyopathy. Nat Genet. 39, 1007-1012 (2007).
- Terrell, E. M., Morrison, D. K. Ras-mediated activation of the Raf family kinases. Cold Spring Harb Perspect Med. 9 (1), 033746(2019).
- Kondo, Y., et al. Cryo-EM structure of a dimeric B-Raf:14-3-3 complex reveals asymmetry in the active sites of B-Raf kinases. Science. 366, 109-115 (2019).
- Park, E., et al. Architecture of autoinhibited and active BRAF-MEK1-14-3-3 complexes. Nature. 575 (7783), 545-550 (2019).
- Tzivion, G., Luo, Z., Avruch, J. A dimeric 14-3-3 protein is an essential cofactor for Raf kinase activity. Nature. 394, 88-92 (1998).
- Spencer-Smith, R., et al. RASopathy mutations provide functional insight into the BRAF cysteine-rich domain and reveal the importance of autoinhibition in BRAF regulation. Mol Cell. 82, 4262-4276 (2022).
- Martinez Fiesco, J. A., Durrant, D. E., Morrison, D. K., Zhang, P. Structural insights into the BRAF monomer-to-dimer transition mediated by RAS binding. Nat Commun. 13, 486(2022).
- Freeman, A. K., Ritt, D. A., Morrison, D. K. The importance of Raf dimerization in cell signaling. Small GTPases. 4, 180-185 (2013).
- Freeman, A. K., Ritt, D. A., Morrison, D. K. Effects of Raf dimerization and its inhibition on normal and disease-associated Raf signaling. Mol Cell. 49, 751-758 (2013).
- Rushworth, L. K., Hindley, A. D., O'Neill, E., Kolch, W. Regulation and role of Raf-1/B-Raf heterodimerization. Mol Cell Biol. 26, 2262-2272 (2006).
- Garnett, M. J., Rana, S., Paterson, H., Barford, D., Marais, R. Wild-type and mutant B-RAF activate C-RAF through distinct mechanisms involving heterodimerization. Mol Cell. 20, 963-969 (2005).
- Tran, N. H., Wu, X., Frost, J. A. B-Raf and Raf-1 are regulated by distinct autoregulatory mechanisms. J Biol Chem. 280, 16244-16253 (2005).
- Chong, H., Guan, K. L. Regulation of Raf through phosphorylation and N terminus-C terminus interaction. J Biol Chem. 278, 36269-36276 (2003).
- Cutler, R. E., Stephens, R. M., Saracino, M. R., Morrison, D. K. Autoregulation of the Raf-1 serine/threonine kinase. Proc Natl Acad Sci U S A. 95, 9214-9219 (1998).
- Park, E., et al. Cryo-EM structure of a RAS/RAF recruitment complex. Nat Commun. 14, 4580(2023).
- Spencer-Smith, R., Morrison, D. K. Protocol for measuring BRAF autoinhibition in live cells using a proximity-based NanoBRET assay. STAR Protoc. 4, 102461(2023).
- Clark, G. J., et al. 14-3-3 zeta negatively regulates raf-1 activity by interactions with the Raf-1 cysteine-rich domain. J Biol Chem. 272, 20990-20993 (1997).
- Machleidt, T., et al. NanoBRET--A novel BRET platform for the analysis of protein-protein interactions. ACS Chem Biol. 10, 1797-1804 (2015).
- Hekman, M., et al. Dynamic changes in C-Raf phosphorylation and 14-3-3 protein binding in response to growth factor stimulation: differential roles of 14-3-3 protein binding sites. J Biol Chem. 279, 14074-14086 (2004).
- Hatzivassiliou, G., et al. RAF inhibitors prime wild-type RAF to activate the MAPK pathway and enhance growth. Nature. 464, 431-435 (2010).
- Bondzi, C., Grant, S., Krystal, G. W. A novel assay for the measurement of Raf-1 kinase activity. Oncogene. 19, 5030-5033 (2000).
- Spencer-Smith, R., et al. Inhibition of RAS function through targeting an allosteric regulatory site. Nat Chem Biol. 13 (1), 62-68 (2016).
- Roy, S., et al. 14-3-3 facilitates Ras-dependent Raf-1 activation in vitro and in vivo. Mol Cell Biol. 18, 3947-3955 (1998).
- Durrant, D. E., et al. Development of a high-throughput NanoBRET screening platform to identify modulators of the RAS/RAF interaction. Mol Cancer Ther. 20, 1743-1754 (2021).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon