Method Article
Одноклеточных Quantitation мРНК и поверхности белков в инфицированных Обезьяний вирус иммунодефицита CD4+ T клетки изолированы от макак резус
В этой статье
Резюме
Описал представляет собой методологию для quantitate экспрессии генов, 96 и 18 поверхностных белков в одиночных клетках ex vivo, позволяя для идентификации дифференциально выразил генов и белков в инфицированных вирусом клеток по отношению к неинфицированных клеток. Мы применяем подход к изучению SIV-инфицированных CD4+ T клетки изолированы от макак резус.
Аннотация
Сингл элементный анализ является важным инструментом для рассечения гетерогенной популяции клеток. Выявление и изоляция редких клеток может быть затруднено. Для решения этой задачи, методология сочетания индексируются проточной цитометрии и высок объём мультиплексированных количественных полимеразной цепной реакции (ПЦР) была разработана. Цель заключалась в выявлении и характеризуют обезьяний иммунодефицита (СИВ)-инфицированные клетки присутствуют в пределах макак резус. Через количественный поверхностных белков путем активации флуоресценции клеток сортируя (FACS) и мРНК, ПЦР инфицированные вирусом клетки определяются выражением вирусных генов, который в сочетании с принимающей генов и белков измерений для создания многомерных профиля . Мы называем подход, целенаправленных одноклеточных Proteo-transcriptional оценки или tSCEPTRE. Для выполнения метода, жизнеспособных клеток запятнаны с флуоресцентных антител специфических для поверхностных маркеров, используемых для изоляции СУИМ подмножества ячеек и/или течению фенотипического анализа. Отдельные ячейки сортируются после немедленного лизиса, мультиплекс обратная транскрипция (RT), предварительно амплификации PCR и ПЦР высокую пропускную способность до 96 стенограмм. СУИМ измерений записываются во время сортировки и впоследствии связан с данными выражения гена хорошо позиции для создания комбинированного белка и транскрипционный анализ профиля. Для изучения SIV-инфицированных клеток непосредственно ex vivo, клетки были определены ПЦР обнаружение нескольких видов вирусной РНК. Сочетание вирусных стенограммы и количество каждого обеспечивают основу для классификации клеток в различных стадиях вирусный жизненного цикла (например, производственной и непроизводственной). Кроме того tSCEPTRE SIV+ клеток были по сравнению с неинфицированных клеток, изолированные от же образца для оценки дифференциально выраженной хост генов и белков. Результаты анализа показали ранее непонятыми вирусной РНК выражение гетерогенность среди инфицированных клеток, а также в естественных условиях регулирования SIV-опосредованной post-transcriptional гена с одной ячейкой резолюции. Метод tSCEPTRE является актуальной для анализа любой популяции клеток, поддаются идентификации выражением поверхности белка marker(s), принимающих или патогена gene(s) или их комбинации.
Введение
Многих внутриклеточных возбудителей полагаются на принимающей ячейки механизма репликации, часто изменяя хост клеточной биологии или ориентация весьма специфические субпопуляции клеток хозяина, чтобы максимизировать шансы распространения. В результате биологические процессы клеток обычно разрушаются, пагубные последствия для общего состояния здоровья хоста. Понимание взаимодействий между вирусами и клеток хозяина, в которых они повторить будет выяснить механизмы заболеваний, которые могут помочь в разработке улучшения лечения и стратегий для предотвращения инфекции. Прямые аналитические инструменты, которые позволяют изучение взаимодействия хост возбудитель важное значение этой цели. Сингл элементный анализ обеспечивает единственным средством однозначно отнести клеточном фенотипу в частности генотип, или инфекции статус1. К примеру патогенных инфекций часто вызывают прямые и косвенные изменения в клетки хозяина. Таким образом, различие инфицированных клеток от их неинфицированных партнеров необходимо для изменения атрибутов принимающей ячейки прямой инфекции или вторичные эффекты, такие как обобщенные воспаления. Кроме того для многих патогенов, таких как Сив и вирусам иммунодефицита человека (ВИЧ), принимающей ячейки инфекции проходит через несколько этапов, таких как рано, поздно, или скрытая, каждый из которых может характеризоваться отдельных генов и белков выражение профили2 , 3 , 4 , 5. анализ сыпучих смесей клеток удастся захватить это неоднородность6. Напротив, весьма мультиплексированных одноклеточных анализирует возможность количественно оценить выражение как вирусный и принимающих гены предлагают средства для устранения инфекции конкретных клеточных возмущений, включая вариации через стадии инфекции. Кроме того анализ взаимодействия хост возбудителя в физиологически соответствующие параметры имеет решающее значение для выявления событий, которые происходят в зараженных организмов. Таким образом методы, которые могут быть применены непосредственно ex vivo , скорее всего, в лучший захват в естественных условиях процессы.
Сив и ВИЧ целевых CD4+ T клетки, в которых они противодействовать хост противовирусное «ограничение» факторы и помощью антиген представляющих молекул по созданию продуктивной инфекции и избежать иммунной наблюдения7,8, 9,10,11. Без лечения, инфекция приводит к массовой гибели CD4 клеток+ T, в конечном итоге кульминацией приобретенного иммунодефицита (СПИД)12. В параметре антиретровирусной терапии, скрытно инфицированной клетки водохранилищ сохраняются на протяжении десятилетий, что создает серьезное препятствие для лечебной стратегиями. Понимание свойств в vivo ВИЧ/SIV-инфицированных клеток имеет потенциал раскрыть принимающей ячейки особенности важную роль в патогенезе и настойчивость. Однако это весьма сложным, прежде всего благодаря low frequency инфицированных клеток и нехватка реагентов возможность легко идентифицировать их. Клетки, которые транскрибировать вирусной РНК, оцениваются присутствовать на 0,01 – 1% CD4+ T-клеток в крови и лимфоидной ткани13,14,15. При подавляющей терапии скрытно инфицированные клетки даже реже, 10-3– 10-7 16,17,18. Вирусных белков пятнать assays, что хорошо работать для изучения в пробирке инфекций, таких как внутриклеточных кляп, субоптимальный вследствие окрашивания фона 0,01 – 0,1%, аналогичные или больше, чем частота инфицированных клеток13, 14. Пятнать поверхности для Env протеина используя хорошо изученных SIV ВИЧ/Env-моноклональные антитела также было доказано быть трудно, вероятно, по тем же причинам. Недавно, Роман инструменты призваны улучшить обнаружение клеток, выражая кляп, либо включения assays для кляп РНК или с помощью альтернативных изображений технологий14,15,19. Однако такие подходы по-прежнему ограничены в число количественных измерений выполняется для каждой ячейки.
Здесь мы описываем методологии (1) идентифицирует отдельные инфицированные вирусом клетки непосредственно ex vivo чувствительных и конкретных вирусных генов Количественная ПЦР и (2) количественное выражение до 18 поверхностных белков и 96 генов для каждого инфицированных (и неинфицированным) клеток. Эта методология сочетает поверхности белка одноклеточных измерение СУИМ, следуют лизис немедленного клеток и с помощью анализа выражения гена мультиплексированных целевых ПЦР на системе Biomark. Микросхема аэрогидродинамических (МФК) технология позволяет мультиплексированных количественный генов 96 из 96 образцов одновременно, проделанную матрицу 9216 камер, в которых выполняются отдельные ПЦР-реакции. Живой клетки СУИМ Сортировка записей высоким содержанием белка изобилие измерений при сохранении всей транскриптом для анализа сразу же вниз по течению. Для выявления инфицированных вирусом клеток, конкретных анализов для вирусной РНК альтернативно сращивания и unspliced (vRNA), включены в анализ ПЦР, вместе с группой определяемых пользователем анализов, на общую сумму до 96 генов, максимальное количество анализов, в настоящее время размещены в МФК. Экспрессия генов и белков информацию, собранную для каждой ячейки связаны также позиции. Мы сообщалось ранее результаты этого анализа других20. Здесь, мы предоставляем более подробные методологические руководящие принципы, а также дополнительные описательные фенотипирование SIV-инфицированных CD4+ T клетки.
Этот подход, который мы называем tSCEPTRE, могут применяться к суспензии любых жизнеспособных клеток населения реактивной дневно обозначенные антитела и выражая транскриптом совместим с доступных ПЦР-анализов. Например она может использоваться для характеристики дифференциальных генов и белков в редких ячейки или ячейки, не легко отличают поверхности белка маркеров. Подготовка образца опирается на стандарт пятнать протокола с использованием коммерчески доступных антител. Цитофлуориметрами с возможностью сортировки одноклеточных коммерчески доступны, но биобезопасности дополнительные меры предосторожности, необходимые для обработки инфекционных живых клеток. Запись одноклеточного белка выражение профиль для каждой ячейки по позиции, а также упомянутые в настоящем документе как индексированных сортировки, является общей чертой всех коммерчески доступных СУИМ, сортировка программного обеспечения. Вычислительный анализ генов дифференциально выраженной хост среди популяции клеток интерес не описано здесь, но ссылки предоставляются для опубликованных ранее методов.
протокол
Примечание: Схема протокола рабочего процесса показан на рисунке 1. Он состоит из трех основных этапов: СУИМ, RT и предварительно амплификация cDNA и ПЦР для до 96 генов одновременно. Две версии протокола, сортировка клеток в ограничении разведений и сортировка единичных клеток, описаны более подробно в шаге 5 и 6, соответственно. Эти стратегии различные исследовательские вопросы, но следовать аналогичным процедурам.
1. предварительные или предварительного анализа
- Проверка всех анализов выражение гена использоваться как описано6.
Примечание: Этот шаг делается заблаговременно эксперимент. Проверка всех анализов, коммерческих и пользовательских, необходимые для обеспечения эффективного и линейные усилители соответствующих РНК до уровня одной ячейки. Многие коммерчески доступных и пользовательских анализов не отвечают этим требованиям. Обработка и автоматизированного построения кривой для одновременного квалификации выражение анализов до 96 генов предоставляются в Дополнительного кодирования файлов 1 – 5, но отдельные анализы могут быть квалифицированы с помощью R2 и наклон линейной fit. Представитель успешные и неудачные пробы квалификации участков приведены на рисунке 2. -
Разработка потока гранулярных группа антител к запятнать клетки поверхностных маркеров, представляющих интерес.
- Титруйте антитела путем пятнать соответствующий образец, например, резус периферической крови мононуклеаров (получения), с каждого антитела. Начать с 20 мкл антител на тест в 100 мкл пятнать реакции и создание восьми серийных разведений два раза. Определите оптимальную концентрацию, обладающая максимальной пятнать интенсивности при сохранении четкое разделение между населением негативные и позитивные.
- Оцените, комбинированные окрашивания на представленном(ых) дополнительные ячейки, используя смесь всех антител в оптимальной концентрации, определенный на шаге 1.2.1. Убедитесь, что окрашивание близка к соотношению для отдельных антител пятна. Если окрашивание меньше, чем то, что наблюдается, когда любой антитела был использован в изоляции, рассмотрите альтернативные флюрохром конъюгатов для замены таких антител.
2. ген выражение анализа подготовка
- Комбинат 96 экспрессии генов assays в РНКазы/DNase бесплатно 1-15 мл (размер может варьироваться с количество сортировка пластин). Группа анализов, используемые в данном исследовании указывается в таблице 1 и таблице 2. Полученный материал называется «Assay микс». Добавить калибровочных в конечной концентрации 180 Нм праймеров вперед и назад. Добавьте буфер подвески ДНК для достижения соответствующей разбавления смеси Assay.
Примечание: Для практических целей, пользовательские (пользователя генерируемые) пробирного запасы могут быть готовы на 18 мкм согласовываться с концентрацией коммерчески доступных «x 20» ген выражение ПЦР анализов (Таблица материалов). 18 мкм смеси пользовательских прямого и обратного грунтовка для каждого гена, сделаны из запасов решений в буфере подвески ДНК. Коммерчески доступных анализов (Таблица материалов) также включают датчики, но зонды не требуются для RT или предварительно амплификация cDNA и может таким образом быть опущен для пользовательских анализов. Рекомендуется включать один или более уборка генов для использования контроля качества для оценки эффективности сортировки, восстановления клеток и синтез cDNA. Использование случайных праймеров для создания cDNA не была определена, но, как ожидается, будет менее эффективным, чем гена специфические праймеры. - Подготовьте 2 x пробирного пластины для использования на этапе 7.1 (мультиплексной ПЦР). Для каждого массива чип 96 x 96, предполагалось Пипетка 6 мкл калибровочных в каждый места для хорошо из 96-луночных ПЦР-планшете. Например для 5 фишек, 30 мкл калибровочных будет занимать один колодец в 96-луночных пластины. При использовании 96 анализов, каждый хорошо 96-луночных пластины будет содержать assay. Уплотнение пластину с клеевой прокладкой.
Примечание: В идеале, 2.1 и 2.2 выполняются действия одновременно, чтобы избежать нескольких циклов замораживания оттаивания для анализов выражения гена. Все гены в пластину пробирного должны также были в Assay Mix (шаг 2.1). Сочетание пробирного и пробирного пластина может храниться при-20 ° C или 4 ° C для долгосрочного или краткосрочного использования, соответственно.
3. поверхностные пятна жизнеспособных клеток
Примечание: Внутриклеточный пятнать, permeabilization и фиксация не совместимы с помощью этого метода как они компромисс РНК.
- Подготовьте образцы компенсации путем добавления каждого антитела, перечисленных в Таблица материалов до 40 мкл компенсации бусины в 2,5 раза более высокой концентрации, чем используется для окрашивания клеток. Проинкубируйте втечение 20 мин при 25 ° C, защищать от света. Добавить 3 мл PBS бусы и центрифуги на 500 x g 3 мин при 25 ° C. Аспирационная PBS и Ресуспензируйте бисер в ~ 300 мкл PBS.
- Подготовьте потока гранулярных клеток сортировщик для обработки образца: получить компенсацию трубы, создание матрицы компенсации и применять матрицы на приобретение файлы для экспериментальных образцов.
- Подготовка мастер смесь флуоресцентные антитела коктейль, объединяя соответствующие объем каждого антитела, как указано в Таблице материалов, в 1,5 мл янтаря трубки для всех образцов быть запятнано. Вортекс и центрифуги коктейль в 21000 х g на 2 мин при 25 ° C для пеллет антитела агрегатов.
Примечание: Антитела, используемые здесь перечислены в Таблице материалов. - Криоконсервированных клеток в ванну воды 37 ° C на 2 мин добавить 0,5-2 мл суспензии клеток до 12 мл PBS в 15 мл трубку, центрифуги на 500 x g 3 мин при 25 ° C, оттепель и аспирационная PBS. Ресуспензируйте в 3 мл PBS и передачи в 5 мл трубку из полистирола. Центрифуга как выше и аспирационная PBS, оставляя ~ 20 мкл остаточного PBS.
Примечание: Окрашивание температура может быть адаптирована к теплее или холоднее температуры, необходимые для конкретных приложений, изменив антитела титрования, окрашивание условий соответственно (см. шаг 1.2). - Ресуспензируйте до 2 x 107 промывают клетки в 80 мкл антител коктейль и проинкубируйте втечение 20 мин при 25 ° C, защищать от света. Для проб, превышающих 2 x 107 клеток, увеличьте окрашивание реакции соответственно для поддержания < 2 x 107 клеток/100 мкл.
- Вымыть клетки путем добавления 3 мл PBS, центрифугирование на 500 x g на 3 мин и аспирационных супернатант.
- Тщательно Ресуспензируйте клетки в 300 – 500 мкл PBS и фильтра, закупорить через крышкой ситечко нейлон ячейки 35 мкм. Держите клетки на льду и защищены от света до сортировки.
4. Подготовьте ячейки коллекции пластины, выполнения сортировки СУИМ и генерировать cDNA
- Объединить компоненты смеси реакции RT-предусилитель (Таблица 3), дозирование в единый стерильную пробирку РНКазы/DNAse бесплатно.
Примечание: Этот шаг может производиться до или во время окрашивания в шаге 3. Чтобы определить вклад шаблон ДНК, ПЦР сигнал здесь можно опускать RT фермента. - Используйте многоканальные пипетки обойтись 10 мкл RT-предусилитель реакция смеси в желаемое количество 96-луночных ПЦР сортировка коллекции пластины. Печать пластины с липкой пленкой и поместите пластины на предварительно охлажденные 96-луночных алюминиевых блоков цилиндров.
- Создание ячеек, сортировка стробирования схема на проточный цитометр путем приобретения данных из примерно 20 000 клеток окрашенных образца. Убедитесь, что матрица компенсации применяется для собранных данных. Нарисуйте ворота и определить стробирования дерево, которое идентифицирует ячейку группе(ах) интерес быть изолированы для анализа выражения гена.
Примечание: Стробирования дерево, используемое для коллекции потенциальных SIV vRNA+ клеток показана на рисунке 3. - Введите соответствующий инструмент настройки для указания числа и подмножество ячеек должен быть отсортирован в каждой скважине. Дополнительные подробные инструкции для сортировки либо ограничивающих серии разрежения ячейки или отдельные ячейки предоставляются в шаги 5 и 6, соответственно.
- СУИМ сортировать клетки в подготовленных 96-луночных ПЦР коллекции пластины. Удалить клей уплотнение до сортировки и заменить свежим печать после сортировки.
Примечание: Держите пластины на предварительно охлажденные алюминиевых блоков во все времена, в том числе во время сортировки. - Сразу же после сортировки, вихрь и центрифуги коллекции пластины на 2000 x g 1 мин при 4 ° C.
- Thermocycle пластины в машину ПЦР с разогретой крышкой, с использованием следующих условий: 50 ° C 15 мин (RT), 95 ° C в течение 2 мин, после чего 18 циклов 95 ° c 15 s и 60 ° C в течение 4 мин (предварительного усиления).
- Разбавьте cDNA 1:5 путём перевода 5 мкл cDNA в 20 мкл буфера подвески ДНК в новой 96-луночных ПЦР-планшете. Разреженных cDNA могут храниться при 4 ° С или 20 ° C на неопределенный срок на данный момент. CDNA теперь готов для использования в качестве шаблона для ПЦР (шаги 5.2, 7,4).
Примечание: Этот разрежения гарантирует, что в реакции RT-предусилитель праймеры не способствуют течению ПЦР.
5. вариант A: СУИМ сортировки клеток в серию разрежения ограничения для определения частоты vRNA+ клеток или выполнить экспериментальный контроль качества
Примечание: Перед выполнением сортировки одной ячейки, он может быть полезным для определения частоты клеток интерес, путем сортировки клеток в серийных разведений в репликации. Этот шаг также предоставляет ценные качества контроля для сортировки эффективность, лизис клеток, восстановления РНК и синтез cDNA, как описано в шаге 5.3. Предварительное определение частоты vRNA+ клетки позволяет для более точной оценки числа одиночных камер, которые должны быть отсортированы для достижения достаточного размера выборки для анализа выражения гена надлежащим питанием vRNA+ клеток.
- СУИМ сортировать клетки в 96-луночных пластины, подготовленных как в шагах 4.1-4.2 и собирать 1 – 1000 ячеек на хорошо в нескольких реплицирует.
Примечание: Количество реплицировать скважин при разбавлении каждой ячейки обычно обратно, связанные с концентрацией клеток в колодец. Когда частота инфицированной клетки значительно ниже 1%, ячейка разведений должна сосредоточиться на 100 – 1000 ячеек на хорошо. Пример сортировки пластины карты приводится в Рисунок 1, слева сверху. Свыше 1000 ячеек на колодец следует избегать из-за результирующего увеличения объема реакции и вмешательства с синтеза cDNA вниз по течению и количественной оценки. - Объединить ПЦР реактивы в растворе мастер смесь в таблице 4. Для тома реакции 25 мкл 22,5 мкл мастер смеси в сочетании с 2,5 мкл разбавленного cDNA шаблон с шагом 3,9. Выполнять ПЦР с использованием стандартных Велоспорт условий (например, 94 ° C за 5 мин, после чего 40 циклов 94 ° C 15 s и 60 ° C в течение 1 мин).
Примечание: Singleplex реакций ПЦР с помощью обычных ПЦР в реальном времени инструмент рекомендуются как экономичный предварительный анализ одного или нескольких анализов для демонстрации эффективного ячейку Сортировка, восстановления РНК и синтез cDNA. Он также может использоваться для вычисления частоты vRNA+ клеток. Мультиплексной ПЦР-реакции, с помощью Biomark обычно более подходящим для крупномасштабных одноклеточных анализов. - Для контроля качества, участок Et значения (Et = CtМакс − Ct) по сравнению с количеством клеток сортируются в а в масштабе10 журнала и применять линейный регрессионный анализ.
Примечание: Последовательное реплицирует, линейной регрессии склоне 3.3 (± 0,3) и R2 > 0,9 свидетельствует об эффективной эксперимента. На рисунке 4приведены примеры оптимальных и субоптимальные рода, RT-предусилитель эксперименты. - Чтобы определить частоту vRNA+ клеток, участок числа клеток, отсортированных в колодец на оси x (журнал10 масштаба) и часть скважин позитивным для vRNA в каждой ячейке разрежения на оси y. В качестве примера см. Рисунок 1 (нижний левый, распределение Пуассона). Применить модель линейной регрессии к данным определить количество ячеек, которые гавани один положительный клеточный в среднем соответствует 63,2% скважин положительный (0.632 на оси y)21. Преобразуйте это число клеток разрежения (оси x перехват) частота в процентах. Например одна клетка vRNA+ за 48 клетки эквивалентен частотой 2,1%.
6. вариант B: СУИМ сортировки клеток для анализа одной ячейки
- Выполните шаги 4.1 – 4,8 и укажите одну ячейку Сортировка в хорошо функция проточный цитометр индекс сортировки для создания отдельных файлов FCS для каждой ячейке Сортировка, сопоставлены хорошо позиции.
Примечание: Если количество пластин сортировки коллекции превышает количество имеющихся thermocyclers, Велоспорт может быть остановлена после этапа инактивации обратной транскриптазы (95 ° C на 2 мин) и cDNA могут храниться при температуре 4 ° C до thermocyclers доступны. В этом случае начать предварительное усиление в первом цикле 95 ° C 15 s. - Дополнительно: Создание cDNA «бассейны» состоящий из cDNA от определяемых пользователем партий одиночных клеток экран для редких клетки интереса, используя остаточного неразбавленном cDNA. Перенести 2 мкл неразбавленном одноклеточных предварительно усиленный cDNA, многоканальные пипетки в новую плиту 96-луночных. Повторите, дозирования 2 мкл cDNA от всех дополнительных единичных клеток назначенный бассейн в то же хорошо. Экран cDNA бассейны для gene(s) интерес (например, vRNA) для определения тех, которые содержат позитивные клетки, с использованием обычных ПЦР. Поскольку пул требуется только небольшая Алиготе cDNA от каждой ячейки, оставшиеся ~ 8 мкл cDNA-прежнему доступен для анализа одной ячейки.
Примечание: Объединение стратегий, рекомендуется перед выполнением анализа выражение гена одноклеточных в попытке уменьшить количество единичных клеток, допрашивали ресурсоемких мультиплексной ПЦР. Это подходит для ситуаций, в которых клетки интереса (например, Сив мРНК +) могут быть определены путем анализа ПЦР предварительного сингл plex. Простой стратегии высокой пропускной способности для создания cDNA бассейны, сочетающие 2 мкл неразбавленном cDNA от пластины сортировка коллекции строк (то есть, все 12 ячеек в строке A) или столбцы (то есть, все 8 ячеек в столбце 1), в одну скважину в новой пластинкой. Чтобы определить оптимальную стратегию объединения, рассмотрим ожидаемая частота клетки интереса от шага 5.4. Например если 10% клеток, как ожидается, будет положительным, бассейны, состоит из шести образцов одноклеточных cDNA часто будет отрицательным и клетки, представленных в этом пуле таким образом могут быть исключены из вниз по течению одноклеточных анализы.
7. мультиплексной ПЦР на платформе Biomark
Примечание: Этот раздел может следовать версии A или B, описанных выше. В исследовании, описанные здесь он применяется исключительно к анализу одной ячейки.
- Подготовьте пластину анализа ПЦР дозирования 4 мкл калибровочных от 2 x пробирного пластины (подготовленных на шаге 2.2) в новой 96-луночных ПЦР-планшете, содержащий 4 мкл пробирного загрузки реагента в каждой скважине. Поддерживать пробирного пластины при 4 ° C.
Примечание: Пластину пробирного стабилен при 4 ° C до одной недели и при-20 ° C на один месяц. Таким образом это может быть полезным для подготовки достаточного материала для фишек и храните надлежащим образом. - Отказаться от управления линии жидкости от грунтовки шприцы в двух впускных клапанов чипа. Удалите защитные пластиковые из-под плиты. Установите фишку на контроллере МФК с зубчатой стороной на позиции A1. Из главного меню выберите сценарий «Prime». Запустите сценарий.
- Подготовка смесь реального времени реакции путем смешивания 50 мкл пример загрузки реагента с 500 мкл ПЦР Мастер микс (Таблица материалов) для каждого microfluidic чипа. Пипетка 4.4 мкл в каждой скважине новой пластинкой 96-луночных PCR, отныне назначен пластину «образец».
- Пипетка 3.6 мкл разбавленного cDNA 1:5 от шага 4.8 в пластину образцы, содержащие смесь реального времени реакции.
Примечание: Если вниз выбор ПЦР была выполнена на экран для редких (например, vRNA+) клетки для анализа ниже по течению, как описано в шаге 6.2, включают только те ячейки, которые представлены в положительных бассейнов. - После завершения чип грунтовки Загрузите чип заливов дозирования 5 мкл от пробирного пластины в соответствующий хорошо на v-образный вырез (проба) стороне чипа, и 5 мкл из образца пластины в соответствующий хорошо на другой стороне (образец) чипа. Вставьте контроллер МФК чип и запустите сценарий «Загрузить набор».
- Передача чип на Biomark платформу для выполнения мультиплексной ПЦР. Продолжите инструмент установки и программирования ПЦР, следуя шаг за шагом инструкции, предоставляемые Real-Time PCR анализа программного обеспечения и с использованием стандартных V.1 выражение гена (GE) 96.96 протокола с 40 циклов PCR. Сохраните файл ChipRun в папку.
Примечание: Может выполняться несколько фишек в день и в течение нескольких дней. - Проанализируйте данные и ПЦР.
- Откройте программное обеспечение для анализа ПЦР в реальном времени. Откройте файл «ChipRun.bml» от «файл | Откройте» меню.
- Найти «Чип исследователь» и «Чип запустить резюме» в левом верхнем углу окна программного обеспечения. Определить три компоненты запуска резюме чип: анализ представления, образец установки и установки детектора.
- Нажмите на «Детектор Setup». «Задача» нажмите кнопку «Создать» и выберите тип контейнера «SBS пластины» и формат контейнера «SBS96». Рядом с «Сопоставление», щелкните на... и выбрать «M96-Assay-SBS96.dsp».
- Дополнительно: Назначьте каждое детектор (проба) номер или имя в разделе «Имя» каждой скважины, дважды щелкнув на 1st хорошо. Перейти к следующей хорошо, нажав клавишу «F2».
- Нажмите на «Установка образцов». Под «Задачи» рядом с «Сопоставление», нажмите кнопку... и выбрать «M96-образец-SBS96.dsp».
- Нажмите на «Анализ мнения». В разделе «Задачи» в закладке «ПЦР», выберите «Коррекция базовой линии для линейных (производная)» и «Ct порог метод для пользователя (детекторы)». На вкладке «Ct пороги» проверьте «Initialize с автоматической коробкой». Нажмите на кнопку «Анализировать» выше.
- В верхнем правом квадранте «Анализ взглядов» нажмите кнопку на второй вкладке «Таблица результатов». Из раскрывающегося меню выберите «Вид карты тепла». Тепловая карта с данными появится.
- Дополнительно: Для обеспечения единообразного ROX флуоресценции через чип, выберите «Просмотр изображения» вместо «Тепловой карте вид» из того же меню. Во втором из правой части окна выше тепловой карте выберите «ROX». В первом из правой части окна выберите один из 1-40 циклов. Нажмите на четвертый от окна справа для переключения в черно-белый дисплей ROX флуоресценции. Изображение будет отображаться, сообщает брызжет, частицы, или дефектов на чипе. Если грубо закрывается ROX единообразия, повторно запустите чипа.
- Под тепловые карты нажмите кнопку «Порог» и «Журнал граф». Настройте пороги Ct вручную для каждого детектора, нажав на анализы (столбцы тепла карта) и перетащив порог, необходимые для пересечения кривых амплификации в экспоненциальной фазе. Когда это сделано, нажмите кнопку «Анализировать».
- Экспорт данных ПЦР в виде файла .csv. Импорт данных в таблицу или статистический анализ программного обеспечения (например, СПМ) и сопоставить результаты выборки и анализа позиций на чипе. Группировать ячейки основе экспрессии вирусных генов, создав новый столбец и с помощью условной формулы. В разделе «Анализировать» выберите «Fit Y от X» и участок экспрессии генов против группы. Применить статистического анализа.
Примечание: Представитель одноклеточных количественное выражение четырех видов РНК SIV изображен в двумерных участков в Рисунок 5A. Количественные хост экспрессии генов в клетках SIV РНК+ показано на рисунке 5B. - Извлечение количественных белка выражение значения из одной ячейки данных СУИМ.
- Открыть .fcs файлов с СУИМ сортировки (шаг 6.1) соответствует 96-луночных пластину, используя FlowJo версии 9. С выделенным именем файла, выберите «платформа | Число событий ворота | Создание индексированного сортировки ворота». Отдельные ячейки появится отображаются построчно.
- Выделите все 96 клетки (не строк) и выберите «область | Экспорт | Выберите все компенсируется fluors.» Под «Тип данных» выберите «FCS файл», нажмите «Экспорт» и выберите нужную папку.
- Перетащите новый .fcs файлов для отдельных ячеек в новой рабочей области FlowJo. Выделить все ячейки, нажмите кнопку «Добавить статистика» («Σ» кнопка в левом верхнем углу) «| Значит | Все параметры Флуор».
- Откройте «редактор таблиц», нажав кнопку четвертый слева в верхнем левом углу. Выделите все тей первой ячейки и перетащить их в окно редактора таблиц. В окне Редактор таблицы нажмите ту же кнопку в верхней части «Создание и просмотр таблицы». Это создаст таблицу 96 клеток и числовой параметр для каждого Флуор.
- Скопируйте вывод в программное обеспечение базы данных (например, MS Excel, JMP), либо копирования/вставки или, нажав кнопку «Сохранить и запустить приложение» (четвертый из левой кнопкой над таблицей).
Примечание: Эта процедура является специфичным для FlowJo версии 9. FlowJo версии 10 использует различные процедуры для импорта индексированных данных. Индексированные потока данных можно также скопировать/вставить в JMP непосредственно из CSV-файлов, созданных клеток сортировщик.
- Слияние данных СУИМ одноклеточных и ПЦР по количеству пластины и хорошо позиции. Выполнить графический и статистический анализ на комбинированных одноклеточных ген (ПЦР) и белок выражение (FACS) данных.
Примечание: Примеры одной ячейки объединены ПЦР и СУИМ данные приведены на рисунке 6 (принимающей поверхности белка выражение профили для SIV-инфицированных, сращивания vRNA+ резус макака клетки), рис (CD4 экспрессии генов по сравнению с поверхности CD4 белков в клетках макак резус сращивания vRNA+ ) и ранее опубликована20. Для выявления дифференциально выразил генов в клетки группе(ах) интереса, одноклеточных анализ методов описанных ранее рекомендованные20,22,23,24, которые приходится Доля положительных генов, а также значение выражения непрерывной гена клеток.
Результаты
Рабочий процесс для весь протокол изображен на рисунке 1. Он состоит из двух вариантов определяется количество клеток сортировка: либо ограничивающих разрежения или как одной клетки, как описано в тексте. На рисунке 2приведены примеры анализа квалификации праймер зонд 2 раза серийных разведений РНК. Шлюзовые стратегию для выявления потенциальных клетки SIV+ показано на рисунке 3. Контроль качества успешного, неоптимальной и неудачных ПЦР для уборки гена GAPDH на СУИМ Сортировка ячеек в ограничении разрежения показаны на рисунке 4. Количественное выражение четырех SIV РНК видов и резус макака генов, которые высказывались дифференцировано в инфицированных клетках одноклеточных показаны на рисунке 5. Представитель двумерных, гистограммы и scatterplots изображают выражение профиль поверхности белка SIV РНК+ CD4+ T клеток измеряется проточной цитометрии на рисунке 6. Участок trivariate пузырь (рис. 7) Отображает связь между белка поверхности CD3 или CD4, CD3 или CD4 мРНК и количественных вирусных генов (ТАТ/rev) выражение в одиночных клетках.

Рисунок 1 : Схема экспериментальной рабочего процесса показывает три основных компонента: поток гранулярных сортировки, обратная транскрипция плюс на основе ПЦР предварительно амплификация cDNA (RT, предварительного усилителя) и ПЦР. Сортировка может быть выполнена как либо ограничивающих разрежения (A, зеленый фон) или как отдельные клетки (B, оранжевый фон). Сразу же после сортировки СУИМ клетки лизированы и РНК вспять транскрибируется в cDNA и предварительно усиленный (RT, предусилитель ПЦР) подготовить ПЦР шаблон. Ограничение разрежения сорта определить частоту положительных клеток для вирусной РНК с помощью статистики распределения Пуассона, а также экспериментальных эффективность и пример восстановления. Головку зеленая стрелка указывает предполагаемое количество клеток отсортированы в хорошо содержащие одну ячейку положительные вирусные гена (соответствующий 63,2% вероятность таких скважин, геном положительных), который преобразуется в ячейке частоты. Оценки частоты может использоваться сообщить количество единичных клеток, собранные в последующей сортировки (B). Индексированные одноклеточных СУИМ сортировки депозитов один ячейки в колодец и генерирует файлы данных для каждой ячейки, аннотированный хорошо позиции в течение 96-луночных пластины. В мультиплексе для 96 генов одновременно выполняется одной ячейки ПЦР. Сочетание поверхности белка (FACS) и выражение mRNA позволяет профилирование отдельных клеток (правая колонка). Необязательный ПЦР для вирусных генов может осуществляться между предварительной амплификации PCR и мультиплексной ПЦР (B, средний) экран отдельные ячейки или бассейны кДНК одной ячейки вниз выберите вирусной РНК+ клетки или бассейны для мультиплексной ПЦР анализа. Тепловая карта иллюстрирует экспрессии генов (Ct значение) для 96 анализов (столбцы) и 96 единичных клеток (строк). Пожалуйста, нажмите здесь, чтобы посмотреть большую версию этой фигуры.

Рисунок 2: Приводятся данные представителя ПЦР из экспериментов грунт квалификации для успешной (левый и средний) и не (справа) коммерчески доступных анализов ( CD6, Сив ТАТ/rev, и TLR3, соответственно). Для CD6 и TLR3, РНК было извлечено от 106 сортировка СУИМ резус макака КСДОР CD4 Т-клеток с использованием коммерческих комплект+ . Восемь реплицирует серии 12-точка РНК два раза разрежения (0,023-48 нг РНК, соответствующий 1.2 – 2400 клеток эквиваленты предполагая 20 pg РНК на CD4+ Т-клеток) были подвергнуты RT-предусилитель и ПЦР. Для SIV ТАТ/revРНК было извлечено от резус макака получения инфицированных в пробирке с SIVmac239. РНК разведений были подготовлены связующего РНК эквиваленты 6 – 12 000 ячеек. ET (40-Ct) значения, которые увеличивают с экспрессии генов, заговор против число оценкам клеток. Разбавление серии выставке R2 > 0.97 и наклон 3.32 ±0, 3 указывают успешный грунтовка квалификации. Пожалуйста, нажмите здесь, чтобы посмотреть большую версию этой фигуры.
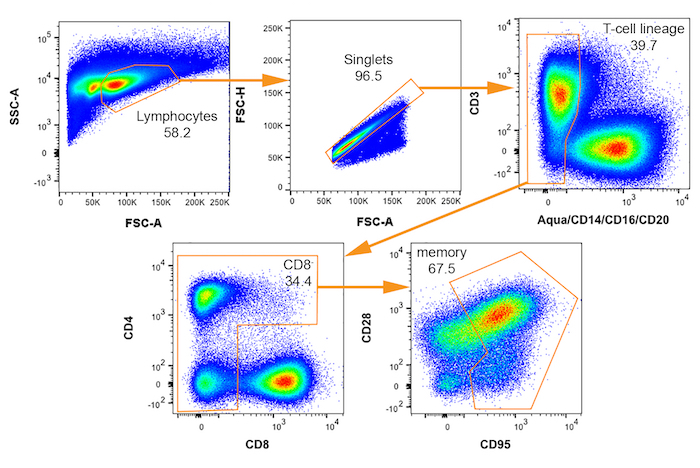
Рисунок 3: Двумерных СУИМ участки с стробирования схема для изоляции резус макака клеток потенциально инфицированных SIV. Последовательные ворота для выделения памяти (CD95 +) CD4+ T клетки отображаются от верхнего левого угла в нижний правый с каждым именем населения указал. Указывается процент родительского сюжета, который падает в пределах каждой ворот. Пожалуйста, нажмите здесь, чтобы посмотреть большую версию этой фигуры.

Рисунок 4 : Представитель экспериментальной проверки ПЦР осуществляется на СУИМ Сортировка ячеек в ограничении разрежения с помощью GAPDH уборка гена для трех независимых экспериментов: успешный (слева), неоптимальной (в центре) и не (справа). Реплицирует 10-100 ячеек на хорошо должен охватывать не более чем 2 Ets и 300 – 1000 ячеек скважин в пределах 1 Et. Наклон линейной регрессии должна быть 3.32 ± 0,3, R2 > 0,95. Неспособность достичь эти спецификации указывает технические трудности в шаги 1, 2, 3 или их комбинации. Пожалуйста, нажмите здесь, чтобы посмотреть большую версию этой фигуры.

Рисунок 5 : Одноклеточных количественных вирусных и принимающих экспрессии генов в СУИМ сортировка резус макака CD4+ Т-клеток от верхней брыжеечной Лимфоузел 10 дней пост SIVmac251 инфекции. (A) экспрессии вирусных генов двумерных графиков умножить сращивания (ТАТ/rev) и поодиночке сращивания (env) SIV мРНК, одной клетки (слева). ТАТ/rev+env– (клетки светло зеленый вдоль оси x) Экспресс меньше копий ТАТ/rev РНК чем env+ клетки (тёмно-зелёный), в соответствии с ранним стадии инфекции и до Rev белка-опосредованной стабилизации и ядерного экспорта частично сращивания vRNAs как env. Клетки, которые не выражают сращивания вирусной РНК изображены в серый (не вирусной РНК), коричневый (кляп+ и LTR+) или Тан ( кляп+ или LTR+). Unspliced (кляп+) и общее выражение mRNA SIV (LTR+) показан для тех же ячейках (справа). Высокое обилие unspliced кляп РНК в ТАТ/rev++ envклеток соответствует поздней стадии продуктивной инфекции во время которого обильные геномной РНК выражается для упаковки в подающий надежды Вирионы. (B) скрипка участков резус макака генов дифференциально выражена в по крайней мере одно подмножество SIV-инфицированных клеток, по сравнению с неинфицированных клеток (серый). Статистический анализ проводился описанный ранее20,,2223,24. Звездочка указывает ложные обнаружения ставка < 10% в комбинированных вероятность коэффициент сравнения теста относительно неинфицированных клеток. Линия соединяет средние значения ячеек групп для каждого гена. Этот показатель был изменен из Болтона и др. 20 Пожалуйста, нажмите здесь, чтобы посмотреть большую версию этой фигуры.

Рисунок 6: Представитель принимающей поверхности белка выражение профили СУИМ сортировка клеток от резус макака брыжеечных Лимфоузел 10 дней пост SIVmac251 инфекции. (A) рассеяния показ CD3, CD4 и ИКОС поверхности белков в незараженных (серый), кляп,+ТАТ/rev– (коричневый) и кляп++ ТАТ/revклетки (зеленый). Интенсивность флуоресценции рассчитывается для каждой ячейки (точка). Останец прямоугольные изображают межквартильный диапазон (ИКР) и средний (коробка), дальней точки в пределах 1,5 x IQR из коробки (усы), а также потенциал выбросов (отключенных точек). Турники в верхней указывают существенные различия (p < 0,05, непараметрический тест ранга Вилкоксон). (B) отображение Бивариантный и гистограммы поверхности CD3 и CD4 выражения протеина для ячеек в (A). Точка участок (слева) указывает процент населения каждой ячейки в квадранте. CD3 и CD4 гистограммы (в центре, справа) изображают поверхности белка Даунрегуляция среди кляп+ТАТ/rev+ по отношению к незараженным и кляп+ТАТ/rev– ячейки. (C) двенадцать представитель ТАТ/rev+ единичных клеток (A – B) показываются через три двумерных участков поверхности выражение CD3/CD4 (слева), CD69/CD38 (в середине) и ИКОС/HLA-DR (справа). Пожалуйста, нажмите здесь, чтобы посмотреть большую версию этой фигуры.
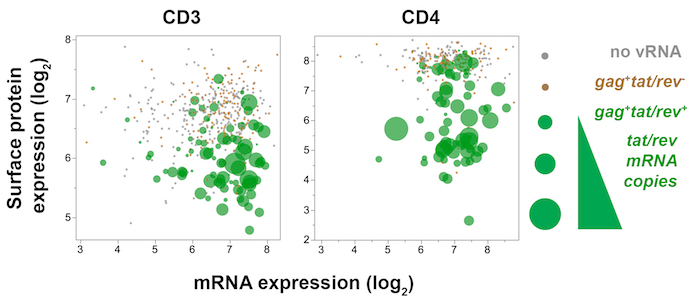
Рисунок 7 : Trivariate участок отображения одноклеточных вирусных генов (ТАТ/rev), принимающих гена (CD3 или CD4) и принимающих поверхности белка (CD3 или CD4) выражение в памяти SIV-инфицированных CD4+ T клеток от верхней брыжеечной Лимфоузел 10 дней пост SIVmac251 инфекции. Выражение протеина CD4 (флуоресценции) заговор против мРНК CD4 (ПЦР), в то время как количество ТАТ/rev выраженные каждой ячейки выражается точка размер. В ТАТ/rev+ клетки (зеленый), снижение поверхности CD4 и CD3 выражение протеина с устойчивой CD4 и CD3 стенограммы, соответственно, указывают, что выражение поверхности белка модулируется вниз по течению экспрессии генов. Пожалуйста, нажмите здесь, чтобы посмотреть большую версию этой фигуры.
Таблица 1: Грунты и датчиков, используемых для обнаружения SIV нуклеиновых кислот. Когда две последовательности указаны для грунтовки или зонд, использовались эквимолярных количествах обеих последовательностей. Пожалуйста, нажмите здесь, чтобы загрузить этот файл.
Таблица 2: 96-ген группа используется для количественный ампликонов на Biomark инструменте. С синим фоном, указаны четыре SIV анализов. Пожалуйста, нажмите здесь, чтобы загрузить этот файл.
Таблица 3: Реакции смесь используется для обратной транскрипции и предварительного усиления. Пожалуйста, нажмите здесь, чтобы загрузить этот файл.
Таблица 4: смесь реакции ПЦР для ПЦР в реальном времени выполнена на инструменте Квант Studio 6. Пожалуйста, нажмите здесь, чтобы загрузить этот файл.
Дополнительного кодирования файла 1. Инструкции для квалификации анализов выражение гена. Пожалуйста, нажмите здесь, чтобы загрузить этот файл.
Дополнительного кодирования файла 2: пример карты шаблон в СПМ. Пожалуйста, нажмите здесь, чтобы загрузить этот файл.
Дополнительного кодирования файла 3: зонд карта шаблон в СПМ. Пожалуйста, нажмите здесь, чтобы загрузить этот файл.
Дополнительного кодирования файла 4: грунтовка анализ сценария для JMP. Пожалуйста, нажмите здесь, чтобы загрузить этот файл.
Дополнительного кодирования файла 5. Кусочно-анализ сценария для JMP. Пожалуйста, нажмите здесь, чтобы загрузить этот файл.
Обсуждение
Протокол описано здесь, называется tSCEPTRE, интегрирует поверхности белка одноклеточных количественный, многопараметрических проточной цитометрии с количественное выражение mRNA одноклеточных, высоко мультиплексированных RT-ПЦР. Объединение этих двух технологий позволяет высоким содержанием снимки комбинированных транскрипционный анализ и профиль белка из одной клетки в формате высокой пропускной способности. Мы используем метод для идентификации до сих пор недостижимой клетки, инфицированные с SIV в естественных условияхи описать дифференциально выраженной хост генов и белков. Протокол может быть адаптирована для изучения любой популяции клеток интерес различимых выражением поверхности белки, мРНК, или их комбинации. Описанные методы полагаются на точной сортировки одноклеточных и записи подачей cytometry данных в паре с коммерчески доступных ПЦР реактивы и мультиплексированных реальном масштабе времени PCR инструментария. Результатом является чувствительным, количественные оценки одноклеточных комбинированных данных белков и ген выражение.
Другие подходы, связывание белка и ген выражение в одиночных клетках были описаны1,25,,2627. Индексированные СУИМ сортировки следуют RNAseq, успешно применяется для характеризации гемопоэтических стволовых клеток, представляет собой особенно многообещающим подходом28. Однако в то время как RNAseq имеет ряд преимуществ над целевых генов выражение анализы, а именно анализ беспристрастной полным транскриптом, он подлежит более высокой несколько сравнений статистическая стоимость и могут быть менее чувствительным для количественной оценки низкая копирования стенограммы. Кроме того, это не в настоящее время экономически целесообразно выполнять RNAseq на тысячи клеток в поисках редких зараженные клетки присутствуют на частотах < 1% (например, ВИЧ, Сив). Другие новые технологии одноклеточных цель сформировать единый индикация, т.е. СУИМ или ПЦР, для обнаружения белков и нуклеиновых кислот в той же ячейке. К ним относятся обнаружение белков, флуоресцентных антител и мРНК, флуоресцентный олигонуклеотидных зондов, следуют СУИМ анализа (мРНК поток-рыба)14,15,29,30. Кроме того белки могут быть обнаружены пар антител олигонуклеотида конъюгатов, которые распознаются ПЦР параллельно с наоборот трансляции мРНК31,32,33,34 . Эти подходы «гибридные» обеспечивают одновременное нуклеиновых кислот и белков измерения с одной ячейкой резолюции похож на tSCEPTRE, но они ограничены где они либо не количественные (мРНК поток-рыба) или может потребоваться настроить антитела или мРНК зонды. Наш подход tSCEPTRE количественные измерения протеина и мРНК и все реагенты являются коммерчески доступных, с возможным исключением возбудителя конкретных анализов.
Особое внимание должны применяться к несколько шагов в протоколе до анализа экспериментальных образцов. Во-первых, высокий параметр проточной цитометрии требует тщательной оптимизации группы флуоресцентных антител для обеспечения чувствительных обнаружение и разрешение35. Каждое антитело должны индивидуально титруют определить концентрацию, которая достигает оптимального разделения и минимальным фон окраски, следуют оценки пятнать когда все конъюгатов антитела используются в сочетании. Во-вторых, экспериментальное определение пробирного выражение гена соответствующие ПЦР для измерения каждого гена интереса имеет важное значение. Несколько коммерчески доступных анализов обычно доступны для каждого гена, но по нашему опыту, многие из них не количественным одну ячейку уровня6. Таким образом важно претендовать все предложенные анализов на серийно разреженных РНК до использования для экспрессии генов количественный. Оптимизация этих реагентов отнимает много времени, но значение надежных панелей конъюгатов антител и анализов выражение гена которая позволяет для чувствительных обнаружения всех молекул интерес оправдывает время инвестиции. Кроме того первоочередное внимание должно уделяться коммерчески доступных анализов, содержащие датчики, которые охватывают Экзон экзона развязок (обозначено с суффиксом «М1») для повышения специфичности мРНК, хотя альтернативные assays способны обнаруживать геномной ДНК (суффикс «s1» или «g1» ), считаются маловероятным повлиять на результаты выражения гена из-за эквивалент хромосомные копии в каждой ячейке. Сохранение РНК, вверх по течению РТ критических и достигается, сохраняя образцы охлажденные, как указано в протоколе. Аналогично, продолжительность сортировки СУИМ и интервал между сортировкой и RT-предусилитель должны быть сведены к минимуму. Для ограничения разрежения и одну ячейку Сортировка cDNA может быть впервые проанализированы обычных ПЦР для оценки восстановления РНК сильно выражена уборка генов. Если наблюдаемое выражение не является единообразной среди реплицирует как числа клеток, или наклон линейной подходят для ограничения разведениях происходит сбой проверки, устранение неполадок следует выполнять на ячейку процедуры сортировки и вниз по течению.
Потенциальные ограничения подход, описанный здесь включают (1) обнаружение ДНК в дополнение к РНК, особенно для анализов не конкретные мРНК сращивания узлов, и (2) ограниченное количество поверхностных белков и гены измеряется. Однако обнаружение ДНК вряд ли внести существенный вклад анализ выражения гена разностного узла по причине, говорилось выше. Кроме того, мы непосредственно измерить степень которой ДНК способствует ПЦР сигнал в этом протоколе, два подхода:) за исключением обратной транскриптазы и b) изменение протокол лизис клетки для повышения ядерной мембраны lysis. В отсутствие обратной транскриптазы, оба сращивания и unspliced вирусных генов по-прежнему были обнаружены, но в значительно снизить частоту, чем при наличии обратной транскриптазы (~ бриллиантов и 2 раза сокращения, соответственно)20. Следовательно выражение анализов вирусных генов может переоценить количество вирусных РНК в клетке вследствие наличия связанной ячейки вирусной ДНК. Мы связываем этот вывод цитоплазматических RT продуктов, образующихся при принимающей ячейки инфекции, известный для сращивания и unspliced РНК ВИЧ/Сив, возникая от входящих вирионы36. Таким образом возможно, целесообразно посвятить часть экспериментов в условиях, не имея обратной транскриптазы чтобы quantitate ДНК производный шаблон для некоторых приложений. Следует также отметить, что unspliced РНК SIV/ВИЧ, производные от входящих вирус нельзя отличить от de novo синтезированных вирусной РНК. Во-вторых, мы включены ядерной лизис шаг в протоколе RT-предусилитель и наблюдается экспоненциальный рост в комплексной SIV ДНК (Alu-LTR) копии (Рисунок S3 Болтон и др.) 20. геномной ДНК вряд ли таким образом вносить существенный вклад в шаблоне нуклеиновых кислот, извлеченные из метода лизис клеток, используемый здесь. Следует отметить помимо ядерной лизис шаг может быть полезные исследования в будущем одноклеточных, стремящихся расследовать SIV или другой вирус ДНК положительных клеток, включая латентно инфицированных клеток.
Количество поверхностных белков проанализирована диктуется возможности потока гранулярных клеток сортировщик. Текущий коммерчески доступных инструментов не превышает 30 параметров. Будущие исследования, используя более продвинутые проточной цитометрии далее расширит белок, профилирования потенциал этого подхода. Количество стенограммы может также распространяться за рамки 96, предоставляемых вспомогательных реагентов (например, грунтовки высокой концентрации), оборудования и приборов. В конечном счете новые технологии одной ячейки, которые сочетают в себе анализ протеома (масс-спектрометрия), транскриптом (RNAseq) и генома (DNAseq) заменит целенаправленные подходы для обнаружения исследований31,37, 38 , 39. Однако, целенаправленных ПЦР скорее всего будет оставаться ценным инструментом для проверки таких «омику» подходов, как золотой стандарт для количественного выражения анализа.
Сводный анализ белков и транскрипции, tSCEPTRE является мощным инструментом для изучения редких или трудно определить клеток, таких как тех патогенных укрывательство, содержащие онкогенов или иным образом экспонирование аберрантных фенотип. Новые маркеры для транскрипционно активных SIV/ВИЧ инфицированных клеток могут быть определены таким образом, а также открытие новых механизмов, участвующих в патогенезе этих вирусных инфекций. Идентификация латентно инфицированных клеток потребует дальнейшего развития протокола к вирусной ДНК интегрированы в геноме принимающей ослов. Следует отметить частота ВИЧ/SIV инфицированных клеток значительно ниже в хронической viremic или лечение инфекции, которая представит практических проблемы в изучении инфицированных клеток, полученных от этих параметров. Наш подход закладывает основу для оценки ранее неразрешимыми механизм: что post-transcriptional регулирования на уровне одной ячейки, и имеет широкую применимость к хост возбудитель взаимодействий, а также более общих клеточных процессов.
Раскрытие информации
Эта работа была поддержана соглашением о сотрудничестве (W81XWH-07-2-0067) между Генри м. Джексон фонда для улучшения военной медицины, Inc. и Департамент обороны США (DOD). Мнения являются мнениями авторов и не должно быть истолковано как представлять позиции армии США или Министерство обороны. Исследование было проведено по протоколу утвержденных использование животных в объекте AAALACi аккредитованных в соответствии с Закон о благосостоянии животных и других федеральных законов и нормативных положений, касающихся животных и экспериментов с участием животных и придерживается принципов говорится в руководстве для ухода и использования лабораторных животных, СРН публикации, издание 2011.
Благодарности
Авторы хотели бы поблагодарить NIAID VRC потока Cytometry Core и Cytometry Core потока MHRP Услуги для технического обслуживания и эксплуатации инструментов СУИМ и сортировочное оборудование; Мария Монтеро, Вишакха Шарма, песня имён для экспертной технической помощи; Майкл Пятак, младший (покойный) для помощи с SIV ПЦР анализа дизайн; и Брэндон Кил и Мэтью Scarlotta для SIV изолировать последовательности. Мнения являются мнениями авторов и не должно быть истолковано как представлять позиции армии США или Министерство обороны. Исследование было проведено по протоколу утвержденных использование животных в соответствии с Закон о благосостоянии животных и других федеральных законов и нормативных положений, касающихся животных АААЛЖ аккредитованных объекта и экспериментов с участием животных и придерживается принципов говорится в руководстве для ухода и использования лабораторных животных, СРН публикации, издание 2011.
Материалы
| Name | Company | Catalog Number | Comments |
| RNA extraction and PCR reagents and consumables | |||
| Genemate 96-Well Semi-Skirted PCR Plate | BioExpress/VWR | T-3060-1 | |
| Adhesive PCR Plate Seals | ThermoFisher | AB0558 | |
| Armadillo 384-well PCR Plate | ThermoFisher | AB2384 | |
| MicroAmp Optical Adhesive Film | Applied Biosystems/ThermoFisher | 4311971 | |
| DEPC Water | Quality Biological | 351-068-101 | |
| Glass Distilled Water | Teknova | W3345 | |
| Superscript III Platinum One-Step qRT-PCR Kit | Invitrogen/ThermoFisher | 11732088 | |
| SUPERase-In Rnase Inhibitor | Invitrogen/ThermoFisher | AM2696 | |
| Platinum Taq | Invitrogen/ThermoFisher | 10966034 | |
| dNTP Mix | Invitrogen/ThermoFisher | 18427088 | |
| ROX Reference Dye (if separate from kit) | Invitrogen/ThermoFisher | 12223012 | |
| DNA Suspension Buffer | Teknova | T0223 | |
| RNAqueous kit | Invitrogen/ThermoFisher | AM1931 | |
| TaqMan gene expression assays not listed in Table 2 | |||
| CD6 | Applied Biosystems/ThermoFisher | Hs00198752_m1 | |
| TLR3 | Applied Biosystems/ThermoFisher | Hs1551078_m1 | |
| Biomark reagents | |||
| Control Line Fluid Kit | Fluidigm | 89000021 | |
| TaqMan Universal PCR Mix | Applied Biosystems/ThermoFisher | 4304437 | |
| Assay Loading Reagent | Fluidigm | 85000736 | |
| Sample Loading Reagent | Fluidigm | 85000735 | |
| Dynamic Array 96.96 (chip) | Fluidigm | BMK-M-96.96 | |
| FACS reagents | |||
| SPHERO COMPtrol Goat anti-mouse (lambda) | Spherotech Inc. | CMIgP-30-5H | |
| CompBeads Anti-Mouse Ig,k | BD Biosciences | 51-90-9001229 | |
| 5 ml Polystyrene tube with strainer cap | FALCON | 352235 | |
| Aqua Live/Dead stain | Invitrogen/ThermoFisher | L34976 | dilute 1:800 |
| Mouse Anti-Human CD3 BV650 clone SP34-2 | BD Biosciences | 563916 | dilute 1:40 |
| Mouse Anti-Human CD4 BV786 clone L200 | BD Biosciences | 563914 | dilute 1:20 |
| Mouse Anti-Human CD8 BUV496 clone RPA-T8 | BD Biosciences | 564804 | dilute 1:10 |
| Mouse Anti-Human CD28 BV711 clone CD28.2 | Biolegend | 302948 | dilute 1:20 |
| Mouse Anti-Human CD95 BUV737 clone DX2 | BD Biosciences | 564710 | dilute 1:10 |
| Mouse Anti-Human CD14 BV510 clone M5E2 | Biolegend | 301842 | dilute 1:83 |
| Mouse Anti-Human CD16 BV510 clone 3G8 | Biolegend | 302048 | dilute 1:167 |
| Mouse Anti-Human CD20 BV510 clone 2H7 | Biolegend | 302340 | dilute 1:37 |
| Anti-CD38-R PE clone OKT10 | NHP reagent recource | N/A | dilute 1:100 |
| Mouse Anti-Human CD69 BUV395 clone FN50 | BD Biosciences | 564364 | dilute 1:10 |
| Mouse Anti-Human HLA-DR APC-H7 clone G46-6 | BD Biosciences | 561358 | dilute 1:20 |
| Mouse Anti-Human ICOS Alexa Fluor 700 clone C398.4A | Biolegend | 313528 | dilute 1:80 |
| Instruments | |||
| BioPrptect Containment Enclosure | Baker | ||
| BD FACS Aria | BD Biosciences | ||
| ProtoFlex Dual 96-well PCR system | Applied Biosystems/ThermoFisher | 4484076 | |
| Quant Studio 6 qPCR instrument | Applied Biosystems/ThermoFisher | 4485694 | |
| IFC controller HX | Fluidigm | IFC-HX | |
| Biomark HD | Fluidigm | BMKHD-BMKHD |
Ссылки
- Macaulay, I. C., Ponting, C. P., Voet, T. Single-Cell Multiomics: Multiple Measurements from Single Cells. Trends Genet. 33 (2), 155-168 (2017).
- Pasternak, A. O., Lukashov, V. V., Berkhout, B. Cell-associated HIV RNA: a dynamic biomarker of viral persistence. Retrovirology. 10, 41(2013).
- Mailler, E., et al. The Life-Cycle of the HIV-1 Gag-RNA Complex. Viruses. 8 (9), (2016).
- Varmus, H. Retroviruses. Science. 240 (4858), 1427-1435 (1988).
- Frankel, A. D., Young, J. A. HIV-1: fifteen proteins and an RNA. Annu Rev Biochem. 67, 1-25 (1998).
- Dominguez, M. H., et al. Highly multiplexed quantitation of gene expression on single cells. J Immunol Methods. 391 (1-2), 133-145 (2013).
- Tokarev, A., Guatelli, J. Misdirection of membrane trafficking by HIV-1 Vpu and Nef: Keys to viral virulence and persistence. Cell Logist. 1 (3), 90-102 (2011).
- Malim, M. H., Bieniasz, P. D. HIV Restriction Factors and Mechanisms of Evasion. Cold Spring Harb Perspect Med. 2 (5), a006940(2012).
- Sugden, S. M., Bego, M. G., Pham, T. N., Cohen, E. A. Remodeling of the Host Cell Plasma Membrane by HIV-1 Nef and Vpu: A Strategy to Ensure Viral Fitness and Persistence. Viruses. 8 (3), 67(2016).
- Simon, V., Bloch, N., Landau, N. R. Intrinsic host restrictions to HIV-1 and mechanisms of viral escape. Nat Immunol. 16 (6), 546-553 (2015).
- Kirchhoff, F. Immune evasion and counteraction of restriction factors by HIV-1 and other primate lentiviruses. Cell Host Microbe. 8 (1), 55-67 (2010).
- Wigzell, H. Immunopathogenesis of HIV infection. J Acquir Immune Defic Syndr. 1 (6), 559-565 (1988).
- Reynolds, M. R., et al. Ex vivo analysis of SIV-infected cells by flow cytometry. Cytometry A. 77 (11), 1059-1066 (2010).
- Baxter, A. E., et al. Single-Cell Characterization of Viral Translation-Competent Reservoirs in HIV-Infected Individuals. Cell Host Microbe. 20 (3), 368-380 (2016).
- Grau-Exposito, J., et al. A Novel Single-Cell FISH-Flow Assay Identifies Effector Memory CD4+ T cells as a Major Niche for HIV-1 Transcription in HIV-Infected Patients. MBio. 8 (4), (2017).
- Eriksson, S., et al. Comparative analysis of measures of viral reservoirs in HIV-1 eradication studies. PLoS Pathog. 9 (2), e1003174(2013).
- Finzi, D., et al. Identification of a reservoir for HIV-1 in patients on highly active antiretroviral therapy. Science. 278 (5341), 1295-1300 (1997).
- Chomont, N., et al. HIV reservoir size and persistence are driven by T cell survival and homeostatic proliferation. Nat Med. 15 (8), 893-900 (2009).
- DeMaster, L. K., et al. A Subset of CD4/CD8 Double-Negative T Cells Expresses HIV Proteins in Patients on Antiretroviral Therapy. J Virol. 90 (5), 2165-2179 (2015).
- Bolton, D. L., et al. Combined single-cell quantitation of host and SIV genes and proteins ex vivo reveals host-pathogen interactions in individual cells. PLoS Pathog. 13 (6), e1006445(2017).
- Quintans, J., Lefkovits, I. Precursor cells specific to sheep red cells in nude mice. Estimation of frequency in the microculture system. Eur J Immunol. 3 (7), 392-397 (1973).
- Finak, G., et al. MAST: a flexible statistical framework for assessing transcriptional changes and characterizing heterogeneity in single-cell RNA sequencing data. Genome Biol. 16, 278(2015).
- McDavid, A., et al. Modeling bi-modality improves characterization of cell cycle on gene expression in single cells. PLoS Comput Biol. 10 (7), e1003696(2014).
- McDavid, A., Finak, G., Gottardo, R. The contribution of cell cycle to heterogeneity in single-cell RNA-seq data. Nat Biotechnol. 34 (6), 591-593 (2016).
- Kok, Y. L., Ciuffi, A., Metzner, K. J. Unravelling HIV-1 Latency, One Cell at a Time. Trends Microbiol. , (2017).
- Rato, S., Golumbeanu, M., Telenti, A., Ciuffi, A. Exploring viral infection using single-cell sequencing. Virus Res. 239, 55-68 (2017).
- Wagner, A., Regev, A., Yosef, N. Revealing the vectors of cellular identity with single-cell genomics. Nat Biotechnol. 34 (11), 1145-1160 (2016).
- Nestorowa, S., et al. A single-cell resolution map of mouse hematopoietic stem and progenitor cell differentiation. Blood. 128 (8), e20-e31 (2016).
- Soh, K. T., et al. Simultaneous, Single-Cell Measurement of Messenger RNA, Cell Surface Proteins, and Intracellular Proteins. Curr Protoc Cytom. 75, 41-47 (2016).
- Kochan, J., Wawro, M., Kasza, A. Simultaneous detection of mRNA and protein in single cells using immunofluorescence-combined single-molecule RNA FISH. Biotechniques. 59 (4), 209-212 (2015).
- Stahlberg, A., Thomsen, C., Ruff, D., Aman, P. Quantitative PCR analysis of DNA, RNAs, and proteins in the same single cell. Clin Chem. 58 (12), 1682-1691 (2012).
- Assarsson, E., et al. Homogenous 96-plex PEA immunoassay exhibiting high sensitivity, specificity, and excellent scalability. PLoS One. 9 (4), e95192(2014).
- Fredriksson, S., et al. Protein detection using proximity-dependent DNA ligation assays. Nat Biotechnol. 20 (5), 473-477 (2002).
- Genshaft, A. S., et al. targeted profiling of single-cell proteomes and transcriptomes in a single reaction. Genome Biol. 17 (1), 188(2016).
- Mahnke, Y. D., Roederer, M. Optimizing a multicolor immunophenotyping assay. Clin Lab Med. 27 (3), 469-485 (2007).
- Liang, C., Hu, J., Russell, R. S., Kameoka, M., Wainberg, M. A. Spliced human immunodeficiency virus type 1 RNA is reverse transcribed into cDNA within infected cells. AIDS Res Hum Retroviruses. 20 (2), 203-211 (2004).
- Stegle, O., Teichmann, S. A., Marioni, J. C. Computational and analytical challenges in single-cell transcriptomics. Nat Rev Genet. 16 (3), 133-145 (2015).
- Wu, M., Singh, A. K. Single-cell protein analysis. Curr Opin Biotechnol. 23 (1), 83-88 (2012).
- Haque, A., Engel, J., Teichmann, S. A., Lonnberg, T. A practical guide to single-cell RNA-sequencing for biomedical research and clinical applications. Genome Med. 9 (1), 75(2017).
Перепечатки и разрешения
Запросить разрешение на использование текста или рисунков этого JoVE статьи
Запросить разрешениеСмотреть дополнительные статьи
This article has been published
Video Coming Soon
ISSN 2689-3649
Авторские права © 2025 MyJoVE Corporation. Все права защищены
Мы используем файлы cookie для улучшения качества работы на нашем веб-сайте.
Продолжая пользоваться нашим веб-сайтом или нажимая кнопку «Продолжить», вы соглашаетесь принять наши файлы cookie.