Method Article
Einzellige Quantifizierung der mRNA und Oberfläche Protein-Expression in Simian Immunodeficiency Virus-infizierten CD4+ T Zellen isoliert von Rhesus-Makaken
In diesem Artikel
Zusammenfassung
Beschrieben ist eine Methodik, die Expression von Genen 96 quantitate und 18 Oberflächenproteine von einzelnen Zellen Ex Vivo, ermöglicht die Identifizierung von differenziell ausgedrückt, Genen und Proteinen in Virus-infizierten Zellen im Verhältnis zu nicht infizierten Zellen. Wir wenden das Konzept Studie SIV-infizierten CD4+ T Zellen isoliert von Rhesus-Makaken.
Zusammenfassung
Einzellige Analyse ist ein wichtiges Instrument für heterogene Populationen von Zellen zu sezieren. Die Identifizierung und Isolierung von seltenen Zellen können schwierig sein. Um diese Herausforderung zu meistern, eine Methodik kombinieren indiziert Durchflusszytometrie und Hochdurchsatz-Multiplex quantitative Polymerase-Kettenreaktion (qPCR) entwickelt wurde. Ziel war es, zu identifizieren und zu charakterisieren, simian immunodeficiencyvirus (SIV)-infizierten Zellen in Rhesus-Makaken. Durch Quantifizierung der Oberfläche Protein durch Fluoreszenz-aktivierte Zellsortierung (FACS) und mRNA von qPCR sind Virus-infizierten Zellen durch virale Genexpression identifiziert, kombiniert mit Host-gen und Protein Messungen ein mehrdimensionales Profil erstellen . Wir bezeichnen den Ansatz, gezielte einzellige Proteo-transcriptional Bewertung oder tSCEPTRE. Um die Methode auszuführen, sind vitaler Zellen spezifisch für oberflächenmarker verwendet für FACS Isolierung einer Zelle Teilmenge und/oder nachgeschalteten phänotypische Analyse mit fluoreszierenden Antikörpern gefärbt. Einzelne Zellen werden gefolgt von sofortige Lyse, Multiplex-reverse Transkription (RT), PCR Vorverstärkung und hohem Durchsatz qPCR bis zu 96 Transkripte sortiert. FACS-Messungen sind aufgenommen in die Zeit des Aussortierens und anschließend mit Genexpressionsdaten von gut Lage, einem kombinierten Protein und transkriptionellen Profil zu schaffen. Zur Untersuchung der SIV-infizierten Zellen direkt Ex Vivo, Zellen wurden von qPCR Erkennung von mehrere virale RNA-Spezies identifiziert. Die Kombination von viralen Transkripte und die Menge der einzelnen bilden den Rahmen für die Klassifizierung von Zellen in verschiedene Phasen des viralen Lebenszyklus (z.B.im Vergleich zu nicht-produktiven produktiv). Darüber hinaus wurden tSCEPTRE von SIV+ Zellen im Vergleich zu nicht infizierten Zellen isoliert von der gleichen Probe Host differentiell exprimierten Genen und Proteinen zu beurteilen. Die Analyse ergab bisher verkannte virale RNA Ausdruck Heterogenität unter den infizierten Zellen als auch in Vivo SIV-vermittelten posttranskriptionelle Genregulation mit einzelligen Auflösung. Die tSCEPTRE Methode ist relevant für die Analyse einer Zellpopulation Identifizierung durch Ausdruck der Oberfläche Protein Marker(s), Host oder Erreger ursächlich oder Kombinationen davon zugänglich.
Einleitung
Viele intrazelluläre Erreger abhängig von Host Cell Maschinen zu replizieren, oft Host Zellbiologie zu verändern oder gezielt ganz bestimmte Subpopulationen von Wirtszellen zu maximieren Sie ihre Chancen auf Fortpflanzung. Infolgedessen sind biologischer Zellprozesse häufig gestört, mit schädlichen Folgen für die allgemeine Gesundheit des Wirts. Verständnis der Wechselwirkungen zwischen Viren und den Wirtszellen, in denen sie replizieren, wird Krankheitsmechanismen aufzuklären, die in die Entwicklung von verbesserten Therapien und Strategien helfen kann, um Infektionen zu vermeiden. Direkte Analyse Tools, mit denen das Studium der Wirt-Pathogen Interaktionen sind zu diesem Zweck unerlässlich. Einzellige Analyse bietet die einzige Möglichkeit, eindeutig einen zellulären Phänotyp einer bestimmten Genotyp oder einer Infektion Wartestatus1zuzuschreiben. Beispielsweise bedeuten pathogene Infektionen häufig sowohl direkte als auch indirekte Änderungen in Wirtszellen. Daher unterscheiden infizierte Zellen von nicht infizierten Gegenstücke ist notwendig, um Host Zelle Attributänderungen entweder direkte Infektion oder Nebenwirkungen, wie z. B. generalisierte Entzündung. Darüber hinaus für viele Krankheitserreger wie SIV und Human-Immundefizienz-Viren (HIV), Host Zelle Infektion durchläuft mehrere Phasen, wie früh, spät oder latent, von denen jedes kann durch verschiedene Gene und Protein-Expression profile2 gekennzeichnet werden , 3 , 4 , 5. Bulk Analysen der Zelle Mischungen fehl, diese Heterogenität6zu erfassen. Dagegen sehr gemultiplext einzellige Analysen den Ausdruck beider Viren quantifizieren und Host Gene bieten ein Mittel zur Infektion-spezifische zelluläre Störungen, einschließlich Variationen über Stadien der Infektion zu beheben. Darüber hinaus analysieren Wirt-Pathogen Interaktionen in physiologisch relevante Einstellungen ist entscheidend für die Identifizierung der Ereignisse, die in infizierten Organismen auftreten. Also Methoden, die angewendet werden können, direkt ex Vivo sind wahrscheinlich beste Aufnahme in Vivo verarbeitet.
SIV und HIV Ziel CD4+ T-Zellen, in denen sie Host antivirale "Einschränkung" Faktoren und Downregulate antigenpräsentierende Moleküle, um produktive Infektion zu etablieren und zu vermeiden immunüberwachung7,8, entgegenwirken, 9,10,11. Ohne Behandlung führt die Infektion zu massiven Verlust von CD4+ T Zellen letztlich gipfelte in erworbene Immunschwäche-Syndrom (AIDS)12. In der Einstellung der antiretroviralen Therapie bestehen bleiben latent infizierten Zelle Stauseen jahrzehntelang posiert eine beeindruckende Sperre zu kurativen Strategien. Verständnis der Eigenschaften von in Vivo HIV/SIV-infizierten Zellen hat das Potenzial, Host Zelle Funktionen in der Pathogenese und Beharrlichkeit zu offenbaren. Allerdings ist dies sehr, vor allem aufgrund der Low frequency der infizierten Zellen und Reagenzien in der Lage, sie leicht zu identifizieren eine Herausforderung gewesen. Zellen, die virale RNA transkribiert werden voraussichtlich bei 0,01-1 % der CD4-Zellen im Blut und Lymphgewebe13,14,15+ T. Unter suppressive Therapie sind latent infizierte Zellen sogar seltener um 10-3– 10-7 16,17,18. Virale Protein Beflecken assays, dass Arbeit auch für ein Studium in Vitro Infektionen, wie z. B. intrazellulären Gag sind suboptimale aufgrund Hintergrundfärbung von 0,01-0,1 %, ähnlich wie oder größer als die Häufigkeit der infizierten Zellen13, 14. Oberfläche Beflecken für Env Protein mit gut charakterisierten SIV von HIV/AIDS Env-spezifischen monoklonalen Antikörpern ist auch nachgewiesen worden, wahrscheinlich aus ähnlichen Gründen schwierig sein. Vor kurzem, neuartige Werkzeuge wollen die Erkennung von Zellen Gag zu äußern, indem entweder verbessern Integration Tests speziell für Gag RNA oder durch die Verwendung alternativer bildgebender Technologien14,15,19. Solche Ansätze bleiben jedoch beschränkt auf jede Zelle die Anzahl der quantitativen Messungen durchgeführt.
Wir beschreiben hier, Methodik, die (1) einzige Virus-infizierten Zellen identifiziert direkt ex Vivo von empfindlichen und spezifischen viralen Gens quantitative qPCR und (2) den Ausdruck von bis zu 18 Oberflächenproteine und 96 Gene für jeden infizierten quantifiziert (und nicht infizierte) Zelle. Diese Methode kombiniert einzelne Zelle Oberfläche Protein Messung durch FACS gefolgt von unmittelbaren Zelle Lysis und Gen-Ausdruck Analyse mit gezielten qPCR auf dem Biomark System gemultiplext. Die integrierte fluidische Schaltung (IFC) Technologie ermöglicht Multiplex Quantifizierung 96 Gene von 96 Proben gleichzeitig erreicht, indem eine Matrix von 9.216 Kammern, in denen die einzelnen qPCR Reaktionen durchgeführt werden. Die live FACS Zellsortierung zeichnet High-Content Protein Fülle Messungen während Erhaltung der gesamten transkriptoms für Analyse sofort durchgeführt stromabwärts. Um Virus-infizierten Zellen zu identifizieren, sind Tests speziell für Alternativ gespleißt und unspliced viralen RNAs (vRNA) enthalten in der qPCR-Analyse, zusammen mit einer Gruppe von benutzerdefinierten Assays in Höhe von bis zu 96 Gene, die Höchstzahl Assays, die derzeit untergebracht die IFC. Die Genexpression und Protein erfassten Daten für die einzelnen Zellen sind durch gut Position verbunden. Wir berichteten bereits Ergebnisse aus dieser Analyse an anderer Stelle20. Hier bieten wir detaillierte methodische Richtlinien sowie weitere beschreibende Phänotypisierung von SIV-infizierten CD4+ T Zellen.
Dieser Ansatz, den wir tSCEPTRE nennen, kann an den Aufhängungen lebensfähigen Zellpopulation reaktiv, eindringmittel beschriftete Antikörper und mit dem Ausdruck einer Transkriptom kompatibel zur Verfügung qPCR Tests angewendet werden. Beispielsweise kann es für die Charakterisierung von differenziellen gen- und Proteinexpression in seltenen Zellen oder Zellen, die nicht leicht zu unterscheiden durch Oberfläche Protein-Marker verwendet werden. Die Probenvorbereitung stützt sich auf einen Standard Färbeprotokoll mit handelsüblichen Antikörpern. Cytometers mit einzelligen Sortier-Funktion sind auch im Handel erhältlich, aber zusätzliche biologische Sicherheit Vorsichtsmaßnahmen sind erforderlich für die Bearbeitung von infektiöser lebenden Zellen. Aufnahme der Single – Cell Protein Expressionsprofil für jede Zelle durch gut Lage, hierin ist als indiziert, Sortierung, ein gemeinsames Merkmal der im Handel erhältlichen FACS sortieren Software. Computergestützte Analyse von Host differentiell exprimierten Gene zwischen Zell-Populationen von Interesse ist hier nicht beschrieben, sondern Verweise werden bereitgestellt, um zuvor veröffentlichten Methoden.
Protokoll
Hinweis: Eine schematische Darstellung des Protokoll-Workflow ist in Abbildung 1dargestellt. Es besteht aus drei wesentlichen Schritten: FACS, RT und cDNA Vorverstärkung und qPCR für bis zu 96 Gene gleichzeitig. Zwei Versionen des Protokolls, Zellen in Verdünnungen zu begrenzen und einzelne Zellen Sortieren Sortierung werden bzw. in Schritt 5 und 6, ausführlicher beschrieben. Diese Strategien befassen sich verschiedene Forschungsfragen doch ähnliche Verfahren zu folgen.
1. Voraussetzung oder vorherige Analysen
- Überprüfen Sie alle Gen-Ausdruck-Assays als zuvor beschrieben6verwendet werden.
Hinweis: Dieser Schritt ist gut vor dem Experiment durchgeführt. Überprüfen alle Assays, ist kommerzielle und Brauch, erforderlich, um effizient und Lineare Verstärkung der entsprechenden RNA bis auf die einzelne Zelle Ebene zu gewährleisten. Viele kommerziell verfügbar und benutzerdefinierte Tests fehl, um diese Anforderungen zu erfüllen. Verarbeitung und automatische Kurvenanpassung für die gleichzeitige Qualifikation Ausdruck Proben von bis zu 96 Gene werden in Zusätzliche Codierung Dateien 1 – 5zur Verfügung gestellt, aber einzelne Assays qualifizierbaren mit R2 und Steigung des linearen Fit. Repräsentative erfolgreiche und fehlgeschlagene Assay Qualifikation Parzellen sind in Abbildung 2dargestellt. -
Entwickeln Sie ein Fluss durchflusszytometrischen Panel von Antikörpern, Zelle oberflächenmarker von Interesse zu beflecken.
- Titrieren Sie Antikörper durch eine entsprechende Probe, z. B. Rhesus peripheren mononukleären Blutzellen (PBMCs), Färbung, mit jeder Antikörper. Beginnen Sie mit 20 µL pro Test in 100 µL Antikörper Färbung Reaktion und acht zweifache Verdünnungsreihen zu schaffen. Ermitteln Sie die optimale Konzentration, die die maximale Intensität und gleichzeitig eine klare Trennung zwischen den negativen und positiven Populationen Färbung aufweist.
- Werten Sie die kombinierte Färbung auf zusätzliche Zelle Probe(n) mit der Mischung aus alle Antikörper bei der optimalen Konzentration in Schritt 1.2.1 ermittelten. Stellen Sie sicher, dass die Färbung ähnelt, für einzelne Antikörper Flecken beobachtet. Wenn die Färbung weniger ist als was beobachtet wird, wenn Antikörper isoliert benutzt wurde, betrachten Sie alternative Fluorochrom konjugiert, um solche Antikörper zu ersetzen.
(2) Gen Ausdruck Test Vorbereitung
- Genexpression kombinieren 96 Proben in ein RNase/DNase-frei 1 – 15 mL Tube (Größe variieren mit der Anzahl der Art Platten). Das Panel in dieser Studie verwendeten Assays wird in Tabelle 1 und Tabelle 2angegeben. Das resultierende Material ist als die "Test-Mix" bezeichnet. Fügen Sie jedem Assay, eine Endkonzentration von 180 nM von vorwärts- und Grundierungen. Fügen Sie DNA-Suspension Puffer um die entsprechende Verdünnung der Assay-Mix zu erreichen.
Hinweis: Aus praktischen Gründen können benutzerdefinierte (User generated) Assay Bestände bei 18 µM bereit sein, die Konzentration der im Handel erhältlichen "20 X" Gen Ausdruck qPCR Assays (Table of Materials) entsprechen. 18 µM Mischungen von benutzerdefinierten vorwärts- und Primer für jedes Gen bestehen aus Stammlösungen in der DNA-Suspension-Puffer. Kommerziell erhältliche Tests (Table of Materials) Sonden unter anderem auch Sonden sind nicht erforderlich für RT oder cDNA Vorverstärkung und kann somit für benutzerdefinierte Assays weggelassen werden. Es empfiehlt sich, einen oder mehr Housekeeping-Gene für Verwendung in der Qualitätskontrolle für die Beurteilung der Effizienz der Sortierung, Zelle Erholung und cDNA Synthese. Verwendung von zufälligen Primern, cDNA zu generieren ist nicht festgestellt worden, aber ist weniger effizient als gen-spezifische Primer erwartet. - Bereiten Sie die 2 x Assay Platte für Schritt 7.1 (Multiplex-qPCR). Für jedes 96 x 96 Chip Array erwartet pipette 6 µL jedes Assay in jede gut von einer 96-Well-PCR-Platte bezeichnet. Beispielsweise wird für 5 Chips 30 µL pro Test einen einzigen Brunnen in 96-Well-Platte zu besetzen. Wenn 96 Tests verwenden, wird jede Vertiefung des 96-Well-Platte einen Test enthalten. Dichtplatte mit selbstklebenden Siegel.
Hinweis: Im Idealfall Schritte 2.1 und 2.2 gleichzeitig durchgeführt, um mehrere Gefrier-tau-Zyklen für Gen-Ausdruck-Assays zu vermeiden. Aller Gene innerhalb der Assay-Platte müssen auch in der Assay-Mix (Schritt 2.1) vorhanden gewesen sein. Der Assay-Mix und der Assay-Platte können bei-20 ° C oder 4 ° C für lang- oder kurzfristige Verwendung gespeichert werden.
3. Oberfläche Fleck lebensfähige Zellen
Hinweis: Die intrazelluläre Färbung, Permeabilisierung und Fixierung sind nicht kompatibel mit dieser Methode, da sie RNA beeinträchtigen.
- Bereiten Sie die Entschädigung Proben durch Zugabe von jeder Antikörper aufgeführt in der Tabelle der Materialien zu 40 µL von Entschädigung Perlen an 2,5-fache höhere Konzentration als bei Zelle färben vor. Inkubieren Sie für 20 min bei 25 ° C, vor Licht geschützt werden. Hinzu kommen 3 mL PBS die Perlen und Zentrifuge bei 500 X g für 3 min bei 25 ° C. Aspirieren der PBS und die Perlen in ~ 300 µL PBS aufzuwirbeln.
- Probenverarbeitung Fluss durchflusszytometrischen Zelle Sorter vorbereiten: Entschädigung Röhren zu erwerben, erstellen vergütungsmatrix und Matrix auf der Erwerb Dateien für die experimentellen Proben anwenden.
- Bereiten Sie den master-Mix cocktail fluoreszierende Antikörper durch die Kombination des entsprechenden Volumens der jeder Antikörper wie angegeben in der Tabelle der Materialien, in der Bernstein 1,5 mL Tube für alle Proben gefärbt werden. Wirbel und Zentrifuge Aggregate der Cocktail bei 21.000 X g für 2 min bei 25 ° C, pellet-Antikörpers.
Hinweis: Die hier verwendeten Antikörper sind in der Tabelle der Materialienaufgeführt. - Die kryokonservierten Zellen in einem 37 ° C Wasserbad für 2 min. Add 0,5 – 2 mL Zellsuspension auf 12 mL PBS in einer 15 mL Tube, Zentrifuge bei 500 X g für 3 min bei 25 ° C, Auftauen und Aspirieren der PBS. In 3 mL PBS und Transfer zu einem 5 mL Polystyrol Schlauch aufzuwirbeln. Wie oben beschrieben Zentrifugieren Sie und Aspirieren Sie PBS, ~ 20 µL residual PBS zu verlassen.
Hinweis: Die Färbung Temperatur kann wärmere oder kältere Temperaturen angepasst werden, Bedarf für spezifische Anwendungen durch Ändern der Antikörper-Titration Färbung Bedingungen entsprechend (siehe Schritt 1.2). - Aufschwemmen Sie bis zu 2 x 107 Zellen in 80 µL Antikörper cocktail gewaschen und inkubieren Sie für 20 min bei 25 ° C, vor Licht geschützt. Für Proben, die von mehr als 2 x 107 Zellen, erhöhen die Färbung Reaktionsvolumen entsprechend zur Aufrechterhaltung < 2 x 107 Zellen/100 µL.
- Waschen der Zellen durch Zugabe von 3 mL PBS, 500 X g für 3 min zentrifugieren und Absaugen des Überstands.
- Gründlich Aufschwemmen der Zellen in 300 – 500 µL PBS und Filter durch pipettieren durch eine 35 µm Nylon Sieb Zellenabdeckung. Halten Sie die Zellen auf Eis und vor Licht geschützt werden, bis die Art.
4. bereiten Sie Sammlung Zellplatten, führen FACS sortieren und generieren cDNA
- Kombinieren Sie die RT-Preamp Reaktion Mix-Komponenten (Tabelle 3), indem in einem einzigen RNAse/DNAse-freie steriles Röhrchen pipettieren.
Hinweis: Dieser Schritt kann vor oder während der Färbung in Schritt 3 ausgeführt werden. Das RT-Enzym kann hier entfallen, zu bestimmen, den Beitrag der DNA Schablone qPCR-Signal. - Verwenden Sie eine Mehrkanal-Pipette 10 µL RT-Preamp-Reaktion-Mix in der gewünschten Anzahl der 96-Well-PCR-Art-Sammlung-Platten zu verzichten. Versiegeln Sie die Platten mit Klebefolie, und legen Sie die Platten auf vorgekühlt 96-Well-Aluminium Blöcke.
- Die Zelle Sortieren gating Schema auf das Durchflusszytometer durch die Übernahme von Daten aus rund 20.000 Zellen der gefärbten Probe zu etablieren. Sicherstellen Sie, dass die Entschädigung-Matrix auf die gesammelten Daten angewendet wird. Zeichnen Sie Tore und definieren Sie die gating Baum, der die Zelle Bevölkerungsgruppen von Interesse für Gen Expressionsanalyse isoliert werden identifiziert.
Hinweis: Die gating Baum verwendet für die Auflistung der möglichen SIV vRNA+ Zellen ist in Abbildung 3dargestellt. - Geben Sie die geeignete Instrumenteinstellungen angeben, die Anzahl und die Teilmenge von Zellen in jedes gut sortiert werden. Weitere detaillierte Anleitung für die Sortierung entweder sind eine begrenzende Zelle Verdünnungsreihen oder einzelne Zellen in den Schritten 5 und 6, jeweils vorgesehen.
- FACS sortieren Sie die Zellen in vorbereiteten 96-Well-PCR-Sammlung-Platten. Entfernen Sie die selbstklebende Dichtung vor der Sortierung und ersetzen Sie durch eine neue Dichtung nach der Art.
Hinweis: Halten Sie die Platten auf vorgekühlt Aluminium Blöcke zu allen Zeiten, einschließlich während der Sortierung. - Unmittelbar nach der Art, die Wirbel und die Zentrifuge die Sammlung Platte bei 2.000 X g für 1 min bei 4 ° C.
- Thermocycle der Platte in einer PCR-Maschine mit einem vorgeheizten Deckel mit den folgenden Bedingungen: 50 ° C für 15 min (RT), 95 ° C für 2 min, gefolgt von 18 Zyklen von 95 ° C für 15 s und 60 ° C für 4 min (Vorverstärkung).
- Verdünnen Sie die cDNA 1:5 von 5 μl cDNA in 20 µL DNA Aussetzung Puffer in einer neuen 96-Well-PCR-Platte zu übertragen. Verdünnte cDNA kann bei 4 ° C oder-20 ° C auf unbestimmte Zeit an dieser Stelle gespeichert werden. Die cDNA kann nun als Vorlage für qPCR (Schritte 5.2, 7.4) verwendet werden.
Hinweis: Diese Verdünnung sorgt dafür, dass die Primer in der RT-Preamp-Reaktion vorhanden nicht zur nachgelagerten qPCR beitragen.
5. Variante A: FACS Art Zellen in eine Begrenzung der Verdünnungsreihe zu bestimmen, die Häufigkeit von vRNA+ Zellen oder die experimentelle Qualitätskontrolle durchführen
Hinweis: Bevor Sie eine einzelne Zelle Art durchführen, kann es für die Häufigkeit der Zellen von Interesse, zu bestimmen, durch die Sortierung der Zellen in seriellen Verdünnungen in Wiederholung von Nutzen sein. Dieser Schritt bietet auch wertvolle Qualitätskontrolle für Art Effizienz, Zelle Lysis, RNA-Recovery und cDNA Synthese, wie unter Punkt 5.3 beschrieben. Vorherige Ermittlung der vRNA+ Zelle Frequenz ermöglicht eine genauere Schätzung der Anzahl der Einzelzellen, die sortiert werden müssen, um ausreichende Stichprobengröße für entsprechend betriebenen vRNA+ Zellanalyse Gen Ausdruck zu erreichen.
- FACS sortieren Sie die Zellen in der 96-Well-Platten, wie in den Schritten 4.1-4.2, vorbereitet und 1 – 1.000 Zellen pro Bohrloch in mehreren Wiederholungen zu sammeln.
Hinweis: Die Anzahl der replizieren Wells in jeder Zelle Verdünnung ist in der Regel umgekehrt die Zellkonzentration pro Bohrloch zugeordnet. Wenn die infizierte Zelle Frequenz weit unter 1 % ist, sollte auf 100 – 1.000 Zellen pro Bohrloch Zelle Verdünnungen konzentrieren. In Abbildung 1, oben links ist eine Beispiel Art Platte Karte kostenfrei. Mehr als 1.000 Zellen pro Bohrloch sollte vermieden werden, aufgrund der daraus resultierenden steigt in das Reaktionsvolumen und Interferenzen mit nachgeschalteten cDNA Synthese und Quantifizierung. - Kombinieren Sie die qPCR-Reagenzien in einer master-Mix-Lösung in Tabelle 4. Für eine 25 µL Reaktionsvolumen ist 22,5 µL des master-Mix mit 2,5 µL verdünnter cDNA-Vorlage aus Schritt 3,9 kombiniert. Durchführen der qPCR mit Radsport Standardbedingungen (z. B.94 ° C für 5 min, gefolgt von 40 Zyklen von 94 ° C für 15 s und 60 ° C für 1 min).
Hinweis: Singleplex qPCR Reaktionen mit einem herkömmlichen Echtzeit-qPCR-Instrument sind als vorläufige wirtschaftliche Analyse von einem oder wenigen Assays empfohlen, um effiziente Zellsortierung, RNA-Recovery und cDNA Synthese zu demonstrieren. Es kann auch verwendet werden, um die Häufigkeit von vRNA+ Zellen zu berechnen. Multiplex-qPCR Reaktionen durch die Biomark sind in der Regel besser geeignet für großflächige einzellige Analysen. - Für die Qualitätskontrolle, plot Et Werte (Et = Ctmax − Ct) versus Zahl von Zellen sortiert pro Bohrloch auf einer Skala von10 Log und anwenden eine linearen Regressionsanalyse.
Hinweis: Konsequente Wiederholungen, lineare Regression Hang von 3,3 (± 0,3) und R2 > 0,9 sind kennzeichnend für eine effiziente Experiment. Beispiele für optimale und suboptimale Art, sind RT-Preamp Experimente in Abbildung 4dargestellt. - Zur Bestimmung der Häufigkeit von vRNA+ Zellen, plot Zellzahlen sortiert pro Bohrloch auf der x-Achse (Log10 Skala) und der Anteil der Brunnen positiv für vRNA bei jeder Zelle Verdünnung auf der y-Achse. Ein Beispiel finden Sie in Abbildung 1 (untere linke, Poisson-Verteilung). Wenden Sie ein lineares Regressionsmodell auf die Daten, die Anzahl der Zellen zu bestimmen, die eine positive Zelle im Durchschnitt Hafen von 63,2 % des Brunnen positiv (0.632 auf der y-Achse)21 an. Wandeln Sie diese Verdünnung Zellzahl (x-Achse abfangen) in Frequenz ausgedrückt als Prozentsatz. Beispielsweise entspricht eine vRNA+ Zelle pro 48 Zellen einer Frequenz von 2,1 %.
6. Variante B: FACS Art Zellen für einzellige Analyse
- Führen Sie die Schritte 4.1 – 4,8, und geben Sie eine Zelle pro nun mit dem Durchflusszytometer Index Sortierfunktion erstelle ich einzelne FCS-Dateien für jede Zelle sortiert, sortiert durch gut Position zugeordnet.
Hinweis: Übersteigt die Anzahl der Art Sammlung Platten die Anzahl der verfügbaren Thermocycler, Radfahren kann gestoppt werden, nach dem Reverse-Transkriptase-Inaktivierung Schritt (95 ° C für 2 min) und cDNA kann bei 4 ° C gelagert werden, bis Thermocycler verfügbar sind. In diesem Fall beginnen Vorverstärkung im ersten Zyklus der 95 ° C für 15 s. - Optional: CDNA "Pools" bestehend aus cDNA von Benutzer-definierten Chargen von Einzelzellen zu Bildschirm für seltene Zellen mit residual unverdünnt cDNA zu erstellen. Übertragen Sie 2 µL unverdünnte einzellige vorverstärkt cDNA von Mehrkanal-Pipette in eine neue 96-Well-Platte. Wiederholen Sie, indem pipettieren 2 µL cDNA aus alle zusätzlichen einzelnen Zellen eines bestimmten Pools in gleich gut. Bildschirm cDNA-Pools für ursächlich für diejenigen bestimmen, die positive Zellen unter Verwendung von herkömmlichen qPCR enthalten (z.B. vRNA) interessant. Da nur eine kleine aliquoten cDNA von jeder Zelle Bündelung benötigt werden, steht die restlichen ~ 8 µL cDNA für einzellige Analysen.
Hinweis: Pooling Strategien sind empfohlen, vor der Durchführung von einzelligen Genexpressionsanalysen in dem Bemühen, die Zahl der Einzelzellen von ressourcenintensiven multiplex qPCR verhört. Es eignet sich für Situationen, in denen die Zellen von Interesse (z.B. SIV mRNA +) durch eine vorläufige Single-Plex qPCR Assay identifiziert werden können. Einfachere Hochdurchsatz-Strategien cDNA-Pools zu erstellen gehört die Zusammenführung 2 µL unverdünnte cDNA aus einer Sammlung Art Platte (d. h. alle 12 in der Zeile A Zellen) Zeilen oder Spalten (d. h. alle 8 in Spalte 1 Zellen) in einem einzigen Brunnen in einer neuen Platte. Um die beste Strategie bündeln zu bestimmen, betrachten Sie die erwartete Häufigkeit der Zellen von Interesse aus Schritt 5.4. Zum Beispiel wenn 10 % der Zellen positiv sein sollen, Pools bestehend aus sechs einzellige cDNA Proben häufig negativ sein werden und die Zellen in diesem Pool repräsentiert somit aus nachgelagerten einzellige Analysen ausgeschlossen werden können.
(7) multiplex qPCR auf der Biomark Plattform
Hinweis: Dieser Abschnitt kann entweder Version A oder B, die oben beschriebenen folgen. In der hier beschriebenen Studie wurde es ausschließlich auf einzellige Analyse angewendet.
- Bereiten Sie die qPCR-Assay-Platte durch pipettieren 4 µL jedes Assay von 2 x Assay-Platte (vorbereitet in Schritt 2.2 vor) in eine neue 96-Well-PCR-Platte mit 4 µL Assays laden Reagenz in jedem gut. Pflegen der Assay-Platte bei 4 ° C.
Hinweis: Die Test-Platte ist stabil bei 4 ° C bis zu einer Woche und bei-20 ° C für einen Monat. So ist es möglicherweise sinnvoll, um genügend Material für mehrere Chips vorzubereiten und entsprechend zu speichern. - Die Kontrolle Linie Flüssigkeiten von der Grundierung Spritzen in zwei Einlassventile des Chips zu verzichten. Entfernen Sie die Schutzfolie unterhalb der Platte. Platzieren Sie den Chip auf einem IFC-Controller mit der eingekerbten Seite an der A1-Position. Wählen Sie im Hauptmenü das "Prime"-Skript. Führen Sie das Skript.
- Bereiten Sie die Real-Time Reaktion Mischung durch Mischen von 50 µL Probe laden Reagenz mit 500 µL des PCR Master Mix (Table of Materials) für jeden Mikrofluidik-Chip. Pipette 4.4 µL in jede Vertiefung eine neue 96-Well-PCR-Platte, fortan als "Probenteller" bezeichnet.
- Pipette 3,6 µL der 1:5 verdünnt cDNA von 4.8 betreten Probenteller mit Real-Time Reaktion Mischung.
Hinweis: Wenn eine PCR-Down-Auswahl für selten (z. B.vRNA+) Zellen für nachgelagerte Analyse auf dem Bildschirm wie im Schritt 6.2 beschrieben durchgeführt wurde, enthalten Sie nur die Zellen in die positive Pools vertreten. - Laden Sie nach der Fertigstellung des Chip-Grundierung die Chip-Eingänge durch Abgabe 5 µL aus der Assay-Platte in das entsprechende gut auf die eingekerbte (Assay) Seite des Chips und 5 µL aus Probenteller in das entsprechende gut auf der anderen Seite der (Muster) des Chips. Legen Sie den Chip in der IFC-Controller und führen Sie das Skript "Load Mix".
- Übertragen Sie den Chip auf der Biomark Plattform gemultiplexten qPCR durchführen. Fahren Sie mit dem Instrument-Setup und qPCR Programmierung nach Schritt für Schritt Anleitung zur Verfügung gestellt von der Real-Time-PCR-Analyse-Software und die Gene Expression (GE) 96.96 Standard v. 1-Protokoll mit 40 Zyklen der PCR. Speichern Sie die ChipRun-Datei in einem bestimmten Ordner.
Hinweis: Mehrere Chips können pro Tag und über mehrere Tage ausgeführt werden. - Die qPCR-Daten analysieren.
- Öffnen Sie die Real-Time PCR-Analyse-Software. Öffnen Sie die Datei "ChipRun.bml" aus dem "Datei | Menü öffnen".
- Suchen Sie "Chip-Explorer" und "Chip laufen Zusammenfassung" in der oberen linken Ecke des Fensters Software. Identifizieren Sie drei Komponenten der Chip laufen Zusammenfassung: Analyseansichten, Beispiel-Setup und Detector Setup.
- Klicken Sie auf "Detector Setup". Klicken Sie unter "Aufgabe" auf "Neu" und wählen Sie Container-Typ "SBS-Platte" und Container-Format "SBS96". Klicken Sie neben "Mapping", auf die..., und wählen Sie "M96-Assay-SBS96.dsp".
- Optional: Weisen Sie jeder Detektor (Assay) eine Anzahl oder Namen im Abschnitt "Name" jedes gut durch Doppelklick auf die 1St gut. Auch mit "F2" zum nächsten wechseln.
- Klicken Sie auf "Beispiel-Setup". Klicken Sie unter "Aufgabe" neben "Mapping", die..., und wählen Sie "M96-Probe-SBS96.dsp".
- Klicken Sie auf "Analysesichten". Wählen Sie unter "Aufgabe" im Register "qPCR", "Basislinienkorrektur für Linear (Derivat)" und "Ct-Schwellenwert-Methode für Benutzer (Detektoren)". Überprüfen Sie im Register "Ct Schwellenwerte" "Initialize mit Auto-Box". Klicken Sie oben auf die Schaltfläche "Analysieren".
- Klicken Sie in der oberen rechten Quadranten "Analyseansichten" auf der zweiten Registerkarte "Tabelle". Wählen Sie aus dem Dropdown-Menü "Heat Map View". Die Heatmap mit Daten wird angezeigt.
- Optional: Um einheitliche ROX Fluoreszenz auf dem Chip zu gewährleisten, wählen Sie "Bild anzeigen" anstelle von "Heat Map View" aus dem gleichen Menü. Wählen Sie in der zweiten aus dem rechten Fenster oberhalb der Heatmap "ROX". Wählen Sie in der ersten aus dem rechten Fenster eine der 1 – 40 Zyklen. Klicken Sie auf die vierte aus dem rechten Fenster Umstieg auf schwarz-weiß-Darstellung von ROX Fluoreszenz. Ein Bild erscheint, die Spritzer, Partikel, informiert oder Defekte auf dem Chip. Wenn die ROX-Einheitlichkeit grob verdeckt wird, erneut führen Sie aus, den Chip.
- Klicken Sie unter der Heatmap "Threshold" und "Log Graph". Passen Sie die Ct-Schwellenwerte manuell für jeden Melder durch Assays (Spalten der Heat map) anklicken und ziehen der Schwellenwerts wie nötig, um die Verstärkung Kurven in der exponentiellen Phase Schneiden an. Wenn fertig, klicken Sie auf "Analysieren".
- Exportieren Sie die qPCR-Daten als CSV-Datei. Importieren Sie die Daten in einem Arbeitsblatt oder einer statistischen Analyse-Software (z. B.JMP) und ordnen Sie die Ergebnisse von Probe und Assay Positionen auf dem Chip. Die Zellen basierend auf die Expression viraler Gene durch eine neue Spalte erstellen und Verwenden einer bedingten Formel in Gruppen zu organisieren. Wählen Sie unter "Analyze" "Fit Y von X", und plot-Genexpression im Vergleich zu Gruppe. Statistische Analyse anwenden.
Hinweis: Repräsentative einzellige quantitativer Ausdruck der vier SIV RNA-Spezies ist in bivariate Grundstücke in Abbildung 5Adargestellt. Quantitative Host Genexpression in SIV RNA+ Zellen ist in Abbildung 5 bgezeigt. - Extrahieren Sie quantitative Protein Ausdruckswerte aus einzelligen FACS Daten.
- Öffnen Sie die .fcs-Dateien von der FACS sortieren (Schritt 6.1), 96-Well-Platte mit FlowJo Version 9 entspricht. Wählen Sie die Datei Name markiert, "Plattform | Event Nr. Tor | Erstellen Sie indizierte Art Tore". Einzelne Zellen erscheinen zeilenweise angezeigt.
- Markieren Sie alle 96 Zellen (nicht Zeilen), und wählen Sie "Arbeitsbereich | Exportieren | Wählen Sie alle kompensierten Fluors". Klicken Sie unter "Datentyp" wählen Sie "FCS-Datei", "Exportieren" und wählen Sie einen bestimmten Ordner.
- Ziehen Sie neue .fcs-Dateien für einzelne Zellen in einen neuen FlowJo-Arbeitsbereich. Alle Zellen zu markieren, klicken Sie auf "Hinzufügen Statistik" (die "Σ"-Schaltfläche in der oberen linken Ecke) "| Meine | Alle Fluor-Parameter".
- Öffnen Sie "Table Editor", indem die vierte Schaltfläche von Links in der oberen linken Ecke. Markieren Sie alle Fluores der ersten Zelle und ziehen Sie sie in der Tabelle-Editor-Fenster. Klicken Sie in der Tabelle-Editor-Fenster auf die gleichen oben auf die Schaltfläche "Erstellen und View-Tabelle". Dies erstellt eine Tabelle mit 96 Zellen und ein numerischer Parameter für jedes Fluor.
- Kopieren Sie die Ausgabe in einer Datenbank-Software (z.B.MS Excel, JMP), entweder kopieren/einfügen oder indem Sie auf "Speichern und Launch Application" (vierter von die linke Taste oberhalb der Tabelle).
Hinweis: Dieses Verfahren ist speziell für FlowJo Version 9. FlowJo Version10 verwendet ein anderes Verfahren indizierte Daten importieren. Indizierte Bewegungsdaten können auch in JMP direkt aus CSV-Dateien, die von der Zelle Sorter kopiert/eingefügt werden.
- Einzellige FACS und qPCR Daten durch das Kennzeichen zu verschmelzen und gut zu positionieren. Grafische und statistische Analysen auf dem kombinierten einzellige gen (qPCR) und Protein-Expression (FACS)-Daten.
Hinweis: Beispiele für einzellige kombiniert qPCR und FACS-Daten sind in Abbildung 6 dargestellt (host Oberfläche Protein expressionsprofile für SIV-infizierten, gespleißte vRNA+ Rhesus-Makaken-Zellen), Abbildung 7 (CD4 -gen-Expression versus Oberfläche CD4 Proteinexpression in gespleißte vRNA+ Rhesus-Makaken-Zellen), und zuvor veröffentlicht20. Ermittlung differenziell ausgedrückt Gene in Zellen Bevölkerungsgruppen von Interesse sind einzellige Analyse Methoden zuvor beschriebenen empfohlenen20,22,23,24, welches Konto für der Anteil der Zellen positiv für ein Gen sowie die kontinuierliche gen Ausdruckswert.
Ergebnisse
Der Workflow für das gesamte Protokoll wird in Abbildung 1dargestellt. Es besteht aus zwei Varianten definiert durch die Anzahl der Zellen sortiert: entweder begrenzende Verdünnung oder als einzelne Zellen, wie im Text beschrieben. Beispiele für Primer-Sonden Qualifikation Analysen auf 2-fold RNA Verdünnungsreihen sind in Abbildung 2dargestellt. Die gating Strategie identifizieren potenzielle SIV+ -Zellen ist in Abbildung 3dargestellt. Eine erfolgreiche, suboptimale und fehlgeschlagene Qualitätskontrolle qPCR für das Housekeeping gen GAPDH auf FACS sortiert Zellen bei der Begrenzung Verdünnung sind in Abbildung 4dargestellt. Einzellige quantitativer Ausdruck der vier SIV RNA Arten und Rhesus Makake Gene, die differentiell in infizierten Zellen exprimiert wurden sind in Abbildung 5dargestellt. Vertreter bivariate, Histogramm und Scatterplots zeigen die Oberfläche Protein Expressionsprofil SIV RNA+ CD4+ T Zellen durch Durchflusszytometrie in Abbildung 6gemessen. Das Blasendiagramm Trivariate (Abbildung 7) zeigt die Beziehung zwischen Oberfläche CD3 oder CD4-Protein, CD3 oder CD4 -mRNA und quantitative virale Gene (Tat/Rev) Ausdruck in einzelnen Zellen.

Abbildung 1 : Schematische Darstellung der experimentellen Workflow zeigt drei Hauptkomponenten: fließen durchflusszytometrischen Art, reverse Transkription sowie PCR-basierter Vorverstärkung der cDNA (RT, Pre-Amp), und qPCR. Die Sortierung kann entweder eine begrenzende Verdünnung (A, grünen Hintergrund) oder als Einzelzellen (B, orange Hintergrund) durchgeführt werden. Unmittelbar nach der FACS-Art Zellen lysiert und RNA ist reverse Transkription in cDNA und vorverstärkt (RT, Pre-Amp-PCR) qPCR Vorlage vorbereiten. Begrenzende Verdünnung Arten bestimmen Sie die Frequenz der Zellen positiv für virale RNA mit Poisson-Verteilungsstatistiken sowie experimentelle Effizienz und probieren Sie Erholung. Die grünen Pfeilspitze zeigt die geschätzte Anzahl der Zellen pro auch mit einer Zelle positiv für eine virale gen (entsprechend einer 63,2 % Wahrscheinlichkeit von solchen Brunnen wird gen positiv), sortiert, die in einer Zelle Frequenz umgewandelt wird. Schätzungen der Häufigkeit können verwendet werden, um die Anzahl der Einzelzellen gesammelt in einer nachfolgenden Art (B) zu informieren. Indizierte einzellige FACS Sortierung Ablagerungen eine Zelle pro Bohrloch und generiert Datendateien für jede Zelle von gut Position innerhalb der 96-Well-Platte kommentiert. Einzellige qPCR ist in Multiplex für 96 Gene gleichzeitig durchgeführt. Kombination der Oberfläche Protein (FACS) und die mRNA Ausdruck ermöglicht eine Profilierung der einzelnen Zellen (Rechte Spalte). Eine optionale qPCR für eine virale gen durchgeführt werden kann zwischen der Vorverstärkung PCR und Multiplex qPCR (B, Mitte) Bildschirm Einzelzellen oder Pools von einzelligen cDNA Down-Select virale RNA+ Zellen oder Pools für Multiplex-qPCR-Analyse. Die Heatmap zeigt Genexpression (Ct-Wert) für 96 Assays (Spalten) und 96 Einzelzellen (Zeilen). Bitte klicken Sie hier für eine größere Version dieser Figur.

Abbildung 2: Vertreter qPCR Daten aus Grundierung Qualifikation Experimente gezeigt, für erfolgreiche (Links und Mitte) und (rechts) im Handel erhältlichen Tests fehlgeschlagen (CD6, SIV Tat/Rev., und TLR3, bzw.). Für CD6 und TLR3, RNA aus 106 FACS sortiert Rhesus-Makaken PBMC CD4 extrahiert wurde+ T-Zellen mit einem kommerziellen Kit. Acht von einem zwölf-Punkt RNA zweifache Verdünnungsreihen repliziert (0,023-48 ng RNA, entspricht 1,2 – 2.400 Zelle äquivalente vorausgesetzt 20 Pg RNA pro CD4+ T-Zellen) RT-Preamp und qPCR ausgesetzt waren. SIV Tat/Revwurde RNA aus Rhesus-Makaken PBMCs infiziert in Vitro mit SIVmac239 gewonnen. RNA-Verdünnungen wurden spanning RNA-Äquivalente von 6 – 12.000 Zellen vorbereitet. Et (40-Ct) Werte, die mit Gen-Expression zu erhöhen, sind im Vergleich zu geschätzten Zellzahlen geplottet. Verdünnungsreihe ausstellenden R2 > 0,97 und Neigung von 3,32 ±0.3 zeigen erfolgreiche Grundierung Qualifikation. Bitte klicken Sie hier für eine größere Version dieser Figur.
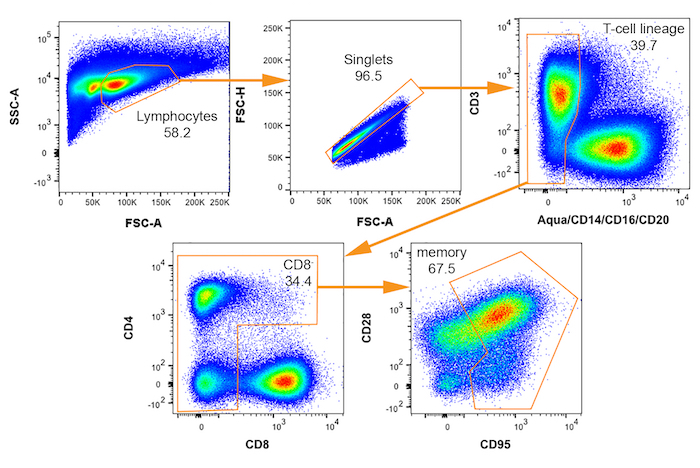
Abbildung 3: Bivariate FACS Grundstücke mit Anspritzung Schema zur Isolation von Rhesus-Makaken-Zellen möglicherweise mit SIV infiziert. Sequentielle Tore für die Auswahl von Speicher (CD95 +) CD4+ T Zellen erscheinen von links oben, rechts mit jedem angegebenen Namen der Bevölkerung zu senken. Prozent des übergeordneten Handlung, die in jedes Tor fällt wird angezeigt. Bitte klicken Sie hier für eine größere Version dieser Figur.

Abbildung 4 : Repräsentative experimenteller Validierung der qPCR durchgeführt auf FACS sortiert Zellen bei der Begrenzung Verdünnung mit GAPDH Hauswirtschaft gen für drei unabhängigen Experimenten: erfolgreiche (links), suboptimale (Mitte), und scheiterte (rechts). Wiederholungen von 10-100 Zellen pro Bohrloch sollte umfassen nicht mehr als 2 Ets und 300 – 1.000 Zelle Brunnen innerhalb 1 Et. Die lineare Regression Hang sollte 3,32 ± 0,3, R2 > 0,95. Die Nichtbeachtung dieser Vorgaben zu erreichen gibt technische Schwierigkeiten in Schritten 1, 2, 3, oder eine Kombination davon. Bitte klicken Sie hier für eine größere Version dieser Figur.

Abbildung 5 : Einzellige quantitative virale und Host Genexpression in FACS sortiert Rhesus-Makaken CD4+ T-Zellen von mesenterialen Lymphknoten 10 Tage Post-SIVmac251 Infektion. (A) virale Genexpression bivariate Grundstücke multiplizieren gespleißt (tat/Rev) und einzeln gespleißt (Env) SIV mRNA durch einzelne Zellen (links). Tat/Rev+Env– Zellen (Licht grün entlang der x-Achse) express weniger Kopien der tat/Rev RNA als Env+ Zellen (dunkelgrün), mit einer frühen Stadium der Infektion und vor dem Rev-Protein-vermittelte Stabilisierung und nukleare Export von teilweise gespleißte vRNAs wie Env. Zellen, die nicht gespleißt virale RNA Ausdrücken werden in grau (keine viralen RNA), Braun (+ Knebelund LTR+) oder tan (+ Knebeloder LTR+) dargestellt. Unspliced (gag+) und total (LTR+) SIV mRNA Ausdruck wird für die gleichen Zellen (rechts) angezeigt. Hohe Fülle von unspliced gag RNA in tat/Rev+Env+ Zellen steht im Einklang mit späten Stadium produktive Infektion während dessen drückt sich reichlich genomische RNA für die Verpackung in angehende Virionen. (B) Violine Grundstücke Rhesus-Makaken-Gene differenziell ausgedrückt in mindestens eine Teilmenge von SIV-infizierten Zellen im Vergleich zu nicht infizierten Zellen (grau). Statistische Auswertung erfolgte wie zuvor beschrieben,20,22,23,24. Sternchen gibt false Discovery Rate < 10 % in kombinierten Wahrscheinlichkeit-Verhältnis Testvergleiche im Verhältnis zu nicht infizierten Zellen. Die Linie verbindet Mittelwerte über Zellgruppen für jedes Gen. Diese Zahl wurde von Bolton Et Al. modifiziert 20 Bitte klicken Sie hier für eine größere Version dieser Figur.

Abbildung 6: Repräsentative Host Oberfläche Protein expressionsprofile von FACS sortiert Zellen von Rhesus-Makaken mesenterialen Lymphknoten 10 Tage Post-SIVmac251 Infektion. (A) Streudiagramm von CD3, CD4 und ICOS Oberfläche Protein-Expression in nicht infizierten (grau),+ KnebelTat/Rev– (braun) und+ KnebelTat/Rev+ Zellen (grün) angezeigt. Fluoreszenzintensität ist für jede Zelle (Dot) aufgetragen. Ausreißer-Box-Plots zeigen die interquartilbereich (IQR) und Median (Box), die am weitesten entfernte Punkte innerhalb 1,5 X IQR aus der Box (Schnurrhaare) und Potenzial Ausreißer (getrennte Punkte). Horizontale Balken oben zeigen signifikante Unterschiede (p < 0,05, Nichtparametrische Wilcoxon-Rank-Test). (B) Bivariate und Histogramm Anzeige der Oberfläche CD3 und CD4-Protein-Expression für Zellen, die in (A) gezeigt. Dot-Plot (links) zeigt Prozentsätze jede Zellpopulation in einem Quadranten. CD3 und CD4 Histogramme (Mitte, rechts) zeigen Oberfläche Protein Herabregulation unter+ KnebelTat/Rev+ Zellen im Vergleich zu nicht infizierten und knebeln+Tat/Rev– . (C) zwölf Vertreter Tat/Rev+ einzelne Zellen von (A – B) sind über drei bivariate Grundstücke für oberflächenexpression von CD3/CD4 (links), CD69/CD38 (Mitte) und ICOS/HLA-DR (rechts) gezeigt. Bitte klicken Sie hier für eine größere Version dieser Figur.
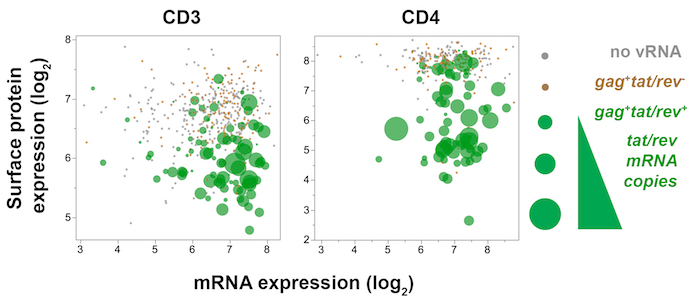
Abbildung 7 : Trivariate Grundstück Anzeige einzellige viralen Gens (Tat/Rev), Host-gen (CD3 und CD4) und Host Oberfläche (CD3 und CD4) Proteinexpression in Erinnerung SIV-infizierten CD4+ T Zellen von mesenterialen Lymphknoten 10 Tage Post-SIVmac251 Infektion. CD4-Protein-Expression (Fluoreszenz) wird geplottet gegen CD4 mRNA (qPCR), während die Menge der Tat/Rev ausgedrückt durch jede Zelle von Punktgröße reflektiert wird. In der tat/Rev+ -Zellen (grün), verminderte CD4 und CD3 Protein oberflächenexpression mit nachhaltigen CD4 und CD3 Transkripte, beziehungsweise, darauf hinweisen, dass die Oberfläche Proteinexpression flussabwärts moduliert ist der Genexpression. Bitte klicken Sie hier für eine größere Version dieser Figur.
Tabelle 1: Primer und Sonden für die Erkennung von SIV Nukleinsäuren verwendet. Wenn zwei Sequenzen für eine Grundierung oder Sonde angegeben sind, wurden äquimolaren Mengen an beiden Sequenzen verwendet. Bitte klicken Sie hier, um diese Datei herunterladen.
Tabelle 2: 96-gen-Panel für die Quantifizierung der Amplifikate auf Biomark Instrument verwendet. Vier SIV-Assays sind mit blauem Hintergrund gekennzeichnet. Bitte klicken Sie hier, um diese Datei herunterladen.
Tabelle 3: Reaktion Mischung für die reverse Transkription und Vorverstärkung. Bitte klicken Sie hier, um diese Datei herunterladen.
Tabelle 4: qPCR Reaktion Mischung verwendet für Real-Time PCR durchgeführt auf einem Instrument Quant Studio 6. Bitte klicken Sie hier, um diese Datei herunterladen.
Zusätzliche Codierung Datei 1. Anweisungen für das qualifying Gen Ausdruck Assays. Bitte klicken Sie hier, um diese Datei herunterladen.
Zusätzliche Codierung Datei 2: Beispielkarte Vorlage in JMP. Bitte klicken Sie hier, um diese Datei herunterladen.
Zusätzliche Codierung Datei 3: Sonde Kartenvorlage in JMP. Bitte klicken Sie hier, um diese Datei herunterladen.
Zusätzliche Codierung Datei 4: Primer Analyse Skript für JMP. Bitte klicken Sie hier, um diese Datei herunterladen.
Zusätzliche Codierung Datei 5. Stückweise Analyse Skript für JMP. Bitte klicken Sie hier, um diese Datei herunterladen.
Diskussion
Das Protokoll hier beschriebenen, genannt tSCEPTRE, einzellige Oberfläche Protein Quantifizierung von multiparameter Durchflusszytometrie mit quantitativen einzellige mRNA Expression von hoch Multiplex RT-qPCR integriert. Die Vereinigung dieser beiden Technologien ermöglicht High-Content Momentaufnahmen des kombinierten transkriptionelle und Protein Profil einzelner Zellen in einem Hochdurchsatz-Format. Wir verwenden die Methode bisher schwer mit SIV in Vivoinfizierte Zellen identifizieren und beschreiben Host differentiell exprimierten Genen und Proteinen. Das Protokoll kann für die Untersuchung von jeder Zellenbevölkerung von Interesse zu unterscheiden durch den Ausdruck der Oberfläche benutzt, mRNA oder eine Kombination davon angepasst werden. Die beschriebenen Methoden beruhen auf genaue einzellige sortieren und Datenaufzeichnung durch Durchflusszytometrie gepaart mit handelsüblichen qPCR Reagenzien und Multiplex Real-Time PCR-Instrumentierung. Die Ausgabe ist empfindlich, quantitative Auswertung von einzelligen kombinierten Protein und Gen Ausdruck.
Andere Ansätze, die Verknüpfung von Protein und Gen Ausdruck in Einzelzellen wurden1,25,26,27beschrieben. Indizierte FACS sortieren gefolgt von RNAseq, bis hin zur Charakterisierung von Hämatopoetischen Stammzellen erfolgreich angewendet, stellt eine besonders viel versprechende Ansatz28. Jedoch während RNAseq mehrere Vorteile gegenüber gezielte Genexpressionsanalysen, nämlich unvoreingenommene voll Transkriptom-Analyse hat, unterliegt einer höheren Mehrfachvergleiche statistische Kosten und möglicherweise weniger empfindlich für die Quantifizierung von niedrigen Kopie Transkripte. Darüber hinaus ist es nicht gerade wirtschaftlich durchführen RNAseq auf Tausenden von Zellen auf der Suche nach seltenen infizierte Zellen bei Frequenzen < 1 % (z. B.HIV, SIV). Andere Einzeller-Technologien wollen eine einzelne auslesen, d. h. durch FACS oder PCR für den Nachweis von Proteinen und Nukleinsäuren in derselben Zelle generieren. Dazu gehören die Erkennung von Proteinen durch fluoreszierende Antikörper und mRNA durch fluoreszierende Oligonukleotid Sonden gefolgt von FACS Analyse (mRNA-Flow-FISH)14,15,29,30. Alternativ können Proteine durch Paare von Antikörper-Oligonukleotid-Konjugate nachgewiesen werden, die durch qPCR parallel umgekehrt transkribierte mRNA31,32,33,34 erkannt werden . Diese "Hybrid"-Ansätze bieten simultane Nukleinsäure und Protein-Messungen mit einzelligen Auflösung ähnlich wie tSCEPTRE, aber sie beschränken sich dort sind entweder nicht quantitative (mRNA-Flow-Fish) oder erfordern maßgeschneiderte Antikörper oder mRNA Sonden. Unser tSCEPTRE Ansatz ist für Protein- und mRNA Messungen quantitativ und alle Reagenzien sind im Handel erhältlich, mit der möglichen Ausnahme von Erreger-spezifischen Assays.
Besondere Aufmerksamkeit sollte auf mehrere Schritte in das Protokoll vor der Analyse der experimentellen Proben angewendet werden. Erste, hoch-Parameter Durchflusszytometrie erfordert sorgfältige Optimierung eines Panels von fluoreszierenden Antikörpern gegen empfindliche Erkennung und Auflösung35zu gewährleisten. Jeder Antikörper sollten individuell titriert werden, um die Konzentration zu identifizieren, die optimale Trennung und minimale Hintergrundfärbung erzielt, gefolgt von der Bewertung der Färbung, wenn alle Antikörper-Konjugate in Kombination verwendet werden. Zweite, experimentelle Bestimmung des geeigneten qPCR Gen Ausdruck Assays, jedes Gen des Interesses zu messen ist wichtig. Mehreren im Handel erhältlichen Tests gibt es in der Regel für jedes Gen, aber nach unserer Erfahrung sind viele nicht quantitativ an der einzelligen Ebene6. Daher ist es wichtig, alle vorgeschlagenen Assays auf seriell verdünnt RNA vor der Verwendung für Quantitative Genexpression zu qualifizieren. Optimierung dieser Reagenzien ist zeitaufwendig, aber der Wert der zuverlässige Platten von Antikörper-Konjugate und Gen-Ausdruck-Assays, der empfindliche Detektion aller Moleküle des Interesses ermöglicht rechtfertigt die Investition. Darüber hinaus sollte Vorrang eingeräumt werden handelsübliche Assays mit Sonden, die Exon-Exon-Kreuzungen (gekennzeichnet mit Suffix "m1") zur Verbesserung der Spezifität für mRNA, obwohl alternative Erkennung von genomischer DNA (Suffix "s1" oder "g1" Tests umfassen ) werden als unwahrscheinlich gen Ausdrucksergebnisse durch gleichwertige chromosomalen Kopien in jeder Zelle zu beeinflussen. Erhaltung der RNA stromaufwärts von RT ist entscheidend und wird erreicht, indem man Proben gekühlt, wie im gesamten Protokoll erwähnt. Ebenso die Dauer der FACS sortieren und das Intervall zwischen sortieren und RT-Preamp sollte auf ein Minimum gehalten werden. Für beide begrenzende Verdünnung und einzellige sortieren kann cDNA zuerst durch konventionelle qPCR RNA Erholung der Bewertung analysiert werden, stark ausgedrückt housekeeping Gene. Wenn der beobachteten Ausdruck nicht einheitlich Wiederholungen von wie Zellzahlen oder die Steigung der linearen Fit für die Begrenzung der Verdünnungen fehlschlägt Validierung ist, sollte Fehlerbehebung bei Zelle Sortier- und nachgelagerten Verfahren durchgeführt werden.
Mögliche Einschränkungen der hier beschriebene Ansatz umfassen (1) Nachweis von DNA neben RNA, besonders für Assays nicht spezifisch für mRNA Splice Verbindungen und (2) eingeschränkte Anzahl der Oberflächenproteine und Gene gemessen. DNA-Nachweis ist jedoch unwahrscheinlich, einen wesentlichen Beitrag zur differenziellen Host Genanalyse Ausdruck aus dem oben genannten Grund. Darüber hinaus messen wir direkt das Ausmaß, in dem DNA, das qPCR-Signal in diesem Protokoll durch zwei Ansätze beiträgt: (a) ohne Reverse Transkriptase, und (b) Änderung der Zelle Lysis Protokoll zur Verbesserung der Kernmembran Lyse. In Abwesenheit von Reverse Transkriptase, beide gespleißt und unspliced viraler Gene wurden noch erkannt, aber zu deutlich niedrigeren Frequenz als in Anwesenheit von Reverse Transkriptase (~ 6-fold und 2-fold-Reduktion bzw.)20. Daher können die virale Gen Ausdruck Assays die Höhe der viralen RNA in eine Zelle aufgrund des Vorhandenseins von Zelle-assoziierten viralen DNA überschätzen. Wir führen diese Feststellung zu zytoplasmatischen RT Produkte erzeugt während der Host Zelle Infektion bekanntermaßen für gespleißt und unspliced SIV/HIV-RNA aus eingehenden Virionen36auftreten. So ist es ratsam, einen Teil von Experimenten widmen Bedingungen fehlen Reverse Transkriptase um DNA abgeleitet Vorlage für einige Anwendungen quantitate möglicherweise. Anzumerken ist, dass unspliced SIV/HIV-RNA aus eingehenden Virus abgeleitet aus de Novo synthetisiert virale RNA unterschieden werden können. Zweitens, wir das RT-Preamp-Protokoll einen nuklearen Lyse Schritt eingearbeitet und beobachteten eine exponentielle Zunahme integrierte SIV DNA (Alu-LTR) Kopien (Abbildung S3 von Bolton Et Al.) 20. Genomic DNA ist daher unwahrscheinlich, einen wesentlichen Beitrag an der Nukleinsäure-Vorlage aus der Zelle Lysis Methode hier gewonnen. Der Hinweis möglicherweise die Zugabe von einem nuklearen Lyse Schritt nützlich in Zukunft einzellige Studien SIV oder anderen Virus DNA-positiven Zellen, einschließlich der latent infizierte Zellen zu untersuchen.
Die Anzahl der Oberflächenproteine analysiert richtet sich von der Leistungsfähigkeit der den Fluss durchflusszytometrischen Zelle Sorter. Aktuellen im Handel erhältlichen Instrumente überschreiten 30 Parameter nicht. Zukünftige Studien beschäftigt erweiterte Durchflusszytometrie werden das Protein Profilerstellung Fähigkeit dieses Ansatzes weiter auszubauen. Die Anzahl der Transkripte auch über 96, bereitgestellte Unterstützung Reagenzien (z.B. Grundierungen höherer Konzentration), Ausrüstung und Instrumentierung erweiterbar. Letztlich wird neue einzellige Technologien, die Analyse des Proteoms (Massenspektrometrie) kombinieren, Transkriptom (RNAseq) und Genom (DNAseq) die gezielte Ansätze zur Entdeckung Forschung31,37, ablösen 38 , 39. gezielte qPCR bleibt jedoch wahrscheinlich ein wertvolles Werkzeug für die Validierung solcher "Omics"-Ansätze als Goldstandard für quantitative Expressionsanalysen.
Kombinierte Protein und Transkription Analyse von tSCEPTRE ist ein leistungsfähiges Werkzeug für die Untersuchung von seltenen oder schwer zu identifizieren Zellen wie diese Krankheitserreger beherbergen, mit Onkogene oder sonst eine aberrante Phänotyp auszustellen. Neue Marker für transcriptionally aktiv SIV/HIV-infizierten Zellen können in dieser Art und Weise sowie die Entdeckung von neuen Mechanismen in der Pathogenese dieser viralen Infektionen erkannt werden. Identifizierung von latent infizierten Zellen benötigen Weiterentwicklung eines Protokolls zum Esel, virale DNA in das wirtsgenom integriert. Hervorzuheben ist die Häufigkeit von HIV/SIV-infizierten Zellen deutlich niedriger in chronischen virämische oder behandelten Infektion, die eine praktische Herausforderung im Studium infizierten Zellen aus diesen Einstellungen präsentieren wird. Unser Ansatz legt den Grundstein für die Beurteilung eines zuvor unlösbaren Mechanismus: das posttranskriptionelle Verordnung Ebene der einzelligen und hat breite Anwendbarkeit zu Wirt-Pathogen Interaktionen sowie weitere allgemeine zelluläre Prozesse.
Offenlegungen
Diese Arbeit wurde durch einen Kooperationsvertrag unterzeichnet (W81XWH-07-2-0067) zwischen die Henry M. Jackson Foundation für die Weiterentwicklung der militärischen Medizin, Inc., und das US Department of Defense (DOD) unterstützt. Geäußerten Meinungen sind diejenigen der Autoren und sind nicht zur Darstellung der Positionen von der US-Armee oder das Department of Defense auszulegen. Forschung wurde durchgeführt unter einem genehmigten Tierversuche-Protokoll in einer AAALACi akkreditierten Einrichtung im Einklang mit dem Tierschutzgesetz und andere Bundesgesetze und Verordnungen in Bezug auf Tiere und Experimente mit Tieren und hält sich an Prinzipien in der Anleitung für die Pflege und Verwendung der Versuchstiere, NRC Publikation, Ausgabe 2011 angegeben.
Danksagungen
Die Autoren möchten NIAID VRC Flow Cytometry Kern und die MHRP Flow Cytometry Kern Einrichtungen für Wartung und Betrieb von FACS Instrumenten und Sortieranlagen danken; Maria Montero, Vishakha Sharma, Kaimei Song für kompetente technische Unterstützung; Michael Piatak, Jr. (verstorben), für die Unterstützung mit SIV qPCR Assay Design; und Brandon Keele und Matthew Scarlotta für SIV isolieren Sequenzen. Geäußerten Meinungen sind diejenigen der Autoren und sind nicht zur Darstellung der Positionen von der US-Armee oder das Department of Defense auszulegen. Forschung wurde durchgeführt unter einem genehmigten Tierversuche-Protokoll in einer AAALAC akkreditierten Einrichtung im Einklang mit dem Tierschutzgesetz und andere Bundesgesetze und Verordnungen in Bezug auf Tiere und Experimente mit Tieren und hält sich an Prinzipien in der Anleitung für die Pflege und Verwendung der Versuchstiere, NRC Publikation, Ausgabe 2011 angegeben.
Materialien
| Name | Company | Catalog Number | Comments |
| RNA extraction and PCR reagents and consumables | |||
| Genemate 96-Well Semi-Skirted PCR Plate | BioExpress/VWR | T-3060-1 | |
| Adhesive PCR Plate Seals | ThermoFisher | AB0558 | |
| Armadillo 384-well PCR Plate | ThermoFisher | AB2384 | |
| MicroAmp Optical Adhesive Film | Applied Biosystems/ThermoFisher | 4311971 | |
| DEPC Water | Quality Biological | 351-068-101 | |
| Glass Distilled Water | Teknova | W3345 | |
| Superscript III Platinum One-Step qRT-PCR Kit | Invitrogen/ThermoFisher | 11732088 | |
| SUPERase-In Rnase Inhibitor | Invitrogen/ThermoFisher | AM2696 | |
| Platinum Taq | Invitrogen/ThermoFisher | 10966034 | |
| dNTP Mix | Invitrogen/ThermoFisher | 18427088 | |
| ROX Reference Dye (if separate from kit) | Invitrogen/ThermoFisher | 12223012 | |
| DNA Suspension Buffer | Teknova | T0223 | |
| RNAqueous kit | Invitrogen/ThermoFisher | AM1931 | |
| TaqMan gene expression assays not listed in Table 2 | |||
| CD6 | Applied Biosystems/ThermoFisher | Hs00198752_m1 | |
| TLR3 | Applied Biosystems/ThermoFisher | Hs1551078_m1 | |
| Biomark reagents | |||
| Control Line Fluid Kit | Fluidigm | 89000021 | |
| TaqMan Universal PCR Mix | Applied Biosystems/ThermoFisher | 4304437 | |
| Assay Loading Reagent | Fluidigm | 85000736 | |
| Sample Loading Reagent | Fluidigm | 85000735 | |
| Dynamic Array 96.96 (chip) | Fluidigm | BMK-M-96.96 | |
| FACS reagents | |||
| SPHERO COMPtrol Goat anti-mouse (lambda) | Spherotech Inc. | CMIgP-30-5H | |
| CompBeads Anti-Mouse Ig,k | BD Biosciences | 51-90-9001229 | |
| 5 ml Polystyrene tube with strainer cap | FALCON | 352235 | |
| Aqua Live/Dead stain | Invitrogen/ThermoFisher | L34976 | dilute 1:800 |
| Mouse Anti-Human CD3 BV650 clone SP34-2 | BD Biosciences | 563916 | dilute 1:40 |
| Mouse Anti-Human CD4 BV786 clone L200 | BD Biosciences | 563914 | dilute 1:20 |
| Mouse Anti-Human CD8 BUV496 clone RPA-T8 | BD Biosciences | 564804 | dilute 1:10 |
| Mouse Anti-Human CD28 BV711 clone CD28.2 | Biolegend | 302948 | dilute 1:20 |
| Mouse Anti-Human CD95 BUV737 clone DX2 | BD Biosciences | 564710 | dilute 1:10 |
| Mouse Anti-Human CD14 BV510 clone M5E2 | Biolegend | 301842 | dilute 1:83 |
| Mouse Anti-Human CD16 BV510 clone 3G8 | Biolegend | 302048 | dilute 1:167 |
| Mouse Anti-Human CD20 BV510 clone 2H7 | Biolegend | 302340 | dilute 1:37 |
| Anti-CD38-R PE clone OKT10 | NHP reagent recource | N/A | dilute 1:100 |
| Mouse Anti-Human CD69 BUV395 clone FN50 | BD Biosciences | 564364 | dilute 1:10 |
| Mouse Anti-Human HLA-DR APC-H7 clone G46-6 | BD Biosciences | 561358 | dilute 1:20 |
| Mouse Anti-Human ICOS Alexa Fluor 700 clone C398.4A | Biolegend | 313528 | dilute 1:80 |
| Instruments | |||
| BioPrptect Containment Enclosure | Baker | ||
| BD FACS Aria | BD Biosciences | ||
| ProtoFlex Dual 96-well PCR system | Applied Biosystems/ThermoFisher | 4484076 | |
| Quant Studio 6 qPCR instrument | Applied Biosystems/ThermoFisher | 4485694 | |
| IFC controller HX | Fluidigm | IFC-HX | |
| Biomark HD | Fluidigm | BMKHD-BMKHD |
Referenzen
- Macaulay, I. C., Ponting, C. P., Voet, T. Single-Cell Multiomics: Multiple Measurements from Single Cells. Trends Genet. 33 (2), 155-168 (2017).
- Pasternak, A. O., Lukashov, V. V., Berkhout, B. Cell-associated HIV RNA: a dynamic biomarker of viral persistence. Retrovirology. 10, 41(2013).
- Mailler, E., et al. The Life-Cycle of the HIV-1 Gag-RNA Complex. Viruses. 8 (9), (2016).
- Varmus, H. Retroviruses. Science. 240 (4858), 1427-1435 (1988).
- Frankel, A. D., Young, J. A. HIV-1: fifteen proteins and an RNA. Annu Rev Biochem. 67, 1-25 (1998).
- Dominguez, M. H., et al. Highly multiplexed quantitation of gene expression on single cells. J Immunol Methods. 391 (1-2), 133-145 (2013).
- Tokarev, A., Guatelli, J. Misdirection of membrane trafficking by HIV-1 Vpu and Nef: Keys to viral virulence and persistence. Cell Logist. 1 (3), 90-102 (2011).
- Malim, M. H., Bieniasz, P. D. HIV Restriction Factors and Mechanisms of Evasion. Cold Spring Harb Perspect Med. 2 (5), a006940(2012).
- Sugden, S. M., Bego, M. G., Pham, T. N., Cohen, E. A. Remodeling of the Host Cell Plasma Membrane by HIV-1 Nef and Vpu: A Strategy to Ensure Viral Fitness and Persistence. Viruses. 8 (3), 67(2016).
- Simon, V., Bloch, N., Landau, N. R. Intrinsic host restrictions to HIV-1 and mechanisms of viral escape. Nat Immunol. 16 (6), 546-553 (2015).
- Kirchhoff, F. Immune evasion and counteraction of restriction factors by HIV-1 and other primate lentiviruses. Cell Host Microbe. 8 (1), 55-67 (2010).
- Wigzell, H. Immunopathogenesis of HIV infection. J Acquir Immune Defic Syndr. 1 (6), 559-565 (1988).
- Reynolds, M. R., et al. Ex vivo analysis of SIV-infected cells by flow cytometry. Cytometry A. 77 (11), 1059-1066 (2010).
- Baxter, A. E., et al. Single-Cell Characterization of Viral Translation-Competent Reservoirs in HIV-Infected Individuals. Cell Host Microbe. 20 (3), 368-380 (2016).
- Grau-Exposito, J., et al. A Novel Single-Cell FISH-Flow Assay Identifies Effector Memory CD4+ T cells as a Major Niche for HIV-1 Transcription in HIV-Infected Patients. MBio. 8 (4), (2017).
- Eriksson, S., et al. Comparative analysis of measures of viral reservoirs in HIV-1 eradication studies. PLoS Pathog. 9 (2), e1003174(2013).
- Finzi, D., et al. Identification of a reservoir for HIV-1 in patients on highly active antiretroviral therapy. Science. 278 (5341), 1295-1300 (1997).
- Chomont, N., et al. HIV reservoir size and persistence are driven by T cell survival and homeostatic proliferation. Nat Med. 15 (8), 893-900 (2009).
- DeMaster, L. K., et al. A Subset of CD4/CD8 Double-Negative T Cells Expresses HIV Proteins in Patients on Antiretroviral Therapy. J Virol. 90 (5), 2165-2179 (2015).
- Bolton, D. L., et al. Combined single-cell quantitation of host and SIV genes and proteins ex vivo reveals host-pathogen interactions in individual cells. PLoS Pathog. 13 (6), e1006445(2017).
- Quintans, J., Lefkovits, I. Precursor cells specific to sheep red cells in nude mice. Estimation of frequency in the microculture system. Eur J Immunol. 3 (7), 392-397 (1973).
- Finak, G., et al. MAST: a flexible statistical framework for assessing transcriptional changes and characterizing heterogeneity in single-cell RNA sequencing data. Genome Biol. 16, 278(2015).
- McDavid, A., et al. Modeling bi-modality improves characterization of cell cycle on gene expression in single cells. PLoS Comput Biol. 10 (7), e1003696(2014).
- McDavid, A., Finak, G., Gottardo, R. The contribution of cell cycle to heterogeneity in single-cell RNA-seq data. Nat Biotechnol. 34 (6), 591-593 (2016).
- Kok, Y. L., Ciuffi, A., Metzner, K. J. Unravelling HIV-1 Latency, One Cell at a Time. Trends Microbiol. , (2017).
- Rato, S., Golumbeanu, M., Telenti, A., Ciuffi, A. Exploring viral infection using single-cell sequencing. Virus Res. 239, 55-68 (2017).
- Wagner, A., Regev, A., Yosef, N. Revealing the vectors of cellular identity with single-cell genomics. Nat Biotechnol. 34 (11), 1145-1160 (2016).
- Nestorowa, S., et al. A single-cell resolution map of mouse hematopoietic stem and progenitor cell differentiation. Blood. 128 (8), e20-e31 (2016).
- Soh, K. T., et al. Simultaneous, Single-Cell Measurement of Messenger RNA, Cell Surface Proteins, and Intracellular Proteins. Curr Protoc Cytom. 75, 41-47 (2016).
- Kochan, J., Wawro, M., Kasza, A. Simultaneous detection of mRNA and protein in single cells using immunofluorescence-combined single-molecule RNA FISH. Biotechniques. 59 (4), 209-212 (2015).
- Stahlberg, A., Thomsen, C., Ruff, D., Aman, P. Quantitative PCR analysis of DNA, RNAs, and proteins in the same single cell. Clin Chem. 58 (12), 1682-1691 (2012).
- Assarsson, E., et al. Homogenous 96-plex PEA immunoassay exhibiting high sensitivity, specificity, and excellent scalability. PLoS One. 9 (4), e95192(2014).
- Fredriksson, S., et al. Protein detection using proximity-dependent DNA ligation assays. Nat Biotechnol. 20 (5), 473-477 (2002).
- Genshaft, A. S., et al. targeted profiling of single-cell proteomes and transcriptomes in a single reaction. Genome Biol. 17 (1), 188(2016).
- Mahnke, Y. D., Roederer, M. Optimizing a multicolor immunophenotyping assay. Clin Lab Med. 27 (3), 469-485 (2007).
- Liang, C., Hu, J., Russell, R. S., Kameoka, M., Wainberg, M. A. Spliced human immunodeficiency virus type 1 RNA is reverse transcribed into cDNA within infected cells. AIDS Res Hum Retroviruses. 20 (2), 203-211 (2004).
- Stegle, O., Teichmann, S. A., Marioni, J. C. Computational and analytical challenges in single-cell transcriptomics. Nat Rev Genet. 16 (3), 133-145 (2015).
- Wu, M., Singh, A. K. Single-cell protein analysis. Curr Opin Biotechnol. 23 (1), 83-88 (2012).
- Haque, A., Engel, J., Teichmann, S. A., Lonnberg, T. A practical guide to single-cell RNA-sequencing for biomedical research and clinical applications. Genome Med. 9 (1), 75(2017).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten