Method Article
Unicellulaire quantification des ARNm et Expression de protéines de Surface CD4 infectées par le Virus d’immunodéficience simien+ T les cellules isolées de macaques rhésus
Dans cet article
Résumé
Décrit est une méthodologie pour quantifier l’expression des 96 gènes et 18 protéines de surface de cellules uniques ex vivo, permettant d’identifier de façon différentielle exprimée des gènes et des protéines dans les cellules infectées par le virus par rapport aux cellules non infectées. Nous appliquons la démarche d’étude CD4 infectés par le SIV+ T cellules isolées de macaques rhésus.
Résumé
Analyse de cellules individuelles est un important outil de dissection des populations hétérogènes de cellules. L’identification et l’isolement de cellules rares peuvent être difficiles. Pour relever ce défi, une combinaison de méthodologie indexé de cytométrie en flux et haut débit multiplex quantitative PCR (qPCR) a été développé. L’objectif était d’identifier et de caractériser le virus de l’immunodéficience simienne (vis)-infecté les cellules présentes au sein de macaques rhésus. Par dosage de la protéine de surface par un tri de cellule activée par fluorescence (FACS) et de l’ARNm par qPCR, cellules infectées par le virus sont identifiés par l’expression des gènes viraux, qui est combinée avec les mesures de gènes et des protéines hôte pour créer un profil multidimensionnel . Nous appelons l’approche, évaluation unicellulaires Proteo-transcriptional ciblée ou tSCEPTRE. Pour exécuter la méthode, des cellules viables sont colorées avec des anticorps fluorescents spécifiques de marqueurs de surface utilisés pour isoler les FACS d’un sous-ensemble de cellules et/ou d’analyse phénotypique en aval. Cellules individuelles sont triés suivis par lyse immédiate, multiplexe transcription inverse (RT), les pré amplification par PCR et haut débit qPCR de jusqu'à 96 transcrits. Les mesures de FACS sont enregistrées au moment du tri et liés ultérieurement pour les données d’expression de gène en position bien à créer une protéine combinée et le profil transcriptionnel. Pour étudier les infectés par le SIV cellules directement ex vivo, les cellules ont été identifiés par la détection de qPCR de multiples espèces d’ARN virales. La combinaison des transcriptions virales et la quantité de chaque fournissent un cadre pour la classification des cellules en étapes distinctes du cycle de vie viral (p. ex., productives et non productives). En outre, tSCEPTRE des cellules SIV+ ont été par rapport à des cellules isolées du même spécimen pour évaluer des protéines et gènes différentiellement exprimés hôtes. L’analyse a révélé précédemment incomprises hétérogénéité d’expression de RNA virale chez les cellules infectées comme in vivo la régulation des gènes post-transcriptionnels induite par le SIV avec résolution unicellulaire. La méthode tSCEPTRE est pertinente pour l’analyse d’une population cellulaire se prête à l’identification par l’expression du marqueur de la protéine de surface, ou des gènes pathogène ou d’un hôte ou une combinaison.
Introduction
De nombreux pathogènes intracellulaires dépendent de machinerie cellulaire hôte à répliquer, souvent modifier la biologie cellulaire hôte ou ciblage très certaines sous-populations de cellules de l’hôte pour maximiser leurs chances de propagation. Ainsi, les processus biologiques de cellule sont communément perturbés, avec des conséquences délétères sur la santé globale de l’hôte. Compréhension des interactions entre les virus et les cellules de l’hôte dans lequel ils répliquent élucidera les mécanismes de la maladie qui pourraient contribuer à l’élaboration de stratégies et d’amélioration des traitements pour prévenir l’infection. Les outils d’analyse directes qui permettent l’étude des interactions hôte-pathogène sont essentiels à cette fin. Analyse de cellule unique fournit le seul moyen d’attribuer sans ambiguïté un phénotype cellulaire à un génotype particulier, ou infection état1. Par exemple, les infections pathogènes induisent fréquemment des changements tant directes qu’indirectes dans les cellules de l’hôte. Par conséquent, distinguer les cellules infectées de leurs homologues non infectés est nécessaire à l’attribut change de cellule hôte à une infection directe ou des effets secondaires, tels que généralisé l’inflammation. En outre, de nombreux agents pathogènes, tels que SIV et des virus de l’immunodéficience (VIH), infection de cellule hôte procède par étapes multiples, tels que dès le début, fin, ou latente, dont chacun peut être caractérisée par gènes distincts et protein expression profils2 , 3 , 4 , 5. analyses en vrac des mélanges de cellules vont échouer à capturer cette hétérogénéité6. En revanche, fortement multiplexés analyses unicellulaires capables de quantifier l’expression des deux virale et gènes hôtes offrent un moyen de résoudre les perturbations cellulaires spécifiques à l’infection, y compris les variations à travers des stades de l’infection. En outre, analyse des interactions hôte-pathogène chez physiologiquement paramètres pertinents est essentiel pour l’identification des événements qui se produisent dans les organismes infectés. Ainsi, les méthodes pouvant être appliquées directement ex vivo sont susceptibles de la meilleure capture en vivo traite.
SIV et VIH cible CD4+ T des cellules, dans lesquelles ils lutter contre les facteurs antiviraux « restriction » de l’hôte et atténuent présentatrices d’antigènes molécules afin d’établir une infection productive et éviter la surveillance immunitaire7,8, 9,10,11. Sans traitement, l’infection se traduit par une perte massive de CD4+ T cells, finalement sanctionnée par acquis d’immunodéficience (sida)12. Dans le cadre d’une thérapie antirétrovirale, réservoirs de cellules infectées de façon latente persistent depuis des décennies, posant un obstacle redoutable à stratégies curatives. Comprendre les propriétés de in vivo des cellules infectées par le VIH/SIV a le potentiel pour révéler les caractéristiques des cellules hôte joué un rôle dans la pathogénie et la persistance. Toutefois, cela a été très difficile, en raison surtout de la low frequency des cellules infectées et manque de réactifs capables de les identifier facilement. Les cellules qui transcrivent l’ARN viral, sont estimés à présent à 0,01 – 1 % de CD4 des cellules dans le sang et les tissus lymphoïdes13,14,15+ T. Sous un traitement suppressif, cellules infectées de façon latente sont encore moins fréquentes à 10-3-10-7 16,17,18. Coloration de la protéine virale dosages que bien pour l’étude des infections in vitro , par exemple pour des Gag intracellulaire, sont sous-optimaux en raison de la coloration de fond de 0,01 à 0,1 %, similaire ou supérieure à la fréquence des cellules infectées13, 14. Surface de coloration pour protéine Env en utilisant des anticorps monoclonaux spécifiques au SIV/VIH Env bien caractérisés aussi a été prouvée que sera difficile, probablement pour des raisons similaires. Récemment, les nouveaux outils visent à améliorer la détection des cellules exprimant le Gag soit par intégrant des analyses spécifiques pour gag RNA ou à l’aide de moyens d’imagerie technologies14,15,19. Cependant, ces approches restent limités dans le nombre de mesures quantitatives effectuées sur chaque cellule.
Nous décrivons ici la méthodologie qui (1) identifie les cellules infectées par le virus directement ex vivo de qPCR quantitative sensible et spécifique des gènes viraux et (2) quantifie l’expression des protéines de surface jusqu'à 18 et 96 gènes pour chaque infecté (et cellules non infectées). Cette méthode combine la mesure de la protéine de surface monocellulaire de FACS, suivie de la lyse cellulaire immédiate et à l’aide de gene expression analyse multiplexés qPCR ciblée sur le système Biomark. La technologie intégrée circuit fluidique (IFC) permet multiplexé dosage des 96 gènes 96 échantillons simultanément, accompli par une matrice de 9 216 chambres dans lesquelles sont effectuées les réactions individuelles qPCR. Le tri des cellules vivantes FACS enregistre les mesures d’abondance haute teneur protéique tout en préservant le transcriptome de tout analyse effectuée immédiatement en aval. Pour identifier les cellules infectées par le virus, dosages spécifiques pour l’ARN viral alternativement épissé et unspliced (vRNA) sont inclus dans l’analyse de qPCR, avec un panel de tests définis par l’utilisateur totalisant jusqu'à 96 gènes, le nombre maximal d’essais actuellement logés dans la SFI. Les l’expression des gènes et les protéines informations collectées pour chaque cellule sont reliées par une position bien. Nous avons déjà indiqué les résultats de cette analyse ailleurs20. Ici, nous fournissons plus d’orientations méthodologiques, mais aussi plus descriptive phénotypage des CD4 infectés par le SIV+ T des cellules.
Cette approche, que nous appelons tSCEPTRE, peut être appliquée à des suspensions d’une population de cellules viables réactive anticorps fluorescent étiquetés et exprimant un transcriptome compatible avec des essais de qPCR disponible. Par exemple, il peut être utilisé pour caractériser les gènes différentiel et expression de la protéine dans les cellules rares ou pas facilement distingués par des marqueurs de la protéine de surface des cellules. La préparation de l’échantillon se fonde sur une norme souillant le protocole utilisant des anticorps disponibles dans le commerce. Cytomètres de flux avec capacité de tri unicellulaires sont également disponibles dans le commerce, mais des précautions biosécurité supplémentaires sont nécessaires pour le traitement des cellules vivantes infectieuses. Enregistrer le profil d’expression de protéines monocellulaires pour chaque cellule de position bien, mentionné aux présentes comme indexée tri, est une caractéristique commune des FACS disponibles dans le commerce, logiciel de tri. Analyse computationnelle de gènes différentiellement exprimés hôte parmi les populations de cellules d’intérêt n’est pas décrit ici, mais les références sont fournies aux méthodes publiées antérieurement.
Protocole
Remarque : Un schéma du flux de protocole est illustré à la Figure 1. Il se compose de trois étapes principales : FACS, RT et préamplification ADNc et qPCR jusqu'à 96 gènes simultanément. Deux versions du protocole, tri des cellules en limitant les dilutions et le tri des cellules individuelles, sont décrites en détail dans les étapes 5 et 6, respectivement. Ces stratégies aborder des questions de recherche différents mais suivent des procédures similaires.
1. prérequis ou préalables des Analyses
- Valider tous les épreuves d’expression de gène pour être utilisé comme précédemment décrit6.
Remarque : Cette étape est faite bien avant la date de l’expérience. Valider tous les essais, commerciale et personnalisé, est nécessaire pour assurer l’amplification linéaire et efficace de l’ARN pertinente jusqu’au niveau de single-cell. Nombreux essais commercialement disponibles et personnalisés ne satisfont pas à ces spécifications. Traitement et ajustement de courbe automatisé pour la qualification simultanée des tests d’expression des gènes jusqu'à 96 sont fournis dans Les fichiers de codage supplémentaires 1 – 5, mais les essais individuels peuvent être qualifiés à l’aide de R2 et la pente de l’ajustement linéaire. Parcelles de qualification test représentatif de réussite ou un échec sont indiquées à la Figure 2. -
Développer un panel de cytométrie en flux d’anticorps pour colorer les marqueurs de surface de cellules d’intérêt.
- Titrer les anticorps par coloration un exemple pertinent, par exemple, rhésus mononucléaires cellules du sang périphérique (PBMC), avec chacun des anticorps. Commencez par 20 µL d’anticorps par test dans 100 µL de réaction de coloration et de créer huit double série de dilutions. Identifier la concentration optimale qui présente le maximum d’intensité de la coloration tout en conservant une séparation claire entre les populations négatives et positives.
- Évaluer la coloration combinées sur les échantillons de cellules supplémentaires en utilisant le mélange de tous les anticorps à la concentration optimale déterminée à l’étape 1.2.1. Veiller à ce que la coloration est similaire à celle observée pour les taches de différents anticorps. Si la coloration est inférieur à ce qui est observé quand n’importe quel anticorps a été utilisé dans l’isolement, envisager autre fluorochrome conjugués pour remplacer ces anticorps.
2. Gene Expression MODE OPERATOIRE Préparation
- 96 de combiner l’expression des gènes dosages dans une RNase/DNase-free tube 1 – 15 mL (taille peut varier selon le nombre de plateaux de tri). Le panneau des essais utilisés dans cette étude est spécifié dans le tableau 1 et tableau 2. Le produit est dénommé le Mix de « test ». Ajouter chaque série de tests à une concentration finale de 180 nM, des amorces et inverses. Ajouter le tampon de Suspension d’ADN pour réaliser la dilution appropriée de la combinaison de test.
Remarque : Pour des raisons pratiques, les stocks personnalisés (généré par l’utilisateur) dosage peuvent être réalisés à 18 µM soit compatible avec la concentration de disponible dans le commerce « x 20 » gene expression qPCR dosages (Table des matières). 18 mélanges µM d’amorce de marche avant et arrière personnalisé pour chaque gène sont fabriqués à partir de solutions mères dans le tampon de Suspension d’ADN. Tests disponibles dans le commerce (Table des matières) incluent également les sondes, mais les sondes ne sont pas obligatoires pour RT ou cDNA de pré-amplification et peut donc être omis pour les analyses personnalisées. Il est recommandé d’inclure un ou plusieurs gènes domestiques pour les utilisent dans le contrôle de la qualité pour évaluer l’efficacité du tri, récupération de la cellule et synthèse de cDNA. Utilisation des amorces aléatoires pour générer des ADNc n’a pas été déterminée, mais devrait être moins efficaces que les amorces gène-spécifiques. - Préparer le 2 x plaque d’essai à utiliser dans l’étape 7.1 (qPCR multiplex). Pour chaque tableau de 96 x 96 puce prévu, Pipetter 6 µL de chaque série de tests dans chacun désigné bien d’une plaque PCR 96 puits. Par exemple, pour 5 jetons, 30 µL de chaque essai occupera un puits unique dans la plaque à 96 puits. Si vous utilisez 96 essais, chaque puits de la plaque à 96 puits contiendra un dosage. Sceller la plaque avec sceau adhésif.
Remarque : Dans l’idéal, étapes 2.1 et 2.2 sont effectuées simultanément, afin d’éviter de multiples cycles de gel-dégel pour essais d’expression de gène. Tous les gènes au sein de la plaque de test doivent avoir été aussi présents dans le mélange d’essai (étape 2.1). Tant le mélange d’essai et la plaque de test peuvent être stockés à-20 ° C ou 4 ° C pour une utilisation à court ou à long terme, respectivement.
3. surface tache cellules viables
NOTE : Intracellulaire coloration perméabilisation et fixation ne sont pas compatibles avec cette méthode car ils compromettent RNA.
- Préparer les échantillons de compensation en ajoutant chaque anticorps énumérés dans la Table des matières à 40 µL de perles de compensation à 2,5 concentration plus élevée que celle utilisée pour la coloration de la cellule. Incuber pendant 20 min à 25 ° C, abri de la lumière. Ajouter 3 mL de PBS aux perles et centrifuger à 500 x g pendant 3 min à 25 ° C. Aspirer le PBS et remettre en suspension les perles à ~ 300 µL de PBS.
- Préparer le trieur de cellules de cytométrie en flux pour le traitement des échantillons : acquérir des tubes d’indemnisation, créer la matrice de compensation et appliquer la matrice aux fichiers d’acquisition pour les spécimens expérimentales.
- Préparer le mélange maître d’immunofluorescence cocktail en combinant le volume approprié de chaque anticorps comme spécifié dans la Table des matières, dans un tube de verre ambré de 1,5 mL pour tous les échantillons à colorer. Vortex et centrifuger le cocktail à 21 000 x g pendant 2 min à 25 ° C à l’anticorps de granule agrégats.
Remarque : Les anticorps utilisés ici sont répertoriés dans la Table des matières. - Décongeler les cellules cryoconservés dans un bain-marie à 37 ° C pendant 2 min. Ajouter 0,5 à 2 mL de la suspension cellulaire à 12 mL de PBS dans un tube de 15 mL, centrifuger à 500 x g pendant 3 min à 25 ° C et aspirer le PBS. Resuspendre dans 3 mL de PBS et transférez dans un tube de polystyrène de 5 mL. Centrifuger comme ci-dessus et aspirer le PBS, laissant environ 20 µL résiduelle PBS.
Remarque : La température de coloration peut-être être adaptée à des températures plus chaudes ou plus froides au besoin pour des applications spécifiques en modifiant le titrage d’anticorps coloration des conditions en conséquence (Voir l’étape 1.2). - Remettre en suspension jusqu'à 2 x 107 lavé les cellules dans 80 µL d’anticorps cocktail et incuber pendant 20 min à 25 ° C, abri de la lumière. Pour les échantillons dépassant 2 x 107 cellules, augmenter le volume de réaction coloration en conséquence pour maintenir < 2 x 107 cellules/100 µL.
- Laver les cellules en ajoutant 3 mL de PBS, centrifugation à 500 g pendant 3 min et aspirer le surnageant.
- Bien remettre en suspension les cellules de 300 à 500 µL de PBS et filtre en pipettant également, en établissant un plafond tamis cellulaire de 35 µm en nylon. Garder les cellules sur la glace et l’abri de la lumière jusqu'à ce que le tri.
4. préparer les plaques Collection cellulaires, exécuter un tri FACS et Generate cDNA
- Combiner les composants du mélange réactionnel RT-préampli (tableau 3) en pipettant également, dans un seul tube stérile RNAse/DNAse-libre.
Remarque : Cette étape peut être réalisée avant ou pendant la coloration à l’étape 3. L’enzyme RT peut-être être omise ici pour déterminer la contribution de la matrice d’ADN au signal de qPCR. - Utiliser une pipette multicanaux pour distribuer 10 µL du mélange réactionnel RT-préampli dans le nombre de plaques de 96 puits PCR tri collection désiré. Sceller les plaques avec du film adhésif et placer les plaques sur des blocs d’aluminium préalablement réfrigérées de 96 puits.
- Mettre en place la cellule Tri schéma de blocage sur le cytomètre en flux par l’acquisition de données d’environ 20 000 cellules de l’échantillon coloré. Veiller à ce que la matrice de rémunération s’applique aux données collectées. Dessiner les portes et définir l’arborescence gating qui identifie la population dans la cellule de l’intérêt à isoler pour l’analyse de l’expression génique.
Remarque : L’arborescence gating utilisé pour le prélèvement de cellules de vRNA+ SIV potentiels est illustré Figure 3. - Entrez les paramètres de l’instrument approprié pour spécifier le nombre et le sous-ensemble de cellules à trier dans chaque puits. Des instructions détaillées supplémentaires pour le tri soit une série de dilution limite cellule ou des cellules individuelles sont fournis aux étapes 5 et 6, respectivement.
- FACS trier les cellules en prêts plaques collectrices de PCR 96 puits. Enlever le joint adhésif avant tri et remplacer par un joint frais après le tri.
NOTE : Gardez les plaques sur les blocs d’aluminium pré réfrigéré en tout temps, même pendant le tri. - Immédiatement après le tri, le vortex et la centrifugeuse sur la collection plaque à 2 000 x g pendant 1 min à 4 ° C.
- Thermocycle la plaque dans une machine PCR avec un couvercle préchauffé en utilisant les conditions suivantes : 50 ° C pendant 15 min (RT), 95 ° C pendant 2 min, suivie de 18 cycles de 95 ° C pour 15 s et 60 ° C pendant 4 min (pré amplification).
- Diluer le cDNA 1:5 en transférant 5 μL d’ADNc dans 20 µL de tampon de suspension de l’ADN dans une nouvelle plaque PCR 96 puits. ADNc dilué peut-être être conservé à 4 ° C ou -20 ° C indéfiniment à ce stade. L’ADNc est maintenant prêt à être utilisé comme modèle pour qPCR (étapes 5.2, 7.4).
Remarque : Cette dilution s’assure que les amorces présents dans la réaction de RT-préampli ne contribuent pas à qPCR en aval.
5. variation A: FACS trier des cellules dans une série de Dilution limite pour déterminer la fréquence des vRNA+ cellules ou effectuer le contrôle de la qualité expérimentale
Remarque : Avant d’effectuer une sorte de cellule unique, il peut être d’utilité pour déterminer la fréquence des cellules d’intérêt, en triant les cellules dans des dilutions à répliquer. Cette étape prévoit aussi précieux contrôle qualité Tri efficacité, lyse cellulaire, la récupération de RNA et synthèse de cDNA, comme indiqué au point 5.3. Permet de déterminer au préalable fréquence cellulaire vRNA+ pour une estimation plus précise du nombre de cellules individuelles doivent être triés pour atteindre suffisamment taille de l’échantillon pour analyse l’expression génique cellulaire vRNA convenablement alimenté+ .
- FACS trier les cellules dans les plaques de 96 puits préparés tout comme aux étapes 4.1, 4.2 et collectez 1 – 1 000 cellules par puits dans des répétitions multiples.
Remarque : Le nombre de puits répétées à chaque dilution cellulaire est généralement inversement associé à la concentration en cellules par puits. Lorsque la fréquence de la cellule infectée est nettement inférieur à 1 %, les dilutions de cellule devraient se concentrer sur 100 – 1 000 cellules par puits. Une carte de plaque de tri exemple est présentée à la Figure 1, en haut à gauche. Supérieure à 1 000 cellules / puits doit être évitée en raison de l’augmentation qui en résulte dans le volume réactionnel et d’ingérence dans la synthèse de cDNA en aval et de la quantification. - Mélanger les réactifs de qPCR dans une solution de mélange principal au tableau 4. Pour un volume de réaction 25 µL, 22,5 µL du mélange maître est combiné avec 2,5 µL de matrice d’ADNc dilués à l’étape 3.9. Effectuer le qPCR en utilisant les conditions de vélo standards (p. ex., 94 ° C pendant 5 min, suivi de 40 cycles de 94 ° C pour 15 s et 60 ° C pendant 1 min).
Remarque : Singleplex qPCR réactions à l’aide d’un instrument conventionnel qPCR en temps réel sont recommandées comme une analyse économique préliminaire d’un ou plusieurs essais pour démontrer efficace cellule Tri, récupération de RNA et synthèse de cDNA. Il peut également être utilisé pour calculer la fréquence des cellules vRNA+ . Réactions de qPCR multiplex à l’aide de la Biomark sont généralement plus appropriées pour des analyses à grande échelle de cellule unique. - Pour le contrôle de la qualité, tracer Et valeurs (Et = −max Ct Ct) par rapport à un nombre de cellules triées par puits sur une échelle de10 logarithmique et procéder à une analyse de régression linéaire.
NOTE : Compatibles réplicats, pente de la régression linéaire de 3,3 (± 0,3) et R2 > 0,9 témoignent d’une expérience efficace. Exemples de tri optimal et sous-optimal, expériences RT-préampli sont indiquées à la Figure 4. - Pour déterminer la fréquence des cellules vRNA+ , tracer le nombre de cellules trié par puits sur l’axe des x (log10 échelle) et la fraction de puits positif pour vRNA à chaque dilution de cellule sur l’axe y. Pour obtenir un exemple, voir la Figure 1 (inférieure gauche, distribution de Poisson). Appliquer un modèle de régression linéaire aux données afin de déterminer le nombre de cellules qui hébergent une cellule positive en moyenne, correspondant à 63,2 % des puits positif (0,632 sur l’axe des ordonnées)21. Convertir ce nombre de dilution de cellule (intersection de l’axe des abscisses) en fréquence, exprimée en pourcentage. Par exemple, une cellule de vRNA+ par 48 cellules équivaut à une fréquence de 2.1 %.
6. variation B: FACS trier des cellules pour l’analyse des cellules individuelles
- Suivez les étapes 4.1 – 4,8 et spécifier une cellule triée par bien en utilisant la nouvelle fonctionnalité de tri du cytomètre pour créer des fichiers individuels de FCS pour chaque cellule triés, mappé par poste bien.
Remarque : Si le nombre d’assiettes de collection sorte dépasse le nombre des thermocycleurs disponible, vélo peut être arrêté après l’étape d’inactivation de la transcriptase inverse (95 ° C pendant 2 min) et ARNC peut être stocké à 4 ° C jusqu'à ce qu’il existe des thermocycleurs. Dans ce cas, commencer pré-amplification au premier cycle de 95 ° C pendant 15 s. - En option : Créer cDNA « pools » consistant en ADNc provenant de lots définis par l’utilisateur des cellules individuelles pour dépister les cellules rares d’intérêt using cDNA non dilué résiduelle. Transférer 2 µL de non dilué unicellulaires pré amplifié ADNc par pipette multicanaux dans une nouvelle plaque de 96 puits. Répéter en pipettant : 2 l du cDNA de toutes les autres cellules individuelles d’un groupe désigné dans le même bien. Pools d’ADNc pour un ou des gènes d’intérêt (p. ex., vRNA) afin de déterminer celles qui contiennent des cellules positives à l’aide de qPCR classique de l’écran. Depuis la mise en commun nécessite seulement une petite partie aliquote du cDNA de chaque cellule, le µL de ~ 8 restants de l’ADNc est toujours disponible pour analyses unicellulaires.
NOTE : Les stratégies de mise en commun sont recommandées avant d’effectuer des analyses d’expression génique de la cellule unique dans le but de réduire le nombre de cellules individuelles, interrogé par qPCR multiplex gourmandes en ressources. Il convient pour les situations dans lesquelles les cellules d’intérêt (p. ex., SIV mRNA +) peuvent être identifiés par un dosage de qPCR préliminaire single-plex. Les stratégies de haut débit simples pour créer des pools de cDNA comprennent combinant 2 µL du cDNA non dilué de lignes (c'est-à-dire 12 toutes les cellules dans la ligne A) ou des colonnes (c.-à-d. 8 toutes les cellules dans la colonne 1), de la plaque sort une collection dans un puits unique dans une nouvelle plaque. Pour déterminer la meilleure stratégie de mutualisation, examiner la fréquence prévue des cellules d’intérêt de l’étape 5.4. Par exemple, si 10 % des cellules sont susceptibles d’être positif, piscines composés de six échantillons de cellule unique cDNA sera souvent négatifs et peuvent donc d’exclure les cellules représentées dans ce pool du aval analyses unicellulaires.
7. multiplex qPCR sur la plate-forme Biomark
Remarque : Cette section peut suivre le version A ou B ci-dessus. Dans l’étude décrite dans les présentes, il a été appliqué exclusivement à l’analyse de cellules individuelles.
- Préparer la plaque de test de qPCR en pipettant : 4 l de chaque série de tests de 2 x plaque de test (préparé à l’étape 2.2) dans une nouvelle plaque PCR 96 puits contenant 4 µL de réactif dans chaque puits de chargement de dosage. Maintenir la plaque d’essai à 4 ° C.
Remarque : La plaque de test est stable à 4 ° C pendant une semaine et à-20 ° C pendant un mois. Ainsi, il peut être utile de préparer suffisamment de matière pour plusieurs jetons et stocker de manière appropriée. - Distribuer les fluides de ligne de commande de seringues d’amorçage dans les soupapes de deux admission de la puce. Retirez la pellicule protectrice en plastique sous la plaque. Placez le jeton sur un contrôleur de l’IFC avec le côté échancré postées sur la A1. Dans le menu principal, sélectionnez le script de « Prime ». Exécutez le script.
- Préparer le mélange de réaction en temps réel en mélangeant 50 µL de réactif avec 500 µL de PCR Master Mix (Table des matières) pour chaque puce microfluidique de chargement. Pipetter 4,4 µL dans chaque puits d’une nouvelle plaque PCR 96 puits, désormais désignée comme la « plaque d’échantillon ».
- Pipetter 3,6 µL du cDNA dilué 1:5 de l’étape 4,8 dans la plaque d’échantillon contenant le mélange de réaction en temps réel.
Remarque : Si une sélection PCR a été réalisée afin de dépister les rares (p. ex., vRNA+) cellules pour analyse en aval comme indiqué dans l’étape 6.2, inclure seulement les cellules représentées dans les pools positifs. - Après l’achèvement de l’amorçage de la puce, charger des entrées de la puce de distribution 5 µL de la plaque de test dans la correspond bien du côté de la puce cranté (essai) et 5 µL de la plaque d’échantillon dans la correspond bien sur l’autre côté (échantillon) de la puce. Insérez la puce dans le contrôleur de la SFI et exécutez le script de « Mélange de charger ».
- Transférer la puce sur la plate-forme Biomark pour effectuer le qPCR multiplexé. Aller de l’avant avec le réglage de l’instrument et la programmation de qPCR suite d’instructions détaillées fournies par le logiciel d’analyse de PCR en temps réel et utilise le protocole de Gene Expression (GE) 96,96 V.1 Standard avec 40 cycles de PCR. Enregistrez le fichier ChipRun dans un dossier désigné.
Remarque : Plusieurs puces peuvent être exécutées par jour et Pendant plusieurs jours. - Analyser les données de qPCR.
- Ouvrez le logiciel d’analyse PCR en temps réel. Ouvrez le fichier « ChipRun.bml » de la « fichier | Menu Open ».
- Localisez « Chip Explorer » et « Résumé de Run de puce » dans le coin supérieur gauche de la fenêtre du logiciel. Identifier les trois composantes de la puce Run Résumé : vues d’analyse, échantillon d’installation et installation de détecteur.
- Cliquez sur « Détecteur Setup ». En vertu de la « Tâche », cliquez sur « Nouveau » et sélectionnez le type de conteneur « Plaque de SBS » et le format de conteneur « SBS96 ». À côté de « Cartographie », cliquez sur le... et sélectionnez « M96-dosage-SBS96.dsp ».
- Facultatif : Assigner chaque détecteur (dosage) un nombre ou un nom dans la section « nom » de chaque puits en double-cliquant sur le 1st bien. Passer à la suivante en appuyant sur « F2 ».
- Cliquez sur « Setup de l’échantillon ». Sous « Tâche » à côté de « Cartographie », cliquez sur le... et sélectionnez « M96-échantillon-SBS96.dsp ».
- Cliquez sur « Vues d’analyse ». Sous « Tâche » dans l’onglet « qPCR », sélectionnez « Baseline Correction Linear (dérivé) » et « Ct seuil Method for User (détecteurs) ». Dans l’onglet « Ct seuils », vérifiez la « Initialize avec boîte automatique ». Cliquez sur le bouton « Analyser » ci-dessus.
- Dans le quadrant supérieur droit de « Vues d’analyse », cliquez sur le deuxième onglet « Tableau des résultats ». Dans le menu déroulant, sélectionnez « Affichage de carte de chaleur ». La carte thermique avec des données s’affiche.
- Facultatif : Pour garantir la puce uniforme fluorescence ROX, sélectionnez « Image View » au lieu de « Chaleur carte View » dans le même menu. Dans le second de la fenêtre de droite au-dessus de la carte de chaleur, sélectionnez « ROX ». Dans la première de la fenêtre de droite, sélectionnez un des cycles de 1 à 40. Cliquez sur la quatrième de la fenêtre de droite pour passer à un affichage en noir et blanc de la fluorescence de ROX. Une image s’affiche qui informe des éclaboussures, des particules, ou les défauts sur la puce. Si l’uniformité ROX est grossièrement obscurcie, ré-exécuter la puce.
- Sous la carte thermique, cliquez sur « Seuil » et « Journal graphique ». Ajuster les seuils de Ct manuellement pour chaque détecteur en faisant glisser le seuil nécessaire au point d’intersection des courbes d’amplification dans la phase exponentielle sur les dosages (colonnes de la feuille de chaleur). Une fois terminé, cliquez sur « Analyser ».
- Exportez les données de qPCR comme un fichier .csv. Importer les données dans un tableur ou un logiciel d’analyse statistique (p. ex., JMP) et cartographier les résultats par échantillon et analyse des positions sur la puce. Organiser les cellules en groupes basés sur l’expression des gènes viraux, en créant une nouvelle colonne et en utilisant une formule conditionnelle. Sous « Analyze », sélectionnez « Fit Y par X » et tracer l’expression des gènes contre le groupe. Appliquer l’analyse statistique.
NOTE : Représentante unicellulaires l’expression quantitative de quatre espèces d’ARN de SIV est représentée dans les graphiques à deux variables dans la Figure 5 a. L’expression des gènes dans les cellules de SIV RNA+ hôte quantitative est montrée dans la Figure 5 b. - Extraire valeurs d’expression de protéine quantitative des données de FACS unicellulaires.
- Ouvrez le .fcs fichiers à partir de la FACS Trier (étape 6.1) correspondant à la plaque à 96 puits à l’aide de FlowJo version 9. Avec le nom du fichier en surbrillance, sélectionnez « plate-forme | Porte numéro événement | Créer tri indexée gates ». Les cellules individuelles apparaîtra affichées par rangée.
- Sélectionnez toutes les 96 cellules (pas de lignes), puis sélectionnez « espace de travail | Export | Sélectionnez des fluors tout compensés ». Sous « Type de données », sélectionnez « Fichier FCS », cliquez sur « Exporter » et sélectionnez un dossier désigné.
- Faites glisser les nouveaux fichiers .fcs pour les cellules individuelles dans un nouvel espace de travail FlowJo. Mettre en évidence toutes les cellules, cliquez sur « Ajouter des statistiques » (le bouton « Σ » dans le coin supérieur gauche) » | Veux dire | Tous les paramètres de fluor ».
- Ouvrez « Table Editor » en cliquant sur la quatrième case de gauche à droite dans le coin supérieur gauche. Sélectionnez tous les fluores de la première cellule, puis faites-les glisser dans la fenêtre de l’éditeur de table. Dans la fenêtre de l’éditeur de table, cliquez sur le bouton même au sommet « Table de créer et de vue ». Cela créera un tableau de 96 cellules et un paramètre numérique pour chaque fluor.
- Copiez la sortie dans un logiciel de base de données (p. ex., MS Excel, JMP), soit copier/coller ou en cliquant sur « Enregistrer et lancer des Application » ((quatrième de la touche gauche au-dessus de la table).
Remarque : Cette procédure est spécifique pour FlowJo version 9. FlowJo version 10 utilise une procédure différente pour importer les données indexées. Données sur le débit indexée peuvent également être copié/collé dans JMP directement à partir de fichiers .csv créé par le trieur de cellules.
- Fusionner les données de FACS unicellulaires et qPCR données par le numéro de plaque et bien positionner. Effectuer des analyses graphiques et statistiques sur le gène de la cellule unique combiné (qPCR) et données d’expression (FACS) de protéines.
Remarque : Exemples de cellule unique combiné qPCR et FACS données sont indiquées à la Figure 6 (accueillir les profils d’expression de la protéine de surface de cellules de macaques rhésus infectés par le SIV, épissé vRNA+ ), Figure 7 (expression du gèneCD4 par rapport à la surface CD4 expression de la protéine dans les cellules de macaques rhésus épissés vRNA+ ) et précédemment publié20. Pour identifier différentiellement exprimés gènes dans les cellules ou les populations d’intérêt, seule cellule analyse les méthodes décrites précédemment sont recommandée20,22,23,24, qui représentent la proportion de cellules positives pour un gène ainsi que la valeur de l’expression du gène continue.
Résultats
Le flux de travail pour l’ensemble du protocole est représenté dans la Figure 1. Il se compose de deux variations définies par le nombre de cellules triées : chaque dilution limitante ou en tant que simple de cellules, comme décrit dans le texte. Exemples d’apprêt-sonde analyse de qualification sur 2 fois des dilutions sériées de RNA sont indiquées à la Figure 2. La stratégie de blocage pour identifier les éventuelles cellules SIV+ est illustrée à la Figure 3. Un qPCR contrôle qualité suboptimales, réussite ou un échec pour le gène de ménage GAPDH FACS-tri des cellules en limitant la dilution sont indiquées à la Figure 4. Unicellulaire l’expression quantitative de quatre SIV espèces et rhésus macaque gènes d’ARN qui ont été exprimés dans les cellules infectées sont indiquées à la Figure 5. Représentant deux variables, histogramme et nuages de points représentent le profil d’expression protéine de surface du SIV RNA+ CD4+ T cellules mesurée par cytométrie de flux à la Figure 6. Le graphique en bulles trivarié (Figure 7) affiche la relation entre la protéine de surface CD3 ou CD4, CD3 ou ARNm CD4 et des gènes viraux quantitative (tat/rev) expression dans des cellules individuelles.

Figure 1 : Représentation schématique du flux de travail expérimental illustre trois grands volets : flux Tri par cytométrie en flux, transcription inverse plus basées sur la PCR pré-amplification d’ADNc (RT, préampli) et qPCR. Le tri peut être effectué soit une dilution limite (A, toile de fond vert) ou comme des cellules individuelles (B, fond orange). Immédiatement après le tri de FACS, les cellules sont lysées et RNA est reverse transcrit en ADNc et pré amplifié (RT, PCR pré-amp) pour préparer le modèle de qPCR. Sortes de dilution limite déterminent la fréquence des cellules positives pour l’ARN viral en utilisant les statistiques de distribution de Poisson ainsi que l’efficacité expérimentale et récupération de l’échantillon. La tête de la flèche verte indique le nombre de cellules triées par bien contenant une seule cellule positive pour un gène viral (correspondant à une probabilité de 63,2 % de ces puits Bingen positif), qui se transforme en une fréquence de cellules. Les estimations de fréquence peuvent servir à informer le nombre de cellules individuelles recueillies dans un tri ultérieur (B). Indexée unicellulaires FACS tri des dépôts un cellulaire / puits et génère des fichiers de données pour chaque cellule annotée par poste bien au sein de la plaque à 96 puits. QPCR unicellulaire est réalisée en multiplex pour 96 gènes simultanément. Combinant la protéine de surface (FACS) et l’expression de l’ARNm permet pour le profilage des cellules individuelles (colonne de droite). Un qPCR facultatif pour un gène viral peut être effectuée entre la pré amplification PCR et multiplexé qPCR (B, middle) monocellules écran ou piscines de cDNA monocellulaires à bas-sélectionner des cellules RNA+ virales ou piscines pour analyse qPCR multiplex. La carte thermique illustre l’expression des gènes (valeur de l’EC) pour 96 essais (colonnes) et 96 cellules individuelles (lignes). S’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.

Figure 2 : Représentant qPCR données des amorces qualification expériences sont indiquées pour réussie (gauche et milieu) et n’a pas de tests (à droite) dans le commerce ( CD6, SIV tat/rev, et TLR3, respectivement). Pour CD6 et TLR3, ARN a été extrait de macaque de rhésus FACS-tri 106 PBMC CD4+ T-cellules à l’aide d’une trousse commerciale. Huit réplique d’une série de douze-point ARN double dilution (0,023-48 ng d’ARN, correspondant à 1,2 – 2 400 équivalents de cellule en supposant que 20 pg RNA par CD4+ T-cellule) ont été soumis à la RT-préampli et qPCR. Pour tat/revSIV, l’ARN a été extrait du macaque rhésus PBMC infectés in vitro avec SIVmac239. Les dilutions de RNA ont été préparées équivalents de RNA couvrant de 6 – 12 000 cellules. Et valeurs (40-Ct), qui augmentent avec l’expression des gènes, sont tracés par rapport à un nombre estimé de cellules. Des séries de dilutions présentant R2 > 0,97 et pente de ±0, 3 3,32 indiquent qualification amorce réussie. S’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.
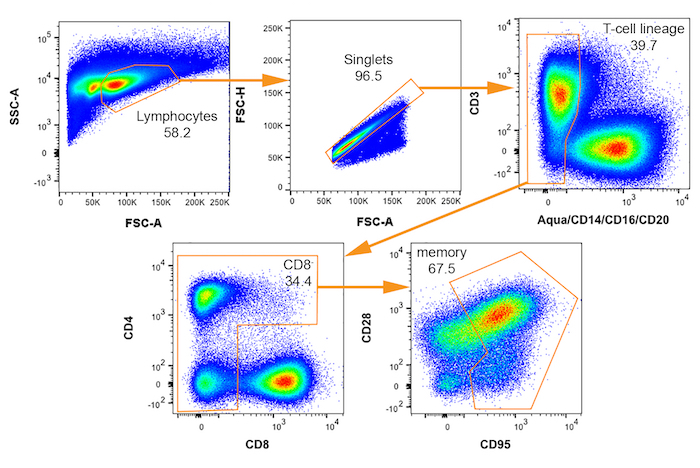
Figure 3 : Deux variables FACS parcelles dont le régime d’isolement de cellules de macaques rhésus potentiellement infectés par le SIV. Portes séquentielles pour sélection de mémoire (CD95 +) CD4+ T cellules sont montrés du coin supérieur gauche pour abaisser le droit avec chaque nom de la population a indiqué. Pourcentage de parcelle de parent qui relève de chaque porte est indiquée. S’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.

Figure 4 : Représentante validation expérimentale de qPCR exécutées sur des FACS-tri des piles pour limiter la dilution à l’aide GAPDH gène de ménage pour trois expériences indépendantes : réussi (à gauche), sous-optimal (au milieu) et a échoué (à droite). Réplique de 10 à 100 cellules / puits devrait s’étendent aux Ets pas plus de 2 et 300 – 1 000 puits de cellule dans 1 Et. La pente de la régression linéaire doit être 3,32 ± 0,3, R2 > 0,95. L’incapacité d’atteindre ces spécifications indique des difficultés techniques dans les étapes 1, 2, 3 ou une combinaison des deux. S’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.

Figure 5 : Monocellulaires quantitative virale et hôte expression génique dans macaque rhésus FACS-tri CD4+ Cellules T des ganglions lymphatiques mésentériques 10 jours post-SIVmac251 infection. (A) expression de gènes viraux bivariée des parcelles de multiplier-épissé (tat/rev) et séparément épissé (env) SIV ARNm par single cells (à gauche). Tat/rev+env– (cellules lumière verte le long de l’axe des abscisses) expriment moins de copies de tat/rev RNA que les cellules d' env+ (vert foncé), conformes à des premiers stade de l’infection et avant stabilisation induite par la protéine Rev et exportations nucléaires de vRNAs partiellement épissage telles que env. Les cellules qui n’expriment pas d’épissage des ARN viral sont représentés en gris (pas l’ARN viral), brown (gag+ et LTR+) ou tan ( gag+ ou LTR+). Unspliced (gag+) et la totale expression de l’ARNm SIV (LTR+) est indiquée pour les mêmes cellules (à droite). Grande abondance d’unspliced gag RNA dans tat/ cellulesenv+ rev+est compatible avec infection productive scène tardive au cours de laquelle l’ARN génomique abondante s’exprime pour l’emballage en herbe virions. (B) violon tracés des gènes de macaque rhésus différentiellement exprimés au moins un sous-ensemble de cellules infectées par le SIV par rapport aux cellules non infectées (gris). L’analyse statistique a été réalisée comme décrit précédemment20,22,23,24. Astérisque indique les taux de fausse découverte < 10 % en probabilité combinée ratio test des comparaisons par rapport aux cellules non infectées. Ligne relie les valeurs moyennes dans l’ensemble des groupes de cellules pour chaque gène. Ce chiffre a été modifié par Bolton et al. 20 S’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.

Figure 6 : Profils d’expression de protéine de surface hôte représentatif des FACS-trier des cellules de rhésus macaque ganglions lymphatiques mésentériques 10 jours post-SIVmac251 infection. Nuage de points (A) l’affichage de l’expression de la protéine de surface CD3, CD4 et ICOS en non infectées (gris), gag+tat/rev– (brun) et les cellules detat/rev+ gag+(vert). Intensité de la fluorescence est tracée pour chaque cellule (dot). Diagrammes en boîte aberrantes dépeignent l’intervalle interquartile (IQR) et la médiane (boîte), les points plus éloignés au sein de 1,5 x IQR des boîte (moustaches) et le potentiel des valeurs aberrantes (points déconnectées). Les barres horizontales en haut montrent des différences significatives (p < 0,05, test de rang non paramétriques Wilcoxon). (B) affichage d’histogramme et bivariés de surface CD3 et CD4 expression des protéines des cellules montré en (A). Plot point (à gauche) indique le pourcentage de la population de chaque cellule dans un quadrant. Les histogrammes CD3 et CD4 (milieu, droit) dépeignent downregulation de protéine de surface entre les cellules detat/rev+ gag+par rapport aux non-infectées et gag+tat/rev– . (C), douze représentant tat/rev+ des cellules individuelles de (A – B) figurent dans l’ensemble de trois graphiques à deux variables pour l’expression en surface de CD3/CD4 (à gauche), CD69/CD38 (au milieu) et ICOS/HLA-DR (à droite). S’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.
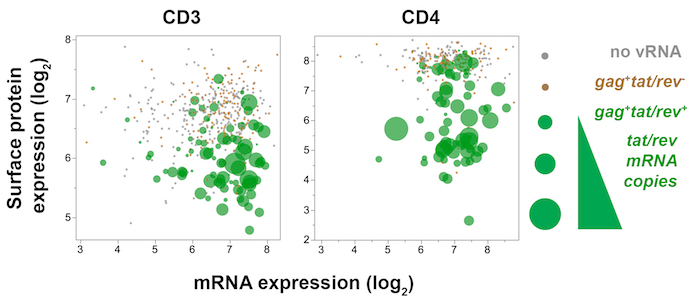
Figure 7 : Intrigue trivarié affichant des gènes viraux unicellulaires (tat/rev), gène de l’hôte (CD3 ou CD4) et l’expression d’une protéine de surface (CD3 ou CD4) hôte en mémoire infectés par le SIV CD4+ T les cellules des ganglions lymphatiques mésentériques 10 jours infection post-SIVmac251. Expression de la protéine CD4 (fluorescence) est tracée contre ARNm CD4 (qPCR), tandis que la quantité de tat/rev exprimées par chaque cellule est reflété par la taille de la dot. En tat/ cellulesrev+ (vert), a diminué de surface CD4 et CD3 expression des protéines avec des transcriptions CD4 et CD3 soutenues, respectivement, indiquent que l’expression de la protéine de surface est modulée en aval de l’expression génique. S’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.
Tableau 1 : Des amorces et sondes utilisées pour la détection des acides nucléiques SIV. Lorsque deux séquences sont indiqués pour une sonde ou un apprêt, quantités équimolaires de deux séquences ont été utilisées. S’il vous plaît cliquez ici pour télécharger ce fichier.
Tableau 2 : un panel de 96-génétique pour le dosage des amplicons Biomark instrument. Quatre essais SIV sont indiqués avec fond bleu. S’il vous plaît cliquez ici pour télécharger ce fichier.
Tableau 3 : Mélange de réaction utilisée pour la transcription inverse et préamplification. S’il vous plaît cliquez ici pour télécharger ce fichier.
Tableau 4 : mélange réactionnel de qPCR utilisé pour PCR en temps réel effectuée sur un instrument Quant Studio 6. S’il vous plaît cliquez ici pour télécharger ce fichier.
Fichier de codage supplémentaires 1. Instructions pour qualification gene expression assays. S’il vous plaît cliquez ici pour télécharger ce fichier.
Fichier de codage supplémentaire 2 : modèle de l’exemple de mappage de JMP. S’il vous plaît cliquez ici pour télécharger ce fichier.
Fichier de codage supplémentaire 3 : modèle de carte de sonde de JMP. S’il vous plaît cliquez ici pour télécharger ce fichier.
Fichier de codage supplémentaire 4 : script d’analyse d’apprêt pour JMP. S’il vous plaît cliquez ici pour télécharger ce fichier.
Fichier de codage supplémentaires 5. Script d’analyse par morceaux pour JMP. S’il vous plaît cliquez ici pour télécharger ce fichier.
Discussion
Le protocole décrit ici, appelé tSCEPTRE, intègre le dosage de la protéine de surface monocellulaire par cytométrie multiparamétrique avec l’expression de l’ARNm unicellulaires quantitative par RT-qPCR hautement multiplexé. L’union de ces deux technologies permet à haute teneur instantanés du combiné transcriptionnelles et profil protéique des cellules individuelles dans un format haut débit. Nous utilisons la méthode pour identifier les cellules jusqu’ici insaisissables infectés par le SIV en vivoet décrire les gènes différentiellement exprimés des hôtes et des protéines. Le protocole peut être adapté pour l’étude d’une population de cellules d’intérêt distingue par l’expression des protéines de surface, ARNm ou une combinaison des deux. Les méthodes décrites dépendent précise unicellulaires tri et enregistrement par cytométrie de flux de données jumelé avec des réactifs disponibles dans le commerce de qPCR et multiplexés instrumentation de PCR en temps réel. La sortie est sensible, quantitative évaluation de données expression protéique et gène combinées unicellulaires.
Autres approches reliant les protéines et l’expression génique dans des cellules individuelles ont été décrit1,25,26,27. Indexée FACS Trier suivie RNAseq, appliquée avec succès à la caractérisation des cellules souches hématopoïétiques, représente un particulièrement prometteuse approche28. Cependant, tandis que RNAseq a plusieurs avantages par rapport aux analyses d’expression de gène ciblé, nommément l’analyse impartiale transcriptome complet, il est soumis à un niveau plus élevé de comparaisons multiples statistique coût et peut être moins sensible pour la quantification de faibles copies relevés de notes. En outre, il n’est pas actuellement économiquement possible d’effectuer des RNAseq sur des milliers de cellules à la recherche de cellules infectées rares présents aux fréquences < 1 % (p. ex., VIH, SIV). Autres technologies émergentes de la cellule unique visent à générer une lecture unique, c'est-à-dire, de FACS ou PCR pour la détection des protéines et des acides nucléiques dans la même cellule. Il s’agit de la détection des protéines par des anticorps fluorescents et des ARNm de sondes fluorescentes suivie de FACS analysis (ARNm-flux-poisson)14,15,29,30. Alternativement, les protéines peuvent être détectées par paires de conjugués d’anticorps-oligonucléotides qui sont détectés par qPCR en parallèle avec inversement transcrit mRNA31,32,33,34 . Ces approches « hybride » fournissent simultanée d’acide nucléique et de mesures de protéines monocellulaires résolution similaire à tSCEPTRE, mais ils sont limités où ils sont soit non quantitatif (ARNm-flux-poisson) ou nécessitent personnalisés anticorps ou ARNm les sondes. Notre approche tSCEPTRE est quantitative pour les mesures de l’ARNm et la protéine, et tous les réactifs sont disponibles dans le commerce, à l’exception possible des dosages d’agents pathogènes spécifiques.
Une attention particulière doit être appliquée à plusieurs étapes du protocole avant l’analyse des échantillons expérimentaux. Tout d’abord, haute-paramètre cytométrie nécessite optimisation minutieuse d’un panel d’anticorps fluorescents pour assurer sensibles de détection et résolution35. Chaque anticorps devrait être réglée individuellement pour déterminer la concentration qui permet d’obtenir une séparation optimale et des taches de fond minimal, suivie de l’évaluation de coloration lorsque tous les anticorps conjugués sont utilisés en combinaison. Deuxième détermination expérimentale du test de l’expression génique qPCR approprié pour mesurer chaque gène d’intérêt est essentielle. Plusieurs essais disponibles dans le commerce sont généralement disponibles pour chaque gène, mais d’après notre expérience, beaucoup ne sont pas quantitatives à la cellule unique niveau6. Ainsi, il est important de se qualifier tout projets essais sur dilués en série RNA avant toute utilisation de l’expression génétique quantitative. Optimisation de ces réactifs prend du temps, mais la valeur des panneaux fiables de scientifiques et de tests d’expression de gène qui permet la détection sensible de toutes les molécules d’intérêt justifie l’investissement de temps. En outre, la priorité devrait être accordée aux déterminations commercialement disponibles contenant les sondes qui s’étendent sur les jonctions exon-exon (désignées avec le suffixe « m1 ») afin d’améliorer la spécificité des ARNm, bien que remplaçant tests capables de détecter l’ADN génomique (suffixe « s1 » ou « g1 » ) sont considérés comme peu susceptibles d’influencer les résultats d’expression de gène en raison des copies chromosomiques équivalents dans chaque cellule. Préservation de l’ARN en amont de la RT est critique et est réalisée en gardant échantillons réfrigérés, tel qu’indiqué dans le protocole. De même, la durée des FACS trier et l’intervalle entre tri et RT-préamplificateur doit être maintenue à un minimum. Pour les limitant dilution et monocellulaires tri, cDNA peut être tout d’abord analysé par qPCR conventionnel pour évaluer la récupération de RNA de fortement exprimés gènes de ménage. Si l’expression observée n’est pas uniforme entre les répétitions comme le nombre de cellules, ou la pente de l’ajustement linéaire pour limiter la validation échoue dilutions, dépannage doit être effectuée à des procédures de tri et en aval de cellules.
Les limites potentielles de l’approche décrite ici incluent (1) la détection de l’ADN en plus de l’ARN, notamment pour les tests non spécifiques d’ARNm épissure jonctions et (2) restreint nombre de gènes mesurées et les protéines de surface. Toutefois, détection de l’ADN est peu probable à contribuer considérablement à l’analyse de l’expression génique différentielle hôte pour la raison indiquée ci-dessus. En outre, nous avons mesuré directement la mesure dans laquelle l’ADN contribue au signal qPCR dans le présent protocole par deux approches : une) à l’exclusion de la transcriptase inverse et b) modifiant le protocole de lyse cellulaire pour améliorer la lyse de la membrane nucléaire. En l’absence de la transcriptase inverse, les deux épissé et unspliced de gènes viraux ont été encore détectée, mais significativement plus faible fréquence qu’en présence de la transcriptase inverse (~ 6 fois et 2 fois les réductions, respectivement)20. Par conséquent, les essais d’expression de gènes viraux peuvent surestimer la quantité d’ARN viral dans une cellule en raison de la présence de l’ADN viral à la cellule. Nous attribuons cette constatation à cytoplasmiques produits RT générée lors de l’infection de cellule hôte, se manifester fois épissés et unspliced SIV/HIV ARN provenant de virions entrant36. Ainsi, il peut être opportun de consacrer une partie des expériences aux conditions manque la transcriptase inverse pour quantifier l’ADN dérivé de modèle pour certaines applications. Il convient également de noter qu’unspliced SIV/charge virale provenant de virus entrants ne peuvent être distinguée de l’ARN viral de novo synthétisé. Deuxièmement, nous avons incorporé une étape nucléaire lyse dans le protocole RT-préampli et a observé une augmentation exponentielle des copies d’ADN SIV (Alu-LTR) intégrées (Figure S3 de Bolton et al.) 20. l’ADN génomique est donc peu probable à contribuer considérablement à l’acide nucléique modèle extrait de la méthode de lyse cellulaire utilisée ici. À noter l’ajout d’une étape de lyse nucléaire peut être utiles unicellulaire à l’avenir études cherchant à enquêter sur le SIV ou autres cellules d’ADN positif de virus, y compris les cellules infectées de façon latente.
Le nombre de protéines de surface analysée est dicté par la capacité du trieur de cellules de cytométrie en flux. Les instruments disponibles dans le commerce actuels n’excèdent pas 30 paramètres. Futures études employant plus avancés de la cytométrie élargira également la protéine profilage des possibilités de cette approche. Le nombre de relevés de notes peut être étendu au-delà de 96, réactifs de justificatifs fournis (p. ex., des amorces de concentration plus élevée), équipements et instruments. En bout de ligne, nouvelles technologies de cellule unique qui combine l’analyse du protéome (spectrométrie de masse), transcriptome (RNAseq) et le génome (DNAseq) remplaceront les approches ciblées pour découverte recherche31,37, 38 , 39. Cependant, qPCR ciblée restera probablement un outil précieux pour la validation de ces approches « omiques » comme un étalon-or pour les analyses d’expression quantitative.
Analyse combinée de protéine et de transcription de tSCEPTRE est un outil puissant pour enquêter sur les rares ou difficiles à identifier les cellules comme ceux contenant des agents pathogènes, contenant des oncogènes ou autrement présentant un phénotype aberrant. Nouveaux marqueurs pour les cellules infectées par le VIH/SIV transcriptionnellement actifs peuvent être identifiés dans cette voie, ainsi que la découverte de nouveaux mécanismes impliqués dans la pathogenèse de ces infections virales. Identification des cellules infectées de façon latente nécessitera la poursuite du développement d’un protocole pour évaluer l’ADN viral intégré dans le génome hôte. À noter, la fréquence des cellules HIV/SIV infecté est considérablement plus faible dans une infection chronique virémique ou traitée, qui présentera un défi pratique en étudiant les cellules infectées provenant de ces paramètres. Notre approche jette les bases d’évaluation un mécanisme auparavant insoluble : celle de post-transcriptionnelle niveau unicellulaire, et a large applicabilité aux interactions hôte-pathogène, mais aussi les processus cellulaires plus générales.
Déclarations de divulgation
Ce travail a été soutenu par un accord de coopération (W81XWH-07-2-0067) entre le Henry M. Jackson Foundation pour la promotion de la médecine militaire, Inc et le département américain de la défense (DOD). Les opinions exprimées sont celles des auteurs et ne doivent pas être interprétées pour représenter les positions de l’armée américaine ou le ministère de la défense. Recherche a été effectuée en vertu d’un protocole approuvé l’utilisation des animaux dans une installation AAALACi accrédité dans le respect de l’Animal Welfare Act et autres lois fédérales et les règlements relatifs aux animaux et expériences impliquant des animaux et adhère aux principes indiqué dans le Guide pour l’édition 2011 de soins et utilisation des animaux de laboratoire, Publication du CNRC.
Remerciements
Les auteurs aimeraient remercier le NIAID VRC Flow Cytometry Core et les installations du MHRP Flow Cytometry Core pour l’entretien et le fonctionnement des instruments de FACS et des appareils à tri ; Maria Montero, Vishakha Sharma, Kaimei Song for expertise assistance technique ; Michael Piatak, Jr. (décédé) d’assistance avec un modèle SIV qPCR dosage ; et Brandon Keele et Matthew Scarlotta pour SIV isoler les séquences. Les opinions exprimées sont celles des auteurs et ne doivent pas être interprétées pour représenter les positions de l’armée américaine ou le ministère de la défense. Recherche a été effectuée en vertu d’un protocole approuvé l’utilisation des animaux dans une installation AAALAC accrédité dans le respect de l’Animal Welfare Act et autres lois fédérales et les règlements relatifs aux animaux et expériences impliquant des animaux et adhère aux principes indiqué dans le Guide pour l’édition 2011 de soins et utilisation des animaux de laboratoire, Publication du CNRC.
matériels
| Name | Company | Catalog Number | Comments |
| RNA extraction and PCR reagents and consumables | |||
| Genemate 96-Well Semi-Skirted PCR Plate | BioExpress/VWR | T-3060-1 | |
| Adhesive PCR Plate Seals | ThermoFisher | AB0558 | |
| Armadillo 384-well PCR Plate | ThermoFisher | AB2384 | |
| MicroAmp Optical Adhesive Film | Applied Biosystems/ThermoFisher | 4311971 | |
| DEPC Water | Quality Biological | 351-068-101 | |
| Glass Distilled Water | Teknova | W3345 | |
| Superscript III Platinum One-Step qRT-PCR Kit | Invitrogen/ThermoFisher | 11732088 | |
| SUPERase-In Rnase Inhibitor | Invitrogen/ThermoFisher | AM2696 | |
| Platinum Taq | Invitrogen/ThermoFisher | 10966034 | |
| dNTP Mix | Invitrogen/ThermoFisher | 18427088 | |
| ROX Reference Dye (if separate from kit) | Invitrogen/ThermoFisher | 12223012 | |
| DNA Suspension Buffer | Teknova | T0223 | |
| RNAqueous kit | Invitrogen/ThermoFisher | AM1931 | |
| TaqMan gene expression assays not listed in Table 2 | |||
| CD6 | Applied Biosystems/ThermoFisher | Hs00198752_m1 | |
| TLR3 | Applied Biosystems/ThermoFisher | Hs1551078_m1 | |
| Biomark reagents | |||
| Control Line Fluid Kit | Fluidigm | 89000021 | |
| TaqMan Universal PCR Mix | Applied Biosystems/ThermoFisher | 4304437 | |
| Assay Loading Reagent | Fluidigm | 85000736 | |
| Sample Loading Reagent | Fluidigm | 85000735 | |
| Dynamic Array 96.96 (chip) | Fluidigm | BMK-M-96.96 | |
| FACS reagents | |||
| SPHERO COMPtrol Goat anti-mouse (lambda) | Spherotech Inc. | CMIgP-30-5H | |
| CompBeads Anti-Mouse Ig,k | BD Biosciences | 51-90-9001229 | |
| 5 ml Polystyrene tube with strainer cap | FALCON | 352235 | |
| Aqua Live/Dead stain | Invitrogen/ThermoFisher | L34976 | dilute 1:800 |
| Mouse Anti-Human CD3 BV650 clone SP34-2 | BD Biosciences | 563916 | dilute 1:40 |
| Mouse Anti-Human CD4 BV786 clone L200 | BD Biosciences | 563914 | dilute 1:20 |
| Mouse Anti-Human CD8 BUV496 clone RPA-T8 | BD Biosciences | 564804 | dilute 1:10 |
| Mouse Anti-Human CD28 BV711 clone CD28.2 | Biolegend | 302948 | dilute 1:20 |
| Mouse Anti-Human CD95 BUV737 clone DX2 | BD Biosciences | 564710 | dilute 1:10 |
| Mouse Anti-Human CD14 BV510 clone M5E2 | Biolegend | 301842 | dilute 1:83 |
| Mouse Anti-Human CD16 BV510 clone 3G8 | Biolegend | 302048 | dilute 1:167 |
| Mouse Anti-Human CD20 BV510 clone 2H7 | Biolegend | 302340 | dilute 1:37 |
| Anti-CD38-R PE clone OKT10 | NHP reagent recource | N/A | dilute 1:100 |
| Mouse Anti-Human CD69 BUV395 clone FN50 | BD Biosciences | 564364 | dilute 1:10 |
| Mouse Anti-Human HLA-DR APC-H7 clone G46-6 | BD Biosciences | 561358 | dilute 1:20 |
| Mouse Anti-Human ICOS Alexa Fluor 700 clone C398.4A | Biolegend | 313528 | dilute 1:80 |
| Instruments | |||
| BioPrptect Containment Enclosure | Baker | ||
| BD FACS Aria | BD Biosciences | ||
| ProtoFlex Dual 96-well PCR system | Applied Biosystems/ThermoFisher | 4484076 | |
| Quant Studio 6 qPCR instrument | Applied Biosystems/ThermoFisher | 4485694 | |
| IFC controller HX | Fluidigm | IFC-HX | |
| Biomark HD | Fluidigm | BMKHD-BMKHD |
Références
- Macaulay, I. C., Ponting, C. P., Voet, T. Single-Cell Multiomics: Multiple Measurements from Single Cells. Trends Genet. 33 (2), 155-168 (2017).
- Pasternak, A. O., Lukashov, V. V., Berkhout, B. Cell-associated HIV RNA: a dynamic biomarker of viral persistence. Retrovirology. 10, 41 (2013).
- Mailler, E., et al. The Life-Cycle of the HIV-1 Gag-RNA Complex. Viruses. 8 (9), (2016).
- Varmus, H. Retroviruses. Science. 240 (4858), 1427-1435 (1988).
- Frankel, A. D., Young, J. A. HIV-1: fifteen proteins and an RNA. Annu Rev Biochem. 67, 1-25 (1998).
- Dominguez, M. H., et al. Highly multiplexed quantitation of gene expression on single cells. J Immunol Methods. 391 (1-2), 133-145 (2013).
- Tokarev, A., Guatelli, J. Misdirection of membrane trafficking by HIV-1 Vpu and Nef: Keys to viral virulence and persistence. Cell Logist. 1 (3), 90-102 (2011).
- Malim, M. H., Bieniasz, P. D. HIV Restriction Factors and Mechanisms of Evasion. Cold Spring Harb Perspect Med. 2 (5), a006940 (2012).
- Sugden, S. M., Bego, M. G., Pham, T. N., Cohen, E. A. Remodeling of the Host Cell Plasma Membrane by HIV-1 Nef and Vpu: A Strategy to Ensure Viral Fitness and Persistence. Viruses. 8 (3), 67 (2016).
- Simon, V., Bloch, N., Landau, N. R. Intrinsic host restrictions to HIV-1 and mechanisms of viral escape. Nat Immunol. 16 (6), 546-553 (2015).
- Kirchhoff, F. Immune evasion and counteraction of restriction factors by HIV-1 and other primate lentiviruses. Cell Host Microbe. 8 (1), 55-67 (2010).
- Wigzell, H. Immunopathogenesis of HIV infection. J Acquir Immune Defic Syndr. 1 (6), 559-565 (1988).
- Reynolds, M. R., et al. Ex vivo analysis of SIV-infected cells by flow cytometry. Cytometry A. 77 (11), 1059-1066 (2010).
- Baxter, A. E., et al. Single-Cell Characterization of Viral Translation-Competent Reservoirs in HIV-Infected Individuals. Cell Host Microbe. 20 (3), 368-380 (2016).
- Grau-Exposito, J., et al. A Novel Single-Cell FISH-Flow Assay Identifies Effector Memory CD4+ T cells as a Major Niche for HIV-1 Transcription in HIV-Infected Patients. MBio. 8 (4), (2017).
- Eriksson, S., et al. Comparative analysis of measures of viral reservoirs in HIV-1 eradication studies. PLoS Pathog. 9 (2), e1003174 (2013).
- Finzi, D., et al. Identification of a reservoir for HIV-1 in patients on highly active antiretroviral therapy. Science. 278 (5341), 1295-1300 (1997).
- Chomont, N., et al. HIV reservoir size and persistence are driven by T cell survival and homeostatic proliferation. Nat Med. 15 (8), 893-900 (2009).
- DeMaster, L. K., et al. A Subset of CD4/CD8 Double-Negative T Cells Expresses HIV Proteins in Patients on Antiretroviral Therapy. J Virol. 90 (5), 2165-2179 (2015).
- Bolton, D. L., et al. Combined single-cell quantitation of host and SIV genes and proteins ex vivo reveals host-pathogen interactions in individual cells. PLoS Pathog. 13 (6), e1006445 (2017).
- Quintans, J., Lefkovits, I. Precursor cells specific to sheep red cells in nude mice. Estimation of frequency in the microculture system. Eur J Immunol. 3 (7), 392-397 (1973).
- Finak, G., et al. MAST: a flexible statistical framework for assessing transcriptional changes and characterizing heterogeneity in single-cell RNA sequencing data. Genome Biol. 16, 278 (2015).
- McDavid, A., et al. Modeling bi-modality improves characterization of cell cycle on gene expression in single cells. PLoS Comput Biol. 10 (7), e1003696 (2014).
- McDavid, A., Finak, G., Gottardo, R. The contribution of cell cycle to heterogeneity in single-cell RNA-seq data. Nat Biotechnol. 34 (6), 591-593 (2016).
- Kok, Y. L., Ciuffi, A., Metzner, K. J. Unravelling HIV-1 Latency, One Cell at a Time. Trends Microbiol. , (2017).
- Rato, S., Golumbeanu, M., Telenti, A., Ciuffi, A. Exploring viral infection using single-cell sequencing. Virus Res. 239, 55-68 (2017).
- Wagner, A., Regev, A., Yosef, N. Revealing the vectors of cellular identity with single-cell genomics. Nat Biotechnol. 34 (11), 1145-1160 (2016).
- Nestorowa, S., et al. A single-cell resolution map of mouse hematopoietic stem and progenitor cell differentiation. Blood. 128 (8), e20-e31 (2016).
- Soh, K. T., et al. Simultaneous, Single-Cell Measurement of Messenger RNA, Cell Surface Proteins, and Intracellular Proteins. Curr Protoc Cytom. 75, 41-47 (2016).
- Kochan, J., Wawro, M., Kasza, A. Simultaneous detection of mRNA and protein in single cells using immunofluorescence-combined single-molecule RNA FISH. Biotechniques. 59 (4), 209-212 (2015).
- Stahlberg, A., Thomsen, C., Ruff, D., Aman, P. Quantitative PCR analysis of DNA, RNAs, and proteins in the same single cell. Clin Chem. 58 (12), 1682-1691 (2012).
- Assarsson, E., et al. Homogenous 96-plex PEA immunoassay exhibiting high sensitivity, specificity, and excellent scalability. PLoS One. 9 (4), e95192 (2014).
- Fredriksson, S., et al. Protein detection using proximity-dependent DNA ligation assays. Nat Biotechnol. 20 (5), 473-477 (2002).
- Genshaft, A. S., et al. targeted profiling of single-cell proteomes and transcriptomes in a single reaction. Genome Biol. 17 (1), 188 (2016).
- Mahnke, Y. D., Roederer, M. Optimizing a multicolor immunophenotyping assay. Clin Lab Med. 27 (3), 469-485 (2007).
- Liang, C., Hu, J., Russell, R. S., Kameoka, M., Wainberg, M. A. Spliced human immunodeficiency virus type 1 RNA is reverse transcribed into cDNA within infected cells. AIDS Res Hum Retroviruses. 20 (2), 203-211 (2004).
- Stegle, O., Teichmann, S. A., Marioni, J. C. Computational and analytical challenges in single-cell transcriptomics. Nat Rev Genet. 16 (3), 133-145 (2015).
- Wu, M., Singh, A. K. Single-cell protein analysis. Curr Opin Biotechnol. 23 (1), 83-88 (2012).
- Haque, A., Engel, J., Teichmann, S. A., Lonnberg, T. A practical guide to single-cell RNA-sequencing for biomedical research and clinical applications. Genome Med. 9 (1), 75 (2017).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon