Method Article
Célula única quantificação de mRNA e expressão de proteínas de superfície em CD4 infectados por vírus de imunodeficiência simiana+ T células isoladas de macacos Rhesus
Neste Artigo
Resumo
Descrito é uma metodologia para quantificar a expressão dos 96 genes e 18 proteínas de superfície por células únicas ex vivo, permitindo a identificação de diferencialmente expressaram genes e proteínas em células infectadas por vírus em relação as células não infectadas. Aplicamos a abordagem de estudo CD4 infectadas SIV+ T células isoladas de macacos rhesus.
Resumo
Análise de célula única é uma ferramenta importante para dissecar heterogêneas populações de células. A identificação e isolamento de células raras podem ser difícil. Para superar este desafio, combinando uma metodologia indexados citometria de fluxo e elevado-throughput multiplexado quantitativa cadeia da polimerase (qPCR) foi desenvolvido. O objetivo foi identificar e caracterizar o vírus da imunodeficiência símia (SIV)-infectados células presentes dentro macaques do rhesus. Através da quantificação da proteína classificando fluorescência-ativado da pilha (FACS) e mRNA por qPCR, células infectadas por vírus são identificadas pela expressão do gene viral, que é combinada com medidas de gene e proteína de anfitrião para criar um perfil multidimensional . Chamamos a abordagem, avaliação de célula única Proteo-transcriptional alvo ou tSCEPTRE. Para executar o método, células viáveis estão manchadas com anticorpos fluorescentes específicas para marcadores de superfície utilizados para isolamento de FACS de um subconjunto de células e/ou análise fenotípica a jusante. Células individuais são classificadas seguido de Lise imediata, multiplex transcrição reversa (RT), pré-amplificação por PCR e qPCR alto throughput de até 96 transcrições. Medições de FACS são registradas no momento da triagem e posteriormente relacionadas com os dados de expressão do gene por posição bem para criar uma proteína combinada e perfil transcricional. Para estudar infectados com SIV diretamente células ex vivo, as células foram identificadas por qPCR detecção de múltiplas espécies de RNA virais. A combinação de transcrição viral e a quantidade de cada fornecem uma estrutura para classificar as células em estágios diferentes do ciclo de vida viral (por exemplo, produtivo versus não-produtivos). Além disso, tSCEPTRE de células SIV+ foram em relação ao não infectados células isoladas do mesmo espécime para avaliar genes diferencialmente expressos anfitrião e proteínas. A análise revelou anteriormente apreciada heterogeneidade de expressão do RNA viral entre células infectadas, assim como na vivo mediada por SIV pós-transcricional gene regulamento com resolução de célula única. O método tSCEPTRE é relevante para a análise de qualquer população de célula passível de identificação pela expressão da proteína de superfície marker(s), host ou patógeno genes ou suas combinações.
Introdução
Muitos patógenos intracelulares contam com máquinas de célula de acolhimento para replicar, muitas vezes alterando a biologia de pilha de anfitrião ou direcionamento especifico subpopulações de células hospedeiras para maximizar suas chances de propagação. Como resultado, processos biológicos células comumente são rompidos, com consequências deletérias para a saúde geral do hospedeiro. Compreender as interações entre os vírus e as células hospedeiras, na qual eles replicar irá elucidar mecanismos de doença que podem auxiliar no desenvolvimento de terapias melhoradas e estratégias para prevenir a infecção. Diretas ferramentas analíticas que permitem o estudo das interações patógeno-hospedeiro são essenciais para este fim. Análise de célula única fornece os meios apenas atribuem inequivocamente um fenótipo celular a um determinado genótipo ou infecção status1. Por exemplo, infecções patogênicas frequentemente induzem alterações directas e indirectas em células hospedeiras. Por conseguinte, distinguir células infectadas de seus homólogos não infectados é necessário atributo anfitrião célula alterações ou infecção direta ou efeitos secundários, tais como generalizada inflamação. Além disso, para muitos patógenos, como SIV e vírus de imunodeficiência humana (HIV), infecção de célula de acolhimento prossegue através de múltiplos estágios, tais como cedo, tarde, ou latente, cada uma delas pode ser caracterizada por gene distinto e de perfis de expressão da proteína2 , 3 , 4 , 5. as análises em massa das misturas de célula irão falhar capturar esta heterogeneidade6. Por outro lado, altamente multiplexado análises de célula única capazes de quantificar a expressão dos dois viral e genes de host oferecem um meio para resolver perturbações celulares específicos de infecção, incluindo variações em fases de infecção. Além disso, analisando interações patógeno-hospedeiro em fisiologicamente configurações relevantes é fundamental para a identificação dos eventos que ocorrem em organismos infectados. Assim, processos de métodos que podem ser aplicados diretamente ex vivo são susceptíveis de melhor captura na vivo .
SIV e HIV CD4-alvo+ T células, no qual eles neutralizar fatores do hospedeiro antiviral "restrição" e downregulate apresentadoras de moléculas para estabelecer infecção produtiva e evitar a vigilância imune7,8, 9,10,11. Sem tratamento, a infecção resulta em enormes perdas de CD4+ T células, em última análise, culminando na adquirida da imunodeficiência síndrome (AIDS)12. No cenário da terapia anti-retroviral, reservatórios de células Latentemente infectadas persistirem por décadas, colocando uma barreira formidável para estratégias curativas. Compreender as propriedades na vivo de células infectadas pelo VIH/SIV tem potencial para revelar características de célula de acolhimento fundamental na patogênese e persistência. No entanto, isto tem sido altamente desafiador, principalmente devido a low frequency de células infectadas e falta de reagentes prontamente reconhecê-los. Células que transcrevem RNA viral, estima-se que para estar presente no 1 – 0,01% de CD4+ T células no sangue e tecido linfoide13,14,15. Sob terapia supressiva, células Latentemente infectadas são ainda menos frequentes em 10-3– 10-7 16,17,18. Coloração da proteína viral ensaios que funcionam bem para estudar em vitro infecções, tais como para Gag intracelular, são suboptimal devido a coloração de fundo de 0.01-0.1%, similar ou maior do que a frequência de células infectadas13, 14. Coloração da superfície para proteína Env usando bem caracterizadas SIV/HIV Env específicas os anticorpos monoclonais também provou ser difícil, provavelmente por razões semelhantes. Recentemente, novas ferramentas que visam melhorar a detecção de células expressando mordaça por qualquer incorporando os ensaios específicos para mordaça RNA ou usando tecnologias alternativas de imagem14,15,19. No entanto, essas abordagens permanecem limitadas no número de medições quantitativas executadas em cada célula.
Aqui, descrevemos a metodologia que (1) identifica células infectadas por vírus única diretamente ex vivo por qPCR quantitativo sensível e específica do gene viral e (2) quantifica a expressão de proteínas de superfície até 18 e 96 genes para cada infectado (e célula não infectada). Esta metodologia combina a medição da proteína de superfície de célula única por FACS seguido de lise celular imediata e expressão de gene análise usando multiplexado qPCR alvo no sistema Biomark. A tecnologia de circuito integrado de fluídico (IFC) permite a quantificação multiplexada de 96 genes de 96 amostras simultaneamente, realizado por uma matriz de 9.216 câmaras em que são realizadas as reações individuais qPCR. A classificação de células vivas FACS registra medições de abundância de alto teor de proteína, enquanto preservar a transcriptoma inteira para análise realizada imediatamente a jusante. Para identificar as células infectadas por vírus, ensaios específicos para RNAs virais alternativamente emendados e unspliced (vRNA) estão incluídos na análise qPCR, juntamente com um painel de ensaios definidos pelo usuário, totalizando até 96 genes, o número máximo de ensaios atualmente acomodadas em a IFC. A expressão de gene e proteína informações coletadas para cada célula são ligados por posição bem. Anteriormente, informou20resultados desta análise em outro lugar. Aqui, nós fornecemos mais detalhadas orientações metodológicas, bem como mais descritiva fenotipagem de CD4 infectadas SIV+ T células.
Esta abordagem, que nós chamamos de tSCEPTRE, pode ser aplicada para as suspensões de qualquer população de célula viável reativa para anticorpos fluorescente etiquetados e expressando um transcriptome compatível com ensaios de qPCR disponível. Por exemplo, ele pode ser usado para caracterizar gene diferencial e expressão de proteínas em raras células ou células não facilmente distinguidas por marcadores de proteína. A preparação da amostra se baseia em um padrão que mancha o protocolo usando anticorpos comercialmente disponíveis. Cytometers com capacidade de classificação de célula única também estão disponíveis comercialmente, mas precauções de biossegurança adicionais são necessárias para o processamento de células vivas infecciosas. Gravar o perfil de expressão de proteínas de célula única – para cada célula pela posição bem, referida aqui como indexados a classificação, é uma característica comum de FACS comercialmente disponível software de classificação. Análise computacional de genes diferencialmente expressos anfitrião entre populações de células de interesse não é descrito aqui, mas as referências são fornecidas métodos publicados anteriormente.
Protocolo
Nota: Um esquema de fluxo de trabalho do protocolo é mostrado na Figura 1. Consiste em três etapas principais: FACS, RT e pré-amplificação do cDNA e qPCR para até 96 genes simultaneamente. Duas versões do protocolo, classificação de células em limitar as diluições e classificação células únicas, são descritas mais detalhadamente nas etapas 5 e 6, respectivamente. Estas estratégias abordar questões de investigação diferentes, mas seguem procedimentos semelhantes.
1. pré-requisito ou prévia análise
- Valide todos os ensaios de expressão de gene para ser usado como descrito anteriormente,6.
Nota: Este passo é feito bem antes da data do experimento. Validando todos os ensaios, comercial e personalizado, é necessário para garantir o eficiente e linear de amplificação do RNA relevante a nível de célula única. Muitos ensaios comercialmente disponíveis e personalizados não cumprem essas especificações. Processamento e encaixe de curva automatizado para qualificação simultânea dos ensaios de expressão de genes até 96 são fornecidos em Arquivos de codificação suplementar 1 – 5, mas ensaios individuais podem ser qualificados usando R2 e inclinação do ajuste linear. Representante do ensaio de sucesso e falha qualificação parcelas são mostradas na Figura 2. -
Desenvolva um painel de fluxo cytometric de anticorpos para mancha de marcadores de superfície celular de interesse.
- Titule anticorpos manchando uma amostra relevante, por exemplo, reso mononucleares células de sangue periférico (PBMC), com cada anticorpo. Começar com 20 µ l de anticorpo por teste em 100 µ l reação de coloração e criar oito diluições de série duas vezes. Identifica a concentração ideal que apresenta a máxima intensidade de coloração, mantendo uma separação clara entre as populações de negativas e positivas.
- Avalie a coloração combinada na amostra de célula adicional usando a mistura de todos os anticorpos na concentração ideal determinada na etapa 1.2.1. Certifique-se de que a coloração é semelhante à observada para manchas individuais de anticorpo. Se a coloração for menor que o que é observado quando qualquer anticorpo foi usado em isolamento, considere a alternativo fluorocromo conjuga para substituir tais anticorpos.
2. Gene Expression ensaio preparação
- Expressão gênica de combinar 96 ensaios em um RNase DNase-livre/tubo de 1 – 15 mL (o tamanho pode variar com o número de placas de classificação). O painel de ensaios utilizado neste estudo é especificado na tabela 1 e tabela 2. O material resultante é referido como o "Mix de ensaio". Adicionar a cada ensaio para uma concentração final de 180 nM de primers para diante e reversos. Adicione DNA suspensão Buffer para alcançar a diluição adequada da mistura do ensaio.
Nota: Para fins práticos, ações personalizadas do ensaio (gerado pelo usuário) podem ser preparadas em 18 µM para coadunar-se com a concentração dos ensaios (Tabela de materiais) qPCR expressão do gene comercialmente disponível "x 20". 18 misturas µM de primer de frente e verso personalizado para cada gene são feitas de soluções estoque no Buffer de suspensão de DNA. Ensaios disponíveis comercialmente (Tabela de materiais) também incluem sondas, mas sondas não são necessárias para RT ou pré-amplificação do cDNA e pode, portanto, ser omitidos para ensaios personalizados. Recomenda-se incluir um ou mais genes de limpeza para usam em controle de qualidade para avaliar a eficiência de classificação, recuperação celular e síntese de cDNA. Uso de primers aleatórios para gerar cDNA não foi determinado, mas é esperado para ser menos eficiente do que as primeiras demão gene-específico. - Prepare o 2 x placa de ensaio para uso em passo 7.1 (multiplex qPCR). Para cada matriz de 96 x 96 microplaqueta antecipado, pipete 6 µ l de cada ensaio para cada um designado a bem de uma placa PCR de 96 poços. Por exemplo, para 5 chips, 30 µ l de cada ensaio ocupará um único poço da placa de 96 poços. Se usar 96 ensaios, cada poço da placa de 96 poços irá conter um ensaio. A placa do selo com selo adesivo.
Nota: Idealmente, passos 2.1 e 2.2 são executados simultaneamente, para evitar vários ciclos de congelamento e descongelamento para ensaios de expressão do gene. Todos os genes dentro da placa de ensaio devem também estiveram presentes no ensaio Mix (passo 2.1). Tanto a mistura de ensaio e a placa de ensaio podem ser armazenadas a-20 ° C ou 4 ° C, para uso de curto ou longo prazo, respectivamente.
3. superfície mancha de células viáveis
Nota: Coloracao intracelular, permeabilização e fixação não são compatíveis com este método como elas comprometer o RNA.
- Prepare as amostras de compensação adicionando cada anticorpo listado na Tabela de materiais a 40 µ l de contas de compensação no 2.5-fold com concentrações mais elevada do que o utilizado para coloracao celular. Incube durante 20 min a 25 ° C, protegido da luz. Adicionar 3 mL de PBS para os grânulos e centrifugar 500 x g por 3 min a 25 ° C. Aspirar a PBS e ressuspender os grânulos em ~ 300 µ l de PBS.
- Classificador de célula de fluxo cytometric Prepare-se para o processamento da amostra: adquirir tubos de compensação, criar matriz de compensação e aplicar a matriz para os arquivos de aquisição para os espécimes experimentais.
- Prepare a mistura mestre de coquetel de anticorpos fluorescentes, combinando o volume apropriado de cada anticorpo conforme especificado na Tabela de materiais, em um tubo de 1,5 mL âmbar para todas as amostras ser manchado. Vórtice e centrifugar o coquetel a 21.000 x g por 2 min a 25 ° C para o anticorpo de pelotas agrega.
Nota: Anticorpos usados aqui são listados na Tabela de materiais. - Descongelar as células criopreservadas em banho-maria 37 ° C por 2 min. Adicionar 0,5-2 mL de suspensão de células para 12 mL de PBS em um tubo de 15 mL, centrifugar 500 x g por 3 min a 25 ° C e aspire a PBS. Resuspenda em 3 mL de PBS e transferir para um tubo de poliestireno de 5 mL. Centrifugar como acima e aspire a PBS, deixando ~ 20 µ l PBS residual.
Nota: A temperatura de coloração pode ser adaptada a temperaturas mais quentes ou mais frias conforme necessário para aplicações específicas, modificando a titulação de anticorpos coloração condições nesse sentido (consulte a etapa 1.2). - Resuspenda até 2 x 107 lavado células em 80 µ l de anticorpo cocktail e incube por 20 min a 25 ° C, protegido da luz. Para amostras superiores a 2 x 107 células, aumentar o volume de reação a coloração conformemente para manter < 2 x 107 células/100 µ l.
- Lavar as células, adicionando-se 3 mL de PBS, centrifugação a 500 x g durante 3 min e aspirar o sobrenadante.
- Exaustivamente Ressuspender as células em 300 – 500 µ l de PBS e filtro pipetando através de uma tampa de filtro de célula de nylon de 35 µm. Manter as células no gelo e protegido da luz, até o tipo.
4. preparar pratos de coleção de célula, executar FACS classificar e gerar cDNA
- Combine os componentes da mistura de reação de RT-pré-amplificador (tabela 3) pipetando igualmente para um único tubo estéril de RNAse/DNAse-livre.
Nota: Esta etapa pode ser executada antes ou durante a coloração na etapa 3. A enzima RT pode ser omitida aqui para determinar a contribuição do modelo de DNA para qPCR sinal. - Use uma pipeta multicanal para dispensar 10 µ l da mistura de reação de RT-pré-amplificador para o número desejado de placas de coleção de tipo PCR 96 poços. Selar as placas com película adesiva e colocar as placas em blocos de alumínio previamente refrigeradas de 96 poços.
- Estabelece a célula classificação associada do regime o citômetro de fluxo através da aquisição de dados de aproximadamente 20.000 células da amostra manchada. Certifique-se de que a matriz de compensação é aplicada aos dados coletados. Portões de desenhar e definir a árvore associada que identifica ou as células populações de interesse para ser isolado para análise de expressão do gene.
Nota: A árvore associada, usada para a coleção de células de vRNA+ potenciais SIV é mostrada na Figura 3. - Insira as configurações de instrumento adequado para especificar o número e o subconjunto de células a serem classificados em cada poço. Instruções detalhadas adicionais para classificação ou uma série de diluição limitante célula ou células individuais são fornecidas nas etapas 5 e 6, respectivamente.
- FACS classificar as células em placas de coleção de PCR de 96 poços preparadas. Remova o selo adesivo antes da classificação e substituir por um selo fresco, seguindo a classificação.
Nota: Manter as placas em blocos de alumínio previamente refrigerados em todos os momentos, inclusive durante a classificação. - Imediatamente após a classificação, vórtice e centrifugar a coleção da placa a 2.000 x g por 1 min a 4 ° C.
- Thermocycle a placa em uma máquina PCR com uma tampa pré-aquecido usando as seguintes condições: 50 ° C por 15 min (RT), 95 ° C por 2 min, seguido de 18 ciclos de 95 ° C por 15 s e 60 ° C durante 4 min (pré-amplificação).
- Dilua a 1:5 do cDNA Transferindo 5 μL de cDNA em 20 µ l de tampão de suspensão de DNA em uma nova placa PCR de 96 poços. CDNA diluído pode ser armazenado em 4 ° C ou a-20 ° C indefinidamente neste ponto. O cDNA é agora pronto para ser usado como um modelo para qPCR (medidas 5.2, 7,4).
Nota: Esta diluição garante que os primers presentes na reação de RT-pré-amplificador não contribuem para a jusante qPCR.
5. variação r: FACS classificar as células em uma série de diluição limitar para determinar a frequência de vRNA+ células ou realizar o controle de qualidade Experimental
Nota: Antes de executar um tipo de célula única, pode ser útil para determinar a frequência de células de interesse, classificando-se as células em diluições em série em replicar. Esta etapa também fornece controle de qualidade valiosa para classificar eficiência, lise celular, recuperação de RNA e síntese de cDNA, conforme descrito na etapa 5.3. Determinação prévia de vRNA+ frequência de celulares permite uma estimativa mais precisa do número de células únicas que devem ser classificados para atingir o tamanho de amostra suficiente para análise de expressão de gene de células de vRNA adequadamente alimentado+ .
- FACS classificar as células nas placas de 96 poços, preparadas como etapas 4.1 – 4.2 e coletar 1 – 1.000 células por poço em múltiplas repetições.
Nota: O número de poços replicar a cada diluição de celular normalmente é inversamente associado com a concentração de células por poço. Quando a frequência de células infectadas é bem abaixo de 1%, diluições de célula devem focar 100 – 1.000 células por poço. Um mapa de placa do tipo de exemplo é fornecido na Figura 1, canto superior esquerdo. Superior a 1.000 células por poço deve ser evitado devido o aumento resultante no volume de reação e interferência com a síntese do cDNA a jusante e quantificação. - Combine os reagentes qPCR em uma solução de mistura de mestre na tabela 4. Para um volume de reação 25 µ l, 22,5 µ l do mix mestre é combinado com 2,5 µ l de cDNA diluídos modelo da etapa 3,9. Executar o qPCR usando condições de ciclismo padrão (por exemplo, 94 ° C por 5 min, seguido de 40 ciclos de 94 ° C por 15 s e 60 ° C por 1 min).
Nota: Singleplex qPCR reações usando um instrumento convencional em tempo real qPCR são recomendadas como uma análise econômica preliminar de um ou alguns ensaios para demonstrar eficiente célula classificação, recuperação de RNA e síntese de cDNA. Ele também pode ser usado para calcular a frequência de células vRNA+ . Reações de qPCR multiplex usando a Biomark são tipicamente mais apropriadas para análises em larga escala de célula única. - Para controle de qualidade, lote Et valores (Et = −máxima do Ct Ct) versus números de células ordenadas por bem em uma escala de10 log e aplicar uma análise de regressão linear.
Nota: Réplicas consistentes, regressão linear inclinação de 3.3 (± 0,3) e R2 > 0.9 são indicativos de uma experiência eficiente. Exemplos de tipo ideal e de qualidade inferior, experimentos de RT-pré-amplificador são mostrados na Figura 4. - Para determinar a frequência de células vRNA+ , plotar o número de células ordenado por alvéolo no eixo x (escala de log10 ) e a fração de poços positivos para vRNA em cada diluição de célula no eixo y. Para obter um exemplo, veja a Figura 1 (inferior esquerda, distribuição de Poisson). Aplica um modelo de regressão linear para os dados para determinar o número de células que abrigam uma célula positiva, em média, correspondente a 63,2% dos poços positivo (0.632 no eixo y)21. Converta este número de diluição de célula (intercepção do eixo x) em frequência, expressada em percentagem. Por exemplo, uma célula de vRNA+ por 48 células é equivalente a uma frequência de 2,1%.
6. variação b: tipo de FACS células para análise de célula única
- Siga os passos 4.1 – 4.8 e especificar uma célula classificada por bem utilizando o recurso de tipo de índice do citômetro de fluxo para criar arquivos de FCS individuais para cada célula classificados, mapeado por posição bem.
Nota: Se o número de placas de coleção do tipo excede o número de thermocyclers disponíveis, andar de bicicleta pode ser interrompido após a etapa de inativação de transcriptase reversa (95 ° C por 2 min) e cDNA pode ser armazenado a 4 ° C até thermocyclers estão disponíveis. Neste caso, iniciar o pré-amplificação no primeiro ciclo de 95 ° C por 15 s. - Opcional: Crie cDNA "pools", consistindo de cDNA de lotes definidos pelo usuário de células únicas para triagem de raras células de interesse usando residual do cDNA não diluído. Transferi 2 µ l do cDNA de pre-amplificado não diluído unicelulares com uma pipeta multicanal para uma nova placa de 96 poços. Repita por pipetagem 2 μL de cDNA de células única todos adicionais de um pool designado para o mesmo bem. Tela do cDNA piscinas para genes de interesse (por exemplo, vRNA) para determinar aqueles que contêm células positivas usando qPCR convencional. Desde pool requer apenas uma pequena alíquota do cDNA de cada célula, os restantes µ l ~ 8 de cDNA ainda está disponível para análises de célula única.
Nota: Pool de estratégias são recomendadas antes de realizar as análises de expressão de gene de célula única em um esforço para reduzir o número de células únicas interrogado por qPCR multiplex de uso intensivo de recursos. É adequado para situações em que as células de interesse (por exemplo, SIV mRNA +) podem ser identificadas por um ensaio de qPCR preliminar single-plex. Estratégias simples de alta produtividade para criar pools de cDNA incluem combinando 2 µ l de cDNA não diluído de linhas (ou seja, todos os 12 células na linha A) ou colunas (ou seja, 8 todas as células na coluna 1), de uma placa tipo de coleção em um único bem em um prato novo. Para determinar a melhor estratégia de pool, considere a frequência esperada das células de interesse da etapa 5.4. Por exemplo, se 10% das células são esperados para ser positivo, piscinas, compostas de seis amostras de cDNA de célula única frequentemente será negativas e as células representadas naquela piscina, portanto, podem ser excluídas das análises de célula única a jusante.
7. multiplex qPCR na plataforma Biomark
Nota: Esta seção pode seguir ou a versão A ou a B descrito acima. No estudo aqui descrito, aplicou-se exclusivamente à análise de célula única.
- Prepare a placa de ensaio de qPCR pipetagem µ l 4 de cada ensaio do 2 x placa de ensaio (preparado no passo 2.2) em uma nova placa PCR de 96 poços contendo 4 µ l de reagente em cada cavidade de carga de ensaio. Manter a placa de ensaio a 4 ° C.
Nota: A placa de ensaio é estável a 4 ° C, durante uma semana e a-20 ° C, durante um mês. Assim, pode ser útil preparar material suficiente para vários chips e armazenar adequadamente. - Dispense os fluidos de linha controle de seringas de escorva para as válvulas de dois entrada do chip. Retire o plástico de protecção debaixo da placa. Coloque o chip em um controlador de IFC, com o lado entalhado na posição A1. No menu principal, selecione o script "Prime". Execute o script.
- Prepare a mistura de reação em tempo real pela mistura de 50 µ l de amostra reagente com 500 µ l de PCR Master Mix (Mesa de materiais) para cada chip microfluidic de carregamento. Pipete 4,4 µ l em cada poço de uma nova placa PCR de 96 poços, doravante designada como a "placa de amostra".
- Pipete 3,6 µ l do cDNA diluído 1:5 da etapa 4.8 na placa da amostra contendo a mistura de reação em tempo real.
Nota: Se uma seleção para baixo de PCR foi realizada para tela para raros (por exemplo, vRNA+) células para análise a jusante, como discutido na etapa 6.2, inclua apenas as células representadas nas piscinas positivas. - Na sequência da conclusão de escorva da microplaqueta, carrega as entradas de chip pela distribuidora 5 µ l da placa de ensaio para a correspondente bem no lado entalhado (ensaio) do chip e 5 µ l da placa de amostra para a correspondente bem do outro lado (amostra) do chip. Insira o chip controlador da IFC e execute o script "Mistura de carga".
- Transferi o chip para a plataforma Biomark para executar o qPCR multiplexado. Prosseguir com a instalação do instrumento e qPCR programação seguindo as instruções passo a passo fornecido pelo software de análise de PCR em tempo real e usando o protocolo v. padrão 1 Gene Expression (GE) 96.96 com 40 ciclos de PCR. Salve o arquivo ChipRun em uma pasta designada.
Nota: Fichas múltiplas podem ser executadas por dia e durante vários dias. - Analise os dados de qPCR.
- Abra o Software de análise PCR em tempo real. Abra o arquivo "ChipRun.bml" do "arquivo | No menu aberto".
- Localize o "Chip Explorer" e "Chip Run Resumo" no canto superior esquerdo da janela do software. Identificar os três componentes do Chip executar Resumo: análise de pontos de vista, amostra, Detector de instalação e.
- Clique em "Instalação do Detector". Em "Tarefa", clique em "Novo" e selecione o tipo de recipiente "Placa SBS" e formato de contêiner "SBS96". Ao lado de "Mapeamento", clique sobre o... e seleccione "M96-ensaio-SBS96.dsp".
- Opcional: Atribua cada Detector (ensaio) de um número ou nome na seção "nome" de cada poço, clicando duas vezes sobre o 1st bem. Mover para o próximo bem pressionando "F2".
- Clique em "Configuração de amostra". Em "Tarefa" ao lado de "Mapeamento", clique no... e seleccione "M96-amostra-SBS96.dsp".
- Clique em "Pontos de vista de análise". Em "Tarefa" na aba "qPCR", selecione "Correção de linha de base para Linear (derivado)," e "Método de limiar de Ct para usuário (detectores)". Na aba "Limiares de Ct", verifique o "inicializar com caixa automática". Clique no botão "Analisar" acima.
- No quadrante superior direito de "Análise de pontos de vista", clique na guia segundo "Tabela de resultados". No menu suspenso, selecione "Exibição de mapa de calor". O mapa de calor com dados aparecerão.
- Opcional: Para garantir a fluorescência de ROX uniforme entre o chip, selecione "Exibir imagem" em vez de "Vista de mapa de calor" no mesmo menu. Na segunda da janela da direita acima o mapa de calor, selecione "ROX". No primeiro da janela da direita, selecione um dos ciclos de 1 a 40. Clique no quarto da janela da direita para alternar para exibição em preto e branco da fluorescência de ROX. Uma imagem aparecerá que informa splatters, partículas, ou defeitos no chip. Se a uniformidade ROX grosseiramente é obscurecida, execute novamente o chip.
- No mapa de calor, clique em "Limite" e "Gráfico de Log". Ajuste os limites de Ct manualmente para cada detector clicando em ensaios (colunas do mapa de calor) e arrastando o limiar necessário para se cruzam as curvas de amplificação na fase exponencial. Quando terminar, clique em "Analisar".
- Exporte os dados de qPCR como um arquivo. csv. Importar os dados em uma planilha ou software de análise estatística (por exemplo, JMP) e mapear os resultados por amostra e ensaio posições no chip. Organize as células em grupos com base na expressão de genes virais, criando uma nova coluna e usando uma fórmula condicional. Em "Analisar", selecione "Fit Y por X" e traçar a expressão gênica contra o grupo. Aplica a análise estatística.
Nota: Representante célula única expressão quantitativa das quatro espécies de RNA SIV é retratado em parcelas bivariadas na Figura 5A. Expressão do gene hospedeiro quantitativa em células RNA SIV+ é mostrada na Figura 5B. - Extrai valores de expressão quantitativa de proteína de célula única FACS dados.
- Abra o .fcs arquivos a partir do FACS classificar (etapa 6.1) correspondente à placa de 96 poços, usando FlowJo versão 9. Com destaque o nome do arquivo, selecione "plataforma | Portão número de evento | Crie tipo indexado gates". Células individuais aparecerá exibidas por linha.
- Destacar todas as 96 células (não linhas) e selecione "área de trabalho | Exportação | Selecione o fluors tudo compensadas". Em "Tipo de dados", selecione "Arquivo de FCS", clique em "Exportar" e selecione uma pasta designada.
- Arraste os arquivos de .fcs novo para células individuais em um novo espaço de trabalho FlowJo. Destacar todas as células, clique em "Adicionar estatísticas" (o botão de "Σ" no canto superior esquerdo) "| Quer dizer | Todos os parâmetros de fluor".
- Abra o "Editor de tabela" clicando no quarto botão da esquerda no canto esquerdo superior. Destacar todas 3U da primeira célula e arrastá-los para a janela do editor de tabela. Na janela do editor de tabela, clique no mesmo botão na parte superior, "Criar e visualização da mesa". Isto irá criar uma tabela de 96 células e um parâmetro numérico para cada fluor.
- Copie a saída para um software de banco de dados (por exemplo, MS Excel, JMP), ou copiando/colando ou clicando em "Salvar e lançar aplicativo" (quarto do botão esquerdo acima da tabela).
Nota: Este procedimento é específico para FlowJo versão 9. FlowJo versão 10 usa um procedimento diferente para importar dados indexados. Dados de fluxo indexada também podem ser copiados/colados em JMP diretamente de arquivos. csv criado pelo classificador de célula.
- Mesclar dados de célula única FACS e qPCR pelo número da placa e posicionar bem. Realizar análises gráficas e estatísticas sobre o gene de célula única combinado (qPCR) e dados de expressão (FACS) da proteína.
Nota: Exemplos de célula única combinada qPCR e dados de FACS são mostrados na Figura 6 (hospedar perfis de expressão da proteína de superfície de células de macaco rhesus infectados com SIV, emendados vRNA+ ), Figura 7 (CD4 expressão gênica contra a superfície CD4 expressão de proteínas nas células de macaco rhesus emendados vRNA+ ) e anteriormente publicado20. Para identificar diferencialmente expressos genes em células ou populações de interesse, célula única análise métodos descritos anteriormente são recomendados20,22,23,24, que respondem por a proporção de células positivas para um gene, bem como o valor da expressão de gene contínua.
Resultados
O fluxo de trabalho para o protocolo inteiro é representado na Figura 1. Consiste em duas variações definidas pelo número de células ordenadas: qualquer diluição limitante ou como único células, conforme descrito no texto. Exemplos de análises de qualificação da primeira demão-sonda em 2 vezes diluições em série do RNA são mostrados na Figura 2. A estratégia associada para identificar potenciais células SIV+ é mostrada na Figura 3. Um qPCR bem sucedida, qualidade inferior e falha de controle de qualidade para o gene de limpeza GAPDH em células FACS-classificados na limitação de diluição são mostrados na Figura 4. Célula única expressão quantitativa de quatro SIV RNA espécies e rhesus macaque genes diferencialmente expressas em células infectadas são mostrados na Figura 5. Representante bivariada, histograma e scatterplots retratam o perfil de expressão de proteínas de superfície do RNA SIV+ CD4+ T medido de células por citometria de fluxo na Figura 6. O enredo de bolha trivariate (Figura 7) exibe a relação entre o gene viral quantitativa, CD3 ou CD4 mRNA e proteína CD3 ou CD4 (tat/rev) expressão em células únicas.

Figura 1 : Diagrama de fluxo de trabalho experimental ilustra três componentes principais: fluxo cytometric tipo, transcrição reversa mais baseados em PCR pré-amplificação do cDNA (RT, pre-amp) e qPCR. A classificação pode ser executada como uma diluição de qualquer limitação (A, pano de fundo verde) ou como células únicas (B, pano de fundo laranja). Imediatamente após a classificação de FACS, células lysed e RNA é reverter transcritos em cDNA e pre-amplificado (RT, pre-amp PCR) para preparar o modelo qPCR. Os tipos de diluição limitante determinam a frequência de células positivas para o RNA viral usando as estatísticas de distribuição de Poisson, bem como a eficiência experimental e recuperação da amostra. A cabeça da seta verde indica que o número estimado de células classificados por bem que contém uma célula positiva para um gene viral (correspondente a uma probabilidade de 63,2% de tais poços sendo gene positivo), que é convertido em uma frequência de cela. Estimativas de frequência podem ser usadas para informar o número de células únicas coletadas em uma classificação subsequente (B). Indexado monocelulares FACS classificação depósitos um celular por alvéolo e gera arquivos de dados para cada célula anotada por posição bem dentro da placa de 96 poços. Célula única qPCR é executada no multiplex para 96 genes simultaneamente. Combinando a proteína de superfície (FACS) e a expressão de RNAm permite a caracterização das células individuais (coluna direita). Um opcional qPCR para um gene viral pode ser realizada entre o pré-amplificação do PCR e qPCR multiplexado (B, meio) de células única tela ou piscinas de cDNA de célula única para baixo-selecione as células de RNA+ virais ou piscinas para análise de qPCR multiplex. O mapa de calor ilustra a expressão dos genes (valor de Ct) para 96 ensaios (colunas) e 96 células únicas (linhas). Clique aqui para ver uma versão maior desta figura.

Figura 2: Representante qPCR dados de experiências de qualificação da primeira demão são mostrados para bem sucedida (esquerda e meio) e falhou (à direita) comercialmente disponíveis ensaios (CD6, SIV tat/rev, e TLR3, respectivamente). Para CD6 e TLR3, RNA foi extraído de macaca de FACS-classificados do rhesus 106 PBMC CD4+ T-células usando um kit comercial. Oito repetições de uma série de doze-ponto do RNA dupla diluição (0,023-48 ng RNA, correspondente a 1.2 – 2.400 equivalentes de célula assumindo pg 20 RNA por CD4+ células T) foram submetidos a RT-pré-amplificador e qPCR. Para SIV tat/rev, o RNA foi extraído das rhesus macaque PBMCs infectados em vitro com SIVmac239. Diluições de RNA foram preparadas abrangência equivalentes de RNA de células 6 – 12.000. Et valores (40-Ct), que aumentam com a expressão de gene, são representados graficamente em função estimada de celulares. Série de diluição exibindo R2 > 0.97 e inclinação de ± 0,3 3.32 indicam qualificação bem sucedida da primeira demão. Clique aqui para ver uma versão maior desta figura.
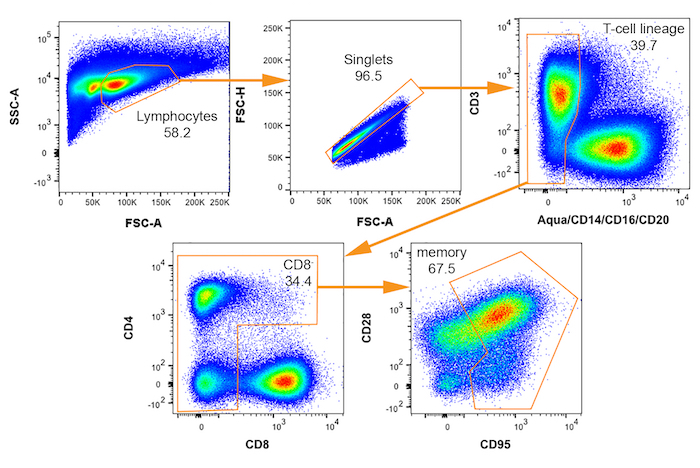
Figura 3: FACS bivariada parcelas com retenção de esquema para o isolamento de células de macaco rhesus potencialmente infectados pelo SIV. Gates sequenciais para selecionar memória (CD95 +) CD4+ T células são mostradas da esquerda superior para inferior direito com o nome de cada população indicada. Por cento de trama de pai que cai dentro de cada porta é indicado. Clique aqui para ver uma versão maior desta figura.

Figura 4 : Representante validação experimental de qPCR executada em células FACS-classificados em limitar a diluição usando GAPDH gene das tarefas domésticas para três experimentos independentes: sucesso (esquerda), qualidade inferior (meio) e falhou (à direita). Repetições de 10 a 100 células por poço devem abranger Ets não superior a 2 e poços de célula 300 – 1.000 dentro 1 Et. A inclinação da regressão linear deve ser 3.32 ± 0,3, R2 > 0,95. Impossibilidade de atingir essas especificações indica dificuldades técnicas nas etapas 1, 2, 3 ou uma combinação destes. Clique aqui para ver uma versão maior desta figura.

Figura 5 : Monocelulares quantitativa viral e host de expressão gênica em macaco rhesus FACS-classificados CD4+ As pilhas de T do nó de linfa mesentérica infecção de post-SIVmac251 de 10 dias. (A) expressão de gene Viral bivariada parcelas de multiplicar-emendados (tat/rev) e emendados isoladamente (env) SIV mRNA pelo single células (à esquerda). Tat/rev+env– células (luz verde ao longo do eixo x) expressam menos cópias de tat/rev RNA do que as células env+ (verde escuro), consistentes com um início estágio de infecção e antes da estabilização de proteínas Rev-mediada e exportação nuclear de vRNAs parcialmente emendados como env. Células que não expressam RNA viral emendado são representadas em cinza (não do RNA viral), brown (amordaçá-+ e LTR+) ou tan ( amordaçá-+ ou LTR+). Unspliced (amordaçá-+) e a total expressão de RNAm SIV (LTR+) é indicado para as mesmas células (direito). Elevada abundância de unspliced mordaça RNA em tat/rev+env+ células é consistente com tarde infecção produtiva fase durante a qual abundante RNA genômico é expressa para embalagens em brotamento virions. (B) violino parcelas de genes de macaco rhesus diferencialmente expressados pelo menos um subconjunto de células infectadas com SIV em comparação com células não infectadas (cinza). Análise estatística foi realizada conforme descrito anteriormente,20,22,23,24. Asterisco indica a taxa de falsa descoberta < 10% em comparações de teste de relação de probabilidade combinada em relação as células não infectadas. Linha conecta-se a valores médios entre grupos de célula para cada gene. Esta figura foi modificada em Bolton et al 20 Clique aqui para ver uma versão maior desta figura.

Figura 6: Perfis de expressão representante anfitrião proteína de superfície de células FACS-classificados da infecção pós-SIVmac251 do rhesus macaque linfonodos mesentéricos 10 dias. Gráfico de dispersão (A) Exibir de expressão de proteínas de superfície CD3, CD4 e ICOS em não infectados (cinza), amordaçá-+tat/rev.– (marrom) e células detat/rev+ amordaçá-+(verde). Intensidade da fluorescência é plotada para cada célula (ponto). Outlier caixa enredos retratam o intervalo interquartil (IQR) e mediana (caixa), os pontos mais distantes dentro de 1,5 x IQR das caixa (bigodes) e potencial outliers (pontos desconectados). Barras horizontais na parte superior indicam diferenças significativas (p < 0.05, teste não paramétrico Wilcoxon rank). (B) exibir bivariada e histograma de superfície CD3 e CD4 expressão de proteínas para as células, mostrado em (A). Trama do ponto (à esquerda) indica percentagens de cada população de células dentro de um quadrante. CD3 e CD4 histogramas (centro, direita) retratam downregulation de proteína entre amordaçá-+tat/rev+ células em relação a não infectados e amordaçá-+células detat/rev.– . (C) doze representante tat/rev+ células únicas de (A – B) são mostradas em três parcelas bivariadas para expressão de superfície de CD3/CD4 (à esquerda), CD69/CD38 (médio) e ICOS/HLA-DR (à direita). Clique aqui para ver uma versão maior desta figura.
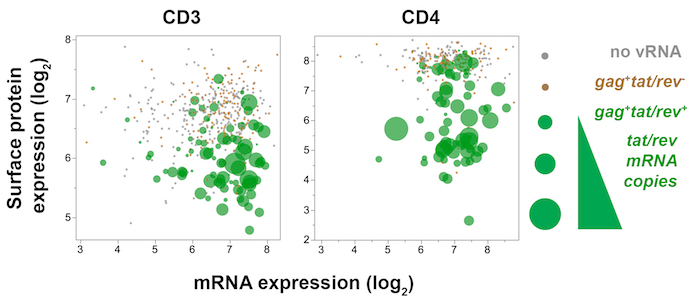
Figura 7 : Trivariate enredo exibindo monocelulares gene viral (tat/rev), gene hospedeiro (CD3 ou CD4) e expressão de proteínas de superfície (CD3 ou CD4) de anfitrião na memória infectados com SIV CD4+ T células do nó de linfa mesentérico 10 dias infecção pós-SIVmac251. Expressão da proteína de CD4 (fluorescência) é plotada contra CD4 mRNA (qPCR), enquanto a quantidade de tat/rev expressado por cada célula é refletida pelo tamanho do ponto. Em tat/rev+ células (verde), diminuição da superfície CD4 e CD3 expressão da proteína com transcrições de CD4 e CD3 sustentadas, respectivamente, indicam que a expressão da proteína de superfície modulada a jusante da expressão do gene. Clique aqui para ver uma versão maior desta figura.
Tabela 1: Primers e sondas utilizadas para a detecção de ácidos nucleicos SIV. Quando duas sequências são indicadas para uma primeira demão ou sonda, montantes equimolar de ambas as sequências foram usados. Clique aqui para baixar este arquivo.
Tabela 2: um painel de 96-gene utilizado para quantificação de amplicons Biomark instrumento. Quatro ensaios SIV são indicados com fundo azul. Clique aqui para baixar este arquivo.
Tabela 3: Mistura de reação, usada para a transcrição reversa e pré-amplificação. Clique aqui para baixar este arquivo.
Tabela 4: mistura de reação de qPCR usada por PCR em tempo real realizado em um instrumento de Quant Studio 6. Clique aqui para baixar este arquivo.
Arquivo suplementar codificação 1. Instruções para ensaios de expressão do gene de qualificação. Clique aqui para baixar este arquivo.
Arquivo de codificação suplementar 2: modelo de mapa de amostra na JMP. Clique aqui para baixar este arquivo.
Arquivo de codificação complementar 3: modelo de Probe mapa na JMP. Clique aqui para baixar este arquivo.
Arquivo de codificação suplementar 4: roteiro de análise de Primer para JMP. Clique aqui para baixar este arquivo.
Suplementares codificação arquivo 5. Roteiro de análise em trechos para JMP. Clique aqui para baixar este arquivo.
Discussão
O protocolo descrito aqui, denominado tSCEPTRE, integra a quantificação da proteína de superfície de célula única por citometria de fluxo multiparâmetros com expressão de RNAm de célula única quantitativa por RT-qPCR altamente multiplexado. A União dessas duas tecnologias permite snapshots de alto teor do combinado transcriptional e perfil de proteína de células individuais em um formato de alta produtividade. Usamos o método para identificar células até então indescritíveis infectadas com SIV na vivoe descrever genes diferencialmente expressos anfitrião e proteínas. O protocolo pode ser adaptado para o estudo de qualquer população de células de interesse distinguível pela expressão de superfície antisentido, mRNA ou uma combinação destes. Os métodos descritos dependem de classificação exata de célula única e gravação por citometria de fluxo de dados emparelhados com os reagentes de qPCR comercialmente disponíveis e multiplexagem Instrumentação de PCR em tempo real. A saída é sensível avaliação quantitativa de célula única proteína e gene expressão dados combinados.
Outras abordagens, vinculando a expressão da proteína e do gene em células individuais têm sido descritas,1,25,26,27. Indexado FACS classificação seguido por RNAseq, aplicado com sucesso para a caracterização de células-tronco hematopoiéticas, representa um particularmente promissora abordagem28. No entanto, enquanto RNAseq tem várias vantagens sobre análises de expressão do gene alvo, ou seja imparcial completo transcriptome análise, é sujeito a uma maior comparações múltiplas estatístico custo e podem ser menos sensível para a quantificação de cópia baixa transcrições. Além disso, não é atualmente economicamente viável para realizar RNAseq em milhares de células em busca de raras células infectadas presentes nas frequências < 1% (por exemplo, HIV, SIV). Outras tecnologias emergentes de célula única visam gerar uma leitura única, ou seja, pela FACS ou PCR, para a deteção de proteínas e ácidos nucleicos na mesma cela. Estes incluem a detecção de proteínas por anticorpos fluorescentes e mRNA por sondas fluorescentes do oligonucleotide, seguido de análise FACS (rnam-fluxo-peixe)14,15,29,de30. Alternativamente, as proteínas podem ser detectadas por pares de conjugados de anticorpo-do oligonucleotide que são detectados por qPCR em paralelo com mRNA transcrito inversamente31,32,33,34 . Essas abordagens "híbrido" fornecem simultânea do ácido nucleico e proteína medições com célula única resolução semelhante ao tSCEPTRE, mas eles são limitados onde eles são ou não quantitativos (rnam-fluxo-peixe) ou podem exigir personalizados anticorpo ou mRNA sondas. Nossa abordagem tSCEPTRE é quantitativa para medições tanto proteínas e RNAm e todos os reagentes estão disponíveis comercialmente, com a possível exceção dos ensaios específicos do patógeno.
Atenção especial deve ser aplicada a vários passos no protocolo antes da análise das amostras experimentais. Primeiro, citometria de fluxo alto-parâmetro requer cuidadosa otimização de um painel de anticorpos fluorescentes para garantir sensível deteção e resolução35. Cada anticorpo deve ser titulado individualmente para identificar a concentração que atinge a separação ideal e coloração de fundo mínimo, seguindo-se a avaliação da coloração quando todos o anticorpo cojugado é usado em combinação. Experimental, segunda determinação do ensaio de expressão do gene qPCR apropriado para medir cada gene de interesse é essencial. Vários ensaios disponíveis comercialmente são normalmente disponíveis para cada gene, mas em nossa experiência, muitos não são quantitativos no monocelulares nível6. Assim, é importante qualificar tudo propostos ensaios sobre serialmente diluído antes do RNA uso para expressão gênica quantitativa. Otimização destes reagentes é demorada, mas o valor de confiança painéis de anticorpos conjugados e ensaios de expressão do gene que permite detecção sensível de todas as moléculas de interesse justifica o investimento de tempo. Além disso, deve ser dada prioridade para ensaios comercialmente disponíveis contendo sondas que abrangem as junções exon-exon (designadas com sufixo "m1") para melhorar a especificidade para mRNA, embora suplente ensaios capazes de detectar o DNA genômico (sufixo "s1" ou "g1" ) são considerados susceptíveis de influenciar os resultados de expressão de gene devido equivalentes cópias de cromossomos em cada célula. Preservação do RNA o montante de RT é fundamental e é conseguida mantendo amostras refrigeradas, como observado em todo o protocolo. Da mesma forma, a duração da classificação de FACS e o intervalo entre a classificação e RT-pré-amplificador deve ser mantida a um mínimo. Para tanto limitante diluição e de célula única classificação, cDNA pode ser primeiro analisado por qPCR convencional para avaliar a recuperação de RNA de altamente expressa housekeeping genes. Se a expressão observada não é uniforme entre repetições de números de celular, ou a inclinação do ajuste linear para limitar a validação falhar de diluições, solução de problemas deve ser realizada em procedimentos de classificação e a jusante da pilha.
Potenciais limitações da abordagem descrita aqui incluem (1) detecção de DNA além de RNA, particularmente para os ensaios não específicos para as junções da tala do mRNA e (2) restrito número de proteínas de superfície e genes medidos. No entanto, a detecção de DNA é improvável contribuir substancialmente para análise de expressão de gene hospedeiro diferencial pelo motivo dito acima. Além disso, medimos diretamente na medida em que DNA contribui para o sinal de qPCR neste protocolo por duas abordagens: a) excluindo a transcriptase reversa e b) modificar o protocolo de lise celular para realçar a lise da membrana nuclear. Na ausência de transcriptase reversa, ambos emendados e unspliced de genes virais foram detectadas ainda, mas em diminuir significativamente a frequência do que na presença de transcriptase reversa (~ reduções 6-fold e 2 vezes, respectivamente)20. Portanto, os ensaios de expressão do gene viral podem superestimar a quantidade de RNA viral presente em uma célula devido à presença de DNA viral associada a célula. Atribuímos este achado para produtos de RT citoplasmáticos gerado durante a infecção de célula de acolhimento, conhecida deve ocorrer para emendados e unspliced SIV/HIV RNA proveniente de entrada virions36. Assim, pode ser aconselhável dedicar uma parte das experiências de condições falta transcriptase reversa para dosar DNA-derivado de modelo para algumas aplicações. Também deve ser notado que não possa ser distinguida unspliced SIV/HIV RNA derivado entrada vírus RNA viral de novo sintetizado. Em segundo lugar, nós incorporada uma etapa de Lise nuclear o protocolo de RT-pré-amplificador e observou um aumento exponencial nas cópias de DNA SIV (Alu-LTR) integrados (Figura S3 de Bolton et al) 20. DNA genômico, portanto, é improvável que contribuem substancialmente para o modelo de ácidos nucleicos extraído do método de lise celular usado aqui. Digno de nota, a adição de uma etapa de Lise nuclear pode ser útil no futuro monocelulares estudos buscando investigar SIV ou outras células de DNA positivo de vírus, incluindo células Latentemente infectadas.
O número de proteínas de superfície analisada é determinado pela capacidade de classificador de célula de fluxo cytometric. Instrumentos disponíveis no mercado atuais não exceda 30 parâmetros. Futuros estudos empregando mais avançada citometria de fluxo ampliará ainda mais a capacidade desta abordagem de criação de perfil de proteína. O número de transcrições também pode ser estendido para 96, reagentes de suporte fornecidos (por exemplo, as primeiras demão de maior concentração), além de equipamentos e instrumentação. Em última análise, emergentes tecnologias de célula única que combinam a análise do proteoma (espectrometria de massa), transcriptoma (RNAseq) e o genoma (DNAseq) substituirá as abordagens orientadas para descoberta pesquisa31,37, 38 , 39. no entanto, qPCR alvo provavelmente continuará a ser uma ferramenta valiosa para validar tais abordagens "omics" como um padrão de ouro para análises quantitativas de expressão.
Análise combinada de proteína e transcrição por tSCEPTRE é uma poderosa ferramenta para investigar raras ou difíceis de identificar células como aqueles abrigar patógenos, contendo oncogenes ou caso contrário, exibindo um fenótipo aberrante. Novos marcadores para células transcricionalmente ativos SIV/HIV infectados podem ser identificados desta forma, bem como a descoberta de novos mecanismos envolvidos na patogênese destas infecções virais. Identificação de células Latentemente infectadas exigirá o desenvolvimento de um protocolo para avaliar DNA viral integrado no genoma do hospedeiro. Digno de nota, a frequência de células infectada HIV/SIV é consideravelmente menor em infecção crônica virémicos ou tratada, que apresentará um desafio prático em estudar células infectadas derivado essas configurações. Nossa abordagem estabelece as bases para avaliar um mecanismo anteriormente intratável: a de regulamento pós-transcricional a nível de célula única, e tem ampla aplicabilidade para interações patógeno-hospedeiro, bem como processos celulares mais gerais.
Divulgações
Este trabalho foi apoiado por um acordo de cooperação (W81XWH-07-2-0067) entre M. Henry Jackson Foundation para o avanço dos militares medicina, Inc. e o departamento E.U. de defesa (DOD). As opiniões expressadas são as dos autores e não devem ser interpretadas para representar as posições do exército dos EUA ou o departamento de defesa. Pesquisa foi conduzida sob um protocolo aprovado uso de animais em uma instalação de AAALACi credenciado em conformidade com o Animal Welfare Act e outros estatutos federais e regulamentos relativos a animais e experimentos envolvendo animais e adere aos princípios indicado no guia para o cuidado e uso de animais de laboratório, publicação do NRC, edição 2011.
Agradecimentos
Os autores gostaria de agradecer o NIAID VRC Flow Cytometry núcleo e as instalações do núcleo de citometria de fluxo MHRP para manutenção e operação de instrumentos de FACS e classificação de equipamentos; Maria Montero, Vishakha Sharma, Kaimei Song para assistência técnica especializada; Michael Piatak, Jr. (falecido) para obter assistência com projeto de ensaio qPCR SIV; e Brandon Keele e Matthew Scarlotta para SIV isolar sequências. As opiniões expressadas são as dos autores e não devem ser interpretadas para representar as posições do exército dos EUA ou o departamento de defesa. Pesquisa foi conduzida sob um protocolo aprovado uso de animais em uma facilidade AAALAC credenciado em conformidade com o Animal Welfare Act e outros estatutos federais e regulamentos relativos a animais e experimentos envolvendo animais e adere aos princípios indicado no guia para o cuidado e uso de animais de laboratório, publicação do NRC, edição 2011.
Materiais
| Name | Company | Catalog Number | Comments |
| RNA extraction and PCR reagents and consumables | |||
| Genemate 96-Well Semi-Skirted PCR Plate | BioExpress/VWR | T-3060-1 | |
| Adhesive PCR Plate Seals | ThermoFisher | AB0558 | |
| Armadillo 384-well PCR Plate | ThermoFisher | AB2384 | |
| MicroAmp Optical Adhesive Film | Applied Biosystems/ThermoFisher | 4311971 | |
| DEPC Water | Quality Biological | 351-068-101 | |
| Glass Distilled Water | Teknova | W3345 | |
| Superscript III Platinum One-Step qRT-PCR Kit | Invitrogen/ThermoFisher | 11732088 | |
| SUPERase-In Rnase Inhibitor | Invitrogen/ThermoFisher | AM2696 | |
| Platinum Taq | Invitrogen/ThermoFisher | 10966034 | |
| dNTP Mix | Invitrogen/ThermoFisher | 18427088 | |
| ROX Reference Dye (if separate from kit) | Invitrogen/ThermoFisher | 12223012 | |
| DNA Suspension Buffer | Teknova | T0223 | |
| RNAqueous kit | Invitrogen/ThermoFisher | AM1931 | |
| TaqMan gene expression assays not listed in Table 2 | |||
| CD6 | Applied Biosystems/ThermoFisher | Hs00198752_m1 | |
| TLR3 | Applied Biosystems/ThermoFisher | Hs1551078_m1 | |
| Biomark reagents | |||
| Control Line Fluid Kit | Fluidigm | 89000021 | |
| TaqMan Universal PCR Mix | Applied Biosystems/ThermoFisher | 4304437 | |
| Assay Loading Reagent | Fluidigm | 85000736 | |
| Sample Loading Reagent | Fluidigm | 85000735 | |
| Dynamic Array 96.96 (chip) | Fluidigm | BMK-M-96.96 | |
| FACS reagents | |||
| SPHERO COMPtrol Goat anti-mouse (lambda) | Spherotech Inc. | CMIgP-30-5H | |
| CompBeads Anti-Mouse Ig,k | BD Biosciences | 51-90-9001229 | |
| 5 ml Polystyrene tube with strainer cap | FALCON | 352235 | |
| Aqua Live/Dead stain | Invitrogen/ThermoFisher | L34976 | dilute 1:800 |
| Mouse Anti-Human CD3 BV650 clone SP34-2 | BD Biosciences | 563916 | dilute 1:40 |
| Mouse Anti-Human CD4 BV786 clone L200 | BD Biosciences | 563914 | dilute 1:20 |
| Mouse Anti-Human CD8 BUV496 clone RPA-T8 | BD Biosciences | 564804 | dilute 1:10 |
| Mouse Anti-Human CD28 BV711 clone CD28.2 | Biolegend | 302948 | dilute 1:20 |
| Mouse Anti-Human CD95 BUV737 clone DX2 | BD Biosciences | 564710 | dilute 1:10 |
| Mouse Anti-Human CD14 BV510 clone M5E2 | Biolegend | 301842 | dilute 1:83 |
| Mouse Anti-Human CD16 BV510 clone 3G8 | Biolegend | 302048 | dilute 1:167 |
| Mouse Anti-Human CD20 BV510 clone 2H7 | Biolegend | 302340 | dilute 1:37 |
| Anti-CD38-R PE clone OKT10 | NHP reagent recource | N/A | dilute 1:100 |
| Mouse Anti-Human CD69 BUV395 clone FN50 | BD Biosciences | 564364 | dilute 1:10 |
| Mouse Anti-Human HLA-DR APC-H7 clone G46-6 | BD Biosciences | 561358 | dilute 1:20 |
| Mouse Anti-Human ICOS Alexa Fluor 700 clone C398.4A | Biolegend | 313528 | dilute 1:80 |
| Instruments | |||
| BioPrptect Containment Enclosure | Baker | ||
| BD FACS Aria | BD Biosciences | ||
| ProtoFlex Dual 96-well PCR system | Applied Biosystems/ThermoFisher | 4484076 | |
| Quant Studio 6 qPCR instrument | Applied Biosystems/ThermoFisher | 4485694 | |
| IFC controller HX | Fluidigm | IFC-HX | |
| Biomark HD | Fluidigm | BMKHD-BMKHD |
Referências
- Macaulay, I. C., Ponting, C. P., Voet, T. Single-Cell Multiomics: Multiple Measurements from Single Cells. Trends Genet. 33 (2), 155-168 (2017).
- Pasternak, A. O., Lukashov, V. V., Berkhout, B. Cell-associated HIV RNA: a dynamic biomarker of viral persistence. Retrovirology. 10, 41(2013).
- Mailler, E., et al. The Life-Cycle of the HIV-1 Gag-RNA Complex. Viruses. 8 (9), (2016).
- Varmus, H. Retroviruses. Science. 240 (4858), 1427-1435 (1988).
- Frankel, A. D., Young, J. A. HIV-1: fifteen proteins and an RNA. Annu Rev Biochem. 67, 1-25 (1998).
- Dominguez, M. H., et al. Highly multiplexed quantitation of gene expression on single cells. J Immunol Methods. 391 (1-2), 133-145 (2013).
- Tokarev, A., Guatelli, J. Misdirection of membrane trafficking by HIV-1 Vpu and Nef: Keys to viral virulence and persistence. Cell Logist. 1 (3), 90-102 (2011).
- Malim, M. H., Bieniasz, P. D. HIV Restriction Factors and Mechanisms of Evasion. Cold Spring Harb Perspect Med. 2 (5), a006940(2012).
- Sugden, S. M., Bego, M. G., Pham, T. N., Cohen, E. A. Remodeling of the Host Cell Plasma Membrane by HIV-1 Nef and Vpu: A Strategy to Ensure Viral Fitness and Persistence. Viruses. 8 (3), 67(2016).
- Simon, V., Bloch, N., Landau, N. R. Intrinsic host restrictions to HIV-1 and mechanisms of viral escape. Nat Immunol. 16 (6), 546-553 (2015).
- Kirchhoff, F. Immune evasion and counteraction of restriction factors by HIV-1 and other primate lentiviruses. Cell Host Microbe. 8 (1), 55-67 (2010).
- Wigzell, H. Immunopathogenesis of HIV infection. J Acquir Immune Defic Syndr. 1 (6), 559-565 (1988).
- Reynolds, M. R., et al. Ex vivo analysis of SIV-infected cells by flow cytometry. Cytometry A. 77 (11), 1059-1066 (2010).
- Baxter, A. E., et al. Single-Cell Characterization of Viral Translation-Competent Reservoirs in HIV-Infected Individuals. Cell Host Microbe. 20 (3), 368-380 (2016).
- Grau-Exposito, J., et al. A Novel Single-Cell FISH-Flow Assay Identifies Effector Memory CD4+ T cells as a Major Niche for HIV-1 Transcription in HIV-Infected Patients. MBio. 8 (4), (2017).
- Eriksson, S., et al. Comparative analysis of measures of viral reservoirs in HIV-1 eradication studies. PLoS Pathog. 9 (2), e1003174(2013).
- Finzi, D., et al. Identification of a reservoir for HIV-1 in patients on highly active antiretroviral therapy. Science. 278 (5341), 1295-1300 (1997).
- Chomont, N., et al. HIV reservoir size and persistence are driven by T cell survival and homeostatic proliferation. Nat Med. 15 (8), 893-900 (2009).
- DeMaster, L. K., et al. A Subset of CD4/CD8 Double-Negative T Cells Expresses HIV Proteins in Patients on Antiretroviral Therapy. J Virol. 90 (5), 2165-2179 (2015).
- Bolton, D. L., et al. Combined single-cell quantitation of host and SIV genes and proteins ex vivo reveals host-pathogen interactions in individual cells. PLoS Pathog. 13 (6), e1006445(2017).
- Quintans, J., Lefkovits, I. Precursor cells specific to sheep red cells in nude mice. Estimation of frequency in the microculture system. Eur J Immunol. 3 (7), 392-397 (1973).
- Finak, G., et al. MAST: a flexible statistical framework for assessing transcriptional changes and characterizing heterogeneity in single-cell RNA sequencing data. Genome Biol. 16, 278(2015).
- McDavid, A., et al. Modeling bi-modality improves characterization of cell cycle on gene expression in single cells. PLoS Comput Biol. 10 (7), e1003696(2014).
- McDavid, A., Finak, G., Gottardo, R. The contribution of cell cycle to heterogeneity in single-cell RNA-seq data. Nat Biotechnol. 34 (6), 591-593 (2016).
- Kok, Y. L., Ciuffi, A., Metzner, K. J. Unravelling HIV-1 Latency, One Cell at a Time. Trends Microbiol. , (2017).
- Rato, S., Golumbeanu, M., Telenti, A., Ciuffi, A. Exploring viral infection using single-cell sequencing. Virus Res. 239, 55-68 (2017).
- Wagner, A., Regev, A., Yosef, N. Revealing the vectors of cellular identity with single-cell genomics. Nat Biotechnol. 34 (11), 1145-1160 (2016).
- Nestorowa, S., et al. A single-cell resolution map of mouse hematopoietic stem and progenitor cell differentiation. Blood. 128 (8), e20-e31 (2016).
- Soh, K. T., et al. Simultaneous, Single-Cell Measurement of Messenger RNA, Cell Surface Proteins, and Intracellular Proteins. Curr Protoc Cytom. 75, 41-47 (2016).
- Kochan, J., Wawro, M., Kasza, A. Simultaneous detection of mRNA and protein in single cells using immunofluorescence-combined single-molecule RNA FISH. Biotechniques. 59 (4), 209-212 (2015).
- Stahlberg, A., Thomsen, C., Ruff, D., Aman, P. Quantitative PCR analysis of DNA, RNAs, and proteins in the same single cell. Clin Chem. 58 (12), 1682-1691 (2012).
- Assarsson, E., et al. Homogenous 96-plex PEA immunoassay exhibiting high sensitivity, specificity, and excellent scalability. PLoS One. 9 (4), e95192(2014).
- Fredriksson, S., et al. Protein detection using proximity-dependent DNA ligation assays. Nat Biotechnol. 20 (5), 473-477 (2002).
- Genshaft, A. S., et al. targeted profiling of single-cell proteomes and transcriptomes in a single reaction. Genome Biol. 17 (1), 188(2016).
- Mahnke, Y. D., Roederer, M. Optimizing a multicolor immunophenotyping assay. Clin Lab Med. 27 (3), 469-485 (2007).
- Liang, C., Hu, J., Russell, R. S., Kameoka, M., Wainberg, M. A. Spliced human immunodeficiency virus type 1 RNA is reverse transcribed into cDNA within infected cells. AIDS Res Hum Retroviruses. 20 (2), 203-211 (2004).
- Stegle, O., Teichmann, S. A., Marioni, J. C. Computational and analytical challenges in single-cell transcriptomics. Nat Rev Genet. 16 (3), 133-145 (2015).
- Wu, M., Singh, A. K. Single-cell protein analysis. Curr Opin Biotechnol. 23 (1), 83-88 (2012).
- Haque, A., Engel, J., Teichmann, S. A., Lonnberg, T. A practical guide to single-cell RNA-sequencing for biomedical research and clinical applications. Genome Med. 9 (1), 75(2017).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados