Method Article
Sola célula cuantificación del mRNA y la expresión de la proteína de superficie de CD4 infectados por el Virus de la inmunodeficiencia de los simios+ T las células aisladas de macaques del macaco de la India
En este artículo
Resumen
Descrito es una metodología para cuantificar la expresión de 96 genes y 18 proteínas de la superficie de células ex vivo, lo que permite la identificación de diferencialmente expresan genes y proteínas en células virus-infectadas en comparación con las células no infectadas. Aplicamos el enfoque de estudio CD4 infectados por SIV+ T las células aisladas de macaques del macaco de la India.
Resumen
El análisis unicelular es una herramienta importante para poblaciones heterogéneas de células de disección. La identificación y aislamiento de células pueden ser difíciles. Para superar este desafío, una combinación de metodología indexadas citometría de flujo y reacción en cadena de alto rendimiento multiplexados cuantitativa de la polimerasa (qPCR) fue desarrollada. El objetivo fue identificar y caracterizar el virus de la inmunodeficiencia símica (VIS)-infectó las células presentes en macaques del macaco de la India. A través de la cuantificación de la proteína de la superficie de separación de células activado por fluorescencia (FACS) y mRNA por qPCR, células infectadas por virus se identifican por la expresión génica viral, que se combina con medidas gen y la proteína de host para crear un perfil multidimensional . Llamamos el enfoque de evaluación unicelulares Proteo-transcripcional dirigida y tSCEPTRE. Para realizar el método, células viables se tiñen con anticuerpos fluorescentes específicas para marcadores de superficie utilizados para el aislamiento de la FACS de un subconjunto de células y análisis fenotípico aguas abajo. Las células se clasifican seguido de lisis inmediata, multiplex transcripción reversa (RT), la amplificación por PCR y qPCR de alto rendimiento de hasta 96 transcripciones. Mediciones de FACS se registran en el momento de la clasificación y posteriormente vinculadas a los datos de expresión genética por posición bien para crear un perfil transcripcional y proteína combinada. Para estudiar infectados por SIV directamente células ex vivo, las células fueron identificadas por la detección de la qPCR de múltiples especies de RNA virales. La combinación de la transcripción viral y la cantidad de cada uno proporcionan un marco para clasificar las células en distintas etapas del ciclo de vida viral (p. ej., productivo versus no-productivos). Además, tSCEPTRE de las células SIV+ eran en comparación con no infectado células aisladas de la misma muestra para evaluar diferencialmente expresados anfitrión genes y proteínas. El análisis reveló previamente poco apreciado heterogeneidad de expresión RNA viral en células infectadas como en vivo gen postranscripcional mediada por SIV Reglamento con resolución unicelular. El método tSCEPTRE es relevante para el análisis de cualquier población de células susceptible de identificación por la expresión del marcador de la proteína de la superficie, host o patógeno genes o combinaciones de las mismas.
Introducción
Muchos patógenos intracelulares dependen de maquinaria de la célula huésped para replicar, a menudo alterando la biología de la célula huésped o dirigidas a subpoblaciones muy específicas de las células del huésped para maximizar sus posibilidades de propagación. Como resultado, los procesos biológicos celulares comúnmente se interrumpen, con consecuencias deletéreas para la salud general del hospedero. Comprensión de las interacciones entre el virus y las células hospedadoras que replican a aclarar mecanismos de la enfermedad que pueden ayudar en el desarrollo de mejores terapias y estrategias para prevenir la infección. Directas herramientas analíticas que permiten el estudio de las interacciones huésped-patógeno son esenciales para este fin. Sola célula análisis proporciona el único medio para atribuir inequívocamente un fenotipo celular a un genotipo particular o infección estado1. Por ejemplo, infecciones patógenas frecuentemente inducen cambios directos e indirectos en las células del huésped. Por lo tanto, distinguir las células infectadas de sus contrapartes no infectadas es necesario cambios de atributo anfitrión célula infección directa o efectos secundarios, tales como generalizada inflamación. Por otra parte, para muchos patógenos, como el SIV y el virus de inmunodeficiencia humana (VIH), infección de la célula huésped procede a través de etapas múltiples, tales como temprano, tarde, o latente, que puede caracterizarse por genes distintos y de perfiles de expresión de la proteína2 , 3 , 4 , 5. Análisis de bulk de mezclas celular dejará de capturar esta heterogeneidad6. Por el contrario, altamente multiplexadas en análisis unicelulares capaces de cuantificar la expresión de ambos virus y genes de host ofrecen un medio para resolver las perturbaciones celulares específicos de infección, incluyendo las variaciones en distintas etapas de la infección. Además, análisis huésped-patógeno en fisiológicamente ajustes pertinentes es fundamental para la identificación de eventos que se producen en los organismos infectados. Así, los procesos de métodos que pueden aplicarse directamente ex vivo son probablemente mejor captura en vivo .
SIV y VIH objetivo CD4+ T las células, en el cual ellos contrarrestar factores antivirales "restricción" de host y downregulate antígeno que presenta las moléculas para establecer infección productiva y evitar la vigilancia inmune7,8, 9,10,11. Sin tratamiento, la infección resulta en la pérdida masiva de CD4+ T de las células, en última instancia culminando en adquirido de la inmunodeficiencia adquirida (SIDA)12. En el marco de la terapia antiretroviral, célula latente infectada depósitos persisten durante décadas, planteando una barrera formidable a estrategias curativas. Comprender las propiedades de en vivo células infectadas por el VIH/SIV tiene el potencial para revelar características de la célula de host en la patogenia y persistencia. Sin embargo, esto ha sido muy difícil, debido principalmente a la obviados de las células infectadas y la falta de reactivos capaces de identificarlos fácilmente. Las células que transcriben el ARN viral, se estima que en 0.01 – 1% de CD4+ T las células en la sangre y tejido linfoide13,14,15. Bajo tratamiento supresivo, células latente infectadas son aún menos frecuentes en 10-3-10-7 16,17,18. La coloración de la proteína viral ensayos que trabajo bien para el estudio de las infecciones in vitro , tales como Gag intracelular, son subóptimos debido a la tinción de fondo de 0.01 – 0.1%, similar o mayor que la frecuencia de las células infectadas13, 14. Coloración superficial para proteína Env bien caracterizados anticuerpos monoclonales SIV y VIH Env-específicas también ha demostrado ser difícil, probablemente por razones similares. Recientemente, nuevas herramientas tienen por objeto mejorar la detección de las células que expresan la mordaza ya sea por la incorporación de ensayos específicos para gag RNA o usando proyección de imagen alternativas tecnologías14,15,19. Sin embargo, estos enfoques siguen siendo limitados en el número de mediciones cuantitativas realizadas en cada célula.
Aquí, describimos la metodología que (1) identifica las células virus-infectadas directamente ex vivo por gene viral sensible y específico cuantitativo qPCR y (2) cuantifica la expresión de proteínas de la superficie hasta 18 y 96 genes por cada infectado (y célula no infectada). Esta metodología combina la medición de la proteína unicelular de la superficie por FACS seguido de lisis celular inmediata y análisis de expresión génica usando multiplexado qPCR específica en el sistema de Biomark. La tecnología integrado circuito fluídico (IFC) permite cuantificación multiplexada de 96 genes de 96 muestras al mismo tiempo, logrado por una matriz de 9.216 cámaras en las que se realizan las reacciones individuales de la qPCR. La clasificación de células vivas FACS registra las mediciones de abundancia de proteína de alto contenido mientras que preserva el transcriptoma completo para análisis realizados inmediatamente aguas abajo. Para identificar las células virus-infectadas, ensayos específicos para RNAs virales o bien empalmados y unspliced (vRNA) están incluidos en el análisis de qPCR, junto a un panel de análisis definidos por el usuario por un total de hasta 96 genes, el número máximo de ensayos actualmente alojados en la IFC. La expresión de genes y proteína información recogido para cada celda están vinculados por la posición bien. Divulgamos previamente los resultados de este análisis en otro lugar20. Presentamos más detallada lineamientos metodológicos así como más descriptivo phenotyping de CD4 infectados por SIV+ T las células.
Este enfoque, que llamamos tSCEPTRE, puede aplicarse a las suspensiones de cualquier población de células viables reactiva a anticuerpos fluorescente etiquetados y expresando un transcriptoma compatible con ensayos de qPCR disponibles. Por ejemplo, puede utilizarse para la caracterización génica diferencial y expresión de proteínas en las células raras o no fácilmente distinguidas por marcadores de la proteína de la superficie de las células. La preparación de la muestra se basa en un estándar de protocolo utilizando anticuerpos comercialmente disponibles de tinción. Citómetros con capacidad clasificación unicelulares están también disponibles comercialmente, pero medidas de bioseguridad adicionales son necesarios para el procesamiento de células vivas infecciosas. Registrar el perfil de expresión – una proteína de la célula para cada celda por bien puesto, contemplado en el presente como indexadas clasificación, es una característica común de FACS comercialmente disponible software de clasificación. Análisis computacional de genes diferencialmente expresados host entre poblaciones celulares de interés no se describen aquí, pero se proporcionan referencias a los métodos previamente publicados.
Protocolo
Nota: Un esquema del flujo de trabajo de protocolo se muestra en la figura 1. Consta de tres pasos principales: FACS, RT y pre amplificación de cDNA y qPCR para hasta 96 genes simultáneamente. Dos versiones del Protocolo, clasificar las células en la limitación de las diluciones y clasificación de las células, se describen en mayor detalle en el paso 5 y paso 6, respectivamente. Estas estrategias de investigación diferentes cuestiones pero seguir procedimientos similares.
1. requisitos previos o previo análisis
- Validar los ensayos de expresión genética para ser utilizado como se describió anteriormente6.
Nota: Este paso se realiza antes de la fecha del experimento. Validación de todos los ensayos, comercial y personalizada, es necesaria para asegurar la amplificación eficiente y lineal de ARN correspondiente hasta el nivel unicelular. Muchos ensayos comercialmente disponibles y personalizados no cumplan con estas especificaciones. Procesamiento y ajuste de curvas automatizado para calificación simultánea de ensayos de expresión de los genes hasta 96 se ofrecen en Archivos de codificación complementaria 1 – 5, pero los ensayos individuales se pueden calificar con R2 y la pendiente del ajuste lineal. Parcelas de calificación representativa ensayo éxito y fracaso se muestran en la figura 2. -
Desarrollar un panel de citometría de flujo de anticuerpos para marcadores de la superficie de la célula de interés de la mancha.
- Valorar anticuerpos por tinción de una muestra relevante, por ejemplo, macaco de la India células mononucleares sangre periférica (PBMCs), con cada anticuerpo. Iniciar con 20 μl de anticuerpo por prueba en 100 μl de la reacción de tinción y crear ocho diluciones dobles en serie. Determinar la concentración óptima que exhibe la máxima intensidad de tinción manteniendo una separación clara entre las poblaciones positivas y negativas.
- Evaluar la coloración combinada en muestras adicionales de la célula mediante la mezcla de todos los anticuerpos a la concentración óptima determinada en el paso 1.2.1. Asegúrese de que la tinción es similar a la observada para las manchas de los anticuerpos. Si la tinción es menos de lo que se observa cuando cualquier anticuerpo se utilizó en el aislamiento, considerar alternativa fluorocromo conjuga para reemplazar dichos anticuerpos.
2. preparación del análisis de expresión génica
- Expresión del gen de la cosechadora 96 ensayos en una Rnasa DNasa libre/tubo de 1 – 15 mL (el tamaño puede variar con el número de placas de tipo). El panel de análisis utilizados en este estudio se especifica en la tabla 1 y tabla 2. El material resultante se conoce como la "mezcla de ensayo". Añadir cada ensayo a una concentración final de 180 nM de cartillas de avance y retroceso. Añadir el tampón de suspensión de ADN para lograr la dilución de la mezcla de ensayo.
Nota: Para fines prácticos, las poblaciones de ensayo personalizados (generado por el usuario) pueden prepararse a 18 μm concordar con la concentración de ensayos de qPCR de expresión de genética disponible en el mercado "x 20" (Tabla de materiales). 18 mezclas μm del primer avance y retroceso personalizado para cada gen se hacen de soluciones en el Buffer de suspensión de ADN. Ensayos disponibles en el mercado (Tabla de materiales) también incluyen sondas y sondas no son necesarias para la RT o la amplificación de cDNA y puede así ser omitido para ensayos personalizados. Se recomienda incluir uno o más genes de limpieza para usan en control de calidad para evaluar la eficiencia de clasificación, recuperación de la célula y síntesis de cDNA. Uso de los primers al azar para generar cDNA no ha sido determinada, pero se espera que sea menos eficiente que cartillas gene-específicas. - Preparar 2 x placa de ensayo para su uso en el paso 7.1 (multiplex qPCR). Para cada matriz de 96 x 96 chip esperado, pipetear 6 μl de cada ensayo en cada uno señalado de una placa de PCR de 96 pocillos. Por ejemplo, para 5 chips, 30 μl de cada ensayo ocupará un único pozo en la placa de 96 pocillos. Si usa 96 ensayos, cada pocillo de la placa de 96 pocillos contendrá un análisis. Selle la placa con el sello adhesivo.
Nota: Idealmente, los pasos 2.1 y 2.2 se realizan simultáneamente, para evitar múltiples ciclos de congelación y descongelación para ensayos de expresión genética. Todos los genes dentro de la placa de ensayo deben también están presentes en la mezcla de ensayo (paso 2.1). La mezcla de ensayo y la placa de ensayo pueden conservarse a-20 ° C o 4 ° C para uso de corto o largo plazo, respectivamente.
3. células viables de mancha superficial
Nota: La coloración, permeabilización y fijación intracelular no son compatibles con este método como comprometen RNA.
- Preparar las muestras de compensación mediante la adición de cada anticuerpo que aparece en la Tabla de materiales a 40 μl de cuentas de compensación en 2,5 mayor concentración que el utilizado para la tinción celular. Incubar por 20 min a 25 ° C protegido de la luz. Añadir 3 mL de PBS para los granos y centrifugar a 500 x g durante 3 min a 25 ° C. Aspirar el PBS y resuspender los granos en ~ 300 μL de PBS.
- Preparar el clasificador de celular de citometría de flujo para el procesamiento de las muestras: adquirir tubos de compensación, crear matriz de compensación y aplicar matriz a los archivos de adquisición de las muestras experimentales.
- Preparar la mezcla principal de anticuerpos fluorescentes cóctel combinando el volumen apropiado de cada anticuerpo tal como se especifica en la Tabla de materiales, en un tubo de 1,5 mL de color ámbar para todas las muestras a teñir. Vortex y centrifugar el cóctel a 21.000 x g durante 2 min a 25 ° C para el anticuerpo de la pelotilla agregados.
Nota: Anticuerpos utilizados aquí se enumeran en la Tabla de materiales. - Descongelar las células cryopreserved en un baño de agua de 37 ° C durante 2 minutos Añadir 0.5 – 2 mL de la suspensión celular en 12 mL de PBS en un tubo de 15 mL, centrifugar a 500 x g durante 3 min a 25 ° C y aspirar el PBS. Resuspender en 3 mL de PBS y transfiéralo a un tubo de poliestireno de 5 mL. Centrifugar como arriba y aspirar el PBS, dejando unos 20 μl PBS residual.
Nota: La temperatura de tinción se puede adaptar a las temperaturas más cálidas o más frías según sea necesario para aplicaciones específicas mediante la modificación de la valoración de anticuerpos condiciones de tinción en consecuencia (véase el paso 1.2). - Suspender hasta 2 x 107 lavadas las células en 80 μl de anticuerpo cóctel e incubar por 20 min a 25 ° C protegido de la luz. Para muestras superiores a 2 x 107 células, aumentar el volumen de reacción de tinción en consecuencia para mantener < 2 x 107 células/100 μl.
- Lavar las células añadiendo 3 mL de PBS, centrifugar a 500 x g durante 3 min y aspirar el sobrenadante.
- Bien resuspender las células en 300 – 500 μl de PBS y filtro transfiriendo a través de una tapa de filtro 35 de μm nylon celular. Mantener las células en hielo y protegidos de la luz hasta la clase.
4. prepare placas de colección de células, tipo de FACS realizar y generar cDNA
- Pipetear en un tubo estéril libre de DNasa/Rnasa único para combinar los componentes de la mezcla de reacción RT-preamplificador (tabla 3).
Nota: Este paso puede realizarse antes o durante la coloración en el paso 3. La enzima RT puede omitirse aquí para determinar la contribución de la plantilla de la DNA a la señal de la qPCR. - Utilice una pipeta multicanal para dispensar 10 μl de la mezcla de reacción RT-preamplificador en el número de placas de colección de tipo PCR de 96 pocillos. Sellar las placas con la película adhesiva y colocar las placas en los bloques de aluminio de 96 pocillos previamente enfriada.
- Establecer la célula clasificación esquema bloquea en el citómetro de flujo con la adquisición de datos de aproximadamente 20.000 células de la muestra teñida. Asegúrese de que la matriz de compensación se aplica a los datos recogidos. Dibujar puertas y definir el árbol bloquea que identifica a la población de células de interés para ser aisladas para análisis de expresión génica.
Nota: El árbol bloquea utilizado para la recogida de posibles células de vRNA+ SIV se muestra en la figura 3. - Introduzca la configuración de instrumento adecuado para especificar el número y el subconjunto de células a ser clasificado en cada pocillo. Instrucción detallada adicional para clasificar ya sea una serie de diluciones limitantes de la célula o las células se proporcionan en los pasos 5 y 6, respectivamente.
- FACS clasificar las células en placas de colección de PCR de 96 pocillos preparadas. Retire el sello adhesivo antes de la clasificación y reemplácelo con un sello fresco después de la clase.
Nota: Mantenga las placas en los bloques de aluminio previamente refrigerado en todo momento, incluso durante la clase. - Inmediatamente después de la clase, vortex y centrifugar la placa de la colección a 2.000 x g durante 1 min a 4 ° C.
- Thermocycle la placa en una máquina PCR con tapa precalentada utilizando las siguientes condiciones: 50 ° C por 15 min (RT), 95 ° C por 2 min, seguido por 18 ciclos de 95 ° C por 15 s y 60 ° C durante 4 min (pre-amplificación).
- Diluir el cDNA 1:5 por transferencia 5 μL de cDNA en 20 μl de buffer de suspensión de ADN en una nueva placa de PCR de 96 pocillos. CDNA diluido puede ser almacenada a 4 ° C o -20 ° C por tiempo indefinido en este punto. El cDNA está ahora listo para ser utilizado como plantilla para qPCR (pasos 5.2, 7.4).
Nota: Esta dilución asegura que los iniciadores presentes en la reacción de RT-preamplificador no contribuyan a la qPCR aguas abajo.
5. variación A: FACS tipo células en una serie de diluciones limitantes para determinar la frecuencia de vRNA+ células o realizar el Control de calidad Experimental
Nota: Antes de realizar a una especie de unicelulares, pueden ser de utilidad para determinar la frecuencia de las células de interés, por clasificar las células en diluciones seriadas en repetición. Este paso también proporciona valiosos de control de calidad tipo eficiencia, lisis celular, recuperación de RNA y síntesis de cDNA, como se describe en el paso 5.3. Previa determinación de la frecuencia de célula vRNA+ permite la estimación más precisa del número de células que se debe ordenar para alcanzar suficiente tamaño de muestra para análisis de expresión de gene de vRNA adecuadamente alimentado+ celular.
- FACS clasificar las células en las placas de 96 pocillos preparadas como en los pasos 4.1, 4.2 y recoger 1 – 1.000 células por pocillo en múltiples repeticiones.
Nota: El número de pozos de replicar a cada dilución de células suele ser inversamente asociado con la concentración de células por pocillo. Cuando la frecuencia de la célula infectada está muy por debajo de 1%, diluciones de células deben centrarse en 100 – 1.000 células por pocillo. Un mapa de la placa de tipo de ejemplo se proporciona en la figura 1, arriba a la izquierda. Superior a 1.000 células por pocillo debe evitarse debido a aumentos resultantes en el volumen de reacción y la interferencia con la síntesis de cDNA aguas abajo y cuantificación. - Se combinan los reactivos de qPCR en una solución de mezcla principal en la tabla 4. Para un volumen de reacción 25 μl, 22,5 μl de la mezcla principal se combina con 2,5 μl de cDNA diluidos plantilla de paso 3.9. Realizar la qPCR utilizando condiciones de ciclo estándar (p. ej., 94 ° C por 5 min, seguido de 40 ciclos de 94 ° C por 15 s y 60 ° C por 1 min).
Nota: Se recomiendan Singleplex qPCR reacciones usando un instrumento convencional qPCR en tiempo real como un análisis económico preliminar de uno o pocos ensayos para demostrar la célula eficiente clasificación, recuperación de RNA y síntesis de cDNA. También puede utilizarse para calcular la frecuencia de las células de vRNA+ . Reacciones Multiplex qPCR utilizando el Biomark son típicamente más apropiadas para el análisis unicelular a gran escala. - Control de calidad, trama y valores (Et = Ctmax − Ct) versus el número de células ordenadas por pozo en una escala de10 registro y aplicar un análisis de regresión lineal.
Nota: Repeticiones constantes, pendiente de la regresión lineal de 3,3 (± 0,3) y R2 > 0,9 son indicativos de un experimento eficaz. En la figura 4se muestran ejemplos de tipo óptimo y subóptimo, experimentos de RT-preamplificador. - Para determinar la frecuencia de las células de vRNA+ , parcela ordenado por pozo en el eje x (escala logarítmica de10 ) y la fracción de un número de células los pozos positivos de vRNA a cada dilución de células en el eje y. Por ejemplo, vea la figura 1 (inferior izquierda, la distribución de Poisson). Aplicar un modelo de regresión lineal a los datos para determinar el número de células que una célula positiva en promedio, correspondiente al 63,2% de pozos positivos (0.632 en el eje y)21. Convertir a este número de dilución de la célula (intercepto del eje x) frecuencia expresada como un porcentaje. Por ejemplo, una célula de vRNA+ por 48 células equivale a una frecuencia de 2,1%.
6. variación B: clase FACS células para el análisis unicelular
- Siga los pasos 4.1-4.8 y especifique una célula ordenada por bien con función de tipo de índice de citómetro de flujo para crear archivos FCS individuales para cada celda ordenadas, asignada por posición bien.
Nota: Si el número de placas de tipo colección supera el número de termocicladores disponible, ciclismo puede interrumpirse después de la etapa de inactivación de la transcriptasa reversa (95 ° C por 2 min) y cDNA puede almacenarse a 4 ° C hasta termocicladores están disponibles. En este caso, iniciar la amplificación en el primer ciclo de 95 ° C por 15 s. - Opcional: Crear cDNA "pools" de cDNA de lotes definidos por el usuario de células individuales para detectar células de interés utilizando cDNA no diluido residual. Transferencia de 2 μl de puro unicelular previamente amplificado cDNA por pipeta multicanal en una nueva placa de 96 pocillos. Repita por pipeteo 2 μl de cDNA de las células todo adicionales de un grupo designado en el mismo bien. Piscinas de cDNA de los genes de interés (e.g., vRNA) para determinar aquellos que contienen las células positivas utilizando qPCR convencional de la pantalla. Desde agrupación requiere sólo una pequeña alícuota de cDNA de cada célula, el restante 8 μl de cDNA es todavía disponibles para el análisis unicelular.
Nota: Puesta en común de estrategias se recomiendan antes de realizar el análisis de expresión del gene de unicelular en un esfuerzo por reducir el número de células interrogado por qPCR multiplex consumen muchos recursos. Es adecuado para situaciones en que las células de interés (e.g., SIV ARNm +) pueden identificarse mediante un análisis de qPCR preliminar solo-plex. Sencillas estrategias de alto rendimiento para generar cDNA incluyen combinar 2 μl de cDNA sin diluir de una placa de tipo de colección (es decir, 12 todas las células en fila A) las filas o columnas (es decir, 8 todas las células en la columna 1), en un solo pozo en una nueva placa. Para determinar la mejor estrategia de puesta en común, tener en cuenta la frecuencia de las células de interés en el paso 5.4. Por ejemplo, si se espera que el 10% de células positivas, piscinas compuestos de seis muestras de cDNA de unicelular con frecuencia será negativos y así se pueden excluir las células en eso piscina de posteriores análisis unicelular.
7. multiplex qPCR en la plataforma Biomark
Nota: Esta sección puede seguir versión A o B descritos anteriormente. En el estudio descrito en este documento, se aplica exclusivamente en análisis unicelulares.
- Preparar la placa de ensayo qPCR pipeteo 4 μL de cada ensayo de la 2 placa de ensayo (preparada en el paso 2.2) en una nueva placa PCR 96 pocillos que contienen 4 μL del ensayo carga del reactivo en cada pozo. Mantener la placa de ensayo a 4 ° C.
Nota: La placa de ensayo es estable a 4 ° C hasta una semana y a-20 ° C durante un mes. Por lo tanto, puede ser útil preparar suficiente material para varios chips y almacenar adecuadamente. - Verter los líquidos de la línea de control de las jeringas de cebado en las válvulas del dos producto de la viruta. Retire el plástico protector por debajo de la placa. Coloque el chip en un controlador IFC con el lado muescado en la posición A1. En el menú principal, seleccione el guión de "Primer". Ejecute el script.
- Preparar la mezcla de reacción en tiempo real mediante la mezcla de 50 μl de la muestra carga reactivo con 500 μl de PCR Master Mix (Tabla de materiales) para cada chip de microfluidos. Pipetear 4,4 μL en cada pocillo de una placa PCR de 96 pocillos nuevo, de aquí en adelante designada como la "placa de la muestra".
- Pipetee 3.6 μl de cDNA diluidos 1:5 en el paso 4.8 en la placa de la muestra que contiene la mezcla de reacción en tiempo real.
Nota: Si se realizó una selección abajo PCR para detectar raros (e.g., vRNA+) células para análisis posteriores como se explica en el paso 6.2, incluye sólo las células en las piscinas positivo. - Tras la realización de cebado de chip, cargar las entradas de chip por dispensación 5 μl de la placa de ensayo a los correspondientes en el lado de la muesca (ensayo) de la viruta y 5 μl de la placa de la muestra en el correspondiente bien en el otro lado (muestra) de la viruta. Inserte el chip en el controlador IFC y ejecutar el script "Load mix".
- Transferir el chip a la plataforma Biomark para llevar a cabo la qPCR multiplexado. Proceder con la configuración del instrumento y a la programación de qPCR siguiendo instrucciones paso a paso proporciona el software de análisis de PCR en tiempo real y utilizando el protocolo estándar V.1 de Gene expresión (GE) 96.96 con 40 ciclos de PCR. Guarde el archivo ChipRun en una carpeta designada.
Nota: Pueden ejecutar fichas múltiples por día y durante varios días. - Analizar los datos qPCR.
- Abra el Software de análisis PCR en tiempo real. Abra el archivo "ChipRun.bml" desde el "archivo | Menú que se abre".
- Busque "Chip Explorer" y "Chip ejecutar Resumen" en la esquina superior izquierda de la ventana del software. Identificar los tres componentes del Resumen de funcionamiento de Chip: vistas de análisis, instalación de muestra y configuración del Detector.
- Haga clic en "Configuración del Detector". En "Tarea", haga clic en "Nuevo" y seleccione el contenedor tipo "Placa" de SBS y formato contenedor "SBS96". Junto a la "Asignación", haga clic en el... botón y seleccione "M96-ensayo-SBS96.dsp".
- Opcional: Asignar cada Detector (ensayo) un número o nombre en la sección "nombre" de cada bien haga doble clic en la 1st bien. Pasar a la siguiente bien pulsando "F2".
- Haga clic en "Configuración de la muestra". En la "Tarea" al lado de "Mapping", haga clic en el... botón y seleccione "M96-muestra-SBS96.dsp".
- Haga clic en "Vistas de análisis". En la "Tarea" en la pestaña "qPCR", seleccione "Corrección de línea base para Linear (derivado de)" y "Método de umbral de Ct para usuario (detectores)". En la pestaña de "Umbrales de Ct", compruebe el "inicializar con la caja automática". Haga clic en el botón "Analizar".
- En el cuadrante superior derecho de"análisis", haga clic en la segunda pestaña "Tabla de resultados". En el menú desplegable, seleccione "Vista de mapa de calor". Aparece el mapa de calor con datos.
- Opcional: Para asegurar la fluorescencia uniforme de ROX en el chip, seleccionar "Ver imagen" en lugar de "Vista de mapa de calor" desde el mismo menú. En el segundo de la ventana derecha arriba del mapa de calor, seleccione "ROX". En el primero de la ventana derecha, seleccione uno de los ciclos 1-40. Haga clic en el cuarto de la ventana derecha para cambiar a la pantalla en blanco y negro de la fluorescencia de ROX. Aparecerá una imagen que informa salpicaduras, partículas, o defectos en el chip. Si la uniformidad ROX es muy oscurecida, vuelva a ejecutar el chip.
- En el mapa, haga clic en «Umbral» y «Registro gráfico». Ajustar los umbrales de Ct manualmente para cada detector haciendo clic en ensayos (columnas del mapa de calor) y arrastrando el umbral como necesario se cruzan las curvas de amplificación en la fase exponencial. Cuando termine, haga clic en "Analizar".
- Exportar los datos qPCR como un archivo .csv. Importar los datos en una hoja de cálculo o software de análisis estadístico (por ejemplo, JMP) y mapa de los resultados de muestra y análisis de puestos en el chip. Organizar las células en grupos basados en la expresión de genes virales, creando una nueva columna y utilizando una fórmula condicional. En "Analizar", seleccione "Y ajuste por X" y trama de la expresión génica frente a grupo. Aplicar el análisis estadístico.
Nota: Representante unicelular expresión cuantitativa de cuatro especies de SIV ARN se representa en diagramas bivariados en la figura 5A. Expresión de genes cuantitativos host en células SIV ARN+ se muestra en la figura 5B. - Extraer valores de expresión de proteína cuantitativa de los datos de FACS unicelular.
- Abrir el .fcs ordenar los archivos de la FACS (paso 6.1) correspondiente a la placa de 96 pozos utilizando FlowJo versión 9. Con el nombre resaltado, seleccione "plataforma | Puerta número evento | Crear a ordenación indexada puertas". Células individuales aparecerán mostradas por fila.
- Resaltar 96 celdas todos (no filas) y seleccione "espacio de trabajo | De exportación | Seleccione fluors todo compensados". En "Tipo de datos", seleccione "Archivo de FCS", haga clic en "Exportar" y seleccione una carpeta designada.
- Arrastre archivos nuevos de .fcs para las células individuales en un nuevo espacio de trabajo FlowJo. Resaltar todas las celdas, haga clic en "Añadir estadísticas" (el botón de "Σ" en la esquina superior izquierda) "| Significa | Todos los parámetros de fluor".
- Abrir el "Editor de tabla" haciendo clic en el cuarto botón de la izquierda en la esquina superior izquierda. Resalte todos Grad de la primera celda y arrastrar en la ventana del editor de mesa. En la ventana del editor de tabla, haga clic en el botón en la parte superior "Crear y tabla". Esto creará una tabla de 96 celdas y un parámetro numérico para cada fluor.
- Copia de la salida a un software de base de datos (por ejemplo, MS Excel, JMP), o copiar/pegar o haciendo clic en "Guardar y lanzar la aplicación" (cuarto desde el botón izquierdo encima de la mesa).
Nota: Este procedimiento es específico para FlowJo versión 9. FlowJo versión 10 utiliza un procedimiento distinto para importar datos indexados. Indexadas flujo de datos también puede copiar/pegar en JMP directamente desde archivos CSV creado por el clasificador de células.
- Combinar datos de FACS unicelular y qPCR por el número de placa y coloque bien. Realizar análisis gráficos y estadísticos en el gene de unicelular combinado (qPCR) y datos de la expresión (FACS) de proteína.
Nota: Ejemplos de unicelular combinan qPCR y datos FACS se muestran en la figura 6 (sede de perfiles de expresión de la proteína de la superficie de células de macaco rhesus infectados con SIV, empalmado vRNA+ ), figura 7 (expresión de genesCD4 versus superficie Expresión de proteínas de CD4 en células de macaco rhesus empalmado vRNA+ ) y publicado anteriormente20. Para identificar diferencialmente expresados genes en la población de células de interés, análisis unicelular métodos descritos anteriormente son recomendadas20,22,23,24, que representan el la proporción de células positivas para un gen como el valor de la expresión del gen continuo.
Resultados
El flujo de trabajo para el conjunto del Protocolo se muestra en la figura 1. Consta de dos variantes definidas por el número de células ordenadas: cualquier dilución limitante o como solo las células, como se describe en el texto. En la figura 2se muestran ejemplos de análisis de calificación primer-probe 2 veces diluciones seriadas de la RNA. La estrategia de bloquea para identificar posibles células SIV+ se muestra en la figura 3. Un control de calidad acertado, subóptimas y no qPCR para el gen housekeeping GAPDH en las células ordenadas de FACS en la limitación de dilución se muestran en la figura 4. Expresión cuantitativa unicelular de cuatro SIV ARN especies y macaco de la India macaque genes que se expresan diferencialmente en células infectadas se muestran en la figura 5. Representante bivariado, histograma y diagramas de dispersión muestran el perfil de expresión de proteína de la superficie del SIV ARN+ CD4+ T células medición por citometría de flujo en la figura 6. El diagrama de burbuja trivariate (figura 7) muestra la relación entre proteína CD3 o CD4 de la superficie, CD3 o CD4 mRNA y cuantitativa gen viral (tat/rev) expresión en las células.

Figura 1 : Esquema de flujo de trabajo experimental ilustra tres componentes principales: flujo cytometric tipo, transcripción inversa además de pre-amplificación basadas en PCR de cDNA (RT, pre-amp) y qPCR. El tipo se puede realizar ya sea una dilución limitante (A, Telón de fondo verde) o como las células (B, fondo naranja). Inmediatamente después de la clase FACS, las células son sometidas a lisis y RNA es inversa transcrito a cDNA y pre amplificado (RT, PCR pre-amp) para preparar la plantilla de la qPCR. Tipo de dilución limitante determinar la frecuencia de células positivas para el ARN viral usando la distribución de Poisson estadísticas así como eficacia experimental y recuperación de la muestra. La cabeza de la flecha verde indica el número de células ordenadas por bien que contienen una célula positiva para un gen viral (correspondiente a una probabilidad de 63.2% de estos pozos es gene positivo), que se convierte en una frecuencia de celda. Las estimaciones de frecuencia pueden utilizarse para informar el número de células recogidas en una especie posterior (B). Indexadas unicelular FACS clasificación depósitos uno de células por pocillo y genera archivos de datos para cada celda anotado por bien puesto dentro de la placa de 96 pocillos. Sola célula qPCR se realiza en múltiplex para 96 genes simultáneamente. La combinación de la proteína de la superficie (FACS) y la expresión de mRNA permite perfiles de células individuales (columna derecha). Una qPCR opcional para un gene viral puede realizarse entre la amplificación de la PCR y qPCR multiplexado (B, intermedia) a células individuales de la pantalla o piscinas de cDNA sola célula selecciona células virales de RNA+ o piscinas para análisis de qPCR multiplex. El mapa ilustra la expresión génica (valor de Ct) para 96 ensayos (columnas) y 96 células (filas). Haga clic aquí para ver una versión más grande de esta figura.

Figura 2: Se muestran datos de qPCR representante de cartilla calificación experimentos exitosa (izquierda y centro) y ensayos (derecha) comercialmente disponibles (CD6, SIV tat/rev, y TLR3, respectivamente). Para CD6 y TLR3, ARN fue extraído de 10 macaco rhesus FACS-clasificados de6 PBMC CD4+ las células de T, utilizando un kit comercial. Ocho repeticiones de una serie de doce-punto ARN doble dilución (0.023-48 ng ARN, correspondientes a 1.2 – 2.400 equivalentes de celular asumiendo 20 pg de RNA por CD4+ T-cell) fueron sometidos a RT-preamplificador y qPCR. Para SIV tat/rev, se extrajo RNA de macaque del macaco de PBMCs infectados en vitro con SIVmac239. Se preparan diluciones de RNA expansión equivalentes de RNA de las células 6 – 12.000. Y valores (40-Ct), que aumentan con la expresión génica, se representan frente al número estimado de células. Serie de diluciones que R2 > 0.97 y pendiente de ±0. 3 3.32 indican calificación primer éxito. Haga clic aquí para ver una versión más grande de esta figura.
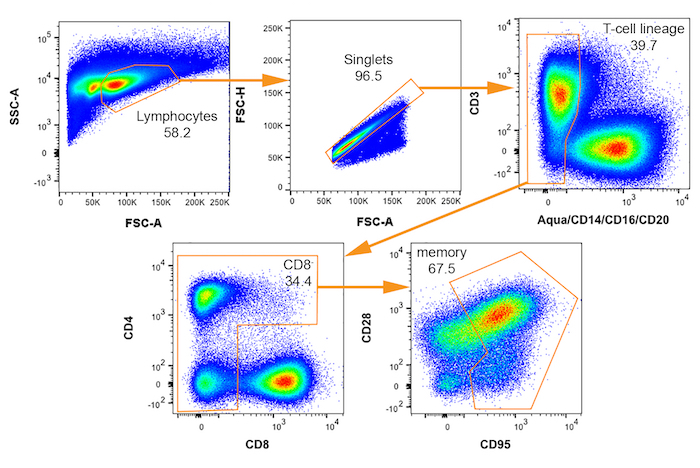
Figura 3: Bivariado FACS parcelas con gating esquema para el aislamiento de las células de macaque del macaco de la India potencialmente infectados por SIV. Puertas secuenciales de memoria (CD95 +) CD4+ T células aparecen desde la parte superior izquierda a inferior derecha con cada nombre de población indicada. Por ciento de la trama de padre que cae dentro de cada puerta está indicado. Haga clic aquí para ver una versión más grande de esta figura.

Figura 4 : Validación experimental representativa de qPCR realizados en células ordenadas de FACS para limitar uso de dilución GAPDH gen housekeeping de tres experimentos independientes: éxito (izquierda), subóptima (medio) y no (derecha). Repeticiones de 10-100 células por pocillo deben abarcar Ets no más de 2, 300 – 1.000 células pozos y 1 Et. La pendiente de la regresión lineal debe ser 3.32 ± 0.3, R2 > 0.95. Incumplimiento de estas especificaciones indica dificultades técnicas en los pasos 1, 2, 3 o una combinación de los mismos. Haga clic aquí para ver una versión más grande de esta figura.

Figura 5 : Unicelular cuantitativa viral y host expresión génica en macacos rhesus FACS-clasificado CD4+ Las células de T de infección de nodo de linfa mesentérico 10 días post-SIVmac251. Expresión de genes virales (A) bivariado parcelas de multiplicar-empalmado (tat/rev) y solo empalmado (env) SIV mRNA por solo células (izquierda). TAT/rev+env– (células luz verde a lo largo del eje x) expresan menos copias de tat/rev RNA de las células env+ (verde oscuro), consistentes con un temprano etapa de la infección y antes de la estabilización mediada por la proteína Rev y exportación nuclear de vRNAs parcialmente empalmado como env. Las células que no expresan ARN viral empalmado se representan en gris (sin ARN viral), marrón (gag+ y l+) o tan ( gag+ o l+). Unspliced (gag+) y total expresión del mRNA SIV (LTR+) se muestra de las células del mismo (derecha). Gran abundancia de unspliced mordaza RNA en tatrev+env+ células es consistente con la última infección productiva etapa durante la cual se expresa abundante RNA genómico para embalaje en ciernes viriones. (B) violín parcelas de genes de macaque del macaco de la India diferencialmente expresados en al menos un subconjunto de células infectadas por el SIV, en comparación con las células no infectadas (gris). Análisis estadístico se realizó como se describió anteriormente20,22,23,24. Asterisco indica false discovery rate < 10% en comparaciones de prueba de cociente de probabilidad combinado en relación con las células no infectadas. La línea conecta valores medios a través de grupos de células para cada gen. Esta figura ha sido modificada desde Bolton et al. 20 Haga clic aquí para ver una versión más grande de esta figura.

Figura 6: Representativo anfitrión perfiles de expresión de proteína de la superficie de las células ordenadas de FACS de macaco de la India macaque nodo de linfa mesentérico 10 días post-SIVmac251 una infección. Diagrama de dispersión (A) muestra de expresión de proteína de la superficie de CD3, CD4 y ICOS en no infectadas (gris), mordaza+tat/rev– (marrón) y célulastat/rev+ mordaza+(verde). Intensidad de la fluorescencia se traza para cada celda (dot). Diagramas de caja aislados representan el rango intercuartil (IQR) y mediana (caja), los puntos más lejos dentro de 1,5 x IQR de las caja (bigotes) y el potencial outliers (puntos desconectados). Barras horizontales en la parte superior indican diferencias significativas (p < 0.05, prueba de rango de Wilcoxon no paramétrica). (B) Mostrar bifactorial e histograma de CD3 y CD4 proteína la expresión superficial de las células que se muestra en (A). Trama de puntos (izquierda) indica porcentajes de cada población celular dentro de un cuadrante. Histogramas de CD3 y CD4 (medio derecha) representan downregulation de la proteína de la superficie entretat/rev– mordaza+tat/rev+ células en relación con la no infectada y mordaza+. (C) doce representante tat/rev+ las células de (A – B) se muestran a través de tres parcelas bivariados para la superficial expresión de CD3/CD4 (izquierda), CD69/CD38 (medio) y ICOS/HLA-DR (derecha). Haga clic aquí para ver una versión más grande de esta figura.
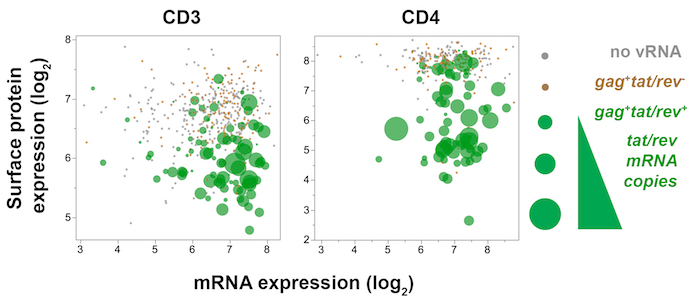
Figura 7 : Trama Trivariate mostrando unicelular gen viral (tat/rev), gene del anfitrión (CD3 o CD4) y anfitrión superficial (CD3 o CD4) expresión de la proteína en memoria infectados con SIV CD4+ T de las células del nodo de linfa mesentérico 10 días infección post-SIVmac251. Expresión de la proteína CD4 (fluorescencia) se traza contra CD4 mRNA (qPCR), mientras que la cantidad de tat/rev expresadas por cada celular se refleja en el tamaño de punto. En tat/rev+ las células (verde), disminución de superficie CD4 y CD3 proteínas expresión sostenidas transcripciones CD4 y CD3 , respectivamente, indicando que la expresión de la proteína de la superficie está modulada aguas abajo de la expresión génica. Haga clic aquí para ver una versión más grande de esta figura.
Tabla 1: Cebadores y sondas para la detección de ácidos nucleicos SIV. Cuando dos secuencias están indicadas para una primer o sonda, se utilizaron cantidades equimolares de ambas secuencias. Haga clic aquí para descargar este archivo.
Tabla 2: un panel de 96-gen utilizado para la cuantificación de amplicones Biomark instrumento. Cuatro ensayos SIV se indican con fondo azul. Haga clic aquí para descargar este archivo.
Tabla 3: Mezcla de reacción utilizado para transcripción reversa y amplificación previa. Haga clic aquí para descargar este archivo.
Tabla 4: qPCR de mezcla de reacción para PCR en tiempo real se realiza en un instrumento de Quant Studio 6. Haga clic aquí para descargar este archivo.
Archivo codificación complementario 1. Instrucciones para la calificación de ensayos de expresión genética. Haga clic aquí para descargar este archivo.
Archivo de codificación complementaria 2: plantilla de mapa muestra en JMP. Haga clic aquí para descargar este archivo.
Archivo de codificación complementaria 3: plantilla de mapa sonda en JMP. Haga clic aquí para descargar este archivo.
Archivo de codificación adicional 4: guión de Primer análisis de JMP. Haga clic aquí para descargar este archivo.
Archivo codificación complementario 5. Guión de análisis por trozos de JMP. Haga clic aquí para descargar este archivo.
Discusión
El protocolo aquí descrito, denominado tSCEPTRE, integra la cuantificación de la proteína unicelular de la superficie por citometría de flujo multiparamétrico con la expresión de mRNA de unicelular cuantitativa por RT-qPCR altamente multiplexado. La Unión de estas dos tecnologías permite instantáneas de alto contenido de combinado transcripcionales y perfil proteico de las células en un formato de alto rendimiento. Utilice el método para identificar células hasta ahora esquivos infectadas con SIV en vivoy describir host diferencialmente expresados genes y proteínas. El protocolo puede ser adaptado para el estudio de cualquier población de la célula de interés distinguibles por la expresión de la proteína superficial, mRNA o una combinación de éstos. Los métodos descritos dependen precisa sola célula clasificación y grabación mediante citometría de flujo de datos con reactivos comercialmente disponibles qPCR y había multiplexado instrumentación de PCR en tiempo real. La salida es evaluación cuantitativa sensible de sola célula proteína y gene expresión datos combinados.
Otros enfoques que enlazan a la proteína y expresión genética en las células han sido descritas1,25,26,27. Indexadas de FACS clasificación seguida RNAseq, aplicado con éxito a la caracterización de células madre hematopoyéticas, representa un particularmente prometedor enfoque28. Sin embargo, aunque RNAseq tiene varias ventajas sobre el análisis de expresión del gene específicos, a saber: Análisis de transcriptoma completo imparcial, es sujeto a un mayor comparaciones múltiples costos estadística y puede ser menos sensible para la cuantificación de copia bajo transcripciones. Por otra parte, en la actualidad no es económicamente factible realizar RNAseq en miles de células en busca de células infectadas presentes en frecuencias < 1% (p. ej., VIH, SIV). Otras tecnologías emergentes unicelular pretenden generar una sola lectura, es decir, por FACS o PCR, para la detección de proteínas y ácidos nucleicos en la célula misma. Estos incluyen la detección de proteínas por anticuerpos fluorescentes y mRNA por sondas de oligonucleótidos fluorescentes seguido por FACS análisis (mRNA-flujo-FISH)14,15,29,30. Por otra parte, las proteínas pueden ser detectadas por pares conjugados de anticuerpos de oligonucleótidos que son detectados por qPCR en paralelo con revés transcrito mRNA31,32,33,34 . Estos enfoques "híbrido" proporcionan simultánea de ácido nucleico y las mediciones de proteína unicelular resolución similares a tSCEPTRE, pero están limitados donde están o no cuantitativos (mRNA flujo-peces) o puede requerir modificado para requisitos particulares anticuerpo o mRNA puntas de prueba. Nuestro enfoque tSCEPTRE es cuantitativa para las mediciones de proteína y de mRNA y todos los reactivos están comercialmente disponibles, con la posible excepción de ensayos específicos del patógeno.
Especial atención se debe aplicar a varios pasos en el protocolo antes del análisis de las muestras experimentales. Primero, citometría de flujo alto parámetro requiere optimización cuidadosa de un panel de anticuerpos fluorescentes para sensibles de la detección y resolución de35. Cada anticuerpo debe titularse individualmente para determinar la concentración que logra una óptima separación y tinción de fondo mínimo, seguido de la evaluación de la coloración cuando todos los conjugados de anticuerpos se usan en combinación. Segunda determinación experimental del ensayo qPCR correspondiente gene expresión para medir cada gen de interés es esencial. Múltiples ensayos disponibles en el mercado están típicamente disponibles para cada gen, pero en nuestra experiencia, muchos no son cuantitativas en el nivel unicelular6. Por lo tanto, es importante calificar todo propuestos ensayos en serie diluido antes de RNA para expresión génica cuantitativa. Optimización de estos reactivos es mucho tiempo, pero el valor de paneles confiables de los conjugados de anticuerpos y ensayos de expresión genética que permite la detección sensible de todas las moléculas de interés justifica la inversión de tiempo. Además, debe darse prioridad a ensayos disponibles comercialmente que contienen sondas que abarcan las uniones exón-exón (señalados con sufijo "m1") para mejorar la especificidad de ARNm, aunque suplente ensayos capaces de detectar el ADN genómico (sufijo "s1" o "g1" ) se consideran poco probable que influir en los resultados de expresión génica debido a copias cromosómicas equivalente en cada célula. Preservación de ARN antes de RT es fundamental y se logra mantener las muestras refrigeradas, como se señala en el protocolo. Del mismo modo, la duración de la clasificación de FACS y el intervalo entre clasificación y RT-preamplificador debe mantenerse al mínimo. Limitación dilución tanto unicelulares clasificación, cDNA puede ser primero analizado por qPCR convencional para evaluar recuperación de RNA de altamente expresada housekeeping genes. Si la expresión observada no es uniforme entre repeticiones de igual número de células, o la pendiente del ajuste lineal para limitar la validación falla de diluciones, solución de problemas debe realizarse procedimientos de clasificación y aguas abajo de la célula.
Potenciales limitaciones de la aproximación descrita aquí incluyen (1) detección de la DNA además de ARN, particularmente para ensayos no específicos para uniones de empalme del mRNA y (2) limitado número de proteínas de la superficie y genes medidos. Sin embargo, detección de ADN es probable contribuir substancialmente al análisis de expresión génica diferencial host por la razón anterior. Por otra parte, medimos directamente la medida en que ADN contribuye a la señal de qPCR en este protocolo por dos enfoques: a) excluyendo de la transcriptasa reversa y b) modificando el protocolo de lisis celular para mejorar la lisis de la membrana nuclear. En la ausencia de la transcriptasa inversa, ambos empalmados y unspliced genes virales se detectaron todavía, pero con significativamente menos frecuencia que en presencia de la transcriptasa reversa (~ reducción de 6-fold y 2 veces, respectivamente)20. Por lo tanto, los ensayos de expresión génica viral sobrestiman la cantidad de RNA viral presente en una célula debido a la presencia de ADN viral asociada a la célula. Atribuimos este resultado a productos de RT citoplásmicos generados durante la infección de célula anfitrión, sabida que se presenta para el empalmado y unspliced SIV y VIH ARN origina viriones entrante36. Así, puede ser recomendable para dedicar una parte de los experimentos en condiciones carentes de la transcriptasa inversa para cuantificar la plantilla derivados de ADN para algunas aplicaciones. Cabe señalar que no se pueden distinguir unspliced SIV/VIH ARN derivados de virus entrante de novo de sintetizado el ARN viral. Segundo incorporado a un paso de lisis nuclear en el protocolo de RT-preamplificador y observado un incremento exponencial en copias de DNA del SIV (Alu-LTR) integrados (Figura S3 de Bolton et al.) 20. extracción de ADN genómico por lo tanto es poco probable que contribuyen sustancialmente a la plantilla de ácido nucleico extraída por el método de lisis celular utilizado aquí. De la nota, la adición de un paso de lisis nuclear puede ser útiles estudios en futuro unicelular intentando investigar SIV u otros virus DNA positivos células, incluyendo células latente infectadas.
El número de proteínas de la superficie analizada se basa en la capacidad de la clasificadora de celular de citometría de flujo. Instrumentos disponibles en el mercado actuales no excedan 30 parámetros. Estudios futuros utilizando citometría de flujo más avanzado ampliará aún más la proteína perfiles de capacidad de este enfoque. El número de las transcripciones también puede extenderse más allá de 96, reactivos apoyo proporcionados (por ejemplo, cartillas de mayor concentración), equipos e instrumental. En última instancia, unicelular tecnologías emergentes que combinan análisis del proteoma (espectrometría), transcriptoma (RNAseq) y genoma (DNAseq) sustituirá a los enfoques dirigidos por descubrimiento investigación31,37, 38 , 39. sin embargo, qPCR dirigida probablemente seguirá siendo una valiosa herramienta para la validación de estos enfoques "ómicas" como un estándar de oro para análisis de expresión cuantitativa.
Análisis combinado de proteínas y la transcripción de tSCEPTRE es una herramienta poderosa para investigar raros o difíciles de identificar células tales como ésos albergar patógenos, contienen oncogenes o lo contrario, exhibiendo un fenotipo aberrante. Nuevos marcadores para células SIV/VIH transcripcionalmente activas pueden ser identificados en esta forma, así como el descubrimiento de nuevos mecanismos implicados en la patogenia de estas infecciones virales. Identificación de las células infectadas latente requerirá el desarrollo de un protocolo para evaluar la DNA viral integrada en el genoma del anfitrión. De nota, la frecuencia de las células infectada VIH/SIV es considerablemente menor en infección crónica viremic o tratada, que presentará un desafío práctico en estudio células infectadas derivado de estos ajustes. Nuestro enfoque sienta las bases para la evaluación de un mecanismo previamente insuperable: la de regulación postranscripcional en el nivel unicelular, y tiene amplia aplicabilidad huésped-patógeno, así como procesos celulares más general.
Divulgaciones
Este trabajo fue apoyado por un acuerdo cooperativo (W81XWH-07-2-0067) entre el Henry M. Jackson Foundation para el avance de la medicina militar, Inc. y el Departamento estadounidense de defensa (DOD). Las opiniones expresadas son las de los autores y no deben ser interpretadas para representar las posiciones del ejército de Estados Unidos o el Departamento de defensa. Investigación se llevó a cabo bajo un protocolo aprobado uso de animales en un centro de AAALACi acreditado en conformidad con la ley de Bienestar Animal y otros estatutos federales y reglamentos relativos a los animales y experimentos con animales y se adhiere a los principios indicado en la guía para el cuidado y uso de animales de laboratorio, publicación del NRC, edición 2011.
Agradecimientos
Los autores desean agradecer a la base de citometría de flujo de NIAID VRC y las instalaciones de la base de Cytometry del flujo MHRP para mantenimiento y operación de FACS instrumentos y equipos de clasificación; María Montero, Vishakha Sharma, Kaimei Song expertos asistencia técnica; Michael Piatak, Jr. (fallecido) para obtener ayuda con diseño de ensayo qPCR SIV; y Brandon Keele y Matthew Scarlotta para SIV aislar secuencias. Las opiniones expresadas son las de los autores y no deben ser interpretadas para representar las posiciones del ejército de Estados Unidos o el Departamento de defensa. Investigación se llevó a cabo bajo un protocolo aprobado uso de animales en una instalación AAALAC acreditado conforme a la ley de Bienestar Animal y otros estatutos federales y reglamentos relativos a los animales y experimentos con animales y se adhiere a los principios indicado en la guía para el cuidado y uso de animales de laboratorio, publicación del NRC, edición 2011.
Materiales
| Name | Company | Catalog Number | Comments |
| RNA extraction and PCR reagents and consumables | |||
| Genemate 96-Well Semi-Skirted PCR Plate | BioExpress/VWR | T-3060-1 | |
| Adhesive PCR Plate Seals | ThermoFisher | AB0558 | |
| Armadillo 384-well PCR Plate | ThermoFisher | AB2384 | |
| MicroAmp Optical Adhesive Film | Applied Biosystems/ThermoFisher | 4311971 | |
| DEPC Water | Quality Biological | 351-068-101 | |
| Glass Distilled Water | Teknova | W3345 | |
| Superscript III Platinum One-Step qRT-PCR Kit | Invitrogen/ThermoFisher | 11732088 | |
| SUPERase-In Rnase Inhibitor | Invitrogen/ThermoFisher | AM2696 | |
| Platinum Taq | Invitrogen/ThermoFisher | 10966034 | |
| dNTP Mix | Invitrogen/ThermoFisher | 18427088 | |
| ROX Reference Dye (if separate from kit) | Invitrogen/ThermoFisher | 12223012 | |
| DNA Suspension Buffer | Teknova | T0223 | |
| RNAqueous kit | Invitrogen/ThermoFisher | AM1931 | |
| TaqMan gene expression assays not listed in Table 2 | |||
| CD6 | Applied Biosystems/ThermoFisher | Hs00198752_m1 | |
| TLR3 | Applied Biosystems/ThermoFisher | Hs1551078_m1 | |
| Biomark reagents | |||
| Control Line Fluid Kit | Fluidigm | 89000021 | |
| TaqMan Universal PCR Mix | Applied Biosystems/ThermoFisher | 4304437 | |
| Assay Loading Reagent | Fluidigm | 85000736 | |
| Sample Loading Reagent | Fluidigm | 85000735 | |
| Dynamic Array 96.96 (chip) | Fluidigm | BMK-M-96.96 | |
| FACS reagents | |||
| SPHERO COMPtrol Goat anti-mouse (lambda) | Spherotech Inc. | CMIgP-30-5H | |
| CompBeads Anti-Mouse Ig,k | BD Biosciences | 51-90-9001229 | |
| 5 ml Polystyrene tube with strainer cap | FALCON | 352235 | |
| Aqua Live/Dead stain | Invitrogen/ThermoFisher | L34976 | dilute 1:800 |
| Mouse Anti-Human CD3 BV650 clone SP34-2 | BD Biosciences | 563916 | dilute 1:40 |
| Mouse Anti-Human CD4 BV786 clone L200 | BD Biosciences | 563914 | dilute 1:20 |
| Mouse Anti-Human CD8 BUV496 clone RPA-T8 | BD Biosciences | 564804 | dilute 1:10 |
| Mouse Anti-Human CD28 BV711 clone CD28.2 | Biolegend | 302948 | dilute 1:20 |
| Mouse Anti-Human CD95 BUV737 clone DX2 | BD Biosciences | 564710 | dilute 1:10 |
| Mouse Anti-Human CD14 BV510 clone M5E2 | Biolegend | 301842 | dilute 1:83 |
| Mouse Anti-Human CD16 BV510 clone 3G8 | Biolegend | 302048 | dilute 1:167 |
| Mouse Anti-Human CD20 BV510 clone 2H7 | Biolegend | 302340 | dilute 1:37 |
| Anti-CD38-R PE clone OKT10 | NHP reagent recource | N/A | dilute 1:100 |
| Mouse Anti-Human CD69 BUV395 clone FN50 | BD Biosciences | 564364 | dilute 1:10 |
| Mouse Anti-Human HLA-DR APC-H7 clone G46-6 | BD Biosciences | 561358 | dilute 1:20 |
| Mouse Anti-Human ICOS Alexa Fluor 700 clone C398.4A | Biolegend | 313528 | dilute 1:80 |
| Instruments | |||
| BioPrptect Containment Enclosure | Baker | ||
| BD FACS Aria | BD Biosciences | ||
| ProtoFlex Dual 96-well PCR system | Applied Biosystems/ThermoFisher | 4484076 | |
| Quant Studio 6 qPCR instrument | Applied Biosystems/ThermoFisher | 4485694 | |
| IFC controller HX | Fluidigm | IFC-HX | |
| Biomark HD | Fluidigm | BMKHD-BMKHD |
Referencias
- Macaulay, I. C., Ponting, C. P., Voet, T. Single-Cell Multiomics: Multiple Measurements from Single Cells. Trends Genet. 33 (2), 155-168 (2017).
- Pasternak, A. O., Lukashov, V. V., Berkhout, B. Cell-associated HIV RNA: a dynamic biomarker of viral persistence. Retrovirology. 10, 41(2013).
- Mailler, E., et al. The Life-Cycle of the HIV-1 Gag-RNA Complex. Viruses. 8 (9), (2016).
- Varmus, H. Retroviruses. Science. 240 (4858), 1427-1435 (1988).
- Frankel, A. D., Young, J. A. HIV-1: fifteen proteins and an RNA. Annu Rev Biochem. 67, 1-25 (1998).
- Dominguez, M. H., et al. Highly multiplexed quantitation of gene expression on single cells. J Immunol Methods. 391 (1-2), 133-145 (2013).
- Tokarev, A., Guatelli, J. Misdirection of membrane trafficking by HIV-1 Vpu and Nef: Keys to viral virulence and persistence. Cell Logist. 1 (3), 90-102 (2011).
- Malim, M. H., Bieniasz, P. D. HIV Restriction Factors and Mechanisms of Evasion. Cold Spring Harb Perspect Med. 2 (5), a006940(2012).
- Sugden, S. M., Bego, M. G., Pham, T. N., Cohen, E. A. Remodeling of the Host Cell Plasma Membrane by HIV-1 Nef and Vpu: A Strategy to Ensure Viral Fitness and Persistence. Viruses. 8 (3), 67(2016).
- Simon, V., Bloch, N., Landau, N. R. Intrinsic host restrictions to HIV-1 and mechanisms of viral escape. Nat Immunol. 16 (6), 546-553 (2015).
- Kirchhoff, F. Immune evasion and counteraction of restriction factors by HIV-1 and other primate lentiviruses. Cell Host Microbe. 8 (1), 55-67 (2010).
- Wigzell, H. Immunopathogenesis of HIV infection. J Acquir Immune Defic Syndr. 1 (6), 559-565 (1988).
- Reynolds, M. R., et al. Ex vivo analysis of SIV-infected cells by flow cytometry. Cytometry A. 77 (11), 1059-1066 (2010).
- Baxter, A. E., et al. Single-Cell Characterization of Viral Translation-Competent Reservoirs in HIV-Infected Individuals. Cell Host Microbe. 20 (3), 368-380 (2016).
- Grau-Exposito, J., et al. A Novel Single-Cell FISH-Flow Assay Identifies Effector Memory CD4+ T cells as a Major Niche for HIV-1 Transcription in HIV-Infected Patients. MBio. 8 (4), (2017).
- Eriksson, S., et al. Comparative analysis of measures of viral reservoirs in HIV-1 eradication studies. PLoS Pathog. 9 (2), e1003174(2013).
- Finzi, D., et al. Identification of a reservoir for HIV-1 in patients on highly active antiretroviral therapy. Science. 278 (5341), 1295-1300 (1997).
- Chomont, N., et al. HIV reservoir size and persistence are driven by T cell survival and homeostatic proliferation. Nat Med. 15 (8), 893-900 (2009).
- DeMaster, L. K., et al. A Subset of CD4/CD8 Double-Negative T Cells Expresses HIV Proteins in Patients on Antiretroviral Therapy. J Virol. 90 (5), 2165-2179 (2015).
- Bolton, D. L., et al. Combined single-cell quantitation of host and SIV genes and proteins ex vivo reveals host-pathogen interactions in individual cells. PLoS Pathog. 13 (6), e1006445(2017).
- Quintans, J., Lefkovits, I. Precursor cells specific to sheep red cells in nude mice. Estimation of frequency in the microculture system. Eur J Immunol. 3 (7), 392-397 (1973).
- Finak, G., et al. MAST: a flexible statistical framework for assessing transcriptional changes and characterizing heterogeneity in single-cell RNA sequencing data. Genome Biol. 16, 278(2015).
- McDavid, A., et al. Modeling bi-modality improves characterization of cell cycle on gene expression in single cells. PLoS Comput Biol. 10 (7), e1003696(2014).
- McDavid, A., Finak, G., Gottardo, R. The contribution of cell cycle to heterogeneity in single-cell RNA-seq data. Nat Biotechnol. 34 (6), 591-593 (2016).
- Kok, Y. L., Ciuffi, A., Metzner, K. J. Unravelling HIV-1 Latency, One Cell at a Time. Trends Microbiol. , (2017).
- Rato, S., Golumbeanu, M., Telenti, A., Ciuffi, A. Exploring viral infection using single-cell sequencing. Virus Res. 239, 55-68 (2017).
- Wagner, A., Regev, A., Yosef, N. Revealing the vectors of cellular identity with single-cell genomics. Nat Biotechnol. 34 (11), 1145-1160 (2016).
- Nestorowa, S., et al. A single-cell resolution map of mouse hematopoietic stem and progenitor cell differentiation. Blood. 128 (8), e20-e31 (2016).
- Soh, K. T., et al. Simultaneous, Single-Cell Measurement of Messenger RNA, Cell Surface Proteins, and Intracellular Proteins. Curr Protoc Cytom. 75, 41-47 (2016).
- Kochan, J., Wawro, M., Kasza, A. Simultaneous detection of mRNA and protein in single cells using immunofluorescence-combined single-molecule RNA FISH. Biotechniques. 59 (4), 209-212 (2015).
- Stahlberg, A., Thomsen, C., Ruff, D., Aman, P. Quantitative PCR analysis of DNA, RNAs, and proteins in the same single cell. Clin Chem. 58 (12), 1682-1691 (2012).
- Assarsson, E., et al. Homogenous 96-plex PEA immunoassay exhibiting high sensitivity, specificity, and excellent scalability. PLoS One. 9 (4), e95192(2014).
- Fredriksson, S., et al. Protein detection using proximity-dependent DNA ligation assays. Nat Biotechnol. 20 (5), 473-477 (2002).
- Genshaft, A. S., et al. targeted profiling of single-cell proteomes and transcriptomes in a single reaction. Genome Biol. 17 (1), 188(2016).
- Mahnke, Y. D., Roederer, M. Optimizing a multicolor immunophenotyping assay. Clin Lab Med. 27 (3), 469-485 (2007).
- Liang, C., Hu, J., Russell, R. S., Kameoka, M., Wainberg, M. A. Spliced human immunodeficiency virus type 1 RNA is reverse transcribed into cDNA within infected cells. AIDS Res Hum Retroviruses. 20 (2), 203-211 (2004).
- Stegle, O., Teichmann, S. A., Marioni, J. C. Computational and analytical challenges in single-cell transcriptomics. Nat Rev Genet. 16 (3), 133-145 (2015).
- Wu, M., Singh, A. K. Single-cell protein analysis. Curr Opin Biotechnol. 23 (1), 83-88 (2012).
- Haque, A., Engel, J., Teichmann, S. A., Lonnberg, T. A practical guide to single-cell RNA-sequencing for biomedical research and clinical applications. Genome Med. 9 (1), 75(2017).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados