Method Article
MRNA およびサル免疫不全ウイルス感染 cd4 表面タンパク質発現の単一細胞定量+ T 細胞孤立したアカゲザルから
要約
96 遺伝子の発現を量的に方法論が説明し、単一セルがex vivo、差動の同定を可能にする 18 の表面タンパク質は感染していない細胞を基準にしてウイルスに感染した細胞の遺伝子そして蛋白質を表明しました。+ T 研究 SIV 感染 CD4 にアプローチを適用アカゲザルから分離した細胞。
要約
単一細胞解析は、細胞の異種個体集団を解剖するための重要なツールです。同定や希少細胞の分離は困難することができます。このような課題を克服するためにインデックス付き cytometry 流れの手法の組み合わせと高スループット多重量的なポリメラーゼの連鎖反応 (qPCR) を開発しました。目的は、識別し、特徴付けるサル免疫不全ウイルス (SIV)-感染細胞アカゲザル内に存在します。多次元のプロファイルを作成するホスト遺伝子および蛋白測定と組み合わせてウイルスの遺伝子によって識別がウイルス感染細胞表面タンパク質蛍光活性化セル (FACS) を並べ替えると qPCR による mRNA の定量を.アプローチ、対象の単一細胞タンパク質転写評価または tSCEPTRE と呼びます。メソッドを実行するには、実行可能なセルは細胞サブセットおよび/または下流の表現型解析の FACS 分離に使用される表面マーカーの特定蛍光抗体で染色が。単一のセルは即時換散、多重の逆のトランスクリプション (RT)、pcr 前、最大 96 の転写産物の高スループット qPCR の順で並べ替えられます。FACS 測定選別の時に記録、その後結合蛋白質と転写プロファイルを作成するよく位置によって遺伝子発現データにリンクします。Ex vivoセル直接 SIV 感染を研究するのには、セルが複数のウイルス RNA 種の qPCR の検出によって識別されました。それぞれの量とウイルスの転写産物の組み合わせ (例えば、非生産性対生産的な) ウイルスのライフ サイクルの異なる段階に細胞を分類するためのフレームワークを提供します。また、SIV+細胞の tSCEPTRE に比べて感染していない細胞発現宿主遺伝子とタンパク質を評価する同じ標本から分離したしていた。分析では、感染細胞だけでなく体内の SIV を介した転写後遺伝子発現制御単一セルの解像度の以前功績のウイルス RNA 式異質性を明らかにしました。TSCEPTRE メソッドは表面蛋白質のマーカー、ホストまたは病原体の遺伝子、またはこれらの組み合わせの式による識別に従う任意のセルの人口の分析のため。
概要
多くの細胞内の病原体は頻繁にホスト細胞生物学を変更すること、または伝播の彼らのチャンスを最大化するために宿主細胞の非常に特定の集団を対象と、レプリケートする宿主の細胞機構に依存します。その結果、細胞生物学的プロセスがホストの全体的な健康に有害な影響をよく破壊され。ウイルス、複製する、ホスト細胞間の相互作用を理解することは感染を防ぐために改良された治療法と戦略の開発に役立つ可能性があります病気のメカニズムを解明します。宿主-病原体相互作用の研究を可能にする直接の分析ツールこの年末に向けて不可欠です。単一細胞解析は、明確に特定の遺伝子または感染状態1に細胞の表現型を属性する唯一の手段を提供します。たとえば、病原性感染症はよくホスト細胞の直接と間接の両方の変化を誘発します。したがって、よう一般化感染していない相手からの感染細胞は直接感染する二次的影響属性ホスト セルの変更に必要な区別する炎症。また、SIV とヒト免疫不全ウイルス (HIV) のような多くの病原体を早く、遅く、ホスト細胞への感染のように複数の段階を進行または潜在的なそれぞれの特徴付けることができる個別の遺伝子、タンパク質発現プロファイル2で,3,4,5. 細胞の混合物の分析がこの不均質性6をキャプチャに失敗します。対照的に、両方のウイルスの発現を定量化することができる単一細胞解析の高多重し、宿主遺伝子が感染の段階でバリエーションを含む感染特異的細胞摂動を解決する手段を提供します。さらに、ホスト病原体の相互作用を生理学的分析関連する設定は、感染した生物で発生するイベントを識別するために重要です。したがって前のヴィヴォ、生体内で最高の可能性が高いキャプチャ直接適用できるメソッドを処理します。
SIV と HIV は、CD4 をターゲット+ T 細胞レセプターの抗原提示分子感染を確立し、免疫監視7、8を避けるためとホスト抗ウイルス「制限」の要因に対抗します。 9,10,11。治療せずに、感染は CD4 の大規模な損失の結果最終的に+ T 細胞免疫不全症候群 (AIDS)12を取得で最高潮に達する。抗レトロ ウイルス療法の設定、潜伏感染細胞の貯蔵所は、何十年も治療戦略に手強い障壁をポーズ保持されます。SIV 感染細胞の生体内での特性を理解病因と永続化に尽力のホスト細胞機能を明らかにする可能性があります。ただし、これは非常に挑戦して、感染した細胞のユーザヘとそれらを簡単に識別することができる試薬の不足のために主に。ウイルスの RNA を転写細胞は 0.01-1 に存在すると推定される CD4 の %+ T 細胞血液とリンパ組織13,14,15。抑制療法下で潜伏感染細胞は 10-3– 10-7 16,17,18より頻繁に。体外感染細胞内のギャグなどを学ぶためによく働く、背景は 0.01-0.1 染色による最適な試金のウイルス蛋白質の汚損 %、感染細胞13、周波数以上のよう 14。よ特徴付けられた SIV/HIV Env-特異的モノクローナル抗体を使用して Env 蛋白質の表面の汚損は、おそらく同様の理由から困難であること実証されています。最近では、新規ツールをいずれかでギャグを発現する細胞の検出率の向上目指すギャグ RNA または代替イメージング技術14,15,19を使用して特定の試金を組み込みます。しかし、このようなアプローチは限られている残る量的測定値の数で各セルに対して実行されます。
ここでは、(1)前のヴィヴォ特異的かつ高感度のウイルス遺伝子定量 qPCR、(2) 感染ごとに最大 18 の表面タンパク質および 96 の遺伝子の発現を定量化直接単一のウイルスに感染した細胞を識別する方法論について述べる (感染していない) のセルです。この方法論を組み合わせた即時セル換散によって続いて FACS による単一細胞表面タンパク質測定および Biomark システムにターゲットを絞った qPCR を多重遺伝子発現解析を使用しています。集積流体回路 (IFC) 技術は、9,216 室個別 qPCR 反応が実行されますのマトリックスによって達成、96 のサンプルから 96 遺伝子の多重化の定量を同時にできます。すぐに実行全体のトランスクリプトーム解析を維持しながら高蛋白質豊富な測定を記録ライブ セル FACS 並べ替え下流。ウイルスに感染した細胞を識別するための代わりにスプライシングと, ウイルス Rna (vRNA) 特定のアッセイまで 96 遺伝子で現在収容される試金の最大数の合計ユーザー定義アッセイのパネルと一緒に、qPCR 解析に含まれています。IFC。遺伝子発現とタンパク質情報を各セルには、よく位置によってリンクされています。我々 は以前20をこの分析結果を報告しました。ここで、SIV に感染した CD4 のさらにわかりやすい表現と同様に、方法論のガイドラインの詳細を提供+ T 細胞。
TSCEPTRE 長期的なこのアプローチは、蛍光に分類された抗体とトランスクリプトーム利用 qPCR アッセイと互換性を表現に反応の任意の実行可能なセル人口の懸濁液に適用できます。たとえば、差動遺伝子と希少な細胞または細胞表面蛋白質のマーカーによって簡単に区別の蛋白質の表現の特性評価に使用できます。サンプル調製は、市販の抗体を用いてのプロトコルを汚す標準に依存します。単一セルの並べ替え機能を備えた Cytometers も、市販されているが、追加安全予防策が感染細胞を処理するため必要。称するよく位置によって各セルの単細胞タンパク質の発現プロファイルを記録の並べ替え、インデックスとしては市販 FACS 選別ソフトウェアの一般的な機能です。興味の細胞集団間発現宿主遺伝子の解析はここでは、説明しませんが、参照は、以前に発行されたメソッドに提供されます。
プロトコル
注: プロトコル ワークフローの模式図は、図 1に示すです。3 つの主要手順で構成されています: FACS、RT と cDNA 中古増幅、qPCR 最大 96 の遺伝子を同時に。希釈を制限することで単一のセルを並べ替えセルを並べ替え、プロトコルの 2 つのバージョンは、それぞれ手順 5 と手順 6 で詳しく説明します。これらの戦略は様々 な研究の質問に対処が、同様の手順に従ってください。
1. 前提条件や事前分析
- 前述6として使用するすべての遺伝子発現アッセイを検証します。
注: この手順は、試験日の前に実行されます。すべてのアッセイを検証、商業とカスタム、単一細胞レベルまでの関連する RNA の効率的かつ線形増幅を確保するため必要です。多くの商業的利用とカスタムの試金は、これらの仕様を満たすために失敗します。処理と最大 96 遺伝子の発現アッセイの同時の資格のための自動化されたカーブ フィット補足符号化ファイル 1-5で提供されますが、R2と直線近似の傾斜を使用して、個々 のアッセイを修飾できます。代表的な成功および失敗したアッセイ資格プロットは図 2のとおりです。 -
興味のセル表面のマーカーを染色する抗体の流れフローサイト メーター パネルを開発します。
- 各抗体と関連するサンプルは、たとえば、アカゲザル末梢血単核球 (PBMCs) の汚損によって抗体滴定しなさい。100 μ L のテストあたり抗体の 20 μ L で開始反応を染色し、8 の 2 倍のシリアル希薄を作成します。最大の正および負の集団間の明確な分離を維持しながら染色を示す最適な濃度を特定します。
- 1.2.1 ステップで決定した最適な濃度ですべての抗体の混合物を使用して追加のセル試料に結合された染色を評価します。染色が個々 の抗体の汚れの観察に似ていることを確認します。染色抗体が単独で使用されたときに観察されたものよりも小さいですが、このような抗体を交換する抱合体代替螢光色素を検討してください。
2. 遺伝子発現の分析の準備
- 1-15 mL チューブ、RNase/DNase 自由に試金する結合 96年遺伝子発現 (並べ替え板の数とサイズが異なる場合があります)。本研究で使用される試金のパネルは、表 1と表 2で指定されます。得られた材料は、「アッセイ ミックス」と呼ばれます。180 の最終的な集中に各試験を追加 nM の前方および逆のプライマー。アッセイ ミックスの適切な希釈を達成するために DNA の懸濁液のバッファーを追加します。
注: 実用的な目的は、カスタム (ユーザー生成) 分析の株式準備かもしれない 18 μ M で市販"20 x"遺伝子発現 qPCR 試金 (材料表) の濃度と一致します。各遺伝子のカスタムの前方および逆のプライマーの 18 μ M ミックスは、DNA サスペンション バッファー内の貯蔵液から作られています。市販の試金 (材料表) もなくほかのプローブ、プローブ RT または cDNA 中古増幅することができます、必要なカスタム試金のため省略します。それは 1 つを含めることが推奨または並べ替え、細胞回復および cDNA 合成の効率を評価するため品質管理でのハウスキーピング遺伝子を使用します。CDNA を生成するランダムなプライマーの使用は決定されていないが、遺伝子特定のプライマーより効率が低下する予定です。 - 手順 7.1 (多重 qPCR) で使用するアッセイ プレート × 2 を用意します。96 x 96 チップ アレイが予想されるごとに 96 ウェル PCR プレートの指定ごとに各試験の 6 μ L をピペットします。たとえば、5 チップの各アッセイの 30 μ L、96 ウェル プレートで単一の井戸を占有します。96 の試金を使用して、96 ウェル プレートの各ウェルにアッセイが含まれます。プレートをシール粘着シールを。
注: 理想的には、手順 2.1 と 2.2 は同時に実行、遺伝子発現の試金のための複数の凍結融解サイクルを避けるために。すべて遺伝子アッセイ プレート内する必要がありますもに存在しているアッセイ ミックス (ステップ 2.1)。アッセイ ミックスとアッセイ プレートの両方は、それぞれの長期または短期使用のための 4 ° C または-20 ° C で保存できます。
3. 表面汚れの進行生菌
注: 細胞内染色、透過、および固定、互換性がないこの方法で RNA を危険にさらします。
- 補正のサンプルを準備するには、細胞染色に使用される 2.5-fold の高い濃度で補正ビーズを 40 μ l 添加する材料の表に記載されている各抗体を追加します。光から保護された 25 の ° C で 20 分間インキュベートします。3 mL の PBS を追加ビーズ 500 x gで 25 ° C で 3 分間遠心しPBS を吸引し、〜 300 μ L の PBS のビードを再停止しなさい。
- サンプル処理のフロー フローサイトメトリー セルソーターを準備: 補償管を取得、補償行列を作成し、実験供試体のマトリックスを取得ファイルに適用します。
- 各抗体の材料表、染色されるすべてのサンプル オレンジ 1.5 mL チューブで指定されている適切なボリュームを組み合わせることにより蛍光抗体カクテルのマスターの組合せを準備します。渦と遠心分離機抗体をペレットに 25 ° C で 2 分間 21,000 x gでカクテルを集計します。
注: ここで使用される抗体の材料表のとおりです。 - 2 分追加 0.5-2 mL の PBS の 12 ml 500 x gで 25 ° C で 3 分間遠心 15 mL チューブに細胞懸濁液のための 37 ° C の水浴で凍結保存した細胞を解凍し、PBS を吸引します。PBS 3 mL、5 mL のポリスチレン管への転送で再懸濁します。上記の遠心し、残して 〜 20 μ L 残留 PBS PBS を吸引します。
注: 染色条件に応じて (ステップ 1.2 参照) 抗体価を変更することによって特定のアプリケーションの必要に応じて、染色温度は暖かいまたは冷たい温度に適応する場合があります。 - 最大 2 x 107カクテル 80 μ L 抗体の細胞を洗浄再懸濁し、光から保護された 25 の ° C で 20 分間インキュベートします。2 x 10 の7セルを超えるサンプル増加染色反応ボリュームに応じて維持するために < 2 x 107セル/100 μ L。
- 3 mL の PBS を追加することによって、細胞を洗って 3 分 500 × gで遠心分離し、上清を吸引します。
- 徹底的に 35 μ m ナイロン セル ストレーナー キャップをピペッティングで 300-500 μ L の PBS とフィルターで細胞を再懸濁します。氷の上の細胞を維持し、並べ替えまで光から保護します。
4. セル コレクション プレート、FACS 並べ替えの実行、および生成 cDNA を準備します。
- 単一 RNAse 空き DNAse の生殖不能の管にピペッティングして RT プリアンプ反応混合物のコンポーネント (表 3) を組み合わせます。
注: 手順 3 で染色時に前に、この手順を実行できます。RT 酵素は、qPCR 信号に DNA テンプレートの寄与を決定するここに省略されるかもしれません。 - 96 ウェル PCR 並べ替えコレクション プレートの希望数に RT プリアンプ反作用の組合せの 10 μ L を分注するのにマルチ チャンネル ピペットを使用します。粘着フィルムでプレートをシールし、中古冷蔵 96 ウェル アルミ ブロックにプレートを置きます。
- ステンド グラスのサンプルのおよそ 20,000 のセルからデータを取得することによって流れの cytometer でゲート方式を並べ替えセルを確立します。補償行列が収集されたデータに適用されたことを確認します。ゲートを描画し、遺伝子発現解析のため分離する興味のセル population(s) を識別するゲートのツリーを定義します。
注: 潜在的な SIV vRNA+セルのコレクションに使用されるゲートのツリーは図 3に示すです。 - 各ウェルに分類番号とセルのサブセットを指定する適切な機器の設定を入力します。いずれかの並べ替えのための追加の詳細な命令制限セル希釈系列または単一の細胞それぞれが用意されて手順 5 と 6 で。
- FACS は準備された 96 ウェル PCR コレクション プレートにセルを並べ替えます。並べ替えの前に粘着シールを取り外して、新鮮なシールを次のように置き換えます。
注: は、すべての回、並べ替えの間を含む事前冷却アルミ ブロックにプレートを保ちます。 - 並べ替え、渦および遠心分離機の直後にコレクションが 4 ° C で 1 分間 2,000 x gでプレートします。
- 陶歯, 次の条件を使用して予熱した蓋な PCR 機械で板: 15 分 (RT)、95 ° C 2 分は 50 ° C に続いて 18 サイクル 95 ° C の 15 s の 60 ° C で 4 分 (中古増幅)。
- 20 μ L の DNA 新しい 96 ウェル PCR プレートにバッファーが懸濁液に cDNA の 5 μ L を転送することにより cDNA 1:5 を希釈します。希薄化後の cDNA が 4 ° C または-20 ° C で不明確に保存されるこの時点で。CDNA は、qPCR (ステップ 5.2 7.4) のテンプレートとして使用する準備が整いました。
注: この希釈により RT プリアンプ反応に存在のプライマーが下流 qPCR に寄与しません。
5. 変動 a: FACS 並べ替えセル制限希釈系列内の vRNA+の周波数を決定するために細胞や実験の品質管理を行う
メモ: 単一セルの並べ替えを実行する前にことを使用して複製のシリアル希薄にセルを並べ替えによって興味のセルの周波数を決定のです。この手順も提供します貴重な品質管理並べ替え cDNA 合成 RNA 回復、セル換散効率ステップ 5.3 で説明したよう。VRNA+セル周波数による事前決定適切に動力を与えられた vRNA+細胞遺伝子発現解析のための十分なサンプル サイズを達成するために並べ替える必要があります単一セルの数のより正確な推定が可能です。
- FACS は、4.1-4.2 の手順のように 96 ウェル プレートにセルを並べ替え、複数のレプリケートの井戸あたり 1-1,000 細胞を収集しています。
注: 各セルの希釈でレプリケート井戸の数は一般的にウェルあたり細胞濃度と逆相関します。感染した細胞の頻度は 1% を下回るが、セル希釈する必要がありますウェルあたり 100-1,000 の細胞に焦点を当てます。並べ替えプレート マップの例は、図 1、左上で提供されます。結果の増加を避けるべき井戸ごとのセル数 1,000 を超える反応体積と下流 cDNA 合成と定量化に干渉。 - 表 4のマスター ミックス ソリューションの qPCR 試薬を組み合わせます。25 μ L 反応ボリュームのマスター ミックスの 22.5 μ L は 3.9 のステップからの希薄化後の cDNA テンプレートの 2.5 μ L と組み合わされます。QPCR 標準サイクリング条件を使用してを実行 (94 ° C の 40 のサイクルが続くなど、94 ° C、5 分 15 秒/1 分の 60 ° C)。
注: 従来のリアルタイム qPCR 器具を使用して Singleplex qPCR 反応は、効率的な細胞選別、RNA 回復と cDNA 合成を示す 1 つまたはいくつかのアッセイの経済的な予備的分析として推奨されます。VRNA+細胞の頻度の計算にも使えます。多重 qPCR 反応、Biomark を使用して、通常より大規模な単一細胞分析適しています。 - 品質管理、プロット Et 値 (Et = Ct最大− Ct) あたりもよく、ログスケール10並べ替えセルの数と、線形回帰分析を適用。
注: 一貫した複製、3.3 (± 0.3) の線形回帰斜面と R2 > 0.9 効率的な実験を示唆しています。および並べ替えの例については、RT プリアンプの実験は図 4のとおりです。 - VRNA+細胞の頻度を決定する、プロットの x 軸 (ログ10スケール) との割合によくあたりソート セル番号に肯定的な y 軸に各セル希釈 vRNA の井戸します。例については、図 1 (下左、ポアソン分布) を参照してください。港 1 つの肯定的なセル平均、井戸正 (y 軸の 0.632)21の 63.2% に対応するセルの数を決定するデータを線形回帰モデルを適用されます。このセル希釈数 (x 軸切片) を割合で表した周波数に変換します。たとえば、48 セルあたり 1 つの vRNA+セルは 2.1% の周波数と同じです。
6. 変化 b: 単一細胞解析のため細胞の FACS 並べ替え
- 手順 4.1-4.8 してよく位置によってマップも流れの cytometer のインデックスの並べ替え機能を使用して並べ替え、各セルの個々 の FCS ファイルを作成するごとに並べ替え 1 つのセルを指定します。
注: 並べ替えコレクション板の数は、利用可能な thermocyclers の数を超えた場合、サイクリングを停止できる逆転写酵素不活性化ステップ (2 分の 95 ° C) の後、thermocyclers が利用可能になるまで、4 ° C で cDNA を格納することができます。この場合、15 の 95 ° C の最初のサイクルで中古増幅を開始 s。 - オプション: 残留原液 cDNA を使用して興味の希少な細胞のためのスクリーンに単一細胞のバッチをユーザー定義から cDNA から成る「プール」cDNA を作成します。マルチ チャンネル ピペットで新しい 96 ウェル プレートに 2 μ L 原液単一セル中古増幅 cDNA を転送します。同じに指定されたプールのすべての追加の単一セルからの cDNA の 2 μ L 分注してを繰り返します。従来の qPCR を使用して陽性の細胞を含むものを決定するため (例えば、内の vRNA) 興味の遺伝子の cDNA プールを画面します。プールには、各セルからの cDNA の小さい因数だけする必要がある、ので cDNA の残り 〜 8 μ L が単一細胞解析できます。
注: リソース集中型多重 qPCR による尋問単一セルの数を減らすために単一細胞遺伝子発現解析を実行する前に推奨される戦略にプールします。予備単一プレックス qPCR 法により (例えば、SIV mRNA +) 興味のセルが識別される状況に適したです。単純な高スループット戦略を cDNA のプールを作成する新しい板の単一の井戸に原液 cDNA コレクションの並べ替えプレートの (すなわち行 A のセルをすべて 12) 行または列 (すなわち列 1 のセルをすべて 8)、2 μ L を組み合わせるもあります。最高のプールの方法するには、ステップ 5.4 から興味のセルの予想される頻度を検討してください。たとえば、セルの 10% がプラスになる場合は、六つの単一細胞 cDNA サンプルから成るプールが頻繁にマイナスになるし、そのプールで表されるセルは、こうして下流の単一細胞解析から除外することができます。
7. Biomark プラットフォーム上の qPCR の多重化します。
注: このセクションは、バージョン A または B 上記に続くかもしれない。記載の研究でそれは単一細胞解析にのみ適用されました。
- QPCR アッセイ プレート (2.2 の手順で準備) アッセイ プレート x 2 から各分析の 4 μ L 分注 4 μ L の各ウェルに試薬を読み込みアッセイを含む新しい 96 ウェル PCR プレートに準備します。4 ° C でアッセイ プレートを維持します。
メモ: アッセイ プレートは 4 ° C、1 週間で 1 ヶ月-20 ° C で安定しました。したがって、複数のチップのための十分な材料を準備し、適切に保管すると便利場合があります。 - チップの 2 つの吸気バルブにプライミング注射器からコントロール ライン流体を分配します。プレートの下からプラスチック製の保護を削除します。場所 A1 位置に切り欠き側と IFC コント ローラーのチップ。メイン メニューから「プライム」スクリプトを選択します。スクリプトを実行します。
- 50 μ L の試料 500 μ L PCR マスター ミックス (材料表) の各マイクロ チップと試薬を混合することによってリアルタイム反応混合物を準備します。今後「サンプル プレート」として示される新しい 96 ウェル PCR プレートの各ウェルに 4.4 μ l のピペット。
- 1:5 ステップ 4.8 リアルタイム反応混合物を含むサンプル プレートに希釈 cDNA の 3.6 μ L をピペットします。
注: 場合、PCR ダウン選択したステップ 6.2 で説明したように稀である (例えば、vRNA+) の下流解析のためのセルを画面で、肯定的なプールで表されるセルのみが含まれます。 - チップ プライミングが完了した後、チップの他の (サンプル) 側にも対応するのにサンプル板からチップの切欠き (アッセイ) 側も対応にアッセイ プレートから 5 μ L 分注し、5 μ L チップ入り江をロードします。IFC コント ローラーにチップを挿入し、「ロード ミックス」スクリプトを実行します。
- チップを多重 qPCR を実行する Biomark プラットフォームに転送します。計測器のセットアップと qPCR プログラミング リアルタイム PCR 解析ソフトウェアによって提供される、PCR の 40 のサイクルと遺伝子発現 (GE) 96.96 標準 V.1 プロトコルを使用してステップバイ ステップの命令を次に進みます。指定したフォルダーに ChipRun ファイルを保存します。
注: 1 日あたり、複数日にわたる複数のチップを実行できます。 - QPCR データを分析します。
- リアルタイム PCR 解析ソフトウェアを開きます。「ChipRun.bml」ファイルを開くと、"ファイル |メニューを開きます"。
- ソフトウェアのウィンドウの左上隅に「エクスプ ローラー チップ」と「チップの実行の概要」を検索します。チップ実行概要の 3 つのコンポーネントを識別する: 分析ビュー、サンプルのセットアップ、および検出器のセットアップ。
- 「検出器のセットアップ」をクリックしてします。「タスク」の下で「新規」をクリックし、コンテナー型「SBS プレート」とコンテナ フォーマット"SBS96"を選択します。「マッピング」横あるをクリックして、... ボタンをクリックし、、"M96-分析-SBS96.dsp"を選択します。
- (省略可能) は、各検出器 (アッセイ) 番号または各ウェルの「名」セクションの名を割り当てるも 1stをダブルクリックしています。"F2"を押しても次に移動します。
- 「サンプルのセットアップ」をクリックします。「タスク」「マッピング」の横にある、をクリックして、... ボタンをクリックし、"M96-サンプル-SBS96.dsp"を選択します。
- 「分析ビュー」をクリックします。「QPCR」タブの"タスク"、[選択「リニア (派生物) のベースライン補正」、および"ユーザー (探知機) の Ct しきい値法"。「Ct しきい値」タブで「自動ボックスを初期化」をチェックします。上記の「分析」ボタンをクリックします。
- 「分析ビュー」の右上に、2 番目のタブ「結果表」をクリックしてします。ドロップ ダウン メニューから「ヒート マップ ビュー」を選択します。ヒート マップにデータが表示されます。
- オプション: 制服の ROX 蛍光するチップを渡って流れるように、同じメニューからの「ヒート マップ ビュー」ではなく「イメージ ビュー」を選択します。ヒート マップ上右側のウィンドウから 2 番目、「ロックス」を選択します。最初は右側のウィンドウから、1-40 のサイクルの 1 つを選択します。ROX 蛍光の白黒表示に切り替える右側のウィンドウから 4 番目をクリックします。粒子、飛び散っを通知またはチップ上の欠陥のイメージが表示されます。ROX の均一性が著しく隠されている場合は、チップを再実行します。
- ヒート マップでは、「しきい値」と「ログ グラフ」をクリックします。アッセイ (ヒート マップの列) をクリックして指数段階の増幅曲線が交差する場合は必要に応じてしきい値をドラッグ手動で、各検出器 Ct しきい値を調整します。完了したら、「分析」をクリックします。
- QPCR データを .csv ファイルとしてエクスポートします。スプレッドシートまたは統計解析ソフトウェア (例えばJMP) にデータをインポートし、チップ上のサンプルと試験位置によって結果をマップします。セルを新しい列を作成すると、条件付き数式を使用してウイルスの遺伝子発現に基づくグループにまとめます。「分析」の下で"フィット Y で X"を選択し、グループ対遺伝子発現をプロットします。統計分析を適用します。
注: 4 SIV RNA 種の代表的な単一細胞の定量的表現は図 5 aの二変量プロットで描かれています。SIV RNA+細胞における定量的ホスト遺伝子発現を図 5Bに示します。 - 単一細胞の FACS データから定量的なタンパク質発現値を抽出します。
- .Fcs を開いて、FACS からファイルを並べ替える FlowJo バージョン 9 を使用して 96 ウェル プレートに対応する (ステップ 6.1)。強調表示されているファイル名を選択」プラットフォーム |イベント番号ゲート |インデックス付きの並べ替えを作成ゲート」。個々 のセルは、行で表示で表示されます。
- すべて 96 セル (行ではない) を強調表示し、選択"ワークスペース |エクスポート |「すべて補償 fluors をを選択します。「データ型」の下「FCS ファイル」、「エクスポート」をクリックし、指定フォルダーを選択します。
- 新しい FlowJo ワークスペースに個々 の細胞の新しい .fcs ファイルをドラッグします。すべてのセルを強調表示、「追加統計」(左上隅にある「Σ」ボタン) をクリックして"|意味 |すべての蛍光パラメーター"。
- 左上隅の左から 4 番目のボタンをクリックして「表エディター」を開きます。最初のセルのすべての蛍光灯を選択し、テーブル エディター ウィンドウにドラッグします。テーブル エディター ウィンドウの上部「を作成し、ビュー テーブル」に同じボタンをクリックします。これは 96 セルと各フローの数値パラメーターのテーブルが作成されます。
- 出力 (4 からテーブルの上に左ボタン) いずれかのコピー/貼り付けまたは「アプリケーションの保存および起動」をクリックしてデータベースのソフトウェア (例えば、エクセル、JMP)、にコピーします。
注: この手順は、FlowJo バージョン 9 の特定です。FlowJo バージョン 10 では、インデックス付きデータをインポートするのに別の手順を使用します。インデックス付きデータのセルソーターで作成した .csv ファイルから直接 JMP にコピー/貼り付けることができます。
- 単一細胞の FACS データとナンバー プレートの番号によって qPCR データをマージし、よく配置します。結合された単一細胞遺伝子 (qPCR) でグラフィカルな統計分析を実行およびタンパク質発現 (FACS) データ。
注: 単一のセルの例を組み合わせて qPCR、FACS データは図 6に示す (SIV 感染、スプライシング vRNA+アカゲザル サル細胞の表面蛋白質発現プロファイルのホスト)、図 7 (対表面CD4遺伝子発現CD4 細胞におけるタンパク質発現スプライシング vRNA+アカゲザル サル)、以前20を公開。特異的に識別する興味のセル population(s) の遺伝子を表現、単一細胞解析メソッドが前に説明した推奨20,22,23,24, 占める連続遺伝子の式の値と同様、遺伝子陽性細胞の割合。
結果
プロトコル全体のワークフローは、図 1に描かれています。細胞の数によって定義された 2 つのバリエーションで構成されています: いずれかの制限の希釈または単一のセル、テキストで説明されているようです。2 倍シリアル RNA 希釈プライマー プローブ資格解析の例を図 2に示します。SIV+の潜在的な細胞を識別するためにゲートの戦略を図 3に示します。ハウスキーピング遺伝子GAPDH限界希釈における細胞の FACS ソートのため、最適ではない成功と失敗の品質管理 qPCR は図 4のとおりです。単一セル量的遺伝子の発現 4 SIV RNA 種およびリーサス サル感染細胞で発現したことは、図 5のとおりです。代表二変量、ヒストグラム、および散布図を描く表面蛋白質発現プロファイル SIV RNA+ CD4 の+ T 細胞は図 6にフローサイトメトリーによって測定します。変数バブル プロット (図 7) 表面の CD3、CD4 蛋白質、 CD3やCD4 mRNA 定量的ウイルス遺伝子の関係が表示されます (tat/rev)単一セルの式。

図 1: 実験的ワークフローの模式図は、3 つの主要なコンポーネントを示しています: フローサイトメトリーによる並べ替え、逆のトランスクリプション プラス cDNA の前拡大は PCR に基づくフロー (RT、プリアンプ) と qPCR 。並べ替えは、いずれ限界希釈 (A, 緑の背景)、または単一細胞 (B、オレンジ背景) として実行できます。すぐに FACS 並べ替えセルを分離、cDNA にあらかじめ増幅 (RT、プリアンプ PCR) qPCR テンプレートを準備する RNA は転写リバースします。制限希釈ソートは、ポアソン分布統計情報として実験効率を使用してウイルスの rna 陽性細胞の頻度を決定して、サンプルの回復。緑の矢印は、細胞数の推定がセルの周波数に変換 (肯定的な遺伝子があることそのような井戸の 63.2% の確率に対応する) ウイルスの遺伝子、陽性も含む 1 つのセルあたり並べ替えを示します。周波数推定を使用 (B) その後の並べ替えで集められる単一セルの数を通知する場合があります。インデックス付き単一セル FACS 並べ替え堆積物 1 ウェルあたりのセルし、96 ウェル プレート内でよく位置で注釈が付けられたセルごとにデータ ファイルを生成します。単一セルの qPCR、96 遺伝子の多重同時に実行されます。個々 のセル (右側の列) のプロファイリング表面蛋白質 (FACS) および mRNA の発現を組み合わせることができます。ウイルス遺伝子のオプション qPCR を実行可能性があります中古増幅 PCR と多重 qPCR (B真ん中) 画面の単一細胞またはダウン選択ウイルス RNA+セルに単一細胞 cDNA のプールまたは多重 qPCR 解析用のプールに。ヒート マップは、96 試金 (列) のための遺伝子発現 (Ct 値) と 96 単一セル (行) を示しています。この図の拡大版を表示するのにはここをクリックしてください。

図 2:の入門資格実験から代表 qPCR データが表示されます成功 (左、中央)、(右) 市販の試金を失敗した (CD6、 SIV tat/rev と TLR3、それぞれ).CD6 、 TLR3、 RNA 抽出された 106 FACS ソート アカゲザル PBMC CD4 の+ T 細胞商業キットを使用しています。12 ポイントの RNA の 2 倍希釈系列の複製 8 (0.023 48 ng CD4 あたり 20 pg RNA と仮定すると 1.2-2,400 セル同等に対応する RNA+ T 細胞) RT プリアンプと qPCR に服従しました。SIV tat/revRNA は感染した PBMCsの in vitro SIVmac239 アカゲザルから抽出します。RNA の希釈は、6-12,000 セルのスパンの RNA 同等を用意しました。ら推定される細胞数対してプロットする遺伝子発現を増加、40 Ct 値。希釈系列の R2を展示 > 0.97 と 3.32 ± 0.3 の傾きを示す成功の入門資格。この図の拡大版を表示するのにはここをクリックしてください。
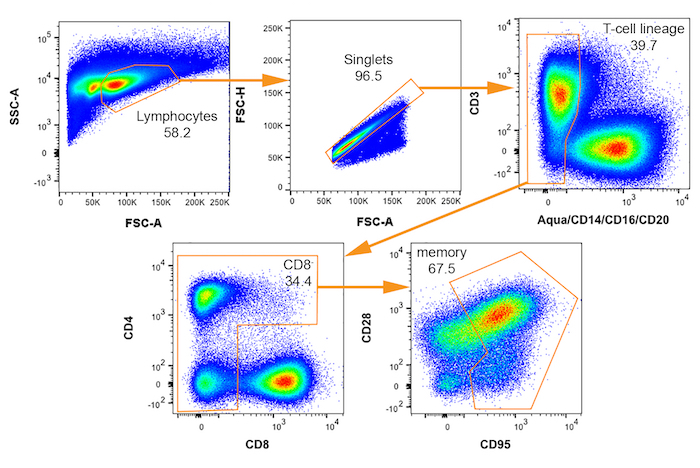
図 3:ゲーティング機構の SIV に感染した可能性のあるアカゲザル サル細胞を分離して二変量 FACS を印刷します。メモリ (CD95 +) を選択するシーケンシャル ゲート CD4+ T 細胞は示された各人口名と右を下に左上から表示されます。各ゲート内親プロットの割合が示されます。この図の拡大版を表示するのにはここをクリックしてください。

図 4: QPCR の代表的な実験による検証を希釈を使用して制限することで細胞の FACS ソート実行GAPDH3 つの独立実験のためのハウスキーピング遺伝子: 成功 (左)、最適ではない (中央)、(右) に失敗しました。以上 2 Ets と 300-1,000 セル井戸 1 ウェルあたり 10-100 個の細胞の複製がまたがる Et。線形回帰斜面は 3.32 ± 0.3 R2をする必要があります > 0.95。これらの仕様を達成するために失敗は、ステップ 1、2、3、またはその組み合わせで技術的な問題を示します。この図の拡大版を表示するのにはここをクリックしてください。

図 5: 単一細胞定量ウイルスとホストにおける遺伝子発現 FACS ソート アカゲザル CD4+ 腸間膜リンパ節 10 日ポスト SIVmac251 感染 T 細胞。(A) ウイルスの遺伝子発現の変量を乗算スプライスのプロット (tat/rev) 単独でスプライス (env) 単一 SIV mRNA 細胞 (左) と。Tat/rev+env-細胞 (ライト グリーン x 軸に沿って) エクスプレスtatの少ないコピー/rev RNA env+の細胞 (ダーク グリーン)、初期に一貫したよりも感染と Rev 蛋白による安定化と環境変数などのスプライシングは、部分的に vRNAs の核輸出前のステージ。スプライシングは、ウイルスの RNA を発現していない細胞はグレー (ないウイルス RNA)、ブラウン (+をギャグとLTR+) またはタン (ギャグ+またはLTR+のいずれか) で描かれています。(ギャグ+), と合計 (LTR+) SIV mRNA 発現が (右) の同じセルに表示されます。ギャグRNA tatでの高い豊富/rev+環境変数+細胞が出芽に包装中に豊富なゲノム RNA を表明した後期段階感染に一貫性のあります。ウイルス粒子。(B) ヴァイオリンがアカゲザル サル遺伝子発現の SIV 感染細胞の感染していない細胞 (グレー) と比較して、少なくとも 1 つのサブセットにプロットされます。統計解析は、20,22,23,24を前述のように行った。アスタリスクは、感染していない細胞を基準にして組み合わせた可能性比率テスト比較に虚偽の発見率 < 10% を示します。行は、各遺伝子の細胞グループ間で平均値を接続します。この図は、ボルトンらから変更されています。20この図の拡大版を表示するのにはここをクリックしてください。

図 6:アカゲザル サル腸間膜リンパ節 10 日ポスト SIVmac251 感染からの細胞の FACS ソートの代表ホスト表面蛋白質発現プロファイル。感染していない (グレー)+をギャグのtat/rev- (ブラウン) の CD3、CD4 およびアイコス表面蛋白質の表現および+をギャグのtat/rev+細胞 (緑) の表示を (A) 散布図。蛍光強度は、(ドット) をセルごとにプロットされます。外れ箱ひげ図を描く四分範囲 (IQR) と中央 (ボックス)、1.5 以内遠いポイント ボックス (ひげ)、潜在的な外れ値 (切断されたポイント) から x IQR。上部の水平バーを示す有意差 (p < 0.05、ノンパラ メトリック Wilcoxon 順位検定)。(B) (A) に示すように細胞の表面の CD3 および CD4 蛋白質の表現の変量とヒストグラムの表示。ドット プロット (左) は、作業領域内の各セル人口の割合を示します。CD3 および CD4 ヒストグラム (中、右) には、感染していない相対的tat/rev+細胞をギャグ+と+をギャグのtat/rev-間の表面蛋白質ダウンレギュレーションが描かれています。(A – B) から 12 (C) 代表的なtat/rev+単一細胞は CD3/CD4 (左)、CD69/CD38 (中央)、(右) アイコス/HLA-博士の表面表現のための 3 つの変量プロットで表示されます。この図の拡大版を表示するのにはここをクリックしてください。
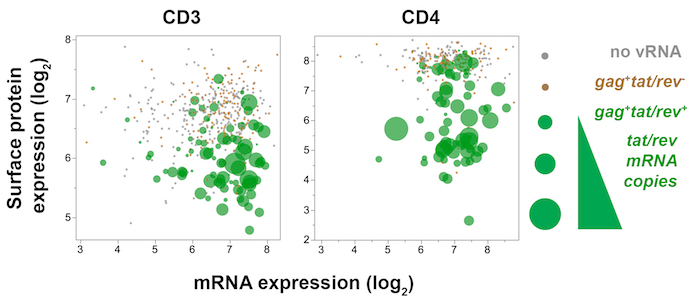
図 7: 10 日腸間膜リンパ節から細胞の単一細胞のウイルスの遺伝子 (tat/回転)、ホスト遺伝子 (CD3 、 CD4) とホスト (CD3、CD4) 表面のタンパク質発現 SIV 感染メモリー cd4 陽性+ T を表示する変数プロットポスト SIVmac251 感染。CD4 蛋白質の表現 (蛍光) は、 tat/回転の各セルで表される量はドットのサイズによって反映されるCD4 mRNA (qPCR) に対してプロットされます。Tatで/rev+細胞 (緑) がそれぞれ持続的なCD4およびCD3の成績、表面の CD3 および CD4 蛋白質の表現を減少、表面蛋白質の表現は下流に変調を示す遺伝子発現。この図の拡大版を表示するのにはここをクリックしてください。
表 1:のプライマーとプローブの SIV 核酸の検出に使用されます。プライマーやプローブは、2 つのシーケンスが示され、両方のシーケンスの等モルの量が使用されました。このファイルをダウンロードするここをクリックしてください。
表 2: Biomark 楽器の産物の定量用 96 遺伝子パネル。4 つの SIV アッセイは、青色の背景色で示されます。このファイルをダウンロードするここをクリックしてください。
テーブル 3:逆のトランスクリプションと中古増幅に使用される反作用の組合せ。このファイルをダウンロードするここをクリックしてください。
表 4:クワント スタジオ 6 楽器に対してリアルタイム PCR 用 qPCR 反応ミックス。このファイルをダウンロードするここをクリックしてください。
の補足的なコーディングのファイル 1。遺伝子発現アッセイを予選について。このファイルをダウンロードするここをクリックしてください。
補足符号化ファイル 2: JMP のサンプル マップ テンプレート。このファイルをダウンロードするここをクリックしてください。
補足符号化ファイル 3: JMP でプローブのマップ テンプレート。このファイルをダウンロードするここをクリックしてください。
補足符号化ファイル 4: JMP のプライマー解析スクリプト。このファイルをダウンロードするここをクリックしてください。
の補足的なコーディングのファイル 5。JMP の区分の分析] スクリプト。このファイルをダウンロードするここをクリックしてください。
ディスカッション
プロトコルは、ここで説明、tSCEPTRE と呼ばれる、高多重 RT qPCR による単一細胞 mRNA の発現を定量的に多項目フローサイトメトリーによる単一細胞表面タンパク質定量を統合します。これら 2 つのテクノロジの連合有効高コンテンツ スナップショットを組み合わせて転写および高スループット形式で単一のセルの蛋白質のプロファイルにします。我々 はこれまでとらえどころのない細胞を生体内SIV、感染を識別するためにメソッドを使用し、発現宿主遺伝子およびタンパク質について説明。プロトコルは表面蛋白、mRNA、またはそれらの組み合わせの式によって区別可能な関心の任意の細胞集団の研究に適応することができます。説明の方法は正確な単一セルの並べ替えに依存とフローサイトメトリーによる記録データ市販 qPCR 試薬とペアになって、リアルタイム PCR のインストルメンテーションを多重化します。出力は単一セル結合タンパク質と遺伝子発現データの機密、定量的な評価です。
単一細胞におけるタンパク質と遺伝子発現のリンク他のアプローチがされている1,25,26,27を説明しました。インデックス付き FACS 並べ替え RNAseq、造血幹細胞の評価に適用した正常に続いて特に有望なアプローチ28を表します。しかし、RNAseq は目標とされた遺伝子発現解析、すなわち公平なにおけるトランスクリプトーム解析をいくつかメリットがありますが高い対象となる複数の比較統計コスト、低コピーの定量化のためより少なく敏感であること成績証明書。また、現在経済的に不可能ですまれな感染細胞の周波数で存在を求めて細胞の何千もの RNAseq を実行する < 1% (例えばHIV、SIV)。他の単一セル技術は、1 つの読み出し、すなわち、 FACS または PCR によってタンパク質と同じ細胞の核酸の検出のための生成を目指しています。FACS 解析 (mRNA 流れ魚)14,15,29,30続いて蛍光オリゴヌクレオチド プローブによる mRNA および蛍光抗体によるタンパク質の検出が含まれます。また、タンパク質は逆転写された mRNA31,32,33,34と並行 qPCR によって検出される抗体オリゴヌクレオチド抱合体のペアで検出できます。.これらの「ハイブリッド」アプローチを提供同時核酸と蛋白質測定単一セルの解像度、tSCEPTRE のように、限定されます、必要がありますをどちらかない定量的 (mRNA-流れ-魚) をされている抗体や mRNA をカスタマイズプローブ。私たちの tSCEPTRE アプローチはタンパクと mRNA の測定の定量的とすべての試薬が市販、病原体固有の試金の可能な例外を除いて。
特別な注意は、実験試料の分析の前にプロトコルのいくつかのステップに適用されるべき。最初、高パラメーター フローサイトメトリーように敏感な検出と解決35蛍光抗体パネルの慎重に最適化が必要です。各抗体は後染色の組み合わせですべての抗体の複合体を使用する場合の評価に最適な分離と最小限の背景を汚すを実現する濃度を識別するために個別に滴定する必要があります。興味の各遺伝子を測定する適切な qPCR の遺伝子発現アッセイの第 2 実験的決定が不可欠です。複数の市販の試金は多くは単一セルのレベル6で定量的ではありません我々 の経験では各遺伝子の通常使用。したがって、定量的遺伝子発現に使用する希釈した RNA の前にすべての提案されたアッセイを修飾することが重要です。これらの試薬の最適化は時間がかかる、しかし、関心のすべての分子の高感度検出できる抗体複合体および遺伝子発現アッセイの信頼性の高いパネルの値は、時間の投資を正当化します。また、優先順位は、(指定"m1"のサフィックス) mrna、特異性を改善するために代替の (サフィックス"s1"または「g1」のゲノム DNA を検出可能な試金がエクソン エキソン接合にまたがるプローブを含む市販のアッセイに与えられるべき) 各セルに同じ染色体コピーによる遺伝子の式の結果に影響を与える可能性は低いと見なされます。RT の RNA を上流の保全は非常に重要で、前述のプロトコルを通じて、チルドのサンプルを保つことによって達成。同様に、FACS の並べ替えと並べ替えの間隔の持続期間と RT プリアンプを最小限に抑える必要があります。両方制限の希釈および単一セルの並べ替え、cDNA 最初分析できる RNA の回復を評価する従来の qPCR でハウスキーピング遺伝子高発現します。観測された式がセル番号の複製または希釈が失敗した検証を制限するため直線近似の傾斜の中で一様ではない場合、セルの並べ替えと下流の手順でトラブルシューティングを実行する必要があります。
ここで説明したアプローチの潜在的な制限には、(1) 試金 mRNA スプライス接合、表面蛋白質の遺伝子の測定 (2) 限られた数の固有ではないため特に rna や DNA の検出が含まれます。ただし、DNA 検出は上記の理由により差分ホスト遺伝子発現解析に大幅に貢献する可能性がありません。さらに、我々 は直接 2 つのアプローチによりする DNA がこのプロトコルで qPCR 信号に貢献する程度を測定:)、逆転写酵素を除くと b) 核膜の換散を強化するセル換散のプロトコルを変更します。大幅に低くより逆転写酵素の存在下での周波数がスプライス両方逆転写酵素がない場合や, ウイルス遺伝子が検出されたまだ (~ 6 と 2 倍の削減、それぞれ)20。したがって、ウイルス遺伝子発現アッセイは、ウイルス RNA ウイルスの DNA を細胞の存在のためのセルの量を過大評価可能性があります。我々 は、この発見受信ウイルス粒子36から SIV/HIV rna がスプライシングと, 発生すること知られているホスト細胞感染中に生成された細胞質の RT 製品を属性します。したがって、いくつかのアプリケーションのためのテンプレートの DNA 由来を量的に逆転写酵素に欠けている条件に実験の一部を捧げることをお勧め場合があります。また、デノボ合成されたウイルス RNA から, SIV/HIV RNA 由来の着信ウイルスを区別できないことに注意する必要があります。第二に、我々 は RT プリアンプ プロトコルに核散のステップを組み込むし、統合された SIV DNA (Alu LTR) コピーの急激な増加を観察 (図 S3ボルトンのら)20. ゲノム DNA は、したがってここで使用されるセル換散方法から抽出した核酸テンプレートに大幅に貢献するそうです。注記のうち、核散のステップの追加は SIV または潜伏感染細胞を含む他のウイルス DNA 陽性細胞を調査しようとして有用な将来の単一細胞研究にあります。
表面タンパク質の数は流れフローサイトメトリー セルソーターの機能によって決まります。現在の市販商品は、30 パラメーターを超えていません。高度なフローサイトメトリーを用いた研究は将来さらにプロファイリングこのアプローチの機能タンパク質が広がります。成績証明書の数は機器や計装 96、提供されているサポート試薬 (例えば、高い濃度のプライマー), を超えて拡張もできます。最終的には、新興プロテオーム (質量分析法) の解析を組み合わせて単一セル技術、トランスクリプトーム (RNAseq)、(DNAseq) のゲノムに取って代わる探索研究31,37,のためのターゲットを絞ったアプローチ38,39します。 ただし、ターゲットを絞った qPCR、おそらく定量的発現解析のためのゴールド スタンダードとしてこのようなオミクスのアプローチを検証するための貴重なツールのまま。
TSCEPTRE による結合タンパク質や転写の解析は、希少またはそれら病原体を抱いて、癌遺伝子, 細胞を含むまたはそれ以外の場合異常な表現型を示すなどの細胞を識別することは困難を調査するための強力なツールです。転写活性状態の SIV/HIV 感染細胞の新しいマーカーは、これらのウイルス感染症の発症機序に関与する新しいメカニズムの発見と同様、この方法で識別できます。潜伏感染細胞の同定ロバ ウイルス DNA は宿主のゲノムに統合するためのプロトコルのさらなる開発が必要になります。注記のうち、HIV/SIV 感染細胞の頻度は、これらの設定から派生した勉強の感染細胞内の実用的な挑戦が存在慢性潜伏または扱われる感染でかなり低いです。我々 のアプローチを産む以前難治性メカニズムを評価するための基礎: 単一細胞レベルでの転写後調節の宿主-病原体相互作用だけでなくより一般的な細胞プロセスの幅広い適用性であり。
開示事項
この仕事は、軍事医学の進歩, inc. のヘンリー ・ m ・ ジャクソン財団と米国国防総省 (DOD) の間の協力協定 (W81XWH-07-2-0067) によって支えられました。見解はこれらの者、米国陸軍や国防総省の位置を表す解釈すべきではないです。研究動物福祉法その他の連邦法と動物に関する規制に準拠して AAALACi 認定施設における動物の使用を承認されたプロトコルの下で実施された実験含む動物と原則を遵守してケアと NRC 文書実験動物使用の 2011 年版のためのガイドに記載されています。
謝辞
著者は NIAID VRC 流れ Cytometry コアと FACS 器具及び分別装置のメンテナンスおよび操作のための MHRP 流れ Cytometry 中核施設を感謝したいです。マリア ・ モンテロ、Vishakha シャルマ、開明歌の専門テクニカル サポート;マイケル ・ Piatak ・ ジュニア (故人) については SIV の qPCR の試金のデザイン。ブランドン メルディと SIV のマシュー Scarlotta 分離シーケンス。見解はこれらの者、米国陸軍や国防総省の位置を表す解釈すべきではないです。研究動物福祉法その他の連邦法と動物に関する規制に準拠して AAALAC 認定施設における動物の使用を承認されたプロトコルの下で実施された実験含む動物と原則を遵守してケアと NRC 文書実験動物使用の 2011 年版のためのガイドに記載されています。
資料
| Name | Company | Catalog Number | Comments |
| RNA extraction and PCR reagents and consumables | |||
| Genemate 96-Well Semi-Skirted PCR Plate | BioExpress/VWR | T-3060-1 | |
| Adhesive PCR Plate Seals | ThermoFisher | AB0558 | |
| Armadillo 384-well PCR Plate | ThermoFisher | AB2384 | |
| MicroAmp Optical Adhesive Film | Applied Biosystems/ThermoFisher | 4311971 | |
| DEPC Water | Quality Biological | 351-068-101 | |
| Glass Distilled Water | Teknova | W3345 | |
| Superscript III Platinum One-Step qRT-PCR Kit | Invitrogen/ThermoFisher | 11732088 | |
| SUPERase-In Rnase Inhibitor | Invitrogen/ThermoFisher | AM2696 | |
| Platinum Taq | Invitrogen/ThermoFisher | 10966034 | |
| dNTP Mix | Invitrogen/ThermoFisher | 18427088 | |
| ROX Reference Dye (if separate from kit) | Invitrogen/ThermoFisher | 12223012 | |
| DNA Suspension Buffer | Teknova | T0223 | |
| RNAqueous kit | Invitrogen/ThermoFisher | AM1931 | |
| TaqMan gene expression assays not listed in Table 2 | |||
| CD6 | Applied Biosystems/ThermoFisher | Hs00198752_m1 | |
| TLR3 | Applied Biosystems/ThermoFisher | Hs1551078_m1 | |
| Biomark reagents | |||
| Control Line Fluid Kit | Fluidigm | 89000021 | |
| TaqMan Universal PCR Mix | Applied Biosystems/ThermoFisher | 4304437 | |
| Assay Loading Reagent | Fluidigm | 85000736 | |
| Sample Loading Reagent | Fluidigm | 85000735 | |
| Dynamic Array 96.96 (chip) | Fluidigm | BMK-M-96.96 | |
| FACS reagents | |||
| SPHERO COMPtrol Goat anti-mouse (lambda) | Spherotech Inc. | CMIgP-30-5H | |
| CompBeads Anti-Mouse Ig,k | BD Biosciences | 51-90-9001229 | |
| 5 ml Polystyrene tube with strainer cap | FALCON | 352235 | |
| Aqua Live/Dead stain | Invitrogen/ThermoFisher | L34976 | dilute 1:800 |
| Mouse Anti-Human CD3 BV650 clone SP34-2 | BD Biosciences | 563916 | dilute 1:40 |
| Mouse Anti-Human CD4 BV786 clone L200 | BD Biosciences | 563914 | dilute 1:20 |
| Mouse Anti-Human CD8 BUV496 clone RPA-T8 | BD Biosciences | 564804 | dilute 1:10 |
| Mouse Anti-Human CD28 BV711 clone CD28.2 | Biolegend | 302948 | dilute 1:20 |
| Mouse Anti-Human CD95 BUV737 clone DX2 | BD Biosciences | 564710 | dilute 1:10 |
| Mouse Anti-Human CD14 BV510 clone M5E2 | Biolegend | 301842 | dilute 1:83 |
| Mouse Anti-Human CD16 BV510 clone 3G8 | Biolegend | 302048 | dilute 1:167 |
| Mouse Anti-Human CD20 BV510 clone 2H7 | Biolegend | 302340 | dilute 1:37 |
| Anti-CD38-R PE clone OKT10 | NHP reagent recource | N/A | dilute 1:100 |
| Mouse Anti-Human CD69 BUV395 clone FN50 | BD Biosciences | 564364 | dilute 1:10 |
| Mouse Anti-Human HLA-DR APC-H7 clone G46-6 | BD Biosciences | 561358 | dilute 1:20 |
| Mouse Anti-Human ICOS Alexa Fluor 700 clone C398.4A | Biolegend | 313528 | dilute 1:80 |
| Instruments | |||
| BioPrptect Containment Enclosure | Baker | ||
| BD FACS Aria | BD Biosciences | ||
| ProtoFlex Dual 96-well PCR system | Applied Biosystems/ThermoFisher | 4484076 | |
| Quant Studio 6 qPCR instrument | Applied Biosystems/ThermoFisher | 4485694 | |
| IFC controller HX | Fluidigm | IFC-HX | |
| Biomark HD | Fluidigm | BMKHD-BMKHD |
参考文献
- Macaulay, I. C., Ponting, C. P., Voet, T. Single-Cell Multiomics: Multiple Measurements from Single Cells. Trends Genet. 33 (2), 155-168 (2017).
- Pasternak, A. O., Lukashov, V. V., Berkhout, B. Cell-associated HIV RNA: a dynamic biomarker of viral persistence. Retrovirology. 10, 41(2013).
- Mailler, E., et al. The Life-Cycle of the HIV-1 Gag-RNA Complex. Viruses. 8 (9), (2016).
- Varmus, H. Retroviruses. Science. 240 (4858), 1427-1435 (1988).
- Frankel, A. D., Young, J. A. HIV-1: fifteen proteins and an RNA. Annu Rev Biochem. 67, 1-25 (1998).
- Dominguez, M. H., et al. Highly multiplexed quantitation of gene expression on single cells. J Immunol Methods. 391 (1-2), 133-145 (2013).
- Tokarev, A., Guatelli, J. Misdirection of membrane trafficking by HIV-1 Vpu and Nef: Keys to viral virulence and persistence. Cell Logist. 1 (3), 90-102 (2011).
- Malim, M. H., Bieniasz, P. D. HIV Restriction Factors and Mechanisms of Evasion. Cold Spring Harb Perspect Med. 2 (5), a006940(2012).
- Sugden, S. M., Bego, M. G., Pham, T. N., Cohen, E. A. Remodeling of the Host Cell Plasma Membrane by HIV-1 Nef and Vpu: A Strategy to Ensure Viral Fitness and Persistence. Viruses. 8 (3), 67(2016).
- Simon, V., Bloch, N., Landau, N. R. Intrinsic host restrictions to HIV-1 and mechanisms of viral escape. Nat Immunol. 16 (6), 546-553 (2015).
- Kirchhoff, F. Immune evasion and counteraction of restriction factors by HIV-1 and other primate lentiviruses. Cell Host Microbe. 8 (1), 55-67 (2010).
- Wigzell, H. Immunopathogenesis of HIV infection. J Acquir Immune Defic Syndr. 1 (6), 559-565 (1988).
- Reynolds, M. R., et al. Ex vivo analysis of SIV-infected cells by flow cytometry. Cytometry A. 77 (11), 1059-1066 (2010).
- Baxter, A. E., et al. Single-Cell Characterization of Viral Translation-Competent Reservoirs in HIV-Infected Individuals. Cell Host Microbe. 20 (3), 368-380 (2016).
- Grau-Exposito, J., et al. A Novel Single-Cell FISH-Flow Assay Identifies Effector Memory CD4+ T cells as a Major Niche for HIV-1 Transcription in HIV-Infected Patients. MBio. 8 (4), (2017).
- Eriksson, S., et al. Comparative analysis of measures of viral reservoirs in HIV-1 eradication studies. PLoS Pathog. 9 (2), e1003174(2013).
- Finzi, D., et al. Identification of a reservoir for HIV-1 in patients on highly active antiretroviral therapy. Science. 278 (5341), 1295-1300 (1997).
- Chomont, N., et al. HIV reservoir size and persistence are driven by T cell survival and homeostatic proliferation. Nat Med. 15 (8), 893-900 (2009).
- DeMaster, L. K., et al. A Subset of CD4/CD8 Double-Negative T Cells Expresses HIV Proteins in Patients on Antiretroviral Therapy. J Virol. 90 (5), 2165-2179 (2015).
- Bolton, D. L., et al. Combined single-cell quantitation of host and SIV genes and proteins ex vivo reveals host-pathogen interactions in individual cells. PLoS Pathog. 13 (6), e1006445(2017).
- Quintans, J., Lefkovits, I. Precursor cells specific to sheep red cells in nude mice. Estimation of frequency in the microculture system. Eur J Immunol. 3 (7), 392-397 (1973).
- Finak, G., et al. MAST: a flexible statistical framework for assessing transcriptional changes and characterizing heterogeneity in single-cell RNA sequencing data. Genome Biol. 16, 278(2015).
- McDavid, A., et al. Modeling bi-modality improves characterization of cell cycle on gene expression in single cells. PLoS Comput Biol. 10 (7), e1003696(2014).
- McDavid, A., Finak, G., Gottardo, R. The contribution of cell cycle to heterogeneity in single-cell RNA-seq data. Nat Biotechnol. 34 (6), 591-593 (2016).
- Kok, Y. L., Ciuffi, A., Metzner, K. J. Unravelling HIV-1 Latency, One Cell at a Time. Trends Microbiol. , (2017).
- Rato, S., Golumbeanu, M., Telenti, A., Ciuffi, A. Exploring viral infection using single-cell sequencing. Virus Res. 239, 55-68 (2017).
- Wagner, A., Regev, A., Yosef, N. Revealing the vectors of cellular identity with single-cell genomics. Nat Biotechnol. 34 (11), 1145-1160 (2016).
- Nestorowa, S., et al. A single-cell resolution map of mouse hematopoietic stem and progenitor cell differentiation. Blood. 128 (8), e20-e31 (2016).
- Soh, K. T., et al. Simultaneous, Single-Cell Measurement of Messenger RNA, Cell Surface Proteins, and Intracellular Proteins. Curr Protoc Cytom. 75, 41-47 (2016).
- Kochan, J., Wawro, M., Kasza, A. Simultaneous detection of mRNA and protein in single cells using immunofluorescence-combined single-molecule RNA FISH. Biotechniques. 59 (4), 209-212 (2015).
- Stahlberg, A., Thomsen, C., Ruff, D., Aman, P. Quantitative PCR analysis of DNA, RNAs, and proteins in the same single cell. Clin Chem. 58 (12), 1682-1691 (2012).
- Assarsson, E., et al. Homogenous 96-plex PEA immunoassay exhibiting high sensitivity, specificity, and excellent scalability. PLoS One. 9 (4), e95192(2014).
- Fredriksson, S., et al. Protein detection using proximity-dependent DNA ligation assays. Nat Biotechnol. 20 (5), 473-477 (2002).
- Genshaft, A. S., et al. targeted profiling of single-cell proteomes and transcriptomes in a single reaction. Genome Biol. 17 (1), 188(2016).
- Mahnke, Y. D., Roederer, M. Optimizing a multicolor immunophenotyping assay. Clin Lab Med. 27 (3), 469-485 (2007).
- Liang, C., Hu, J., Russell, R. S., Kameoka, M., Wainberg, M. A. Spliced human immunodeficiency virus type 1 RNA is reverse transcribed into cDNA within infected cells. AIDS Res Hum Retroviruses. 20 (2), 203-211 (2004).
- Stegle, O., Teichmann, S. A., Marioni, J. C. Computational and analytical challenges in single-cell transcriptomics. Nat Rev Genet. 16 (3), 133-145 (2015).
- Wu, M., Singh, A. K. Single-cell protein analysis. Curr Opin Biotechnol. 23 (1), 83-88 (2012).
- Haque, A., Engel, J., Teichmann, S. A., Lonnberg, T. A practical guide to single-cell RNA-sequencing for biomedical research and clinical applications. Genome Med. 9 (1), 75(2017).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
ISSN 2689-3649
Copyright © 2023 MyJoVE Corporation. All rights reserved
当社はcookieを使用しています。
「続行」をクリックすることで、当社のcookieへの同意となります。