Method Article
Ensaios de fluorescência para o estudo da interação do Mycobacterium tuberculosis com o receptor imunológico SLAMF1
Neste Artigo
Resumo
Este estudo fornece um protocolo para avaliar a interação do Mycobacterium tuberculosis com o sensor microbiano SLAMF1. Os ensaios foram conduzidos em macrófagos derivados de monócitos humanos usando citometria de fluxo e microscopia de fluorescência. As ferramentas descritas são relevantes para o estudo das interações entre patógenos e imunorreceptores.
Resumo
A avaliação da interação direta entre patógenos e receptores imunes geralmente envolve técnicas sofisticadas ou implica o uso de cepas transgênicas e células geneticamente modificadas. Aqui, um método alternativo para detectar a interação bioquímica entre o sensor microbiano de macrófagos SLAMF1 e Mycobacterium tuberculosis é descrito. Duas abordagens técnicas empregando citometria de fluxo e microscopia de fluorescência foram desenvolvidas. Extratos de proteínas celulares totais de macrófagos humanos foram gerados, depois incubados com células inteiras de antígenos de M. tuberculosis (WCMtb) ou M. tuberculosis (Mtb Ags) durante a noite a 4 ° C e, finalmente, reticulados usando formaldeído / glicina / etilenoglicol bis (succinimidil succinato). A interação SLAMF1 com WCMtb por citometria de fluxo foi detectada com um anticorpo anti-SLAMF1 específico para PE. A existência de interação por microscopia de fluorescência foi realizada anexando-se Mtb Ags corados com rodamina-PE a lâminas revestidas com poli-D-lisina, que foram incubadas com o extrato proteico total de macrófagos derivados de monócitos. Após o tratamento de reticulação, o SLAMF1 foi visualizado usando anticorpos primários (anti-SLAMF1) e secundários (Alexa Fluor 488). Os ensaios forneceram uma forte ferramenta bioquímica para medir as interações patógeno-imunorreceptor, superando as dificuldades associadas a linhagens celulares transgênicas e experimentos de modulação da expressão gênica de proteínas.
Introdução
Mycobacterium tuberculosis, o patógeno causador da tuberculose identificado há 142 anos, continua sendo um desafio global, infectando atualmente pelo menos um quarto da população mundial1. Transmitido por gotículas transportadas pelo ar de pessoas infectadas, o M. tuberculosis atinge macrófagos alveolares nos pulmões, onde pode sobreviver por longos períodos em estado latente 2,3. Não apenas a resposta macrofágica local é ativada, mas, nos últimos anos, tem sido descrito que monócitos periféricos podem ser recrutados para o trato respiratório e se diferenciar em macrófagos alveolares, gerando uma resposta ainda mais robusta contra M. tuberculosis do que sua contraparte de origem fetal 2,4.
Os macrófagos são atores fundamentais da imunidade inata. Após a ingestão de M. tuberculosis, os macrófagos exibem inúmeras funções microbicidas, como a secreção de citocinas pró-inflamatórias, a fusão do fagolisossomo e a ativação de outras células imunes para matar o M. tuberculosis 2,3. No entanto, a complexa estrutura arquitetônica desse patógeno fornece efetores (por exemplo, proteínas e lipídios) capazes de regular o metabolismo e as funções dos macrófagos e, consequentemente, o processo inflamatório3. Essa manipulação das respostas dos macrófagos, por meio da secreção de fatores de virulência ou da exploração de fatores do hospedeiro, leva a diferentes estratégias de escape exercidas pelo M. tuberculosis. Alguns dos principais mecanismos de evasão incluem indução de citocinas anti-inflamatórias, inibição da maturação e acidificação do fagolisossomo, alterações do estresse oxidativo, interrupção da autofagia e deficiências durante o processamento e apresentação do antígeno 3,5.
Uma interação rigidamente regulada entre macrófagos e M. tuberculosis é crucial para o desenvolvimento de uma resposta imune adequada. Portanto, estudar essas sinapses é fundamental para identificar mecanismos imunoprotetores ou imunopatogênicos induzidos pela diafonia patógeno-hospedeiro, bem como identificar potenciais alvos terapêuticos. Muitos receptores medeiam o reconhecimento e/ou internalização de M. tuberculosis, incluindo TLRs 6,7,8, NLRs 7,8, receptores de complemento 6,8, receptores de lectina tipo C 6,8 e receptores de limpeza 6,8. Dados acumulados indicam que tanto os PRRs ligados à superfície quanto os intracelulares desempenham um papel importante durante a infecção, reconhecendo M. tuberculosis opsonizado e não opsonizado.
Zihad e Sifat et al. revisaram recentemente a participação dos PRRs nas respostas induzidas por M. tuberculosis por células inatas9. Em particular, a maioria dos membros da família TLR foi implicada na interação com ligantes de M. tuberculosis 6,7. O TLR2 de superfície reconhece antígenos micobacterianos, como lipoproteínas aciladas, lipoproteína de 19 kDa, lipoproteína LprA, lipoproteína LprG, antígeno de 30 kDa, antígeno de 38 kDa, MymA, prolina-prolina-ácido glutâmico (PPE)-57, LAM, LM, PIM, proteína de choque térmico 60, proteína de assinatura Rv1509 e a proteína secretada ESAT-67. A membrana TLR4 interage com proteínas de choque térmico, antígeno de 38 kDa, RpfE, Rv0652, Rv0335c, Rv2659c, Rv1738, Rv2627c, Rv2628, GrpE e HBHA. Por outro lado, os ligantes para TLRs endossômicos (TLR 3, 7, 8 e 9) incluem dsRNA, tRNA, ssRNA, RNA fagossomal e dsDNA de M. tuberculosis. Além disso, o TLR2 tem um papel ativo no início de respostas imunes ao atuar em conjunto com TLR1 e TLR6, TLR4 e TLR9 6,7. NOD2 e NLRP3 são os NLRs citoplasmáticos mais bem caracterizados na tuberculose. Embora seus ligantes específicos permaneçam em estudo, esses receptores são ativados por dipeptídeo muramil ou ESAT-6, respectivamente 7,8. Os receptores de lectina do tipo C estão classicamente envolvidos na endocitose de M. tuberculosis. Enquanto a Dectina-2 atua como um PRR direto para ManLAM da parede celular de M. tuberculosis, o ligante Dectina-1 ainda não foi descoberto8. Mincle e MCL reconhecem o glicolipídio trealose-6,6′-dimicolato (TDM), também chamado de fator de corda7. O DCAR interage com os glicolipídios micobacterianos fosfatidil-mio-inositol manosídeos (PIMs)7. CR3 reconhece micobactérias LAM e PIM, DC-SIGN liga ManLAM, MR reconhece vários componentes de M. tuberculosis, incluindo ManLAM, PIM, LM, glicoproteína de 38 kDa, antígeno de 19 kDa e outras proteínas manosiladas, enquanto o ligante DCIR ainda não foi identificado.
Os CLRs solúveis incluem SP-A, que reconhece ManLam, LM, glicoproteína de 60 kDa e glicoproteína Apa; SP-D interagindo com LAM, LM e PILAM; e MBL, especializada no reconhecimento ManLAM 6,7. Os receptores de limpeza também são PRRs fagocíticos. SR-A e MARCO têm TDM como ligante, SR-B1 reconhece ESAT-6 e CD36 se liga a ManLAM e LM 7,8. Além disso, Dectin-1, Mintle e MARCO também podem se combinar com TLR2 ou TLR4 para disparar sinais após a detecção de PAMPs de M. tuberculosis 6,7. O CD14 é um receptor de superfície que tem a capacidade de internalizar bactérias não opsonizadas e também reconhece a proteína de choque térmico Chaperonina 60.1. Em particular, o CD14 funciona como um co-receptor junto com MARCO e TLR26. AIM2 é um receptor citosólico que pode detectar ssDNA em M. tuberculosis escapa do fagossomo8. Finalmente, AhR é um fator de transcrição ativado por ligante que se liga ao fator de virulência pigmentado naftoquinona ftiocol de M. tuberculosis6.
Muitas das interações descritas acima foram postuladas e não estritamente demonstradas. Mesmo os ligantes para certos receptores permanecem desconhecidos, o que reforça a necessidade de entender melhor o campo de imunoreconhecimento do M. tuberculosis. Nesse contexto, a molécula coestimulatória SLAMF1 (Signaling Lymphocytic Activation Molecule) foi recentemente descrita como um receptor de M. tuberculosis por Barbero et al.10. Ao atuar não apenas como uma molécula de sinalização, mas também como um sensor de M. tuberculosis, o SLAMF1 tem um papel particularmente intrigante na tuberculose. O SLAMF1 pode induzir a ativação de células imunes modulando funções protetoras, como a produção de IFN-γ por células T por meio da fosforilação de Erk / CREB11 , 12 , 13 , autofagia em neutrófilos14 e depuração bacteriana em macrófagos15.
As interações receptor-ligante têm sido estudadas ao longo dos anos utilizando técnicas como ELISA, Ressonância Plasmônica de Superfície (SPR), Calorimetria de Titulação Isotérmica (ITC), Polarização de Fluorescência (FP), Cristalografia de Raios-X, Espectroscopia de Ressonância Magnética Nuclear (RMN), Termoforese em Microescala (MST), Microscopia Confocal de Transferência de Energia de Ressonância (por exemplo, BRET ou FRET), Microscopia Eletrônica, Microscopia Crioeletrônica (Cryo-EM) e Microscopia de Força Atômica (AFM)16,17, 18,19,20,21,22,23,24,25,26,27,28. Algumas dessas abordagens implicam o uso de genes repórteres, proteínas recombinantes rotuladas ou moléculas quiméricas, modelos de nocaute, knockdown ou superexpressão. Alternativamente, as ferramentas computacionais podem prever interações receptor-ligante e locais de ligação e são frequentemente usadas em combinação com abordagens biológicas para obter uma compreensão abrangente das interações 29,30,31. Aqui, são descritas duas alternativas para detectar interação bioquímica e também uma seção sobre como marcar bactérias fluorescentemente. Um protocolo que permite o estudo das interações receptor-patógeno in vitro é apresentado, avaliando particularmente o envolvimento SLAMF1-M. tuberculosis por citometria de fluxo e microscopia de fluorescência, duas técnicas geralmente disponíveis e usadas rotineiramente.
Protocolo
Todos os procedimentos envolvendo macrófagos derivados de monócitos humanos foram realizados de acordo com a Declaração de Helsinque (2013) e de acordo com o Comitê de Ética da UNNOBA (COENOBA). O consentimento informado por escrito foi obtido antes da coleta da amostra. A distribuição do grupo masculino/feminino foi de 13/6 e a mediana de idade foi de 32 anos, com intervalo interquartil (IIQ) de 18 a 75 anos. A presença de patologias prévias, comorbidades ou diagnóstico positivo para tuberculose foram definidos como critérios de exclusão. Os detalhes dos reagentes e equipamentos estão listados na Tabela de Materiais.
1. Cultura e estimulação de macrófagos derivados de monócitos
- Coleta de sangue
- Obtenha 60 mL de sangue de doadores saudáveis por punção venosa em uma seringa heparinizada.
NOTA: A extração de sangue deve ser realizada por um bioquímico ou técnico de flebotomia. - Descarte a agulha no recipiente de descarte de perfurocortantes e qualquer material que tenha estado em contato com o sangue dos doadores no recipiente de resíduos patológicos.
- Obtenha 60 mL de sangue de doadores saudáveis por punção venosa em uma seringa heparinizada.
- Isolamento de monócitos e geração de macrófagos
- Transfira cuidadosamente o sangue para tubos de 50 mL e dilua-o pela metade com solução salina.
- Realize centrifugação em um meio de gradiente de densidade para obter células mononucleares do sangue periférico (PBMCs).
- Colete os PBMCs do halo esbranquiçado.
- Realize duas lavagens com solução salina a 400 x g por 10 min e uma lavagem final a 200 x g por 15 min a 20 ° C para eliminar as plaquetas.
- Determine o número de PBMCs em uma câmara de contagem de células e execute a seleção magnética positiva de CD14 com esferas de CD14.
NOTA: Durante a seleção magnética, siga cuidadosamente as instruções do fabricante. Além disso, pode-se verificar a pureza de monócitos isolados usando citometria de fluxo. - Ressuspenda as células isoladas em RPMI 1640 e determine o número de monócitos em uma câmara de contagem de células.
- Semear 500 μL de 1 × 106/mL de monócitos selecionados CD14 positivos por poço em uma placa de cultura de células de 24 poços na ausência de soro para promover a aderência.
- Após 2 h, remova as células não aderentes lavando-as com RPMI 1640 pré-aquecido.
- Para diferenciar os macrófagos, cultive os monócitos aderentes por mais 16-18 h (durante a noite) em 1 mL de meio completo (meio RPMI 1640 suplementado com L-glutamina, 10% de soro fetal bovino (FBS), 100 U / mL de penicilina e 100 μg / mL de estreptomicina).
- No dia seguinte, mude o meio de cultura. Remova o meio, lave com 1 mL de 1x PBS e adicione 1 mL de meio completo.
- Estimular os macrófagos com M. tuberculosis sonicado (antigénios de M. tuberculosis (Ags)) durante 24 h. Use 10 μL de M. tuberculosis Ags (10 μL = 1 × 106 bactérias) por 1 × 106 macrófagos derivados de monócitos.
NOTA: 10 μL de M. tuberculosis Ags (Mtb Ags) = 1 × 106 bactérias, quantificado por O.D. 600nm em espectrofotômetro considerando um O.D. de 1 como 1 x 108 bactérias/mL. Use cabines de biossegurança BSL2 e uma incubadora a 37 °C em uma atmosfera com 5% de CO2 para todas as etapas da cultura.
- Corroboração da expressão SLAMF1
- Colete os macrófagos de um único poço. Coloque a placa de cultura em uma superfície fria. Retire as células usando movimentos para cima e para baixo adicionando 1 mL de FACS frio (1x PBS + 2% FBS) duas vezes consecutivas. Use um tubo de fundo redondo de citometria para a coleta.
- Centrifugue as células a 500 x g durante 5 min a 4 °C. Descarte o sobrenadante despejando-o.
- Manchar as células com um anticorpo anti-SLAMF1 humano acoplado a fluoróforo durante 30 min a 4 °C no escuro. Vórtice após a adição de 1,25 μL do anticorpo.
- Lave as células para eliminar o excesso de anticorpos usando FACS, ressuspenda o pellet em FACS e adquira a amostra usando um citômetro de fluxo.
NOTA: 1,25 μL de anticorpo anti-SLAMF1 corresponde a 0,0625 μg que é usado para marcar 0,5 x 106 macrófagos. No entanto, a titulação do anticorpo é fortemente recomendada, bem como a realização do isotipo correspondente ou controle de fluorescência menos um (FMO).
2. Preparação de extrato de proteína celular total
- Preparação do tampão de lise
- Prepare tampão de ensaio de imunoprecipitação de rádio (RIPA) com NaCl 150 mM, Tris 10 mM, EDTA 5 mM, SDS 1%, Triton X-100 1% e desoxicolato de sódio 1%.
- Suplemente o tampão RIPA com fluoreto de fenilmetilsulfonil (PMSF) 1 mM e inibidores de protease, como pepstatina A e leupeptina.
- Coleta de células e isolamento de proteínas
- Descarte o RPMI completo dos poços da placa usando uma pipeta P1000 e colha macrófagos por pipetagem para cima e para baixo com PBS 1x frio.
- Transfira as células para tubos de microcentrífuga de 1,5 mL. Colete um poço por microtubo.
- Centrifugue a 500 x g durante 5 min a 4 °C. Descarte o sobrenadante.
- Adicione 1 mL de PBS frio e repita as etapas 2.2.2 e 2.2.3 coletando as células no mesmo tubo de microcentrífuga.
- Ressuspenda os pellets de células em 100 μL de 1x PBS e agrupe quatro tubos. Repita a etapa 2.2.3. Cada tubo conterá 2 x 106 macrófagos.
- Adicione 100 μL de tampão RIPA suplementado e incube a suspensão em gelo por 1 h, vórtice a cada 10 min.
- Centrifugue a 14.000 x g por 15 min. Recolher o sobrenadante e conservar até ser utilizado a -20 °C.
3. Interação M. tuberculosis-SLAMF1 por citometria de fluxo
- Reticulação proteínas-bactérias
- Incubar 1 × 106 células inteiras inativadas de M. tuberculosis (WCMtb, M. tuberculosis inativadas por irradiação gama) com extrato de proteína de 1 x 106 macrófagos (50 μL) durante a noite a 4 ° C em um suporte de microtubo rotativo.
NOTA: 10 μL de WCMtb = 1 × 106 bactérias, quantificado por O.D. 600nm em espectrofotômetro considerando um O.D. de 1 como 1 x 108 bactérias/mL. - No dia seguinte, adicione 500 μL de formaldeído a 1% diluído em 1x PBS. Incubar por 15 min sob agitação (agitador) à temperatura ambiente (RT).
- Adicionar 25 μL de glicina 0,125 M diluída em água. Incubar durante 5 min sob agitação (agitador) à RT.
- Lave duas vezes com 1 mL de 1x PBS a 14.000 x g por 5 min. Descarte o sobrenadante entre as etapas de lavagem.
- Ressuspender o sedimento e incubar a suspensão em 500 μL de 2 mM de reticulador de etilenoglicol bis (succinato de succinimidil) (EGS) diluído numa mistura de 1:1 de ácido acético glacial:água durante 1 h a RT.
NOTA: A preparação dos reagentes no momento do uso é altamente recomendada. Para preparar o EGS, o ácido acético glacial e a água devem ser misturados em proporções iguais e aquecidos a 70 °C. Se a solução não estiver quente, o EGS não é completamente ressuspenso.
- Incubar 1 × 106 células inteiras inativadas de M. tuberculosis (WCMtb, M. tuberculosis inativadas por irradiação gama) com extrato de proteína de 1 x 106 macrófagos (50 μL) durante a noite a 4 ° C em um suporte de microtubo rotativo.
- Coloração SLAMF1
- Repita a etapa 3.1.4 usando 500 μL de 1x PBS na primeira lavagem e 1 mL na segunda.
- Manchar o complexo proteína-bactéria durante 30 minutos a 4 °C no escuro com um anticorpo anti-SLAMF1 humano (como no passo 1.3.3) adicionando o anticorpo ao microtubo e ao vórtice. Use a quantidade de anticorpo adequada para 1 x 106 células (2,5 μL ou 0,125 μg).
- Lave as células para eliminar o excesso de anticorpos usando FACS, ressuspenda o pellet em FACS e adquira a amostra em um citômetro de fluxo (como na etapa 1.3.4).
NOTA: Execute o isotipo correspondente ou controle FMO.
4. Marcação de antígenos de M. tuberculosis
- Ressuspenda a rodamina B em etanol a uma concentração de estoque de 5 mg / mL (1000x). Conservar a -20 °C até à utilização.
- Adicione 1 μL de rodamina B por 100 μL de M. tuberculosis Ags em um tubo de microcentrífuga de 1,5 mL.
- Incubar por 45 min em RT sob agitação contínua (vórtice).
- Lave pelo menos 3 vezes a 14.000 x g por 5 min a 24 ° C usando 1 mL de 1x PBS até que o sobrenadante fique transparente. O pellet (Mtb-R Ags) ficará rosa claro.
- Ressuspenda o Mtb-R Ags em 100 μL de 1x PBS para restaurar o volume inicial.
NOTA: Recomenda-se trabalhar em condições estéreis. Certifique-se de que a coloração seja realizada imediatamente antes de realizar experimentos.
5. Interação M. tuberculosis-SLAMF1 por microscopia de fluorescência
- Reticulação bactéria-proteína
- Introduzir lamínulas redondas de 12 mm em placas de cultura de 24 poços (1 lamínula por alvéolo) utilizando uma pinça cirúrgica de ponta redonda.
NOTA: Limpe bem as lamínulas com etanol a 70%. Se desejar, eles podem ser autoclavados ou esterilizados sob luz ultravioleta por 30 min. - Incubar a lamínula com 400 μL de 10 μg/ml de poli-D-lisina durante a noite a 4 °C. Cubra a placa com papel alumínio para protegê-la da luz.
- No dia seguinte, retire a placa da geladeira e lave a lamínula duas vezes com 1 mL de água.
- Deixe a lamínula secar.
- Diluir 20 μL (2 x 106) de Mtb-R Ags em 180 μL de 1x PBS. Os 200 μL finais são suficientes para cobrir completamente uma lamínula.
- Incubar a lamínula com Mtb-R Ags durante 1 h a 37 °C numa incubadora de células.
- Descarte o líquido restante e lave duas vezes com 1mL de 1x PBS.
NOTA: Nesta etapa, a correta adesão do Mtb-R à lamínula pode ser verificada com um microscópio de fluorescência usando os filtros correspondentes ao canal vermelho. - Adicione 400 μL de tampão de bloqueio (10% FBS em 1x PBS) por 30 min em RT em agitação (agitador).
- Lave duas vezes com 400 μL de tampão de bloqueio.
- Incubar com 100 μL de extrato proteico diluído em 300 μL de 1x PBS por 2 h em RT em agitação (shaker).
- Adicione 400 μL de formaldeído a 1% diluído em 1x PBS. Incubar durante 15 min sob agitação (agitador) em RT.
- Adicione 500 μL de glicina 0,125 M diluída em água. Incubar durante 5 min sob agitação (agitador) à RT.
- Lave duas vezes com 1 mL de 1x PBS.
- Adicionar 400 μL de EGS 2 mM durante 1 h a RT em agitação (agitador).
- Repita a etapa 5.1.13
NOTA: A preparação dos reagentes no momento do uso é altamente recomendada.
- Introduzir lamínulas redondas de 12 mm em placas de cultura de 24 poços (1 lamínula por alvéolo) utilizando uma pinça cirúrgica de ponta redonda.
- Coloração SLAMF1
- Enrole a tampa da placa de cultura em filme de parafina.
- Adicione uma gota de 60 μL de anticorpo primário anti-SLAMF1 previamente titulado (diluição 1:75) na tampa coberta com película de parafina.
- Remova cuidadosamente a lamínula da placa usando pinças cirúrgicas curvas de ponta fina e agulhas.
NOTA: Para facilitar a remoção das lamínulas, a agulha pode ser dobrada em um ângulo aproximado de 45°. - Inverter a lamínula sobre a gota que contém o anticorpo. Incube por 30 min em RT no escuro.
- Levante a lamínula e lave duas vezes com o amortecedor de bloqueio.
- Inverter a lamínula sobre uma nova gota contendo o anticorpo secundário (diluição 1:200). Incube por 30 min em RT no escuro.
- Repita a etapa 5.2.5.
- Remova o excesso de líquido e monte a lamínula em uma gota de líquido de montagem colocada em uma lâmina de vidro.
- Observar ao microscópio de fluorescência com filtros adequados.
NOTA: Recomenda-se trabalhar em condições estéreis. Dilua os anticorpos no tampão de bloqueio. Manuseie a lamínula com cuidado para evitar arranhões que possam dificultar a observação ao microscópio. Um fluido de montagem adequado para fluorescência é altamente recomendado.
Resultados
Neste trabalho, é fornecido um protocolo que permite a avaliação da interação de M. tuberculosis com o receptor imune SLAMF1 em macrófagos humanos (Figura 1). Para tanto, foi obtido sangue periférico de doadores saudáveis. Em seguida, as PBMCs foram separadas por centrifugação sobre o meio de gradiente de densidade e os monócitos foram isolados por seleção magnética positiva (≥95% de pureza, Figura 2A,B). Os monócitos isolados foram aderidos a placas de cultura plástica por 2 h para obter macrófagos derivados de monócitos e, em seguida, cultivados com ON. Posteriormente, os macrófagos foram estimulados com M. tuberculosis sonicado (Mtb Ags) para induzir a expressão de superfície de SLAMF1 conforme relatado antesde 10, e os níveis de SLAMF1 de superfície foram confirmados por citometria de fluxo (Figura 2C). Os macrófagos foram finalmente lisados usando tampão de lise RIPA combinado com vórtice e incubação em gelo para facilitar a lise celular. O extrato proteico total gerado foi utilizado nos ensaios de interação.
No primeiro conjunto de procedimentos experimentais, a citometria de fluxo foi empregada para estudar a interação de SLAMF1 com células inteiras de M. tuberculosis (WCMtb) (Figura 3). Primeiro, a interação física entre SLAMF1 (conteúdo no extrato proteico) e WCMtb foi promovida incubando-os juntos ON em rotação contínua a 4 ° C. O tratamento de reticulação foi então realizado e, usando um anticorpo anti-SLAMF1 específico, o receptor SLAMF1 que estava ligado ao WCMtb foi detectado. Neste ensaio, o WCMtb sozinho foi usado para definir as voltagens correspondentes ao tamanho e granularidade de M. tuberculosis no citômetro de fluxo, que era a população de interesse para detectar a ligação de SLAMF1.
Na segunda abordagem experimental, a microscopia de fluorescência foi empregada para detectar a interação SLAMF1-M. tuberculosis (Figura 4). Este ensaio, no qual foi utilizado M. tuberculosis sonicado (Mtb Ags), complementa o anterior. Mtb Ags corados com rodamina B (Mtb-R Ags) foram usados para facilitar a detecção de fluorescência no canal/filtro correspondente ao fluorocromo PE. As Ags Mtb-R foram anexadas a lamínulas revestidas com poli-D-lisina, e a coloração bem-sucedida foi verificada por observação microscópica (Figura 4A). Em seguida, as lâminas revestidas com Ags Mtb-R foram incubadas com os extratos proteicos contendo SLAMF1. Após o tratamento de reticulação, o SLAMF1 foi detectado usando um anticorpo anti-SLAMF1 específico primário seguido por um anticorpo secundário acoplado a um fluorocromo compatível para ser co-observado com PE. Após a obtenção de imagens individuais de microscopia fluorescente nos dois canais para Mtb-R Ags e SLAMF1, a interação foi corroborada pela fusão de imagens (Figura 4B).
A realização dessas abordagens demonstrou a existência de uma interação entre SLAMF1 e M. tuberculosis. Foi detectado o crosstalk de SLAMF1 com bactérias inteiras e sonicadas, o que deu força à hipótese que aponta SLAMF1 como um novo receptor inato de macrófagos humanos para M. tuberculosis10.

Figura 1: Fluxo de trabalho para detectar a interação SLAMF1-M. tuberculosis . O sangue periférico foi obtido de doadores saudáveis por punção venosa e centrifugado em um meio de gradiente de densidade para isolar as células mononucleares do sangue periférico (PBMCs). Após a determinação do número de células, as PBMCs foram submetidas à seleção magnética positiva para purificar os monócitosCD14 pos . Os monócitos foram então aderidos a placas de cultura por 2 h na ausência de soro fetal bovino e, em seguida, cultivados durante a noite em RPMI suplementado (etapa de repouso) para obter macrófagos derivados de monócitos. Posteriormente, os macrófagos foram estimulados com M. tuberculosis sonicado (Mtb Ags) por 24 h para induzir a expressão de superfície de SLAMF1. Os níveis de SLAMF1 foram testados por citometria de fluxo usando um anticorpo humano anti-SLAMF1 específico em uma fração da população de macrófagos. Os macrófagos restantes foram usados para obter extratos proteicos totais. A lise celular foi realizada combinando vórtice e incubações em gelo em um tampão de lise. Finalmente, a interação entre SLAMF1 e M. tuberculosis foi avaliada por citometria de fluxo e microscopia de fluorescência após a reticulação de proteínas de macrófagos com Mtb Ags ou células inteiras de M. tuberculosis (WCMtb). Clique aqui para ver uma versão maior desta figura.

Figura 2: Controles experimentais e técnicos. (A) Os monócitosCD14 pos foram isolados de PBMCs por seleção magnética positiva. As frações de células positivas (monócitos, azuis) e negativas (vermelho) resultantes foram avaliadas por citometria de fluxo (SSC-A vs. FSC-A) para confirmar uma seleção bem-sucedida. (B) A pureza da fração positiva foi corroborada por citometria de fluxo analisando a granularidade e o tamanho da célula (SSC-A vs. FSC-A). (C) Macrófagos derivados de monócitos foram selecionados por SSC-A vs. FSC-A e, em seguida, fechado para excluir dupletos por uma estratégia de singletos duplos (FSC-A vs. FSC-H seguido por SSC-A vs. SSC-H). A expressão de superfície de SLAMF1 foi avaliada em células únicas. Em todos os casos (A-C), gráficos representativos ou histogramas são mostrados. Clique aqui para ver uma versão maior desta figura.
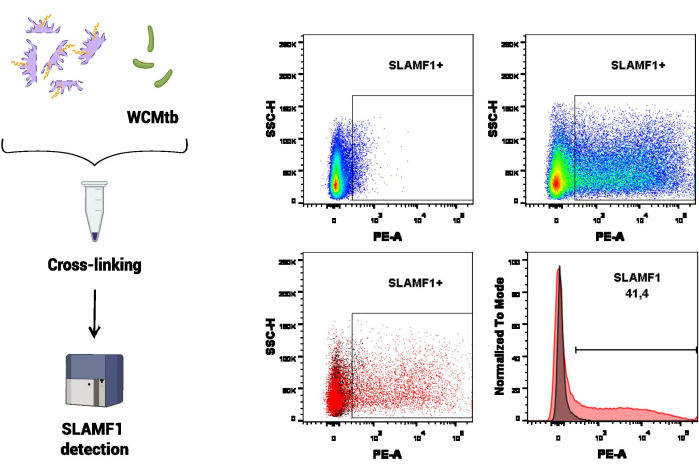
Figura 3: Interação SLAMF1-M. tuberculosis por citometria de fluxo. A reticulação entre proteínas de macrófagos e células inteiras de M. tuberculosis (WCMtb) foi realizada por tratamento com formaldeído/glicina/etilenoglicol bis, conforme descrito na seção do protocolo. Em seguida, a interação foi avaliada por citometria de fluxo, detectando um sinal SLAMF1 (PE) positivo após selecionar a população de interesse (SSC-A vs. FSC-A). O painel superior mostra um sinal positivo para SLAMF1, comparando o controle de fluorescência menos um (FMO) (esquerda) com a coloração positiva (direita). O painel inferior mostra a sobreposição (esquerda, dot plot e direita, histograma) entre o sinal SLAMF1 e o controle FMO. Clique aqui para ver uma versão maior desta figura.

Figura 4: Interação SLAMF1-M. tuberculosis por microscopia de fluorescência. (A) Mtb Ags foram corados com Rodamina B (Mtb-R Ags) e fixados a uma lamínula redonda. A correta adesão e fluorescência das Ags Mtb-R foram corroboradas por microscopia de fluorescência utilizando filtros correspondentes ao canal vermelho. (B) A reticulação entre proteínas de macrófagos e Mtb-R Ags foi realizada por tratamento com formaldeído/glicina/etilenoglicol bis, conforme descrito na seção do protocolo, adicionando o extrato de proteína sobre os Ags Mtb-R anexados. A interação foi avaliada por microscopia de fluorescência usando um anticorpo humano anti-SLAMF1 seguido de anticorpo secundário marcado com Alexa Fluor 488. As micrografias superiores mostram sinais positivos SLAMF1 (esquerda, verde) e Mtb-R Ags (direita, vermelho). A micrografia baixa evidencia a interação em amarelo (fusão de ambos os canais). As barras de escala correspondem a 10 μm. Clique aqui para ver uma versão maior desta figura.
Discussão
Este estudo fornece um guia útil para estudar a interação bioquímica entre M. tuberculosis e sensores microbianos expressos em macrófagos humanos, um tipo de célula chave envolvida na resposta do hospedeiro durante a tuberculose. Os protocolos fornecidos serão relevantes para decifrar moléculas que desempenham um papel na entrada de M. tuberculosis em fagócitos.
A caracterização de interações biomoleculares, como a entre patógenos e imunorreceptores, é crucial para entender os mecanismos imunoprotetores e as estratégias de evasão provocadas por M. tuberculosis. Muitas vezes, demonstrar uma interação direta do receptor com seu ligante bacteriano pode ser complexo e pode exigir técnicas sofisticadas ou sistemas sem versatilidade. Driessen et al. estudaram o papel dos manosídeos de fosfatidilinositol na interação entre micobactérias e DC-SIGN usando cepas deficientes mutantes de M. bovis BCG32. Embora tenham conseguido estudar essas interações, os autores não encontraram diferenças para as cepas mutantes e discutiram que a criação de bactérias mutantes que apresentam ligação reduzida seria uma tarefa enorme32. Outros autores realizaram um excelente e trabalhoso trabalho para demonstrar a interação entre TLR2 e algumas proteínas PE/PPE de M. tuberculosis33,34. Esses estudos exigiram o uso de camundongos knockout para o receptor, transfecção de células e purificação das proteínas acima mencionadas ou de certos domínios proteicos33,34. Além disso, a função dos receptores é frequentemente inferida, mas a interação não é demonstrada de forma confiável. Um exemplo é o receptor solúvel PTX3, para o qual foram encontrados haplótipos associados ao desfecho da doença, mas cuja interação específica com M. tuberculosis não foi rigorosamente estudada35. Em relação a essas deficiências, nosso método permite avaliar a interação do receptor-alvo em estudo com o M. tuberculosis de forma simples e com técnicas acessíveis e de fácil interpretação.
Em relação aos pontos anteriores, e particularmente para SLAMF1, as interações com OmpC e OmpF de E. coli36 e Omp25 de B. abortus37 foram previamente avaliadas. Degos et al.37 usaram células COS-7 transfectadas com um plasmídeo que codifica apenas o domínio extracelular de SLAMF1 para então purificar SLAMF1 e analisar a interação com Omp25 por Western blot. Eles também realizaram experimentos usando camundongos SLAMF1-/- e B. abortus do tipo selvagem ou um mutante defeituoso de Omp25. Berger et al. 36 também mostraram evidências convincentes de uma interação direta entre SLAMF1 e E. coli Omp e S. typhimurium SseB-. Eles usaram diferentes abordagens, incluindo camundongos SLAMF1-/- , macrófagos RAW264.7 codificando uma construção SLAMF1mCherry e também um ensaio de amplificação usando células T Jurkat transfectadas com SLAMF1. Eles desenvolveram um ensaio de amplificação de sinal sensível com um repórter de luciferase usando 1 x 108 E. coli. A este respeito, um ponto forte dos ensaios de interação bioquímica apresentados neste relatório é que a interação foi realmente encontrada usando células do tipo selvagem e a mesma proporção de células: bactérias usadas para avaliações funcionais em um trabalho anterior10, sem a necessidade de superexpressar ou revogar a expressão de SLAMF1.
O protocolo fornecido envolve algumas etapas críticas. Uma delas é a corroboração da expressão de SLAMF1 em macrófagos humanos antes da realização dos ensaios de interação. Isso é crucial para evitar a obtenção de um resultado falso negativo, que na verdade seria devido à falta de expressão do receptor. Aqui, os níveis de SLAMF1 foram verificados por citometria de fluxo, pois é uma técnica rápida que permite obter esses dados em menos de 2 h. No entanto, outras técnicas, como microscopia ou Western blot, podem ser usadas. Não recomendamos metodologias como PCR em tempo real que identificam os níveis de mRNA quando o objetivo final é estudar um receptor no nível da proteína. Outra etapa fundamental é a etapa de reticulação. O uso de reticuladores busca estabilizar as interações proteína-proteína. Durante o ensaio baseado em citometria de fluxo, não fomos capazes de detectar um sinal para SLAMF1 sem aplicar a etapa de reticulação. No caso da abordagem microscópica, observamos interação sem o uso de tratamento de reticulação, mas com sensibilidade muito baixa. Portanto, a etapa de reticulação é altamente recomendada em todos os casos.
O protocolo descrito fornece evidências de interação entre os antígenos SLAMF1 e M. tuberculosis acessíveis na superfície do patógeno, ajudando a demonstrar que SLAMF1 reconhece uma assinatura molecular presente em M. tuberculosis. No entanto, este modelo apresenta algumas limitações. O ponto fraco é que esse protocolo não permite identificar qual antígeno específico do M. tuberculosis é o que interage com o SLAMF1. Apesar disso, o protocolo pode ser modificado para atingir esse fim, por exemplo, usando antígenos purificados da bactéria. Outra limitação é a necessidade de saber qual estímulo induz a expressão de SLAMF1 em macrófagos. Isso pode implicar uma complicação se o receptor escolhido não for expresso em condições basais ou para o qual o padrão de expressão é desconhecido. Finalmente, essas abordagens não discriminam se SLAMF1 requer outras moléculas para interagir com M. tuberculosis. Nesse caso, ensaios de imunoprecipitação poderiam ser realizados ou mais anticorpos poderiam ser usados no esquema metodológico.
A metodologia proposta neste relato pode ser facilmente adaptada para o estudo de outros receptores imunes, do mesmo receptor em outros tipos celulares em que se expressa, ou para avaliar a interação com outras bactérias ou cepas de diferentes micobactérias. Além disso, aqui, um protocolo curto e simples é fornecido para corar fluorescentemente M . tuberculosis sonicado, uma rotulagem que pode ser aplicada a outras bactérias e até mesmo cepas vivas. No entanto, este procedimento se concentra na diafonia SLAMF1-M. tuberculosis , outros usos potenciais incluem a compreensão do resultado de uma interação bloqueada, atrasada ou inadequada, estudando mecanismos de evasão ou revelando potenciais alvos moleculares envolvidos no reconhecimento de patógenos. Da mesma forma, este protocolo pode ser aplicado ao estudo de novas estratégias terapêuticas e imunoterapias para entender a coevolução de M. tuberculosis e macrófagos ou outras células hospedeiras e também pode ser usado em diferentes campos que buscam entender a dinâmica ligante-receptor.
Divulgações
Os autores declaram não haver conflitos de interesse.
Agradecimentos
Este trabalho foi apoiado pela Universidad Nacional del Noroeste de la Provincia de Buenos Aires (processo SIB 0618/2019, SIB 2582/2012 a V.P.), Agencia Nacional de Promoción Científica y Tecnológica (ANPCyT, processo PICT-2012-2459 e PICT A 2017-1896 a V.P. e PICT-2021-I-INVI-00584 a A.B.); Fundação Florencio Fiorini; e Consejo Nacional de Investigaciones Científicas y Técnicas (CONICET, processo número PIO 15720150100010CO para V.P.). Agradecemos a Natalia Menite e Gastón Villafañe pelo apoio técnico. Agradecemos à Dra. Paula Barrionuevo e à Dra. Luciana Balboa pela discussão científica durante a publicação que deu origem ao desenvolvimento dos ensaios apresentados neste trabalho. Por fim, agradecemos ao Dr. Estermann por seus conselhos sobre protocolos de reticulação, Lic. Morôni por seus conselhos sobre como trabalhar com Polilisina e Lic. Moriconi por sua ajuda com figuras esquemáticas.
Materiais
| Name | Company | Catalog Number | Comments |
| Alexa Fluor 488 secondary antibody | Invitrogen | A21121 | For fluorescence microscopy |
| anti-SLAMF1 FITC antibody | eBioscience | 11-1509-42 | For flow cytometry |
| anti-SLAMF1 PE antibody | BioLegend | 306308 | For flow cytometry |
| anti-SLAMF1 primary antibody | BioLegend | 306302 | For fluorescence microscopy |
| Aqua-Poly/Mount | Polysciences | 18606-20 | Mounting media |
| CD14 MicroBeads | Miltenyi Biotec | 130-097-052 | For monocytes isolation |
| Coverslips 12mm | HDA | - | For interaction assay by microscopy |
| EGS | ThermoFisher Scientific | 21565 | For crosslinking treatment |
| FACSCanto II | BD Biosciences | 338960 | Flow cytometer with BD FACSDiva software |
| Fetal Bovine Serum | Natocor | - | Inactivated and irradiated, for macrophages culture |
| Ficoll-Paque PLUS | Cytiva | 17144003 | For PBMCs separation |
| Fiji/ImageJ | Open Source software | - | For micrographs analysis |
| FlowJo 7.6.2 | Tree Star | - | For flow cytometry analysis |
| Formaldehyde | Merck | K47740803613 | For crosslinking treatment |
| Glass slides | Glass Klass | - | For interaction assay by microscopy |
| Glycine | Sigma | G8898 | For crosslinking treatment |
| Imager.A2 | Carl Zeiss | 430005-9901-000 | Fluorescence microscope with Colibri 7 illumination module |
| iMark | BIO-RAD | 1681130 | Microplate absorbance reader |
| L-glutamine | Sigma Aldrich | 49419 | For macrophages culture |
| M. tuberculosis, strain H37Rv, gamma-irradiated whole cells | BEI Resources, NIAID, NIH | NR-14819 | For interaction assay |
| M. tuberculosis, strain H37Rv, whole cell lysate | BEI Resources, NIAID, NIH | NR-14822 | For macrophages stimulation and interaction assay |
| Neofuge 13R | Heal Force | Neofuge 13R | High Speed Refrigerated Centrifuge for protein extraction |
| Penicillin/Streptomycin | Gibco | 15140122 | For macrophages culture |
| PMSF | ThermoFisher Scientific | 36978 | For proteins isolation |
| Poly-D-Lysine | Sigma Aldrich | A-003-M | For coverslips treatment |
| Protease Inhibitor Cocktail | Sigma Aldrich | P8340 | For proteins isolation |
| Rhodamine B | Sigma Aldrich | 21955 | For M. tuberculosis staining |
| RPMI 1640 | Gibco | 11875093 | For macrophages culture |
| Sorvall ST 16/16R centrifuge | ThermoFisher Scientific | 75004240 | For PBMCs and monocytes isolation |
Referências
- Global Tuberculosis Report 2023. , World Health Organization. Available at: https://www.who.int/tb/publications/global_report/en/ (2023).
- Ahmad, F., et al. Macrophage: A cell with many faces and functions in tuberculosis. Front Immunol. 13, 882130(2022).
- Bo, H., et al. Mycobacterium tuberculosis-macrophage interaction: Molecular updates. Front Cell Infect Microbiol. 13, 1187205(2023).
- Papp, A. C., et al. AmpliSeq transcriptome analysis of human alveolar and monocyte-derived macrophages over time in response to Mycobacterium tuberculosis infection. PLoS One. 13 (5), e0198069(2018).
- Zhai, W., Wu, F., Zhang, Y., Fu, Y., Liu, Z. The immune escape mechanisms of Mycobacterium tuberculosis. Int J Mol Sci. 20 (2), 340(2019).
- Van Crevel, R., Ottenhoff, T. H. M., Van der Meer, J. W. M. Innate immunity to Mycobacterium tuberculosis. Clin Microbiol Rev. 15 (2), 294-309 (2002).
- Mortaz, E., et al. Interaction of pattern recognition receptors with Mycobacterium tuberculosis. J Clin Immunol. 35 (1), 1-10 (2015).
- Stamm, C. E., Collins, A. C., Shiloh, M. U. Sensing of Mycobacterium tuberculosis and consequences to both host and bacillus. Immunol Rev. 264 (1), 204-219 (2015).
- Zihad, S. M. N. K., et al. Role of pattern recognition receptors in sensing Mycobacterium tuberculosis. Heliyon. 9 (10), e20636(2023).
- Barbero, A. M., et al. SLAMF1 signaling induces Mycobacterium tuberculosis uptake leading to endolysosomal maturation in human macrophages. J Leukoc Biol. 109 (8), 257-273 (2021).
- Pasquinelli, V., et al. Expression of signaling lymphocytic activation molecule-associated protein interrupts IFN-γ production in human tuberculosis. J Immunol. 172 (2), 1177-1185 (2004).
- Pasquinelli, V., et al. Phosphorylation of mitogen-activated protein kinases contributes to interferon-γ production in response to Mycobacterium tuberculosis. J Infect Dis. 207 (2), 340-350 (2012).
- Pasquinelli, V., et al. IFN-γ production during active tuberculosis is regulated by mechanisms that involve IL-17, SLAM, and CREB. J Infect Dis. 199 (5), 661-665 (2009).
- Pellegrini, J. M., et al. Neutrophil autophagy during human active tuberculosis is modulated by SLAMF1. Autophagy. 17 (2), 423-426 (2021).
- Song, T., Dong, C., Xiong, S. Signaling lymphocyte-activation molecule SLAMF1 augments mycobacteria BCG-induced inflammatory response and facilitates bacterial clearance. Int J Med Microbiol. 305 (6), 572-580 (2015).
- Eble, J. A. Titration ELISA as a method to determine the dissociation constant of receptor-ligand interaction. J Vis Exp. (132), e57032(2018).
- Patching, S. G. Surface plasmon resonance spectroscopy for characterization of membrane protein-ligand interactions and its potential for drug discovery. Biochim Biophys Acta Biomembr. 1838 (1), 43-55 (2014).
- Draczkowski, P., Matosiuk, D., Jozwiak, K. Isothermal titration calorimetry in membrane protein research. J Pharm Biomed Anal. 87, 296-304 (2014).
- Smith, D. S., Eremin, S. A. Fluorescence polarization immunoassays and related methods for simple, high-throughput screening of small molecules. Anal Bioanal Chem. 391 (5), 1499-1507 (2008).
- García-Nafría, J., Tate, C. G. Cryo-electron microscopy: Moving beyond X-ray crystal structures for drug receptors and drug development. Annu Rev Pharmacol Toxicol. 60, 51-71 (2020).
- Asami, J., Shimizu, T. Structural and functional understanding of the toll-like receptors. Protein Sci. 30 (4), 761-772 (2021).
- Phạm, T. T. T., Rainey, J. K. On-cell nuclear magnetic resonance spectroscopy to probe cell surface interactions. Biochem Cell Biol. 99 (6), 683-692 (2021).
- El Deeb, S., et al. Microscale thermophoresis as a powerful growing analytical technique for the investigation of biomolecular interaction and the determination of binding parameters. Methods Appl Fluoresc. 10 (4), 045001(2022).
- El Khamlichi, C., et al. Bioluminescence resonance energy transfer as a method to study protein-protein interactions: Application to G protein-coupled receptor biology. Molecules. 24 (3), 505(2019).
- Schneider, P., Willen, L., Smulski, C. R. Tools and techniques to study ligand-receptor interactions and receptor activation by TNF superfamily members. Methods Enzymol. 545, 103-125 (2014).
- Park, A. M. W., Schirmer, P. S. H. Atomic force microscopy: A multifaceted tool to study membrane proteins and their interactions with ligands. Biochim Biophys Acta Biomembr. 1838 (1), 74-89 (2014).
- Zalejski, J., Sun, J., Sharma, A. Unravelling the mystery inside cells by using single-molecule fluorescence imaging. J Imaging. 9 (9), 183(2023).
- Zheng, S., Zou, M., Shao, Y., Wu, H., Wang, X. Two-dimensional measurements of receptor-ligand interactions. Front Mol Biosci. 10, 1075587(2023).
- Zlotnikov, I. D., Kudryashova, E. V. Computer simulation of the receptor-ligand interactions of mannose receptor CD206 in comparison with the lectin concanavalin A model. Biochemistry (Moscow). 87 (1), 54-69 (2022).
- Fu, Y., Zhao, J., Chen, Z. Insights into the molecular mechanisms of protein-ligand interactions by molecular docking and molecular dynamics simulation: A case of oligopeptide binding protein. Comput Math Methods Med. 2018, 7604567(2018).
- Farina, B., et al. A novel approach for studying receptor-ligand interactions on living cells' surface by using NUS/T1ρ-NMR methodologies combined with computational techniques: The RGDechi15D-αvβ5 integrin complex. Comput Struct Biotechnol J. 19, 3303-3318 (2021).
- Driessen, N. N., et al. Role of phosphatidylinositol mannosides in the interaction between mycobacteria and DC-SIGN. Infect Immun. 77 (10), 4538-4547 (2009).
- Bansal, K., et al. Src homology 3-interacting domain of Rv1917c of Mycobacterium tuberculosis induces selective maturation of human dendritic cells by regulating PI3K-MAPK-NF-κB signaling and drives Th2 immune responses. J Biol Chem. 285 (47), 36511-36522 (2010).
- Xu, Y., et al. PPE57 induces activation of macrophages and drives Th1-type immune responses through TLR2. J Mol Med. 93 (6), 645-662 (2015).
- Olesen, R., et al. DC-SIGN (CD209), pentraxin 3 and vitamin D receptor gene variants associate with pulmonary tuberculosis risk in West Africans. Genes Immun. 8, 456-467 (2007).
- Berger, S. B., et al. SLAM is a microbial sensor that regulates bacterial phagosome functions in macrophages. Nat Immunol. 11 (10), 920-927 (2010).
- Degos, C., et al. Omp25-dependent engagement of SLAMF1 by Brucella abortus in dendritic cells limits acute inflammation and favours bacterial persistence in vivo. Cell Microbiol. 22 (4), e13156(2020).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoExplore Mais Artigos
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados