Method Article
Fluoreszenz-Assays zur Untersuchung der Interaktion von Mycobacterium tuberculosis mit dem Immunrezeptor SLAMF1
In diesem Artikel
Zusammenfassung
Diese Studie liefert ein Protokoll zur Bewertung der Interaktion von Mycobacterium tuberculosis mit dem mikrobiellen Sensor SLAMF1. Die Assays wurden an humanen Monozyten-abgeleiteten Makrophagen mittels Durchflusszytometrie und Fluoreszenzmikroskopie durchgeführt. Die beschriebenen Werkzeuge sind relevant für die Untersuchung von Wechselwirkungen zwischen Krankheitserregern und Immunrezeptoren.
Zusammenfassung
Die Bewertung der direkten Interaktion zwischen Krankheitserregern und Immunrezeptoren erfordert in der Regel ausgeklügelte Techniken oder impliziert den Einsatz von transgenen Stämmen und gentechnisch veränderten Zellen. Hier wird eine alternative Methode zum Nachweis der biochemischen Interaktion zwischen dem mikrobiellen Makrophagensensor SLAMF1 und Mycobacterium tuberculosis beschrieben. Es wurden zwei technische Ansätze entwickelt, die Durchflusszytometrie und Fluoreszenzmikroskopie. Es wurden Gesamtzellproteinextrakte aus menschlichen Makrophagen erzeugt, dann über Nacht bei 4 °C mit ganzen Zellen von M. tuberculosis (WCMtb) oder M. tuberculosis Antigenen (Mtb Ags) inkubiert und schließlich mittels Formaldehyd/Glycin/Ethylenglykol-Bis-Behandlung (Succinimidylsuccinat) vernetzt. Eine SLAMF1-Wechselwirkung mit WCMtb wurde mittels Durchflusszytometrie mit einem PE-spezifischen Anti-SLAMF1-Antikörper nachgewiesen. Das Vorhandensein einer Wechselwirkung durch Fluoreszenzmikroskopie wurde durch Anhängen von Rhodamin-PE-gefärbten Mtb-Ags an Poly-D-Lysin-beschichtete Objektträger durchgeführt, die mit dem Gesamtproteinextrakt aus Monozyten-abgeleiteten Makrophagen inkubiert wurden. Nach der Vernetzungsbehandlung wurde SLAMF1 mit primären (anti-SLAMF1) und sekundären (Alexa Fluor 488) Antikörpern sichtbar gemacht. Die Assays stellten ein starkes biochemisches Werkzeug zur Messung von Erreger-Immunrezeptor-Interaktionen dar und überwanden die Schwierigkeiten, die mit transgenen Zelllinien und Experimenten zur Modulation der Proteingenexpression verbunden sind.
Einleitung
Mycobacterium tuberculosis, der vor 142 Jahren identifizierte Tuberkulose-Erreger, ist nach wie vor eine globale Herausforderung und infiziert derzeit mindestens ein Viertel der Weltbevölkerung1. M. tuberculosis wird durch Tröpfchen in der Luft von infizierten Personen übertragen und gelangt in die Alveolarmakrophagen in der Lunge, wo es über lange Zeiträume in einem latenten Zustand überleben kann 2,3. Nicht nur wird die lokale makrophagische Reaktion aktiviert, sondern in den letzten Jahren wurde beschrieben, dass periphere Monozyten in die Atemwege rekrutiert werden und sich zu Alveolarmakrophagen differenzieren können, was eine noch robustere Reaktion gegen M. tuberculosis hervorruft als ihr Gegenstück fetalen Ursprungs 2,4.
Makrophagen sind grundlegende Akteure der angeborenen Immunität. Bei der Aufnahme von M. tuberculosis zeigen Makrophagen zahlreiche mikrobizide Funktionen, wie z.B. die Sekretion von proinflammatorischen Zytokinen, die Fusion des Phagolysosoms und die Aktivierung anderer Immunzellen, um M. tuberculosis abzutöten 2,3. Die komplexe architektonische Struktur dieses Erregers stellt jedoch Effektoren (z. B. Proteine und Lipide) zur Verfügung, die in der Lage sind, den Stoffwechsel und die Funktionen der Makrophagen und damit den Entzündungsprozess zu regulieren3. Diese Manipulation der Makrophagenreaktionen durch die Sekretion von Virulenzfaktoren oder die Ausnutzung von Wirtsfaktoren führt zu unterschiedlichen Fluchtstrategien von M. tuberculosis. Zu den wichtigsten Evasionsmechanismen gehören die Induktion von entzündungshemmenden Zytokinen, die Hemmung der Phagolysosomenreifung und -ansäuerung, Veränderungen des oxidativen Stresses, Störungen der Autophagie und Defizite während der Antigenprozessierung und -präsentation 3,5.
Eine streng regulierte Interaktion zwischen Makrophagen und M. tuberculosis ist entscheidend für die Entwicklung einer richtigen Immunantwort. Daher ist die Untersuchung dieser Synapsen der Schlüssel zur Identifizierung immunprotektiver oder immunpathogener Mechanismen, die durch Wirt-Pathogen-Crosstalk induziert werden, sowie zur Identifizierung potenzieller therapeutischer Ziele. Viele Rezeptoren vermitteln die Erkennung und/oder Internalisierung von M. tuberculosis, darunter TLRs 6,7,8, NLRs 7,8, Komplementrezeptoren 6,8, C-Typ-Lektinrezeptoren 6,8 und Scavenger-Rezeptoren 6,8. Immer mehr Daten deuten darauf hin, dass sowohl oberflächengebundene als auch intrazelluläre PRRs eine wichtige Rolle während der Infektion spielen, wobei opsonisierte und nicht-opsonisierte M. tuberculosis erkannt werden können.
Zihad und Sifat et al. haben kürzlich die Beteiligung der PRRs an M. tuberculosis-induzierten Reaktionen angeborener Zellen überprüft9. Insbesondere sind die meisten Mitglieder der TLR-Familie an der Interaktion mit M. tuberculosis-Liganden beteiligt 6,7. Surface TLR2 erkennt mykobakterielle Antigene wie acylierte Lipoproteine, 19-kDa-Lipoproteine, LprA-Lipoproteine, LprG-Lipoproteine, 30-kDa-Antigene, 38-kDa-Antigene, MymA, Prolin-Prolin-Glutaminsäure (PPE)-57, LAM, LM, PIM, Hitzeschockprotein 60, Signaturprotein Rv1509 und das sezernierte Protein ESAT-67. Membran TLR4 interagiert mit Hitzeschockproteinen, 38-kDa-Antigen, RpfE, Rv0652, Rv0335c, Rv2659c, Rv1738, Rv2627c, Rv2628, GrpE und HBHA. Auf der anderen Seite umfassen Liganden für endosomale TLRs (TLR 3, 7, 8 und 9) dsRNA, tRNA, ssRNA, phagosomale RNA und dsDNA von M. tuberculosis. Darüber hinaus spielt TLR2 eine aktive Rolle bei der Initiierung von Immunantworten, wenn es in Verbindung mit TLR1 und TLR6, TLR4 und TLR9wirkt 6,7. NOD2 und NLRP3 sind die am besten charakterisierten zytoplasmatischen NLRs bei Tuberkulose. Obwohl ihre spezifischen Liganden noch untersucht werden, werden diese Rezeptoren durch Muramyldipeptid bzw. ESAT-6 aktiviert 7,8. C-Typ-Lektinrezeptoren sind klassischerweise an der Endozytose von M. tuberculosis beteiligt. Während Dectin-2 als direkter PRR für ManLAM der M. tuberculosis-Zellwand wirkt, wurde der Dectin-1-Ligand bisher nicht entdeckt8. Mincle und MCL erkennen beide das Glykolipid Trehalose-6,6′-dimycolat (TDM), auch Nabelschnurfaktor7 genannt. DCAR interagiert mit den mykobakteriellen Glykolipiden Phosphatidyl-Myo-Inositol-Mannoside (PIMs)7. CR3 erkennt mykobakterielle LAM und PIM, DC-SIGN ligiert ManLAM, MR erkennt eine Reihe von M. tuberculosis-Komponenten, einschließlich ManLAM, PIM, LM, 38-kDa-Glykoprotein, 19-kDa-Antigen und andere mannosylierte Proteine, während der DCIR-Ligand noch nicht identifiziert ist.
Zu den löslichen CLRs gehören SP-A, das ManLam, LM, 60-kDa-Glykoprotein und Glykoprotein Apa erkennt; SP-D interagiert mit LAM, LM und PILAM; und MBL, das sich auf die ManLAM-Erkennungspezialisiert hat 6,7. Scavenger-Rezeptoren sind auch phagozytäre PRRs. SR-A und MARCO haben TDM als Liganden, SR-B1 erkennt ESAT-6 und CD36 bindet ManLAM und LM 7,8. Darüber hinaus können Dectin-1, Mincle und MARCO auch mit TLR2 oder TLR4 kombiniert werden, um Signale nach dem Nachweis von M. tuberculosis PAMPs 6,7 auszulösen. CD14 ist ein Oberflächenrezeptor, der die Fähigkeit hat, nicht-opsonisierte Bakterien zu internalisieren und auch das Hitzeschockprotein Chaperonin 60.1 erkennt. Insbesondere fungiert CD14 zusammen mit MARCO und TLR2 als Co-Rezeptor6. AIM2 ist ein zytosolischer Rezeptor, der ssDNA erkennen kann, wenn M. tuberculosis aus dem Phagosomentweicht 8. Schließlich ist AhR ein ligandenaktivierter Transkriptionsfaktor, der den pigmentierten Virulenzfaktor Naphthochinon Phthiocol von M. tuberculosisbindet 6.
Viele der oben beschriebenen Wechselwirkungen wurden postuliert und nicht streng nachgewiesen. Selbst die Liganden für bestimmte Rezeptoren sind noch unbekannt, was die Notwendigkeit verstärkt, das Gebiet der Immunerkennung von M. tuberculosis besser zu verstehen. In diesem Zusammenhang wurde kürzlich das kostimulatorische Molekül SLAMF1 (Signaling Lymphocytic Activation Molecule) von Barbero et al.10 als M. tuberculosis-Rezeptor beschrieben. Da SLAMF1 nicht nur als Signalmolekül, sondern auch als M. tuberculosis-Sensor fungiert, spielt es eine besonders interessante Rolle bei der Tuberkulose. SLAMF1 kann die Aktivierung von Immunzellen induzieren, indem es Schutzfunktionen wie die Produktion von IFN-γ durch T-Zellen durch Erk/CREB-Phosphorylierung 11,12,13, Autophagie in Neutrophilen14 und bakterielle Clearance in Makrophagen15 moduliert.
Rezeptor-Liganden-Wechselwirkungen wurden im Laufe der Jahre mit Techniken wie ELISA, Oberflächenplasmonenresonanz (SPR), isothermer Titrationskalorimetrie (ITC), Fluoreszenzpolarisation (FP), Röntgenkristallographie, Kernspinresonanzspektroskopie (NMR), mikroskaliger Thermophorese (MST), Resonanzenergietransfer (z. B. BRET oder FRET) konfokaler Mikroskopie, Elektronenmikroskopie, Kryo-Elektronenmikroskopie (Kryo-EM) und Rasterkraftmikroskopie (AFM) untersucht16,17, 18,19,20,21,22,23,24,25,26,27,28. Einige dieser Ansätze implizieren die Verwendung von Reportergenen, markierten rekombinanten Proteinen oder chimären Molekülen, Knockout-, Knockdown- oder Überexpressionsmodellen. Alternativ können computergestützte Werkzeuge Rezeptor-Liganden-Wechselwirkungen und Bindungsstellen vorhersagen und werden häufig in Kombination mit biologischen Ansätzen verwendet, um ein umfassendes Verständnis der Wechselwirkungen zu erlangen 29,30,31. Hier werden zwei Alternativen zum Nachweis biochemischer Wechselwirkungen und auch ein Abschnitt zur fluoreszierenden Markierung von Bakterien beschrieben. Es wird ein Protokoll vorgestellt, das die Untersuchung von Rezeptor-Pathogen-Wechselwirkungen in vitro ermöglicht, insbesondere die Bewertung des SLAMF1-M. tuberculosis-Engagements durch Durchflusszytometrie und Fluoreszenzmikroskopie, zwei üblicherweise verfügbare und routinemäßig verwendete Techniken.
Protokoll
Alle Verfahren mit humanen Monozyten-abgeleiteten Makrophagen wurden in Übereinstimmung mit der Deklaration von Helsinki (2013) und in Übereinstimmung mit der Ethikkommission der UNNOBA (COENOBA) durchgeführt. Vor der Probenentnahme wurde eine schriftliche Einverständniserklärung eingeholt. Die Verteilung der männlichen/weiblichen Gruppe betrug 13/6 und das mediane Alter 32 Jahre, mit einem Interquartilsabstand (IQR) von 18-75 Jahren. Das Vorliegen früherer Pathologien, Komorbiditäten oder eine positive Diagnose für Tuberkulose wurden als Ausschlusskriterien definiert. Die Details zu den Reagenzien und Geräten sind in der Materialtabelle aufgeführt.
1. Aus Monozyten gewonnene Makrophagenkultur und Stimulation
- Blutabnahme
- Gewinnen Sie 60 ml Blut von gesunden Spendern durch Venenpunktion in einer heparinisierten Spritze.
HINWEIS: Die Blutentnahme muss von einem Biochemiker oder Phlebotomietechniker durchgeführt werden. - Entsorgen Sie die Nadel in den Entsorgungsbehälter für scharfe Gegenstände und sämtliches Material, das mit dem Blut der Spender in Berührung gekommen ist, in den Behälter für pathologische Abfälle.
- Gewinnen Sie 60 ml Blut von gesunden Spendern durch Venenpunktion in einer heparinisierten Spritze.
- Monozytenisolierung und Makrophagengenerierung
- Füllen Sie das Blut vorsichtig in 50-ml-Röhrchen und verdünnen Sie es mit Kochsalzlösung zur Hälfte.
- Führen Sie eine Zentrifugation über einem Dichtegradientenmedium durch, um mononukleäre Zellen (PBMCs) des peripheren Blutes zu erhalten.
- Sammle die PBMCs aus dem weißlichen Halo.
- Führen Sie zwei Waschgänge mit Kochsalzlösung bei 400 x g für 10 min und eine letzte Wäsche bei 200 x g für 15 min bei 20 °C durch, um Blutplättchen zu entfernen.
- Bestimmen Sie die Anzahl der PBMCs in einer Zellzählkammer und führen Sie eine CD14-positive magnetische Selektion mit CD14-Kügelchen durch.
HINWEIS: Befolgen Sie bei der magnetischen Auswahl sorgfältig die Anweisungen des Herstellers. Zusätzlich kann man die Reinheit von isolierten Monozyten mittels Durchflusszytometrie überprüfen. - Resuspendieren Sie die isolierten Zellen in RPMI 1640 und bestimmen Sie die Anzahl der Monozyten in einer Zellzählkammer.
- 500 μl von 1 × 106/ml CD14-positiven ausgewählten Monozyten pro Well in einer 24-Well-Zellkulturplatte in Abwesenheit des Serums aussäen, um die Adhärenz zu fördern.
- Entfernen Sie nach 2 Stunden nicht anhaftende Zellen, indem Sie sie mit vorgewärmtem RPMI 1640 waschen.
- Zur Unterscheidung von Makrophagen kultivieren Sie die adhärenten Monozyten für weitere 16-18 Stunden (über Nacht) in 1 ml vollständigem Medium (RPMI 1640 Medium, ergänzt mit L-Glutamin, 10 % fötales Rinderserum (FBS), 100 U/ml Penicillin und 100 μg/ml Streptomycin).
- Wechseln Sie am nächsten Tag das Kulturmedium. Entfernen Sie das Medium, waschen Sie es mit 1 mL 1x PBS und fügen Sie 1 mL des vollständigen Mediums hinzu.
- Stimulieren Sie die Makrophagen mit beschalltem M. tuberculosis (M. tuberculosis Antigene (Ags)) für 24 h. Es werden 10 μl M . tuberculosis Ags (10 μl = 1 × 106 Bakterien) pro 1 × 106 Monozyten-abgeleitete Makrophagen verwendet.
HINWEIS: 10 μl M. tuberculosis Ags (Mtb Ags) = 1 × 106 Bakterien, quantifiziert durch O.D. 600nm im Spektralphotometer unter Berücksichtigung eines O.D. von 1 als 1 x 108 Bakterien/ml. Verwenden Sie für alle Kulturschritte BSL2-Biosicherheitswerkbänke und einen Inkubator bei 37 °C in einer Atmosphäre mit 5 % CO2 .
- Bestätigung der SLAMF1-Expression
- Sammeln Sie die Makrophagen aus einer einzigen Vertiefung. Stellen Sie die Kulturplatte auf eine kalte Oberfläche. Trennen Sie die Zellen durch Auf- und Abbewegungen, indem Sie zweimal hintereinander 1 ml kaltes FACS (1x PBS + 2% FBS) hinzufügen. Verwenden Sie für die Entnahme ein Zytometrie-Röhrchen mit rundem Boden.
- Die Zellen bei 500 x g für 5 min bei 4 °C zentrifugieren. Entsorge den Überstand, indem du ihn entwirfst.
- Färben Sie die Zellen mit einem Fluorophor-gekoppelten Anti-Human-Antikörper SLAMF1 für 30 min bei 4 °C im Dunkeln. Vortex nach Zugabe von 1,25 μl des Antikörpers.
- Waschen Sie die Zellen, um den Überschuss an Antikörpern mit FACS zu beseitigen, resuspendieren Sie das Pellet in FACS und entnehmen Sie die Probe mit einem Durchflusszytometer.
HINWEIS: 1,25 μl Anti-SLAMF1-Antikörper entsprechen 0,0625 μg, die zur Markierung von 0,5 x 106 Makrophagen verwendet werden. Es wird jedoch dringend empfohlen, den Antikörper zu titrieren, ebenso wie die Durchführung des entsprechenden Isotyps oder der Fluoreszenz-Minus-Eins-Kontrolle (FMO).
2. Zubereitung des Gesamtzellproteinextrakts
- Vorbereitung des Lysepuffers
- Bereiten Sie einen RIPA-Puffer (Radio Immunoprecipitation Assay) mit NaCl 150 mM, Tris 10 mM, EDTA 5 mM, SDS 1 %, Triton X-100 1 % und Natriumdesoxycholat 1 % vor.
- Ergänzen Sie den RIPA-Puffer mit 1 mM Phenylmethylsulfonylfluorid (PMSF) und Proteaseinhibitoren wie Pepstatin A und Leupeptin.
- Zellentnahme und Proteinisolierung
- Entsorgen Sie das komplette RPMI mit einer P1000-Pipette aus den Plattenvertiefungen und ernten Sie Makrophagen durch Auf- und Abpipettieren mit kaltem 1x PBS.
- Übertragen Sie die Zellen in 1,5 mL Mikrozentrifugenröhrchen. Sammeln Sie eine Vertiefung pro Mikroröhrchen.
- Zentrifugieren Sie bei 500 x g für 5 min bei 4 °C. Entsorgen Sie den Überstand.
- Fügen Sie 1 ml kaltes PBS hinzu und wiederholen Sie die Schritte 2.2.2 und 2.2.3, indem Sie die Zellen im selben Mikrozentrifugenröhrchen sammeln.
- Resuspendieren Sie die Zellpellets in 100 μl 1x PBS und poolen Sie vier Röhrchen. Wiederholen Sie Schritt 2.2.3. Jedes Röhrchen enthält 2 x 106 Makrophagen.
- Fügen Sie 100 μl supplementierten RIPA-Puffer hinzu und inkubieren Sie die Suspension 1 h lang auf Eis, wobei Sie alle 10 Minuten vortexen.
- Zentrifugieren bei 14.000 x g für 15 min. Den Überstand auffangen und bis zur Verwendung bei -20 °C aufbewahren.
3. M . tuberculosis-SLAMF1-Wechselwirkung durch Durchflusszytometrie
- Proteine-Bakterien-Vernetzung
- 1 × 106 ganze inaktivierte M. tuberculosis-Zellen (WCMtb, M. tuberculosis durch Gammabestrahlung inaktiviert) mit Proteinextrakt von 1 x 106 Makrophagen (50 μL) über Nacht bei 4 °C in einem rotierenden Mikroröhrchenhalter inkubieren.
HINWEIS: 10 μl WCMtb = 1 × 106 Bakterien, quantifiziert durch einen Außendurchmesser von 600nm im Spektralphotometer unter Berücksichtigung eines Außendurchmessers von 1 als 1 x 108 Bakterien/ml. - Am nächsten Tag fügen Sie 500 μL 1% Formaldehyd zu, verdünnt in 1x PBS. 15 min unter Rühren (Shaker) bei Raumtemperatur (RT) inkubieren.
- Fügen Sie 25 μl 0,125 M Glycin in Wasser verdünnt hinzu. 5 min unter Rühren (Shaker) bei RT inkubieren.
- Zweimal mit 1 mL 1x PBS bei 14.000 x g für 5 min waschen. Entsorgen Sie den Überstand zwischen den Waschschritten.
- Resuspendieren Sie das Pellet und inkubieren Sie die Suspension in 500 μl 2 mM Ethylenglykol-Bis-(Succinimidylsuccinat) (EGS)-Vernetzer, verdünnt in einer 1:1-Mischung aus Eisessig: Wasser für 1 h bei RT.
HINWEIS: Die Vorbereitung der Reagenzien zum Zeitpunkt der Anwendung wird dringend empfohlen. Zur Herstellung des EGS müssen Eisessig und Wasser zu gleichen Anteilen vermischt und auf 70 °C erhitzt werden. Wenn die Lösung nicht heiß ist, wird das EGS nicht vollständig resuspendiert.
- 1 × 106 ganze inaktivierte M. tuberculosis-Zellen (WCMtb, M. tuberculosis durch Gammabestrahlung inaktiviert) mit Proteinextrakt von 1 x 106 Makrophagen (50 μL) über Nacht bei 4 °C in einem rotierenden Mikroröhrchenhalter inkubieren.
- SLAMF1-Färbung
- Wiederholen Sie Schritt 3.1.4 mit 500 μl 1x PBS in der ersten Wäsche und 1 mL in der zweiten Wäsche.
- Färben Sie den Protein-Bakterien-Komplex für 30 min bei 4 °C im Dunkeln mit einem Anti-Human-SLAMF1-Antikörper (wie in Schritt 1.3.3), indem Sie den Antikörper in das Mikroröhrchen geben und vortexen. Verwenden Sie die für 1 x 106 Zellen (2,5 μL oder 0,125 μg) geeignete Antikörpermenge.
- Waschen Sie die Zellen, um den Überschuss an Antikörpern mit FACS zu eliminieren, resuspendieren Sie das Pellet in FACS und entnehmen Sie die Probe in einem Durchflusszytometer (wie in Schritt 1.3.4).
HINWEIS: Führen Sie die entsprechende Isotyp- oder FMO-Steuerung durch.
4. Markierung von M. tuberculosis-Antigenen
- Resuspendieren Sie Rhodamin B in Ethanol in einer Stammkonzentration von 5 mg/ml (1000x). Bis zur Verwendung bei -20 °C lagern.
- 1 μl Rhodamin B pro 100 μl M. tuberculosis Ags in ein 1,5 ml-Mikrozentrifugenröhrchen geben.
- 45 min bei RT unter kontinuierlichem Rühren (Vortex) inkubieren.
- Mindestens 3 Mal bei 14.000 x g für 5 min bei 24 °C mit 1 mL 1x PBS waschen, bis der Überstand transparent ist. Das Pellet (Mtb-R Ags) sieht hellrosa aus.
- Resuspendieren Sie das Mtb-R Ags in 100 μl 1x PBS, um das ursprüngliche Volumen wiederherzustellen.
HINWEIS: Es wird empfohlen, unter sterilen Bedingungen zu arbeiten. Stellen Sie sicher, dass die Färbung unmittelbar vor der Durchführung von Experimenten durchgeführt wird.
5. M . tuberculosis-SLAMF1-Interaktion durch Fluoreszenzmikroskopie
- Bakterien-Protein-Vernetzung
- 12 mm runde Deckgläser mit einer chirurgischen Pinzette mit runder Nase in 24-Well-Kulturplatten (1 Deckglas pro Well) einführen.
HINWEIS: Reinigen Sie die Deckgläser gründlich mit 70% Ethanol. Auf Wunsch können sie autoklaviert oder unter UV-Licht für 30 min sterilisiert werden. - Inkubieren Sie das Deckglas mit 400 μl 10 μg/ml Poly-D-Lysin über Nacht bei 4 °C. Decken Sie die Platte mit Alufolie ab, um sie vor Licht zu schützen.
- Nehmen Sie am nächsten Tag die Platte aus dem Kühlschrank und waschen Sie das Deckglas zweimal mit 1 ml Wasser.
- Lassen Sie das Deckglas trocknen.
- 20 μl (2 x 106) Mtb-R Ags in 180 μl 1x PBS verdünnen. Die letzten 200 μl reichen aus, um ein Deckglas vollständig abzudecken.
- Inkubieren Sie das Deckglas mit Mtb-R Ags für 1 h bei 37 °C in einem Zellinkubator.
- Entsorgen Sie die restliche Flüssigkeit und waschen Sie sie zweimal mit 1 ml 1x PBS.
HINWEIS: In diesem Schritt kann die korrekte Haftung von Mtb-R auf dem Deckglas mit einem Fluoreszenzmikroskop unter Verwendung der Filter überprüft werden, die dem Rotkanal entsprechen. - 400 μl Blockierungspuffer (10 % FBS in 1x PBS) für 30 min bei RT in Bewegung (Schüttler) zugeben.
- Zweimal mit 400 μl Blockierungspuffer waschen.
- Inkubieren Sie mit 100 μl Proteinextrakt, verdünnt in 300 μl 1x PBS für 2 h bei RT in Bewegung (Shaker).
- Fügen Sie 400 μL 1% Formaldehyd zu, verdünnt in 1x PBS. 15 min unter Rühren (Shaker) bei RT inkubieren.
- 500 μl Glycin 0,125 M in Wasser verdünnt zugeben. 5 min unter Rühren (Shaker) bei RT inkubieren.
- Zweimal mit 1 mL 1x PBS waschen.
- 400 μl 2 mM EGS für 1 h bei RT in Bewegung (Shaker) geben.
- Schritt 5.1.13 wiederholen
HINWEIS: Die Vorbereitung der Reagenzien zum Zeitpunkt der Anwendung wird dringend empfohlen.
- 12 mm runde Deckgläser mit einer chirurgischen Pinzette mit runder Nase in 24-Well-Kulturplatten (1 Deckglas pro Well) einführen.
- SLAMF1-Färbung
- Wickeln Sie den Deckel der Kulturplatte in Paraffinfolie ein.
- Geben Sie einen Tropfen mit 60 μl eines zuvor titrierten Anti-SLAMF1-Primärantikörpers (1:75 Verdünnung) auf den mit Paraffinfilm bedeckten Deckel.
- Entfernen Sie das Deckglas vorsichtig mit einer gebogenen chirurgischen Pinzette und Nadeln mit feiner Spitze von der Platte.
HINWEIS: Um das Entfernen von Deckgläsern zu erleichtern, kann die Nadel in einem Winkel von ca. 45° gebogen werden. - Drehen Sie das Deckglas über den Tropfen, der den Antikörper enthält. 30 min bei RT im Dunkeln inkubieren.
- Heben Sie den Deckglas an und waschen Sie ihn zweimal mit dem Blockpuffer.
- Drehen Sie das Deckglas über einen neuen Tropfen, der den Sekundärantikörper enthält (1:200 Verdünnung). 30 min bei RT im Dunkeln inkubieren.
- Wiederholen Sie Schritt 5.2.5.
- Entfernen Sie die überschüssige Flüssigkeit und montieren Sie das Deckglas auf einen Tropfen Montageflüssigkeit, der auf einen Objektträger gelegt wird.
- Betrachten Sie unter dem Fluoreszenzmikroskop mit geeigneten Filtern.
HINWEIS: Es wird empfohlen, unter sterilen Bedingungen zu arbeiten. Verdünnen Sie die Antikörper im Blockierungspuffer. Gehen Sie vorsichtig mit dem Deckglas um, um Kratzer zu vermeiden, die die Beobachtung unter dem Mikroskop behindern könnten. Eine für die Fluoreszenz geeignete Einbettflüssigkeit wird dringend empfohlen.
Ergebnisse
In dieser Arbeit wird ein Protokoll zur Verfügung gestellt, das die Bewertung der Interaktion von M. tuberculosis mit dem Immunrezeptor SLAMF1 in humanen Makrophagen ermöglicht (Abbildung 1). Zu diesem Zweck wurde peripheres Blut von gesunden Spendern gewonnen. Anschließend wurden die PBMCs durch Zentrifugation über dem Dichtegradientenmedium getrennt und die Monozyten durch magnetische positive Selektion isoliert (≥95% Reinheit, Abbildung 2A,B). Die isolierten Monozyten wurden 2 Stunden lang auf plastische Kulturplatten geklebt, um von Monozyten abgeleitete Makrophagen zu erhalten, und dann ON kultiviert. Danach wurden Makrophagen mit beschallter M. tuberculosis (Mtb Ags) stimuliert, um die SLAMF1-Oberflächenexpression zu induzieren, wie vor10 berichtet, und die Oberflächen-SLAMF1-Spiegel wurden durch Durchflusszytometrie bestätigt (Abbildung 2C). Die Makrophagen wurden schließlich mit RIPA-Lysepuffer in Kombination mit Vortexing und Inkubation auf Eis lysiert, um die Zelllyse zu erleichtern. Der erzeugte Gesamtproteinextrakt wurde in den Interaktionsassays verwendet.
In der ersten Reihe von experimentellen Verfahren wurde die Durchflusszytometrie eingesetzt, um die Interaktion von SLAMF1 mit ganzen Zellen von M. tuberculosis (WCMtb) zu untersuchen (Abbildung 3). Zunächst wurde die physikalische Wechselwirkung zwischen SLAMF1 (Gehalt im Proteinextrakt) und WCMtb gefördert, indem sie zusammen ON in kontinuierlicher Rotation bei 4 °C inkubiert wurden. Anschließend wurde eine Cross-Linking-Behandlung durchgeführt und unter Verwendung eines spezifischen Anti-SLAMF1-Antikörpers der SLAMF1-Rezeptor nachgewiesen, der an WCMtb gebunden war. In diesem Assay wurde WCMtb allein verwendet, um die Spannungen entsprechend der Größe und Granularität von M. tuberculosis im Durchflusszytometer einzustellen, das die Population von Interesse war, in der die SLAMF1-Bindung nachgewiesen werden sollte.
Im zweiten experimentellen Ansatz wurde die Fluoreszenzmikroskopie eingesetzt, um die Interaktion zwischen SLAMF1 und M tuberkulose nachzuweisen (Abbildung 4). Dieser Assay, bei dem beschallte M. tuberculosis (Mtb Ags) verwendet wurde, ergänzt den vorhergehenden. Rhodamin B-gefärbte Mtb-Ags (Mtb-R Ags) wurden verwendet, um die Fluoreszenzdetektion im Kanal/Filter, der dem PE-Fluorochrom entspricht, zu erleichtern. Mtb-R Ags wurden auf Poly-D-Lysin-beschichtete Deckgläser aufgebracht, und die erfolgreiche Färbung wurde durch mikroskopische Beobachtung überprüft (Abbildung 4A). Danach wurden die mit Mtb-R Ags beschichteten Objektträger mit den SLAMF1-haltigen Proteinextrakten inkubiert. Nach der Vernetzungsbehandlung wurde SLAMF1 unter Verwendung eines primären spezifischen Anti-SLAMF1-Antikörpers nachgewiesen, gefolgt von einem sekundären Antikörper, der an ein kompatibles Fluorochrom gekoppelt war, um zusammen mit PE beobachtet zu werden. Nach der Aufnahme einzelner fluoreszierender mikroskopischer Bilder in den beiden Kanälen für Mtb-R Ags und SLAMF1 wurde die Wechselwirkung durch Bildzusammenführung bestätigt (Abbildung 4B).
Die Durchführung dieser Ansätze zeigte die Existenz einer Interaktion zwischen SLAMF1 und M. tuberculosis. Es wurde die Wechselwirkung von SLAMF1 sowohl mit ganzen als auch mit beschallten Bakterien nachgewiesen, was die Hypothese stützte, dass SLAMF1 ein neuer Rezeptor für M. tuberculosis10 ist.

Abbildung 1: Arbeitsablauf zum Nachweis der SLAMF1-M. tuberculosis-Interaktion. Peripheres Blut wurde von gesunden Spendern durch Venenpunktion gewonnen und über ein Dichtegradientenmedium zentrifugiert, um mononukleäre Zellen des peripheren Blutes (PBMCs) zu isolieren. Nach Bestimmung der Anzahl der Zellen wurden die PBMCs einer positiven magnetischen Selektion unterzogen, um CD14-pos-Monozyten zu reinigen. Monozyten wurden dann 2 Stunden lang in Abwesenheit von fötalem Rinderserum auf Kulturplatten geklebt und dann über Nacht in supplementiertem RPMI (Ruheschritt) kultiviert, um von Monozyten abgeleitete Makrophagen zu erhalten. Danach wurden Makrophagen 24 h lang mit beschalltem M. tuberculosis (Mtb Ags) stimuliert, um die SLAMF1-Oberflächenexpression zu induzieren. Die SLAMF1-Spiegel wurden mittels Durchflusszytometrie unter Verwendung eines humanen Anti-SLAMF1-spezifischen Antikörpers in einer Fraktion der Makrophagenpopulation getestet. Die restlichen Makrophagen wurden verwendet, um Gesamtproteinextrakte zu erhalten. Die Zelllyse wurde durch die Kombination von Vortexing und Inkubationen auf Eis in einem Lysepuffer durchgeführt. Schließlich wurde die Interaktion zwischen SLAMF1 und M. tuberculosis mittels Durchflusszytometrie und Fluoreszenzmikroskopie nach Vernetzung von Makrophagenproteinen mit Mtb Ags oder ganzen Zellen von M. tuberculosis (WCMtb) untersucht. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.

Abbildung 2: Experimentelle und technische Kontrollen. (A) CD14-pos-Monozyten wurden durch positive magnetische Selektion aus PBMCs isoliert. Die resultierenden positiven (Monozyten, blau) und negativen (roten) Zellfraktionen wurden durchflusszytometrisch ausgewertet (SSC-A vs. FSC-A), um eine erfolgreiche Auswahl zu bestätigen. (B) Die Reinheit der positiven Fraktion wurde durch Durchflusszytometrie unter Analyse der Zellgranularität und -größe bestätigt (SSC-A vs. FSC-A). (C) Monozyten-abgeleitete Makrophagen wurden durch SSC-A vs. FSC-A und dann durch eine Double-Singulett-Strategie zum Ausschluss von Dubletten (FSC-A vs. FSC-H gefolgt von SSC-A vs. SSC-H). Die Oberflächenexpression von SLAMF1 wurde in einzelnen Zellen ausgewertet. In allen Fällen (A-C) werden repräsentative Diagramme oder Histogramme angezeigt. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
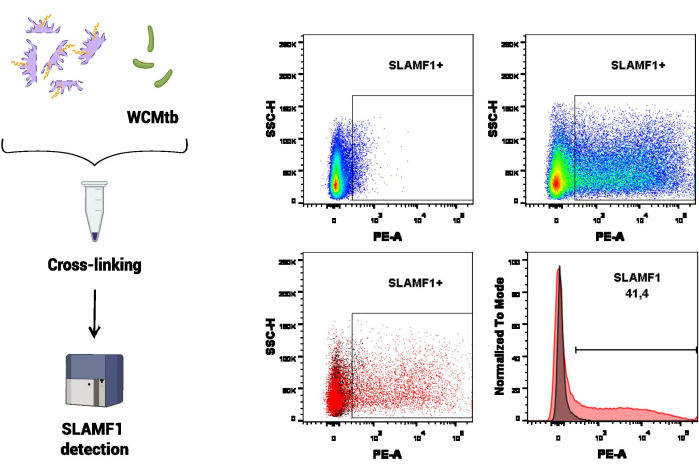
Abbildung 3: SLAMF1-M. tuberkulose-Interaktion mittels Durchflusszytometrie. Die Vernetzung zwischen Makrophagenproteinen und ganzen Zellen von M. tuberculosis (WCMtb) erfolgte durch Behandlung mit Formaldehyd/Glycin/Ethylenglykol bis, wie im Protokollabschnitt beschrieben. Anschließend wurde die Interaktion mittels Durchflusszytometrie bewertet, indem ein positives SLAMF1 (PE)-Signal nach Auswahl der interessierenden Population (SSC-A vs. FSC-A). Das obere Bild zeigt ein positives Signal für SLAMF1, wobei die Fluoreszenz-Minus-Eins-Kontrolle (FMO) (links) mit der positiven Färbung (rechts) verglichen wird. Das untere Feld zeigt die Überlagerung (links, Punktdiagramm und rechts, Histogramm) zwischen dem SLAMF1-Signal und der FMO-Steuerung. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.

Abbildung 4: SLAMF1-M. tuberculosis-Interaktion mittels Fluoreszenzmikroskopie. (A) Mtb-Ags wurden mit Rhodamin B (Mtb-R Ags) gefärbt und auf einem runden Deckglas befestigt. Die korrekte Adhäsion und Fluoreszenz von Mtb-R Ags wurde durch Fluoreszenzmikroskopie mit Filtern bestätigt, die dem roten Kanal entsprechen. (B) Die Vernetzung zwischen Makrophagenproteinen und Mtb-R-Ags erfolgte durch Behandlung mit Formaldehyd/Glycin/Ethylenglykol-Bis, wie im Protokollabschnitt beschrieben, durch Zugabe des Proteinextrakts über die angehängten Mtb-R-Ags. Die Interaktion wurde durch Fluoreszenzmikroskopie mit einem humanen Anti-SLAMF1-Antikörper untersucht, gefolgt von einem Alexa Fluor 488-markierten Sekundärantikörper. Die oberen Mikroaufnahmen zeigen positive Signale von SLAMF1 (links, grün) und Mtb-R Ags (rechts, rot). Die niedrige Mikroskopaufnahme zeigt die Wechselwirkung in gelb (Verschmelzung beider Kanäle). Die Maßstabsbalken entsprechen 10 μm. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Diskussion
Diese Studie bietet einen nützlichen Leitfaden für die Untersuchung der biochemischen Interaktion zwischen M. tuberculosis und mikrobiellen Sensoren, die in menschlichen Makrophagen exprimiert werden, einem Schlüsselzelltyp, der an der Wirtsreaktion während der Tuberkulose beteiligt ist. Die bereitgestellten Protokolle werden relevant sein, um Moleküle zu entschlüsseln, die eine Rolle beim Eintritt von M. tuberculosis in Phagozyten spielen.
Die Charakterisierung biomolekularer Interaktionen, wie z. B. zwischen Krankheitserregern und Immunrezeptoren, ist entscheidend für das Verständnis sowohl der immunprotektiven Mechanismen als auch der Evasionsstrategien, die durch M. tuberculosis ausgelöst werden. Oft kann der Nachweis einer direkten Interaktion des Rezeptors mit seinem bakteriellen Liganden komplex sein und ausgeklügelte Techniken oder Systeme erfordern, denen es an Vielseitigkeit mangelt. Driessen et al. untersuchten die Rolle von Phosphatidylinositol-Mannoside in der Interaktion zwischen Mykobakterien und DC-SIGN unter Verwendung mutierter defizienter Stämme von M. bovis BCG32. Obwohl es ihnen gelang, diese Wechselwirkungen zu untersuchen, fanden die Autoren keine Unterschiede für die mutierten Stämme und diskutierten, dass die Schaffung von mutierten Bakterien, die eine verminderte Bindung zeigen, eine enorme Aufgabe darstellen würde32. Andere Autoren haben hervorragende und mühsame Arbeit geleistet, um die Interaktion zwischen TLR2 und einigen PE/PPE-Proteinen von M. tuberculosis zu demonstrieren 33,34. Diese Studien erforderten die Verwendung von Knockout-Mäusen für den Rezeptor, die Transfektion von Zellen und die Reinigung der oben genannten Proteine oder bestimmter Proteindomänen33,34. Darüber hinaus wird oft auf die Funktion der Rezeptoren geschlossen, aber die Wechselwirkung wird nicht zuverlässig nachgewiesen. Ein Beispiel ist der lösliche Rezeptor PTX3, für den Haplotypen gefunden wurden, die mit dem Krankheitsverlauf assoziiert sind, dessen spezifische Interaktion mit M. tuberculosis jedoch nicht streng untersucht wurde35. In Bezug auf diese Unzulänglichkeiten ermöglicht unsere Methode, die Interaktion des untersuchten Zielrezeptors mit M. tuberculosis auf einfache Weise und mit zugänglichen Techniken der einfachen Interpretation zu bewerten.
Hinsichtlich der vorstehenden Punkte, und insbesondere für SLAMF1, wurden zuvor die Wechselwirkungen mit OmpC und OmpF aus E. coli36 und Omp25 aus B. abortus37 evaluiert. Degos et al.37 verwendeten COS-7-Zellen, die mit einem Plasmid transfiziert wurden, das nur für die extrazelluläre Domäne von SLAMF1 kodiert, um dann SLAMF1 zu reinigen und die Interaktion mit Omp25 durch Western Blot zu analysieren. Sie führten auch Experimente mit SLAMF1-/- Mäusen und Wildtyp B. abortus oder einer Omp25-defekten Mutante durch. Berger et al. 36 Arbeiten zeigten auch überzeugende Beweise für eine direkte Interaktion zwischen SLAMF1 und E. coli Omp und S. typhimurium SseB-. Sie verwendeten verschiedene Ansätze, darunter SLAMF1-/- Mäuse, RAW264.7-Makrophagen, die für ein SLAMF1mCherry-Konstrukt kodieren, sowie einen Amplifikationsassay mit SLAMF1-transfizierten Jurkat-T-Zellen. Sie entwickelten einen sensitiven Signalamplifikationsassay mit einem Luciferase-Reporter unter Verwendung von 1 x 108 E. coli. In dieser Hinsicht besteht eine Stärke der in diesem Bericht vorgestellten biochemischen Interaktionsassays darin, dass die Interaktion tatsächlich durch die Verwendung von Wildtyp-Zellen und dem gleichen Verhältnis von Zellen zu Bakterien gefunden wurde, das in einer früheren Arbeit10 für funktionelle Bewertungen verwendet wurde, ohne dass die SLAMF1-Expression überexprimiert oder aufgehoben werden musste.
Das bereitgestellte Protokoll umfasst einige kritische Schritte. Eine davon ist die Bestätigung der SLAMF1-Expression in humanen Makrophagen vor der Durchführung der Interaktionsassays. Dies ist entscheidend, um ein falsch negatives Ergebnis zu vermeiden, das eigentlich auf einen Mangel an Rezeptorexpression zurückzuführen wäre. Hier wurden die SLAMF1-Spiegel mit Hilfe der Durchflusszytometrie überprüft, da es sich um eine schnelle Technik handelt, die es ermöglicht, diese Daten in weniger als 2 Stunden zu erhalten. Es könnten aber auch andere Techniken wie Mikroskopie oder Western Blot zum Einsatz kommen. Wir empfehlen keine Methoden wie die Echtzeit-PCR zur Identifizierung von mRNA-Spiegeln, wenn das ultimative Ziel darin besteht, einen Rezeptor auf Proteinebene zu untersuchen. Ein weiterer grundlegender Schritt ist die Vernetzungsstufe. Durch den Einsatz von Vernetzern sollen Protein-Protein-Wechselwirkungen stabilisiert werden. Während des Assays auf Basis der Durchflusszytometrie konnten wir ohne Anwendung des Vernetzungsschritts kein Signal für SLAMF1 nachweisen. Im Falle des Mikroskopie-Ansatzes haben wir eine Interaktion ohne den Einsatz einer Vernetzungsbehandlung, aber mit sehr geringer Sensitivität beobachtet. Daher ist der Vernetzungsschritt in allen Fällen sehr zu empfehlen.
Das beschriebene Protokoll liefert Hinweise auf eine Interaktion zwischen SLAMF1 und M. tuberculosis-Antigenen , die auf der Oberfläche des Erregers zugänglich sind, und hilft zu zeigen, dass SLAMF1 eine molekulare Signatur erkennt, die in M. tuberculosis vorhanden ist. Dieses Modell weist jedoch einige Einschränkungen auf. Der Schwachpunkt ist, dass dieses Protokoll es uns nicht ermöglicht, zu identifizieren, welches spezifische M. tuberculosis-Antigen dasjenige ist, das mit SLAMF1 interagiert. Trotzdem könnte das Protokoll modifiziert werden, um dieses Ziel zu erreichen, z. B. durch die Verwendung von gereinigten Antigenen aus den Bakterien. Eine weitere Einschränkung ist die Notwendigkeit zu wissen, welcher Stimulus die Expression von SLAMF1 in Makrophagen induziert. Dies könnte eine Komplikation bedeuten, wenn der gewählte Rezeptor unter basalen Bedingungen nicht exprimiert wird oder für den das Expressionsmuster unbekannt ist. Schließlich unterscheiden diese Ansätze nicht, ob SLAMF1 andere Moleküle benötigt, um mit M. tuberculosis zu interagieren. In diesem Fall könnten Immunpräzipitationsassays durchgeführt werden, oder es könnten mehr Antikörper im methodischen Schema verwendet werden.
Die in diesem Bericht vorgeschlagene Methodik kann leicht an die Untersuchung anderer Immunrezeptoren angepasst werden, an denselben Rezeptor in anderen Zelltypen, in denen er exprimiert wird, oder an die Bewertung der Interaktion mit anderen Bakterien oder Stämmen verschiedener Mykobakterien. Darüber hinaus wird hier ein kurzes und einfaches Protokoll zur Fluoreszenzfärbung von beschalltem M. tuberculosis bereitgestellt, eine Markierung, die auf andere Bakterien und sogar lebende Stämme angewendet werden könnte. Dieses Verfahren konzentriert sich jedoch auf den SLAMF1-M. tuberculosis-Crosstalk , andere mögliche Anwendungen sind das Verständnis des Ergebnisses einer blockierten, verzögerten oder unangemessenen Interaktion, die Untersuchung von Umgehungsmechanismen oder die Aufdeckung potenzieller molekularer Ziele, die an der Erkennung von Krankheitserregern beteiligt sind. In ähnlicher Weise könnte dieses Protokoll auf die Untersuchung neuartiger therapeutischer Strategien und Immuntherapien angewendet werden, um die Koevolution von M. tuberculosis und Makrophagen oder anderen Wirtszellen zu verstehen, und könnte auch in verschiedenen Bereichen eingesetzt werden, die versuchen, die Liganden-Rezeptor-Dynamik zu verstehen.
Offenlegungen
Die Autoren erklären, dass keine Interessenkonflikte bestehen.
Danksagungen
Diese Arbeit wurde unterstützt von der Universidad Nacional del Noroeste de la Provincia de Buenos Aires (Fördernummern SIB 0618/2019, SIB 2582/2012 an V.P.), Agencia Nacional de Promoción Científica y Tecnológica (ANPCyT, Förderkennzeichen PICT-2012-2459 und PICT A 2017-1896 an V.P. und PICT-2021-I-INVI-00584 an A.B.); Florencio Fiorini Stiftung; und Consejo Nacional de Investigaciones Científicas y Técnicas (CONICET, Fördernummer PIO 15720150100010CO an V.P.). Wir danken Natalia Menite und Gastón Villafañe für die technische Unterstützung. Wir danken Dr. Paula Barrionuevo und Dr. Luciana Balboa für die wissenschaftliche Diskussion während der Publikation, die zur Entwicklung der in dieser Arbeit vorgestellten Assays geführt hat. Abschließend danken wir Dr. Estermann für seine Ratschläge zu Cross-Linking-Protokollen, Lic. Moroni für seine Ratschläge zur Arbeit mit Poly-Lysin und Lic. Moriconi für seine Hilfe bei der Erstellung schematischer Figuren.
Materialien
| Name | Company | Catalog Number | Comments |
| Alexa Fluor 488 secondary antibody | Invitrogen | A21121 | For fluorescence microscopy |
| anti-SLAMF1 FITC antibody | eBioscience | 11-1509-42 | For flow cytometry |
| anti-SLAMF1 PE antibody | BioLegend | 306308 | For flow cytometry |
| anti-SLAMF1 primary antibody | BioLegend | 306302 | For fluorescence microscopy |
| Aqua-Poly/Mount | Polysciences | 18606-20 | Mounting media |
| CD14 MicroBeads | Miltenyi Biotec | 130-097-052 | For monocytes isolation |
| Coverslips 12mm | HDA | - | For interaction assay by microscopy |
| EGS | ThermoFisher Scientific | 21565 | For crosslinking treatment |
| FACSCanto II | BD Biosciences | 338960 | Flow cytometer with BD FACSDiva software |
| Fetal Bovine Serum | Natocor | - | Inactivated and irradiated, for macrophages culture |
| Ficoll-Paque PLUS | Cytiva | 17144003 | For PBMCs separation |
| Fiji/ImageJ | Open Source software | - | For micrographs analysis |
| FlowJo 7.6.2 | Tree Star | - | For flow cytometry analysis |
| Formaldehyde | Merck | K47740803613 | For crosslinking treatment |
| Glass slides | Glass Klass | - | For interaction assay by microscopy |
| Glycine | Sigma | G8898 | For crosslinking treatment |
| Imager.A2 | Carl Zeiss | 430005-9901-000 | Fluorescence microscope with Colibri 7 illumination module |
| iMark | BIO-RAD | 1681130 | Microplate absorbance reader |
| L-glutamine | Sigma Aldrich | 49419 | For macrophages culture |
| M. tuberculosis, strain H37Rv, gamma-irradiated whole cells | BEI Resources, NIAID, NIH | NR-14819 | For interaction assay |
| M. tuberculosis, strain H37Rv, whole cell lysate | BEI Resources, NIAID, NIH | NR-14822 | For macrophages stimulation and interaction assay |
| Neofuge 13R | Heal Force | Neofuge 13R | High Speed Refrigerated Centrifuge for protein extraction |
| Penicillin/Streptomycin | Gibco | 15140122 | For macrophages culture |
| PMSF | ThermoFisher Scientific | 36978 | For proteins isolation |
| Poly-D-Lysine | Sigma Aldrich | A-003-M | For coverslips treatment |
| Protease Inhibitor Cocktail | Sigma Aldrich | P8340 | For proteins isolation |
| Rhodamine B | Sigma Aldrich | 21955 | For M. tuberculosis staining |
| RPMI 1640 | Gibco | 11875093 | For macrophages culture |
| Sorvall ST 16/16R centrifuge | ThermoFisher Scientific | 75004240 | For PBMCs and monocytes isolation |
Referenzen
- Global Tuberculosis Report 2023. , World Health Organization. Available at: https://www.who.int/tb/publications/global_report/en/ (2023).
- Ahmad, F., et al. Macrophage: A cell with many faces and functions in tuberculosis. Front Immunol. 13, 882130(2022).
- Bo, H., et al. Mycobacterium tuberculosis-macrophage interaction: Molecular updates. Front Cell Infect Microbiol. 13, 1187205(2023).
- Papp, A. C., et al. AmpliSeq transcriptome analysis of human alveolar and monocyte-derived macrophages over time in response to Mycobacterium tuberculosis infection. PLoS One. 13 (5), e0198069(2018).
- Zhai, W., Wu, F., Zhang, Y., Fu, Y., Liu, Z. The immune escape mechanisms of Mycobacterium tuberculosis. Int J Mol Sci. 20 (2), 340(2019).
- Van Crevel, R., Ottenhoff, T. H. M., Van der Meer, J. W. M. Innate immunity to Mycobacterium tuberculosis. Clin Microbiol Rev. 15 (2), 294-309 (2002).
- Mortaz, E., et al. Interaction of pattern recognition receptors with Mycobacterium tuberculosis. J Clin Immunol. 35 (1), 1-10 (2015).
- Stamm, C. E., Collins, A. C., Shiloh, M. U. Sensing of Mycobacterium tuberculosis and consequences to both host and bacillus. Immunol Rev. 264 (1), 204-219 (2015).
- Zihad, S. M. N. K., et al. Role of pattern recognition receptors in sensing Mycobacterium tuberculosis. Heliyon. 9 (10), e20636(2023).
- Barbero, A. M., et al. SLAMF1 signaling induces Mycobacterium tuberculosis uptake leading to endolysosomal maturation in human macrophages. J Leukoc Biol. 109 (8), 257-273 (2021).
- Pasquinelli, V., et al. Expression of signaling lymphocytic activation molecule-associated protein interrupts IFN-γ production in human tuberculosis. J Immunol. 172 (2), 1177-1185 (2004).
- Pasquinelli, V., et al. Phosphorylation of mitogen-activated protein kinases contributes to interferon-γ production in response to Mycobacterium tuberculosis. J Infect Dis. 207 (2), 340-350 (2012).
- Pasquinelli, V., et al. IFN-γ production during active tuberculosis is regulated by mechanisms that involve IL-17, SLAM, and CREB. J Infect Dis. 199 (5), 661-665 (2009).
- Pellegrini, J. M., et al. Neutrophil autophagy during human active tuberculosis is modulated by SLAMF1. Autophagy. 17 (2), 423-426 (2021).
- Song, T., Dong, C., Xiong, S. Signaling lymphocyte-activation molecule SLAMF1 augments mycobacteria BCG-induced inflammatory response and facilitates bacterial clearance. Int J Med Microbiol. 305 (6), 572-580 (2015).
- Eble, J. A. Titration ELISA as a method to determine the dissociation constant of receptor-ligand interaction. J Vis Exp. (132), e57032(2018).
- Patching, S. G. Surface plasmon resonance spectroscopy for characterization of membrane protein-ligand interactions and its potential for drug discovery. Biochim Biophys Acta Biomembr. 1838 (1), 43-55 (2014).
- Draczkowski, P., Matosiuk, D., Jozwiak, K. Isothermal titration calorimetry in membrane protein research. J Pharm Biomed Anal. 87, 296-304 (2014).
- Smith, D. S., Eremin, S. A. Fluorescence polarization immunoassays and related methods for simple, high-throughput screening of small molecules. Anal Bioanal Chem. 391 (5), 1499-1507 (2008).
- García-Nafría, J., Tate, C. G. Cryo-electron microscopy: Moving beyond X-ray crystal structures for drug receptors and drug development. Annu Rev Pharmacol Toxicol. 60, 51-71 (2020).
- Asami, J., Shimizu, T. Structural and functional understanding of the toll-like receptors. Protein Sci. 30 (4), 761-772 (2021).
- Phạm, T. T. T., Rainey, J. K. On-cell nuclear magnetic resonance spectroscopy to probe cell surface interactions. Biochem Cell Biol. 99 (6), 683-692 (2021).
- El Deeb, S., et al. Microscale thermophoresis as a powerful growing analytical technique for the investigation of biomolecular interaction and the determination of binding parameters. Methods Appl Fluoresc. 10 (4), 045001(2022).
- El Khamlichi, C., et al. Bioluminescence resonance energy transfer as a method to study protein-protein interactions: Application to G protein-coupled receptor biology. Molecules. 24 (3), 505(2019).
- Schneider, P., Willen, L., Smulski, C. R. Tools and techniques to study ligand-receptor interactions and receptor activation by TNF superfamily members. Methods Enzymol. 545, 103-125 (2014).
- Park, A. M. W., Schirmer, P. S. H. Atomic force microscopy: A multifaceted tool to study membrane proteins and their interactions with ligands. Biochim Biophys Acta Biomembr. 1838 (1), 74-89 (2014).
- Zalejski, J., Sun, J., Sharma, A. Unravelling the mystery inside cells by using single-molecule fluorescence imaging. J Imaging. 9 (9), 183(2023).
- Zheng, S., Zou, M., Shao, Y., Wu, H., Wang, X. Two-dimensional measurements of receptor-ligand interactions. Front Mol Biosci. 10, 1075587(2023).
- Zlotnikov, I. D., Kudryashova, E. V. Computer simulation of the receptor-ligand interactions of mannose receptor CD206 in comparison with the lectin concanavalin A model. Biochemistry (Moscow). 87 (1), 54-69 (2022).
- Fu, Y., Zhao, J., Chen, Z. Insights into the molecular mechanisms of protein-ligand interactions by molecular docking and molecular dynamics simulation: A case of oligopeptide binding protein. Comput Math Methods Med. 2018, 7604567(2018).
- Farina, B., et al. A novel approach for studying receptor-ligand interactions on living cells' surface by using NUS/T1ρ-NMR methodologies combined with computational techniques: The RGDechi15D-αvβ5 integrin complex. Comput Struct Biotechnol J. 19, 3303-3318 (2021).
- Driessen, N. N., et al. Role of phosphatidylinositol mannosides in the interaction between mycobacteria and DC-SIGN. Infect Immun. 77 (10), 4538-4547 (2009).
- Bansal, K., et al. Src homology 3-interacting domain of Rv1917c of Mycobacterium tuberculosis induces selective maturation of human dendritic cells by regulating PI3K-MAPK-NF-κB signaling and drives Th2 immune responses. J Biol Chem. 285 (47), 36511-36522 (2010).
- Xu, Y., et al. PPE57 induces activation of macrophages and drives Th1-type immune responses through TLR2. J Mol Med. 93 (6), 645-662 (2015).
- Olesen, R., et al. DC-SIGN (CD209), pentraxin 3 and vitamin D receptor gene variants associate with pulmonary tuberculosis risk in West Africans. Genes Immun. 8, 456-467 (2007).
- Berger, S. B., et al. SLAM is a microbial sensor that regulates bacterial phagosome functions in macrophages. Nat Immunol. 11 (10), 920-927 (2010).
- Degos, C., et al. Omp25-dependent engagement of SLAMF1 by Brucella abortus in dendritic cells limits acute inflammation and favours bacterial persistence in vivo. Cell Microbiol. 22 (4), e13156(2020).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenWeitere Artikel entdecken
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten