Method Article
Ensayos de fluorescencia para el estudio de la interacción de Mycobacterium tuberculosis con el receptor inmunitario SLAMF1
En este artículo
Resumen
Este estudio proporciona un protocolo para evaluar la interacción de Mycobacterium tuberculosis con el sensor microbiano SLAMF1. Los ensayos se llevaron a cabo en macrófagos derivados de monocitos humanos mediante citometría de flujo y microscopía de fluorescencia. Las herramientas descritas son relevantes para estudiar las interacciones entre patógenos e inmunorreceptores.
Resumen
La evaluación de la interacción directa entre patógenos y receptores inmunitarios suele implicar técnicas sofisticadas o el uso de cepas transgénicas y células modificadas genéticamente. En este trabajo se describe un método alternativo para detectar la interacción bioquímica entre el sensor microbiano de macrófagos SLAMF1 y Mycobacterium tuberculosis . Se desarrollaron dos enfoques técnicos que emplean citometría de flujo y microscopía de fluorescencia. Se generaron extractos de proteínas celulares totales de macrófagos humanos, luego se incubaron con células enteras de antígenos de M. tuberculosis (WCMtb) o M. tuberculosis (Mtb Ags) durante la noche a 4 °C y finalmente se reticularon utilizando un tratamiento con formaldehído/glicina/etilenglicol bis (succinato de succinimidil). La interacción de SLAMF1 con WCMtb mediante citometría de flujo se detectó con un anticuerpo anti-SLAMF1 específico de PE. La existencia de interacción por microscopía de fluorescencia se realizó mediante la unión de Mtb Ags teñidos con rodamina-PE a portaobjetos recubiertos de poli-D-lisina, que se incubaron con el extracto proteico total de macrófagos derivados de monocitos. Después del tratamiento con reticulación, SLAMF1 se visualizó utilizando anticuerpos primarios (anti-SLAMF1) y secundarios (Alexa Fluor 488). Los ensayos proporcionaron una sólida herramienta bioquímica para medir las interacciones entre patógenos e inmunorreceptores, superando las dificultades asociadas con las líneas celulares transgénicas y los experimentos de modulación de la expresión génica de proteínas.
Introducción
Mycobacterium tuberculosis, el patógeno causante de la tuberculosis identificado hace 142 años, sigue siendo un desafío global, infectando actualmente al menos a una cuarta parte de la población mundial1. Transmitida a través de gotitas en el aire de personas infectadas, M. tuberculosis alcanza los macrófagos alveolares en los pulmones, donde puede sobrevivir durante largos períodos en estado latente 2,3. No solo se activa la respuesta macrófagica local, sino que en los últimos años se ha descrito que los monocitos periféricos pueden ser reclutados hacia el tracto respiratorio y diferenciarse en macrófagos alveolares, generando una respuesta aún más robusta contra M. tuberculosis que su contraparte de origen fetal 2,4.
Los macrófagos son actores fundamentales de la inmunidad innata. Tras la ingestión de M. tuberculosis, los macrófagos muestran numerosas funciones microbicidas, como la secreción de citocinas proinflamatorias, la fusión del fagolisosoma y la activación de otras células inmunitarias para matar M. tuberculosis 2,3. Sin embargo, la compleja estructura arquitectónica de este patógeno proporciona efectores (por ejemplo, proteínas y lípidos) capaces de regular el metabolismo y las funciones de los macrófagos y, en consecuencia, el proceso inflamatorio3. Esta manipulación de las respuestas de los macrófagos, a través de la secreción de factores de virulencia o la explotación de factores del huésped, conduce a diferentes estrategias de escape ejercidas por M. tuberculosis. Algunos mecanismos de evasión clave incluyen la inducción de citocinas antiinflamatorias, la inhibición de la maduración y acidificación de los fagolisosomas, las alteraciones del estrés oxidativo, la interrupción de la autofagia y las deficiencias durante el procesamiento y la presentación del antígeno 3,5.
Una interacción estrechamente regulada entre los macrófagos y M. tuberculosis es crucial para el desarrollo de una respuesta inmunitaria adecuada. Por lo tanto, el estudio de estas sinapsis es clave para identificar los mecanismos inmunoprotectores o inmunopatogénicos inducidos por la diafonía huésped-patógeno, así como para identificar posibles dianas terapéuticas. Muchos receptores median el reconocimiento y/o internalización de M. tuberculosis, incluyendo los TLRs 6,7,8, NLRs 7,8, los receptores del complemento 6,8, los receptores de lectina tipo C 6,8 y los receptores carroñeros 6,8. Los datos acumulados indican que tanto los PRR unidos a la superficie como los intracelulares desempeñan un papel importante durante la infección, reconociendo M. tuberculosis opsonizada y no opsonizada.
Zihad y Sifat et al. han revisado recientemente la participación de los PRRs en las respuestas inducidas por M. tuberculosis por células innatas9. En particular, la mayoría de los miembros de la familia TLR han sido implicados en la interacción con los ligandos de M. tuberculosis 6,7. Surface TLR2 reconoce antígenos micobacterianos como lipoproteínas aciladas, lipoproteína de 19 kDa, lipoproteína LprA, lipoproteína LprG, antígeno de 30 kDa, antígeno de 38 kDa, MymA, ácido prolina-prolina-glutámico (PPE)-57, LAM, LM, PIM, proteína de choque térmico 60, proteína de firma Rv1509 y la proteína secretada ESAT-67. La membrana TLR4 interactúa con las proteínas de choque térmico, el antígeno de 38 kDa, RpfE, Rv0652, Rv0335c, Rv2659c, Rv1738, Rv2627c, Rv2628, GrpE y HBHA. Por otro lado, los ligandos de los TLR endosomales (TLR 3, 7, 8 y 9) incluyen dsRNA, tRNA, ssRNA, ARN fagosomal y dsDNA de M. tuberculosis. Además, TLR2 tiene un papel activo en el inicio de respuestas inmunitarias cuando actúa junto con TLR1 y TLR6, TLR4 y TLR9 6,7. NOD2 y NLRP3 son los NLRs citoplasmáticos mejor caracterizados en la tuberculosis. Aunque sus ligandos específicos siguen siendo objeto de estudio, estos receptores son activados por el dipéptido de muramilo o ESAT-6, respectivamente 7,8. Los receptores de lectina de tipo C están clásicamente implicados en la endocitosis por M. tuberculosis. Mientras que la Dectina-2 actúa como PRR directo para ManLAM de la pared celular de M. tuberculosis, el ligando Dectina-1 aún no ha sido descubierto8. Tanto Mincle como MCL reconocen el glicolípido trehalosa-6,6′-dimycolato (TDM), también llamado factor de cordón7. El DCAR interactúa con los glicolípidos micobacterianos fosfatidil-mio-inositol maniósidos (PIMs)7. CR3 reconoce las micobacterias LAM y PIM, DC-SIGN liga ManLAM, MR reconoce una serie de componentes de M. tuberculosis, incluidos ManLAM, PIM, LM, glicoproteína de 38 kDa, antígeno de 19 kDa y otras proteínas manosiladas, mientras que el ligando DCIR aún no está identificado.
Los CLR solubles incluyen SP-A, que reconoce ManLam, LM, glicoproteína de 60 kDa y glicoproteína Apa; SP-D interactuando con LAM, LM y PILAM; y MBL, que se especializa en el reconocimiento ManLAM 6,7. Los receptores scavenger también son PRRs fagocíticos: SR-A y MARCO tienen TDM como ligando, SR-B1 reconoce ESAT-6 y CD36 se une a ManLAM y LM 7,8. Además, Dectin-1, Mincle y MARCO también pueden combinarse con TLR2 o TLR4 para activar señales después de detectar PAMPs de M. tuberculosis 6,7. CD14 es un receptor de superficie que tiene la capacidad de internalizar bacterias no opsonizadas y también reconoce la proteína de choque térmico Chaperonin 60.1. En particular, CD14 funciona como correceptor junto con MARCO y TLR26. AIM2 es un receptor citosólico que puede detectar el ssDNA cuando M. tuberculosis escapa del fagosoma8. Por último, AhR es un factor de transcripción activado por ligando que se une al factor de virulencia pigmentado naftoquinona phthiocol de M. tuberculosis6.
Muchas de las interacciones descritas anteriormente han sido postuladas y no estrictamente demostradas. Incluso los ligandos de ciertos receptores siguen siendo desconocidos, lo que refuerza la necesidad de comprender mejor el campo del inmunorreconocimiento de M. tuberculosis. En este contexto, la molécula coestimuladora SLAMF1 (Signaling Lymphocytic Activation Molecule) ha sido descrita recientemente como un receptor de M. tuberculosis por Barbero et al.10. Al actuar no solo como una molécula de señalización, sino también como un sensor de M. tuberculosis, SLAMF1 tiene un papel particularmente intrigante en la tuberculosis. SLAMF1 puede inducir la activación de las células inmunitarias modulando las funciones protectoras como la producción de IFN-γ por las células T a través de la fosforilación de Erk/CREB 11,12,13, la autofagia en neutrófilos 14 y el aclaramiento bacteriano en macrófagos15.
Las interacciones receptores-ligando se han estudiado a lo largo de los años utilizando técnicas como ELISA, Resonancia de Plasmón de Superficie (SPR), Calorimetría de Titulación Isotérmica (ITC), Polarización de Fluorescencia (FP), Cristalografía de Rayos X, Espectroscopía de Resonancia Magnética Nuclear (RMN), Termoforesis a Microescala (MST), Transferencia de Energía de Resonancia (por ejemplo, BRET o FRET), Microscopía Confocal, Microscopía Electrónica, Criomicroscopía Electrónica (Cryo-EM) y Microscopía de Fuerza Atómica (AFM)16,17, 18,19,20,21,22,23,24,25,26,27,28. Algunos de estos enfoques implican el uso de genes reporteros, proteínas recombinantes marcadas o moléculas quiméricas, modelos knockout, knockdown o de sobreexpresión. Alternativamente, las herramientas computacionales pueden predecir las interacciones receptor-ligando y los sitios de unión y, a menudo, se utilizan en combinación con enfoques biológicos para obtener una comprensión integral de las interacciones 29,30,31. Aquí, se describen dos alternativas para detectar la interacción bioquímica y también una sección sobre cómo marcar bacterias con fluorescencia. Se presenta un protocolo que permite el estudio de las interacciones receptor-patógeno in vitro, evaluando particularmente el compromiso de SLAMF1-M. tuberculosis mediante citometría de flujo y microscopía de fluorescencia, dos técnicas habitualmente disponibles y de uso rutinario.
Protocolo
Todos los procedimientos con macrófagos derivados de monocitos humanos se realizaron de acuerdo con la Declaración de Helsinki (2013) y de acuerdo con el Comité de Ética de la UNNOBA (COENOBA). Se obtuvo el consentimiento informado por escrito antes de la recolección de la muestra. La distribución del grupo masculino/femenino fue de 13/6 y la mediana de edad fue de 32 años, con un rango intercuartílico (RIC) de 18-75 años. Se definió como criterio de exclusión la presencia de patologías previas, comorbilidades o diagnóstico positivo de Tuberculosis. Los detalles de los reactivos y el equipo se enumeran en la Tabla de Materiales.
1. Cultivo y estimulación de macrófagos derivados de monocitos
- Extracción de sangre
- Obtener 60 mL de sangre de donantes sanos por venopunción en una jeringa heparinizada.
NOTA: La extracción de sangre debe ser realizada por un bioquímico o un técnico en flebotomía. - Deseche la aguja en el contenedor de eliminación de objetos punzocortantes y cualquier material que haya estado en contacto con la sangre de los donantes en el contenedor de desechos patológicos.
- Obtener 60 mL de sangre de donantes sanos por venopunción en una jeringa heparinizada.
- Aislamiento de monocitos y generación de macrófagos
- Transfiera cuidadosamente la sangre a tubos de 50 ml y dilúyala a la mitad con solución salina.
- Realice la centrifugación sobre un medio de gradiente de densidad para obtener células mononucleares de sangre periférica (PBMC).
- Recoge los PBMC del halo blanquecino.
- Realizar dos lavados con suero fisiológico a 400 x g durante 10 min y un lavado final a 200 x g durante 15 min a 20 °C para eliminar las plaquetas.
- Determine el número de PBMC en una cámara de recuento de células y realice una selección magnética positiva de CD14 con perlas de CD14.
NOTA: Durante la selección magnética, siga cuidadosamente las instrucciones del fabricante. Además, se puede comprobar la pureza de los monocitos aislados mediante citometría de flujo. - Vuelva a suspender las células aisladas en RPMI 1640 y determine el número de monocitos en una cámara de recuento de células.
- Siembra 500 μL de monocitos seleccionados CD14 positivos de 1 ×10 6/mL por pocillo en una placa de cultivo celular de 24 pocillos en ausencia de suero para promover la adherencia.
- Después de 2 h, retire las células no adherentes lavándolas con RPMI 1640 precalentado.
- Para diferenciar los macrófagos, se deben cultivar los monocitos adherentes durante 16-18 h adicionales (durante la noche) en 1 mL de medio completo (medio RPMI 1640 suplementado con L-glutamina, 10% de suero fetal bovino (FBS), 100 U/mL de penicilina y 100 μg/mL de estreptomicina).
- Al día siguiente, cambia el medio de cultivo. Retire el medio, lave con 1 mL de 1x PBS y agregue 1 mL de medio completo.
- Estimular los macrófagos con M. tuberculosis sonicado (antígenos de M. tuberculosis (Ags)) durante 24 h. Utilice 10 μL de Ags de M. tuberculosis (10 μL = 1 × 106 bacterias) por 1 × 106 macrófagos derivados de monocitos.
NOTA: 10 μL de M. tuberculosis Ags (Mtb Ags) = 1 × 106 bacterias, cuantificadas por D.O. 600nm en espectrofotómetro considerando una Õ.D. de 1 como 1 x 108 bacterias/mL. Utilice cabinas de bioseguridad BSL2 y una incubadora a 37 °C en una atmósfera con 5% deCO2 para todas las etapas de cultivo.
- Corroboración de la expresión de SLAMF1
- Recoge los macrófagos de un solo pocillo. Coloque la placa de cultivo sobre una superficie fría. Separe las celdas usando movimientos hacia arriba y hacia abajo agregando 1 mL de FACS frío (1x PBS + 2% FBS) dos veces consecutivas. Utilice un tubo de citometría de fondo redondo para la recolección.
- Centrifugar las células a 500 x g durante 5 min a 4 °C. Deseche el sobrenadante tirándolo.
- Teñir las células con un anticuerpo SLAMF1 antihumano acoplado a fluoróforos durante 30 min a 4 °C en la oscuridad. Vórtice después de añadir 1,25 μL del anticuerpo.
- Lavar las células para eliminar el exceso de anticuerpos mediante FACS, resuspender el pellet en FACS y adquirir la muestra mediante un citómetro de flujo.
NOTA: 1,25 μL de anticuerpo anti-SLAMF1 corresponde a 0,0625 μg que se utilizan para marcar 0,5 x 106 macrófagos. Sin embargo, se recomienda encarecidamente la titulación del anticuerpo, así como la realización del correspondiente control del isotipo o de fluorescencia menos uno (FMO).
2. Preparación del extracto de proteína celular total
- Preparación del tampón de lisis
- Prepare un tampón de ensayo de radioinmunoprecipitación (RIPA) con NaCl 150 mM, Tris 10 mM, EDTA 5 mM, SDS 1%, Triton X-100 1% y desoxicolato de sodio 1%.
- Complemente el tampón RIPA con fluoruro de fenilmetilsulfonilo (PMSF) de 1 mM e inhibidores de la proteasa como la pepstatina A y la leupeptina.
- Recolección celular y aislamiento de proteínas
- Deseche el RPMI completo de los pocillos de placa con una pipeta P1000 y recoja los macrófagos mediante pipeteo ascendente y descendente con PBS 1x en frío.
- Transfiera las células a tubos de microcentrífuga de 1,5 mL. Recoja un pocillo por microtubo.
- Centrifugar a 500 x g durante 5 min a 4 °C. Deseche el sobrenadante.
- Añadir 1 mL de PBS frío y repetir los pasos 2.2.2 y 2.2.3 recogiendo las células en el mismo tubo de microcentrífuga.
- Vuelva a suspender los gránulos de celda en 100 μL de 1x PBS y agrupe cuatro tubos. Repita el paso 2.2.3. Cada tubo contendrá 2 x 106 macrófagos.
- Añadir 100 μL de tampón RIPA suplementado e incubar la suspensión en hielo durante 1 h, vórtice cada 10 min.
- Centrifugar a 14.000 x g durante 15 min. Recoja el sobrenadante y consérvelo hasta su uso a -20 °C.
3. Interacción M. tuberculosis-SLAMF1 por citometría de flujo
- Reticulación proteínas-bacterias
- Incubar 1 × 106 células enteras inactivadas de M. tuberculosis (WCMtb, M. tuberculosis inactivadas por irradiación gamma) con extracto proteico de 1 x 106 macrófagos (50 μL) durante la noche a 4 °C en un soporte de microtubos giratorio.
NOTA: 10 μL de WCMtb = 1 × 106 bacterias, cuantificadas por D.O. 600nm en espectrofotómetro considerando una Ø.D. de 1 como 1 x 108 bacterias/mL. - Al día siguiente, agregue 500 μL de formaldehído al 1% diluido en 1x PBS. Incubar durante 15 min bajo agitación (agitador) a temperatura ambiente (RT).
- Añadir 25 μL de glicina 0,125 M diluida en agua. Incubar durante 5 min bajo agitación (agitador) a RT.
- Lavar dos veces con 1 ml de 1x PBS a 14.000 x g durante 5 min. Deseche el sobrenadante entre las etapas de lavado.
- Vuelva a suspender el pellet e incube la suspensión en 500 μL de 2 mM de reticulado de etilenglicol bis (succinimidil succinimidato) (EGS) diluido en una mezcla 1:1 de ácido acético glacial:agua durante 1 h a RT.
NOTA: Se recomienda encarecidamente la preparación de los reactivos en el momento de su uso. Para preparar el EGS, el ácido acético glacial y el agua deben mezclarse en proporciones iguales y calentarse a 70 °C. Si la solución no está caliente, el EGS no se vuelve a suspender por completo.
- Incubar 1 × 106 células enteras inactivadas de M. tuberculosis (WCMtb, M. tuberculosis inactivadas por irradiación gamma) con extracto proteico de 1 x 106 macrófagos (50 μL) durante la noche a 4 °C en un soporte de microtubos giratorio.
- Tinción SLAMF1
- Repita el paso 3.1.4 usando 500 μL de 1x PBS en el primer lavado y 1 mL en el segundo.
- Teñir el complejo proteína-bacteria durante 30 min a 4 °C en la oscuridad con un anticuerpo anti-SLAMF1 humano (como en el paso 1.3.3) añadiendo el anticuerpo al microtubo y vórtice. Utilice la cantidad de anticuerpo adecuada para 1 x 106 células (2,5 μL o 0,125 μg).
- Lavar las células para eliminar el exceso de anticuerpos mediante FACS, resuspender el pellet en FACS y adquirir la muestra en un citómetro de flujo (como en el paso 1.3.4).
NOTA: Realizar el control de isotipo o FMO correspondiente.
4. Marcaje de antígenos de M. tuberculosis
- Vuelva a suspender la rodamina B en etanol a una concentración de 5 mg/mL (1000x). Conservar a -20 °C hasta su uso.
- Añadir 1 μL de Rhodamina B por cada 100 μL de M . tuberculosis Ags en un tubo de microcentrífuga de 1,5 mL.
- Incubar durante 45 min en RT bajo agitación continua (vórtice).
- Lavar al menos 3 veces a 14.000 x g durante 5 min a 24 °C con 1 mL de 1x PBS hasta que el sobrenadante esté transparente. El pellet (Mtb-R Ags) se verá de color rosa claro.
- Vuelva a suspender el Mtb-R Ags en 100 μL de 1x PBS para restaurar el volumen inicial.
NOTA: Se recomienda trabajar en condiciones estériles. Asegúrese de que la tinción se realice inmediatamente antes de realizar experimentos.
5. Interacción M. tuberculosis-SLAMF1 por microscopía de fluorescencia
- Reticulación bacteria-proteína
- Introducir cubreobjetos redondos de 12 mm en placas de cultivo de 24 pocillos (1 cubreobjetos por pocillo) utilizando pinzas quirúrgicas de punta redonda.
NOTA: Limpie a fondo los cubreobjetos con etanol al 70%. Si se desea, se pueden esterilizar en autoclave o bajo luz ultravioleta durante 30 minutos. - Incubar el cubreobjetos con 400 μL de 10 μg/mL de Poly-D-Lisina durante la noche a 4 °C. Cubra la placa con papel de aluminio para protegerla de la luz.
- Al día siguiente, retire la placa del refrigerador y lave el cubreobjetos dos veces con 1 ml de agua.
- Deja que el cubreobjetos se seque.
- Diluir 20 μL (2 x 106) de Mtb-R Ags en 180 μL de 1x PBS. Los últimos 200 μL son suficientes para cubrir completamente un cubreobjetos.
- Incubar el cubreobjetos con Mtb-R Ags durante 1 h a 37 °C en una incubadora de células.
- Deseche el líquido restante y lave dos veces con 1 ml de 1x PBS.
NOTA: En este paso, la correcta adherencia de Mtb-R al cubreobjetos se puede comprobar con un microscopio de fluorescencia utilizando los filtros correspondientes al canal rojo. - Añadir 400 μL de tampón de bloqueo (10% FBS en 1x PBS) durante 30 min a RT en agitación (agitador).
- Lavar dos veces con 400 μL de tampón de bloqueo.
- Incubar con 100 μL de extracto proteico diluido en 300 μL de 1x PBS durante 2 h a RT en agitación (agitador).
- Añadir 400 μL de formaldehído al 1% diluido en 1x PBS. Incubar durante 15 min bajo agitación (agitador) a RT.
- Añadir 500 μL de glicina 0,125 M diluida en agua. Incubar durante 5 min bajo agitación (agitador) a RT.
- Lavar dos veces con 1 mL de 1x PBS.
- Añadir 400 μL de 2 mM de EGS durante 1 h a RT en agitación (agitador).
- Repita el paso 5.1.13
NOTA: Se recomienda encarecidamente la preparación de los reactivos en el momento de su uso.
- Introducir cubreobjetos redondos de 12 mm en placas de cultivo de 24 pocillos (1 cubreobjetos por pocillo) utilizando pinzas quirúrgicas de punta redonda.
- Tinción SLAMF1
- Envuelva la tapa de la placa de cultivo en una película de parafina.
- Añadir una gota de 60 μL de anticuerpo primario anti-SLAMF1 previamente titulado (dilución 1:75) en el párpado cubierto con película de parafina.
- Retire con cuidado el cubreobjetos de la placa con pinzas quirúrgicas curvas de punta fina y agujas.
NOTA: Para facilitar la extracción de los cubreobjetos, la aguja se puede doblar en un ángulo aproximado de 45°. - Invierta el cubreobjetos sobre la gota que contiene el anticuerpo. Incubar durante 30 min en RT en la oscuridad.
- Levante el cubreobjetos y lávelo dos veces con el tampón de bloqueo.
- Invierta el cubreobjetos sobre una nueva gota que contenga el anticuerpo secundario (dilución 1:200). Incubar durante 30 min en RT en la oscuridad.
- Repita el paso 5.2.5.
- Retire el exceso de líquido y monte el cubreobjetos sobre una gota de líquido de montaje colocada en un portaobjetos de vidrio.
- Observar bajo el microscopio de fluorescencia utilizando filtros adecuados.
NOTA: Se recomienda trabajar en condiciones estériles. Diluir los anticuerpos en el tampón de bloqueo. Manipule el cubreobjetos con cuidado para evitar arañazos que puedan dificultar la observación bajo el microscopio. Se recomienda encarecidamente un líquido de montaje adecuado para la fluorescencia.
Resultados
En este trabajo se proporciona un protocolo que permite evaluar la interacción de M. tuberculosis con el receptor inmune SLAMF1 en macrófagos humanos (Figura 1). Para ello, se obtuvo sangre periférica de donantes sanos. A continuación, los PBMC se separaron por centrifugación sobre el medio de gradiente de densidad, y los monocitos se aislaron por selección magnética positiva (≥95% de pureza, Figura 2A,B). Los monocitos aislados se adhirieron a placas de cultivo plásticas durante 2 h para obtener macrófagos derivados de monocitos y luego se cultivaron ON. Posteriormente, los macrófagos se estimularon con M. tuberculosis sonicado (Mtb Ags) para inducir la expresión de superficie de SLAMF1 como se informó anteriormente10, y los niveles de superficie de SLAMF1 se confirmaron mediante citometría de flujo (Figura 2C). Finalmente, los macrófagos se lisaron utilizando el tampón de lisis RIPA combinado con vórtice e incubación en hielo para facilitar la lisis celular. El extracto proteico total generado se utilizó en los ensayos de interacción.
En el primer conjunto de procedimientos experimentales, se empleó la citometría de flujo para estudiar la interacción de SLAMF1 con células enteras de M. tuberculosis (WCMtb) (Figura 3). En primer lugar, se promovió la interacción física entre SLAMF1 (contenido en el extracto proteico) y WCMtb incubándolos juntos en ON en rotación continua a 4 °C. A continuación, se realizó un tratamiento de reticulación y, mediante el uso de un anticuerpo anti-SLAMF1 específico, se detectó el receptor SLAMF1 que se unió a WCMtb. En este ensayo, se utilizó WCMtb solo para establecer los voltajes correspondientes al tamaño y la granularidad de M. tuberculosis en el citómetro de flujo, que fue la población de interés en la que se detectó la unión de SLAMF1.
En el segundo enfoque experimental, se empleó microscopía de fluorescencia para detectar la interacción SLAMF1-M. tuberculosis (Figura 4). Este ensayo, en el que se utilizó M. tuberculosis sonicada (Mtb Ags), complementa al anterior. Se utilizaron Mtb Ags teñidos con rodamina B (Mtb-R Ags) para facilitar la detección de fluorescencia en el canal/filtro correspondiente al fluorocromo PE. Los Ags de Mtb-R se adhirieron a cubreobjetos recubiertos de poli-D-lisina, y la tinción exitosa se verificó mediante observación microscópica (Figura 4A). A continuación, los portaobjetos recubiertos con Mtb-R Ags se incubaron con los extractos de proteínas que contenían SLAMF1. Después del tratamiento con reticulación, SLAMF1 se detectó mediante el uso de un anticuerpo anti-SLAMF1 específico primario seguido de un anticuerpo secundario acoplado a un fluorocromo compatible para ser observado conjuntamente con la EP. Después de obtener imágenes individuales de microscopía fluorescente en los dos canales para Mtb-R Ags y SLAMF1, la interacción se corroboró mediante fusión de imágenes (Figura 4B).
La realización de estos abordajes demostró la existencia de una interacción entre SLAMF1 y M. tuberculosis. Se detectó la diafonía de SLAMF1 con bacterias enteras y sonicadas, lo que reforzó la hipótesis que apunta a SLAMF1 como un nuevo receptor innato de macrófagos humanos para M. tuberculosis10.

Figura 1: Flujo de trabajo para detectar la interacción entre el SLAMF1 y el M de tuberculosis . La sangre periférica se obtuvo de donantes sanos por venopunción y se centrifugó sobre un medio de gradiente de densidad para aislar células mononucleares de sangre periférica (PBMC). Después de determinar el número de células, las PBMC se sometieron a selección magnética positiva para purificar los monocitosCD14 pos . A continuación, los monocitos se adhirieron a placas de cultivo durante 2 h en ausencia de suero fetal bovino y luego se cultivaron durante la noche en RPMI suplementado (paso de reposo) para obtener macrófagos derivados de monocitos. Posteriormente, los macrófagos se estimularon con M. tuberculosis sonicada (Mtb Ags) durante 24 h para inducir la expresión de superficie de SLAMF1. Los niveles de SLAMF1 se analizaron mediante citometría de flujo utilizando un anticuerpo humano anti-SLAMF1 específico en una fracción de la población de macrófagos. El resto de los macrófagos se utilizaron para obtener extractos de proteínas totales. La lisis celular se realizó combinando vórtices e incubaciones en hielo en un tampón de lisis. Finalmente, se evaluó la interacción entre SLAMF1 y M. tuberculosis mediante citometría de flujo y microscopía de fluorescencia después de la reticulación de proteínas de macrófagos con Mtb Ags o células enteras de M. tuberculosis (WCMtb). Haga clic aquí para ver una versión más grande de esta figura.

Figura 2: Controles experimentales y técnicos. (A) Los monocitosCD14 pos se aislaron de PBMC por selección magnética positiva. Las fracciones de células positivas (monocitos, azules) y negativas (rojos) resultantes se evaluaron mediante citometría de flujo (SSC-A vs. FSC-A) para confirmar una selección exitosa. (B) La pureza de la fracción positiva se corroboró mediante citometría de flujo que analizó la granularidad y el tamaño de la célula (SSC-A vs. FSC-A). (C) Los macrófagos derivados de monocitos fueron seleccionados por SSC-A vs. FSC-A y luego se limita para excluir dobletes mediante una estrategia de dobles singletes (FSC-A vs. FSC-H seguido de SSC-A vs. SSC-H). Se evaluó la expresión superficial de SLAMF1 en células individuales. En todos los casos (A-C), se muestran gráficos o histogramas representativos. Haga clic aquí para ver una versión más grande de esta figura.
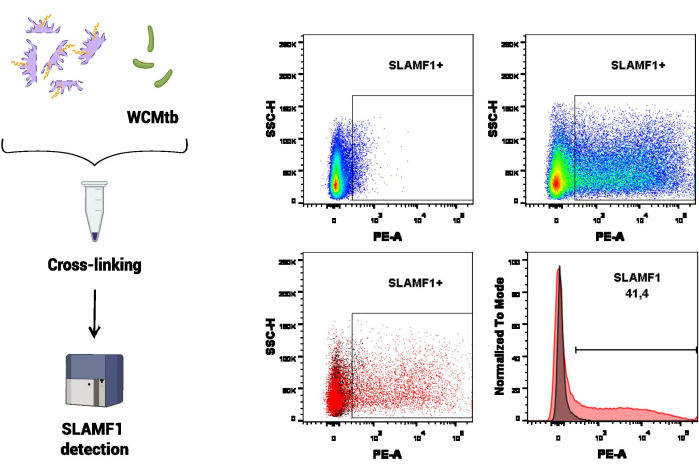
Figura 3: Interacción SLAMF1-M. tuberculosis por citometría de flujo. La reticulación entre las proteínas de los macrófagos y las células enteras de M. tuberculosis (WCMtb) se realizó mediante tratamiento con formaldehído/glicina/etilenglicol bis, como se describe en la sección del protocolo. Luego, la interacción se evaluó por citometría de flujo mediante la detección de una señal positiva de SLAMF1 (PE) después de seleccionar la población de interés (SSC-A vs. FSC-A). El panel superior muestra una señal positiva para SLAMF1, comparando el control de fluorescencia menos uno (FMO) (izquierda) con la tinción positiva (derecha). El panel inferior muestra la superposición (izquierda, diagrama de puntos y derecha, histograma) entre la señal SLAMF1 y el control FMO. Haga clic aquí para ver una versión más grande de esta figura.

Figura 4: Interacción SLAMF1-M. tuberculosis por microscopía de fluorescencia. (A) Los Mtb Ags se tiñeron con Rhodamina B (Mtb-R Ags) y se unieron a un cubreobjetos redondo. La correcta adherencia y fluorescencia de los Ags de Mtb-R se corroboró mediante microscopía de fluorescencia utilizando filtros correspondientes al canal rojo. (B) La reticulación entre las proteínas de los macrófagos y los Ags de Mtb-R se realizó mediante un tratamiento con formaldehído/glicina/etilenglicol bis como se describe en la sección del protocolo, añadiendo el extracto de proteína sobre los Ags de Mtb-R adjuntos. La interacción se evaluó mediante microscopía de fluorescencia utilizando un anticuerpo humano anti-SLAMF1 seguido de un anticuerpo secundario marcado con Alexa Fluor 488. Las micrografías superiores muestran las señales positivas SLAMF1 (izquierda, verde) y Mtb-R Ags (derecha, rojo). La micrografía baja evidencia la interacción en amarillo (fusión de ambos canales). Las barras de escala corresponden a 10 μm. Haga clic aquí para ver una versión más grande de esta figura.
Discusión
Este estudio proporciona una guía útil para estudiar la interacción bioquímica entre M. tuberculosis y los sensores microbianos expresados en los macrófagos humanos, un tipo de célula clave implicada en la respuesta del huésped durante la tuberculosis. Los protocolos proporcionados serán relevantes para descifrar las moléculas que juegan un papel en la entrada de M. tuberculosis en los fagocitos.
La caracterización de las interacciones biomoleculares, como la que existe entre patógenos e inmunorreceptores, es crucial para comprender tanto los mecanismos inmunoprotectores como las estrategias de evasión provocadas por M. tuberculosis. Muchas veces, demostrar una interacción directa del receptor con su ligando bacteriano puede ser complejo y puede requerir técnicas sofisticadas o sistemas carentes de versatilidad. Driessen et al. estudiaron el papel de los maniósidos de fosfatidilinositol en la interacción entre micobacterias y DC-SIGN utilizando cepas deficientes mutantes de M. bovis BCG32. A pesar de que lograron estudiar estas interacciones, los autores no encontraron diferencias para las cepas mutantes y discutieron que la creación de bacterias mutantes que muestren una unión reducida sería una tarea enorme32. Otros autores realizaron un excelente y laborioso trabajo para demostrar la interacción entre TLR2 y algunas proteínas PE/PPE de M. tuberculosis33,34. Estos estudios requirieron el uso de ratones knockout para el receptor, la transfección de células y la purificación de las proteínas antes mencionadas o de ciertos dominios proteicos33,34. Además, a menudo se infiere la función de los receptores, pero la interacción no se demuestra de forma fiable. Un ejemplo es el receptor soluble PTX3, para el cual se han encontrado haplotipos asociados con el desenlace de la enfermedad, pero cuya interacción específica con M. tuberculosis no ha sido estrictamente estudiada35. En relación con estas carencias, nuestro método permite evaluar la interacción del receptor diana en estudio con M. tuberculosis de forma sencilla y con técnicas accesibles y de fácil interpretación.
Respecto a los puntos anteriores, y en particular para SLAMF1, se han evaluado previamente las interacciones con OmpC y OmpF de E. coli36 y Omp25 de B. abortus37 . Degos et al.37 utilizaron células COS-7 transfectadas con un plásmido que codifica solo el dominio extracelular de SLAMF1 para luego purificar SLAMF1 y analizar la interacción con Omp25 por Western blot. También realizaron experimentos con ratones SLAMF1-/- y B. abortus de tipo salvaje o un mutante defectuoso de Omp25. Berger y cols. 36 trabajos también mostraron evidencia convincente de una interacción directa entre SLAMF1 y E. coli Omp y S. typhimurium SseB-. Utilizaron diferentes enfoques, incluidos ratones SLAMF1-/- , macrófagos RAW264.7 que codifican una construcción SLAMF1mCherry y también un ensayo de amplificación con células Jurkat T transfectadas con SLAMF1. Desarrollaron un ensayo de amplificación de señal sensible con un indicador de luciferasa utilizando 1 x 108 E. coli. En este sentido, una fortaleza de los ensayos de interacción bioquímica presentados en este informe es que la interacción se encontró realmente mediante el uso de células de tipo salvaje y la misma proporción de células:bacterias utilizada para las evaluaciones funcionales en un trabajo previo10, sin la necesidad de sobreexpresar o abrogar la expresión de SLAMF1.
El protocolo proporcionado implica algunos pasos críticos. Una de ellas es la corroboración de la expresión de SLAMF1 en macrófagos humanos antes de realizar los ensayos de interacción. Esto es crucial para evitar obtener un resultado falso negativo, que en realidad se debería a una falta de expresión del receptor. En este caso, se comprobaron los niveles de SLAMF1 mediante citometría de flujo, ya que es una técnica rápida que permite obtener estos datos en menos de 2 h. Sin embargo, se podrían utilizar otras técnicas, como la microscopía o el Western blot. No recomendamos metodologías como la PCR en tiempo real que identifica los niveles de ARNm cuando el objetivo final es estudiar un receptor a nivel de proteína. Otro paso fundamental es la etapa de reticulación. El uso de reticulantes busca estabilizar las interacciones proteína-proteína. Durante el ensayo basado en citometría de flujo, no pudimos detectar una señal para SLAMF1 sin aplicar el paso de reticulación. En el caso del abordaje de microscopía, hemos observado interacción sin el uso de tratamiento de reticulación pero con muy baja sensibilidad. Por lo tanto, el paso de reticulación es muy recomendable en todos los casos.
El protocolo descrito proporciona evidencia de la interacción entre SLAMF1 y los antígenos de M. tuberculosis accesibles en la superficie del patógeno, lo que ayuda a demostrar que SLAMF1 reconoce una firma molecular presente en M. tuberculosis. Sin embargo, este modelo presenta algunas limitaciones. El punto débil es que este protocolo no permite identificar qué antígeno concreto de M. tuberculosis es el que interactúa con el SLAMF1. A pesar de ello, el protocolo podría modificarse para lograr este fin, por ejemplo, utilizando antígenos purificados de las bacterias. Otra limitación es la necesidad de saber qué estímulo induce la expresión de SLAMF1 en los macrófagos. Esto podría implicar una complicación si el receptor elegido no se expresa en condiciones basales o para el que se desconoce el patrón de expresión. Finalmente, estos enfoques no discriminan si SLAMF1 requiere otras moléculas para interactuar con M. tuberculosis. En este caso, se podrían realizar ensayos de inmunoprecipitación, o se podrían utilizar más anticuerpos en el esquema metodológico.
La metodología propuesta en este informe puede ser fácilmente adaptable al estudio de otros receptores inmunes, del mismo receptor en otros tipos celulares en los que se expresa, o para evaluar la interacción con otras bacterias o cepas de diferentes micobacterias. Además, aquí se proporciona un protocolo breve y sencillo para teñir con fluorescencia M . tuberculosis sonicada, un etiquetado que podría aplicarse a otras bacterias e incluso a cepas vivas. Sin embargo, este procedimiento se centra en la diafonía de SLAMF1-M. tuberculosis , otros usos potenciales incluyen la comprensión del resultado de una interacción bloqueada, retrasada o inapropiada, el estudio de los mecanismos de evasión o la revelación de posibles objetivos moleculares involucrados en el reconocimiento de patógenos. Del mismo modo, este protocolo podría aplicarse al estudio de nuevas estrategias terapéuticas e inmunoterapias para comprender la coevolución de M. tuberculosis y macrófagos u otras células huésped y también podría utilizarse en diferentes campos que buscan comprender la dinámica ligando-receptor.
Divulgaciones
Los autores declaran no tener conflictos de intereses.
Agradecimientos
Este trabajo contó con el apoyo de la Universidad Nacional del Noroeste de la Provincia de Buenos Aires (números de beca SIB 0618/2019, SIB 2582/2012 a V.P.), Agencia Nacional de Promoción Científica y Tecnológica (ANPCyT, números de beca PICT-2012-2459 y PICT A 2017-1896 a V.P. y PICT-2021-I-INVI-00584 a A.B.); Fundación Florencio Fiorini; y el Consejo Nacional de Investigaciones Científicas y Técnicas (CONICET, subvención número PIO 15720150100010CO a V.P.). Agradecemos a Natalia Menite y Gastón Villafañe por el apoyo técnico. Agradecemos a la Dra. Paula Barrionuevo y a la Dra. Luciana Balboa por la discusión científica durante la publicación que dio origen al desarrollo de los ensayos presentados en este trabajo. Por último, agradecemos al Dr. Estermann por sus consejos sobre los protocolos de reticulación, Lic. Moroni por sus consejos sobre cómo trabajar con Poly-Lysine y Lic. Moriconi por su ayuda con las figuras esquemáticas.
Materiales
| Name | Company | Catalog Number | Comments |
| Alexa Fluor 488 secondary antibody | Invitrogen | A21121 | For fluorescence microscopy |
| anti-SLAMF1 FITC antibody | eBioscience | 11-1509-42 | For flow cytometry |
| anti-SLAMF1 PE antibody | BioLegend | 306308 | For flow cytometry |
| anti-SLAMF1 primary antibody | BioLegend | 306302 | For fluorescence microscopy |
| Aqua-Poly/Mount | Polysciences | 18606-20 | Mounting media |
| CD14 MicroBeads | Miltenyi Biotec | 130-097-052 | For monocytes isolation |
| Coverslips 12mm | HDA | - | For interaction assay by microscopy |
| EGS | ThermoFisher Scientific | 21565 | For crosslinking treatment |
| FACSCanto II | BD Biosciences | 338960 | Flow cytometer with BD FACSDiva software |
| Fetal Bovine Serum | Natocor | - | Inactivated and irradiated, for macrophages culture |
| Ficoll-Paque PLUS | Cytiva | 17144003 | For PBMCs separation |
| Fiji/ImageJ | Open Source software | - | For micrographs analysis |
| FlowJo 7.6.2 | Tree Star | - | For flow cytometry analysis |
| Formaldehyde | Merck | K47740803613 | For crosslinking treatment |
| Glass slides | Glass Klass | - | For interaction assay by microscopy |
| Glycine | Sigma | G8898 | For crosslinking treatment |
| Imager.A2 | Carl Zeiss | 430005-9901-000 | Fluorescence microscope with Colibri 7 illumination module |
| iMark | BIO-RAD | 1681130 | Microplate absorbance reader |
| L-glutamine | Sigma Aldrich | 49419 | For macrophages culture |
| M. tuberculosis, strain H37Rv, gamma-irradiated whole cells | BEI Resources, NIAID, NIH | NR-14819 | For interaction assay |
| M. tuberculosis, strain H37Rv, whole cell lysate | BEI Resources, NIAID, NIH | NR-14822 | For macrophages stimulation and interaction assay |
| Neofuge 13R | Heal Force | Neofuge 13R | High Speed Refrigerated Centrifuge for protein extraction |
| Penicillin/Streptomycin | Gibco | 15140122 | For macrophages culture |
| PMSF | ThermoFisher Scientific | 36978 | For proteins isolation |
| Poly-D-Lysine | Sigma Aldrich | A-003-M | For coverslips treatment |
| Protease Inhibitor Cocktail | Sigma Aldrich | P8340 | For proteins isolation |
| Rhodamine B | Sigma Aldrich | 21955 | For M. tuberculosis staining |
| RPMI 1640 | Gibco | 11875093 | For macrophages culture |
| Sorvall ST 16/16R centrifuge | ThermoFisher Scientific | 75004240 | For PBMCs and monocytes isolation |
Referencias
- Global Tuberculosis Report 2023. , World Health Organization. Available at: https://www.who.int/tb/publications/global_report/en/ (2023).
- Ahmad, F., et al. Macrophage: A cell with many faces and functions in tuberculosis. Front Immunol. 13, 882130(2022).
- Bo, H., et al. Mycobacterium tuberculosis-macrophage interaction: Molecular updates. Front Cell Infect Microbiol. 13, 1187205(2023).
- Papp, A. C., et al. AmpliSeq transcriptome analysis of human alveolar and monocyte-derived macrophages over time in response to Mycobacterium tuberculosis infection. PLoS One. 13 (5), e0198069(2018).
- Zhai, W., Wu, F., Zhang, Y., Fu, Y., Liu, Z. The immune escape mechanisms of Mycobacterium tuberculosis. Int J Mol Sci. 20 (2), 340(2019).
- Van Crevel, R., Ottenhoff, T. H. M., Van der Meer, J. W. M. Innate immunity to Mycobacterium tuberculosis. Clin Microbiol Rev. 15 (2), 294-309 (2002).
- Mortaz, E., et al. Interaction of pattern recognition receptors with Mycobacterium tuberculosis. J Clin Immunol. 35 (1), 1-10 (2015).
- Stamm, C. E., Collins, A. C., Shiloh, M. U. Sensing of Mycobacterium tuberculosis and consequences to both host and bacillus. Immunol Rev. 264 (1), 204-219 (2015).
- Zihad, S. M. N. K., et al. Role of pattern recognition receptors in sensing Mycobacterium tuberculosis. Heliyon. 9 (10), e20636(2023).
- Barbero, A. M., et al. SLAMF1 signaling induces Mycobacterium tuberculosis uptake leading to endolysosomal maturation in human macrophages. J Leukoc Biol. 109 (8), 257-273 (2021).
- Pasquinelli, V., et al. Expression of signaling lymphocytic activation molecule-associated protein interrupts IFN-γ production in human tuberculosis. J Immunol. 172 (2), 1177-1185 (2004).
- Pasquinelli, V., et al. Phosphorylation of mitogen-activated protein kinases contributes to interferon-γ production in response to Mycobacterium tuberculosis. J Infect Dis. 207 (2), 340-350 (2012).
- Pasquinelli, V., et al. IFN-γ production during active tuberculosis is regulated by mechanisms that involve IL-17, SLAM, and CREB. J Infect Dis. 199 (5), 661-665 (2009).
- Pellegrini, J. M., et al. Neutrophil autophagy during human active tuberculosis is modulated by SLAMF1. Autophagy. 17 (2), 423-426 (2021).
- Song, T., Dong, C., Xiong, S. Signaling lymphocyte-activation molecule SLAMF1 augments mycobacteria BCG-induced inflammatory response and facilitates bacterial clearance. Int J Med Microbiol. 305 (6), 572-580 (2015).
- Eble, J. A. Titration ELISA as a method to determine the dissociation constant of receptor-ligand interaction. J Vis Exp. (132), e57032(2018).
- Patching, S. G. Surface plasmon resonance spectroscopy for characterization of membrane protein-ligand interactions and its potential for drug discovery. Biochim Biophys Acta Biomembr. 1838 (1), 43-55 (2014).
- Draczkowski, P., Matosiuk, D., Jozwiak, K. Isothermal titration calorimetry in membrane protein research. J Pharm Biomed Anal. 87, 296-304 (2014).
- Smith, D. S., Eremin, S. A. Fluorescence polarization immunoassays and related methods for simple, high-throughput screening of small molecules. Anal Bioanal Chem. 391 (5), 1499-1507 (2008).
- García-Nafría, J., Tate, C. G. Cryo-electron microscopy: Moving beyond X-ray crystal structures for drug receptors and drug development. Annu Rev Pharmacol Toxicol. 60, 51-71 (2020).
- Asami, J., Shimizu, T. Structural and functional understanding of the toll-like receptors. Protein Sci. 30 (4), 761-772 (2021).
- Phạm, T. T. T., Rainey, J. K. On-cell nuclear magnetic resonance spectroscopy to probe cell surface interactions. Biochem Cell Biol. 99 (6), 683-692 (2021).
- El Deeb, S., et al. Microscale thermophoresis as a powerful growing analytical technique for the investigation of biomolecular interaction and the determination of binding parameters. Methods Appl Fluoresc. 10 (4), 045001(2022).
- El Khamlichi, C., et al. Bioluminescence resonance energy transfer as a method to study protein-protein interactions: Application to G protein-coupled receptor biology. Molecules. 24 (3), 505(2019).
- Schneider, P., Willen, L., Smulski, C. R. Tools and techniques to study ligand-receptor interactions and receptor activation by TNF superfamily members. Methods Enzymol. 545, 103-125 (2014).
- Park, A. M. W., Schirmer, P. S. H. Atomic force microscopy: A multifaceted tool to study membrane proteins and their interactions with ligands. Biochim Biophys Acta Biomembr. 1838 (1), 74-89 (2014).
- Zalejski, J., Sun, J., Sharma, A. Unravelling the mystery inside cells by using single-molecule fluorescence imaging. J Imaging. 9 (9), 183(2023).
- Zheng, S., Zou, M., Shao, Y., Wu, H., Wang, X. Two-dimensional measurements of receptor-ligand interactions. Front Mol Biosci. 10, 1075587(2023).
- Zlotnikov, I. D., Kudryashova, E. V. Computer simulation of the receptor-ligand interactions of mannose receptor CD206 in comparison with the lectin concanavalin A model. Biochemistry (Moscow). 87 (1), 54-69 (2022).
- Fu, Y., Zhao, J., Chen, Z. Insights into the molecular mechanisms of protein-ligand interactions by molecular docking and molecular dynamics simulation: A case of oligopeptide binding protein. Comput Math Methods Med. 2018, 7604567(2018).
- Farina, B., et al. A novel approach for studying receptor-ligand interactions on living cells' surface by using NUS/T1ρ-NMR methodologies combined with computational techniques: The RGDechi15D-αvβ5 integrin complex. Comput Struct Biotechnol J. 19, 3303-3318 (2021).
- Driessen, N. N., et al. Role of phosphatidylinositol mannosides in the interaction between mycobacteria and DC-SIGN. Infect Immun. 77 (10), 4538-4547 (2009).
- Bansal, K., et al. Src homology 3-interacting domain of Rv1917c of Mycobacterium tuberculosis induces selective maturation of human dendritic cells by regulating PI3K-MAPK-NF-κB signaling and drives Th2 immune responses. J Biol Chem. 285 (47), 36511-36522 (2010).
- Xu, Y., et al. PPE57 induces activation of macrophages and drives Th1-type immune responses through TLR2. J Mol Med. 93 (6), 645-662 (2015).
- Olesen, R., et al. DC-SIGN (CD209), pentraxin 3 and vitamin D receptor gene variants associate with pulmonary tuberculosis risk in West Africans. Genes Immun. 8, 456-467 (2007).
- Berger, S. B., et al. SLAM is a microbial sensor that regulates bacterial phagosome functions in macrophages. Nat Immunol. 11 (10), 920-927 (2010).
- Degos, C., et al. Omp25-dependent engagement of SLAMF1 by Brucella abortus in dendritic cells limits acute inflammation and favours bacterial persistence in vivo. Cell Microbiol. 22 (4), e13156(2020).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoExplorar más artículos
This article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados