Method Article
結核菌と免疫受容体SLAMF1との相互作用の研究のための蛍光アッセイ
要約
この研究は、 結核菌 とSLAMF1微生物センサーとの相互作用を評価するためのプロトコルを提供します。アッセイは、フローサイトメトリーおよび蛍光顕微鏡法を用いて、ヒト単球由来マクロファージで実施しました。記載されているツールは、病原体と免疫受容体との間の相互作用の研究に関連しています。
要約
病原体と免疫受容体との間の直接的な相互作用の評価には、通常、高度な技術が含まれるか、トランスジェニック株や遺伝子操作細胞の使用を意味します。ここでは、マクロファージ微生物センサーSLAMF1と 結核菌 との間の生化学的相互作用を検出するための代替方法について説明する。フローサイトメトリーと蛍光顕微鏡法を用いた2つの技術的アプローチが開発されました。ヒトマクロファージからの全細胞タンパク質抽出物を作製し、 結核菌 (WCMtb)または 結核菌 抗原(Mtb Ag)の全細胞と4°Cで一晩インキュベートし、最後にホルムアルデヒド/グリシン/エチレングリコールビス(コハク酸スクシンイミジル)処理を用いて架橋しました。 フローサイトメトリーによるSLAMF1とWCMtbとの相互作用は、PE特異的抗SLAMF1抗体で検出されました。蛍光顕微鏡による相互作用の存在は、Rhodamine-PEで染色したMtb Agsをpoly-D-lysineでコーティングしたスライドに付着させ、単球由来マクロファージの全タンパク質抽出物とインキュベートすることによって行いました。架橋処理後、一次抗体(抗SLAMF1抗体)および二次抗体(Alexa Fluor 488)を用いてSLAMF1を可視化しました。このアッセイは、病原体と免疫受容体の相互作用を測定するための強力な生化学的ツールを提供し、トランスジェニック細胞株やタンパク質遺伝子発現調節実験に関連する困難を克服しました。
概要
結核菌は、142年前に特定された結核の原因病原体であり、現在でも世界人口の少なくとも4分の1が感染しており、世界的な課題となっています1。結核菌は、感染者からの空気中の飛沫を介して感染し、肺の肺胞マクロファージに到達し、潜伏状態で長期間生存することができます2,3。局所的なマクロファージ応答が活性化されるだけでなく、近年では、末梢単球を気道に動員して肺胞マクロファージに分化させることができ、結核菌に対して胎児由来のものよりもさらに強力な応答を引き起こすことができることが説明されています2,4。
マクロファージは自然免疫の基本的なプレーヤーです。結核菌を摂取すると、マクロファージは、炎症誘発性サイトカインの分泌、ファゴリソソームの融合、結核菌を殺すための他の免疫細胞の活性化など、多数の殺菌機能を示します2,3。しかし、この病原体の複雑な構造は、マクロファージの代謝と機能を調節できるエフェクター(タンパク質や脂質など)を提供し、その結果、炎症過程3を調節します。このマクロファージ応答の操作は、病原性因子の分泌または宿主因子の利用を通じて、結核菌によって発揮されるさまざまな回避戦略につながります。いくつかの主要な回避メカニズムには、抗炎症性サイトカインの誘導、ファゴリソソームの成熟と酸性化の阻害、酸化ストレスの変化、オートファジーの混乱、および抗原処理と提示中の欠損が含まれます3,5。
マクロファージと結核菌との間の厳密に制御された相互作用は、適切な免疫応答の発達に不可欠です。したがって、これらのシナプスを研究することは、宿主と病原体のクロストークによって誘発される免疫防御的または免疫病原性のメカニズムを特定し、潜在的な治療標的を特定するための鍵となります。結核菌の認識および/または内在化を媒介する受容体は、TLR6,7,8、NLR7,8、補体受容体6,8、C型レクチン受容体6,8、スカベンジャー受容体6,8など、多くの受容体が結核菌の認識および/または内在化を媒介します.蓄積されたデータは、表面結合PRRと細胞内PRRの両方が感染中に重要な役割を果たし、オプソニン化および非オプソニン化M.tuberculosisを認識することを示しています。
ZihadとSifatらは最近、自然細胞による結核菌誘発応答におけるPRRの関与についてレビューした9。特に、TLRファミリーのほとんどのメンバーは、結核菌リガンドとの相互作用に関与している6,7。表面TLR2は、アシル化リポタンパク質、19-kDaリポタンパク質、LprAリポタンパク質、LprGリポタンパク質、30-kDa抗原、38-kDa抗原、MymA、プロリン-プロリン-グルタミン酸(PPE)-57、LAM、LM、PIM、熱ショックタンパク質60、シグネチャータンパク質Rv1509、分泌タンパク質ESAT-6などのマイコバクテリア抗原を認識します7。メンブレンTLR4は、熱ショックタンパク質、38-kDa抗原、RpfE、Rv0652、Rv0335c、Rv2659c、Rv1738、Rv2627c、Rv2628、GrpE、およびHBHAと相互作用します。一方、エンドソームTLR(TLR3、7、8、9)のリガンドには、結核菌のdsRNA、tRNA、ssRNA、ファゴソームRNA、dsDNAなどがあります。さらに、TLR2は、TLR1およびTLR6、TLR4、およびTLR9と連動して作用する場合、免疫応答を開始する上で積極的な役割を果たします6,7。NOD2 と NLRP3 は、結核で最も特徴付けられた細胞質 NLR です。それらの特異的なリガンドはまだ研究中ですが、これらの受容体はムラミルジペプチドまたはESAT-6によってそれぞれ活性化されます7,8。C型レクチン受容体は、古典的に結核菌のエンドサイトーシスに関与しています。Dectin-2は結核菌の細胞壁のManLAMの直接的なPRRとして機能しますが、Dectin-1リガンドはまだ発見されていません8。MincleとMCLはどちらも、コードファクター7とも呼ばれる糖脂質トレハロース-6,6′-ジミコレート(TDM)を認識します。DCARは、マイコバクテリアの糖脂質であるホスファチジル-ミオ-イノシトールマンノシド(PIM)と相互作用します7。CR3はマイコバクテリアのLAMとPIMを認識し、DCサインはManLAMを結紮し、MRはManLAM、PIM、LM、38-kDa糖タンパク質、19-kDa抗原、その他のマンノシル化タンパク質を含む多くの結核菌成分を認識しますが、DCIRリガンドはまだ同定されていません。
可溶性CLRには、ManLam、LM、60-kDa糖タンパク質、および糖タンパク質Apaを認識するSP-Aが含まれます。SP-DがLAM、LM、およびPILAMと相互作用する。MBLは、ManLAM認識6,7を専門としています。SR-AとMARCOはTDMをリガンドとして持ち、SR-B1はESAT-6を認識し、CD36はManLAMとLM7,8に結合します。さらに、Dectin-1、Mincle、およびMARCOは、TLR2またはTLR4と組み合わせて、結核菌PAMPを検出した後に信号をトリガーすることもできます6,7。CD14は、非オプソニン化細菌を内在化する能力を持ち、熱ショックタンパク質であるシャペロニン60.1も認識する表面受容体です。特に、CD14はMARCOおよびTLR2とともに共受容体として機能します6。AIM2は、結核菌がファゴソーム8から脱出する際にssDNAを感知できる細胞質受容体である。最後に、AhRは、結核菌6由来の色素性病原性因子ナフトキノンフチオコールに結合するリガンド活性化転写因子です。
上記の相互作用の多くは仮定されており、厳密には示されていません。特定の受容体のリガンドでさえ不明のままであり、結核菌の免疫認識の分野をより深く理解する必要性が高まっています。これに関連して、共刺激分子SLAMF1(Signaling Lymphocytic Activation Molecule)は、最近、Barberoらによって結核菌受容体として報告されました10。シグナル伝達分子としてだけでなく、結核菌のセンサーとしても働くことで、SLAMF1は結核において特に興味深い役割を果たしています。SLAMF1は、Erk/CREBリン酸化11,12,13、好中球のオートファジー14、マクロファージ15の細菌クリアランスを通じて、T細胞によるIFN-γ産生などの防御機能を調節することにより、免疫細胞の活性化を誘導することができる。
受容体-リガンド相互作用は、ELISA、表面プラズモン共鳴(SPR)、等温滴定型熱量測定(ITC)、蛍光偏光(FP)、X線結晶構造解析、核磁気共鳴(NMR)分光法、マイクロスケール熱泳動(MST)、共鳴エネルギー移動(BRETやFRETなど)共焦点顕微鏡、電子顕微鏡、クライオ電子顕微鏡(クライオEM)および原子間力顕微鏡(AFM)などの技術を使用して長年にわたって研究されてきました16,17。18、19、20、21、22、23、24、25、26、27、28。これらのアプローチの一部は、レポーター遺伝子、標識組換えタンパク質またはキメラ分子、ノックアウト、ノックダウン、または過剰発現モデルの使用を意味します。あるいは、計算ツールは、受容体-リガンド相互作用および結合部位を予測することができ、相互作用の包括的な理解を得るために生物学的アプローチと組み合わせて使用されることが多い29,30,31。ここでは、生化学的相互作用を検出するための2つの選択肢と、細菌を蛍光標識する方法に関するセクションについて説明します。in vitroでの受容体と病原体の相互作用の研究を可能にするプロトコルが提示され、特にフローサイトメトリーと蛍光顕微鏡法によるSLAMF1-M.結核の関与の評価が提示されます。
プロトコル
ヒト単球由来マクロファージを含むすべての手順は、ヘルシンキ宣言(2013年)に従って、UNNOBAの倫理委員会(COENOBA)の合意に基づいて実施されました。サンプル収集前に書面によるインフォームドコンセントが得られました。男性/女性グループの分布は13/6で、年齢の中央値は32歳で、四分位範囲(IQR)は18〜75歳でした。以前の病状、併存疾患の存在、または結核の陽性診断は、除外基準として定義されました。試薬と機器の詳細は 、材料表に記載されています。
1. 単球由来マクロファージの培養と刺激
- 採血
- 健康なドナーから 60 mL の血液を、ヘパリン注射器での静脈穿刺により採取します。
注:採血は、生化学者または瀉血技術者が行う必要があります。 - 鋭利物廃棄容器に針を捨て、ドナーの血液と接触した材料は病理学的廃棄物容器に捨てます。.
- 健康なドナーから 60 mL の血液を、ヘパリン注射器での静脈穿刺により採取します。
- 単球単離とマクロファージ生成
- 血液を慎重に50mLのチューブに移し、生理食塩水で半分に希釈します。
- 密度勾配培地で遠心分離を行い、末梢血単核細胞(PBMC)を取得します。
- 白っぽいハローからPBMCを収集します。
- 生理食塩水(400 x g )で10分間2回洗浄し、最終洗浄(200 x g で20°Cで15分間)を行い、血小板を除去します。
- 細胞計数チャンバー内のPBMCの数を決定し、CD14ビーズを用いてCD14ポジティブ磁気選択を行います。
注意: 磁気選択中は、製造元の指示に注意深く従ってください。さらに、フローサイトメトリーを使用して単離された単球の純度を確認できます。 - 単離した細胞をRPMI 1640に再懸濁し、細胞計数チャンバー内の単球の数を決定します。
- 24ウェル細胞培養プレートに1ウェルあたり1個×10個6/ mLのCD14陽性選択単球500μLを播種し、付着を促進します。
- 2時間後、非接着性細胞を予熱したRPMI 1640で洗浄して除去します。
- マクロファージを分化するには、接着性単球を1 mLの完全培地(RPMI 1640培地にL-グルタミン、10%ウシ胎児血清(FBS)、100 U/mLのペニシリン、100 μg/mLのストレプトマイシンを添加)でさらに16〜18時間(一晩)培養します。
- 翌日、培地を交換します。培地を取り出し、1x PBS1 mLで洗浄し、1 mLの完全培地を加えます。
- 超音波処理された結核菌(結核菌抗原(Ags))でマクロファージを24時間刺激します。単球由来マクロファージ10×10×6個につき10μLの結核菌Ags(10μL=1010.6個の細菌)を使用してください。
注:10 μLの 結核菌 Ags(Mtb Ags)= 1 × 106 細菌、分光光度計でO.D. 600nm で定量、O.D. 1を1 x 108 bacteria/mLとみなして定量。BSL2バイオセーフティキャビネットとインキュベーターを37°Cの5%CO2 の雰囲気ですべての培養ステップに使用します。
- SLAMF1発現の確証
- 1つのウェルからマクロファージを採取します。培養プレートを冷たい面に置きます。1 mLのコールドFACS(1x PBS + 2% FBS)を2回連続して加えて、上下の動きで細胞を剥離します。コレクションにはサイトメトリー丸底チューブを使用してください。
- 細胞を500 x g で4°Cで5分間遠心分離します。 上清を捨てて捨てます。
- 蛍光色素共役抗ヒトSLAMF1抗体で、暗所4°Cで30分間細胞を染色します。1.25 μLの抗体を添加した後、ボルテックスします。
- FACSを使用して細胞を洗浄して過剰な抗体を排除し、ペレットをFACSに再懸濁し、フローサイトメーターを使用してサンプルを採取します。
注:1.25 μLの抗SLAMF1抗体は、0.5 x 106 マクロファージの標識に使用される0.0625 μgに相当します。ただし、抗体の滴定を行い、対応するアイソタイプまたは蛍光マイナス1(FMO)コントロールを行うことを強く推奨します。
2. 全細胞タンパク質抽出物の調製
- 溶解バッファーの調製
- NaCl 150 mM、Tris 10 mM、EDTA 5 mM、SDS 1%、Triton X-100 1%、およびデオキシコール酸ナトリウム1%を含むRadio Immunoprecipitation Assay(RIPA)バッファーを調製します。
- RIPAバッファーに1 mMフェニルメチルスルホニルフッ化物(PMSF)と、ペプスタチンAやロイペプチンなどのプロテアーゼ阻害剤を補充します。
- 細胞収集とタンパク質の単離
- P1000ピペットを使用してプレートウェルから完全なRPMIを廃棄し、コールド1x PBSで上下にピペッティングしてマクロファージを回収します。
- 細胞を1.5 mLの微量遠心チューブに移します。マイクロチューブごとに1つのウェルを収集します。
- 500 x g で4°Cで5分間遠心分離します。 上清を捨てます。
- 1 mLの冷たいPBSを加え、ステップ2.2.2と2.2.3を繰り返して、同じ微量遠心チューブに細胞を回収します。
- 細胞ペレットを1x PBSの100 μLに再懸濁し、4本のチューブをプールします。手順2.2.3を繰り返します。各チューブには、2 x 106 個のマクロファージが含まれます。
- 100 μLのRIPAバッファーを添加し、懸濁液を氷上で1時間インキュベートし、10分ごとにボルテックスします。
- 14,000 x g で15分間遠心分離します。上清を回収し、-20°Cで使用するまで保存します。
3. 結核菌-フローサイトメトリーによるSLAMF1相互作用
- タンパク質-細菌架橋
- 1 × 106 個の不活化 結核菌 細胞 (WCMtb, M . tuberculosis inactivated by gamma iljunction) を 1 x 106 マクロファージ (50 μL) のタンパク質抽出物と共に、回転マイクロチューブホルダーで 4 °C で一晩インキュベートします。
注:10 μL の WCMtb = 1 × 106 バクテリア、分光光度計で O.D. 600nm で定量、O.D. 1 を 1 x 108 バクテリア/mL と見なします。 - 翌日、1x PBSで希釈した1%ホルムアルデヒド500μLを加えます。室温(RT)で撹拌(シェーカー)下で15分間インキュベートします。
- 水で希釈した0.125 Mグリシン25 μLを加えます。RTで撹拌(シェーカー)下で5分間インキュベートします。
- 1x PBS1 mLを14,000 x g で5分間2回洗浄します。洗浄ステップの間に上清を捨てます。
- ペレットを再懸濁し、懸濁液を500 μLの2 mMエチレングリコールビス(コハク酸スクシンイミジル)(EGS)架橋剤にインキュベートし、氷酢酸:水の1:1混合物で希釈し、室温で1時間インキュベートします。
注:使用時の試薬の調製を強くお勧めします。EGSを調製するには、氷酢酸と水を同じ割合で混合し、70°Cに加熱する必要があります。 溶液が高温でない場合、EGSは完全には再懸濁されません。
- 1 × 106 個の不活化 結核菌 細胞 (WCMtb, M . tuberculosis inactivated by gamma iljunction) を 1 x 106 マクロファージ (50 μL) のタンパク質抽出物と共に、回転マイクロチューブホルダーで 4 °C で一晩インキュベートします。
- SLAMF1染色
- 1回目の洗浄では500 μLの1x PBSを、2回目の洗浄では1 mLを使用して、ステップ3.1.4を繰り返します。
- タンパク質-バクテリア複合体を暗所4°Cで30分間、抗ヒトSLAMF1抗体(ステップ1.3.3と同様)で染色し、抗体をマイクロチューブに添加してボルテックスします。1 x 106 細胞に適した量の抗体を使用してください(2.5 μLまたは0.125 μg)。
- FACSを使用して細胞を洗浄して過剰な抗体を除去し、ペレットをFACSに再懸濁し、フローサイトメーターでサンプルを採取します(ステップ1.3.4を参照)。
注意: 対応するアイソタイプまたはFMOコントロールを実行します。
4. 結核菌 抗原標識
- ローダミンBをエタノールにストック濃度5 mg/mL(1000x)で再懸濁します。使用するまで-20°Cで保管してください。
- 1.5 mLの微量遠心チューブに、 結核菌 Ags100 μLあたり1 μLのローダミンBを添加します。
- 連続撹拌(ボルテックス)下でRTで45分間インキュベートします。
- 上清が透明になるまで、1x PBS1 mLを使用して、14,000 x g 、24°C、5分間、少なくとも3回洗浄します。ペレット(Mtb-R Ags)は淡いピンク色に見えます。
- Mtb-R Ags を 1x PBS 100 μL に再懸濁して、初期容量を回復します。
注:無菌条件下での作業をお勧めします。染色は実験を行う直前に行ってください。
5. 結核菌-SLAMF1の蛍光顕微鏡による相互作用
- 細菌-タンパク質架橋
- 丸い鼻の外科用ピンセットを使用して、12 mmの丸いカバースリップを24ウェル培養プレート(ウェルごとに1枚のカバースリップ)に導入します。
注意: カバースリップを70%エタノールで完全に清掃します。必要に応じて、UV光の下で30分間オートクレーブまたは滅菌することができます。 - カバーガラスを400 μLの10 μg/mL Poly-D-Lysineと4°Cで一晩インキュベートします。 プレートをアルミホイルで覆い、光から保護します。
- 翌日、プレートを冷蔵庫から取り出し、カバーガラスを1mLの水で2回洗います。
- カバースリップを乾かします。
- 20 μL (2 x 106) の Mtb-R Ags を 1x PBS の 180 μL で希釈します。最終的な200μLは、カバーガラスを完全に覆うのに十分です。
- カバーガラスをMtb-R AGsと共溶かし、細胞インキュベーター内で37°Cで1時間インキュベートします。
- 残った液体を捨て、1mLの1x PBSで2回洗浄します。
注:このステップでは、赤チャンネルに対応するフィルターを使用して、蛍光顕微鏡でMtb-Rのカバーガラスへの正しい接着を確認できます。 - 400 μLのブロッキングバッファー(1x PBS中の10% FBS)をRTで30分間、撹拌(シェーカー)で加えます。
- 400 μLのブロッキングバッファーで2回洗浄します。
- 1x PBSの300 μLで希釈したタンパク質抽出物100 μLと室温で2時間インキュベートします(シェーカー)。
- 1x PBSで希釈した1%ホルムアルデヒド400 μLを加えます。RTで撹拌(シェーカー)下で15分間インキュベートします。
- 500 μLのグリシン0.125 Mを水で希釈して加えます。RTで撹拌(シェーカー)下で5分間インキュベートします。
- 1x PBS1 mLで2回洗浄します。
- 2 mM EGSを400 μLをRTで1時間加えます(シェーカー)。
- 手順5.1.13を繰り返します
注:使用時の試薬の調製を強くお勧めします。
- 丸い鼻の外科用ピンセットを使用して、12 mmの丸いカバースリップを24ウェル培養プレート(ウェルごとに1枚のカバースリップ)に導入します。
- SLAMF1染色
- 培養プレートの蓋をパラフィンフィルムで包みます。
- パラフィンフィルムで覆われた蓋に、以前に滴定した抗SLAMF1一次抗体60 μL (希釈度1:75) を滴下します。
- 湾曲した細い先端の外科用ピンセットと針を使用して、カバースリップをプレートから慎重に取り外します。
注意: カバースリップの取り外しを容易にするために、針は約45°の角度で曲げることができます。 - 抗体を含む液滴の上にカバースリップを反転させます。暗闇の中でRTで30分間インキュベートします。
- カバースリップを持ち上げ、ブロッキングバッファーで2回洗います。
- カバースリップを裏返して、二次抗体を含む新しい滴(1:200希釈)にかけます。暗闇の中でRTで30分間インキュベートします。
- 手順5.2.5を繰り返します。
- 余分な液体を取り除き、スライドガラスの上に置いた取り付け液の滴にカバースリップを取り付けます。
- 適切なフィルターを使用して蛍光顕微鏡で観察します。
注:無菌条件下での作業をお勧めします。ブロッキングバッファーで抗体を希釈します。カバーガラスは、顕微鏡での観察を妨げる可能性のある傷を避けるために慎重に取り扱ってください。蛍光に適した埋込液を強くお勧めします。
結果
この研究では、ヒトマクロファージにおける 結核菌 と免疫受容体SLAMF1との相互作用の評価を可能にするプロトコルが提供されます(図1)。この目的のために、健康なドナーから末梢血が得られた。次に、PBMCを密度勾配培地上で遠心分離して分離し、単球を磁気ポジティブセレクション(純度≥95%、 図2A、B)によって単離しました。単離された単球をプラスチック培養プレートに2時間接着して単球由来マクロファージを得た後、ONで培養した。その後、マクロファージを超音波処理した 結核菌 (Mtb Ags)で刺激し、10以前に報告されたSLAMF1表面発現を誘導し、表面SLAMF1レベルをフローサイトメトリーで確認した(図2C)。).マクロファージは、最終的にRIPA溶解バッファーとボルテックスおよび氷上でのインキュベーションを組み合わせて溶解し、細胞溶解を促進しました。生成された全タンパク質抽出物を相互作用アッセイに使用しました。
最初の一連の実験手順では、フローサイトメトリーを使用して、結核菌(WCMtb)の全細胞とのSLAMF1相互作用を研究しました(図3)。まず、SLAMF1(タンパク質抽出物中の含有量)とWCMtbとの間の物理的相互作用を、4°Cの連続回転で一緒にインキュベートすることにより促進しました。 その後、架橋治療を行い、特異的な抗SLAMF1抗体を用いて、WCMtbに結合したSLAMF1受容体を検出しました。このアッセイでは、WCMtbのみを使用して、SLAMF1結合を検出する対象集団であるフローサイトメーターで結核菌のサイズと粒度に対応する電圧を設定しました。
2番目の実験的アプローチでは、蛍光顕微鏡を使用してSLAMF1-M.結核 の相互作用を検出しました(図4)。超音波処理された結 核菌 (Mtb Ags)を使用したこのアッセイは、前のものを補完するものです。ローダミンB染色Mtb Ags(Mtb-R Ags)を使用して、PE蛍光色素に対応するチャネル/フィルターでの蛍光検出を容易にしました。ポリ-D-リジンコーティングされたカバースリップにMtb-R Agを付着させ、顕微鏡観察により染色成功を確認しました(図4A)。その後、Mtb-R Agsでコーティングしたスライドを、SLAMF1を含むタンパク質抽出物とインキュベートしました。架橋処理後、SLAMF1は、一次特異的抗SLAMF1抗体と続いて、PEと共観察する適合蛍光色素に結合した二次抗体を用いて検出した。Mtb-R AgsとSLAMF1の2つのチャンネルで個々の蛍光顕微鏡画像を取得した後、画像マージによって相互作用が確認されました(図4B)。
これらのアプローチを行ったところ、SLAMF1と結核菌との間に相互作用が存在することが実証されました。SLAMF1と全菌および超音波処理細菌の両方とのクロストークが検出され、SLAMF1が結核菌10の新たなヒト自然マクロファージ受容体であることを示す仮説に力を与えました。

図1:SLAMF1-M.結核 の相互作用を検出するためのワークフロー。 末梢血は、静脈穿刺によって健康なドナーから採取され、密度勾配培地で遠心分離されて末梢血単核細胞(PBMC)を単離されました。細胞数を決定した後、PBMCを正の磁気選択にかけ、CD14pos 単球を精製した。次いで、単球をウシ胎児血清の非存在下で培養プレートに2時間接着し、次いで補充されたRPMI(休止ステップ)で一晩培養して、単球由来マクロファージを得た。その後、マクロファージを超音波処理した 結核菌 (Mtb Ags)で24時間刺激し、SLAMF1表面発現を誘導した。SLAMF1レベルは、マクロファージ集団の一部でヒト抗SLAMF1特異的抗体を使用したフローサイトメトリーによって試験されました。残りのマクロファージを使用して、総タンパク質抽出物を取得しました。細胞溶解は、ボルテックスと氷上でのインキュベーションを溶解バッファー内で組み合わせることによって行いました。最後に、SLAMF1と 結核菌 との相互作用を、マクロファージタンパク質をMtb Agsまたは結核 菌 の全細胞(WCMtb)と架橋した後、フローサイトメトリーおよび蛍光顕微鏡法によって評価しました。 この図の拡大版を表示するには、ここをクリックしてください。

図2:実験的および技術的対照 (A)CD14pos単球を正の磁気選択によりPBMCから単離した。得られた陽性(単球、青)および陰性(赤)の細胞画分をフローサイトメトリー(SSC-A対FSC-A)を使用して、選択が成功したことを確認します。(B)陽性画分の純度は、細胞の粒度とサイズを分析するフローサイトメトリーによって裏付けられました(SSC-A対。FSC-A)です。(C)単球由来マクロファージは、SSC-A対SSC-Aによって選択されました。FSC-Aとゲート化してダブルシングレット戦略(FSC-A対.FSC-Hに続いてSSC-AとSSC-H)です。SLAMF1表面発現をシングルセルで評価しました。すべての場合(A-C)で、代表的なプロットまたはヒストグラムが表示されます。この図の拡大版を表示するには、ここをクリックしてください。
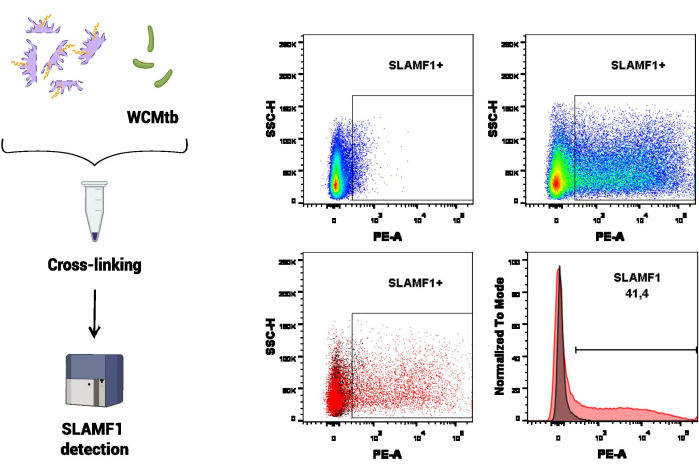
図3:フローサイトメトリーによるSLAMF1-M.結核 の相互作用。 マクロファージタンパク質と 結核菌 の全細胞(WCMtb)との間の架橋は、プロトコールのセクションに記載されているように、ホルムアルデヒド/グリシン/エチレングリコールビスによる治療によって行われました。次に、目的の集団を選択した後に陽性のSLAMF1(PE)シグナルを検出することにより、フローサイトメトリーによって相互作用を評価しました(SSC-A 対。FSC-A)です。上段のパネルは、蛍光マイナス1(FMO)コントロール(左)とポジティブ染色(右)を比較したSLAMF1の陽性シグナルを示しています。下のパネルには、SLAMF1信号とFMO制御器の間のオーバーレイ(左、ドットプロット、右、ヒストグラム)が表示されます。 この図の拡大版を表示するには、ここをクリックしてください。

図4:蛍光顕微鏡によるSLAMF1-M.結核 の相互作用。 (A)Mtb AgをローダミンB(Mtb-R Ags)で染色し、丸いカバースリップに貼り付けました。Mtb-R Agsの正しい接着性と蛍光性は、赤色チャネルに対応するフィルターを用いた蛍光顕微鏡法によって確認されました。(B)マクロファージタンパク質とMtb-R Agsとの間の架橋は、プロトコールのセクションに記載されているように、ホルムアルデヒド/グリシン/エチレングリコールビスで処理することにより、付着したMtb-R Ags上にタンパク質抽出物を添加することにより行った。この相互作用は、ヒト抗SLAMF1抗体と続いてAlexa Fluor 488標識二次抗体を用いた蛍光顕微鏡法で評価しました。上の顕微鏡写真は、SLAMF1(左、緑)とMtb-R Ags(右、赤)の陽性シグナルを示しています。低い顕微鏡写真は、黄色の相互作用(両方のチャネルのマージ)を示しています。スケールバーは10μmに対応しています。 この図の拡大版を表示するには、ここをクリックしてください。
ディスカッション
この研究は、 結核菌 と、結核中の宿主応答に関与する主要な細胞タイプであるヒトマクロファージに発現する微生物センサーとの間の生化学的相互作用を研究するための有用なガイドを提供します。提供されるプロトコルは、 結核菌 の食細胞への侵入に関与する分子の解読に関連しています。
病原体と免疫受容体との間の相互作用など、生体分子相互作用を特徴づけることは、結核菌が誘発する免疫保護メカニズムと回避戦略の両方を理解するために重要です。多くの場合、受容体とその細菌リガンドとの直接的な相互作用を実証することは複雑であり、高度な技術や汎用性に欠けるシステムが必要になる場合があります。Driessenらは、M. bovis BCG32の変異体欠損株を用いて、マイコバクテリアとDC-SIGNとの間の相互作用におけるホスファチジルイノシトールマンノシドの役割を研究しました。彼らはこれらの相互作用を研究することに成功したが、著者らは変異株の違いを見つけられず、結合の減少を示す突然変異細菌の作製が大きな課題になると議論した32。他の著者は、TLR2と結核菌の一部のPE/PPEタンパク質との間の相互作用を実証するために、優れた骨の折れる研究を行いました33,34。これらの研究では、受容体のためのノックアウトマウスの使用、細胞のトランスフェクション、および前述のタンパク質または特定のタンパク質ドメインの精製が必要であった33,34。さらに、受容体の機能はしばしば推測されますが、相互作用は確実に示されません。一例は可溶性受容体PTX3であり、疾患転帰に関連するハプロタイプが発見されていますが、結核菌との特異的な相互作用は厳密には研究されていません35。これらの欠点に関連して、私たちの方法は、研究中の標的受容体と結核菌との相互作用を簡単な方法で、簡単に解釈できるアクセス可能な技術で評価することを可能にします。
前述の点、特にSLAMF1については、大腸菌36のOmpCおよびOmpF、およびB.abortus37のOmp25との相互作用が以前に評価されている。Degosら37は、SLAMF1の細胞外ドメインのみをコードするプラスミドをトランスフェクトしたCOS-7細胞を使用してSLAMF1を精製し、ウェスタンブロットによるOmp25との相互作用を解析しました。また、SLAMF1-/-マウスと野生型B.abortusまたはOmp25欠損変異体を用いた実験も行った。Berger et al.36の研究はまた、SLAMF1と大腸菌OmpおよびS.typhimuriumSseB-との間の直接的な相互作用の説得力のある証拠を示しました。彼らは、SLAMF1-/-マウス、SLAMF1mCherryコンストラクトをコードするRAW264.7マクロファージ、SLAMF1トランスフェクトJurkat T細胞を使用した増幅アッセイなど、さまざまなアプローチを使用しました。彼らは、1 x 108E. coliを使用して、ルシフェラーゼレポーターによる高感度シグナル増幅アッセイを開発しました。この点で、この報告で提示された生化学的相互作用アッセイの1つの強みは、相互作用が実際に野生型細胞と、以前の研究10で機能評価に使用された細胞:細菌の同じ比率を使用して、SLAMF1発現を過剰発現または無効化することなく発見されたことです。
提供されるプロトコルには、いくつかの重要な手順が伴います。その1つは、相互作用アッセイを実施する前のヒトマクロファージにおけるSLAMF1発現の確証です。これは、実際には受容体の発現不足による偽陰性の結果を避けるために重要です。ここでは、SLAMF1レベルをフローサイトメトリーを使用してチェックしました。これは、このデータを2時間未満で取得できる迅速な手法であるためです。ただし、顕微鏡法やウェスタンブロットなどの他の手法を使用することもできます。タンパク質レベルで受容体を研究することが最終目標である場合に、mRNAレベルを特定するリアルタイムPCRなどの方法論はお勧めしません。もう1つの基本的なステップは、架橋段階です。架橋剤の使用は、タンパク質間相互作用の安定化を目指しています。フローサイトメトリーに基づくアッセイでは、架橋ステップを適用しなければSLAMF1のシグナルを検出することはできませんでした。顕微鏡法の場合、架橋治療を使用せずに、非常に低い感度で相互作用が観察されました。したがって、架橋ステップはすべての場合に強くお勧めします。
記載されたプロトコルは、SLAMF1と病原体の表面でアクセス可能な 結核菌 抗原との間の相互作用の証拠を提供し、SLAMF1が 結核菌に存在する分子シグネチャを認識することを実証するのに役立ちます。ただし、このモデルにはいくつかの制限があります。弱点は、このプロトコルでは、どの特定の 結核菌 抗原がSLAMF1と相互作用する抗原であるかを特定できないことです。それにもかかわらず、この目的を達成するために、例えば、細菌から精製された抗原を使用して、プロトコルを変更することができる。別の制限は、どの刺激がマクロファージでSLAMF1の発現を誘導するかを知る必要があることです。これは、選択された受容体が基礎状態で発現しない場合、または発現パターンが不明な場合に合併症を意味する可能性があります。最後に、これらのアプローチは、SLAMF1が結 核菌と相互作用するために他の分子を必要とするかどうかを区別するものではありません。この場合、免疫沈降アッセイを実施するか、方法論的スキームでより多くの抗体を使用することができます。
このレポートで提案されている方法論は、他の免疫受容体の研究、それが発現する他の細胞タイプの同じ受容体の研究、または他の細菌や異なるマイコバクテリアの株との相互作用の評価に容易に適応できます。さらに、ここでは、超音波処理された 結核菌を蛍光染色するための短くて簡単なプロトコルが提供されており、他の細菌や生きた株にも適用できる標識です。しかし、この手順ではSLAMF1-M.結核 のクロストークに焦点を当てており、他の潜在的な用途には、ブロックされた、遅延した、または不適切な相互作用の結果を理解すること、回避メカニズムを研究すること、または病原体の認識に関与する潜在的な分子標的を明らかにすることが含まれます。同様に、このプロトコルは、 結核菌 とマクロファージまたは他の宿主細胞の共進化を理解するための新しい治療戦略および免疫療法の研究に適用でき、リガンド受容体ダイナミクスの理解を求めるさまざまな分野でも使用できます。
開示事項
著者は、利益相反を宣言しません。
謝辞
本研究は、ブエノスアイレス国立州立大学(グラント番号SIB 0618/2019、SIB 2582/2012からV.P.)、Agencia Nacional de Promoción Científica y Tecnológica(ANPCyT、グラント番号PICT-2012-2459およびPICT A 2017-1896からV.P.、PICT-2021-I-INVI-00584からA.B.)の支援を受けました。フロレンシオ・フィオリーニ財団;Consejo Nacional de Investigaciones Científicas y Técnicas (CONICET、助成金番号 PIO 15720150100010CO to V.P.)。技術サポートを提供してくれたNatalia MeniteとGastón Villafañeに感謝します。この研究で提示されたアッセイの開発を引き起こした出版中の科学的な議論に対して、Paula Barrionuevo博士とLuciana Balboa博士に感謝します。最後に、クロスリンクプロトコルに関するアドバイスをいただいたEstermann博士に感謝します。Moroniは、Poly-LysineとLicでの作業に関するアドバイスをいただきました。モリコーニは、概略図の協力をしてくれました。
資料
| Name | Company | Catalog Number | Comments |
| Alexa Fluor 488 secondary antibody | Invitrogen | A21121 | For fluorescence microscopy |
| anti-SLAMF1 FITC antibody | eBioscience | 11-1509-42 | For flow cytometry |
| anti-SLAMF1 PE antibody | BioLegend | 306308 | For flow cytometry |
| anti-SLAMF1 primary antibody | BioLegend | 306302 | For fluorescence microscopy |
| Aqua-Poly/Mount | Polysciences | 18606-20 | Mounting media |
| CD14 MicroBeads | Miltenyi Biotec | 130-097-052 | For monocytes isolation |
| Coverslips 12mm | HDA | - | For interaction assay by microscopy |
| EGS | ThermoFisher Scientific | 21565 | For crosslinking treatment |
| FACSCanto II | BD Biosciences | 338960 | Flow cytometer with BD FACSDiva software |
| Fetal Bovine Serum | Natocor | - | Inactivated and irradiated, for macrophages culture |
| Ficoll-Paque PLUS | Cytiva | 17144003 | For PBMCs separation |
| Fiji/ImageJ | Open Source software | - | For micrographs analysis |
| FlowJo 7.6.2 | Tree Star | - | For flow cytometry analysis |
| Formaldehyde | Merck | K47740803613 | For crosslinking treatment |
| Glass slides | Glass Klass | - | For interaction assay by microscopy |
| Glycine | Sigma | G8898 | For crosslinking treatment |
| Imager.A2 | Carl Zeiss | 430005-9901-000 | Fluorescence microscope with Colibri 7 illumination module |
| iMark | BIO-RAD | 1681130 | Microplate absorbance reader |
| L-glutamine | Sigma Aldrich | 49419 | For macrophages culture |
| M. tuberculosis, strain H37Rv, gamma-irradiated whole cells | BEI Resources, NIAID, NIH | NR-14819 | For interaction assay |
| M. tuberculosis, strain H37Rv, whole cell lysate | BEI Resources, NIAID, NIH | NR-14822 | For macrophages stimulation and interaction assay |
| Neofuge 13R | Heal Force | Neofuge 13R | High Speed Refrigerated Centrifuge for protein extraction |
| Penicillin/Streptomycin | Gibco | 15140122 | For macrophages culture |
| PMSF | ThermoFisher Scientific | 36978 | For proteins isolation |
| Poly-D-Lysine | Sigma Aldrich | A-003-M | For coverslips treatment |
| Protease Inhibitor Cocktail | Sigma Aldrich | P8340 | For proteins isolation |
| Rhodamine B | Sigma Aldrich | 21955 | For M. tuberculosis staining |
| RPMI 1640 | Gibco | 11875093 | For macrophages culture |
| Sorvall ST 16/16R centrifuge | ThermoFisher Scientific | 75004240 | For PBMCs and monocytes isolation |
参考文献
- Global Tuberculosis Report 2023. , World Health Organization. Available at: https://www.who.int/tb/publications/global_report/en/ (2023).
- Ahmad, F., et al. Macrophage: A cell with many faces and functions in tuberculosis. Front Immunol. 13, 882130(2022).
- Bo, H., et al. Mycobacterium tuberculosis-macrophage interaction: Molecular updates. Front Cell Infect Microbiol. 13, 1187205(2023).
- Papp, A. C., et al. AmpliSeq transcriptome analysis of human alveolar and monocyte-derived macrophages over time in response to Mycobacterium tuberculosis infection. PLoS One. 13 (5), e0198069(2018).
- Zhai, W., Wu, F., Zhang, Y., Fu, Y., Liu, Z. The immune escape mechanisms of Mycobacterium tuberculosis. Int J Mol Sci. 20 (2), 340(2019).
- Van Crevel, R., Ottenhoff, T. H. M., Van der Meer, J. W. M. Innate immunity to Mycobacterium tuberculosis. Clin Microbiol Rev. 15 (2), 294-309 (2002).
- Mortaz, E., et al. Interaction of pattern recognition receptors with Mycobacterium tuberculosis. J Clin Immunol. 35 (1), 1-10 (2015).
- Stamm, C. E., Collins, A. C., Shiloh, M. U. Sensing of Mycobacterium tuberculosis and consequences to both host and bacillus. Immunol Rev. 264 (1), 204-219 (2015).
- Zihad, S. M. N. K., et al. Role of pattern recognition receptors in sensing Mycobacterium tuberculosis. Heliyon. 9 (10), e20636(2023).
- Barbero, A. M., et al. SLAMF1 signaling induces Mycobacterium tuberculosis uptake leading to endolysosomal maturation in human macrophages. J Leukoc Biol. 109 (8), 257-273 (2021).
- Pasquinelli, V., et al. Expression of signaling lymphocytic activation molecule-associated protein interrupts IFN-γ production in human tuberculosis. J Immunol. 172 (2), 1177-1185 (2004).
- Pasquinelli, V., et al. Phosphorylation of mitogen-activated protein kinases contributes to interferon-γ production in response to Mycobacterium tuberculosis. J Infect Dis. 207 (2), 340-350 (2012).
- Pasquinelli, V., et al. IFN-γ production during active tuberculosis is regulated by mechanisms that involve IL-17, SLAM, and CREB. J Infect Dis. 199 (5), 661-665 (2009).
- Pellegrini, J. M., et al. Neutrophil autophagy during human active tuberculosis is modulated by SLAMF1. Autophagy. 17 (2), 423-426 (2021).
- Song, T., Dong, C., Xiong, S. Signaling lymphocyte-activation molecule SLAMF1 augments mycobacteria BCG-induced inflammatory response and facilitates bacterial clearance. Int J Med Microbiol. 305 (6), 572-580 (2015).
- Eble, J. A. Titration ELISA as a method to determine the dissociation constant of receptor-ligand interaction. J Vis Exp. (132), e57032(2018).
- Patching, S. G. Surface plasmon resonance spectroscopy for characterization of membrane protein-ligand interactions and its potential for drug discovery. Biochim Biophys Acta Biomembr. 1838 (1), 43-55 (2014).
- Draczkowski, P., Matosiuk, D., Jozwiak, K. Isothermal titration calorimetry in membrane protein research. J Pharm Biomed Anal. 87, 296-304 (2014).
- Smith, D. S., Eremin, S. A. Fluorescence polarization immunoassays and related methods for simple, high-throughput screening of small molecules. Anal Bioanal Chem. 391 (5), 1499-1507 (2008).
- García-Nafría, J., Tate, C. G. Cryo-electron microscopy: Moving beyond X-ray crystal structures for drug receptors and drug development. Annu Rev Pharmacol Toxicol. 60, 51-71 (2020).
- Asami, J., Shimizu, T. Structural and functional understanding of the toll-like receptors. Protein Sci. 30 (4), 761-772 (2021).
- Phạm, T. T. T., Rainey, J. K. On-cell nuclear magnetic resonance spectroscopy to probe cell surface interactions. Biochem Cell Biol. 99 (6), 683-692 (2021).
- El Deeb, S., et al. Microscale thermophoresis as a powerful growing analytical technique for the investigation of biomolecular interaction and the determination of binding parameters. Methods Appl Fluoresc. 10 (4), 045001(2022).
- El Khamlichi, C., et al. Bioluminescence resonance energy transfer as a method to study protein-protein interactions: Application to G protein-coupled receptor biology. Molecules. 24 (3), 505(2019).
- Schneider, P., Willen, L., Smulski, C. R. Tools and techniques to study ligand-receptor interactions and receptor activation by TNF superfamily members. Methods Enzymol. 545, 103-125 (2014).
- Park, A. M. W., Schirmer, P. S. H. Atomic force microscopy: A multifaceted tool to study membrane proteins and their interactions with ligands. Biochim Biophys Acta Biomembr. 1838 (1), 74-89 (2014).
- Zalejski, J., Sun, J., Sharma, A. Unravelling the mystery inside cells by using single-molecule fluorescence imaging. J Imaging. 9 (9), 183(2023).
- Zheng, S., Zou, M., Shao, Y., Wu, H., Wang, X. Two-dimensional measurements of receptor-ligand interactions. Front Mol Biosci. 10, 1075587(2023).
- Zlotnikov, I. D., Kudryashova, E. V. Computer simulation of the receptor-ligand interactions of mannose receptor CD206 in comparison with the lectin concanavalin A model. Biochemistry (Moscow). 87 (1), 54-69 (2022).
- Fu, Y., Zhao, J., Chen, Z. Insights into the molecular mechanisms of protein-ligand interactions by molecular docking and molecular dynamics simulation: A case of oligopeptide binding protein. Comput Math Methods Med. 2018, 7604567(2018).
- Farina, B., et al. A novel approach for studying receptor-ligand interactions on living cells' surface by using NUS/T1ρ-NMR methodologies combined with computational techniques: The RGDechi15D-αvβ5 integrin complex. Comput Struct Biotechnol J. 19, 3303-3318 (2021).
- Driessen, N. N., et al. Role of phosphatidylinositol mannosides in the interaction between mycobacteria and DC-SIGN. Infect Immun. 77 (10), 4538-4547 (2009).
- Bansal, K., et al. Src homology 3-interacting domain of Rv1917c of Mycobacterium tuberculosis induces selective maturation of human dendritic cells by regulating PI3K-MAPK-NF-κB signaling and drives Th2 immune responses. J Biol Chem. 285 (47), 36511-36522 (2010).
- Xu, Y., et al. PPE57 induces activation of macrophages and drives Th1-type immune responses through TLR2. J Mol Med. 93 (6), 645-662 (2015).
- Olesen, R., et al. DC-SIGN (CD209), pentraxin 3 and vitamin D receptor gene variants associate with pulmonary tuberculosis risk in West Africans. Genes Immun. 8, 456-467 (2007).
- Berger, S. B., et al. SLAM is a microbial sensor that regulates bacterial phagosome functions in macrophages. Nat Immunol. 11 (10), 920-927 (2010).
- Degos, C., et al. Omp25-dependent engagement of SLAMF1 by Brucella abortus in dendritic cells limits acute inflammation and favours bacterial persistence in vivo. Cell Microbiol. 22 (4), e13156(2020).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved