Method Article
Um método de limpeza de tecidos para imagens neuronais de escalas mesoscópicas a microscópicas
Neste Artigo
Resumo
O protocolo fornece um método detalhado de imagem neuronal na fatia cerebral usando um método de limpeza tecidual, ScaleSF. O protocolo inclui preparação de tecidos cerebrais, esclarecimento de tecidos, manuseio de fatias limpas e microscopia de varredura a laser confocal de estruturas neuronais de níveis mesoscópicos a microscópicos.
Resumo
Um protocolo detalhado é fornecido aqui para visualizar estruturas neuronais de níveis mesoscópicos a microscópicos em tecidos cerebrais. Estruturas neuronais que vão desde circuitos neurais até estruturas neuronais subcelulares são visualizadas em fatias cerebrais de camundongos adequadamente limpas com ScaleSF. Este método de compensação é uma versão modificada do ScaleS e é um método de limpeza de tecido hidrofílico para fatias teciduais que alcança potente capacidade de limpeza, bem como um alto nível de preservação de sinais de fluorescência e integridade estrutural. Uma câmara de imagem digital 3D digitalizado (3D) personalizável é projetada para uma montagem confiável de tecidos cerebrais limpos. Cérebros de camundongos injetados com um vetor de vírus associado ao adeno carregando um gene de proteína fluorescente verde aprimorado foram corrigidos com 4% de paraformaldeído e cortados em fatias de espessura de 1 mm com um cortador de tecido vibrante. As fatias cerebrais foram limpas seguindo o protocolo de compensação, que incluem incubações sequenciais em três soluções: solução ScaleS0, soro fisco tampão de fosfato (–), e solução ScaleS4, totalizando 10,5-14,5 h. As fatias cerebrais limpas foram montadas na câmara de imagem e embutidas em gel de 1,5% de agarose dissolvido na solução ScaleS4D25(0). A aquisição de imagem 3D das fatias foi realizada utilizando um microscópio de varredura a laser confocal equipado com uma lente objetiva de multi-imersão de uma longa distância de trabalho. Começando com imagens neuronais mesoscópicas, conseguimos visualizar estruturas neuronais subcelulares finas, como espinhas dendríticas e boutons axonais, nas fatias cerebrais adequadamente limpas. Este protocolo facilitaria a compreensão das estruturas neuronais desde circuito até escalas de componentes subcelulares.
Introdução
Os métodos de limpeza tecidual melhoraram a imagem independente de profundidade de amostras biológicas e clínicas com microscopia leve, permitindo a extração de informações estruturais em tecidos intactos 1,2. Técnicas de compensação óptica também poderiam potencialmente acelerar e reduzir o custo para análise histológica. Atualmente, três grandes abordagens de compensação estão disponíveis: métodos à base de hidrofílico, hidrofóbico e hidrogel 1,2. As abordagens hidrofílicas superam na preservação de sinais de fluorescência e integridade tecidual e são menos tóxicas em comparação com as outras duas abordagens 3,4.
Um método de compensação hidrofílica, ScaleS, mantém uma posição distinta com sua preservação da integridade estrutural e molecular, bem como potente capacidade de compensação (espectro de preservação de desmatamento)5. Em um estudo anterior, desenvolvemos um protocolo de compensação rápida e isométrica, ScaleSF, para fatias de tecido (~1 mm de espessura) modificando o procedimento de compensação do ScaleS6. Este protocolo de compensação requer incubações sequenciais de fatias cerebrais em três soluções para 10,5-14,5 h. O método é apresentado com um espectro de alta preservação de compensação, que é compatível mesmo com a análise de microscopia eletrônica (EM) (Figura Suplementar 1), permitindo imagens tridimensionais (3D) de alta resolução em larga escala com reconstrução precisa do sinal6. Assim, o ScaleSF deve ser eficaz especialmente no cérebro, onde as células neuronais elaboram processos exuberantes de comprimento tremendo, e organizam estruturas subcelulares finas especializadas para transmitir e receber informações. Extrair informações estruturais com escalas de circuito para níveis subcelulares em células neuronais é bastante útil para uma melhor compreensão das funções cerebrais.
Aqui, fornecemos um protocolo detalhado para visualizar estruturas neuronais com escamas do mesoscópico/circuito ao nível microscópico/subcelular usando ScaleSF. O protocolo inclui preparação tecidual, esclarecimento de tecidos, manuseio de tecidos limpos e microscopia de escaneamento a laser confocal (CLSM) de tecidos limpos. Nosso protocolo se concentra em interrogar estruturas neuronais de circuito para escalas de componentes subcelulares. Para um procedimento detalhado para a preparação das soluções e injeção estereotaxica de vetores de vírus associados ao adeno (AAV) em cérebros de camundongos, consulte Miyawaki et al. 20167 e Okamoto et al. 20218, respectivamente.
Protocolo
Todos os experimentos foram aprovados pelos Comitês Institucionais de Cuidados e Uso de Animais da Universidade de Juntendo (Aprovação nº 2021245, 2021246) e realizados de acordo com as Diretrizes Fundamentais para a Conduta Adequada dos Experimentos Animais pelo Conselho Científico do Japão (2006). Aqui, camundongos C57BL/6J machos injetados com vetor AAV que transportam genes de proteína fluorescente verde aumentada (EGFP) e parvalbumina (PV)/myristoylation-EGFP-low-density lipoprotein receptor C-terminal bacteriano bacteriano camundongos transgênicos (BAC) camundongos transgênicos (pv-FGL mice)9 . Os camundongos PV-FGL foram mantidos em fundo C57BL/6J. Não foram encontradas diferenças baseadas em sexo em relação a este estudo.
1. Preparação tecidual
- Fixação porfusão
NOTA: Realizar as etapas 1.1.1 a 1.1.3 em um capô de fumaça para limitar a exposição ao paraformaldeído (PFA).- Anestesiar camundongos machos adultos (8 a 16 semanas de idade) por uma injeção intraperitoneal de overdose de pentobarbital de sódio (200 mg/kg). Confirme a adequação da anestesia pela ausência de abstinência do dedo e reflexos piscando os olhos.
- Abra a cavidade torácica e corte o apêndice atrial direito com uma tesoura cirúrgica. Perfume os camundongos com 20 mL de soro fisco de fosfato gelado (PBS) usando uma agulha de 23 G presa a uma seringa de 20 mL, seguida por perfusão de 20 mL de PFA gelada de 4% em tampão fosfato de 0,1 M (PB) usando outra seringa de 20 mL.
ATENÇÃO: O PFA é tóxico e teratogênico. Evite a inalação ou contato com a pele, olhos e membrana mucosa. - Remova os tecidos cerebrais do crânio com pinças. Transfira os tecidos cerebrais para um tubo de 15 mL contendo 4% de PFA em 0,1 M PB, proteja as amostras da luz e arrase suavemente durante a noite a 4 °C em um shaker a 50-100 rpm.
- NOTA: Os tecidos cerebrais colhidos podem ser armazenados por várias semanas em azida de sódio de 0,02% (NaN3) na PBS a 4 °C.
ATENÇÃO: A NaN3 é tóxica. Evite a inalação ou contato com a pele, olhos e membrana mucosa. Manuseie-o dentro de um capô de fumaça.
- Preparação de fatias cerebrais
- Prepare 4% de ágar em PBS adicionando 2 g de ágar a 50 mL de PBS. Micro-ondas a mistura até que o ágar esteja totalmente dissolvido. Deixe a solução esfriar para 40-45 °C.
NOTA: A propriedade viscosa fornecida pela agaropectina, um componente importante do ágar, melhora a facilidade de corte de fatias de tecido. - Adicione 10 mL da solução de ágar a uma placa de cultura de 6 poços. Submergir o tecido cerebral na solução de ágar usando fórceps. Deixe o ágar solidificar no gelo.
- Remova o tecido cerebral incorporado do poço e corte o ágar com uma lâmina de barbear. Fixar o bloco de ágar na parte inferior do banho de vibratome com supercola e despeje 0,1 M PB na bandeja de tampão.
- Limpe outra lâmina de barbear usando um papel de tecido sem fiapos embebido em etanol e conecte a lâmina ao suporte da lâmina do cortador de tecido vibratório.
- Defina a velocidade de secção para 0,14 mm/s com amplitude de 1,4 mm e a frequência para 75-77 Hz. Corte o tecido cerebral em fatias de 1 mm de espessura e colete as fatias em uma placa de cultura celular de 6 poços contendo PBS.
NOTA: As fatias cerebrais podem ser armazenadas por várias semanas em 0,02% naN3 em PBS a 4 °C.
- Prepare 4% de ágar em PBS adicionando 2 g de ágar a 50 mL de PBS. Micro-ondas a mistura até que o ágar esteja totalmente dissolvido. Deixe a solução esfriar para 40-45 °C.
2. Esclarecimento de tecidos
NOTA: As composições das soluções ScaleS utilizadas estão listadas na Tabela 1. As amostras devem ser protegidas da luz cobrindo com uma folha. As etapas de compensação são mostradas na Figura 1A.
- Adicione 8 mL de solução ScaleS0 a um poço de uma placa de cultura celular de 6 poços e adicione 8 mL de solução ScaleS4 a outro poço da placa e pré-aqueça a 37 °C em uma incubadora.
- Transfira as fatias cerebrais para a solução ScaleS0 pré-aquecida com uma espátula e incubar por 2h a 37 °C em uma incubadora de agitação a 90 rpm.
- Transfira as fatias cerebrais permeabilizadas em 8 mL de PBS(–) em uma placa de cultura celular de 6 poços com uma espátula e lave por 15 minutos mantendo-se em um agitador orbital a 40-60 rpm. Repita isso duas vezes.
- Transfira as fatias cerebrais na solução pré-aquecida de 8 mL de ScaleS4 com uma espátula e limpe-as incubando em uma incubadora a 90 rpm por 8-12 h a 37 °C. Uma fatia de cérebro limpa pode ser vista na Figura 1.
3. Montagem de fatias cerebrais
NOTA: Uma câmara de imagem personalizável é usada para uma montagem confiável de fatias cerebrais limpas (Figura 2)6. A câmara consiste no quadro da câmara e na tampa inferior. Os adaptadores de estágio do microscópio também são projetados para montar a câmara de imagem em estágios de microscópio diretamente (Figura 2A,B). Os adaptadores de estágio de quadro de câmara e microscópio podem ser impressos em 3D usando serviços internos ou terceirizados de impressão 3D. Os dados de design com auxílio ao computador 3D da câmara de imagem são fornecidos em Furuta et al. 20226.
- Para preparação da câmara, conecte o quadro da câmara a um deslizamento de tampa usando um adesivo sensível à pressão.
- Prepare 1,5% de agarose na solução ScaleS4D25(0) (ScaleS4 gel) adicionando 1,5 g de agarose a 100 mL da solução em uma garrafa. Misture bem a solução mexendo e micro-ondas a solução até que a agarose esteja totalmente dissolvida. Uma vez feito, deixe a solução esfriar a 37 °C.
- Monte a fatia do cérebro limpa na tampa inferior da câmara de imagem com uma espátula. Limpe a solução em excesso da fatia limpa usando um papel de tecido limpo sem fiapos.
- Adicione o gel ScaleS4 na fatia cerebral usando uma micropipette para encher a câmara de imagem. Coloque outra mancha de cobertura em cima com fórceps e coloque um pedaço de papel de tecido sem fiapos e um deslizamento de vidro na tampa nesta ordem.
- Transfira a câmara de imagem para uma geladeira a 4 °C. Coloque pesos metálicos no escorregador de vidro e deixe-os por 30 minutos.
- Remova os pesos metálicos, o deslizamento de vidro, o papel de tecido sem fiapos e o deslizamento da câmara de imagem e limpe o excesso de gel (Figura 2A,B).
- Coloque a câmara de imagem em uma placa de Petri de vidro de 60 mm e conecte a borda da câmara de imagem à placa com um adesivo sensível à pressão. Anexe a câmara em vários pontos à placa de Petri.
- Despeje a solução ScaleS4 no prato e agite suavemente por 1 h a 20-25 °C em um agitador orbital a 40-60 rpm. Substitua com uma solução fresca e remova bolhas de ar na superfície do gel raspando suavemente a superfície usando uma ponta de pipeta de 200 μL. Monte a câmara de imagem imersa em um estágio de microscópio (Figura 2C).
4. Imagem CLSM
- Adquira imagens utilizando um CLSM equipado com uma lente objetiva de multi-imersão de uma longa distância de trabalho (WD) (abertura numérica de 16x/0,60 [NA], WD = 2,5 mm).
NOTA: As lentes objetivas de NA altas podem fornecer uma resolução limitada à alta difração. - Ligue todos os equipamentos de imagem relevantes (estação de trabalho, microscópio, scanner, lasers e lâmpada de mercúrio) e lance um software de imagem CLSM.
- Coloque a coleira de correção da lente objetiva de multi-imersão para 1,47. A solução ScaleS4 tem um índice de refração (RI) de cerca de 1,47 5,7. Aberrações induzidas por incompatibilidade de RI podem perturbar a formação de imagem (Figura 3).
- Mergulhe a lente objetiva na solução e deixe-a aproximar-se lentamente da fatia. Remova quaisquer bolhas de ar presas na ponta da lente objetiva. Encontre regiões de interesse (ROIs) nos tecidos limpos usando epifluorescência.
- Defina parâmetros de aquisição de imagens testando as configurações apropriadas.
- Determine a profundidade do bit para aquisição de imagens. O tamanho dos dados da imagem aumenta com a profundidade do bit.
- Defina o comprimento de onda de detecção. Ajuste o portão apropriado para o detector de acordo com o espectro de emissões. Certifique-se de que o comprimento de onda de detecção não cobre nenhuma linha laser.
- Defina a resolução xy . Formatos maiores fornecem melhores resoluções de xy , mas leva mais tempo para coletar as imagens.
- Defina a velocidade de varredura. Uma velocidade de varredura mais lenta fornece uma alta relação sinal-ruído. No entanto, também aumenta o tempo de moradia dos pixels e o risco de fotobleaching. Escolha de acordo.
- Ajuste o tamanho do orifício. O tamanho do pinhole controla a espessura da seção óptica. Um tamanho menor do pinhole cria uma seção óptica mais fina e, portanto, melhor resolução z , mas reduz o sinal de fluorescência. Tornar o tamanho do orifício maior fornece uma seção óptica mais espessa com sinal de fluorescência mais forte.
- Defina a potência do laser, o ganho do detector/amplificador e o deslocamento. Aumente gradualmente a potência do laser e o ganho do detector/amplificador até que uma imagem adequada seja obtida. Uma alta potência laser traz o risco de fotobleaching. Ajuste o deslocamento (contraste) adequadamente para obter uma alta relação sinal-ruído.
- Determine a área de plantio necessária com base no tamanho do ROI. Certifique-se de que todo o comprimento e largura do ROI seja capturado.
- Navegue pelos tecidos limpos em todos os planos e defina os pontos de partida e fim da pilha. Defina o tamanho da etapa z de acordo com a resolução z desejada.
- Colete imagens quando satisfeito com as configurações de aquisição de imagens e grave imagens capturadas. Processe as imagens usando um software de análise de imagem.
Resultados
A limpeza óptica de uma fatia cerebral do rato de 1 mm de espessura foi alcançada usando este protocolo. A Figura 1B representa imagens de transmissão de uma fatia cerebral de camundongos antes e depois do tratamento de compensação. O método de limpeza tecidual tornou transparente uma fatia cerebral de camundongo de 1 mm de espessura. Uma ligeira expansão nos tamanhos finais das fatias cerebrais foi encontrada após a incubação na solução de compensação por 12h (expansão linear: 102,5% ± 1,3%). A preservação da fluorescência e integridade estrutural dos tecidos foi avaliada com expressão EGFP direcionada na membrana plasmática em camundongos PV-FGL (Figura 1C). Nestes camundongos, o EGFP alvo de membrana somatodendritica é expresso em neurônios fotovoltaicospositivos 9. A expressão EGFP direcionada à membrana plasmática na região somatodendritica foi mantida após o tratamento (Figura 1C). Além disso, o estudo anterior da EM mostra integridade estrutural bem preservada nos tecidos cerebrais limpos com ScaleSF (Figura Suplementar 1)6.
As aberrações induzidas por incompatibilidade de RI causaram uma perda notável de brilho e resolução da imagem (Figura 3). Uma fatia cerebral de 1 mm de espessura do mouse PV-FGL foi limpa e imagem sob um CLSM equipado com uma lente objetiva de multi-imersão de um longo WD. Ajuste da coleira de correção da lente objetiva para a posição da água (RI 1.33) dificultou a visualização clara de neurônios positivos EGFP localizados nas profundezas de 400 μm e 800 μm devido ao baixo brilho e baixo contraste. (Figura 3A,C). Esses neurônios foram claramente visualizados com o mesmo CLSM, quando a coleira de correção foi ajustada para corresponder à solução ScaleS4 (RI 1.47; Figura 3B,D). A correspondência de RI entre um fluido de imersão e uma lente objetiva é fundamental para imagens 3D precisas em tecidos eliminados opticamente.
Por fim, foram utilizados neurônios neocorticais do camundongo para demonstrar a viabilidade do protocolo. Um cérebro de camundongo injetado com vetor AAV2/1-SynTetOff-EGFP10 no córtex somatosensorial primário (S1) foi fixado com 4% de PFA em 0,1 M PB. Fatias coronais de 1 mm de espessura foram preparadas do cérebro com um cortador de tecido vibratório. Após a limpeza e montagem na câmara de imagem, foi realizada a imagem neuronal direcionada aos neurônios neocorticais (Figura 4). Uma reconstrução 3D de neurônios rotulados por EGFP na fatia cerebral de 1 mm de espessura está representada na Figura 4A. Uma imagem de ampliação mais elevada mostra arboris dendríticas individuais decoradas com espinhas dendríticas (Figura 4B). Mostramos ainda arborizações terminais de axônio e boutons axonal no córtex contralateral (Figura 4C).
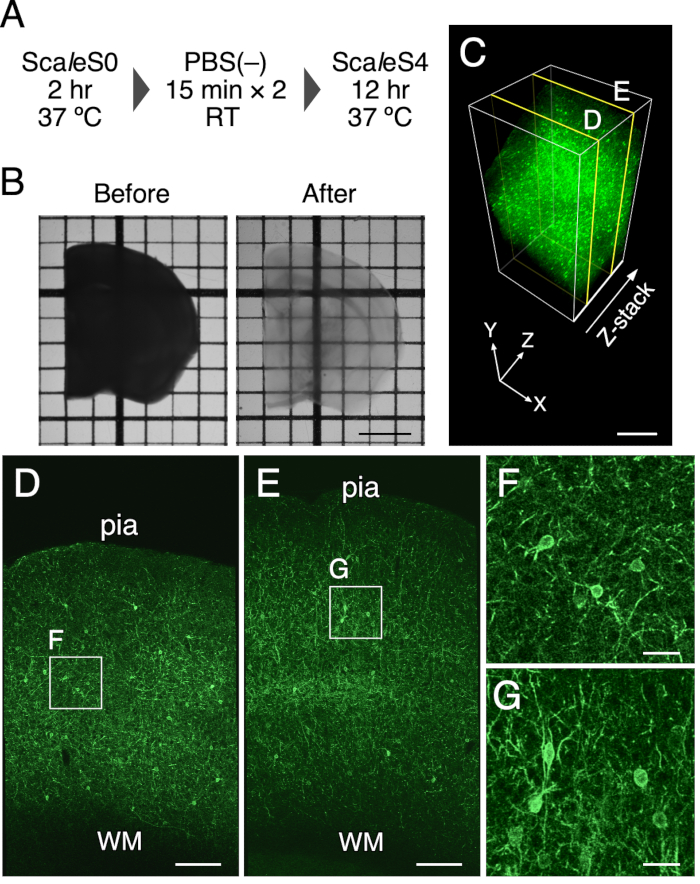
Figura 1: Limpeza óptica de fatias cerebrais do rato de 1 mm de espessura. (A) O cronograma para limpeza de tecido ScaleSF. (B) Imagens de transmissão de um cérebro de 1 mm de espessura corta antes (esquerda) e depois (direita). (C) Uma renderização de volume 3D do córtex cerebral de um mouse PV-FGL limpo utilizando o método de limpeza tecidual. (D,E) xy imagens em (C) nas profundezas de 250 μm (D) e 750 μm (E). (F,G) Visão ampliada dos retângulos delineados em (D) e (E). As imagens que aparecem em (C-G) são desconvoludas antes do processo de renderização. Abreviaturas: pia = pia mater, WM = matéria branca. Barra de escala: 2 mm in (B), 500 μm in (C), 200 μm in (D,E) e 40 μm in (F,G). Este número foi modificado a partir de Furata et al. 20226. Clique aqui para ver uma versão maior desta figura.
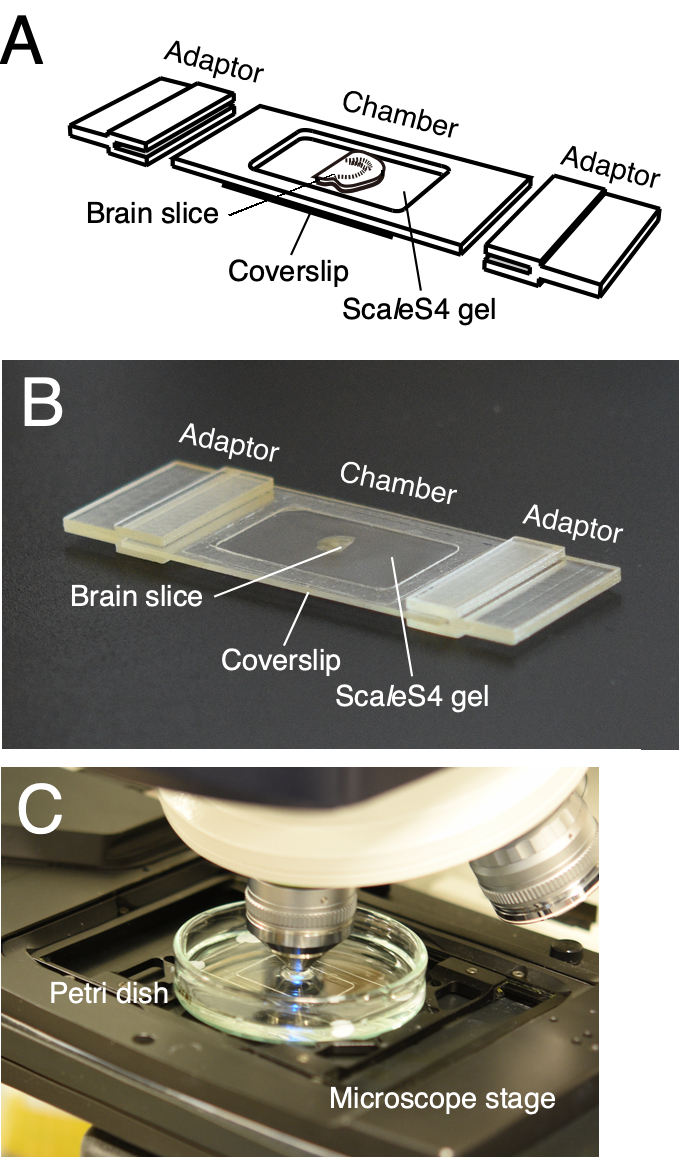
Figura 2: Uma câmara de imagem impressa em 3D personalizável para visualização de fatias de tecido. (A,B) Um desenho de esquema (A) e imagem (B) de uma câmara de imagem impressa em 3D personalizável. A câmara de imagem é composta por um quadro de câmara, um deslizamento de tampa inferior e adaptadores de estágio de microscópio. As fatias de tecido limpas são colocadas na tampa inferior e embutidas em gel ScaleS4. O quadro da câmara, os adaptadores de fase de tampa inferior e microscópio são personalizáveis de acordo com o tamanho e espessura das fatias de tecido. (C) Uma configuração de imagem com a câmara de imagem. A câmara de imagem está imersa na solução ScaleS4 em uma placa de Petri e montada em um palco de um CLSM vertical. Este número foi modificado a partir de Furata et al. 20226. Clique aqui para ver uma versão maior desta figura.

Figura 3: Imagens profundas comprometidas causadas por uma incompatibilidade de RI entre uma lente objetiva e a solução ScaleS4. (A-D) xy imagens do córtex cerebral de um mouse PV-FGL a profundidades de 400 μm (A,B) e 800 μm (C,D). A correção da coleira de uma lente objetiva multi-imersão é ajustada para 1,33 polegadas (A,C) e 1,47 em (B,D). As imagens são adquiridas com os mesmos parâmetros, exceto para RIs da lente objetiva. Barra de escala: 50 μm. Clique aqui para ver uma versão maior desta figura.

Figura 4: Imagem neuronal em uma fatia cerebral de camundongo de 1 mm de espessura limpa com ScaleSF. (A) Renderização de volume 3D de neurônios neocortical de camundongos rotulados por EGFP no S1. Os neurônios neocorticais são rotulados com o vetor SynTetOff-EGFP AAV2/1. (B) Arbóreos dendráticos de neurônios neocorticais rotulados pela EGFP. Uma imagem de projeção de intensidade máxima (MIP) de uma profundidade de 39 μm a 48 μm é representada. Pontas de flecha indicam espinhas dendríticas. (C) Terminais de axônio com etiqueta EGFP no córtex contralateral. Uma imagem MIP de uma profundidade de 481,5 μm a 513 μm é representada. Pontas de flecha indicam ataques axonais. As imagens que aparecem em (B) e (C) são desconvoludas. Barras de escala: 300 μm in (A) e 10 μm in (C). A barra em (C) também se aplica a (B). Clique aqui para ver uma versão maior desta figura.
Figura suplementar 1: Ultraestrutura em fatias cerebrais limpas com ScaleSF, CUBIC e PACT. (A-C) Transmissão em imagens do córtex cerebral do rato limpo com ScaleSF (A), CUBIC (B) e PACT (C). Cérebros de camundongos são fixados com 4% de PFA contendo 1% de glutaraldeído. Seções ultrathinas são preparadas a partir de fatias cerebrais limpas. As estruturas de membrana são severamente danificadas em fatias cerebrais limpas com CUBIC (B) e PACT (C). Pontas de flecha indicam membranas postinas. Barra de escala: 500 nm. Este número foi modificado a partir de Furata et al. 20226. Clique aqui para baixar este Arquivo.
| Recipies para soluções ScaleS | |
| Solução ScaleS0 | |
| Reagente | Concentração final |
| D-sorbitol | 20% (w/v) |
| Glicerol | 5% (w/v) |
| Metil-β-ciclodextrina | 1 mM |
| γ ciclodextrina | 1 mM |
| Sulfoxida de dimetila | 3% (v/v) |
| 10x PBS(–) | 1x |
| Solução ScaleS4 | |
| Reagente | Concentração final |
| Ureia | 4 M |
| D-sorbitol | 40% (w/v) |
| Glicerol | 10% (w/v) |
| Tritão X-100 | 0,2% (w/v) |
| Sulfoxida de dimetila | 25% (v/v) |
| Solução ScaleS4D25(0) | |
| Reagente | Concentração final |
| Ureia | 4 M |
| D-sorbitol | 40% (w/v) |
| Glicerol | 10% (w/v) |
| Sulfoxida de dimetila | 25% (v/v) |
Tabela 1: Composição das três soluções ScaleS. Estão listadas as composições das soluções ScaleS0, ScaleS4 e ScaleS4D25(0). Para um procedimento detalhado para a elaboração dessas soluções, consulte Miyawaki et al. 20167.
Discussão
Passos críticos dentro do protocolo
Existem alguns passos críticos no protocolo que devem ser conduzidos com a máxima cautela para obter resultados significativos. A fixação uniforme das amostras é imprescindível para imagens 3D dentro de tecidos de grande escala. A lente objetiva, a amostra e o fluido de imersão devem ter ri correspondente. A incompatibilidade de RI entre eles levará a imagens altamente perturbadas de células expressas por EGFP dentro das fatias cerebrais limpas (Figura 3). O ajuste da coleira de correção da lente objetiva para o fluido de imersão minimiza aberrações esféricas induzidas por profundidade para maximizar o sinal, contraste e resolução espacial em imagens 3D. Os RIs das soluções preparadas podem ser medidos usando um refratômetro.
Solução de problemas da técnica
O armazenamento mais longo das soluções pode afetar a capacidade de compensação e sua capacidade de preservação de sinais de fluorescência e integridade estrutural. Devem ser utilizadas soluções recém-preparadas. Estas soluções podem ser armazenadas até 1 mês a 4 °C. A isometricidade é fundamental para imagens neuronais eficazes e eficientes com reconstrução precisa do sinal. Embora tenha sido observada uma ligeira expansão nos tamanhos amostrais após a incubação por 12 h (Figura 1B), a expansão pode ser controlada diminuindo o período de incubação entre 8-12 h. Para imagens de profundidade 3D mais precisas, a coleira de correção pode precisar ser ajustada em um determinado plano devido à aberração esférica induzida por profundidade.
Modificações da técnica
No presente estudo, utilizamos tecidos cerebrais de camundongos para demonstrar a viabilidade do protocolo. No entanto, o protocolo descrito aqui também pode ser usado para animais de cérebro grande, como primatas. De fato, este protocolo tem sido usado em tecidos cerebrais de sagui comum (Callithrix jacchus), e conseguiu visualizar simultaneamente circuito neural e estruturas subcelulares de seus circuitos corticostriais6. Os adaptadores do estágio do microscópio são projetados para montar a câmara de imagem nos estágios do microscópio diretamente6 (Figura 2A,B). As fatias de tecido limpo são observáveis usando um microscópio invertido através da tampa inferior da câmara de imagem. Após a restauração de tecidos cerebrais limpos com PBS(-) (deScaling)5,11, podemos preparar seções teciduais de 20-μm a 50-μm de espessura de tecidos cerebrais que foram limpos com ScaleSF (re-secção)6. Estruturas subcelulares capturadas dentro de tecidos limpos podem ser imagens novamente em re-seções com uma lente objetiva na alta de uma pequena WD. A limpeza de fatias cerebrais perfumadas com fixações contendo glutaraldeído foi alcançada usando este protocolo, proporcionando preservação superior da ultraestrutura6. O SCAleSF alcança um alto nível de preservação da ultraestrutura que permite a análise de EM em tecidos adequadamente limpos6 (Figura Suplementar 1). A compatibilidade EM deste método é particularmente útil para estruturas de imagem com as escalas do nível macroscópico ao nanoscópico.
Limitações da técnica
O protocolo descrito aqui nos permite visualizar estruturas neuronais do circuito às escalas subcelulares em fatias cerebrais de 1 mm de espessura. No entanto, três limitações permanecem no protocolo. A primeira é a capacidade de compensação do protocolo de compensação. ScaleSF é um protocolo de compensação para fatias cerebrais, não para todo o cérebro. Embora as fatias cerebrais de 1 mm de espessura possam fornecer um bom conhecimento das arbores axonais dendríticas e locais12, a informação sobre projeções axonas que abrangem todo o cérebro é fragmentária e incompleta nas fatias 13,14,15,16. A segunda é a resolução de imagens. Usando o protocolo aqui descrito, conseguimos visualizar estruturas neuronais subcelulares, como espinhos dendráticos e boutons axonais, em uma fatia cerebral opticamente limpa (Figura 4). No entanto, a resolução da lente objetiva utilizada neste estudo, resolução xiy de 400-750 nm, não é suficiente para resolver estruturas mais finas das células neuronais. Dado que as lentes objetivas na altas são tipicamente projetadas para imersão em óleo (RI 1.52), a incompatibilidade de RI com as soluções (RI 1.47) pode impedir imagens de alta resolução com essas lentes objetivas. A terceira é a rotulagem de proteína fluorescente de células neuronais. O método de rotulagem limita as aplicações amplas de nossa técnica de imagem. Técnicas histoquímicas e/ou imunohistoquímicas que rotulam tecidos de grande escala, mantendo a integridade tecidual, avançariam significativamente o protocolo aqui fornecido.
Significância em relação aos métodos existentes e aplicações futuras da técnica
No presente estudo, descrevemos um protocolo de detalhes para imagens neuronais de estruturas mesoscópicas a microscópicas utilizando a limpeza tecidual ScaleSF. O protocolo aqui descrito possibilita visualizar estruturas neuronais de circuitos a níveis subcelulares em uma quantidade razoável de tempo sem equipamento especializado, facilitando a compreensão das estruturas neuronais do circuito às escalas de componentes. Os neurônios elaboram processos exuberantes de comprimento tremendo e organizam estruturas finas especializadas para transmitir e receber informações. Assim, a imagem neuronal requer um método de limpeza tecidual que exerça potente capacidade de limpeza, bem como um alto nível de preservação do tecido para visualização simultânea de estruturas de grande e pequena escala. No entanto, os métodos de limpeza tecidual apresentados com altas capacidades de limpeza removem agressivamente lipídios e pigmentos para esclarecimento extensivo de tecido 3,4, comprometendo a integridade tecidual 5,6,17 (Figura Suplementar 1). Isso contrasta fortemente com o protocolo de compensação aqui utilizado que alcança um alto nível de preservação da estrutura6 (Figura Suplementar 1). Assim, a limpeza do tecido ScaleSF permite uma imagem neuronal eficaz e eficiente que requer imagens 3D de alta resolução em larga escala com reconstrução precisa do sinal.
Divulgações
Os autores não têm nada a revelar.
Agradecimentos
Os autores agradecem a Yoko Ishida (Universidade de Juntendo) pela produção de vetores AAV e kisara Hoshino (Universidade de Juntendo) pela assistência técnica. Este estudo foi apoiado por JSPS KAKENHI (JP20K07231 a K.Y.; JP21H03529 a T.F.; JP20K07743 a M.K.; JP21H02592 a H.H.) e Pesquisa Científica sobre Área Inovadora "Ressonância Bio" (JP18H04743 a H.H.). Este estudo também foi apoiado pela Agência japonesa de Pesquisa e Desenvolvimento Médico (AMED) (JP21dm0207112 a T.F. e H.H.), Moonshot P&D da Agência de Ciência e Tecnologia do Japão (JST) (JPMJMS2024 a H.H.), Fusion Oriented Research for disruptive Science and Technology (FOREST) da JST (JPMJFR204D a H.H.), Grants-in-Aid do Instituto de Pesquisa para Doenças da Velhice na Escola de Medicina da Universidade de Juntendo (X2016 a K.Y.; X2001 para H.H.), e o Projeto de Branding de Escolas Particulares.
Materiais
| Name | Company | Catalog Number | Comments |
| 16x multi-immersion objective lens | Leica Microsystems | HC FLUOTAR 16x/0.60 IMM CORR VISIR | |
| Agar | Nacalai Tesque | 01028-85 | |
| Agarose | TaKaRa Bio | L03 | |
| Dimethyl sulfoxide | Nacalai Tesque | 13407-45 | |
| D-Sorbitol | Nacalai Tesque | 06286-55 | |
| γ-cyclodextrin | Wako Pure Chemical Industries | 037-10643 | |
| Glycerol | Sigma-Aldrich | G9012 | |
| Huygens Essential | Scientific Volume Imaging | ver. 18.10.0p8/21.10.1p0 64b | |
| Imaris | Bitplane | ver. 9.0.0 | |
| Leica Application Suite X | Leica Microsystems | LAS X, ver. 3.5.5.19976 | |
| Methyl-β-cyclodextrin | Tokyo Chemical Industry | M1356 | |
| Paraformaldehyde | Merck Millipore | 1.04005.1000 | |
| Phosphate Buffered Saline (10x; pH 7.4) | Nacalai Tesque | 27575-31 | 10x PBS(–) |
| Sodium azide | Nacalai Tesque | 31233-55 | |
| Sodium pentobarbital | Kyoritsu Seiyaku | N/A | |
| TCS SP8 | Leica Microsystems | N/A | |
| Triton X-100 | Nacalai Tesque | 35501-15 | |
| Urea | Nacalai Tesque | 35940-65 | |
| Vibrating tissue slicer | Dosaka EM | PRO7N |
Referências
- Susaki, E. A., Ueda, H. R. Whole-body and whole-organ clearing and imaging techniques with single-cell resolution: Toward organism-level systems biology in mammals. Cell Chemical Biology. 23 (1), 137-157 (2016).
- Tainaka, K., Kuno, A., Kubota, S. I., Murakami, T., Ueda, H. R. Chemical principles in tissue clearing and staining protocols for whole-body cell profiling. Annual Reviews of Cell and Developmental Biology. 32, 713-741 (2016).
- Ueda, H. R., et al. Whole-brain profiling of cells and circuits in mammals by tissue clearing and light-sheet microscopy. Neuron. 106 (3), 369-387 (2020).
- Ueda, H. R., et al. Tissue clearing and its applications in neuroscience. Nature Reviews. Neuroscience. 21 (2), 61-79 (2020).
- Hama, H., et al. ScaleS: an optical clearing palette for biological imaging. Nature Neuroscience. 18 (10), 1518-1529 (2015).
- Furuta, T., et al. Multi-scale light microscopy/electron microscopy neuronal imaging from brain to synapse with a tissue clearing method, ScaleSF. iScience. 25 (1), 103601(2022).
- Miyawaki, A., et al. Deep imaging of cleared brain by confocal laser-scanning microscopy. Protocol Exchange. , (2016).
- Okamoto, S., et al. Exclusive labeling of direct and indirect pathway neurons in the mouse neostriatum by an adeno-associated virus vector with Cre/lox system. STAR Protocols. 2 (1), 100230(2021).
- Kameda, H., et al. Parvalbumin-producing cortical interneurons receive inhibitory inputs on proximal portions and cortical excitatory inputs on distal dendrites. The European Journal of Neuroscience. 35 (6), 838-854 (2012).
- Sohn, J., et al. A single vector platform for high-level gene transduction of central neurons: Adeno-associated virus vector equipped with the Tet-off system. PLoS One. 12 (1), 0169611(2017).
- Hama, H., et al. Scale: a chemical approach for fluorescence imaging and reconstruction of transparent mouse brain. Nature Neuroscience. 14 (11), 1481-1488 (2011).
- Stepanyants, A., Martinez, L. M., Ferecsko, A. S., Kisvarday, Z. F. The fractions of short- and long-range connections in the visual cortex. Proceedings of the National Academy of Sciences of the United States of America. 106 (9), 3555-3560 (2009).
- Kuramoto, E., et al. Two types of thalamocortical projections from the motor thalamic nuclei of the rat: a single neuron-tracing study using viral vectors. Cerebral Cortex. 19 (9), New York, N.Y. 2065-2077 (2009).
- Matsuda, W., et al. Single nigrostriatal dopaminergic neurons form widely spread and highly dense axonal arborizations in the neostriatum. The Journal of Neuroscience: The Official Journal of the Society for Neuroscience. 29 (2), 444-453 (2009).
- Lin, R., et al. Cell-type-specific and projection-specific brain-wide reconstruction of single neurons. Nature Methods. 15 (12), 1033-1036 (2018).
- Winnubst, J., et al. Reconstruction of 1,000 projection neurons reveals new cell types and organization of long-range connectivity in the mouse brain. Cell. 179 (1), 268-281 (2019).
- Neckel, P. H., Mattheus, U., Hirt, B., Just, L., Mack, A. F. Large-scale tissue clearing (PACT): Technical evaluation and new perspectives in immunofluorescence, histology, and ultrastructure. Scientific Reports. 6, 34331(2016).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoExplore Mais Artigos
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados