Method Article
Un metodo di compensazione dei tessuti per l'imaging neuronale dalle scale mesoscopiche a quelle microscopiche
In questo articolo
Riepilogo
Il protocollo fornisce un metodo dettagliato di imaging neuronale nella fetta di cervello utilizzando un metodo di compensazione dei tessuti, ScaleSF. Il protocollo include la preparazione del tessuto cerebrale, la chiarificazione dei tessuti, la manipolazione di fette ripulite e l'imaging al microscopio a scansione laser confocale di strutture neuronali da mesoscopiche a livelli microscopici.
Abstract
Qui viene fornito un protocollo dettagliato per visualizzare le strutture neuronali dai livelli mesoscopici a quelli microscopici nei tessuti cerebrali. Le strutture neuronali che vanno dai circuiti neurali alle strutture neuronali subcellulari sono visualizzate in fette di cervello di topo otticamente cancellate con ScaleSF. Questo metodo di compensazione è una versione modificata di ScaleS ed è un metodo di compensazione del tessuto idrofilo per fette di tessuto che raggiunge una potente capacità di compensazione e un alto livello di conservazione dei segnali di fluorescenza e integrità strutturale. Una camera di imaging tridimensionale (3D) personalizzabile è progettata per il montaggio affidabile di tessuti cerebrali eliminati. I cervelli di topo iniettati con un vettore virale adeno-associato che trasporta un gene proteico fluorescente verde potenziato sono stati fissati con paraformaldeide al 4% e tagliati in fette di spessore di 1 mm con un'affettatrice di tessuto vibrante. Le fette di cervello sono state eliminate seguendo il protocollo di compensazione, che include incubazioni sequenziali in tre soluzioni, vale a dire, soluzione ScaleS0, soluzione salina tampone fosfato (–) e soluzione ScaleS4, per un totale di 10,5-14,5 ore. Le fette di cervello eliminate sono state montate sulla camera di imaging e incorporate in gel di agarosio all'1,5% disciolto nella soluzione ScaleS4D25(0). L'acquisizione dell'immagine 3D delle fette è stata effettuata utilizzando un microscopio a scansione laser confocale dotato di una lente obiettivo multi-immersione di una lunga distanza di lavoro. Iniziando con l'imaging neuronale mesoscopico, siamo riusciti a visualizzare le strutture neuronali subcellulari fini, come le spine dendritiche e i bouton assonali, nelle fette di cervello otticamente cancellate. Questo protocollo faciliterebbe la comprensione delle strutture neuronali dalle scale dei circuiti ai componenti subcellulari.
Introduzione
I metodi di compensazione dei tessuti hanno migliorato l'imaging indipendente dalla profondità di campioni biologici e clinici con microscopia ottica, consentendo l'estrazione di informazioni strutturali su tessuti intatti 1,2. Le tecniche di compensazione ottica potrebbero anche potenzialmente accelerare e ridurre i costi per l'analisi istologica. Attualmente, sono disponibili tre principali approcci di compensazione: metodi idrofili, idrofobici e a base di idrogel 1,2. Gli approcci idrofili superano nel preservare i segnali di fluorescenza e l'integrità dei tessuti e sono meno tossici rispetto agli altri due approcci 3,4.
Un metodo di compensazione idrofila, ScaleS, occupa una posizione distintiva per la sua conservazione dell'integrità strutturale e molecolare e per la potente capacità di compensazione (spettro di compensazione-conservazione)5. In uno studio precedente, abbiamo sviluppato un protocollo di compensazione rapida e isometrica, ScaleSF, per fette di tessuto (~ 1 mm di spessore) modificando la procedura di compensazione di ScaleS6. Questo protocollo di compensazione richiede incubazioni sequenziali di fette di cervello in tre soluzioni per 10,5-14,5 ore. Il metodo è caratterizzato da un elevato spettro di conservazione della compensazione, che è compatibile anche con l'analisi al microscopio elettronico (EM) (Figura supplementare 1), consentendo l'imaging tridimensionale (3D) ad alta risoluzione su più scale con un'accurata ricostruzione del segnale6. Pertanto, ScaleSF dovrebbe essere efficace soprattutto nel cervello, dove le cellule neuronali elaborano processi esuberanti di lunghezza enorme e organizzano strutture subcellulari fini specializzate per trasmettere e ricevere informazioni. Estrarre informazioni strutturali con scale dal circuito ai livelli subcellulari sulle cellule neuronali è molto utile per una migliore comprensione delle funzioni cerebrali.
Qui, forniamo un protocollo dettagliato per visualizzare le strutture neuronali con scale dal mesoscopico / circuito al livello microscopico / subcellulare utilizzando ScaleSF. Il protocollo include la preparazione dei tessuti, la chiarificazione dei tessuti, la manipolazione dei tessuti eliminati e l'imaging al microscopio a scansione laser confocale (CLSM) dei tessuti cancellati. Il nostro protocollo si concentra sull'interrogazione delle strutture neuronali dalle scale dei circuiti ai componenti subcellulari. Per una procedura dettagliata per la preparazione delle soluzioni e l'iniezione stereotassica di vettori di virus adeno-associati (AAV) nel cervello dei topi, fare riferimento rispettivamente a Miyawaki et al. 20167 e Okamoto et al. 20218.
Protocollo
Tutti gli esperimenti sono stati approvati dai comitati istituzionali per la cura e l'uso degli animali dell'Università juntendo (approvazione n. 2021245, 2021246) ed eseguiti in conformità con le linee guida fondamentali per una corretta conduzione degli esperimenti sugli animali dal Consiglio scientifico del Giappone (2006). Qui sono stati utilizzati topi maschi C57BL / 6J iniettati con vettore AAV che trasporta il gene della proteina fluorescente verde potenziata (EGFP) e parvalbumina (PV) / miristoilazione-EGFP-recettore delle lipoproteine a bassa densità C-terminale cromosoma artificiale batterico (BAC) topi transgenici (topi PV-FGL)9 . I topi PV-FGL sono stati mantenuti in background C57BL / 6J. Non sono state riscontrate differenze basate sul sesso per quanto riguarda questo studio.
1. Preparazione dei tessuti
- Fissazione della perfusione
NOTA: eseguire i passaggi da 1.1.1 a 1.1.3 in una cappa aspirante per limitare l'esposizione alla paraformaldeide (PFA).- Anestetizzare topi maschi adulti (8-16 settimane di età) mediante iniezione intraperitoneale di sovradosaggio di pentobarbital di sodio (200 mg/kg). Confermare l'adeguatezza dell'anestesia dall'assenza di astinenza da pizzicamento delle dita dei piedi e riflessi lampeggianti.
- Aprire la cavità toracica e tagliare l'appendice atriale destra con le forbici chirurgiche. Perfondere i topi con 20 mL di soluzione salina tampone fosfato ghiacciato (PBS) utilizzando un ago da 23 G attaccato a una siringa da 20 mL, seguito da una perfusione di 20 mL di PFA al 4% ghiacciato in tampone fosfato da 0,1 M (PB) utilizzando un'altra siringa da 20 mL.
ATTENZIONE: il PFA è tossico e teratogeno. Evitare l'inalazione o il contatto con la pelle, gli occhi e le mucose. - Rimuovere i tessuti cerebrali dal cranio con una pinzetta. Trasferire i tessuti cerebrali in un tubo da 15 ml contenente il 4% di PFA in 0,1 M PB, proteggere i campioni dalla luce e oscillare delicatamente durante la notte a 4 °C su uno shaker a 50-100 giri / min.
- NOTA: I tessuti cerebrali raccolti possono essere conservati per diverse settimane in azide di sodio allo 0,02% (NaN3) in PBS a 4 °C.
ATTENZIONE: NaN3 è tossico. Evitare l'inalazione o il contatto con la pelle, gli occhi e le mucose. Maneggialo all'interno di una cappa aspirante.
- Preparazione della fetta di cervello
- Preparare il 4% di agar in PBS aggiungendo 2 g di agar a 50 ml di PBS. Microonde la miscela fino a quando l'agar è completamente sciolto. Lasciare raffreddare la soluzione a 40-45 °C.
NOTA: La proprietà viscosa fornita dall'agaropectina, un componente importante dell'agar, migliora la facilità di taglio delle fette di tessuto. - Aggiungere 10 ml di soluzione di agar a una piastra di coltura a 6 pozzetti. Immergere il tessuto cerebrale nella soluzione di agar usando una pinza. Lascia che l'agar si solidifichi sul ghiaccio.
- Rimuovere il tessuto cerebrale incorporato dal pozzo e tagliare l'agar con una lama di rasoio. Fissare il blocco di agar sul fondo del bagno vibratome con supercolla e versare 0,1 M PB nel vassoio tampone.
- Pulire un'altra lama di rasoio utilizzando una carta velina priva di lanugine imbevuta di etanolo e fissare la lama al supporto della lama dell'affettatrice vibrante.
- Impostare la velocità di sezionamento a 0,14 mm/s con un'ampiezza di 1,4 mm e la frequenza a 75-77 Hz. Tagliare il tessuto cerebrale in fette spesse 1 mm e raccogliere le fette in una piastra di coltura cellulare a 6 pozzetti contenente PBS.
NOTA: Le fette di cervello possono essere conservate per diverse settimane nello 0,02% di NaN3 in PBS a 4 °C.
- Preparare il 4% di agar in PBS aggiungendo 2 g di agar a 50 ml di PBS. Microonde la miscela fino a quando l'agar è completamente sciolto. Lasciare raffreddare la soluzione a 40-45 °C.
2. Chiarificazione dei tessuti
NOTA: Le composizioni delle soluzioni ScaleS utilizzate sono elencate nella Tabella 1. I campioni devono essere protetti dalla luce coprendo con un foglio. I passaggi di compensazione sono illustrati nella Figura 1A.
- Aggiungere 8 mL di soluzione ScaleS0 in un pozzetto di una piastra di coltura cellulare a 6 pozzetti e aggiungere 8 mL di soluzione ScaleS4 ad un altro pozzetto della piastra e preriscaldare a 37 °C in un incubatore.
- Trasferire le fette di cervello nella soluzione ScaleS0 preriscaldata con una spatola e incubare per 2 ore a 37 °C in un incubatore vibrante a 90 giri/min.
- Trasferire le fette di cervello permeabilizzate in 8 ml di PBS (–) in una piastra di coltura cellulare a 6 pozzetti con una spatola e lavare per 15 minuti mantenendo uno shaker orbitale a 40-60 giri / min. Ripeti l'operazione due volte.
- Trasferire le fette di cervello negli 8 mL preriscaldati della soluzione di ScaleS4 con una spatola e cancellarle incubandole in un incubatore vibrante a 90 giri/min per 8-12 ore a 37 °C. Una fetta di cervello cancellata può essere vista nella Figura 1.
3. Montaggio della fetta di cervello
NOTA: una camera di imaging personalizzabile viene utilizzata per il montaggio affidabile di fette di cervello cancellate (Figura 2)6. La camera è costituita dal telaio della camera e dal coperchio inferiore. Gli adattatori per stadio del microscopio sono inoltre progettati per montare direttamente la camera di imaging sugli stadi del microscopio (Figura 2A,B). Gli adattatori per telaio della camera e stadio del microscopio possono essere stampati in 3D utilizzando servizi di stampa 3D interni o in outsourcing. I dati CAD (Computer-Aided Design) 3D della camera di imaging sono forniti in Furuta et al. 20226.
- Per la preparazione della camera, collegare il telaio della camera a un coperchio utilizzando un adesivo sensibile alla pressione.
- Preparare l'1,5% di agarosio in soluzione di ScaleS4D25(0) (gel ScaleS4) aggiungendo 1,5 g di agarosio a 100 ml di soluzione in flacone. Mescolare bene la soluzione mescolando e microonde la soluzione fino a quando l'agarosio è completamente sciolto. Una volta fatto, lasciare raffreddare la soluzione a 37 °C.
- Montare la fetta di cervello cancellata sul coperchio inferiore della camera di imaging con una spatola. Pulire la soluzione in eccesso dalla fetta eliminata utilizzando una carta velina pulita e priva di lanugine.
- Aggiungere il gel ScaleS4 sulla fetta di cervello utilizzando una micropipetta per riempire la camera di imaging. Posiziona un altro coverslip sopra con una pinza e posiziona un pezzo di carta velina priva di lanugine e un vetrino sul coperchio in questo ordine.
- Trasferire la camera di imaging in frigorifero a 4 °C. Posizionare i pesi metallici sul vetrino e lasciarli per 30 minuti.
- Rimuovere i pesi metallici, il vetrino, la carta velina priva di lanugine e il coperchio dalla camera di imaging e pulire il gel in eccesso (Figura 2A, B).
- Posizionare la camera di imaging in una capsula di Petri di vetro da 60 mm e fissare il bordo della camera di imaging alla parabola con un adesivo sensibile alla pressione simile a uno stucco. Attaccare la camera in più punti alla capsula di Petri.
- Versare la soluzione di ScaleS4 nella parabola e agitare delicatamente per 1 ora a 20-25 °C su uno shaker orbitale a 40-60 giri/min. Sostituire con soluzione fresca e rimuovere le bolle d'aria sulla superficie del gel raschiando delicatamente la superficie utilizzando una punta della pipetta da 200 μL. Montare la camera di imaging immersa su uno stadio di microscopio (Figura 2C).
4. Imaging CLSM
- Acquisire immagini utilizzando un CLSM dotato di un obiettivo multi-immersione a lunga distanza di lavoro (WD) (apertura numerica 16x/0,60 [NA], WD = 2,5 mm).
NOTA: le lenti ad alto NA possono fornire un'elevata risoluzione limitata alla diffrazione. - Accendi tutte le apparecchiature di imaging pertinenti (workstation, microscopio, scanner, laser e lampada al mercurio) e avvia un software di imaging CLSM.
- Impostare il collare di correzione dell'obiettivo multi-immersione su 1,47. La soluzione ScaleS4 ha un indice di rifrazione (RI) di circa 1,47 5,7. Le aberrazioni indotte da mancata corrispondenza del RI possono disturbare la formazione dell'immagine (Figura 3).
- Immergere l'obiettivo nella soluzione e lasciarlo avvicinare lentamente alla fetta. Rimuovere eventuali bolle d'aria intrappolate sulla punta della lente dell'obiettivo. Trova le regioni di interesse (ROI) nei tessuti eliminati usando l'epifluorescenza.
- Impostare i parametri di acquisizione delle immagini testando le impostazioni appropriate.
- Determinare la profondità di bit per l'acquisizione delle immagini. La dimensione dei dati dell'immagine aumenta con la profondità di bit.
- Impostare la lunghezza d'onda di rilevamento. Regolare il gate appropriato per il rilevatore in base allo spettro di emissione. Assicurarsi che la lunghezza d'onda di rilevamento non copra le linee laser.
- Impostare la risoluzione xy . I formati più grandi offrono risoluzioni xy migliori, ma ci vuole più tempo per raccogliere le immagini.
- Impostare la velocità di scansione. Una velocità di scansione più lenta fornisce un elevato rapporto segnale-rumore. Tuttavia, aumenta anche il tempo di permanenza dei pixel e il rischio di fotosbiancamento. Scegli di conseguenza.
- Regolare la dimensione del foro stenopeico. La dimensione del foro stenopeico controlla lo spessore della sezione ottica. Una dimensione stenopeica più piccola crea una sezione ottica più sottile e quindi una migliore risoluzione z , ma riduce il segnale di fluorescenza. L'ingrandimento delle dimensioni del foro stenopeico fornisce una sezione ottica più spessa con un segnale di fluorescenza più forte.
- Impostare la potenza del laser, il guadagno del rilevatore/amplificatore e l'offset. Aumentare gradualmente la potenza del laser e il guadagno del rilevatore/amplificatore fino ad ottenere un'immagine adatta. Un'elevata potenza laser comporta il rischio di fotosbiancamento. Regolare l'offset (contrasto) in modo appropriato per ottenere un elevato rapporto segnale-rumore.
- Determinare l'area di lavorazione necessaria in base alle dimensioni del ROI. Assicurati che l'intera lunghezza e larghezza del ROI venga acquisita.
- Naviga tra i tessuti eliminati su tutti i piani e imposta i punti iniziale e finale della pila. Impostare la dimensione z-step in base alla risoluzione z desiderata.
- Raccogli le immagini quando sei soddisfatto delle impostazioni di acquisizione delle immagini e registra le immagini acquisite. Elaborare le immagini utilizzando un software di analisi delle immagini.
Risultati
La compensazione ottica di una fetta di cervello di topo di 1 mm di spessore è stata ottenuta utilizzando questo protocollo. La Figura 1B rappresenta le immagini di trasmissione di una fetta di cervello di topo prima e dopo il trattamento di compensazione. Il metodo di pulizia dei tessuti ha reso trasparente una fetta di cervello di topo spessa 1 mm. Una leggera espansione nelle dimensioni finali delle fette di cervello è stata trovata dopo l'incubazione nella soluzione di compensazione per 12 ore (espansione lineare: 102,5% ± 1,3%). La conservazione della fluorescenza e dell'integrità strutturale dei tessuti è stata valutata con espressione mirata di EGFP nella membrana plasmatica nei topi PV-FGL (Figura 1C). In questi topi, l'EGFP mirato alla membrana somatodendritica è espresso nei neuroniPV-positivi 9. L'espressione di EGFP mirata alla membrana plasmatica nella regione somatodendritica è stata mantenuta dopo il trattamento (Figura 1C). Inoltre, il precedente studio EM mostra un'integrità strutturale ben conservata nei tessuti cerebrali eliminati con ScaleSF (Figura supplementare 1)6.
Le aberrazioni indotte da discrepanze RI hanno causato una notevole perdita di luminosità e risoluzione dell'immagine (Figura 3). Una fetta di cervello di 1 mm di spessore di topo PV-FGL è stata cancellata e fotografata sotto un CLSM dotato di una lente obiettivo multi-immersione di un lungo WD. La regolazione del collare di correzione della lente dell'obiettivo alla posizione dell'acqua (RI 1.33) ha ostacolato la chiara visualizzazione dei neuroni positivi EGFP situati alle profondità di 400 μm e 800 μm a causa della bassa luminosità e del basso contrasto. (Figura 3A,C). Questi neuroni sono stati chiaramente visualizzati con lo stesso CLSM, quando il collare di correzione è stato regolato per corrispondere alla soluzione ScaleS4 (RI 1.47; Figura 3B,D). La corrispondenza RI tra un fluido ad immersione e una lente obiettivo è fondamentale per un'accurata imaging 3D nei tessuti otticamente eliminati.
Infine, i neuroni neocorticali di topo sono stati utilizzati per dimostrare la fattibilità del protocollo. Un cervello di topo iniettato con il vettore AAV2/1-SynTetOff-EGFP10 nella corteccia somatosensoriale primaria (S1) è stato fissato con il 4% di PFA in 0,1 M PB. Fette coronali di spessore di 1 mm sono state preparate dal cervello con un'affettatrice di tessuto vibrante. Dopo la pulizia e il montaggio sulla camera di imaging, è stata condotta l'imaging neuronale mirato ai neuroni neocorticali (Figura 4). Una ricostruzione 3D dei neuroni marcati con EGFP nella fetta di cervello spessa 1 mm è rappresentata nella Figura 4A. Un'immagine a ingrandimento più elevato mostra singoli pergole dendritiche decorate con spine dendritiche (Figura 4B). Abbiamo inoltre mostrato arborizzazioni terminali assonali e bouton assonali nella corteccia controlaterale (Figura 4C).
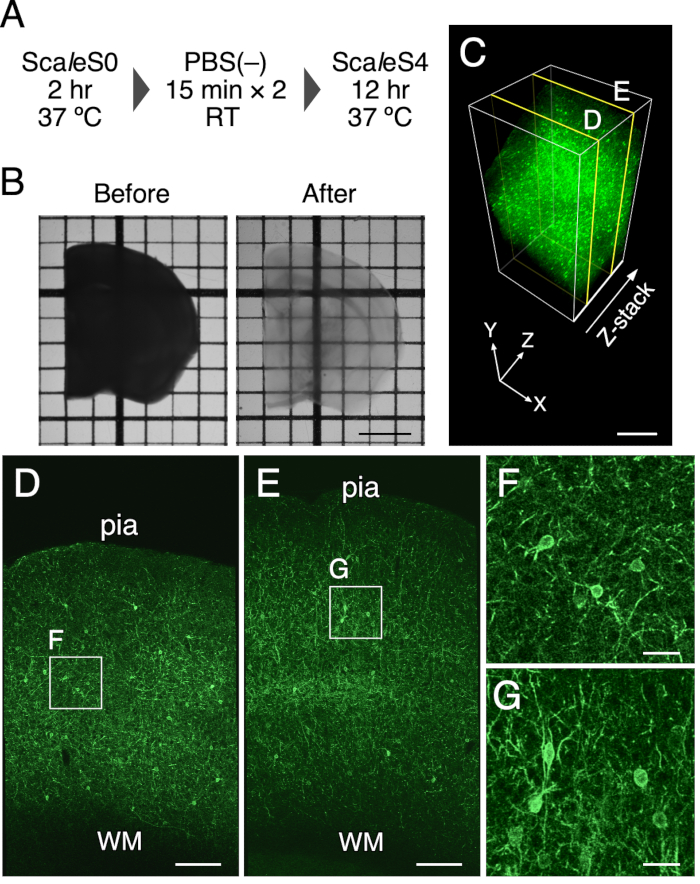
Figura 1: Clearing ottico di fette di cervello di topo di 1 mm di spessore. (A) Il programma perlapulizia dei tessuti Sca l eSF. (B) Immagini di trasmissione di fette di cervello spesse 1 mm prima (a sinistra) e dopo (a destra) il trattamento. (C) Un rendering in volume 3D della corteccia cerebrale di un topo PV-FGL eliminato utilizzando il metodo di compensazione dei tessuti. (D,E) immagini xy in (C) alle profondità di 250 μm (D) e 750 μm (E). (F,G) Vista ingrandita dei rettangoli di cui alle lettere (D) ed (E). Le immagini visualizzate in (C-G) vengono deconvolute prima del processo di rendering. Abbreviazioni: pia = pia mater, WM = sostanza bianca. Barra della scala: 2 mm in (B), 500 μm in (C), 200 μm in (D,E) e 40 μm in (F,G). Questa cifra è stata modificata da Furata et al. 20226. Fare clic qui per visualizzare una versione più grande di questa figura.
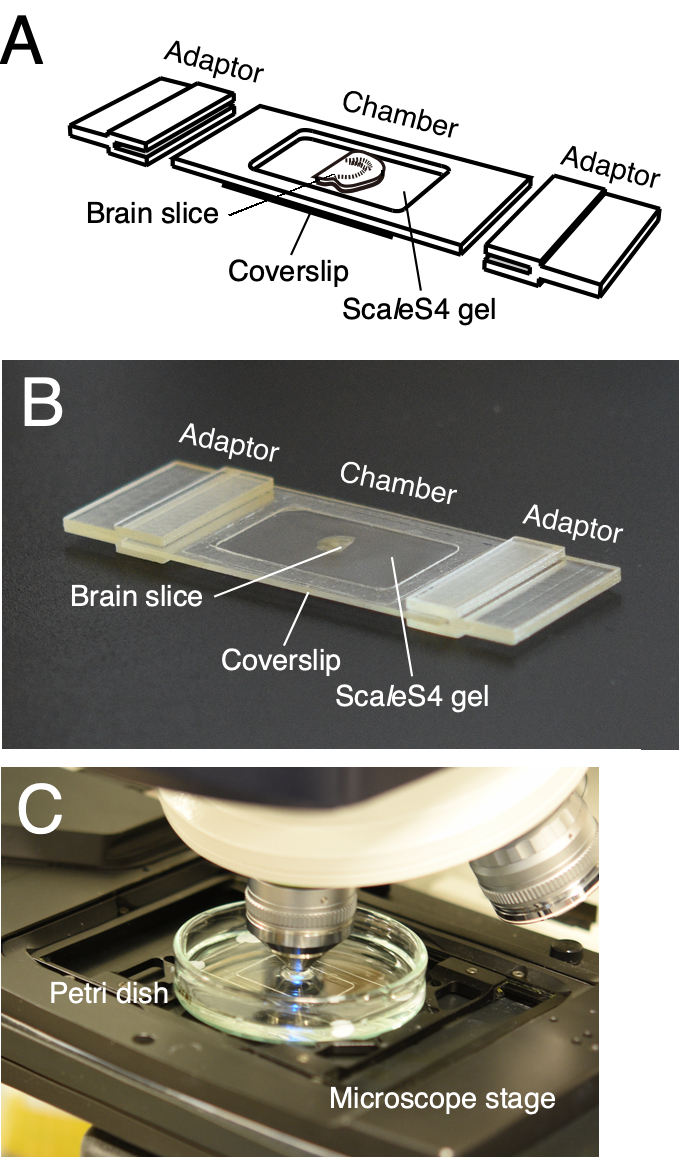
Figura 2: Una camera di imaging personalizzabile stampata in 3D per la visualizzazione di fette di tessuto. (A, B) Un disegno dello schema (A) e immagine (B) di una camera di imaging stampata in 3D personalizzabile. La camera di imaging è composta da un telaio della camera, un coperchio inferiore e adattatori per stadio del microscopio. Le fette di tessuto ripulite vengono posizionate sul coperchio inferiore e incorporate nel gel ScaleS4. Il telaio della camera, il coperchio inferiore e gli adattatori dello stadio del microscopio sono personalizzabili in base alle dimensioni e allo spessore delle fette di tessuto. (C) Una configurazione di imaging con la camera di imaging. La camera di imaging è immersa nella soluzione ScaleS4 in una capsula di Petri e montata su uno stadio di un CLSM verticale. Questa cifra è stata modificata da Furata et al. 20226. Fare clic qui per visualizzare una versione più grande di questa figura.

Figura 3: Imaging profondo compromesso causato da una mancata corrispondenza RI tra una lente obiettiva e una soluzione scaleS4. (A-D) immagini xy della corteccia cerebrale di un topo PV-FGL a profondità di 400 μm (A,B) e 800 μm (C,D). La correzione del collare di una lente multi-immersione è regolata a 1,33 in (A,C) e 1,47 in (B,D). Le immagini vengono acquisite con gli stessi parametri ad eccezione dei RI dell'obiettivo. Barra della scala: 50 μm. Fare clic qui per visualizzare una versione più grande di questa figura.

Figura 4: Imaging neuronale in una fetta di cervello di topo spessa 1 mm cancellata con ScaleSF. (A) Rendering del volume 3D dei neuroni neocorticali di topo marcati EGFP nell'S1. I neuroni neocorticali sono etichettati con il vettore AAV2/1 SynTetOff-EGFP. (B) Pergole dendritiche di neuroni neocorticali marcati EGFP. Viene rappresentata un'immagine di proiezione a intensità massima (MIP) da una profondità di 39 μm a 48 μm. Le punte di freccia indicano spine dendritiche. (C) Terminali assoni marcati EGFP nella corteccia controlaterale. Viene rappresentata un'immagine MIP da una profondità di 481,5 μm a 513 μm. Le punte di freccia indicano i bouton assonali. Le immagini che appaiono in (B) e (C) sono deconvolute. Barre di scala: 300 μm in (A) e 10 μm in (C). La barra di cui al punto (C) si applica anche a (B). Fare clic qui per visualizzare una versione più grande di questa figura.
Figura supplementare 1: Ultrastruttura in fette di cervellocancellatecon Sca l eSF, CUBIC e PACT. (A-C) Trasmissione EM immagini della corteccia cerebrale di topo cancellate con ScaleSF (A), CUBIC (B) e PACT (C). I cervelli dei topi sono fissati con il 4% di PFA contenente l'1% di glutaraldeide. Le sezioni ultrasottili sono preparate da fette di cervello cancellate. Le strutture della membrana sono gravemente danneggiate nelle fette di cervello eliminate con CUBIC (B) e PACT (C). Le punte di freccia indicano le membrane postsinaptiche. Barra scala: 500 nm. Questa cifra è stata modificata da Furata et al. 20226. Fare clic qui per scaricare questo file.
| Ricette per le soluzioni ScaleS | |
| Soluzione Sca leS0 | |
| Reagente | Concentrazione finale |
| D-sorbitolo | 20% (p/v) |
| Glicerolo | 5% (p/v) |
| Metil-β-ciclodestrina | 1 mM |
| γ-ciclodestrina | 1 mM |
| Dimetil solfossido | 3% (v/v) |
| 10x PBS(–) | 1x |
| Soluzione Sca leS4 | |
| Reagente | Concentrazione finale |
| Urea | 4 Metri |
| D-sorbitolo | 40% (p/v) |
| Glicerolo | 10% (p/v) |
| Triton X-100 | 0,2% (p/v) |
| Dimetil solfossido | 25% (v/v) |
| ScaleS4D25(0) soluzione | |
| Reagente | Concentrazione finale |
| Urea | 4 Metri |
| D-sorbitolo | 40% (p/v) |
| Glicerolo | 10% (p/v) |
| Dimetil solfossido | 25% (v/v) |
Tabella 1: Composizione delle tre soluzioni ScaleS. Sono elencate le composizioni delle soluzioni ScaleS0, Sca l eS4 e ScaleS4D25(0). Per una procedura dettagliata per la preparazione di queste soluzioni, fare riferimento a Miyawaki et al. 20167.
Discussione
Passaggi critici all'interno del protocollo
Ci sono alcuni passaggi critici nel protocollo che dovrebbero essere condotti con la massima cautela per ottenere risultati significativi. La fissazione uniforme dei campioni è indispensabile per l'imaging 3D all'interno di tessuti su larga scala. La lente dell'obiettivo, il campione e il fluido di immersione devono avere RI corrispondente. La mancata corrispondenza ri tra loro porterà a un'imaging altamente disturbato delle cellule che esprimono EGFP all'interno delle fette di cervello cancellate (Figura 3). La regolazione del collare di correzione della lente dell'obiettivo sul fluido di immersione riduce al minimo le aberrazioni sferiche indotte dalla profondità per massimizzare il segnale, il contrasto e la risoluzione spaziale nell'imaging 3D. Le IR delle soluzioni preparate possono essere misurate utilizzando un rifrattometro.
Risoluzione dei problemi della tecnica
L'archiviazione più lunga delle soluzioni può influire sulla capacità di compensazione e sulla sua capacità di conservazione dei segnali di fluorescenza e sull'integrità strutturale. Dovrebbero essere utilizzate soluzioni preparate al momento. Queste soluzioni possono essere conservate fino a 1 mese a 4 °C. L'isometria è fondamentale per un'imaging neuronale efficace ed efficiente con un'accurata ricostruzione del segnale. Sebbene sia stata osservata una leggera espansione delle dimensioni del campione dopo l'incubazione per 12 ore (Figura 1B), l'espansione può essere controllata diminuendo il periodo di incubazione tra 8-12 ore. Per un'imaging di profondità 3D più accurato, potrebbe essere necessario regolare il collare di correzione su un determinato piano a causa dell'aberrazione sferica indotta dalla profondità.
Modifiche della tecnica
Nel presente studio, abbiamo utilizzato tessuti cerebrali di topo per dimostrare la fattibilità del protocollo. Eppure il protocollo qui descritto può essere utilizzato anche per animali dal cervello grande, come i primati. In effetti, questo protocollo è stato utilizzato nei comuni tessuti cerebrali dell'uistitì (Callithrix jacchus) ed è riuscito nella visualizzazione simultanea del circuito neurale e delle strutture subcellulari dei suoi circuiti corticostriattili6. Gli adattatori per stadio del microscopio sono progettati per montare la camera di imaging sugli stadi del microscopio direttamente6 (Figura 2A, B). Le fette di tessuto ripulite sono osservabili utilizzando un microscopio invertito attraverso il coperchio inferiore della camera di imaging. Dopo il ripristino dei tessuti cerebrali eliminati con PBS(-) (deScaling)5,11, possiamo preparare sezioni di tessuto di spessore da 20 μm a 50 μm da tessuti cerebrali che sono stati eliminati con ScaleSF (ri-sezionamento)6. Le strutture subcellulari catturate all'interno dei tessuti eliminati possono essere nuovamente riprese su sezioni con una lente obiettivo NA alta di un breve WD. La pulizia delle fette di cervello perfuse con fissativi contenenti glutaraldeide è stata ottenuta utilizzando questo protocollo, fornendo una conservazione dell'ultrastruttura superiore6. ScaleSF raggiunge un elevato livello di conservazione dell'ultrastruttura che consente l'analisi EM in tessuti otticamente eliminati6 (Figura supplementare 1). La compatibilità EM di questo metodo è particolarmente utile per l'imaging di strutture con le scale dal livello macroscopico a quello nanoscopico.
Limitazioni della tecnica
Il protocollo qui descritto ci consente di visualizzare le strutture neuronali dal circuito alle scale subcellulari in fette di cervello di 1 mm di spessore. Tuttavia, rimangono tre limitazioni nel protocollo. Il primo è la capacità di compensazione del protocollo di compensazione. ScaleSF è un protocollo di compensazione per le fette di cervello, non per l'intero cervello. Sebbene fette di cervello di spessore di 1 mm possano fornire una buona conoscenza dei pergolati dendritici e assonali locali12, le informazioni sulle proiezioni assonali che coprono l'intero cervello sono frammentarie e incomplete nelle fette 13,14,15,16. Il secondo è la risoluzione di imaging. Utilizzando il protocollo qui descritto, siamo riusciti a visualizzare le strutture neuronali subcellulari, come le spine dendritiche e i bouton assonali, in una fetta di cervello otticamente cancellata (Figura 4). Tuttavia, la risoluzione della lente obiettivo utilizzata in questo studio, la risoluzione xy di 400-750 nm, non è sufficiente per risolvere strutture più fini delle cellule neuronali. Dato che le lenti ad alto NA sono tipicamente progettate per l'immersione in olio (RI 1.52), la mancata corrispondenza RI con le soluzioni (RI 1.47) potrebbe impedire l'imaging ad alta risoluzione con queste lenti obiettivo. Il terzo è l'etichettatura delle proteine fluorescenti delle cellule neuronali. Il metodo di etichettatura limita ampie applicazioni della nostra tecnica di imaging. Le tecniche istochimiche e / o immunoistochimiche che etichettano i tessuti su larga scala mantenendo l'integrità dei tessuti farebbero progredire significativamente il protocollo fornito qui.
Importanza rispetto ai metodi esistenti e alle future applicazioni della tecnica
Nel presente studio, descriviamo un protocollo dettagliato per l'imaging neuronale dalle strutture mesoscopiche a quelle microscopiche utilizzando la pulizia tissutale ScaleSF. Il protocollo qui descritto consente di visualizzare le strutture neuronali dal circuito ai livelli subcellulari in un ragionevole lasso di tempo senza attrezzature specializzate, facilitando la comprensione delle strutture neuronali dal circuito alle scale dei componenti. I neuroni elaborano processi esuberanti di enorme lunghezza e organizzano strutture fini specializzate per trasmettere e ricevere informazioni. Pertanto, l'imaging neuronale richiede un metodo di pulizia dei tessuti che esercita una potente capacità di compensazione e un alto livello di conservazione dei tessuti per la visualizzazione simultanea di strutture sia grandi che su piccola scala. Tuttavia, i metodi di compensazione dei tessuti caratterizzati da elevate capacità di compensazione rimuovono in modo aggressivo lipidi e pigmenti per un'ampia chiarificazione dei tessuti 3,4, compromettendo l'integrità dei tessuti 5,6,17 (Figura supplementare 1). Ciò è in netto contrasto con il protocollo di compensazione utilizzato qui che raggiunge un alto livello di conservazione della struttura6 (Figura supplementare 1). Pertanto,lapulizia dei tessuti Sca l eSF consente un'imaging neuronale efficace ed efficiente che richiede l'imaging 3D ad alta risoluzione su più scale con un'accurata ricostruzione del segnale.
Divulgazioni
Gli autori non hanno nulla da rivelare.
Riconoscimenti
Gli autori ringraziano Yoko Ishida (Juntendo University) per la produzione di vettori AAV e Kisara Hoshino (Juntendo University) per l'assistenza tecnica. Questo studio è stato supportato da JSPS KAKENHI (JP20K07231 to K.Y.; JP21H03529 a T.F.; JP20K07743 a M.K.; JP21H02592 a H.H.) e Ricerca Scientifica sull'Area Innovativa "Risonanza Bio" (jp18H04743 a H.H.). Questo studio è stato supportato anche dall'Agenzia giapponese per la ricerca e lo sviluppo medico (AMED) (JP21dm0207112 a T.F. e H.H.), Moonshot R& D della Japan Science and Technology Agency (JST) (JPMJMS2024 a H.H.), Fusion Oriented Research for disruptive Science and Technology (FOREST) da JST (JPMJFR204D a H.H.), Grants-in-Aid dall'Istituto di ricerca per le malattie della vecchiaia presso la Juntendo University School of Medicine (X2016 a K.Y.; X2001 a H.H.), e il Private School Branding Project.
Materiali
| Name | Company | Catalog Number | Comments |
| 16x multi-immersion objective lens | Leica Microsystems | HC FLUOTAR 16x/0.60 IMM CORR VISIR | |
| Agar | Nacalai Tesque | 01028-85 | |
| Agarose | TaKaRa Bio | L03 | |
| Dimethyl sulfoxide | Nacalai Tesque | 13407-45 | |
| D-Sorbitol | Nacalai Tesque | 06286-55 | |
| γ-cyclodextrin | Wako Pure Chemical Industries | 037-10643 | |
| Glycerol | Sigma-Aldrich | G9012 | |
| Huygens Essential | Scientific Volume Imaging | ver. 18.10.0p8/21.10.1p0 64b | |
| Imaris | Bitplane | ver. 9.0.0 | |
| Leica Application Suite X | Leica Microsystems | LAS X, ver. 3.5.5.19976 | |
| Methyl-β-cyclodextrin | Tokyo Chemical Industry | M1356 | |
| Paraformaldehyde | Merck Millipore | 1.04005.1000 | |
| Phosphate Buffered Saline (10x; pH 7.4) | Nacalai Tesque | 27575-31 | 10x PBS(–) |
| Sodium azide | Nacalai Tesque | 31233-55 | |
| Sodium pentobarbital | Kyoritsu Seiyaku | N/A | |
| TCS SP8 | Leica Microsystems | N/A | |
| Triton X-100 | Nacalai Tesque | 35501-15 | |
| Urea | Nacalai Tesque | 35940-65 | |
| Vibrating tissue slicer | Dosaka EM | PRO7N |
Riferimenti
- Susaki, E. A., Ueda, H. R. Whole-body and whole-organ clearing and imaging techniques with single-cell resolution: Toward organism-level systems biology in mammals. Cell Chemical Biology. 23 (1), 137-157 (2016).
- Tainaka, K., Kuno, A., Kubota, S. I., Murakami, T., Ueda, H. R. Chemical principles in tissue clearing and staining protocols for whole-body cell profiling. Annual Reviews of Cell and Developmental Biology. 32, 713-741 (2016).
- Ueda, H. R., et al. Whole-brain profiling of cells and circuits in mammals by tissue clearing and light-sheet microscopy. Neuron. 106 (3), 369-387 (2020).
- Ueda, H. R., et al. Tissue clearing and its applications in neuroscience. Nature Reviews. Neuroscience. 21 (2), 61-79 (2020).
- Hama, H., et al. ScaleS: an optical clearing palette for biological imaging. Nature Neuroscience. 18 (10), 1518-1529 (2015).
- Furuta, T., et al. Multi-scale light microscopy/electron microscopy neuronal imaging from brain to synapse with a tissue clearing method, ScaleSF. iScience. 25 (1), 103601(2022).
- Miyawaki, A., et al. Deep imaging of cleared brain by confocal laser-scanning microscopy. Protocol Exchange. , (2016).
- Okamoto, S., et al. Exclusive labeling of direct and indirect pathway neurons in the mouse neostriatum by an adeno-associated virus vector with Cre/lox system. STAR Protocols. 2 (1), 100230(2021).
- Kameda, H., et al. Parvalbumin-producing cortical interneurons receive inhibitory inputs on proximal portions and cortical excitatory inputs on distal dendrites. The European Journal of Neuroscience. 35 (6), 838-854 (2012).
- Sohn, J., et al. A single vector platform for high-level gene transduction of central neurons: Adeno-associated virus vector equipped with the Tet-off system. PLoS One. 12 (1), 0169611(2017).
- Hama, H., et al. Scale: a chemical approach for fluorescence imaging and reconstruction of transparent mouse brain. Nature Neuroscience. 14 (11), 1481-1488 (2011).
- Stepanyants, A., Martinez, L. M., Ferecsko, A. S., Kisvarday, Z. F. The fractions of short- and long-range connections in the visual cortex. Proceedings of the National Academy of Sciences of the United States of America. 106 (9), 3555-3560 (2009).
- Kuramoto, E., et al. Two types of thalamocortical projections from the motor thalamic nuclei of the rat: a single neuron-tracing study using viral vectors. Cerebral Cortex. 19 (9), New York, N.Y. 2065-2077 (2009).
- Matsuda, W., et al. Single nigrostriatal dopaminergic neurons form widely spread and highly dense axonal arborizations in the neostriatum. The Journal of Neuroscience: The Official Journal of the Society for Neuroscience. 29 (2), 444-453 (2009).
- Lin, R., et al. Cell-type-specific and projection-specific brain-wide reconstruction of single neurons. Nature Methods. 15 (12), 1033-1036 (2018).
- Winnubst, J., et al. Reconstruction of 1,000 projection neurons reveals new cell types and organization of long-range connectivity in the mouse brain. Cell. 179 (1), 268-281 (2019).
- Neckel, P. H., Mattheus, U., Hirt, B., Just, L., Mack, A. F. Large-scale tissue clearing (PACT): Technical evaluation and new perspectives in immunofluorescence, histology, and ultrastructure. Scientific Reports. 6, 34331(2016).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneEsplora altri articoli
This article has been published
Video Coming Soon