Method Article
Un método de limpieza de tejidos para imágenes neuronales de escalas mesoscópicas a microscópicas
En este artículo
Resumen
El protocolo proporciona un método detallado de imágenes neuronales en cortes cerebrales utilizando un método de limpieza de tejidos, ScaleSF. El protocolo incluye la preparación del tejido cerebral, la clarificación del tejido, el manejo de rodajas despejadas y la microscopía de escaneo láser confocal de las estructuras neuronales desde los niveles mesoscópicos hasta los microscópicos.
Resumen
Aquí se proporciona un protocolo detallado para visualizar las estructuras neuronales desde los niveles mesoscópicos hasta los microscópicos en los tejidos cerebrales. Las estructuras neuronales que van desde los circuitos neuronales hasta las estructuras neuronales subcelulares se visualizan en rodajas cerebrales de ratón limpiadas ópticamente con ScaleSF. Este método de limpieza es una versión modificada de ScaleS y es un método de limpieza de tejido hidrófilo para rodajas de tejido que logra una potente capacidad de limpieza, así como un alto nivel de preservación de las señales de fluorescencia y la integridad estructural. Una cámara de imágenes tridimensional (3D) personalizable está diseñada para un montaje confiable de tejidos cerebrales despejados. Los cerebros de ratón inyectados con un vector de virus adenoasociado que transportaba un gen de proteína fluorescente verde mejorado se fijaron con paraformaldehído al 4% y se cortaron en rodajas de 1 mm de espesor con una cortadora de tejido vibrante. Las rebanadas cerebrales se eliminaron siguiendo el protocolo de limpieza, que incluye incubaciones secuenciales en tres soluciones, a saber, solución ScaleS0, solución salina tampón de fosfato (–) y solución ScaleS4, para un total de 10.5-14.5 h. Las rodajas cerebrales despejadas se montaron en la cámara de imágenes y se incrustaron en gel de agarosa al 1,5% disuelto en la solución ScaleS4D25 (0). La adquisición de imágenes 3D de las rodajas se llevó a cabo utilizando un microscopio de barrido láser confocal equipado con una lente de objetivo de inmersión múltiple de una larga distancia de trabajo. Comenzando con imágenes neuronales mesoscópicas, logramos visualizar estructuras neuronales subcelulares finas, como espinas dendríticas y boutons axonales, en las rebanadas cerebrales ópticamente despejadas. Este protocolo facilitaría la comprensión de las estructuras neuronales desde el circuito hasta las escalas de componentes subcelulares.
Introducción
Los métodos de limpieza de tejidos han mejorado la obtención de imágenes independientes de la profundidad de muestras biológicas y clínicas con microscopía de luz, lo que permite extraer información estructural sobre tejidos intactos 1,2. Las técnicas de limpieza óptica también podrían acelerar y reducir el costo del análisis histológico. Actualmente, hay tres enfoques principales de limpieza disponibles: métodos hidrófilos, hidrófobos y basados en hidrogel 1,2. Los enfoques hidrófilos superan en la preservación de las señales de fluorescencia y la integridad de los tejidos y son menos tóxicos en comparación con los otros dos enfoques 3,4.
Un método de limpieza hidrofílica, ScaleS, ocupa una posición distintiva con su preservación de la integridad estructural y molecular, así como una potente capacidad de limpieza (espectro de limpieza-preservación)5. En un estudio anterior, desarrollamos un protocolo de limpieza rápido e isométrico, ScaleSF, para rodajas de tejido (~ 1 mm de espesor) modificando el procedimiento de limpieza de ScaleS6. Este protocolo de limpieza requiere incubaciones secuenciales de cortes cerebrales en tres soluciones durante 10.5-14.5 h. El método se presenta con un alto espectro de limpieza y preservación, que es compatible incluso con el análisis de microscopía electrónica (EM) (Figura suplementaria 1), lo que permite obtener imágenes tridimensionales (3D) de alta resolución a múltiples escalas con reconstrucción precisa de la señal6. Por lo tanto, ScaleSF debe ser eficaz especialmente en el cerebro, donde las células neuronales elaboran procesos exuberantes de tremenda longitud, y organizan estructuras subcelulares finas especializadas para transmitir y recibir información. La extracción de información estructural con escalas desde el circuito hasta los niveles subcelulares en las células neuronales es bastante útil para una mejor comprensión de las funciones cerebrales.
Aquí, proporcionamos un protocolo detallado para visualizar estructuras neuronales con escalas desde el nivel mesoscópico / circuito hasta el nivel microscópico / subcelular utilizando ScaleSF. El protocolo incluye la preparación de tejidos, la clarificación de tejidos, el manejo de tejidos despejados y la microscopía de barrido láser confocal (CLSM) de tejidos despejados. Nuestro protocolo se centra en interrogar estructuras neuronales desde el circuito hasta las escalas de componentes subcelulares. Para obtener un procedimiento detallado para la preparación de las soluciones y la inyección estereotáxica de vectores de virus adenoasociados (AAV) en cerebros de ratón, consulte Miyawaki et al. 20167 y Okamoto et al. 20218, respectivamente.
Protocolo
Todos los experimentos fueron aprobados por los Comités Institucionales de Cuidado y Uso de Animales de la Universidad de Juntendo (Aprobación No. 2021245, 2021246) y realizados de acuerdo con las Directrices Fundamentales para la Conducta Adecuada de Experimentos con Animales por el Consejo científico de Japón (2006). Aquí, se utilizaron ratones machos C57BL/6J inyectados con vector AAV portador del gen de proteína fluorescente verde mejorada (EGFP) y ratones transgénicos de parvalbúmina (PV)/miristoilación-EGFP-receptor de lipoproteína de baja densidad C-terminal del cromosoma artificial bacteriano (BAC) (ratones PV-FGL)9 . Los ratones PV-FGL se mantuvieron en segundo plano C57BL/6J. No se encontraron diferencias basadas en el sexo con respecto a este estudio.
1. Preparación de tejidos
- Fijación de perfusión
NOTA: Realice los pasos 1.1.1 a 1.1.3 en una campana extractora de humos para limitar la exposición al paraformaldehído (PFA).- Anestesiar ratones machos adultos (8-16 semanas de edad) mediante una inyección intraperitoneal de sobredosis de pentobarbital sódico (200 mg/kg). Confirmar la adecuación de la anestesia por la ausencia de retirada del dedo del pie y reflejos de parpadeo.
- Abra la cavidad torácica y corte el apéndice auricular derecho con tijeras quirúrgicas. Perfundir los ratones con 20 ml de solución salina tampón de fosfato helada (PBS) utilizando una aguja de 23 G unida a una jeringa de 20 ml, seguida de una perfusión de 20 ml de PFA al 4% helado en tampón de fosfato (PB) de 0,1 M utilizando otra jeringa de 20 ml.
PRECAUCIÓN: El PFA es tóxico y teratogénico. Evite la inhalación o el contacto con la piel, los ojos y la membrana mucosa. - Extraiga los tejidos cerebrales del cráneo con pinzas. Transfiera los tejidos cerebrales a un tubo de 15 ml que contenga 4% de PFA en 0,1 M PB, proteja las muestras de la luz y mece suavemente durante la noche a 4 °C en un agitador a 50-100 rpm.
- NOTA: Los tejidos cerebrales cosechados se pueden almacenar durante varias semanas en azida de sodio al 0,02% (NaN3) en PBS a 4 °C.
PRECAUCIÓN: NaN3 es tóxico. Evite la inhalación o el contacto con la piel, los ojos y la membrana mucosa. Manejarlo dentro de una campana extractora de humos.
- Preparación de rebanadas cerebrales
- Prepare agar al 4% en PBS agregando 2 g de agar a 50 ml de PBS. Microondas la mezcla hasta que el agar esté completamente disuelto. Deje que la solución se enfríe a 40-45 °C.
NOTA: La propiedad viscosa proporcionada por la agaropectina, un componente importante del agar, mejora la facilidad de corte de las rodajas de tejido. - Agregue 10 ml de la solución de agar a una placa de cultivo de 6 pocillos. Sumerja el tejido cerebral en la solución de agar usando fórceps. Deja que el agar se solidifique sobre hielo.
- Retire el tejido cerebral incrustado del pozo y recorte el agar con una cuchilla de afeitar. Asegure el bloque de agar en la parte inferior del baño de vibratomo con superpegamento y vierta 0,1 M pb en la bandeja tampón.
- Limpie otra cuchilla de afeitar con un papel de seda sin pelusa empapado en etanol y conecte la cuchilla al soporte de la cuchilla de la cortadora de tejido vibratorio.
- Establezca la velocidad de seccionamiento en 0,14 mm/s con una amplitud de 1,4 mm y la frecuencia a 75–77 Hz. Corte el tejido cerebral en rodajas de 1 mm de grosor y recoja las rebanadas en una placa de cultivo celular de 6 pocillos que contenga PBS.
NOTA: Las rebanadas cerebrales se pueden almacenar durante varias semanas en NaN3 al 0,02% en PBS a 4 °C.
- Prepare agar al 4% en PBS agregando 2 g de agar a 50 ml de PBS. Microondas la mezcla hasta que el agar esté completamente disuelto. Deje que la solución se enfríe a 40-45 °C.
2. Clarificación tisular
NOTA: Las composiciones de las soluciones ScaleS utilizadas se enumeran en la Tabla 1. Las muestras deben protegerse de la luz cubriendo con una lámina. Los pasos de borrado se muestran en la Figura 1A.
- Agregue 8 ml de solución ScaleS0 a un pocillo de una placa de cultivo celular de 6 pocillos y agregue 8 ml de solución ScaleS4 a otro pozo de la placa y precaliente a 37 ° C en una incubadora.
- Transfiera las rodajas cerebrales a la solución ScaleS0 precalentada con una espátula e incube durante 2 h a 37 °C en una incubadora de agitación a 90 rpm.
- Transfiera las rodajas cerebrales permeabilizadas en 8 ml de PBS(–) en una placa de cultivo celular de 6 pocillos con una espátula y lávelas durante 15 minutos manteniéndolas en un agitador orbital a 40–60 rpm. Repita esto dos veces.
- Transfiera las rodajas cerebrales en los 8 ml precalentados de la solución ScaleS4 con una espátula y límpielas incubando en una incubadora de agitación a 90 rpm durante 8-12 h a 37 °C. Una rebanada de cerebro despejada se puede ver en la Figura 1.
3. Montaje de cortes cerebrales
NOTA: Se utiliza una cámara de imágenes personalizable para el montaje fiable de cortes cerebrales despejados (Figura 2)6. La cámara consiste en el marco de la cámara y la cubierta inferior. Los adaptadores de etapa del microscopio también están diseñados para montar la cámara de imágenes en las etapas del microscopio directamente (Figura 2A, B). El marco de la cámara y los adaptadores de etapa del microscopio se pueden imprimir en 3D utilizando servicios de impresión 3D internos o subcontratados. Los datos de diseño asistido por computadora (CAD) 3D de la cámara de imágenes se proporcionan en Furuta et al. 20226.
- Para la preparación de la cámara, conecte el marco de la cámara a una cubierta con un adhesivo sensible a la presión.
- Preparar agarosa al 1,5% en solución ScaleS4D25(0) (Gel ScaleS4) añadiendo 1,5 g de agarosa a 100 ml de la solución en un frasco. Mezcle bien la solución revolviendo y microondas la solución hasta que la agarosa se disuelva por completo. Una vez hecho esto, deje que la solución se enfríe a 37 °C.
- Monte la rebanada cerebral despejada en la cubierta inferior de la cámara de imágenes con una espátula. Limpie el exceso de solución de la rodaja limpia con un papel de seda limpio y sin pelusa.
- Agregue el gel ScaleS4 en la rebanada cerebral con una micropipeta para llenar la cámara de imágenes. Coloque otro cubrebocas en la parte superior con fórceps y coloque un trozo de papel de seda sin pelusa y un portaobjetos de vidrio en el cobertor en este orden.
- Transfiera la cámara de imágenes a un refrigerador a 4 °C. Coloque pesas de metal en el portaobjetos de vidrio y déjelas durante 30 minutos.
- Retire las pesas metálicas, el portaobjetos de vidrio, el papel de seda sin pelusa y el cubrehojas de la cámara de imágenes, y limpie el exceso de gel (Figura 2A, B).
- Coloque la cámara de imágenes en una placa de Petri de vidrio de 60 mm y conecte el borde de la cámara de imágenes a la placa con un adhesivo sensible a la presión similar a una masilla. Conecte la cámara en varios puntos a la placa de Petri.
- Vierta la solución ScaleS4 en el plato y agite suavemente durante 1 h a 20-25 °C en un agitador orbital a 40-60 rpm. Sustitúyalo con una solución fresca y elimine las burbujas de aire en la superficie del gel raspando suavemente la superficie con una punta de pipeta de 200 μL. Monte la cámara de imágenes sumergida en una etapa de microscopio (Figura 2C).
4. Imágenes CLSM
- Adquiera imágenes utilizando un CLSM equipado con una lente de objetivo de inmersión múltiple de larga distancia de trabajo (WD) (apertura numérica de 16x/0.60 [NA], WD = 2.5 mm).
NOTA: Las lentes de alto objetivo NA pueden proporcionar una alta resolución limitada por difracción. - Encienda todos los equipos de imágenes relevantes (estación de trabajo, microscopio, escáner, láseres y lámpara de mercurio) y lance un software de imágenes CLSM.
- Ajuste el collar de corrección de la lente del objetivo de inmersión múltiple a 1.47. La solución ScaleS4 tiene un índice de refracción (RI) de alrededor de 1,47 5,7. Las aberraciones inducidas por el desajuste de RI pueden perturbar la formación de la imagen (Figura 3).
- Sumerja la lente del objetivo en la solución y deje que se acerque lentamente a la rebanada. Retire las burbujas de aire atrapadas en la punta de la lente del objetivo. Encuentre regiones de interés (ROI) en los tejidos despejados utilizando epifluorescencia.
- Establezca parámetros de adquisición de imágenes probando la configuración adecuada.
- Determine la profundidad de bits para la adquisición de imágenes. El tamaño de los datos de la imagen aumenta con la profundidad de bits.
- Establezca la longitud de onda de detección. Ajuste la compuerta apropiada para el detector de acuerdo con el espectro de emisión. Asegúrese de que la longitud de onda de detección no cubra ninguna línea láser.
- Establezca la resolución xy . Los formatos más grandes proporcionan mejores resoluciones xy , pero lleva más tiempo recopilar las imágenes.
- Establezca la velocidad de escaneo. Una velocidad de escaneo más lenta proporciona una alta relación señal-ruido. Sin embargo, también aumenta el tiempo de permanencia de los píxeles y el riesgo de fotoblanqueo. Elija en consecuencia.
- Ajuste el tamaño del agujero de alfiler. El tamaño del agujero de alfiler controla el grosor de la sección óptica. Un tamaño de agujero de alfiler más pequeño crea una sección óptica más delgada y, por lo tanto, una mejor resolución z , pero reduce la señal de fluorescencia. Hacer que el tamaño del agujero de alfiler sea más grande proporciona una sección óptica más gruesa con una señal de fluorescencia más fuerte.
- Ajuste la potencia del láser, la ganancia del detector/amplificador y el desplazamiento. Aumente gradualmente la potencia del láser y la ganancia del detector / amplificador hasta que se obtenga una imagen adecuada. Una alta potencia láser conlleva el riesgo de fotoblanqueo. Ajuste el desplazamiento (contraste) adecuadamente para obtener una alta relación señal-ruido.
- Determine el área de labranza necesaria en función del tamaño del ROI. Asegúrese de que se captura toda la longitud y el ancho del ROI.
- Navegue por los tejidos despejados en todos los planos y establezca los puntos de inicio y final de la pila. Establezca el tamaño del paso z de acuerdo con la resolución z deseada.
- Recopile imágenes cuando esté satisfecho con la configuración de adquisición de imágenes y grabe las imágenes capturadas. Procese las imágenes utilizando un software de análisis de imágenes.
Resultados
La limpieza óptica de una rebanada de cerebro de ratón de 1 mm de espesor se logró utilizando este protocolo. La Figura 1B representa imágenes de transmisión de una rebanada de cerebro de ratón antes y después del tratamiento de limpieza. El método de limpieza de tejido hizo que una rebanada de cerebro de ratón de 1 mm de grosor fuera transparente. Se encontró una ligera expansión en los tamaños finales de las rebanadas cerebrales después de la incubación en la solución de limpieza durante 12 h (expansión lineal: 102,5% ± 1,3%). La preservación de la fluorescencia y la integridad estructural de los tejidos se evaluó con la expresión dirigida de EGFP en la membrana plasmática en ratones PV-FGL (Figura 1C). En estos ratones, el EGFP dirigido a la membrana somatodendrítica se expresa en neuronas PV-positivas9. La expresión de EGFP dirigida a la membrana plasmática en la región somatodendrítica se mantuvo después del tratamiento (Figura 1C). Además, el estudio EM anterior muestra una integridad estructural bien conservada en los tejidos cerebrales limpiados con ScaleSF (Figura suplementaria 1)6.
Las aberraciones inducidas por el desajuste de RI causaron una pérdida notable del brillo y la resolución de la imagen (Figura 3). Una rebanada cerebral de 1 mm de espesor de ratón PV-FGL fue despejada y fotografiada bajo un CLSM equipado con una lente de objetivo de inmersión múltiple de un WD largo. El ajuste del collar de corrección de la lente del objetivo a la posición del agua (RI 1.33) dificultó la visualización clara de las neuronas EGFP positivas ubicadas a las profundidades de 400 μm y 800 μm debido al bajo brillo y bajo contraste. (Figura 3A,C). Estas neuronas se visualizaron claramente con el mismo CLSM, cuando se ajustó el collar de corrección para que coincidiera con la solución ScaleS4 (RI 1,47; Figura 3B,D). La coincidencia de RI entre un fluido de inmersión y una lente objetiva es fundamental para obtener imágenes 3D precisas en tejidos ópticamente despejados.
Por último, se utilizaron neuronas neocorticales de ratón para demostrar la viabilidad del protocolo. Un cerebro de ratón inyectado con AAV2/1-SynTetOff-EGFP vector10 en la corteza somatosensorial primaria (S1) se fijó con 4% de PFA en 0,1 M PB. Se prepararon rebanadas coronales de 1 mm de espesor desde el cerebro con una cortadora de tejido vibratorio. Después de despejar y montar en la cámara de imágenes, se realizaron imágenes neuronales dirigidas a las neuronas neocorticales (Figura 4). Una reconstrucción 3D de neuronas marcadas con EGFP en la rebanada cerebral de 1 mm de espesor está representada en la Figura 4A. Una imagen de mayor aumento muestra cenadores dendríticos individuales decorados con espinas dendríticas (Figura 4B). Además, mostramos arborizaciones terminales de axones y boutones axonales en la corteza contralateral (Figura 4C).
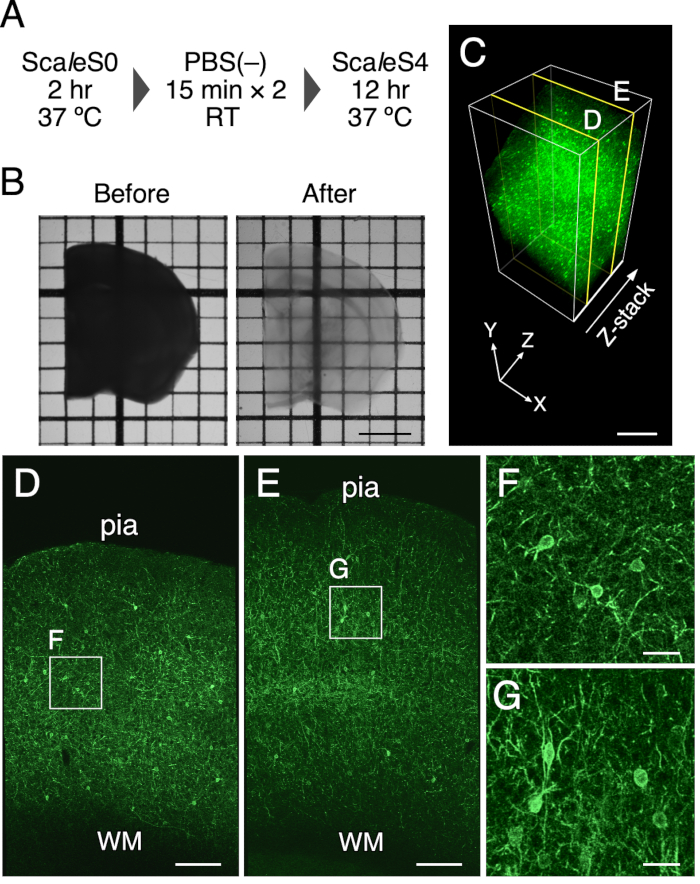
Figura 1: Limpieza óptica de cortes cerebrales de ratón de 1 mm de espesor. (A) El programa para la limpieza de tejido ScaleSF. (B) Imágenes de transmisión de un cerebro de 1 mm de grosor antes (izquierda) y después (derecha) del tratamiento. (C) Una representación de volumen 3D de la corteza cerebral de un ratón PV-FGL despejada utilizando el método de limpieza de tejidos. (D,E) xy imágenes en (C) a profundidades de 250 μm (D) y 750 μm (E). (F,G) Vista ampliada de los rectángulos descritos en (D) y (E). Las imágenes que aparecen en (C-G) se descontorbanican antes del proceso de renderizado. Abreviaturas: pia = pia mater, WM = materia blanca. Barra de escala: 2 mm en (B), 500 μm en (C), 200 μm en (D,E) y 40 μm en (F,G). Esta cifra ha sido modificada a partir de Furata et al. 20226. Haga clic aquí para ver una versión más grande de esta figura.
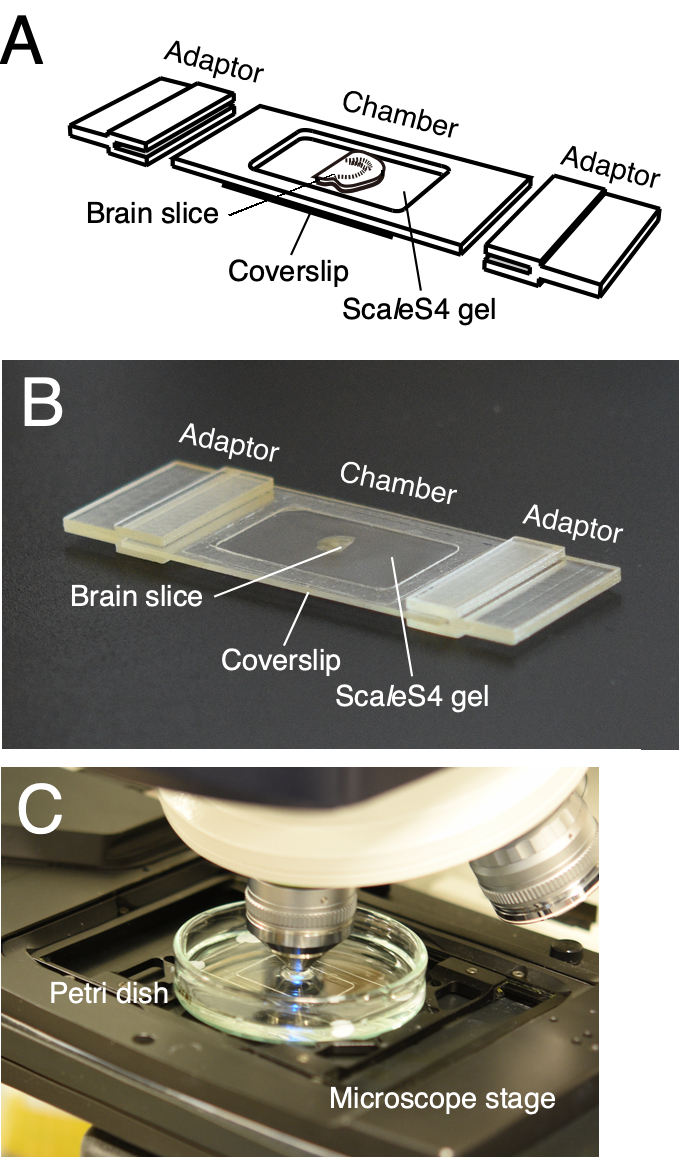
Figura 2: Una cámara de imágenes personalizable impresa en 3D para la visualización de cortes de tejido. (A, B) Un dibujo de esquema (A) e imagen (B) de una cámara de imágenes personalizable impresa en 3D. La cámara de imagen se compone de un marco de cámara, un labio de cubierta inferior y adaptadores de etapa de microscopio. Las rodajas de tejido despejado se colocan en la cubierta inferior y se incrustan en el gel ScaleS4. El marco de la cámara, la cubierta inferior y los adaptadores de etapa de microscopio son personalizables de acuerdo con el tamaño y el grosor de las rodajas de tejido. (C) Una configuración de imágenes con la cámara de imágenes. La cámara de imagen se sumerge en la solución ScaleS4 en una placa de Petri y se monta en un escenario de un CLSM vertical. Esta cifra ha sido modificada a partir de Furata et al. 20226. Haga clic aquí para ver una versión más grande de esta figura.

Figura 3: Imágenes profundas comprometidas causadas por un desajuste de RI entre una lente objetivo y la solución ScaleS4. (A-D) Imágenes xy de la corteza cerebral de un ratón PV-FGL a profundidades de 400 μm (A, B) y 800 μm (C, D). La corrección del collar de una lente de objetivo de inmersión múltiple se ajusta a 1.33 in (A, C) y 1.47 in (B, D). Las imágenes se adquieren con los mismos parámetros a excepción de los RI de la lente del objetivo. Barra de escala: 50 μm. Haga clic aquí para ver una versión más grande de esta figura.

Figura 4: Imágenes neuronales en una rebanada de cerebro de ratón de 1 mm de espesor despejada con ScaleSF. (A) Representación de volumen 3D de neuronas neocorticales de ratón marcadas con EGFP en el S1. Las neuronas neocorticales están marcadas con el vector AAV2/1 SynTetOff-EGFP. (B) Cenadores dendríticos de neuronas neocorticales marcadas con EGFP. Se representa una imagen de proyección de máxima intensidad (MIP) desde una profundidad de 39 μm a 48 μm. Las puntas de flecha indican espinas dendríticas. (C) Terminales axónicos marcados con EGFP en la corteza contralateral. Se representa una imagen MIP de una profundidad de 481,5 μm a 513 μm. Las puntas de flecha indican boutons axonales. Las imágenes que aparecen en (B) y (C) son desenrevesadas. Barras de escala: 300 μm en (A) y 10 μm en (C). La barra en (C) también se aplica a (B). Haga clic aquí para ver una versión más grande de esta figura.
Figura suplementaria 1: Ultraestructura en cortes cerebrales despejados con ScaleSF, CUBIC y PACT. (A-C) Transmisión de imágenes EM de la corteza cerebral del ratón borradas con ScaleSF (A), CUBIC (B) y PACT (C). Los cerebros de ratón se fijan con 4% de PFA que contiene 1% de glutaraldehído. Las secciones ultrafinas se preparan a partir de rodajas cerebrales despejadas. Las estructuras de la membrana están severamente dañadas en cortes cerebrales limpiados con CUBIC (B) y PACT (C). Las puntas de flecha indican membranas postsinápticas. Barra de escala: 500 nm. Esta cifra ha sido modificada a partir de Furata et al. 20226. Haga clic aquí para descargar este archivo.
| Recipies para las soluciones ScaleS | |
| Solución ScaleS0 | |
| Reactivo | Concentración final |
| D-sorbitol | 20% (p/v) |
| Glicerol | 5% (p/v) |
| Metil-β-ciclodextrina | 1 mM |
| γ-ciclodextrina | 1 mM |
| Dimetilsulfóxido | 3% (v/v) |
| 10x PBS(–) | 1x |
| Solución ScaleS4 | |
| Reactivo | Concentración final |
| Urea | 4 M |
| D-sorbitol | 40% (p/v) |
| Glicerol | 10% (p/v) |
| Tritón X-100 | 0,2% (p/v) |
| Dimetilsulfóxido | 25% (v/v) |
| Solución ScaleS4D25(0) | |
| Reactivo | Concentración final |
| Urea | 4 M |
| D-sorbitol | 40% (p/v) |
| Glicerol | 10% (p/v) |
| Dimetilsulfóxido | 25% (v/v) |
Tabla 1: Composición de las tres soluciones ScaleS. Se enumeran las composiciones de las soluciones ScaleS0, ScaleS4 y ScaleS4D25(0). Para un procedimiento detallado para la preparación de estas soluciones, consulte Miyawaki et al. 20167.
Discusión
Pasos críticos dentro del protocolo
Hay algunos pasos críticos en el protocolo que deben llevarse a cabo con la máxima precaución para obtener resultados significativos. La fijación uniforme de muestras es imprescindible para la obtención de imágenes 3D dentro de tejidos a gran escala. La lente del objetivo, la muestra y el fluido de inmersión deben tener ri correspondiente. El desajuste de RI entre ellos conducirá a imágenes altamente perturbadas de las células que expresan EGFP dentro de las rebanadas cerebrales despejadas (Figura 3). El ajuste del collar de corrección de la lente del objetivo al fluido de inmersión minimiza las aberraciones esféricas inducidas por la profundidad para maximizar la señal, el contraste y la resolución espacial en las imágenes 3D. Los RI de las soluciones preparadas se pueden medir utilizando un refractómetro.
Solución de problemas de la técnica
El almacenamiento más prolongado de las soluciones puede afectar la capacidad de limpieza y su capacidad para la preservación de las señales de fluorescencia y la integridad estructural. Se deben utilizar soluciones recién preparadas. Estas soluciones se pueden almacenar hasta 1 mes a 4 °C. La isometría es fundamental para obtener imágenes neuronales efectivas y eficientes con una reconstrucción precisa de la señal. Aunque se observó una ligera expansión en el tamaño de la muestra después de la incubación durante 12 h (Figura 1B), la expansión se puede controlar disminuyendo el período de incubación entre 8-12 h. Para obtener imágenes de profundidad 3D más precisas, es posible que sea necesario ajustar el collar de corrección en un plano determinado debido a la aberración esférica inducida por la profundidad.
Modificaciones de la técnica
En el presente estudio, utilizamos tejidos cerebrales de ratón para demostrar la viabilidad del protocolo. Sin embargo, el protocolo descrito aquí también se puede usar para animales de cerebro grande, como los primates. De hecho, este protocolo se ha utilizado en tejidos cerebrales de tití común (Callithrix jacchus), y ha tenido éxito en la visualización simultánea del circuito neuronal y las estructuras subcelulares de sus circuitos corticoestriatales6. Los adaptadores de etapa de microscopio están diseñados para montar la cámara de imágenes en etapas de microscopio directamente6 (Figura 2A, B). Las rodajas de tejido despejado son observables utilizando un microscopio invertido a través de la cubierta inferior de la cámara de imágenes. Después de la restauración de los tejidos cerebrales despejados con PBS(-) (deScaling)5,11, podemos preparar secciones de tejido de 20-μm a 50-μm de espesor a partir de tejidos cerebrales que fueron limpiados con ScaleSF (re-sección)6. Las estructuras subcelulares capturadas dentro de los tejidos despejados se pueden obtener nuevamente en re-secciones con una lente de alto objetivo NA de un WD corto. La limpieza de cortes cerebrales perfundidas con fijadores que contienen glutaraldehído se ha logrado utilizando este protocolo, proporcionando una preservación superior de la ultraestructura6. ScaleSF logra un alto nivel de preservación de la ultraestructura que permite el análisis EM en tejidos ópticamente despejados6 (Figura suplementaria 1). La compatibilidad EM de este método es particularmente útil para obtener imágenes de estructuras con escalas desde el nivel macroscópico hasta el nanoscópico.
Limitaciones de la técnica
El protocolo descrito aquí nos permite visualizar estructuras neuronales desde el circuito hasta las escalas subcelulares en rebanadas cerebrales de 1 mm de espesor. Sin embargo, quedan tres limitaciones en el protocolo. El primero es la capacidad de compensación del protocolo de compensación. ScaleSF es un protocolo de limpieza para cortes de cerebro, no para todo el cerebro. Aunque las rebanadas cerebrales de 1 mm de espesor pueden proporcionar un buen conocimiento de los cenadores axonales dendríticos y locales12, la información sobre las proyecciones axonales que abarcan todo el cerebro es fragmentaria e incompleta en las rebanadas 13,14,15,16. El segundo es la resolución de la imagen. Usando el protocolo descrito aquí, logramos visualizar estructuras neuronales subcelulares, como espinas dendríticas y boutons axonales, en una rebanada cerebral ópticamente despejada (Figura 4). Sin embargo, la resolución de la lente objetivo utilizada en este estudio, resolución xy de 400-750 nm, no es suficiente para resolver estructuras más finas de las células neuronales. Dado que las lentes de alto objetivo NA suelen estar diseñadas para la inmersión en aceite (RI 1.52), el desajuste de RI con las soluciones (RI 1.47) podría evitar la obtención de imágenes de alta resolución con estas lentes de objetivo. El tercero es el marcado de proteínas fluorescentes de las células neuronales. El método de etiquetado limita las amplias aplicaciones de nuestra técnica de imagen. Las técnicas histoquímicas y / o inmunohistoquímicas que etiquetan los tejidos a gran escala mientras mantienen la integridad del tejido avanzarían significativamente el protocolo proporcionado aquí.
Importancia con respecto a los métodos existentes y las aplicaciones futuras de la técnica
En el presente estudio, describimos un protocolo detallado para la obtención de imágenes neuronales desde estructuras mesoscópicas hasta estructuras microscópicas utilizando la limpieza de tejidos ScaleSF. El protocolo descrito aquí permite visualizar estructuras neuronales desde el circuito hasta los niveles subcelulares en un período de tiempo razonable sin equipo especializado, facilitando la comprensión de las estructuras neuronales desde el circuito hasta las escalas de componentes. Las neuronas elaboran procesos exuberantes de tremenda longitud y organizan estructuras finas especializadas para transmitir y recibir información. Por lo tanto, las imágenes neuronales requieren un método de limpieza de tejidos que ejerza una potente capacidad de limpieza, así como un alto nivel de preservación de tejidos para la visualización simultánea de estructuras a gran y pequeña escala. Sin embargo, los métodos de limpieza de tejidos presentados con altas capacidades de limpieza eliminan agresivamente los lípidos y pigmentos para una amplia clarificación de tejidos 3,4, comprometiendo la integridad del tejido 5,6,17 (Figura suplementaria 1). Esto está en marcado contraste con el protocolo de limpieza utilizado aquí que logra un alto nivel de preservación de la estructura6 (Figura suplementaria 1). Por lo tanto, la limpieza de tejidos ScaleSF permite una imagen neuronal efectiva y eficiente que requiere imágenes 3D de alta resolución multiescala con reconstrucción de señal precisa.
Divulgaciones
Los autores no tienen nada que revelar.
Agradecimientos
Los autores agradecen a Yoko Ishida (Universidad de Juntendo) por la producción de vectores AAV y a Kisara Hoshino (Universidad de Juntendo) por la asistencia técnica. Este estudio fue apoyado por JSPS KAKENHI (JP20K07231 a K.Y.; JP21H03529 a T.F.; JP20K07743 a M.K.; JP21H02592 a S.S.) e Investigación Científica en el Área Innovadora "Resonancia Bio" (JP18H04743 a H.H.). Este estudio también fue apoyado por la Agencia Japonesa de Investigación y Desarrollo Médico (AMED) (JP21dm0207112 a T.F. y H.H.), Moonshot R&D de la Agencia de Ciencia y Tecnología de Japón (JST) (JPMJMS2024 a H.H.), Investigación Orientada a la Fusión para Ciencia y Tecnología disruptiva (FOREST) de JST (JPMJFR204D a H.H.), Becas en Ayuda del Instituto de Investigación para Enfermedades de la Vejez en la Facultad de Medicina de la Universidad de Juntendo (X2016 a K.Y.; X2001 a H.H.), y el Private School Branding Project.
Materiales
| Name | Company | Catalog Number | Comments |
| 16x multi-immersion objective lens | Leica Microsystems | HC FLUOTAR 16x/0.60 IMM CORR VISIR | |
| Agar | Nacalai Tesque | 01028-85 | |
| Agarose | TaKaRa Bio | L03 | |
| Dimethyl sulfoxide | Nacalai Tesque | 13407-45 | |
| D-Sorbitol | Nacalai Tesque | 06286-55 | |
| γ-cyclodextrin | Wako Pure Chemical Industries | 037-10643 | |
| Glycerol | Sigma-Aldrich | G9012 | |
| Huygens Essential | Scientific Volume Imaging | ver. 18.10.0p8/21.10.1p0 64b | |
| Imaris | Bitplane | ver. 9.0.0 | |
| Leica Application Suite X | Leica Microsystems | LAS X, ver. 3.5.5.19976 | |
| Methyl-β-cyclodextrin | Tokyo Chemical Industry | M1356 | |
| Paraformaldehyde | Merck Millipore | 1.04005.1000 | |
| Phosphate Buffered Saline (10x; pH 7.4) | Nacalai Tesque | 27575-31 | 10x PBS(–) |
| Sodium azide | Nacalai Tesque | 31233-55 | |
| Sodium pentobarbital | Kyoritsu Seiyaku | N/A | |
| TCS SP8 | Leica Microsystems | N/A | |
| Triton X-100 | Nacalai Tesque | 35501-15 | |
| Urea | Nacalai Tesque | 35940-65 | |
| Vibrating tissue slicer | Dosaka EM | PRO7N |
Referencias
- Susaki, E. A., Ueda, H. R. Whole-body and whole-organ clearing and imaging techniques with single-cell resolution: Toward organism-level systems biology in mammals. Cell Chemical Biology. 23 (1), 137-157 (2016).
- Tainaka, K., Kuno, A., Kubota, S. I., Murakami, T., Ueda, H. R. Chemical principles in tissue clearing and staining protocols for whole-body cell profiling. Annual Reviews of Cell and Developmental Biology. 32, 713-741 (2016).
- Ueda, H. R., et al. Whole-brain profiling of cells and circuits in mammals by tissue clearing and light-sheet microscopy. Neuron. 106 (3), 369-387 (2020).
- Ueda, H. R., et al. Tissue clearing and its applications in neuroscience. Nature Reviews. Neuroscience. 21 (2), 61-79 (2020).
- Hama, H., et al. ScaleS: an optical clearing palette for biological imaging. Nature Neuroscience. 18 (10), 1518-1529 (2015).
- Furuta, T., et al. Multi-scale light microscopy/electron microscopy neuronal imaging from brain to synapse with a tissue clearing method, ScaleSF. iScience. 25 (1), 103601 (2022).
- Miyawaki, A., et al. Deep imaging of cleared brain by confocal laser-scanning microscopy. Protocol Exchange. , (2016).
- Okamoto, S., et al. Exclusive labeling of direct and indirect pathway neurons in the mouse neostriatum by an adeno-associated virus vector with Cre/lox system. STAR Protocols. 2 (1), 100230 (2021).
- Kameda, H., et al. Parvalbumin-producing cortical interneurons receive inhibitory inputs on proximal portions and cortical excitatory inputs on distal dendrites. The European Journal of Neuroscience. 35 (6), 838-854 (2012).
- Sohn, J., et al. A single vector platform for high-level gene transduction of central neurons: Adeno-associated virus vector equipped with the Tet-off system. PLoS One. 12 (1), 0169611 (2017).
- Hama, H., et al. Scale: a chemical approach for fluorescence imaging and reconstruction of transparent mouse brain. Nature Neuroscience. 14 (11), 1481-1488 (2011).
- Stepanyants, A., Martinez, L. M., Ferecsko, A. S., Kisvarday, Z. F. The fractions of short- and long-range connections in the visual cortex. Proceedings of the National Academy of Sciences of the United States of America. 106 (9), 3555-3560 (2009).
- Kuramoto, E., et al. Two types of thalamocortical projections from the motor thalamic nuclei of the rat: a single neuron-tracing study using viral vectors. Cerebral Cortex. 19 (9), 2065-2077 (2009).
- Matsuda, W., et al. Single nigrostriatal dopaminergic neurons form widely spread and highly dense axonal arborizations in the neostriatum. The Journal of Neuroscience: The Official Journal of the Society for Neuroscience. 29 (2), 444-453 (2009).
- Lin, R., et al. Cell-type-specific and projection-specific brain-wide reconstruction of single neurons. Nature Methods. 15 (12), 1033-1036 (2018).
- Winnubst, J., et al. Reconstruction of 1,000 projection neurons reveals new cell types and organization of long-range connectivity in the mouse brain. Cell. 179 (1), 268-281 (2019).
- Neckel, P. H., Mattheus, U., Hirt, B., Just, L., Mack, A. F. Large-scale tissue clearing (PACT): Technical evaluation and new perspectives in immunofluorescence, histology, and ultrastructure. Scientific Reports. 6, 34331 (2016).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoExplorar más artículos
This article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados