Method Article
Uso do Análogo da Pirimidina 5-Iodo-2′-Desoxiuridina (IdU) com Marcadores do Ciclo Celular para Estabelecer Fases do Ciclo Celular em uma Plataforma de Citometria de Massa
Neste Artigo
Resumo
Este protocolo adapta medidas do ciclo celular para uso em uma plataforma de citometria de massa. Com as capacidades multiparamétricas da citometria de massa, a medição direta da incorporação de iodo permite a identificação de células em fase S, enquanto marcadores de ciclagem intracelular permitem a caracterização de cada estado do ciclo celular em uma variedade de condições experimentais.
Resumo
A regulação da fase do ciclo celular é um aspecto importante da proliferação celular e da homeostase. A interrupção dos mecanismos regulatórios que regem o ciclo celular é uma característica de uma série de doenças, incluindo o câncer. O estudo do ciclo celular requer a capacidade de definir o número de células em cada porção da progressão do ciclo celular, bem como delinear claramente entre cada fase do ciclo celular. O advento da citometria de massa (MCM) fornece um enorme potencial para análise de célula única de alto rendimento através de medições diretas de isótopos elementares, e o desenvolvimento de um método para medir o estado do ciclo celular por MCM amplia ainda mais a utilidade da MCM. Descrevemos aqui um método que mede diretamente a 5-iodo-2′-desoxiuridina (IdU), semelhante à 5-bromo-2'-desoxiuridina (BrdU), em um sistema MCM. O uso desse MCM baseado em IdU oferece várias vantagens. Primeiro, o IdU é rapidamente incorporado ao DNA durante sua síntese, permitindo a medição confiável de células na fase S com incubações tão curtas quanto 10-15 minutos. Em segundo lugar, o IdU é medido sem a necessidade de anticorpos secundários ou a necessidade de degradação do DNA. Terceiro, a coloração de IdU pode ser facilmente combinada com a medição de ciclina B1, proteína fosforilada de retinoblastoma (pRb) e histona fosforilada H3 (pHH3), que coletivamente fornece delineamento claro das cinco fases do ciclo celular. A combinação desses marcadores de ciclo celular com o alto número de parâmetros possíveis com a MCM permite a combinação com inúmeras outras métricas.
Introdução
A citometria de massa permite a detecção de aproximadamente 40 parâmetros, aproveitando a alta resolução e a natureza quantitativa da espectroscopia de massas. Anticorpos marcados com metais são usados em vez de anticorpos conjugados com fluoróforos que permitem um maior número de canais e produzem mínimo spillover 1,2. A MCM apresenta vantagens e desvantagens em relação à análise do ciclo celular em comparação à citometria de fluxo. Uma grande vantagem da MCM é que o grande número de parâmetros permite a medição simultânea do estado do ciclo celular em um grande número de tipos de células T imunofenotipicamente distintos em amostras altamente heterogêneas. A MCM tem sido usada com sucesso para medir o estado do ciclo celular durante a hematopoese normal na medula óssea humana3 e modelos murinos transgênicos de deficiência de telomerase4. A análise do estado do ciclo celular na leucemia mieloide aguda (LMA) mostrou que o ciclo celular correlacionou-se com respostas conhecidas a terapias clínicas, fornecendo uma visão in vivo das características funcionais que podem informar as seleções terapêuticas5. Uma segunda vantagem da análise do ciclo celular por citometria de massa é a capacidade de medir um grande número de outros marcadores funcionais que podem ser correlacionados com o estado do ciclo celular. Trabalhos recentes têm sido capazes de correlacionar a síntese de proteínas e RNA com o estado do ciclo celular através do uso de anticorpos marcados com IdU e metal para BRU e rRNA6. Esse tipo de análise altamente paramétrica medindo o estado do ciclo celular em várias populações em um continuum de diferenciação seria quase impossível com a tecnologia atual de citometria de fluxo. A principal desvantagem da MCM é a falta de colorações de DNA ou RNA comparáveis às usadas em citometria de fluxo fluorescente (por exemplo, DAPI, Hoechst, Pyronin Y, etc.). Corantes fluorescentes podem fornecer medidas relativamente precisas do conteúdo de DNA e RNA, mas essa precisão só é possível devido às mudanças nas propriedades fluorescentes desses corantes que ocorrem na intercalação entre bases nucleotídicas. A análise de MCM é, portanto, incapaz de medir o conteúdo de DNA ou RNA com precisão semelhante. Em vez disso, a análise do ciclo celular por citometria de massa baseia-se em medições de proteínas relacionadas ao estado do ciclo celular, como ciclina B1, proteína de retinoblastoma fosforilado (pRb) e histona H3 fosforilada (pHH3) combinadas com a medição direta do átomo de iodo da incorporação de IdU em células de fase S. Essas duas abordagens de medição produzem resultados altamente semelhantes durante a proliferação celular normal, mas podem ser potencialmente discordantes quando a progressão do ciclo celular é interrompida.
A medição do número de células em cada fase do ciclo celular é importante para entender o desenvolvimento normal do ciclo celular, bem como a interrupção do ciclo celular, que é comumente observada em cânceres e doenças imunológicas. MCM fornece medição confiável de fatores extracelulares e intracelulares usando anticorpos marcados com metal; no entanto, a medição da fase S foi limitada, pois o intercalador de DNA baseado em irídio foi incapaz de diferenciar entre DNA 2N e 4N. Para definir as fases do ciclo celular, Behbehani desenvolveu um método que utiliza IdU com massa de 127, que se enquadra na faixa do citômetro de massa e permite a medição direta de células na fase S3. Essa medição direta contorna a necessidade de anticorpos secundários ou o uso de agentes desnaturantes do DNA, como ácido ou DNase. Em conjunto com marcadores de ciclagem intracelular, permite alta resolução da distribuição do ciclo celular em modelos experimentais.
Este protocolo adapta medidas do ciclo celular de protocolos comuns de citometria de fluxo para MCM. Nossos métodos fornecem uma maneira conveniente e simples de incluir parâmetros do ciclo celular. A incorporação de amostras in vitro por IdU requer apenas 10 a 15 minutos de incubação a 37 °C, o que é mais curto do que a maioria dos protocolos de coloração de BrdU, que recomendam tempos de incubação de várias horas 3,7. As amostras incorporadas de IdU e BrdU podem ser fixadas usando um estabilizador proteômico e, em seguida, armazenadas por algum tempo em um freezer de -80 °C. Isso permite que um grande número de amostras coradas com IdU seja arquivado para análise em lote sem redução na qualidade da amostra.
Protocolo
1. Preparação das existências de IdU
- Dissolver 5-iodo-2'-desoxiuridina (IdU) em DMSO até uma concentração de 50 mM. Filtro estéril, alíquota em tubos de 10-50 μL e conservar a -80 °C
- Retire o IdU do congelador e descongele à temperatura ambiente. Diluir IdU em RPMI-1640 para fazer uma solução de trabalho a uma concentração final de 1 mM. Pipetar para cima e para baixo ou vórtice para misturar.
- Normalmente, diluir o IdU concentrado no meio em que as células estão sendo cultivadas (por exemplo, DMEM, IMDM, etc.) ou diluí-lo em PBS para adição direta a amostras de sangue periférico ou aspirado de medula óssea. Esta etapa de pré-diluição facilita a mistura do DMSO com o meio aquoso das células de interesse.
NOTA: A concentração final de IdU durante a incubação deve ser de 10 μM; uma solução de 1 mM pode ser adicionada na proporção de 10 μL para cada 1 mL de meio.
- Normalmente, diluir o IdU concentrado no meio em que as células estão sendo cultivadas (por exemplo, DMEM, IMDM, etc.) ou diluí-lo em PBS para adição direta a amostras de sangue periférico ou aspirado de medula óssea. Esta etapa de pré-diluição facilita a mistura do DMSO com o meio aquoso das células de interesse.
2. Incubação de IdU e preservação da amostra
- Manter as amostras numa incubadora humidificada a 37 °C. Retire a amostra da incubadora e mova-a para uma capela de biossegurança.
- Adicionar 10 μL de 1 mM IdU a cada 1 mL de amostra.
- Para uma placa de 6 poços, adicionar 30 μL de 1 mM IdU aos 3 mL de meio de cultura em cada poço. A IdU também pode ser usada diretamente em aspirados de medula óssea, bem como em estudos murinos4.
- Colocar a amostra de volta na incubadora a 37 °C durante 10-15 min. Manter as células sob as condições ideais de crescimento de interesse durante a exposição ao IdU, a fim de obter a medição mais precisa da fase S.
- Após a incubação do IdU, remova a amostra e transfira para um tubo cônico.
- Gire a amostra a 400 x g durante 10 minutos à temperatura ambiente.
- Aspirar o sobrenadante e ressuspender em 200 μL de PBS.
- Se necessário, realizar uma coloração viva/morta (usando cisplatina) nesta etapa para marcar as células mortas antes da fixação e congelamento.
NOTA: A coloração de ródio vivo/morto não tem um bom desempenho após a permeabilização com metanol, por isso não é recomendada para uso em análises do ciclo celular.
- Se necessário, realizar uma coloração viva/morta (usando cisplatina) nesta etapa para marcar as células mortas antes da fixação e congelamento.
- Adicionar 18,75 μL de paraformaldeído (PFA) a 16% ao PBS para uma concentração final de 1,5% de PFA. Incubar à temperatura ambiente durante 10 min. Gire a amostra para baixo a 400 x g durante 10 minutos à temperatura ambiente.
- Aspirar a solução de PBS/PFA e ressuspender a amostra em 500 μL de Cell Staining Media (CSM; 1x PBS com BSA a 0,5% e azida sódica a 0,02%) + DMSO a 10% antes do congelamento.
- Se estiver usando Estabilizante Proteômico comercial, adicionar 280 μL de Estabilizante Proteômico à amostra ressuspensa em 200 μL de PBS (1:1.4). Incubar as amostras à temperatura ambiente durante 10 min e, em seguida, colocar directamente na temperatura -80 °C.
NOTA: Foi demonstrado que o IdU incorpora eficazmente dentro de 10-15 min de incubação a 37 °C. Incubações de IdU por mais de 10-15 min reduzirão progressivamente a resolução das populações das fases S e G2, à medida que as células marcadas com IdU saem da fase S e progridem para a fase G2 ou M. Também observamos que a incubação a longo prazo com IdU pode causar morte celular e artefatos do ciclo celular. O processamento celular subsequente e a coloração de anticorpos após a incorporação de IdU são suficientes para lavar o IdU residual que não foi incorporado às células da fase S. Nós não observamos fundo significativo de iodo ao usar o protocolo in vitro descrito aqui; no entanto, muito raramente observamos contaminação por iodo em amostras clínicas. Isso pode ocorrer a partir de procedimentos médicos, como contraste iodado em uma tomografia computadorizada, ou de medicamentos contendo iodo. Se grandes quantidades de fundo de IdU forem observadas, a amostra não deve ser executada para evitar danos ao detector do citômetro de massa.
- Se estiver usando Estabilizante Proteômico comercial, adicionar 280 μL de Estabilizante Proteômico à amostra ressuspensa em 200 μL de PBS (1:1.4). Incubar as amostras à temperatura ambiente durante 10 min e, em seguida, colocar directamente na temperatura -80 °C.
3. Coloração de amostras para citometria de massa
- Retire as amostras dos -80 °C e deixe descongelar antes da coloração superficial.
- Se utilizar o método de fixação SmartTube, descongelar as amostras a 0-4 °C para evitar fixação adicional à medida que as amostras aquecem.
- Depois que as amostras forem descongeladas, transfira aproximadamente 1-2 milhões de células para um tubo FACS de 5 mL.
- Centrifugar o tubo FACS a 600 x g por 5 min e encher o tubo FACS com meio corante celular (CSM) para lavar as células. Repita mais uma vez.
- Se as células são conhecidas por ficarem juntas, adicione 400 U/mL de heparina às lavagens CSM para evitar o contato célula a célula, mas isso não é estritamente necessário.
- Incubar as células com agente bloqueador de FC, 5 μL do agente por 100 μL de células, por 10 min à temperatura ambiente.
- Prepare uma mistura de anticorpos que manchará a superfície, ou porção extracelular, das células. A mistura total de coloração será de 100 μL por 1-2 milhões de células em cada teste. A mistura de coloração será balanceada de acordo com CSM e heparina no coquetel e tubo FACS.
NOTA: A adição de CSM nesta etapa também reduzirá artefatos de coloração inespecíficos8. Essa mistura de coloração é inteiramente dependente de alvos de interesse e fenótipo de superfície (por exemplo, um estudo envolvendo células T usará uma mistura de superfície de CD45, CD3, CD4, CD8, etc.). Um protocolo detalhado de processamento e coloração das amostras pode ser encontrado em Behbehani e McCarthy et al.9,10. - Adicione a mistura de coloração superficial às células e incube à temperatura ambiente com agitação contínua por 30-60 min.
- Após a coloração, preencher o tubo FACS com CSM e girar para baixo a 600 x g por 5 min.
- Lavar mais duas vezes com CSM, girando a amostra a 600 x g por 5 min e aspirando cada lavagem CSM.
- Corrigir os anticorpos extracelulares adicionando 1 mL de PBS com MSC a 10% e PFA a 1,5%.
- Encha o tubo FACS contendo a mistura PBS/CSM/PFA com CSM. Gire para baixo a 600 x g por 5 min e aspirar o sobrenadante.
- Adicionar metanol a -20 °C.
- Vórtice a amostra por 1-2 min para obter uma suspensão de célula única e verifique se todos os aglomerados celulares foram ressuspensos.
- Enquanto a amostra estiver vomitando lentamente, adicione rapidamente 1 mL de metanol gelado usando uma pipeta de 1.000 μL com uma ponta de filtro.
- Segure o tubo FACs até a luz e certifique-se de que não haja aglomerações visíveis; A nebulosidade é esperada. Quaisquer aglomerações tornarão a amostra inutilizável para análises subsequentes de MCM.
- Conservar a amostra a -20 °C durante 10-20 min.
- Prepare a mistura de coloração intracelular durante este tempo. A mistura corante intracelular será dependente dos alvos de interesse. Para análise do ciclo celular, inclua CyclinB1, pRb, Ki67 e pHH3 nesta mistura de coloração, mas outros marcadores intracelulares podem ser adicionados conforme necessário.
- Após 10-20 min a -20 °C, retirar a amostra, adicionar 1,5 mL de PBS e preencher o restante com CSM.
- Centrifugar a amostra 600 x g por 5 min e aspirar o sobrenadante.
- Lavar mais duas vezes com CSM, girando a amostra para baixo a 600 x g durante 5 min e aspirando o sobrenadante de cada vez.
- Após a última lavagem CSM, centrifugar a amostra e deixar um volume residual de aproximadamente 50 μL.
- Adicione a mistura de anticorpos preparada (normalmente adicione 50 μL de cocktail de coloração de anticorpos para atingir um volume de coloração final de 100 μL) à amostra e incube numa plataforma de agitação durante 30 a 60 minutos à temperatura ambiente.
- Após a coloração adicionar CSM e centrifugar a 600 x g por 5 min.
- Aspirar o MSC, lavar novamente com MSC, girando a 600 x g por 5 min, aspirando o MSC, em seguida, adicionar PBS.
- Após a conclusão da coloração intracelular, coloque as células em uma solução intercaladora que fixa os anticorpos nas células e cora o DNA de cada célula para permitir a identificação. A solução intercaladora contém intercalador de irídio não isotopicamente puro (pentametilciclopentadienil-Ir(III)-dipiridofenazina) adicionado da solução-mãe do fabricante a uma concentração de 500 μM. Diluir o estoque de Irídio 1:4000 em uma solução de PBS e PFA a 1,5%. Adicionar a solução intercaladora de irídio a 100-200 μL por milhão de células para manchar uniformemente e evitar a sobrecoloração.
NOTA: O irídio nesta solução intercalador destina-se a identificar células para gating singlete, não deve ser usado para manchas vivas/mortas. Se as colorações vivas/mortas forem desejadas, elas precisam ser realizadas antes da fixação, como observado acima e em McCarthy et al.9. - Conservar as amostras em solução intercaladora num frigorífico a 4 °C durante um período máximo de duas semanas antes da aquisição das amostras no CyTOF.
4. Operação do citômetro de massa
NOTA: A operação de citometria de massa pode ser específica da máquina. É sempre aconselhável verificar o manual do usuário do CyTOF antes da operação. Além disso, existem atualmente dois artigos do JoVE que tratam de partida e manutenção de máquinas 9,11.
- Verifique se há entupimentos, rachaduras e outras irregularidades no nebulizador antes de acionar o citômetro de massa.
- Conecte o nebulizador ao citômetro e inicie o procedimento de aquecimento. Não inicie o citômetro de massa sem o nebulizador no lugar.
- Passe água através das linhas de amostra assim que o citômetro de massa terminar de aquecer. A câmara de pulverização precisa atingir aproximadamente 200 °C antes de realizar o ajuste ou a análise da amostra.
- Corra água por 5-10 min. Após 5-10 min, carregue a solução de ajuste e selecione o gerenciador de ajuste. A solução de afinação é uma solução contendo concentrações fixas de metais e usada para otimizar o citômetro de massa antes da aquisição da amostra
- No gerenciador de ajuste, selecione Visualizar quando a solução de ajuste atingir um registro de acertos em estado estacionário para iniciar o processo de ajuste automatizado.
- Uma vez terminado o ajuste, carregue o amostrador com água e permita que a água percorra as linhas de amostragem durante o processamento da amostra. O protocolo detalhado para a operação e sintonia diária do citômetro pode ser encontrado em Leipold11.
- Ocasionalmente, o ajuste automatizado não será ajustado ao desempenho ideal da máquina. Repita o procedimento de ajuste para corrigir isso.
- Lavar a amostra com CSM uma vez e com água pura deionizada duas vezes antes da aquisição da amostra. A lavagem com água é importante para remover o sal residual do PBS/CSM.
- Verifique a sensibilidade e o fluxo da amostra usando esferas de equilíbrio fornecidas pelo fabricante, esferas de poliestireno carregadas com concentrações de metal conhecidas.
- Altere o modo de aquisição de ajuste para o modo de captura de eventos. Defina o prazo para interromper a aquisição em 120 s. Aguarde 45 s antes de selecionar Gravar.
- O citômetro de massa interromperá a aquisição da amostra automaticamente após 120 segundos. Use o visualizador de gráfico de chuva para verificar a intensidade de Eu151 e Eu153.
- Diluir esferas de equilíbrio em água pura deionizada na proporção de 1:20.
- Antes da aquisição da amostra, verifique o gerenciador de experimentos. Use o gerenciador de experimentos para atribuir nomes a canais e adicionar canais a serem gravados.
- Verifique se o canal 127-I foi adicionado se estiver usando IdU.
- Observe que é essencial ajustar o citômetro de massa para medir os parâmetros necessários (por exemplo, IdU) antes da aquisição da amostra. Se os canais não forem selecionados com antecedência, os dados não serão coletados de nenhum canal não selecionado e não poderão ser recuperados.
- Diluir as células para uma concentração de aproximadamente 1-2 x 106/mL usando a mistura de água deionizada pura 1:20 e cordões de equilíbrio. Passe as células através do tubo FACS com tampa do filtro para remover quaisquer aglomerados residuais.
- Carregue a amostra e altere o tempo de aquisição.
- Pressione Visualizar e aguarde a estabilização da contagem de eventos por segundo.
- Não execute eventos acima de 400 eventos por segundo, isso levará a quantidades significativas de duplicações e detritos. Normalmente, coletamos pelo menos 20.000 a 50.000 eventos celulares, mas o número ideal dependerá do desenho experimental. A coloração de até 2 milhões de células normalmente produzirá de 300.000 a 400.000 eventos celulares. Observe que nem todos os eventos serão células (haverá detritos e eventos de contas incluídos na contagem de eventos).
- Uma vez que a aquisição da amostra é feita carregar uma solução de lavagem, iniciar a indução da amostra e executar por 5-10 minutos. Após 5-10 minutos, pare a indução da amostra e corra água por 10-20 minutos. A solução de lavagem é uma solução fraca de ácido fluorídrico projetada para retirar o metal residual das linhas de amostra.
- Desligue o citômetro de massa e remova o nebulizador. O nebulizador estará quente, tome cuidado durante o manuseio.
5. Análise dos dados
- A fim de remover contas e também corrigir a deriva de sinal durante a aquisição da amostra, normalize os arquivos FCS usando o software Fluidigm ou o aplicativo desenvolvido por Finck12.
- Carregue o FCS no Cytobank ou em outro software de análise de citometria de fluxo. Os arquivos FCS podem ser usados em qualquer software compatível, para os fins deste protocolo todo o gating e análise adicional foi feita no Cytobank13.
- Antes que as portas do ciclo celular possam ser desenhadas, exclua quaisquer duplos ou detritos celulares da análise a jusante, isso pode ser feito usando o gráfico biaxial de Comprimento do Evento vs 191-Ir (Figura 1a). As células formarão uma população distinta ebrilhante que pode ser usada para excluir duplos e detritos. Esta é a porta única. Esse método de gating normalmente remove cerca de 50-60% dos eventos de célula dupla, portanto, estratégias adicionais podem ser necessárias para remover eventos de célula dupla restantes.
- Altere a escala de comprimento do evento (mínimo e máximo) para fazer com que as células pareçam mais proeminentes para ajudar no gating singlete.
- Além disso, remova duplicatas e detritos usando parâmetros Gaussianos, Residual e Offset. Um Residual mais alto com menor Offset também são detritos e duplos, e o confinamento em torno dessa população pode remover ainda mais duplos e detritos (Figura 1b,c).
- Gating da fase S – A fase S é a porta mais fácil de desenhar, mas também a mais importante. Desenhe esta porta usando um gráfico biaxial de IdU vs pRb, Ki67 ou ciclina B1. As células IdU+ da fase S formarão uma população distinta ao observar esses gráficos biaxiais (Figura 2b).
- G0/G1-fase, G2/M-fase gating – Estabeleça as portas de fase G0/G1 e G2/M no gráfico IdU vs CyclinB1 e o uso da incorporação de IdU é crucial para estabelecer o limite entre as portas de fase G0/G1 e G2/M. A fase G0/G1 será a ciclina B1baixa/IdU- e a fase G2/M será a ciclinaB1 alta/IdU-. A boa coloração de ciclina B1 mostrará uma população natural entre as populações G0-G1 e G2-M; no entanto, isso irá variar entre amostras e tipos celulares sob condições experimentais. Em condições experimentais onde a distribuição do ciclo celular pode ser afetada e há menor separação entre a ciclina B1 G0/G1-fase e G2/M-fase utilizando a fase S permitirá o gating consistente para tipos celulares particulares em cada experimento específico. Este método é detalhado abaixo.
- Plotar apenas as células da fase S na ciclina B1 vs IdU para ajudar a estabelecer a separação entre a fase G0/G1 e a fase G2/M. Desenhe um portão naalta população de ciclina B1 e ajuste até que aproximadamente os 5% superiores da população da fase S estejam dentro do portão (Figura 2c). Este estabelece o ponto de quebra entre as comportas de fase G0/G1 e G2/M (Figura 2e,f). A população ativa será alterada para a população de interesse e a parcela residente dentro do portão anterior será a população da fase G2/M, enquanto o restante será a população da fase G0/G1.
- G0-phase gating – Estabeleça a fase G0 no gráfico pRb vs IdU. A fase G0 será representada por uma população pRbbaixa/IdU- . A população ciclável ativa terá alta expressão de incorporação de pRb e IdU, a porta de fase G0 pode ser desenhada neste limite, pois tipicamente se expressa em duas populações distintas (Figura 2g,i).
- Defina a fase G0 fazendo da população da fase S desenhada anteriormente (Figura 2b) a população ativa e desenhando um portão incorporando os 90-100% superiores da populaçãoalta de pRb. Esta é a população ciclável pRb+ (Figura 2i), a populaçãopRb baixa fora deste portão é a população G0 (Figura 2j).
- Se o pRb não estiver disponível ou não puder ser registrado, use Ki67 vs IdU para estabelecer a população da fase G0. Desenhando um portão representando a maioria da população da fase S e usando esse portão como limite para o Ki67 vs IdU, o restante da população será a fase G0 (Figura 3a).
- Gating de fase M – Estabelecer a fase M no gráfico biaxial IdU vs pH3. A fase M representa uma fração muito pequena de células e está fechada na populaçãode pH 3 alto (Figura 2d).
- A incorporação de IdU falhou ou não foi possível – Se a IdU não estiver disponível, defina a ciclagem de células e não as frações de ciclagem usando Ki67 e pRb. Ki67 e pRb, em condições normais, formam duas populações distintas: Ki67 alto/pRbalto e Ki67 baixo/pRbbaixo. A população dupla positiva representa a população ciclista ativa, correlacionando-se com as fases G1, S, G2 e M. A dupla população baixa representa a população não ciclante, correlacionando-se com a fase G0 (Figura 3b).
NOTA: Não é possível delinear cada fase individual usando o Ki67 vs pRb, mas os efeitos experimentais sobre as populações relativas ciclando/não ciclando podem ser determinados. - Análise do ciclo celular – Uma vez que as comportas tenham sido estabelecidas, exporte os valores numéricos das portas para análise posterior. As porcentagens em cada ciclo podem ser obtidas subtraindo-se as populações individuais das populações combinadas. A fase G0, desenhada na porcentagem de porta deIdU baixa de pRb pode ser subtraída da fase G0/G1, desenhada na Ciclina B1baixaIdU neg, para encontrar a porcentagem de fase G1. Da mesma forma, a porcentagem de fase G2 é derivada da subtração da porta de fase M da porta de fase G2/M. Isso gerará valores numéricos para cada fase do ciclo celular individual; G0, G1, S, G2 e M. Os valores numéricos gerados para cada fase do ciclo celular individual podem ser usados para análises posteriores, como gráficos e análises estatísticas.
Resultados
Utilizando células HL-60 e um aspirado de medula óssea humana é possível mostrar como as condições experimentais podem afetar a distribuição e análise do ciclo celular. Primeiro, a estratégia de gating deve ser estabelecida para demonstrar como as fases do ciclo celular são derivadas. Na Figura 1 mostramos o estabelecimento da porta singlete, que é importante na separação de restos celulares e duplos, estabelecendo uma população de células únicas. Para linhagens celulares, a porta singlete é tudo o que é necessário para passar para a análise do ciclo celular (Figura 2a). Para amostras humanas, populações imunofenotípicas normalmente precisam ser estabelecidas antes da análise do ciclo celular, uma vez que os limites exatos de cada porta do ciclo celular podem variar entre diferentes tipos celulares. Uma vez que as populações tenham sido estabelecidas (geralmente por meio de marcadores de superfície que definem essa população), as portas do ciclo celular precisarão então ser estabelecidas. A Figura 2b demonstra o estabelecimento da fase S no gráfico biaxial IdU vs CyclinB1. Este gráfico também é usado para estabelecer o limite da porta de fase G2/M (Figura 2f). Uma vez estabelecida a fase G2/M, o restante é a porta da fase G0/G1 (Figura 2f). O IdU vs pRb é usado para estabelecer a população cicladora de pRb+ primeiro, estabelecendo uma porta em células incorporadoras de IdU (Figura 2g,i). Apopulação neg pRb+/IdU fora dessa porta é a fase G0 (Figura 2j). A fase M é estabelecida na IdU vs pHH3, onde as células da fase M expressam altos níveis de pHH3 e não exibem incorporação de IdU (Figura 2k). Caso o pRb não seja incluído, a fase G0 pode ser replicada usando Ki67 de forma semelhante ao método descrito acima (Figura 3a). Se a incorporação de IdU falhou ou não foi realizada, ainda é possível determinar frações relativas de ciclagem usando Ki67 e pRb. Usando o Ki67 e pRb biaxial duas populações distintas, uma pRb+/Ki67+ dupla positiva e uma pRb baixa/Ki67baixa população. A população dupla positiva representa células em ciclo, enquanto a baixa representa células não em ciclo (Figura 3b). Usando células incorporadoras de IdU e célulasbaixas de pRb sem incorporação de IdU, mostramos que a fase S está primariamente na população pRb+/Ki67+, enquanto a fase G0 está primariamente na população pRb baixa/Ki67baixa.
A análise do ciclo celular depende de boa técnica experimental, especialmente durante a etapa de incubação do IdU. Embora a incorporação de IdU seja flexível (sendo aplicável em cultura celular, aspirados de medula óssea e até mesmo em estudos murinos), é necessário realizar a incorporação e fixação de IdU sem interromper o estado de interesse experimental do ciclo celular. A marcação de IdU e, portanto, a análise do ciclo celular a jusante podem ser significativamente afetadas pelo tempo e temperatura, como indicado pela Figura 4. As células que permanecerem por muito tempo em recipientes fechados ou que possam ser encontradas no envio de amostras ou no transporte de amostras entre locais, terão fração de fase S reduzida e não serão precisas para análise do ciclo celular (Figura 4a). No entanto, períodos curtos de tempo, aqueles com menos de uma hora no total, terão distribuição normal do ciclo celular, indicando que o transporte rápido pode não prejudicar a análise do ciclo celular (Figura 4b). Outro importante modificador é a criopreservação, que é rotineiramente utilizada na maioria dos laboratórios. Ao examinar o estado do ciclo celular em células criopreservadas, um longo período de equilíbrio pode ser necessário antes que as células retornem à ciclagem celular ativa, o que ainda pode não refletir o estado do ciclo celular pré-cirepreservação (Figura 4c).
Amostras humanas primárias são frequentemente compósitos de vários tipos de células diferentes, esses diferentes tipos de células podem ter diferentes sensibilidades ao processamento, levando a diferentes ciclos celulares. Em dois aspirados de medula óssea marcados com IdU imediatamente, armazenados por 30 minutos antes da marcação de IdU ou armazenados criogenicamente após a separação de Ficoll há diferenças entre cada amostra e população (Figura 5a,b). Duas populações imunofenotípicas foram examinadas para diferenças na incorporação de IdU; Células T (CD45 alto/CD3alto) e monoblastos (CD33+, HLADR+, CD11bbaixo, CD14neg). Com a combinação correta de marcas de superfície, é possível examinar outras populações imunofenotípicas. Na medula #2 houve um efeito de ativação de células T perceptível após 30 minutos de armazenamento, que não foi visto nos monoblastos do mesmo paciente (Figura 5b). Assim como as células cultivadas, houve mudanças perceptíveis na marcação de IdU após o armazenamento criogênico, que também foi dependente da população. A medula #1 apresentou redução na população de células T, mas aumento nas frações marcadas com IdU de monoblastos quando comparada à linha de base (Figura 5 a,b), a medula #2 mostrou redução tanto em células T quanto em monoblastos quando comparada à linha de base (Figura 5a,b). As células congeladas requerem então um período de incubação notável antes de retornar ao estado normal do ciclo celular e isso pode influenciar estudos que se baseiam na modificação do estado do ciclo celular ou do estado do ciclo celular como uma métrica do efeito da droga ou experimental.
Outro benefício da MCM é a capacidade de discriminar células em parada do ciclo celular ou que tenham distribuição anormal do ciclo celular. Embora os corantes de DNA comumente usados em citometria de fluxo sejam capazes de discriminar entre o conteúdo de DNA de 2N e 4N, eles são muito brilhantes, o que pode complicar muito a medição de outros parâmetros desse laser. IdU, no entanto, leva apenas um canal de massa e tem derramamento mínimo, permitindo que outros marcadores sejam usados na determinação do ciclo celular. As células MOLM13 irradiadas apresentam diminuição da incorporação de IdU e diminuição da fase M quando comparadas às células controle (Figura 6). A interrupção dos pontos de verificação normais do ciclo celular pode alterar o estado aparente do ciclo celular pela MCM. Observando as populações de pH2AX e cPARP nas células não irradiadas, a população de pH2AXbaixa e cPARPbaixa mostra distribuição normal do ciclo celular, enquanto as células que expressam níveis mais altos de pH2AX ou cPARP localizam-se principalmente na fase G0/G1, o que é esperado (Figura 6a). Nas células irradiadas o pH2AX baixo e cPARPbaixo, entretanto, as células estão quase inteiramente localizadas na fase G0, enquanto as células pH2AXalta e cPARPbaixa apresentam fenótipo de parada do ciclo celular com incorporação de IdU e localização para a fase G0/G1 e fase G2 com ausência de fase M. As células altas de pH2AX e cPARP tambémmostram células incorporando algum IdU e localizando-se na fase G0/G1 indicativasde dano radioativo (Figura 6b).
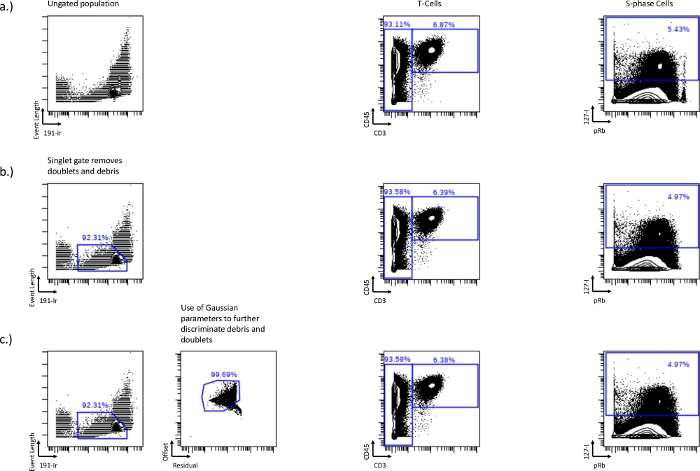
Figura 1: Estabelecimento das portas singlet usando 191-Ir por Comprimento de Evento e também parâmetros Gaussianos, Residual e Offset.
As diferenças nas células T (CD45+/CD3+) e na fase S (IdU+) entre uma amostra ungada (a), um comprimento de evento vs porta singlet 191-Ir (b), ou uma porta singlet combinada com parâmetros gaussianos, residual e offset (c). O gating singlete remove detritos, duplos e contas mostrados na perda da populaçãoalta de pRb no canto direito do biaxial. Esta porta singlet pode ser otimizada ainda mais, incluindo parâmetros gaussianos, como residual e offset, removendo mais detritos. Clique aqui para ver uma versão maior desta figura.
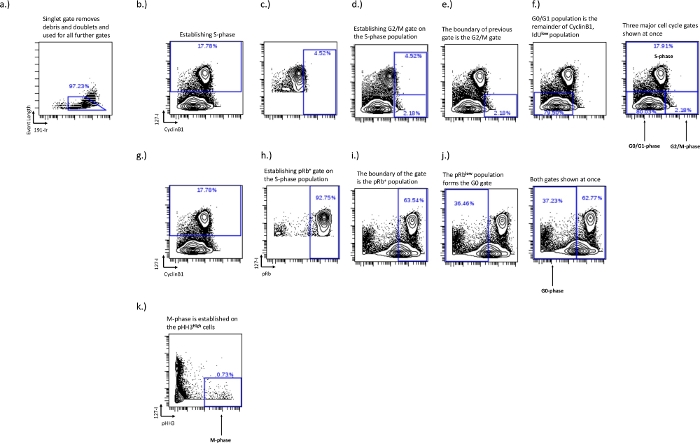
Figura 2: Esquema de gating para estabelecer portas de ciclo celular para as fases G0, G1, S, G2 e M usando IdU, CyclinB1, pRb e pHH3.
O portão único é estabelecido para remover duplos e detritos (a). A fase S deve ser estabelecida (b), uma vez estabelecida a fase S, a população IdU+ pode ser usada para estabelecer o limite da fase G2/M (c,d). O estabelecimento do limite da fase G2/M estabelece os limites da população da fase G0/G1 (f). A população das fases pRb+ e G0 é estabelecida no biaxial IdU vs pRb. As células IdU+ (h) são usadas para estabelecer o limite para a população pRb+ (i). O limite da população pRb+ estabelece o limite da população da fase G0 (j). A fase M é estabelecida em células pHH3+ que são IdU- (k). Clique aqui para ver uma versão maior desta figura.
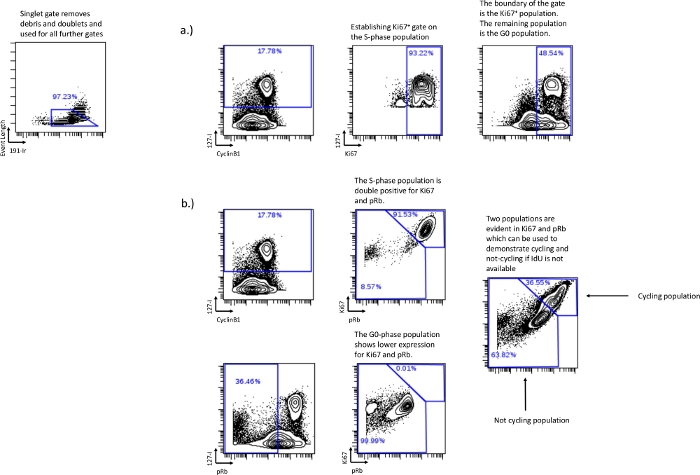
Figura 3: Estabelecimento de portas de ciclo celular sem o uso de pRb ou sem o uso de incorporação de IdU.
O desenho da fase G0 também pode ser feito com o Ki-67 seguindo a mesma estratégia de gating que foi utilizada com o pRb, caso o pRb não seja incluído no experimento (a). Se a incorporação de IdU falhou ou não foi realizada, ainda é possível recuperar as frações relativas de ciclagem celular através do uso de Ki67 e pRb. A expressão dupla positiva de Ki67 e pRb é correlacionada com células em ciclo, como evidenciado pela demonstração de que as células IdU+ são encontradas principalmente na população de duplo-positivos (b). A população baixa de Ki67 e pRb correlaciona-se com a população cicladora de fase G0 ou não demonstradapor células neg de pRb baixo/IdU encontradas na população de Ki67 e pRbbaixo. Este método não pode discriminar fases individuais do ciclo celular, mas ainda pode ser usado para determinar frações relativas de ciclagem em condições experimentais. Clique aqui para ver uma versão maior desta figura.

Figura 4: Figuras representativas do efeito de diferentes condições de armazenamento na distribuição do ciclo celular das células HL-60.
As células foram incubadas por uma hora seguida de uma hora de repouso nas condições de temperatura indicada em tubos selados (a). Há efeitos perceptíveis na distribuição do ciclo celular quando comparado ao controle. As células HL60 foram mantidas em tubos selados à temperatura ambiente por 30 minutos, situação que pode ocorrer em ambiente clínico, antes da incorporação de IdU (b). Os tubos selados mantidos à temperatura ambiente por 30 minutos não apresentaram diferenças apreciáveis no ciclo celular. O efeito do armazenamento criogênico foi investigado sobre o ciclo celular em células HL60, onde uma amostra foi coletada antes do armazenamento criogênico e uma amostra após uma hora de repouso após uma semana no armazenamento criogênico (c). Uma hora após o descongelamento, a distribuição do ciclo celular é afetada e a distribuição do ciclo celular não retorna ao normal até aproximadamente uma semana após o descongelamento. Clique aqui para ver uma versão maior desta figura.

Figura 5: O processamento pode ter um efeito nas amostras dos pacientes e imagens representativas são mostradas entre dois pacientes diferentes mostrando as (a) células T (CD45 alto/CD3alto) e (b) monoblastos (CD33+, HLADR+, CD11bbaixo, CD14neg).
Entre a medula um e a medula dois, fica claro que na medula 2 houve um efeito de ativação de células T durante os 30 minutos de repouso, enquanto na medula não houve tal efeito. A medula um mostrou possível efeito de ativação na população de monoblastos após 30 minutos, enquanto a medula dois não. Em ambas as medulas, no entanto, ficou claro que o armazenamento criogênico impactou a distribuição do ciclo celular, independentemente do tipo celular. Clique aqui para ver uma versão maior desta figura.

Figura 6: As células MOLM13 foram deixadas como controle ou irradiadas com irradiador de raios X a 10Gy.
As células MOLM13 mostram a distribuição do ciclo celular em quatro populações diferentes de expressão de pH2AX e cPARP. As células controle apresentam coloração mínima de pH2AX e cPARP, com características normais do ciclo celular sendo mostradasna população baixa de pH2AX e baixa de cPARP (a). Enquanto as células irradiadas apresentam uma distribuição anormal do ciclo celular, com a maioria das células cicladoras localizando-se no pH2AX alto e cPARPalto, indicando ruptura do ciclo celular (b). As células não danificadas, com pH2AX baixo e cPARPbaixo, apresentam falta de características de ciclagem são encontradas principalmente na fase G0. Sem esses marcadores, essas células apareceriam como células 4N e 2N em citometria de fluxo normal, possivelmente confundindo a análise do ciclo celular a jusante. Clique aqui para ver uma versão maior desta figura.
Discussão
Os exemplos aqui apresentados demonstram como usar uma plataforma MCM para analisar a distribuição do ciclo celular. Também foi demonstrado que a análise do ciclo celular é sensível a condições experimentais como tempo e temperatura, o que é uma consideração importante que os pesquisadores devem ter ao considerar a MCM para sua análise do ciclo celular14. As amostras deixadas em armazenamento por um curto período de tempo, não superior a uma hora, terão incorporação de IdU comparável ao seu estado normal. Amostras em sistema fechado por longos períodos de tempo, aproximadamente 2 horas, terão incorporação de IdU reduzida, no entanto, as frações relativas cicladoras e não cicladoras não serão alteradas permitindo a análise do ciclo celular grosso. O armazenamento criogênico e o subsequente descongelamento interrompem a distribuição normal do ciclo celular por um período significativo de tempo. Observou-se anteriormente que o armazenamento criogênico interrompe a distribuição de proteínas e RNAs, mas só recentemente foi demonstrado que interrompe o ciclo celular14,15,16,17. Em conjunto, estes indicam que, se longos tempos de armazenamento são antecipados ou preservação criogênica de amostras, seria melhor corar as células com IdU antes do armazenamento ou armazenamento criogênico, fixar as células e armazená-las até que a análise possa ser feita. Como a análise do ciclo celular por MCM não requer células vivas, isso permitiria que os pesquisadores depositassem amostras preciosas e realizassem análises precisas do ciclo celular a jusante.
A análise do ciclo celular por MCM é um sistema robusto capaz de interrogar em profundidade modelos experimentais. Devido à alta contagem de parâmetros da MCM, aproximadamente 40-50 canais de massa, a análise do ciclo celular pode ser combinada com outros marcadores intracelulares ou extracelulares, ignorando a necessidade de classificação de células com base no imunofenótipo, o que pode causar efeitos significativos no ciclo celular. A natureza de alta parametrização do MCM presta-se a examinar os efeitos em aplicações de mapeamento de alta dimensão, como SPADE e viSNE. Enquanto SPADE e viSNE são tipicamente usados para definir populações imunofenotípicas que podem então ser examinadas para alterações do ciclo celular, também seria possível mapear marcadores do ciclo celular. Dependendo das condições experimentais, o mapeamento de marcadores do ciclo celular em um espaço de alta dimensão pode mostrar correlação do ciclo celular com o efeito da droga ou quais populações imunofenotípicas podem estar localizando para cada estado do ciclo 3,5. Enquanto a MCM pode ser limitada pela falta de corantes fluorescentes de ligação ao DNA, isso é compensado pela incorporação direta de IdU durante as células da fase S e proteínas do ciclo celular intracelular podem ser usadas para determinar o estado do ciclo celular. Essas proteínas do ciclo celular também podem ajudar a discriminar entre os estágios de parada do ciclo celular que, de outra forma, apareceriam como 3N ou 4N no fluxo tradicional usando corantes de ligação ao DNA. Tais sistemas altamente paramétricos não estão isentos de desvantagens, no entanto, e é sensível a interrupções no ciclo celular. Nós mostramos que longos tempos de armazenamento e armazenamento criogênico podem afetar significativamente a distribuição do ciclo celular. Isso é especialmente importante quando se tenta racionalizar os efeitos experimentais de drogas que podem afetar a distribuição do ciclo celular. O tratamento de drogas projetadas para afetar o ciclo celular em amostras primárias congeladas pode fornecer dados falsos sobre o efeito do ciclo celular quando usado imediatamente após o descongelamento do armazenamento criogênico. MCM é uma tecnologia versátil para análise de ciclo celular que pode ser aplicada a uma série de modelos experimentais e é especialmente adequada para perfis profundos de sistemas heterogêneos. Como acontece com outros métodos altamente paramétricos, é necessário ter um experimento cuidadosamente projetado com considerações apropriadas sobre como o processamento e os efeitos experimentais afetarão a análise do ciclo celular.
Divulgações
Dr. Behbehani recebe apoio de viagem da Fluidigm. A Fluidigm também adquiriu reagentes e materiais para uso em laboratório.
Agradecimentos
Os autores gostariam de agradecer os esforços de Palak Sekhri, Hussam Alkhalaileh, Hsiaochi Chang e Justin Lyeberger por seu apoio experimental. Este trabalho foi apoiado pelo Programa de Bolsas Pelotonia. Quaisquer opiniões, descobertas e conclusões expressas neste material são do(s) autor(es) e não refletem necessariamente as do Programa de Bolsas Pelotonia."
Materiais
| Name | Company | Catalog Number | Comments |
| Bovine Serum Albumin (BSA) | Sigma | A3059 | Component of CSM |
| Centrifuge | Thermo Scientific | 75-217-420 | Sample centrifugation |
| Cleaved-PARP (D214) | BD Biosciences | F21-852 | Identification of apoptotic cells |
| Cyclin B1 | BD Biosciences | GNS-1 | G2 Resolution |
| Dimethylsulfoxide (DMSO) | Sigma | D2650 | Cryopreservative |
| EQ Four Element Calibration Beads | Fluidigm | 201078 | Internal metal standard for CyTOF performance |
| FACS Tube w/ mesh strainer | Corning | 08-771-23 | Cell strainer to remove clumps/debris before CyTOF run |
| Fetal Bovine Serum (FBS) | VWR | 97068-085 | Cell culture growth supplement |
| Helios | Fluidigm | CyTOF System/Platform | |
| Heparin | Sigma | H3393 | Staining additive to prevent non-specific staining |
| IdU (5-Iodo-2′-deoxyuridine) | Sigma | I7125 | Incorporates in S-phase |
| Ki-67 | eBiosciences | SolA15 | Confirmation of G0/G1 |
| MaxPar Multi Label Kit | Fluidigm | 201300 | Metal labeling kit, attaches metals to antibodies |
| Microplate Shaker | Thermo Scientific | 88880023 | Mixing samples during staining |
| Paraformaldehyde (PFA) | Electron Microscopy Services | 15710 | Fixative |
| pentamethylcyclopentadienyl-Ir(III)-dipyridophenazine | Fluidigm | 201192 | Cell identification during CyTOF acquisition |
| p-H2AX (S139) | Millipore | JBW301 | Detection of DNA damage |
| p-HH3 (S28) | Biolegend | HTA28 | M-phase Resolution |
| Phosphate Buffered Saline (PBS) | Gibco | 14190-144 | Wash solution for cell culture and component of fixative solution |
| p-Rb (S807/811) | BD Biosciences | J112906 | G0/G1 Resolution |
| Proteomic Stabilizer | SmartTube Inc | PROT1 | Sample fixative |
| RPMI 1640 | Gibco | 21870-076 | Cell culture growth medium |
| Sodium Azide | Acros Organics | AC447810250 | Component of CSM/Antibody buffer, biocide |
Referências
- Ornatsky, O., Baranov, V. I., Bandura, D. R., Tanner, S. D., Dick, J. Multiple cellular antigen detection by ICP-MS. Journal of Immunological Methods. 308 (1-2), 68-76 (2006).
- Ornatsky, O. I., et al. Study of cell antigens and intracellular DNA by identification of element-containing labels and metallointercalators using inductively coupled plasma mass spectrometry. Analytical Chemistry. 80 (7), 2539-2547 (2008).
- Behbehani, G. K., Bendall, S. C., Clutter, M. R., Fantl, W. J., Nolan, G. P. Single-cell mass cytometry adapted to measurements of the cell cycle. Cytometry A. 81 (7), 552-566 (2012).
- Raval, A., et al. Reversibility of Defective Hematopoiesis Caused by Telomere Shortening in Telomerase Knockout Mice. PLoS One. 10 (7), 0131722 (2015).
- Behbehani, G. K., et al. Mass Cytometric Functional Profiling of Acute Myeloid Leukemia Defines Cell-Cycle and Immunophenotypic Properties That Correlate with Known Responses to Therapy. Cancer Discovery. 5 (9), 988-1003 (2015).
- Kimmey, S. C., Borges, L., Baskar, R., Bendall, S. C. Parallel analysis of tri-molecular biosynthesis with cell identity and function in single cells. Nature Communications. 10 (1), 1185 (2019).
- Rabinovitch, P. S., Kubbies, M., Chen, Y. C., Schindler, D., Hoehn, H. BrdU-Hoechst flow cytometry: a unique tool for quantitative cell cycle analysis. Experimental Cell Research. 174 (2), 309-318 (1988).
- Rahman, A. H., Tordesillas, L., Berin, M. C. Heparin reduces nonspecific eosinophil staining artifacts in mass cytometry experiments. Cytometry A. 89 (6), 601-607 (2016).
- McCarthy, R. L., Duncan, A. D., Barton, M. C. Sample Preparation for Mass Cytometry Analysis. Journal of Visualized Experiments. (122), e54394 (2017).
- Behbehani, G. K. Immunophenotyping by Mass Cytometry. Methods in Molecular Biology. 2032, 31-51 (2019).
- Leipold, M. D., Maecker, H. T. Mass cytometry: protocol for daily tuning and running cell samples on a CyTOF mass cytometer. Journal of Visualized Experiments. (69), e4398 (2012).
- Finck, R., et al. Normalization of mass cytometry data with bead standards. Cytometry A. 83 (5), 483-494 (2013).
- Kotecha, N., Krutzik, P. O., Irish, J. M. Web-based analysis and publication of flow cytometry experiments. Current Protocols in Cytometry. , 17 (2010).
- Devine, R. D., Sekhri, P., Behbehani, G. K. Effect of storage time and temperature on cell cycle analysis by mass cytometry. Cytometry A. 93 (11), 1141-1149 (2018).
- Campos, L., et al. Expression of immunological markers on leukemic cells before and after cryopreservation and thawing. Cryobiology. 25 (1), 18-22 (1988).
- Kadic, E., Moniz, R. J., Huo, Y., Chi, A., Kariv, I. Effect of cryopreservation on delineation of immune cell subpopulations in tumor specimens as determinated by multiparametric single cell mass cytometry analysis. BMC Immunology. 18 (1), 6 (2017).
- Shabihkhani, M., et al. The procurement, storage, and quality assurance of frozen blood and tissue biospecimens in pathology, biorepository, and biobank settings. Clinical Biochemistry. 47 (4-5), 258-266 (2014).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados