Method Article
Utilisation de l’analogue de la pyrimidine, la 5-iod-2′-désoxyuridine (IdU) avec des marqueurs du cycle cellulaire pour établir les phases du cycle cellulaire dans une plateforme de cytométrie de masse
Dans cet article
Résumé
Ce protocole adapte les mesures du cycle cellulaire pour une utilisation dans une plateforme de cytométrie de masse. Grâce aux capacités multiparamétriques de la cytométrie de masse, la mesure directe de l’incorporation d’iode permet l’identification des cellules en phase S, tandis que les marqueurs du cycle intracellulaire permettent de caractériser chaque état du cycle cellulaire dans une gamme de conditions expérimentales.
Résumé
La régulation de la phase du cycle cellulaire est un aspect important de la prolifération cellulaire et de l’homéostasie. La perturbation des mécanismes de régulation régissant le cycle cellulaire est une caractéristique d’un certain nombre de maladies, y compris le cancer. L’étude du cycle cellulaire nécessite la capacité de définir le nombre de cellules dans chaque partie de la progression du cycle cellulaire ainsi que de délimiter clairement chaque phase du cycle cellulaire. L’avènement de la cytométrie de masse (MCM) offre un potentiel énorme pour l’analyse unicellulaire à haut débit grâce à des mesures directes des isotopes élémentaires, et le développement d’une méthode pour mesurer l’état du cycle cellulaire par MCM étend encore l’utilité de MCM. Nous décrivons ici une méthode qui mesure directement la 5-iode-2′-désoxyuridine (IdU), similaire à la 5-bromo-2'-désoxyuridine (BrdU), dans un système MCM. L’utilisation de ce MCM basé sur IdU offre plusieurs avantages. Tout d’abord, l’IdU est rapidement incorporé dans l’ADN au cours de sa synthèse, ce qui permet une mesure fiable des cellules en phase S avec des incubations aussi courtes que 10-15 minutes. Deuxièmement, l’IdU est mesurée sans avoir besoin d’anticorps secondaires ou de dégradation de l’ADN. Troisièmement, la coloration de l’IdU peut être facilement combinée avec la mesure de la cycline B1, de la protéine du rétinoblastome phosphorylé (pRb) et de l’histone phosphorylée H3 (pHH3), qui fournissent collectivement une délimitation claire des cinq phases du cycle cellulaire. La combinaison de ces marqueurs de cycle cellulaire avec le nombre élevé de paramètres possibles avec MCM permet la combinaison avec de nombreuses autres métriques.
Introduction
La cytométrie de masse permet la détection d’environ 40 paramètres en tirant parti de la haute résolution et de la nature quantitative de la spectroscopie de masse. Les anticorps marqués au métal sont utilisés à la place des anticorps conjugués fluorophores qui permettent un plus grand nombre de canaux et produisent un débordement minimal 1,2. MCM présente des avantages et des inconvénients en ce qui concerne l’analyse du cycle cellulaire par rapport à la cytométrie en flux. L’un des principaux avantages de la MCM est que le grand nombre de paramètres permet la mesure simultanée de l’état du cycle cellulaire sur un grand nombre de types de lymphocytes T immunophénotypiquement distincts dans des échantillons très hétérogènes. MCM a été utilisé avec succès pour mesurer l’état du cycle cellulaire pendant l’hématopoïèse normale dans la moelle osseuse humaine3 et les modèles murins transgéniques de déficit en télomérase4. L’analyse de l’état du cycle cellulaire dans la leucémie myéloïde aiguë (LAM) a montré que le cycle cellulaire était corrélé aux réponses connues aux thérapies cliniques, fournissant un aperçu in vivo des caractéristiques fonctionnelles qui peuvent éclairer les choix de traitement5. Un deuxième avantage de l’analyse du cycle cellulaire cytométrique de masse est la capacité de mesurer un grand nombre d’autres marqueurs fonctionnels qui peuvent être corrélés avec l’état du cycle cellulaire. Des travaux récents ont permis de corréler la synthèse des protéines et de l’ARN avec l’état du cycle cellulaire grâce à l’utilisation d’anticorps IdU et d’anticorps marqués par des métaux dirigés contre BRU et l’ARNr6. Ce type d’analyse hautement paramétrique mesurant l’état du cycle cellulaire dans de nombreuses populations dans un continuum de différenciation serait presque impossible avec la technologie actuelle de cytométrie en flux. Le principal inconvénient de la MCM est l’absence de colorations d’ADN ou d’ARN comparables à celles utilisées en cytométrie en flux fluorescent (par exemple, DAPI, Hoechst, Pyronin Y, etc.). Les colorants fluorescents peuvent donner des mesures relativement précises de la teneur en ADN et en ARN, mais cette précision n’est possible qu’en raison des changements dans les propriétés fluorescentes de ces colorants qui se produisent lors de l’intercalation entre les bases nucléotidiques. L’analyse MCM est donc incapable de mesurer la teneur en ADN ou en ARN avec une précision similaire. Au lieu de cela, l’analyse du cycle cellulaire cytométrique de masse repose sur des mesures de protéines liées à l’état du cycle cellulaire telles que la cycline B1, la protéine du rétinoblastome phosphorylé (pRb) et l’histone phosphorylée H3 (pHH3) combinées à la mesure directe de l’atome d’iode provenant de l’incorporation d’IdU dans les cellules en phase S. Ces deux approches de mesure donnent des résultats très similaires pendant la prolifération cellulaire normale, mais peuvent potentiellement être discordantes lorsque la progression du cycle cellulaire est perturbée.
La mesure du nombre de cellules dans chaque phase du cycle cellulaire est importante pour comprendre le développement normal du cycle cellulaire ainsi que la perturbation du cycle cellulaire, ce qui est couramment observé dans les cancers et les maladies immunologiques. MCM fournit une mesure fiable des facteurs extracellulaires et intracellulaires à l’aide d’anticorps marqués par des métaux; cependant, la mesure de la phase S était limitée car l’intercalateur d’ADN à base d’iridium était incapable de différencier l’ADN 2N et 4N. Afin de définir les phases du cycle cellulaire, Behbehani a développé une méthode qui utilise l’IdU avec une masse de 127, ce qui se situe dans la plage du cytomètre de masse et permet une mesure directe des cellules en phase S3. Cette mesure directe contourne le besoin d’anticorps secondaires ou l’utilisation d’agents dénaturants de l’ADN tels que l’acide ou la DNase. En conjonction avec les marqueurs de cycle intracellulaire, il permet une haute résolution de la distribution du cycle cellulaire dans des modèles expérimentaux.
Ce protocole adapte les mesures du cycle cellulaire à partir des protocoles courants de cytométrie en flux pour MCM. Nos méthodes fournissent un moyen pratique et simple d’inclure des paramètres de cycle cellulaire. L’incorporation d’échantillons in vitro par IdU ne nécessite que 10 à 15 minutes d’incubation à 37 °C, ce qui est plus court que la plupart des protocoles de coloration BrdU qui recommandent des temps d’incubation de plusieurs heures 3,7. Les échantillons incorporés d’IdU et de BrdU peuvent être fixés à l’aide d’un stabilisateur protéomique, puis conservés pendant un certain temps dans un congélateur à -80 °C. Cela permet d’archiver un grand nombre d’échantillons colorés IdU pour analyse par lots sans réduction de la qualité des échantillons.
Protocole
1. Préparation des stocks d’IdU
- Dissoudre la 5-iodo-2'-désoxyuridine (IdU) dans le DMSO à une concentration de 50 mM. Filtre stérile, aliquote dans des tubes de 10 à 50 μL et conserver à -80 °C
- Retirer l’IdU du congélateur et le décongeler à température ambiante. Diluer l’IdU dans le RPMI-1640 pour obtenir une solution de travail à une concentration finale de 1 mM. Pipeter de haut en bas ou vortex pour mélanger.
- En règle générale, diluer l’IdU concentré dans le milieu dans lequel les cellules sont cultivées (p. ex. DMEM, IMDM, etc.) ou l’avoir dilué dans du PBS pour l’ajouter directement aux échantillons d’aspiration de sang périphérique ou de moelle osseuse. Cette étape de pré-dilution facilite le mélange du DMSO avec le milieu aqueux des cellules d’intérêt.
NOTA : La concentration finale d’IdU pendant l’incubation doit être de 10 μM; une solution de 1 mM peut être ajoutée dans un rapport de 10 μL pour 1 mL de média.
- En règle générale, diluer l’IdU concentré dans le milieu dans lequel les cellules sont cultivées (p. ex. DMEM, IMDM, etc.) ou l’avoir dilué dans du PBS pour l’ajouter directement aux échantillons d’aspiration de sang périphérique ou de moelle osseuse. Cette étape de pré-dilution facilite le mélange du DMSO avec le milieu aqueux des cellules d’intérêt.
2. Incubation des IdU et préservation des échantillons
- Conserver les échantillons dans un incubateur humidifié à 37 °C. Retirer l’échantillon de l’incubateur et le déplacer dans une hotte de biosécurité.
- Ajouter 10 μL d’IdU 1 mM à chaque 1 mL d’échantillon.
- Pour une plaque de 6 puits, ajouter 30 μL d’IdU 1 mM aux 3 mL de milieux de culture dans chaque puits. L’IdU peut également être utilisé directement dans les prélèvements de moelle osseuse ainsi que dans les études sur les murins4.
- Replacez l’échantillon dans l’incubateur à 37 °C pendant 10-15 min. Maintenir les cellules dans les conditions de croissance optimales d’intérêt pendant l’exposition à l’IdU afin d’obtenir la mesure la plus précise de la phase S.
- Après l’incubation de l’IdU, prélever l’échantillon et le transférer dans un tube conique.
- Faire tourner l’échantillon à 400 x g pendant 10 min à température ambiante.
- Aspirer le surnageant et remettre en suspension dans 200 μL de PBS.
- Si nécessaire, effectuez une coloration vivante / morte (en utilisant du cisplatine) à cette étape pour marquer les cellules mortes avant la fixation et la congélation.
REMARQUE: La coloration au rhodium vivant / mort ne fonctionne pas bien après perméabilisation au méthanol, elle n’est donc pas recommandée pour une utilisation dans les analyses du cycle cellulaire.
- Si nécessaire, effectuez une coloration vivante / morte (en utilisant du cisplatine) à cette étape pour marquer les cellules mortes avant la fixation et la congélation.
- Ajouter 18,75 μL de paraformaldéhyde (PFA) à 16 % au PBS pour une concentration finale de 1,5 % de PFA. Incuber à température ambiante pendant 10 min. Faire tourner l’échantillon à 400 x g pendant 10 min à température ambiante.
- Aspirer la solution PBS/PFA et remettre l’échantillon en suspension dans 500 μL de milieu de coloration cellulaire (CSM; 1x PBS avec 0,5 % de BSA et 0,02 % d’azoture de sodium) + 10 % de DMSO avant la congélation.
- Si vous utilisez un stabilisant protéomique commercial, ajouter 280 μL de stabilisateur protéomique à l’échantillon remis en suspension dans 200 μL de PBS (1:1,4). Incuber les échantillons à température ambiante pendant 10 min, puis les placer directement dans les -80 °C.
REMARQUE : Il a été démontré que l’IdU s’incorpore efficacement en 10 à 15 minutes d’incubation à 37 °C. Les incubations d’IdU de plus de 10 à 15 minutes réduiront progressivement la résolution des populations en phase S et G2, à mesure que les cellules marquées par l’IdU quittent la phase S et progressent vers la phase G2 ou M. Nous avons également observé que l’incubation à long terme avec IdU peut provoquer la mort cellulaire et des artefacts du cycle cellulaire. Le traitement cellulaire ultérieur et la coloration des anticorps après l’incorporation de l’IdU sont suffisants pour éliminer les IdU résiduels qui n’ont pas été incorporés dans les cellules en phase S. Nous n’avons pas observé de fond iodé significatif lors de l’utilisation du protocole in vitro décrit ici ; Cependant, nous avons très rarement observé une contamination à l’iode dans les échantillons cliniques. Cela peut se produire à partir de procédures médicales, telles que le contraste d’iode dans un scanner, ou de produits pharmaceutiques contenant de l’iode. Si de grandes quantités de bruit de KidU sont observées, l’échantillon ne doit pas être exécuté pour éviter d’endommager le détecteur du cytomètre de masse.
- Si vous utilisez un stabilisant protéomique commercial, ajouter 280 μL de stabilisateur protéomique à l’échantillon remis en suspension dans 200 μL de PBS (1:1,4). Incuber les échantillons à température ambiante pendant 10 min, puis les placer directement dans les -80 °C.
3. Coloration des échantillons pour la cytométrie de masse
- Retirer les échantillons à -80 °C et laisser décongeler avant la coloration de surface.
- Si vous utilisez la méthode de fixation SmartTube, décongelez les échantillons à 0-4 °C pour éviter une fixation supplémentaire lorsque les échantillons se réchauffent.
- Une fois les échantillons décongelés, transférer environ 1 à 2 millions de cellules dans un tube FACS de 5 mL.
- Centrifuger le tube FACS à 600 x g pendant 5 min, et remplir le tube FACS avec un produit de coloration cellulaire (CSM) pour laver les cellules. Répétez une fois de plus.
- Si les cellules sont connues pour coller ensemble, ajouter 400 U/mL d’héparine aux lavages CSM afin d’éviter tout contact de cellule à cellule, mais ce n’est pas strictement nécessaire.
- Incuber les cellules avec un agent bloquant le FC, 5 μL de l’agent pour 100 μL de cellules, pendant 10 min à température ambiante.
- Préparez un mélange d’anticorps qui tachera la surface, ou la partie extracellulaire, des cellules. Le mélange total de coloration s’élèvera à 100 μL pour 1 à 2 millions de cellules dans chaque test. Le mélange de coloration sera équilibré en conséquence avec CSM et héparine dans le cocktail et le tube FACS.
REMARQUE: L’ajout de CSM à cette étape réduira également les artefacts de colorationnon spécifiques 8. Ce mélange de coloration dépend entièrement des cibles d’intérêt et du phénotype de surface (par exemple, une étude impliquant des lymphocytes T utilisera un mélange de surface de CD45, CD3, CD4, CD8, etc.). Un protocole détaillé de traitement et de coloration des échantillons peut être trouvé dans Behbehani et McCarthy et al.9,10. - Ajouter le mélange de coloration de surface aux cellules et incuber à température ambiante en agitant continuellement pendant 30-60 min.
- Après coloration, remplir le tube FACS avec CSM et faire tourner vers le bas à 600 x g pendant 5 min.
- Laver deux fois de plus avec CSM, en faisant tourner l’échantillon à 600 x g pendant 5 min et en aspirant chaque lavage CSM.
- Fixez les anticorps extracellulaires en ajoutant 1 mL de PBS avec 10% CSM et 1,5% PFA.
- Remplir le tube FACS contenant le mélange PBS/CSM/PFA avec CSM. Faire tourner vers le bas à 600 x g pendant 5 min et aspirer le surnageant.
- Ajouter le méthanol à -20 °C.
- Tourbillonner l’échantillon pendant 1-2 minutes pour obtenir une suspension à une seule cellule et vérifier que tous les amas de cellules ont été remis en suspension.
- Pendant que l’échantillon tourbillonne lentement, ajoutez rapidement 1 mL de méthanol glacé à l’aide d’une pipette de 1 000 μL munie d’un embout filtrant.
- Tenez le tube FAC à la lumière et assurez-vous qu’il n’y a pas de touffes visibles; Il faut s’attendre à une nébulosité. Tout amas rendra l’échantillon inutilisable pour une analyse MCM ultérieure.
- Conserver l’échantillon à -20 °C pendant 10-20 min.
- Préparez le mélange de coloration intracellulaire pendant ce temps. Le mélange de coloration intracellulaire dépendra des cibles d’intérêt. Pour l’analyse du cycle cellulaire, inclure CyclinB1, pRb, Ki67 et pHH3 dans ce mélange de coloration, mais d’autres marqueurs intracellulaires peuvent être ajoutés au besoin.
- Après 10-20 min à -20 °C, prélever l’échantillon, ajouter 1,5 mL de PBS et remplir le reste avec CSM.
- Centrifuger l’échantillon 600 x g pendant 5 min, et aspirer le surnageant.
- Laver deux fois de plus avec CSM, en faisant tourner l’échantillon à 600 x g pendant 5 minutes et en aspirant le surnageant à chaque fois.
- Après le dernier lavage CSM, centrifuger l’échantillon et laisser un volume résiduel d’environ 50 μL.
- Ajouter le mélange d’anticorps préparé (ajouter généralement 50 μL de cocktail de coloration d’anticorps pour obtenir un volume de coloration final de 100 μL) à l’échantillon et incuber sur une plate-forme d’agitation pendant 30 à 60 minutes à température ambiante.
- Après coloration, ajouter CSM et centrifuger à 600 x g pendant 5 min.
- Aspirer le MSC, laver à nouveau avec le MSC, en tournant à 600 x g pendant 5 min, en aspirant le MSC, puis ajouter du PBS.
- Une fois la coloration intracellulaire terminée, placez les cellules dans une solution intercalatrice qui fixe les anticorps dirigés contre les cellules et colore l’ADN de chaque cellule pour permettre l’identification. La solution d’intercalateur contient un intercalateur d’iridium non isotopiquement pur (pentaméthylcyclopentadiényl-Ir(III)-dipyridophénazine) ajouté à partir de la solution mère du fabricant à une concentration de 500 μM. Diluer le stock d’iridium 1:4000 dans une solution de PBS et de PFA à 1,5 %. Ajouter la solution d’intercalateur d’iridium à 100-200 μL par million de cellules afin de colorer uniformément et d’éviter la surcoloration.
REMARQUE: L’iridium dans cette solution d’intercalateur est destiné à identifier les cellules pour le gating singulet, il ne doit pas être utilisé pour les taches vivantes / mortes. Si des taches vivantes ou mortes sont souhaitées, elles doivent être effectuées avant la fixation, comme indiqué ci-dessus et dans McCarthy et al.9. - Conservez les échantillons dans une solution intercalatrice dans un réfrigérateur à 4 °C jusqu’à deux semaines avant l’acquisition de l’échantillon sur le CyTOF.
4. Fonctionnement du cytomètre de masse
REMARQUE: L’opération de cytométrie de masse peut être spécifique à la machine. Il est toujours conseillé de consulter le manuel d’utilisation de CyTOF avant l’opération. De plus, il existe actuellement deux articles JoVE traitant du démarrage et de la maintenancedes machines 9,11.
- Vérifiez que le nébuliseur ne présente pas de colmatages, de fissures et d’autres irrégularités avant d’utiliser le cytomètre de masse.
- Connectez le nébuliseur au cytomètre et commencez la procédure de préchauffage. Ne démarrez pas le cytomètre de masse sans le nébuliseur en place.
- Faites couler de l’eau à travers les lignes d’échantillonnage une fois que le cytomètre de masse a fini de se réchauffer. La chambre de pulvérisation doit atteindre environ 200 °C avant d’effectuer le réglage ou l’analyse de l’échantillon.
- Eau courante pendant 5-10 min. Après 5-10 min, chargez la solution de réglage et sélectionnez le gestionnaire de réglage. La solution de réglage est une solution contenant des concentrations fixes de métaux et utilisée pour optimiser le cytomètre de masse avant l’acquisition de l’échantillon
- Dans le gestionnaire de réglage, sélectionnez Aperçu une fois que la solution de réglage a atteint un enregistrement d’accès à l’état stable pour commencer le processus de réglage automatisé.
- Une fois le réglage terminé, chargez l’échantillonneur avec de l’eau et laissez l’eau s’écouler à travers les lignes d’échantillonnage pendant le traitement de l’échantillon. Le protocole détaillé pour le fonctionnement quotidien du cytomètre et le réglage peut être trouvé à Leipold11.
- Parfois, le réglage automatisé ne s’adapte pas aux performances optimales de la machine. Répétez la procédure de réglage pour corriger cela.
- Laver l’échantillon avec CSM une fois et avec de l’eau désionisée pure deux fois avant l’acquisition de l’échantillon. Le lavage à l’eau est important pour éliminer le sel résiduel du PBS / CSM.
- Vérifiez la sensibilité et le débit de l’échantillon à l’aide de billes d’équilibrage fournies par le fabricant, de billes de polystyrène chargées de concentrations de métaux connues.
- Modifiez le mode d’acquisition du réglage au mode de capture d’événements. Fixer le délai d’arrêt de l’acquisition à 120 s. Attendez 45 s avant de sélectionner Enregistrer.
- Le cytomètre de masse arrêtera automatiquement l’acquisition de l’échantillon après 120 secondes. Utilisez la visionneuse de parcelles de pluie pour vérifier l’intensité Eu151 et Eu153.
- Perles d’équilibrage diluées dans de l’eau désionisée pure dans un rapport de 1:20.
- Avant l’acquisition de l’échantillon, vérifiez le gestionnaire d’expériences. Utilisez le gestionnaire d’expériences pour attribuer des noms aux canaux et ajouter des canaux à enregistrer.
- Assurez-vous que le canal 127-I est ajouté si vous utilisez IdU.
- Notez qu’il est essentiel de régler le cytomètre de masse pour mesurer les paramètres nécessaires (par exemple, IdU) avant l’acquisition de l’échantillon. Si les canaux ne sont pas sélectionnés à l’avance, les données ne seront pas collectées à partir d’un canal non sélectionné et ne pourront pas être récupérées.
- Diluer les cellules à une concentration d’environ 1-2 x 106/mL en utilisant l’eau désionisée pure 1:20 et le mélange de billes d’équilibre. Passez les cellules à travers le tube FACS surmonté d’un filtre afin d’éliminer les amas résiduels.
- Chargez l’échantillon et modifiez l’heure d’acquisition.
- Appuyez sur Aperçu et attendez que le nombre d’événements par seconde se stabilise.
- N’exécutez pas d’événements supérieurs à 400 événements par seconde, cela entraînera des quantités importantes de doublets et de débris. Nous recueillons généralement au moins 20 000 à 50 000 événements cellulaires, mais le nombre optimal dépendra de la conception expérimentale. La coloration de jusqu’à 2 millions de cellules produira généralement 300 000 à 400 000 événements cellulaires. Notez que tous les événements ne seront pas des cellules (il y aura des événements de débris et de perles inclus dans le nombre d’événements).
- Une fois l’acquisition de l’échantillon terminée, chargez une solution de lavage, commencez l’induction de l’échantillon et faites fonctionner pendant 5 à 10 minutes. Après 5-10 minutes, arrêtez l’induction de l’échantillon et faites couler l’eau pendant 10-20 minutes. La solution de lavage est une solution faible d’acide fluorhydrique conçue pour éliminer le métal résiduel des lignes d’échantillonnage.
- Fermez le cytomètre de masse et retirez le nébuliseur. Le nébuliseur sera chaud, faites attention pendant la manipulation.
5. Analyse des données
- Afin d’enlever les billes et de corriger la dérive du signal lors de l’acquisition d’échantillons, normalisez les fichiers FCS à l’aide du logiciel Fluidigm ou de l’application développée par Finck12.
- Téléchargez le FCS sur Cytobank ou un autre logiciel d’analyse de cytométrie en flux. Les fichiers FCS peuvent être utilisés dans n’importe quel logiciel compatible, pour les besoins de ce protocole, toutes les analyses et analyses supplémentaires ont été effectuées dans Cytobank13.
- Avant de pouvoir dessiner les portes du cycle cellulaire, exclure tout doublet ou débris cellulaire de l’analyse en aval, cela peut être fait en utilisant le tracé biaxial de la longueur de l’événement par rapport à 191-Ir (Figure 1a). Les cellules formeront une population distincte et brillante Irhaute qui peut être utilisée pour exclure les doublets et les débris. C’est la porte singlet. Cette méthode de contrôle élimine généralement environ 50 à 60% des événements de cellules doublets, de sorte que des stratégies supplémentaires peuvent être nécessaires pour éliminer les événements de cellules doublet restants.
- Modifiez l’échelle de longueur de l’événement (minimum et maximum) pour que les cellules apparaissent plus proéminentes afin de faciliter le contrôle singulet.
- Enlevez davantage les doublets et les débris en utilisant les paramètres gaussiens, résiduels et offset. Un résidu plus élevé avec un décalage plus faible est également constitué de débris et de doublets, et les points de contrôle autour de cette population peuvent éliminer davantage les doublets et les débris (figure 1b, c).
- Gating en phase S – La phase S est la porte la plus facile à dessiner, mais aussi la plus importante. Dessinez cette porte en utilisant un tracé biaxial de IdU vs pRb, Ki67 ou cyclinB1. Les cellules IdU+ en phase S formeront une population distincte lorsque l’on examinera ces diagrammes biaxiaux (Figure 2b).
- G0/G1, phase G2/M – Établir les portes de phase G0/G1 et G2/M sur le tracé IdU vs CyclinB1 et l’utilisation de l’incorporation d’IdU est cruciale pour établir la limite entre les portes de phase G0/G1 et G2/M. La phase G0/G1 seraCyclinB1 faible/IdU- et la phase G2/M sera CyclinB1élevée/IdU-. Une bonne coloration de CyclinB1 montrera une population naturelle entre les populations G0-G1 et G2-M; Cependant, cela variera selon les types d’échantillons et de cellules dans des conditions expérimentales. Dans des conditions expérimentales où la distribution du cycle cellulaire peut être affectée et où il y a moins de séparation entre la phase CyclinB1 G0 / G1 et la phase G2 / M, l’utilisation de la phase S permettra un contrôle cohérent pour des types de cellules particuliers dans chaque expérience spécifique. Cette méthode est détaillée ci-dessous.
- Tracez uniquement les cellules de phase S sur la CyclinB1 vs IdU pour aider à établir la séparation entre la phase G0 / G1 et la phase G2 / M. Dessinez une barrière sur la populationélevée de CyclinB1 et ajustez-la jusqu’à ce qu’environ les 5 % supérieurs de la population de la phase S soient à l’intérieur de la barrière (Figure 2c). Ceci établit le point de rupture entre les portes de phase G0/G1 et G2/M (figure 2e,f). La population active sera remplacée par la population d’intérêt et la partie résidant à l’intérieur de la porte précédente sera la population de phase G2/M, tandis que le reste sera la population de phase G0/G1.
- Gating en phase G0 – Établissez la phase G0 sur le tracé pRb vs IdU. La phase G0 sera représentée par une populationpRb faible/IdU- . La population cycliste active aura une expression élevée de l’incorporation de pRb et d’IdU, la porte de phase G0 peut être dessinée sur cette limite car elle s’exprime généralement à deux populations distinctes (Figure 2g,i).
- Définir la phase G0 en faisant de la population de phase S dessinée précédemment (Figure 2b) la population active et en dessinant une porte incorporant les 90-100% supérieurs de la populationélevée pRb. Il s’agit de la population cycliste pRb+ (Figure 2i), la populationpRb faible en dehors de cette porte est la population G0 (Figure 2j).
- Si pRb n’est pas disponible ou ne peut pas être enregistré, utilisez Ki67 vs IdU pour établir la population en phase G0. En dessinant une porte représentant la majorité de la population de la phase S et en utilisant cette porte comme limite pour le Ki67 vs IdU, le reste de la population sera la phase G0 (Figure 3a).
- Gating de la phase M – Établir la phase M dans le tracé biaxial IdU vs pH3. La phase M représente une très petite fraction des cellules et est fermée sur la populationà pH élevé (Figure 2d).
- L’incorporation de l’IdU a échoué ou n’a pas été possible – Si l’IdU n’est pas disponible, définissez les fractions cycliques et non cycliques à l’aide de Ki67 et pRb. Ki67 et pRb, dans des conditions normales, forment deux populations distinctes : Ki67élevé/pRbélevé et Ki67faible/pRbbas. La population doublement positive représente la population cycliste active, corrélée à la phase G1, à la phase S, à la phase G2 et à la phase M. La double population faible représente la population non cyclique, corrélée à la phase G0 (Figure 3b).
NOTE: Il n’est pas possible de délimiter chaque phase individuelle en utilisant le Ki67 vs pRb, mais les effets expérimentaux sur les populations cyclistes / non cyclistes relatives peuvent être déterminés. - Analyse du cycle cellulaire – Une fois les portes établies, exportez les valeurs numériques des portes pour une analyse plus approfondie. Les pourcentages de chaque cycle peuvent être obtenus en soustrayant les populations individuelles des populations combinées. La phase G0, dessinée sur le pourcentage de portepRbfaible IdU faible, peut être soustraite de la phase G0/G1, tirée sur CyclinB1faible IdUneg, pourtrouver le pourcentage de phase G1. De même, le pourcentage de phase G2 est dérivé de la soustraction de la porte phase M de la porte G2/phase M. Cela générera des valeurs numériques pour chaque phase individuelle du cycle cellulaire; G0, G1, S, G2 et M. Les valeurs numériques générées pour chaque phase individuelle du cycle cellulaire peuvent être utilisées pour des analyses plus approfondies telles que la représentation graphique et l’analyse statistique.
Résultats
En utilisant des cellules HL-60 et une aspiration de moelle osseuse humaine, il est possible de montrer comment les conditions expérimentales peuvent affecter la distribution et l’analyse du cycle cellulaire. Tout d’abord, la stratégie de contrôle doit être établie pour démontrer comment les phases du cycle cellulaire sont dérivées. Dans la figure 1, nous montrons l’établissement de la porte singulet, qui est importante pour séparer les débris cellulaires et les doublets, établissant une population unicellulaire. Pour les lignées cellulaires, la porte singulet est tout ce qui est nécessaire pour passer à l’analyse du cycle cellulaire (Figure 2a). Pour les échantillons humains, les populations immunophénotypiques doivent généralement être établies avant l’analyse du cycle cellulaire, car les limites exactes de chaque porte du cycle cellulaire peuvent varier selon les différents types de cellules. Une fois que les populations ont été établies (généralement en gisant sur des marqueurs de surface qui définissent cette population), les portes du cycle cellulaire devront alors être établies. La figure 2b montre l’établissement de la phase S sur le graphique biaxial IdU vs CyclinB1. Ce tracé est également utilisé pour établir la limite de la porte de phase G2/M (Figure 2f). Une fois la phase G2/M établie, le reste est la porte de phase G0/G1 (Figure 2f). L’IdU vs pRb est utilisé pour établir d’abord la population cyclique pRb+ en établissant une porte sur les cellules IdU incorporant (Figure 2g,i). La populationpRb+/IdU neg à l’extérieur de cette porte est la phase G0 (Figure 2j). La phase M est établie sur l’IdU vs pHH3 où les cellules de phase M expriment des niveaux élevés de pHH3 et ne présentent aucune incorporation d’IdU (Figure 2k). Dans le cas où pRb n’est pas inclus, la phase G0 peut être répliquée à l’aide de Ki67 d’une manière similaire à la méthode décrite ci-dessus (Figure 3a). Si l’incorporation de l’IdU a échoué ou n’a pas été effectuée, il est toujours possible de déterminer les fractions de cycle relatives en utilisant Ki67 et pRb. En utilisant les populations biaxiales Ki67 et pRb, deux populations distinctes se forment, une population pRb+/Ki67+ double positive et une population pRb faible/Ki67faible. La population doublement positive représente les cellules en cycle, tandis que la population faible représente les cellules non en cycle (Figure 3b). En utilisant des cellules incorporant des IdU et des cellulespRb faibles sans incorporation d’IdU, nous montrons que la phase S est principalement dans la population pRb + / Ki67 + tandis que la phase G0 est principalement dans la population pRb faible / Ki67faible.
L’analyse du cycle cellulaire repose sur une bonne technique expérimentale, en particulier pendant l’étape d’incubation de l’IdU. Bien que l’incorporation d’IdU soit flexible (applicable dans la culture cellulaire, les aspirations de moelle osseuse et même les études sur les murins), il est nécessaire d’effectuer l’incorporation et la fixation de l’IdU sans perturber l’état du cycle cellulaire d’intérêt expérimental. Le marquage des IdU et donc l’analyse du cycle cellulaire en aval peuvent être affectés de manière significative par le temps et la température, comme indiqué à la figure 4. Les cellules qui restent trop longtemps dans des récipients fermés ou qui pourraient être rencontrées lors de l’expédition ou du transport des échantillons entre les emplacements, auront une fraction de phase S réduite et ne seront pas précises pour l’analyse du cycle cellulaire (Figure 4a). Cependant, les périodes courtes, celles de moins d’une heure au total, auront une distribution normale du cycle cellulaire, ce qui indique qu’un transport rapide peut ne pas nuire à l’analyse du cycle cellulaire (Figure 4b). Un autre modificateur important est la cryoconservation qui est couramment utilisée dans la plupart des laboratoires. Lors de l’examen de l’état du cycle cellulaire dans des cellules cryoconservées, une longue période d’équilibration peut être nécessaire avant que les cellules ne reviennent au cycle cellulaire actif, ce qui peut encore ne pas refléter l’état du cycle cellulaire pré-cyropréservation (Figure 4c).
Les échantillons humains primaires sont souvent des composites de plusieurs types de cellules différents, ces différents types de cellules peuvent avoir des sensibilités différentes au traitement, ce qui conduit à différents cycles cellulaires. Dans deux aspistations de moelle osseuse marquées immédiatement, stockées pendant 30 minutes avant le marquage de l’IdU ou cryogéniques après séparation Ficoll, il existe des différences entre chaque échantillon et population (Figure 5a, b). Deux populations immunophénotypiques ont été examinées pour déterminer les différences dans l’incorporation d’IdU; lymphocytes T (CD45 élevé/CD3élevé) et monoblastes (CD33+, HLADR+, CD11bfaible, CD14neg). Avec la combinaison correcte de marques de surface, il est possible d’examiner d’autres populations immunophénotypiques. Dans la moelle #2, il y avait un effet d’activation notable des lymphocytes T après 30 minutes de stockage qui n’a pas été observé dans les monoblastes du même patient (Figure 5b). Comme pour les cellules en culture, il y avait des changements notables dans le marquage des IdU après le stockage cryogénique qui dépendait également de la population. La moelle #1 avait une réduction de la population de lymphocytes T, mais une augmentation des fractions marquées par l’IdU des monoblastes par rapport à la ligne de base (Figure 5 a, b), la moelle #2 a montré une réduction des cellules T et des monoblastes par rapport à la ligne de base (Figure 5a, b). Les cellules congelées nécessitent alors une période d’incubation notable avant de revenir à l’état normal du cycle cellulaire, ce qui peut influencer les études qui reposent sur la modification de l’état du cycle cellulaire ou de l’état du cycle cellulaire comme mesure de l’effet médicamenteux ou expérimental.
Un autre avantage de MCM est la capacité de discriminer les cellules dans l’arrêt du cycle cellulaire ou qui ont une distribution anormale du cycle cellulaire. Bien que les colorants à ADN couramment utilisés en cytométrie de flux soient capables de distinguer la teneur en ADN 2N et 4N, ils sont très brillants, ce qui peut grandement compliquer la mesure d’autres paramètres de ce laser. L’IdU, cependant, ne prend qu’un seul canal de masse et a un débordement minimal, ce qui permet d’utiliser d’autres marqueurs dans la détermination du cycle cellulaire. Les cellules MOLM13 irradiées montrent une diminution de l’incorporation d’IdU et une diminution de la phase M par rapport aux cellules témoins (Figure 6). La perturbation des points de contrôle du cycle cellulaire normal peut modifier l’état apparent du cycle cellulaire par MCM. En examinant les populations pH2AX et cPARP dans les cellules non irradiées, la population pH2AXfaible et cPARP faible montre une distribution normale du cycle cellulaire tandis que les cellules exprimant des niveaux plus élevés de pH2AX oucPARP se localisent principalement dans la phase G0 / G1 attendue (Figure 6a). Dans les cellules irradiées, le pH2AX bas et le cPARP bas, cependant, les cellules sont presque entièrement localisées dans la phase G0, tandis que les cellules pH2AXhaut etcPARP bas montrent un phénotype d’arrêt du cycle cellulaire avec incorporation IdU et localisation à la phase G0 / G1 et à la phase G2 avec une absence de phase M. Les cellules pH2AX high et cPARPhigh montrent également des cellules incorporant des IdU et se localisant à la phase G0/G1 indiquant des dommages causés par le rayonnement (Figure 6b).
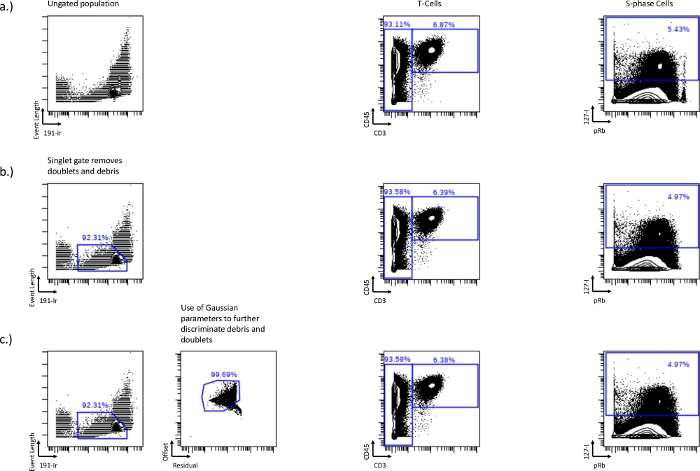
Figure 1: Établissement des portes singulet en utilisant 191-Ir par longueur d’événement et également les paramètres gaussiens, résiduels et décalés.
Les différences de lymphocytes T (CD45+/CD3+) et de phase S (IdU+) entre un échantillon non fermé (a), une longueur d’événement par rapport à une porte singulet 191-Ir (b) ou une porte singulet combinée à des paramètres gaussiens, résiduels et décalés (c). Le singlet gating élimine les débris, les doublets et les billes montrés dans la perte de la populationélevée pRb sur le coin droit de la biaxiale. Cette porte singulet peut être encore optimisée en incluant des paramètres gaussiens tels que les résidus et le décalage, éliminant ainsi plus de débris. Veuillez cliquer ici pour voir une version agrandie de cette figure.
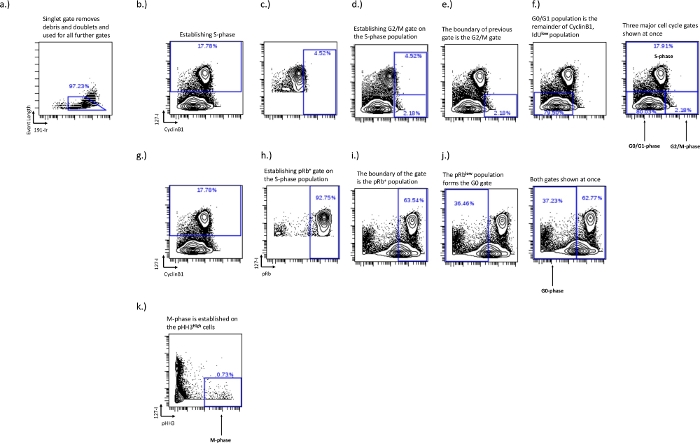
Figure 2 : Schéma de contrôle pour l’établissement de portes de cycle cellulaire pour les phases G0, G1, S, G2 et M à l’aide de IdU, CyclinB1, pRb et pHH3.
La porte singulet est établie pour enlever les doublets et les débris (a). La phase S doit être établie (b), une fois la phase S établie, la population IdU+ peut être utilisée pour établir la limite de la phase G2/M (c, d). L’établissement de la limite de phase G2/M établit les limites de la population de phase G0/G1 (f). La population en phase pRb+ et G0 est établie sur la biaxiale IdU vs pRb. Les cellules IdU+ (h) sont utilisées pour établir la limite de la population pRb+ (i). La limite de la population pRb+ établit la limite de la population de phase G0 (j). La phase M est établie sur les cellules pHH3+ qui sont IdU- (k). Veuillez cliquer ici pour voir une version agrandie de cette figure.
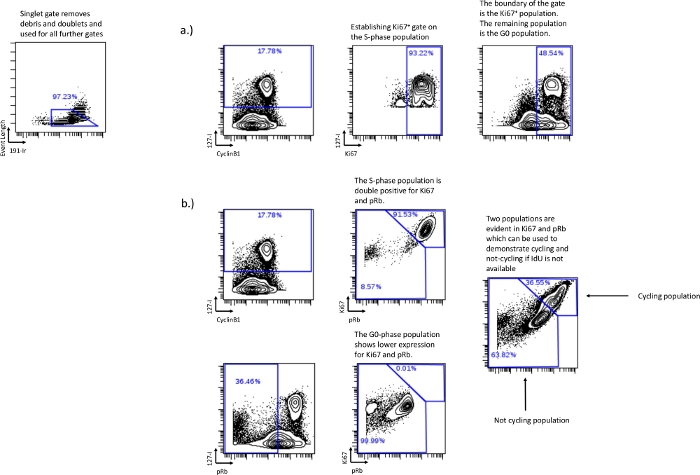
Figure 3 : Établissement de portes de cycle cellulaire sans l’utilisation de pRb ou sans l’utilisation de l’incorporation d’IdU.
Le dessin de la phase G0 peut également être effectué en utilisant Ki-67 en suivant la même stratégie de déclenchement que celle utilisée avec pRb, si pRb n’est pas inclus dans l’expérience (a). Si l’incorporation d’IdU a échoué ou n’a pas été effectuée, il est possible de récupérer les fractions relatives du cycle cellulaire grâce à l’utilisation de Ki67 et de pRb. L’expression double positive de Ki67 et pRb est corrélative aux cellules en cycle, comme en témoigne la démonstration que les cellules IdU+ se trouvent principalement dans la population double positive (b). La population de Ki67 et de pRb faible est en corrélation avec la population en phase G0 ou non cyclique démontrée par les cellulespRb faibles/IdU neg trouvées dans la population Ki67 et pRb faible. Cette méthode ne peut pas discriminer les phases individuelles du cycle cellulaire, mais peut toujours être utilisée pour déterminer les fractions de cycle relatives dans des conditions expérimentales. Veuillez cliquer ici pour voir une version agrandie de cette figure.

Figure 4 : Figures représentatives de l’effet de différentes conditions de stockage sur la distribution du cycle cellulaire des cellules HL-60.
Les cellules ont été incubées pendant une heure, suivies d’une heure de repos aux conditions de température indiquées dans des tubes scellés (a). Il y a des effets notables sur la distribution du cycle cellulaire par rapport au témoin. Les cellules HL60 ont été conservées dans des tubes scellés à température ambiante pendant 30 minutes, une situation qui peut se produire en milieu clinique, avant l’incorporation de l’IdU (b). Les tubes scellés conservés à température ambiante pendant 30 minutes n’ont pas montré de différences appréciables dans le cycle cellulaire. L’effet du stockage cryogénique a été étudié sur le cycle cellulaire dans les cellules HL60, où un échantillon a été prélevé avant le stockage cryogénique et un échantillon prélevé après une heure de repos après une semaine en stockage cryogénique (c). Une heure après le dégel, la distribution du cycle cellulaire est affectée et la distribution du cycle cellulaire ne revient à la normale qu’environ une semaine après la décongélation. Veuillez cliquer ici pour voir une version agrandie de cette figure.

Figure 5 : Le traitement peut avoir un effet sur les échantillons de patients et des images représentatives sont montrées entre deux patients différents montrant (a) les lymphocytes T (CD45 élevé/CD3élevé) et (b) les monoblastes (CD33+, HLADR+, CD11bfaible, CD14neg).
Entre la moelle un et la moelle deux, il est clair que dans la moelle 2, il y avait un effet d’activation des lymphocytes T pendant les 30 minutes de repos alors que la moelle une n’avait pas un tel effet. La moelle un a montré un effet d’activation possible dans la population de monoblastes après 30 minutes, tandis que la moelle deux n’a pas montré. Dans les deux moelles, cependant, il était clair que le stockage cryogénique avait un impact sur la distribution du cycle cellulaire, quel que soit le type de cellule. Veuillez cliquer ici pour voir une version agrandie de cette figure.

Figure 6 : Les cellules MOLM13 ont été laissées comme témoins ou irradiées à l’aide d’un irradiateur à rayons X à 10 Gy.
Les cellules MOLM13 montrent la distribution du cycle cellulaire dans quatre populations différentes d’expression de pH2AX et cPARP. Les cellules témoins présentent une coloration minimale du pH2AX et du cPARP, les caractéristiques normales du cycle cellulaire étant montrées dans la populationà faible pH2AX et la populationcPARP faible (a). Alors que les cellules irradiées montrent une distribution anormale du cycle cellulaire, la majorité des cellules cycliques étant situées dans le pH2AX élevé et cPARPélevé, indiquant une perturbation du cycle cellulaire (b). Les cellules non endommagées, pH2AX faible etcPARP faible, montrent un manque de caractéristiques de cycle se trouvent principalement dans la phase G0. Sans ces marqueurs, ces cellules apparaîtraient comme des cellules 4N et 2N dans la cytométrie en flux normal, ce qui pourrait confondre l’analyse du cycle cellulaire en aval. Veuillez cliquer ici pour voir une version agrandie de cette figure.
Discussion
Les exemples présentés ici montrent comment utiliser une plateforme MCM pour analyser la distribution du cycle cellulaire. Il a également été démontré que l’analyse du cycle cellulaire est sensible aux conditions expérimentales telles que le temps et la température, ce qui est une considération importante que les chercheurs doivent prendre en compte lorsqu’ils envisagent la MCM pour leur analyse du cycle cellulaire14. Les échantillons laissés entreposés pendant une courte période, pas plus d’une heure, auront une incorporation d’IdU comparable à leur état normal. Les échantillons prélevés dans un système fermé pendant de longues périodes, environ 2 heures, auront réduit l’incorporation d’IdU, mais les fractions cycliques et non cycliques relatives ne changeront pas, ce qui permettra une analyse du cycle cellulaire grossier. Le stockage cryogénique et la décongélation subséquente perturbent la distribution normale du cycle cellulaire pendant une période de temps significative. Il a été noté précédemment que le stockage cryogénique perturbe la distribution des protéines et de l’ARN, mais ce n’est que récemment qu’il a été démontré qu’il perturbait le cycle cellulaire14,15,16,17. Pris ensemble, cela indique que si de longues durées de stockage sont prévues ou si l’on prévoit une conservation cryogénique des échantillons, il serait préférable de colorer les cellules avec de l’IdU avant le stockage ou le stockage cryogénique, de fixer les cellules et de les stocker jusqu’à ce que l’analyse puisse être effectuée. Comme l’analyse du cycle cellulaire par MCM ne nécessite pas de cellules vivantes, cela permettrait aux chercheurs de mettre en banque des échantillons précieux et d’effectuer une analyse précise du cycle cellulaire en aval.
L’analyse du cycle cellulaire par MCM est un système robuste capable d’interroger en profondeur des modèles expérimentaux. En raison du nombre élevé de paramètres MCM, environ 40-50 canaux de masse, l’analyse du cycle cellulaire peut être combinée avec d’autres marqueurs intracellulaires ou extracellulaires en contournant la nécessité de trier les cellules en fonction de l’immunophénotype, ce qui peut entraîner une perte significative des effets du cycle cellulaire. La nature hautement paramétrique de MCM se prête à l’examen des effets dans les applications de cartographie de grande dimension telles que SPADE et viSNE. Bien que SPADE et viSNE soient généralement utilisés pour définir des populations immunophénotypiques qui peuvent ensuite être examinées pour les changements du cycle cellulaire, il serait également possible de cartographier les marqueurs du cycle cellulaire. Selon les conditions expérimentales, la cartographie des marqueurs du cycle cellulaire dans un espace de grande dimension peut montrer une corrélation du cycle cellulaire avec l’effet du médicament ou quelles populations immunophénotypiques peuvent localiser à chaque étatde cycle 3,5. Bien que la MCM puisse être limitée par l’absence de colorants fluorescents liant l’ADN, elle est compensée par l’incorporation directe d’IdU pendant les cellules en phase S et les protéines du cycle cellulaire intracellulaire peuvent être utilisées pour déterminer l’état du cycle cellulaire. Ces protéines du cycle cellulaire peuvent également aider à distinguer les étapes de l’arrêt du cycle cellulaire qui apparaîtraient autrement comme 3N ou 4N dans le flux traditionnel utilisant des colorants de liaison à l’ADN. De tels systèmes hautement paramétriques ne sont cependant pas sans inconvénients, et ils sont sensibles aux perturbations du cycle cellulaire. Nous avons montré que les longs temps de stockage et le stockage cryogénique peuvent affecter de manière significative la distribution du cycle cellulaire. Ceci est particulièrement important lorsque l’on tente de rationaliser les effets expérimentaux des médicaments susceptibles d’affecter la distribution du cycle cellulaire. Le traitement des médicaments conçus pour affecter le cycle cellulaire sur des échantillons primaires congelés peut donner de fausses données sur l’effet du cycle cellulaire lorsqu’ils sont utilisés immédiatement après la décongélation du stockage cryogénique. MCM est une technologie polyvalente pour l’analyse du cycle cellulaire qui peut être appliquée à un certain nombre de modèles expérimentaux et est particulièrement adaptée au profilage approfondi de systèmes hétérogènes. Comme pour d’autres méthodes hautement paramétriques, il est nécessaire d’avoir une expérience soigneusement conçue avec des considérations appropriées sur la façon dont le traitement et les effets expérimentaux affecteront l’analyse du cycle cellulaire.
Déclarations de divulgation
Le Dr Behbehani reçoit une aide au voyage de Fluidigm. Fluidigm a également acheté des réactifs et des matériaux pour une utilisation en laboratoire.
Remerciements
Les auteurs tiennent à remercier Palak Sekhri, Hussam Alkhalaileh, Hsiaochi Chang et Justin Lyeberger pour leur soutien expérimental. Ce travail a été soutenu par le programme de bourses Pelotonia Fellowship. Toutes les opinions, constatations et conclusions exprimées dans ce document sont celles de l’auteur (s) et ne reflètent pas nécessairement celles du programme de bourses Pelotonia Fellowship.
matériels
| Name | Company | Catalog Number | Comments |
| Bovine Serum Albumin (BSA) | Sigma | A3059 | Component of CSM |
| Centrifuge | Thermo Scientific | 75-217-420 | Sample centrifugation |
| Cleaved-PARP (D214) | BD Biosciences | F21-852 | Identification of apoptotic cells |
| Cyclin B1 | BD Biosciences | GNS-1 | G2 Resolution |
| Dimethylsulfoxide (DMSO) | Sigma | D2650 | Cryopreservative |
| EQ Four Element Calibration Beads | Fluidigm | 201078 | Internal metal standard for CyTOF performance |
| FACS Tube w/ mesh strainer | Corning | 08-771-23 | Cell strainer to remove clumps/debris before CyTOF run |
| Fetal Bovine Serum (FBS) | VWR | 97068-085 | Cell culture growth supplement |
| Helios | Fluidigm | CyTOF System/Platform | |
| Heparin | Sigma | H3393 | Staining additive to prevent non-specific staining |
| IdU (5-Iodo-2′-deoxyuridine) | Sigma | I7125 | Incorporates in S-phase |
| Ki-67 | eBiosciences | SolA15 | Confirmation of G0/G1 |
| MaxPar Multi Label Kit | Fluidigm | 201300 | Metal labeling kit, attaches metals to antibodies |
| Microplate Shaker | Thermo Scientific | 88880023 | Mixing samples during staining |
| Paraformaldehyde (PFA) | Electron Microscopy Services | 15710 | Fixative |
| pentamethylcyclopentadienyl-Ir(III)-dipyridophenazine | Fluidigm | 201192 | Cell identification during CyTOF acquisition |
| p-H2AX (S139) | Millipore | JBW301 | Detection of DNA damage |
| p-HH3 (S28) | Biolegend | HTA28 | M-phase Resolution |
| Phosphate Buffered Saline (PBS) | Gibco | 14190-144 | Wash solution for cell culture and component of fixative solution |
| p-Rb (S807/811) | BD Biosciences | J112906 | G0/G1 Resolution |
| Proteomic Stabilizer | SmartTube Inc | PROT1 | Sample fixative |
| RPMI 1640 | Gibco | 21870-076 | Cell culture growth medium |
| Sodium Azide | Acros Organics | AC447810250 | Component of CSM/Antibody buffer, biocide |
Références
- Ornatsky, O., Baranov, V. I., Bandura, D. R., Tanner, S. D., Dick, J. Multiple cellular antigen detection by ICP-MS. Journal of Immunological Methods. 308 (1-2), 68-76 (2006).
- Ornatsky, O. I., et al. Study of cell antigens and intracellular DNA by identification of element-containing labels and metallointercalators using inductively coupled plasma mass spectrometry. Analytical Chemistry. 80 (7), 2539-2547 (2008).
- Behbehani, G. K., Bendall, S. C., Clutter, M. R., Fantl, W. J., Nolan, G. P. Single-cell mass cytometry adapted to measurements of the cell cycle. Cytometry A. 81 (7), 552-566 (2012).
- Raval, A., et al. Reversibility of Defective Hematopoiesis Caused by Telomere Shortening in Telomerase Knockout Mice. PLoS One. 10 (7), 0131722 (2015).
- Behbehani, G. K., et al. Mass Cytometric Functional Profiling of Acute Myeloid Leukemia Defines Cell-Cycle and Immunophenotypic Properties That Correlate with Known Responses to Therapy. Cancer Discovery. 5 (9), 988-1003 (2015).
- Kimmey, S. C., Borges, L., Baskar, R., Bendall, S. C. Parallel analysis of tri-molecular biosynthesis with cell identity and function in single cells. Nature Communications. 10 (1), 1185 (2019).
- Rabinovitch, P. S., Kubbies, M., Chen, Y. C., Schindler, D., Hoehn, H. BrdU-Hoechst flow cytometry: a unique tool for quantitative cell cycle analysis. Experimental Cell Research. 174 (2), 309-318 (1988).
- Rahman, A. H., Tordesillas, L., Berin, M. C. Heparin reduces nonspecific eosinophil staining artifacts in mass cytometry experiments. Cytometry A. 89 (6), 601-607 (2016).
- McCarthy, R. L., Duncan, A. D., Barton, M. C. Sample Preparation for Mass Cytometry Analysis. Journal of Visualized Experiments. (122), e54394 (2017).
- Behbehani, G. K. Immunophenotyping by Mass Cytometry. Methods in Molecular Biology. 2032, 31-51 (2019).
- Leipold, M. D., Maecker, H. T. Mass cytometry: protocol for daily tuning and running cell samples on a CyTOF mass cytometer. Journal of Visualized Experiments. (69), e4398 (2012).
- Finck, R., et al. Normalization of mass cytometry data with bead standards. Cytometry A. 83 (5), 483-494 (2013).
- Kotecha, N., Krutzik, P. O., Irish, J. M. Web-based analysis and publication of flow cytometry experiments. Current Protocols in Cytometry. , 17 (2010).
- Devine, R. D., Sekhri, P., Behbehani, G. K. Effect of storage time and temperature on cell cycle analysis by mass cytometry. Cytometry A. 93 (11), 1141-1149 (2018).
- Campos, L., et al. Expression of immunological markers on leukemic cells before and after cryopreservation and thawing. Cryobiology. 25 (1), 18-22 (1988).
- Kadic, E., Moniz, R. J., Huo, Y., Chi, A., Kariv, I. Effect of cryopreservation on delineation of immune cell subpopulations in tumor specimens as determinated by multiparametric single cell mass cytometry analysis. BMC Immunology. 18 (1), 6 (2017).
- Shabihkhani, M., et al. The procurement, storage, and quality assurance of frozen blood and tissue biospecimens in pathology, biorepository, and biobank settings. Clinical Biochemistry. 47 (4-5), 258-266 (2014).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon