Method Article
Uso del análogo de pirimidina, 5-yodo-2′-desoxiuridina (IdU) con marcadores de ciclo celular para establecer fases del ciclo celular en una plataforma de citometría de masas
En este artículo
Resumen
Este protocolo adapta las mediciones del ciclo celular para su uso en una plataforma de citometría de masas. Con las capacidades multiparamétricas de la citometría de masas, la medición directa de la incorporación de yodo permite la identificación de células en fase S, mientras que los marcadores de ciclo intracelular permiten la caracterización de cada estado del ciclo celular en una variedad de condiciones experimentales.
Resumen
La regulación de la fase del ciclo celular es un aspecto importante de la proliferación celular y la homeostasis. La interrupción de los mecanismos reguladores que rigen el ciclo celular es una característica de una serie de enfermedades, incluido el cáncer. El estudio del ciclo celular requiere la capacidad de definir el número de células en cada porción de la progresión del ciclo celular, así como de delinear claramente entre cada fase del ciclo celular. El advenimiento de la citometría de masas (MCM) proporciona un enorme potencial para el análisis de células individuales de alto rendimiento a través de mediciones directas de isótopos elementales, y el desarrollo de un método para medir el estado del ciclo celular por MCM amplía aún más la utilidad de MCM. Aquí describimos un método que mide directamente la 5-yodo-2′-desoxiuridina (IdU), similar a la 5-bromo-2'-desoxiuridina (BrdU), en un sistema MCM. El uso de este MCM basado en IdU proporciona varias ventajas. En primer lugar, IdU se incorpora rápidamente en el ADN durante su síntesis, lo que permite una medición fiable de las células en la fase S con incubaciones tan cortas como 10-15 minutos. En segundo lugar, IdU se mide sin la necesidad de anticuerpos secundarios o la necesidad de degradación del ADN. En tercer lugar, la tinción de IdU se puede combinar fácilmente con la medición de ciclina B1, proteína de retinoblastoma fosforilada (pRb) e histona fosforilada H3 (pHH3), que colectivamente proporciona una delineación clara de las cinco fases del ciclo celular. La combinación de estos marcadores de ciclo celular con el alto número de parámetros posibles con MCM permite la combinación con muchas otras métricas.
Introducción
La citometría de masas permite la detección de aproximadamente 40 parámetros aprovechando la alta resolución y la naturaleza cuantitativa de la espectroscopia de masas. Se utilizan anticuerpos marcados con metales en lugar de anticuerpos conjugados fluoróforos que permiten un mayor número de canales y producen un derrame mínimo 1,2. MCM tiene ventajas y desventajas con respecto al análisis del ciclo celular en comparación con la citometría de flujo. Una ventaja importante de MCM es que el gran número de parámetros permite la medición simultánea del estado del ciclo celular a través de un gran número de tipos de células T inmunofenotípicamente distintos en muestras altamente heterogéneas. MCM se ha utilizado con éxito para medir el estado del ciclo celular durante la hematopoyesis normal en médula ósea humana3 y modelos murinos transgénicos de deficiencia de telomerasa4. El análisis del estado del ciclo celular en la leucemia mieloide aguda (LMA) mostró que el ciclo celular se correlacionó con respuestas conocidas a terapias clínicas, proporcionando una visión in vivo de las características funcionales que pueden informar las selecciones de terapia5. Una segunda ventaja del análisis del ciclo celular citométrico masivo es la capacidad de medir un gran número de otros marcadores funcionales que pueden estar correlacionados con el estado del ciclo celular. Trabajos recientes han podido correlacionar la síntesis de proteínas y ARN con el estado del ciclo celular mediante el uso de IdU y anticuerpos marcados con metales contra BRU y rRNA6. Este tipo de análisis altamente paramétrico que mide el estado del ciclo celular en numerosas poblaciones en un continuo de diferenciación sería casi imposible con la tecnología actual de citometría de flujo. La principal desventaja de MCM es la falta de tinciones de ADN o ARN comparables a las utilizadas en la citometría de flujo fluorescente (por ejemplo, DAPI, Hoechst, Pyronin Y, etc.). Los colorantes fluorescentes pueden dar mediciones relativamente precisas del contenido de ADN y ARN, pero esta precisión solo es posible debido a los cambios en las propiedades fluorescentes de estos colorantes que ocurren al intercalar entre las bases de nucleótidos. Por lo tanto, el análisis MCM no puede medir el contenido de ADN o ARN con una precisión similar. En cambio, el análisis del ciclo celular citométrico masivo se basa en mediciones de proteínas relacionadas con el estado del ciclo celular, como la ciclina B1, la proteína de retinoblastoma fosforilada (pRb) y la histona fosforilada H3 (pHH3) combinada con la medición directa del átomo de yodo a partir de la incorporación de IdU en células de fase S. Estos dos enfoques de medición producen resultados muy similares durante la proliferación celular normal, pero pueden ser potencialmente discordantes cuando se interrumpe la progresión del ciclo celular.
La medición del número de células en cada fase del ciclo celular es importante para comprender el desarrollo normal del ciclo celular, así como la interrupción del ciclo celular, que se observa comúnmente en cánceres y enfermedades inmunológicas. MCM proporciona una medición confiable de factores extracelulares e intracelulares utilizando anticuerpos marcados con metales; sin embargo, la medición de la fase S fue limitada ya que el intercalador de ADN basado en iridio no pudo diferenciar entre el ADN 2N y 4N. Para definir las fases del ciclo celular, Behbehani desarrolló un método que utiliza IdU con una masa de 127, que cae dentro del rango del citómetro de masas y permite la medición directa de células en la fase S3. Esta medición directa evita la necesidad de anticuerpos secundarios o el uso de agentes desnaturalizantes del ADN como ácido o DNasa. Junto con marcadores de ciclo intracelular, permite una alta resolución de la distribución del ciclo celular en modelos experimentales.
Este protocolo adapta las mediciones del ciclo celular de los protocolos comunes de citometría de flujo para MCM. Nuestros métodos proporcionan una manera conveniente y sencilla de incluir parámetros del ciclo celular. La incorporación de IdU de muestras in vitro requiere solo de 10 a 15 minutos de incubación a 37 °C, que es más corto que la mayoría de los protocolos de tinción BrdU que recomiendan tiempos de incubación de varias horas 3,7. Las muestras incorporadas a IdU y BrdU pueden fijarse utilizando un estabilizador proteómico y luego almacenarse durante algún tiempo en un congelador de -80 °C. Esto permite archivar un gran número de muestras teñidas con IdU para el análisis por lotes sin reducir la calidad de la muestra.
Protocolo
1. Preparación de las existencias de IdU
- Disuelva la 5-yodo-2'-desoxiuridina (IdU) en DMSO a una concentración de 50 mM. Filtro estéril, alícuota en tubos de 10-50 μL y conservar a -80 °C
- Retire la IdU del congelador y descongele a temperatura ambiente. Diluir IdU en RPMI-1640 para hacer una solución de trabajo a una concentración final de 1 mM. Pipetear hacia arriba y hacia abajo o vórtice para mezclar.
- Por lo general, diluya la IdU concentrada en el medio en el que se cultivan las células (por ejemplo, DMEM, IMDM, etc.) o la diluya en PBS para agregarla directamente a muestras de aspirado de sangre periférica o médula ósea. Este paso de predilución facilita la mezcla del DMSO con los medios acuosos de las células de interés.
NOTA: La concentración final de IdU durante la incubación debe ser de 10 μM; se puede agregar una solución de 1 mM en una proporción de 10 μL por cada 1 ml de medio.
- Por lo general, diluya la IdU concentrada en el medio en el que se cultivan las células (por ejemplo, DMEM, IMDM, etc.) o la diluya en PBS para agregarla directamente a muestras de aspirado de sangre periférica o médula ósea. Este paso de predilución facilita la mezcla del DMSO con los medios acuosos de las células de interés.
2. Incubación de IdU y conservación de muestras
- Mantener las muestras en una incubadora humidificada a 37 °C. Retire la muestra de la incubadora y muévala a una campana de bioseguridad.
- Añadir 10 μL de IdU de 1 mM a cada 1 ml de muestra.
- Para una placa de 6 pocillos, agregue 30 μL de IdU de 1 mM a los 3 ml de medios de cultivo en cada pocillo. La IdU también se puede utilizar directamente en aspirados de médula ósea, así como en estudios murinos4.
- Vuelva a colocar la muestra en la incubadora a 37 °C durante 10-15 min. Mantener las células bajo las condiciones óptimas de crecimiento de interés durante la exposición a IdU para obtener la medición más precisa de la fase S.
- Después de la incubación de IdU, retire la muestra y transfiérala a un tubo cónico.
- Girar la muestra a 400 x g durante 10 min a temperatura ambiente.
- Aspirar el sobrenadante y resuspender en 200 μL de PBS.
- Si es necesario, realice una tinción viva/muerta (usando cisplatino) en este paso para marcar las células muertas antes de la fijación y congelación.
NOTA: La tinción de rodio vivo / muerto no funciona bien después de la permeabilización del metanol, por lo que no se recomienda su uso en análisis del ciclo celular.
- Si es necesario, realice una tinción viva/muerta (usando cisplatino) en este paso para marcar las células muertas antes de la fijación y congelación.
- Agregue 18.75 μL de paraformaldehído (PFA) al 16% al PBS para una concentración final de 1.5% PFA. Incubar a temperatura ambiente durante 10 min. Girar la muestra a 400 x g durante 10 min a temperatura ambiente.
- Aspirar la solución de PBS/PFA y volver a suspender la muestra en 500 μL de medios de tinción celular (CSM; 1x PBS con BSA al 0,5% y azida sódica al 0,02%) + DMSO al 10% antes de la congelación.
- Si utiliza un estabilizador proteómico comercial, añadir 280 μL de estabilizador proteómico a la muestra resuspendida en 200 μL de PBS (1:1.4). Incubar las muestras a temperatura ambiente durante 10 min y luego colocarlas directamente en -80 °C.
NOTA: Se ha demostrado que IdU se incorpora eficazmente dentro de 10-15 minutos de incubación a 37 °C. Las incubaciones de IdU de más de 10-15 min reducirán progresivamente la resolución de las poblaciones de las fases S y G2, ya que las células marcadas con IdU abandonan la fase S y progresan a la fase G2 o M. También hemos observado que la incubación a largo plazo con IdU puede causar la muerte celular y artefactos del ciclo celular. El procesamiento celular posterior y la tinción de anticuerpos después de la incorporación de IdU es suficiente para eliminar la IdU residual que no se incorporó a las células de fase S. No hemos observado antecedentes significativos de yodo cuando se utiliza el protocolo in vitro descrito aquí; Sin embargo, muy raramente hemos observado contaminación por yodo en muestras clínicas. Esto puede ocurrir a partir de procedimientos médicos, como el contraste de yodo en una tomografía computarizada, o de productos farmacéuticos que contienen yodo. Si se observan grandes cantidades de fondo de IdU, la muestra no debe ejecutarse para evitar daños en el detector del citómetro de masas.
- Si utiliza un estabilizador proteómico comercial, añadir 280 μL de estabilizador proteómico a la muestra resuspendida en 200 μL de PBS (1:1.4). Incubar las muestras a temperatura ambiente durante 10 min y luego colocarlas directamente en -80 °C.
3. Tinción de muestras para citometría de masas
- Retirar las muestras de los -80 °C y dejar descongelar antes de la tinción superficial.
- Si utiliza el método de fijación SmartTube, descongele las muestras a 0-4 °C para evitar una fijación adicional a medida que las muestras se calientan.
- Después de descongelar las muestras, transfiera aproximadamente 1-2 millones de células a un tubo FACS de 5 ml.
- Centrifugar el tubo FACS a 600 x g durante 5 min, y llenar el tubo FACS con medios de tinción celular (CSM) para lavar las células. Repita una vez más.
- Si se sabe que las células se adhieren entre sí, agregue 400 U / ml de heparina a los lavados CSM para evitar el contacto de célula a célula, pero esto no es estrictamente necesario.
- Incubar las células con agente bloqueador de FC, 5 μL del agente por 100 μL de células, durante 10 min a temperatura ambiente.
- Prepare una mezcla de anticuerpos que mancharán la superficie, o porción extracelular, de las células. La mezcla total de tinción ascenderá a 100 μL por 1-2 millones de células en cada prueba. La mezcla de tinción se equilibrará en consecuencia con CSM y heparina en el cóctel y el tubo FACS.
NOTA: La adición de CSM en este paso también reducirá los artefactos de tinción no específicos8. Esta mezcla de tinción depende completamente de los objetivos de interés y del fenotipo superficial (por ejemplo, un estudio con células T utilizará una mezcla superficial de CD45, CD3, CD4, CD8, etc.). Un protocolo detallado de procesamiento y tinción de muestras se puede encontrar en Behbehani y McCarthy et al.9,10. - Agregue la mezcla de tinción superficial a las celdas e incube a temperatura ambiente con agitación continua durante 30-60 min.
- Después de la tinción, llene el tubo FACS con CSM y gire a 600 x g durante 5 min.
- Lavar dos veces más con CSM, girando la muestra a 600 x g durante 5 min y aspirando cada lavado CSM.
- Arregle los anticuerpos extracelulares agregando 1 ml de PBS con 10% de CSM y 1.5% de PFA.
- Llene el tubo FACS que contiene la mezcla PBS/CSM/PFA con CSM. Girar a 600 x g durante 5 min y aspirar el sobrenadante.
- Añadir metanol a -20 °C.
- Vortex la muestra durante 1-2 min para lograr una suspensión de una sola célula y verificar que todos los grupos celulares hayan sido resuspendidos.
- Mientras la muestra está girando lentamente, agregue rápidamente 1 ml de metanol helado usando una pipeta de 1,000 μL con una punta de filtro.
- Sostenga el tubo de FACs hacia la luz y asegúrese de que no haya grumos visibles; Es de esperar nubosidad. Cualquier grumo hará que la muestra sea inutilizable para el posterior análisis de MCM.
- Conservar la muestra a -20 °C durante 10-20 min.
- Prepare la mezcla de tinción intracelular durante este tiempo. La mezcla de tinción intracelular dependerá de los objetivos de interés. Para el análisis del ciclo celular, incluya CyclinB1, pRb, Ki67 y pHH3 en esta mezcla de tinción, pero se pueden agregar otros marcadores intracelulares según sea necesario.
- Después de 10-20 min a -20 °C, retire la muestra, agregue 1,5 ml de PBS y llene el resto con CSM.
- Centrifugar la muestra 600 x g durante 5 min, y aspirar el sobrenadante.
- Lavar dos veces más con CSM, girando la muestra a 600 x g durante 5 min y aspirando el sobrenadante cada vez.
- Después del último lavado CSM, centrifugar la muestra y dejar un volumen residual de aproximadamente 50 μL.
- Agregue la mezcla de anticuerpos preparada (típicamente agregue 50 μL de cóctel de tinción de anticuerpos para lograr un volumen de tinción final de 100 μL) a la muestra e incube en una plataforma de agitación durante 30 a 60 minutos a temperatura ambiente.
- Después de la tinción, agregue CSM y centrifugar a 600 x g durante 5 min.
- Aspirar el CSM, lavar de nuevo con CSM, girar a 600 x g durante 5 min, aspirar el CSM, luego añadir PBS.
- Después de completar la tinción intracelular, coloque las células en una solución intercaladora que fije los anticuerpos a las células y tiñe el ADN de cada célula para permitir la identificación. La solución intercaladora contiene intercalador de iridio no isotópicamente puro (pentametilciclopentadienil-Ir(III)-dipiridofenazina) añadido de la solución madre del fabricante a una concentración de 500 μM. Diluir la cepa de iridio 1:4000 en una solución de PBS y PFA al 1,5%. Agregue la solución intercaladora de iridio a 100-200 μL por millón de células para teñir uniformemente y evitar la tinción excesiva.
NOTA: El iridio en esta solución intercaladora está destinado a identificar células para la activación singlete, no debe usarse para manchas vivas / muertas. Si se desean manchas vivas/muertas, deben realizarse antes de la fijación como se señaló anteriormente y en McCarthy et al.9. - Conservar las muestras en solución intercaladora en nevera a 4 °C durante un máximo de dos semanas antes de la adquisición de la muestra en el CyTOF.
4. Funcionamiento del citómetro de masas
NOTA: La operación de citometría de masas puede ser específica de la máquina. Siempre es aconsejable consultar el manual del usuario de CyTOF antes de operar. Además, actualmente hay dos artículos de JoVE que tratan sobre la puesta en marcha y el mantenimiento de máquinas 9,11.
- Revise el nebulizador en busca de obstrucciones, grietas y otras irregularidades antes de operar el citómetro de masas.
- Conecte el nebulizador al citómetro y comience el procedimiento de calentamiento. No encienda el citómetro de masas sin el nebulizador en su lugar.
- Deje correr agua a través de las líneas de muestra una vez que el citómetro de masas haya terminado de calentarse. La cámara de pulverización debe alcanzar aproximadamente 200 °C antes de realizar el ajuste o el análisis de la muestra.
- Agua corriente durante 5-10 min. Después de 5-10 minutos, cargue la solución de ajuste y seleccione el administrador de ajuste. La solución de ajuste es una solución que contiene concentraciones fijas de metales y se utiliza para optimizar el citómetro de masa antes de la adquisición de la muestra.
- En el administrador de ajuste, seleccione Vista previa una vez que la solución de ajuste haya alcanzado un registro de aciertos de estado estable para comenzar el proceso de ajuste automatizado.
- Una vez finalizado el ajuste, cargue el muestreador con agua y permita que el agua corra a través de las líneas de muestra durante el procesamiento de la muestra. El protocolo detallado para la operación y sintonización diaria del citómetro se puede encontrar en Leipold11.
- Ocasionalmente, el ajuste automatizado no se ajustará al rendimiento óptimo de la máquina. Repita el procedimiento de ajuste para corregir esto.
- Lave la muestra con CSM una vez y con agua desionizada pura dos veces antes de la adquisición de la muestra. El lavado con agua es importante para eliminar la sal residual del PBS/CSM.
- Verifique la sensibilidad y el flujo de muestra utilizando perlas de equilibrio suministradas por el fabricante, perlas de poliestireno cargadas con concentraciones de metal conocidas.
- Cambie el modo de adquisición de ajuste al modo de captura de eventos. Establezca el límite de tiempo para detener la adquisición en 120 s. Espere 45 s antes de seleccionar Grabar.
- El citómetro de masas detendrá la adquisición de muestras automáticamente después de 120 segundos. Utilice el visor de gráficos de lluvia para comprobar la intensidad de Eu151 y Eu153.
- Perlas de equilibrio diluidas en agua desionizada pura en una proporción de 1:20.
- Antes de la adquisición de muestras, compruebe el administrador de experimentos. Utilice el administrador de experimentos para asignar nombres a los canales y agregar canales para grabar.
- Asegúrese de que se agrega el canal 127-I si usa IdU.
- Tenga en cuenta que es esencial configurar el citómetro de masa para medir los parámetros necesarios (por ejemplo, IdU) antes de la adquisición de la muestra. Si los canales no se seleccionan de antemano, no se recopilarán datos de ningún canal no seleccionado y no se podrán recuperar.
- Diluir las células a una concentración de aproximadamente 1-2 x 106/ml utilizando el agua desionizada pura 1:20 y la mezcla de perlas de equilibrio. Pase las células a través del tubo FACS con tapa del filtro para eliminar cualquier grumo residual.
- Cargue la muestra y cambie el tiempo de adquisición.
- Presione Vista previa y espere a que el recuento de eventos por segundo se estabilice.
- No ejecute eventos que excedan los 400 eventos por segundo, esto conducirá a cantidades significativas de dobletes y escombros. Por lo general, recolectamos al menos 20,000 a 50,000 eventos celulares, pero el número óptimo dependerá del diseño experimental. La tinción de hasta 2 millones de células producirá típicamente de 300,000 a 400,000 eventos celulares. Tenga en cuenta que no todos los eventos serán celdas (habrá eventos de escombros y cuentas incluidos en el recuento de eventos).
- Una vez que se realiza la adquisición de la muestra, cargue una solución de lavado, comience la inducción de la muestra y corra durante 5-10 minutos. Después de 5-10 minutos, detenga la inducción de la muestra y haga correr el agua durante 10-20 minutos. La solución de lavado es una solución débil de ácido fluorhídrico diseñada para eliminar el metal residual de las líneas de muestra.
- Apague el citómetro de masas y retire el nebulizador. El nebulizador estará caliente, tenga cuidado durante el manejo.
5. Análisis de datos
- Para eliminar las perlas y también para corregir la desviación de la señal durante la adquisición de muestras, normalice los archivos FCS utilizando el software Fluidigm o la aplicación desarrollada por Finck12.
- Cargue el FCS en Cytobank u otro software de análisis de citometría de flujo. Los archivos FCS se pueden usar en cualquier software compatible, para los fines de este protocolo, se han realizado todos los análisis de acceso y análisis adicionales en Cytobank13.
- Antes de que se puedan dibujar las puertas del ciclo celular, excluya cualquier doblete o desecho celular del análisis posterior, esto se puede hacer utilizando la gráfica biaxial de Longitud del evento vs 191-Ir (Figura 1a). Las células formarán una población distinta ybrillante que se puede usar para excluir dobletes y desechos. Esta es la puerta singlete. Este método de activación generalmente elimina alrededor del 50-60% de los eventos de celdas dobles, por lo que es posible que se requieran estrategias adicionales para eliminar los eventos de celda doblete restantes.
- Cambie la escala de longitud del evento (mínimo y máximo) para que las celdas aparezcan más prominentes para ayudar en la clasificación singlete.
- Elimine aún más los dobletes y los residuos mediante el uso de parámetros gaussianos, Residual y Offset. Un residuo más alto con un desplazamiento más bajo también es escombros y dobletes, y la activación alrededor de esta población puede eliminar aún más dobletes y escombros (Figura 1b, c).
- Puerta de fase S: la fase S es la puerta más fácil de dibujar, pero también la más importante. Dibuje esta puerta usando una gráfica biaxial de IdU vs pRb, Ki67 o cyclinB1. Las células IdU+ de fase S formarán una población distinta al observar estos gráficos biaxiales (Figura 2b).
- G0/G1-phase, G2/M-phase gating – Establezca las puertas de fase G0/G1 y G2/M en la gráfica IdU vs CyclinB1 y el uso de la incorporación IdU es crucial para establecer el límite entre las puertas de fase G0/G1 y G2/M. La fase G0/G1 será CyclinB1 baja/IdU- y la fase G2/M seráCyclinB1 alta/IdU-. Una buena tinción de CyclinB1 mostrará una población natural entre las poblaciones G0-G1 y G2-M; Sin embargo, esto variará según los tipos de muestra y células en condiciones experimentales. En condiciones experimentales donde la distribución del ciclo celular puede verse afectada y hay menos separación entre la fase G0 / G1 de CyclinB1 y la fase G2 / M, el uso de la fase S permitirá una activación consistente para tipos de células particulares en cada experimento específico. Este método se detalla a continuación.
- Trazar solo las células de fase S en CyclinB1 vs IdU para ayudar a establecer la separación entre la fase G0 / G1 y la fase G2 / M. Dibuje una puerta en la poblaciónalta de CyclinB1 y ajuste hasta que aproximadamente el 5% superior de la población de la fase S esté dentro de la puerta (Figura 2c). Esto establece el punto de ruptura entre las compuertas de fase G0/G1 y G2/M (Figura 2e,f). La población activa se cambiará a la población de interés y la porción que reside dentro de la puerta anterior será la población de la fase G2 / M, mientras que el resto será la población de la fase G0 / G1.
- G0-phase gating – Establezca la fase G0 en la gráfica pRb vs IdU. La fase G0 estará representada por una poblaciónpRb baja/IdU- . La población ciclista activa tendrá una alta expresión de incorporación de pRb e IdU, la puerta de fase G0 se puede dibujar en este límite, ya que normalmente se expresa en dos poblaciones distintas (Figura 2g,i).
- Defina la fase G0 haciendo que la población de fase S dibujada previamente (Figura 2b) sea la población activa y dibujando una puerta que incorpore el 90-100% superior de la poblaciónalta de pRb. Esta es la población ciclista de pRb+ (Figura 2i), la poblaciónbaja de pRb fuera de esta puerta es la población G0 (Figura 2j).
- Si pRb no está disponible o no se puede registrar, use Ki67 vs IdU para establecer la población de fase G0. Dibujando una puerta que represente a la mayoría de la población de la fase S y usando esa puerta como límite para el Ki67 vs IdU, el resto de la población será la fase G0 (Figura 3a).
- Activación de fase M: establezca la fase M en la gráfica biaxial IdU vs pH3. La fase M representa una fracción muy pequeña de las células y está cerrada en la poblaciónalta de pH 3 (Figura 2d).
- La incorporación de IdU falló o no fue posible: si IdU no está disponible, defina el ciclo de celdas y no las fracciones de ciclo utilizando Ki67 y pRb. Ki67 y pRb, en condiciones normales, forman dos poblaciones distintas: Ki67 alto/pRbalto y Ki67 bajo/pRbbajo. La población doble positiva representa la población ciclista activa, correlacionándose con la fase G1, la fase S, la fase G2 y la fase M. La población doble baja representa la población no ciclista, correlacionándose con la fase G0 (Figura 3b).
NOTA: No es posible delinear cada fase individual utilizando el Ki67 vs pRb, pero se pueden determinar los efectos experimentales en las poblaciones relativas de ciclismo / no ciclismo. - Análisis del ciclo celular: una vez que se hayan establecido las puertas, exporte los valores numéricos de las puertas para su posterior análisis. Los porcentajes en cada ciclo se pueden lograr restando las poblaciones individuales de las poblaciones combinadas. La fase G0, dibujada en el porcentaje de puertade IdU bajo bajo de pRb, se puede restar de la fase G0 / G1, dibujada en CyclinB1bajoIdU neg, para encontrar el porcentajede fase G1. Del mismo modo, el porcentaje de fase G2 se deriva de la sustracción de la compuerta de fase M de la puerta de fase G2/M. Esto generará valores numéricos para cada fase individual del ciclo celular; G0, G1, S, G2 y M. Los valores numéricos generados para cada fase individual del ciclo celular se pueden utilizar para análisis adicionales, como gráficos y análisis estadísticos.
Resultados
Utilizando células HL-60 y un aspirado de médula ósea humana, es posible mostrar cómo las condiciones experimentales pueden afectar la distribución y el análisis del ciclo celular. En primer lugar, se debe establecer la estrategia de compuerta para demostrar cómo se derivan las fases del ciclo celular. En la Figura 1 mostramos el establecimiento de la puerta singlete, que es importante para separar los desechos celulares y los dobletes, estableciendo una población unicelular. Para las líneas celulares, la puerta singlete es todo lo que se necesita para pasar al análisis del ciclo celular (Figura 2a). Para las muestras humanas, las poblaciones inmunofenotípicas generalmente deben establecerse antes del análisis del ciclo celular, ya que los límites exactos de cada puerta del ciclo celular pueden variar según los diferentes tipos de células. Una vez que se han establecido las poblaciones (generalmente mediante marcadores de superficie que definen esa población) será necesario establecer las puertas del ciclo celular. La Figura 2b muestra el establecimiento de la fase S en la gráfica biaxial IdU vs CyclinB1. Esta gráfica también se utiliza para establecer el límite de la puerta de fase G2/M (Figura 2f). Una vez establecida la fase G2/M, el resto es la compuerta de fase G0/G1 (Figura 2f). El IdU vs pRb se utiliza para establecer primero la población ciclista pRb+ estableciendo una puerta en IdU que incorpora celdas (Figura 2g,i). La población de pRb+/IdUneg fuera de esta puerta es la fase G0 (Figura 2j). La fase M se establece en la IdU vs pHH3 donde las células de la fase M expresan altos niveles de pHH3 y no exhiben incorporación de IdU (Figura 2k). En el caso de que no se incluya pRb, la fase G0 se puede replicar utilizando Ki67 de manera similar al método descrito anteriormente (Figura 3a). Si la incorporación de IdU falló o no se realizó, todavía es posible determinar las fracciones cíclicas relativas utilizando Ki67 y pRb. Mediante el uso del Ki67 y pRb biaxial se forman dos poblaciones distintas, una población pRb+/Ki67+ doble positiva y una poblaciónpRb baja/Ki67baja. La población doble positiva representa células en ciclo, mientras que la baja representa células que no están en ciclo (Figura 3b). Usando IdU que incorpora células y célulasbajas de pRb sin incorporación de IdU, mostramos que la fase S se encuentra principalmente en la población pRb + / Ki67 +, mientras que la fase G0 se encuentra principalmente en la población baja de pRb / Ki67 baja.
El análisis del ciclo celular se basa en una buena técnica experimental, especialmente durante la etapa de incubación de IdU. Si bien la incorporación de IdU es flexible (siendo aplicable en cultivos celulares, aspirados de médula ósea e incluso estudios murinos), es necesario realizar la incorporación y fijación de IdU sin interrumpir el estado del ciclo celular de interés experimental. El etiquetado de IdU y, por lo tanto, el análisis del ciclo celular posterior pueden verse afectados significativamente por el tiempo y la temperatura, como se indica en la Figura 4. Las células que permanecen demasiado tiempo en recipientes cerrados o que podrían encontrarse en el envío de muestras o en el transporte de muestras entre ubicaciones, tendrán una fracción reducida de fase S y no serán precisas para el análisis del ciclo celular (Figura 4a). Sin embargo, los períodos de tiempo cortos, aquellos de menos de una hora en total, tendrán una distribución normal del ciclo celular, lo que indica que el transporte rápido puede no ser un análisis negativo del ciclo celular (Figura 4b). Otro modificador importante es la criopreservación que se utiliza rutinariamente en la mayoría de los laboratorios. Al examinar el estado del ciclo celular en células criopreservadas, puede ser necesario un largo período de equilibrio antes de que las células vuelvan al ciclo celular activo, que aún puede no reflejar el estado del ciclo celular previo a la ciropreservación (Figura 4c).
Las muestras humanas primarias a menudo son compuestos de múltiples tipos de células diferentes, estos diferentes tipos de células pueden tener diferentes sensibilidades al procesamiento, lo que lleva a diferentes ciclos celulares. En dos aspirados de médula ósea que fueron marcados inmediatamente con IdU, almacenados durante 30 minutos antes del etiquetado de IdU o almacenados criogénicamente después de la separación de Ficoll, existen diferencias entre cada muestra y población (Figura 5a, b). Se examinaron dos poblaciones inmunofenotípicas para detectar diferencias en la incorporación de IdU; Células T (CD45 alto/CD3alto) y monoblastos (CD33+, HLADR+, CD11bbajo, CD14neg). Con la combinación correcta de marcas de superficie, es posible examinar más poblaciones inmunofenotípicas. En la médula #2 hubo un notable efecto de activación de células T después de 30 minutos de almacenamiento que no se observó en los monoblastos del mismo paciente (Figura 5b). Al igual que las células cultivadas, hubo cambios notables en el etiquetado de IdU después del almacenamiento criogénico que también dependía de la población. La médula # 1 tuvo una reducción en la población de células T, pero un aumento en las fracciones marcadas con IdU de monoblastos en comparación con la línea de base (Figura 5 a, b), la médula # 2 mostró una reducción tanto en las células T como en los monoblastos en comparación con la línea de base (Figura 5a, b). Las células congeladas requieren un período de incubación notable antes de volver al estado normal del ciclo celular y esto puede influir en los estudios que se basan en la modificación del estado del ciclo celular o el estado del ciclo celular como una métrica del fármaco o efecto experimental.
Otro beneficio de MCM es la capacidad de discriminar células en detención del ciclo celular o que tienen una distribución anormal del ciclo celular. Si bien los colorantes de ADN comúnmente utilizados en la citometría de flujo pueden discriminar entre el contenido de ADN 2N y 4N, son muy brillantes, lo que puede complicar enormemente la medición de otros parámetros de ese láser. IdU, sin embargo, sólo toma un canal de masa y tiene un derrame mínimo, lo que permite que se utilicen otros marcadores en la determinación del ciclo celular. Las células MOLM13 que fueron irradiadas muestran una disminución de la incorporación de IdU y una disminución en la fase M en comparación con las células de control (Figura 6). La interrupción de los puntos de control normales del ciclo celular podría alterar el estado aparente del ciclo celular por MCM. Al observar las poblaciones de pH2AX y cPARP en las células no irradiadas, la población de pH2AX baja y cPARPbaja muestra una distribución normal del ciclo celular, mientras que las células que expresan niveles más altosde pH2AX o cPARP se localizan principalmente en la fase G0 / G1 que se espera (Figura 6a). Sin embargo, en las células irradiadas, el pH2AX bajo y el cPARPbajo, las células están casi completamente localizadas en la fase G0, mientras que las células pH2AXalta y cPARPbaja muestran un fenotipo de detención del ciclo celular con incorporación de IdU y localización a la fase G0 / G1 y la fase G2 con ausencia de fase M. Las células pH2AXalta y cPARPalta también muestran células que incorporan algo de IdU y se localizan en la fase G0 / G1 indicativo de daño por radiación (Figura 6b).
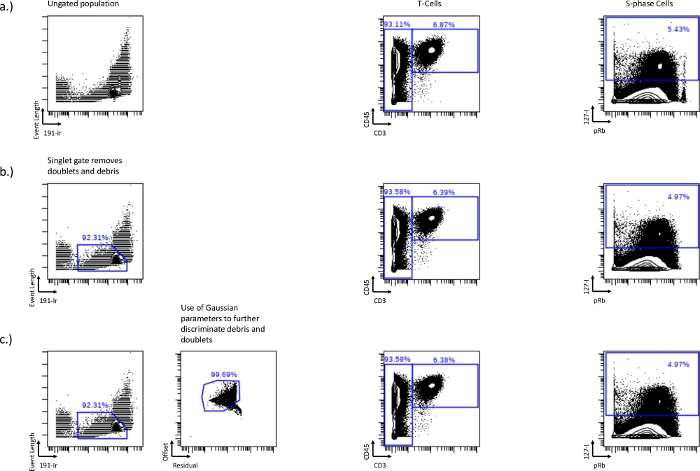
Figura 1: Establecimiento de las puertas singlete usando 191-Ir por longitud de evento y también parámetros gaussianos, residual y offset.
Las diferencias en células T (CD45+/CD3+) y fase S (IdU+) entre una muestra no cubierta (a), una longitud de evento vs 191-Ir puerta singlete (b), o una puerta singlete combinada con parámetros gaussianos, residual y offset (c). La puerta singlete elimina los desechos, dobletes y cuentas que se muestran en la pérdida de la poblaciónalta de pRb en la esquina derecha del biaxial. Esta puerta singlete se puede optimizar aún más mediante la inclusión de parámetros gaussianos como residual y offset, eliminando más residuos. Haga clic aquí para ver una versión más grande de esta figura.
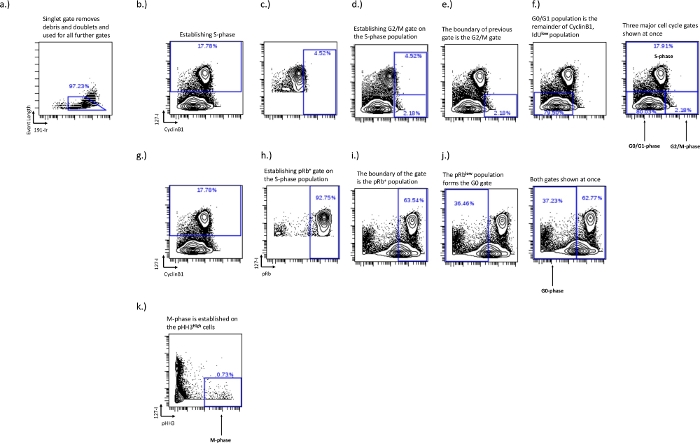
Figura 2: El esquema de compuerta para establecer puertas de ciclo celular para las fases G0, G1, S, G2 y M utilizando IdU, CyclinB1, pRb y pHH3.
La puerta singlete se establece para eliminar dobletes y escombros (a). La fase S debe establecerse (b), una vez establecida la fase S, la población IdU+ se puede utilizar para establecer el límite de la fase G2/M (c,d). El establecimiento del límite de la fase G2/M establece los límites de la población de la fase G0/G1 (f). La población de fase pRb+ y G0 se establece en el IdU vs pRb biaxial. Las celdas IdU+ (h) se utilizan para establecer el límite para la población pRb+ (i). El límite de la población pRb+ establece el límite de la población de fase G0 (j). La fase M se establece en células pHH3+ que son IdU- (k). Haga clic aquí para ver una versión más grande de esta figura.
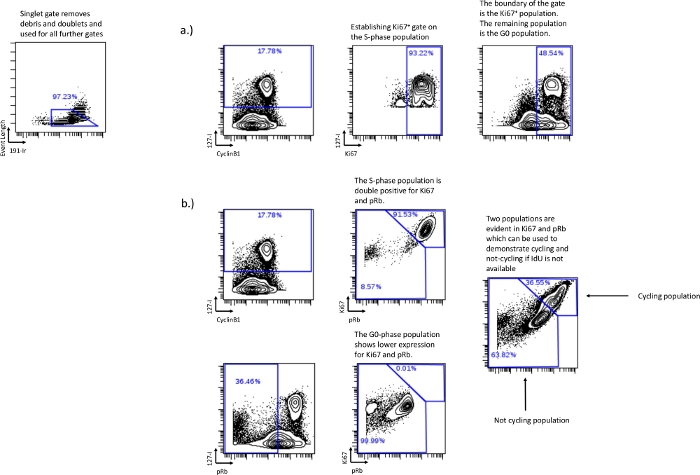
Figura 3: Establecimiento de puertas de ciclo celular sin el uso de pRb o sin el uso de la incorporación de IdU.
El dibujo de la fase G0 también se puede hacer usando Ki-67 siguiendo la misma estrategia de compuerta que se usó con pRb, si pRb no está incluido en el experimento (a). Si la incorporación de IdU falló o no se realizó, es posible recuperar las fracciones de ciclo de células relativas mediante el uso de Ki67 y pRb. La expresión doble positiva de Ki67 y pRb es correlativa a las células en ciclo, como lo demuestra la demostración de que las células IdU + se encuentran principalmente en la población doble positiva (b). La población baja de Ki67 y pRb se correlaciona con la población de fase G0 o no ciclista demostrada por las célulasneg de pRb baja / IdU que se encuentran en la poblaciónbaja de Ki67 y pRb. Este método no puede discriminar fases individuales del ciclo celular, pero aún puede usarse para determinar fracciones cíclicas relativas en condiciones experimentales. Haga clic aquí para ver una versión más grande de esta figura.

Figura 4: Figuras representativas del efecto de diferentes condiciones de almacenamiento en la distribución del ciclo celular de las células HL-60.
Las células se incubaron durante una hora seguida de una hora de reposo a las condiciones de temperatura establecidas en tubos sellados (a). Hay efectos notables en la distribución del ciclo celular en comparación con el control. Las células HL60 se mantuvieron en tubos sellados a temperatura ambiente durante 30 minutos, una situación que puede ocurrir en un entorno clínico, antes de la incorporación de IdU (b). Los tubos sellados mantenidos a temperatura ambiente durante 30 minutos no mostraron diferencias apreciables en el ciclo celular. El efecto del almacenamiento criogénico se investigó sobre el ciclo celular en células HL60, donde se tomó una muestra antes del almacenamiento criogénico y una muestra tomada después de una hora de reposo después de una semana en almacenamiento criogénico (c). Una hora después del deshielo, la distribución del ciclo celular se ve afectada y la distribución del ciclo celular no vuelve a la normalidad hasta aproximadamente una semana después del deshielo. Haga clic aquí para ver una versión más grande de esta figura.

Figura 5: El procesamiento puede tener un efecto en las muestras de pacientes y se muestran imágenes representativas entre dos pacientes diferentes que muestran (a) células T (CD45 alto/CD3alto) y (b) monoblastos (CD33+, HLADR+, CD11bbajo, CD14neg).
Entre la médula uno y la médula dos, está claro que en la médula 2 hubo un efecto de activación de las células T durante los 30 minutos de descanso, mientras que la médula uno no tuvo tal efecto. La médula uno mostró un posible efecto de activación en la población de monoblastos después de 30 minutos, mientras que la médula dos no lo hizo. En ambos tuétanos, sin embargo, estaba claro que el almacenamiento criogénico afectaba la distribución del ciclo celular independientemente del tipo de célula. Haga clic aquí para ver una versión más grande de esta figura.

Figura 6: Las células MOLM13 se dejaron como controles o se irradiaron utilizando un irradiador de rayos X a 10Gy.
Las células MOLM13 muestran la distribución del ciclo celular en cuatro poblaciones diferentes de expresión de pH2AX y cPARP. Las células de control muestran tinción mínima de pH2AX y cPARP, con características normales del ciclo celular que se muestran en la población baja de pH2AX y cPARPbaja (a). Mientras que las células irradiadas muestran una distribución anormal del ciclo celular con la mayoría de las células cíclicas ubicadas en elpH 2AX alto y cPARPalto, lo que indica la interrupción del ciclo celular (b). Las células no dañadas, pH2AX bajo y cPARPbajo, muestran una falta de características cíclicas que se encuentran principalmente en la fase G0. Sin estos marcadores, estas células aparecerían como células 4N y 2N en la citometría de flujo normal, posiblemente confundiendo el análisis del ciclo celular aguas abajo. Haga clic aquí para ver una versión más grande de esta figura.
Discusión
Los ejemplos presentados aquí demuestran cómo utilizar una plataforma MCM para analizar la distribución del ciclo celular. También se ha demostrado que el análisis del ciclo celular es sensible a condiciones experimentales como el tiempo y la temperatura, lo cual es una consideración importante que los investigadores deben tener al considerar MCM para su análisis del ciclo celular14. Las muestras almacenadas durante un corto período de tiempo, no más de una hora, tendrán una incorporación de IdU comparable a su estado normal. Las muestras en un sistema cerrado durante largos períodos de tiempo, aproximadamente 2 horas, tendrán una incorporación reducida de IdU, sin embargo, las fracciones cíclicas y no cíclicas relativas no cambiarán permitiendo el análisis del ciclo celular grueso. El almacenamiento criogénico y la posterior descongelación son perjudiciales para la distribución normal del ciclo celular durante un período de tiempo significativo. Se ha observado previamente que el almacenamiento criogénico interrumpe la distribución de proteínas y ARN, pero solo recientemente se ha demostrado que interrumpe el ciclo celular14,15,16,17. Tomados en conjunto, estos indican que si se anticipan largos tiempos de almacenamiento o preservación criogénica de muestras, sería mejor teñir las células con IdU antes del almacenamiento o almacenamiento criogénico, arreglar las células y almacenarlas hasta que se pueda realizar el análisis. Como el análisis del ciclo celular por MCM no requiere células vivas, esto permitiría a los investigadores almacenar muestras preciosas y realizar un análisis preciso del ciclo celular aguas abajo.
El análisis del ciclo celular por MCM es un sistema robusto capaz de interrogar profundamente modelos experimentales. Debido al alto recuento de parámetros de MCM, aproximadamente 40-50 canales de masa, el análisis del ciclo celular se puede combinar con otros marcadores intracelulares o extracelulares evitando la necesidad de clasificar las células en función del inmunofenotipo, lo que puede causar la pérdida de efectos significativos del ciclo celular. La naturaleza de alto parámetro de MCM se presta para examinar los efectos en aplicaciones de mapeo de alta dimensión como SPADE y viSNE. Si bien SPADE y viSNE se usan típicamente para definir poblaciones inmunofenotípicas que luego pueden examinarse para detectar cambios en el ciclo celular, también sería posible mapear los marcadores del ciclo celular. Dependiendo de las condiciones experimentales, el mapeo de marcadores del ciclo celular en un espacio de alta dimensión puede mostrar la correlación del ciclo celular con el efecto del fármaco o qué poblaciones inmunofenotípicas pueden estar localizando en cada estado del ciclo 3,5. Si bien MCM puede estar limitado por la falta de colorantes fluorescentes de unión al ADN, esto se compensa con la incorporación directa de IdU durante las células de fase S y las proteínas del ciclo celular intracelular se pueden usar para determinar el estado del ciclo celular. Estas proteínas del ciclo celular también pueden ayudar a discriminar entre las etapas de la detención del ciclo celular que de otro modo aparecerían como 3N o 4N en el flujo tradicional utilizando tintes de unión al ADN. Sin embargo, tales sistemas altamente paramétricos no están exentos de desventajas, y son sensibles a las interrupciones en el ciclo celular. Hemos demostrado que los largos tiempos de almacenamiento y el almacenamiento criogénico pueden afectar significativamente la distribución del ciclo celular. Esto es especialmente importante cuando se trata de racionalizar los efectos experimentales de los fármacos que pueden afectar la distribución del ciclo celular. El tratamiento de fármacos diseñados para afectar el ciclo celular en muestras primarias congeladas puede proporcionar datos falsos sobre el efecto del ciclo celular cuando se usa inmediatamente después de la descongelación del almacenamiento criogénico. MCM es una tecnología versátil para el análisis del ciclo celular que se puede aplicar a una serie de modelos experimentales y es especialmente adecuada para el perfil profundo de sistemas heterogéneos. Al igual que con otros métodos altamente paramétricos, es necesario tener un experimento cuidadosamente diseñado con consideraciones apropiadas sobre cómo el procesamiento y los efectos experimentales afectarán el análisis del ciclo celular.
Divulgaciones
El Dr. Behbehani recibe apoyo de viaje de Fluidigm. Fluidigm también ha comprado reactivos y materiales para uso en laboratorio.
Agradecimientos
Los autores desean agradecer los esfuerzos de Palak Sekhri, Hussam Alkhalaileh, Hsiaochi Chang y Justin Lyeberger por su apoyo experimental. Este trabajo fue apoyado por el Programa de Becas Pelotonia. Todas las opiniones, hallazgos y conclusiones expresadas en este material son las del autor (s) y no reflejan necesariamente las del Programa de Becas Pelotonia.
Materiales
| Name | Company | Catalog Number | Comments |
| Bovine Serum Albumin (BSA) | Sigma | A3059 | Component of CSM |
| Centrifuge | Thermo Scientific | 75-217-420 | Sample centrifugation |
| Cleaved-PARP (D214) | BD Biosciences | F21-852 | Identification of apoptotic cells |
| Cyclin B1 | BD Biosciences | GNS-1 | G2 Resolution |
| Dimethylsulfoxide (DMSO) | Sigma | D2650 | Cryopreservative |
| EQ Four Element Calibration Beads | Fluidigm | 201078 | Internal metal standard for CyTOF performance |
| FACS Tube w/ mesh strainer | Corning | 08-771-23 | Cell strainer to remove clumps/debris before CyTOF run |
| Fetal Bovine Serum (FBS) | VWR | 97068-085 | Cell culture growth supplement |
| Helios | Fluidigm | CyTOF System/Platform | |
| Heparin | Sigma | H3393 | Staining additive to prevent non-specific staining |
| IdU (5-Iodo-2′-deoxyuridine) | Sigma | I7125 | Incorporates in S-phase |
| Ki-67 | eBiosciences | SolA15 | Confirmation of G0/G1 |
| MaxPar Multi Label Kit | Fluidigm | 201300 | Metal labeling kit, attaches metals to antibodies |
| Microplate Shaker | Thermo Scientific | 88880023 | Mixing samples during staining |
| Paraformaldehyde (PFA) | Electron Microscopy Services | 15710 | Fixative |
| pentamethylcyclopentadienyl-Ir(III)-dipyridophenazine | Fluidigm | 201192 | Cell identification during CyTOF acquisition |
| p-H2AX (S139) | Millipore | JBW301 | Detection of DNA damage |
| p-HH3 (S28) | Biolegend | HTA28 | M-phase Resolution |
| Phosphate Buffered Saline (PBS) | Gibco | 14190-144 | Wash solution for cell culture and component of fixative solution |
| p-Rb (S807/811) | BD Biosciences | J112906 | G0/G1 Resolution |
| Proteomic Stabilizer | SmartTube Inc | PROT1 | Sample fixative |
| RPMI 1640 | Gibco | 21870-076 | Cell culture growth medium |
| Sodium Azide | Acros Organics | AC447810250 | Component of CSM/Antibody buffer, biocide |
Referencias
- Ornatsky, O., Baranov, V. I., Bandura, D. R., Tanner, S. D., Dick, J. Multiple cellular antigen detection by ICP-MS. Journal of Immunological Methods. 308 (1-2), 68-76 (2006).
- Ornatsky, O. I., et al. Study of cell antigens and intracellular DNA by identification of element-containing labels and metallointercalators using inductively coupled plasma mass spectrometry. Analytical Chemistry. 80 (7), 2539-2547 (2008).
- Behbehani, G. K., Bendall, S. C., Clutter, M. R., Fantl, W. J., Nolan, G. P. Single-cell mass cytometry adapted to measurements of the cell cycle. Cytometry A. 81 (7), 552-566 (2012).
- Raval, A., et al. Reversibility of Defective Hematopoiesis Caused by Telomere Shortening in Telomerase Knockout Mice. PLoS One. 10 (7), 0131722 (2015).
- Behbehani, G. K., et al. Mass Cytometric Functional Profiling of Acute Myeloid Leukemia Defines Cell-Cycle and Immunophenotypic Properties That Correlate with Known Responses to Therapy. Cancer Discovery. 5 (9), 988-1003 (2015).
- Kimmey, S. C., Borges, L., Baskar, R., Bendall, S. C. Parallel analysis of tri-molecular biosynthesis with cell identity and function in single cells. Nature Communications. 10 (1), 1185 (2019).
- Rabinovitch, P. S., Kubbies, M., Chen, Y. C., Schindler, D., Hoehn, H. BrdU-Hoechst flow cytometry: a unique tool for quantitative cell cycle analysis. Experimental Cell Research. 174 (2), 309-318 (1988).
- Rahman, A. H., Tordesillas, L., Berin, M. C. Heparin reduces nonspecific eosinophil staining artifacts in mass cytometry experiments. Cytometry A. 89 (6), 601-607 (2016).
- McCarthy, R. L., Duncan, A. D., Barton, M. C. Sample Preparation for Mass Cytometry Analysis. Journal of Visualized Experiments. (122), e54394 (2017).
- Behbehani, G. K. Immunophenotyping by Mass Cytometry. Methods in Molecular Biology. 2032, 31-51 (2019).
- Leipold, M. D., Maecker, H. T. Mass cytometry: protocol for daily tuning and running cell samples on a CyTOF mass cytometer. Journal of Visualized Experiments. (69), e4398 (2012).
- Finck, R., et al. Normalization of mass cytometry data with bead standards. Cytometry A. 83 (5), 483-494 (2013).
- Kotecha, N., Krutzik, P. O., Irish, J. M. Web-based analysis and publication of flow cytometry experiments. Current Protocols in Cytometry. , 17 (2010).
- Devine, R. D., Sekhri, P., Behbehani, G. K. Effect of storage time and temperature on cell cycle analysis by mass cytometry. Cytometry A. 93 (11), 1141-1149 (2018).
- Campos, L., et al. Expression of immunological markers on leukemic cells before and after cryopreservation and thawing. Cryobiology. 25 (1), 18-22 (1988).
- Kadic, E., Moniz, R. J., Huo, Y., Chi, A., Kariv, I. Effect of cryopreservation on delineation of immune cell subpopulations in tumor specimens as determinated by multiparametric single cell mass cytometry analysis. BMC Immunology. 18 (1), 6 (2017).
- Shabihkhani, M., et al. The procurement, storage, and quality assurance of frozen blood and tissue biospecimens in pathology, biorepository, and biobank settings. Clinical Biochemistry. 47 (4-5), 258-266 (2014).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ISSN 2578-6326
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados
Utilizamos cookies para mejorar su experiencia en nuestra página web.
Al continuar usando nuestro sitio web o al hacer clic en 'Continuar', está aceptando nuestras cookies.