Method Article
Combinação de Optogenetics com microRNAs Artificial para caracterizar os efeitos do Gene Knockdown na função pré-sináptica dentro circuitos neuronais intacta
Neste Artigo
Resumo
Este protocolo fornece um fluxo de trabalho sobre como combinar artificial microRNA mediada por RNA interferência com optogenetics para estimular especificamente pré-sináptica boutons com reduzida expressão de genes seletiva dentro de circuitos neuronais intactos.
Resumo
O propósito do presente protocolo é caracterizar o efeito de knockdown do gene na função pré-sináptica dentro circuitos neuronais intactas. Descrevemos um fluxo de trabalho sobre como combinar microRNA artificial (miR)-mediada por RNA interferência com optogenetics para conseguir a estimulação seletiva de boutons pré-sináptica manipulados em fatias cerebral aguda. A abordagem experimental envolve o uso de um único construto viral e um único promotor de neurônio-específico para dirigir a expressão de uma sonda de optogenetic e o miR(s) artificial contra genes pré-sináptica. Quando Estereotaxia injetado na região do cérebro de interesse, a construção expressa torna possível estimular com luz exclusivamente os neurônios com expressão reduzida dos genes sob investigação. Esta estratégia não exige o desenvolvimento e manutenção de linhas de rato geneticamente modificado e pode em princípio ser aplicada a outros organismos e a qualquer gene neuronal de escolha. Temos recentemente aplicado para investigar como o nocaute de isoformas de splice alternativas de canais de cálcio dependentes de voltagem P/Q-tipo pré-sináptica (VGCCs) regula a curto prazo plasticidade sináptica no CA3 para CA1 sinapses excitatórias em fatias hippocampal agudas. Uma abordagem semelhante também pode ser usada para manipular e sonda o circuito neuronal em vivo.
Introdução
Este protocolo descreve uma nova abordagem para caracterizar o efeito de knockdown do gene na função pré-sináptica dentro circuitos neuronais intactas. Investigando a função pré-sináptica em circuitos neuronais intactos é um desafio porque muitos boutons pré-sináptica são muito pequenas e longe do soma para permitir intervenções moleculares e eletrofisiológicas combinadas. Apesar de interferência do RNA oferece um meio poderoso e flexível para proteínas sinápticas "knockdown"1, esta abordagem tem sido usada frugalmente para investigar a função pré-sináptica, porque é difícil detectar os efeitos da derrubada usando tradicional estímulos elétricos, que não distinguem entre manipularam e ingênuo boutons pré-sináptica2. Aqui, descrevemos como combinar microRNA artificial (miR)-mediada por RNA interferência com optogenetic recentemente desenvolvido a tecnologia para obter estimulação seletiva de boutons pré-sináptica manipulados em fatias cerebral aguda.
Enquanto camundongos knockout condicional em combinação com a eletrofisiologia também podem ser usados para investigar a função das proteínas pré-sináptica3,4, nossa estratégia não exige o desenvolvimento e manutenção de geneticamente modificado em linhas de rato e também podem ser facilmente empregadas para derrubar isoformas específicas de um gene. Relativo a mais comumente usado gancho de cabelo curto RNAs (shRNAs), miRs artificiais, que empregamos aqui, oferecem importantes vantagens para a nocaute em neurônios. Ao contrário de shRNAs, possam ser expressos sob o controle de uma polimerase II promotor1. Assim, um único promotor pode ser usado para dirigir a expressão da miR e uma sonda de optogenetic, junto com um repórter fluorescente. Desta forma, o tamanho da construção pode ser mantido dentro dos limites de empacotamento de vírus adeno-associado recombinantes (rAAV, figura 1A). Além disso, o uso de uma construção única e um único promotor reduz a variabilidade experimental porque permite a expressão da miR, a sonda de optogenetic e o repórter fluorescente em uma proporção fixa.
Temos recentemente aplicado essa tecnologia para examinar o papel das isoformas alternativamente emendados de canais de cálcio pré-sináptica no hipocampo5. Tal estratégia é geralmente aplicável para estudar a relevância fisiológica de outras proteínas pré-sinapticamente expressas em qualquer circuito de cérebro de interesse.
Protocolo
Todos os experimentos foram realizados em conformidade com as diretrizes estabelecidas pelo Conselho das Comunidades Europeias (Directiva 2010/63/UE, de 4 de março de 2014) e foram aprovados pelo Ministério da saúde italiano.
1. projeto de microRNAs para avaliação de sua eficiência em sistemas de expressão heteróloga e interferência do RNA
Nota: Este protocolo requer o conhecimento dos métodos a seguir bem estabelecidos: clonagem molecular, sequenciamento de DNA, manutenção de linhas celulares, fosfato de cálcio transfeccao, quantitativa real tempo PCR (qRT-PCR), preparação de lisados celulares da linha celular e mancha ocidental.
- Determine se o gene de interesse é objecto de splicing alternativo. Para um nocaute geral do gene, selecionar exclusivamente constitutivas exões; para um nocaute de uma isoforma específica alternativamente emendado, selecione o exon alternativamente emendado relevante.
- Use software dedicado (por exemplo, https://rnaidesigner.thermofisher.com/rnaiexpress/) para a concepção artificiais miRs para RNA interferência contra a sequência de interesse.
Nota: É importante usar uma base Local alinhamento pesquisa ferramenta (explosão) para verificar para fora-alvos possíveis dentro da mesma espécie. - Selecione os top três miRs classificados para o gene alvo.
- Encomendar os fios superior e inferior dos miRs selecionados de uma empresa adequada. Para controle negativo, use uma versão codificada de um os miRs selecionados ou um miR previu não para atingir qualquer gene conhecido da espécie utilizada no experimento (ex. para genes de vertebrados, o miR presente no pcDNA6.2-GW/EmGFP-miR-neg).
- Recoze os respectivos fios superior e inferior dos miRs selecionados e cloná-los em um plasmídeo projetado para expressão de miRs (por exemplo, pcDNA6.2-GW/EmGFP-miR) utilizando estratégias de clonagem padrão.
Nota: O objectivo é criar uma gaveta de expressão miR consistindo de uma região flanqueando do miR 5', a sequência selecionada miR e uma 3' miR flanqueando região que pode ser expressa da 3' UTR de um gene repórter sob o controle de um promotor II de tipo de RNA polimerase. - Use o sequenciamento de DNA para validar a inserção correta dos miRs selecionados.
- Desenvolvem-se células HEK293 em uma incubadora de cultura celular umidificado (37 ° C, 5% CO2) usando DMEM meio suplementado com 10% de soro bovino fetal, 1mm NaPyruvate, 10 mM HEPES (pH 7.39) e 1 x penicilina/estreptomicina (caneta/Strep).
- Quando as células HEK293 são ~ 70% confluente, co transfect-los com cada um dos vetores de expressão a miR e um vetor que expressa o gene alvo na proporção 1:1 DNA usando o método de fosfato de cálcio6. Incluir os seguintes controles: (i) untransfected células, (ii) células transfectadas com o vetor que expressa o gene alvo juntamente com qualquer operadora de DNA (por exemplo, pBluescript II SK(+)) ou (iii) controle de miR.
Nota: (i) o gene alvo expressado deve ser da mesma espécie para a qual foram projetados os miRs, a menos que as sequências alvejadas pelos miRs são absolutamente conservadas no nível do nucleotide entre as duas espécies. (ii) célula linhas diferente HEK293 podem ser usadas. (iii) alternativos métodos do transfection, tais como electroporation ou métodos baseados em lipossomas, podem ser usados. -
48 h após a transfeccao, lisar as células, funcione o gel de proteína, executar o borrão ocidental e analisar o teor de proteínas com um anticorpo que reconhece a proteína do alvo. Aponta para uma "knockdown" eficiência de ≥ 50%.
Nota: Knockdown eficiência pode, em alternativa, ou além disso, ser avaliados (i) pela análise de ELISA, (ii), medindo a atividade da proteína alvo, se for o caso (por exemplo, densidades de corrente para canais iônicos) ou (iii) por qRT-PCR no nível de mRNA se apropriado os anticorpos não estão disponíveis (etapa 3 do protocolo).- Coloque os pratos de cultura no gelo e lavar as células uma vez com gelada fosfato salino (PBS).
- Aspire a PBS, em seguida, adicione o amortecedor de RIPA gelado (50 mM Tris-HCl pH 7,4, 150 mM NaCl, 2 mM EDTA, 1% NP40, 0,1% sulfato dodecyl de sódio (SDS), contendo inibidores de protease e fosfatase; 1 mL para um prato de Ø 100 mm, 0,5 mL para um prato de Ø 60 mm).
- Raspar o prato com uma espátula de célula células aderentes e suavemente transferir a suspensão de células para um tubo de microcentrifugadora pre-cooled.
- Centrifugar o tubo a 15.000 x g, durante 15 min a 4 ° C, transferir o sobrenadante em um novo tubo de microcentrifuga pre-cooled e descartar a pelota.
- Determine a concentração de proteína usando o kit de ensaio da proteína de BCA ou outro método adequado (por exemplo, ensaio de Bradford).
Nota: As amostras podem ser congeladas a-20 ° C ou -80 ° C para uso posterior, ou processadas imediatamente. - Transferi um volume adequado de lisados para microcentrifuga tubos para que todas as amostras têm a mesma concentração de proteína e adicionar tampão de Lise gelada adequada para compensar todos os lysates para o mesmo volume.
Nota: 30 a 50 µ g de proteína total deve ser suficiente para a maioria das proteínas, mas a quantidade adequada a ser carregado deve ser determinada de acordo com a abundância da proteína de interesse. - Adicionar uma quantidade adequada de buffer de x Laemmli 2 (SDS de 4%, 20% de glicerol, 10% 2-Mercaptoetanol, 0,004% bromofenol, 0.125 M Tris-HCl, pH 6,8) e ferver as amostras a 95 ° C por 5 min.
- Carrega as amostras em um gel do acrilamido juntamente com um marcador de peso molecular. Funcione o gel a 100 V para 1-2 h.
Nota: A percentagem do gel depende do tamanho da proteína de interesse. - Montar o sanduíche de transferência e transferir as proteínas do gel para uma membrana a 100 V por 2 h. A membrana pode ser nitrocelulose ou PVDF. Ativar o PVDF com metanol para 1 min e enxague com buffer de transferência antes de preparar a pilha.
- Bloquear a membrana por 1h à temperatura ambiente usando tampão de bloqueio (5% de leite em PBST (PBS + 0.1% Tween-20)), então incubar a membrana durante a noite a 4 ° C com anticorpo primário, diluído em tampão de bloqueio.
- Lavar a membrana por 10 min em PBST três vezes e incube-lo com anticorpo secundário conjugado HRP em tampão de bloqueio por 1h à temperatura ambiente.
- Lavar a membrana por 10 min em PBS quatro vezes, em seguida, aplicar o substrato quimioluminescente para a membrana e capturar os sinais quimioluminescente usando um sistema de câmera com base em imagem CCD.
- Use o software de análise de imagem para quantificar a eficiência "knockdown".
- Selecione os dois miRs mais eficientes visando sequências não-sobreposição para mais ensaios funcionais.
Nota: Efeitos Off-alvos podem nunca ser inteiramente descartados por qualquer miR. No entanto, se dois miRs independentes têm um efeito semelhante, é extremamente improvável que isso é por causa de uma mesma derrubada fora do alvo. - Se a eficiência knockdown os miRs selecionados não é satisfatória (< 50%), tela para outras miRs. Alternativamente, expressam o miR(s) mais eficiente em conjunto, isto é, expressá-los em várias cópias da mesma cassete de expressão, que pode resultar no aumento da eficiência "knockdown"7.
2. construção de vetores recombinantes Adeno-associado para expressão combinada de Optogenetic sondas e microRNAs
Nota: Este protocolo requer o conhecimento dos métodos a seguir bem estabelecidos: clonagem molecular, a sequenciação de ADN e a produção de rAAV.
- Cada clone das fitas de expressão miR mais eficientes para o 3' UTR de construções projetadas para produção de rAAVs de recombinação e expressão de excitatório optogenetic sondas, como em pAAV-Syn-ChETA-TdT-miR-X (figura 1A), onde o synapsin promotor neurônio específico conduz a expressão da channelrhodopsin ultra rápida ChETA, o repórter fluorescente vermelha TdTomato e miR(s), inserido entre os sites NheI e EcoRI5.
Nota: (i) não ultrapasse o limite de empacotamento de rAAVs, que é aproximadamente 4,7 Kb no comprimento do ITR (repetição invertida de terminal) para ITR (figura 1A, laranja). (ii) a fita de expressão miR precisa ser inserido entre o códon de parada e WPRE (elemento regulamentar Posttranscriptional do vírus de hepatite marmota; Figura 1A). (iii) a inclusão de uma proteína fluorescente, tais como TdTomato, é extremamente útil, pois permite facilmente monitoramento da localização e intensidade da infecção (etapa 4 do protocolo). - Use o sequenciamento de DNA para validar a inserção correta das fitas da miR.
- Produzir e título rAAV1/2, de acordo com o protocolo anteriormente publicado8, rAAV1/um 2 para cada selecionado miR. Um experimento típico para a derrubada de um gene que incluem dois miRs independentes contra o mesmo gene e o controle de um miR. Apontar para um título de vírus de ≥ 109 genomas virais (vg) / µ l.
Atenção: rAAVs precisa ser manipulado em uma instalação de biossegurança nível 1 (BSL-1). Por favor, verifique com a Comissão de biossegurança institucional para obter informações detalhadas.
Nota: Se o laboratório não tiver uma instalação viral ou títulos virais não são satisfatórios, rAAV produção pode ser terceirizada. Por exemplo, visite https://www.med.upenn.edu/gtp/vectorcore/ ou https://vcf.charite.de/en/metas/.
3. extração de RNA de culturas Neuronal preliminares para avaliação da eficiência de Genes endógenos de nocaute de miR por qRT-PCR
Nota: (i) Este protocolo requer o conhecimento dos métodos a seguir bem estabelecidos: preparação e manutenção de culturas primárias neuronais e qRT-PCR. (ii) repetir a quantificação da eficiência knockdown (passos 3.1 – 3.14) pelo menos 3 vezes (Replica biológica). (iii) estimativa da eficiência "knockdown" no nível de mRNA por qRT-PCR é adequada quando uma análise do conteúdo da proteína é impedida, como quando derrubando alternativamente emendados isoformas para que os anticorpos específicos não são disponíveis5.
-
Prepare-se culturas neuronal preliminares da região de interesse do cérebro. Siga o protocolo para culturas corticais dado na anterior publicação9, com as seguintes modificações:
- Neurônios de placa em 6 pratos bem em uma densidade de 500.000 neurônios por bem. Use a 2,5 mL/poço do meio de fixação e 3,3 mL/bem do meio de manutenção.
- Se for observado crescimento excessivo astrocyte, adicione 0,5 mL/poço de meio de manutenção suplementado com 7,5 µM de citosina β-D-arabinofuranoside (para obter uma concentração final de 1 µM) no 3 – 4 dias em vitro (DIV).
-
Infectar três poços por miR (técnica Replica) no 5-6 DIV. uso da menor dose infecciosa que irá infectar ≥ 99% dos neurônios.
- Remover 2 mL do meio de cada poço e recolhê-la em um tubo falcon de 50ml.
- Adicionar o vírus diretamente para os neurônios, misture delicadamente e coloque as placas de volta na incubadora a 37 ° C.
- Armazene o médio coletado na incubadora. Solta a tampa do tubo falcon para permitir o equilíbrio do gás.
- Após 24 h, adicione a média removido de volta em cada poço (1,9 mL/poço).
-
No 17-18 DIV, lyse os neurônios para extração do RNA.
Atenção: Reagente de Lise e clorofórmio são altamente tóxicos. Trabalhar sob uma capa de química e usar equipamento de proteção; Descarte de resíduos de acordo com as diretrizes nacionais e institucionais.
Nota: Para obter as etapas 3.3 – 3.13, trabalhar em condições de livre de RNAse. Use luvas e usar vidro livre de RNAse e plasticware descartável. Precauções gerais sobre como identificador do RNA, consulte por exemplo apêndice A do manual do Micro RNeasy disponível on-line.- Incube vidro que Pasteur pipetas durante a noite em um forno a 180 ° C.
- Incline as placas e aspire o meio completamente de cada poço usando um pipeta Pasteur, ligada a uma bomba de vácuo de vidro.
- Imediatamente adicione 700 µ l de reagente de Lise a cada poço.
- Espalhe uniformemente a solução por balançar e tremer a placa com cuidado e brevemente com a mão.
- Adicionar a solução acima e para baixo 4 a 5 vezes ou até obter uma suspensão homogênea.
- Transferi a solução de cada poço em um separado 1,5 mL tubo Eppendorf.
Nota: Os lisados celulares podem ser armazenados a-80 ° C ou processados imediatamente.
- Se necessário, descongelar célula os lysates no RT e proceder de imediato à etapa 3.5.
- Adicione 140 µ l de clorofórmio para cada amostra de lisados celulares.
Nota: Clorofórmio é volátil e difícil Pipetar, fechar os tubos de Eppendorf logo que possível. - Agitar os tubos de Eppendorf vigorosamente por 15 s ou até que as amostras são totalmente emulsionadas.
- Manter a amostra em RT por 1 – 2 min ou até começarem a separar as fases líquidas.
- Centrifugar as amostras a 12.000 x g por 15 min a 4 ° C.
- Transferir a fase aquosa superior (~ 320 µ l), contendo RNA para um novo tubo de Eppendorf e descarte o líquido restante.
Nota: Não toque a fase orgânica rosa inferior ou o anel branco entre as duas fases com a ponta da pipeta. - Adicione 1,5 volumes de 100% de etanol (480 µ l) para a fase aquosa, e adicionar a solução lentamente e descer 3 vezes para misturar.
- Purifica o RNA usando um kit disponível comercialmente projetado para a purificação do RNA das amostras pequenas.
- Quantificar a pureza de concentração e amostra de RNA com um espectrofotômetro.
Nota: (i) esperar rendimentos de ≥3.5 µ g/poço; a 260/280 e 260/230 rácios para pureza sobre proteínas e compostos orgânicos deve ambos ser ≥1.9. (ii) as amostras de RNA podem ser armazenadas a-80 ° C ou processadas imediatamente. - Retrotranscribe 250, 500 ou 1000 ng de RNA com um kit disponível comercialmente.
- Quantificar a eficiência "knockdown" para o gene endógeno de interesse por qRT-PCR. Para um protocolo detalhado, consulte referência de10. Aponta para uma "knockdown" eficiência de ≥60% (Figura 2). Normalizar os dados para ≥2 genes de limpeza (por exemplo. GAPDH, ACTB, TUBB3, LPPIA) usando o múltiplo controle interno gene método11.
Nota: Se trabalhando no rato, use os seguintes primers PCR para os genes de limpeza: GAPDH-fwd: 5' GGTGCTGAGTATGTCGTGGA 3' e GAPDH-rev: 5' GATGATGACCCTTTTGGC 3'; ACTB-fwd: 5' CATCACTATCGGCAATGAGC 3' e ACTB-rev: 5' TCATGGATGCCACAGGATT 3'; TUBB3-fwd: 5' GCCTTTGGACACCTATTCAG 3' e TUBB3-rev: 5' TCACATTCTTTCCTCACGAC 3'; LPPIA-fwd: 5' CACTGGGGAGAAAGGATTTG 3' e LPPIA-rev 5' CCATTATGGCGTGTGAAGTC 3'.
4. avaliar o papel das proteínas pré-sináptica em circuitos neuronais intactas por alvo estimulação de neurônios Knocked-down com Optogenetics
Nota: O seguinte protocolo requer experiência anterior com gravações eletrofisiológicas em fatias cerebral aguda e acesso a uma instalação eletrofisiológico.
- Estereotaxia injete o cérebro seguindo um protocolo detalhado no anterior publicação12rAAV1/2. Determine as coordenadas estereotáxica para a região do cérebro de interesse usando Atlas estereotáxica para rato13 ou rato cérebro14.
- Use um extrator de micropipeta para puxar Micropipetas de injeção com longas hastes de Ø 7 – 9 µm; clipe de Micropipetas de injeção as canelas com uma tesoura. Use um marcador fina e papel milimetrado para colocar marcas de calibração nas Micropipetas de injeção cada 2 mm.
- Anestesiar o animal com isoflurano e corrigi-lo no aparelho estereotáxico.
- Aquecer o animal durante toda a operação com uma almofada de aquecimento definida a 37 ° C.
- Protege os olhos com lubrificante ocular.
- Raspe o pelo na cabeça com um barbeador elétrico.
- Espalhe a iodopovidona na cabeça raspada utilizando um cotonete.
- Sob um microscópio de dissecação, fazer uma incisão.
- Limpe a superfície do crânio com um cotonete, então para tornar o bregma e o lambda visível.
- Coloque a micropipeta de injeção no seu suporte. Anexe o titular para o braço estereotáxico.
- Determinar o x e y coordenadas do local da injeção em relação a bregma e/ou lambda.
- Use uma broca para diluir o crânio sobre a área alvo.
Nota: Use movimentos circulares suaves e evite perfurar através do crânio, como isto irá danificar a superfície do cérebro. - Se lá está sangrando, absorção excessiva de sangue com papel toalha.
Nota: Excessiva de sangue tornará difícil determinar corretamente a coordenada z. - Carga ≤ 2 µ l do vírus na micropipeta de injeção por ação capilar.
- Trazer a micropipeta de injeção para o x e y coordenadas do local da injeção.
- Calcular a coordenada z da dura-máter e abaixe a pipeta lentamente para o cérebro.
- Quando a coordenada z é alcançada, espere 3 min para permitir o ajuste de tecido.
- Aplica baixa pressão positiva, utilizando uma seringa de 1 mL, conectada através de um tubo flexível na parte traseira da micropipeta de injeção. Visualmente, monitore a velocidade de ejeção do vírus da micropipeta de injeção através do microscópio de dissecação e usando as marcas de calibração como pontos de referência.
Nota: O vírus precisa ser injetado em ritmo lento (< 100 nL/min) para evitar danos aos tecidos. - Quando a injeção é terminada, espere 5 min, retirar a micropipeta de injeção de 0,2 mm, esperar por mais 5 minutos, para evitar o refluxo do vírus.
- Resguardar a micropipeta de injeção lentamente e completamente do cérebro e elimine-os em um recipiente com água sanitária.
- Molhe o crânio com solução fisiológica e sutura da pele com 3-4 pontos.
- Aplica a pomada gentamicina na ferida.
- Single-casa animal em uma gaiola limpa com alimentos de pelotas e sob uma lâmpada de calor até que ele se recupere totalmente.
- Injeção de pós de ≥ 15 dias, decapitar animais sob anestesia profunda isoflurano.
- Prepare fatias de cérebro aguda da região de interesse do cérebro com um Vibratome usando gás (95% O2, 5% CO2) solução gelada aCSF, contendo (em mM): NaCl 123, 1.25 KCl, 1.25 KH2PO4, 1.5 MgCl2, 1 CaCl2, 25 NaHCO3, NaPyruvate 2 e 18 glicose (osmolaridade ajustada para 300 mOsm). Por exemplo, se o objetivo é analisar a transmissão sináptica de CA3 para os neurônios piramidais do CA1, prepare fatias sagitais da formação hippocampal (350 µm de espessura).
Nota: Desta etapa em diante trabalha sob condições de pouca luz, para evitar a ativação da sonda optogenetic pela luz ambiental. - Deixe as fatias recuperar por 30 min a 37 ° C, na mesma aCSF numa câmara projetada para a realização de fatias de cérebro. Manter as fatias na câmara de fatia de cérebro mesmo à temperatura ambiente até a gravação.
Nota: Usando essas condições, fatias podem ser mantidas saudáveis para até 6-8 h. - Transferência um corta para uma câmara de gravação submerso e superfuse com 2 mL/min do mesmo aCSF usado para recuperação suplementada com 1,5 mM CaCl2 (total de Ca2 +: 2,5 mM) e sem NaPyruvate.
Nota: Fatias são preparadas e mantidas em baixa concentração de Ca2 + (1 mM) para minimizar a toxicidade. As gravações são realizadas em 2,5 mM Ca2 + para favorecer a liberação de vesículas. - Verifique rapidamente o sinal do repórter fluorescente expressa (por exemplo. TdTomato; Figura 3B) para determinar a localização e a intensidade da infecção.
- Encha um eletrodo de remendo com uma solução intracelular que contém (em mM): 110 K-gluconato, 22 KCl, NaCl 5, EGTA 0,5 3 MgCl2, 4 Mg-ATP, 0,5 at3-GTP, 20 K2-fosfato de creatina, HEPES-KOH 10 (pH 7.28, 290 mΩ).
Nota: Use eletrodo de remendo com resistência de pipeta de 5-6 mΩ. - Sob iluminação infravermelha, chegar a configuração de células inteiras-selo apertado de um neurônio receber entradas sinápticas de neurônios infectados. Por exemplo, se os neurônios piramidais CA3 foram infectados, remendo neurônios piramidais no proximal ao trato medial da região CA1 (Figura 3).
Observação: A resistência série pode ficar descompensada, mas deve ser constante e baixa (≤20 MΩ). - Use farmacologia para isolar as correntes sinápticas sob investigação. Por exemplo, se o objetivo é investigar a transmissão sináptica excitatória, bloquear a transmissão sináptica inibitória com 10 µM prosencefálico.
- Evocar correntes sinápticas, por exemplo, excitatórias pós-sináptico correntes (EPSCs), usando um laser azul nm 473, acoplado a uma fibra óptica (Ø ≤250 µm) posicionada sobre o somata dos neurônios infectados (por exemplo. Neurônios piramidais de CA3).
- Ajuste o comprimento de estimulação ao mínimo, para reduzir a possibilidade de evocar mais do que um potencial de ação por pulso de luz. Se usando o ChETA, defina-a 2 ms15.
- Ajustar a força de estimulação do laser para produzir correntes sinápticas pequenas mas claramente detectáveis (amplitude de pico de pA de ≤50 para EPSCs gravado em um potencial de exploração de -70 mV entre neurônios pyramidal CA3 e CA1; Figura 3D). Se usando o ChETA e fibra óptica de 250 µm de diâmetro, estimulação de pontos fortes de 1 – 3 mW em fibra de saída deve ser adequada.
Nota: se a força do laser não pode ser regulada, use filtros de densidade neutra. - Brilhe a luz de laser 473 nm no somata dos neurônios infectados, mas não em seus axônios. Por exemplo, brilhe a luz em CA3 somata e longe os colaterais de Schaffer, para evitar a despolarização direta de axônios.
- Aplicar a tetrodotoxina (TTX; 0,5 µM), um bloqueador dos canais de sódio, para a amostra para confirmar os canais são orientados para o potencial de ação.
- Em gravações diferentes, aplica um bloqueador seletivo à corrente sináptica sob investigação para confirmar que a estimulação é seletiva.
Nota: por exemplo, aplicar NBQX (10 µM) para Leandro-tipo glutamato mediada EPSCs. - Compare correntes sinápticas opticamente e eletricamente evocadas na mesma fatia cerebral aguda em resposta à estimulação repetitiva (estímulos de ≥2 a ≤20 Hz). Um grau semelhante de facilitação sináptica ou depressão deve ser observado entre a estimulação elétrica e óptica quando usando um miR controle, sugerindo que os mecanismos celulares similares são ativados pelos dois tipos de estímulos.
Nota: Os passos acima são necessários para assegurar que estimulação óptica não induz uma despolarização direta de boutons pré-sináptica, ignorando assim alguns mecanismos de transmissão sináptica.
- Compare a transmissão sináptica em resposta a estímulos simples e repetitivos (estímulos de ≥2 a ≤20 Hz) entre um miR controle e miRs como alvo as proteínas pré-sináptica sob investigação.
Resultados
Os procedimentos descritos acima fornecem um método robusto para avaliar a transmissão sináptica como é afetado pelo nocaute de proteínas sinápticas nos neurônios pré-sináptica. Resultados representativos sobre como o nocaute de alternativa splice isoformas de pré-sináptica Cav2.1 (tipo P/Q) VGCCs regula a curto prazo plasticidade sináptica no CA3 para CA1 sinapses excitatórias são dadas abaixo como exemplo.
CAv2.1 (tipo P/Q) canais são os VGCCs pré-sináptica predominantes em sinapses mais rápidas no sistema nervoso central. Alternativas de splicing os exões mutuamente exclusivas 37a e 37b de formadoras de poros α1 subunidade de Cav2.1 (α1A) produz duas variantes principais, Cav2.1 [EFa] e Cav2.1 [EFb]16,17 ,18. Para determinar se Cav2.1 [EFa] e Cav2.1 [EFb] diferencialmente regulam a transmissão sináptica e plasticidade em neurônios piramidais hippocampal do rato, primeiro desenvolvemos miRs isoforma específica para nocaute seletivamente Cav 2.1 [EFa] ou Cav2.1 [EFb]5. Apesar do pequeno tamanho (97 bp) e alta similaridade (61.86% identidade no nível do nucleotide) entre exões 37a e 37b, nós poderíamos projetar três sequências de miR contra rato Cav2.1 [EFa] (miR EFa1: TCCTTATAGTGAATGCGGCCG; miR EFa2: ATGTCCTTATAGTGAATGCGG; miR EFa3: TTGCAAGCAACCCTATGAGGA) e dois contra rato Cav2.1 [EFb] (miR EFb1: ATACATGTCCGGGTAAGGCAT; miR EFb2: ATCTGATACATGTCCGGGTAA) com previsto alta eficiência knockdown. Como controlo negativo (miR controle), usamos o plasmídeo pcDNA6.2-GW/EmGFP-miR-neg contendo uma sequência que não tem como alvo qualquer gene conhecido de vertebrados. Com base em uma primeira tela em células HEK 293 contra heterólogos canais, selecionamos miR EFa1, miR EFa3 e miR EFb2 e clonou suas fitas de expressão para o 3' UTR do vetor pAAV-Syn-ChETA-TdT-miR-X, que é projetado para a produção de rAAVs e onde o synapsin promotor conduz a expressão da channelrhodopsin ultra rápida ChETA, a proteína fluorescente vermelha TdTomato e a miR inserida (Figura 1). Também Duplicamos a gaveta de expressão de miR EFb2 para aumentar a eficiência "knockdown" deste miR.
Em seguida, preparados rAAV1/2 para as construções de quatro acima e quantificada sua eficiência knockdown e seletividade em culturas neuronal de rato primário usando isoforma específica qRT-PCR. miR EFa1 e miR EFa3 reduzida mRNA do nativo Cav2.1 [EFa] ~ 70%, mas não o da Cav2.1 [EFb], enquanto miR EFb reduzido mRNA do nativo Cav2.1 [EFb] ~ 60%, mas não o da Cav2.1 [EFa] (Figura 2).
Nós Estereotaxia injetou cada um dos quatro rAAV1/2 na área CA3 do hipocampo de ratos P18 (Figura 3A-C), com coordenadas de −2.6 (A-P/M-L/D-V de Bregma) / ± 2,9 / −2.9. Pós-injeção de quinze para vinte e quatro dias, estamos preparados fatias hippocampal agudas de ratos rAAV1/2-injetado e usado TdTomato fluorescência para confirmar a expressão e a localização de rAAVs (Figura 3B). Para investigar se pré-sináptica nocaute de Cav2.1 [EFa] ou Cav2.1 [EFb] afetadas a curto prazo plasticidade sináptica no CA3 para CA1 sinapses, nós estimulada seletivamente infectados CA3 neurônios com breve 473 nm laser pulsos de luz (2 ms-long ; A Figura 3) e gravou os EPSCs resultantes emendando neurônios piramidais no proximal ao trato medial da região CA1 (Figura 3). Nós achamos que o knockdown de Cav2.1 da tala isoformas afetou as respostas à estimulação emparelhado-pulso em direções opostas: nocaute de Cav2.1 [EFa] (miR EFa1 ou miR EFa3) impulsionou emparelhado-pulso facilitação (PPF) Considerando que o knockdown de Ca v2.1 [EFb] (miR EFb2) aboliu (Figura 3D, E).
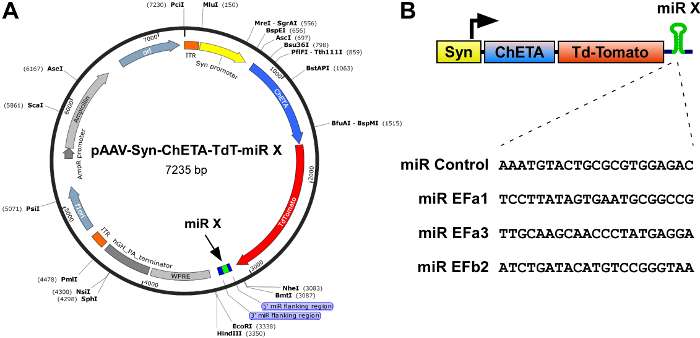
Figura 1: Esquema das construções para expressão combinada da sonda optogenetic ultra rápida ChETA e miRs isoforma específica contra Cav2.1 [EFa] e Cav2.1 [EFb]. (A) mapa de rAAV a construir pAAV-Syn-ChETA-TdT-miR-X, contendo um promotor synapsin (Syn), a channelrhodopsin ultra rápida ChETA fundido para TdTomato e, no proximal 3' UTR, miR de isoforma específica um Cav2.1 da tala. Enzimas de restrição mostradas são cortadores único. (B) superior, regime de gaveta a expressão. O promotor synapsin drives expressão de ChETA-TdTomato e de uma isoforma específica miR. No fundo, complemento reverso das sequências alvo 21-nucleotide, que formam parte da fita miR. Para miR EFb2 duas fitas idênticas miR foram expressos em tandem, um após o outro. Clique aqui para ver uma versão maior desta figura.
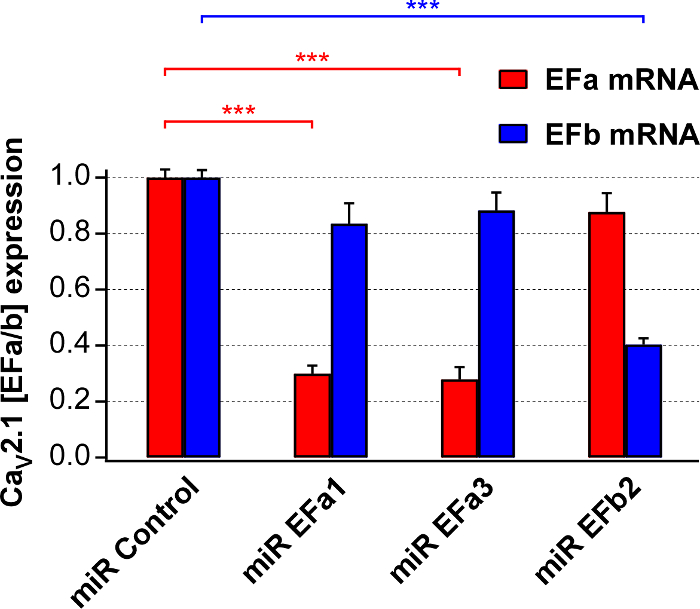
Figura 2. Avaliação da eficiência knockdown e seletividade de miRs isoforma específica para Cav2.1 [EFa] e Cav2.1 [EFb]. Análise de isoforma específica qRT-PCR RNA isolado de culturas primárias de DIV de 17-18 infectadas ao DIV 6 com rAAVs expressando miRs direcionamento de qualquer Cav2.1 [EFa] (miR EFa1 e miR EFa3) ou Cav2.1 [EFb] (miR. EFb2). Dados são normalizados para o controle negativo (miR controle). miR EFa1 e miR EFa3 significativamente e seletivamente reduzem mRNA do Cav2.1 [EFa] (n = 8 e 7 culturas, respectivamente), enquanto miR EFb significativamente e seletivamente reduz o mRNA do Cav2.1 [EFb] (n = 4 culturas; * * * p < 0,001; unidirecional análise de teste de variância seguido do pós-teste de Tukey-Kramer). Os dados são apresentados como média ± SEM. Esta figura foi adaptada da Thalhammer, a. et al. 5. clique aqui para ver uma versão maior desta figura.

Figura 3. Avaliar o papel da Cav2.1 [EFa] e Cav2.1 [EFb] no hipocampo nativo por direcionados a estimulação dos neurônios batido para baixo com optogenetics. (A) esquema da fita expressão das construções rAAV usado para infecção na vivo . (B) seção Hippocampal mostrando essa fluorescência TdTomato é limitada à região CA3 e suas projeções. (C) configuração Experimental: feixe de laser foi dirigido para somata CA3, e gravações de braçadeira do remendo foram realizadas entre os neurônios piramidais do CA1. (D) 2 ms-long azul (473 nm) pulsos de laser luz brilhados a 20 Hz evocam EPSCs cujo PPF é aumentado pela miRs direcionamento Cav2.1 [EFa] e abolida pelo miR para Cav2.1 [EFb]. (E) Resumo do rácio emparelhado-pulso para experiências como em (D), mostrando um aumento em PPF para miR EFa1 e miR EFa3 e uma diminuição para miR EFb2, relativo à miR controle (n = 9-11 gravações; * p = 0,02; * * p = 0,01; * * * p < 0,0004; análise de covariância). Os dados são apresentados como média ± SEM. Esta figura foi adaptada da Thalhammer, a. et al. 5. clique aqui para ver uma versão maior desta figura.
Discussão
Co a expressão de uma sonda de optogenetic e um miR contra um gene pré-sináptica de interesse oferece uma abordagem poderosa para caracterizar o efeito de knockdown do gene na função pré-sináptica dentro circuitos neuronais intactas. Para esta abordagem experimental, é importante identificar e caracterizar miRs que são altamente eficientes e seletivas em derrubar o gene de interesse em sistemas nativos. Se possível, dois ou mais miRs independentes contra o mesmo mRNA de interesse devem ser usado para controle para que os eventuais efeitos fora do alvo. Experiências de resgate, no qual um gene resistente a miR foi reintroduzido no sistema, podem ser usadas como um controle para a especificidade.
Usando a mesma construção para expressar um optogenetic sonda e um miR permite estimulando opticamente apenas os neurônios pré-sináptica que tem sido manipulados. Isto não é possível com estímulos elétricos, porque eles não fazem distinção entre neurônios infectados e não infectados, assim produzindo resultados mistos e diluídos. Porque estímulos ópticos podem induzir vários potenciais de ação15, é importante escolher apenas as sondas mais rápidas de optogenetic, entre uma expansão paleta15,19,20,21. Além disso, é essencial para assegurar que a estimulação óptica não induz uma despolarização direta de boutons pré-sináptica, para evitar algumas das etapas da transmissão sináptica ignorando um deseja investigar22.
A abordagem experimental que descrevemos aqui, torna possível avaliar em paralelo a relevância fisiológica de múltiplos genes pré-sináptica de interesse dentro de um período de tempo limitado (4 – 6 meses). No entanto, é importante ter em mente que knockdowns raramente atingir 100%. Além disso, o rAAVs precisa ser expressa pelo menos duas semanas para permitir a máxima expressão knockdown e cheio da sonda optogenetic, que pode representar uma restrição de tempo, ao investigar os processos de desenvolvimento precoce. Embora mais ratos do knockout condicional, consumindo tempo limitados de neurônios pré-sináptica, geralmente resultam em completar a remoção do gene de interesse e, portanto, oferecer uma abordagem complementar válida.
Uma vantagem específica da tecnologia miR é que ele permite que a expressão de vários miRs o mesmo promotor. Essa propriedade tem sido usada principalmente para aumentar a eficiência "knockdown" inserindo várias cópias da mesma miR ou miRs diferentes contra o mesmo gene alvo. No entanto pode ser usada também para "knockdown" múltiplos genes expressando miRs contra alvo diferentes genes7,23. Esta propriedade pode ser usada para silenciar várias proteínas pré-sináptica para dissecar as vias de sinalização pré-sináptica.
Aqui, nós combinamos optogenetics com miRs artificiais para caracterizar os efeitos do knockdown do gene na função pré-sináptica em fatias hippocampal agudas. Uma abordagem semelhante também pode ser usada para manipular e sondar circuito neuronal na vivo. Além disso, combinando miRs artificiais com chemogenetic abordagens permitiria um circuito neuronal de escalas de tempo mais longos de interrogar.
Divulgações
Os autores não têm nada para divulgar.
Agradecimentos
Agradecemos a F. Benfenati (Istituto Italiano di Tecnologia, IIT) para suporte e Carmela Vitale para ajudar com a demonstração. Este trabalho foi financiado pelo IIT e Compagnia San Paolo (grant n º 9734 para LAC).
Materiais
| Name | Company | Catalog Number | Comments |
| Acrylamide | Sigma | Acrylamide/Bis-acrylamide, 30% solution | Toxic |
| Animal Temperature Controller with heat pad | WPI | ATC2000 | |
| BCA protein assay kit | ThermoFisher Scientific | 23225 | |
| BLOCK-iT Pol II miR RNAi Expression Vector kit | ThermoFisher Scientific | K4936-00 | |
| Brain slices Prechamber | Harvard Apparatus | BSC-PC | |
| CCD camera-based imager | Bio-Rad | ChemiDoc™ MP | |
| Cell Culture reagents | Life Technologies | ||
| Chemiluminescent substrate | GE Helthcare | ECL Western Blotting Reagents, RPN2106 | |
| Chloroform | Sigma | C2432 | Toxic |
| Cytosine β-D-arabinofuranoside | Sigma | C6645 | |
| Drill | Foredom | K.1030 | |
| EGTA | Sigma | E4378 | |
| Gentamicin ointment | Local pharmacy | ||
| Glucose | Sigma | G7021 | |
| Hepes | Sigma | 54459 | |
| Injection micropipettes | Narishige | GD1 | |
| Inorganic salts & detergents | Sigma | ||
| K2-creatine phosphate | Calbiochem | 237911 | |
| KGluconate | Fluka | 60245 | |
| Laser | Laserglow Technologies | LRS-0473-GFM-00100-03 | |
| Membrane | Amersham | Protran™ 0.2 µm NC | |
| MgATP | Sigma | A9187 | |
| Micropipette holder | Narishige | IM-H1 | |
| Micropipette puller | Narishige | PC-100 | |
| Na3GTP | Sigma | G8877 | |
| NaPyruvate | Sigma | P5280 | |
| Ocular lubricant | Local pharmacy | Lacrigel | |
| Phosphate inhibitors | Sigma | P0044, P5726 | |
| Protease inhibitors | Sigma | cOmplete™, EDTA-free Protease Inhibitor Cocktail | |
| Protein gel electrophoresis and blotting devices | Bio-Rad | Mini-Protean III Cell | |
| Providone iodine | Local pharmacy | Betadine | |
| Qiazol Lysis Reagent | Qiagen | 79306 | Toxic |
| QuantiTect Reverse Transcription Kit | Qiagen | 205311 | |
| RNeasy Micro Kit | Qiagen | 74004 | |
| Spectrophotometer | ThermoFisher Scientific | Nanodrop 2000 | |
| Stereotactic apparatus | WPI | ||
| Tetrodotoxin | Tocris | 1069 | Toxic |
| Vibratome | Leica | VT1200S |
Referências
- Lam, J. K., Chow, M. Y., Zhang, Y., Leung, S. W. siRNA Versus miRNA as Therapeutics for Gene Silencing. Mol Ther-Nucl Acids. 4, e252 (2015).
- Kohl, M. M., et al. Hemisphere-specific optogenetic stimulation reveals left-right asymmetry of hippocampal plasticity. Nat Neurosci. 14 (11), 1413-1415 (2011).
- Maejima, T., et al. Postnatal loss of P/Q-type channels confined to rhombic-lip-derived neurons alters synaptic transmission at the parallel fiber to purkinje cell synapse and replicates genomic Cacna1a mutation phenotype of ataxia and seizures in mice. J Neurosci. 33 (12), 5162-5174 (2013).
- Mark, M. D., et al. Delayed postnatal loss of P/Q-type calcium channels recapitulates the absence epilepsy, dyskinesia, and ataxia phenotypes of genomic Cacna1a mutations. J Neurosci. 31 (11), 4311-4326 (2011).
- Thalhammer, A., et al. Alternative Splicing of P/Q-Type Ca2+ Channels Shapes Presynaptic Plasticity. Cell Rep. 20 (2), 333-343 (2017).
- Sambrook, J., Russell, D. W. Calcium-phosphate-mediated Transfection of Eukaryotic Cells with Plasmid DNAs. CSH Protoc. 2006 (1), (2006).
- Chung, K. H., et al. Polycistronic RNA polymerase II expression vectors for RNA interference based on BIC/miR-155. Nucleic Acids Res. 34 (7), e53 (2006).
- McClure, C., Cole, K. L., Wulff, P., Klugmann, M., Murray, A. J. Production and titering of recombinant adeno-associated viral vectors. J Vis Exp. (57), e3348 (2011).
- Cingolani, L. A., et al. Activity-dependent regulation of synaptic AMPA receptor composition and abundance by beta3 integrins. Neuron. 58 (5), 749-762 (2008).
- Huang, H., Xu, Y., Cheng, C. Detection of alternative splicing during epithelial-mesenchymal transition. J Vis Exp. (92), e51845 (2014).
- Vandesompele, J., et al. Accurate normalization of real-time quantitative RT-PCR data by geometric averaging of multiple internal control genes. Genome Biol. 3 (7), (2002).
- Cetin, A., Komai, S., Eliava, M., Seeburg, P. H., Osten, P. Stereotaxic gene delivery in the rodent brain. Nat Protoc. 1 (6), 3166-3173 (2006).
- Paxinos, G., Franklin, K. B. . The Mouse Brain in Stereotaxic Coordinates. , (2012).
- Paxinos, G., Watson, C. . The Rat Brain in Stereotaxic Coordinates. , (2013).
- Gunaydin, L. A., et al. Ultrafast optogenetic control. Nat Neurosci. 13 (3), 387-392 (2010).
- Bourinet, E., et al. Splicing of alpha 1A subunit gene generates phenotypic variants of P- and Q-type calcium channels. Nat Neurosci. 2 (5), 407-415 (1999).
- Chaudhuri, D., et al. Alternative splicing as a molecular switch for Ca2+/calmodulin-dependent facilitation of P/Q-type Ca2+ channels. J Neurosci. 24 (28), 6334-6342 (2004).
- Soong, T. W., et al. Systematic identification of splice variants in human P/Q-type channel alpha1(2.1) subunits: implications for current density and Ca2+-dependent inactivation. J Neurosci. 22 (23), 10142-10152 (2002).
- Berndt, A., et al. High-efficiency channelrhodopsins for fast neuronal stimulation at low light levels. P Natl Acad Sci USA. 108 (18), 7595-7600 (2011).
- Klapoetke, N. C., et al. Independent optical excitation of distinct neural populations. Nat Methods. 11 (3), 338-346 (2014).
- Lin, J. Y., Lin, M. Z., Steinbach, P., Tsien, R. Y. Characterization of engineered channelrhodopsin variants with improved properties and kinetics. Biophys J. 96 (5), 1803-1814 (2009).
- Zhang, Y. P., Oertner, T. G. Optical induction of synaptic plasticity using a light-sensitive channel. Nat Methods. 4 (2), 139-141 (2007).
- Fowler, D. K., Williams, C., Gerritsen, A. T., Washbourne, P. Improved knockdown from artificial microRNAs in an enhanced miR-155 backbone: a designer's guide to potent multi-target RNAi. Nucleic Acids Res. 44 (5), e48 (2016).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados