Method Article
Combinando optogenetica con microRNA artificiali per caratterizzare gli effetti del Gene atterramento su funzione presinaptica all'interno di circuiti neuronali intatto
In questo articolo
Riepilogo
Questo protocollo fornisce un flusso di lavoro su come combinare artificiale microRNA-mediata RNA interference con optogenetica per stimolare specificamente i boutons presinaptici con ridotta espressione dei geni selettivi all'interno di circuiti neuronali intatti.
Abstract
Lo scopo del presente protocollo è quello di caratterizzare l'effetto di atterramento di gene sulla funzione presinaptica all'interno di circuiti neuronali intatti. Descriviamo un flusso di lavoro su come combinare artificiale microRNA (miR)-mediata di interferenza del RNA con optogenetica raggiungere stimolazione selettiva dei boutons presinaptici manipolato nelle fette del cervello acuto. L'approccio sperimentale prevede l'utilizzo di un singolo costrutto virale e un singolo promotore di neurone-specific per guidare l'espressione di un optogenetica sonda e il miR(s) artificiale contro geni presinaptici. Quando stereotactically iniettato nella regione del cervello di interesse, il costrutto espresso rende possibile stimolare con luce esclusivamente i neuroni con ridotta espressione dei geni sotto indagine. Questa strategia non richiede lo sviluppo e la manutenzione delle linee di topi geneticamente modificati per e in linea di principio può essere applicata ad altri organismi e per ogni gene di un neurone di scelta. Recentemente abbiamo applicato per indagare come l'atterramento di isoforme di splicing alternativo dei canali tensione-gated del calcio di tipo P/Q presinaptici (VGCCs) regola la plasticità sinaptica a breve termine presso CA3 di sinapsi eccitatorie CA1 nelle fette hippocampal acute. Un simile approccio potrebbe essere utilizzato anche per manipolare e sonda il circuito neuronale in vivo.
Introduzione
Questo protocollo descrive un nuovo approccio per caratterizzare l'effetto di atterramento di gene sulla funzione presinaptica all'interno di circuiti neuronali intatti. Indagando funzione presinaptica in circuiti neuronali intatti è difficile perché molte boutons presinaptici sono troppo piccoli e lontano dal soma per consentire interventi combinati elettrofisiologici e molecolari. Anche se l'interferenza del RNA offre uno strumento potente e flessibile per atterramento proteine sinaptiche1, questo approccio è stato usato con parsimonia per indagare la funzione presinaptica perché è difficile da rilevare gli effetti di atterramento utilizzando tradizionali gli stimoli elettrici, che non fanno distinzione tra manipolato e ingenuo boutons presinaptici2. Qui, descriviamo come combinare artificiale microRNA (miR)-mediata di interferenza del RNA con optogenetica recentemente sviluppato la tecnologia per ottenere stimolazione selettiva dei boutons presinaptici manipolato nelle fette del cervello acuto.
Mentre topi knockout condizionale in combinazione con elettrofisiologia potrebbero anche essere usati per studiare la funzione di proteine presinaptiche3,4, la nostra strategia non richiede lo sviluppo e la manutenzione di geneticamente modificato mouse linee e può essere facilmente impiegato anche per abbattere isoforme specifiche di un gene. Relativo a più comunemente usate breve tornante RNAs (shRNA), miRs artificiale, che impieghiamo qui, offrono vantaggi chiave per atterramento in neuroni. A differenza di shRNA, possono essere espressi sotto il controllo di una polimerasi II promotore1. Così, un singolo promotore può essere utilizzato per guidare l'espressione di miR e di una sonda di optogenetica, insieme a un reporter fluorescente. In questo modo, la dimensione del costrutto può essere mantenuta entro i limiti di imballaggio dei ricombinanti virus adeno-associato (rAAV, Figura 1A). Inoltre, l'uso di un singolo costrutto e un singolo promotore riduce la variabilità sperimentale perché permette per l'espressione di miR, la sonda optogenetica e il reporter fluorescente in un rapporto fisso.
Recentemente abbiamo applicato questa tecnologia per esaminare il ruolo delle isoforme alternativamente impiombate dei canali del calcio presinaptici in ippocampo5. Tale strategia è generalmente applicabile a studiare la rilevanza fisiologica delle altre proteine presynaptically espresse in qualsiasi circuito del cervello di interesse.
Protocollo
Tutti gli esperimenti sono stati effettuati conformemente agli orientamenti stabiliti dal Consiglio delle Comunità europee (direttiva 2010/63/UE del 4 marzo 2014) e sono stati approvati dal Ministero della salute.
1. progettazione dei microRNA per la valutazione della loro efficienza in sistemi di espressione eterologhi e RNA Interference
Nota: Questo protocollo richiede la conoscenza dei seguenti metodi consolidati: clonazione molecolare, sequenziamento del DNA, manutenzione di linee cellulari, di transfezione del fosfato di calcio, quantitativa real time PCR (qRT-PCR), preparazione dei lisati cellulari da linee cellulari e Western blotting.
- Determinare se il gene di interesse è soggetto a splicing alternativo. Per un atterramento generale del gene, selezionare esclusivamente costitutivi esoni; per un atterramento di un'isoforma specifica, selezionare l'essone alternativamente impiombato pertinente.
- Utilizzare un software dedicato (ad es. https://rnaidesigner.thermofisher.com/rnaiexpress/) per progettare artificiale miRs per interferenza del RNA contro la sequenza di interesse.
Nota: È importante utilizzare una base locale allineamento Cerca strumento (BLAST) per controllare i possibili i bersagli all'interno della stessa specie. - Selezionare il miRs n tre top per il gene dell'obiettivo.
- Ordinare i fili superiore e inferiore di Mir selezionato da una società adatta. Per il controllo negativo, utilizzare una versione strapazzata di uno il miRs selezionato o un miR predetto non a qualsiasi gene conosciuto delle specie utilizzate nell'esperimento di destinazione (ad es. per geni vertebrati, il miR presente in pcDNA6.2-GW/EmGFP-miR-neg).
- Temprare i rispettivi fili superiore e inferiore di Mir selezionati e li Duplica in un plasmide progettato per l'espressione di Mir (ad es. , pcDNA6.2-GW/EmGFP-miR) utilizzando strategie di clonazione standard.
Nota: L'obiettivo è di creare una cassetta di espressione di miR che consiste di una 5' regione fiancheggiante miR, la sequenza selezionata miR e una 3' miR regione di accompagnamento che possa essere espressi dal 3' UTR di un gene del reporter sotto il controllo di un promotore II tipo di RNA polimerasi. - Utilizzare sequenziamento del DNA per convalidare il corretto inserimento di Mir selezionato.
- Crescere le cellule HEK293 in un'incubatrice di coltura cellulare umidificata (37 ° C, 5% CO2) utilizzando DMEM medium completato con 10% siero bovino fetale, 1 mM NaPyruvate, 10 mM HEPES (pH 7,39) e 1 x penicillina/streptomicina (Pen/Strep).
- Quando le cellule HEK293 sono ~ 70% confluenti, co-li transfect con ciascuno dei vettori di espressione di miR e un vettore che esprime il gene bersaglio in un rapporto di 1:1 del DNA utilizzando il metodo di fosfato di calcio6. Includono i seguenti controlli: (i) untransfected cellule, (ii) le cellule transfected con il vettore che esprime il gene mirato insieme o vettore di DNA (ad esempio pBluescript II SK(+)) o (iii) miR controllo.
Nota: (i) il gene bersaglio espresso deve essere della stessa specie, verso il quale sono stati progettati il miRs, a meno che le sequenze bersaglio il miRs sono assolutamente conservate a livello del nucleotide fra le due specie. (ii) linee cellulari diversi da HEK293 possono essere utilizzate. (iii) alternativi metodi di transfezione, ad esempio l'elettroporazione o metodi basati su liposoma, possono essere utilizzati. -
48 ore dopo la trasfezione lisare le cellule, esegua il gel di proteina, eseguire il Western blot e analizzare il contenuto proteico con un anticorpo riconosce la proteina bersaglio. Scopo per un atterramento efficienza ≥ 50%.
Nota: Atterramento efficienza può in alternativa o in più, essere valutato (i) di analisi di ELISA, (ii) misurando l'attività della proteina bersaglio se applicabile (ad es. densità di corrente per canali ionici) o (iii) mediante qRT-PCR al livello del mRNA se adatto gli anticorpi non sono disponibili (punto 3 del protocollo).- Mettere i piatti di cultura sul ghiaccio e lavare le cellule una volta con gelida tampone fosfato salino (PBS).
- Aspirare il PBS, quindi aggiungere ghiacciata RIPA di tampone (pH 50 mM Tris-HCl 7.4, 150 mM NaCl, 2 mM EDTA, 1% NP40, 0.1% sodio dodecil solfato (SDS), contenente inibitori di proteasi e fosfatasi; 1 mL per un piatto di Ø 100 mm, 0,5 mL per un piatto di Ø 60 mm).
- Raschiare le cellule aderenti il piatto con un raschietto in cella e delicatamente trasferisca la sospensione cellulare in un tubo del microcentrifuge pre-raffreddata.
- Centrifugare la provetta a 15.000 x g per 15 min a 4 ° C, trasferire il surnatante in un nuovo tubo del microcentrifuge pre-raffreddato e scartare il pellet.
- Determinare la concentrazione di proteina utilizzando il kit di analisi della proteina di BCA o altro metodo adatto (ad es. analisi di Bradford).
Nota: Campioni possono essere congelati a-20 ° C o da-80 ° C per un uso successivo o elaborati immediatamente. - Trasferire un volume adeguato di lisati per microcentrifuga in modo che tutti i campioni hanno la stessa concentrazione di proteina e aggiungere tampone di lisi ghiacciata adeguate per compensare tutti i lisati allo stesso volume.
Nota: 30 a 50 µ g di proteina totale dovrebbe essere sufficiente per la maggior parte delle proteine, ma la quantità appropriata da caricare deve essere determinata secondo l'abbondanza della proteina di interesse. - Aggiungere una quantità appropriata di 2 x Laemmli buffer (4% SDS, 2-mercaptoetanolo 10%, 20% glicerolo, 0,004% bromofenolo, 0,125 M Tris-HCl, pH 6.8) e far bollire i campioni a 95 ° C per 5 min.
- Caricare i campioni su un gel di acrilammide insieme a un marcatore di peso molecolare. Esegua il gel a 100 V per 1-2 h.
Nota: La percentuale di gel dipende dalle dimensioni della proteina di interesse. - Montare il panino di trasferimento e trasferire le proteine dal gel su una membrana a 100 V per 2 h. La membrana può essere nitrocellulosa o PVDF. Attivare PVDF con metanolo per 1 min e risciacquare con tampone di trasferimento prima di preparare lo stack.
- Bloccare la membrana per 1 h a temperatura ambiente mediante tampone bloccante (5% di latte in PBST (PBS + 0.1% Tween-20)), quindi incubare la membrana durante la notte a 4 ° C con l'anticorpo primario diluito in tampone bloccante.
- Lavare la membrana per 10 min in PBST tre volte e viene quindi incubato con anticorpo secondario coniugato HRP in tampone bloccante per 1 h a temperatura ambiente.
- Lavare la membrana per 10 min in PBS quattro volte, quindi applicare il substrato chemiluminescente alla membrana e catturare i segnali a chemiluminescenza utilizzando un sensore basato su telecamera CCD.
- Utilizzare software di analisi di immagine per quantificare l'efficienza atterramento.
- Selezionare il miRs più efficiente due sequenze non sovrapposte per ulteriori saggi funzionali di targeting.
Nota: Gli effetti Off-target non possono mai essere completamente esclusa per qualsiasi miR. Tuttavia, se due miRs indipendenti hanno un effetto simile, è estremamente improbabile che questo è a causa di un colpo fuori bersaglio stesso. - Se l'efficienza atterramento di Mir selezionato non è soddisfacente (< 50%), schermo per altri miRs. In alternativa, esprimere la miR(s) più efficiente in tandem, cioè esprimerli in più copie dalla stessa cassetta di espressione, che può provocare una maggiore efficienza di atterramento7.
2. costruzione di vettori ricombinante Adeno-associato per espressione combinata di optogenetica sonde e microRNA
Nota: Questo protocollo richiede la conoscenza dei seguenti metodi consolidati: clonazione molecolare, il sequenziamento del DNA e la produzione di rAAV.
- Sonde di Clone ogni di cassette di espressione del miR più efficiente nel 3' UTR di costrutti progettati per la produzione di rAAVs ricombinante ed espressione di eccitatorio optogenetica, ad esempio in pAAV-Syn-ChETA-TdT-miR-X (Figura 1A), dove il espressione della channelrhodopsin ultraveloce ChETA, il reporter fluorescente rosso TdTomato e miR(s) inserito tra i siti NheI ed EcoRI5unità promotore della sinapsina di neurone-specific.
Nota: (i) non superare il limite di imballaggio di rAAVs, che è di circa 4,7 Kb lunghezza da ITR (ripetizione terminale invertito) a ITR (Figura 1A, arancione). (ii) la cassetta di espressione di miR deve essere inserito tra il codone di arresto e WPRE (Marmotta epatite Virus Posttranscriptional elemento normativo; Figura 1A). (iii) l'inclusione di una proteina fluorescente, come TdTomato, è estremamente utile in quanto consente di monitorare facilmente la localizzazione e l'intensità dell'infezione (punto 4 del protocollo). - Utilizzare sequenziamento del DNA per convalidare il corretto inserimento delle cassette miR.
- Produrre e titolo rAAV1/2 secondo il protocollo precedentemente pubblicato8, uno rAAV1/2 per ogni miR selezionato. Un tipico esperimento per il colpo di un gene includerebbe due miRs indipendente contro la stesso gene e il controllo di un miR. Puntare ad un titolo del virus di ≥ 109 genomi virali (vg) / µ l.
Attenzione: rAAVs devono essere gestite in una struttura di livello di biosicurezza 1 (BSL-1). Si prega di controllare con Comitato istituzionale biosicurezza per informazioni dettagliate.
Nota: Se il laboratorio non ha una struttura virale o titoli virali non sono soddisfacenti, rAAV produzione possa essere esternalizzate. Ad esempio, visitare https://www.med.upenn.edu/gtp/vectorcore/ o https://vcf.charite.de/en/metas/.
3. estrazione di RNA da colture neuronali primarie per la valutazione di miR efficienza atterramento di geni endogeni mediante qRT-PCR
Nota: (i) questo protocollo richiede la conoscenza dei seguenti metodi consolidati: preparazione e manutenzione delle colture neuronali primarie e qRT-PCR. (ii) ripetere la quantificazione dell'atterramento efficienza (passaggi 3.1 – 3,14) almeno 3 volte (replica biologici). (iii) la stima di atterramento efficienza a livello di mRNA mediante qRT-PCR è adatta quando un'analisi del contenuto proteico è preclusa, ad esempio quando abbattendo alternativamente impiombato isoforme per cui anticorpi specifici non sono disponibili5.
-
Preparare colture neuronali primarie della regione del cervello di interesse. Seguire il protocollo per colture corticali dato nella precedente pubblicazione9, con le seguenti modifiche:
- Neuroni di piastra a 6 pozzetti ad una densità di 500.000 neuroni per pozzetto. Utilizzare 2,5 mL/pozzetto di attaccamento medio e 3,3 mL/bene di mezzo di manutenzione.
- Se si osserva la crescita eccessiva di astrociti, aggiungere 0,5 mL/pozzetto di mezzo di manutenzione completato con 7,5 µM di citosina β-D-arabinofuranoside (per una concentrazione finale di 1 µM) al 3 – 4 giorni in vitro (DIV).
-
Infettare tre pozzi a miR (tecnico replica) alle 5–6 DIV. uso della più bassa dose infettiva che infetterà ≥ 99% dei neuroni.
- Rimuovere 2 mL di terreno da ogni pozzetto e raccoglierlo in un tubo falcon 50 mL.
- Aggiungere virus direttamente i neuroni, mescolare delicatamente e rimettere le piastre nell'incubatore a 37 ° C.
- Conservare il mezzo raccolto nell'incubatrice. Allentato il tappo della provetta falcon per consentire equilibrazione di gas.
- Dopo 24 h, è necessario aggiungere il medium retro rimosso in ogni pozzetto (1,9 mL/pozzetto).
-
Alle 17–18 DIV, lisare i neuroni per estrazione del RNA.
Attenzione: Il reagente di lisi e cloroformio sono altamente tossici. Lavorare sotto una cappa chimica e indossare indumenti protettivi; smaltire i rifiuti secondo le vostre linee guida nazionali e istituzionali.
Nota: Per la procedura 3.3 – 3,13, lavorare in condizioni di RNAsi-libera. Indossare guanti e usare vetreria RNAsi-libera e plasticware USA e getta. Per le precauzioni generali su come maniglia RNA, consultare ad esempio Appendice A del manuale Micro RNeasy disponibili online.- Incubare il vetro che pipetta Pasteur durante la notte in forno a 180 ° C.
- Inclinare le piastre e aspirare il mezzo completamente da ogni pozzetto usando un pipetta Pasteur collegato ad una pompa a vuoto di vetro.
- Immediatamente aggiungere 700 µ l di reagente di lisi in ciascun pozzetto.
- Distribuire uniformemente la soluzione a dondolo e scuotendo la piastra con attenzione e brevemente a mano.
- Dispensare la soluzione su e giù 4-5 volte o fino ad ottenuta una sospensione omogenea.
- Trasferire la soluzione da ogni pozzetto in un separato provetta Eppendorf da 1,5 mL.
Nota: I lisati possono essere conservati a-80 ° C o elaborati immediatamente.
- Se necessario, sbrinamento cella i lisati a RT e passa direttamente al punto 3.5.
- Aggiungere 140 µ l di cloroformio per ciascun campione di lisati cellulari.
Nota: Il cloroformio è volatile e difficile pipettare, chiudere le provette Eppendorf appena possibile. - Agitare le provette Eppendorf vigorosamente per 15 s o fino a quando i campioni sono completamente emulsionati.
- Mantenere il campione a RT per 1 – 2 min o fino a quando il liquido fasi iniziano a separare.
- Centrifugare i campioni a 12.000 x g per 15 min a 4 ° C.
- Trasferire la fase acquosa superiore (~ 320 µ l) contenente RNA in una nuova provetta Eppendorf e scartare il liquido rimanente.
Nota: Non toccare la fase organica rosa inferiore o l'anello bianco tra le due fasi con la punta della pipetta. - Aggiungere 1,5 volumi di etanolo al 100% (480 µ l) per la fase acquosa e dispensare la soluzione lentamente su e giù per 3 volte per mescolare.
- Purificare il RNA usando un kit disponibile in commercio progettato per la purificazione di RNA da piccoli campioni.
- Quantificare la purezza di concentrazione e campione di RNA con uno spettrofotometro.
Nota: (i) si aspettano rendimenti di ≥ 3,5 µ g/pozzo; 260/280 e 260/230 rapporti per purezza oltre proteine e composti organici dovrebbe entrambi essere ≥1.9. (ii) i campioni di RNA possono essere conservati a-80 ° C o elaborati immediatamente. - Retrotranscribe 250, 500 o 1000 ng di RNA con un kit disponibile in commercio.
- Quantificare l'atterramento efficienza per il gene endogeno di interesse mediante qRT-PCR. Per un protocollo dettagliato, vedere riferimento10. Obiettivo per un atterramento efficienza ≥ 60% (Figura 2). Normalizzare i dati per ≥ 2 geni housekeeping (ad es. GAPDH, ACTB, TUBB3, PPIA) utilizzando il più controllo interno gene metodo11.
Nota: Se lavora nel ratto, utilizzare i seguenti primer PCR per i geni housekeeping: GAPDH-fwd: 5' GGTGCTGAGTATGTCGTGGA 3' e GAPDH-rev: 5' GATGATGACCCTTTTGGC 3'; ACTB-fwd: 5' CATCACTATCGGCAATGAGC 3' e ACTB-rev: 5' TCATGGATGCCACAGGATT 3'; TUBB3-fwd: 5' GCCTTTGGACACCTATTCAG 3' e TUBB3-rev: 5' TCACATTCTTTCCTCACGAC 3'; PPIA-fwd: 5' CACTGGGGAGAAAGGATTTG 3' e PPIA-rev 5' CCATTATGGCGTGTGAAGTC 3'.
4. valutare il ruolo delle proteine presinaptiche in circuiti neuronali intatti dalla stimolazione mirata dei neuroni abbattuto con optogenetica
Nota: Il seguente protocollo richiede precedente esperienza con le registrazioni elettrofisiologiche in fettine di cervello acuto e accesso a un'installazione elettrofisiologica.
- Iniettare stereotactically rAAV1/2 nel cervello che segue un protocollo dettagliato nella precedente pubblicazione12. Determinare le coordinate stereotactic per la regione del cervello di interesse utilizzando atlanti stereotactic per mouse13 o ratto cervello14.
- Utilizzare un estrattore micropipetta per tirare Micropipette di iniezione con lunghi steli di Ø 7-9 µm; agganciare le micropipette di iniezione gli stinchi con le forbici. Utilizzare un pennarello sottile e carta millimetrata per inserire segni di taratura sulle Micropipette di iniezione ogni 2 mm.
- Anestetizzare l'animale con isoflurano e fissarlo nell'apparato stereotassica.
- Mantenere l'animale caldo in tutta l'operazione con un rilievo di riscaldamento impostato a 37 ° C.
- Proteggere gli occhi con il lubrificante oculare.
- Radere il pelo sulla testa con un rasoio elettrico.
- Diffondere lo iodio povidone sulla testa rasata utilizzando un batuffolo di cotone.
- Sotto un microscopio per dissezione, praticare un'incisione del midline.
- Pulire la superficie del cranio con un cotton fioc, in modo da rendere visibile il bregma e lambda.
- Posto la micropipetta di iniezione nel suo supporto. Fissare il supporto al braccio stereotassica.
- Determinare gli assi x e y le coordinate del sito di iniezione rispetto il bregma e/o l'espressione lambda.
- Usare un trapano per fluidificare il cranio sopra la zona di destinazione.
Nota: Utilizzare delicati movimenti circolari ed evitare di forare il cranio, onde evitare di danneggiare la superficie del cervello. - Se c'è sanguinamento, assorbire sangue eccessivo con carta asciugamani.
Nota: Sangue eccessivo renderà difficile determinare correttamente la coordinata z. - Caricare la micropipetta iniezione ≤ 2 µ l di virus per azione capillare.
- Portare la micropipetta di iniezione per x e y le coordinate del sito di iniezione.
- Calcolare la coordinata z da dura e abbassare lentamente la pipetta nel cervello.
- Quando viene raggiunta la coordinata z, attendere 3 minuti per permettere aggiustamenti del tessuto.
- Applicare una pressione positiva basso usando una siringa da 1 mL, collegata tramite un tubo flessibile alla parte posteriore della micropipetta iniezione. Controllare visivamente la velocità di espulsione del virus dalla micropipetta iniezione attraverso il microscopio di dissezione e utilizzando la calibratura contrassegna come punti di riferimento.
Nota: Virus deve essere iniettato a velocità lenta (< 100 nL/min) per evitare danni ai tessuti. - Al termine dell'iniezione, attendere 5 minuti, ritirare la micropipetta iniezione di 0,2 mm, attendere per un altro 5 minuti, per evitare il riflusso del virus.
- Ritirare lentamente e completamente la micropipetta iniezione dal cervello e smaltirla in un contenitore riempito con candeggina.
- Bagnare il cranio con soluzione fisiologica e la pelle con 3-4 punti di sutura.
- Applicare pomata gentamicina alla ferita.
- Singole case l'animale in una gabbia pulita con cibo pellet e sotto una lampada di calore fino a che non recupera completamente.
- ≥ 15 giorni post-iniezione, decapitare animali sotto anestesia profonda isoflurano.
- Preparare fette di cervello acuto della regione cerebrale di interesse con un Vibratome utilizzando gassificato (95% O2, 5% CO2) ghiacciata aCSF soluzione, contenente (in mM): 123 NaCl, 1.25 KCl, 1,25 KH2PO4, 1,5 MgCl2, 1 CaCl2, 25 NaHCO3, NaPyruvate 2 e 18 del glucosio (osmolarità regolato a 300 mOsm). Ad esempio, se l'obiettivo è quello di analizzare la trasmissione sinaptica da CA3 di neuroni piramidali CA1, preparare fette sagittali della formazione hippocampal (350 µm di spessore).
Nota: Da questo punto in poi funziona in condizioni di scarsa illuminazione per evitare l'attivazione della sonda optogenetica dalla luce ambientale. - Lasciate le fette a recuperare per 30 min a 37 ° C nella stessa aCSF in un alloggiamento progettato per lo svolgimento di fette del cervello. Tenere le fette la stessa camera di fetta di cervello a temperatura ambiente fino a registrazione.
Nota: Con queste condizioni, fette possono essere mantenuti sani per fino a 6 – 8 h. - Trasferire una fetta di una camera di registrazione sommerso e mantenere con la stessa aCSF utilizzati per il ripristino completato con 1,5 mM CaCl2 a 2 mL/min (Totale Ca2 +: 2,5 mM) e senza NaPyruvate.
Nota: Fette sono preparati e mantenuti a bassa concentrazione di Ca2 + (1 mM) per minimizzare la tossicità. Le registrazioni vengono eseguite in 2,5 mM Ca2 + per favorire il rilascio delle vescicole. - Controllare brevemente il segnale del reporter fluorescente espressa (ad es. TdTomato; Figura 3B) per accertare la localizzazione e l'intensità dell'infezione.
- Riempire un elettrodo di patch con un contenente soluzione intracellulare (in mM): 110 K-gluconato, 22 5 NaCl, KCl, 0,5 EGTA, 3 MgCl2, 4 Mg-ATP, Na 0,53-GTP, 20 K2-fosfato della creatina, 10 HEPES-KOH (pH 7.28, 290 mΩ).
Nota: Utilizzare patch elettrodo con una resistenza di pipetta di 5 – 6 mΩ. - Sotto illuminazione infrarossa, raggiungere Tenuta configurazione di cellule intere da un neurone ricevendo l'input sinaptico dai neuroni infetti. Ad esempio, se i neuroni piramidali CA3 sono stati infettati, patch di neuroni piramidali nella prossimale al tratto mediale della regione CA1 (Figura 3).
Nota: Resistenza in serie può essere lasciato non compensata, ma dovrebbe essere costante e bassa (≤20 MΩ). - Utilizzare farmacologia per isolare le correnti sinaptiche sotto inchiesta. Ad esempio, se l'obiettivo è quello di indagare la trasmissione sinaptica eccitatoria, bloccare la trasmissione sinaptica inibitoria con 10 µM bicucullina.
- Evocano delle correnti sinaptiche, ad esempio, eccitatori correnti postsinaptiche (EPSC), utilizzando un laser blu nm 473 accoppiato ad una fibra ottica (Ø ≤ 250 µm) posizionata sul somata dei neuroni infetti (ad es. Neuroni piramidali CA3).
- Regolare la lunghezza di stimolazione al minimo, per ridurre la possibilità di evocare più di un potenziale di azione per impulso di luce. Se si utilizza ChETA, impostarlo a 2 ms15.
- Regolare la forza di stimolazione del laser per produrre delle correnti sinaptiche piccole ma chiaramente rilevabile (ampiezza di picco ≤ 50 pA per EPSC registrati a un potenziale di holding di -70 mV tra neuroni piramidali CA3 e CA1; Figura 3D). Se si utilizza ChETA e una fibra ottica di 250 µm di diametro, stimolazione punti di forza di 1 – 3 mW a fibra uscita dovrebbe essere adatta.
Nota: se la forza di laser non può essere regolata, utilizzare filtri a densità neutra. - Risplendere la luce di laser 473 nm sul somata dei neuroni infetti, ma non su loro assoni. Ad esempio, splendere la luce sul CA3 somata e lontano dai collaterali di Schaffer, per evitare la depolarizzazione diretta degli assoni.
- Applicare la tetrodotossina (TTX; 0,5 µM), un bloccante dei canali del sodio, al campione per confermare i canali sono basati sul potenziale d'azione.
- In diverse registrazioni, applicare un bloccante selettivo alla corrente sinaptica sotto inchiesta per confermare che la stimolazione è selettiva.
Nota: ad esempio, applicare NBQX (10 µM) al glutammato di tipo AMPA EPSC ricevitore-mediata. - Confrontare otticamente ed elettricamente evocate delle correnti sinaptiche nel cervello acuto stessa fetta in risposta alla stimolazione ripetitiva (≥ 2 stimoli a ≤20 Hz). Un simile grado di facilitazione sinaptica o depressione dovrebbe essere osservato fra stimolazione elettrica ed ottica quando si utilizza un miR di controllo, suggerendo così che simili meccanismi cellulari sono attivati da due tipi di stimolazioni.
Nota: I passaggi sopra riportati sono necessari per garantire che la stimolazione ottica non induce una depolarizzazione diretta dei boutons presinaptici, aggirando così alcuni meccanismi della trasmissione sinaptica.
- Confrontare la trasmissione sinaptica in risposta a stimolazioni singole e ripetitive (≥ 2 stimoli a ≤20 Hz) tra un miR di controllo e miRs targeting per le proteine presinaptiche sotto inchiesta.
Risultati
Le procedure sopra descritte forniscono un metodo affidabile per valutare la trasmissione sinaptica come risente l'atterramento di proteine sinaptiche nei neuroni presinaptici. Risultati rappresentativi su come l'atterramento di alternativa della giuntura isoforme di presinaptici Cav2.1 VGCCs (tipo P/Q) regola la plasticità sinaptica a breve termine presso CA3 di CA1 sinapsi eccitatorie sono riportate di seguito come esempio.
CAv2.1 (P/Q-type) canali sono il VGCCs presinaptici predominante a più veloce sinapsi nel sistema nervoso centrale. Alternativi di splicing degli esoni mutuamente esclusivi 37a e 37b della subunità1 poro-formare α della Cav2.1 (α1A) produce due varianti principali, Cav2.1 [EFa] e Cav2.1 [EFb]16,17 ,18. Per determinare se Cav2.1 [EFa] e Cav2.1 [EFb] differenziale regolano trasmissione sinaptica e la plasticità in neuroni piramidali hippocampal del ratto, abbiamo sviluppato in primo luogo miRs isoforma-specifici per atterramento selettivamente Cav 2.1 [EFa] o Cav2.1 [EFb]5. Malgrado il formato breve (97 bp) e alta somiglianza (61,86% identità a livello del nucleotide) tra gli esoni 37a e 37b, potremmo progettare tre sequenze di miR contro ratto Cav2.1 [EFa] (miR EFa1: TCCTTATAGTGAATGCGGCCG; miR EFa2: ATGTCCTTATAGTGAATGCGG; miR EFa3: TTGCAAGCAACCCTATGAGGA) e due contro ratto Cav2.1 [EFb] (miR EFb1: ATACATGTCCGGGTAAGGCAT; miR EFb2: ATCTGATACATGTCCGGGTAA) con predetto ad alta efficienza di atterramento. Come controllo negativo (miR controllo), abbiamo usato il plasmide pcDNA6.2-GW/EmGFP-miR-neg contenente una sequenza che non mira ad alcun gene di vertebrati conosciuti. Basato su una prima schermata in cellule HEK 293 contro eterologhe canali, abbiamo selezionato miR EFa1, miR EFa3 e miR EFb2 e clonato loro cassette di espressione nel 3' UTR del vettore pAAV-Syn-ChETA-TdT-miR-X, che è stato progettato per la produzione di rAAVs e dove la promotore della sinapsina unità espressione della channelrhodopsin ultraveloce ChETA, la proteina fluorescente rossa TdTomato e il miR inserito (Figura 1). Abbiamo anche ripetuto la cassetta di espressione di miR EFb2 per aumentare l'efficienza atterramento di questo miR.
Successivamente, abbiamo preparato rAAV1/2 per i costrutti di quattro sopra e quantificati loro atterramento efficienza e selettività in colture neuronali primarie del ratto usando isoforma-specifici qRT-PCR. miR EFa1 e miR EFa3 ridotto mRNA di nativo Cav2.1 [EFa] ~ 70% ma non quella di Cav2.1 [EFb], mentre miR EFb ridotto mRNA di nativo Cav2.1 [EFb] ~ 60% ma non quella di Cav2.1 [EFa] (Figura 2).
Abbiamo quindi stereotactically iniettato ognuna delle quattro rAAV1/2 nella zona CA3 dell'ippocampo dei ratti P18 (Figura 3A-C), con le coordinate di −2.6 (A-P/M-L/D-V da Bregma) / ± 2,9 / −2.9. Post-iniezione di quindici a venti-quattro giorni, abbiamo preparato acute fette hippocampal dai ratti rAAV1/2-iniettato e usato TdTomato fluorescenza per confermare l'espressione e la localizzazione di rAAVs (Figura 3B). Per studiare se presinaptico atterramento di Cav2.1 [EFa] o Cav2.1 [EFb] interessato plasticità sinaptica a breve termine presso CA3 alle sinapsi CA1, abbiamo stimolato infettati selettivamente neuroni CA3 con breve 473 nm luce impulsi laser (2 ms-lungo ; Figura 3) e registrato l'EPSC risultante dalla rattoppatura neuroni piramidali in prossimale al tratto mediale della regione CA1 (Figura 3). Abbiamo trovato che l'atterramento di isoforme di splicing di Cav2.1 influenzato le risposte alla stimolazione di accoppiare-impulso in direzioni opposte: atterramento di Cav2.1 [EFa] (miR EFa1 o miR EFa3) potenziato facilitazione di accoppiare-impulso (PPF) mentre atterramento di Ca v2.1 [EFb] (miR EFb2) abolita (Figura 3D, E).
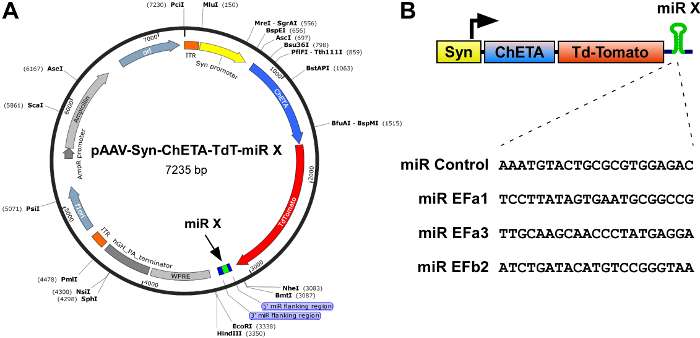
Figura 1: Schema dei costrutti per l'espressione combinata della sonda ultraveloce optogenetica ChETA e miRs isoforma-specifici contro Cav2.1 [EFa] e Cav2.1 [EFb]. (A) mappa della rAAV costruire pAAV-Syn-ChETA-TdT-miR-X, contenente un promotore della sinapsina (Syn), il channelrhodopsin ultraveloce ChETA fusa per TdTomato e, nel prossimale 3' UTR, un miR isoforma-specifici di Cav2.1 della giuntura. Enzimi di restrizione mostrati sono singole frese. (B) schema Top, di cassetta di espressione. Il promotore della sinapsina unità espressione di ChETA-TdTomato e di un miR isoforma-specifici. Fondo, complemento inverso delle sequenze bersaglio di 21 nucleotidi, che formano parte della cassetta miR. Per miR EFb2 due cassette di miR identici sono stati espressi in tandem, uno dopo l'altro. Clicca qui per visualizzare una versione più grande di questa figura.
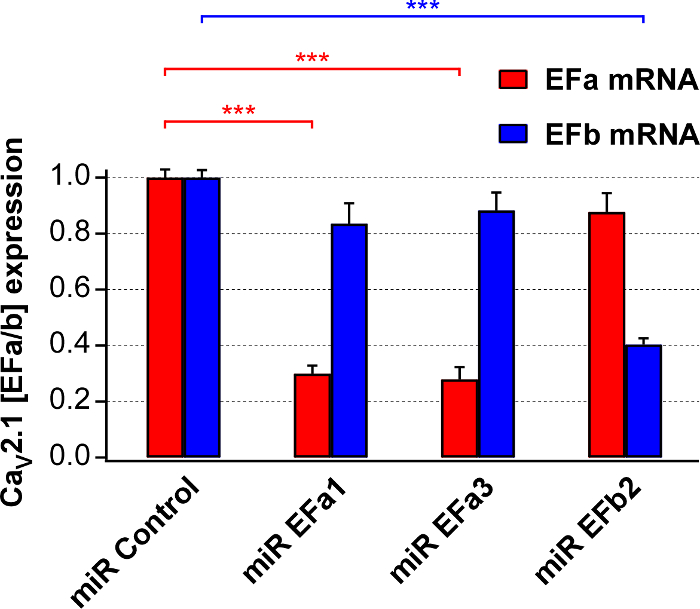
Nella figura 2. Valutazione dell'efficienza di atterramento e selettività dell'isoforma-specifici miRs per Cav2.1 [EFa] e Cav2.1 [EFb]. Analisi specifica isoforma qRT-PCR su RNA isolato da colture primarie di 17-18 DIV infettati a 6 DIV rAAVs esprimendo miRs targeting sia Cav2.1 [EFa] (miR EFa1 e miR EFa3) o Cav2.1 [EFb] (miR EFb2). I dati vengono normalizzati per il controllo negativo (miR controllo). miR EFa1 e miR EFa3 significativamente e selettivamente ridurre mRNA di Cav2.1 [EFa] (n = 8 e 7 culture, rispettivamente), mentre miR EFb significativamente e in modo selettivo riduce mRNA di Cav2.1 [EFb] (n = 4 culture; * * * p < 0,001; unidirezionale analisi di test della varianza seguita dal post-test di Tukey-Kramer). I dati sono presentati come media ± SEM Questa figura è stata adattata da Thalhammer, a. et al. 5. per favore clicca qui per visualizzare una versione più grande di questa figura.

Nella figura 3. Valutazione del ruolo di Cav2.1 [EFa] e Cav2.1 [EFb] nell'ippocampo nativo di mirati stimolazione dei neuroni abbattuto con optogenetica. (A) schema della cassetta espressione dei costrutti rAAV usati per l'infezione in vivo . (B) sezione Hippocampal risultati quello fluorescenza TdTomato è limitata alla regione CA3 e le sue proiezioni. Configurazione sperimentale (C) : raggio laser è stata convogliata sulla CA3 somata e patch morsetto registrazioni sono state eseguite da neuroni piramidali CA1. (D) 2 ms-lungo blu (473 nm) impulsi di luce laser ha brillati a 20 Hz evocano EPSC cui PPF è aumentato del miRs Cav2.1 [EFa] di targeting e abolito da miR per Cav2.1 [EFb]. (E) Riepilogo del rapporto di accoppiare-impulso per esperimenti come in (D), mostrando un aumento in PPF per miR EFa1 e miR EFa3 e una diminuzione per miR EFb2, relativo a miR controllo (n = 9-11 registrazioni; * p = 0,02; * * p = 0,01; * * * p < 0,0004; analisi della covarianza). I dati sono presentati come media ± SEM Questa figura è stata adattata da Thalhammer, a. et al. 5. per favore clicca qui per visualizzare una versione più grande di questa figura.
Discussione
Co-espressione di una sonda di optogenetica e un miR contro un gene presinaptico di interesse offre un approccio potente per caratterizzare l'effetto di atterramento di gene sulla funzione presinaptica all'interno di circuiti neuronali intatti. Per questo approccio sperimentale, è importante identificare e caratterizzare miRs che sono altamente efficienti e selettivi nell'abbattere il gene di interesse in sistemi nativi. Se possibile, due o più indipendenti miRs contro stesso mRNA di interesse deve essere utilizzato per controllo per eventuali effetti fuori bersaglio. Esperimenti di salvataggio, in cui un gene miR-resistente viene reintrodotto nel sistema, possono essere utilizzati come controllo per specificità.
Utilizzando lo stesso costrutto per esprimere un optogenetica sonda e un miR consente per stimolare otticamente solo i neuroni presinaptici che sono stati manipolati. Questo non è possibile con gli stimoli elettrici perché essi non fanno distinzione tra neuroni infetti e non infetti, così producendo risultati misti e diluiti. Perché stimolazioni ottiche possono indurre più potenziali d'azione15, è importante scegliere solo le sonde più veloci di optogenetica, tra un'espansione tavolozza15,19,20,21. Inoltre, è essenziale per garantire che la stimolazione ottica non induce una depolarizzazione diretta dei boutons presinaptici, per evitare di ignorare alcuni dei passaggi della trasmissione sinaptica si vuole indagare22.
L'approccio sperimentale che descriviamo qui, rende possibile valutare in parallelo la rilevanza fisiologica dei geni multipli presinaptici di interesse all'interno di un periodo di tempo limitato (4 – 6 mesi). Tuttavia, è importante tenere a mente che atterramenti raramente raggiungono il 100%. Inoltre, rAAVs bisogno di essere espresso per almeno due settimane consentire massima espressione di atterramento e completo della sonda optogenetica, che può rappresentare un vincolo di tempo durante l'analisi di processi di sviluppo precoce. Anche se più topi knockout richiede tempo, condizionale limitati ai neuroni presinaptici, provocano generalmente completa rimozione del gene di interesse e offrono pertanto un valido approccio complementare.
Un vantaggio specifico della tecnologia miR è che permette l'espressione di Mir multiple dal promotore stesso. Questa proprietà è stata utilizzata principalmente per aumentare l'efficienza di atterramento con l'inserimento di più copie della stessa miR o miRs diverse contro lo stesso gene bersaglio. Può tuttavia essere utilizzata anche per atterramento geni multipli esprimendo miRs contro diversi target geni7,23. Questa proprietà potrebbe essere utilizzata per mettere a tacere più proteine presinaptiche per sezionare le vie di segnalazione presinaptiche.
Qui, abbiamo combinato optogenetica con artificiale miRs per caratterizzare gli effetti di atterramento di gene sulla funzione presinaptica in fettine ippocampali acute. Un simile approccio potrebbe essere utilizzato anche per manipolare e sonda circuito neuronale in vivo. Inoltre, combinando miRs artificiale con chemogenetic approcci consentirebbe uno interrogare i circuiti neuronali su scale di tempo più lungo.
Divulgazioni
Gli autori non hanno nulla a rivelare.
Riconoscimenti
Ringraziamo F. Benfenati (Istituto Italiano di Tecnologia, IIT) per supporto e Carmela Vitale per aiutare con la dimostrazione. Questo lavoro è stato finanziato da IIT e la Compagnia di San Paolo (concessione n. 9734 al LAC).
Materiali
| Name | Company | Catalog Number | Comments |
| Acrylamide | Sigma | Acrylamide/Bis-acrylamide, 30% solution | Toxic |
| Animal Temperature Controller with heat pad | WPI | ATC2000 | |
| BCA protein assay kit | ThermoFisher Scientific | 23225 | |
| BLOCK-iT Pol II miR RNAi Expression Vector kit | ThermoFisher Scientific | K4936-00 | |
| Brain slices Prechamber | Harvard Apparatus | BSC-PC | |
| CCD camera-based imager | Bio-Rad | ChemiDoc™ MP | |
| Cell Culture reagents | Life Technologies | ||
| Chemiluminescent substrate | GE Helthcare | ECL Western Blotting Reagents, RPN2106 | |
| Chloroform | Sigma | C2432 | Toxic |
| Cytosine β-D-arabinofuranoside | Sigma | C6645 | |
| Drill | Foredom | K.1030 | |
| EGTA | Sigma | E4378 | |
| Gentamicin ointment | Local pharmacy | ||
| Glucose | Sigma | G7021 | |
| Hepes | Sigma | 54459 | |
| Injection micropipettes | Narishige | GD1 | |
| Inorganic salts & detergents | Sigma | ||
| K2-creatine phosphate | Calbiochem | 237911 | |
| KGluconate | Fluka | 60245 | |
| Laser | Laserglow Technologies | LRS-0473-GFM-00100-03 | |
| Membrane | Amersham | Protran™ 0.2 µm NC | |
| MgATP | Sigma | A9187 | |
| Micropipette holder | Narishige | IM-H1 | |
| Micropipette puller | Narishige | PC-100 | |
| Na3GTP | Sigma | G8877 | |
| NaPyruvate | Sigma | P5280 | |
| Ocular lubricant | Local pharmacy | Lacrigel | |
| Phosphate inhibitors | Sigma | P0044, P5726 | |
| Protease inhibitors | Sigma | cOmplete™, EDTA-free Protease Inhibitor Cocktail | |
| Protein gel electrophoresis and blotting devices | Bio-Rad | Mini-Protean III Cell | |
| Providone iodine | Local pharmacy | Betadine | |
| Qiazol Lysis Reagent | Qiagen | 79306 | Toxic |
| QuantiTect Reverse Transcription Kit | Qiagen | 205311 | |
| RNeasy Micro Kit | Qiagen | 74004 | |
| Spectrophotometer | ThermoFisher Scientific | Nanodrop 2000 | |
| Stereotactic apparatus | WPI | ||
| Tetrodotoxin | Tocris | 1069 | Toxic |
| Vibratome | Leica | VT1200S |
Riferimenti
- Lam, J. K., Chow, M. Y., Zhang, Y., Leung, S. W. siRNA Versus miRNA as Therapeutics for Gene Silencing. Mol Ther-Nucl Acids. 4, e252(2015).
- Kohl, M. M., et al. Hemisphere-specific optogenetic stimulation reveals left-right asymmetry of hippocampal plasticity. Nat Neurosci. 14 (11), 1413-1415 (2011).
- Maejima, T., et al. Postnatal loss of P/Q-type channels confined to rhombic-lip-derived neurons alters synaptic transmission at the parallel fiber to purkinje cell synapse and replicates genomic Cacna1a mutation phenotype of ataxia and seizures in mice. J Neurosci. 33 (12), 5162-5174 (2013).
- Mark, M. D., et al. Delayed postnatal loss of P/Q-type calcium channels recapitulates the absence epilepsy, dyskinesia, and ataxia phenotypes of genomic Cacna1a mutations. J Neurosci. 31 (11), 4311-4326 (2011).
- Thalhammer, A., et al. Alternative Splicing of P/Q-Type Ca2+ Channels Shapes Presynaptic Plasticity. Cell Rep. 20 (2), 333-343 (2017).
- Sambrook, J., Russell, D. W. Calcium-phosphate-mediated Transfection of Eukaryotic Cells with Plasmid DNAs. CSH Protoc. 2006 (1), (2006).
- Chung, K. H., et al. Polycistronic RNA polymerase II expression vectors for RNA interference based on BIC/miR-155. Nucleic Acids Res. 34 (7), e53(2006).
- McClure, C., Cole, K. L., Wulff, P., Klugmann, M., Murray, A. J. Production and titering of recombinant adeno-associated viral vectors. J Vis Exp. (57), e3348(2011).
- Cingolani, L. A., et al. Activity-dependent regulation of synaptic AMPA receptor composition and abundance by beta3 integrins. Neuron. 58 (5), 749-762 (2008).
- Huang, H., Xu, Y., Cheng, C. Detection of alternative splicing during epithelial-mesenchymal transition. J Vis Exp. (92), e51845(2014).
- Vandesompele, J., et al. Accurate normalization of real-time quantitative RT-PCR data by geometric averaging of multiple internal control genes. Genome Biol. 3 (7), (2002).
- Cetin, A., Komai, S., Eliava, M., Seeburg, P. H., Osten, P. Stereotaxic gene delivery in the rodent brain. Nat Protoc. 1 (6), 3166-3173 (2006).
- Paxinos, G., Franklin, K. B. The Mouse Brain in Stereotaxic Coordinates. , Academic Press. (2012).
- Paxinos, G., Watson, C. The Rat Brain in Stereotaxic Coordinates. , Academic Press. (2013).
- Gunaydin, L. A., et al. Ultrafast optogenetic control. Nat Neurosci. 13 (3), 387-392 (2010).
- Bourinet, E., et al. Splicing of alpha 1A subunit gene generates phenotypic variants of P- and Q-type calcium channels. Nat Neurosci. 2 (5), 407-415 (1999).
- Chaudhuri, D., et al. Alternative splicing as a molecular switch for Ca2+/calmodulin-dependent facilitation of P/Q-type Ca2+ channels. J Neurosci. 24 (28), 6334-6342 (2004).
- Soong, T. W., et al. Systematic identification of splice variants in human P/Q-type channel alpha1(2.1) subunits: implications for current density and Ca2+-dependent inactivation. J Neurosci. 22 (23), 10142-10152 (2002).
- Berndt, A., et al. High-efficiency channelrhodopsins for fast neuronal stimulation at low light levels. P Natl Acad Sci USA. 108 (18), 7595-7600 (2011).
- Klapoetke, N. C., et al. Independent optical excitation of distinct neural populations. Nat Methods. 11 (3), 338-346 (2014).
- Lin, J. Y., Lin, M. Z., Steinbach, P., Tsien, R. Y. Characterization of engineered channelrhodopsin variants with improved properties and kinetics. Biophys J. 96 (5), 1803-1814 (2009).
- Zhang, Y. P., Oertner, T. G. Optical induction of synaptic plasticity using a light-sensitive channel. Nat Methods. 4 (2), 139-141 (2007).
- Fowler, D. K., Williams, C., Gerritsen, A. T., Washbourne, P. Improved knockdown from artificial microRNAs in an enhanced miR-155 backbone: a designer's guide to potent multi-target RNAi. Nucleic Acids Res. 44 (5), e48(2016).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon