Method Article
Kombination von Optogenetik mit künstlichen Mikro-RNAs, die Effekte gen Knockdown präsynaptischen Funktion innerhalb intakte neuronale Schaltkreise zu charakterisieren
In diesem Artikel
Zusammenfassung
Dieses Protokoll enthält einen Workflow, künstliche MicroRNA-vermittelten RNA-Interferenz mit Optogenetik Förderung insbesondere präsynaptischen Boutons mit reduzierten Expression von selektiven-Gens in intakte neuronale Schaltkreise zu kombinieren.
Zusammenfassung
Dieses Protokoll soll die Auswirkungen von gen-Knockdown auf präsynaptischen Funktion innerhalb intakte neuronale Schaltkreise zu charakterisieren. Wir beschreiben einen Workflow wie künstliche MicroRNA (MiR) zu kombinieren-vermittelten RNA-Interferenz mit Optogenetik, selektive Stimulierung der manipulierten präsynaptischen Boutons in akuten Hirnschnitten zu erreichen. Der experimentelle Ansatz beinhaltet die Verwendung einer einzigen virale Konstrukt und einzelne Neuron-spezifische Förderer, den Ausdruck einer optogenetische Sonde und künstliche MiR(s) gegen präsynaptischen ursächlich zu fahren. Wenn hierbei in der Hirnregion Interesse injiziert, macht das ausdrückliche Konstrukt es möglich mit Licht ausschließlich die Neuronen mit reduzierten Ausdruck der Gene untersucht zu stimulieren. Diese Strategie erfordert die Entwicklung und Wartung von gentechnisch veränderten Mauslinien und kann im Prinzip auf andere Organismen und gelten alle neuronalen gen Wahl. Wir haben vor kurzem angewendet, um zu untersuchen, wie die Knockdown von alternativen Spleiß Isoformen von präsynaptischen P/Q-Typ Voltage-gated Calciumkanäle (VGCCs) kurzfristige synaptische Plastizität bei CA3, CA1 exzitatorischen Synapsen in akuten hippocampal Scheiben regelt. Ein ähnlicher Ansatz könnte auch verwendet werden, zu manipulieren und Sonde die neuronalen Schaltkreise in Vivo.
Einleitung
Dieses Protokoll beschreibt einen neuen Ansatz, um die Auswirkungen von gen-Knockdown auf präsynaptischen Funktion innerhalb intakte neuronale Schaltkreise zu charakterisieren. Untersuchung von präsynaptischen Funktion in intakte neuronale Schaltkreise ist schwierig, weil viele präsynaptischen Boutons zu klein sind und weit entfernt von der Soma kombinierte molekulare und elektrophysiologische Interventionen zu ermöglichen. RNA-Interferenz bietet ein leistungsstarkes und flexibles Mittel, um Knockdown synaptischen Proteine1, wurde dieser Ansatz sparsam verwendet, um präsynaptischen Funktion zu untersuchen, denn es schwer ist zu erkennen, die Auswirkungen der Zuschlag mit herkömmlichen elektrische Stimulationen, die nicht zwischen unterscheiden manipuliert und naiv präsynaptischen Boutons2. Wir beschreiben hier, wie künstliche MicroRNA (MiR) zu kombinieren-vermittelten RNA-Interferenz mit neu entwickelten optogenetische-Technologie, selektive Stimulierung der manipulierten präsynaptischen Boutons in akuten Hirnschnitten zu erreichen.
Während bedingte Knockout-Mäusen in Kombination mit Elektrophysiologie auch genutzt werden, könnte zu untersuchen, die Funktion der präsynaptischen Proteine3,4, erfordert unsere Strategie der Entwicklung und Wartung von genetisch keine Mauslinien geändert und kann auch leicht abzureißen spezifische Isoformen eines Gens eingesetzt werden. Relativ zum häufiger verwendeten kurze Haarnadel RNAs (ShRNAs), künstliche MiRs, die wir hier einsetzen, bieten entscheidende Vorteile für Zuschlag in Neuronen. Im Gegensatz zu ShRNAs können sie unter der Kontrolle einer Polymerase II Förderer1ausgedrückt werden. So kann einzelne Förderer verwendet werden, um den Ausdruck von MiR und eine optogenetische Sonde, zusammen mit einem fluoreszierenden Reporter zu fahren. Auf diese Weise kann die Größe des Konstrukts Verpackungen im Rahmen der recombinant Adeno-assoziierten Viren (rAAV, Abb. 1A) gehalten werden. Die Verwendung einer einzigen Konstrukt und einzelne Förderer verringert ebenfalls experimentelle Variabilität, weil sie Ausdruck der MiR, die optogenetische-Sonde und der fluoreszierenden Reporter in einem festen Verhältnis ermöglicht.
Vor kurzem haben wir diese Technologie, um die Rolle der alternativ gespleißten Isoformen von präsynaptischen Kalziumkanäle in den Hippocampus5untersuchen eingesetzt. Eine solche Strategie ist generell für die physiologische Relevanz anderer presynaptically exprimierten Proteine in jedem Gehirn Schaltung von Interesse zu studieren.
Protokoll
Alle Experimente wurden gemäß den Richtlinien der Europäischen Gemeinschaften Rat (Richtlinie 2010/63/EU des 4. März 2014) durchgeführt und wurde vom italienischen Gesundheitsministerium genehmigt.
1. Aufbau von mikroRNAs für RNA-Interferenz und Bewertung ihrer Effizienz im heterologen Expressionssysteme
Hinweis: Dieses Protokoll erfordert Kenntnisse über die folgenden bewährten Methoden: molekulare Klonierung, DNA-Sequenzierung, Wartung von Zelllinien, Transfektion Kalziumphosphat, quantitative Real time PCR (qRT-PCR), Vorbereitung der Zelle Lysates von Zelllinien und westliche Beflecken.
- Bestimmen Sie, ob das gen des Interesses alternatives Spleißen unterliegt. Wählen Sie für eine allgemeine Zuschlag des Gens ausschließlich konstitutive Exons; Wählen Sie für einen Zuschlag von einem bestimmten alternativ gespleißten Isoform das entsprechende Alternativ gespleißte Exon.
- Verwenden Sie speziellen Software (z.B. https://rnaidesigner.thermofisher.com/rnaiexpress/), künstliche MiRs für RNA-Interferenz gegen die Reihenfolge des Interesses zu entwerfen.
Hinweis: Es ist wichtig, eine grundlegende lokale Ausrichtung Suche Tool (BLAST) verwenden, um mögliche off-Ziele innerhalb der gleichen Art überprüfen. - Wählen Sie die oberen drei gewerteten MiRs für das Zielgen.
- Bestellen Sie die oberen und unteren Stränge der ausgewählten MiRs von einem geeigneten Unternehmen. Für negative Kontrolle, verwenden Sie eine verschlüsselte Version von einem der ausgewählten MiRs oder eine MiR vorhergesagt nicht gegen alle bekannten gen der Gattung im Experiment verwendet (zB. für Wirbeltiere Gene, die MiR im pcDNA6.2-GW/EmGFP-MiR-Neg).
- Tempern Sie die jeweiligen oberen und unteren Stränge der ausgewählten MiRs und Klonen sie in ein Plasmid für Ausdruck der MiRs (z. B. pcDNA6.2-GW/EmGFP-MiR) mit standard-Klonen-Strategien entwickelt.
Hinweis: Das Ziel ist die Schaffung einer MiR Expressionskassette bestehend aus einer 5' MiR flankierenden Region, die ausgewählten MiR Sequenz und ein 3' MiR flankierenden Region, die von der 3' UTR von einem Reporter-gen unter der Kontrolle eines RNA-Polymerase-Typ-II-Promotors ausgedrückt werden kann. - Verwenden Sie DNA-Sequenzierung, um das korrekte Einfügen der ausgewählten MiRs zu validieren.
- HEK293 Zellen in einem befeuchteten Zelle Kultur Inkubator (37 ° C, 5 % CO2) wachsen mit DMEM Medium ergänzt mit 10 % fötalen Rinderserum, 1 mM NaPyruvate, 10 mM HEPES (pH 7.39) und 1 X Penicillin/Streptomycin (Pen/Strep).
- Wenn HEK293 Zellen ~ 70 % Zusammenfluss, Co transfizieren sie mit jedem der MiR Expressionsvektoren und einen Vektor mit dem Ausdruck des gezielte Gens im Verhältnis 1:1 DNA mit der Calcium-Phosphat-Methode6. Die folgenden Steuerelemente enthalten: (i) untransfected Zellen, (Ii) Zellen transfiziert mit der Vektor mit dem Ausdruck des gezielte Gens gemeinsam mit beiden Träger DNA (z. B. pBluescript II SK(+)) oder (Iii) MiR Kontrolle.
Hinweis: (i) das ausdrückliche gezielte gen muss aus der gleichen Spezies, in denen denen welche Richtung die MiRs entworfen wurden, es sei denn, die Sequenzen gezielt durch die MiRs unbedingt auf die Nukleotid-Ebene zwischen den beiden Arten konserviert sind. (Ii) Zelllinien als HEK293 einsetzbar. (Iii) alternative Methoden der Transfektion, wie Elektroporation oder Liposomen-basierte Methoden können verwendet werden. -
48 h nach Transfektion, lösen Sie die Zellen, laufen Protein Gel, Western-Blot durchzuführen und Eiweißgehalt mit einem Antikörper erkennen das Zielprotein zu analysieren. Streben Sie nach einem Knockdown Wirkungsgrad von ≥50 %.
Hinweis: Knockdown Effizienz kann alternativ oder zusätzlich (i) von ELISA Analyse, (Ii) durch Messung der Aktivität des Zielproteins falls zutreffend bewertet werden (z. B. Stromdichten für Ionenkanäle) oder (Iii) durch qRT-PCR auf der mRNA-Ebene, wenn geeignete Antikörper sind nicht verfügbar (Protokoll (Schritt 3).- Legen Sie die Kultur-Gerichte auf Eis und waschen Sie die Zellen einmal mit eiskalten Phosphat-gepufferte Kochsalzlösung (PBS).
- Fügen Sie die PBS Aspirieren eiskalte RIPA-Puffer (50 mM Tris-HCl pH 7.4, 150 mM NaCl, 2 mM EDTA, 1 % NP40, 0,1 % Sodium Dodecyl Sulfat (SDS), Phosphatase, Protease-Inhibitoren enthalten; 1 mL für eine Schüssel mit Ø 100 mm, 0,5 mL für eine Schüssel mit Ø 60 mm).
- Die Schüssel mit einem Schaber Zelle adhärente Zellen abkratzen und sanft die Zellsuspension in einem vorgekühlten Microcentrifuge Schlauch übertragen.
- Zentrifugieren Sie das Rohr auf 15.000 x g für 15 min bei 4 ° C, übertragen Sie den Überstand in ein neues vorgekühlte Microcentrifuge Schlauch und entsorgen Sie das Pellet.
- Bestimmen Sie die Konzentration des Proteins mit dem BCA Protein Assay Kit oder andere geeignete Methode (z.B. Bradford-Test).
Hinweis: Proben werden bei-20 ° C oder-80 ° C für eine spätere Verwendung eingefroren oder sofort bearbeitet. - Übertragen Sie einem entsprechenden Volumen von Lysaten auf Mikrozentrifugenröhrchen, so dass alle Proben die gleichen Proteinkonzentration haben und angemessene eiskalte Lyse Puffer zu machen alle Lysates auf derselben Festplatte hinzufügen.
Hinweis: 30 bis 50 µg des Gesamt-Protein sollte für die meisten Proteine ausreichen, aber die entsprechende Menge zu ladende festgelegt werden, nach der Fülle des Proteins des Interesses. - Fügen Sie einen angemessenen Betrag von 2 X Laemmli-Puffer (4 % SDS, 2-Mercaptoethanol 10 %, 20 % Glycerin, 0,004 % Bromophenol blue, 0,125 M Tris-HCl, pH 6,8) und die Proben bei 95 ° C für 5 min kochen.
- Laden Sie die Proben auf einem Acrylamid Gel zusammen mit einem Molekulargewicht Marker. Führen Sie das Gel bei 100 V für 1-2 h.
Hinweis: Die Gel-Prozentsatz hängt von der Größe des Proteins des Interesses. - Montieren Sie das Transfer-Sandwich und übertragen Sie die Proteine aus dem Gel auf eine Membran bei 100 V 2 h zu. Die Membran kann entweder Nitrozellulose oder PVDF. Aktivieren Sie PVDF mit Methanol für 1 min und spülen Sie ihn mit Transfer-Puffer vor der Vorbereitung des Stapels.
- Die Membran für 1 h bei Raumtemperatur mit blockierenden Puffer zu blockieren (5 % Milch in PBST (PBS + 0,1 % Tween-20)), dann die Membran über Nacht bei 4 ° C inkubieren Sie mit Primärantikörper in blocking-Puffer verdünnt.
- Die Membran für 10 min in PBST dreimal waschen und mit HRP-konjugierten Sekundärantikörper in blocking-Puffer für 1 h bei Raumtemperatur inkubieren.
- Waschen Sie die Membran für 10 min in PBS viermal, dann gelten Sie die Chemolumineszenz Substrat für die Membran und erfassen Sie die Chemolumineszenz Signale mit einer CCD Kamera-basierte Imager.
- Verwenden Sie Bildanalyse-Software, um die Knockdown Effizienz zu quantifizieren.
- Wählen Sie die zwei leistungsfähigsten MiRs targeting nicht überlappende Sequenzen für weitere funktionelle Assays.
Hinweis: Ab Ziele Effekte können nie für alle MiR ausgeschlossen werden. Wenn zwei unabhängige MiRs eine ähnliche Wirkung haben, ist es jedoch äußerst unwahrscheinlich, dass dies wegen einer gleichen Ziel Knockdown. - Wenn die Knockdown Effizienz der ausgewählten MiRs nicht zufriedenstellend ist (< 50 %), Bildschirm für andere MiRs. Alternativ Ausdrücken zum Ausdruck bringen das effizienteste MiR(s) im Tandem, d. h. sie in mehrere Kopien der gleichen Expressionskassette, Knockdown Effizienzsteigerung7verursachen.
2. Konstruktion des Recombinant Adeno-assoziierten Vektoren für kombiniert Ausdruck von optogenetische Sonden und Micro-RNAs
Hinweis: Dieses Protokoll erfordert Kenntnisse über die folgenden bewährten Methoden: molekulare Klonierung, DNA-Sequenzierung und rAAV-Produktion.
- Klon der effizienteste MiR Expressionskassetten in der 3' UTR von Konstrukten, die für die Produktion von rekombinanten rAAVs und Ausdruck der exzitatorischen optogenetische Sonden, z. B. in pAAV-Syn-ChETA-TdT-miR-X (Abbildung 1A), wo die Neuron-spezifische Wirbeltiere Promotor treibt Ausdruck der ultraschnellen Channelrhodopsin ChETA rot fluoreszierende Reporter TdTomato und der MiR(s) zwischen den NheI und EcoRI Sites5eingefügt.
Hinweis: (i) nicht übertreffen der Verpackung maximal rAAVs, das ist ca. 4,7 Kb Länge von ITR (inverted terminal Repeat) bis ITR (Abbildung 1A, Orange). (Ii) die MiR Expressionskassette muss zwischen Stopp-Codon und WPRE (Murmeltier Hepatitis Virus posttranskritionelle regulatorisches Element; eingefügt werden Abbildung 1A). (Iii) die Aufnahme eines fluoreszierenden Proteins, wie TdTomato, ist sehr nützlich, da es ermöglicht leicht Überwachung der Lokalisierung und der Intensität der Infektion (Protokoll (Schritt 4). - Verwenden Sie DNA-Sequenzierung, um das korrekte Einfügen der MiR Kassetten zu überprüfen.
- Produzieren und Titer rAAV1/2 gemäß den zuvor veröffentlichten Protokoll8, ein rAAV1/2 für jedes ausgewählte MiR. Ein typisches Experiment für den Zuschlag von einem Gen würde zwei unabhängige MiRs gegen die gleichen gen und einem MiR Kontrolle umfassen. Streben nach einem Virustiter ≥109 viraler Genome (Vg) / µL.
Achtung: rAAVs in einer Einrichtung Biosafety Level 1 (BSL-1) behandelt werden müssen. Bitte überprüfen Sie mit institutionellen Biosafety Committee für detaillierte Informationen.
Hinweis: Wenn das Labor eine virale Anlage keinen oder virale Titer nicht zufriedenstellend sind, kann rAAV Produktion ausgelagert werden. Besuchen Sie zum Beispiel https://www.med.upenn.edu/gtp/vectorcore/ oder https://vcf.charite.de/en/metas/.
3. Gewinnung von RNA aus neuronalen Primärkulturen für die Bewertung von MiR Knockdown Effizienz des endogenen Genen durch qRT-PCR
Hinweis: (i) dieses Protokoll erfordert Kenntnisse über die folgenden bewährten Methoden: Erstellung und Pflege von neuronalen Primärkulturen und qRT-PCR. (Ii) die Quantifizierung der Knockdown Effizienz (Schritte 3.1 – 3.14) mindestens 3 Mal wiederholen (biologische Wiederholungen). (Iii) Schätzung der Knockdown Effizienz auf mRNA-Ebene durch die qRT-PCR ist geeignet, wenn eine Analyse des Eiweißgehalts ausgeschlossen ist, z. B. wenn Isoformen umzuwerfen alternativ gespleißt für die spezifische Antikörper nicht verfügbar5sind.
-
Bereiten Sie neuronale Primärkulturen aus der Region des Gehirns von Interesse. Führen Sie das Protokoll für kortikale Kulturen in früheren Veröffentlichung9, mit den folgenden Änderungen gegeben:
- Platte Neuronen im 6-well-Platten bei einer Dichte von 500.000 Neuronen pro Bohrloch. Verwenden Sie 2,5 mL/Well Anlage Medium und 3,3 mL/auch der Wartung Medium.
- Wenn Astrozyten Überwucherung zu beobachten ist, fügen Sie 0,5 mL/Well Wartung Medium ergänzt mit 7,5 µM Cytosin β-D-Arabinofuranoside (für eine Endkonzentration von 1 µM) bei 3 – 4 Tage in Vitro (DIV).
-
Drei Brunnen pro MiR (technische Wiederholungen) zu infizieren, bei 5–6 DIV. Verwendung der niedrigsten infektiöse Dosis die ≥99 % der Nervenzellen befallen wird.
- 2 mL des Mediums aus jedem Brunnen zu entfernen und in ein 50 mL Falcon-Röhrchen zu sammeln.
- Fügen Sie Virus der Neuronen direkt hinzu, mischen Sie vorsichtig und legen Sie die Platten wieder in den Inkubator bei 37 ° C.
- Speichern Sie die gesammelten Mittel im Inkubator. Verlieren Sie die Kappe von der Falcon-Röhrchen Gasgleichgewichtherstellung zuzulassen.
- Fügen Sie nach 24 h die entfernten mittlere zurück in jede Vertiefung (1,9 mL/Na).
-
Bei 17–18 DIV, lösen Sie die Neuronen für die RNA-Extraktion.
Achtung: Lyse-Reagenz und Chloroform sind hochgiftig. Arbeiten unter einer chemischen Haube und Schutzausrüstung tragen; Entsorgen von Abfällen nach Ihren nationalen und institutionellen Vorgaben.
Hinweis: Schritte 3.3 – 3.13 in RNAse-freie Bedingungen erarbeiten. Tragen Sie Handschuhe und RNAse-freie Glaswaren und Einweg Plastikware. Allgemeine Vorsichtsmaßnahmen wie Sie Griff RNA finden Sie zum Beispiel in Anhang A der RNeasy Micro Handbuch verfügbar online.- Inkubieren Sie Glas, das Pasteur über Nacht im Ofen bei 180 ° c Pipetten
- Kippen Sie die Platten und aspirieren Sie das Medium vollständig aus jeder nun mit einem Glas Pasteurpipette mit einer Vakuumpumpe verbunden.
- Fügen Sie sofort 700 µL Lyse-Reagenz in jede Vertiefung.
- Die Lösung von Schaukeln und schütteln die Platte vorsichtig und kurz mit der hand gleichmäßig.
- Pipette die Lösung oben und unten 4 – 5 Mal oder bis eine homogene Suspension erreicht ist.
- Übertragen Sie die Lösung von jedem Bohrloch in einer separaten 1,5 mL Eppendorf Rohr.
Hinweis: Die Zelle Lysates können bei-80 ° C gelagert oder sofort bearbeitet.
- Bei Bedarf tauen Sie Zelle Lysates bei RT auf, und gehen Sie sofort zum Schritt 3.5.
- Jede Probe der Zelle Lysates 140 µL Chloroform hinzufügen.
Hinweis: Chloroform ist flüchtig und schwer, pipette, die Eppendorf-Röhrchen so bald wie möglich zu schließen. - Schütteln Sie die Eppendorf-Röhrchen energisch für 15 s oder bis die Proben voll emulgiert werden.
- Halten Sie die Probe bei RT, für 1 – 2 min. oder bis der flüssigen Phasen anfangen zu trennen.
- Zentrifugieren Sie die Proben bei 12.000 x g für 15 min bei 4 ° C.
- Transfer der oberen wässrigen Phase (~ 320 µL), RNA, eine neue Eppendorf Tube enthält, und verwerfen die restliche Flüssigkeit.
Hinweis: Berühren Sie nicht die untere rosa organische Phase oder der weisse Ring zwischen den zwei Phasen mit der Spitze der Pipette. - Die wässrige Phase 1,5 Volumen von 100 % Ethanol (480 µL) hinzu, und pipette die Lösung langsam nach oben und unten 3 Mal zu mischen.
- Reinigen Sie RNA mit einem kommerziell erhältlichen Kit für Reinigung von RNA aus kleinen Proben entwickelt.
- RNA-Konzentration und Probe-Reinheit mit einem Spektrophotometer zu quantifizieren.
Hinweis: (i) erwarten Sie Renditen ≥3.5 µg/gut; 260/280 und 260/230 Verhältnisse für Reinheit über Proteine und organische Verbindungen sollten beide sein ≥1.9. (Ii) die RNA-Proben werden bei-80 ° C gelagert oder sofort bearbeitet. - Retrotranscribe 250, 500 oder 1000 ng RNA mit einem kommerziell erhältlichen Kit.
- Quantifizieren Sie die Knockdown Effizienz des endogenen Gens von Interesse durch qRT-PCR. Ein detailliertes Protokoll finden Sie unter Referenz10. Streben Sie nach einem Knockdown Wirkungsgrad von ≥60 % (Abbildung 2). Normalisieren Sie die Daten ≥2 Housekeeping Gene (zB. ACTB, TUBB3, GAPDH, PPIA) verwenden mehrere interne Kontrolle gen Methode11.
Hinweis: Wenn in der Ratte arbeiten, verwenden die folgenden PCR-Primer für die Housekeeping-Gene: GAPDH-fwd: 5' GGTGCTGAGTATGTCGTGGA 3' und GAPDH-Rev: 5' GATGATGACCCTTTTGGC 3'; ACTB-fwd: 5' CATCACTATCGGCAATGAGC 3' und ACTB-Rev: 5' TCATGGATGCCACAGGATT 3'; TUBB3-fwd: 5' GCCTTTGGACACCTATTCAG 3' und TUBB3-Rev: 5' TCACATTCTTTCCTCACGAC 3'; PPIA-fwd: 5' CACTGGGGAGAAAGGATTTG 3' und PPIA-Rev 5' CCATTATGGCGTGTGAAGTC 3'.
4. Bewertung der Rolle der präsynaptischen Proteine in intakte neuronale Schaltkreise durch gezielte Stimulation der geworfen Neuronen mit Optogenetik
Hinweis: Das folgende Protokoll erfordert Vorkenntnisse mit elektrophysiologische Aufnahmen in akuten Hirnschnitten und Zugriff auf eine elektrophysiologische Setup.
- Injizieren Sie rAAV1/2 in das Gehirn nach ein detailliertes Protokoll in früheren Publikation12hierbei. Bestimmen Sie die stereotaktischen Koordinaten für die Region des Gehirns mit stereotaktischen Atlanten für Maus13 oder Ratte Gehirn14.
- Verwenden einer Mikropipette Puller Injektion Mikropipetten mit langen Schäften ø 7 – 9 µm ziehen; die Schäfte der Injektion Mikropipetten mit einer Schere aufstecken. Verwenden Sie eine dünne Marker und Millimeterpapier Kalibrierer Markierungen auf die Injektion Mikropipetten alle 2 mm zu platzieren.
- Das Tier mit Isofluran zu betäuben und in die stereotaktische Apparatur befestigen.
- Halten Sie das Tier warm während der Operation mit einem Heizkissen gesetzt bei 37 ° C.
- Schützen Sie die Augen mit okulärer Schmiermittel.
- Rasieren Sie das Fell auf dem Kopf mit einen elektrischen Rasierapparat.
- Povidon-Jod auf den rasierten Kopf mit einem Wattestäbchen zu verbreiten.
- Machen Sie unter dem sezierenden Mikroskop einen Mittellinie Einschnitt.
- Reinigen Sie die Oberfläche der Schädel mit einem Wattestäbchen, um die Bregma und Lambda sichtbar zu machen.
- Ort der Injektion Mikropipette in die Halterung. Die stereotaktische Arm befestigen Sie Halterung.
- Bestimmen Sie die x und y Koordinaten von der Injektionsstelle im Verhältnis zu den Bregma und/oder der Lambda-Ausdruck.
- Verwenden Sie einen Bohrer auf um den Schädel über dem Zielgebiet zu dünn.
Hinweis: Verwenden Sie sanftere kreisenden Bewegungen und zu vermeiden Sie, durch den Schädel Bohren, da dadurch die Oberfläche des Gehirns beschädigt wird. - Wenn es blutet, absorbieren Sie übermäßigen Blut mit Handtuch Papier.
Hinweis: Übermäßige Blut wird korrekt die Z-Koordinate bestimmen erschweren. - Laden Sie ≤2 µL des Virus in die Injektion Mikropipette durch Kapillarwirkung.
- Bringen die Injektion Mikropipette in x- und y-Koordinaten der Injektionsstelle.
- Berechnen Sie die Z-Koordinate aus der Dura und senken Sie die Pipette langsam ins Gehirn.
- Wenn die Z-Koordinate erreicht ist, warten Sie 3 Minuten, um Gewebe Anpassung zu ermöglichen.
- Gelten Sie positive Niederdruck mit einer 1 mL Spritze über einen flexiblen Schlauch an das Ende der Injektion Mikropipette verbunden. Visuell überwacht die Geschwindigkeit des Auswurf des Virus von der Injektion Mikropipette durch die Dissektion Mikroskop und mit Hilfe der Kalibrierung als Referenzpunkte markiert.
Hinweis: Virus muss langsam injiziert werden (< 100 nL/min), Gewebeschäden zu vermeiden. - Wenn Injektion abgeschlossen ist, warten Sie 5 min, zurückziehen Sie der Injektion Mikropipette um 0,2 mm, warten Sie noch 5 min, Rückfluss des Virus zu vermeiden.
- Zurückziehen der Injektion Mikropipette langsam und vollständig aus dem Gehirn und entsorgen Sie es in einen Behälter mit Bleichmittel gefüllt.
- Benetzen Sie den Schädel mit physiologischer Lösung und Naht der Haut mit 3 bis 4 Maschen.
- Gentamicin-Salbe auf die Wunde auftragen
- Single-Haus das Tier in einem sauberen Käfig mit Essen pellets und unter einer Wärmelampe bis es vollständig erholt.
- ≥15 Tage nach der Injektion, Tiere in tiefen Isoflurane Narkose zu enthaupten.
- Bereiten Sie akuten Hirnschnitten von der Region des Gehirns von Interesse mit einem Vibratome vergast (95 % O2, 5 % CO2) eiskalte ACFS Lösung, enthält (in mM): 123 NaCl, 1,25 KCl, 1,25 KH2PO4, 1,5 MgCl2, 1 CaCl2, 25 Nahco3-3, 2 NaPyruvate und 18 Glukose (auf 300 mOsm eingestellt Osmolarität). Zum Beispiel wenn synaptische Übertragung von CA3 CA1 pyramidale Neuronen analysieren soll, bereiten Sie sagittale Scheiben der hippocampalen Bildung (350 µm dick).
Hinweis: Von diesem Schritt ab arbeiten Sie unter schlechten Lichtverhältnissen, Aktivierung der optogenetische Sonde durch Umgebungslicht zu vermeiden. - Lassen Sie die Scheiben für 30 min bei 37 ° C in der gleichen ACFS in einer Kammer für die Abhaltung Gehirnscheiben entworfen zu erholen. Halten Sie die Scheiben in der gleichen Gehirn Slice Kammer bei Raumtemperatur bis Aufnahme.
Hinweis: Wenn Sie diese Bedingungen, können Scheiben für bis zu 6 – 8 h gesund gepflegt werden. - Übertragung einer Scheibe zu einem versunkenen Aufnahme Kammer und Superfuse es mit 2 mL/min von der gleichen ACFS verwendet für die Wiederherstellung mit 1,5 mM CaCl2 ergänzt (Gesamt Ca2 +: 2,5 mM) und ohne NaPyruvate.
Hinweis: Scheiben sind vorbereitet und gepflegt in geringer Konzentration von Ca2 + (1 mM), Toxizität zu minimieren. Aufnahmen erfolgen in 2,5 mM Ca2 + , Vesikel Version zu bevorzugen. - Überprüfen Sie kurz das Signal von der zum Ausdruck gebrachten fluoreszierende Reporter (zB. TdTomato; Abb. 3 b) die Lokalisation und Intensität der Infektion festzustellen.
- Füllen Sie eine Patch-Elektrode mit einem intrazellulären Lösung enthält (in mM): 110 K-Gluconat, 22 5 NaCl, KCl, 0,5 EGTA, 3 MgCl2, 4 Mg-ATP, 0,5 Na3-GTP, 20 K2-Kreatin-Phosphat, 10 HEPES-KOH (pH 7.28, 290 mΩ).
Hinweis: Verwenden Sie Patch-Elektrode mit einer Pipette Widerstand von 5 – 6 mΩ. - Erreichen Sie unter Infrarot-Beleuchtung dicht ganz-Zell Konfiguration von einem Neuron synaptische Eingänge von den infizierten Nervenzellen empfangen. Beispielsweise wenn CA3 pyramidale Neuronen infiziert wurden, patch-pyramidale Neuronen in den proximalen mit medialen Trakt der CA1-Region (Abbildung 3).
Hinweis: Vorwiderstand kann gelassen werden nicht entschädigt sollte, es jedoch konstant und niedrig (≤20 MΩ). - Verwenden Sie Pharmakologie, um die synaptische Ströme unter Untersuchung zu isolieren. Zum Beispiel wenn exzitatorische synaptische Übertragung untersuchen soll, blockieren Sie inhibitorischen synaptischen Übertragung mit 10 µM Bicuculline.
- Evozieren Sie synaptische Ströme, z. B. exzitatorischen postsynaptischen Ströme (EPSCs), mit einem 473 nm blau Laser gekoppelt mit einer optischen Faser (Ø ≤250 µm) positioniert auf die Somata der infizierten Nervenzellen (zB. CA3 pyramidale Neuronen).
- Stellen Sie Anregung Länge auf ein Minimum zu reduzieren Sie die Möglichkeit, mehr als ein Aktionspotential pro Lichtimpuls hervorzurufen. Wenn ChETA verwenden, setzen Sie ihn auf 2 ms15.
- Passen Sie Anregung Stärke des Lasers zu klein, aber eindeutig nachweisbar synaptische Ströme führen (≤50 pA Peak Amplitude für EPSCs aufgenommen in eine Holding-Potenzial von-70 mV zwischen CA3 und CA1 pyramidale Neuronen; Abbildung 3D). Wenn ChETA und einer optischen Faser von 250 µm im Durchmesser zu verwenden, sollte Anregung, die Stärken von 1 – 3 mW an Faser beenden geeignet sein.
Hinweis: Wenn Laser Stärke geregelt werden kann, verwenden Sie Neutralfilter. - Leuchten Sie das 473 nm Laserlicht, auf die Somata der infizierten Nervenzellen, aber nicht auf ihre Axone. Zum Beispiel Leuchten Sie das Licht auf CA3 Somata und Weg von der Schaffer Sicherheiten, um direkten Depolarisation der Axone zu vermeiden.
- Tetrodotoxin (TTX; 0,5 µM) anwenden, ein Blocker von Natriumkanälen, zum Beispiel um die Kanäle zu bestätigen sind Aktionspotential getrieben.
- Gelten Sie in verschiedenen Aufnahmen einen selektiveren Blocker des synaptischen Stroms unter Untersuchung zu bestätigen, dass die Stimulation selektiv ist.
Hinweis: Z. B. NBQX anwenden (10 µM), AMPA-Typ Glutamat-Rezeptor-vermittelten EPSCs. - Optisch und elektrisch evozierten synaptische Ströme in der gleichen akuten Gehirn-Scheibe in Reaktion auf die repetitive Stimulation (≥2 Reize bei ≤20 Hz) zu vergleichen. Ein ähnliches Maß an synaptischen Erleichterung oder Depression sollten beachtet werden, zwischen elektrischen und optischen Stimulation bei der Verwendung einer Kontrolle MiR, hindeutet, dass ähnliche zelluläre Mechanismen durch die beiden Arten von Stimulationen aktiviert werden.
Hinweis: Die oben genannten Schritte sind notwendig, um sicherzustellen, dass optische Stimulation nicht dazu, eine direkte Depolarisation der präsynaptischen Boutons veranlassen, also unter Umgehung einige Mechanismen der synaptischen Übertragung.
- Vergleichen Sie synaptischen Übertragung in Reaktion auf Einzel- und sich wiederholende Stimulationen (≥2 Reize bei ≤20 Hz) zwischen einer Kontrolle MiR und MiRs Ausrichtung der präsynaptischen Proteine untersucht.
Ergebnisse
Die oben beschriebenen Verfahren bieten eine robuste Methode zu beurteilen, wie synaptische Übertragung von Knockdown von synaptischen Proteine in den präsynaptischen Neuronen betroffen ist. Repräsentative Ergebnisse auf wie die Knockdown von Alternative splice Isoformen von präsynaptischen CaV2.1 (P/Q-Typ) VGCCs regelt kurzfristige synaptische Plastizität bei CA3, CA1 exzitatorische Synapsen unten als Beispiel angegeben sind.
CaV2.1 (P/Q-Typ) Kanäle sind die vorherrschenden präsynaptischen VGCCs an die meisten schnellen Synapsen im zentralen Nervensystem. Alternatives Spleißen von sich gegenseitig ausschließenden Exons 37a und 37 b der Pore-forming α-1 -Untereinheit von CaV2.1 (α1A) produziert zwei große Varianten, CaV2.1 [EFa] und CaV2.1 [EFb]16,17 ,18. Um festzustellen, ob CaV2.1 [EFa] und CaV2.1 [EFb] differentiell synaptische Übertragung und Plastizität im Hippokampus pyramidale Rattenneuronen regulieren, wir zuerst entwickelt Isoform-spezifische MiRs, Knockdown selektiv CaV 2.1 [EFa] oder CaV2.1 [EFb]5. Trotz der kurzen Größe (97 bp) und hohe Ähnlichkeit (61.86 % Identität auf der Nukleotid-Ebene) zwischen Exons 37a und 37 b, wir könnten design drei MiR Sequenzen gegen Ratte CaV2.1 [EFa] (MiR EFa1: TCCTTATAGTGAATGCGGCCG; MiR EFa2: ATGTCCTTATAGTGAATGCGG; MiR EFa3: TTGCAAGCAACCCTATGAGGA) und zwei gegen Ratte CaV2.1 [EFb] (MiR EFb1: ATACATGTCCGGGTAAGGCAT; MiR EFb2: ATCTGATACATGTCCGGGTAA) mit einem prognostizierten hohen Knockdown Wirkungsgrad. Als Negativkontrolle (MiR Kontrolle) haben wir das pcDNA6.2-GW/EmGFP-MiR-Neg-Plasmid enthält eine Sequenz, die keines bekannte Wirbeltier gen gerichtet ist. Basierend auf einer ersten Bildschirm in HEK-293-Zellen gegen heterologe Kanäle, wir MiR EFa1, MiR EFa3 und MiR EFb2 ausgewählt und in der 3' UTR des Vektors pAAV-Syn-ChETA-TdT-miR-X, die für die Produktion von rAAVs soll ihre Expressionskassetten kloniert und wo die Wirbeltiere Promotor treibt Ausdruck von ultraschnellen Channelrhodopsin ChETA, das rot fluoreszierende Protein TdTomato und der eingefügten MiR (Abbildung 1). Der Expressionskassette der MiR EFb2, Knockdown Effizienz der dieser MiR dupliziert
Als nächstes wir bereit rAAV1/2 für die oben genannten vier Konstrukte und quantifiziert Knockdown Effizienz und Selektivität in primären Ratte neuronalen Kulturen mittels Isoform-spezifische qRT-PCR. MiR EFa1 und MiR reduziert EFa3 mRNA von native CaV2.1 [EFa] ~ 70 % aber nicht, dass der CaV2.1 [EFb], während MiR EFb mRNA von native CaV2.1 [EFb] von ~ 60 % aber nicht, dass der CaV2.1 [EFa] (Abbildung 2) reduziert.
Wir dann hierbei injiziert jeder der vier rAAV1/2 Bereich CA3 des Hippocampus P18 Ratten (Abb. 3A-C), mit den Koordinaten (A-P/M-L/D-V von Bregma) −2.6 / ± 2,9 / −2.9. Fünfzehn bis zwanzig-vier Tage nach der Injektion, wir akute hippocampale Scheiben aus rAAV1/2-injiziert Ratten vorbereitet und TdTomato Fluoreszenz verwendet, um den Ausdruck und die Lokalisierung von rAAVs (Abb. 3 b) bestätigen. Um zu untersuchen, ob präsynaptischen Knockdown von CaV2.1 [EFa] oder CaV2.1 [EFb] kurzfristige synaptische Plastizität bei CA3, CA1 Synapsen betroffen, stimulieren wir gezielt infizierte CA3 Neuronen mit kurzen 473 nm Laser Lichtpulse (2 ms langen ; Abbildung 3), und die sich daraus ergebenden EPSCs von Patchen pyramidale Neuronen in den proximalen mit medialen Trakt der CA1-Region (Abbildung 3) aufgenommen. Wir fanden, dass der Zuschlag von CaV2.1 Spleiß Isoformen Antworten gepaart-Puls-Stimulation in entgegengesetzte Richtungen betroffen: Knockdown von CaV2.1 [EFa] (MiR EFa1 oder MiR EFa3) verstärkt gepaart-Pulse Facilitation (PPF), während Knockdown von Ca V2.1 [EFb] (MiR EFb2) abgeschafft (Abbildung 3D, E).
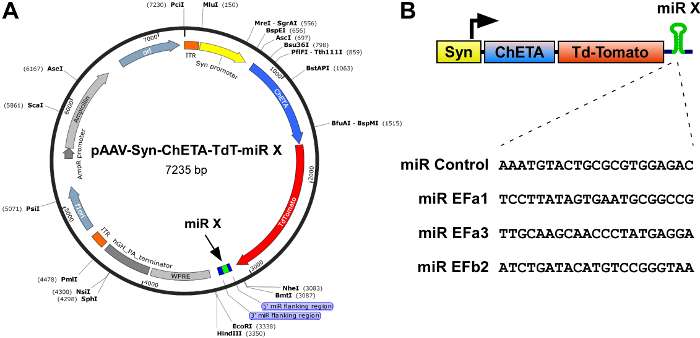
Abbildung 1: Schema der Konstrukte für kombinierte Ausdruck von ultraschnellen optogenetische Sonde ChETA und Isoform-spezifische MiRs gegen CaV2.1 [EFa] und CaV2.1 [EFb]. (A) Karte von den rAAV konstruieren pAAV-Syn-ChETA-TdT-miR-X, mit synapsin Förderer (Syn), die ultraschnelle Channelrhodopsin ChETA verschmolzen, TdTomato und in den proximalen 3' UTR, eine CaV2.1 Spleiß Isoform-spezifische MiR. Restriktionsenzyme gezeigt werden einzelne Messer. (B) oben, Schema der Expressionskassette. Die synapsin Promotor treibt Ausdruck ChETA-TdTomato und eine Isoform-spezifische MiR. Unten, umgekehrte Ergänzung des 21-Nukleotid Zielsequenzen, die Teil der MiR Kassette. Für MiR EFb2 wurden zwei identische MiR Kassetten im Tandem, einer nach dem anderen ausgedrückt. Bitte klicken Sie hier für eine größere Version dieser Figur.
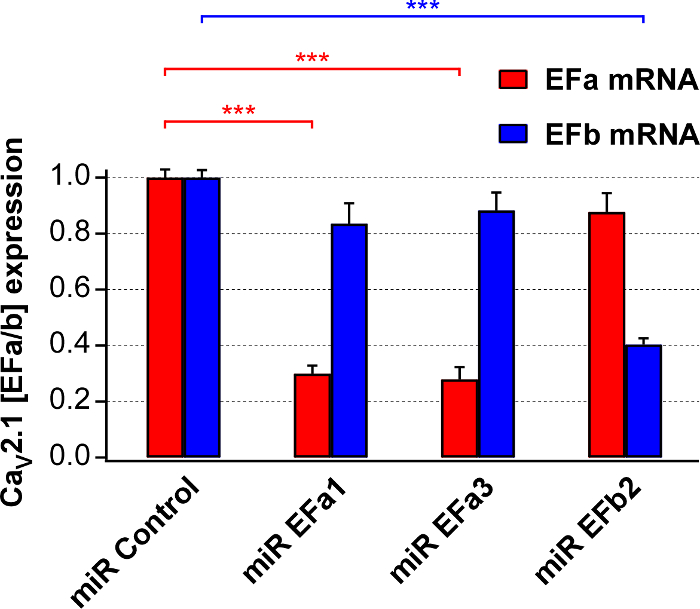
Abbildung 2. Bewertung der Knockdown Effizienz und Selektivität der Isoform-spezifische MiRs für CaV2.1 [EFa] und CaV2.1 [EFb]. Isoform-spezifische qRT-PCR-Analyse auf RNA isoliert vom 17. / 18. DIV Primärkulturen bei 6 DIV mit rAAVs mit dem Ausdruck MiRs Ausrichtung entweder CaV2.1 [EFa] (MiR EFa1 und MiR EFa3) infiziert oder CaV2.1 [EFb] (MiR EFb2). Daten sind auf die negativ-Kontrolle (MiR Kontrolle) normalisiert. MiR EFa1 und MiR EFa3 deutlich und selektiv reduzieren mRNA von CaV2.1 [EFa] (n = 8 und 7 Kulturen bzw.), während MiR EFb deutlich und selektiv mRNA von CaV2.1 [EFb] reduziert (n = 4 Kulturen; *** p < 0,001; One-Way-Analyse Varianz Test gefolgt von der Tukey-Kramer Post-Test). Daten werden als ± SEM bedeuten präsentiert Diese Zahl wurde angepasst von Thalhammer, A. Et al. 5. Bitte klicken Sie hier für eine größere Version dieser Figur.

Abbildung 3. Bewertung der Rolle von CaV2.1 [EFa] und CaV2.1 [EFb] im nativen Hippocampus durch gezielte Stimulation der geworfen Neuronen mit Optogenetik. (A) Regelung der Expressionskassette der rAAV Konstrukte zur in-Vivo -Infektion. (B) Hippocampal Abschnitt zeigen, dass TdTomato Fluoreszenz beschränkt sich der CA3-Region und seine Prognosen. (C) experimentelle Konfiguration: Laserstrahl richtete sich auf CA3 Somata und Patch-Clamp-Aufnahmen wurden von CA1 pyramidale Neuronen durchgeführt. (D) 2 ms langen blau (473 nm) Licht Laserpulse glänzte bei 20 Hz evozieren EPSCs deren PPF ist MiRs targeting CaV2.1 [EFa] gestiegen und von MiR für CaV2.1 [EFb] abgeschafft. (E) Zusammenfassung der gepaart-Puls-Verhältnis für Experimente wie in (D), mit einem Anstieg in PPF MiR EFa1 und MiR EFa3 und einen Rückgang bei MiR EFb2, im Verhältnis zu MiR Kontrolle (n = 9 / 11 Aufnahmen; * p = 0,02; ** p = 0,01; *** p < 0,0004; Analyse der Kovarianz). Daten werden als ± SEM bedeuten präsentiert Diese Zahl wurde angepasst von Thalhammer, A. Et al. 5. Bitte klicken Sie hier für eine größere Version dieser Figur.
Diskussion
Co Ausdruck einer optogenetische-Sonde und eine MiR gegen ein präsynaptischen gen des Interesses bietet einen leistungsfähigen Ansatz um die Auswirkungen von gen-Knockdown auf präsynaptischen Funktion innerhalb intakte neuronale Schaltkreise zu charakterisieren. Für diesen experimentellen Ansatz ist es wichtig, zu identifizieren und zu charakterisieren, MiRs, die höchst effizient und selektiv in das gen des Interesses an nativen Systemen umzuwerfen. Wenn möglich, sollten zwei oder mehr unabhängige MiRs gegen die gleichen mRNA von Interesse zu steuern für eventuelle Ziel Effekte verwendet werden. Rettung-Experimente, in denen ein MiR beständig gen in das System wieder eingeführt wird als ein Steuerelement Spezifität einsetzbar.
Das gleiche Konstrukt ermöglicht ein optogenetische ausdrücken Sonde und eine MiR anregende optisch nur die präsynaptischen Neuronen, die manipuliert worden sind. Dies ist nicht möglich mit elektrischen Stimulationen, weil sie nicht zwischen infizierten und nicht infizierten Nervenzellen, wodurch gemischt und verdünnt Ergebnisse unterscheiden. Da optische Reize mehrere Aktionspotentiale15induzieren können, ist es wichtig, nur die schnellste optogenetische Sonden unter einer wachsenden Palette15,19,20,21zu wählen. Darüber hinaus ist es wichtig sicherzustellen, dass die optische Stimulation nicht dazu veranlassen, eine direkte Depolarisation der präsynaptischen Boutons, um zu vermeiden, einige der Schritte der synaptischen Übertragung unter Umgehung einer22untersuchen möchte.
Der experimentelle Ansatz, den wir hier beschreiben macht es möglich, parallel die physiologische Relevanz mehrere präsynaptischen Gene des Interesses innerhalb eines begrenzten Zeitraums (4 – 6 Monate) zu bewerten. Es ist jedoch wichtig zu beachten, dass Niederschlägen selten 100 % erreichen. Darüber hinaus müssen rAAVs ausgedrückt werden, für mindestens zwei Wochen zu ermöglichen maximale Knockdown und vollen Ausdruck der optogenetische Sonde, die eine zeitliche Begrenzung darstellen können, wenn Sie frühe Entwicklungsprozesse zu untersuchen. Obwohl mehr zeitaufwendig, bedingte Knockout-Mäusen beschränkt auf präsynaptischen Neuronen führen in der Regel vollständige Entfernung des Gens von Interesse und bieten somit einen gültigen komplementären Ansatz.
Ein besonderen Vorteil der MiR-Technologie ist, dass dadurch die Expression von mehreren MiRs vom gleichen Veranstalter. Diese Eigenschaft wurde hauptsächlich zur Knockdown Effizienz zu steigern, indem Sie mehrere Kopien der gleichen MiR oder verschiedene MiRs gegen das gleiche Zielgen einfügen. Jedoch kann es auch verwendet, um Zuschlag mehrere Gene MiRs gegen unterschiedliche Zielgruppen Gene7,23zum Ausdruck zu bringen. Diese Eigenschaft kann verwendet werden, um mehrere präsynaptischen Proteine um präsynaptischen Signalwege sezieren zum Schweigen zu bringen.
Hier haben wir Optogenetik mit künstlichen MiRs zu charakterisieren die Auswirkungen von gen-Knockdown präsynaptischen Funktion im akuten hippocampal Scheiben kombiniert. Ein ähnlicher Ansatz könnte auch verwendet werden, zu manipulieren und Sonde neuronale Schaltkreise in Vivo. Darüber hinaus würde künstliche MiRs mit Chemogenetic kombinieren Ansätze einer neuronalen Schaltkreise auf längeren Zeitskalen Verhören ermöglichen.
Offenlegungen
Die Autoren haben nichts preisgeben.
Danksagungen
Wir danken F. Benfenati (Istituto Italiano di Tecnologia, IIT) für Unterstützung und Carmela Vitale für die Hilfe bei der Demonstration. Diese Arbeit wurde von IIT und die Compagnia San Paolo (Grant Nr. 9734 Lac) finanziert.
Materialien
| Name | Company | Catalog Number | Comments |
| Acrylamide | Sigma | Acrylamide/Bis-acrylamide, 30% solution | Toxic |
| Animal Temperature Controller with heat pad | WPI | ATC2000 | |
| BCA protein assay kit | ThermoFisher Scientific | 23225 | |
| BLOCK-iT Pol II miR RNAi Expression Vector kit | ThermoFisher Scientific | K4936-00 | |
| Brain slices Prechamber | Harvard Apparatus | BSC-PC | |
| CCD camera-based imager | Bio-Rad | ChemiDoc™ MP | |
| Cell Culture reagents | Life Technologies | ||
| Chemiluminescent substrate | GE Helthcare | ECL Western Blotting Reagents, RPN2106 | |
| Chloroform | Sigma | C2432 | Toxic |
| Cytosine β-D-arabinofuranoside | Sigma | C6645 | |
| Drill | Foredom | K.1030 | |
| EGTA | Sigma | E4378 | |
| Gentamicin ointment | Local pharmacy | ||
| Glucose | Sigma | G7021 | |
| Hepes | Sigma | 54459 | |
| Injection micropipettes | Narishige | GD1 | |
| Inorganic salts & detergents | Sigma | ||
| K2-creatine phosphate | Calbiochem | 237911 | |
| KGluconate | Fluka | 60245 | |
| Laser | Laserglow Technologies | LRS-0473-GFM-00100-03 | |
| Membrane | Amersham | Protran™ 0.2 µm NC | |
| MgATP | Sigma | A9187 | |
| Micropipette holder | Narishige | IM-H1 | |
| Micropipette puller | Narishige | PC-100 | |
| Na3GTP | Sigma | G8877 | |
| NaPyruvate | Sigma | P5280 | |
| Ocular lubricant | Local pharmacy | Lacrigel | |
| Phosphate inhibitors | Sigma | P0044, P5726 | |
| Protease inhibitors | Sigma | cOmplete™, EDTA-free Protease Inhibitor Cocktail | |
| Protein gel electrophoresis and blotting devices | Bio-Rad | Mini-Protean III Cell | |
| Providone iodine | Local pharmacy | Betadine | |
| Qiazol Lysis Reagent | Qiagen | 79306 | Toxic |
| QuantiTect Reverse Transcription Kit | Qiagen | 205311 | |
| RNeasy Micro Kit | Qiagen | 74004 | |
| Spectrophotometer | ThermoFisher Scientific | Nanodrop 2000 | |
| Stereotactic apparatus | WPI | ||
| Tetrodotoxin | Tocris | 1069 | Toxic |
| Vibratome | Leica | VT1200S |
Referenzen
- Lam, J. K., Chow, M. Y., Zhang, Y., Leung, S. W. siRNA Versus miRNA as Therapeutics for Gene Silencing. Mol Ther-Nucl Acids. 4, e252(2015).
- Kohl, M. M., et al. Hemisphere-specific optogenetic stimulation reveals left-right asymmetry of hippocampal plasticity. Nat Neurosci. 14 (11), 1413-1415 (2011).
- Maejima, T., et al. Postnatal loss of P/Q-type channels confined to rhombic-lip-derived neurons alters synaptic transmission at the parallel fiber to purkinje cell synapse and replicates genomic Cacna1a mutation phenotype of ataxia and seizures in mice. J Neurosci. 33 (12), 5162-5174 (2013).
- Mark, M. D., et al. Delayed postnatal loss of P/Q-type calcium channels recapitulates the absence epilepsy, dyskinesia, and ataxia phenotypes of genomic Cacna1a mutations. J Neurosci. 31 (11), 4311-4326 (2011).
- Thalhammer, A., et al. Alternative Splicing of P/Q-Type Ca2+ Channels Shapes Presynaptic Plasticity. Cell Rep. 20 (2), 333-343 (2017).
- Sambrook, J., Russell, D. W. Calcium-phosphate-mediated Transfection of Eukaryotic Cells with Plasmid DNAs. CSH Protoc. 2006 (1), (2006).
- Chung, K. H., et al. Polycistronic RNA polymerase II expression vectors for RNA interference based on BIC/miR-155. Nucleic Acids Res. 34 (7), e53(2006).
- McClure, C., Cole, K. L., Wulff, P., Klugmann, M., Murray, A. J. Production and titering of recombinant adeno-associated viral vectors. J Vis Exp. (57), e3348(2011).
- Cingolani, L. A., et al. Activity-dependent regulation of synaptic AMPA receptor composition and abundance by beta3 integrins. Neuron. 58 (5), 749-762 (2008).
- Huang, H., Xu, Y., Cheng, C. Detection of alternative splicing during epithelial-mesenchymal transition. J Vis Exp. (92), e51845(2014).
- Vandesompele, J., et al. Accurate normalization of real-time quantitative RT-PCR data by geometric averaging of multiple internal control genes. Genome Biol. 3 (7), (2002).
- Cetin, A., Komai, S., Eliava, M., Seeburg, P. H., Osten, P. Stereotaxic gene delivery in the rodent brain. Nat Protoc. 1 (6), 3166-3173 (2006).
- Paxinos, G., Franklin, K. B. The Mouse Brain in Stereotaxic Coordinates. , Academic Press. (2012).
- Paxinos, G., Watson, C. The Rat Brain in Stereotaxic Coordinates. , Academic Press. (2013).
- Gunaydin, L. A., et al. Ultrafast optogenetic control. Nat Neurosci. 13 (3), 387-392 (2010).
- Bourinet, E., et al. Splicing of alpha 1A subunit gene generates phenotypic variants of P- and Q-type calcium channels. Nat Neurosci. 2 (5), 407-415 (1999).
- Chaudhuri, D., et al. Alternative splicing as a molecular switch for Ca2+/calmodulin-dependent facilitation of P/Q-type Ca2+ channels. J Neurosci. 24 (28), 6334-6342 (2004).
- Soong, T. W., et al. Systematic identification of splice variants in human P/Q-type channel alpha1(2.1) subunits: implications for current density and Ca2+-dependent inactivation. J Neurosci. 22 (23), 10142-10152 (2002).
- Berndt, A., et al. High-efficiency channelrhodopsins for fast neuronal stimulation at low light levels. P Natl Acad Sci USA. 108 (18), 7595-7600 (2011).
- Klapoetke, N. C., et al. Independent optical excitation of distinct neural populations. Nat Methods. 11 (3), 338-346 (2014).
- Lin, J. Y., Lin, M. Z., Steinbach, P., Tsien, R. Y. Characterization of engineered channelrhodopsin variants with improved properties and kinetics. Biophys J. 96 (5), 1803-1814 (2009).
- Zhang, Y. P., Oertner, T. G. Optical induction of synaptic plasticity using a light-sensitive channel. Nat Methods. 4 (2), 139-141 (2007).
- Fowler, D. K., Williams, C., Gerritsen, A. T., Washbourne, P. Improved knockdown from artificial microRNAs in an enhanced miR-155 backbone: a designer's guide to potent multi-target RNAi. Nucleic Acids Res. 44 (5), e48(2016).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten