Method Article
Combinando Optogenetics con microRNAs artificiales para caracterizar los efectos de Gene caída en la función presináptica en circuitos neuronales intactas
En este artículo
Resumen
Este protocolo proporciona un flujo de trabajo sobre cómo combinar artificial microRNA mediada por ARN de interferencia con optogenetics estimular específicamente botones presinápticos con menor expresión de genes selectivos dentro de circuitos neuronales intactos.
Resumen
El propósito de este protocolo es caracterizar el efecto de la precipitación del gene en la función presináptica en circuitos neuronales intactas. Se describe un flujo de trabajo sobre cómo combinar artificial microARN (miR)-mediada por ARN de interferencia con optogenetics para lograr la estimulación selectiva de manipuladas botones presinápticos en rebanadas de cerebro agudo. El enfoque experimental involucra el uso de una sola construcción viral y solo enolase promotor para conducir la expresión de una sonda de optogenetic y miR(s) artificial contra genes presinápticos. Cuando stereotactically inyectado en la región cerebral de interés, el concepto expresado hace posible estimular con luz exclusivamente las neuronas con la reducida expresión de los genes investigados. Esta estrategia no requiere el desarrollo y mantenimiento de líneas de ratón modificados genéticamente y puede en principio aplicarse a otros organismos y a cualquier gen neuronal de elección. Recientemente hemos aplicado para investigar cómo la caída de isoformas de empalme alternativos de canales de calcio voltaje-bloqueado de P/Q-type presinápticos (VGCCs) regula la plasticidad sináptica a corto plazo en CA3 a CA1 sinapsis excitatorias en rebanadas hippocampal agudas. Un enfoque similar podría utilizarse también para manipular y probe el circuito neuronal in vivo.
Introducción
Este protocolo describe un nuevo enfoque para caracterizar el efecto de la precipitación del gene en la función presináptica en circuitos neuronales intactas. Investigar la función presináptica en circuitos neuronales intactas es difícil porque muchos botones presinápticos son demasiado pequeños y lejos del soma para permitir intervenciones moleculares y electrofisiológicas combinadas. A pesar de interferencia de ARN ofrece un medio potente y flexible a la precipitación de las proteínas sinápticas1, este enfoque se ha utilizado con moderación para investigar la función presináptica porque es difícil detectar los efectos de la precipitación utilizando estímulos eléctricos, que no distinguen entre manipularon e ingenuo botones presinápticos2. Aquí, describimos cómo combinar artificial microARN (miR)-mediada por ARN de interferencia con tecnología de optogenetic recientemente desarrollados para lograr la estimulación selectiva de manipuladas botones presinápticos en rebanadas de cerebro agudo.
Mientras que los ratones condicionales en combinación con electrofisiología podrían utilizarse también para investigar la función de proteínas presinápticas3,4, nuestra estrategia no requiere el desarrollo y mantenimiento de genéticamente modificar líneas de ratón y también se puede emplear fácilmente derribar a isoformas específicas de un gen. Relativa más comúnmente usado horquilla corta RNAs (shRNAs), miRs artificiales, que empleamos aquí, ofrecen ventajas fundamentales para la caída en las neuronas. A diferencia de shRNAs, puede ser expresados bajo el control de una polimerasa II promotor1. Así, un promotor solo puede utilizarse para impulsar la expresión de miR y una sonda de optogenetic, junto con un reportero fluorescente. De esta forma, el tamaño de la construcción se puede mantener dentro de los límites de empaquetado de virus adeno-asociado recombinantes (rAAV, figura 1A). Además, el uso de una construcción única y un único promotor reduce variabilidad experimental porque permite la expresión de la miR, la sonda de optogenetic y el reportero fluorescente en un cociente fijo.
Recientemente hemos aplicado esta tecnología para examinar el papel de los isoforms alternativomente empalmada de canales de calcio presinápticos en el hipocampo5. Esta estrategia es generalmente aplicable al estudio de la relevancia fisiológica de otras proteínas presynaptically expresadas en los circuitos cerebrales de interés.
Protocolo
Todos los experimentos se llevaron a cabo conforme a los lineamientos establecidos por el Consejo de las comunidades europeas (Directiva 2010/63/UE del 04 de marzo de 2014) y fueron aprobados por el Ministerio italiano de salud.
1. diseño de microRNAs para ARN de interferencia y la evaluación de la eficiencia en sistemas de expresión heteróloga
Nota: Este protocolo requiere un conocimiento de los siguientes métodos bien establecidos: clonación molecular, secuenciación de ADN, mantenimiento de líneas celulares, transfección del fosfato de calcio, cuantitativa real tiempo de PCR (qRT-PCR), preparación de Lisados celulares de líneas celulares y Western blotting.
- Determinar si el gen de interés es objeto de splicing alternativo. Para una caída general del gene, seleccionar exclusivamente constitutivos exones; para una caída de una isoforma específica alternativomente empalmada, seleccione el exón alternativamente empalmado pertinente.
- Utilizar software dedicado (por ejemplo, https://rnaidesigner.thermofisher.com/rnaiexpress/) para diseñar miRs artificiales para ARN de interferencia contra la secuencia de interés.
Nota: Es importante utilizar una herramienta básica de búsqueda alineación Local (BLAST) para comprobar posibles de objetivos dentro de la misma especie. - Seleccione las superior tres miRs ordenadas para el gene de la blanco.
- Ordenar los hilos superior e inferior de las miRs seleccionados de una empresa idónea. Para el control negativo, utilice una versión codificada de una las miRs seleccionados o un miR predijo no tratar ningún gen conocido de las especies utilizadas en el experimento (por ej. para los genes de vertebrados, el miR en pcDNA6.2-GW/EmGFP-miR-neg).
- Templar los respectivos hilos superior e inferior de miRs seleccionados y clonar en un plásmido diseñado para la expresión del miRs (e.g. pcDNA6.2-GW/EmGFP-miR) utilizando estrategias de clonación estándar.
Nota: El objetivo es crear un casete de expresión miR consistente en una 5' miR región que flanqueaba, la secuencia de las miR y una 3' miR que flanquean la región que puede expresarse del 3' UTR de un gen reportero bajo el control de un promotor de RNA polimerasa tipo II. - Secuencia de la DNA del uso para validar la correcta inserción de las miRs seleccionadas.
- Cultivar células HEK293 en una incubadora de cultivo celular humidificado (37 ° C, 5% CO2) con DMEM medio suplementado con suero bovino fetal 10%, 1 mM NaPyruvate, 10 mM HEPES (pH 7.39) y 1 x penicilina/estreptomicina (Pen/Strep).
- Cuando las células HEK293 confluentes de ~ 70%, co transfectar con cada uno de los vectores de expresión de la miR y un vector expresando el gen objetivo en una relación 1:1 de ADN mediante el método de fosfato de calcio6. Incluyen los siguientes controles: () untransfected las células, (ii) las células transfectadas con el vector expresando el gen objetivo junto con cualquier portador de ADN (por ejemplo, pBluescript II SK(+)) o (iii) Control de miR.
Nota: (i) el gen objetivo expresado debe ser de la misma especie hacia la cual fueron diseñados los miRs, a menos que las secuencias dirigidas por las miRs se conservan absolutamente en el nivel de nucleótidos entre las dos especies. (ii) pueden utilizar líneas de la célula que no sea HEK293. (iii) otros métodos de transfección, como electroporación o métodos basados en liposomas, pueden ser utilizados. -
48 h después de la transfección, lyse las células, ejecutar gel de proteínas, realizar Western blot y analizar contenido proteínico con un anticuerpo que reconoce la proteína diana. Objetivo para una eficiencia de precipitación de ≥50%.
Nota: Eficiencia de precipitación puede alternativamente, o además, se evaluó (i) por análisis de ELISA, (ii) mediante la medición de la actividad de la proteína diana en su caso (por ejemplo densidades de corriente para canales iónicos) o (iii) por qRT-PCR a nivel de mRNA si conveniente los anticuerpos no están disponibles (paso 3 del Protocolo).- Coloque los platos de la cultura en hielo y lavar las células una vez con helada solución salina con tampón fosfato (PBS).
- Aspirar el PBS, luego agregar helado almacenador intermediario RIPA (50 mM Tris-HCl de pH 7.4, 150 mM NaCl, 2 mM EDTA, 1% NP40, 0.1% sodio dodecil sulfato (SDS), que contiene inhibidores de la proteasa y fosfatasa; 1 mL para un plato de Ø 100 mm, 0,5 mL para un plato de Ø 60 mm).
- Raspar las células adherentes el plato con un raspador celular y transferir suavemente la suspensión de células en un tubo de microcentrífuga previamente enfriado.
- Centrifugar el tubo a 15.000 x g durante 15 min a 4 ° C, transferir el sobrenadante en un tubo nuevo de microcentrífuga previamente enfriado y descartar el precipitado.
- Determinar la concentración de proteína usando el kit de ensayo de proteína BCA u otro método adecuado (por ejemplo análisis de Bradford).
Nota: Muestras pueden ser congeladas a-20 ° C o -80 ° C para su uso posterior, o procesadas inmediatamente. - Transferir un volumen adecuado de lisados para tubos de microcentrífuga para que todas las muestras tienen la misma concentración de proteína y añadir tampón de lisis helada adecuada para compensar todos los lisados al mismo volumen.
Nota: 30 a 50 μg de proteína total debe ser suficiente para la mayoría de las proteínas, pero la cantidad que se cargue debe determinarse según la abundancia de la proteína de interés. - Agregar una cantidad apropiada de búfer de x Laemmli 2 (4% SDS, 10% 2-Mercaptoetanol, 20% glicerol, 0.004% bromofenol azul, 0.125 M Tris-HCl, pH 6,8) y hervir las muestras a 95 ° C durante 5 minutos.
- Cargar las muestras en un gel de acrilamida junto con un marcador de peso molecular. Correr el gel a 100 V por 1-2 h.
Nota: El porcentaje del gel depende del tamaño de la proteína de interés. - Montar el sándwich de traslado y transferencia de las proteínas desde el gel hacia una membrana a 100 V durante 2 h. Puede ser la membrana de nitrocelulosa o PVDF. Activar PVDF con metanol durante 1 min y lavar con tampón de transferencia antes de preparar la pila.
- Bloquear la membrana por 1 h a temperatura ambiente utilizando solución amortiguadora de bloqueo (5% de leche en SAFT (PBS + 0,1% Tween-20)), luego incubar la membrana durante la noche a 4 ° C con anticuerpo primario diluido en solución amortiguadora de bloqueo.
- Lavar la membrana por 10 min en SAFT tres veces e incubar con el anticuerpo secundario conjugado con HRP en tampón de bloqueo durante 1 h a temperatura ambiente.
- Lavar la membrana por 10 min en PBS cuatro veces, luego el sustrato quimioluminiscente se aplica a la membrana y capturar las señales quimioluminiscencia utilizando a un sensor de cámara CCD.
- Utilizar software de análisis de imagen para cuantificar la eficacia de la precipitación.
- Seleccione los dos miRs más eficientes dirigidos a secuencias no superpuestos para ensayos más funcionales.
Nota: Efectos de objetivos no pueden nunca ser totalmente descartadas por cualquier miR. Sin embargo, si dos miRs independiente tienen un efecto similar, es muy poco probable que esto es debido a una caída fuera del objetivo mismo. - Si la eficiencia de precipitación de las miRs seleccionados no es satisfactoria (< 50%), la pantalla para otras miRs. Alternativamente, expresa la miR(s) más eficiente en tándem, es decir, expresarlos en copias múltiples del mismo cassette de expresión, que puede resultar en mayor eficiencia de precipitación7.
2. construcción de vectores combinados expresión de Optogenetic sondas y microRNAs recombinante Adeno-asociado
Nota: Este protocolo requiere un conocimiento de los siguientes métodos bien establecidos: clonación molecular, secuenciación de DNA y producción de rAAV.
- Copia de los cassettes de expresión miR más eficientes en el 3' UTR de construcciones diseñadas para producción de recombinantes rAAVs y expresión de los optogenetic las puntas de prueba, tales como pAAV-Syn-ChETA-TdT-miR-X (figura 1A), donde el promotor sinapsina enolase unidades de expresión de la ultrarrápida channelrhodopsin ChETA, reportero fluorescente rojo TdTomato y miR(s) entre los sitios de NheI y EcoRI5.
Nota: (i) no superan el límite de empaquetado de rAAVs, que es aproximadamente 4,7 Kb en longitud de ITR (repetición terminal invertida) a ITR (figura 1A, naranja). (ii) el casete de expresión miR tiene que ser insertado entre el codón de parada y WPRE (elemento postranscripcional del regulador de la Virus de la Hepatitis de marmota; Figura 1A). (iii) inclusión de una proteína fluorescente, como TdTomato, es muy útil ya que permite para monitorear fácilmente la localización y la intensidad de la infección (paso 4 del Protocolo). - Utilice la secuencia de la DNA para validar la correcta inserción de los casetes miR.
- Productos y título rAAV1/2 según el protocolo anteriormente publicado8, rAAV1/2 para cada seleccionado miR. Un experimento típico para la caída de un gene incluiría dos miRs independiente contra el mismo gen y el control de un miR. Busca un título de virus de genomas virales de ≥ 109 (vg) / μl.
PRECAUCIÓN: rAAVs necesita ser manejado en una instalación de nivel de bioseguridad 1 (BSL-1). Para obtener información detallada, consulte con Comité de bioseguridad institucional.
Nota: Si el laboratorio no cuenta con un servicio viral o títulos virales no son satisfactorios, producción de rAAV puede ser subcontratado. Por ejemplo, visite https://www.med.upenn.edu/gtp/vectorcore/ o https://vcf.charite.de/en/metas/.
3. extracción de RNA a partir de cultivos primarios neuronales para la evaluación del miR eficiencia Knockdown de Genes endógenos por qRT-PCR
Nota: (i) este protocolo requiere un conocimiento de los siguientes métodos bien establecidos: preparación y mantenimiento de cultivos neuronales primarios y qRT-PCR. (ii) repetir la cuantificación de la eficiencia de precipitación (pasos 3.1 – 3.14) al menos 3 veces (Replica biológica). (iii) estimación de la eficiencia de precipitación a nivel de mRNA por qRT-PCR es conveniente cuando un análisis del contenido proteínico queda excluido, como cuando tumbaron alternativamente empalmados isoformas que anticuerpos específicos no están disponibles5.
-
Preparar cultivos neuronales primarios de la región cerebral de interés. Seguir el protocolo para las culturas corticales en anterior publicación9, con las siguientes modificaciones:
- Neuronas de la placa en 6 placas de pozos a una densidad de 500.000 neuronas por pozo. Utilizar 2,5 mL/pozo del medio de fijación y 3,3 mL/pozo de medio de mantenimiento.
- Si se observa un crecimiento excesivo de astrositos, Añadir 0,5 mL/pozo de medio de mantenimiento suplementado con 7.5 μm de citosina β-D-arabinofuranoside (para una concentración final de 1 μm) en 3-4 días en vitro (DIV).
-
Infectar a tres pozos por miR (técnica replica) en 5-6 DIV. utilizar la menor dosis infecciosa que infectará el ≥99% de las neuronas.
- Retirar 2 mL del medio de cada pozo y recoger en un tubo falcon de 50 mL.
- Añadir virus directamente a las neuronas, mezclar suavemente y volver a colocar las placas en la incubadora a 37 ° C.
- Almacenar el medio recogido en la incubadora. Flojo el tapón del tubo del halcón para permitir el equilibrio del gas.
- Después de 24 h, añadir el medio quitado de nuevo en cada pozo (1,9 mL/pocillo).
-
En 17,18 DIV, lyse las neuronas para la extracción de RNA.
PRECAUCIÓN: Reactivo de lisis y cloroformo son altamente tóxicos. Trabajar bajo una campana química y usar equipo de protección; disponer de los residuos según sus directrices nacionales e institucionales.
Nota: Para medidas 3.3 – 3.13, trabajo en condiciones libres de Rnasa. Utilice guantes y utilizar libre de Rnasa cristalería y recipientes de plástico desechables. Precauciones generales sobre cómo mango RNA, consulte por ejemplo Apéndice A del manual Micro RNeasy disponibles en línea.- Incubar de vidrio, que pipetas de Pasteur durante la noche en un horno a 180 º C.
- Inclinación de las placas y aspirar el medio de cada pocillo con un pipeta Pasteur conectada a una bomba de vacío de vidrio totalmente.
- Inmediatamente añadir 700 μl de reactivo de lisis para cada pozo.
- Distribuir uniformemente la solución por balanceo y agitar la placa con cuidado y brevemente con la mano.
- Pipetear la solución hacia arriba y hacia abajo 4 a 5 veces o hasta obtener una suspensión homogénea.
- Transferir la solución de cada pozo en un separado tubo Eppendorf de 1,5 mL.
Nota: Los lysates de la célula pueden ser almacenadas a-80 ° C o procesado inmediatamente.
- Si es necesario, descongelar células lisados en RT y proceder de inmediato a paso 3.5.
- Añadir 140 μl de cloroformo a cada muestra de lysates de la célula.
Nota: El cloroformo es volátil y difícil de la pipeta, cerrar los tubos Eppendorf tan pronto como sea posible. - Agite los tubos Eppendorf vigorosamente durante 15 s o hasta que las muestras son totalmente emulsionadas.
- Mantener la muestra a temperatura ambiente durante 1-2 minutos o hasta que el líquido fases comienzan a separarse.
- Centrifugar las muestras a 12.000 x g durante 15 min a 4 ° C.
- Transferir la fase acuosa superior (~ 320 μL) que contienen RNA a un tubo Eppendorf nuevo y deseche el líquido restante.
Nota: No toque la fase orgánica rosa inferior o el anillo blanco entre las dos fases con la punta de la pipeta. - Añadir 1,5 volúmenes de etanol al 100% (480 μL) a la fase acuosa, y pipetear la solución lentamente arriba y abajo 3 veces para mezclar.
- Purificar el RNA usando un kit disponible en el mercado diseñado para la purificación del ARN de muestras pequeñas.
- Cuantificar la pureza de concentración y de la muestra de RNA con un espectrofotómetro.
Nota: (i) esperar rendimientos de ≥3.5 μg/pocillo; 260/280 y 260/230 proporciones de pureza sobre proteínas y compuestos orgánicos debe ambos ser ≥1.9. (ii) las muestras de RNA pueden ser almacenadas a-80 ° C o procesadas inmediatamente. - Retrotranscribe 250, 500 o 1000 ng de ARN con un kit disponible comercialmente.
- Cuantificar la eficiencia de precipitación para el gen endógeno de interés por qRT-PCR. Para un protocolo detallado, ver referencia10. Aspirar a una eficiencia de precipitación de ≥60% (figura 2). Normalizar los datos a los genes housekeeping ≥2 (por ej. GAPDH, ACTB, TUBB3 PPIA) utilizando los múltiples control interno gene método11.
Nota: Si trabaja en rata, utilice los siguientes iniciadores PCR para los genes housekeeping: GAPDH fwd: 5' GGTGCTGAGTATGTCGTGGA 3' y GAPDH-rev: 5' GATGATGACCCTTTTGGC 3'; ACTB-fwd: 5' CATCACTATCGGCAATGAGC 3' y ACTB-rev: 5' TCATGGATGCCACAGGATT 3'; TUBB3-fwd: 5' GCCTTTGGACACCTATTCAG 3' y TUBB3-rev: 5' TCACATTCTTTCCTCACGAC 3'; PPIA-fwd: 5' CACTGGGGAGAAAGGATTTG 3' y PPIA-rev 5' CCATTATGGCGTGTGAAGTC 3'.
4. evaluación del papel de las proteínas presinápticas en circuitos neuronales intactas por estimulación específica de las neuronas derribado con Optogenetics
Nota: El siguiente protocolo requiere experiencia previa con las grabaciones electrofisiológicas en rebanadas de cerebro agudo y el acceso a una instalación electrofisiológico.
- Stereotactically inyectar rAAV1/2 cerebro siguiendo un protocolo detallado en la publicación anterior12. Determinar las coordenadas stereotactic de la región cerebral de interés utilizando Atlas estereotácticos para ratón13 o rata cerebro14.
- Utilizar un extractor de la micropipeta para Micropipetas de inyección con caña larga de Ø 7 – 9 μm; clip las Micropipetas de inyección en la caña con unas tijeras. Use un marcador fino y papel cuadriculado para colocar marcas de calibración de las Micropipetas de inyección cada 2 mm.
- Anestesiar los animales con isoflurano y fijar en el aparato estereotáctico.
- Mantener el animal caliente a lo largo de la operación con una almohadilla térmica a 37 ° C.
- Proteger los ojos con lubricante ocular.
- Afeitar el pelo en la cabeza con una afeitadora eléctrica.
- Difundir el yodo povidona en la cabeza rapada con un bastoncillo de algodón.
- Con un microscopio de disección, haga una incisión de línea media.
- Limpie la superficie del cráneo con un bastoncillo de algodón, para visibilizar el bregma y lambda.
- Colocar la micropipeta de inyección en su soporte. Instale el soporte al brazo estereotáctico.
- Determinar x e y coordenadas del sitio de la inyección en comparación con el bregma y el lambda.
- Use un taladro para diluir el cráneo sobre el área de destino.
Nota: Utiliza movimientos circulares suaves y evitar la perforación a través del cráneo, esto dañará la superficie del cerebro. - Si hay sangrado, absorber el exceso de sangre con papel toalla.
Nota: Exceso de sangre hará difícil determinar correctamente la coordenada z. - Carga ≤ 2 μl de virus en la micropipeta de inyección por la acción capilar.
- Traer la micropipeta de inyección a x e y coordenadas del sitio de la inyección.
- Calcular la coordenada z de la duramadre y baje lentamente la pipeta en el cerebro.
- Al llega a la coordenada z, esperar 3 minutos para permitir el ajuste del tejido.
- Aplicar baja presión positiva utilizando una jeringa de 1 mL conectada mediante un tubo flexible en la parte posterior de la micropipeta de inyección. Controlar visualmente la velocidad de eyección del virus de la micropipeta de inyección a través del microscopio de disección y uso de las marcas de calibración de puntos de referencia.
Nota: Virus tiene que ser inyectado a ritmo lento (< 100 nL/min) para evitar daño tisular. - Una vez terminada la inyección, esperar 5 min, retirarse de la micropipeta de inyección de 0,2 mm, espere otros 5 minutos, para evitar el reflujo de los virus.
- Retirarse la micropipeta de inyección lenta y completamente el cerebro y disponer de él en un recipiente llenado de blanqueador.
- Moje el cráneo con solución fisiológica y suturar la piel con 3-4 puntos.
- Aplicar ungüento de gentamicina a la herida.
- Solo el animal en una jaula limpia con alimento pellets y bajo una lámpara de calor hasta que se recupere completamente.
- ≥ 15 días después de la inyección, decapitar a los animales bajo anestesia isoflurano profundo.
- Elaborar rebanadas de cerebro agudo de la región cerebral de interés con un Vibratome usando gas (95% O2, 5% CO2) solución helada aCSF, que contiene (en mM): NaCl 123, 1.25 1.25 KH2PO4, 1.5 MgCl2, KCl, 1 CaCl2, 25 NaHCO3, NaPyruvate 2 y 18 glucosa (osmolaridad ajustada a 300 mOsm). Por ejemplo, si el objetivo es analizar la transmisión sináptica de CA3 a neuronas piramidales de CA1, prepare rebanadas sagitales de la formación hipocámpica (350 μm de espesor).
Nota: De este paso adelante trabajan bajo condiciones de poca luz para evitar la activación de la sonda de optogenetic por la luz ambiental. - Deje que las rebanadas de recuperar durante 30 min a 37 ° C en la aCSF mismo en un compartimiento diseñado para la celebración de cortes de cerebro. Mantener las rodajas en la misma cámara de trozo de cerebro a temperatura ambiente hasta la grabación.
Nota: Con estas condiciones, rebanadas pueden mantenerse saludables para un máximo de 6-8 h. - Transferencia una rebanada y una cámara de grabación sumergida superfuse con 2 mL/min de la aCSF mismo utilizado para la recuperación con 1,5 mM de CaCl2 (total de Ca2 +: 2, 5 mM) y sin NaPyruvate.
Nota: Rebanadas son preparadas y mantenidas en baja concentración de Ca2 + (1 mM) para reducir al mínimo la toxicidad. Las grabaciones se realizan en 2,5 mM Ca2 + para favorecer la liberación de la vesícula. - Comprobar brevemente la señal del reportero fluorescente expresa (por ejemplo. TdTomato; Figura 3B) para determinar la localización y la intensidad de la infección.
- Se llenan de un electrodo parche una solución intracelular que contiene (en mM): 110 K-gluconato, 22 3 MgCl2, 4 Mg-ATP, Na 0,5, 0,5 EGTA, KCl, NaCl 53-GTP, 20 K2-fosfato de creatina, 10 HEPES-KOH (pH 7.28, 290 mΩ).
Nota: Use parche electrodo con una resistencia de la pipeta de 5-6 mΩ. - Bajo iluminación infrarroja, llegar a sello hermético celulares configuración desde una neurona recibir entradas sinápticas de las neuronas infectadas. Por ejemplo, si se infectaron neuronas piramidales CA3, remiendo de neuronas piramidales en la proximal a la zona medial de la región CA1 (figura 3).
Nota: Resistencia en serie se puede dejar sin compensación, pero debe ser constante y baja (≤20 MΩ). - Use farmacología para aislar las corrientes sinápticas bajo investigación. Por ejemplo, si el objetivo es investigar la transmisión sináptica excitatoria, bloquean la transmisión sináptica inhibitoria con 10 μm bicuculina.
- Evocan las corrientes sinápticas, por ejemplo, corrientes postsinápticas excitatorias (EPSCs), usando un laser de nm azul 473 acoplada a una fibra óptica (Ø ≤250 μm), colocada en los Somas de las neuronas infectadas (por ejemplo. Neuronas piramidales CA3).
- Ajustar longitud de estimulación al mínimo, para reducir la posibilidad de que más de un potencial de acción por pulso de luz. Si usa el ChETA, colóquela a 2 ms15.
- Ajustar la fuerza de la estimulación del láser para producir las corrientes sinápticas pequeñas pero claramente perceptibles (≤50 amplitud de pico de pA para EPSCs grabado en un potencial de explotación de -70 mV entre neuronas piramidales CA3 y CA1; Figura 3D). Si usa ChETA y una fibra óptica de 250 μm de diámetro, estimulación salir fuerzas de 1 a 3 mW en la fibra debe ser adecuada.
Nota: si fuerza de láser no puede ser regulado, usar filtros de densidad neutra. - Iluminar los 473 nm láser en los Somas de las neuronas infectadas, pero no en sus axones. Por ejemplo, brillar la luz en CA3 somata y lejos de los collaterals de Schaffer, para evitar la despolarización directa de los axones.
- Tetrodotoxin (TTX; 0,5 μm) se aplica, un bloqueador de canales de sodio, a la muestra para confirmar los canales están impulsados por el potencial de acción.
- En diferentes grabaciones, aplicar un bloqueador selectivo a la corriente sináptica bajo investigación para confirmar la estimulación selectiva.
Nota: por ejemplo, aplicar NBQX (10 μm) a glutamato tipo AMPA receptor mediada por EPSCs. - Comparar las corrientes sinápticas ópticamente y eléctricamente evocadas en el mismo segmento cerebral aguda en respuesta a la estimulación repetitiva (≥ 2 estímulos en ≤20 Hz). Un grado similar de facilitación sináptica o depresión debe observarse entre la estimulación eléctrica y óptica cuando se utiliza un miR de control, por lo tanto, lo que sugiere que mecanismos similares de celulares son activados por los dos tipos de estímulos.
Nota: Los pasos anteriores son necesarios para asegurar que la estimulación óptica no induce una despolarización directa de botones presinápticos, obviando algunos mecanismos de transmisión sináptica.
- Comparar la transmisión sináptica en respuesta a estímulos simples y repetitivos (≥ 2 estímulos en ≤20 Hz) entre un miR control y miRs a las proteínas presinápticas bajo investigación.
Resultados
Los procedimientos descritos anteriormente proporcionan un método robusto para evaluar la transmisión sináptica cómo se ve afectado por la caída de proteínas sinápticas de las neuronas presinápticas. Resultados representativos de cómo la caída de alternativa empalme isoformas de presinápticos de Cav2.1 (P/Q-type) VGCCs regula plasticidad sináptica a corto plazo en CA3 a CA1 las sinapsis excitatorias se indican a continuación como ejemplo.
CAv2.1 (P/Q-type) los canales son los VGCCs presinápticas predominantes en las sinapsis más rápidas en el sistema nervioso central. Alternativa de empalme de los exones excluyentes 37a y 37b de la formación de poros α1 subunidad de Cav2.1 (α1A) produce dos variantes principales, Cav2.1 [EPT] y Cav2.1 [EFb]16,17 ,18. Para determinar si Cav2.1 [EPT] y Cav2.1 [EFb] diferencialmente regulan la transmisión sináptica y plasticidad en las neuronas piramidales hipocampales de rata, primero desarrollamos específica de isoforma miRs a caída selectiva de Cav 2.1 [EPT] o Cav2.1 [EFb]5. A pesar del tamaño corto (97 bp) y la alta semejanza (61.86% de identidad en el nivel de nucleótido) entre los exones 37a y 37b, podríamos diseñar tres secuencias miR contra rata Cav2.1 [EPT] (miR EFa1: TCCTTATAGTGAATGCGGCCG; miR EFa2: ATGTCCTTATAGTGAATGCGG; miR EFa3: TTGCAAGCAACCCTATGAGGA) y dos contra rata Cav2.1 [EFb] (miR EFb1: ATACATGTCCGGGTAAGGCAT; miR EFb2: ATCTGATACATGTCCGGGTAA) con eficacia prevista de precipitación alta. Como control negativo (Control de la miR), se utilizó el plásmido pcDNA6.2-GW/EmGFP-miR-neg que contiene una secuencia que no dirige ningún gen vertebrado conocido. En base a una primera pantalla en células HEK 293 contra canales heterólogos, miR EFa1, miR EFa3 y miR EFb2 seleccionados y clonados sus cassettes de expresión en el 3' UTR del vector pAAV-Syn-ChETA-TdT-miR-X, que está diseñado para la producción de rAAVs y donde la promotor sinapsina unidades de expresión de la ultrarrápida channelrhodopsin ChETA, la proteína fluorescente roja TdTomato y el miR insertado (figura 1). También duplica el cassette de la expresión de miR EFb2 para aumentar la eficiencia de la precipitación de este miR.
A continuación, preparamos rAAV1/2 para las construcciones de cuatro arriba y cuantificó su precipitación eficiencia y selectividad en cultivos neuronales de rata primaria mediante qRT-PCR específica de isoforma. miR EFa1 miR EFa3 reducido y mRNA de nativa de Cav2.1 [EPT] ~ 70% pero no de Cav2.1 [EFb], mientras que miR EFb reducido mRNA de nativa de Cav2.1 [EFb] ~ 60% pero no de Cav2.1 [EPT] (figura 2).
Entonces stereotactically inyectamos cada uno de los cuatro rAAV1/2 en el área CA3 del hipocampo de ratas P18 (Figura 3A-C), con coordenadas de −2.6 (A-P/M-L/D-V de Bregma) / ± 2.9 / −2.9. Después de la inyección quince a veinte y cuatro días, preparamos rebanadas hippocampal agudas de ratas rAAV1/2-inyectado y utiliza fluorescencia TdTomato para confirmar la expresión y localización de rAAVs (figura 3B). Para investigar si caída presináptico de Cav2.1 [EPT] o Cav2.1 [EFb] afectó la plasticidad sináptica a corto plazo en CA3 a CA1 sinapsis, nos estimulan selectivamente infectadas neuronas CA3 con breve 473 nm láser pulsos ligeros (2 ms de duración ; Figura 3) y las resultantes EPSCs por parches de neuronas piramidales en la proximal a la zona medial de la región CA1 (figura 3). Encontramos que la caída de Cav2.1 isoformas de empalme afectado las respuestas a la estimulación de junto-pulso en direcciones opuestas: caída de Cav2.1 [EPT] (miR EFa1 o miR EFa3) impulsado facilitación de junto-pulso (PPF) Considerando que la precipitación de Ca v2.1 [EFb] (miR EFb2) había abolido (figura 3D, E).
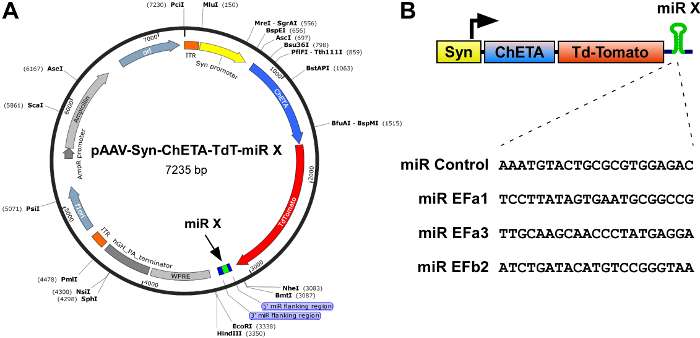
Figura 1: Esquema de las construcciones de la expresión combinada de la sonda de optogenetic ultrarrápida ChETA y miRs de la isoforma específica contra Cav2.1 [EPT] y Cav2.1 [CEE]. (A) mapa de la rAAV construir pAAV-Syn-ChETA-TdT-miR-X, que contiene un promotor sinapsina (Syn), channelrhodopsin ultrarrápida ChETA fundido a TdTomato y en el proximal 3' UTR, un miR de Cav2.1 empalme isoforma específica. Enzimas de restricción que se muestra son solo fresas. Esquema superior, (B) del casete de expresión. El promotor sinapsina unidades de expresión de una isoforma específica miR y ChETA-TdTomato. Fondo, complemento reverso de las secuencias diana de 21 nucleótidos, que forman parte de la cinta de miR. Para miR EFb2 dos casetes miR idénticos fueron expresados en tándem uno tras otro. Haga clic aquí para ver una versión más grande de esta figura.
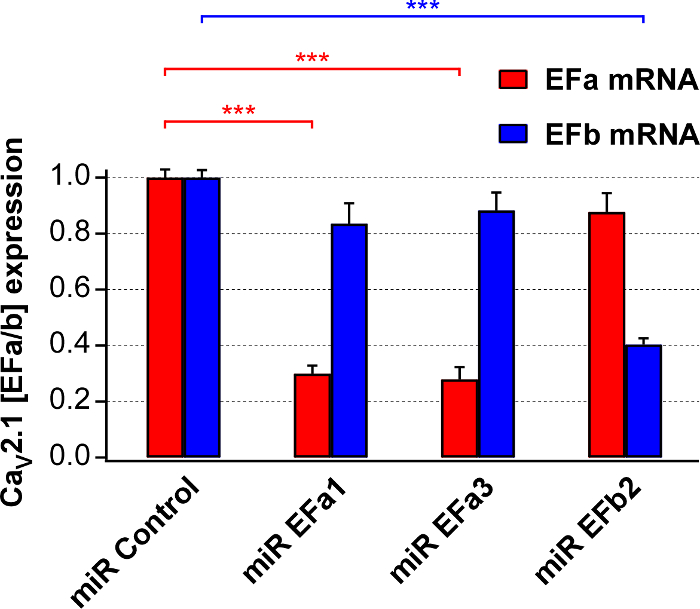
Figura 2. Evaluación de la eficacia de derribo y selectividad de miRs de la isoforma específica de Cav2.1 [EPT] y Cav2.1 [EFb]. Análisis específica de isoforma qRT-PCR de RNA aislado de cultivos primarios de 17-18 DIV infectados en DIV 6 con rAAVs expresando miRs a cualquiera de los dos Cav2.1 [EPT] (miR EFa1 y miR EFa3) o Cav2.1 [EFb] (miR EFb2). Datos son normalizados para el control negativo (Control de la miR). miR EFa1 y miR EFa3 selectiva y significativamente reducen el mRNA de Cav2.1 [EPT] (n = 8 y 7 culturas, respectivamente), mientras que miR EFb significativamente y de forma selectiva reduce el mRNA de Cav2.1 [EFb] (n = 4 culturas; *** p < 0.001; una vía análisis de prueba de varianza seguida por el post-test de Tukey-Kramer). Los datos se presentan como media ± SEM. Esta figura ha sido adaptada de Thalhammer, a. et al. 5. haga clic aquí para ver una versión más grande de esta figura.

Figura 3. Evaluación del papel de Cav2.1 [EPT] y Cav2.1 [EFb] en el hipocampo nativo por dirigida la estimulación de las neuronas derribado con optogenetics. (A) esquema de la cassette de expresión de las construcciones de rAAV utilizado para la infección en vivo . (B) sección hipocampo mostrando esa fluorescencia TdTomato se limita a la región CA3 y sus proyecciones. Configuración Experimental (C) : rayo láser fue dirigido en CA3 somata, y realizaron grabaciones de abrazadera del remiendo de neuronas piramidales de CA1. (D) 2 ms de duración azul (473 nm) pulsos de luz láser brillados en 20 Hz evocan EPSCs cuya PPF es aumentado por miRs a Cav2.1 [EPT] y suprimido por el miR para Cav2.1 [CEE]. Extracto (E) de la relación de junto-pulso para experimentos como en (D), mostrando un incremento en PPF para miR EFa1 y miR EFa3 y una disminución para miR EFb2, comparado con miR Control (n = 9-11 grabaciones; * p = 0.02; ** p = 0,01; *** p < 0,0004; análisis de covarianza). Los datos se presentan como media ± SEM. Esta figura ha sido adaptada de Thalhammer, a. et al. 5. haga clic aquí para ver una versión más grande de esta figura.
Discusión
Coexpresión de una sonda de optogenetic y un miR contra un gene presináptico de interés ofrece un poderoso enfoque para caracterizar el efecto de la precipitación del gene en la función presináptica en circuitos neuronales intactas. Para este enfoque experimental, es importante identificar y caracterizar miRs que son altamente eficientes y selectivos en derribar el gen de interés en sistemas nativos. Si es posible, miRs independientes dos o más contra el mismo ARNm de interés puede usarse para control de posibles efectos off-target. Experimentos de rescate, en la que un gen resistente a la miR es reintroducido en el sistema, pueden utilizarse como un control de especificidad.
Utilizando la misma construcción para expresar una optogenetic sonda y un miR permite estimular ópticamente sólo las neuronas presinápticas que han sido manipuladas. Esto no es posible con estímulos eléctricos porque no distinguen entre las neuronas infectadas y no infectadas, produciendo resultados mezclados y diluidos. Debido a estímulos ópticos pueden inducir múltiples potenciales de acción15, es importante elegir sólo las sondas de optogenetic más rápidas, entre una creciente paleta15,19,20,21. Además, es esencial para asegurar que la estimulación óptica no induce una despolarización directa de botones presinápticos, para evitar pasar por algunos de los pasos de la transmisión sináptica se desea investigar22.
El enfoque experimental que se describe aquí, es posible evaluar en paralelo la relevancia fisiológica de genes múltiples presinápticos de interés dentro de un período de tiempo limitado (4 a 6 meses). Sin embargo, es importante tener en cuenta que caídas rara vez llega a 100%. Además, rAAVs necesitan expresarse durante al menos dos semanas permitir la máxima precipitación y completa expresión de la sonda de optogenetic, que puede representar una restricción de tiempo, la investigación de procesos de desarrollo tempranos. Aunque más ratones knockout condicional, desperdiciador de tiempo limitados a las neuronas presinápticas, generalmente como resultado completar la eliminación del gen de interés y, por tanto, ofrecer un enfoque complementario válido.
Una ventaja específica de la tecnología de miR es que permite la expresión de múltiples miRs de la promotora de la misma. Esta propiedad se ha utilizado principalmente para aumentar la eficiencia de precipitación mediante la inserción de copias múltiples del mismo miR o miRs diferentes contra el mismo gene de la blanco. Sin embargo puede ser utilizado también para caída múltiples genes expresando miRs contra diferentes genes7,23. Esta propiedad podría usarse para silenciar a múltiples proteínas presinápticas para diseccionar las vías de señalización presinápticas.
Aquí hemos combinado optogenetics con miRs artificiales para caracterizar los efectos de la precipitación del gene en la función presináptica en rebanadas hippocampal agudas. Un enfoque similar podría utilizarse también para manipular y la punta de prueba circuitos neuronales en vivo. Además, combinando miRs artificiales con chemogenetic enfoques permitiría a interrogar los circuitos neuronales en escalas de tiempo más largo.
Divulgaciones
Los autores no tienen nada que revelar.
Agradecimientos
Agradecemos a F. Benfenati (Istituto Italiano di Tecnologia, IIT) para apoyo y Carmela Vitale para ayudar con la demostración. Este trabajo fue financiado por el IIT y la Compagnia San Paolo (beca Nº 9734 a LAC).
Materiales
| Name | Company | Catalog Number | Comments |
| Acrylamide | Sigma | Acrylamide/Bis-acrylamide, 30% solution | Toxic |
| Animal Temperature Controller with heat pad | WPI | ATC2000 | |
| BCA protein assay kit | ThermoFisher Scientific | 23225 | |
| BLOCK-iT Pol II miR RNAi Expression Vector kit | ThermoFisher Scientific | K4936-00 | |
| Brain slices Prechamber | Harvard Apparatus | BSC-PC | |
| CCD camera-based imager | Bio-Rad | ChemiDoc™ MP | |
| Cell Culture reagents | Life Technologies | ||
| Chemiluminescent substrate | GE Helthcare | ECL Western Blotting Reagents, RPN2106 | |
| Chloroform | Sigma | C2432 | Toxic |
| Cytosine β-D-arabinofuranoside | Sigma | C6645 | |
| Drill | Foredom | K.1030 | |
| EGTA | Sigma | E4378 | |
| Gentamicin ointment | Local pharmacy | ||
| Glucose | Sigma | G7021 | |
| Hepes | Sigma | 54459 | |
| Injection micropipettes | Narishige | GD1 | |
| Inorganic salts & detergents | Sigma | ||
| K2-creatine phosphate | Calbiochem | 237911 | |
| KGluconate | Fluka | 60245 | |
| Laser | Laserglow Technologies | LRS-0473-GFM-00100-03 | |
| Membrane | Amersham | Protran™ 0.2 µm NC | |
| MgATP | Sigma | A9187 | |
| Micropipette holder | Narishige | IM-H1 | |
| Micropipette puller | Narishige | PC-100 | |
| Na3GTP | Sigma | G8877 | |
| NaPyruvate | Sigma | P5280 | |
| Ocular lubricant | Local pharmacy | Lacrigel | |
| Phosphate inhibitors | Sigma | P0044, P5726 | |
| Protease inhibitors | Sigma | cOmplete™, EDTA-free Protease Inhibitor Cocktail | |
| Protein gel electrophoresis and blotting devices | Bio-Rad | Mini-Protean III Cell | |
| Providone iodine | Local pharmacy | Betadine | |
| Qiazol Lysis Reagent | Qiagen | 79306 | Toxic |
| QuantiTect Reverse Transcription Kit | Qiagen | 205311 | |
| RNeasy Micro Kit | Qiagen | 74004 | |
| Spectrophotometer | ThermoFisher Scientific | Nanodrop 2000 | |
| Stereotactic apparatus | WPI | ||
| Tetrodotoxin | Tocris | 1069 | Toxic |
| Vibratome | Leica | VT1200S |
Referencias
- Lam, J. K., Chow, M. Y., Zhang, Y., Leung, S. W. siRNA Versus miRNA as Therapeutics for Gene Silencing. Mol Ther-Nucl Acids. 4, e252(2015).
- Kohl, M. M., et al. Hemisphere-specific optogenetic stimulation reveals left-right asymmetry of hippocampal plasticity. Nat Neurosci. 14 (11), 1413-1415 (2011).
- Maejima, T., et al. Postnatal loss of P/Q-type channels confined to rhombic-lip-derived neurons alters synaptic transmission at the parallel fiber to purkinje cell synapse and replicates genomic Cacna1a mutation phenotype of ataxia and seizures in mice. J Neurosci. 33 (12), 5162-5174 (2013).
- Mark, M. D., et al. Delayed postnatal loss of P/Q-type calcium channels recapitulates the absence epilepsy, dyskinesia, and ataxia phenotypes of genomic Cacna1a mutations. J Neurosci. 31 (11), 4311-4326 (2011).
- Thalhammer, A., et al. Alternative Splicing of P/Q-Type Ca2+ Channels Shapes Presynaptic Plasticity. Cell Rep. 20 (2), 333-343 (2017).
- Sambrook, J., Russell, D. W. Calcium-phosphate-mediated Transfection of Eukaryotic Cells with Plasmid DNAs. CSH Protoc. 2006 (1), (2006).
- Chung, K. H., et al. Polycistronic RNA polymerase II expression vectors for RNA interference based on BIC/miR-155. Nucleic Acids Res. 34 (7), e53(2006).
- McClure, C., Cole, K. L., Wulff, P., Klugmann, M., Murray, A. J. Production and titering of recombinant adeno-associated viral vectors. J Vis Exp. (57), e3348(2011).
- Cingolani, L. A., et al. Activity-dependent regulation of synaptic AMPA receptor composition and abundance by beta3 integrins. Neuron. 58 (5), 749-762 (2008).
- Huang, H., Xu, Y., Cheng, C. Detection of alternative splicing during epithelial-mesenchymal transition. J Vis Exp. (92), e51845(2014).
- Vandesompele, J., et al. Accurate normalization of real-time quantitative RT-PCR data by geometric averaging of multiple internal control genes. Genome Biol. 3 (7), (2002).
- Cetin, A., Komai, S., Eliava, M., Seeburg, P. H., Osten, P. Stereotaxic gene delivery in the rodent brain. Nat Protoc. 1 (6), 3166-3173 (2006).
- Paxinos, G., Franklin, K. B. The Mouse Brain in Stereotaxic Coordinates. , Academic Press. (2012).
- Paxinos, G., Watson, C. The Rat Brain in Stereotaxic Coordinates. , Academic Press. (2013).
- Gunaydin, L. A., et al. Ultrafast optogenetic control. Nat Neurosci. 13 (3), 387-392 (2010).
- Bourinet, E., et al. Splicing of alpha 1A subunit gene generates phenotypic variants of P- and Q-type calcium channels. Nat Neurosci. 2 (5), 407-415 (1999).
- Chaudhuri, D., et al. Alternative splicing as a molecular switch for Ca2+/calmodulin-dependent facilitation of P/Q-type Ca2+ channels. J Neurosci. 24 (28), 6334-6342 (2004).
- Soong, T. W., et al. Systematic identification of splice variants in human P/Q-type channel alpha1(2.1) subunits: implications for current density and Ca2+-dependent inactivation. J Neurosci. 22 (23), 10142-10152 (2002).
- Berndt, A., et al. High-efficiency channelrhodopsins for fast neuronal stimulation at low light levels. P Natl Acad Sci USA. 108 (18), 7595-7600 (2011).
- Klapoetke, N. C., et al. Independent optical excitation of distinct neural populations. Nat Methods. 11 (3), 338-346 (2014).
- Lin, J. Y., Lin, M. Z., Steinbach, P., Tsien, R. Y. Characterization of engineered channelrhodopsin variants with improved properties and kinetics. Biophys J. 96 (5), 1803-1814 (2009).
- Zhang, Y. P., Oertner, T. G. Optical induction of synaptic plasticity using a light-sensitive channel. Nat Methods. 4 (2), 139-141 (2007).
- Fowler, D. K., Williams, C., Gerritsen, A. T., Washbourne, P. Improved knockdown from artificial microRNAs in an enhanced miR-155 backbone: a designer's guide to potent multi-target RNAi. Nucleic Acids Res. 44 (5), e48(2016).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados