Method Article
Divisão celular dos mamíferos em matrizes 3D através de microscopia de reflexão Confocal quantitativa
* Estes autores contribuíram igualmente
Neste Artigo
Resumo
Este protocolo eficientemente estuda divisão de células de mamíferos em matrizes de colágeno 3D, integrando a sincronização da divisão celular, monitoramento de eventos de divisão em matrizes 3D usando a técnica de imagem live-célula, microscopia de reflexão confocal tempo-resolvido e análise quantitativa de imagem.
Resumo
O estudo de mamífero como a divisão celular é regulamentado em um 3D ambiente permanece largamente inexplorado, apesar de sua relevância fisiológica e significado terapêutico. Possíveis razões para a falta de exploração são as limitações experimentais e desafios técnicos que tornam o estudo da divisão celular em 3D cultura ineficiente. Aqui, descrevemos um método baseado em imagem para estudar eficientemente a divisão de células de mamíferos e célula-matriz interações em matrizes de colágeno 3D. Células rotuladas com H2B fluorescentes são sincronizadas usando a combinação de tratamento de bloqueio e nocodazole timidina, seguido por uma técnica mecânica de agitar-se. Células sincronizadas depois são incorporadas a uma matriz de colagénio 3D. Divisão celular é monitorada usando microscopia de viver-pilha. A deformação das fibras de colágeno durante e após a divisão celular, que é um indicador de interação célula-matriz, pode ser monitorizada e quantificados usando microscopia de reflexão confocal quantitativa. O método fornece uma abordagem eficiente e geral para estudar a divisão de células de mamíferos e célula-matriz interações em um ambiente 3D fisiologicamente relevante. Esta abordagem não só fornece novos insights sobre a base molecular do desenvolvimento do tecido normal e doenças, mas também permite o desenho de novas abordagens diagnósticas e terapêuticas.
Introdução
Mitose da célula é um evento crítico na vida celular, o Regulamento das quais tem um papel crucial no desenvolvimento de tecidos e órgãos. Mitose anormal está implicada na variação genética natural, os processos de envelhecimento humano e a progressão do câncer1,2,3,4,5. O aumento da taxa de proliferação de células tumorais em comparação com células normais é uma das marcas registradas do cancro, apesar do fato de que comportamentos de célula são bastante heterogêneos entre os diferentes tipos de tumores e até mesmo entre os pacientes. Apesar de resultados promissores pré-clínicos, alguns medicamentos antimitóticos recém-desenvolvidos não têm demonstrado para ser eficaz em testes clínicos6,7,8,9,10 ,11. A relevância de modelos experimentais e pré-clínicos tem que ser considerado. Muitos tipos de células de mamíferos e câncer normais dividem-se em matrizes de tridimensionais (3D), como fibroblastos e células de Fibrossarcoma em colágeno-rico 3D tecidos conjuntivos e células de câncer metastático na matriz extracelular do estroma 3D (ECM). No entanto, a grande maioria dos experimentos de divisão de células de mamíferos e os ensaios foram realizada em células cultivadas em substratos de bidimensional (2D). Uma matriz 3D projetado melhor pode recapitular a microestrutura, propriedades mecânicas e bioquímicos sinais de ECM 3D de ambos os tecidos normais e patológicos12,13,14, 15,16,17.
O estudo de mamífero como a divisão celular é regulado em 3D ambientes permanece largamente inexplorado, apesar de tanto a relevância fisiológica e o significado terapêutico18,19. Possíveis razões incluem a dificuldades técnicas e desafios experimentais associados a estudar a divisão celular em matrizes 3D. Mitose celular constitui uma pequena fração temporal no ciclo celular completo20. Trabalho anterior demonstrou que a taxa de proliferação de muitas células de mamíferos, como o adenocarcinoma de mama humano MCF-7, osteossarcoma humana U2OS e fígado humano HepG2, é muito menor em matrizes 3D em comparação com suas contrapartes em substratos 2D21, 22. Além disso, células incorporado em matrizes 3D entram e saem de foco durante a imagem latente da viver-pilha. Todos esses fatores contribuem para a extremamente baixa eficiência de captura de eventos de divisão celular em 3D cultura usando técnicas de imagem.
Interações entre o ECM e células desempenham um papel crítico na regulação de divisões celulares. Aqui, descrevemos uma abordagem para estudar a divisão de células de mamíferos em matrizes de colágeno 3D eficientemente. O método inclui a incorporação de marcadores mitóticas para as células, sincronização da divisão celular, bem como o acompanhamento dos eventos de divisão em matrizes 3D usando a técnica de imagem live-célula, microscopia de reflexão confocal tempo-resolvido, e análise quantitativa de imagem. Proteína de fluorescência-etiquetada histona H2B é introduzido pela primeira vez dentro das células, como um marcador para diferenciar mitose e interfase as células. Em seguida, as células são sincronizadas usando a combinação de tratamento de bloqueio e nocodazole timidina, seguido por uma técnica mecânica de agitar-se. Células sincronizadas então são encapsuladas diretamente em matrizes de colágeno 3D. Eventos de divisão celular de células múltiplas são monitorados com eficiência usando imagens de viver-pilha baixa-ampliação lapso de tempo. A deformação das fibras de colágeno, que é um indicador de interação célula-matriz, é monitorada usando microscopia confocal reflexão em alta ampliação.
Anteriormente usamos essa técnica para monitorar e quantificar a interação célula-matriz antes, durante e após a mitose de duas linhas de células de câncer metastático, carcinoma ductal invasivo humano MDA-MB-231 e Fibrossarcoma humana HT1080 células, colágeno 3D matrizes de19. Os métodos aqui apresentados fornecem uma abordagem eficiente e geral para estudar os dois mamíferos divisão celular em um 3D interações de ambiente e célula-matriz. A linha de celular MDA-MB-231 é usada como um exemplo em todo o papel. Este protocolo fornece novos insights sobre a base molecular do desenvolvimento do tecido normal e doenças e também pode permitir para a concepção de novas abordagens diagnósticas e terapêuticas.
Protocolo
O protocolo fornecido segue as diretrizes de Homewood The das revisão institucional na placa (HIRB).
1. expressao de H2B-mCherry como um marcador para a mitose da célula
-
Geração de particulas de Lentivirus de rim embrionário humano 293T (HEK 293T) células
- As células HEK 293T numa placa de cultura de células de 10 cm em uma densidade populacional de 5 x 106 células/prato em meio de cultura celular (de Dulbecco médio (DMEM) contendo alta glicose modificado águia (4,5 g/L), piruvato de sódio, 10% de soro Fetal bovino (FBS) e 1% da placa penicilina-estreptomicina (caneta/Strep)). Incube por 24 h a 37 ° C e 5% de dióxido de carbono (CO2). A desejado confluência das células no dia do transfection é cerca de 70-80%.
Nota: As células de 293T HEK são usadas aqui para produzir partículas virais, que serão usadas posteriormente para transduce MDA-MB-231 células a fim de gerar uma linhagem de células expressando estàvel H2B-mCherry. Certifique-se que as células estão distribuídas uniformemente através da placa. Adição das células largas sábio à placa e suavemente movendo a placa e para trás pode ajudar na distribuição uniforme. Células também podem ser semeadas em placas ou frascos de outros tamanhos, o que resultarão na coleção de diferentes volumes de partículas virais. - Transfect as células HEK 293T usando um reagente de transfeccao com três plasmídeos (vetor de Lentivirus, CMV ΔR 8,91 e pMDG-VSVG). O plasmídeo codificação H2B-mCherry é clonado em um vetor de Lentivirus com promotor de fosfoglicerato quinase (PGK). ΔR CMV 8,91 contém três necessários HIV genes, gag, pole rev. pMDG-VSVG contém o gene de envelope de VSV-G.
Nota: Existem vários agentes de Transfeccao para escolher.- Permitir que o frasco de reagente de Transfeccao para atingir a temperatura (RT) antes do uso. Invertido ou vórtice do frasco brevemente.
- Mix 16 µ g de DNA, que consiste de 6 µ g de plasmídeo H2B-mCherry, 8 µ g de ΔR CMV 8,91 e 2 µ g do pMDG-VSVG, em 1 mL de mídia soro reduzida. Incube a RT por 5 min.
Nota: A quantidade e a proporção dos três vetores é variado e otimizado para vetores de transferência Lentivirus diferentes. - Adicione 48 µ l (use a proporção de 1:3 do DNA: reagente de transfeccao) do reagente de Transfeccao para a solução de DNA acima e então incubar a RT pelo menos 15 min.
Nota: A relação do DNA vs reagente de transfeccao é variada e otimizada para vetores de transferência Lentivirus diferentes. Certifique-se que o reagente de transfeccao não entra em contacto com o lado do tubo de centrífuga de 1,5 mL. - Adicione a solução de complexos lipídios-DNA feita na etapa 1.1.2.3 drop-wise para as células. Agite suavemente a placa para garantir a distribuição uniforme do complexo no prato.
- Aproximadamente 6 h após a transfeccao, aspirar a médio e adicionar o meio de cultura celular fresco (DMEM com a glicose alta (4,5 g/L), piruvato de sódio, 10% FBS e 1% caneta/Strep).
- Recolher o sobrenadante 24, 48 e pós-transfecção 72 h. Substituir o médio após cada colheita (DMEM, 4,5 g/L glicose, piruvato de sódio, com 10% FBS e 1% caneta/Strep).
- Filtre as Particulas de Lentivirus através de um filtro de 0,45 µm para remover os restos celulares. Alternativamente, spin para baixo o sobrenadante para separar para fora os detritos de células. O sobrenadante pode ser usado para transdução imediatamente, ou pode ser armazenado a-80 ° C.
- As células HEK 293T numa placa de cultura de células de 10 cm em uma densidade populacional de 5 x 106 células/prato em meio de cultura celular (de Dulbecco médio (DMEM) contendo alta glicose modificado águia (4,5 g/L), piruvato de sódio, 10% de soro Fetal bovino (FBS) e 1% da placa penicilina-estreptomicina (caneta/Strep)). Incube por 24 h a 37 ° C e 5% de dióxido de carbono (CO2). A desejado confluência das células no dia do transfection é cerca de 70-80%.
-
Geração de célula linhas estàvel expressando H2B-mCherry
Nota: Este protocolo é descrito em detalhes para células MDA-MB-231. Antes de utilizar no experimento, células de carcinoma de mama humano MDA-MB-231 (centro de Oncologia de ciências físicas, NIH) são cultivadas em DMEM com glicose alta (4,5 g/L), piruvato de sódio, 10% FBS e 1% caneta/Strep.- As células de MDA-MB-231 em uma densidade populacional de 1 x 105 células numa placa de cultura de 35 mm da placa.
Nota: A densidade do chapeamento para linhas de célula diferente deve ser otimizada e, portanto, pode ser diferente a densidade ideal para as células de MDA-MB-231. - 24 h após o chapeamento das células, adicionar 1 mL de vírus e 1 mL de meio fresco (DMEM, glicose alta (4,5 g/L), piruvato de sódio, 10% FBS e 1% caneta/Strep).
- Incube as células para um período de 6 h durante a noite.
Nota: Tanto o volume do vírus e o tempo de incubação precisa ser otimizado para linhas celulares diferentes. Por exemplo, se as células não parece saudáveis algumas horas após a adição do vírus, utilize 1 mL de vírus e 2 mL de meio fresco (DMEM, glicose alta (4,5 g/L), piruvato de sódio, 10% FBS e 1% caneta/Strep) para etapa 1.2.2. Pode também ser reduzido o tempo de incubação na etapa 1.2.3. - Aspire o meio e adicionar 10 mL de meio fresco (DMEM, glicose alta (4,5 g/L), piruvato de sódio, 10% FBS e 1% caneta/Strep). Encube por entre 24 a 72 horas a 37 ° C e 5% de CO2.
- Verifique se a expressão de H2B-mCherry nas células usando um microscópio de epifluorescência.
Nota: Se a eficiência de transdução é baixa nas células, o volume de vírus na etapa 2.2 e o tempo de incubação na etapa 2.3 poderia ser aumentado.
- As células de MDA-MB-231 em uma densidade populacional de 1 x 105 células numa placa de cultura de 35 mm da placa.
2. sincronização das células expressando estàvel H2B-mCherry
- Placa em cada poço de uma placa de 24 células as células em 50 a 60% confluencia, ou seja, placa de 2 x 104 MDA-MB-231.
- Incube a cultura em 37 ° C e 5% CO2 por 24 h.
- Substituir o meio de crescimento com 0,5 mL de meio (DMEM, glicose alta (4,5 g/L), piruvato de sódio, 10% FBS e 1% caneta/Strep) contendo timidina 2mm e deixar na incubadora durante 24 h.
Nota: As células expostas a timidina são presos na fase de crescimento celular (G1) / transição de síntese (S) de DNA e durante a fase S devido a inibição da síntese de DNA. A duração da incubação deve ser variada e otimizada para linhas de célula diferente. - Libere as células da exposição de timidina lavando-os com solução salina tamponada fosfato (PBS) três vezes. Incubar em seguida, as células em meio de cultura celular normal (DMEM, glicose alta (4,5 g/L), piruvato de sódio, 10% FBS e 1% caneta/Strep) por 5h.
Nota: A liberação das células a partir da exposição de timidina permite que as células avançar para o crescimento de células (G2) / mitótica (M) fase para células anteriormente preso na fase G1/S e à fase G1 para células anteriormente preso ao S de fase. O comprimento do tempo de liberação deve ser variado e otimizado para linhas de célula diferente. - Bloquear as células com 250 ng/mL de nocodazole para 12 h.
Nota: Todas as células expostas a nocodazole são presos na fase G2/M. Nocodazole é citotóxica. A exposição prolongada ao nocodazole pode causar apoptose. Ajuste o período ou concentração de exposição para linhas de célula diferente se mortes de célula são observados. As células que são sincronizadas com êxito irão expor uma morfologia esférica. - Agitar as células para 45 s a 1 min usando um agitador orbital em 150 a 200 rpm.
Nota: Células mitóticas, que têm pouca aderência ao substrato, serão abaladas durante o processo. - Remover o meio para extrair células, pipetando o meio para um tubo de centrifugação e em seguida, adicionar 0,5 mL de meio fresco (DMEM, glicose alta (4,5 g/L), piruvato de sódio, 10% FBS e 1% caneta/Strep) para cada poço da placa.
- Repita os passos de 2,6 e 2,7 três vezes.
- Centrifugue a mídia coletada que contém as células mitóticas na 800 x g, durante 3 min.
Nota: Este passo é usado para remover o nocodazole do meio celular.
3. incorporação do sincronizado células em colágeno matrizes
Nota: Tipo I colágeno é a proteína mais abundante no organismo humano e na ECM dos tecidos conjuntivos e, portanto, é amplamente utilizado para investigar a célula eucariótica como funções são moduladas por um ambiente 3D17,23,24 . Colágeno é solúvel em ácido acético. Após a neutralização e o aquecimento da solução de colágeno para 20-37 ° C, monômeros de colágeno polimerizam em uma malha de fibras de colágeno.
- Prepare a solução de x DMEM a 10 pela dissolução de um pacote de pó DMEM, 3,7 g de bicarbonato de sódio (NaHCO3) e 1 g de 4-(2-hydroxyethyl)-1-piperazineethanesulfonic acid (HEPES) em 50 mL de água destilada. Filtrar a solução e então prepare-se 1 M de hidróxido de sódio (NaOH) pela dissolução de 2 g de NaOH pelotas em 50 mL de água destilada. Filtro e alíquota da solução em tubos de centrífuga de 1,5 mL.
Nota: A solução DMEM Normal não deve ser usada nesta etapa. A adição de um volume significativo da solução de colágeno diluirá o meio. Portanto, a solução concentrada de DMEM é preparada para garantir que a concentração final de DMEM na matriz de colágeno será o mesmo que o DMEM normal. - Continue a trabalhar com as células coletadas de passo 2.9. Aspire a médio e ressuspender as células em cerca de 0,25 - 0,5 mL de meio de cultura celular fresco (DMEM, glicose alta (4,5 g/L), piruvato de sódio, 10% FBS e 1% caneta/Strep).
Nota: Para chegar a uma densidade de célula específica na matriz de colagénio, a densidade inicial de células em suspensão não pode ser muito baixa. Assim, o volume do meio usado para re-suspender as células dependerá do número total de células disponíveis. - Lugar de 10 µ l da solução de célula re-suspenso da etapa 3.2 em um hemocytometer e contagem a densidade das células na solução.
-
Determine os volumes necessários para todos os componentes fazer a matriz de colagénio 3D. 500 µ l de gel de colágeno µ g / µ l 2 é usado aqui como um exemplo.
- Calcule o volume da solução de células necessária para obter a 40.000 células/mL (ou 20.000 células para a 500 µ l de gel de colágeno). Este número em µ l é X. 50 µ l de 10 x DMEM será necessária. 50 µ l de FBS serão necessários.
- Calcule o volume de colágeno que armazeno necessário. A concentração de colágeno I é em torno de 4 µ g / µ l. A quantidade de colagénio necessário em µ l é Y.
- Calcular o volume de hidróxido de sódio precisa neutralizar o ácido acético na solução de colágeno: colágeno de µ l Y * 0,023 * 1000 = Z µ l NaOH necessária.
- Determinar o volume da solução de enchimento: (500 µ l total gel - células de X µL - 50 µ l 10 X colágeno para µ l de Y do 50 µ l de FBS - DMEM - Z µ l NaOH) = R µ l
Nota: A solução de enchimento é destilada água. Se o volume calculado de solução o enchimento é um número negativo, a densidade celular original na suspensão de célula é muito baixa. As células precisam ser girado para baixo novamente e re-suspenso em um menor volume de meio (DMEM, glicose alta (4,5 g/L), piruvato de sódio, 10% FBS e 1% caneta/Strep).
- Adicionar os componentes na seguinte ordem para um tubo de centrifugação limpa 1,5 mL: células em média, 10 x DMEM, FBS, deionizada (DI), colágeno, NaOH. Coloque todos os componentes no gelo para abrandar a gelificação da solução de colágeno. Use técnicas de pipetagem apropriadas para evitar a formação de bolhas.
- Depois de adicionar o NaOH, misture cuidadosamente a solução com uma pipeta de 1 mL.
Nota: Gelificação do colágeno vai começar certa imediatamente após a adição de NaOH. Mistura deve ser feito com cuidado e rapidamente. - Uma vez que a solução é bem misturada, adicione 500 µ l da solução a cada poço da placa de 24 poços. Coloque a placa de 24 na incubadora 37 ° C. Coloque o meio de cultura celular fresco em banho-maria a 37 ° C.
Nota: Placas de plástico inferior são utilizadas para viver-pilha de baixa ampliação microcopy em que a distância de trabalho da lente é acima de 1 mm. fundo de vidro as placas são usadas para microscopia de viver-pilha alta ampliação devido a curta distância de trabalho de alto lente de ampliação. - Adicionar 500 µ l de meio pré-aquecido (DMEM, glicose alta (4,5 g/L), piruvato de sódio, 10% FBS e 1% caneta/Strep) ao topo do gel 30 min depois de concluir a etapa 3.7.
4. live Cell Imaging das células se dividindo em matrizes de colágeno 3D (baixa ampliação)
Nota: Imagens de células são coletadas em intervalos de 2 min, usando uma câmera de dispositivo acoplado (CCD) de carga montada em um microscópio de contraste de fase que é equipado com um objetivo X 10 e controlado pelo software de imagem.
- Liga a unidade de célula viva montada em cima da lente objetiva. Espere até que a temperatura se estabilize a 37 ° C, a concentração de CO2 é de 5% e a umidade é de 75%.
- Colocar a placa de gel 24 na unidade de célula viva no microscópio. Encontrar o fundo do prato e em seguida, mova a lente objetiva até o microscópio centra-se em cerca de 500 µm da parte inferior da placa.
Nota: As células na matriz do colágeno que estão muito perto do fundo do prato não são completamente incorporados na matriz 3D. As células que são 500 µm longe da parte inferior da placa de imagem irá garantir que as células não são afetadas pelos efeitos de borda16,17,25. - Mova o palco ao redor para encontrar as células e selecione várias posições para a imagem latente.
- Configure o experimento de lapso de tempo em intervalos de 2 min.
Nota: O número máximo de posições que podem ser tomadas durante o intervalo de 2 min é limitado pela velocidade de movimento do palco motorizado e o comprimento do tempo de exposição para captura de imagens.
5. o colágeno rede deformação durante a divisão celular (microscopia de alta magnificação)
- Liga a unidade de célula viva montada em cima da lente objetiva. Espere até que a temperatura se estabilize a 37 ° C, a concentração de CO2 é de 5% e a umidade é de 75%.
- Para visualizar as fibras de colágeno em matrizes de colágeno 3D imaculado, configure um microscópio confocal para capturar a luz apenas refletida (488 nm) do 488 nm laser usado para iluminar a amostra. O laser usa um 60 X objetivo de água-imersão, at = 1.2, WD = 200 µm e é controlado pelo software de imagem.
- Colocar a amostra (células em matrizes de colágeno) na unidade de células vivas. Encontrar o fundo do prato e em seguida, mova a lente objetiva até o microscópio centra-se em cerca de 100 µm do fundo da placa. As células que são 100 µm longe da parte inferior da placa de imagem irá garantir que as células não são afetadas pelos efeitos de borda.
Nota: Aqui um objectivo de imersão de água é usado em vez de uma lente de imersão de óleo porque a distância de trabalho da lente de imersão de óleo é apenas cerca de 100 µm. O uso da lente óleo-imersão coloca um problema como a espessura do vidro na parte inferior da placa é normalmente cerca de 100 µm. - Mova o palco ao redor para encontrar as células e selecione várias posições para a imagem latente.
Nota: Microscopia Confocal opticamente seções a amostra e só capta os sinais do plano focal. Células vivas se podem estar dentro e fora de foco durante o experimento de lapso de tempo. Para habilitar a captura de imagens da célula, durante longos períodos de tempo, Z-pilha é usada para coletar imagens de sucessivas fatias em intervalos de 5 µm. São imagens de um total de 5 fatias, abrangendo 25 µm. - Configure o experimento de lapso de tempo em intervalos de 5 min.
Nota: 5 minutos é usado aqui como o intervalo em vez de 2 min, usado na seção 4, porque múltiplos modos de varredura, incluindo digitalização para H2B-mCherry, reflexo de digitalização para colágeno e Z-pilha de varredura, de fluorescência são aplicados para cada posição. O uso de múltiplos modos de digitalização reduz a velocidade de varredura de uma posição quando comparado com a imagem de baixa ampliação. - Quantificar a deformação da matriz de colágeno na proximidade de uma célula, devido à força exercida por essa célula usando a partícula velocimetry (PIV) com sub-pixel resolução20de imagem. Realizando esta análise em imagens 2D permite a visualização clara de ambos os sinais do H2B-mCherry e as fibras de colágeno. Aplique a passa-baixa anisotropic filtragem para melhorar o sinal do colágeno rede20.
- A fim de medir o deslocamento local de uma região sub imagem de interesse localizado em (x,y) do quadro k k+ 1 do quadro, extraia uma janela regional de 15 × 15 pixels centrado em (x,y) no quadro k. Em seguida, identificar a melhor correspondência locais (x,y) * através os locais que apresentam um coeficiente de correlação cruzada normalizado máximo na imagem obtida no quadro k+ 1. O vetor de deformação é calculado como (x,y) *-(x,y).
Resultados
O objetivo deste artigo é apresentar um método baseado em imagem para estudar processos de divisão de células de mamíferos em matrizes 3D e para quantificar as interações entre a célula e a matriz extracelular 3D durante e após a divisão celular. Para facilitar a geração de imagens da mitose celular, incorporamos H2B-mCherry em células de MDA-MB-231 usando Lentivirus transdução. H2B conjugados com proteínas fluorescentes é usado como um marcador mitótico para distinguir células mitóticas das células de interfase e para definir diferentes estágios durante celular mitose19,20,26. Usando esse método, fomos capazes de monitorar o processo de divisão das células MDA-MB-231 estàvel expressando H2B-mCherry nas matrizes de colágeno 3D (figura 1A). Fases mitóticas começaram com a dissolução da membrana nuclear (prófase, quadro 1, figura 1A); a re-organização dos cromossomos (prometafase: quadro 2); o alinhamento dos cromossomos no meio do corpo celular (metáfase; quadro 3); a separação dos cromossomos (anáfase; quadro 4); a reorganização da membrana nuclear e os cromossomos e a separação dos corpos das duas células-filhas (telófase/citocinese, quadro 5).
A taxa de proliferação de células em uma matriz 3D é geralmente muito menor do que suas contrapartes em um substrato 2D, o que torna o controlo da divisão celular em 3D menos eficiente19. Para aumentar a eficiência de estudar a divisão celular 3D, usamos um método combinando timidina, nocodazole e uma técnica de agitação para sincronizar e selecione células MDA-MB-231 que estão na fase mitótica. Sincronizada de células foram então incorporadas em matrizes de colágeno. O processo de divisão de várias células foi monitorado em tempo real usando imagem latente da viver-pilha na ampliação de 10x. Cerca de 70% das células dividido até a primeira 2 horas após a formação da matriz de colágeno (Figura 2), permitindo assim um controlo eficaz da mitose em 3D (Figura 2).
Para monitorar a interação entre as células e suas matrizes de colágeno circundante, nós combinamos a microscopia confocal de reflexão de fibras de colágeno de imagem e microscopia de fluorescência para as células de imagem. Uma lente X 60 permite a captura de imagens de alta qualidade das fibras de colágeno. De imagem usando uma lente de alta ampliação, que capta menos células em cada campo de vista, é muito baixa produtividade em comparação com o uso de baixa ampliação de 10x ou 20x. Sincronização bem-sucedida das células realça extremamente a eficiência e a taxa de transferência de tal experiência, desde que a maioria das células sincronizadas divide dentro as primeiras 2 horas após a formação da matriz de colágeno. A deformação da matriz durante e após a divisão celular é visualizada (representante instantâneos são mostrados na figura 1B) e quantificado usando um software personalizado do PIV. Nós quantificados e comparados a deformação da matriz durante a interfase e fase mitótica para as células sincronizados na fase mitótica. Observamos que a deformação de matriz mudou muito pouco durante a mitose fase (Figura 3A), e é menor do que a deformação observada nas fases pós mitóticas (Figura 3B). Este resultado mostra que células de mamíferos têm apego mínimo e interações com a matriz circundante, enquanto eles estão na fase mitótica.
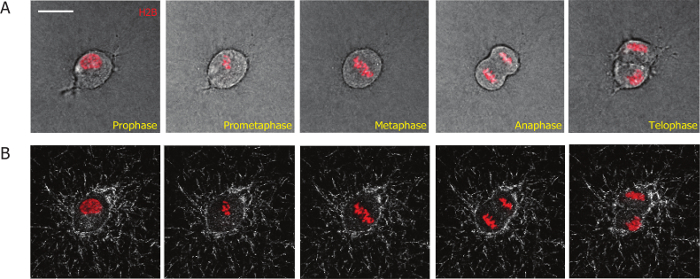
Figura 1: representante micrografias obtidas a partir de um vídeo da imagem latente da viver-pilha alta ampliação de uma célula de MDA-MB-231 incorporado em uma matriz de colagénio que expressa estàvel H2B-mCherry. (A) as diferentes fases da progressão mitótica da célula MDA-MB-231 são definidas pelo H2B-mCherry, como marcado em vermelho. (B) as fibras de colágeno (brancas) durante o processo mitótico são visualizadas por microscopia confocal reflexão dependente do tempo. Barra de escala = 20 µm. clique aqui para ver uma versão maior desta figura.

Figura 2: divisão das células sincronizadas em matrizes de colágeno 3D. Células foram sincronizadas para fase G2/M e incorporadas em uma matriz de colagénio. Cerca de 70% das células sincronizadas dividir dentro as primeiras 2 horas, Considerando que o controle de células sem divisão de sincronização aleatoriamente. Barra de erro = SEM (erro padrão da média). Clique aqui para ver uma versão maior desta figura.
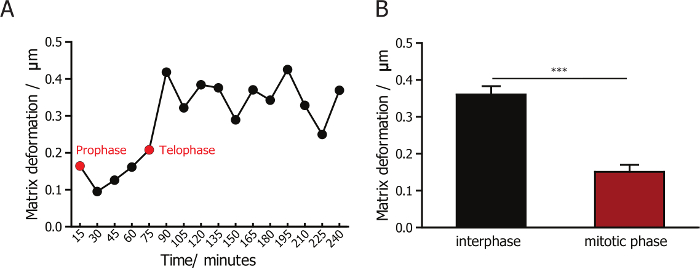
Figura 3: quantificação da deformação matriz para MDA-MB-231 células durante a interfase e mitose. (A) a mudança na magnitude da deformação de matriz para células de MDA-MB-231 matriz-incorporado. A seta verde indica a prófase e a seta vermelha indica a telófase. (B) a quantificação da deformação de matriz para células MDA-MB-231 durante a interfase e fase mitótica, indicando que a deformação de matriz é mínima durante a divisão celular. Barra de erro = SEM (erro padrão da média). * p < 0,05. Clique aqui para ver uma versão maior desta figura.
Discussão
O estudo prévio de divisão celular em 3D não era eficiente devido às limitações experimentais e desafios técnicos18,19. Os passos críticos para o estudo eficiente de divisão de células de mamíferos em matrizes de colágeno 3D são: (1) a incorporação de fluorescência-etiquetada marcadores mitóticas para as células; (2) a sincronização da divisão celular; e (3) o acompanhamento dos eventos de divisão em matrizes 3D usando a técnica de imagem live-célula, microscopia de reflexão confocal tempo-resolvido e análise quantitativa de imagem.
Células mitóticas em substratos 2D distingue as células de interfase com base na sua morfologia, ou seja, células mitóticas são redondas e mal anexar ao substrato, Considerando que a interfase células espalhem-se e unir-se firmemente ao substrato. Em matrizes 3D, no entanto, a morfologia da célula não é um marcador confiável para células mitóticas desde algumas células mal espalhadas-se e permanecer redonda na matriz27,28. Assim, é essencial apresentar um marcador mitótico das células para o estudo da divisão celular em matrizes 3D. Manifestámos estàvel H2B-mCherry nas células do MDA-MB-231, que serviu como um marcador confiável para as diferentes fases mitóticas incluindo prófase, prometafase, metáfase, anáfase e telófase/citocinese. Anteriormente, usamos esta abordagem para distinguir células mitóticas das células de interfase em matrizes 3D. Com a ajuda deste marcador, fomos também capazes de medir a duração da fase mitótica para outra linha de celular, células HT1080, dividindo-se em substratos de 2D e 3D matrizes19.
Há várias maneiras para sincronizar as células, incluindo soro fome29, mitótica agitar-se30,31, timidina dupla bloqueio32e nocodazole32. Nós combinamos o tratamento de timidina e o tratamento de nocodazole para sincronizar eficientemente MDA-MB-231 células na fase G2/M. Células expostas a timidina são presos na transição G1/S e em toda a fase S, devido a inibição da síntese de DNA pela timidina. A liberação das células a partir da exposição de timidina deixe o progresso de células para fase G2/M para células preso na fase G1/S e G1 para células preso na fase S. Todas as células expostas a nocodazole são presos na fase G2/M. As células arredondada, entrando na fase mitótica foram então abalada-fora da placa e encapsuladas diretamente em matrizes de colágeno. Nós mostramos que cerca de 70% das células dividir até 2 horas depois que eles são incorporados em matrizes de colágeno (Figura 2). Alternativamente, as células pudessem ser incorporadas em matrizes de colágeno antes da sincronização, no entanto, a rede reticulada da matriz de colágeno apresenta uma barreira física e reduz a taxa de biomolecular difusão e convecção33, 34. com efeito, nós tentou sincronizar células em matrizes de colágeno utilizando timidina e nocodazole, mas não conseguiram obter sincronização eficiente. Este resultado pode ser devido à ineficiente difusão e convecção das drogas através da matriz de colagénio.
A reflexão é uma propriedade óptica intrínseca de muitos biopolímeros, incluindo o colágeno. A técnica de microscopia confocal reflexão visualiza e dosa o microtopography de biomateriais porosos preparados a partir de polímeros sintéticos e colágeno 3D matrizes23,24,35,36 ,,37. Em nosso laboratório, nós estabelecemos técnicas para monitorar mudanças na polaridade de fibras de colágeno, como a concentração de colágeno varia de38. Aqui, descrevemos o método para monitorar a deformação das fibras de colágeno, baseado no vídeo lapso de tempo das imagens confocal reflexivos para denotar a interação célula-matriz. Os representante resultados aqui apresentados mostram que a deformação de matriz para as células mitóticas do MDA-MB-231 é significativamente menor do que as células em interfase, que sugere que células de mamíferos têm apego mínimo e interações com o matriz circundante quando eles entram a fase mitótica19.
Anteriormente, nós costumávamos microscopia confocal reflexão monitorar e quantificar a interação célula-matriz antes, durante e após a mitose das células HT1080. Nós também monitorada a deformação de matriz por β1-integrina knock-down HT1080 células durante a interfase e fase mitótica. Empobrecimento β1-integrina significativamente reduz a deformação da matriz pela célula durante a interfase. No entanto, não há nenhuma diferença na deformação da matriz durante a fase mitótica de células redonda β1-integrina nocaute (KD) e de células de tipo selvagem o HT108019.
Uma abordagem alternativa para visualizar as fibras de colágeno é empregar conjugados a fluorescência colágeno tipo I. Anteriormente, usamos esta abordagem para faixas de imagem em matrizes de colágeno, geradas por células19. Esta abordagem, no entanto, exige a rotulagem de colágeno com corante fluorescente como isotiocianato de fluoresceína (FITC), que é demorado e menos eficiente. Por outro lado, microscopia confocal reflexão pode ser aplicada diretamente a colágeno não modificado para economizar tempo e recursos e excluir as questões associadas a fluorescência fotobranqueamento. Além disso, este método não requer um canal fluorescente individual e, portanto, é compatível com todos os corantes fluorescentes.
O método apresentado neste trabalho pode potencialmente ser aplicado a qualquer tipo de células de mamíferos que dividem-se em uma matriz de colagénio 3D. A incorporação do marcador H2B-mCherry em outras células de mamíferos por transdução Lentivirus seguirá exatamente os mesmos procedimentos descritos no livro, embora diferentes tipos de células podem ter variado eficiências para transdução por lentivirus 39. tanto a densidade das células em cima de transdução e o título do vírus poderiam ser otimizadas para a eficiência. Se não pode ser alcançada a eficiência elevada de transdução, células poderiam ser selecionadas pelo celular assistida de fluorescência classificação (FACS). Bloqueio de timidina e nocodazole são aplicadas para sincronizar com êxito vários outros tipos de células de mamíferos, tais como HeLa40. Agitação mecânica-fora poderia ser aplicada a qualquer tipo de célula que reúne e mal anexar ao substrato durante a fase mitótica30,31. Além disso, a imagem das fibras de colágeno, utilizando microscopia confocal de reflexão e a quantificação da deformação matriz pode diretamente ser aplicada a todos os outros tipos da divisão de células de mamíferos.
O método apresentado aqui é uma abordagem eficiente e geral para estudar a divisão de células de mamíferos e célula-matriz interações em um ambiente 3D. A abordagem facilita nossa sonda sobre a base molecular do desenvolvimento do tecido normal e doenças e potencializa o design do livro diagnóstico e abordagens terapêuticas no futuro.
Divulgações
Os autores declaram que eles têm não tem interesses financeiro concorrente.
Agradecimentos
Este trabalho foi financiado pelo NIH concede R01CA174388 e U54CA143868. Os autores gostaria de reconhecer o prêmio PURA da Universidade Johns Hopkins para suporte de Chen Wei-tong. Este material é baseado em trabalho suportado pela nacional Science Foundation Graduate bolsa de pesquisa sob Grant no. 1232825.
Materiais
| Name | Company | Catalog Number | Comments |
| Human embryonic kidney 293T | ATCC | ||
| MDA-MB-231 | Physical Sciences Oncology Center, NIH | ||
| DMEM | Corning | 10-013-CV | |
| DMEM powder | ThermoFisher Scientific | 12100-046 | |
| Fetal bovine serum | Hyclone | SH30910.03 | |
| Penicillin-Streptomycin 100X | Sigma-Aldrich | P0781 | |
| Fugene HD | Promega | E2311 | |
| Lipofectamine 2000 | Life technologies | 11668-07 | |
| Plasmid encoding H2B-mCherry in a lentiviral vector | Addgene | plasmid 21217 | |
| Thymidine | Sigma-Aldrich | T1895 | |
| Nocodazole | Sigma-Aldrich | M1404 | |
| Opti-MEM | Life Technologies | 31985-070 | |
| Sodium bicarbonate | GibcoBRL | 11810-025 | |
| HEPES | Sigma-Aldrich | 113375-100 | |
| Collagen | Corning | 354236 | |
| NaOH | J.T. Bake | 3722-01 | |
| Millex-HV syringe filter unit, 0.45-μm, PVDF, 33 mm | Millipore | SLHVM33RS | |
| Nikon TE2000E epifluorescence microscope | Nikon | TE2000E | |
| Cascade 1K CCD camera | Roper Scientific | ||
| NIS-Elements AR imaging software | Nikon | ||
| Nikon A1 confocal microscope | Nikon | A1 |
Referências
- Ly, D. H., Lockhart, D. J., Lerner, R. A., Schultz, P. G. Mitotic misregulation and human aging. Science. 287 (5462), 2486-2492 (2000).
- Gascoigne, K. E., Taylor, S. S. How do anti-mitotic drugs kill cancer cells?. J Cell Sci. 122 (Pt 15), 2579-2585 (2009).
- Brinkley, B. R. Managing the centrosome numbers game: from chaos to stability in cancer cell division. Trends Cell Biol. 11 (1), 18-21 (2001).
- Phillip, J. M., Aifuwa, I., Walston, J., Wirtz, D. The Mechanobiology of Aging. Annu Rev Biomed Eng. 17, 113-141 (2015).
- Hanahan, D., Weinberg, R. A. Hallmarks of cancer: the next generation. Cell. 144 (5), 646-674 (2011).
- Martin, M. D., et al. Effect of ablation or inhibition of stromal matrix metalloproteinase-9 on lung metastasis in a breast cancer model is dependent on genetic background. Cancer Res. 68 (15), 6251-6259 (2008).
- Knox, J. J., Hotte, S. J., Kollmannsberger, C., Winquist, E., Fisher, B., Eisenhauer, E. A. Phase II study of Triapine in patients with metastatic renal cell carcinoma: a trial of the National Cancer Institute of Canada Clinical Trials Group (NCIC IND.161). Invest New Drugs. 25 (5), 471-477 (2007).
- Komlodi-Pasztor, E., Sackett, D. L., Fojo, A. T. Inhibitors targeting mitosis: tales of how great drugs against a promising target were brought down by a flawed rationale. Clin Cancer Res. 18 (1), 51-63 (2012).
- Tong, W. G., et al. Phase I and pharmacologic study of SNS-032, a potent and selective Cdk2, 7, and 9 inhibitor, in patients with advanced chronic lymphocytic leukemia and multiple myeloma. J Clin Oncol. 28 (18), 3015-3022 (2010).
- Matulonis, U. A., et al. Phase II study of MLN8237 (alisertib), an investigational Aurora A kinase inhibitor, in patients with platinum-resistant or -refractory epithelial ovarian, fallopian tube, or primary peritoneal carcinoma. Gynecol Oncol. 127 (1), 63-69 (2012).
- Boss, D. S., et al. Clinical evaluation of AZD1152, an i.v. inhibitor of Aurora B kinase, in patients with solid malignant tumors. Ann Oncol. 22 (2), 431-437 (2011).
- Griffith, L. G., Swartz, M. A. Capturing complex 3D tissue physiology in vitro. Nat Rev Mol Cell Biol. 7 (3), 211-224 (2006).
- Cukierman, E., Pankov, R., Stevens, D. R., Yamada, K. M. Taking cell-matrix adhesions to the third dimension. Science. 294 (5547), 1708-1712 (2001).
- Lu, P., Weaver, V. M., Werb, Z. The extracellular matrix: a dynamic niche in cancer progression. J Cell Biol. 196 (4), 395-406 (2012).
- Giri, A., et al. The Arp2/3 complex mediates multigeneration dendritic protrusions for efficient 3-dimensional cancer cell migration. FASEB J. 27 (10), 4089-4099 (2013).
- Fraley, S. I., Feng, Y., Giri, A., Longmore, G. D., Wirtz, D. Dimensional and temporal controls of three-dimensional cell migration by zyxin and binding partners. Nat Commun. 3, 719 (2012).
- Fraley, S. I., et al. A distinctive role for focal adhesion proteins in three-dimensional cell motility. Nat Cell Biol. 12 (6), 598-604 (2010).
- Lesman, A., Notbohm, J., Tirrell, D. A., Ravichandran, G. Contractile forces regulate cell division in three-dimensional environments. J Cell Biol. 205 (2), 155-162 (2014).
- He, L., et al. Local 3D matrix confinement determines division axis through cell shape. Oncotarget. 7 (6), 6994-7011 (2016).
- Held, M., et al. CellCognition: time-resolved phenotype annotation in high-throughput live cell imaging. Nat Methods. 7 (9), 747-754 (2010).
- Fallica, B., Maffei, J. S., Makin, G., Zaman, M. Alteration of cellular behavior and response to PI3K pathway inhibition by culture in 3D collagen gels. PLoS One. 7 (10), e48024 (2012).
- Meli, L., Jordan, E. T., Clark, D. S., Linhardt, R. J., Dordick, J. S. Influence of a three-dimensional, microarray environment on human Cell culture in drug screening systems. Biomaterials. 33 (35), 9087-9096 (2012).
- Artym, V. V., Matsumoto, K. Imaging cells in three-dimensional collagen matrix. Curr Protoc Cell Biol. 10, 1-20 (2010).
- Gunzer, M., Kampgen, E., Brocker, E. B., Zanker, K. S., Friedl, P. Migration of dendritic cells in 3D-collagen lattices. Visualisation of dynamic interactions with the substratum and the distribution of surface structures via a novel confocal reflection imaging technique. Adv Exp Med Biol. 417, 97-103 (1997).
- Geraldo, S., Simon, A., Vignjevic, D. M. Revealing the cytoskeletal organization of invasive cancer cells in 3D. J Vis Exp. (80), e50763 (2013).
- Lleres, D., James, J., Swift, S., Norman, D. G., Lamond, A. I. Quantitative analysis of chromatin compaction in living cells using FLIM-FRET. J Cell Biol. 187 (4), 481-496 (2009).
- Poincloux, R., et al. Contractility of the cell rear drives invasion of breast tumor cells in 3D Matrigel. Proc Natl Acad Sci U S A. 108 (5), 1943-1948 (2011).
- Carey, S. P., Kraning-Rush, C. M., Williams, R. M., Reinhart-King, C. A. Biophysical control of invasive tumor cell behavior by extracellular matrix microarchitecture. Biomaterials. 33 (16), 4157-4165 (2012).
- Langan, T. J., Chou, R. C. Synchronization of mammalian cell cultures by serum deprivation. Methods Mol Biol. 761, 75-83 (2011).
- Jackman, J., O'Connor, P. M. Methods for synchronizing cells at specific stages of the cell cycle. Curr Protoc Cell Biol. 8, (2001).
- Zwanenburg, T. S. Standardized shake-off to synchronize cultured CHO cells. Mutat Res. 120 (2-3), 151-159 (1983).
- Harper, J. V. Synchronization of cell populations in G1/S and G2/M phases of the cell cycle. Methods Mol Biol. 296, 157-166 (2005).
- Kihara, T., Ito, J., Miyake, J. Measurement of biomolecular diffusion in extracellular matrix condensed by fibroblasts using fluorescence correlation spectroscopy. PLoS One. 8 (11), e82382 (2013).
- Ramanujan, S., et al. Diffusion and convection in collagen gels: implications for transport in the tumor interstitium. Biophys J. 83 (3), 1650-1660 (2002).
- Harjanto, D., Maffei, J. S., Zaman, M. H. Quantitative analysis of the effect of cancer invasiveness and collagen concentration on 3D matrix remodeling. PLoS One. 6 (9), e24891 (2011).
- Wolf, K., et al. Collagen-based cell migration models in vitro and in vivo. Semin Cell Dev Biol. 20 (8), 931-941 (2009).
- Petroll, W. M. Differential interference contrast and confocal reflectance imaging of collagen organization in three-dimensional matrices. Scanning. 28 (6), 305-310 (2006).
- Fraley, S. I., et al. Three-dimensional matrix fiber alignment modulates cell migration and MT1-MMP utility by spatially and temporally directing protrusions. Sci Rep. 5, 14580 (2015).
- Ikeda, Y., Collins, M. K., Radcliffe, P. A., Mitrophanous, K. A., Takeuchi, Y. Gene transduction efficiency in cells of different species by HIV and EIAV vectors. Gene Ther. 9 (14), 932-938 (2002).
- Ma, H. T., Poon, R. Y. Synchronization of HeLa cells. Methods Mol Biol. 761, 151-161 (2011).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados