Method Article
Division cellulaire chez les mammifères dans des Matrices 3D par microscopie Quantitative réflexion Confocal
* Ces auteurs ont contribué à parts égales
Dans cet article
Résumé
Ce protocole a efficacement étudie la division cellulaire chez les mammifères dans les matrices de collagène 3D en intégrant la synchronisation de la division cellulaire, la surveillance des manifestations de la division dans des matrices 3D à l’aide de la technique d’imagerie de cellules vivantes, microscopie de réflexion confocal résolution temporelle et analyse d’imagerie quantitative.
Résumé
L’étude de la division cellulaire chez les mammifères comment est réglementée dans une 3D environnement demeure largement inexploré malgré sa pertinence physiologique et sa signification thérapeutique. Les raisons possibles de l’absence d’exploration sont les limitations expérimentales et les défis techniques qui rendent l’étude de la division cellulaire en culture 3D inefficace. Nous décrivons ici une méthode axée sur l’imagerie pour étudier efficacement la division cellulaire chez les mammifères et les interactions cellule-matrice dans des matrices 3D de collagène. Cellules marquées avec la H2B fluorescents sont synchronisées à l’aide de la combinaison du traitement de blocage et la nocodazole thymidine, suivie d’une technique mécanique de shake-off. Cellules synchrones sont ensuite incorporées dans une matrice de collagène 3D. La division cellulaire est contrôlée au moyen de la microscopie de cellules vivantes. La déformation des fibres de collagène pendant et après la division cellulaire, qui est un indicateur de l’interaction cellule-matrice, peut être surveillée et quantifiées à l’aide de la microscopie quantitative réflexion confocale. La méthode fournit une approche efficace et générale pour étudier la division cellulaire chez les mammifères et les interactions cellule-matrice dans un environnement 3D physiologiquement pertinent. Cette approche non seulement fournit de nouveaux aperçus de la base moléculaire du développement des tissus normaux et les maladies, mais permet également à la conception de nouvelles approches diagnostiques et thérapeutiques.
Introduction
Mitose de la cellule est un événement critique dans la vie cellulaire, dont la réglementation joue un rôle crucial dans le développement de tissus et organes. Une mitose anormale est impliquée dans des variations génétiques naturelles, les processus de vieillissement humain et la progression du cancer1,2,3,4,5. L’augmentation du taux de prolifération des cellules tumorales par rapport aux cellules normales est une des caractéristiques du cancer, malgré le fait que les comportements cellulaires sont assez hétérogènes entre les différents types de tumeurs et même chez les patients. En dépit des résultats précliniques prometteurs, certaines drogues antimitotiques nouvellement développé n’ont pas démontré d’être efficaces dans les essais cliniques6,7,8,9,10 ,11. La pertinence des modèles expérimentaux et précliniques doit être considérée. Plusieurs types de cellules normales de mammifères et cancer divisent dans des matrices en trois dimensions (3D), tels que les fibroblastes et cellules de Fibrosarcome en collagène riche j’ai 3D des tissus conjonctifs et cellules cancéreuses métastatiques dans la 3D stromales la matrice extracellulaire (MEC). Cependant, la grande majorité de la division cellulaire chez les mammifères des expériences et des essais ont été réalisée sur les cellules cultivées sur des substrats à deux dimensions (2D). Une matrice 3D ingénierie pourrait mieux récapituler la microstructure et propriétés mécaniques des signaux biochimiques de l’ECM 3D de deux tissus normaux et pathologiques12,13,14, 15,16,17.
L’étude des mammifère comment la division cellulaire est régulée en 3D des environnements demeure largement inexploré malgré la pertinence physiologique tant la signification thérapeutique18,19. Les raisons possibles incluent les difficultés techniques et expérimentales défis associés à étudier la division cellulaire dans des matrices 3D. La mitose cellulaire constitue une petite fraction temporelle dans le cycle cellulaire complet20. Travaux antérieurs ont montré que le taux de prolifération de plusieurs cellules de mammifères, tels que l’adénocarcinome du sein MCF-7, ostéosarcome humain U2OS et foie humain HepG2, est beaucoup plus faible dans des matrices 3D par rapport à leurs homologues sur des substrats 2D21, 22. En outre, les cellules incorporés dans des matrices 3D se déplacent dans et hors de discussion au cours de l’imagerie de cellules vivantes. Tous ces facteurs contribuent à la très faible efficacité de capture d’événements de la division cellulaire en 3D à l’aide de techniques d’imagerie de la culture.
Interactions entre les cellules et ECM jouent un rôle crucial dans la régulation des divisions cellulaires. Nous décrivons ici une approche pour étudier efficacement la division cellulaire chez les mammifères dans des matrices 3D de collagène. La méthode comprend l’incorporation des marqueurs mitotiques pour les cellules, la synchronisation de la division cellulaire, ainsi que le suivi des événements de la division dans des matrices 3D à l’aide de la technique d’imagerie de cellules vivantes, de la microscopie de réflexion confocal résolution temporelle, et analyse d’imagerie quantitative. Protéine de fluorescence-étiquetée histone H2B est tout d’abord introduit dans les cellules comme marqueurs pour différencier mitose et interphase de cellules. Puis les cellules sont synchronisées à l’aide de la combinaison du traitement de blocage et la nocodazole thymidine, suivie d’une technique mécanique de shake-off. Cellules synchrones sont alors directement encapsulées dans des matrices 3D de collagène. Événements de la division cellulaire des cellules multiples sont surveillées efficacement à l’aide de l’imagerie de cellules vivantes Time-lapse faible grossissement. La déformation des fibres de collagène, qui est un indicateur de l’interaction cellule-matrice, est contrôlée au moyen de la microscopie confocale réflexion à fort grossissement.
Nous avons déjà utilisé cette technique pour surveiller et de quantifier l’interaction cellule-matrice avant, pendant et après la mitose de deux lignées de cellules de cancer métastatique, carcinome canalaire invasif humain MDA-MB-231 et les cellules humaines Fibrosarcome HT1080 en collagène 3D 19de matrices. Les méthodes présentées ici fournissent une approche efficace et générale pour étudier les deux mammifères division cellulaire dans une interaction environnement et cellule-matrice 3D. La lignée de cellules MDA-MB-231 est utilisée à titre d’exemple dans le présent document. Ce protocole prévoit de nouveaux aperçus de la base moléculaire du développement des tissus normaux et les maladies et pourrait également permettre la conception de nouvelles approches diagnostiques et thérapeutiques.
Protocole
Le protocole fourni respecte les directives de la Homewood institutionnels Review Board (HIRB).
1. stable Expression de H2B-mCherry comme un marqueur de cellule mitose
-
Génération de Particules Lentivirales rein embryonnaires humaines 293 t (HEK 293 t) cellules
- Les cellules HEK 293 t sur un plat de culture cellulaire de 10 cm à la densité de 5 x 106 cellules/plat au milieu de culture cellulaire (de Dulbecco Medium (DMEM) contenant hyperglycémie Modified Eagle (4,5 g/L), pyruvate de sodium, 10 % sérum bovin fœtal (SVF) et 1 % de la plaque pénicilline-streptomycine (Pen/Strep)). Incuber 24 h à 37 ° C et 5 % de dioxyde de carbone (CO2). La confluence souhaitée des cellules sur le jour de la transfection est environ 70 à 80 %.
Remarque : Des cellules HEK 293 t sont utilisés ici pour produire des particules virales, qui seront utilisés plus tard pour transmettre les cellules MDA-MB-231 afin de produire une lignée de cellules exprimant de manière stable H2B-mCherry. Assurez-vous que les cellules sont répartis uniformément sur la plaque. Ajout des cellules drop sage de la plaque, et déplace doucement la plaque en arrière peut aider à la distribution uniforme. Les cellules peuvent également être ensemencés en assiettes ou de flacons d’autres tailles, qui seront traduira par la collection de volumes différents des particules virales. - Transfecter les cellules HEK 293 t en utilisant un réactif de transfection avec trois plasmides (vecteur lentiviral, CMV ΔR 8,91 et pMDG-VSVG). Le plasmide codant H2B-mCherry est cloné dans un vecteur lentiviral avec promoteur de phosphoglycérate kinase (PGK). CMV ΔR 8,91 contient trois requis VIH gènes gag, polet rev. pMDG-VSVG contient le gène d’enveloppe de VSV-G.
Remarque : Il existe plusieurs agents de transfection à choisir.- Laisser le flacon de réactif de transfection à température ambiante (RT) avant utilisation. Inverti ou vortex le flacon brièvement.
- Mix 16 µg d’ADN, qui se compose de 6 µg de plasmide H2B-mCherry et 8 µg de CMV ΔR 8,91 2 µg de pMDG-VSVG, dans 1 mL de médias de sérum réduit. Incuber 5 min à RT.
Remarque : La quantité et le rapport des trois vecteurs est variée et optimisé pour les vecteurs de transfert des gènes différents. - Ajouter 48 µL (utiliser le rapport 1:3 d’ADN : réactif de transfection) du réactif de transfection dans la solution ci-dessus de l’ADN et puis incuber à RT pendant au moins 15 minutes.
NOTE : Le rapport entre l’ADN vs réactif de transfection est variée et optimisé pour les vecteurs de transfert des gènes différents. Assurez-vous que le réactif de transfection ne contacte pas le côté du tube de centrifugation de 1,5 mL. - Ajouter la solution complexe ADN-lipides effectuée à l’étape 1.1.2.3 goutte-à-goutte aux cellules. Agiter doucement de la plaque afin d’assurer la distribution uniforme de l’ensemble de la plaque.
- Environ 6 h après la transfection, aspirer le milieu et ajouter le milieu de culture de cellules fraîches (DMEM avec hyperglycémie (4,5 g/L), pyruvate de sodium, 10 % de SVF et 1 % Pen/infection streptococcique).
- Récolter le surnageant 24, 48 et transfection après 72 h. Remplacer le support après chaque récolte (DMEM, 4,5 g/L de glucose, de pyruvate de sodium, avec 10 % de SVF et 1 % Pen/infection streptococcique).
- Les particules Lentivirales filtrer sur un filtre de 0,45 µm pour éliminer les débris cellulaires. Sinon, tournez en bas le surnageant pour séparer les débris cellulaires. Le surnageant peut être utilisé pour la transduction tout de suite, ou peut être stocké à-80 ° C.
- Les cellules HEK 293 t sur un plat de culture cellulaire de 10 cm à la densité de 5 x 106 cellules/plat au milieu de culture cellulaire (de Dulbecco Medium (DMEM) contenant hyperglycémie Modified Eagle (4,5 g/L), pyruvate de sodium, 10 % sérum bovin fœtal (SVF) et 1 % de la plaque pénicilline-streptomycine (Pen/Strep)). Incuber 24 h à 37 ° C et 5 % de dioxyde de carbone (CO2). La confluence souhaitée des cellules sur le jour de la transfection est environ 70 à 80 %.
-
Génération de cellule lignes exprimant stablement H2B-mCherry
Remarque : Ce protocole est décrit en détail pour les cellules MDA-MB-231. Avant de l’utiliser dans l’expérience, les cellules de carcinome mammaire humain MDA-MB-231 (Centre d’oncologie Sciences physique, NIH) sont cultivées en DMEM contenant des taux de glycémie élevé (4,5 g/L), pyruvate de sodium, 10 % SVF et 1 % Pen/Strep.- Les cellules MDA-MB-231 à la densité des cellules5 1 x 10 dans une boîte de Petri de 35 mm de la plaque.
Remarque : La densité de placage de différentes lignées cellulaires doit être optimisée et par conséquent peut être différente de la densité optimale pour les cellules MDA-MB-231. - 24 h après l’ensemencement les cellules, ajouter 1 mL de virus et 1 mL de milieu frais (DMEM, hyperglycémie (4,5 g/L), pyruvate de sodium, 10 % de SVF et 1 % Pen/infection streptococcique).
- Incuber les cellules pendant un laps de temps de 6 h à la nuit.
NOTE : Fois le volume du virus et le temps d’incubation doivent être optimisés pour différentes lignées cellulaires. Par exemple, si les cellules ne regardent pas en bonne santé quelques heures après l’ajout du virus, utiliser 1 mL du virus et 2 mL de milieu frais (DMEM, hyperglycémie (4,5 g/L), pyruvate de sodium, 10 % de SVF et 1 % Pen/Strep) pour l’étape 1.2.2. La durée de l’incubation à l’étape 1.2.3 peut également être réduite. - Aspirer le milieu et ajouter 10 mL de milieu frais (DMEM, hyperglycémie (4,5 g/L), pyruvate de sodium, 10 % de SVF et 1 % Pen/infection streptococcique). Incuber pendant 24 à 72 h à 37 ° C et 5 % de CO2.
- Contrôler l’expression de H2B-mCherry dans les cellules à l’aide d’un microscope à épifluorescence.
Remarque : Si l’efficacité de la transduction est faible dans les cellules, le volume du virus à l’étape 2.2 et le temps d’incubation à l’étape 2.3 pourrait être augmenté.
- Les cellules MDA-MB-231 à la densité des cellules5 1 x 10 dans une boîte de Petri de 35 mm de la plaque.
2. synchronisation des cellules exprimant de manière stable H2B-mCherry
- Plaque de cellules des cellules à 50 à 60 % confluency, c'est-à-dire la plaque de 2 x 104 MDA-MB-231 dans chaque puits d’une plaque 24 puits.
- Incuber la culture à 37 ° C et 5 % de CO2 pendant 24 h.
- Remplacer le milieu de culture avec 0,5 mL de milieu (DMEM, hyperglycémie (4,5 g/L), pyruvate de sodium, 10 % de SVF et 1 % Pen/Strep) contenant la thymidine 2 mM et laisser dans l’incubateur pendant 24 h.
Remarque : Les cellules exposées à la thymidine sont arrêtés lors de la phase de croissance cellulaire (G1) / transition (S) de la synthèse de l’ADN et tout au long de la phase S en raison de l’inhibition de la synthèse de l’ADN. La durée de l’incubation doit être variée et optimisée pour différentes lignées cellulaires. - Libérer les cellules de l’exposition de la thymidine en les lavant avec du sérum physiologique tamponné au phosphate (PBS) trois fois. Puis, incuber les cellules dans un milieu de culture cellulaire normale (DMEM, hyperglycémie (4,5 g/L), pyruvate de sodium, 10 % de SVF et 1 % Pen/Strep) pendant 5 h.
Remarque : La libération des cellules par l’exposition de thymidine permet aux cellules de progresser à la croissance cellulaire (G2) / phase mitotique de (M) pour les cellules précédemment arrêté lors de la phase G1/S et la phase G1 pour les cellules déjà arrêté à la S phase. La durée du congé doit être variée et optimisée pour différentes lignées cellulaires. - Bloquer les cellules avec 250 ng/mL de la nocodazole pendant 12 h.
Remarque : Toutes les cellules exposées à la nocodazole sont arrêtés lors de la phase G2/M. Nocodazole est cytotoxique. Une exposition prolongée à la nocodazole peut provoquer l’apoptose. Ajuster la période ou la concentration d’exposition pour les différentes lignées cellulaires si décès de cellule sont observées. Les cellules qui sont correctement synchronisées exposera une morphologie sphérique. - Secouer les cellules pour 45 s à 1 min à l’aide d’un agitateur orbital à 150 à 200 tr/min.
Remarque : Les cellules mitotiques, qui ont peu d’adhérence au substrat, seront ébranlées au cours du processus. - Retirez le support pour extraire des cellules, en pipettant également, le milieu dans un tube à centrifuger et puis ajouter 0,5 mL de milieu frais (DMEM, hyperglycémie (4,5 g/L), pyruvate de sodium, 10 % de SVF et 1 % Pen/Strep) dans chaque puits de la plaque.
- Répétez les étapes 2.6 et 2.7 trois fois.
- Centrifuger le milieu collecté contenant les cellules mitotiques à 800 g pendant 3 min.
Remarque : Cette étape sert à retirer la nocodazole le milieu cellulaire.
3. l’incorporation de la synchronisation des cellules dans le collagène j’ai Matrices
NOTE : J’ai du Type collagène est la protéine la plus abondante dans le corps humain et dans l’ECM des tissus conjonctifs et ainsi est employé couramment pour enquêter sur des cellules eucaryotes comment fonctions sont modulées par un environnement 3D17,23,24 . Le collagène est soluble dans l’acide acétique. Après neutralisation et la solution de collagène à 20-37 ° C le réchauffement, les monomères de collagène se polymérisent dans un réseau de fibrilles de collagène.
- Préparer la solution de x DMEM 10 en dissolvant un paquet de poudre DMEM, 3,7 g de bicarbonate de sodium (NaHCO3) et 1 g d’acide-(2-hydroxyethyl)-1-piperazineethanesulfonic 4 (HEPES) dans 50 mL d’eau distillée. Filtrer la solution et puis préparer 1 M d’hydroxyde de sodium (NaOH) dissoudre 2 g de pastilles de NaOH dans 50 mL d’eau distillée. Filtre et partie aliquote de la solution dans les tubes à centrifuger 1,5 mL.
NOTE : Solution DMEM Normal ne doit pas être utilisée dans cette étape. L’ajout d’un volume significatif de la solution de collagène va diluer le milieu. La solution concentrée de DMEM est donc disposée à faire en sorte que la concentration finale de DMEM dans la matrice de collagène sera le même que le DMEM normal. - Continuer à travailler avec les cellules recueillies à l’étape 2,9. Aspirez le milieu et remettre en suspension les cellules à environ 0,25 - 0,5 mL de milieu de culture de cellules fraîches (DMEM, hyperglycémie (4,5 g/L), pyruvate de sodium, 10 % de SVF et 1 % Pen/infection streptococcique).
Remarque : Pour atteindre une densité cellulaire spécifique dans la matrice de collagène, la densité initiale des cellules de la suspension ne peut pas être trop basses. Ainsi, le volume du milieu utilisé pour remettre en suspension les cellules dépendra du nombre total de cellules disponibles. - Déposer 10 µL de la solution de remises en suspension cellulaire de l’étape 3.2 sur un hémocytomètre et compte la densité des cellules dans la solution.
-
Déterminer les volumes nécessaires pour tous les composants réaliser la matrice de collagène 3D. 500 µL de 2 gel de collagène µg/µL est utilisé ici comme un exemple.
- Calculer le volume de la solution de cellules nécessaire pour obtenir 40 000 cellules/mL (ou 20 000 cellules pour la 500 µL de gel de collagène). Ce numéro µL est X. 50 µL de 10 x DMEM est nécessaires. 50 µL de FBS sera nécessaire.
- Calculer le volume du collagène que je stock nécessaire. La concentration de collagène I est autour de 4 µg/µL. La quantité de collagène nécessaire en µL est Y.
- Calculer le volume d’hydroxyde de sodium nécessaire neutraliser l’acide acétique dans la solution de collagène : collagène µL Y * 0,023 * 1000 = Z µL NaOH nécessaire.
- Déterminer le volume de la solution de remplissage : (500 µL gel totale - cellules X µL - 50 µL 10 X collagène DMEM - 50 µL de FBS - Y µL - Z µL NaOH) = R µL
Remarque : La solution de remplissage est distillée à l’eau. Si la valeur de la solution de remplissage est un nombre négatif, la densité cellulaire original dans la suspension cellulaire est trop faible. Les cellules ont besoin d’être tourné vers le bas encore une fois et remis en suspension dans un plus petit volume de milieu (DMEM, hyperglycémie (4,5 g/L), pyruvate de sodium, 10 % de SVF et 1 % Pen/infection streptococcique).
- Ajouter les composants dans l’ordre suivant dans un tube à centrifuger propre 1,5 mL : cellules dans un milieu, 10 x DMEM, FBS, l’eau désionisée (DI), collagène, NaOH. Placez tous les composants sur la glace pour ralentir la gélification de la solution de collagène. Utiliser les techniques appropriées de pipetage pour éviter la formation de bulles.
- Après avoir ajouté le NaOH, mélanger la solution soigneusement avec une pipette de 1 mL.
NOTE : Gélification du collagène va commencer droite immédiatement après l’addition de NaOH. Mélange doit être faite avec soin et rapidement. - Une fois que la solution est bien mélangée, ajouter 500 µL de la solution dans chaque puits de la plaque 24 puits. Placer la plaque 24 puits dans un incubateur à 37 ° C. Placez le milieu de culture de cellules fraîches dans le bain-marie à 37 ° C.
Remarque : Les plaques de fond en plastique sont utilisés pour microcopy de cellules vivantes de faible grossissement dans lequel le travail à distance de la lentille est dessus verre fond mm 1. plaques sont utilisées pour la microscopie de cellules vivantes à fort grossissement en raison de la courte distance de travail de la haute lentille de grossissement. - Ajouter 500 µL de milieu préchauffé (DMEM, hyperglycémie (4,5 g/L), pyruvate de sodium, 10 % de SVF et 1 % Pen/Strep) vers le haut du gel 30 min après la fin de l’étape 3,7.
4. vivre l’imagerie cellulaire des cellules divisant dans la matrice de collagène 3D (faible grossissement)
NOTE : Images de cellules sont recueillies à des intervalles de 2 min en utilisant une caméra de dispositif couplé (DAC) frais montée sur un microscope à contraste de phase qui est équipé d’un objectif 10 X et contrôlé par logiciel d’imagerie.
- Allumez le groupe de cellules vivantes monté sur le dessus de l’objectif. Attendez que la température se stabilise à 37 ° C, la concentration de CO2 est à 5 % et le taux d’humidité est à 75 %.
- Mettre la plaque 24 puits de gels dans l’unité de cellules vivantes sur le microscope. Trouver le fond de la plaque, puis déplacez la lentille de l’objectif jusqu'à ce que le microscope se concentre à environ 500 µm de la partie inférieure de la plaque.
Remarque : Les cellules dans la matrice de collagène qui sont trop près de la partie inférieure de la plaque ne sont pas entièrement noyés dans la matrice 3D. Imagerie de cellules qui sont 500 µm loin de la partie inférieure de la plaque fera en sorte que les cellules ne sont pas affectés par les effets de bord16,17,25. - Déplacer la scène autour pour trouver les cellules, puis sélectionnez plusieurs postes pour l’imagerie.
- Mettre en place l’expérience Time-lapse à intervalles de 2 min.
Remarque : Le nombre maximal de postes qui peuvent être prises pendant l’intervalle de 2 min est limité par la vitesse de déplacement de la platine motorisée et la durée de l’exposition pour capturer des images.
5. collagène réseau déformation pendant la Division cellulaire (microscopie fort grossissement)
- Allumez le groupe de cellules vivantes monté sur le dessus de l’objectif. Attendez que la température se stabilise à 37 ° C, la concentration de CO2 est à 5 % et le taux d’humidité est à 75 %.
- Afin de visualiser les fibres de collagène dans les matrices de collagène 3D colorées ou non, configurer un microscope confocal pour capturer seulement lumière réfléchie (488 nm) du 488 nm laser servant à éclairer l’échantillon. Le laser utilise un 60 X objectif immersion dans l’eau, NA = 1.2, WD = 200 µm et est contrôlé par le logiciel d’imagerie.
- Mettre l’échantillon (cellules de matrices de collagène) dans l’unité de cellules vivantes. Trouver le fond de la plaque, puis déplacez la lentille de l’objectif jusqu'à ce que le microscope se concentre à environ 100 µm de la partie inférieure de la plaque. Imagerie de cellules qui sont à 100 µm de la partie inférieure de la plaque fera en sorte que les cellules ne sont pas affectés par les effets de bord.
NOTE : Ici un objectif à immersion dans l’eau est utilisé au lieu d’un objectif à immersion dans l’huile parce que la distance de travail de la lentille immersion dans l’huile est seulement environ 100 µm. L’utilisation de l’objectif à immersion dans l’huile pose un problème car l’épaisseur du verre sur la partie inférieure de la plaque est normalement d’environ 100 µm. - Déplacer la scène autour pour trouver les cellules, puis sélectionnez plusieurs postes pour l’imagerie.
Remarque : La microscopie confocale sections optiquement l’échantillon et seulement capte les signaux du plan focal. Des cellules vivantes peuvent se déplacer dans et hors de discussion pendant l’expérience de Time-lapse. Pour activer la capture d’images de cellules sur de longues périodes de temps, Z-pile est utilisée pour recueillir des images des tranches successives à intervalle de 5 µm. Un total de 5 tranches s’étendant sur 25 µm sont imagés. - Mettre en place l’expérience Time-lapse à intervalles de 5 min.
Remarque : 5 minutes est utilisé ici comme l’intervalle au lieu de 2 min, utilisé à l’article 4, car plusieurs modes de balayage, y compris fluorescence recherchant les H2B-mCherry, de réflexion à balayage pour le collagène et Z-pile numérisation, sont appliqués à chaque position. L’utilisation de plusieurs modes de balayage réduit la vitesse de balayage d’une position par rapport à l’imagerie de faible grossissement. - Quantifier la déformation de la matrice de collagène dans le voisinage une cellule en raison des forces exercées par cette cellule à l’aide de particle imaging velocimetry (PIV) avec résolution de sous-pixel20. Cette analyse sur des images 2D permet la visualisation claire des deux signaux de la H2B-mCherry et les fibres de collagène. Appliquer le passe-bas anisotrope de filtrage afin d’améliorer le signal du réseau collagène20.
- Afin de mesurer le déplacement local d’une région de sous-image d’intérêt situé à (x,y) du châssis k pour k+ 1 du cadre, extraire une fenêtre régionale de 15 × 15 pixels centrée à (x,y) à armature k. Ensuite, identifiez le meilleur appariement des lieux (x,y) * dans les endroits qui présentent un coefficient de corrélation croisée normalisé maximal dans l’image obtenue à armature k+ 1. Le vecteur de la déformation est calculé sous la forme (x,y) *-(x,y).
Résultats
L’objectif de cet article est de présenter une méthode axée sur l’imagerie pour étudier les processus de la division cellulaire chez les mammifères dans des matrices 3D et de quantifier les interactions entre la cellule et la matrice extracellulaire 3D pendant et après la division cellulaire. Afin de faciliter l’imagerie de la mitose de la cellule, nous avons incorporé H2B-mCherry dans les cellules MDA-MB-231 à l’aide de transduction des gènes. H2B conjugué avec protéines fluorescentes est utilisé comme marqueur mitotique de distinguer les cellules mitotiques de cellules en interphase et de définir les différentes étapes au cours de la mitose de cellules19,20,26. En utilisant cette méthode, nous avons été en mesure de suivre le processus d’ensemble de la division des cellules MDA-MB-231, exprimant de manière stable H2B-mCherry dans les matrices de collagène 3D (Figure 1 a). Phases de la mitose a commencé avec la dissolution de la membrane nucléaire (prophase, cadre 1, Figure 1 a) ; la réorganisation des chromosomes (prométaphase : structure 2) ; l’alignement des chromosomes dans le milieu du corps cellulaire (métaphase cadre 3) ; la séparation des chromosomes (anaphase cadre 4) ; la réorganisation des chromosomes et membrane nucléaire et la séparation des corps des deux cellules filles (télophase/cytocinèse, image 5).
Le taux de prolifération des cellules dans une matrice 3D est généralement beaucoup plus faible que leurs homologues sur un support 2D qui restitue le contrôle de la division cellulaire en 3D moins efficace19. Pour augmenter l’efficacité de l’étude de la division cellulaire 3D, nous avons utilisé une méthode combinant thymidine, la nocodazole et une technique de shake-off à synchroniser, puis sélectionnez les cellules MDA-MB-231 qui sont à la phase mitotique. Cellules synchrones ont été ensuite incorporées dans les matrices de collagène. Le processus de division des cellules multiples a été suivi en temps réel en utilisant imagerie de cellules vivantes à un grossissement de 10 X. Environ 70 % des cellules divisée au cours des 2 premières heures après la formation de la matrice de collagène (Figure 2), permettant ainsi un suivi efficace de la mitose en 3D (Figure 2).
Pour surveiller l’interaction entre les cellules et leurs matrices de collagène avoisinantes, nous avons combiné la microscopie confocale réflexion aux fibres de collagène d’image et microscopie de fluorescence à l’image des cellules. Une lentille X 60 permet la capture d’images de haute qualité des fibres de collagène. Imagerie en utilisant un objectif à fort grossissement, qui capte moins de cellules dans chaque champ de vision, est beaucoup plus faible débit comparé à l’utilisation de faible grossissement 10 X ou 20 X. Synchronisation des cellules augmente considérablement l’efficacité et le rendement d’une telle expérience, puisque la plupart des cellules synchronisées divise au cours des 2 premières heures après la formation de la matrice de collagène. La déformation de la matrice pendant et après la division cellulaire est visualisée (représentant instantanés sont indiquées dans la Figure 1 b) et quantifiées à l’aide d’un logiciel personnalisé de PIV. Nous avons quantifié et contre la déformation de la matrice pendant l’interphase et mitose phase pour synchronisé dans la phase mitotique des cellules. Nous avons observé que les déformations de la matrice a très peu changé au cours de la mitose de phase (Figure 3 a), et est inférieure à la déformation observée dans les phases post-mitotiques (Figure 3 b). Ce résultat montre que les cellules de mammifères ont attachement minimal et les interactions avec la matrice environnante alors qu’ils sont dans la phase mitotique.
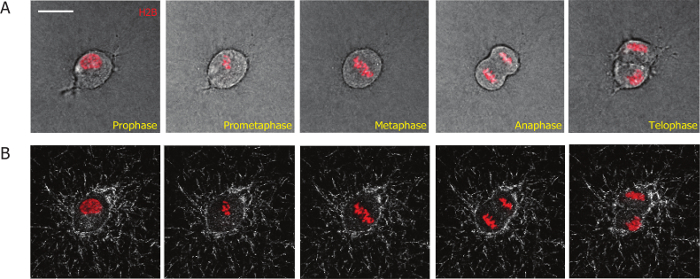
Figure 1 : micrographies représentatives obtenues à partir d’une vidéo d’imagerie de cellules vivantes à fort grossissement d’une cellule de MDA-MB-231 noyées dans une matrice de collagène qui exprime stablement H2B-mCherry. (A) différentes phases de la progression mitotique de la cellule de MDA-MB-231 sont définies par H2B-mCherry comme en rouge. (B) les fibres de collagène (blancs) pendant le processus de mitose sont visualisées par microscopie de réflexion confocal dépendant du temps. Echelle = 20 µm. s’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.

Figure 2 : Division des cellules synchronisées dans des matrices de collagène 3D. Les cellules ont été synchronisées à la phase G2/M et noyées dans une matrice de collagène. Environ 70 % des cellules synchrones diviser au cours des 2 premières heures, alors que le contrôle des cellules sans fracture de synchronisation au hasard. Barre d’erreur = SEM (erreur standard de la moyenne). S’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.
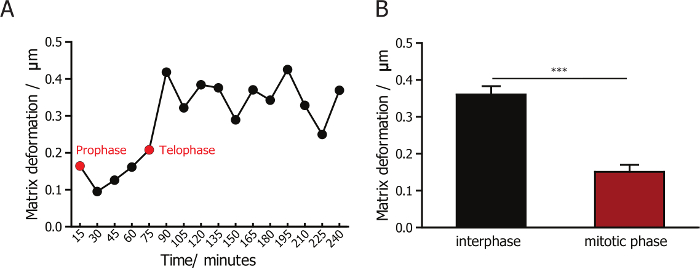
Figure 3 : Quantification de la déformation de la matrice pour MDA-MB-231 cellules en interphase et mitose. (A) les variations d’intensité de la déformation de la matrice pour matrice incorporé les cellules MDA-MB-231. La flèche verte indique la prophase et la flèche rouge indique la télophase. (B) Quantification de la déformation de la matrice pour les cellules MDA-MB-231 pendant l’interphase et de la phase de la mitose, ce qui indique que la déformation de la matrice est minime pendant la division cellulaire. Barre d’erreur = SEM (erreur standard de la moyenne). * p < 0,05. S’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.
Discussion
L’étude précédente de la division cellulaire en 3D n’était pas efficace en raison de limitations expérimentales et défis techniques18,19. Les étapes essentielles pour une étude efficace de la division cellulaire chez les mammifères dans les matrices de collagène 3D sont : (1) l’incorporation de marqueurs mitotiques marqués par fluorescence aux cellules ; (2) la synchronisation de la division cellulaire ; et (3) le suivi des événements de la division dans des matrices 3D à l’aide de la technique d’imagerie de cellules vivantes, réflexion confocal résolution temporelle microscopie et analyse d’imagerie quantitative.
Les cellules mitotiques sur substrats 2D peuvent être distingués des cellules interphasiques basés sur leur morphologie, c'est-à-dire des cellules mitotiques sont ronds et à peine joindre au substrat, tandis que les cellules interphasiques étalent et fixer fermement sur le substrat. Dans des matrices 3D, cependant, la morphologie cellulaire n’est pas un marqueur fiable pour cellules mitotiques depuis certaines cellules à peine étalés et restent dans la matrice27,28. Ainsi, il est essentiel de présenter un marqueur mitotique des cellules pour l’étude de la division cellulaire dans des matrices 3D. Nous avons exprimé stablement H2B-mCherry dans les cellules MDA-MB-231, qui a servi comme un marqueur fiable pour différentes phases mitotiques, y compris la prométaphase, la prophase, la métaphase, l’anaphase et télophase/cytokinèse. Précédemment, nous avons utilisé cette approche pour distinguer les cellules mitotiques de cellules en interphase dans des matrices 3D. Avec l’aide de ce marqueur, nous pouvions également mesurer la longueur de la phase mitotique pour une autre lignée cellulaire, cellules HT1080, divisant sur substrats 2D et 3D de matrices19.
Il existe plusieurs façons pour synchroniser les cellules, y compris le sérum famine29, mitotique shake-off30,31, thymidine double blocage32et32de la nocodazole. Nous avons combiné le traitement de la thymidine et le traitement de la nocodazole pour synchroniser efficacement les cellules MDA-MB-231 de la phase G2/M. Les cellules exposées à la thymidine sont arrêtés lors de la transition G1/S et tout au long de la phase S en raison de l’inhibition de la synthèse de l’ADN par la thymidine. La libération des cellules par l’exposition de thymidine permettre la progression de cellules de phase G2/M pour cellules arrêté à phase G1/S et de G1 pour les cellules, arrêté à la phase S. Toutes les cellules exposées à la nocodazole sont arrêtés lors de la phase G2/M. Les cellules arrondie, ouverture de la phase mitotique étaient alors secoué d’envoi de la plaque et directement encapsulées dans les matrices de collagène. Nous avons montré que près de 70 % des cellules se divisent dans les 2 h après qu’ils sont incorporés dans les matrices de collagène (Figure 2). Par ailleurs, les cellules peuvent être intégrées dans des matrices de collagène avant la synchronisation, cependant, le réseau réticulé de la matrice de collagène présente une barrière physique et réduit le taux de biomoléculaire diffusion et convection33, 34. en effet, nous avons tenté de synchroniser les cellules dans les matrices de collagène à l’aide de la thymidine et la nocodazole, mais n’a pu obtenir une synchronisation efficace. Ce résultat pourrait être dû à la diffusion inefficace et la convection de la drogue à travers la matrice de collagène.
Réflexion est une propriété intrinsèque optique de biopolymères beaucoup, y compris le collagène. La technique de microscopie confocale réflexion visualise et quantitates la microtopographie des biomatériaux poreux préparés à partir de polymères synthétiques et de collagène 3D matrices23,24,35,36 ,,37. Dans notre laboratoire, nous avons établi des techniques pour surveiller les changements dans la polarité de fibre de collagène comme la concentration de collagène varie de38. Nous décrivons ici la méthode pour surveiller la déformation des fibres de collagène, basé sur la vidéo Time-lapse de l’images confocales réfléchissants pour désigner l’interaction cellule-matrice. Les résultats représentatifs présentés ici montrent que la déformation de la matrice pour les cellules MDA-MB-231 mitotiques est significativement plus petite que les cellules en interphase, ce qui suggère que les cellules de mammifères ont attachement minimal et les interactions avec les matrice environnante lorsqu’ils entrent dans la phase mitotique19.
Auparavant, nous avons utilisé la microscopie confocale réflexion de surveiller et de quantifier l’interaction cellule-matrice avant, pendant et après la mitose des cellules HT1080. Nous avons également suivi la déformation de matrice par β1-intégrine précipitation HT1080 cellules en interphase et en phase de mitose. Appauvrissant la couche d’intégrine β1 significativement réduit la déformation de la matrice de la cellule en interphase. Cependant, il n’y a aucune différence dans la déformation de la matrice pendant la phase mitotique des cellules rondes β1-intégrine knockdown (KD) et le de cellules de type sauvage HT108019.
Une autre approche pour visualiser les fibres de collagène est d’employer la fluorescence conjugué du collagène type I. Précédemment, nous avons utilisé cette approche aux pistes de l’image dans la matrice de collagène produite par les cellules19. Cette approche, cependant, exige le marquage de collagène avec un colorant fluorescent tels que l’isothiocyanate de fluorescéine (FITC), qui est la fois fastidieux et moins efficace. En revanche, la microscopie confocale réflexion peut être directement appliquée au collagène non modifié pour gagner du temps et ressources et exclure les questions liées à la fluorescence photoblanchiment. En outre, cette méthode ne nécessite pas un canal fluorescent individuel et est donc compatible avec tous les colorants fluorescents.
La méthode présentée dans cet article peut potentiellement être appliquée à n’importe quel type de cellules de mammifères qui se divisent en une matrice de collagène 3D. L’incorporation du marqueur H2B-mCherry dans les autres cellules mammifères par transduction LENTIVIRAUX suivra exactement les mêmes procédures tel que décrit dans le livre, bien que différents types de cellules a pu varier l’efficacité pour la transduction de lentivirus 39. les deux la densité des cellules à la transduction et le titre du virus pourraient être optimisées pour l’efficacité. Si l’efficacité élevée de transduction ne peut être atteints, on pourraient choisir cellules par cellule assistée à fluorescence triant (FACS). Blocage de la thymidine et la nocodazole sont appliqués pour synchroniser plusieurs autres types de cellules de mammifères, tels que HeLa40. Secousse mécanique d’arrêt pourrait être appliquée à tous les types cellulaires arrondir et à peine joindre au substrat au cours de la phase mitotique30,31. En outre, l’imagerie des fibres de collagène à l’aide de la microscopie confocale par réflexion et la quantification de la déformation de la matrice peut être directement appliqué à tous les autres types de diviser des cellules de mammifères.
La méthode présentée ici est une approche efficace et générale pour étudier la division cellulaire chez les mammifères et les interactions cellule-matrice dans un environnement 3D. L’approche facilite notre sonde dans la base moléculaire du développement des tissus normaux et les maladies et potentialise la conception du roman diagnostique et thérapeutique s’approche à l’avenir.
Déclarations de divulgation
Les auteurs déclarent qu’ils n’ont aucun intérêt financier concurrentes.
Remerciements
Ce travail a été soutenu par les NIH accorde R01CA174388 et U54CA143868. Les auteurs aimerait remercier le prix PURA de l’Université Johns Hopkins, pour le soutien de Chen Wei-tong. Ce matériel est basé sur le travail soutenu par la National Science Foundation recherche bourse d’études supérieures au titre de la subvention no 1232825.
matériels
| Name | Company | Catalog Number | Comments |
| Human embryonic kidney 293T | ATCC | ||
| MDA-MB-231 | Physical Sciences Oncology Center, NIH | ||
| DMEM | Corning | 10-013-CV | |
| DMEM powder | ThermoFisher Scientific | 12100-046 | |
| Fetal bovine serum | Hyclone | SH30910.03 | |
| Penicillin-Streptomycin 100X | Sigma-Aldrich | P0781 | |
| Fugene HD | Promega | E2311 | |
| Lipofectamine 2000 | Life technologies | 11668-07 | |
| Plasmid encoding H2B-mCherry in a lentiviral vector | Addgene | plasmid 21217 | |
| Thymidine | Sigma-Aldrich | T1895 | |
| Nocodazole | Sigma-Aldrich | M1404 | |
| Opti-MEM | Life Technologies | 31985-070 | |
| Sodium bicarbonate | GibcoBRL | 11810-025 | |
| HEPES | Sigma-Aldrich | 113375-100 | |
| Collagen | Corning | 354236 | |
| NaOH | J.T. Bake | 3722-01 | |
| Millex-HV syringe filter unit, 0.45-μm, PVDF, 33 mm | Millipore | SLHVM33RS | |
| Nikon TE2000E epifluorescence microscope | Nikon | TE2000E | |
| Cascade 1K CCD camera | Roper Scientific | ||
| NIS-Elements AR imaging software | Nikon | ||
| Nikon A1 confocal microscope | Nikon | A1 |
Références
- Ly, D. H., Lockhart, D. J., Lerner, R. A., Schultz, P. G. Mitotic misregulation and human aging. Science. 287 (5462), 2486-2492 (2000).
- Gascoigne, K. E., Taylor, S. S. How do anti-mitotic drugs kill cancer cells? J Cell Sci. 122 (Pt 15), 2579-2585 (2009).
- Brinkley, B. R. Managing the centrosome numbers game: from chaos to stability in cancer cell division. Trends Cell Biol. 11 (1), 18-21 (2001).
- Phillip, J. M., Aifuwa, I., Walston, J., Wirtz, D. The Mechanobiology of Aging. Annu Rev Biomed Eng. 17, 113-141 (2015).
- Hanahan, D., Weinberg, R. A. Hallmarks of cancer: the next generation. Cell. 144 (5), 646-674 (2011).
- Martin, M. D., et al. Effect of ablation or inhibition of stromal matrix metalloproteinase-9 on lung metastasis in a breast cancer model is dependent on genetic background. Cancer Res. 68 (15), 6251-6259 (2008).
- Knox, J. J., Hotte, S. J., Kollmannsberger, C., Winquist, E., Fisher, B., Eisenhauer, E. A. Phase II study of Triapine in patients with metastatic renal cell carcinoma: a trial of the National Cancer Institute of Canada Clinical Trials Group (NCIC IND.161). Invest New Drugs. 25 (5), 471-477 (2007).
- Komlodi-Pasztor, E., Sackett, D. L., Fojo, A. T. Inhibitors targeting mitosis: tales of how great drugs against a promising target were brought down by a flawed rationale. Clin Cancer Res. 18 (1), 51-63 (2012).
- Tong, W. G., et al. Phase I and pharmacologic study of SNS-032, a potent and selective Cdk2, 7, and 9 inhibitor, in patients with advanced chronic lymphocytic leukemia and multiple myeloma. J Clin Oncol. 28 (18), 3015-3022 (2010).
- Matulonis, U. A., et al. Phase II study of MLN8237 (alisertib), an investigational Aurora A kinase inhibitor, in patients with platinum-resistant or -refractory epithelial ovarian, fallopian tube, or primary peritoneal carcinoma. Gynecol Oncol. 127 (1), 63-69 (2012).
- Boss, D. S., et al. Clinical evaluation of AZD1152, an i.v. inhibitor of Aurora B kinase, in patients with solid malignant tumors. Ann Oncol. 22 (2), 431-437 (2011).
- Griffith, L. G., Swartz, M. A. Capturing complex 3D tissue physiology in vitro. Nat Rev Mol Cell Biol. 7 (3), 211-224 (2006).
- Cukierman, E., Pankov, R., Stevens, D. R., Yamada, K. M. Taking cell-matrix adhesions to the third dimension. Science. 294 (5547), 1708-1712 (2001).
- Lu, P., Weaver, V. M., Werb, Z. The extracellular matrix: a dynamic niche in cancer progression. J Cell Biol. 196 (4), 395-406 (2012).
- Giri, A., et al. The Arp2/3 complex mediates multigeneration dendritic protrusions for efficient 3-dimensional cancer cell migration. FASEB J. 27 (10), 4089-4099 (2013).
- Fraley, S. I., Feng, Y., Giri, A., Longmore, G. D., Wirtz, D. Dimensional and temporal controls of three-dimensional cell migration by zyxin and binding partners. Nat Commun. 3, 719(2012).
- Fraley, S. I., et al. A distinctive role for focal adhesion proteins in three-dimensional cell motility. Nat Cell Biol. 12 (6), 598-604 (2010).
- Lesman, A., Notbohm, J., Tirrell, D. A., Ravichandran, G. Contractile forces regulate cell division in three-dimensional environments. J Cell Biol. 205 (2), 155-162 (2014).
- He, L., et al. Local 3D matrix confinement determines division axis through cell shape. Oncotarget. 7 (6), 6994-7011 (2016).
- Held, M., et al. CellCognition: time-resolved phenotype annotation in high-throughput live cell imaging. Nat Methods. 7 (9), 747-754 (2010).
- Fallica, B., Maffei, J. S., Makin, G., Zaman, M. Alteration of cellular behavior and response to PI3K pathway inhibition by culture in 3D collagen gels. PLoS One. 7 (10), e48024(2012).
- Meli, L., Jordan, E. T., Clark, D. S., Linhardt, R. J., Dordick, J. S. Influence of a three-dimensional, microarray environment on human Cell culture in drug screening systems. Biomaterials. 33 (35), 9087-9096 (2012).
- Artym, V. V., Matsumoto, K. Imaging cells in three-dimensional collagen matrix. Curr Protoc Cell Biol. 10, 1-20 (2010).
- Gunzer, M., Kampgen, E., Brocker, E. B., Zanker, K. S., Friedl, P. Migration of dendritic cells in 3D-collagen lattices. Visualisation of dynamic interactions with the substratum and the distribution of surface structures via a novel confocal reflection imaging technique. Adv Exp Med Biol. 417, 97-103 (1997).
- Geraldo, S., Simon, A., Vignjevic, D. M. Revealing the cytoskeletal organization of invasive cancer cells in 3D. J Vis Exp. (80), e50763(2013).
- Lleres, D., James, J., Swift, S., Norman, D. G., Lamond, A. I. Quantitative analysis of chromatin compaction in living cells using FLIM-FRET. J Cell Biol. 187 (4), 481-496 (2009).
- Poincloux, R., et al. Contractility of the cell rear drives invasion of breast tumor cells in 3D Matrigel. Proc Natl Acad Sci U S A. 108 (5), 1943-1948 (2011).
- Carey, S. P., Kraning-Rush, C. M., Williams, R. M., Reinhart-King, C. A. Biophysical control of invasive tumor cell behavior by extracellular matrix microarchitecture. Biomaterials. 33 (16), 4157-4165 (2012).
- Langan, T. J., Chou, R. C. Synchronization of mammalian cell cultures by serum deprivation. Methods Mol Biol. 761, 75-83 (2011).
- Jackman, J., O'Connor, P. M. Methods for synchronizing cells at specific stages of the cell cycle. Curr Protoc Cell Biol. 8, (2001).
- Zwanenburg, T. S. Standardized shake-off to synchronize cultured CHO cells. Mutat Res. 120 (2-3), 151-159 (1983).
- Harper, J. V. Synchronization of cell populations in G1/S and G2/M phases of the cell cycle. Methods Mol Biol. 296, 157-166 (2005).
- Kihara, T., Ito, J., Miyake, J. Measurement of biomolecular diffusion in extracellular matrix condensed by fibroblasts using fluorescence correlation spectroscopy. PLoS One. 8 (11), e82382(2013).
- Ramanujan, S., et al. Diffusion and convection in collagen gels: implications for transport in the tumor interstitium. Biophys J. 83 (3), 1650-1660 (2002).
- Harjanto, D., Maffei, J. S., Zaman, M. H. Quantitative analysis of the effect of cancer invasiveness and collagen concentration on 3D matrix remodeling. PLoS One. 6 (9), e24891(2011).
- Wolf, K., et al. Collagen-based cell migration models in vitro and in vivo. Semin Cell Dev Biol. 20 (8), 931-941 (2009).
- Petroll, W. M. Differential interference contrast and confocal reflectance imaging of collagen organization in three-dimensional matrices. Scanning. 28 (6), 305-310 (2006).
- Fraley, S. I., et al. Three-dimensional matrix fiber alignment modulates cell migration and MT1-MMP utility by spatially and temporally directing protrusions. Sci Rep. 5, 14580(2015).
- Ikeda, Y., Collins, M. K., Radcliffe, P. A., Mitrophanous, K. A., Takeuchi, Y. Gene transduction efficiency in cells of different species by HIV and EIAV vectors. Gene Ther. 9 (14), 932-938 (2002).
- Ma, H. T., Poon, R. Y. Synchronization of HeLa cells. Methods Mol Biol. 761, 151-161 (2011).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon
Nous utilisons des cookies afin d'améliorer votre expérience sur notre site web.
En continuant à utiliser notre site ou en cliquant sur le bouton ''continuer'', vous acceptez l'utilisation de cookies.