Method Article
Divisione delle cellule dei mammiferi in matrici 3D tramite microscopia confocale quantitativa di riflessione
* Questi autori hanno contribuito in egual misura
In questo articolo
Riepilogo
Questo protocollo studia in modo efficiente divisione cellulare dei mammiferi in matrici di collagene 3D integrando sincronizzazione della divisione cellulare, monitoraggio degli eventi di divisione in matrici 3D utilizzando la tecnica di imaging cellule vive, microscopia risolta in tempo di riflessione confocale e quantitative analisi di imaging.
Abstract
Lo studio di mammifero come la divisione cellulare è regolato in un 3D ambiente rimane in gran parte inesplorata, nonostante la sua rilevanza fisiologica e significato terapeutico. Motivi possibili per la mancanza di esplorazione sono i limiti sperimentali e le sfide tecniche che rendono lo studio di divisione delle cellule in cultura 3D inefficiente. Qui, descriviamo un metodo basato su formazione immagine per studiare in modo efficiente divisione cellulare dei mammiferi e delle interazioni cellula-matrice in matrici di collagene 3D. Cellule identificate con H2B fluorescenti vengono sincronizzate utilizzando la combinazione di trattamento blocco e nocodazole della timidina, seguita da una tecnica di shake-off meccanica. Sincronizzate le cellule vengono quindi incorporate in una matrice di collagene 3D. Divisione cellulare viene monitorata tramite microscopia di cellule vive. La deformazione delle fibre del collagene durante e dopo la divisione cellulare, che è un indicatore di interazione cellula-matrice, possa essere monitorata e quantificati tramite microscopia confocale quantitativa di riflessione. Il metodo fornisce un approccio efficiente e generale per studiare la divisione delle cellule dei mammiferi e delle interazioni cellula-matrice in un ambiente 3D fisiologicamente rilevante. Questo approccio non solo fornisce le comprensioni novelle nella base molecolare dello sviluppo del tessuto normale e malattie, ma permette anche per la progettazione di nuovi approcci diagnostici e terapeutici.
Introduzione
Mitosi delle cellule sono un evento critico nella vita cellulare, il regolamento di cui svolge un ruolo cruciale nello sviluppo di tessuti ed organi. Mitosi anomale sono implicato in variazioni genetiche naturali, processi di invecchiamento umano e la progressione del cancro1,2,3,4,5. Dell'aumento del tasso di proliferazione delle cellule tumorali rispetto alle cellule normali è uno dei tratti distintivi del cancro, nonostante il fatto che i comportamenti delle cellule sono piuttosto eterogenei tra diversi tipi di tumori e anche fra i pazienti. Nonostante i promettenti risultati preclinici, alcuni farmaci antimitotici recente sviluppato non hanno dimostrato di essere efficace in studi clinici6,7,8,9,10 ,11. La rilevanza dei modelli preclinici e sperimentale deve essere considerato. Molti tipi di cellule di cancro e mammiferi normali si dividono in tridimensionale (3D) matrici, come fibroblasti e cellule di fibrosarcoma in tessuti connettivi 3D-ricchi di collagene e cellule tumorali metastatiche in 3D stromal matrice extracellulare (ECM). Tuttavia, la stragrande maggioranza degli esperimenti di divisione delle cellule dei mammiferi e saggi è stata effettuata su cellule coltivate su substrati di bidimensionale (2D). Una matrice 3D ingegnerizzata potrebbe meglio ricapitolare la microstruttura, proprietà meccaniche e segnali biochimici della ECM 3D di entrambi tessuti normali e patologici12,13,14, 15,16,17.
Lo studio di mammifero come la divisione cellulare è regolato in 3D ambienti rimane in gran parte inesplorata, nonostante sia la rilevanza fisiologica e il significato terapeutico18,19. Possibili cause sono le difficoltà tecniche e sperimentali sfide connesse con lo studio di divisione cellulare in matrici 3D. Mitosi delle cellule costituiscono una piccola frazione temporale nell' intero ciclo cellulare20. Il lavoro precedente ha indicato che il tasso di proliferazione delle cellule di molti mammiferi, come adenocarcinoma umano del seno MCF-7, osteosarcoma umano U2OS e fegato umano HepG2, è molto più bassa in matrici 3D rispetto alle loro controparti su substrati 2D21, 22. Inoltre, cellule incorporate in matrici 3D muovono dentro e fuori la messa a fuoco durante la formazione immagine di cellule vive. Tutti questi fattori contribuiscono all'efficienza estremamente bassa di acquisizione degli eventi di divisione delle cellule in cultura 3D usando tecniche di imaging.
Interazioni tra ECM e cellule giocano un ruolo critico nella regolazione di divisioni cellulari. Qui, descriviamo un metodo per studiare in modo efficiente divisione cellulare dei mammiferi in matrici di collagene 3D. Il metodo comprende l'incorporazione di marcatori mitotici delle cellule, sincronizzazione di divisione delle cellule, come pure il monitoraggio degli eventi di divisione in matrici 3D utilizzando la tecnica di imaging di cellule vive, risolta in tempo di riflessione confocale microscopia, e analisi quantitativa di imaging. Proteina di fluorescenza-labeled istone H2B è introdotto nelle cellule come un marcatore di differenziare mitotica e cellule di interfase. Quindi le cellule vengono sincronizzate utilizzando la combinazione di trattamento blocco e nocodazole della timidina, seguita da una tecnica di shake-off meccanica. Cellule sincronizzate sono quindi direttamente incapsulate in matrici di collagene 3D. Eventi di divisione cellulare delle cellule più sono monitorati in modo efficiente utilizzando basso ingrandimento time-lapse cellule vive imaging. La deformazione delle fibre del collagene, che è un indicatore di interazione cellula-matrice, è controllata mediante microscopia confocale riflessione alle ad alto ingrandimento.
In precedenza abbiamo usato questa tecnica per monitorare e quantificare l'interazione cellula-matrice prima, durante e dopo la mitosi di due linee cellulari di cancro metastatico, il carcinoma ductal dilagante umano MDA-MB-231 e cellule di fibrosarcoma umano HT1080, in collagene 3D matrici19. I metodi qui presentati forniscono un approccio efficiente e generale per studiare entrambi divisione cellulare dei mammiferi in un ambiente 3D interazioni cellula-matrice. La linea di cellule MDA-MB-231 è utilizzata come esempio in tutto il libro. Questo protocollo fornisce le comprensioni novelle nella base molecolare dello sviluppo del tessuto normale e malattie e potrebbe anche consentire per la progettazione di nuovi approcci diagnostici e terapeutici.
Protocollo
Protocollo di cui segue le linee guida di il Homewood The delle revisione istituzionale in Consiglio (HIRB).
1. stabile espressione di H2B-mCherry come indicatore per mitosi delle cellule
-
Generazione di particelle lentivirali da embrionali umane del rene 293T (HEK 293T) cellule
- Le cellule HEK 293T su una piastra di coltura delle cellule di 10 cm alla densità di 5 x 106 cellule/piatto nel mezzo di coltura cellulare (di Dulbecco Medium (DMEM) contenenti elevati del glucosio dell'Aquila per volta (4,5 g/L), piruvato di sodio, 10% siero bovino fetale (FBS) e 1% del piatto penicillina-streptomicina (Pen/Strep)). Incubare per 24 h a 37 ° C e 5% di anidride carbonica (CO2). Il confluency di desiderata delle cellule il giorno di transfezione è circa 70-80%.
Nota: Le cellule HEK 293T sono usate qui per produrre particelle virali, che verranno utilizzate successivamente per trasdurre cellule MDA-MB-231 al fine di generare una linea di cellule che esprimono stabilmente H2B-mCherry. Assicurarsi che le celle siano distribuite uniformemente in tutta la piastra. Aggiunta delle cellule goccia saggio alla piastra, e delicatamente muovendo avanti e indietro la piastra può aiutare nella distribuzione uniforme. Cellule possono anche essere seminate in piastre o boccette di altri formati, che si tradurrà nella raccolta di volumi differenti di particelle virali. - Transfect le cellule HEK 293T utilizzando un reagente di transfezione con tre plasmidi (vettore lentivirale, CMV ΔR 8,91 e pMDG-VSVG). Il plasmide codifica H2B-mCherry è clonato in un vettore lentivirale con promotore di chinasi di phosphoglycerate (PGK). ΔR CMV 8,91 contiene tre richiesto HIV geni gag, pole rev. pMDG-VSVG contiene il gene di busta VSV-G.
Nota: Ci sono più agenti di transfezione da scegliere.- Lasciare riposare il flaconcino di reagente di transfezione di raggiungere temperatura ambiente (TA) prima dell'uso. Inverti o vortex brevemente la fiala.
- Mix 16 µ g di DNA, che consiste di 6 µ g di H2B-mCherry plasmide, 8 µ g di CMV ΔR 8,91 e 2 µ g di pMDG-VSVG, in 1 mL di media riduttori del siero. Incubare a temperatura ambiente per 5 min.
Nota: La quantità e il rapporto dei tre vettori è variato e ottimizzato per vettori lentivirali differenti di trasferimento. - µ L 48 (utilizzare il rapporto 1:3 di DNA: reagente di transfezione) di reagente di transfezione nella soluzione di DNA di cui sopra e poi incubare a temperatura ambiente per almeno 15 min.
Nota: Il rapporto del DNA vs reagente di transfezione è variato e ottimizzato per vettori lentivirali differenti di trasferimento. Assicurarsi che il reagente di transfezione non faccia contatto con il lato della provetta da centrifuga da 1,5 mL. - Aggiungere la soluzione di complessi DNA-lipidico effettuata nel passaggio 1.1.2.3 drop-wise alle cellule. Agitare delicatamente la piastra per garantire la distribuzione uniforme del complesso della piastra.
- Circa 6 ore dopo la trasfezione, aspirare il mezzo e aggiungere mezzo di coltura di cellule fresche (DMEM con alto glucosio (4,5 g/L), piruvato di sodio, 10% FBS e 1% Pen/Strep).
- Raccogliere il surnatante 24, 48 e post-transfezione 72 h. Sostituire il mezzo dopo ogni raccolto (DMEM, 4,5 g/L glucosio, sodio piruvato, con 10% FBS e 1% Pen/Strep).
- Filtrare le particelle lentivirali attraverso un filtro da 0,45 µm per rimuovere i detriti cellulari. In alternativa, di rallentare il supernatante di separare i detriti cellulari. Il supernatante può essere utilizzato per la trasduzione subito, o possa essere conservato a-80 ° C.
- Le cellule HEK 293T su una piastra di coltura delle cellule di 10 cm alla densità di 5 x 106 cellule/piatto nel mezzo di coltura cellulare (di Dulbecco Medium (DMEM) contenenti elevati del glucosio dell'Aquila per volta (4,5 g/L), piruvato di sodio, 10% siero bovino fetale (FBS) e 1% del piatto penicillina-streptomicina (Pen/Strep)). Incubare per 24 h a 37 ° C e 5% di anidride carbonica (CO2). Il confluency di desiderata delle cellule il giorno di transfezione è circa 70-80%.
-
Generazione di cellule linee stabilmente esprimenti H2B-mCherry
Nota: Questo protocollo è descritto in dettaglio per le cellule MDA-MB-231. Prima dell'uso nell'esperimento, cellule di carcinoma mammario umano MDA-MB-231 (centro oncologico di scienze fisiche, NIH) sono coltivate in DMEM contenente alto glucosio (4,5 g/L), piruvato di sodio, 10% FBS e 1% Pen/Strep.- Piastra le cellule MDA-MB-231 alla densità di 1 x 105 celle in una piastra di coltura di 35 mm.
Nota: La densità di placcatura per differenti linee cellulari dovrebbe essere ottimizzata e pertanto potrebbe essere diversa dalla densità ottima per le cellule MDA-MB-231. - 24 h dopo il placcaggio delle cellule, aggiungere 1 mL di virus e 1 mL di terreno nuovo (DMEM, alto glucosio (4,5 g/L), piruvato di sodio, 10% FBS e 1% Pen/Strep).
- Incubare le cellule per un periodo di tempo di 6 ore per una notte.
Nota: Sia il volume del virus e il tempo di incubazione devono essere ottimizzate per differenti linee cellulari. Ad esempio, se le cellule non appaiono sane poche ore dopo l'aggiunta del virus, utilizzare 1 mL di virus e 2 mL di terreno nuovo (DMEM, alto glucosio (4,5 g/L), piruvato di sodio, 10% FBS e 1% Pen/Strep) al punto 1.2.2. La lunghezza del tempo di incubazione nel passaggio 1.2.3 può anche essere ridotto. - Aspirare il mezzo e aggiungere 10 mL di terreno nuovo (DMEM, alto glucosio (4,5 g/L), piruvato di sodio, 10% FBS e 1% Pen/Strep). Incubare per 24-72 h a 37 ° C e 5% CO2.
- Verificare che l'espressione di H2B-mCherry nelle cellule utilizzando un microscopio a epifluorescenza.
Nota: Se l'efficienza di trasduzione è bassa nelle cellule, il volume del virus nel passaggio 2.2 e il tempo di incubazione nel passaggio 2.3 potrebbe essere aumentato.
- Piastra le cellule MDA-MB-231 alla densità di 1 x 105 celle in una piastra di coltura di 35 mm.
2. sincronizzazione delle cellule che esprimono stabilmente H2B-mCherry
- Piastra le cellule a 50-60% confluenza, cioè piastra 2 x 104 MDA-MB-231 cellule in ciascun pozzetto di una piastra a 24 pozzetti.
- Incubare la coltura a 37 ° C e 5% di CO2 per 24 h.
- Sostituire il mezzo di crescita con 0,5 mL di medium (DMEM, alto glucosio (4,5 g/L), piruvato di sodio, 10% FBS e 1% Pen/Strep) contenente timidina 2mm e lasciare nell'incubatrice per 24 h.
Nota: Le cellule esposte a timidina sono arrestate nella fase di crescita delle cellule (G1) / transizione di DNA sintesi (S) e in tutta la fase di S dovuto l'inibizione della sintesi del DNA. La lunghezza del tempo di incubazione dovrebbe essere vario e ottimizzata per differenti linee cellulari. - Rilasciare le cellule dall'esposizione della timidina lavandole con tamponato fosfato salino (PBS) tre volte. Quindi, incubare le cellule in terreno di coltura delle cellule normali (DMEM, alto glucosio (4,5 g/L), piruvato di sodio, 10% FBS e 1% Pen/Strep) per 5 h.
Nota: Il rilascio delle cellule dall'esposizione della timidina permette alle cellule di progresso per la crescita delle cellule (G2) / mitotica fase (M) per le cellule precedentemente arrestato nella fase G1/S e alla fase G1 per cellule precedentemente arrestato al S di fase. La lunghezza del tempo di rilascio dovrebbe essere vario e ottimizzata per differenti linee cellulari. - Bloccare le cellule con 250 ng/mL di nocodazole per 12 h.
Nota: Tutte le cellule esposte a nocodazole vengono arrestate nella fase G2/M. Nocodazole è citotossico. L'esposizione prolungata a nocodazole può causare l'apoptosi. Regolare il periodo o la concentrazione di esposizione per diverse linee cellulari se morti di cella sono osservati. Le cellule che vengono sincronizzate correttamente esporrà una morfologia sferica. - Agitare le cellule per 45 s per 1 min, utilizzando un agitatore orbitale a 150-200 giri/min.
Nota: Cellule mitotiche, che hanno poca aderenza al substrato, saranno scosso durante il processo. - Rimuovere il mezzo per estrarre le cellule, pipettando il mezzo in una provetta da centrifuga e quindi aggiungere 0,5 mL di terreno nuovo (DMEM, alto glucosio (4,5 g/L), piruvato di sodio, 10% FBS e 1% Pen/Strep) a ciascun pozzetto della piastra.
- Ripetere i passaggi da 2.6 e 2.7 tre volte.
- Centrifugare il supporto raccolto che contiene le cellule mitotiche a 800 x g per 3 min.
Nota: Questo passaggio viene utilizzato per rimuovere il nocodazole da medium delle cellule.
3. incorporazione di Synchronized di celle in collagene sono matrici
Nota: Tipo I collagene è la proteina più abbondante nel corpo umano e in ECM dei tessuti connettivi e così è ampiamente usato per studiare delle cellule eucariotiche come funzioni sono modulate da un ambiente 3D17,23,24 . Il collagene è solubile in acido acetico. Dopo la neutralizzazione e la soluzione di collagene a 20-37 ° C il riscaldamento, monomeri di collagene polimerizzano in un reticolo di fibrille di collagene.
- Preparare la soluzione di x DMEM 10 da sciogliere una bustina di polvere DMEM, 3,7 g di bicarbonato di sodio (NaHCO3) e 1 g di acido-(2-hydroxyethyl)-1-piperazineethanesulfonic 4 (HEPES) in 50 mL di acqua distillata. Filtrare la soluzione e quindi preparare 1 M di idrossido di sodio (NaOH) sciogliendo 2 g di NaOH pellet in 50 mL di acqua distillata. Filtro e aliquota della soluzione in provette per centrifuga da 1,5 mL.
Nota: Soluzione DMEM normale non dovrebbe essere usato in questo passaggio. L'aggiunta di volume significativo della soluzione collagene diluirà il mezzo. Quindi la soluzione concentrata di DMEM è preparata per garantire che la concentrazione finale di DMEM nella matrice collagene sarà lo stesso come il normale DMEM. - Continuare a lavorare con le cellule prelevate dal passo 2.9. Aspirare il medio e risospendere le cellule in circa 0,25 - 0,5 mL di terreno di coltura di cellule fresche (DMEM, alto glucosio (4,5 g/L), piruvato di sodio, 10% FBS e 1% Pen/Strep).
Nota: Per raggiungere una densità di cella specifica della matrice di collagene, la densità iniziale delle cellule in sospensione non può essere troppo bassa. Così, il volume del mezzo utilizzato per risospendere le cellule dipenderà il numero totale di celle disponibili. - Posto 10 µ l della soluzione ri-sospensione cellulare da passo 3.2 un emocitometro e il Conte la densità delle cellule nella soluzione.
-
Determinare i volumi necessari per tutti i componenti rendere la matrice del collagene 3D. 500 µ l di gel di collagene µ g / µ l 2 viene utilizzato qui come esempio.
- Calcolare il volume della soluzione cella necessaria ad ottenere la 40.000 cellule/mL (o 20.000 celle per la 500 µ l di gel di collagene). Questo numero in µ l è X. 50 µ l di 10 x DMEM saranno necessari. Saranno necessari 50 µ l di FBS.
- Calcolare il volume di collagene che scorta necessario. La concentrazione di collagene I è intorno a 4 µ g / µ l. La quantità di collagene necessario in µ l è Y.
- Calcolare il volume di idrossido di sodio è necessario neutralizzare l'acido acetico nella soluzione del collagene: collagene µ l Y * 0,023 * 1000 = Z µ l NaOH necessari.
- Determinare il volume della soluzione di riempimento: (500 µ l totale gel - cellule del X µL - 50 µ l 10 X collagene µ l di DMEM - 50 µ l di FBS - Y - Z µ l NaOH) = R µ l
Nota: La soluzione di riempimento è distillata acqua. Se il volume calcolato della soluzione di riempimento è un numero negativo, la densità cellulare originale la sospensione cellulare è troppo bassa. Le cellule hanno bisogno di essere filato giù nuovamente e nuovamente sospeso in un più piccolo volume del mezzo (DMEM, alto glucosio (4,5 g/L), piruvato di sodio, 10% FBS e 1% Pen/Strep).
- Aggiungere i componenti nel seguente ordine a una provetta da centrifuga pulito 1,5 mL: cellule in medium, 10x DMEM, FBS, acqua deionizzata (DI), collagene, NaOH. Mettere tutti i componenti sul ghiaccio per rallentare la gelificazione della soluzione del collagene. Utilizzare tecniche di pipettaggio corretto per prevenire la formazione di bolle.
- Dopo aver aggiunto il NaOH, mescolate con cura la soluzione con una pipetta da 1 mL.
Nota: Gelificazione del collagene partirà proprio subito dopo l'aggiunta di NaOH. Miscelazione dovrebbe essere fatto con cura e rapidamente. - Una volta che la soluzione è ben amalgamata, aggiungere 500 µ l della soluzione in ciascun pozzetto della piastra 24 pozzetti. Posizionare la piastra a 24 pozzetti nell'incubatore 37 ° C. Luogo terreno di coltura di cellule fresche nel bagno d'acqua di 37 ° C.
Nota: Piastre di fondo in plastica sono utilizzate per basso ingrandimento microcopy di cellule vive in cui la distanza di lavoro della lente è sopra vetro inferiore di 1 mm. piastre sono utilizzati per microscopia di cellule vive di alto ingrandimento a causa della breve distanza di lavoro di alta lente di ingrandimento. - Aggiungere 500 µ l di terreno pre-riscaldato (DMEM, alto glucosio (4,5 g/L), piruvato di sodio, 10% FBS e 1% Pen/Strep) alla parte superiore del gel 30 min dopo aver completato il passaggio 3,7.
4. live Cell Imaging delle cellule dividendo nelle matrici di collagene 3D (basso ingrandimento)
Nota: Vengono raccolte le immagini delle cellule a intervalli di 2 minuti utilizzando una fotocamera di charge coupled device (CCD) montata su un microscopio di contrasto di fase che è equipaggiato con un obiettivo da 10x e controllato da software di imaging.
- Accendere l'unità di tensione delle cellule montato sopra la lente dell'obiettivo. Attendere che la temperatura si stabilizza a 37 ° C, la concentrazione di CO2 è al 5% e l'umidità è al 75%.
- Mettere la piastra a 24 pozzetti del gel nell'unità di cellule vive sul microscopio. Trovare il fondo del piatto e quindi spostare la lente dell'obiettivo fino a quando il microscopio si concentra a circa 500 µm dalla parte inferiore della piastra.
Nota: Le cellule della matrice di collagene che sono troppo vicino alla parte inferiore della piastra non sono completamente incorporati nella matrice 3D. Le cellule che sono 500 µm dalla parte inferiore della piastra di imaging farà in modo che le cellule non sono interessate da effetti di bordo16,17,25. - Spostare il palco intorno per trovare le celle e selezionare posizioni multiple per l'imaging.
- Impostare l'esperimento di time-lapse a intervalli di 2 min.
Nota: Il numero massimo di posizioni che possono essere adottate durante l'intervallo di 2 min è limitato la velocità di spostamento di fase motorizzata, e la lunghezza del tempo di posa per catturare le immagini.
5. collagene rete deformazione durante la divisione cellulare (microscopia ad alto ingrandimento)
- Accendere l'unità di tensione delle cellule montato sopra la lente dell'obiettivo. Attendere che la temperatura si stabilizza a 37 ° C, la concentrazione di CO2 è al 5% e l'umidità è al 75%.
- Per visualizzare le fibre di collagene in matrici di collagene 3D non macchiate, configurare un microscopio confocale per catturare solo luce riflessa (488 nm) del laser di 488 nm utilizzato per illuminare il campione. Il laser utilizza un 60 X obiettivo a immersione in acqua, NA = 1.2, WD = 200 µm ed è controllata da software di imaging.
- Mettere il campione (celle in matrici di collagene) nell'unità di cellule vive. Trovare il fondo del piatto e quindi spostare la lente dell'obiettivo fino a quando il microscopio si concentra a circa 100 µm dalla parte inferiore della piastra. Le cellule che sono 100 µm dalla parte inferiore della piastra di imaging farà in modo che le cellule non sono interessate dagli effetti di bordo.
Nota: Qui un obiettivo a immersione in acqua è usato invece di un obiettivo a immersione in olio perché la distanza di lavoro dell'obiettivo d'immersione in olio è solo circa 100 µm. L'uso dell'obiettivo d'immersione in olio pone un problema come lo spessore del vetro sul fondo della piastra è normalmente circa 100 µm. - Spostare il palco intorno per trovare le celle e selezionare posizioni multiple per l'imaging.
Nota: La microscopia confocale otticamente sezioni del campione e acquisisce solo i segnali dal piano focale. Cellule vive, che potrebbero essere in movimento dentro e fuori di messa a fuoco durante l'esperimento di time-lapse. Per consentire l'acquisizione di immagini di cella per lunghi periodi di tempo, Z-stack viene utilizzato per raccogliere immagini da fette successivi a intervalli di 5 µm. Un totale di 5 fette spanning 25 µm sono Imaging. - Impostare l'esperimento di time-lapse a intervalli di 5 min.
Nota: 5 minuti viene utilizzato qui come l'intervallo invece di 2 min utilizzati nella sezione 4 perché molteplici modalità di scansione, tra cui fluorescenza scansione per H2B-mCherry, riflesso di scansione per il collagene e Z-stack scansione, vengono applicate a ogni posizione. L'uso delle diverse modalità di scansione riduce la velocità di scansione una posizione rispetto all'imaging di ingrandimento basso. - Quantificare la deformazione della matrice del collagene nelle vicinanze di una cella a causa di forze esercitate da quella cella utilizzando la particella velocimetry (PIV) con sub-pixel risoluzione20di imaging. L'esecuzione di questa analisi su immagini 2D consente per la chiara visualizzazione di entrambi i segnali di H2B-mCherry e le fibre di collagene. Applicare l'anisotropo passa-basso filtro per migliorare il segnale del collagene rete20.
- Al fine di misurare lo spostamento locale di una regione sub-immagine di interesse che si trova a (x,y) da telaio k a telaio k+ 1, estrarre una finestra regionale di 15 × 15 pixel centrati rispetto a (x,y) al telaio k. Quindi identificare il miglior abbinamento di posizioni (x,y) * attraverso le località che dispongono di un massimo coefficiente di correlazione normalizzato nell'immagine ottenuta a telaio k+ 1. Il vettore di deformazione è calcolato come (x,y) *-(x,y).
Risultati
L'obiettivo di questo articolo è di presentare un metodo basato su imaging per lo studio dei processi di divisione cellulare dei mammiferi in matrici 3D e quantificare le interazioni tra la cella e la matrice extracellulare 3D durante e dopo la divisione cellulare. Per facilitare l'imaging della mitosi delle cellule, abbiamo incorporato H2B-mCherry nelle cellule MDA-MB-231 usando trasduzione lentivirale. H2B coniugati con proteine fluorescenti è utilizzato come marcatore mitotic di distinguere cellule mitotiche da cellule di interfase e di definire diverse fasi durante cellula mitosi19,20,26. Utilizzando questo metodo, siamo stati in grado di monitorare il processo di tutta la divisione delle cellule MDA-MB-231 che esprimono stabilmente H2B-mCherry in matrici di collagene 3D (Figura 1A). Mitotiche fasi iniziato con la dissoluzione della membrana nucleare (profase fotogramma 1, fig. 1A); la riorganizzazione dei cromosomi (Prometafase: telaio 2); l'allineamento dei cromosomi al centro del corpo di cell (metafase telaio 3); la separazione dei cromosomi (anafase telaio 4); la riorganizzazione dei cromosomi e membrana nucleare e la separazione dei corpi di due cellule della figlia (telofase/citochinesi, frame 5).
Il tasso di proliferazione delle cellule in una matrice 3D è solitamente molto più basso rispetto alle loro controparti su un substrato 2D, che rende il controllo della divisione cellulare in 3D meno efficiente19. Per aumentare l'efficienza dello studio 3D divisione cellulare, abbiamo impiegato un metodo di combinazione della timidina, nocodazole e una tecnica shake-off per la sincronizzazione e selezionare le cellule MDA-MB-231 che sono nella fase mitotica. Sincronizzate le cellule erano quindi incorporate in matrici di collagene. Il processo di divisione delle cellule multiple è stato monitorato in tempo reale con immagini di cellule vive a 10 ingrandimenti. Circa il 70% delle cellule diviso entro il primo 2 h dopo la formazione della matrice di collagene (Figura 2), consentendo in tal modo un controllo efficace della mitosi in 3D (Figura 2).
Per monitorare l'interazione tra cellule e loro matrici di collagene circostante, abbiamo combinato la microscopia confocal di riflessione immagine alle fibre di collagene e microscopia a fluorescenza per le celle di immagine. Un obiettivo X 60 permette per la cattura di immagini di alta qualità delle fibre del collagene. Imaging mediante una lente ad alto ingrandimento, che cattura meno cellule in ogni campo di vista, è molto inferiore-rendimento rispetto all'uso di basso ingrandimento 10x o 20x. Corretta sincronizzazione delle cellule aumenta notevolmente l'efficienza e la velocità effettiva di tale esperimento, poiché la maggior parte delle cellule sincronizzate dividere entro i primi 2 h dopo la formazione della matrice del collagene. La deformazione di matrice durante e dopo la divisione cellulare è visualizzato in quel momento (rappresentante istantanee sono mostrati in Figura 1B) e quantificati tramite un software personalizzato di PIV. Abbiamo quantificato e confrontato deformazione della matrice durante l'interfase e fase mitotica per cellule sincronizzate in fase mitotica. Abbiamo osservato che la deformazione della matrice molto poco cambiato durante il mitotic fase (Figura 3A), ed è meno che la deformazione osservata nelle fasi post-mitotici (Figura 3B). Questo risultato dimostra che le cellule di mammiferi hanno allegato minimal e interazioni con la matrice circostante mentre sono in fase mitotica.
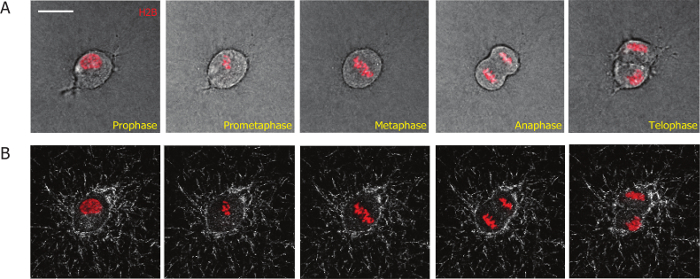
Figura 1: rappresentante micrografie ottenute da un video di formazione immagine di cellule vive ad alto ingrandimento di una cella di MDA-MB-231 incorporati in una matrice di collagene che esprime stabilmente H2B-mCherry. (A) diverse fasi della progressione mitotica della cella MDA-MB-231 sono definiti da H2B-mCherry come indicato in rosso. (B) le fibre di collagene (bianche) durante il processo mitotico sono visualizzate da microscopia di confocal riflesso dipendente dal tempo. Barra della scala = 20 µm. Clicca qui per visualizzare una versione più grande di questa figura.

Figura 2: divisione delle cellule sincronizzate in matrici di collagene 3D. Le cellule erano sincronizzate alla fase G2/M e incorporate in una matrice di collagene. Circa il 70% delle cellule sincronizzate dividere entro le prime 2 ore, considerando che controllo le cellule senza divisione di sincronizzazione in modo casuale. Barra di errore = SEM (errore standard della media). Clicca qui per visualizzare una versione più grande di questa figura.
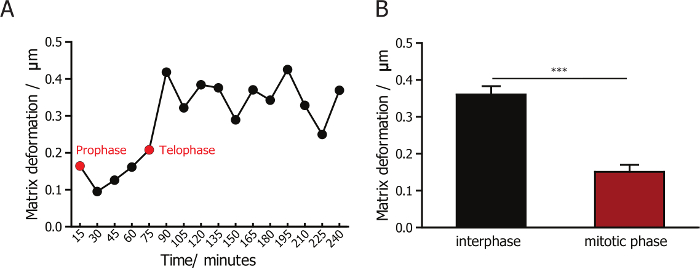
Figura 3: quantificazione della deformazione matrice per MDA-MB-231 cellule durante l'interfase e mitosi. (A) la modifica dell'ordine di deformazione della matrice per matrix-embedded cellule MDA-MB-231. La freccia verde indica la profase e freccia rossa indica telofase. (B) quantificazione della deformazione della matrice per le cellule MDA-MB-231 durante l'interfase e fase mitotica, che indica che la deformazione della matrice è minimo durante la divisione cellulare. Barra di errore = SEM (errore standard della media). * p < 0.05. Clicca qui per visualizzare una versione più grande di questa figura.
Discussione
Il precedente studio di divisione cellulare in 3D non era efficiente a causa di limiti sperimentali e sfide tecniche18,19. I passaggi critici per efficiente studio di divisione delle cellule dei mammiferi in matrici di collagene 3D sono: (1) l'incorporazione di fluorescenza-labeled mitotici marcatori per le cellule; (2) la sincronizzazione della divisione cellulare; e (3) il monitoraggio degli eventi di divisione in matrici 3D utilizzando la tecnica di imaging di cellule vive, risolta in tempo di riflessione confocale microscopia e analisi quantitativa di imaging.
Cellule mitotiche su substrati 2D si possono distinguere dalle cellule di interfase basate sulla loro morfologia, cioè cellule mitotiche sono rotonde e a malapena associare al substrato, mentre cellule di interfase stendere e fissare saldamente al substrato. In matrici 3D, tuttavia, la morfologia delle cellule non è un indicatore affidabile per cellule mitotiche poiché alcune cellule a malapena sparsi e rimangono rotonda nella matrice27,28. Quindi, è essenziale per introdurre un marcatore mitotico alle cellule per lo studio della divisione cellulare in matrici 3D. Abbiamo espresso stabilmente H2B-mCherry in cellule MDA-MB-231, che ha servito come un indicatore affidabile per diverse fasi mitotiche tra cui profase, prometafase, metafase, anafase e telofase/citochinesi. Abbiamo precedentemente usato questo approccio per distinguere cellule mitotiche da cellule di interfase in matrici 3D. Con l'aiuto di questo marcatore, siamo stati anche in grado di misurare la lunghezza della fase mitotica per un'altra linea cellulare, cellule HT1080, dividendo su substrati 2D e in 3D matrici19.
Ci sono diversi modi per sincronizzare le cellule, tra cui siero inedia29, mitotica shake-off30,31, timidina doppia blocco32e nocodazole32. Abbiamo combinato il trattamento di timidina e il trattamento di nocodazole per sincronizzare in modo efficiente le cellule MDA-MB-231 alla fase G2/M. Le cellule esposte a timidina vengono arrestate alla transizione G1/S e durante la fase di S dovuto l'inibizione della sintesi del DNA da timidina. Il rilascio delle cellule dall'esposizione della timidina che i progressi di cellule in fase G2/M per cellule arrestato alla fase G1/S e a G1 per cellule arrestato alla fase S. Tutte le cellule esposte a nocodazole vengono arrestate nella fase G2/M. Le cellule di arrotondato entrando in fase mitotica erano poi scosso-fuori dalla piastra e incapsulate direttamente nelle matrici di collagene. Abbiamo mostrato che circa il 70% delle cellule di dividersi entro 2 h dopo che sono incorporati nelle matrici di collagene (Figura 2). In alternativa, le cellule possono essere incorporate nelle matrici di collagene prima della sincronizzazione, tuttavia, la rete reticolata della matrice del collagene presenta una barriera fisica e riduce il tasso di biomolecolari diffusione e convezione33, 34. infatti, abbiamo tentato di sincronizzare le celle in matrici di collagene utilizzando timidina e nocodazole, ma non è riuscito a ottenere sincronizzazione efficiente. Questo risultato potrebbe essere a causa della diffusione inefficiente e convezione dei farmaci attraverso la matrice di collagene.
Riflessione è una proprietà intrinseca ottica di biopolimeri molti, tra cui collagene. La tecnica di microscopia confocale reflection Visualizza e quantitates la topografia dei biomateriali porosi preparato da polimeri sintetici e collagene 3D matrici23,24,35,36 ,37. Nel nostro laboratorio, abbiamo stabilito le tecniche per monitorare i cambiamenti nella polarità della fibra collagene come la concentrazione di collagene varia38. Qui, descriviamo il metodo per monitorare la deformazione delle fibre del collagene basato sul video time-lapse delle immagini confocale riflettente per denotare l'interazione cellula-matrice. I risultati rappresentativi presentati qui dimostrano che la deformazione della matrice per le cellule MDA-MB-231 mitotiche è significativamente inferiore a quelle cellule in interfase, che suggerisce che le cellule di mammiferi hanno allegato minimal e interazioni con la matrice circostante quando entrano la fase mitotica19.
In precedenza, abbiamo usato la microscopia confocal di riflessione per monitorare e quantificare l'interazione cellula-matrice prima, durante e dopo la mitosi delle cellule HT1080. Abbiamo controllato anche la deformazione della matrice dalle cellule HT1080 knock-down di β1-integrina durante l'interfase sia fase mitotica. Che impoveriscono lo strato di β1-integrina significativamente riduce la deformazione della matrice dalla cella durante l'interfase. Tuttavia, non esiste alcuna differenza di deformazione della matrice durante la fase mitotica delle cellule rotonde β1-integrina atterramento (KD) e tipo selvaggio cellule HT108019.
Un approccio alternativo per visualizzare le fibre di collagene è di impiegare coniugati a fluorescenza collagene di tipo I. In precedenza abbiamo usato questo approccio all'immagine tracce nelle matrici di collagene generate da cellule19. Questo approccio, tuttavia, richiede l'etichettatura del collagene con colorante fluorescente ad esempio isotiocianato di fluorescina (FITC), che è sia in termini di tempo e meno efficiente. D'altra parte, la microscopia confocal di riflessione può essere applicata direttamente al collagene non modificato per risparmiare tempo e risorse ed escludere i problemi associati con fluorescenza photobleaching. Inoltre, questo metodo non richiede un singolo canale fluorescente e pertanto è compatibile con tutti i coloranti fluorescenti.
Il metodo presentato in questa carta potenzialmente applicabile a qualsiasi tipo di cellule di mammifero che dividono in una matrice di collagene 3D. L'incorporazione del marcatore H2B-mCherry in altre cellule di mammiferi attraverso la trasduzione lentivirale seguirà esattamente le stesse procedure come descritto nel libro, sebbene diversi tipi di cellule possono avere varie efficienze per trasduzione da lentivirus 39. sia la densità delle cellule al momento di trasduzione e il titolo del virus potrebbe essere ottimizzati per l'efficienza. Se non è possibile efficienza di trasduzione alta, cellule potrebbero essere selezionate dalla cella assistita fluorescenza ordinano (FACS). Blocco di timidina e nocodazole sono applicate per sincronizzare correttamente diversi altri tipi di cellule di mammifero, come HeLa40. Shake-off meccanico potrebbe essere applicato a qualsiasi tipo di cellula che arrotondare e a malapena allega al substrato durante la fase mitotica30,31. Inoltre, l'imaging delle fibre del collagene usando la microscopia confocal di riflessione e la quantificazione della deformazione matrice può essere direttamente applicato a tutti gli altri tipi di divisione delle cellule dei mammiferi.
Il metodo presentato qui è un approccio efficiente e generale per studiare la divisione delle cellule dei mammiferi e delle interazioni cellula-matrice in un ambiente 3D. L'approccio facilita la nostra sonda nella base molecolare dello sviluppo del tessuto normale e malattie e rafforza il design del romanzo diagnostico e terapeutico si avvicina in futuro.
Divulgazioni
Gli autori dichiarano di non avere nessun concorrenti interessi finanziari.
Riconoscimenti
Questo lavoro è stato supportato da NIH concede R01CA174388 e U54CA143868. Gli autori si desidera ringraziare il premio PURA della Johns Hopkins University per il supporto di Chen Wei-tong. Questo materiale si basa su lavori sostenuta dalla National Science Foundation Graduate Research Fellowship sotto Grant n. 1232825.
Materiali
| Name | Company | Catalog Number | Comments |
| Human embryonic kidney 293T | ATCC | ||
| MDA-MB-231 | Physical Sciences Oncology Center, NIH | ||
| DMEM | Corning | 10-013-CV | |
| DMEM powder | ThermoFisher Scientific | 12100-046 | |
| Fetal bovine serum | Hyclone | SH30910.03 | |
| Penicillin-Streptomycin 100X | Sigma-Aldrich | P0781 | |
| Fugene HD | Promega | E2311 | |
| Lipofectamine 2000 | Life technologies | 11668-07 | |
| Plasmid encoding H2B-mCherry in a lentiviral vector | Addgene | plasmid 21217 | |
| Thymidine | Sigma-Aldrich | T1895 | |
| Nocodazole | Sigma-Aldrich | M1404 | |
| Opti-MEM | Life Technologies | 31985-070 | |
| Sodium bicarbonate | GibcoBRL | 11810-025 | |
| HEPES | Sigma-Aldrich | 113375-100 | |
| Collagen | Corning | 354236 | |
| NaOH | J.T. Bake | 3722-01 | |
| Millex-HV syringe filter unit, 0.45-μm, PVDF, 33 mm | Millipore | SLHVM33RS | |
| Nikon TE2000E epifluorescence microscope | Nikon | TE2000E | |
| Cascade 1K CCD camera | Roper Scientific | ||
| NIS-Elements AR imaging software | Nikon | ||
| Nikon A1 confocal microscope | Nikon | A1 |
Riferimenti
- Ly, D. H., Lockhart, D. J., Lerner, R. A., Schultz, P. G. Mitotic misregulation and human aging. Science. 287 (5462), 2486-2492 (2000).
- Gascoigne, K. E., Taylor, S. S. How do anti-mitotic drugs kill cancer cells? J Cell Sci. 122 (Pt 15), 2579-2585 (2009).
- Brinkley, B. R. Managing the centrosome numbers game: from chaos to stability in cancer cell division. Trends Cell Biol. 11 (1), 18-21 (2001).
- Phillip, J. M., Aifuwa, I., Walston, J., Wirtz, D. The Mechanobiology of Aging. Annu Rev Biomed Eng. 17, 113-141 (2015).
- Hanahan, D., Weinberg, R. A. Hallmarks of cancer: the next generation. Cell. 144 (5), 646-674 (2011).
- Martin, M. D., et al. Effect of ablation or inhibition of stromal matrix metalloproteinase-9 on lung metastasis in a breast cancer model is dependent on genetic background. Cancer Res. 68 (15), 6251-6259 (2008).
- Knox, J. J., Hotte, S. J., Kollmannsberger, C., Winquist, E., Fisher, B., Eisenhauer, E. A. Phase II study of Triapine in patients with metastatic renal cell carcinoma: a trial of the National Cancer Institute of Canada Clinical Trials Group (NCIC IND.161). Invest New Drugs. 25 (5), 471-477 (2007).
- Komlodi-Pasztor, E., Sackett, D. L., Fojo, A. T. Inhibitors targeting mitosis: tales of how great drugs against a promising target were brought down by a flawed rationale. Clin Cancer Res. 18 (1), 51-63 (2012).
- Tong, W. G., et al. Phase I and pharmacologic study of SNS-032, a potent and selective Cdk2, 7, and 9 inhibitor, in patients with advanced chronic lymphocytic leukemia and multiple myeloma. J Clin Oncol. 28 (18), 3015-3022 (2010).
- Matulonis, U. A., et al. Phase II study of MLN8237 (alisertib), an investigational Aurora A kinase inhibitor, in patients with platinum-resistant or -refractory epithelial ovarian, fallopian tube, or primary peritoneal carcinoma. Gynecol Oncol. 127 (1), 63-69 (2012).
- Boss, D. S., et al. Clinical evaluation of AZD1152, an i.v. inhibitor of Aurora B kinase, in patients with solid malignant tumors. Ann Oncol. 22 (2), 431-437 (2011).
- Griffith, L. G., Swartz, M. A. Capturing complex 3D tissue physiology in vitro. Nat Rev Mol Cell Biol. 7 (3), 211-224 (2006).
- Cukierman, E., Pankov, R., Stevens, D. R., Yamada, K. M. Taking cell-matrix adhesions to the third dimension. Science. 294 (5547), 1708-1712 (2001).
- Lu, P., Weaver, V. M., Werb, Z. The extracellular matrix: a dynamic niche in cancer progression. J Cell Biol. 196 (4), 395-406 (2012).
- Giri, A., et al. The Arp2/3 complex mediates multigeneration dendritic protrusions for efficient 3-dimensional cancer cell migration. FASEB J. 27 (10), 4089-4099 (2013).
- Fraley, S. I., Feng, Y., Giri, A., Longmore, G. D., Wirtz, D. Dimensional and temporal controls of three-dimensional cell migration by zyxin and binding partners. Nat Commun. 3, 719(2012).
- Fraley, S. I., et al. A distinctive role for focal adhesion proteins in three-dimensional cell motility. Nat Cell Biol. 12 (6), 598-604 (2010).
- Lesman, A., Notbohm, J., Tirrell, D. A., Ravichandran, G. Contractile forces regulate cell division in three-dimensional environments. J Cell Biol. 205 (2), 155-162 (2014).
- He, L., et al. Local 3D matrix confinement determines division axis through cell shape. Oncotarget. 7 (6), 6994-7011 (2016).
- Held, M., et al. CellCognition: time-resolved phenotype annotation in high-throughput live cell imaging. Nat Methods. 7 (9), 747-754 (2010).
- Fallica, B., Maffei, J. S., Makin, G., Zaman, M. Alteration of cellular behavior and response to PI3K pathway inhibition by culture in 3D collagen gels. PLoS One. 7 (10), e48024(2012).
- Meli, L., Jordan, E. T., Clark, D. S., Linhardt, R. J., Dordick, J. S. Influence of a three-dimensional, microarray environment on human Cell culture in drug screening systems. Biomaterials. 33 (35), 9087-9096 (2012).
- Artym, V. V., Matsumoto, K. Imaging cells in three-dimensional collagen matrix. Curr Protoc Cell Biol. 10, 1-20 (2010).
- Gunzer, M., Kampgen, E., Brocker, E. B., Zanker, K. S., Friedl, P. Migration of dendritic cells in 3D-collagen lattices. Visualisation of dynamic interactions with the substratum and the distribution of surface structures via a novel confocal reflection imaging technique. Adv Exp Med Biol. 417, 97-103 (1997).
- Geraldo, S., Simon, A., Vignjevic, D. M. Revealing the cytoskeletal organization of invasive cancer cells in 3D. J Vis Exp. (80), e50763(2013).
- Lleres, D., James, J., Swift, S., Norman, D. G., Lamond, A. I. Quantitative analysis of chromatin compaction in living cells using FLIM-FRET. J Cell Biol. 187 (4), 481-496 (2009).
- Poincloux, R., et al. Contractility of the cell rear drives invasion of breast tumor cells in 3D Matrigel. Proc Natl Acad Sci U S A. 108 (5), 1943-1948 (2011).
- Carey, S. P., Kraning-Rush, C. M., Williams, R. M., Reinhart-King, C. A. Biophysical control of invasive tumor cell behavior by extracellular matrix microarchitecture. Biomaterials. 33 (16), 4157-4165 (2012).
- Langan, T. J., Chou, R. C. Synchronization of mammalian cell cultures by serum deprivation. Methods Mol Biol. 761, 75-83 (2011).
- Jackman, J., O'Connor, P. M. Methods for synchronizing cells at specific stages of the cell cycle. Curr Protoc Cell Biol. 8, (2001).
- Zwanenburg, T. S. Standardized shake-off to synchronize cultured CHO cells. Mutat Res. 120 (2-3), 151-159 (1983).
- Harper, J. V. Synchronization of cell populations in G1/S and G2/M phases of the cell cycle. Methods Mol Biol. 296, 157-166 (2005).
- Kihara, T., Ito, J., Miyake, J. Measurement of biomolecular diffusion in extracellular matrix condensed by fibroblasts using fluorescence correlation spectroscopy. PLoS One. 8 (11), e82382(2013).
- Ramanujan, S., et al. Diffusion and convection in collagen gels: implications for transport in the tumor interstitium. Biophys J. 83 (3), 1650-1660 (2002).
- Harjanto, D., Maffei, J. S., Zaman, M. H. Quantitative analysis of the effect of cancer invasiveness and collagen concentration on 3D matrix remodeling. PLoS One. 6 (9), e24891(2011).
- Wolf, K., et al. Collagen-based cell migration models in vitro and in vivo. Semin Cell Dev Biol. 20 (8), 931-941 (2009).
- Petroll, W. M. Differential interference contrast and confocal reflectance imaging of collagen organization in three-dimensional matrices. Scanning. 28 (6), 305-310 (2006).
- Fraley, S. I., et al. Three-dimensional matrix fiber alignment modulates cell migration and MT1-MMP utility by spatially and temporally directing protrusions. Sci Rep. 5, 14580(2015).
- Ikeda, Y., Collins, M. K., Radcliffe, P. A., Mitrophanous, K. A., Takeuchi, Y. Gene transduction efficiency in cells of different species by HIV and EIAV vectors. Gene Ther. 9 (14), 932-938 (2002).
- Ma, H. T., Poon, R. Y. Synchronization of HeLa cells. Methods Mol Biol. 761, 151-161 (2011).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon