Method Article
División de célula mamífera en Matrices 3D mediante microscopia de reflexión Confocal cuantitativa
* Estos autores han contribuido por igual
En este artículo
Resumen
Este protocolo eficientemente estudios División de célula mamífera en matrices de colágeno 3D mediante la integración de sincronización de la división celular, monitoreo de eventos de la división de matrices 3D utilizando la técnica de imágenes de células vivas, la microscopia confocal tiempo-resolved de la reflexión cuantitativos y análisis de imágenes.
Resumen
El estudio de mamífero como la división celular está reglamentado en un 3D medio ambiente sigue siendo en gran parte inexplorado a pesar de su importancia fisiológica y significación terapéutica. Posibles razones de la falta de exploración son las limitaciones experimentales y desafíos técnicos que hacen el estudio de la división celular en 3D cultura ineficiente. Aquí, describimos un método basado en la proyección de imagen para estudiar eficientemente mamífera división celular y las interacciones célula-matriz en matrices de colágeno 3D. Las células marcadas con fluorescente H2B se sincronizan mediante la combinación de tratamiento de bloqueo y nocodazole timidina, seguido por una técnica mecánica de agitar. Sincronizado las células entonces están incrustadas en una matriz de colágeno 3D. División de célula se controla mediante microscopía de células vivas. La deformación de las fibras de colágeno durante y después de la división celular, que es un indicador de la célula-matriz de interacción, puede controlarse y cuantificada mediante microscopía de reflexión confocal cuantitativa. El método proporciona un enfoque eficaz y general para estudiar mamífera división celular y las interacciones célula-matriz en un entorno 3D fisiológicamente relevante. Este enfoque no sólo proporciona nuevas penetraciones en la base molecular del desarrollo del tejido normal y las enfermedades, sino que también permite el diseño de nuevos enfoques diagnósticos y terapéuticos.
Introducción
Mitosis de la célula es un evento crítico en la vida celular, la regulación de la que juega un papel crucial en el desarrollo de órganos y tejidos. Mitosis anormales está implicado en las variaciones genéticas naturales, procesos del envejecimiento humano y la progresión del cáncer1,2,3,4,5. El aumento de la tasa de proliferación de células tumorales en comparación con las células normales es una de las características distintivas del cáncer, a pesar de que comportamientos de la célula son muy heterogéneos entre los diferentes tipos de tumores y aún entre los pacientes. A pesar de resultados preclínicos prometedores, algunas drogas antimitótica desarrollado recientemente no han demostrado ser eficaces en ensayos clínicos6,7,8,9,10 ,11. La relevancia de los modelos experimentales y preclínicas debe ser considerada. Muchos tipos de células de mamíferos y cáncer normales se dividen en matrices (3D) tridimensionales, tales como fibroblastos y células de fibrosarcoma en colágeno-ricos 3D tejidos conectivos y células de cáncer metastásico en la matriz extracelular stromal 3D (ECM). Sin embargo, la gran mayoría de mamíferos división celular experimentos y ensayos se han realizado en células cultivadas en substratos bidimensionales de (2D). Una matriz 3D ingeniería mejor podría recapitular la microestructura, propiedades mecánicas y las señales bioquímicas de la ECM 3D de ambos tejidos normales y patológicos12,13,14, 1516,de,17.
El estudio de mamífero como la división celular está reglamentado en 3D entornos sigue siendo en gran parte inexplorado a pesar de la relevancia fisiológica y la importancia terapéutica de18,19. Posibles razones incluyen las dificultades técnicas y desafíos experimentales asociados con el estudio de división celular en matrices 3D. Mitosis de la célula constituye una pequeña fracción temporal en el ciclo celular completo20. Trabajos previos han demostrado que la tasa de proliferación de muchas células mamíferas, tales como adenocarcinoma de mama humano MCF-7 y osteosarcoma humano U2OS hígado humano HepG2, es mucho menor en matrices 3D en comparación con sus contrapartes en sustratos 2D21, 22. Además, las células embebidas en matrices de 3D se mueven dentro y fuera de foco durante la proyección de imagen de células vivas. Todos estos factores contribuyen a la muy baja eficiencia de captura de eventos de la división de célula en cultura 3D utilizando técnicas de imagen.
Interacciones entre las ECM y las células juegan un papel crítico en la regulación de las divisiones de célula. Aquí, describimos un enfoque para el estudio eficiente división de célula mamífera en matrices de colágeno 3D. El método incluye la incorporación de marcadores mitóticas de las células, sincronización de la división celular, así como el seguimiento de eventos de la división de matrices 3D usando una técnica de imágenes de células vivas, la microscopia confocal tiempo-resolved de la reflexión, y Análisis de imagen cuantitativo. Proteína de fluorescencia etiquetada histona H2B se introdujo en las células como marcador para diferenciar mitosis y células de interfase. A continuación, las células están sincronizadas utilizando la combinación de tratamiento de bloqueo y nocodazole timidina, seguido por una técnica mecánica de agitar. Sincronizado las células luego se encapsulan directamente en matrices de colágeno 3D. Eventos de la división celular de células múltiples están controlados eficientemente usando proyección de imagen de células vivas Time-lapse bajo magnificación. La deformación de las fibras de colágeno, que es un indicador de la célula-matriz de interacción, se controla mediante microscopía confocal de la reflexión a alta magnificación.
Anteriormente hemos utilizado esta técnica para controlar y cuantificar la interacción célula-matriz antes, durante y después de la mitosis de dos líneas celulares de cáncer metastásico, carcinoma ductal infiltrante humano MDA-MB-231 y las células humanas de fibrosarcoma HT1080, colágeno 3D matrices de19. Los métodos presentados aquí ofrecen un enfoque eficaz y general para estudiar ambos mamíferos división celular en un 3D interacciones célula-matriz y entorno. La línea de celular MDA-MB-231 se utiliza como un ejemplo en el papel. Este protocolo proporciona nuevas penetraciones en la base molecular del desarrollo del tejido normal y las enfermedades y también podría permitir el diseño de nuevos enfoques diagnósticos y terapéuticos.
Protocolo
El protocolo siempre sigue las pautas de la Homewood institucional Review Board (HIRB).
1. estable expresión de H2B mCherry como marcador de Mitosis celular
-
Generación de partículas lentivirales de riñón embrionario humano 293T (HEK 293T) las células
- Las células HEK 293T en una placa de cultivo celular de 10 cm en la densidad de 5 x 106 células/plato en medio de cultivo celular (de Dulbecco Medium (DMEM) que contienen alta glucosa modificado Eagle (4,5 g/L), piruvato de sodio, 10% suero bovino Fetal (FBS) y 1% de la placa penicilina-estreptomicina (Pen/Strep)). Incubar durante 24 h a 37 ° C y 5% de dióxido de carbono (CO2). La confluencia deseada de las células en el día de la transfección es alrededor del 70-80%.
Nota: Células HEK 293T se utilizan aquí para producir partículas virales, que se utilizará más tarde para transducir las células MDA-MB-231 para generar una línea celular estable expresando mCherry H2B. Asegúrese de que las células se distribuyen uniformemente a lo largo de la placa. Además de las células de la gota sabio a la placa, y suavemente moviendo la placa hacia adelante y hacia atrás puede ayudar en la distribución. Las células también pueden ser sembradas en placas o frascos de otros tamaños, que resultarán en la recogida de diferentes volúmenes de partículas virales. - Transfectar las células HEK 293T con un reactivo de transfección de tres plásmidos (vectores lentivirales, CMV ΔR 8.91 y pMDG VSVG). El plásmido codificar mCherry H2B se clona en un vector lentivirales con el promotor de la cinasa de phosphoglycerate (PGK). Por CMV ΔR 8.91 contiene tres necesaria VIH genes gag, poly rev. pMDG VSVG contiene el gen de la envoltura de VSV-G.
Nota: Hay múltiples agentes de transfección para elegir.- Deje que el frasco de reactivo de transfección para alcanzar la temperatura ambiente (RT) antes de uso. Invertir o vórtex brevemente el frasco.
- Mezcla 16 μg de DNA, que consta de 6 μg de plásmido H2B mCherry, 8 μg de CMV ΔR 8.91 y 2 μg de pMDG-VSVG, en 1 mL de suero reducidos medios. Incubar a temperatura ambiente durante 5 minutos.
Nota: La cantidad y proporción de los tres vectores es variado y optimizado para vectores de transferencia lentivirales diferentes. - Añadir 48 μl (utilizar relación 1:3 del ADN: reactivo de transfección) el reactivo de transfección en la solución anterior de ADN y luego incubar a temperatura ambiente durante al menos 15 minutos.
Nota: La relación entre el ADN vs reactivo de transfección es variada y optimizada para vectores de transferencia lentivirales diferentes. Asegúrese de que el reactivo de transfección no tenga contacto con el lado del tubo de centrífuga de 1.5 mL. - Añadir la solución de ADN-lípido complejo hecha en paso 1.1.2.3 mediante goteo a las células. Agitar suavemente la placa para asegurar la distribución uniforme del complejo de la placa.
- Aproximadamente 6 h después de la transfección, Aspire el medio y agregar medio de cultivo celular fresco (DMEM con glucosa alta (4,5 g/L), piruvato sódico, 10% FBS y 1% pluma/Strep).
- Coseche el sobrenadante 24, 48 y transfección después de 72 h. Reemplazar el medio después de cada cosecha (DMEM, 4,5 g/L glucosa, piruvato de sodio, con 10% FBS y 1% pluma/Strep).
- Filtrar las partículas lentivirales a través de un filtro de 0.45 μm para eliminar los desechos celulares. Por otra parte, girar por el sobrenadante para separar los desechos de la célula. El sobrenadante puede utilizarse para la transducción de inmediato, o puede ser almacenado a-80 ° C.
- Las células HEK 293T en una placa de cultivo celular de 10 cm en la densidad de 5 x 106 células/plato en medio de cultivo celular (de Dulbecco Medium (DMEM) que contienen alta glucosa modificado Eagle (4,5 g/L), piruvato de sodio, 10% suero bovino Fetal (FBS) y 1% de la placa penicilina-estreptomicina (Pen/Strep)). Incubar durante 24 h a 37 ° C y 5% de dióxido de carbono (CO2). La confluencia deseada de las células en el día de la transfección es alrededor del 70-80%.
-
Generación de la célula líneas estable expresando H2B-mCherry
Nota: Este protocolo se describe en detalle para las células MDA-MB-231. Antes de usar en el experimento, las células de carcinoma de mama MDA-MB-231 (centro de Oncología de ciencias física, NIH) son cultivadas en DMEM que contenía glucosa alta (4,5 g/L), piruvato sódico, 10% FBS y 1% pluma/Strep.- Las células MDA-MB-231 en la densidad de 1 x 105 células en una placa de cultivo de 35 mm de la placa.
Nota: La densidad de la galjanoplastia para diferentes líneas celulares debe ser optimizada y por lo tanto puede ser diferente de la densidad óptima para las células MDA-MB-231. - 24 h después de platear las células, agregar 1 mL de virus y 1 mL de medio fresco (DMEM, glucosa alta (4,5 g/L), piruvato sódico, 10% FBS y 1% pluma/Strep).
- Incubar las células durante un período de tiempo de 6 h hasta toda la noche.
Nota: Tanto el volumen del virus y el tiempo de incubación necesario ser optimizado para diferentes líneas celulares. Por ejemplo, si las células no se ven saludables unas horas después de la adición del virus, utilizar 1 mL de virus y 2 mL de medio fresco (DMEM, glucosa alta (4,5 g/L), piruvato sódico, 10% FBS y 1% pluma/Strep) paso 1.2.2. También puede reducirse la duración de la incubación en paso 1.2.3. - Aspire el medio y añadir 10 mL de medio fresco (DMEM, glucosa alta (4,5 g/L), piruvato sódico, 10% FBS y 1% pluma/Strep). Incubar entre 24 a 72 h a 37 ° C y 5% CO2.
- Controlar la expresión de H2B-mCherry en las células usando un microscopio de epifluorescencia.
Nota: Si la eficiencia de transducción es baja en las células, podría incrementarse el volumen de virus en el paso 2.2 y el tiempo de incubación en el paso 2.3.
- Las células MDA-MB-231 en la densidad de 1 x 105 células en una placa de cultivo de 35 mm de la placa.
2. sincronización de las células expresando estable H2B mCherry
- Las celdas de 50 a 60% confluency, es decir, placa de 2 x 104 MDA-MB-231 células en cada pozo de una placa de 24 pocillos de la placa.
- Incubar el cultivo a 37 ° C y 5% de CO2 durante 24 h.
- Reemplazar el medio de crecimiento con 0,5 mL de medio (DMEM, glucosa alta (4,5 g/L), piruvato sódico, 10% FBS y 1% pluma/Strep) que contiene timidina de 2 mM y dejar en la incubadora durante 24 h.
Nota: Las células expuestas a timidina son detenidas en la fase de crecimiento celular (G1) / transición de síntesis (S) de ADN y fase S debido a la inhibición de la síntesis de ADN. La duración de la incubación debe ser variada y optimizada para diferentes líneas celulares. - Liberar las células de la exposición de la timidina por lavarlas tres veces con tampón fosfato salino (PBS). Luego, incubar las células en el medio de cultivo celular normal (DMEM, glucosa alta (4,5 g/L), piruvato sódico, 10% FBS y 1% pluma/Strep) de 5 horas.
Nota: La liberación de las células de la exposición de timidina permite a las células para progresar en el crecimiento de la célula (G2) y mitosis (M) fase de células previamente detenido en la fase G1/S, a la fase G1 de las células previamente detenido en la S de la fase. La longitud de tiempo de liberación debe ser variada y optimizada para diferentes líneas celulares. - Bloquear las células con 250 ng/mL de nocodazole durante 12 h.
Nota: Todas las células expuestas a nocodazole están detenidos en la fase G2/M. Nocodazole es citotóxico. La exposición prolongada a nocodazole puede causar apoptosis. Ajustar el período o la concentración de exposición para diferentes líneas celulares si se observan las muertes celulares. Las células que se sincronizan con éxito presenta una morfología esférica. - Agitar las células para 45 s a 1 min utilizando un agitador orbital a 150 a 200 rpm.
Nota: Las células mitóticas, que tienen poca adherencia al sustrato, serán sacudidas durante el proceso. - Quitarlo del medio para extraer las células, transfiriendo el medio en un tubo de centrífuga y luego añadir 0,5 mL de medio fresco (DMEM, glucosa alta (4,5 g/L), piruvato sódico, 10% FBS y 1% pluma/Strep) a cada pocillo de la placa.
- Repita los pasos tres veces 2.6 y 2.7.
- Centrifugar el medio recogido que contiene las células mitóticas a 800 x g durante 3 minutos.
Nota: Este paso se utiliza para quitar el nocodazole del medio celular.
3. incorporación de la sincronizada células en el colágeno Matrices
Nota: El tipo I colágeno es la proteína más abundante en el cuerpo humano y en la MEC de los tejidos conectivos y por lo tanto es ampliamente utilizado para investigar cómo eukaryotic célula funciones son moduladas por un entorno 3D17,23,24 . El colágeno es soluble en ácido acético. Después de la neutralización y el calentamiento de la solución de colágeno al 20-37 ° C, monómeros de colágeno polimerizan en una red de fibrillas de colágeno.
- Preparar la solución x DMEM 10 por disolver un paquete de polvo DMEM, 3,7 g de bicarbonato de sodio (NaHCO3) y 1 g de ácido de-(2-hydroxyethyl)-1--1-ácido 4 (HEPES) en 50 mL de agua destilada. Filtrar la solución y luego preparar 1 M de hidróxido de sodio (NaOH) disolviendo 2 g de pellets de NaOH en 50 mL de agua destilada. Filtro y la parte alícuota de la solución en tubos de centrífuga de 1.5 mL.
Nota: Solución DMEM Normal no debe ser utilizada en esta etapa. La adición de un volumen significativo de la solución de colágeno diluye el medio. Por lo tanto la solución concentrada de DMEM está preparada para asegurar que la concentración final de DMEM en la matriz de colágeno será el mismo que el DMEM normal. - Continúan trabajando con las células de paso 2.9. Aspire el medio y resuspender las células en unos 0.25 - 0.5 mL de medio de cultivo celular fresco (DMEM, glucosa alta (4,5 g/L), piruvato sódico, 10% FBS y 1% pluma/Strep).
Nota: Para llegar a una densidad específica de la célula en la matriz de colágeno, la densidad inicial de las células en la suspensión no puede ser demasiado baja. Así, el volumen de los medios utilizados para resuspender las células dependerá de la cantidad de células disponibles. - Lugar 10 μl de la solución celular suspende de nuevo desde el paso 3.2 un hemocitómetro y la densidad de las células en la solución.
-
Determinar los volúmenes necesarios para que todos los componentes hacer la matriz de colágeno 3D. 500 μl de 2 gel de colágeno μg/μl se utiliza aquí como ejemplo.
- Calcular el volumen de la solución de células necesaria para la obtención de 40.000 células/mL (o 20.000 células para 500 μl de gel de colágeno). Este número en μL es X. 50 μl de 10 x se necesitarán DMEM. Se necesitarán 50 μl de FBS.
- Calcular el volumen de colágeno que stock necesitada. La concentración de colágeno es alrededor de 4 μg/μl. Es la cantidad de colágeno en μl.
- Calcular el volumen de hidróxido de sodio necesario neutralizar el ácido acético en la solución de colágeno: colágeno de μl Y * 0.023 * 1000 = Z μl de NaOH necesarios.
- Determinar el volumen de la solución de relleno: (500 μl 50 total gel - células X µL - de μl 10 X colágeno μl de DMEM - 50 μl de FBS - Y - Z μl NaOH) = μl de R
Nota: La solución de relleno se destila agua. Si el volumen calculado de la solución de relleno es un número negativo, la densidad de la célula original en la suspensión de células es demasiado baja. Las células deben girar hacia abajo otra vez y resuspendió en un volumen pequeño de medio (DMEM, glucosa alta (4,5 g/L), piruvato sódico, 10% FBS y 1% pluma/Strep).
- Añadir los componentes en el orden siguiente para un tubo de centrífuga limpio de 1,5 mL: células en medio, 10 x DMEM, SBF, agua desionizada (DI), colágeno, NaOH. Coloque todos los componentes en hielo para retrasar la gelificación de la solución de colágeno. Utilizar técnicas apropiadas de pipeteo para evitar la formación de burbujas.
- Después de agregar el NaOH, mezclar la solución cuidadosamente con una pipeta de 1 mL.
Nota: Gelificación del colágeno se iniciará justo inmediatamente después de la adición de NaOH. Mezcla debe hacerse con cuidado y rápidamente. - Una vez que la solución esté bien mezclada, Añadir 500 μl de la solución en cada pocillo de la placa de 24 pocillos. Coloque la placa de 24 pocillos en la incubadora de 37 ° C. Coloque el medio de cultivo celular fresco en el baño de agua de 37 ° C.
Nota: Las placas inferiores plástico se utilizan para baja magnificación Microcopia de células vivas en que la distancia de la lente está por encima de 1 mm. parte inferior de cristal se utilizan las placas para microscopía de células vivas de alta magnificación debido a la corta distancia de trabajo de la alta lente de aumento. - Añadir 500 μl de medio precalentado (DMEM, glucosa alta (4,5 g/L), piruvato sódico, 10% FBS y 1% pluma/Strep) en la parte superior del gel 30 minutos después de completar el paso 3.7.
4. proyección de imagen de célula de las células en división en las Matrices de colágeno 3D (bajos aumentos) en vivo
Nota: Imágenes de las células se recogen a intervalos de 2 minutos usando una cámara de dispositivo acoplado (CCD) de carga montada en un microscopio de contraste de fase que está equipado con un objetivo de X 10 y controlado por el software de imágenes.
- Encienda la unidad de células vivas en la parte superior de la lente del objetivo. Espere hasta que la temperatura se estabiliza a 37 ° C, la concentración de CO2 es 5% y la humedad es 75%.
- Poner la placa de 24 pocillos de geles en la unidad de células vivas en el microscopio. Encontrar la parte inferior de la placa y luego mueva el objetivo hasta que el microscopio se centra en alrededor de 500 μm de la parte inferior de la placa.
Nota: Las células en la matriz de colágeno que están muy cerca la parte inferior de la placa no están totalmente incrustados en la matriz 3D. Las células que son 500 μm de la parte inferior de la placa de la proyección de imagen se asegurarán de que las células no son afectadas por efectos de borde16,17,25. - Mover el escenario para encontrar las células y múltiples posiciones para la proyección de imagen, seleccione.
- Establecer el lapso de tiempo experimento a intervalos de 2 min.
Nota: El número máximo de posiciones que se pueden tomar durante el intervalo de 2 min es limitado por la velocidad de movimiento de la etapa motorizada y la duración de la exposición para capturar imágenes.
5. colágeno red deformación durante la división celular (microscopio de alta magnificación)
- Encienda la unidad de células vivas en la parte superior de la lente del objetivo. Espere hasta que la temperatura se estabiliza a 37 ° C, la concentración de CO2 es 5% y la humedad es 75%.
- Para visualizar las fibras de colágeno en matrices de colágeno 3D sin manchas, configurar un microscopio confocal para capturar solamente luz reflejada (488 nm) de la láser de 488 nm sirve para iluminar la muestra. El láser utiliza un 60 X objetivo de inmersión en agua, NA = 1.2, WD = 200 μm y es controlado por el software de imágenes.
- Poner la muestra (células en matrices de colágeno) en la unidad de células vivas. Encontrar la parte inferior de la placa y luego mueva el objetivo hasta que el microscopio se centra en cerca de 100 μm de la parte inferior de la placa. Las células que son 100 μm de la parte inferior de la placa de la proyección de imagen se asegurarán de que las células no son afectadas por efectos de borde.
Nota: Aquí un objetivo de inmersión en agua se utiliza en lugar de una lente de inmersión en aceite porque la distancia de la lente de inmersión en aceite es solamente cerca de 100 μm. El uso de la lente de inmersión en aceite plantea un problema como el espesor del vidrio en la parte inferior de la placa es normalmente alrededor de 100 μm. - Mover el escenario para encontrar las células y múltiples posiciones para la proyección de imagen, seleccione.
Nota: La microscopia Confocal ópticamente las secciones de la muestra y sólo capta señales desde el plano focal. Células vivas podrían ser móviles dentro y fuera de foco durante el experimento de Time-lapse. Para habilitar la captura de imágenes de células durante largos períodos de tiempo, se utiliza Z-pila para recoger imágenes de rebanadas sucesivas a intervalos 5 μm. Son imágenes de un total de 5 segmentos que abarca 25 μm. - Establecer el lapso de tiempo experimento a intervalos de 5 minutos.
Nota: 5 minutos se utiliza aquí como el intervalo en lugar de 2 min utilizado en la sección 4 porque se aplican múltiples modos de escaneo, incluyendo exploración H2B mCherry, análisis de reflexión para colágeno y exploración de Z-stack, de la fluorescencia para cada posición. El uso de múltiples modos de escaneo reduce la velocidad de exploración de una posición en comparación con la proyección de imagen de ampliación baja. - Cuantificar la deformación de la matriz del colágeno en las cercanías de una célula debido a las fuerzas ejercidas por ese celular usando partículas imaging velocimetry (PIV) con sub-pixel de resolución20. Realizar este análisis en imágenes 2D permite la clara visualización de ambos las señales de la mCherry H2B y las fibras de colágeno. Aplique el paso bajo anisotrópico filtrado para mejorar la señal de la red de colágeno20.
- Para medir el desplazamiento local de una región de subimagen de interés situado en (x,y) de marco k a marco k+ 1, extraer una ventana regional de 15 × 15 píxeles centran en (x,y) en el marco de la k. Identificar la mejor coincidencia de puntos (x,y) * a través de los lugares que cuentan con un coeficiente de correlación cruzada normalizado máxima en la imagen obtenida en el marco de la k+ 1. El vector de deformación se calcula como (x,y) *-(x,y).
Resultados
El objetivo de este artículo es presentar un método basado en la proyección de imagen para el estudio de procesos de división de célula mamífera en matrices 3D y para cuantificar las interacciones entre la célula y la matriz extracelular 3D durante y después de la división celular. Para facilitar la proyección de imagen de la mitosis celular, incorporamos mCherry H2B en las células MDA-MB-231 mediante transducción lentivirales. H2B conjugados con proteínas fluorescentes se utiliza como un marcador mitótico para distinguir las células mitóticas de las células de la interfase y definir diferentes etapas durante la mitosis de la célula19,20,26. Usando este método, hemos sido capaces de controlar el proceso entero de la división de las células MDA-MB-231 estable expresaban H2B-mCherry en matrices de colágeno 3D (figura 1A). Fases mitóticas empezaron con la disolución de la membrana nuclear (profase, cuadro 1, figura 1A); la reorganización de los cromosomas (Prometafase: Capítulo 2); la alineación de los cromosomas en el cuerpo de la célula (metafase; cuadro 3); la separación de los cromosomas (anafase; cuadro 4); la reorganización de los cromosomas, la membrana nuclear y la separación de los cuerpos de las dos células hijas (telofase/citocinesis, cuadro 5).
La tasa de proliferación de células en una matriz 3D es generalmente mucho más baja que sus contrapartes en un sustrato de 2D, que hace el seguimiento de la división celular en 3D menos eficientes19. Para aumentar la eficacia de estudiar 3D la división celular, se empleó un método de combinación de timidina, nocodazole y una técnica de movimiento para sincronizar y seleccionar las células MDA-MB-231 que se encuentran en la fase mitótica. Células sincronizadas fueron encaja entonces en matrices de colágeno. El proceso de división de las células múltiples fue monitoreado en tiempo real usando proyección de imagen de células vivas con 10 aumentos. Alrededor del 70% de las células se dividen en las primeras 2 h después de la formación de la matriz de colágeno (figura 2), lo que permite una supervisión eficaz de la mitosis en 3D (figura 2).
Para monitorear la interacción entre las células y sus matrices de colágeno circundante, hemos combinado la microscopia confocal de la reflexión a las fibras de colágeno de la imagen y microscopía de fluorescencia en las células de la imagen. Una lente X 60 permite la captura de imágenes de alta calidad de fibras de colágeno. Proyección de imagen usando un lente de alta magnificación, que captura las células menos en cada campo de visión, es mucho menor rendimiento en comparación con el uso de la baja magnificación 10 X o 20 X. Sincronización exitosa de las células mejora en gran medida la eficiencia y el rendimiento de un experimento así, ya que la mayoría de las células sincronizadas se divide en las primeras 2 h después de la formación de la matriz de colágeno. La deformación de la matriz durante y después de la división celular es visualizado (representante de instantáneas se muestran en la figura 1B) y cuantificada mediante un software personalizado de PIV. Cuantificado y en comparación con la deformación de la matriz durante la interfase y la fase mitótica de células sincronizadas en la fase mitótica. Se observa que la deformación de la matriz cambia muy poco durante la mitosis de fase (Figura 3A), y es menor la deformación observada en fases poste-mitotic (figura 3B). Este resultado muestra que las células mamíferas tienen apego mínimo y las interacciones con la matriz circundante mientras están en la fase mitótica.
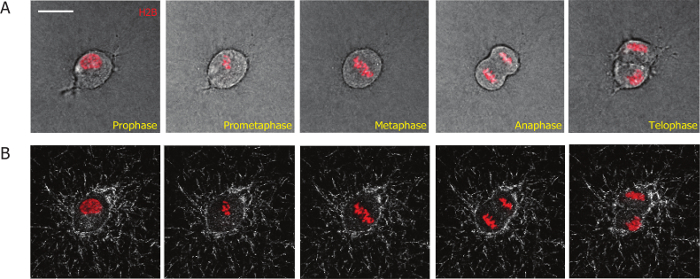
Figura 1: micrografías representativas obtenidas de un video imágenes de células vivas de alta magnificación de un celular MDA-MB-231 embebidas en una matriz de colágeno que expresa estable mCherry H2B. (A) diferentes fases de la progresión mitótica de las células MDA-MB-231 se definen por H2B mCherry tal como se indica en rojo. (B) las fibras de colágeno (blanco) durante el proceso mitótico se visualizan por microscopia de reflexión confocal dependiente del tiempo. Barra de escala = 20 μm. haga clic aquí para ver una versión más grande de esta figura.

Figura 2: División de las células sincronizadas en matrices de colágeno 3D. Las células fueron sincronizadas en fase G2/M y embebidas en una matriz de colágeno. Alrededor del 70% de las células sincronizadas se dividen en las primeras 2 h, mientras que control de células sin división de sincronización al azar. Barra de error = SEM (error estándar de la media). Haga clic aquí para ver una versión más grande de esta figura.
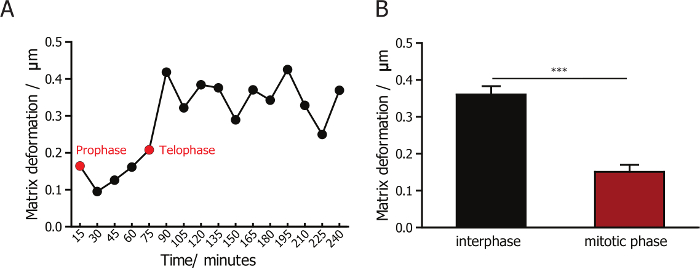
Figura 3: cuantificación de la deformación de la matriz para MDA-MB-231 células en interfase y mitosis. (A) el cambio en la magnitud de la deformación de la matriz para las células MDA-MB-231 embebido en matriz. La flecha verde indica profase y telofase flecha roja. (B) cuantificación de la deformación de la matriz para las células MDA-MB-231 durante la interfase y la fase mitótica, lo que indica que la deformación de la matriz es mínima durante la división celular. Barra de error = SEM (error estándar de la media). * p < 0.05. Haga clic aquí para ver una versión más grande de esta figura.
Discusión
El estudio previo de la división celular en 3D no fue eficiente debido a limitaciones experimentales y desafíos técnicos18,19. Los pasos críticos para el estudio eficaz de la división de célula mamífera en matrices de colágeno 3D son: (1) la incorporación de marcadores mitotic marcados con fluorescencia en las células; (2) la sincronización de la división celular; y (3) el monitoreo de eventos de la división de matrices 3D utilizando la técnica de imágenes de células vivas, la microscopia confocal tiempo-resolved de la reflexión y análisis de imagen cuantitativo.
Las células mitotic en sustratos 2D pueden distinguirse de las células de la interfase basadas en su morfología, es decir, las células mitotic son redondas y apenas fijar al sustrato mientras que las células de la interfase extendieron y sujete firmemente al sustrato. En matrices 3D, sin embargo, la morfología de la célula no es un marcador confiable para las células mitotic desde algunas células apenas estirar y permanecen en la matriz27,28. Por lo tanto, es esencial introducir un marcador mitótico en las células para el estudio de la división celular en matrices de 3D. Estable expresamos H2B-mCherry en las células MDA-MB-231, que sirvió como un marcador confiable para diferentes fases mitóticas incluyendo profase, Prometafase, metafase, anafase y telofase/citocinesis. Anteriormente utilizamos este enfoque para distinguir las células mitóticas de las células de la interfase en matrices 3D. Con la ayuda de este marcador, pudimos también medir la longitud de la fase mitotic para otra línea de celular, las células HT1080, dividiendo en los substratos 2D y en 3D matrices19.
Hay múltiples maneras de sincronizar las células, incluyendo suero hambre29, mitotic shake off30,31, timidina doble bloqueo32y nocodazole32. Hemos combinado el tratamiento de la timidina y el tratamiento nocodazole sincronizar eficientemente las células MDA-MB-231 a la fase G2/M. Las células expuestas a timidina son arrestadas en la transición G1/S y en fase de S debido a la inhibición de la síntesis de ADN por timidina. La liberación de las células de la exposición de timidina permite el avance de las células G1 las células detenidas en fase S y fase G2/M para las células detenidas en fase G1/S. Todas las células expuestas a nocodazole están detenidas en la fase G2/M. Las células redondeadas hacia arriba entrando en fase mitótica fueron entonces sacudarido apagado de la placa y encapsuladas directamente en las matrices de colágeno. Nos mostró que alrededor del 70% de las células se dividen en 2 h después se incorporan en las matrices de colágeno (figura 2). Como alternativa, las células podrían embebidas en matrices de colágeno antes de sincronización, sin embargo, la red entrelazada de la matriz de colágeno presenta una barrera física y reduce la tasa de biomolecular difusión y convección33, 34. de hecho, intentó sincronizar las células en matrices de colágeno con timidina y nocodazole, pero no pudo obtener sincronización eficiente. Este resultado podría ser debido a la ineficiente difusión y convección de las drogas a través de la matriz del colágeno.
La reflexión es una propiedad óptica intrínseca de muchos biopolímeros, incluyendo colágeno. La técnica de microscopía confocal de la reflexión visualiza y quantitates la microtopografía de biomateriales porosos preparados a partir de polímeros sintéticos y colágeno 3D matrices23,24,35,36 ,37. En nuestro laboratorio, hemos establecido técnicas para monitorear los cambios en la polaridad de la fibra de colágeno como la concentración de colágeno varía de38. Aquí, describimos el método para controlar la deformación de las fibras de colágeno basado en el vídeo Time-lapse de las imágenes confocales reflectantes para denotar la célula-matriz de interacción. Los resultados representativos presentados aquí demuestran que la deformación de la matriz para las células MDA-MB-231 mitotic es significativamente menor que las células en interfase, lo que sugiere que las células mamíferas tienen mínimo apego e interacción con el matriz circundante cuando entran en la fase mitotic19.
Anteriormente, se utilizó microscopía confocal de la reflexión para controlar y cuantificar la interacción célula-matriz antes, durante y después de la mitosis de las células HT1080. También supervisamos la deformación de la matriz por las células HT1080 de integrina β1 precipitación durante la interfase y la fase mitótica. Agotamiento de integrina β1 significativamente reduce la deformación de la matriz por la célula durante la interfase. Sin embargo, no hay ninguna diferencia en la deformación de la matriz durante la fase mitótica de las células de caída (KD) ronda β1-integrina y el de células de tipo salvaje HT108019.
Un enfoque alternativo para visualizar las fibras de colágeno es emplear conjugado fluorescencia colágeno tipo I. Anteriormente utilizamos este enfoque a pistas de imagen en las matrices de colágeno generadas por células19. Sin embargo, este enfoque requiere el etiquetado de colágeno con colorante fluorescente como el isotiocianato de fluoresceína (FITC), que es lento y menos eficiente. Por otro lado, la microscopia confocal de la reflexión puede ser aplicada directamente al colágeno sin modificar para ahorrar tiempo y recursos y excluir los problemas asociados con fluorescencia fotoblanqueo. Además, este método no requiere un canal fluorescente individual y por lo tanto es compatible con todos los tintes fluorescentes.
El método presentado en este documento puede aplicarse potencialmente a cualquier tipo de células de mamífero que dividir en una matriz de colágeno 3D. La incorporación del marcador mCherry H2B en otras células de mamífero por transducción lentivirales seguirá exactamente el mismo procedimiento como se describe en el documento, aunque diferentes tipos de células pueden tienen diferentes eficiencias para la transducción por lentivirus 39. tanto la densidad de las células sobre la transducción de señales y el título del virus podrían ser optimizados para la eficiencia. Si no se logra eficiencia de transducción alta, las células puede seleccionarse por célula asistida por fluorescencia (FACS) de clasificación. Bloqueo de timidina y nocodazole se aplican para sincronizar con éxito otros tipos de células de mamífero, como HeLa40. Batido mecánico apagado podría aplicarse a cualquier tipo de célula que redondear y apenas agregar al sustrato durante la fase mitótica30,31. Por otra parte, la proyección de imagen de fibras de colágeno mediante microscopia confocal de la reflexión y la cuantificación de la deformación de la matriz puede ser aplicada directamente a todos los demás tipos de dividir las células mamíferas.
El método presentado aquí es un enfoque eficaz y general para el estudio de mamíferos la división celular y las interacciones célula-matriz en un entorno 3D. El enfoque facilita la sonda en la base molecular del desarrollo del tejido normal y las enfermedades y potencializa el diseño de la novela diagnóstico y enfoques terapéuticos en el futuro.
Divulgaciones
Los autores declaran que no tienen intereses financieros que compiten.
Agradecimientos
Este trabajo fue financiado por los NIH concede R01CA174388 y U54CA143868. Los autores desean reconocer la concesión PURA de la Universidad de Johns Hopkins para la ayuda de Chen Wei tong. Este material está basado en trabajo apoyado por la nacional ciencia Fundación graduados beca de investigación bajo la subvención no. 1232825.
Materiales
| Name | Company | Catalog Number | Comments |
| Human embryonic kidney 293T | ATCC | ||
| MDA-MB-231 | Physical Sciences Oncology Center, NIH | ||
| DMEM | Corning | 10-013-CV | |
| DMEM powder | ThermoFisher Scientific | 12100-046 | |
| Fetal bovine serum | Hyclone | SH30910.03 | |
| Penicillin-Streptomycin 100X | Sigma-Aldrich | P0781 | |
| Fugene HD | Promega | E2311 | |
| Lipofectamine 2000 | Life technologies | 11668-07 | |
| Plasmid encoding H2B-mCherry in a lentiviral vector | Addgene | plasmid 21217 | |
| Thymidine | Sigma-Aldrich | T1895 | |
| Nocodazole | Sigma-Aldrich | M1404 | |
| Opti-MEM | Life Technologies | 31985-070 | |
| Sodium bicarbonate | GibcoBRL | 11810-025 | |
| HEPES | Sigma-Aldrich | 113375-100 | |
| Collagen | Corning | 354236 | |
| NaOH | J.T. Bake | 3722-01 | |
| Millex-HV syringe filter unit, 0.45-μm, PVDF, 33 mm | Millipore | SLHVM33RS | |
| Nikon TE2000E epifluorescence microscope | Nikon | TE2000E | |
| Cascade 1K CCD camera | Roper Scientific | ||
| NIS-Elements AR imaging software | Nikon | ||
| Nikon A1 confocal microscope | Nikon | A1 |
Referencias
- Ly, D. H., Lockhart, D. J., Lerner, R. A., Schultz, P. G. Mitotic misregulation and human aging. Science. 287 (5462), 2486-2492 (2000).
- Gascoigne, K. E., Taylor, S. S. How do anti-mitotic drugs kill cancer cells? J Cell Sci. 122 (Pt 15), 2579-2585 (2009).
- Brinkley, B. R. Managing the centrosome numbers game: from chaos to stability in cancer cell division. Trends Cell Biol. 11 (1), 18-21 (2001).
- Phillip, J. M., Aifuwa, I., Walston, J., Wirtz, D. The Mechanobiology of Aging. Annu Rev Biomed Eng. 17, 113-141 (2015).
- Hanahan, D., Weinberg, R. A. Hallmarks of cancer: the next generation. Cell. 144 (5), 646-674 (2011).
- Martin, M. D., et al. Effect of ablation or inhibition of stromal matrix metalloproteinase-9 on lung metastasis in a breast cancer model is dependent on genetic background. Cancer Res. 68 (15), 6251-6259 (2008).
- Knox, J. J., Hotte, S. J., Kollmannsberger, C., Winquist, E., Fisher, B., Eisenhauer, E. A. Phase II study of Triapine in patients with metastatic renal cell carcinoma: a trial of the National Cancer Institute of Canada Clinical Trials Group (NCIC IND.161). Invest New Drugs. 25 (5), 471-477 (2007).
- Komlodi-Pasztor, E., Sackett, D. L., Fojo, A. T. Inhibitors targeting mitosis: tales of how great drugs against a promising target were brought down by a flawed rationale. Clin Cancer Res. 18 (1), 51-63 (2012).
- Tong, W. G., et al. Phase I and pharmacologic study of SNS-032, a potent and selective Cdk2, 7, and 9 inhibitor, in patients with advanced chronic lymphocytic leukemia and multiple myeloma. J Clin Oncol. 28 (18), 3015-3022 (2010).
- Matulonis, U. A., et al. Phase II study of MLN8237 (alisertib), an investigational Aurora A kinase inhibitor, in patients with platinum-resistant or -refractory epithelial ovarian, fallopian tube, or primary peritoneal carcinoma. Gynecol Oncol. 127 (1), 63-69 (2012).
- Boss, D. S., et al. Clinical evaluation of AZD1152, an i.v. inhibitor of Aurora B kinase, in patients with solid malignant tumors. Ann Oncol. 22 (2), 431-437 (2011).
- Griffith, L. G., Swartz, M. A. Capturing complex 3D tissue physiology in vitro. Nat Rev Mol Cell Biol. 7 (3), 211-224 (2006).
- Cukierman, E., Pankov, R., Stevens, D. R., Yamada, K. M. Taking cell-matrix adhesions to the third dimension. Science. 294 (5547), 1708-1712 (2001).
- Lu, P., Weaver, V. M., Werb, Z. The extracellular matrix: a dynamic niche in cancer progression. J Cell Biol. 196 (4), 395-406 (2012).
- Giri, A., et al. The Arp2/3 complex mediates multigeneration dendritic protrusions for efficient 3-dimensional cancer cell migration. FASEB J. 27 (10), 4089-4099 (2013).
- Fraley, S. I., Feng, Y., Giri, A., Longmore, G. D., Wirtz, D. Dimensional and temporal controls of three-dimensional cell migration by zyxin and binding partners. Nat Commun. 3, 719(2012).
- Fraley, S. I., et al. A distinctive role for focal adhesion proteins in three-dimensional cell motility. Nat Cell Biol. 12 (6), 598-604 (2010).
- Lesman, A., Notbohm, J., Tirrell, D. A., Ravichandran, G. Contractile forces regulate cell division in three-dimensional environments. J Cell Biol. 205 (2), 155-162 (2014).
- He, L., et al. Local 3D matrix confinement determines division axis through cell shape. Oncotarget. 7 (6), 6994-7011 (2016).
- Held, M., et al. CellCognition: time-resolved phenotype annotation in high-throughput live cell imaging. Nat Methods. 7 (9), 747-754 (2010).
- Fallica, B., Maffei, J. S., Makin, G., Zaman, M. Alteration of cellular behavior and response to PI3K pathway inhibition by culture in 3D collagen gels. PLoS One. 7 (10), e48024(2012).
- Meli, L., Jordan, E. T., Clark, D. S., Linhardt, R. J., Dordick, J. S. Influence of a three-dimensional, microarray environment on human Cell culture in drug screening systems. Biomaterials. 33 (35), 9087-9096 (2012).
- Artym, V. V., Matsumoto, K. Imaging cells in three-dimensional collagen matrix. Curr Protoc Cell Biol. 10, 1-20 (2010).
- Gunzer, M., Kampgen, E., Brocker, E. B., Zanker, K. S., Friedl, P. Migration of dendritic cells in 3D-collagen lattices. Visualisation of dynamic interactions with the substratum and the distribution of surface structures via a novel confocal reflection imaging technique. Adv Exp Med Biol. 417, 97-103 (1997).
- Geraldo, S., Simon, A., Vignjevic, D. M. Revealing the cytoskeletal organization of invasive cancer cells in 3D. J Vis Exp. (80), e50763(2013).
- Lleres, D., James, J., Swift, S., Norman, D. G., Lamond, A. I. Quantitative analysis of chromatin compaction in living cells using FLIM-FRET. J Cell Biol. 187 (4), 481-496 (2009).
- Poincloux, R., et al. Contractility of the cell rear drives invasion of breast tumor cells in 3D Matrigel. Proc Natl Acad Sci U S A. 108 (5), 1943-1948 (2011).
- Carey, S. P., Kraning-Rush, C. M., Williams, R. M., Reinhart-King, C. A. Biophysical control of invasive tumor cell behavior by extracellular matrix microarchitecture. Biomaterials. 33 (16), 4157-4165 (2012).
- Langan, T. J., Chou, R. C. Synchronization of mammalian cell cultures by serum deprivation. Methods Mol Biol. 761, 75-83 (2011).
- Jackman, J., O'Connor, P. M. Methods for synchronizing cells at specific stages of the cell cycle. Curr Protoc Cell Biol. 8, (2001).
- Zwanenburg, T. S. Standardized shake-off to synchronize cultured CHO cells. Mutat Res. 120 (2-3), 151-159 (1983).
- Harper, J. V. Synchronization of cell populations in G1/S and G2/M phases of the cell cycle. Methods Mol Biol. 296, 157-166 (2005).
- Kihara, T., Ito, J., Miyake, J. Measurement of biomolecular diffusion in extracellular matrix condensed by fibroblasts using fluorescence correlation spectroscopy. PLoS One. 8 (11), e82382(2013).
- Ramanujan, S., et al. Diffusion and convection in collagen gels: implications for transport in the tumor interstitium. Biophys J. 83 (3), 1650-1660 (2002).
- Harjanto, D., Maffei, J. S., Zaman, M. H. Quantitative analysis of the effect of cancer invasiveness and collagen concentration on 3D matrix remodeling. PLoS One. 6 (9), e24891(2011).
- Wolf, K., et al. Collagen-based cell migration models in vitro and in vivo. Semin Cell Dev Biol. 20 (8), 931-941 (2009).
- Petroll, W. M. Differential interference contrast and confocal reflectance imaging of collagen organization in three-dimensional matrices. Scanning. 28 (6), 305-310 (2006).
- Fraley, S. I., et al. Three-dimensional matrix fiber alignment modulates cell migration and MT1-MMP utility by spatially and temporally directing protrusions. Sci Rep. 5, 14580(2015).
- Ikeda, Y., Collins, M. K., Radcliffe, P. A., Mitrophanous, K. A., Takeuchi, Y. Gene transduction efficiency in cells of different species by HIV and EIAV vectors. Gene Ther. 9 (14), 932-938 (2002).
- Ma, H. T., Poon, R. Y. Synchronization of HeLa cells. Methods Mol Biol. 761, 151-161 (2011).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados