Method Article
해안 습지에서 메탄 순환 미생물 역학 시각화
요약
이 프로토콜은 남부 텍사스 해안 습지에서 주요 메탄 순환 유전자를 검출하고 공간 분포를 시각화하여 이러한 역동적인 생태계에서 메탄 조절과 메탄 조절이 환경에 미치는 영향에 대한 이해를 높입니다.
초록
해안 습지는 메탄의 가장 큰 생물 공급원으로, 메탄 겐은 유기물을 메탄으로 전환하고 메탄 영양 생물은 메탄을 산화시켜 메탄 순환을 조절하는 데 중요한 역할을합니다. 기후 변화로 인해 잦은 기상 이변, 염도 변동 및 인위적 활동의 영향을 받는 남부 텍사스의 습지는 메탄 순환에 영향을 미칩니다. 이러한 과정의 생태학적 중요성에도 불구하고 남부 텍사스 해안 습지의 메탄 순환은 여전히 충분히 조사되지 않은 상태로 남아 있습니다. 이러한 간극을 해소하기 위해 메탄겐 및 메탄영양생물과 관련된 유전자를 검출하는 방법을 개발하고 최적화했으며, 여기에는 메탄겐에 대한 바이오마커인 mcrA 와 메탄영양생물에 대한 바이오마커인 pmoA1, pmoA2 및 mmoX 가 포함되었습니다. 또한 본 연구는 지리정보시스템(GIS) 소프트웨어인 ArcGIS Pro를 활용하여 메탄겐과 메탄영양 풍부도의 공간적, 시간적 분포 패턴을 시각화하는 것을 목표로 하였다. 이러한 분자 기술과 고급 지리 공간 시각화의 통합은 남부 텍사스 습지 전역에 걸쳐 메탄겐 및 메탄 영양 군집의 공간적 및 시간적 분포에 대한 중요한 통찰력을 제공했습니다. 따라서 이 연구에서 확립된 방법론은 습지의 미생물 역학을 매핑하기 위한 강력한 프레임워크를 제공하고, 다양한 환경 조건에서 메탄 순환에 대한 이해를 높이고, 더 광범위한 생태 및 환경 변화 연구를 지원합니다.
서문
연안 습지는 탄소 격리, 증발산, 메탄(CH4) 배출과 같은 과정을 통해 기후 조절, 생물다양성 보전, 수자원 관리에 기여하는 중요한 생태계이다1. 담수 습지와 바닷물 습지2를 포함한 이러한 생태계는 생산성이 높으며 이산화탄소(CO2)를 흡수하고 육상 및 해양 환경에서 유기물을 포집하는 데 중요한 구역 역할을 합니다3 , 4 . 이러한 습지 내의 역동적인 상호작용은 미생물 CH4 생산과 소비5를 자극하여 CH46의 가장 큰 자연 공급원 중 하나로 자리매김한다. 두 번째로 중요한 온실 가스 인 CH4는 CO2 4,7,8,9보다 약 27-30 배 더 큰 지구 온난화 잠재력을 가지고 있으므로 기후 변화 시대에 해안 습지의 CH4 배출에 대한 연구가 필수적입니다. CH4의 배출은 다양한 환경적 요인, 특히 염도의 영향을 받으며, 이는 미생물 과정에 결정적인 역할을 한다10. 담수 습지는 황산염 수준이 낮아 대기 중 메탄에 크게 기여하여 미생물 CH4 생산을 촉진하는 반면, 염수 습지는 일반적으로 황산염 농도가 높기 때문에 CH4를 덜 방출하는 경향이 있습니다 11,12,13.
해안 습지로부터의 CH4 배출은 일반적으로 메탄겐(methanogens)과 메탄영양생물(methanotrophs14)로 알려진 두 그룹의 미생물에 의해 제어된다. 메탄겐은 메탄생성(methanogenesis)15로 알려진 과정을 통해 포름산염, 아세테이트, 수소 또는 메틸화 화합물과 같은 기질을 분해하여 무산소 퇴적물에서 CH4를 생성합니다. 이 경로에서 중요한 효소는 메틸-코엔자임 M 환원효소(MCR)로, 메탄 생성의 최종적이고 속도 제한 단계를 촉매하기 때문입니다 15,16,17. MCR의 알파 소단위체를 암호화하는 mcrA 유전자는 모든 메탄생성 고세균(methanogenic archaea18)에서 발견될 수 있는 기능적 마커이다. 더욱이, 해안 습지에서는 황산염-메탄 전이대(SMTZ)가 메탄생성 지대 위에 형성되며, 여기서 위쪽으로 확산되는 메탄과 아래쪽으로 이동하는 황산염이 수렴하여 고갈된다19. 이 구역 내에서 혐기성 메탄영양 고세균(ANME)은 MCR 효소를 사용하여 메탄을 이산화탄소로 산화시키는 반면, 황산염 환원 박테리아(SRB)는 황산염을 황화물로 환원시킵니다. SRB는 수소와 아세테이트에 대해 메탄 생성보다 경쟁하여 황산염이 고갈될 때까지 메탄 생성을 제한합니다 16,17.
대조적으로, 호기성 메탄영양 박테리아는 다양한 형태의 메탄 모노옥시게나제(MMO)를 사용하여 호기성 환경20에서 CH4를 산화합니다. 여기에는 세포질내 막에 내장된 구리 함유 효소인 미립자 메탄 모노옥시게나제(pMMO)와 세포질에서 발견되는 철 함유 효소인 수용성 메탄 모노옥시게나제(sMMO)가 포함됩니다. 그러나 pMMO의 경우 세 가지 유전자 오페론이 있습니다 pmoCAB21; 그 중 pmoA 유전자는 모든 메탄영양생물에 대해 가장 보수적이다. pmoA에 대한 두 가지 다른 바이오마커 유전자가 있습니다: pmoA1 및 pmoA222. 또한, 메탄영양생물에 대한 포괄적인 이해를 위해 mmoX 유전자는 분자생물학에서 sMMO를 함유한 메탄영양생물을 식별하는 도구로 사용됩니다23. 메탄겐 및 호기성 메탄영양 생물의 대사 경로와 환경 요구 사항의 이러한 구분은 해안 습지 생태계에서 메탄 순환을 조절하는 복잡한 미생물 상호 작용을 강조합니다.
텍사스 남부의 생산적인 바닷물 환경인 보카치카(BC) 습지는 멕시코만(GOM)의 조수 영향을 받아 특히 고염분 Laguna Madre24에 근접하기 때문에 표면 염분 수준이 가변적입니다. 만조와 간조를 번갈아 가며 일어나는 이 조석 작용은 산소 농도가 변동하게 한다25 이는 퇴적물26 의 메탄겐 및 메탄 영양 활성을 변화시킬 수 있다. 대조적으로, 해안 담수 습지는 CH4 플럭스27의 중요한 핫스팟으로 간주됩니다. GOM의 조수 효과에서 멀리 떨어진 Resaca Del Rancho Viejo(RV)와 Lozano Banco(LB)를 포함한 남부 텍사스의 해안 담수 습지는 뚜렷한 수문학적 관리를 가지고 있습니다. RV는 수위가 낮을 때 강물에 의해 보충되는 펄스 흐름을 경험하는 반면 LB는 이러한 보충 없이 오프라인 흐름 시스템으로 작동합니다. 더욱이, RV와 LB는 각각 인위적으로 펌핑된 담수의 높은 배출과 옥스보우 호수이기 때문에 낮은 염도 수준을 유지합니다. 다양한 환경 요인은 남부 텍사스 해안 습지를 가로지르는 메탄 순환에 큰 영향을 미칠 수 있습니다. 그러나 남부 텍사스 해안 습지의 메탄 순환은 아직 철저히 조사되지 않은 영역으로 남아 있습니다.
중합효소연쇄반응(PCR) 및 Real-Time PCR(정량적 PCR[qPCR]이라고도 함)은 환경 샘플에서 특정 유전자의 상대적 풍부도를 검출하고 정량화하기 위해 널리 사용되는 기본적이고 널리 사용되는 기술입니다. 이러한 기술은 DNA의 표적 영역을 특이적으로 증폭하여 CH4 사이클링 관련 유전자의 존재 및 상대적인 양을 표시하여 잠재적인 메탄 사이클링의 지표를 제공합니다. 역시, PCR 뇌관 세트의 유용성 그리고 효험은 추출된 환경 DNA에 있는 각종 억제 요인에 의해 제한될지도 모르다, 환경의 유형에 의해 영향을 받는28,29. 따라서 본 연구는 주로 남부 텍사스 해안 습지에서 CH4 사이클링 관련 유전자의 존재를 검출하기 위한 최적의 PCR 방법을 확립한 다음(그림 1) 이러한 생태계에서 정량화된 상대적 풍부도를 시각화했습니다. 이 연구의 결과는 다양한 해안 생태계에서 CH4 순환 및 미생물 역학에 대한 이해를 높이기 위해 다른 해안 지역에 적용될 수 있습니다.
프로토콜
1. 샘플 수집
- 침전물 그랩 샘플러 또는 삽을 사용하여 침전물 샘플을 수집합니다.
참고: 샘플은 2023년과 2024년의 서늘한 계절(10월-2월, 평균 기온 20°C)과 따뜻한 계절(4월-6월, 평균 기온 27°C) 동안 3개의 뚜렷한 해안 습지의 두 관측소에서 수집되었습니다. 해안 담수 습지에서 샘플을 채취할 때는 퇴적물 채취 샘플러가 사용되었으며(그림 2), 조수의 영향을 받는 해안 염수 습지에는 삽이 사용되었습니다. - 먼저 샘플러를 얕은 수역으로 낮추고 자체 무게로 퇴적물 표면(상단 50cm 이내)으로 가라앉도록 하여 그림 2와 같이 퇴적물 구조에 대한 방해를 최소화합니다.
- 일반적으로 깊이가 60cm에서 215cm30 인 물기둥에서 꺼내 침전물 샘플을 지퍼백에 옮기고 즉시 아이스 박스에 보관하십시오.
알림: 다음 스테이션으로 진행하기 전에 그랩 샘플러를 탈이온(DI) 물로 세척하십시오. - 모든 샘플은 실험실에서 즉시 -20 °C에 보관하십시오.
- 각 샘플링 사이트에서 다중 매개변수 수질 측정기를 사용하여 염도 및 현장 온도와 같은 지표수 품질 매개변수를 측정합니다.
알림: 프로브는 각 스테이션에서 사용한 후 탈이온수(DI water)로 헹궈냈습니다.
2. 게놈 DNA 추출
- 게놈 DNA 추출 절차를 시작하기 전에 실온에서 샘플을 해동합니다.
- 약 500mg의 침전물 샘플을 15mL 튜브에 옮기고 4,250× g 에서 3분 동안 원심분리하여 모든 물을 제거합니다.
- 제조업체의 프로토콜31 에 따라 토양용 DNA 추출 키트를 사용하여 약간의 수정을 가하여 게놈 DNA를 추출하고 즉시 -20°C에서 보관합니다.
참고: 이 변경 사항은 중복성과 효율적인 작업 흐름을 줄이기 위해 이루어졌습니다.- 유리 구슬/세라믹 구체 함유 튜브에 최대 500mg의 토양 샘플을 추가합니다.
- 978 μL의 Sodium Phosphate Buffer를 bead/sphere-containing tube의 샘플에 추가합니다.
- 122μL의 완충액 용해 용액을 비드/구 함유 튜브의 샘플에 추가하여 외부 오염 물질을 용해시킵니다.
- 비드 밀 균질화기를 사용하여 5배 속도 수준에서 20초 동안 균질화하고 2배를 반복합니다.
참고: 이 연구에서는 비드 밀 균질화가 사용되었기 때문에 속도가 조정되었습니다. - 혼합물을 14,000 ×g에서 10분 동안 원심분리합니다 .
- 상층액을 깨끗한 2.0mL 마이크로 원심분리 튜브로 옮깁니다.
- 250μL의 단백질 침전 용액(PPS)을 추가하여 세포 파편 및 용해 매트릭스에서 용해된 핵산을 분리합니다. 튜브를 10배 뒤집어 섞습니다.
- 14,000 × g 에서 5분 동안 원심분리기를 사용하여 펠릿을 침전시키고 세포 파편을 제거하고 매트릭스를 용해합니다.
- 상층액을 깨끗한 15mL 마이크로 원심분리기 튜브로 옮깁니다.
- 1.0mL의 결합 매트릭스 현탁액을 15mL 튜브의 상층액에 추가합니다.
참고: 바인딩 매트릭스 서스펜션을 흔들어 다시 suspend한 후 추가합니다. - 2분 동안 회전기에 튜브를 올려 DNA가 결합 매트릭스에 결합할 수 있도록 합니다.
- 모든 튜브를 랙에 놓고 바인딩 매트릭스가 안정화될 수 있도록 3분 동안 배양합니다.
- 3분 후 750μL를 버리고 피펫을 사용하여 남은 상등액을 펠릿과 부드럽게 혼합합니다.
- 혼합물 750μL를 SPIN 필터로 옮기고 1분 동안 14,000× g 으로 원심분리합니다. 캐치 튜브를 비우고 재사용하십시오. 나머지 혼합물로 반복하십시오.
- 준비된 세척 용액 500μL(적절한 양의 에탄올이 첨가됨)를 첨가하여 불순물을 더욱 용해시킵니다. 피펫 끝에서 액체의 힘을 사용하여 펠릿을 부드럽게 다시 매달립니다.
- 14,000 × g 에서 1분 동안 원심분리기를 사용하여 불순물을 제거합니다. 캐치 튜브를 비우고 재사용하십시오.
- 아무것도 넣지 않고 14,000 × g 에서 2분 동안 다시 원심분리기를 사용합니다.
- 캐치 튜브를 깨끗하고 새 캐치 튜브로 교체하고 SPIN 필터를 실온에서 5분 동안 자연 건조합니다.
- 50μL DNAse 자유수(DES)를 넣고 14,000× g 에서 1분 동안 원심분리합니다.
참고: 이 프로토콜에서는 추출된 환경 DNA(eDNA)의 최적 회수 및 안정성을 보장하기 위해 DNA 용리에 50μL의 DES가 사용되었습니다.
3. DNA 정량화
- 0.5 mL 튜브에 추출된 DNA 1 μL와 형광 염료 200 μL를 넣고 피펫팅으로 철저히 혼합합니다.
- 빛이 투과되지 않도록 즉시 튜브를 알루미늄 호일로 감싸고 실온에서 5분 동안 배양합니다.
- 제조업체의 프로토콜에 따라 ONE DNA 농도 모드에서 형광측정기를 사용하여 DNA 농도를 측정합니다.
4. 종래의 PCR에 의하여 16S rRNA, pmoA1 , pmoA2 , mmoX 및 mcrA 의 검출
- 기존 PCR(cPCR)을 실행하기 전에 모든 샘플과 시약을 얼음 양동이에서 해동합니다.
- 추출된 모든 eDNA 샘플을 10ng/μL로 희석합니다.
참고: 프라이머 목록은 표 1에 나와 있습니다. - 12.5 μL의 2x PCR 마스터 믹스, 0.5 μL의 정방향 및 역방향 프라이머(10 μM)(프라이머 목록은 표 1 참조), 1 μL의 10 ng/μL eDNA 및 10.5 μL의 뉴클레아제-프리 워터를 포함하여 각 샘플에 대해 25 μL cPCR 반응 혼합물을 준비합니다.
참고: 피펫팅 오류를 최소화하기 위해 하나의 추가 샘플에 충분한 부피로 마스터 믹스를 준비합니다. - 2 분 동안 95 °C에 처음 변성으로 이루어져 있는 프로토콜에 따라 cPCR 반응을 실행하고, 45 s 동안 95 °C에 변성의 40 주기, 30 s 동안 72 °C에 연장, 및 5 m를 위한 72 °C에 마지막 연장, 다른 뇌관을 위한 다양한 어닐링 온도와 더불어 (다른 유전자를 위해 이용된 다른 뇌관의 어닐링 온도를 위한 표 1 보십시오).
참고 : mcrA 유전자의 경우, ML 프라이머는 처음 5 사이클32 동안 어닐링과 확장 단계 사이에 0.1 °C / s의 느린 램프 속도를 사용하여 원하는 밴드를 보여주었습니다. - 브롬화 에티듐(EtBr)이 미리 염색된 아가로스 겔에서 cPCR 산물을 시각화합니다.
참고: 50bp 래더에는 2.5% 아가로스 젤을 사용하고 1kb 래더에는 0.9% 젤을 사용합니다. 2.5% 아가로스 젤에는 1x TAE 버퍼를 사용하고 0.9% 아가로스 젤에는 0.5x TAE 버퍼를 사용합니다.
5. 정량적 Real-time PCR에 의한 pmoA1 , pmoA2 , mmoX, mcrA 검출
참고: pmoA1, pmoA2, mmoX 및 mcrA 풍부도와 같은 메탄겐 및 메탄영양 표적 유전자는 Real-Time PCR 시스템을 사용하여 qPCR에서 관찰되었습니다.
- 각 유전자에 대한 표준 곡선을 얻기 위해 각 유전자에 대한 표준물질을 개별적으로 준비합니다.
- 각 유전자의 겔 전기영동 동안 가장 밝은 띠를 생성한 샘플을 사용하여 cPCR로 표적 유전자를 증폭합니다. 섹션 4에 설명된 방법과 입문서를 따릅니다.
- 겔 추출 키트를 사용하여 증폭된 산물을 정제하고 섹션 3에 설명된 방법에 따라 DNA 농도를 측정하고 -20°C에서 보관합니다.
알림: 추가 사용을 위해 정제된 표준물질을 별도의 튜브로 분취합니다. - qPCR에 대한 복제 수 계산 웹사이트를 사용하여 측정된 DNA 농도에서 유전자 사본을 계산합니다.
- qPCR을 실행하기 전에 뉴클레아제가 없는 물로 계산된 표준물질 복제 수를 희석하여 108 - 102 copies/μL 범위의 각 표준물질을 준비합니다.
- 음성 대조군(NTC, 템플릿 DNA 없음)을 포함하여 각 복제 번호의 3회 복제를 사용하여 표준 곡선을 준비합니다.
참고: 각 표준 곡선의 R2 값이 0.99보다 큽니다.
- 모든 샘플, 표준물질 및 NTC에 대해 20μL qPCR 반응 혼합물을 준비합니다. 모든 샘플에 대해 qPCR 분석을 3회씩 수행합니다.
- qPCR 마스터 믹스, 프라이머, 뉴클레아제가 없는 물, 표준물질 및 샘플을 포함한 모든 반응 성분을 시작하기 전에 얼음 선반에 놓습니다.
- SYBR Green 마스터 믹스 10 μL, 각 10 μM 정방향 및 역방향 프라이머 0.5 μL, 뉴클레아제 무함유 정제수 8 μL, 10 ng/μL 템플릿 DNA, 표준물 또는 DI 물 1 μL를 포함하는 각 샘플, 표준물질 및 NTC에 대해 20 μL qPCR 반응 혼합물을 준비합니다.
참고: qPCR에 대한 기존 PCR에서 pmoA1 및 mcrA 유전자에 대한 최적의 결과를 얻으려면 표시된 프라이머 세트를 사용하십시오( 표 1 참조). 정확도를 높이려면 각 샘플을 세 번 실행하십시오. 다음 단계는 샘플당 60μL의 결합 부피로 3중 샘플을 준비하기 위한 효율적인 방법을 제공합니다.- 반응 혼합물을 준비하여 마스터 믹스, 특정 유전자에 대한 정방향 및 역방향 프라이머, 주형 DNA를 제외한 2mL 튜브에 뉴클레아제가 없는 물을 결합합니다.
참고: 피펫팅 오류를 고려하여 반응 혼합물 부피를 준비합니다. 예를 들어, 24개의 샘플과 8개의 표준물질이 있는 경우 32개의 반응 대신 33개의 반응에 대한 총 부피를 계산하여 파이펫팅 오류를 최소화합니다. 이 경우, 삼중 반응에 필요한 총 부피는 다음과 같습니다: 990 μL의 마스터 믹스(33 샘플 x 3 반복 x 10 μL), 49.5 μL의 순방향 프라이머(33 샘플 x 3 반복 x 0.5 μL), 49.5 μL의 역방향 프라이머(33 샘플 x 3 반복 x 0.5 μL) 및 792 μL의 뉴클레아제-프리 물(33 샘플 x 3 반복 x 8 μL). - 샘플 수와 표준물질에 따라 PCR 튜브를 준비합니다.
- 준비된 반응 혼합물 57μL를 각 PCR 튜브에 분배합니다.
참고: 각 샘플에 대해 qPCR을 3회씩 수행하려면 샘플당 57μL의 총 반응 혼합물 부피를 준비합니다(템플릿 DNA, 표준물질 또는 물 제외). 이 부피는 하나의 샘플에 대해 3개의 웰로 균등하게 나뉘며 각 웰에 19μL가 할당됩니다. - 각 튜브에 3μL의 template DNA, standard 또는 nuclease-free water를 추가하고 튜브 바닥을 가볍게 두드려 혼합합니다.
참고: 하나의 샘플에 대해 준비된 반응 혼합물의 총 부피는 현재 각 튜브에서 60μL입니다.
- 반응 혼합물을 준비하여 마스터 믹스, 특정 유전자에 대한 정방향 및 역방향 프라이머, 주형 DNA를 제외한 2mL 튜브에 뉴클레아제가 없는 물을 결합합니다.
- 각 튜브에서 준비된 반응 혼합물 20μL를 96웰 qPCR 플레이트의 지정된 웰로 분취합니다. 어플리케이터를 사용하여 PCR 플레이트를 접착 PCR 밀봉 필름으로 밀봉합니다.
- 밀봉된 플레이트를 1,000 × g 에서 1분 동안 원심분리하여 웰 내의 기포를 제거하기 위해 적절한 혼합을 확인합니다.
- PCR 플레이트를 열 순환기에 놓습니다. qPCR 기계를 켠 다음 관련 소프트웨어를 열어 프로토콜을 설정합니다.
- qPCR 마스터 믹스 가이드라인에 따라 프로토콜을 설정합니다. 다음 프로토콜을 사용하십시오: 95°C에서 10분, 95°C에서 15초, 72°C에서 30초 동안 확장 단계를 수행합니다. 표 1 의 관련 프라이머에 대해 지정된 어닐링 온도에서 어닐링 단계를 45초 동안 수행합니다. 35주기 동안 모든 qPCR 실행을 수행합니다.
- 96웰 qPCR 플레이트를 샘플 함유 플레이트와 동일한 구성으로 표준물질 및 NTC와 함께 설정합니다.
- 절대 정량화 표준 곡선 방법을 사용하여 각 샘플에서 증폭된 산물과 유전자 복제 수를 정량화합니다33.
6. South Texas Coastal Wetlands 지도에서 메탄 순환 유전자 시각화
- 지리 정보 시스템(GIS) 소프트웨어인 ArcGIS Pro 를 열고 컴퓨터의 지정된 폴더에 Study Area 라는 이름으로 프로젝트 파일을 저장합니다.
- 왼쪽 상단의 지도를 클릭합니다 | Basemap 을 선택하고 Terrain with Labels를 베이스맵으로 선택합니다.
- Locate( 찾기)를 클릭합니다. 검색하고 검색 창이 열리면 영역 이름을 입력하여 학습 영역을 찾습니다. 해당 영역이 표시됩니다.
- 지리 참조를 사용하여 특정 영역을 그립니다.
- View( 보기) | 카탈로그 창을 클릭합니다.
- 카탈로그에서 Folder( 폴더 )를 두 번 클릭합니다 . 파일 이름.
- geodatabase (.gdb) 파일을 마우스 오른쪽 버튼으로 클릭한 다음 New(새로 만들기) | 피쳐 클래스 및 피쳐 클래스 생성이 표시됩니다.
- Name and Alias 상자를 입력하고 하단의 Finish를 클릭합니다.
- View( 보기) | 목차. 별칭 이름이 콘텐츠 창에 표시됩니다.
- 상단 레이어에서 편집을 클릭합니다 | 만듭니다. 기능 만들기 창이 열립니다. Create Features 창에서 Alias 를 두 번 클릭하면 Configure Tool Feedback Options 가 나타납니다.
- 선을 선택한 다음 맵에서 선을 스케치하여 연구 영역의 외부 경계를 만듭니다. 완료되면 지도를 두 번 클릭합니다.
- GIS 소프트웨어를 최소화한 다음 스프레드시트를 엽니다. 첫 번째 열에 샘플 이름을 입력하고, 두 번째 열에 위도를 입력하고, 세 번째 열에 경도 를 입력합니다. pmoA1, pmoA2, mmoX 및 mcrA의 qPCR 데이터에 대해 다음 4개의 열을 사용합니다.
- 컴퓨터의 특정 폴더에 파일을 CSV 형식으로 저장합니다.
- GIS 소프트웨어를 다시 열고 Add Data(데이터 추가) | XY 점 데이터.
- 입력 테이블 상자에서 컴퓨터의 폴더에서 CSV 파일을 선택합니다. Output Feature Class에서 파일 이름을 바꾼 다음 Run을 클릭하여 맵에 샘플링 점을 표시합니다.
- 상단의 v검색창을 클릭하고 Kriging을 검색합니다.
- 샘플링 스테이션 파일을 선택한 다음 pmoA1을 선택합니다.
- 환경 | select the sampling station in layer and mask(레이어 및 마스크에서 샘플링 스테이션 선택) | 실행을 클릭합니다.
- 6.9, 6.10 및 6.11단계에서 언급한 프로토콜에 따라 모든 연구 영역에 대해 pmoA2, mmoX 및 mcrA에 대한 Kriging을 생성합니다.
- 맵의 레이아웃을 만듭니다.
- 상단 레이어에서 삽입 을 클릭하십시오 | 새 레이아웃을 선택하고 ANSI - 가로를 선택합니다.
- Map Frame 을 클릭하고, Kriging 이 있는 맵을 선택한 다음, 사각형을 그려 레이아웃에 모든 맵을 배치하십시오. 이렇게 하면 레이아웃에서 지도가 표시됩니다.
- 북쪽 화살표를 선택하고 레이아웃에 배치하여 북쪽 방향을 나타냅니다.
- 축척 막대를 선택하여 지도에 영역의 축척을 표시합니다.
- 범례를 클릭하여 범례를 표시한 다음 레이아웃에 배치합니다.
- 그리드를 클릭하고 검은색 계수선 옵션을 선택합니다. 이렇게 하면 위도와 경도가 있는 그리드가 생성되고 Black Horizontal Label Graticule이라는 레이블이 있는 콘텐츠 창에 표시됩니다.
- Black Horizontal Label Graticule을 두 번 클릭한 다음 Components를 선택합니다. Ticks 1 및 Grid를 클릭하고 오른쪽에 있는 십자 기호를 클릭하여 이러한 구성 요소를 제거합니다.
- 맨 위 레이어에서 Share 를 클릭한 다음 Export Layout을 클릭합니다. 파일 형식을 PDF로 선택하고, 이름 상자를 사용하여 컴퓨터에 파일을 저장하고, 수직 해상도를 500 DPI로 설정하고, 내보내기 를 클릭하여 맵의 PDF 파일을 만듭니다.
결과
남부 텍사스의 해안 습지에서 CH4 사이클링 관련 유전자(mcrA, pmoA1, pmoA2 및 mmoX)의 분포와 풍부함을 이해하기 위해 각 샘플에서 추출한 eDNA를 cPCR 및 qPCR로 분석했습니다. 각 바이오마커에 대한 범용 프라이머를 선택하여 이전 연구(표 1)22,34,35,36,37에서 cPCR을 실행하고 샘플 특성 및 환경 조건에 따라 어닐링 온도 및 농도를 최적화하기 위해 수정했습니다. 예를 들어, 16S rRNA, pmoA1 및 pmoA2는 대부분이 표준 어닐링 온도보다 훨씬 낮게 증폭되었기 때문에 표준 어닐링 온도를 사용하여 증폭하지 않았습니다(데이터는 표시되지 않음). 또한, cPCR 과정에서 10ng/μL DNA의 농도는 pmoA1, pmoA2 및 mmoX 유전자에 대해 뚜렷한 띠를 보인 반면, 5ng/μL의 템플릿 DNA를 사용하면 mcrA 유전자에 대해 더 밝은 띠를 보여주었습니다(보충 파일 1-보충 그림 S1).
cPCR 분석은 텍사스 남부의 세 가지 유형의 해안 습지에서 유전자 검출의 공간적 다양성을 밝혔습니다. 표 2에서 볼 수 있듯이, mcrA 유전자는 연안 담수 습지인 LB와 RV의 모든 샘플에서 검출되었다. 그러나 cPCR은 염수 습지의 샘플링된 퇴적물에서 mcrA 유전자를 검출할 수 없었으며, 이는 염분과 메탄겐 분포 사이의 잠재적인 연관성을 시사합니다(보충 파일 1-보충 그림 S2). mcrA 검출을 위해 본 연구에서는 MLF-MLR, MCRf-MCRr, ME1F-ME2R 및 McrA 159F-McrA 345R을 포함한 4개의 프라이머 쌍을 사용했습니다(표 1). 그 중 ML 프라이머 쌍만이 남부 텍사스 습지에서 메탄겐 군집을 검출할 수 있었습니다(보충 파일 1-보충 그림 S2, 보충 그림 S3 및 보충 그림 S4). 대조적으로, 호기성 메탄영양 생물의 경우, pmoA1 유전자는 모든 RV 샘플에서 검출되었지만 LB 및 BC 샘플에는 없었습니다(보충 파일 1-보충 그림 S5). pmoA1 동정을 위해 A189를 순방향 프라이머로, A682 및 mb661을 역방향 프라이머로 사용하였으며, 여기서 A189-A682 프라이머 쌍은 amoA35,38도 검출할 수 있습니다. 그러나, 이 뇌관 쌍은 젤 심상이 아주 희미한 띠를 보인 이 학문에 있는 cPCR에 있는 유전자의 존재를 검출할 수 있었다, pmoA1 유전자의 낮은 풍부함을 건의하는. 또 다른 역방향 프라이머 mb661은 A189를 순방향 프라이머38로 하여 pmoA1 유전자를 특이적으로 식별하도록 설계되었습니다. mb661을 리버스 프라이머로 사용했을 때, cPCR은 더 밝은 띠(Supplemental File 1-Supplemental Figure S6)를 보였으며, 이는 나중에 qPCR에 사용되었습니다. 흥미롭게도, pmoA2 유전자는 2개의 BC 샘플과 1개의 LB 샘플에서 검출되었지만, 1개의 RV 샘플에서는 없었다(보충 파일 1-보충 그림 S7). 이전 연구에서는 66 °C에서 206f-703b 프라이머가 438 bp 제품 크기22로 pmoA2 만 증폭 할 수있는 반면, 60 °C에서는 245 및 438 bp의 두 가지 크기의 앰플리콘이 관찰 된 pmoA2의 어닐링 온도가 66 °C 및 60 °C임을 발견했습니다. 그러나 이 연구에서는 60°C의 어닐링 온도에서 증폭이 성공하여 LB 및 BC 부위에서 예상되는 크기(438bp)의 대역을 생성했습니다(보충 파일 1-보충 그림 S7). 또한, mmoX 유전자는 LB 샘플에서 검출되었으나 하나의 RV 샘플(보충 파일 1-보충 그림 S8)에서는 없었고 BC 샘플에서는 전혀 없었다. mmoX에 사용된 프라이머 쌍은 이 연구에서 남부 텍사스 습지를 연구하는 데에도 적합했습니다. 55°C36의 어닐링 온도는 cPCR에서 mmoX에 대한 밝은 대역을 생성했습니다(보충 파일 1-보충 그림 S8). 샘플 무결성을 확인하기 위해 16S rRNA를 cPCR 중 모든 샘플에서 16S rRNA를 성공적으로 증폭하는 양성 대조군으로 사용했으며, 이는 샘플이 오염되지 않았음을 시사합니다(보충 파일 1-보충 그림 S9).
유전자 풍부도의 시간적, 공간적 변화를 평가하기 위해 cPCR에서 확인된 최적화된 프라이머 세트를 사용하여 모든 표적 유전자에 대해 qPCR 분석을 수행했습니다. pmoA2 및 mmoX 유전자의 qPCR은 cPCR에 사용된 것과 동일한 primer로 수행되었습니다. 그러나, pmoA1 유전자의 경우 A189-mb661 프라이머 쌍을 사용하였고, mcrA 유전자의 경우 MLF-MLR 프라이머 쌍을 선택하여 둘 다 cPCR에서 양호한 결과를 보였다. 남부 텍사스의 습지 전반에 걸친 계절적 차이와 염분 변화도 미생물 군집 역학에 영향을 미칠 수 있기 때문에 고려되었습니다. 그림 3 및 그림 4에서 볼 수 있듯이, 두 개의 서로 다른 샘플링 기간 사이에 메탄겐과 메탄영양생물의 유전자 풍부도에 차이가 있었습니다. 해안 담수 습지에서는 염도의 계절적 변화가 없었지만, 해안 염수 습지에서는 염도의 계절적 차이가 관찰되었는데39 이는 메탄겐과 메탄영양 풍부도의 변화를 설명할 수 있다. 그림 3 은 서늘한 계절에 mmoX 가 1.46 × 104 copies/μL의 가장 높은 풍부도로 이 모든 습지에서 매우 풍부했음을 보여줍니다. LB, RV 및 BC의 한 스테이션에서 pmoA2 의 비율이 적었습니다. 그러나 qPCR은 서늘한 계절에는 함량이 낮아 pmoA1 및 mcrA 를 검출할 수 없었습니다. 대조적으로, pmoA1 은 따뜻한 계절에 대부분 풍부했으며 2.44 × 103 copies/μL의 가장 높은 풍부도를 보였습니다(그림 4). mmoX 및 mcrA 는 LB(상단 50cm 이내)의 표면 퇴적물 샘플에서도 검출되었으며, 따뜻한 계절에는 상대적으로 적은 양임에도 불구하고 각각 1.92 × 102 copies/μL 및 2.04 × 102 copies/μL의 최대 풍부도를 보여주었습니다. 흥미롭게도, 따뜻한 계절에는 pmoA2 가 전혀 검출되지 않았고, BC 샘플에서 표적 유전자 중 어느 것도 검출되지 않았는데, 이는 남부 텍사스의 따뜻한 계절 동안 이 바닷물 습지39 특유의 환경적 요인에 의해 영향을 받은 것으로 보인다.
이 연구는 해안 습지 전반에 걸친 CH4 사이클링 관련 유전자 분포에서 뚜렷한 공간적, 계절적 변화를 보여줍니다. 특히, mcrA 유전자는 담수 LB 샘플에서만 검출되어 서식지 선호를 시사합니다. mmoX 및 pmoA2 유전자는 서늘한 계절에 모든 샘플에 걸쳐 존재한 반면, pmoA1 은 따뜻한 계절에 담수 환경에서 나타났습니다. 이러한 결과는 환경 조건과 계절적 변화가 CH4 사이클링 관련 미생물 활동에 미치는 영향을 강조합니다. 그림 3 과 그림 4에서 볼 수 있듯이 크리깅 보간법을 사용한 향상된 시각화는 해안 습지 생태계에서 CH4 사이클링 관련 미생물 활동에 대한 이해를 향상시켰습니다.
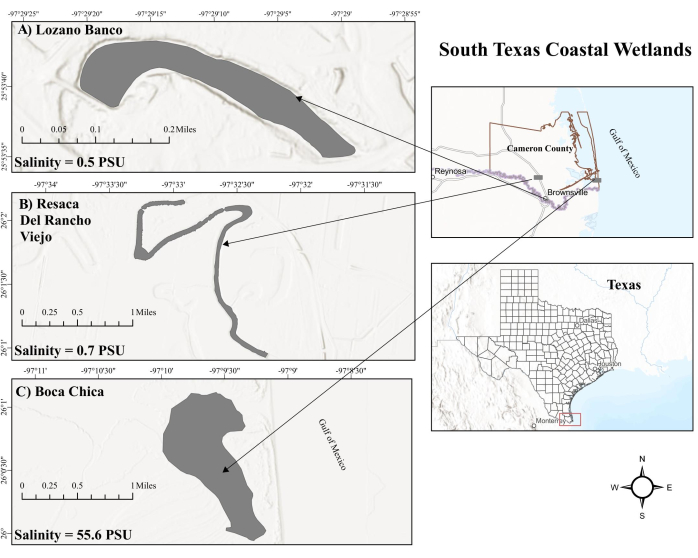
그림 1: 남부 텍사스 해안 습지 지도. (A) 로자노 방코. LB는 해안 담수 습지입니다. (b) Resaca del Rancho Viejo. RV는 또한 해안 담수 습지입니다. (C) 보카치카 습지. BC는 조수의 영향을 받는 고염분 해안 바닷물 습지입니다. 약어: LB = Lozano Banco; RV = Resaca Del Rancho Viejo; BC = 보카 치카; PSU = 실제 염도 단위. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
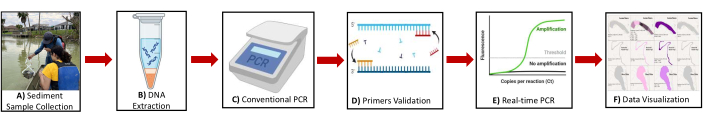
그림 2: 프로토콜의 개요를 보여주는 개략도. 주요 단계의 그림에는 (A) 퇴적물 샘플 수집 (B) DNA 추출 (C) 기존 PCR (D) 프라이머 검증 (E) Real-time PCR (F) 메탄 겐 및 메탄 영양 생물의 바이오 마커 유전자를 식별하기위한 데이터 시각화가 포함됩니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
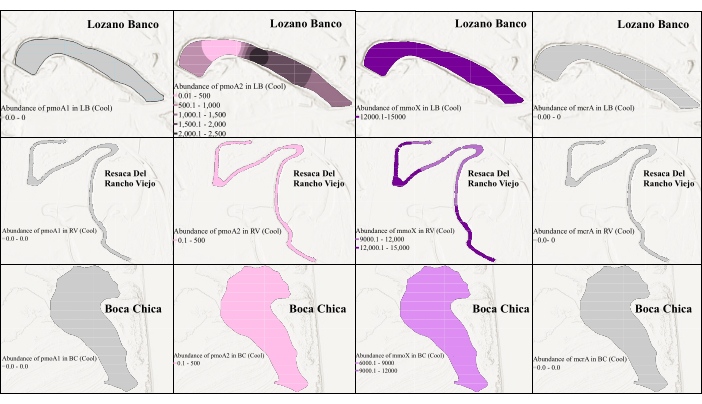
그림 3: 서늘한 계절에 메탄겐 및 메탄영양생물의 바이오마커 유전자의 공간적 분포. 그림은 시원한 계절에 mmoX 가 풍부하다는 것을 보여줍니다. 여기서 회색은 qPCR이 존재량이 낮아 유전자를 검출할 수 없음을 나타냅니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.

그림 4: 따뜻한 계절에 메탄겐 및 메탄영양생물의 바이오마커 유전자의 공간적 분포. 여기서, 회색은 qPCR이 낮은 존재량으로 인해 유전자를 검출할 수 없음을 나타냅니다. 이 그림은 로사노 방코(Lozano Banco)와 레사카 델 란초 비에호(Resaca Del Rancho Viejo)에서 따뜻한 계절에 pmoA1 의 풍부함을 보여주지만, 보카 치카(Boca Chica)에서는 풍부함이 발견되지 않았습니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
| 프라이머 이름 | 유전자 | 어닐링 온도 (°C) 이 연구에서 | 제품 길이 (bp) | 시퀀스 (5'-3') | 참조 | |
| 1055층 | 16S rRNA | 55 | 337 | 아 | (Harms et al., 2003) | |
| 1392R | 엑그gtgtac | |||||
| A189F | pmoA1 | 55 | 525 | GGNGACTGGGACTTCTGG | (Holmes et al., 1995) | |
| A682R | GAASGCNGAGAAGAASGC | |||||
| A189F | 56 | 470 | GGNGACTGGGACTTCTGG | (Costello & Lidstrom, 1999년) | ||
| MB661R | CCG, GMG, CAA, CGT, CYT, TACC | |||||
| 206층 | pmoA2 | 60 | 438 | GGNGACTGGGACTTCTG 가트갓가트카가트CG | (Tchawa Yimga 외, 2003) | |
| 703b | GAASGCNGAGAAGAASGC GGCGACCGGAACGACGT | |||||
| 536층 | mmoX | 55 | 362 | CGCTGTGG캣가그CGG | (Fuse et al., 1998) | |
| 898R | GCTCGACCTTGAACTTGGAGCC | |||||
| 엠씨르프 | 엠크라이 | 46 | ~490명 | 타이게이카라트기트 | (Springer et al., 1995) | |
| 엠씨R | ACRTTCATNGCRTARTT | |||||
| 나1F | 50 | 763 | GCMATGCARATHGGWATGTC | (Hales et al., 1996) | ||
| 나2R | TCATKGCRTAGTTDGGRTAGT | |||||
| MLF (MLF) | 55 | ~470명 | GGTGGTGTMG가트 카카르타이gcwacagc | (Luton et al., 2002) | ||
| MLR | TTCATTGCRTAGTTWGGRTAGTT | |||||
| 엠크라 159F | 62 | 186 | AAAGTGCGGAGCAGCAATCACC | (Vaksmaa 외, 2017) | ||
| MCRA의 345R | TCGTCCCATTCCTGCTGCATTGC | |||||
표 1: 메탄 순환과 관련된 기능적 유전자를 검출하기 위해 이 연구에서 사용된 프라이머 세트. 이러한 모든 프라이머 쌍은 기존 PCR에 사용되었으며, 마지막으로 대담한 쌍은 사우스 텍사스 해안 습지에서 메탄 생성 및 메탄 영양 생물 관련 유전자를 검출하기 위해 설정되었습니다.
| 유전자 | 샘플 | ||||||||
| 로자노 방코 | Resaca Del Rancho 비에호 | 보카치카 | |||||||
| 파운드 -1 | RV-1 시리즈 | RV-2 시리즈 | 엠1 | 엠2 | 지하 1층 | 나2층 | MG1 | MG2 | |
| pmoA1 | - | + | + | - | - | - | - | - | - |
| pmoA2 | + | + | + | - | + | - | - | + | - |
| mmoX | + | - | + | - | - | - | - | - | - |
| 엠크라이 | + | + | + | - | - | - | - | - | - |
표 2: 기존 PCR을 사용하여 서늘한 계절에 세 가지 유형의 수역에서 유전자 검출. '+', 현재; '-', 없습니다. 약어: LB = Lozano Banco, RV = Resaca del Rancho Viejo, M = 진흙, B = 바티스, MG = 맹그로브.
보충 파일 1: mcrA, pmoA1, pmoA2, mmoX 및 16S rRNA 유전자에 대한 겔 전기영동 이미지. 이 파일을 다운로드하려면 여기를 클릭하십시오.
토론
해안 습지는 중요한 온실 가스 인 대기 중 메탄의 중요한 원인으로 인식되고 있습니다40. 습지의 메탄 플럭스와 메탄 생성에 대한 연구가 있었지만 41,42,43 메탄 영양 생물이 다양한 환경 또는 다양한 관리 관행, 특히 수위가 변동하는 습지에서 어떻게 작동하는지에 대해서는 알려진 바가 거의 없습니다 44. 더욱이, eDNA에 부식산(humic acids)과 같은 억제제가 널리 존재하면 추출 및 증폭 과정이 복잡해져(45), 잠재적으로 위음성(false negative) 결과가 나올 수 있습니다46. 이 과제는 eDNA에 작용하는 프라이머의 다양한 효율로 인해 더욱 복잡해지며, 이는 이러한 연구에서 체계적인 검증의 필요성을 시사합니다47. 따라서 이 연구는 연구가 부족한 남부 텍사스 해안 습지에서 메탄 순환을 이해하기 위한 기초 방법론을 확립합니다. PCR 기술의 개발 및 검증에 초점을 맞춤으로써, 이 연구는 이러한 역동적인 생태계에서 주요 메탄 순환 유전자의 검출을 발전시킵니다.
이 연구의 중요한 측면 중 하나는 미생물 메탄 순환 관련 유전자의 시각화이며, 이는 다양한 계절과 염분 구배에 걸쳐 이러한 유전자의 공간 분포 패턴을 관찰하는 데 중요한 역할을 합니다. 이러한 시각화는 단순한 보조 도구가 아닙니다. 이는 잠재적인 메탄 순환이 있을 수 있음을 나타내는 중요한 메탄 생성 및 메탄 영양 미생물의 존재 영역을 식별하는 데 필수적이며, 이를 통해 이러한 과정에 영향을 미치는 환경 조건에 대한 이해를 높이는 데 필수적입니다.
본 연구에서는 cPCR과 qPCR을 이용하여 pmoA1, pmoA2, mmoX, mcrA 등의 메탄겐 및 메탄영양 마커 유전자를 규명하는 방법론을 마련하였다. mcrA 유전자를 표적으로 하는 4개의 primer pair와 pmoA1 유전자를 표적으로 하는 2개의 pair를 사용했으며, 각 set에서 단 한 쌍만이 기존 PCR과 real-time PCR 모두에서 이러한 유전자를 효과적으로 검출했습니다. 이전 연구에서는 pmoA1을 검출하기 위해 3개의 프라이머 쌍을 사용했습니다. 그 중 A189-A682 프라이머는 메탄영양 다양성을 검출할 수 없었다. 대신, 그들은 대부분 amoA 유전자를 증폭시키고 비특이적 PCR 산물을 생성했다48. 이 연구는 사용된 프라이머 쌍이 메탄영양 생물 개체군이 많은 환경에서만 pmoA1 유전자를 검출하는 데 효과적이라는 것을 발견했습니다. 더욱이, 해당 연구에서 A189-mb661 프라이머 쌍을 사용하여 가장 많은 양의 메탄영양 생물을 얻었으며, 이는 이러한 프라이머가 담수 생태계에 적합하다는 것을 나타냅니다48. 따라서, 이들 프라이머 쌍은 추가 분석으로서 qPCR에 사용하였다.
mcrA에 사용된 4개의 프라이머 쌍 중 ML 프라이머는 모든 환경에서 메탄겐 종을 증폭할 수 있었습니다49. ME 프라이머는 증폭 범위가 좁습니다. 그들은 다양한 유형의 자연 환경에서 모든 종류의 메탄겐 종을 감지할 수 없습니다. MCR 프라이머는 축퇴 수준이 높아 PCR 어닐링 온도에 민감합니다. 이러한 퇴화는 광범위한 표적 염기서열에 결합할 수 있게 해주며, 종종 여러 불일치를 수용합니다. 그러나, 이것은 또한 어닐링 온도의 작은 변화가 결합 효율 및 특이성에 상당한 영향을 미쳐 불충분한 증폭을 초래할 수 있으며, 결과적으로 메탄 발생 종49,50에 대한 위음성 결과를 초래할 수 있음을 나타냅니다.
반면에, 저다양성 메탄겐 군집에서는 PCR의 마지막 단계에서 PCR 산물의 높은 축적이 템플릿 재어닐링으로 이어지고 프라이머가 결합하는 것을 방지하며, 이는 또한 원하는 밴드를 검출하지 못합니다49. 흥미롭게도, McrA 159F-McrA 345R 프라이머 쌍은 질산염 의존성 혐기성 메탄영양 Candidatus M. nitroreducens archaea51을 식별하도록 특별히 설계되었으며, 이 연구에서는 이러한 프라이머 쌍을 사용하여 혐기성 메탄영양 생물을 검출할 수 없었습니다. 그러나 메탄(AOM)의 혐기성 산화는 철(Fe), 망간(Mn) 및 황산염(SO42-)52와 같은 대체 전자 수용체의 환원과 결합될 수 있으며, 이는 이 방법을 사용하여 ANME를 검출할 수 없는 이유를 설명할 수 있습니다. 또한 연구 지역의 특정 환경 조건 때문일 수 있습니다. 따라서, qPCR의 경우, ML 프라이머를 사용하여 메탄겐 풍부도를 검출했는데, 이는 군집 다양성이 낮은 조건에서도 신뢰할 수 있는 증폭을 제공했기 때문입니다49.
또한, 부식산 또는 비표적 종 DNA와 같은 PCR 억제제는 환경 샘플에 널리 존재하며, 이는 PCR 효율을 감소시킬 수 있습니다53. 고농도의 DNA는 cPCR에 의해 어떠한 유전자도 검출할 수 없었기 때문에, PCR 억제제의 효과를 줄이기 위해 cPCR을 실행하기 전에 모든 DNA 샘플을 희석하였다54. 박테리아와 고세균에 대한 보편적인 마커인 16S rRNA 유전자는 PCR 조건을 검증하고 모든 샘플에서 DNA 무결성을 확인하기 위한 강력한 기준선을 제공했습니다55,56. 그러나 바닷물 지역은 PCR 억제제의 농도가 더 높은 것으로 알려져 있다46. 더욱이, 염도는 황산염 농도와 관련이 있기 때문에 해안 습지의 CH4 배출에 간접적으로 영향을 미칩니다. 바닷물 습지의 염도가 높으면 황산염 환원 박테리아와 메탄 겐 간의 경쟁으로 인해 메탄 생성을 억제하는 황산염 수치 상승과 관련이 있는 반면, 담수 습지의 황산염 농도가 낮으면 메탄 배출량이 높아집니다11,41. 또한, 바닷물과 담수 습지 사이의 CH4 배출 차이는 또한 바다와의 조석 연결에 의해 영향을 받는데,이는 또한 BC의 cPCR에 의한 메탄 순환 관련 유전자의 실패한 검출을 설명할 수 있다. 이 연구에서 cPCR은 LB 및 RV 샘플에서 모든 표적 유전자를 성공적으로 검출했지만 BC 샘플에서 이러한 유전자의 존재를 검출하지 못했으며 pmoA2 유전자가 유일한 예외였습니다. 더욱이 qPCR은 모든 샘플에서 mmoX를 검출할 수 있었지만 cPCR은 BC에서 mmoX를 검출할 수 없었습니다. 이는 qPCR이 cPCR에 비해 PCR 억제제에 대해 더 큰 회복력을 보이며, 이는 검출 메커니즘의 민감도가 향상되었기 때문이라는 것을 나타냅니다58. 또 다른 이유는 qPCR은 PCR 주기의 기하급수적 단계에서 PCR 산물을 식별하는 반면 cPCR은 고원 단계에서 PCR 산물을 검출하기 때문입니다. 이러한 구분으로 인해 qPCR은 시약이 고갈되는 고급 반응 주기 동안 제품 분해에 대한 민감성이 감소할 수 있습니다47.
이 연구는 PCR 억제제 및 가변 프라이머 효율과 관련된 문제를 극복하기 위해 사우스 텍사스 해안 습지에서 메탄 순환 관련 유전자를 식별하는 PCR 기술을 확립했습니다. mcrA 유전자를 표적으로 하는 4개의 primer pair와 pmoA1 유전자를 표적으로 하는 2개의 pair를 사용했으며, 각 set에서 단 한 쌍만이 기존 PCR과 real-time PCR 모두에서 이러한 유전자를 효과적으로 검출했습니다. 이 연구는 pmoA1에 대한 a189-mb6661, pmoA2에 대한 206f-703b, mmoX에 대한 536F-898R 및 mcrA에 대한 ML을 포함하여 메탄 순환 관련 유전자에 대한 프라이머 쌍 세트를 결론지었습니다. 이는 남부 텍사스의 예를 들어 해안 습지에서 메탄 순환 관련 유전자를 검출하기 위한 적절한 프라이머 선택의 중요성을 시사합니다. 또한, 이 연구는 mmoX 가 시원한 계절에 가장 풍부했던 반면 pmoA1 은 따뜻한 계절에 풍부하다는 것을 보여주었습니다.
따라서 이 연구의 결과는 메탄 순환과 관련된 미생물 과정에 대한 과학적 이해를 위한 토대를 제공하며, 남부 텍사스 해안 습지의 계절 및 공간 변화에 걸친 환경 요인의 영향을 강조합니다. 이러한 결과를 바탕으로 향후 연구에서는 논과 담수 습지와 같은 다른 생태계에서 이 접근 방식을 추가로 검증하여 견고성과 일반화 가능성을 높이는 것을 목표로 합니다. 또한 이러한 연구에는 퇴적물 메탄 농도 및 공극수 황화물과 같은 지구화학적 분석과 메타유전체학 및 전사체학과 같은 분자 기술이 포함되며, 이는 메탄 순환 역학에 대한 보다 포괄적인 이해를 제공하기 위한 이러한 기초 작업을 기반으로 합니다.
공개
저자는 선언할 이해 상충이 없습니다.
감사의 말
현장 관찰 및 실험실 분석에 도움을 주신 C-REAL 회원들에게 감사드립니다.
자료
| Name | Company | Catalog Number | Comments |
| 0.2 mL PCR tubes | ThermoFisher Scientific | AB0620 | https://www.thermofisher.com/order/catalog/product/AB0620?SID=srch-srp-AB0620 |
| 0.5 mL PCR Tubes | Promega | E4941 | https://www.promega.com/products/biochemicals-and-labware/tips-and-accessories/0_5ml-pcr-tubes/?catNum=E4941 |
| 10 μL tips | ThermoFisher Scientific | 05-408-187 | Fisherbrand SureGrip Pipet Tip Racked or Reload System Tips Natural; 10μL; | Fisher Scientific |
| 15 mL centrifuge tube | ThermoFisher Scientific | 14-959-53A | https://www.fishersci.com/shop/products/falcon-15ml-conical-centrifuge-tubes-5/p-193301 |
| 200 μL tips | ThermoFisher Scientific | 05-408-190 | Fisherbrand SureGrip Pipet Tip Racked or Reload System Tips Natural; 200μL; | Fisher Scientific |
| 1000 μL tips | ThermoFisher Scientific | 02-707-402 | https://www.fishersci.com/shop/products/sureone-micropoint-pipette-tips-specific-standard-fit/02707402?gclid=Cj0KCQiAp NW6BhD5ARIsACmEb kUsQ9Lu0YIq5i4vWege 17qPdtxIYZyvmJH1cDo ARuwereO1V4GLz9UaA lDREALw_wcB&ef_id=C j0KCQiApNW6BhD5ARI sACmEbkUsQ9Lu0YIq5i 4vWege17qPdtxIYZyvmJ H1cDoARuwereO1V4GLz 9UaAlDREALw_wcB:G:s &ppc_id=PLA_goog_2175 7693617_171052169911_02 707402__715434303113_1555 377385658230343&ev_chn=sh op&s_kwcid=AL!4428!3!71543430 3113!!!g!2366517300713!&gad_source=1 |
| Applied Biosystem Power SYBR Green Master Mix | ThermoFisher Scientific | 4368577 | https://www.thermofisher.com/order/catalog/product/4368577 |
| ArcGIS Pro | esri | https://www.esri.com/en-us/arcgis/products/arcgis-pro/overview?srsltid=AfmBOopatJ4 JvHJfscHRcAaDx0Jz5_Jrl8l5 vYkkBvfOqE-uNSsMghN1 | |
| CFX Duet Real-Time PCR system | Bio-Rad | 12016265 | https://www.bio-rad.com/en-us/product/cfx-duet-real-time-pcr-system?ID=97722926-9ed9-16a4-1d83-c92f587e427a |
| Corning Lambda plus single channel pipettor volume 0.5-10 μL | Sigma-Aldrich | CLS4071-1EA | https://www.sigmaaldrich.com/US/en/product/sigma/cls4071 |
| Corning Lambda plus single channel pipettor volume 100-1000 μL | Sigma-Aldrich | CLS4075-1EA | https://www.sigmaaldrich.com/US/en/product/sigma/cls4075 |
| Corning Lambda plus single channel pipettor volume 20-200 μL | Sigma-Aldrich | CLS4074-1EA | https://www.sigmaaldrich.com/US/en/product/sigma/cls4074 |
| FastDNA spin kit for soil | MP Biomedical | 116560200-CF | https://www.mpbio.com/us/116560000-fastdna-spin-kit-for-soil-samp-cf?srsltid=AfmBOoqOxxGilzY3IHNIZR ajegGTr9MoX1oMZUh 3dcbJqe0UvvukY128 |
| Gene copy calculator | Science Primer | https://scienceprimer.com/copy-number-calculator-for-realtime-pcr . | |
| High speed benchtop centrifuge | ThermoFisher Scientific | 75004241 | https://newlifescientific.com/products/thermo-scientific-sorvall-st16-high-speed-benchtop-centrifuge-75004241?gad_source=1&gclid=Cj0KCQiApN W6BhD5ARIsACmEbkVC_-cCIN9j 20TvYq8iDsBlUR5cPK_1_wN OBEcjMdv-CYVoGCfeOLYaAv enEALw_wcB |
| High speed microcentrifuge | VWR | 75838-336 | https://us.vwr.com/store/product/20546590/null |
| Lysing Matrix E tube | glass bead/ceramic sphere-containing tube | ||
| Microcentrifuge tube | ThermoFisher Scientific | 02-681-320 | https://www.fishersci.com/shop/products/fisherbrand-low-retention-microcentrifuge-tubes-8/02681320?gclid=Cj0KCQiAp NW6BhD5ARIsACm EbkWbG4_o3oUiGk HJPU-_31-CuexDwQ fmWPnfyhBOf2BHXsy K3fFW1toaAgJbEALw_ wcB&ef_id=Cj0KCQiAp NW6BhD5ARIsACmEb kWbG4_o3oUiGkHJPU- _31-CuexDwQfmWPnfy hBOf2BHXsyK3fFW1toa AgJbEALw_wcB:G:s&ppc _id=PLA_goog_21757693 617_171052169911_0268 1320__715434303113_10 349826094968484711&ev _chn=shop&s_kwcid=AL!4 428!3!715434303113!!!g!23 66517300713!&gad_source=1 |
| PCR Master mix | Promega | M7502 | https://www.promega.com/products/pcr/taq-polymerase/master-mix-pcr/?catNum=M7502 |
| Quantiflour ONE dsDNA system | Promega | E4871 | https://www.promega.com/products/rna-analysis/dna-and-rna-quantitation/quantifluor-one-dsdna-system/?gad_source=1&gbraid=0AAAAAD _rg189yJTY3cxeVqMdu8RPx10 Ma&gclid=CjwKCAjwxNW2BhAk EiwA24Cm9FUgViPNyWq7UfZL VeeoroLAZ5JIP6w07RGK_4D0w oZgAqf-G1XTmxoCxm8QAvD_B wE&catNum=E4871 |
| Quantus Fluorometer | Promega | E6150 | https://www.promega.com/products/microplate-readers-fluorometers-luminometers/fluorometers/quantus-fluorometer/?catNum=E6150 |
| YSI Pro 2030 | YSI a xylem brand | 603174 | https://www.ysi.com/product/id-p2030/pro2030-kits |
참고문헌
- Xu, T., et al. Wetlands of international importance: Status, threats, and future protection. Int J Environ Res Public Health. 16 (10), 1818 (2019).
- Corn, M. L. . Deepwater Horizon oil spill: coastal wetland and wildlife impacts and response. , (2010).
- Hendriks, I. E., Sintes, T., Bouma, T. J., Duarte, C. M. Experimental assessment and modeling evaluation of the effects of the seagrass Posidonia oceanica on flow and particle trapping. Marine Ecology Progress Series. 356, 163-173 (2008).
- Krause, S. J. E., Treude, T. Deciphering cryptic methane cycling: Coupling of methylotrophic methanogenesis and anaerobic oxidation of methane in hypersaline coastal wetland sediment. Geochimica et Cosmochimica Acta. 302, 160-174 (2021).
- Reddy, K. R., DeLaune, R. D., Inglett, P. W. . Biogeochemistry of Wetlands: Science and Applications. , (2022).
- La, W., et al. Sulfate concentrations affect sulfate reduction pathways and methane consumption in coastal wetlands. Water Research. 217, 118441 (2022).
- Derwent, R. G. Global warming potential (GWP) for methane: Monte Carlo analysis of the uncertainties in Global Tropospheric Model predictions. Atmosphere. 11 (5), 486 (2020).
- Potter, C., et al. Methane emissions from natural wetlands in the United States: Satellite-derived estimation based on ecosystem carbon cycling. Earth Interactions. 10 (22), 1-12 (2006).
- . Understanding global warming potentials Available from: https://www.epa.gov/ghgemissions/understanding-global-warming-potentials (2024)
- Wallenius, A. J., Dalcin Martins, P., Slomp, C. P., Jetten, M. S. M. Anthropogenic and environmental constraints on the microbial methane cycle in coastal sediments. Front Microbiol. 12, 631621 (2021).
- Qu, Y., et al. Salinity causes differences in stratigraphic methane sources and sinks. Environmental Science and Ecotechnology. 19, 100334 (2024).
- Vizza, C., West, W. E., Jones, S. E., Hart, J. A., Lamberti, G. A. Regulators of coastal wetland methane production and responses to simulated global change. Biogeosciences. 14 (2), 431-446 (2017).
- van Dijk, G., et al. Salinization lowers nutrient availability in formerly brackish freshwater wetlands; unexpected results from a long-term field experiment. Biogeochemistry. 143 (1), 67-83 (2019).
- Aronson, E., Allison, S., Helliker, B. R. Environmental impacts on the diversity of methane-cycling microbes and their resultant function. Front Microbiol. 4, 225 (2013).
- Reeburgh, W. S. Oceanic methane biogeochemistry. Chem Rev. 107 (2), 486-513 (2007).
- Thauer, R. K. Biochemistry of methanogenesis: a tribute to Marjory Stephenson. Marjory Stephenson Prize Lecture. Microbiology (Reading). 144 (Pt 9), 2377-2406 (1998).
- Thauer, R. K. Anaerobic oxidation of methane with sulfate: on the reversibility of the reactions that are catalyzed by enzymes also involved in methanogenesis from CO2. Curr Opin Microbiol. 14 (3), 292-299 (2011).
- Friedrich, M. W. Methyl-coenzyme M reductase genes: Unique functional markers for methanogenic and anaerobic methane-oxidizing Archaea. Methods in Enzymology. 397, 428-442 (2005).
- Reeburgh, W. S. Oceanic methane biogeochemistry. Chem Rev. 107 (2), 486-513 (2007).
- Rasigraf, O., Schmitt, J., Jetten, M. S. M., Lüke, C. Metagenomic potential for and diversity of N-cycle driving microorganisms in the Bothnian Sea sediment. Microbiologyopen. 6 (4), e00475 (2017).
- McDonald, I. R., Bodrossy, L., Chen, Y., Murrell, J. C. Molecular ecology techniques for the study of aerobic methanotrophs. Appl Environ Microbiol. 74 (5), 1305-1315 (2008).
- Tchawa Yimga, M., Dunfield, P. F., Ricke, P., Heyer, J., Liesack, W. Wide distribution of a novel pmoA-like gene copy among type II methanotrophs, and its expression in Methylocystis strain SC2. Appl Environ Microbiol. 69 (9), 5593-5602 (2003).
- Knief, C. Diversity and habitat preferences of cultivated and uncultivated aerobic methanotrophic bacteria evaluated based on pmoA as molecular marker. Front Microbiol. 6, 1346 (2015).
- Huang, I. -. S., et al. Preliminary assessment of microbial community structure of Wind-Tidal Flats in the Laguna Madre, Texas, USA. Biology. 9 (8), 183 (2020).
- Wilding, T. K., Brown, E., Collier, K. J. Identifying dissolved oxygen variability and stress in tidal freshwater streams of northern New Zealand. Environ Monit Assess. 184 (10), 6045-6060 (2012).
- Roy Chowdhury, T., Mitsch, W. J., Dick, R. P. Seasonal methanotrophy across a hydrological gradient in a freshwater wetland. Ecological Engineering. 72, 116-124 (2014).
- Sun, Q. -. Q., et al. Carbon dioxide and methane fluxes: Seasonal dynamics from inland riparian ecosystems, northeast China. Sci Total Environ. 465, 48-55 (2013).
- Lee, S., et al. Comparison and selection of conventional PCR primer sets for studies associated with nitrogen cycle microorganisms in surface soil. Appl Sci. 12 (20), 10314 (2022).
- Bae, K. -. S., et al. Development of diagnostic systems for wide range and highly sensitive detection of two waterborne hepatitis viruses from groundwater using the conventional reverse transcription nested PCR assay. J Virol Methods. 299, 114344 (2022).
- Lecusay, D. . Assessment and Monitoring of Deltaic Wetlands and Fluvial Systems: Refining and Validating a Multimetric Index of Resaca Ecosystem Health. , (2021).
- . FastDNA SPIN Kit for Soil (Cat No. 116560200) Available from: https://www.mpbio.com/media/productattachment/LS082019-EN-FastDNA-SPIN-Kit-for-Soil-116560200-Manual.pdf (2025)
- Luton, P. E., Wayne, J. M., Sharp, R. J., Riley, P. W. The mcrA gene as an alternative to 16S rRNA in the phylogenetic analysis of methanogen populations in landfill. Microbiology (Reading, England). 148 (Pt 11), 3521-3530 (2002).
- Changsoo, L., Jaai, K., Seung Gu, S., Seokhwan, H. Absolute and relative QPCR quantification of plasmid copy number in Escherichia coli. J Biotechnol. 123 (3), 273-280 (2006).
- Harms, G., et al. Real-time PCR quantification of nitrifying bacteria in a municipal wastewater treatment plant. Environ Sci Technol. 37 (2), 343-351 (2003).
- Holmes, A. J., Costello, A., Lidstrom, M. E., Murrell, J. C. Evidence that particulate methane monooxygenase and ammonia monooxygenase may be evolutionarily related. FEMS Microbiol Lett. 132 (3), 203-208 (1995).
- Fuse, H., et al. Oxidation of trichloroethylene and dimethyl sulfide by a marine Methylomicrobium strain containing soluble methane monooxygenase. Biosci Biotechnol Biochem. 62 (10), 1925-1931 (1998).
- Springer, E., Sachs, M. S., Woese, C. R., Boone, D. R. Partial gene sequences for the A subunit of methyl-coenzyme M reductase (mcrI) as a phylogenetic tool for the family Methanosarcinaceae. Int J Syst Bacteriol. 45 (3), 554-559 (1995).
- Costello, A. M., Lidstrom, M. E. Molecular characterization of functional and phylogenetic genes from natural populations of methanotrophs in lake sediments. Appl Environ Microbiol. 65 (11), 5066-5074 (1999).
- Flores, E. A. . Effects of Nutrient Enrichment on Mangrove and Saltmarsh Habitats. , (2022).
- Minjie, H., Jordi, S., Xianyu, Y., Josep, P., Chuan, T. Patterns and environmental drivers of greenhouse gas fluxes in the coastal wetlands of China: A systematic review and synthesis. Environ Res. 186, 109576 (2020).
- Bridgham, S. D., Cadillo-Quiroz, H., Keller, J. K., Zhuang, Q. Methane emissions from wetlands: biogeochemical, microbial, and modeling perspectives from local to global scales. Glob Chang Biol. 19 (5), 1325-1346 (2013).
- Liu, D. Y., Ding, W. X., Jia, Z. J., Cai, Z. C. Relation between methanogenic archaea and methane production potential in selected natural wetland ecosystems across China. Biogeosciences. 8 (2), 329-338 (2011).
- Ke, Z., et al. Methane emissions and methanogenic community investigation from constructed wetlands in Chengdu City. Urban Climate. 39, 100956 (2021).
- Chowdhury, T. R., Dick, R. P. Ecology of aerobic methanotrophs in controlling methane fluxes from wetlands. Applied Soil Ecology. 65, 8-22 (2013).
- Maja, S., et al. Humic substances cause fluorescence inhibition in real-time polymerase chain reaction. Anal Biochem. 487, 30-37 (2015).
- Sanches, T. M., Schreier, A. D. Optimizing an eDNA protocol for estuarine environments: Balancing sensitivity, cost and time. PLOS ONE. 15 (5), e0233522 (2020).
- Xia, Z., et al. Conventional versus real-time quantitative PCR for rare species detection. Ecol Evol. 8 (23), 11799-11807 (2018).
- Bourne, D. G., McDonald, I. R., Murrell, J. C. Comparison of pmoA PCR primer sets as tools for investigating methanotroph diversity in three Danish soils. Appl Environ Microbiol. 67 (9), 3802-3809 (2001).
- Juottonen, H., Galand, P. E., Yrjala, K. Detection of methanogenic Archaea in peat: comparison of PCR primers targeting the mcrA gene. Res Microbiol. 157 (10), 914-921 (2006).
- Lueders, T., Friedrich, M. W. Evaluation of PCR amplification bias by terminal restriction fragment length polymorphism analysis of small-subunit rRNA and mcrA genes by using defined template mixtures of methanogenic pure cultures and soil DNA extracts. Appl Environ Microbiol. 69 (1), 320-326 (2003).
- Vaksmaa, A., Jetten, M. S., Ettwig, K. F., Luke, C. McrA primers for the detection and quantification of the anaerobic archaeal methanotroph 'Candidatus Methanoperedens nitroreducens'. Appl Microbiol Biotechnol. 101 (4), 1631-1641 (2017).
- Ren, G., et al. Electron acceptors for anaerobic oxidation of methane drive microbial community structure and diversity in mud volcanoes. Environ Microbiol. 20 (7), 2370-2385 (2018).
- Goldberg, C. S., et al. Critical considerations for the application of environmental DNA methods to detect aquatic species. Methods in Ecology and Evolution. 7 (11), 1299-1307 (2016).
- McKee, A. M., Spear, S. F., Pierson, T. W. The effect of dilution and the use of a post-extraction nucleic acid purification column on the accuracy, precision, and inhibition of environmental DNA samples. Biological Conservation. 183, 70-76 (2015).
- Gohl, D. M., et al. Systematic improvement of amplicon marker gene methods for increased accuracy in microbiome studies. Nat Biotechnol. 34 (9), 942-949 (2016).
- Ballarini, A., Segata, N., Huttenhower, C., Jousson, O. Simultaneous quantification of multiple bacteria by the BactoChip microarray designed to target species-specific marker genes. PLOS ONE. 8 (2), e55764 (2013).
- Le Mer, J., Roger, P. Production, oxidation, emission and consumption of methane by soils: A review. Eur J Soil Biol. 37 (1), 25-50 (2001).
- Smith, C. J., Osborn, A. M. Advantages and limitations of quantitative PCR (Q-PCR)-based approaches in microbial ecology. FEMS Microbiol Ecol. 67 (1), 6-20 (2009).
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유