Method Article
Visualizzazione delle dinamiche microbiche del ciclo del metano nelle zone umide costiere
In questo articolo
Riepilogo
Il protocollo rileva i geni chiave del ciclo del metano nelle zone umide costiere del Texas meridionale e visualizza la loro distribuzione spaziale per migliorare la comprensione della regolazione del metano e dei suoi impatti ambientali in questi ecosistemi dinamici.
Abstract
Le zone umide costiere sono la più grande fonte biotica di metano, dove i metanogeni convertono la materia organica in metano e i metanotrofi ossidano il metano, svolgendo così un ruolo fondamentale nella regolazione del ciclo del metano. Le zone umide del Texas meridionale, soggette a frequenti eventi meteorologici, livelli di salinità fluttuanti e attività antropiche dovute ai cambiamenti climatici, influenzano il ciclo del metano. Nonostante l'importanza ecologica di questi processi, il ciclo del metano nelle zone umide costiere del Texas meridionale rimane insufficientemente esplorato. Per colmare questa lacuna, abbiamo sviluppato e ottimizzato un metodo per rilevare i geni correlati ai metanogeni e ai metanotrofi, tra cui mcrA come biomarcatore per i metanogeni e pmoA1, pmoA2 e mmoX come biomarcatori per i metanotrofi. Inoltre, questo studio mirava a visualizzare i modelli di distribuzione spaziale e temporale del metanogeno e dell'abbondanza di metanotropo utilizzando il software GIS (Geographic Information System) ArcGIS Pro. L'integrazione di queste tecniche molecolari con la visualizzazione geospaziale avanzata ha fornito informazioni critiche sulla distribuzione spaziale e temporale delle comunità di metanogeni e metanotrofi nelle zone umide del Texas meridionale. Pertanto, la metodologia stabilita in questo studio offre un solido quadro per mappare le dinamiche microbiche nelle zone umide, migliorando la nostra comprensione del ciclo del metano in condizioni ambientali variabili e supportando studi più ampi sui cambiamenti ecologici e ambientali.
Introduzione
Le zone umide costiere sono ecosistemi vitali che contribuiscono alla regolazione del clima, alla conservazione della biodiversità e alla gestione delle acque attraverso processi come il sequestro del carbonio, l'evapotraspirazione e le emissioni di metano (CH4)1. Questi ecosistemi, comprese le zone umide di acqua dolce e salata2, sono altamente produttivi e fungono da zone critiche per l'assorbimento di anidride carbonica (CO2) e catturano la materia organica dagli ambienti terrestri e marini 3,4. Le interazioni dinamiche all'interno di queste zone umide stimolano la produzione e il consumo microbico di CH4 5, posizionandole come una delle maggiori fonti naturali di CH46. Essendo il secondo gas serra più importante, il CH4 ha un potenziale di riscaldamento globale circa 27-30 volte superiore a quello del CO2 4,7,8,9, rendendo lo studio delle emissioni di CH 4 dalle zone umide costiere essenziale nell'era del cambiamento climatico. L'emissione di CH4 è influenzata da vari fattori ambientali, in particolare dalla salinità, che svolge un ruolo cruciale nei processi microbici10. Le zone umide d'acqua dolce contribuiscono in modo significativo al metano atmosferico a causa dei loro livelli di solfato più bassi, che facilita una maggiore produzione microbica di CH4, mentre le zone umide d'acqua salata tendono generalmente a emettere meno CH4 a causa di concentrazioni di solfato più elevate 11,12,13.
Le emissioni di CH4 dalle zone umide costiere sono generalmente controllate da due gruppi di microrganismi, noti come metanogeni e metanotrofi14. I metanogeni producono CH4 nei sedimenti anossici scomponendo substrati come formiato, acetato, idrogeno o composti metilati attraverso un processo noto come metanogenesi15. L'enzima importante in questa via è la metil-coenzima M reduttasi (MCR), in quanto catalizza la fase finale e limitante della metanogenesi 15,16,17. Il gene mcrA, che codifica per la subunità alfa di MCR, è un marcatore funzionale che può essere trovato in tutti gli archei metanogenici18. Inoltre, nelle zone umide costiere, la zona di transizione solfato-metano (SMTZ) si forma al di sopra della zona metanogenica, dove il metano che si diffonde verso l'alto e il solfato che si muove verso il basso convergono e si esauriscono19. All'interno di questa zona, gli archei metanotrofi anaerobici (ANME) ossidano il metano in anidride carbonica utilizzando l'enzima MCR, mentre i batteri solfato-riduttori (SRB) riducono il solfato in solfuro. Gli SRB superano i metanogeni per l'idrogeno e l'acetato, limitando la produzione di metano fino all'esaurimento del solfato16,17.
Al contrario, i batteri metanotrofici aerobici ossidano CH4 in ambienti aerobici20, utilizzando diverse forme di metano monoossigenasi (MMO). Questi includono il particolato metano monoossigenasi (pMMO), un enzima contenente rame incorporato nella membrana intracitoplasmatica, e la metano monoossigenasi solubile (sMMO), un enzima contenente ferro presente nel citoplasma. Tuttavia, per pMMO, ci sono tre operoni genici pmoCAB21; tra questi, il gene pmoA è il più conservativo per tutti i metanotrofi. Esistono due diversi geni biomarcatori per pmoA: pmoA1 e pmoA222. Inoltre, per una comprensione completa dei metanotrofi, il gene mmoX viene utilizzato come strumento in biologia molecolare per identificare i metanotrofi23 contenenti sMMO. Questa distinzione nelle vie metaboliche e nei requisiti ambientali dei metanogeni e dei metanotrofi aerobici evidenzia le complesse interazioni microbiche che regolano il ciclo del metano negli ecosistemi delle zone umide costiere.
La zona umida di Boca Chica (BC), un ambiente produttivo di acqua salata nel sud del Texas, subisce influenze di marea dal Golfo del Messico (GOM), portando a livelli di salinità superficiale variabili, soprattutto a causa della sua vicinanza alla Laguna Madre24 ipersalina. Questa azione di marea, alternata tra alte e basse maree, provoca fluttuazioni dei livelli di ossigeno25 che potrebbero alterare l'attività del metanogeno e del metanotrofio nei sedimenti26. Al contrario, le zone umide costiere d'acqua dolce sono considerate un hotspot significativo per i flussi CH4 27. Le zone umide costiere d'acqua dolce nel sud del Texas, tra cui Resaca Del Rancho Viejo (RV) e Lozano Banco (LB), distanti dagli effetti delle maree del GOM, hanno una gestione idrologica distinta. RV sperimenta flussi di impulsi integrati dall'acqua del fiume durante i bassi livelli dell'acqua, mentre LB funziona come un sistema di flusso offline senza tale integrazione. Inoltre, RV e LB mantengono livelli di salinità più bassi a causa di un elevato scarico di acqua dolce pompata artificialmente e del fatto di essere un lago oxbow. I diversi fattori ambientali possono influenzare in modo significativo il ciclo del metano nelle zone umide costiere del Texas meridionale. Tuttavia, il ciclo del metano nelle zone umide costiere del Texas meridionale rimane un'area che deve ancora essere indagata a fondo.
La reazione a catena della polimerasi (PCR) e la PCR in tempo reale (chiamata anche PCR quantitativa [qPCR]) rappresentano tecniche fondamentali e ampiamente utilizzate per rilevare e quantificare l'abbondanza relativa di geni specifici in campioni ambientali. Queste tecniche amplificano specificamente regioni mirate del DNA per indicare la presenza e la quantità relativa di geni correlati al ciclo CH4, fornendo indicatori del potenziale ciclo del metano. Tuttavia, la disponibilità e l'efficacia dei set di primer per PCR potrebbero essere limitate da vari fattori inibitori nel DNA ambientale estratto, essendo influenzati dai tipi di ambienti28,29. Pertanto, questo studio ha stabilito principalmente un metodo PCR ottimale per rilevare la presenza di geni correlati al ciclo CH4 nelle zone umide costiere del Texas meridionale (Figura 1) e quindi ha visualizzato la loro abbondanza relativa quantificata in questi ecosistemi. I risultati di questo studio possono essere applicati ad altre regioni costiere per migliorare la comprensione del ciclo CH4 e delle dinamiche microbiche in diversi ecosistemi costieri.
Protocollo
1. Raccolta dei campioni
- Raccogli campioni di sedimenti utilizzando un campionatore a pinza per sedimenti o una pala.
NOTA: I campioni sono stati raccolti da due stazioni di tre distinte zone umide costiere durante le stagioni fresche (ottobre-febbraio, la temperatura media è di 20 °C) e calde (aprile-giugno, la temperatura media è di 27 °C) del 2023 e del 2024. Un campionatore di sedimenti è stato utilizzato quando i campioni sono stati raccolti dalle zone umide costiere d'acqua dolce (Figura 2) e una pala è stata utilizzata per le zone umide costiere di acqua salata influenzate dalle maree. - Abbassare prima il campionatore nel corpo idrico poco profondo e lasciarlo affondare sulla superficie del sedimento (entro i primi 50 cm) sotto il suo stesso peso, riducendo al minimo il disturbo alla struttura del sedimento, come mostrato nella Figura 2.
- Estrarlo dalla colonna d'acqua, dove la profondità era tipicamente compresa tra 60 cm e 215 cme 30, trasferire i campioni di sedimento in sacchetti con chiusura a zip e conservarli immediatamente in una ghiacciaia.
NOTA: Pulire e lavare il campionatore a benna con acqua deionizzata (DI) prima di procedere alle stazioni successive. - Conservare immediatamente tutti i campioni a -20 °C in laboratorio.
- In ogni sito di campionamento, misurare i parametri di qualità delle acque superficiali come la salinità e la temperatura in situ utilizzando un misuratore di qualità dell'acqua multiparametrico.
NOTA: Le sonde sono state risciacquate con acqua deionizzata (acqua deionizzata) dopo l'uso in ciascuna stazione.
2. Estrazione del DNA genomico
- Scongelare i campioni a temperatura ambiente prima di iniziare la procedura per l'estrazione del DNA genomico.
- Trasferire circa 500 mg di campioni di sedimento in una provetta da 15 ml e centrifugare a 4.250 × g per 3 minuti per rimuovere tutta l'acqua.
- Estrarre il DNA genomico utilizzando un kit di estrazione del DNA per il terreno seguendo il protocollo31 del produttore con una piccola modifica e conservare immediatamente a -20 °C.
NOTA: le modifiche sono state apportate per ridurre la ridondanza e rendere efficiente il flusso di lavoro.- Aggiungere fino a 500 mg di campione di terreno in una provetta contenente perle di vetro/sfere di ceramica.
- Aggiungere 978 μl di tampone fosfato di sodio al campione nella provetta contenente il microsfera/sfera.
- Aggiungere 122 μl di soluzione tampone di lisi al campione nella provetta contenente il microsfone/sfera per solubilizzare i contaminanti esterni.
- Omogeneizzare con un omogeneizzatore a 5 volte il livello di velocità per 20 s e ripetere 2 volte.
NOTA: In questo studio è stato utilizzato un omogeneizzatore per macina perline, motivo per cui la velocità è stata regolata. - Centrifugare il composto per 10 minuti a 14.000 × g.
- Trasferire il surnatante in una provetta da microcentrifuga da 2,0 mL pulita.
- Aggiungere 250 μl di soluzione di precipitazione proteica (PPS) per separare gli acidi nucleici solubilizzati dai detriti cellulari e dalla matrice lisizzante. Mescolare capovolgendo il tubo 10 volte.
- Centrifugare a 14.000 × g per 5 min per far precipitare il pellet, rimuovendo i detriti cellulari e la matrice lisiante.
- Trasferire il surnatante in una provetta da microcentrifuga pulita da 15 mL.
- Aggiungere 1,0 mL della sospensione Binding Matrix al surnatante nella provetta da 15 mL.
NOTA: Agitare la sospensione Binding Matrix per risospendere prima di aggiungerla. - Consentire il legame del DNA alla matrice di legame posizionando le provette su un rotatore per 2 minuti.
- Posizionare tutte le provette su un rack e incubare per 3 minuti per consentire la sedimentazione della matrice legante.
- Dopo 3 minuti, scartare 750 μl e mescolare delicatamente il surnatante rimanente con il pellet utilizzando una pipetta.
- Trasferire 750 μl della miscela in un filtro SPIN e centrifugare a 14.000 × g per 1 minuto. Svuotare il tubo di raccolta e riutilizzarlo. Ripetere con il composto rimanente.
- Aggiungere 500 μl della soluzione di lavaggio preparata (con l'aggiunta della quantità appropriata di etanolo) per solubilizzare ulteriormente le impurità. Risospendere delicatamente il pellet utilizzando la forza del liquido dalla punta della pipetta.
- Centrifugare a 14.000 × g per 1 minuto per rimuovere le impurità. Svuotare il tubo di raccolta e riutilizzarlo.
- Centrifugare nuovamente a 14.000 × g per 2 minuti senza aggiungere nulla.
- Sostituire il tubo di raccolta con un tubo di raccolta nuovo e pulito e asciugare all'aria il filtro centrifugo per 5 minuti a temperatura ambiente.
- Aggiungere 50 μL di acqua priva di DNAsi (DES) e centrifugare a 14.000 × g per 1 minuto.
NOTA: In questo protocollo, 50 μL di DES sono stati utilizzati per l'eluizione del DNA per garantire un recupero e una stabilità ottimali del DNA ambientale estratto (eDNA).
3. Quantificazione del DNA
- Aggiungere 1 μl di DNA estratto con 200 μl di colorante fluorescente in una provetta da 0,5 mL e mescolare accuratamente mediante pipettaggio.
- Avvolgere immediatamente la provetta con un foglio di alluminio in modo che la luce non possa penetrare e incubare a temperatura ambiente per 5 minuti.
- Misurare la concentrazione di DNA utilizzando un fluorimetro in modalità di concentrazione del DNA ONE seguendo il protocollo del produttore.
4. Rilevamento di 16S rRNA, pmoA1 , pmoA2 , mmoX e mcrA mediante PCR convenzionale
- Prima di eseguire la PCR convenzionale (cPCR), scongelare tutti i campioni e i reagenti in un secchiello per il ghiaccio.
- Diluire tutti i campioni di eDNA estratti a 10 ng/μL.
NOTA: Un elenco di inneschi è riportato nella Tabella 1. - Preparare una miscela di reazione cPCR da 25 μl per ciascun campione, inclusi 12,5 μl di 2x PCR Master mix, 0,5 μl di primer diretti e inversi (10 μM) (vedere la Tabella 1 per l'elenco dei primer), 1 μl di eDNA da 10 ng/μl e 10,5 μl di acqua priva di nucleasi.
NOTA: Preparare la miscela master con un volume sufficiente per un campione aggiuntivo per ridurre al minimo gli errori di pipettaggio. - Eseguire la reazione di cPCR seguendo il protocollo che consiste in una denaturazione iniziale a 95 °C per 2 minuti, seguita da 40 cicli di denaturazione a 95 °C per 45 s, estensione a 72 °C per 30 s e estensione finale a 72 °C per 5 m, con temperatura di ricottura variabile per diversi primer (vedere la Tabella 1 per le temperature di ricottura di diversi primer utilizzati per diversi geni).
NOTA: Per il gene mcrA , i primer ML hanno mostrato la banda desiderata utilizzando una velocità di rampa lenta di 0,1 °C/s tra le fasi di ricottura ed estensione per i primi cinque cicli32. - Visualizza i prodotti cPCR in un gel di agarosio prestato con bromuro di etidio (EtBr).
NOTA: Utilizzare gel di agarosio al 2,5% per una scala da 50 bp e gel allo 0,9% per una scala da 1 kb. Utilizzare 1x tampone TAE per un gel di agarosio al 2,5% e 0.5x tampone TAE per un gel di agarosio allo 0.9%.
5. Rilevamento di pmoA1 , pmoA2 , mmoX e mcrA mediante PCR quantitativa in tempo reale
NOTA: I geni mirati al metanogeno e al metanotrofio come l'abbondanza di pmoA1, pmoA2, mmoX e mcrA sono stati osservati mediante qPCR utilizzando un sistema di PCR in tempo reale.
- Preparare gli standard per ciascun gene separatamente per ottenere la curva standard per ciascun gene.
- Amplificare il gene bersaglio con la cPCR, utilizzando i campioni che hanno prodotto la banda più luminosa durante l'elettroforesi su gel di ciascun gene. Seguire il metodo e i primer descritti nel paragrafo 4.
- Purificare i prodotti amplificati utilizzando un kit di estrazione su gel e misurare la concentrazione di DNA seguendo il metodo descritto nella sezione 3 e conservare a -20 °C.
NOTA: Aliquotare gli standard purificati in provette separate per un ulteriore utilizzo. - Calcola le copie dei geni dalla concentrazione di DNA misurata utilizzando il sito Web di calcolo del numero di copie per la qPCR.
- Diluire il numero di copie calcolato dello standard con acqua priva di nucleasi per preparare ogni standard, da 108 a 102 copie/μL, prima di eseguire la qPCR.
- Preparare la curva standard utilizzando tre repliche di ciascun numero di copie, incluso un controllo negativo (NTC, senza DNA stampo).
NOTA: Il valore R2 di ciascuna curva standard era maggiore di 0,99.
- Preparare una miscela di reazione qPCR da 20 μl per tutti i campioni, gli standard e le NTC. Eseguire l'analisi qPCR in triplicati per tutti i campioni.
- Posizionare tutti i componenti della reazione, tra cui la master mix qPCR, i primer, l'acqua priva di nucleasi, gli standard e i campioni, su una griglia per il ghiaccio prima di iniziare.
- Preparare una miscela di reazione qPCR da 20 μl per ciascun campione, standard e NTC, contenente 10 μl di master mix SYBR Green, 0,5 μl di ogni primer diretto e inverso da 10 μM, 8 μl di acqua priva di nucleasi e 1 μl di DNA stampo da 10 ng/μl o acqua standard o ID, rispettivamente.
NOTA: Utilizzare i set di primer che hanno dimostrato di produrre risultati ottimali per i geni pmoA1 e mcrA nella PCR convenzionale per qPCR (vedere la Tabella 1). Per migliorare l'accuratezza, eseguire ogni campione in triplice copia. I passaggi seguenti forniscono un metodo efficiente per la preparazione di campioni triplicati con un volume combinato di 60 μl per campione.- Preparare la miscela di reazione per combinare la miscela master, i primer diretti e inversi per il gene specifico e l'acqua priva di nucleasi in una provetta da 2 mL, ad eccezione del DNA stampo.
NOTA: Preparare il volume della miscela di reazione tenendo conto dell'errore di pipettaggio. Ad esempio, se sono presenti 24 campioni e 8 standard, calcolare il volume totale per 33 reazioni anziché 32 reazioni per ridurre al minimo gli errori di pipettaggio. In questo caso, il volume totale richiesto per le reazioni triplicate sarebbe il seguente: 990 μL di master mix (33 campioni x 3 repliche x 10 μL), 49,5 μL di primer diretti (33 campioni x 3 repliche x 0,5 μL), 49,5 μL di primer inversi (33 campioni x 3 repliche x 0,5 μL) e 792 μL di acqua priva di nucleasi (33 campioni x 3 repliche x 8 μL). - Preparare le provette per PCR in base al numero di campioni e agli standard.
- Erogare 57 μl della miscela di reazione preparata in ciascuna provetta per PCR.
NOTA: Per eseguire la qPCR per ciascun campione in triplicato, preparare un volume totale di miscela di reazione di 57 μl per campione (escluso DNA stampo, standard o acqua). Questo volume sarà diviso equamente in tre pozzetti per un campione, con 19 μl assegnati a ciascun pozzetto. - Aggiungere 3 μL di DNA stampo, acqua standard o priva di nucleasi a ciascuna provetta e mescolare picchiettando delicatamente il fondo della provetta.
NOTA: Il volume totale della miscela di reazione preparata per un campione sarebbe di 60 μl in ciascuna provetta.
- Preparare la miscela di reazione per combinare la miscela master, i primer diretti e inversi per il gene specifico e l'acqua priva di nucleasi in una provetta da 2 mL, ad eccezione del DNA stampo.
- Aliquotare 20 μl della miscela di reazione preparata da ciascuna provetta nei pozzetti designati di una piastra qPCR a 96 pozzetti. Sigillare la piastra PCR con una pellicola sigillante PCR adesiva utilizzando un applicatore.
- Centrifugare la piastra sigillata a 1.000 × g per 1 minuto per garantire una miscelazione corretta per eliminare eventuali bolle all'interno dei pozzetti.
- Posizionare la piastra PCR nel termociclatore. Accendere la macchina qPCR e quindi aprire il software correlato per impostare il protocollo.
- Impostare il protocollo in base alle linee guida della miscela master qPCR. Utilizzare il seguente protocollo: 95 °C per 10 minuti, seguiti da 95 °C per 15 s e un passo di estensione a 72 °C per 30 s. Eseguire la fase di ricottura alla temperatura di ricottura specificata per i relativi inneschi nella Tabella 1 per 45 s. Condurre tutte le esecuzioni di qPCR per 35 cicli.
- Impostare la piastra qPCR a 96 pozzetti con standard e NTC nella stessa configurazione della piastra contenente il campione.
- Utilizzare il metodo della curva standard di quantificazione assoluta per quantificare il prodotto amplificato e il numero di copie geniche in ciascun campione33.
6. Visualizzazione dei geni del ciclo del metano nella mappa delle zone umide costiere del Texas meridionale
- Aprire il software GIS (Geographic Information System) ArcGIS Pro e salvare il file di progetto con il nome Area di studio nella cartella specificata sul computer.
- Clicca su Mappa in alto a sinistra | Mappa di base e selezionare Terreno con etichette come mappa di base.
- Fare clic su Localizza | Cerca e quando si apre la barra di ricerca, individua l'area di studio digitando il nome dell'area; L'area verrà visualizzata.
- Disegna l'area specifica utilizzando la georeferenziazione.
- Clicca su Visualizza | Riquadro del catalogo dal livello superiore.
- Fare doppio clic su Cartella dal catalogo | Nome file.
- Fare clic con il pulsante destro del mouse sul file del geodatabase (.gdb) e quindi fare clic su Nuovo | Verranno visualizzati Classe entità geografiche e Crea classe di entità geografiche .
- Digitare la casella Nome e alias e fare clic su Fine in basso.
- Clicca su Visualizza | Contenuto. Il nome dell'alias verrà visualizzato nel riquadro dei contenuti.
- Fare clic su Modifica dal livello superiore | Creare. Verrà visualizzato il riquadro Crea funzionalità . Fare doppio clic sul riquadro Alias in Crea funzionalità per visualizzare Configura opzioni di feedback utensile .
- Seleziona Linee, quindi disegna le linee sulla mappa per creare un confine esterno dell'area di studio. Al termine, fai doppio clic sulla mappa.
- Riduci a icona il software GIS, quindi apri un foglio di calcolo. Digitare il nome del campione nella prima colonna, immettere la latitudine nella seconda colonna e la longitudine nella terza colonna. Utilizzare le quattro colonne successive per i dati qPCR di pmoA1, pmoA2, mmoX e mcrA.
- Salvare il file in formato CSV in una cartella specifica del computer.
- Aprire nuovamente il software GIS e fare clic su Aggiungi dati | Dati punto XY.
- Selezionare il file CSV dalla cartella del computer nella casella Tabella di input . Rinominare il nome del file nella classe di entità geografiche di output, quindi fare clic su Esegui per visualizzare i punti di campionamento sulla mappa.
- Clicca sulbarra di ricerca in alto e cerca Kriging.
- Selezionare il file della stazione di campionamento, quindi selezionare pmoA1.
- Fare clic sul pulsante Ambiente | selezionare la stazione di campionamento in layer e maschera | fare clic su Esegui.
- Seguire i protocolli menzionati nei passaggi 6.9, 6.10 e 6.11 per creare Kriging per pmoA2, mmoX e mcrA per tutta l'area di studio.
- Creare un layout della mappa.
- Fare clic su Inserisci dal livello superiore | Nuovo layout e selezionare ANSI - Orizzontale.
- Fare clic su Cornice mappa, selezionare la mappa con il Kriging e posizionare tutte le mappe nel layout disegnando un rettangolo. In questo modo la mappa sarà visibile nel layout.
- Seleziona la freccia nord e posizionala nel layout per indicare la direzione nord.
- Selezionare Barra scala per visualizzare la scala dell'area sulla mappa.
- Fare clic su Legenda per visualizzare le legende, quindi posizionarla nel layout.
- Fare clic su Griglia e selezionare una delle opzioni del reticolo nero. In questo modo verrà creata la griglia con latitudine e longitudine e visualizzata nel riquadro dei contenuti con l'etichetta Reticolo dell'etichetta orizzontale nera.
- Fare doppio clic su Reticolo orizzontale nero dell'etichetta, quindi selezionare Componenti. Fare clic su Ticks 1 e Grid e rimuovere questi componenti facendo clic sul segno di croce alla loro destra.
- Fai clic su Condividi dal livello superiore, quindi fai clic su Esporta layout. Selezionare il tipo di file come PDF, salvare il file sul computer utilizzando la casella Nome, impostare la risoluzione verticale su 500 DPI e fare clic su Esporta per creare il file PDF della mappa.
Risultati
Per comprendere la distribuzione e l'abbondanza dei geni correlati al ciclo CH4 (mcrA, pmoA1, pmoA2 e mmoX) nelle zone umide costiere del Texas meridionale, l'eDNA estratto da ciascun campione è stato analizzato mediante cPCR e qPCR. I primer universali per ciascun biomarcatore sono stati selezionati per eseguire la cPCR da studi precedenti (Tabella 1)22,34,35,36,37 e sono state apportate modifiche per ottimizzare le temperature e le concentrazioni di ricottura in base alle caratteristiche del campione e alle condizioni ambientali. Ad esempio, l'rRNA 16S, il pmoA1 e il pmoA2 non si sono amplificati utilizzando la temperatura di ricottura standard perché la maggior parte di essi si è amplificata ben al di sotto della temperatura di ricottura standard (dati non mostrati). Inoltre, durante il processo di cPCR, una concentrazione di 10 ng/μL di DNA ha mostrato una banda distinta per i geni pmoA1, pmoA2 e mmoX, mentre l'utilizzo di 5 ng/μL di DNA stampo ha mostrato una banda più luminosa per il gene mcrA (Supplemental File 1-Supplemental Figure S1).
L'analisi cPCR ha rivelato variabilità spaziale nel rilevamento dei geni in tre tipi di zone umide costiere del Texas meridionale. Come mostrato nella Tabella 2, i geni mcrA sono stati rilevati in tutti i campioni di LB e RV, che sono zone umide costiere d'acqua dolce. Tuttavia, la cPCR non è stata in grado di rilevare alcun gene mcrA nel sedimento campionato della zona umida di acqua salata, suggerendo un potenziale legame tra salinità e distribuzione del metanogeno (Supplemental File 1-Supplemental Figure S2). Per il rilevamento di mcrA, in questo studio sono state utilizzate quattro coppie di primer, tra cui MLF-MLR, MCRf-MCRr, ME1F-ME2R e McrA 159F-McrA 345R (Tabella 1); tra questi, solo le coppie di primer ML sono state in grado di rilevare la comunità metanogena nelle zone umide del Texas meridionale (Supplemental File 1-Supplemental Figure S2, Supplemental Figure S3 e Supplemental Figure S4). Al contrario, per i metanotrofi aerobi, il gene pmoA1 è stato rilevato in tutti i campioni di RV, ma era assente nei campioni di LB e BC (Supplemental File 1-Supplemental Figure S5). Per identificare pmoA1, A189 è stato utilizzato come innesco diretto e A682 e mb661 come innesco inverso, dove le coppie di primer A189-A682 possono anche rilevare amoA35,38. Tuttavia, questa coppia di primer è stata in grado di rilevare la presenza di geni nella cPCR in questo studio in cui le immagini del gel hanno mostrato una banda molto debole, suggerendo la bassa abbondanza del gene pmoA1. Un altro primer inverso mb661 è stato progettato per identificare il gene pmoA1 specificamente con A189 come primer diretto38. Quando mb661 è stato utilizzato come innesco inverso, la cPCR ha mostrato una banda più luminosa (Supplemental File 1-Supplemental Figure S6), che è stata successivamente utilizzata nella qPCR. È interessante notare che il gene pmoA2 è stato rilevato in due campioni di BC e un campione di LB, ma era assente in un campione di RV (Supplemental File 1-Supplemental Figure S7). Uno studio precedente ha rilevato che la temperatura di ricottura è di 66 °C e 60 °C per pmoA2, dove a 60 °C sono stati osservati due ampliconi di dimensioni diverse, 245 e 438 bp, mentre a 66 °C, i primer 206f-703b possono amplificare solo pmoA2 con 438 bp di dimensione22. Tuttavia, in questo studio, l'amplificazione ha avuto successo a una temperatura di ricottura di 60 °C, producendo una banda della dimensione prevista (438 bp) nei siti LB e BC (File supplementare 1-Figura supplementare S7). Inoltre, il gene mmoX è stato rilevato in un campione di LB ma era assente in un campione di RV (Supplemental File 1-Supplemental Figure S8) ed era completamente assente nei campioni di BC. Le coppie di primer utilizzate per mmoX erano adatte anche per studiare le zone umide del Texas meridionale in questo studio. Una temperatura di ricottura di 55 °C36 ha prodotto una banda luminosa per mmoX in cPCR (File supplementare 1-Figura supplementare S8). Per verificare l'integrità del campione, l'rRNA 16S è stato utilizzato come controllo positivo con amplificazione riuscita dell'rRNA 16S in tutti i campioni durante la cPCR, suggerendo che i campioni non erano contaminati (File supplementare 1-Figura supplementare S9).
Per valutare i cambiamenti temporali e spaziali nell'abbondanza genica, l'analisi qPCR è stata condotta su tutti i geni bersaglio utilizzando set di primer ottimizzati identificati nella cPCR. La qPCR dei geni pmoA2 e mmoX è stata condotta con gli stessi primer utilizzati nella cPCR. Tuttavia, per il gene pmoA1 , è stata utilizzata la coppia di primer A189-mb661 e per il gene mcrA è stata selezionata la coppia di primer MLF-MLR, poiché entrambi hanno prodotto buoni risultati nella cPCR. Sono state prese in considerazione anche le differenze stagionali e la variazione di salinità nelle zone umide del Texas meridionale, in quanto potrebbero influenzare le dinamiche della comunità microbica. Come mostrato nella Figura 3 e nella Figura 4, c'era una differenza nell'abbondanza genica dei metanogeni e dei metanotrofi tra due diversi periodi di campionamento. Mentre non vi è stata alcuna variazione stagionale della salinità nelle zone umide costiere d'acqua dolce, è stata osservata una differenza stagionale della salinità nelle zone umide costiere di acqua salata39, che potrebbe spiegare la variazione dell'abbondanza di metanogeno e metanotrofi. La Figura 3 mostra che durante la stagione fredda, l'mmoX era molto abbondante in tutte queste zone umide con la più alta abbondanza di 1,46 × 104 copie/μL. C'era una piccola percentuale di pmoA2 in una stazione di LB, RV e BC. Tuttavia, la qPCR non è stata in grado di rilevare pmoA1 e mcrA a causa della bassa abbondanza durante la stagione fredda. Al contrario, pmoA1 era per lo più abbondante nella stagione calda con la massima abbondanza di 2,44 × 103 copie/μL (Figura 4). L'mmoX e l'mcrA sono stati rilevati anche nei campioni di sedimenti superficiali di LB (entro i primi 50 cm), mostrando l'abbondanza massima di 1,92 × 102 copie/μL e 2,04 × 102 copie/μL, rispettivamente, nonostante la loro abbondanza relativamente bassa durante la stagione calda. È interessante notare che, durante la stagione calda, pmoA2 non è stato rilevato affatto e nessuno dei geni bersaglio è stato rilevato nei campioni di BC, probabilmente influenzato da fattori ambientali unici di questa zona umida di acqua salata39 durante la stagione calda nel sud del Texas.
Questo studio dimostra distinte variazioni spaziali e stagionali nella distribuzione dei geni correlati al ciclo CH4 nelle zone umide costiere. In particolare, i geni mcrA sono stati rilevati solo nel campione di LB d'acqua dolce, suggerendo una preferenza per l'habitat. I geni mmoX e pmoA2 erano presenti in tutti i campioni nelle stagioni fredde, mentre pmoA1 si esibiva in ambienti di acqua dolce durante le stagioni calde. Questi risultati evidenziano l'influenza sia delle condizioni ambientali che dei cambiamenti stagionali sulle attività microbiche correlate al ciclo CH4 . La visualizzazione migliorata utilizzando l'interpolazione kriging, come mostrato nella Figura 3 e nella Figura 4, ha migliorato la nostra comprensione delle attività microbiche correlate al ciclo CH4 negli ecosistemi delle zone umide costiere.
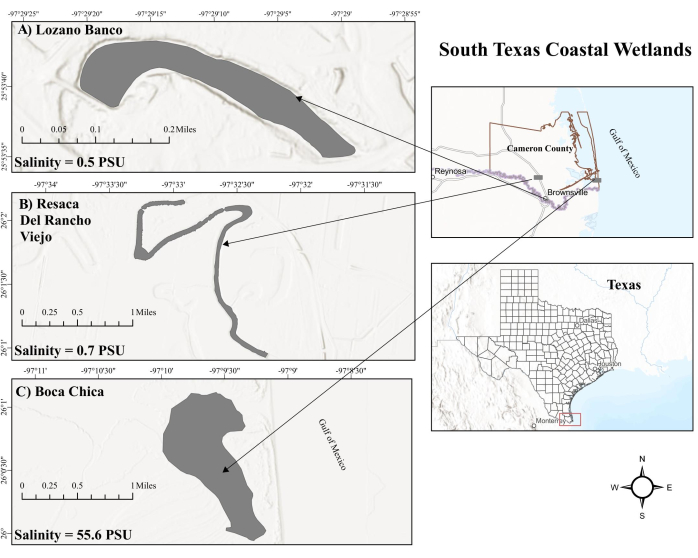
Figura 1: Mappa delle zone umide costiere del Texas meridionale. (A) Lozano Banco. LB è una zona umida costiera d'acqua dolce. (b) Resaca del Rancho Viejo. RV è anche una zona umida costiera d'acqua dolce. (C) Zona umida di Boca Chica. BC è una zona umida costiera ipersalina di acqua salata influenzata dalle maree. Abbreviazioni: LB = Lozano Banco; RV = Resaca Del Rancho Viejo; a.C. = Boca Chica; PSU = unità pratiche di salinità. Clicca qui per visualizzare una versione più grande di questa figura.
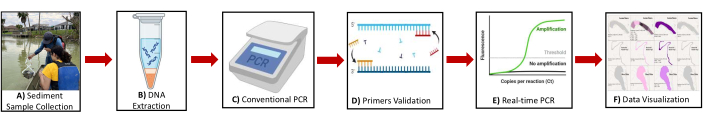
Figura 2: Diagramma schematico che mostra la panoramica del protocollo. L'illustrazione delle fasi chiave include (A) Raccolta di campioni di sedimenti (B) Estrazione del DNA (C) PCR convenzionale (D) Convalida del primer (E) PCR in tempo reale (F) Visualizzazione dei dati per l'identificazione dei geni dei biomarcatori del metanogeno e dei metanotrofi. Clicca qui per visualizzare una versione più grande di questa figura.
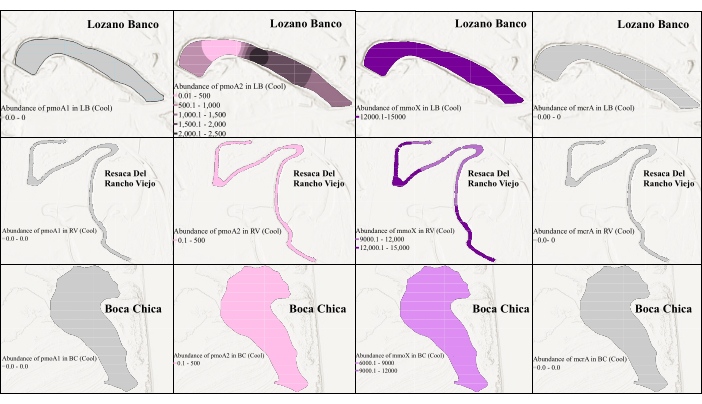
Figura 3: Distribuzione spaziale dei geni dei biomarcatori del metanogeno e dei metanotrofi nella stagione fredda. La figura mostra l'abbondanza di mmoX durante la stagione fredda. Qui, il colore grigio indica che la qPCR non è stata in grado di rilevare il gene a causa della bassa abbondanza. Clicca qui per visualizzare una versione più grande di questa figura.

Figura 4: Distribuzione spaziale dei geni dei biomarcatori del metanogeno e dei metanotrofi nella stagione calda. Qui, il colore grigio rappresenta che la qPCR non è stata in grado di rilevare il gene a causa della sua bassa abbondanza. La figura mostra l'abbondanza di pmoA1 durante la stagione calda a Lozano Banco e Resaca Del Rancho Viejo, mentre non è stata rilevata alcuna abbondanza a Boca Chica. Clicca qui per visualizzare una versione più grande di questa figura.
| Nome del primer | Gene | Temp. di ricottura (°C) in questo studio | Lunghezza del prodotto (bp) | Sequenza (5'-3') | Riferimento | |
| 1055F | 16S rRNA | 55 | 337 | ATGGCTGTCGTCAGCT | (Harms et al., 2003) | |
| 1392R | ACGGGCGGTGTGTAC | |||||
| A189F | pmoA1 | 55 | 525 | GGNGACTGGGACTTCTGG | (Holmes et al., 1995) | |
| A682R | GAASGCNGAGAAGAASGC | |||||
| A189F | 56 | 470 | GGNGACTGGGACTTCTGG | (Costello & Lidstrom, 1999) | ||
| mb661R | CCG GMG CAA CGT CYT TACC | |||||
| 206 e segg | pmoA2 | 60 | 438 | GGNGACTGGGACTTCTG GATCGACTTCAAGGATCG | (Tchawa Yimga et al., 2003) | |
| 703b | GAASGCNGAGAAGAASGC GGCGACCGGAACGACGT | |||||
| 536F | mmoX | 55 | 362 | CGCTGTGGAAGGGCATGAAGCG | (Fuse et al., 1998) | |
| 898R | GCTCGACCTTGAACTTGGAGCC | |||||
| MCRf | mcrA | 46 | ~490 | TAYGAYCARATHTGGYT | (Springer et al., 1995) | |
| MCRr | ACRTTCATNGCRTARTT | |||||
| ME1F | 50 | 763 | GCMATGCARATHGGWATGTC | (Hales et al., 1996) | ||
| ME2R | TCATKGCRTAGTTDGGRTAGT | |||||
| MLF | 55 | ~470 | GGTGGTGTMGGATTCA CACARTAYGCWACAGC | (Luton et al., 2002) | ||
| MLR | TTCATTGCRTAGTTWGGRTAGTT | |||||
| McrA 159F | 62 | 186 | AAAGTGCGGAGCAGCAATCACC | (Vaksmaa et al., 2017) | ||
| McrA 345R | TCGTCCCATTCCTGCTGCATTGC | |||||
Tabella 1: Set di primer utilizzati in questo studio per rilevare geni funzionali correlati al ciclo del metano. Tutte queste coppie di primer sono state utilizzate per la PCR convenzionale e, infine, quelle audaci sono state messe a punto per rilevare i geni correlati al metanogeno e ai metanotrofi nelle zone umide costiere del Texas meridionale.
| Gene | Campioni | ||||||||
| Lozano Banco | Resaca Del Rancho Viejo | Boca Chica | |||||||
| LB-1 | RV-1 | RV-2 | M1 | M2 | B1 | B2 | MG1 | MG2 | |
| pmoA1 | - | + | + | - | - | - | - | - | - |
| pmoA2 | + | + | + | - | + | - | - | + | - |
| mmoX | + | - | + | - | - | - | - | - | - |
| mcrA | + | + | + | - | - | - | - | - | - |
Tabella 2: Rilevamento di geni in tre diversi tipi di corpi idrici nella stagione fredda utilizzando la PCR convenzionale. '+', presente; '-', assente. Abbreviazioni: LB = Lozano Banco, RV = Resaca del Rancho Viejo, M = Fango, B = Batis, MG = Mangrovie.
File supplementare 1: Immagini di elettroforesi su gel per i geni mcrA, pmoA1, pmoA2, mmoX e 16S rRNA. Clicca qui per scaricare questo file.
Discussione
Le zone umide costiere sono riconosciute come fattori significativi per il metano atmosferico, un importante gas serra40. Sebbene ci siano stati studi sul flusso di metano e sui metanogeni nelle zone umide 41,42,43, poco si sa su come i metanotrofi operino in diversi ambienti o in varie pratiche di gestione, specialmente nelle zone umide con livelli d'acqua fluttuanti44. Inoltre, la presenza diffusa di inibitori come gli acidi umici nell'eDNA complica i processi di estrazione e amplificazione45, portando potenzialmente a risultati falsi negativi46. Questa sfida è ulteriormente complicata dall'efficienza variabile dei primer che lavorano sull'eDNA, suggerendo la necessità di una convalida sistematica in tali studi47. Pertanto, questa ricerca stabilisce una metodologia fondamentale per comprendere il ciclo del metano nelle zone umide costiere del Texas meridionale, poco studiate. Concentrandosi sullo sviluppo e la convalida delle tecniche di PCR, questo studio fa progredire l'individuazione dei geni chiave del ciclo del metano in questi ecosistemi dinamici.
Uno degli aspetti critici di questa ricerca è la visualizzazione dei geni microbici correlati al ciclo del metano, che svolge un ruolo cruciale nell'osservazione dei modelli di distribuzione spaziale di questi geni attraverso diverse stagioni e gradienti di salinità. Tale visualizzazione non è solo uno strumento supplementare; È essenziale per identificare le aree di presenza significativa di microbi metanogenici e metanotrofici che indicano che potrebbe esserci un potenziale ciclo del metano, migliorando così la nostra comprensione delle condizioni ambientali che influenzano questi processi.
In questo studio, è stata specificata la metodologia per identificare i geni marcatori del metanogeno e del metanotrofio come pmoA1, pmoA2, mmoX e mcrA utilizzando cPCR e qPCR. Sono state utilizzate quattro coppie di primer che hanno come bersaglio il gene mcrA e due coppie che hanno come bersaglio il gene pmoA1 e solo una coppia di ciascun set ha rilevato efficacemente questi geni sia nella PCR convenzionale che in quella in tempo reale. Uno studio precedente ha utilizzato tre coppie di primer per rilevare pmoA1; tra questi, i primer A189-A682 non sono stati in grado di rilevare la diversità metanotrofica. Invece, hanno amplificato principalmente il gene amoA e hanno prodotto prodotti PCR aspecifici48. Lo studio ha scoperto che la coppia di primer utilizzata era efficace per rilevare il gene pmoA1 solo in ambienti con un'elevata popolazione di metanotrofi. Inoltre, la più alta abbondanza di metanotrofi è stata ottenuta utilizzando le coppie di primer A189-mb661 in quello studio, indicando che questi primer sono adatti per gli ecosistemi d'acqua dolce48. Pertanto, queste coppie di primer sono state utilizzate per la qPCR come ulteriore analisi.
Tra le quattro coppie di primer utilizzate per l'mcrA, i primer ML sono stati in grado di amplificare le specie metanogene in tutti gli ambienti49. I primer ME hanno un intervallo di amplificazione ristretto; Non sono in grado di rilevare tutti i tipi di specie metanogene in diversi tipi di ambienti naturali. I primer MCR hanno un alto livello di degenerazione, che li rende sensibili alla temperatura di ricottura PCR. Questa degenerazione consente loro di legarsi a un'ampia gamma di sequenze bersaglio, spesso adattandosi a più discrepanze. Tuttavia, ciò indica anche che una piccola variazione della temperatura di ricottura può influenzare significativamente la loro efficienza di legame e specificità, portando a un'amplificazione insufficiente e, di conseguenza, a risultati falsi negativi per la specie metanogena49,50.
D'altra parte, nella comunità dei metanogeni a bassa diversità, durante le ultime fasi della PCR, un elevato accumulo di prodotti PCR porta alla ricottura del modello e impedisce ai primer di legarsi, che non riesce nemmeno a rilevare le bande desiderate49. È interessante notare che le coppie di primer McrA 159F-McrA 345R sono state specificamente progettate per identificare il Candidatus metanotrofico anaerobico nitrato-dipendente M. nitroreducens archaea51, e questo studio non è stato in grado di rilevare metanotrofi anaerobici utilizzando queste coppie di primer. Tuttavia, l'ossidazione anaerobica del metano (AOM) può essere accoppiata alla riduzione di accettori di elettroni alternativi come il ferro (Fe), il manganese (Mn) e il solfato (SO42-)52, il che potrebbe spiegare l'incapacità di rilevare l'ANME utilizzando questo metodo. Inoltre, potrebbe essere dovuto alle condizioni ambientali specifiche dell'area di studio. Pertanto, per la qPCR, i primer ML sono stati utilizzati per rilevare l'abbondanza di metanogeno, in quanto forniva un'amplificazione affidabile anche in condizioni di bassa diversità della comunità49.
Inoltre, gli inibitori della PCR come gli acidi umici o il DNA di specie non bersaglio sono ampiamente presenti nei campioni ambientali, il che può ridurre l'efficienza della PCR53. Poiché l'alta concentrazione di DNA non è stata in grado di rilevare alcun gene mediante cPCR, tutti i campioni di DNA sono stati diluiti prima di eseguire la cPCR per ridurre l'effetto degli inibitori della PCR54. Il gene 16S rRNA, come marcatore universale per batteri e archei, ha fornito una solida base per convalidare le nostre condizioni di PCR e confermare l'integrità del DNA in tutti i campioni55,56. Tuttavia, le aree di acqua salata sono note per avere concentrazioni più elevate di inibitori della PCR46. Inoltre, la salinità influenza indirettamente le emissioni di CH4 nelle zone umide costiere, in quanto è associata alle concentrazioni di solfati. Una maggiore salinità nelle zone umide di acqua salata è correlata a livelli elevati di solfato che sopprimono la produzione di metano a causa della competizione tra batteri solfato-riduttori e metanogeni, mentre concentrazioni più basse di solfato nelle zone umide d'acqua dolce provocano maggiori emissioni di metano11,41. Inoltre, le differenze di emissione di CH4 tra le zone umide di acqua salata e quelle d'acqua dolce sono influenzate anche dalle loro connessioni di marea con l'oceano57, il che potrebbe anche spiegare il mancato rilevamento di geni correlati al ciclo del metano mediante cPCR in BC. In questo studio, la cPCR ha rilevato con successo tutti i geni bersaglio nei campioni di LB e RV, ma non è riuscita a rilevare la presenza di questi geni nei campioni di BC, con il gene pmoA2 che è l'unica eccezione. Inoltre, la qPCR è stata in grado di rilevare l'mmoX in tutti i campioni, mentre la cPCR non è stata in grado di rilevarlo nel BC. Ciò indica che la qPCR mostra una maggiore resilienza agli inibitori della PCR rispetto alla cPCR, attribuibile alla sua maggiore sensibilità nei meccanismi di rilevamento58. Un altro motivo è che la qPCR identifica i prodotti PCR durante la fase esponenziale del ciclo PCR, mentre la cPCR li rileva nella fase di plateau. Questa distinzione consente alla qPCR di mostrare una ridotta suscettibilità alla degradazione del prodotto durante i cicli di reazione avanzati, in cui i reagenti sono esauriti47.
Questo studio ha stabilito una tecnica PCR per identificare i geni correlati al ciclo del metano nelle zone umide costiere del Texas meridionale per superare le sfide associate agli inibitori della PCR e alle efficienze variabili dei primer. Sono state utilizzate quattro coppie di primer che hanno come bersaglio il gene mcrA e due coppie che hanno come bersaglio il gene pmoA1 e solo una coppia di ciascun set ha rilevato efficacemente questi geni sia nella PCR convenzionale che in quella in tempo reale. Questo studio conclude l'insieme di coppie di primer per i geni correlati al ciclo del metano, tra cui a189-mb6661 per pmoA1, 206f-703b per pmoA2, 536F-898R per mmoX e ML per mcrA. Ciò suggerisce l'importanza di un'appropriata selezione di primer per rilevare i geni correlati al ciclo del metano nelle zone umide costiere, con quelli nel sud del Texas come esempi. Inoltre, lo studio ha dimostrato che mmoX era più abbondante durante la stagione fredda, mentre pmoA1 era abbondante durante la stagione calda.
Pertanto, i risultati di questo studio forniscono le basi per la comprensione scientifica dei processi microbici coinvolti nel ciclo del metano, evidenziando l'influenza dei fattori ambientali sulle variazioni stagionali e spaziali nelle zone umide costiere del Texas meridionale. Sulla base di questi risultati, gli studi futuri mirano a convalidare ulteriormente questo approccio in altri ecosistemi, come le risaie e le zone umide d'acqua dolce, per aumentare la robustezza e la generalizzabilità. Inoltre, questi studi includeranno anche analisi geochimiche come le concentrazioni di metano nei sedimenti e il solfuro nelle acque interstiziali, e tecniche molecolari come la metagenomica e la trascrittomica, che si baseranno su questo lavoro di base per fornire una comprensione più completa delle dinamiche del ciclo del metano.
Divulgazioni
Gli autori non hanno conflitti di interesse da dichiarare.
Riconoscimenti
Siamo grati ai membri di C-REAL per la loro assistenza nell'osservazione sul campo e nelle analisi di laboratorio.
Materiali
| Name | Company | Catalog Number | Comments |
| 0.2 mL PCR tubes | ThermoFisher Scientific | AB0620 | https://www.thermofisher.com/order/catalog/product/AB0620?SID=srch-srp-AB0620 |
| 0.5 mL PCR Tubes | Promega | E4941 | https://www.promega.com/products/biochemicals-and-labware/tips-and-accessories/0_5ml-pcr-tubes/?catNum=E4941 |
| 10 μL tips | ThermoFisher Scientific | 05-408-187 | Fisherbrand SureGrip Pipet Tip Racked or Reload System Tips Natural; 10μL; | Fisher Scientific |
| 15 mL centrifuge tube | ThermoFisher Scientific | 14-959-53A | https://www.fishersci.com/shop/products/falcon-15ml-conical-centrifuge-tubes-5/p-193301 |
| 200 μL tips | ThermoFisher Scientific | 05-408-190 | Fisherbrand SureGrip Pipet Tip Racked or Reload System Tips Natural; 200μL; | Fisher Scientific |
| 1000 μL tips | ThermoFisher Scientific | 02-707-402 | https://www.fishersci.com/shop/products/sureone-micropoint-pipette-tips-specific-standard-fit/02707402?gclid=Cj0KCQiAp NW6BhD5ARIsACmEb kUsQ9Lu0YIq5i4vWege 17qPdtxIYZyvmJH1cDo ARuwereO1V4GLz9UaA lDREALw_wcB&ef_id=C j0KCQiApNW6BhD5ARI sACmEbkUsQ9Lu0YIq5i 4vWege17qPdtxIYZyvmJ H1cDoARuwereO1V4GLz 9UaAlDREALw_wcB:G:s &ppc_id=PLA_goog_2175 7693617_171052169911_02 707402__715434303113_1555 377385658230343&ev_chn=sh op&s_kwcid=AL!4428!3!71543430 3113!!!g!2366517300713!&gad_source=1 |
| Applied Biosystem Power SYBR Green Master Mix | ThermoFisher Scientific | 4368577 | https://www.thermofisher.com/order/catalog/product/4368577 |
| ArcGIS Pro | esri | https://www.esri.com/en-us/arcgis/products/arcgis-pro/overview?srsltid=AfmBOopatJ4 JvHJfscHRcAaDx0Jz5_Jrl8l5 vYkkBvfOqE-uNSsMghN1 | |
| CFX Duet Real-Time PCR system | Bio-Rad | 12016265 | https://www.bio-rad.com/en-us/product/cfx-duet-real-time-pcr-system?ID=97722926-9ed9-16a4-1d83-c92f587e427a |
| Corning Lambda plus single channel pipettor volume 0.5-10 μL | Sigma-Aldrich | CLS4071-1EA | https://www.sigmaaldrich.com/US/en/product/sigma/cls4071 |
| Corning Lambda plus single channel pipettor volume 100-1000 μL | Sigma-Aldrich | CLS4075-1EA | https://www.sigmaaldrich.com/US/en/product/sigma/cls4075 |
| Corning Lambda plus single channel pipettor volume 20-200 μL | Sigma-Aldrich | CLS4074-1EA | https://www.sigmaaldrich.com/US/en/product/sigma/cls4074 |
| FastDNA spin kit for soil | MP Biomedical | 116560200-CF | https://www.mpbio.com/us/116560000-fastdna-spin-kit-for-soil-samp-cf?srsltid=AfmBOoqOxxGilzY3IHNIZR ajegGTr9MoX1oMZUh 3dcbJqe0UvvukY128 |
| Gene copy calculator | Science Primer | https://scienceprimer.com/copy-number-calculator-for-realtime-pcr . | |
| High speed benchtop centrifuge | ThermoFisher Scientific | 75004241 | https://newlifescientific.com/products/thermo-scientific-sorvall-st16-high-speed-benchtop-centrifuge-75004241?gad_source=1&gclid=Cj0KCQiApN W6BhD5ARIsACmEbkVC_-cCIN9j 20TvYq8iDsBlUR5cPK_1_wN OBEcjMdv-CYVoGCfeOLYaAv enEALw_wcB |
| High speed microcentrifuge | VWR | 75838-336 | https://us.vwr.com/store/product/20546590/null |
| Lysing Matrix E tube | glass bead/ceramic sphere-containing tube | ||
| Microcentrifuge tube | ThermoFisher Scientific | 02-681-320 | https://www.fishersci.com/shop/products/fisherbrand-low-retention-microcentrifuge-tubes-8/02681320?gclid=Cj0KCQiAp NW6BhD5ARIsACm EbkWbG4_o3oUiGk HJPU-_31-CuexDwQ fmWPnfyhBOf2BHXsy K3fFW1toaAgJbEALw_ wcB&ef_id=Cj0KCQiAp NW6BhD5ARIsACmEb kWbG4_o3oUiGkHJPU- _31-CuexDwQfmWPnfy hBOf2BHXsyK3fFW1toa AgJbEALw_wcB:G:s&ppc _id=PLA_goog_21757693 617_171052169911_0268 1320__715434303113_10 349826094968484711&ev _chn=shop&s_kwcid=AL!4 428!3!715434303113!!!g!23 66517300713!&gad_source=1 |
| PCR Master mix | Promega | M7502 | https://www.promega.com/products/pcr/taq-polymerase/master-mix-pcr/?catNum=M7502 |
| Quantiflour ONE dsDNA system | Promega | E4871 | https://www.promega.com/products/rna-analysis/dna-and-rna-quantitation/quantifluor-one-dsdna-system/?gad_source=1&gbraid=0AAAAAD _rg189yJTY3cxeVqMdu8RPx10 Ma&gclid=CjwKCAjwxNW2BhAk EiwA24Cm9FUgViPNyWq7UfZL VeeoroLAZ5JIP6w07RGK_4D0w oZgAqf-G1XTmxoCxm8QAvD_B wE&catNum=E4871 |
| Quantus Fluorometer | Promega | E6150 | https://www.promega.com/products/microplate-readers-fluorometers-luminometers/fluorometers/quantus-fluorometer/?catNum=E6150 |
| YSI Pro 2030 | YSI a xylem brand | 603174 | https://www.ysi.com/product/id-p2030/pro2030-kits |
Riferimenti
- Xu, T., et al. Wetlands of international importance: Status, threats, and future protection. Int J Environ Res Public Health. 16 (10), 1818 (2019).
- Corn, M. L. . Deepwater Horizon oil spill: coastal wetland and wildlife impacts and response. , (2010).
- Hendriks, I. E., Sintes, T., Bouma, T. J., Duarte, C. M. Experimental assessment and modeling evaluation of the effects of the seagrass Posidonia oceanica on flow and particle trapping. Marine Ecology Progress Series. 356, 163-173 (2008).
- Krause, S. J. E., Treude, T. Deciphering cryptic methane cycling: Coupling of methylotrophic methanogenesis and anaerobic oxidation of methane in hypersaline coastal wetland sediment. Geochimica et Cosmochimica Acta. 302, 160-174 (2021).
- Reddy, K. R., DeLaune, R. D., Inglett, P. W. . Biogeochemistry of Wetlands: Science and Applications. , (2022).
- La, W., et al. Sulfate concentrations affect sulfate reduction pathways and methane consumption in coastal wetlands. Water Research. 217, 118441 (2022).
- Derwent, R. G. Global warming potential (GWP) for methane: Monte Carlo analysis of the uncertainties in Global Tropospheric Model predictions. Atmosphere. 11 (5), 486 (2020).
- Potter, C., et al. Methane emissions from natural wetlands in the United States: Satellite-derived estimation based on ecosystem carbon cycling. Earth Interactions. 10 (22), 1-12 (2006).
- . Understanding global warming potentials Available from: https://www.epa.gov/ghgemissions/understanding-global-warming-potentials (2024)
- Wallenius, A. J., Dalcin Martins, P., Slomp, C. P., Jetten, M. S. M. Anthropogenic and environmental constraints on the microbial methane cycle in coastal sediments. Front Microbiol. 12, 631621 (2021).
- Qu, Y., et al. Salinity causes differences in stratigraphic methane sources and sinks. Environmental Science and Ecotechnology. 19, 100334 (2024).
- Vizza, C., West, W. E., Jones, S. E., Hart, J. A., Lamberti, G. A. Regulators of coastal wetland methane production and responses to simulated global change. Biogeosciences. 14 (2), 431-446 (2017).
- van Dijk, G., et al. Salinization lowers nutrient availability in formerly brackish freshwater wetlands; unexpected results from a long-term field experiment. Biogeochemistry. 143 (1), 67-83 (2019).
- Aronson, E., Allison, S., Helliker, B. R. Environmental impacts on the diversity of methane-cycling microbes and their resultant function. Front Microbiol. 4, 225 (2013).
- Reeburgh, W. S. Oceanic methane biogeochemistry. Chem Rev. 107 (2), 486-513 (2007).
- Thauer, R. K. Biochemistry of methanogenesis: a tribute to Marjory Stephenson. Marjory Stephenson Prize Lecture. Microbiology (Reading). 144 (Pt 9), 2377-2406 (1998).
- Thauer, R. K. Anaerobic oxidation of methane with sulfate: on the reversibility of the reactions that are catalyzed by enzymes also involved in methanogenesis from CO2. Curr Opin Microbiol. 14 (3), 292-299 (2011).
- Friedrich, M. W. Methyl-coenzyme M reductase genes: Unique functional markers for methanogenic and anaerobic methane-oxidizing Archaea. Methods in Enzymology. 397, 428-442 (2005).
- Reeburgh, W. S. Oceanic methane biogeochemistry. Chem Rev. 107 (2), 486-513 (2007).
- Rasigraf, O., Schmitt, J., Jetten, M. S. M., Lüke, C. Metagenomic potential for and diversity of N-cycle driving microorganisms in the Bothnian Sea sediment. Microbiologyopen. 6 (4), e00475 (2017).
- McDonald, I. R., Bodrossy, L., Chen, Y., Murrell, J. C. Molecular ecology techniques for the study of aerobic methanotrophs. Appl Environ Microbiol. 74 (5), 1305-1315 (2008).
- Tchawa Yimga, M., Dunfield, P. F., Ricke, P., Heyer, J., Liesack, W. Wide distribution of a novel pmoA-like gene copy among type II methanotrophs, and its expression in Methylocystis strain SC2. Appl Environ Microbiol. 69 (9), 5593-5602 (2003).
- Knief, C. Diversity and habitat preferences of cultivated and uncultivated aerobic methanotrophic bacteria evaluated based on pmoA as molecular marker. Front Microbiol. 6, 1346 (2015).
- Huang, I. -. S., et al. Preliminary assessment of microbial community structure of Wind-Tidal Flats in the Laguna Madre, Texas, USA. Biology. 9 (8), 183 (2020).
- Wilding, T. K., Brown, E., Collier, K. J. Identifying dissolved oxygen variability and stress in tidal freshwater streams of northern New Zealand. Environ Monit Assess. 184 (10), 6045-6060 (2012).
- Roy Chowdhury, T., Mitsch, W. J., Dick, R. P. Seasonal methanotrophy across a hydrological gradient in a freshwater wetland. Ecological Engineering. 72, 116-124 (2014).
- Sun, Q. -. Q., et al. Carbon dioxide and methane fluxes: Seasonal dynamics from inland riparian ecosystems, northeast China. Sci Total Environ. 465, 48-55 (2013).
- Lee, S., et al. Comparison and selection of conventional PCR primer sets for studies associated with nitrogen cycle microorganisms in surface soil. Appl Sci. 12 (20), 10314 (2022).
- Bae, K. -. S., et al. Development of diagnostic systems for wide range and highly sensitive detection of two waterborne hepatitis viruses from groundwater using the conventional reverse transcription nested PCR assay. J Virol Methods. 299, 114344 (2022).
- Lecusay, D. . Assessment and Monitoring of Deltaic Wetlands and Fluvial Systems: Refining and Validating a Multimetric Index of Resaca Ecosystem Health. , (2021).
- . FastDNA SPIN Kit for Soil (Cat No. 116560200) Available from: https://www.mpbio.com/media/productattachment/LS082019-EN-FastDNA-SPIN-Kit-for-Soil-116560200-Manual.pdf (2025)
- Luton, P. E., Wayne, J. M., Sharp, R. J., Riley, P. W. The mcrA gene as an alternative to 16S rRNA in the phylogenetic analysis of methanogen populations in landfill. Microbiology (Reading, England). 148 (Pt 11), 3521-3530 (2002).
- Changsoo, L., Jaai, K., Seung Gu, S., Seokhwan, H. Absolute and relative QPCR quantification of plasmid copy number in Escherichia coli. J Biotechnol. 123 (3), 273-280 (2006).
- Harms, G., et al. Real-time PCR quantification of nitrifying bacteria in a municipal wastewater treatment plant. Environ Sci Technol. 37 (2), 343-351 (2003).
- Holmes, A. J., Costello, A., Lidstrom, M. E., Murrell, J. C. Evidence that particulate methane monooxygenase and ammonia monooxygenase may be evolutionarily related. FEMS Microbiol Lett. 132 (3), 203-208 (1995).
- Fuse, H., et al. Oxidation of trichloroethylene and dimethyl sulfide by a marine Methylomicrobium strain containing soluble methane monooxygenase. Biosci Biotechnol Biochem. 62 (10), 1925-1931 (1998).
- Springer, E., Sachs, M. S., Woese, C. R., Boone, D. R. Partial gene sequences for the A subunit of methyl-coenzyme M reductase (mcrI) as a phylogenetic tool for the family Methanosarcinaceae. Int J Syst Bacteriol. 45 (3), 554-559 (1995).
- Costello, A. M., Lidstrom, M. E. Molecular characterization of functional and phylogenetic genes from natural populations of methanotrophs in lake sediments. Appl Environ Microbiol. 65 (11), 5066-5074 (1999).
- Flores, E. A. . Effects of Nutrient Enrichment on Mangrove and Saltmarsh Habitats. , (2022).
- Minjie, H., Jordi, S., Xianyu, Y., Josep, P., Chuan, T. Patterns and environmental drivers of greenhouse gas fluxes in the coastal wetlands of China: A systematic review and synthesis. Environ Res. 186, 109576 (2020).
- Bridgham, S. D., Cadillo-Quiroz, H., Keller, J. K., Zhuang, Q. Methane emissions from wetlands: biogeochemical, microbial, and modeling perspectives from local to global scales. Glob Chang Biol. 19 (5), 1325-1346 (2013).
- Liu, D. Y., Ding, W. X., Jia, Z. J., Cai, Z. C. Relation between methanogenic archaea and methane production potential in selected natural wetland ecosystems across China. Biogeosciences. 8 (2), 329-338 (2011).
- Ke, Z., et al. Methane emissions and methanogenic community investigation from constructed wetlands in Chengdu City. Urban Climate. 39, 100956 (2021).
- Chowdhury, T. R., Dick, R. P. Ecology of aerobic methanotrophs in controlling methane fluxes from wetlands. Applied Soil Ecology. 65, 8-22 (2013).
- Maja, S., et al. Humic substances cause fluorescence inhibition in real-time polymerase chain reaction. Anal Biochem. 487, 30-37 (2015).
- Sanches, T. M., Schreier, A. D. Optimizing an eDNA protocol for estuarine environments: Balancing sensitivity, cost and time. PLOS ONE. 15 (5), e0233522 (2020).
- Xia, Z., et al. Conventional versus real-time quantitative PCR for rare species detection. Ecol Evol. 8 (23), 11799-11807 (2018).
- Bourne, D. G., McDonald, I. R., Murrell, J. C. Comparison of pmoA PCR primer sets as tools for investigating methanotroph diversity in three Danish soils. Appl Environ Microbiol. 67 (9), 3802-3809 (2001).
- Juottonen, H., Galand, P. E., Yrjala, K. Detection of methanogenic Archaea in peat: comparison of PCR primers targeting the mcrA gene. Res Microbiol. 157 (10), 914-921 (2006).
- Lueders, T., Friedrich, M. W. Evaluation of PCR amplification bias by terminal restriction fragment length polymorphism analysis of small-subunit rRNA and mcrA genes by using defined template mixtures of methanogenic pure cultures and soil DNA extracts. Appl Environ Microbiol. 69 (1), 320-326 (2003).
- Vaksmaa, A., Jetten, M. S., Ettwig, K. F., Luke, C. McrA primers for the detection and quantification of the anaerobic archaeal methanotroph 'Candidatus Methanoperedens nitroreducens'. Appl Microbiol Biotechnol. 101 (4), 1631-1641 (2017).
- Ren, G., et al. Electron acceptors for anaerobic oxidation of methane drive microbial community structure and diversity in mud volcanoes. Environ Microbiol. 20 (7), 2370-2385 (2018).
- Goldberg, C. S., et al. Critical considerations for the application of environmental DNA methods to detect aquatic species. Methods in Ecology and Evolution. 7 (11), 1299-1307 (2016).
- McKee, A. M., Spear, S. F., Pierson, T. W. The effect of dilution and the use of a post-extraction nucleic acid purification column on the accuracy, precision, and inhibition of environmental DNA samples. Biological Conservation. 183, 70-76 (2015).
- Gohl, D. M., et al. Systematic improvement of amplicon marker gene methods for increased accuracy in microbiome studies. Nat Biotechnol. 34 (9), 942-949 (2016).
- Ballarini, A., Segata, N., Huttenhower, C., Jousson, O. Simultaneous quantification of multiple bacteria by the BactoChip microarray designed to target species-specific marker genes. PLOS ONE. 8 (2), e55764 (2013).
- Le Mer, J., Roger, P. Production, oxidation, emission and consumption of methane by soils: A review. Eur J Soil Biol. 37 (1), 25-50 (2001).
- Smith, C. J., Osborn, A. M. Advantages and limitations of quantitative PCR (Q-PCR)-based approaches in microbial ecology. FEMS Microbiol Ecol. 67 (1), 6-20 (2009).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon