Method Article
Wir danken den C-REAL-Mitgliedern für ihre Unterstützung bei der Feldbeobachtung und bei Laboranalysen.Visualisierung der mikrobiellen Dynamik des Methankreislaufs in Küstenfeuchtgebieten
In diesem Artikel
Zusammenfassung
Das Protokoll erkennt wichtige Gene für den Methankreislauf in den Feuchtgebieten der südtexanischen Küste und visualisiert ihre räumliche Verteilung, um das Verständnis der Methanregulierung und ihrer Umweltauswirkungen in diesen dynamischen Ökosystemen zu verbessern.
Zusammenfassung
Küstenfeuchtgebiete sind die größte biotische Quelle für Methan, wo Methanogene organisches Material in Methan umwandeln und Methanotrophe Methan oxidieren und somit eine entscheidende Rolle bei der Regulierung des Methankreislaufs spielen. Die Feuchtgebiete in Südtexas, die aufgrund des Klimawandels häufigen Wetterereignissen, schwankenden Salzgehalten und anthropogenen Aktivitäten ausgesetzt sind, beeinflussen den Methankreislauf. Trotz der ökologischen Bedeutung dieser Prozesse ist der Methankreislauf in den Feuchtgebieten der südtexanischen Küste nach wie vor unzureichend erforscht. Um diese Lücke zu schließen, haben wir eine Methode zum Nachweis von Genen entwickelt und optimiert, die mit Methanogenen und Methanotrophen in Verbindung stehen, einschließlich mcrA als Biomarker für Methanogene und pmoA1, pmoA2 und mmoX als Biomarker für Methanotrophe. Darüber hinaus zielte diese Studie darauf ab, die räumlichen und zeitlichen Verteilungsmuster der Methanogen- und Methanotrophenhäufigkeit mit Hilfe der GIS-Software (Geographic Information System) ArcGIS Pro zu visualisieren. Die Integration dieser molekularen Techniken mit fortschrittlicher räumlicher Visualisierung lieferte wichtige Einblicke in die räumliche und zeitliche Verteilung von Methanogen- und Methanotrophengemeinschaften in den Feuchtgebieten von Südtexas. Daher bietet die in dieser Studie etablierte Methodik einen robusten Rahmen für die Kartierung der mikrobiellen Dynamik in Feuchtgebieten, verbessert unser Verständnis des Methankreislaufs unter unterschiedlichen Umweltbedingungen und unterstützt breitere Studien zu ökologischen und ökologischen Veränderungen.
Einleitung
Küstenfeuchtgebiete sind lebenswichtige Ökosysteme, die durch Prozesse wie Kohlenstoffbindung, Evapotranspiration und Methanemissionen (CH4) zur Klimaregulierung, zum Erhalt der biologischen Vielfalt und zum Wassermanagement beitragen1. Diese Ökosysteme, zu denen sowohl Süß- als auch Salzwasserfeuchtgebiete gehören2, sind hochproduktiv und fungieren als kritische Zonen für die Aufnahme von Kohlendioxid (CO2) und binden organisches Material aus der Land- und Meeresumwelt 3,4. Die dynamischen Wechselwirkungen innerhalb dieser Feuchtgebiete stimulieren die Produktion und den Verbrauch von mikrobiellem CH4 5 und positionieren sie als eine der größten natürlichen Quellen für CH46. Als zweitwichtigstes Treibhausgas hat CH4 ein Treibhauspotenzial, das etwa 27-30x höher ist als das von CO2 4,7,8,9, was die Untersuchung der CH4-Emissionen aus Küstenfeuchtgebieten im Zeitalter des Klimawandels unerlässlich macht. Die Emission von CH4 wird von verschiedenen Umweltfaktoren beeinflusst, insbesondere vom Salzgehalt, der eine entscheidende Rolle bei mikrobiellen Prozessen spielt10. Süßwasserfeuchtgebiete tragen aufgrund ihres niedrigeren Sulfatgehalts erheblich zum atmosphärischen Methan bei, was eine höhere mikrobielle CH4-Produktion ermöglicht, während Salzwasserfeuchtgebiete aufgrund höherer Sulfatkonzentrationen im Allgemeinen weniger CH4 ausstoßen 11,12,13.
CH4-Emissionen aus Küstenfeuchtgebieten werden im Allgemeinen durch zwei Gruppen von Mikroorganismen kontrolliert, die als Methanogene und Methanotrophe bekannt sind14. Methanogene produzieren CH4 in anoxischen Sedimenten, indem sie Substrate wie Formiat, Acetat, Wasserstoff oder methylierte Verbindungen durch einen Prozess abbauen, der als Methanogenesebekannt ist 15. Das wichtige Enzym in diesem Signalweg ist die Methyl-Coenzym-M-Reduktase (MCR), da es den letzten und geschwindigkeitsbestimmenden Schritt der Methanogenese katalysiert 15,16,17. Das mcrA-Gen, das für die Alpha-Untereinheit von MCR kodiert, ist ein funktioneller Marker, der in allen methanogenen Archaeen zu finden ist18. Darüber hinaus bildet sich in küstennahen Feuchtgebieten die Sulfat-Methan-Übergangszone (SMTZ) über der methanogenen Zone, in der das nach oben diffundierende Methan und das sich nach unten bewegende Sulfat zusammenlaufen und abgebaut werden19. Innerhalb dieser Zone oxidieren anaerobe methanotrophe Archaeen (ANME) Methan mit Hilfe des MCR-Enzyms zu Kohlendioxid, während sulfatreduzierende Bakterien (SRB) Sulfat zu Sulfid reduzieren. SRB verdrängt Methanogene um Wasserstoff und Acetat und begrenzt die Methanproduktion, bis Sulfat erschöpft ist16,17.
Im Gegensatz dazu oxidieren aerobe methanotrophe Bakterien CH4 in aeroben Umgebungen20 unter Verwendung verschiedener Formen der Methanmonooxygenase (MMO). Dazu gehören die partikuläre Methanmonooxygenase (pMMO), ein kupferhaltiges Enzym, das in die intrazytoplasmatische Membran eingebettet ist, und die lösliche Methanmonooxygenase (sMMO), ein eisenhaltiges Enzym, das im Zytoplasma vorkommt. Für pMMO gibt es jedoch drei Genoperone: pmoCAB21; Unter ihnen ist das pmoA-Gen das konservativste für alle Methanotrophen. Es gibt zwei verschiedene Biomarker-Gene für pmoA: pmoA1 und pmoA222. Darüber hinaus wird das mmoX-Gen für ein umfassendes Verständnis von Methanotrophen in der Molekularbiologie als Werkzeug zur Identifizierung von sMMO-haltigen Methanotrophen verwendet23. Diese Unterscheidung der Stoffwechselwege und Umweltanforderungen von Methanogenen und aeroben Methanotrophen unterstreicht die komplexen mikrobiellen Wechselwirkungen, die den Methankreislauf in Küstenfeuchtgebietsökosystemen regulieren.
Das Feuchtgebiet Boca Chica (BC), eine produktive Salzwasserumgebung in Südtexas, ist Gezeiteneinflüssen aus dem Golf von Mexiko (GOM) ausgesetzt, was zu unterschiedlichen Salzgehalten an der Oberfläche führt, insbesondere aufgrund seiner Nähe zur hypersalinen Laguna Madre24. Diese Gezeitenwirkung, die zwischen Ebbe und Flut wechselt, führt zu Schwankungen des Sauerstoffgehalts25 , was die Methanogen- und Methanotrophaktivität in den Sedimenten verändern könnte26. Im Gegensatz dazu gelten küstennahe Süßwasserfeuchtgebiete als signifikanter Hotspot für CH4-Flüsse 27. Die küstennahen Süßwasserfeuchtgebiete in Südtexas, einschließlich Resaca Del Rancho Viejo (RV) und Lozano Banco (LB), die weit von den Gezeiteneffekten der GOM entfernt sind, weisen ein ausgeprägtes hydrologisches Management auf. RV erlebt bei niedrigem Wasserstand Impulsströmungen, die durch Flusswasser ergänzt werden, während LB als Offline-Strömungssystem ohne eine solche Ergänzung arbeitet. Darüber hinaus weisen RV und LB einen niedrigeren Salzgehalt bei, da sie einen hohen Abfluss von künstlich gepumptem Süßwasser aufweisen und es sich um einen Altarm handelt. Die verschiedenen Umweltfaktoren können den Methankreislauf in den Küstenfeuchtgebieten von Südtexas erheblich beeinflussen. Der Methankreislauf in den Feuchtgebieten der südtexanischen Küste ist jedoch nach wie vor ein Bereich, der noch nicht gründlich untersucht wurde.
Die Polymerase-Kettenreaktion (PCR) und die real-time PCR (auch quantitative PCR [qPCR] genannt) stellen grundlegende und weit verbreitete Techniken zum Nachweis und zur Quantifizierung der relativen Häufigkeit spezifischer Gene in Umweltproben dar. Diese Techniken amplifizieren gezielt Zielregionen der DNA, um das Vorhandensein und die relative Menge von Genen, diemit dem CH-4-Kreislauf in Verbindung stehen, anzuzeigen und Indikatoren für einen potenziellen Methankreislauf zu liefern. Nichtsdestotrotz kann die Verfügbarkeit und Wirksamkeit von PCR-Primer-Sets durch verschiedene inhibitorische Faktoren in der extrahierten Umwelt-DNA eingeschränkt werden, die von der Art der Umgebungen beeinflusst werden28,29. Daher etablierte diese Studie hauptsächlich eine optimale PCR-Methode zum Nachweis des Vorhandenseins von CH 4-Schaltkreis-bezogenen Genen in südtexanischen Küstenfeuchtgebieten (Abbildung 1) und visualisierte dann deren quantifizierte relative Häufigkeit in diesen Ökosystemen. Die Ergebnisse dieser Studie können auf andere Küstenregionen übertragen werden, um das Verständnis des CH4-Kreislaufs und der mikrobiellen Dynamik in verschiedenen Küstenökosystemen zu verbessern.
Protokoll
1. Probenentnahme
- Sammeln Sie Sedimentproben mit einem Sedimentgreifer oder einer Schaufel.
HINWEIS: Die Proben wurden an zwei Stationen von drei verschiedenen Küstenfeuchtgebieten während der kühlen (Oktober-Februar, Durchschnittstemperatur beträgt 20 °C) und warmen (April-Juni, Durchschnittstemperatur 27 °C) Jahreszeiten 2023 und 2024 entnommen. Bei der Entnahme von Proben aus küstennahen Süßwasserfeuchtgebieten (Abbildung 2) wurde ein Sedimentgreifer verwendet, und für gezeitenbeeinflusste Salzwasserfeuchtgebiete an der Küste wurde eine Schaufel verwendet. - Senken Sie den Probenehmer zuerst in das flache Gewässer ab und lassen Sie ihn unter seinem eigenen Gewicht an die Sedimentoberfläche sinken (innerhalb der oberen 50 cm), um die Störung der Sedimentstruktur zu minimieren, wie in Abbildung 2 gezeigt.
- Ziehen Sie es aus der Wassersäule, wo die Tiefe typischerweise 60 cm bis 215 cm30 betrug, geben Sie die Sedimentproben in Zip-Lock-Beutel und bewahren Sie sie sofort in einer Eisbox auf.
HINWEIS: Reinigen und waschen Sie den Probenehmer mit deionisiertem (DI) Wasser, bevor Sie zu den nächsten Stationen gehen. - Lagern Sie alle Proben sofort bei -20 °C im Labor.
- Messen Sie an jeder Probenahmestelle Parameter der Oberflächenwasserqualität wie Salzgehalt und Temperatur vor Ort mit einem Multiparameter-Wasserqualitätsmessgerät.
HINWEIS: Die Sonden wurden nach dem Gebrauch in jeder Station mit entionisiertem Wasser (DI-Wasser) gespült.
2. Extraktion genomischer DNA
- Tauen Sie die Proben bei Raumtemperatur auf, bevor Sie mit dem Verfahren zur Extraktion genomischer DNA beginnen.
- Übertragen Sie ca. 500 mg Sedimentproben in ein 15-ml-Röhrchen und zentrifugieren Sie sie 3 Minuten lang bei 4.250 × g , um das gesamte Wasser zu entfernen.
- Extrahieren Sie genomische DNA mit einem DNA-Extraktionskit für Böden nach dem Protokoll31 des Herstellers mit einer kleinen Modifikation und lagern Sie es sofort bei -20 °C.
HINWEIS: Die Änderungen wurden vorgenommen, um Redundanz zu reduzieren und einen effizienten Arbeitsablauf zu gewährleisten.- Geben Sie bis zu 500 mg Bodenprobe in ein Röhrchen mit Glasperlen/Keramikkugeln.
- Geben Sie 978 μl Natriumphosphatpuffer in die Probe in das Kügelchen-/Kugel-enthaltende Röhrchen.
- Geben Sie 122 μl Puffer-Lyselösung in die Probe im Bead-/Kugel-haltigen Röhrchen, um externe Verunreinigungen zu lösen.
- Mit einem Kugelmühlen-Homogenisator bei 5-facher Drehzahl für 20 s homogenisieren und 2x wiederholen.
HINWEIS: In dieser Studie wurde ein Kugelmühlen-Homogenisator verwendet, weshalb die Drehzahl angepasst wurde. - Die Mischung 10 min bei 14.000 × g zentrifugieren .
- Übertragen Sie den Überstand in ein sauberes 2,0-ml-Mikrozentrifugenröhrchen.
- Fügen Sie 250 μl Proteinfällungslösung (PPS) hinzu, um die löslichen Nukleinsäuren von den Zelltrümmern und der Lysematrix zu trennen. Mixen, indem man das Röhrchen 10x umdreht.
- Zentrifugieren Sie 5 Minuten lang bei 14.000 × g , um das Pellet auszufällen und die Zelltrümmer und die Lysematrix zu entfernen.
- Übertragen Sie den Überstand in ein sauberes 15-ml-Mikrozentrifugenröhrchen.
- Geben Sie 1,0 mL der Binding Matrix-Suspension in den Überstand im 15-ml-Röhrchen.
HINWEIS: Schütteln Sie die Bindungsmatrix-Suspension, um sie erneut zu suspendieren, bevor Sie sie hinzufügen. - Ermöglichen Sie die Bindung der DNA an die Bindungsmatrix, indem Sie die Röhrchen für 2 Minuten auf einen Rotator legen.
- Legen Sie alle Röhrchen auf ein Gestell und inkubieren Sie sie 3 Minuten lang, damit sich die Bindungsmatrix absetzen kann.
- Nach 3 Minuten werden 750 μl verworfen und der restliche Überstand mit einer Pipette vorsichtig mit dem Pellet vermischt.
- 750 μl der Mischung in einen SPIN-Filter geben und 1 min bei 14.000 × g zentrifugieren. Leeren Sie das Auffangrohr und verwenden Sie es wieder. Wiederholen Sie den Vorgang mit der restlichen Mischung.
- Fügen Sie 500 μl der vorbereiteten Waschlösung (mit der entsprechenden Menge Ethanol) hinzu, um Verunreinigungen weiter aufzulösen. Suspendieren Sie das Pellet vorsichtig mit der Kraft der Flüssigkeit aus der Pipettenspitze.
- Zentrifugieren Sie 1 Minute lang bei 14.000 × g , um Verunreinigungen zu entfernen. Leeren Sie das Auffangrohr und verwenden Sie es wieder.
- Erneut bei 14.000 × g für 2 min zentrifugieren, ohne etwas hinzuzufügen.
- Ersetzen Sie das Auffangrohr durch ein neues, sauberes Auffangrohr und trocknen Sie den SPIN-Filter 5 Minuten lang bei Raumtemperatur an der Luft.
- 50 μl DNAse freies Wasser (DES) zugeben und bei 14.000 × g für 1 min zentrifugieren.
HINWEIS: In diesem Protokoll wurden 50 μl DES für die DNA-Elution verwendet, um eine optimale Rückgewinnung und Stabilität der extrahierten Umwelt-DNA (eDNA) zu gewährleisten.
3. DNA-Quantifizierung
- 1 μl extrahierte DNA mit 200 μl Fluoreszenzfarbstoff in ein 0,5-ml-Röhrchen geben und durch Pipettieren gründlich mischen.
- Wickeln Sie das Röhrchen sofort mit Alufolie ein, damit kein Licht eindringen kann, und inkubieren Sie es 5 Minuten lang bei Raumtemperatur.
- Messen Sie die DNA-Konzentration mit einem Fluorometer im ONE DNA-Konzentrationsmodus gemäß dem Protokoll des Herstellers.
4. Nachweis von 16S rRNA, pmoA1 , pmoA2 , mmoX und mcrA durch konventionelle PCR
- Tauen Sie vor der herkömmlichen PCR (cPCR) alle Proben und Reagenzien in einem Eiskübel auf.
- Verdünnen Sie alle extrahierten eDNA-Proben auf 10 ng/μl.
HINWEIS: Eine Liste der Grundierungen ist in Tabelle 1 enthalten. - Bereiten Sie für jede Probe ein 25-μl-cPCR-Reaktionsgemisch vor, einschließlich 12,5 μl 2x PCR-Mastermix, 0,5 μl Vorwärts- und Rückwärtsprimer (10 μM) (siehe Tabelle 1 für die Primerliste), 1 μl 10 ng/μl eDNA und 10,5 μl nukleasefreies Wasser.
HINWEIS: Bereiten Sie den Mastermix mit genügend Volumen für eine zusätzliche Probe vor, um Pipettierfehler zu minimieren. - Die cPCR-Reaktion nach dem Protokoll besteht aus einer anfänglichen Denaturierung bei 95 °C für 2 Minuten, gefolgt von 40 Denaturierungszyklen bei 95 °C für 45 s, einer Verlängerung bei 72 °C für 30 s und einer abschließenden Verlängerung bei 72 °C für 5 m, mit unterschiedlicher Glühtemperatur für verschiedene Primer (siehe Tabelle 1 für die Glühtemperaturen verschiedener Primer, die für verschiedene Gene verwendet wurden).
HINWEIS: Für das mcrA-Gen zeigten ML-Primer die gewünschte Bande mit einer langsamen Rampenrate von 0,1 °C/s zwischen den Annealing- und Extensionsschritten für die ersten fünf Zyklen32. - Visualisieren Sie cPCR-Produkte in einem Agarosegel, das mit Ethidiumbromid (EtBr) vorgefärbt ist.
HINWEIS: Verwenden Sie 2,5 % Agarosegel für eine 50 bp Leiter und 0,9 % Gel für eine 1 kb Leiter. Verwenden Sie 1x TAE-Puffer für ein 2,5%iges Agarose-Gel und 0,5x TAE-Puffer für ein 0,9%iges Agarose-Gel.
5. Nachweis von pmoA1 , pmoA2 , mmoX und mcrA durch quantitative real-time PCR
HINWEIS: Die Häufigkeit von Methanogen- und Methanotroph-Zielgenen wie pmoA1, pmoA2, mmoX und mcrA wurde mittels qPCR unter Verwendung eines real-Time-PCR-Systems beobachtet.
- Bereiten Sie die Standards für jedes Gen separat vor, um die Standardkurve für jedes Gen zu erhalten.
- Amplifizieren Sie das Zielgen mit cPCR, indem Sie die Proben verwenden, die während der Gelelektrophorese jedes Gens die hellste Bande erzeugt haben. Befolgen Sie die Methode und die Primer, die in Abschnitt 4 beschrieben sind.
- Die amplifizierten Produkte werden mit einem Gelextraktionskit gereinigt und die DNA-Konzentration nach der in Abschnitt 3 beschriebenen Methode gemessen und bei -20 °C gelagert.
HINWEIS: Aliquotieren Sie die gereinigten Standards zur weiteren Verwendung in separate Röhrchen. - Berechnen Sie Genkopien aus der gemessenen DNA-Konzentration unter Verwendung der Website zur Berechnung der Kopienzahl für die qPCR.
- Verdünnen Sie die berechnete Kopienzahl des Standards mit nukleasefreiem Wasser, um jeden Standard herzustellen, und zwar im Bereich von 108 bis 102 Kopien/μl, bevor Sie die qPCR durchführen.
- Bereiten Sie die Standardkurve mit drei Replikaten jeder Kopienzahl vor, einschließlich einer Negativkontrolle (NTC, keine Template-DNA).
HINWEIS: DerR2-Wert jeder Normkurve war größer als 0,99.
- Bereiten Sie ein 20-μl-qPCR-Reaktionsgemisch für alle Proben, Standards und NTC vor. Führen Sie die qPCR-Analyse in dreifacher Ausfertigung für alle Proben durch.
- Legen Sie alle Reaktionskomponenten, einschließlich qPCR-Mastermix, Primer, nukleasefreies Wasser, Standards und Proben, auf ein Eisgitter, bevor Sie beginnen.
- Bereiten Sie für jede Probe, jeden Standard und jeden NTC ein 20-μl-qPCR-Reaktionsgemisch vor, das 10 μl SYBR Green Mastermix, 0,5 μl von jedem 10 μM Forward- und Reverse-Primer, 8 μl nukleasefreies Wasser und 1 μl entweder 10 ng/μl Template-DNA oder Standard- bzw. DI-Wasser enthält.
HINWEIS: Verwenden Sie die gezeigten Primer-Sets, um optimale Ergebnisse für pmoA1 - und mcrA-Gene in der konventionellen PCR für qPCR zu erzielen (siehe Tabelle 1). Um die Genauigkeit zu verbessern, führen Sie jede Probe in dreifacher Ausfertigung aus. Die folgenden Schritte bieten eine effiziente Methode zur Herstellung von dreifachen Proben mit einem kombinierten Volumen von 60 μl pro Probe.- Bereiten Sie das Reaktionsgemisch vor, um den Mastermix, die Vorwärts- und Rückwärtsprimer für das spezifische Gen und nukleasefreies Wasser in einem 2-ml-Röhrchen mit Ausnahme der Matrizen-DNA zu kombinieren.
HINWEIS: Bereiten Sie das Volumen der Reaktionsmischung unter Berücksichtigung eines Pipettierfehlers vor. Wenn z. B. 24 Proben und 8 Standards vorhanden sind, berechnen Sie das Gesamtvolumen für 33 Reaktionen anstelle von 32 Reaktionen, um Pipettierfehler zu minimieren. In diesem Fall wäre das für dreifache Reaktionen erforderliche Gesamtvolumen wie folgt: 990 μl Mastermix (33 Proben x 3 Replikate x 10 μl), 49,5 μl Forward-Primer (33 Proben x 3 Replikate x 0,5 μl), 49,5 μl Reverse-Primer (33 Proben x 3 Replikate x 0,5 μl) und 792 μl nukleasefreies Wasser (33 Proben x 3 Replikate x 8 μl). - Bereiten Sie PCR-Röhrchen entsprechend der Anzahl der Proben und Standards vor.
- Geben Sie 57 μl des vorbereiteten Reaktionsgemisches in jedes PCR-Röhrchen.
HINWEIS: Um die qPCR für jede Probe in dreifacher Ausfertigung durchzuführen, bereiten Sie ein Gesamtvolumen des Reaktionsgemisches von 57 μl pro Probe vor (ohne Template-DNA, Standard oder Wasser). Dieses Volumen wird zu gleichen Teilen in drei Vertiefungen für eine Probe aufgeteilt, wobei jeder Vertiefung 19 μl zugewiesen werden. - Geben Sie 3 μl Matrizen-DNA, Standard- oder nukleasefreies Wasser in jedes Röhrchen und mischen Sie, indem Sie vorsichtig auf den Boden des Röhrchens klopfen.
HINWEIS: Das Gesamtvolumen des für eine Probe vorbereiteten Reaktionsgemisches würde jetzt 60 μl in jedem Röhrchen betragen.
- Bereiten Sie das Reaktionsgemisch vor, um den Mastermix, die Vorwärts- und Rückwärtsprimer für das spezifische Gen und nukleasefreies Wasser in einem 2-ml-Röhrchen mit Ausnahme der Matrizen-DNA zu kombinieren.
- Aliquotieren Sie 20 μl des vorbereiteten Reaktionsgemisches aus jedem Röhrchen in die dafür vorgesehenen Vertiefungen einer 96-Well-qPCR-Platte. Verschließen Sie die PCR-Platte mit einem Applikator mit einer selbstklebenden PCR-Siegelfolie.
- Zentrifugieren Sie die versiegelte Platte 1 Minute lang bei 1.000 × g , um eine ordnungsgemäße Durchmischung zu gewährleisten und Blasen in den Vertiefungen zu entfernen.
- Setzen Sie die PCR-Platte in den Thermocycler ein. Schalten Sie das qPCR-Gerät ein und öffnen Sie dann die zugehörige Software, um das Protokoll einzurichten.
- Richten Sie das Protokoll gemäß den qPCR-Mastermix-Richtlinien ein. Verwenden Sie das folgende Protokoll: 95 °C für 10 Minuten, gefolgt von 95 °C für 15 s und eine Verlängerungsstufe bei 72 °C für 30 s. Der Glühschritt wird bei der für die entsprechenden Primer in Tabelle 1 angegebenen Glühtemperatur 45 s lang durchgeführt. Führen Sie alle qPCR-Läufe über 35 Zyklen durch.
- Richten Sie die 96-Well-qPCR-Platte mit Standards und NTCs in der gleichen Konfiguration wie die probenhaltige Platte ein.
- Es wird die Standardkurvenmethode der absoluten Quantifizierung verwendet, um das amplifizierte Produkt und die Genkopienzahl in jeder Probe zu quantifizieren33.
6. Visualisierung von Genen des Methankreislaufs in der Karte der Feuchtgebiete an der Küste von Südtexas
- Öffnen Sie die GIS-Software (GIS) ArcGIS Pro , und speichern Sie die Projektdatei mit dem Namen Untersuchungsgebiet im angegebenen Ordner auf dem Computer.
- Klicken Sie oben links auf Karte | Grundkarte, und wählen Sie Terrain mit Beschriftungen als Grundkarte aus.
- Klicken Sie auf Suchen | Suchen Sie und wenn sich die Suchleiste öffnet, suchen Sie das Untersuchungsgebiet, indem Sie den Namen des Gebiets eingeben. Der Bereich wird angezeigt.
- Zeichnen Sie den spezifischen Bereich mithilfe der Georeferenzierung.
- Klicken Sie auf Ansicht | Katalogbereich aus der obersten Ebene.
- Doppelklicken Sie auf Ordner aus dem Katalog | Dateiname.
- Klicken Sie mit der rechten Maustaste auf die Geodatabase-Datei (.gdb), und klicken Sie dann auf Neu | Feature-Class und Feature-Class erstellen werden angezeigt.
- Geben Sie das Feld Name und Alias ein und klicken Sie unten auf Fertig stellen .
- Klicken Sie auf Ansicht | Inhalt. Der Aliasname wird im Inhaltsbereich angezeigt.
- Klicken Sie in der obersten Ebene auf Bearbeiten | Kreieren. Der Bereich Features erstellen wird geöffnet. Doppelklicken Sie im Bereich Features erstellen auf den Alias, und die Feedback-Optionen für das Werkzeug konfigurieren werden angezeigt.
- Wählen Sie Linien aus, und skizzieren Sie dann Linien auf der Karte, um eine äußere Begrenzung des Untersuchungsgebiets zu erstellen. Doppelklicken Sie auf die Karte, wenn Sie fertig sind.
- Minimieren Sie die GIS-Software, und öffnen Sie dann eine Tabelle. Geben Sie den Namen des Beispiels in die erste Spalte ein, geben Sie den Breitengrad in der zweiten Spalte und den Längengrad in der dritten Spalte ein. Verwenden Sie die nächsten vier Spalten für die qPCR-Daten von pmoA1, pmoA2, mmoX und mcrA.
- Speichern Sie die Datei im CSV-Format in einem bestimmten Ordner des Computers.
- Öffnen Sie die GIS-Software erneut, und klicken Sie auf Daten hinzufügen | XY-Punkt-Daten.
- Wählen Sie die CSV-Datei aus dem Ordner auf dem Computer im Feld Eingabetabelle aus. Benennen Sie den Dateinamen in der Ausgabe-Feature-Class um, und klicken Sie dann auf Ausführen , um die Probenahmepunkte auf der Karte anzuzeigen.
- Klicken Sie auf dievSuchleiste oben und suchen Sie nach Kriging.
- Wählen Sie die Datei der Probenahmestation und dann pmoA1 aus.
- Klicken Sie auf die Schaltfläche Umgebung | Wählen Sie die Probenahmestation in Layer und Maske aus | Klicken Sie auf Ausführen.
- Befolgen Sie die in den Schritten 6.9, 6.10 und 6.11 genannten Protokolle, um Kriging für pmoA2, mmoX und mcrA für das gesamte Untersuchungsgebiet zu erstellen.
- Erstellen Sie ein Layout der Karte.
- Klicken Sie auf Aus der obersten Ebene einfügen | Neues Layout, und wählen Sie ANSI - Querformat aus.
- Klicken Sie auf Kartenrahmen, wählen Sie die Karte mit dem Kriging aus und platzieren Sie alle Karten im Layout, indem Sie ein Rechteck zeichnen. Dadurch wird die Karte im Layout sichtbar.
- Wählen Sie den Nordpfeil aus, und platzieren Sie ihn im Layout, um die Nordrichtung anzugeben.
- Wählen Sie Maßstabsleiste aus, um den Maßstab des Bereichs auf der Karte anzuzeigen.
- Klicken Sie auf Legende , um die Legenden anzuzeigen, und platzieren Sie sie dann im Layout.
- Klicken Sie auf Raster und wählen Sie eine der Optionen für das schwarze Raster aus. Dadurch wird das Raster mit dem Breiten- und Längengrad erstellt und im Inhaltsbereich mit der Beschriftung Schwarzes horizontales Beschriftungsraster angezeigt.
- Doppelklicken Sie auf Schwarzes horizontales Beschriftungsraster und wählen Sie dann Komponenten. Klicken Sie auf die Häkchen 1 und das Raster und entfernen Sie diese Komponenten, indem Sie auf das Kreuzzeichen rechts davon klicken.
- Klicken Sie in der obersten Ebene auf Freigeben und dann auf Layout exportieren. Wählen Sie den Dateityp als PDF aus, speichern Sie die Datei über das Namensfeld auf dem Computer, stellen Sie die vertikale Auflösung auf 500 DPI ein, und klicken Sie auf Exportieren , um die PDF-Datei der Karte zu erstellen.
Ergebnisse
Um die Verteilung und Häufigkeit von CH 4-Zyklus-bezogenen Genen (mcrA, pmoA1, pmoA2 und mmoX) in den Küstenfeuchtgebieten von Südtexas zu verstehen, wurde die extrahierte eDNA aus jeder Probe mittels cPCR und qPCR analysiert. Für die Durchführung der cPCR aus früheren Studien wurden universelle Primer für jeden Biomarker ausgewählt (Tabelle 1)22,34,35,36,37, und es wurden Modifikationen vorgenommen, um die Glühtemperaturen und -konzentrationen basierend auf den Probeneigenschaften und den Umgebungsbedingungen zu optimieren. Zum Beispiel amplifizierten 16S rRNA, pmoA1 und pmoA2 nicht mit der Standard-Glühtemperatur, da die meisten von ihnen weit unter der Standardglühtemperatur amplifizierten (Daten nicht gezeigt). Darüber hinaus zeigte eine Konzentration von 10 ng/μl DNA während des cPCR-Prozesses eine deutliche Bande für die Gene pmoA1, pmoA2 und mmoX, während die Verwendung von 5 ng/μl Template-DNA eine hellere Bande für das mcrA-Gen aufwies (Supplemental File 1-Supplemental Abbildung S1).
Die cPCR-Analyse zeigte eine räumliche Variabilität beim Gennachweis in drei Arten von Küstenfeuchtgebieten in Südtexas. Wie in Tabelle 2 gezeigt, wurden mcrA-Gene in allen Proben aus LB und RV nachgewiesen, bei denen es sich um küstennahe Süßwasserfeuchtgebiete handelt. Mit der cPCR konnte jedoch kein mcrA-Gen im beprobten Sediment des Salzwasser-Feuchtgebiets nachgewiesen werden, was auf einen möglichen Zusammenhang zwischen dem Salzgehalt und der Methanogenverteilung hindeutet (Ergänzende Datei 1 - Ergänzende Abbildung S2). Für die mcrA-Detektion wurden in dieser Studie vier Primerpaare verwendet, darunter MLF-MLR, MCRf-MCRr, ME1F-ME2R und McrA 159F-McrA 345R (Tabelle 1); unter ihnen waren nur ML-Primerpaare in der Lage, die Methanogengemeinschaft in südtexanischen Feuchtgebieten nachzuweisen (Ergänzende Datei 1 - Ergänzende Abbildung S2, Ergänzende Abbildung S3 und Ergänzende Abbildung S4). Im Gegensatz dazu wurde bei aeroben Methanotrophen das pmoA1-Gen in allen RV-Proben nachgewiesen, fehlte jedoch in LB- und BC-Proben (Supplemental File 1 - Supplemental Abbildung S5). Zur Identifizierung von pmoA1 wurde A189 als Forward-Primer und A682 und mb661 als Reverse-Primer verwendet, wobei A189-A682-Primerpaare auch amoA35,38 detektieren können. Dieses Primerpaar war jedoch in der Lage, das Vorhandensein von Genen in der cPCR in dieser Studie nachzuweisen, wobei die Gelbilder eine sehr schwache Bande zeigten, was auf die geringe Häufigkeit des pmoA1-Gens hindeutet. Ein weiterer reverser Primer mb661 wurde entwickelt, um das pmoA1-Gen spezifisch mit A189 als Forward-Primer38 zu identifizieren. Wenn mb661 als reverser Primer verwendet wurde, zeigte die cPCR eine hellere Bande (Supplemental File 1-Supplemental Figure S6), die später in der qPCR verwendet wurde. Interessanterweise wurde das pmoA2-Gen in zwei BC-Proben und einer LB-Probe nachgewiesen, fehlte jedoch in einer RV-Probe (Supplemental File 1-Supplemental Figure S7). Eine frühere Studie ergab eine Glühtemperatur von 66 °C und 60 °C für pmoA2, wobei bei 60 °C zwei unterschiedlich große Amplikone, 245 und 438 bp, beobachtet wurden, während bei 66 °C 206f-703b-Primer nur pmoA2 mit 438 bp Produktgröße22 amplifizieren können. In dieser Studie war die Amplifikation jedoch bei einer Glühtemperatur von 60 °C erfolgreich, wodurch eine Bande der erwarteten Größe (438 bp) an LB- und BC-Stellen erzeugt wurde (Ergänzende Datei 1 - Ergänzende Abbildung S7). Darüber hinaus wurde das mmoX-Gen in einer LB-Probe nachgewiesen, fehlte jedoch in einer RV-Probe (Supplemental File 1 - Supplemental Figure S8) und fehlte in BC-Proben vollständig. Die für mmoX verwendeten Primerpaare eigneten sich auch für die Untersuchung der südtexanischen Feuchtgebiete in dieser Studie. Eine Glühtemperatur von 55 °C36 erzeugte eine helle Bande für mmoX in der cPCR (Supplemental File 1 - Supplemental Figure S8). Um die Integrität der Probe zu überprüfen, wurde 16S rRNA als Positivkontrolle mit erfolgreicher Amplifikation der 16S rRNA in allen Proben während der cPCR verwendet, was darauf hindeutet, dass die Proben nicht kontaminiert waren (Ergänzende Datei 1 - Ergänzende Abbildung S9).
Um zeitliche und räumliche Veränderungen der Genhäufigkeit zu beurteilen, wurde eine qPCR-Analyse an allen Zielgenen unter Verwendung optimierter Primer-Sets durchgeführt, die in der cPCR identifiziert wurden. Die qPCR der pmoA2 - und mmoX-Gene wurde mit den gleichen Primern durchgeführt, die auch bei der cPCR verwendet werden. Für das pmoA1-Gen wurde jedoch das Primerpaar A189-mb661 verwendet, und für das mcrA-Gen wurde das MLF-MLR-Primerpaar ausgewählt, da beide gute Ergebnisse in der cPCR lieferten. Saisonale Unterschiede und Schwankungen des Salzgehalts in den Feuchtgebieten von Südtexas wurden ebenfalls berücksichtigt, da sie die Dynamik der mikrobiellen Gemeinschaft beeinflussen könnten. Wie in Abbildung 3 und Abbildung 4 gezeigt, gab es einen Unterschied in der Genhäufigkeit von Methanogenen und Methanotrophen zwischen zwei verschiedenen Probenahmezeiträumen. Während es keine saisonale Variation des Salzgehalts in den küstennahen Süßwasserfeuchtgebieten gab, wurde eine saisonale Variation des Salzgehalts in den küstennahen Salzwasserfeuchtgebieten beobachtet39, was die Variation der Methanogen- und Methanotrophenhäufigkeit erklären könnte. Abbildung 3 zeigt, dass der mmoX während der kühlen Jahreszeit in all diesen Feuchtgebieten sehr häufig vorkam, mit der höchsten Abundanz von 1,46 × 104 Kopien/μL. Es gab einen kleinen Prozentsatz von pmoA2 in einer Station von LB, RV und BC. Die qPCR konnte pmoA1 und mcrA jedoch aufgrund der geringen Abundanz in der kühlen Jahreszeit nicht nachweisen. Im Gegensatz dazu war pmoA1 in der warmen Jahreszeit am häufigsten vorhanden, mit der höchsten Abundanz von 2,44 × 103 Kopien/μL (Abbildung 4). Die mmoX und mcrA wurden auch in den Oberflächensedimentproben von LB (innerhalb der oberen 50 cm) nachgewiesen und zeigten die maximale Abundanz von 1,92 × 102 Kopien/μL bzw. 2,04 × 102 Kopien/μL, trotz ihrer relativ geringen Abundanz während der warmen Jahreszeit. Interessanterweise wurde pmoA2 während der warmen Jahreszeit überhaupt nicht nachgewiesen, und keines der Zielgene wurde in BC-Proben nachgewiesen, was wahrscheinlich von Umweltfaktoren beeinflusst wurde, die für dieses Salzwasserfeuchtgebiet während der warmen Jahreszeit in Südtexas einzigartig sind39 .
Diese Studie zeigt deutliche räumliche und saisonale Variationen in der Verteilung von CH 4-Schaltkreis-bezogenen Genen in Küstenfeuchtgebieten. Konkret wurden mcrA-Gene nur in der Süßwasser-LB-Probe nachgewiesen, was auf eine Habitatpräferenz hindeutet. Die mmoX- und pmoA2-Gene waren in allen Proben in kühlen Jahreszeiten vorhanden, während pmoA1 in Süßwasserumgebungen während der warmen Jahreszeiten auftrat. Diese Ergebnisse unterstreichen den Einfluss sowohl von Umweltbedingungen als auch von saisonalen Verschiebungen auf die mikrobiellen Aktivitäten im Zusammenhang mit CH4-Zyklen. Die verbesserte Visualisierung mittels Kriging-Interpolation, wie in Abbildung 3 und Abbildung 4 gezeigt, hat unser Verständnis der mikrobiellen Aktivitäten im Zusammenhang mit CH4-Zyklen in Ökosystemen von Küstenfeuchtgebieten erweitert.
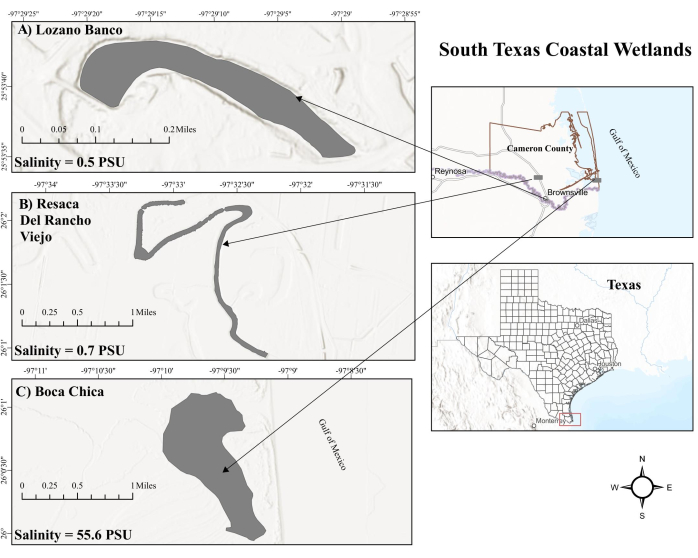
Abbildung 1: Karte der Küstenfeuchtgebiete in Südtexas. a) Lozano Banco. LB ist ein küstennahes Süßwasserfeuchtgebiet. (b) Resaca del Rancho Viejo. RV ist auch ein Süßwasserfeuchtgebiet an der Küste. (C) Feuchtgebiet von Boca Chica. BC ist ein vom Gezeiten beeinflusstes, hypersalines Salzwasser-Feuchtgebiet an der Küste. Abkürzungen: LB = Lozano Banco; RV = Resaca Del Rancho Viejo; BC = Boca Chica; PSU = praktische Einheiten des Salzgehalts. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
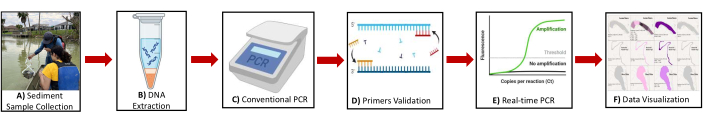
Abbildung 2: Schematische Darstellung der Übersicht über das Protokoll. Die Veranschaulichung der wichtigsten Schritte umfasst: (A) Entnahme von Sedimentproben, (B) DNA-Extraktion, (C) konventionelle PCR, (D) Validierung des Primers, (E) Echtzeit-PCR, (F) Datenvisualisierung zur Identifizierung von Methanogen und Biomarker-Genen von Methanotrophen. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
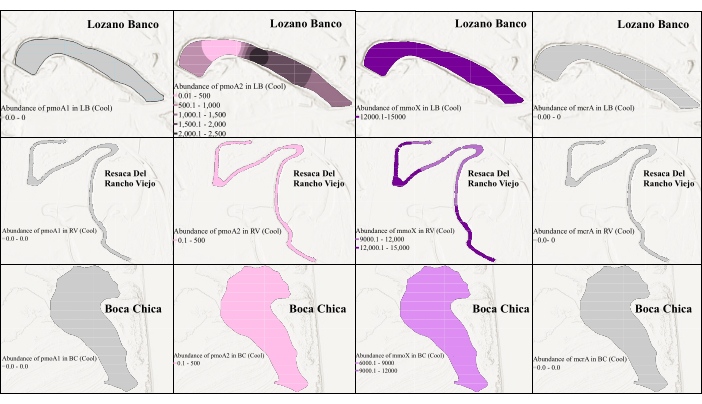
Abbildung 3: Räumliche Verteilung von Methanogen und Biomarker-Genen von Methanotrophen in der kühlen Jahreszeit. Die Abbildung zeigt den Überfluss an mmoX während der kühlen Jahreszeit. Hier deutet die graue Farbe darauf hin, dass die qPCR das Gen aufgrund der geringen Abundanz nicht nachweisen konnte. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.

Abbildung 4: Räumliche Verteilung von Methanogen und den Biomarker-Genen von Methanotrophen in der warmen Jahreszeit. Hier steht die graue Farbe dafür, dass die qPCR das Gen aufgrund seiner geringen Häufigkeit nicht nachweisen konnte. Die Abbildung zeigt die Häufigkeit von pmoA1 während der warmen Jahreszeit in Lozano Banco und Resaca Del Rancho Viejo, während in Boca Chica keine Häufigkeit festgestellt wurde. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
| Name der Grundierung | Gen | Glühtemperatur (°C) in dieser Studie | Produktlänge (bp) | Sequenz (5'-3') | Referenz | |
| 1055F | 16S rRNA | 55 | 337 | ATGGCTGTCGTCAGCT | (Harms et al., 2003) | |
| 1392R | ACGGGCGGTGTGTAC | |||||
| Nr. A189F | pmoA1 | 55 | 525 | GGNGACTGGGACTTCTGG | (Holmes et al., 1995) | |
| A682R | GAASGCNGAGAAGAASGC | |||||
| Nr. A189F | 56 | 470 | GGNGACTGGGACTTCTGG | (Costello & Lidstrom, 1999) | ||
| mb661R | CCG GMG CAA CGT CYT TACC | |||||
| 206f | pmoA2 | 60 | 438 | GGNGACTGGGACTTCTG GATCGACTTCAAGGATCG | (Tchawa Yimga et al., 2003) | |
| 703b | GAASGCNGAGAAGAASGC GGCGACCGGAACGACGT | |||||
| 536F | mmoX (Englisch) | 55 | 362 | CGCTGTGGAAGGGCATGAAGCG | (Fuse et al., 1998) | |
| 898R | GCTCGACCTTGAACTTGGAGCC | |||||
| MCRf | mcrA | 46 | ~490 kg | TAYGAYCARATHTGGYT | (Springer et al., 1995) | |
| MCRr | ACRTTCATNGCRTARTT | |||||
| ME1F | 50 | 763 | GCMATGCARATHGGWATGTC | (Hales et al., 1996) | ||
| ME2R | TCATKGCRTAGTTDGGRTAGT | |||||
| MLF | 55 | ~470 kg | GGTGGTGTGTMGGATTCA CACARTAYGCWACAGC | (Luton et al., 2002) | ||
| MLR | TTCATTGCRTAGTTWGGRTAGTT | |||||
| McrA 159F | 62 | 186 | AAAGTGCGGAGCAGCAATCACC | (Vaksmaa et al., 2017) | ||
| McrA 345R | TCGTCCCATTCCTGCTGCATTGC | |||||
Tabelle 1: Primer-Sets, die in dieser Studie verwendet wurden, um funktionelle Gene zu erkennen, die mit dem Methankreislauf in Verbindung stehen. Alle diese Primerpaare wurden für die konventionelle PCR verwendet, und schließlich wurden die fettgedruckten Paare für den Nachweis von Methanogen und methanotrophen Genen in Feuchtgebieten an der Küste von Südtexas eingerichtet.
| Gen | Proben | ||||||||
| Lozano Banco | Resaca Del Rancho Viejo | Boca Chica | |||||||
| LB-1 | RV-1 | RV-2 | M1 | M2 | B1 | B2 | MG1 | MG2 | |
| pmoA1 | - | + | + | - | - | - | - | - | - |
| pmoA2 | + | + | + | - | + | - | - | + | - |
| mmoX (Englisch) | + | - | + | - | - | - | - | - | - |
| mcrA | + | + | + | - | - | - | - | - | - |
Tabelle 2: Nachweis von Genen in drei verschiedenen Gewässertypen in der kühlen Jahreszeit mittels konventioneller PCR. '+', Gegenwart; '-', abwesend. Abkürzungen: LB = Lozano Banco, RV = Resaca del Rancho Viejo, M = Schlamm, B = Batis, MG = Mangrove.
Ergänzende Datei 1: Gelelektrophorese-Bilder für mcrA-, pmoA1-, pmoA2-, mmoX- und 16S-rRNA-Gene. Bitte klicken Sie hier, um diese Datei herunterzuladen.
Diskussion
Küstenfeuchtgebiete gelten als bedeutende Verursacher von atmosphärischem Methan, einem wichtigen Treibhausgas40. Obwohl es Studien über den Methanfluss und Methanogene in Feuchtgebieten gibt 41,42,43, ist wenig darüber bekannt, wie Methanotrophen in verschiedenen Umgebungen oder unter verschiedenen Bewirtschaftungspraktiken funktionieren, insbesondere in Feuchtgebieten mit schwankenden Wasserständen44. Darüber hinaus erschwert das weit verbreitete Vorhandensein von Inhibitoren wie Huminsäuren in der eDNA die Extraktions- und Amplifikationsprozesse45, was möglicherweise zu falsch negativen Ergebnissenführt 46. Diese Herausforderung wird durch die unterschiedliche Effizienz von Primern, die auf eDNA wirken, noch komplizierter, was auf die Notwendigkeit einer systematischen Validierung in solchen Studien hindeutet47. Daher etabliert diese Forschung eine grundlegende Methodik zum Verständnis des Methankreislaufs in den wenig untersuchten Küstenfeuchtgebieten von Südtexas. Durch den Fokus auf die Entwicklung und Validierung von PCR-Techniken fördert diese Studie den Nachweis wichtiger Gene des Methankreislaufs in diesen dynamischen Ökosystemen.
Einer der kritischen Aspekte dieser Forschung ist die Visualisierung von Genen, die mit dem mikrobiellen Methankreislauf in Verbindung stehen, was eine entscheidende Rolle bei der Beobachtung der räumlichen Verteilungsmuster dieser Gene über verschiedene Jahreszeiten und Salzgehaltsgradienten hinweg spielt. Eine solche Visualisierung ist nicht nur ein ergänzendes Werkzeug; Es ist wichtig, um Gebiete mit signifikanten methanogenen und methanotrophen Mikroben zu identifizieren, die auf einen potenziellen Methankreislauf hinweisen, und damit unser Verständnis der Umweltbedingungen, die diese Prozesse beeinflussen, zu verbessern.
In dieser Studie wurde die Methodik zur Identifizierung von Methanogen und methanotrophen Markergenen wie pmoA1, pmoA2, mmoX und mcrA mittels cPCR und qPCR spezifiziert. Es wurden vier Primerpaare verwendet, die auf das mcrA-Gen abzielen, und zwei Paare, die auf das pmoA1-Gen abzielen, und nur ein Paar aus jedem Satz konnte diese Gene sowohl in der konventionellen als auch in der Echtzeit-PCR effektiv nachweisen. In einer früheren Studie wurden drei Primerpaare zum Nachweis von pmoA1 verwendet; unter ihnen konnten A189-A682-Primer die methanotrophe Diversität nicht nachweisen. Stattdessen amplifizierten sie hauptsächlich das amoA-Gen und lieferten unspezifische PCR-Produkte48. Diese Studie ergab, dass das verwendete Primerpaar nur in Umgebungen mit einer hohen Population von Methanotrophen für den Nachweis des pmoA1-Gens wirksam war. Darüber hinaus wurde die höchste Abundanz von Methanotrophen unter Verwendung der A189-mb661-Primerpaare in dieser Studie erzielt, was darauf hindeutet, dass diese Primer für Süßwasserökosysteme geeignet sind48. Daher wurden diese Primerpaare für die qPCR als weitere Analyse verwendet.
Von den vier Primerpaaren, die für mcrA verwendet wurden, waren ML-Primer in der Lage, Methanogenspezies in allen Umgebungen zu amplifizieren49. ME-Primer haben einen engen Verstärkungsbereich; Sie können nicht alle Arten von Methanogenarten in verschiedenen natürlichen Umgebungen nachweisen. MCR-Primer weisen einen hohen Grad an Degeneration auf, was sie empfindlich gegenüber der PCR-Annealing-Temperatur macht. Diese Entartung ermöglicht es ihnen, an eine breite Palette von Zielsequenzen zu binden, wobei sie oft mehrere Fehlanpassungen berücksichtigen. Dies deutet jedoch auch darauf hin, dass eine kleine Änderung der Glühtemperatur ihre Bindungseffizienz und Spezifität signifikant beeinflussen kann, was zu einer unzureichenden Amplifikation und folglich zu falsch negativen Ergebnissen für die Methanogenspeziesführt 49,50.
Andererseits führt in der Methanogengemeinschaft mit geringer Diversität in den letzten Stadien der PCR eine hohe Akkumulation von PCR-Produkten zu einem erneuten Anlügen der Matrizen und verhindert die Bindung der Primer, wodurch auch die gewünschten Banden nicht nachgewiesen werdenkönnen 49. Interessanterweise wurden McrA 159F-McrA 345R Primerpaare speziell entwickelt, um nitratabhängige anaerobe methanotrophe Candidatus M. nitroreducens archaea51 zu identifizieren, und diese Studie konnte keine anaeroben Methanotrophen mit diesen Primerpaaren nachweisen. Die anaerobe Oxidation von Methan (AOM) kann jedoch mit der Reduktion alternativer Elektronenakzeptoren wie Eisen (Fe), Mangan (Mn) und Sulfat (SO42-)52 gekoppelt werden, was die Unfähigkeit erklären könnte, ANME mit dieser Methode nachzuweisen. Darüber hinaus kann es an den spezifischen Umweltbedingungen des Untersuchungsgebiets liegen. Daher wurden für die qPCR ML-Primer zum Nachweis der Methanogenhäufigkeit verwendet, da sie auch unter Bedingungen geringer Gemeinschaftsdiversität eine zuverlässige Amplifikation ermöglichten49.
Darüber hinaus sind PCR-Inhibitoren wie Huminsäuren oder DNA von Nichtzielspezies in Umweltproben weit verbreitet, was die PCR-Effizienz verringern kann53. Da durch die hohe DNA-Konzentration kein Gen durch cPCR nachgewiesen werden konnte, wurden alle DNA-Proben vor der cPCR verdünnt, um die Wirkung der PCR-Inhibitoren zu verringern54. Das 16S rRNA-Gen als universeller Marker für Bakterien und Archaeen lieferte eine robuste Grundlage für die Validierung unserer PCR-Bedingungen und die Bestätigung der DNA-Integrität in allen Proben 55,56. Es ist jedoch bekannt, dass Salzwassergebiete höhere Konzentrationen von PCR-Inhibitorenaufweisen 46. Darüber hinaus beeinflusst der Salzgehalt indirekt die CH4-Emissionen in küstennahen Feuchtgebieten, da er mit den Sulfatkonzentrationen in Verbindung gebracht wird. Ein höherer Salzgehalt in Salzwasser-Feuchtgebieten korreliert mit erhöhten Sulfatgehalten, die die Methanproduktion aufgrund der Konkurrenz zwischen sulfatreduzierenden Bakterien und Methanogenen unterdrücken, während niedrigere Sulfatkonzentrationen in Süßwasser-Feuchtgebieten zu höheren Methanemissionen führen11,41. Darüber hinaus werden die CH 4-Emissionsunterschiede zwischen Salzwasser- und Süßwasserfeuchtgebieten auch durch ihre Gezeitenverbindungen zum Ozean beeinflusst57, was auch den erfolglosen Nachweis von Genen, die mit dem Methankreislauf in Verbindung stehen, durch cPCR in BC erklären könnte. In dieser Studie wurden mit der cPCR alle Zielgene in LB- und RV-Proben erfolgreich nachgewiesen, das Vorhandensein dieser Gene in BC-Proben jedoch nicht nachgewiesen, wobei das pmoA2-Gen die einzige Ausnahme darstellte. Darüber hinaus war die qPCR in der Lage, mmoX in allen Proben nachzuweisen, während die cPCR es in BC nicht nachweisen konnte. Dies deutet darauf hin, dass die qPCR im Vergleich zur cPCR eine größere Resilienz gegenüber PCR-Inhibitoren aufweist, was auf ihre erhöhte Sensitivität der Nachweismechanismen zurückzuführenist 58. Ein weiterer Grund dafür ist, dass die qPCR PCR-Produkte in der exponentiellen Phase des PCR-Zyklus identifiziert, während sie bei der cPCR in der Plateauphase nachgewiesen werden. Diese Unterscheidung ermöglicht es der qPCR, während fortgeschrittener Reaktionszyklen, in denen die Reagenzien erschöpft sind, eine verringerte Anfälligkeit für den Produktabbau zu zeigen47.
In dieser Studie wurde eine PCR-Technik zur Identifizierung von Genen im Zusammenhang mit dem Methankreislauf in Feuchtgebieten an der Küste von Südtexas etabliert, um die Herausforderungen zu bewältigen, die mit PCR-Inhibitoren und variablen Primereffizienzen verbunden sind. Es wurden vier Primerpaare verwendet, die auf das mcrA-Gen abzielen, und zwei Paare, die auf das pmoA1-Gen abzielen, und nur ein Paar aus jedem Satz konnte diese Gene sowohl in der konventionellen als auch in der Echtzeit-PCR effektiv nachweisen. Diese Studie schließt den Satz von Primerpaaren für Gene ab, die mit dem Methankreislauf in Verbindung stehen, einschließlich a189-mb6661 für pmoA1, 206f-703b für pmoA2, 536F-898R für mmoX und ML für mcrA. Dies deutet auf die Bedeutung einer geeigneten Primerauswahl für den Nachweis von Genen im Zusammenhang mit dem Methankreislauf in Küstenfeuchtgebieten hin, wobei die in Südtexas als Beispiele dienen. Darüber hinaus zeigte die Studie, dass mmoX in der kühlen Jahreszeit am häufigsten vorkam, während pmoA1 in der warmen Jahreszeit reichlich vorhanden war.
Die Ergebnisse dieser Studie bilden somit die Grundlage für ein wissenschaftliches Verständnis der mikrobiellen Prozesse, die am Methankreislauf beteiligt sind, und unterstreichen den Einfluss von Umweltfaktoren auf saisonale und räumliche Schwankungen in den Feuchtgebieten der Küste von Südtexas. Basierend auf diesen Erkenntnissen zielen zukünftige Studien darauf ab, diesen Ansatz in anderen Ökosystemen wie Reisfeldern und Süßwasserfeuchtgebieten weiter zu validieren, um die Robustheit und Generalisierbarkeit zu erhöhen. Darüber hinaus werden diese Studien auch geochemische Analysen wie Methankonzentrationen im Sediment und Porenwassersulfid sowie molekulare Techniken wie Metagenomik und Transkriptomik umfassen, die auf dieser Grundlage basieren, um ein umfassenderes Verständnis der Dynamik des Methankreislaufs zu ermöglichen.
Offenlegungen
Die Autoren haben keine Interessenkonflikte anzugeben.
Danksagungen
Wir danken den C-REAL-Mitgliedern für ihre Unterstützung bei der Feldbeobachtung und bei Laboranalysen.
Materialien
| Name | Company | Catalog Number | Comments |
| 0.2 mL PCR tubes | ThermoFisher Scientific | AB0620 | https://www.thermofisher.com/order/catalog/product/AB0620?SID=srch-srp-AB0620 |
| 0.5 mL PCR Tubes | Promega | E4941 | https://www.promega.com/products/biochemicals-and-labware/tips-and-accessories/0_5ml-pcr-tubes/?catNum=E4941 |
| 10 μL tips | ThermoFisher Scientific | 05-408-187 | Fisherbrand SureGrip Pipet Tip Racked or Reload System Tips Natural; 10μL; | Fisher Scientific |
| 15 mL centrifuge tube | ThermoFisher Scientific | 14-959-53A | https://www.fishersci.com/shop/products/falcon-15ml-conical-centrifuge-tubes-5/p-193301 |
| 200 μL tips | ThermoFisher Scientific | 05-408-190 | Fisherbrand SureGrip Pipet Tip Racked or Reload System Tips Natural; 200μL; | Fisher Scientific |
| 1000 μL tips | ThermoFisher Scientific | 02-707-402 | https://www.fishersci.com/shop/products/sureone-micropoint-pipette-tips-specific-standard-fit/02707402?gclid=Cj0KCQiAp NW6BhD5ARIsACmEb kUsQ9Lu0YIq5i4vWege 17qPdtxIYZyvmJH1cDo ARuwereO1V4GLz9UaA lDREALw_wcB&ef_id=C j0KCQiApNW6BhD5ARI sACmEbkUsQ9Lu0YIq5i 4vWege17qPdtxIYZyvmJ H1cDoARuwereO1V4GLz 9UaAlDREALw_wcB:G:s &ppc_id=PLA_goog_2175 7693617_171052169911_02 707402__715434303113_1555 377385658230343&ev_chn=sh op&s_kwcid=AL!4428!3!71543430 3113!!!g!2366517300713!&gad_source=1 |
| Applied Biosystem Power SYBR Green Master Mix | ThermoFisher Scientific | 4368577 | https://www.thermofisher.com/order/catalog/product/4368577 |
| ArcGIS Pro | esri | https://www.esri.com/en-us/arcgis/products/arcgis-pro/overview?srsltid=AfmBOopatJ4 JvHJfscHRcAaDx0Jz5_Jrl8l5 vYkkBvfOqE-uNSsMghN1 | |
| CFX Duet Real-Time PCR system | Bio-Rad | 12016265 | https://www.bio-rad.com/en-us/product/cfx-duet-real-time-pcr-system?ID=97722926-9ed9-16a4-1d83-c92f587e427a |
| Corning Lambda plus single channel pipettor volume 0.5-10 μL | Sigma-Aldrich | CLS4071-1EA | https://www.sigmaaldrich.com/US/en/product/sigma/cls4071 |
| Corning Lambda plus single channel pipettor volume 100-1000 μL | Sigma-Aldrich | CLS4075-1EA | https://www.sigmaaldrich.com/US/en/product/sigma/cls4075 |
| Corning Lambda plus single channel pipettor volume 20-200 μL | Sigma-Aldrich | CLS4074-1EA | https://www.sigmaaldrich.com/US/en/product/sigma/cls4074 |
| FastDNA spin kit for soil | MP Biomedical | 116560200-CF | https://www.mpbio.com/us/116560000-fastdna-spin-kit-for-soil-samp-cf?srsltid=AfmBOoqOxxGilzY3IHNIZR ajegGTr9MoX1oMZUh 3dcbJqe0UvvukY128 |
| Gene copy calculator | Science Primer | https://scienceprimer.com/copy-number-calculator-for-realtime-pcr . | |
| High speed benchtop centrifuge | ThermoFisher Scientific | 75004241 | https://newlifescientific.com/products/thermo-scientific-sorvall-st16-high-speed-benchtop-centrifuge-75004241?gad_source=1&gclid=Cj0KCQiApN W6BhD5ARIsACmEbkVC_-cCIN9j 20TvYq8iDsBlUR5cPK_1_wN OBEcjMdv-CYVoGCfeOLYaAv enEALw_wcB |
| High speed microcentrifuge | VWR | 75838-336 | https://us.vwr.com/store/product/20546590/null |
| Lysing Matrix E tube | glass bead/ceramic sphere-containing tube | ||
| Microcentrifuge tube | ThermoFisher Scientific | 02-681-320 | https://www.fishersci.com/shop/products/fisherbrand-low-retention-microcentrifuge-tubes-8/02681320?gclid=Cj0KCQiAp NW6BhD5ARIsACm EbkWbG4_o3oUiGk HJPU-_31-CuexDwQ fmWPnfyhBOf2BHXsy K3fFW1toaAgJbEALw_ wcB&ef_id=Cj0KCQiAp NW6BhD5ARIsACmEb kWbG4_o3oUiGkHJPU- _31-CuexDwQfmWPnfy hBOf2BHXsyK3fFW1toa AgJbEALw_wcB:G:s&ppc _id=PLA_goog_21757693 617_171052169911_0268 1320__715434303113_10 349826094968484711&ev _chn=shop&s_kwcid=AL!4 428!3!715434303113!!!g!23 66517300713!&gad_source=1 |
| PCR Master mix | Promega | M7502 | https://www.promega.com/products/pcr/taq-polymerase/master-mix-pcr/?catNum=M7502 |
| Quantiflour ONE dsDNA system | Promega | E4871 | https://www.promega.com/products/rna-analysis/dna-and-rna-quantitation/quantifluor-one-dsdna-system/?gad_source=1&gbraid=0AAAAAD _rg189yJTY3cxeVqMdu8RPx10 Ma&gclid=CjwKCAjwxNW2BhAk EiwA24Cm9FUgViPNyWq7UfZL VeeoroLAZ5JIP6w07RGK_4D0w oZgAqf-G1XTmxoCxm8QAvD_B wE&catNum=E4871 |
| Quantus Fluorometer | Promega | E6150 | https://www.promega.com/products/microplate-readers-fluorometers-luminometers/fluorometers/quantus-fluorometer/?catNum=E6150 |
| YSI Pro 2030 | YSI a xylem brand | 603174 | https://www.ysi.com/product/id-p2030/pro2030-kits |
Referenzen
- Xu, T., et al. Wetlands of international importance: Status, threats, and future protection. Int J Environ Res Public Health. 16 (10), 1818 (2019).
- Corn, M. L. . Deepwater Horizon oil spill: coastal wetland and wildlife impacts and response. , (2010).
- Hendriks, I. E., Sintes, T., Bouma, T. J., Duarte, C. M. Experimental assessment and modeling evaluation of the effects of the seagrass Posidonia oceanica on flow and particle trapping. Marine Ecology Progress Series. 356, 163-173 (2008).
- Krause, S. J. E., Treude, T. Deciphering cryptic methane cycling: Coupling of methylotrophic methanogenesis and anaerobic oxidation of methane in hypersaline coastal wetland sediment. Geochimica et Cosmochimica Acta. 302, 160-174 (2021).
- Reddy, K. R., DeLaune, R. D., Inglett, P. W. . Biogeochemistry of Wetlands: Science and Applications. , (2022).
- La, W., et al. Sulfate concentrations affect sulfate reduction pathways and methane consumption in coastal wetlands. Water Research. 217, 118441 (2022).
- Derwent, R. G. Global warming potential (GWP) for methane: Monte Carlo analysis of the uncertainties in Global Tropospheric Model predictions. Atmosphere. 11 (5), 486 (2020).
- Potter, C., et al. Methane emissions from natural wetlands in the United States: Satellite-derived estimation based on ecosystem carbon cycling. Earth Interactions. 10 (22), 1-12 (2006).
- . Understanding global warming potentials Available from: https://www.epa.gov/ghgemissions/understanding-global-warming-potentials (2024)
- Wallenius, A. J., Dalcin Martins, P., Slomp, C. P., Jetten, M. S. M. Anthropogenic and environmental constraints on the microbial methane cycle in coastal sediments. Front Microbiol. 12, 631621 (2021).
- Qu, Y., et al. Salinity causes differences in stratigraphic methane sources and sinks. Environmental Science and Ecotechnology. 19, 100334 (2024).
- Vizza, C., West, W. E., Jones, S. E., Hart, J. A., Lamberti, G. A. Regulators of coastal wetland methane production and responses to simulated global change. Biogeosciences. 14 (2), 431-446 (2017).
- van Dijk, G., et al. Salinization lowers nutrient availability in formerly brackish freshwater wetlands; unexpected results from a long-term field experiment. Biogeochemistry. 143 (1), 67-83 (2019).
- Aronson, E., Allison, S., Helliker, B. R. Environmental impacts on the diversity of methane-cycling microbes and their resultant function. Front Microbiol. 4, 225 (2013).
- Reeburgh, W. S. Oceanic methane biogeochemistry. Chem Rev. 107 (2), 486-513 (2007).
- Thauer, R. K. Biochemistry of methanogenesis: a tribute to Marjory Stephenson. Marjory Stephenson Prize Lecture. Microbiology (Reading). 144 (Pt 9), 2377-2406 (1998).
- Thauer, R. K. Anaerobic oxidation of methane with sulfate: on the reversibility of the reactions that are catalyzed by enzymes also involved in methanogenesis from CO2. Curr Opin Microbiol. 14 (3), 292-299 (2011).
- Friedrich, M. W. Methyl-coenzyme M reductase genes: Unique functional markers for methanogenic and anaerobic methane-oxidizing Archaea. Methods in Enzymology. 397, 428-442 (2005).
- Reeburgh, W. S. Oceanic methane biogeochemistry. Chem Rev. 107 (2), 486-513 (2007).
- Rasigraf, O., Schmitt, J., Jetten, M. S. M., Lüke, C. Metagenomic potential for and diversity of N-cycle driving microorganisms in the Bothnian Sea sediment. Microbiologyopen. 6 (4), e00475 (2017).
- McDonald, I. R., Bodrossy, L., Chen, Y., Murrell, J. C. Molecular ecology techniques for the study of aerobic methanotrophs. Appl Environ Microbiol. 74 (5), 1305-1315 (2008).
- Tchawa Yimga, M., Dunfield, P. F., Ricke, P., Heyer, J., Liesack, W. Wide distribution of a novel pmoA-like gene copy among type II methanotrophs, and its expression in Methylocystis strain SC2. Appl Environ Microbiol. 69 (9), 5593-5602 (2003).
- Knief, C. Diversity and habitat preferences of cultivated and uncultivated aerobic methanotrophic bacteria evaluated based on pmoA as molecular marker. Front Microbiol. 6, 1346 (2015).
- Huang, I. -. S., et al. Preliminary assessment of microbial community structure of Wind-Tidal Flats in the Laguna Madre, Texas, USA. Biology. 9 (8), 183 (2020).
- Wilding, T. K., Brown, E., Collier, K. J. Identifying dissolved oxygen variability and stress in tidal freshwater streams of northern New Zealand. Environ Monit Assess. 184 (10), 6045-6060 (2012).
- Roy Chowdhury, T., Mitsch, W. J., Dick, R. P. Seasonal methanotrophy across a hydrological gradient in a freshwater wetland. Ecological Engineering. 72, 116-124 (2014).
- Sun, Q. -. Q., et al. Carbon dioxide and methane fluxes: Seasonal dynamics from inland riparian ecosystems, northeast China. Sci Total Environ. 465, 48-55 (2013).
- Lee, S., et al. Comparison and selection of conventional PCR primer sets for studies associated with nitrogen cycle microorganisms in surface soil. Appl Sci. 12 (20), 10314 (2022).
- Bae, K. -. S., et al. Development of diagnostic systems for wide range and highly sensitive detection of two waterborne hepatitis viruses from groundwater using the conventional reverse transcription nested PCR assay. J Virol Methods. 299, 114344 (2022).
- Lecusay, D. . Assessment and Monitoring of Deltaic Wetlands and Fluvial Systems: Refining and Validating a Multimetric Index of Resaca Ecosystem Health. , (2021).
- . FastDNA SPIN Kit for Soil (Cat No. 116560200) Available from: https://www.mpbio.com/media/productattachment/LS082019-EN-FastDNA-SPIN-Kit-for-Soil-116560200-Manual.pdf (2025)
- Luton, P. E., Wayne, J. M., Sharp, R. J., Riley, P. W. The mcrA gene as an alternative to 16S rRNA in the phylogenetic analysis of methanogen populations in landfill. Microbiology (Reading, England). 148 (Pt 11), 3521-3530 (2002).
- Changsoo, L., Jaai, K., Seung Gu, S., Seokhwan, H. Absolute and relative QPCR quantification of plasmid copy number in Escherichia coli. J Biotechnol. 123 (3), 273-280 (2006).
- Harms, G., et al. Real-time PCR quantification of nitrifying bacteria in a municipal wastewater treatment plant. Environ Sci Technol. 37 (2), 343-351 (2003).
- Holmes, A. J., Costello, A., Lidstrom, M. E., Murrell, J. C. Evidence that particulate methane monooxygenase and ammonia monooxygenase may be evolutionarily related. FEMS Microbiol Lett. 132 (3), 203-208 (1995).
- Fuse, H., et al. Oxidation of trichloroethylene and dimethyl sulfide by a marine Methylomicrobium strain containing soluble methane monooxygenase. Biosci Biotechnol Biochem. 62 (10), 1925-1931 (1998).
- Springer, E., Sachs, M. S., Woese, C. R., Boone, D. R. Partial gene sequences for the A subunit of methyl-coenzyme M reductase (mcrI) as a phylogenetic tool for the family Methanosarcinaceae. Int J Syst Bacteriol. 45 (3), 554-559 (1995).
- Costello, A. M., Lidstrom, M. E. Molecular characterization of functional and phylogenetic genes from natural populations of methanotrophs in lake sediments. Appl Environ Microbiol. 65 (11), 5066-5074 (1999).
- Flores, E. A. . Effects of Nutrient Enrichment on Mangrove and Saltmarsh Habitats. , (2022).
- Minjie, H., Jordi, S., Xianyu, Y., Josep, P., Chuan, T. Patterns and environmental drivers of greenhouse gas fluxes in the coastal wetlands of China: A systematic review and synthesis. Environ Res. 186, 109576 (2020).
- Bridgham, S. D., Cadillo-Quiroz, H., Keller, J. K., Zhuang, Q. Methane emissions from wetlands: biogeochemical, microbial, and modeling perspectives from local to global scales. Glob Chang Biol. 19 (5), 1325-1346 (2013).
- Liu, D. Y., Ding, W. X., Jia, Z. J., Cai, Z. C. Relation between methanogenic archaea and methane production potential in selected natural wetland ecosystems across China. Biogeosciences. 8 (2), 329-338 (2011).
- Ke, Z., et al. Methane emissions and methanogenic community investigation from constructed wetlands in Chengdu City. Urban Climate. 39, 100956 (2021).
- Chowdhury, T. R., Dick, R. P. Ecology of aerobic methanotrophs in controlling methane fluxes from wetlands. Applied Soil Ecology. 65, 8-22 (2013).
- Maja, S., et al. Humic substances cause fluorescence inhibition in real-time polymerase chain reaction. Anal Biochem. 487, 30-37 (2015).
- Sanches, T. M., Schreier, A. D. Optimizing an eDNA protocol for estuarine environments: Balancing sensitivity, cost and time. PLOS ONE. 15 (5), e0233522 (2020).
- Xia, Z., et al. Conventional versus real-time quantitative PCR for rare species detection. Ecol Evol. 8 (23), 11799-11807 (2018).
- Bourne, D. G., McDonald, I. R., Murrell, J. C. Comparison of pmoA PCR primer sets as tools for investigating methanotroph diversity in three Danish soils. Appl Environ Microbiol. 67 (9), 3802-3809 (2001).
- Juottonen, H., Galand, P. E., Yrjala, K. Detection of methanogenic Archaea in peat: comparison of PCR primers targeting the mcrA gene. Res Microbiol. 157 (10), 914-921 (2006).
- Lueders, T., Friedrich, M. W. Evaluation of PCR amplification bias by terminal restriction fragment length polymorphism analysis of small-subunit rRNA and mcrA genes by using defined template mixtures of methanogenic pure cultures and soil DNA extracts. Appl Environ Microbiol. 69 (1), 320-326 (2003).
- Vaksmaa, A., Jetten, M. S., Ettwig, K. F., Luke, C. McrA primers for the detection and quantification of the anaerobic archaeal methanotroph 'Candidatus Methanoperedens nitroreducens'. Appl Microbiol Biotechnol. 101 (4), 1631-1641 (2017).
- Ren, G., et al. Electron acceptors for anaerobic oxidation of methane drive microbial community structure and diversity in mud volcanoes. Environ Microbiol. 20 (7), 2370-2385 (2018).
- Goldberg, C. S., et al. Critical considerations for the application of environmental DNA methods to detect aquatic species. Methods in Ecology and Evolution. 7 (11), 1299-1307 (2016).
- McKee, A. M., Spear, S. F., Pierson, T. W. The effect of dilution and the use of a post-extraction nucleic acid purification column on the accuracy, precision, and inhibition of environmental DNA samples. Biological Conservation. 183, 70-76 (2015).
- Gohl, D. M., et al. Systematic improvement of amplicon marker gene methods for increased accuracy in microbiome studies. Nat Biotechnol. 34 (9), 942-949 (2016).
- Ballarini, A., Segata, N., Huttenhower, C., Jousson, O. Simultaneous quantification of multiple bacteria by the BactoChip microarray designed to target species-specific marker genes. PLOS ONE. 8 (2), e55764 (2013).
- Le Mer, J., Roger, P. Production, oxidation, emission and consumption of methane by soils: A review. Eur J Soil Biol. 37 (1), 25-50 (2001).
- Smith, C. J., Osborn, A. M. Advantages and limitations of quantitative PCR (Q-PCR)-based approaches in microbial ecology. FEMS Microbiol Ecol. 67 (1), 6-20 (2009).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten