Method Article
Visualisation de la dynamique microbienne du cycle du méthane dans les zones humides côtières
Dans cet article
Résumé
Le protocole détecte les principaux gènes du cycle du méthane dans les zones humides côtières du sud du Texas et visualise leur distribution spatiale afin d’améliorer la compréhension de la régulation du méthane et de ses impacts environnementaux dans ces écosystèmes dynamiques.
Résumé
Les zones humides côtières sont la plus grande source biotique de méthane, où les méthanogènes convertissent la matière organique en méthane et les méthanotrophes oxydent le méthane, jouant ainsi un rôle essentiel dans la régulation du cycle du méthane. Les zones humides du sud du Texas, qui sont soumises à des événements météorologiques fréquents, à des niveaux de salinité fluctuants et à des activités anthropiques dues au changement climatique, influencent le cycle du méthane. Malgré l’importance écologique de ces processus, le cycle du méthane dans les zones humides côtières du sud du Texas reste insuffisamment exploré. Pour combler cette lacune, nous avons développé et optimisé une méthode de détection des gènes liés aux méthanogènes et aux méthanotrophes, y compris mcrA comme biomarqueur pour les méthanogènes et pmoA1, pmoA2 et mmoX comme biomarqueurs pour les méthanotrophes. De plus, cette étude visait à visualiser les modèles de distribution spatiale et temporelle de l’abondance des méthanogènes et des méthanotrophes à l’aide du logiciel de système d’information géographique (SIG) ArcGIS Pro. L’intégration de ces techniques moléculaires à la visualisation géospatiale avancée a fourni des informations essentielles sur la distribution spatiale et temporelle des communautés de méthanogènes et de méthanotrophes dans les zones humides du sud du Texas. Ainsi, la méthodologie établie dans cette étude offre un cadre solide pour cartographier la dynamique microbienne dans les zones humides, améliorer notre compréhension du cycle du méthane dans des conditions environnementales variables et soutenir des études plus larges sur les changements écologiques et environnementaux.
Introduction
Les zones humides côtières sont des écosystèmes vitaux qui contribuent à la régulation du climat, à la conservation de la biodiversité et à la gestion de l’eau par des processus tels que la séquestration du carbone, l’évapotranspiration et les émissions de méthane (CH4)1. Ces écosystèmes, qui comprennent les zones humides d’eau douce et d’eau salée2, sont très productifs et constituent des zones critiques pour l’absorption de dioxyde de carbone (CO2) et la capture de la matière organique dans les environnements terrestres et marins 3,4. Les interactions dynamiques au sein de ces zones humides stimulent la production et la consommation microbiennes de CH4 5, ce qui les positionne comme l’une des plus grandes sources naturelles de CH46. En tant que deuxième gaz à effet de serre le plus important, le CH4 a un potentiel de réchauffement planétaire environ 27 à 30 fois supérieur à celui du CO2 4,7,8,9, ce qui rend l’étude des émissions de CH 4 des zones humides côtières essentielle à l’ère du changement climatique. L’émission de CH4 est influencée par divers facteurs environnementaux, en particulier la salinité, qui joue un rôle crucial dans les processus microbiens10. Les zones humides d’eau douce contribuent de manière significative au méthane atmosphérique en raison de leurs faibles niveaux de sulfate, ce qui facilite une plus grande production microbienne de CH4, tandis que les zones humides d’eau salée ont généralement tendance à émettre moins de CH4 en raison de concentrations plus élevées de sulfates 11,12,13.
Les émissions de CH4 provenant des zones humides côtières sont généralement contrôlées par deux groupes de micro-organismes, connus sous le nom de méthanogènes et de méthanotrophes14. Les méthanogènes produisent du CH4 dans les sédiments anoxiques en décomposant des substrats tels que le formiate, l’acétate, l’hydrogène ou les composés méthylés par un processus connu sous le nom de méthanogenèse15. L’enzyme importante de cette voie est la méthyl-coenzyme M réductase (MCR), car elle catalyse l’étape finale et limitante de la méthanogenèse 15,16,17. Le gène mcrA, qui code pour la sous-unité alpha de MCR, est un marqueur fonctionnel que l’on retrouve chez toutes les archées méthanogènes18. De plus, dans les zones humides côtières, la zone de transition sulfate-méthane (SMTZ) se forme au-dessus de la zone méthanogène, où le méthane se diffusant vers le haut et le sulfate se déplaçant vers le bas convergent et s’appauvrissent19. Dans cette zone, les archées méthanotrophes anaérobies (ANME) oxydent le méthane en dioxyde de carbone à l’aide de l’enzyme MCR, tandis que les bactéries sulfato-réductrices (SRB) réduisent le sulfate en sulfure. Les SRB surpassent les méthanogènes pour l’hydrogène et l’acétate, limitant la production de méthane jusqu’à ce que le sulfate soit épuisé16,17.
En revanche, les bactéries méthanotrophes aérobies oxydent le CH4 dans les environnements aérobies20, en utilisant différentes formes de méthane monooxygénase (MMO). Il s’agit notamment de la méthane monooxygénase particulaire (pMMO), une enzyme contenant du cuivre intégrée dans la membrane intracytoplasmique, et de la méthane monooxygénase soluble (sMMO), une enzyme contenant du fer présente dans le cytoplasme. Cependant, pour le pMMO, il existe trois opérateurs de gènes pmoCAB21 ; parmi eux, le gène pmoA est le plus conservateur pour tous les méthanotrophes. Il existe deux gènes biomarqueurs différents pour le pmoA : pmoA1 et pmoA222. De plus, pour une compréhension complète des méthanotrophes, le gène mmoX est utilisé comme outil en biologie moléculaire pour identifier les méthanotrophes23 contenant des sMMO. Cette distinction dans les voies métaboliques et les exigences environnementales des méthanogènes et des méthanotrophes aérobies met en évidence les interactions microbiennes complexes qui régulent le cycle du méthane dans les écosystèmes des zones humides côtières.
La zone humide de Boca Chica (C.-B.), un environnement d’eau salée productif dans le sud du Texas, subit des influences de marée du golfe du Mexique (GOM), entraînant des niveaux de salinité de surface variables, notamment en raison de sa proximité avec l’hypersaline Laguna Madre24. Cette action des marées, alternant entre les marées hautes et basses, fait fluctuer les niveaux d’oxygène25 , ce qui pourrait modifier l’activité méthanogène et méthanotrophe dans les sédiments26. En revanche, les zones humides d’eau douce côtières sont considérées comme un point chaud important pour les flux de CH4 27. Les zones humides d’eau douce côtières du sud du Texas, y compris Resaca Del Rancho Viejo (RV) et Lozano Banco (LB), éloignées des effets de marée du GOM, ont une gestion hydrologique distincte. Le VR subit des débits pulsés complétés par de l’eau de rivière pendant les faibles niveaux d’eau, tandis que le LB fonctionne comme un système d’écoulement hors ligne sans une telle supplémentation. De plus, RV et LB maintiennent des niveaux de salinité plus faibles en raison d’un débit élevé d’eau douce pompée artificiellement et du fait qu’il s’agit d’un lac de bras mort, respectivement. Les différents facteurs environnementaux peuvent influencer de manière significative le cycle du méthane dans les zones humides côtières du sud du Texas. Cependant, le cycle du méthane dans les zones humides côtières du sud du Texas reste une zone qui n’a pas encore fait l’objet d’une enquête approfondie.
La réaction en chaîne par polymérase (PCR) et la PCR en temps réel (également appelée PCR quantitative [qPCR]) représentent des techniques fondamentales et largement utilisées pour détecter et quantifier l’abondance relative de gènes spécifiques dans les échantillons environnementaux. Ces techniques amplifient spécifiquement les régions ciblées de l’ADN pour indiquer la présence et la quantité relative de gènes liés au cycle du CH4, fournissant ainsi des indicateurs du cycle potentiel du méthane. Néanmoins, la disponibilité et l’efficacité des ensembles d’amorces PCR peuvent être limitées par divers facteurs inhibiteurs dans l’ADN environnemental extrait, étant influencés par les types d’environnements28,29. Ainsi, cette étude a principalement établi une méthode PCR optimale pour détecter la présence de gènes liés au cycle CH4 dans les zones humides côtières du sud du Texas (Figure 1), puis a visualisé leur abondance relative quantifiée dans ces écosystèmes. Les résultats de cette étude peuvent être appliqués à d’autres régions côtières afin d’améliorer la compréhension du cycle du CH4 et de la dynamique microbienne dans divers écosystèmes côtiers.
Protocole
1. Prélèvement d’échantillons
- Prélever des échantillons de sédiments à l’aide d’un échantillonneur instantané ou d’une pelle.
REMARQUE : Les échantillons ont été prélevés à partir de deux stations de trois milieux humides côtiers distincts pendant les saisons fraîches (octobre-février, la température moyenne est de 20 °C) et chaudes (avril-juin, température moyenne est de 27 °C) de 2023 et 2024. Un échantillonneur instantané de sédiments a été utilisé lors du prélèvement d’échantillons dans les milieux humides d’eau douce côtiers (figure 2) et une pelle a été utilisée pour les milieux humides côtiers d’eau salée influencés par les marées. - Descendez d’abord l’échantillonneur dans le plan d’eau peu profond et laissez-le couler à la surface des sédiments (dans les 50 premiers cm) sous son propre poids, réduisant ainsi au minimum la perturbation de la structure sédimentaire, comme le montre la figure 2.
- Retirez-le de la colonne d’eau où la profondeur était généralement de 60 cm à 215 cm30, transférez les échantillons de sédiments dans des sacs à fermeture éclair et rangez-les immédiatement dans une glacière.
REMARQUE : Nettoyez et lavez l’échantillonneur instantané avec de l’eau déminéralisée (DI) avant de passer aux stations suivantes. - Conservez immédiatement tous les échantillons à -20 °C dans le laboratoire.
- Dans chaque site d’échantillonnage, mesurez les paramètres de qualité de l’eau de surface tels que la salinité et la température in situ à l’aide d’un indicateur de qualité de l’eau multiparamètre.
REMARQUE : Les sondes ont été rincées à l’eau déminéralisée (eau DI) après utilisation dans chaque station.
2. Extraction de l’ADN génomique
- Décongelez les échantillons à température ambiante avant de commencer la procédure d’extraction de l’ADN génomique.
- Transvaser environ 500 mg d’échantillons de sédiments dans un tube de 15 mL et centrifuger à 4 250 × g pendant 3 minutes pour éliminer toute l’eau.
- Extrayez l’ADN génomique à l’aide d’un kit d’extraction d’ADN pour le sol en suivant le protocole31 du fabricant avec une petite modification et stockez-le immédiatement à -20 °C.
REMARQUE : Les modifications ont été apportées pour réduire la redondance et améliorer l’efficacité du flux de travail.- Ajouter jusqu’à 500 mg d’échantillon de sol dans un tube contenant des billes de verre ou des sphères de céramique.
- Ajouter 978 μL de tampon de phosphate de sodium à l’échantillon dans le tube contenant une bille ou une sphère.
- Ajouter 122 μL de solution de lyse tampon à l’échantillon dans le tube contenant des billes ou des sphères pour solubiliser les contaminants externes.
- Homogénéiser à l’aide d’un homogénéisateur de broyeur à billes à 5 fois le niveau de vitesse pendant 20 s et répéter 2 fois.
REMARQUE : Un homogénéisateur de broyeur à billes a été utilisé dans cette étude, c’est pourquoi la vitesse a été ajustée. - Centrifuger le mélange pendant 10 min à 14 000 × g.
- Transférez le surnageant dans un tube de microcentrifugation propre de 2,0 ml.
- Ajouter 250 μL de solution de précipitation des protéines (PPS) pour séparer les acides nucléiques solubilisés des débris cellulaires et de la matrice de lyse. Mélanger en inversant le tube 10x.
- Centrifuger à 14 000 × g pendant 5 min pour précipiter la pastille, éliminer les débris cellulaires et lyser la matrice.
- Transférez le surnageant dans un tube de microcentrifugation propre de 15 ml.
- Ajouter 1,0 mL de suspension Binding Matrix au surnageant dans le tube de 15 mL.
REMARQUE : Secouez la suspension de la matrice de liaison pour la remettre en suspension avant de l’ajouter. - Permettez la liaison de l’ADN à la matrice de liaison en plaçant les tubes sur un rotateur pendant 2 min.
- Placez tous les tubes sur une grille et incubez pendant 3 min pour permettre la stabilisation de la matrice de liaison.
- Après 3 min, jeter 750 μL et mélanger délicatement le surnageant restant avec la pastille à l’aide d’une pipette.
- Transvaser 750 μL du mélange dans un filtre SPIN et centrifuger à 14 000 × g pendant 1 min. Videz le tube de récupération et réutilisez-le. Répétez l’opération avec le reste du mélange.
- Ajouter 500 μL de la solution de lavage préparée (avec la quantité appropriée d’éthanol ajoutée) pour solubiliser davantage les impuretés. Remettez doucement la pastille en suspension en utilisant la force du liquide de la pointe de la pipette.
- Centrifuger à 14 000 × g pendant 1 min pour éliminer les impuretés. Videz le tube de récupération et réutilisez-le.
- Centrifuger à nouveau à 14 000 × g pendant 2 min sans rien ajouter.
- Remplacez le tube de récupération par un tube de récupération neuf et propre et faites sécher à l’air libre le filtre SPIN pendant 5 minutes à température ambiante.
- Ajouter 50 μL d’eau libre d’ADN (DES) et centrifuger à 14 000 × g pendant 1 min.
REMARQUE : Dans ce protocole, 50 μL de DES ont été utilisés pour l’élution de l’ADN afin d’assurer une récupération et une stabilité optimales de l’ADN environnemental extrait (ADNe).
3. Quantification de l’ADN
- Ajouter 1 μL d’ADN extrait avec 200 μL de colorant fluorescent dans un tube de 0,5 mL et bien mélanger par pipetage.
- Enveloppez immédiatement le tube avec du papier d’aluminium pour que la lumière ne puisse pas pénétrer et incubez à température ambiante pendant 5 min.
- Mesurez la concentration d’ADN à l’aide d’un fluorimètre dans UN mode de concentration d’ADN en suivant le protocole du fabricant.
4. Détection de l’ARNr 16S, du pmoA1 , du pmoA2 , du mmoX et du mcrA par PCR conventionnelle
- Avant d’effectuer une PCR conventionnelle (cPCR), décongelez tous les échantillons et réactifs dans un seau à glace.
- Diluer tous les échantillons d’ADNe extraits à 10 ng/μL.
REMARQUE : Une liste d’amorces est donnée dans le tableau 1. - Préparez un mélange réactionnel cPCR de 25 μL pour chaque échantillon, y compris 12,5 μL de 2 mélanges maîtres PCR, 0,5 μL d’amorces directes et inverses (10 μM) (voir le tableau 1 pour la liste des amorces), 1 μL d’ADNe de 10 ng/μL et 10,5 μL d’eau exempte de nucléases.
REMARQUE : Préparez le mélange principal avec un volume suffisant pour un échantillon supplémentaire afin de minimiser les erreurs de pipetage. - Effectuer une réaction cPCR selon le protocole consistant en une dénaturation initiale à 95 °C pendant 2 min, suivie de 40 cycles de dénaturation à 95 °C pendant 45 s, d’extension à 72 °C pendant 30 s et d’extension finale à 72 °C pendant 5 m, avec une température de recuit variable pour différentes amorces (voir le tableau 1 pour les températures de recuit des différentes amorces utilisées pour différents gènes).
REMARQUE : Pour le gène mcrA , les amorces ML ont montré la bande souhaitée en utilisant un taux de rampe lent de 0,1 °C/s entre les étapes de recuit et d’extension pour les cinq premiers cycles32. - Visualisez les produits de cPCR dans un gel d’agarose précoloré au bromure d’éthidium (EtBr).
REMARQUE : Utilisez du gel d’agarose à 2,5 % pour une échelle de 50 pb et du gel à 0,9 % pour une échelle de 1 kb. Utilisez 1 tampon TAE pour un gel d’agarose à 2,5 % et un tampon TAE 0,5 pour un gel d’agarose à 0,9 %.
5. Détection de pmoA1, pmoA2, mmoX et mcrA par PCR quantitative en temps réel
REMARQUE : L’abondance de gènes ciblant les méthanogènes et les méthanotrophes, tels que l’abondance de pmoA1, pmoA2, mmoX et mcrA , a été observée par qPCR à l’aide d’un système de PCR en temps réel.
- Préparez les étalons pour chaque gène séparément afin d’obtenir la courbe standard pour chaque gène.
- Amplifiez le gène ciblé avec la cPCR, en utilisant les échantillons qui ont produit la bande la plus brillante lors de l’électrophorèse sur gel de chaque gène. Suivez la méthode et les amorces décrites à la rubrique 4.
- Purifier les produits amplifiés à l’aide d’un kit d’extraction de gel et mesurer la concentration d’ADN selon la méthode décrite au paragraphe 3 et les conserver à -20 °C.
REMARQUE : Aliquote les étalons purifiés dans des tubes séparés pour une utilisation ultérieure. - Calculez les copies de gènes à partir de la concentration d’ADN mesurée à l’aide du site Web de calcul du nombre de copies pour la qPCR.
- Diluer le nombre de copies calculé de l’étalon avec de l’eau exempte de nucléases pour préparer chaque étalon, allant de 108 à 102 copies/μL, avant d’effectuer la qPCR.
- Préparez la courbe standard à l’aide de trois répétitions de chaque numéro de copie, y compris un témoin négatif (NTC, pas d’ADN matrice).
REMARQUE : La valeur R2 de chaque courbe standard était supérieure à 0,99.
- Préparez un mélange réactionnel qPCR de 20 μL pour tous les échantillons, les étalons et le NTC. Effectuez l’analyse qPCR en trois exemplaires pour tous les échantillons.
- Placez tous les composants de la réaction, y compris le mélange maître qPCR, les amorces, l’eau sans nucléases, les étalons et les échantillons, sur un support à glace avant de commencer.
- Préparez un mélange réactionnel qPCR de 20 μL pour chaque échantillon, étalon et NTC, contenant 10 μL de mélange maître SYBR Green, 0,5 μL de chaque amorce directe et inverse de 10 μM, 8 μL d’eau exempte de nucléases et 1 μL d’ADN matrice de 10 ng/μL, d’eau étalon ou d’eau DI respectivement.
REMARQUE : Utilisez les ensembles d’amorces qui ont montré qu’ils donnaient des résultats optimaux pour les gènes pmoA1 et mcrA dans la PCR conventionnelle pour la qPCR (voir Tableau 1). Pour améliorer la précision, analysez chaque échantillon en trois exemplaires. Les étapes suivantes constituent une méthode efficace pour préparer des échantillons triples d’un volume combiné de 60 μL par échantillon.- Préparez le mélange réactionnel pour combiner le mélange maître, les amorces avant et arrière pour le gène spécifique et de l’eau sans nucléases dans un tube de 2 ml, à l’exception de l’ADN matrice.
REMARQUE : Préparez le volume du mélange réactionnel en tenant compte de l’erreur de pipetage. Par exemple, s’il y a 24 échantillons et 8 étalons, calculez le volume total pour 33 réactions au lieu de 32 réactions afin de minimiser les erreurs de pipetage. Dans ce cas, le volume total requis pour les réactions triples serait le suivant : 990 μL de mélange maître (33 échantillons x 3 répétitions x 10 μL), 49,5 μL d’amorces directes (33 échantillons x 3 répétitions x 0,5 μL), 49,5 μL d’amorces inverses (33 échantillons x 3 répétitions x 0,5 μL) et 792 μL d’eau exempte de nucléases (33 échantillons x 3 répétitions x 8 μL). - Préparer les tubes PCR en fonction du nombre d’échantillons et d’étalons.
- Versez 57 μL du mélange réactionnel préparé dans chaque tube PCR.
REMARQUE : Pour effectuer une qPCR pour chaque échantillon en trois exemplaires, préparez un volume total de mélange réactionnel de 57 μL par échantillon (à l’exclusion de l’ADN matrice, de l’étalon ou de l’eau). Ce volume sera divisé à parts égales en trois puits pour un échantillon, avec 19 μL alloués à chaque puits. - Ajoutez 3 μL d’ADN matrice, d’eau standard ou d’eau sans nucléases dans chaque tube et mélangez en tapotant doucement le fond du tube.
REMARQUE : Le volume total du mélange réactionnel préparé pour un échantillon serait maintenant de 60 μL dans chaque tube.
- Préparez le mélange réactionnel pour combiner le mélange maître, les amorces avant et arrière pour le gène spécifique et de l’eau sans nucléases dans un tube de 2 ml, à l’exception de l’ADN matrice.
- Aliquote 20 μL du mélange réactionnel préparé de chaque tube dans les puits désignés d’une plaque qPCR à 96 puits. Scellez la plaque PCR avec un film d’étanchéité PCR adhésif à l’aide d’un applicateur.
- Centrifugez la plaque scellée à 1 000 × g pendant 1 min pour assurer un bon mélange afin d’éliminer toute bulle dans les puits.
- Placez la plaque PCR dans le thermocycleur. Allumez la machine qPCR, puis ouvrez le logiciel associé pour configurer le protocole.
- Configurez le protocole conformément aux directives du master mix qPCR. Utilisez le protocole suivant : 95 °C pendant 10 min, suivi de 95 °C pendant 15 s et d’un pas d’extension à 72 °C pendant 30 s. Effectuez l’étape de recuit à la température de recuit spécifiée pour les apprêts correspondants dans le tableau 1 pendant 45 s. Effectuez toutes les exécutions qPCR pendant 35 cycles.
- Configurez la plaque qPCR à 96 puits avec des étalons et des NTC dans la même configuration que la plaque contenant l’échantillon.
- Utilisez la méthode de la courbe standard de quantification absolue pour quantifier le produit amplifié et le nombre de copies de gènes dans chaque échantillon33.
6. Visualisation des gènes du cycle du méthane sur la carte des zones humides côtières du sud du Texas
- Ouvrez le logiciel de système d’information géographique (SIG) ArcGIS Pro et enregistrez le fichier de projet sous le nom de Zone d’étude dans le dossier spécifié sur l’ordinateur.
- Cliquez sur la carte en haut à gauche | Fond de carte et sélectionnez Terrain avec étiquettes comme fond de carte.
- Cliquez sur Localiser | Recherchez et lorsque la barre de recherche s’ouvre, localisez la zone d’étude en tapant le nom de la zone ; La zone apparaîtra.
- Dessinez la zone spécifique à l’aide du géoréférencement.
- Cliquez sur Voir | Volet Catalogue à partir de la couche supérieure.
- Double-cliquez sur Dossier dans le catalogue | Nom du fichier.
- Faites un clic droit sur le fichier de géodatabase (.gdb), puis cliquez sur Nouveau | Les options Classe d’entités et Créer une classe d’entités s’affichent.
- Tapez la case Nom et Alias et cliquez sur Terminer en bas.
- Cliquez sur Voir | Contenu de la procédure. Le nom de l’alias s’affiche dans le volet Contenu.
- Cliquez sur Modifier à partir du calque supérieur | Créer. La fenêtre Create Features (Créer des entités ) s’ouvre. Double-cliquez sur le volet Alias dans Créer des fonctionnalités et les options de retour d’outil Configurer apparaîtront.
- Sélectionnez Lignes, puis esquissez des lignes sur la carte pour créer une limite extérieure de la zone d’étude. Double-cliquez sur la carte lorsque vous avez terminé.
- Réduisez le logiciel SIG, puis ouvrez une feuille de calcul. Tapez le nom de l’échantillon dans la première colonne, entrez la latitude dans la deuxième colonne et la longitude dans la troisième colonne. Utilisez les quatre colonnes suivantes pour les données qPCR de pmoA1, pmoA2, mmoX et mcrA.
- Enregistrez le fichier au format CSV dans n’importe quel dossier spécifique de l’ordinateur.
- Ouvrez à nouveau le logiciel SIG et cliquez sur Ajouter des données | Données ponctuelles XY.
- Sélectionnez le fichier CSV dans le dossier de l’ordinateur dans la zone Table d’entrée . Renommez le nom du fichier dans la classe d’entités en sortie, puis cliquez sur Exécuter pour afficher les points d’échantillonnage sur la carte.
- Cliquez sur la barre de recherche virtuelle en haut et recherchez Krigeage.
- Sélectionnez le fichier de la station d’échantillonnage, puis pmoA1.
- Cliquez sur l’onglet Environnement | sélectionnez l’abscisse curviligne d’échantillonnage dans Calque et masque | cliquez sur Exécuter.
- Suivez les protocoles mentionnés aux étapes 6.9, 6.10 et 6.11 pour créer du krigeage pour pmoA2, mmoX et mcrA pour toute la zone d’étude.
- Créez une mise en page de la carte.
- Cliquez sur Insérer à partir de la couche supérieure | Nouvelle mise en page, puis sélectionnez ANSI - Paysage.
- Cliquez sur le cadre de la carte, sélectionnez la carte avec le krigeage et placez toutes les cartes dans la mise en page en dessinant un rectangle. Cela rendra la carte visible dans la mise en page.
- Sélectionnez la flèche vers le nord et placez-la dans la mise en page pour indiquer la direction du nord.
- Sélectionnez Barre d’échelle pour afficher l’échelle de la zone sur la carte.
- Cliquez sur Légende pour afficher les légendes, puis placez-la dans la mise en page.
- Cliquez sur Grille et sélectionnez l’une des options de réticule noir. Cela créera la grille avec la latitude et la longitude et l’affichera dans le volet de contenu avec l’étiquette Noir Horizontal Étiquette Graticule.
- Double-cliquez sur Étiquette horizontale noire Graticule, puis sélectionnez Composants. Cliquez sur les coches 1 et la grille, puis supprimez ces composants en cliquant sur le signe de croix à leur droite.
- Cliquez sur Partager dans le calque supérieur, puis cliquez sur Exporter la mise en page. Sélectionnez le type de fichier PDF, enregistrez le fichier sur l’ordinateur à l’aide de la zone Nom, définissez la résolution verticale sur 500 DPI et cliquez sur Exporter pour créer le fichier PDF de la carte.
Résultats
Pour comprendre la distribution et l’abondance des gènes liés au cycle CH4 (mcrA, pmoA1, pmoA2 et mmoX) dans les zones humides côtières du sud du Texas, l’ADNe extrait de chaque échantillon a été analysé par cPCR et qPCR. Des amorces universelles pour chaque biomarqueur ont été sélectionnées pour exécuter la cPCR à partir d’études antérieures (Tableau 1)22,34,35,36,37, et des modifications ont été apportées pour optimiser les températures et les concentrations de recuit en fonction des caractéristiques de l’échantillon et des conditions environnementales. Par exemple, l’ARNr 16S, le pmoA1 et le pmoA2 n’ont pas été amplifiés à l’aide de la température de recuit standard, car la plupart d’entre eux se sont amplifiés bien en dessous de la température de recuit standard (données non présentées). De plus, au cours du processus de cPCR, une concentration de 10 ng/μL d’ADN a montré une bande distincte pour les gènes pmoA1, pmoA2 et mmoX, tandis que l’utilisation de 5 ng/μL d’ADN matrice a montré une bande plus brillante pour le gène mcrA (Fichier supplémentaire 1-Figure supplémentaire S1).
L’analyse cPCR a révélé une variabilité spatiale dans la détection des gènes dans trois types de zones humides côtières du sud du Texas. Comme le montre le tableau 2, les gènes mcrA ont été détectés dans tous les échantillons de LB et de RV, qui sont des zones humides d’eau douce côtières. Cependant, la cPCR n’a pas permis de détecter de gène mcrA dans les sédiments échantillonnés de la zone humide d’eau salée, ce qui suggère un lien potentiel entre la salinité et la distribution des méthanogènes (Fichier supplémentaire 1-Figure supplémentaire S2). Pour la détection de mcrA, quatre paires d’amorces ont été utilisées dans cette étude, soit MLF-MLR, MCRf-MCRr, ME1F-ME2R et McrA 159F-McrA 345R (tableau 1) ; parmi eux, seules les paires d’amorces ML ont pu détecter la communauté de méthanogènes dans les zones humides du sud du Texas (Fichier supplémentaire 1-Figure supplémentaire S2, Figure supplémentaire S3 et Figure supplémentaire S4). En revanche, pour les méthanotrophes aérobies, le gène pmoA1 a été détecté dans tous les échantillons de VD, mais était absent dans les échantillons LB et BC (Fichier supplémentaire 1-Figure supplémentaire S5). Pour identifier pmoA1, A189 a été utilisé comme amorce directe et A682 et mb661 comme amorces inverses, où les paires d’amorces A189-A682 peuvent également détecter amoA35,38. Cependant, cette paire d’amorces a pu détecter la présence de gènes dans la cPCR dans cette étude où les images de gel ont montré une bande très faible, suggérant la faible abondance du gène pmoA1. Une autre amorce inverse, mb661, a été conçue pour identifier le gène pmoA1 spécifiquement avec A189 comme amorce directe38. Lorsque mb661 a été utilisé comme amorce inverse, la cPCR a montré une bande plus brillante (Fichier supplémentaire 1-Figure supplémentaire S6), qui a ensuite été utilisée dans la qPCR. Il est intéressant de noter que le gène pmoA2 a été détecté dans deux échantillons de BC et un échantillon de LB, mais qu’il était absent dans un échantillon de RV (Fichier supplémentaire 1-Figure supplémentaire S7). Une étude précédente a révélé que la température de recuit était de 66 °C et 60 °C pour le pmoA2, alors qu’à 60 °C, deux amplicons de tailles différentes, 245 et 438 pb, ont été observés, tandis qu’à 66 °C, les amorces 206f-703b ne peuvent amplifier que le pmoA2 avec un produit de 438 pb de taille22. Cependant, dans cette étude, l’amplification a été réussie à une température de recuit de 60 °C, produisant une bande de la taille attendue (438 pb) dans les sites LB et BC (Dossier supplémentaire 1-Figure supplémentaire S7). De plus, le gène mmoX a été détecté dans un échantillon de LB, mais il était absent dans un échantillon de RV (Fichier supplémentaire 1-Figure supplémentaire S8) et était totalement absent dans les échantillons de la Colombie-Britannique. Les paires d’amorces utilisées pour mmoX convenaient également à l’étude des zones humides du sud du Texas dans cette étude. Une température de recuit de 55 °C36 a produit une bande brillante pour mmoX dans la cPCR (Dossier supplémentaire 1-Figure supplémentaire S8). Pour vérifier l’intégrité de l’échantillon, l’ARNr 16S a été utilisé comme témoin positif avec amplification réussie de l’ARNr 16S dans tous les échantillons pendant la cPCR, ce qui suggère que les échantillons n’étaient pas contaminés (Dossier supplémentaire 1-Figure supplémentaire S9).
Pour évaluer les changements temporels et spatiaux de l’abondance des gènes, une analyse qPCR a été effectuée sur tous les gènes ciblés à l’aide d’ensembles d’amorces optimisés identifiés par cPCR. La qPCR des gènes pmoA2 et mmoX a été réalisée avec les mêmes amorces que celles utilisées dans la cPCR. Cependant, pour le gène pmoA1 , la paire d’amorces A189-mb661 a été utilisée, et pour le gène mcrA , la paire d’amorces MLF-MLR a été sélectionnée, car les deux ont donné de bons résultats en cPCR. Les différences saisonnières et les variations de salinité entre les zones humides du sud du Texas ont également été prises en compte, car elles pourraient influencer la dynamique des communautés microbiennes. Comme le montrent les figures 3 et 4, il y avait une différence dans l’abondance des gènes des méthanogènes et des méthanotrophes entre deux périodes d’échantillonnage différentes. Bien qu’il n’y ait pas eu de variation saisonnière de la salinité dans les zones humides d’eau douce côtières, une différence saisonnière de salinité a été observée dans les zones humides d’eau salée côtières39, ce qui pourrait expliquer la variation de l’abondance des méthanogènes et des méthanotrophes. La figure 3 montre que pendant la saison fraîche, le mmoX était très abondant dans tous ces milieux humides, avec une abondance maximale de 1,46 × 104 copies/μL. Il y avait un faible pourcentage de pmoA2 dans une station de LB, RV et BC. Cependant, la qPCR n’a pas pu détecter pmoA1 et mcrA en raison de leur faible abondance pendant la saison fraîche. En revanche, le pmoA1 était surtout abondant pendant la saison chaude, avec une abondance maximale de 2,44 × 103 copies/μL (figure 4). Les mmoX et mcrA ont également été détectés dans les échantillons de sédiments de surface de LB (dans les 50 premiers cm), montrant une abondance maximale de 1,92 × 102 copies/μL et de 2,04 × 102 2 copies/μL, respectivement, malgré leur abondance relativement faible pendant la saison chaude. Il est intéressant de noter que pendant la saison chaude, le pmoA2 n’a pas été détecté du tout, et qu’aucun des gènes cibles n’a été détecté dans les échantillons de la Colombie-Britannique, probablement influencés par des facteurs environnementaux propres à cette zone humide d’eau salée39 pendant la saison chaude dans le sud du Texas.
Cette étude met en évidence des variations spatiales et saisonnières distinctes dans la distribution des gènes liés au cycle CH4 dans les milieux humides côtiers. Plus précisément, les gènes mcrA n’ont été détectés que dans l’échantillon de LB d’eau douce, ce qui suggère une préférence pour l’habitat. Les gènes mmoX et pmoA2 étaient présents dans tous les échantillons pendant les saisons fraîches, tandis que pmoA1 étaient présents dans les environnements d’eau douce pendant les saisons chaudes. Ces résultats mettent en évidence l’influence des conditions environnementales et des changements saisonniers sur les activités microbiennes liées au cycle du CH4 . La visualisation améliorée à l’aide de l’interpolation par krigeage, comme le montrent les figures 3 et 4, a permis de mieux comprendre les activités microbiennes liées au cycle du CH4 dans les écosystèmes des milieux humides côtiers.
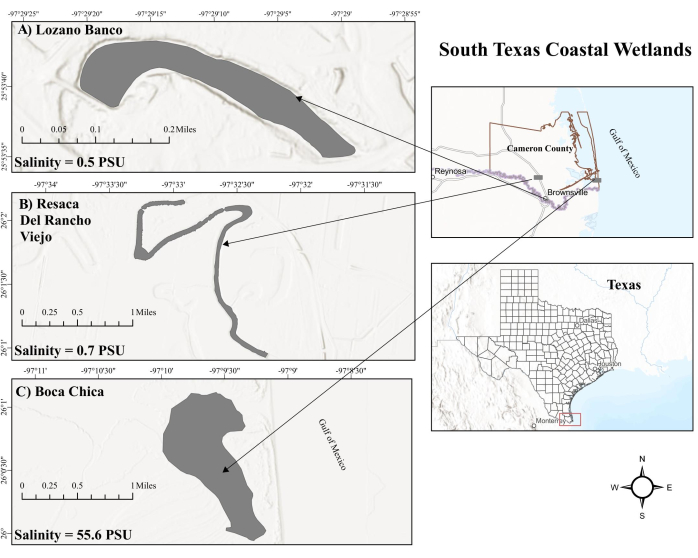
Figure 1 : Carte des zones humides côtières du sud du Texas. a) Lozano Banco. LB est une zone humide côtière d’eau douce. b) Resaca del Rancho Viejo. RV est également une zone humide côtière d’eau douce. (C) Zone humide de Boca Chica. La Colombie-Britannique est une zone humide côtière salée hypersaline influencée par les marées. Abréviations : LB = Lozano Banco ; RV = Resaca Del Rancho Viejo; BC = Boca Chica ; PSU = unités de salinité pratiques. Veuillez cliquer ici pour voir une version agrandie de cette figure.
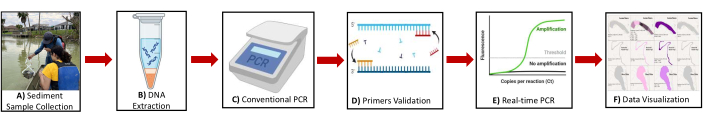
Figure 2 : Schéma de principe montrant la vue d’ensemble du protocole. L’illustration des étapes clés comprend (A) la collecte d’échantillons de sédiments (B) l’extraction de l’ADN (C) la PCR conventionnelle (D) la validation de l’amorce (E) la PCR en temps réel (F) la visualisation des données pour l’identification des gènes biomarqueurs du méthanogène et des méthanotrophes. Veuillez cliquer ici pour voir une version agrandie de cette figure.
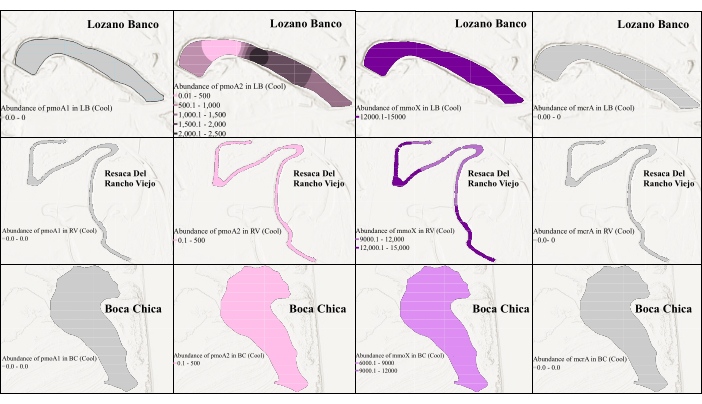
Figure 3 : Distribution spatiale des gènes biomarqueurs du méthanogène et des méthanotrophes en saison fraîche. La figure montre l’abondance de mmoX pendant la saison fraîche. Ici, la couleur grise indique que la qPCR n’a pas pu détecter le gène en raison de sa faible abondance. Veuillez cliquer ici pour voir une version agrandie de cette figure.

Figure 4 : Distribution spatiale des gènes biomarqueurs du méthanogène et des méthanotrophes pendant la saison chaude. Ici, la couleur grise représente que la qPCR n’a pas pu détecter le gène en raison de sa faible abondance. La figure montre l’abondance de pmoA1 pendant la saison chaude à Lozano Banco et Resaca Del Rancho Viejo, alors qu’il n’y a pas eu d’abondance détectée à Boca Chica. Veuillez cliquer ici pour voir une version agrandie de cette figure.
| Nom de l’abécédaire | Gène | Température de recuit (°C) dans cette étude | Longueur du produit (bp) | Séquence (5'-3') | Référence | |
| 1055F | ARNr 16S | 55 | 337 | ATGGCTGTCGTCAGCT | (Harms et coll., 2003) | |
| 1392R | ACGGGCGGTGTAC | |||||
| A189F | pmoA1 | 55 | 525 | GGNGACTGGGACTTCTGG | (Holmes et al., 1995) | |
| A682R | GAASGCNGAGAAGAASGC | |||||
| A189F | 56 | 470 | GGNGACTGGGACTTCTGG | (Costello et Lidstrom, 1999) | ||
| mb661R | CCG GMG CAA CGT CYT TACC | |||||
| 206f | pmoA2 | 60 | 438 | GGNGACTGGGACTTCTG GATCGACTTCAAGGATCG | (Tchawa Yimga et al., 2003) | |
| 703b | GAASGCNGAGAAGAASGC GGCGACCGGAACGACGT | |||||
| 536F | mmoX | 55 | 362 | CGCTGTGGAAGGGCATGAAGCG | (Fuse et al., 1998) | |
| 898R | GCTCGACCTTGAACTTGGAGCC | |||||
| MCRf | mcrA | 46 | ~490 | TAYGAYCARATHTGGYT | (Springer et al., 1995) | |
| MCRr | ACRTTCATNGCRTARTT | |||||
| ME1F | 50 | 763 | GCMATGCARATHGGWATGTC | (Hales et al., 1996) | ||
| ME2R | TCATKGCRTAGTTDGGRTAGT | |||||
| FML | 55 | ~470 | GGTGGTGTMGGATTCA CACARTAYGCWACAGC | (Luton et al., 2002) | ||
| Taux d’escompte officiel | TTCATTGCRTAGTTWGGRTAGTT | |||||
| McrA 159F | 62 | 186 | AAAGTGCGGAGCAGCAATCACC | (Vaksmaa et al., 2017) | ||
| McrA 345R | TCGTCCCATTCCTGCTGCATTGC | |||||
Tableau 1 : Ensembles d’amorces utilisés dans cette étude pour détecter les gènes fonctionnels liés au cycle du méthane. Toutes ces paires d’amorces ont été utilisées pour la PCR conventionnelle, et enfin, les plus audacieuses ont été mises en place pour détecter les méthanogènes et les gènes liés aux méthanotrophes dans les zones humides côtières du sud du Texas.
| Gène | Échantillons | ||||||||
| Lozano Banco | Resaca Del Rancho Viejo | Boca Chica | |||||||
| LB-1 | RV-1 | RV-2 | M1 | M2 | B1 | B2 | MG1 | MG2 | |
| pmoA1 | - | + | + | - | - | - | - | - | - |
| pmoA2 | + | + | + | - | + | - | - | + | - |
| mmoX | + | - | + | - | - | - | - | - | - |
| mcrA | + | + | + | - | - | - | - | - | - |
Tableau 2 : Détection de gènes dans trois types différents de plans d’eau pendant la saison fraîche à l’aide de la PCR conventionnelle. '+', présent ; '-', absent. Abréviations : LB = Lozano Banco, RV = Resaca del Rancho Viejo, M = Boue, B = Batis, MG = Mangrove.
Fichier supplémentaire 1 : Images d’électrophorèse sur gel pour les gènes mcrA, pmoA1, pmoA2, mmoX et 16S. Veuillez cliquer ici pour télécharger ce fichier.
Discussion
Les zones humides côtières sont reconnues comme contribuant de manière importante au méthane atmosphérique, un important gaz à effet de serre40. Bien qu’il y ait eu des études sur les flux de méthane et les méthanogènes dans les zones humides 41,42,43, on sait peu de choses sur la façon dont les méthanotrophes fonctionnent dans différents environnements ou selon diverses pratiques de gestion, en particulier dans les zones humides avec des niveaux d’eau fluctuants44. De plus, la présence généralisée d’inhibiteurs tels que les acides humiques dans l’ADNe complique les processus d’extraction et d’amplification45, conduisant potentiellement à des résultats faussement négatifs46. Ce défi est encore compliqué par l’efficacité variable des amorces travaillant sur l’ADNe, ce qui suggère la nécessité d’une validation systématique dans de telles études47. Ainsi, cette recherche établit une méthodologie fondamentale pour comprendre le cycle du méthane dans les zones humides côtières du sud du Texas, peu étudiées. En se concentrant sur le développement et la validation de techniques de PCR, cette étude fait progresser la détection des gènes clés du cycle du méthane dans ces écosystèmes dynamiques.
L’un des aspects critiques de cette recherche est la visualisation des gènes liés au cycle microbien du méthane, qui joue un rôle crucial dans l’observation des modèles de distribution spatiale de ces gènes à travers différentes saisons et gradients de salinité. Une telle visualisation n’est pas seulement un outil supplémentaire ; Il est essentiel d’identifier les zones de présence significative de microbes méthanogènes et méthanotrophes, indiquant qu’il pourrait y avoir un cycle potentiel du méthane, améliorant ainsi notre compréhension des conditions environnementales qui influencent ces processus.
Dans cette étude, la méthodologie a été spécifiée pour identifier les gènes marqueurs méthanogènes et méthanotrophes tels que pmoA1, pmoA2, mmoX et mcrA à l’aide de la cPCR et de la qPCR. Quatre paires d’amorces ciblant le gène mcrA et deux paires ciblant le gène pmoA1 ont été utilisées, et une seule paire de chaque ensemble a détecté efficacement ces gènes dans la PCR conventionnelle et en temps réel. Une étude précédente a utilisé trois paires d’amorces pour détecter pmoA1 ; parmi eux, les amorces A189-A682 n’ont pas pu détecter la diversité méthanotrophe. Au lieu de cela, ils ont amplifié principalement le gène amoA et ont produit des produits PCR non spécifiques48. Cette étude a révélé que la paire d’amorces utilisée n’était efficace pour détecter le gène pmoA1 que dans des environnements à forte population de méthanotrophes. De plus, la plus grande abondance de méthanotrophes a été obtenue à l’aide des paires d’amorces A189-mb661 dans cette étude, ce qui indique que ces amorces conviennent aux écosystèmes d’eau douce48. Ainsi, ces paires d’amorces ont été utilisées pour la qPCR comme analyse plus approfondie.
Parmi les quatre paires d’amorces utilisées pour la mcrA, les amorces ML ont pu amplifier les espèces de méthanogène dans tous les environnements49. Les amorces ME ont une plage d’amplification étroite ; Ils ne peuvent pas détecter toutes sortes d’espèces de méthanogènes dans différents types d’environnements naturels. Les amorces MCR ont un niveau élevé de dégénérescence, ce qui les rend sensibles à la température de recuit PCR. Cette dégénérescence leur permet de se lier à un large éventail de séquences cibles, s’accommodant souvent de plusieurs décalages. Cependant, cela indique également qu’un petit changement de température de recuit peut influencer de manière significative leur efficacité de liaison et leur spécificité, conduisant à une amplification insuffisante et, par conséquent, à des résultats faussement négatifs pour l’espèce de méthanogène49,50.
D’autre part, dans la communauté des méthanogènes à faible diversité, au cours des dernières étapes de la PCR, une forte accumulation de produits PCR entraîne un recuit de matrice et empêche les amorces de se lier, ce qui ne permet pas non plus de détecter les bandes souhaitées49. Il est intéressant de noter que les paires d’amorces McrA 159F-McrA 345R ont été spécifiquement conçues pour identifier les Candidatus M. nitroreducens archaea51 méthanotrophes anaérobies dépendants des nitrates, et cette étude n’a pas pu détecter les méthanotrophes anaérobies à l’aide de ces paires d’amorces. Cependant, l’oxydation anaérobie du méthane (AOM) peut être couplée à la réduction d’accepteurs d’électrons alternatifs tels que le fer (Fe), le manganèse (Mn) et le sulfate (SO42-)52, ce qui pourrait expliquer l’incapacité à détecter l’ANME à l’aide de cette méthode. De plus, cela peut être dû aux conditions environnementales spécifiques de la zone d’étude. Ainsi, pour la qPCR, des amorces ML ont été utilisées pour détecter l’abondance du méthanogène, car elles fournissaient une amplification fiable même dans des conditions de faible diversité de communauté49.
De plus, les inhibiteurs de la PCR tels que les acides humiques ou l’ADN des espèces non cibles sont largement présents dans les échantillons environnementaux, ce qui peut réduire l’efficacité de la PCR53. Comme la forte concentration d’ADN ne permettait de détecter aucun gène par cPCR, tous les échantillons d’ADN ont été dilués avant l’exécution de la cPCR afin de réduire l’effet des inhibiteurs de la PCR54. Le gène de l’ARNr 16S, en tant que marqueur universel pour les bactéries et les archées, a fourni une base de référence solide pour valider nos conditions de PCR et confirmer l’intégrité de l’ADN dans tous les échantillons55,56. Cependant, les zones d’eau salée sont reconnues pour avoir des concentrations plus élevées d’inhibiteurs de PCR46. De plus, la salinité influence indirectement les émissions de CH4 dans les zones humides côtières, car elle est associée aux concentrations de sulfates. Une salinité plus élevée dans les zones humides d’eau salée est corrélée à des niveaux élevés de sulfate qui suppriment la production de méthane en raison de la compétition entre les bactéries sulfato-réductrices et les méthanogènes, tandis que des concentrations plus faibles de sulfate dans les zones humides d’eau douce entraînent des émissions de méthane plus élevées11,41. De plus, les différences d’émission de CH4 entre les milieux humides d’eau salée et d’eau douce sont également influencées par leurs liens entre les marées et l’océan57, ce qui pourrait également expliquer l’échec de la détection des gènes liés au cycle du méthane par la cPCR en Colombie-Britannique. Dans cette étude, la cPCR a réussi à détecter tous les gènes ciblés dans les échantillons LB et RV, mais n’a pas réussi à détecter la présence de ces gènes dans les échantillons de BC, le gène pmoA2 étant la seule exception. De plus, la qPCR a pu détecter mmoX dans tous les échantillons, alors que la cPCR n’a pas pu le détecter en Colombie-Britannique. Cela indique que la qPCR montre une plus grande résilience aux inhibiteurs de la PCR par rapport à la cPCR, en raison de sa sensibilité accrue dans les mécanismes de détection58. Une autre raison est que la qPCR identifie les produits PCR pendant la phase exponentielle du cycle de PCR, tandis que la cPCR les détecte dans la phase de plateau. Cette distinction permet à la qPCR de présenter une sensibilité réduite à la dégradation du produit au cours de cycles de réaction avancés, où les réactifs sont épuisés47.
Cette étude a permis d’établir une technique de PCR pour identifier les gènes liés au cycle du méthane dans les zones humides côtières du sud du Texas afin de surmonter les défis associés aux inhibiteurs de PCR et à l’efficacité variable des amorces. Quatre paires d’amorces ciblant le gène mcrA et deux paires ciblant le gène pmoA1 ont été utilisées, et une seule paire de chaque ensemble a détecté efficacement ces gènes dans la PCR conventionnelle et en temps réel. Cette étude conclut l’ensemble des paires d’amorces pour les gènes liés au cycle du méthane, y compris a189-mb6661 pour pmoA1, 206f-703b pour pmoA2, 536F-898R pour mmoX et ML pour mcrA. Cela suggère l’importance d’une sélection appropriée d’amorces pour détecter les gènes liés au cycle du méthane dans les zones humides côtières, avec ceux du sud du Texas comme exemples. De plus, l’étude a montré que mmoX était plus abondant pendant la saison fraîche tandis que pmoA1 était abondant pendant la saison chaude.
Ainsi, les résultats de cette étude jettent les bases de la compréhension scientifique des processus microbiens impliqués dans le cycle du méthane, mettant en évidence l’influence des facteurs environnementaux sur les variations saisonnières et spatiales dans les zones humides côtières du sud du Texas. Sur la base de ces résultats, des études futures visent à valider davantage cette approche dans d’autres écosystèmes, tels que les rizières et les zones humides d’eau douce, afin d’accroître la robustesse et la généralisabilité. De plus, ces études comprendront également des analyses géochimiques telles que les concentrations de méthane dans les sédiments et le sulfure d’eau interstitielle, ainsi que des techniques moléculaires telles que la métagénomique et la transcriptomique, qui seront basées sur ce travail de base pour fournir une compréhension plus complète de la dynamique du cycle du méthane.
Déclarations de divulgation
Les auteurs n’ont aucun conflit d’intérêts à déclarer.
Remerciements
Nous sommes reconnaissants envers les membres de C-REAL pour leur aide dans l’observation sur le terrain et les analyses en laboratoire.
matériels
| Name | Company | Catalog Number | Comments |
| 0.2 mL PCR tubes | ThermoFisher Scientific | AB0620 | https://www.thermofisher.com/order/catalog/product/AB0620?SID=srch-srp-AB0620 |
| 0.5 mL PCR Tubes | Promega | E4941 | https://www.promega.com/products/biochemicals-and-labware/tips-and-accessories/0_5ml-pcr-tubes/?catNum=E4941 |
| 10 μL tips | ThermoFisher Scientific | 05-408-187 | Fisherbrand SureGrip Pipet Tip Racked or Reload System Tips Natural; 10μL; | Fisher Scientific |
| 15 mL centrifuge tube | ThermoFisher Scientific | 14-959-53A | https://www.fishersci.com/shop/products/falcon-15ml-conical-centrifuge-tubes-5/p-193301 |
| 200 μL tips | ThermoFisher Scientific | 05-408-190 | Fisherbrand SureGrip Pipet Tip Racked or Reload System Tips Natural; 200μL; | Fisher Scientific |
| 1000 μL tips | ThermoFisher Scientific | 02-707-402 | https://www.fishersci.com/shop/products/sureone-micropoint-pipette-tips-specific-standard-fit/02707402?gclid=Cj0KCQiAp NW6BhD5ARIsACmEb kUsQ9Lu0YIq5i4vWege 17qPdtxIYZyvmJH1cDo ARuwereO1V4GLz9UaA lDREALw_wcB&ef_id=C j0KCQiApNW6BhD5ARI sACmEbkUsQ9Lu0YIq5i 4vWege17qPdtxIYZyvmJ H1cDoARuwereO1V4GLz 9UaAlDREALw_wcB:G:s &ppc_id=PLA_goog_2175 7693617_171052169911_02 707402__715434303113_1555 377385658230343&ev_chn=sh op&s_kwcid=AL!4428!3!71543430 3113!!!g!2366517300713!&gad_source=1 |
| Applied Biosystem Power SYBR Green Master Mix | ThermoFisher Scientific | 4368577 | https://www.thermofisher.com/order/catalog/product/4368577 |
| ArcGIS Pro | esri | https://www.esri.com/en-us/arcgis/products/arcgis-pro/overview?srsltid=AfmBOopatJ4 JvHJfscHRcAaDx0Jz5_Jrl8l5 vYkkBvfOqE-uNSsMghN1 | |
| CFX Duet Real-Time PCR system | Bio-Rad | 12016265 | https://www.bio-rad.com/en-us/product/cfx-duet-real-time-pcr-system?ID=97722926-9ed9-16a4-1d83-c92f587e427a |
| Corning Lambda plus single channel pipettor volume 0.5-10 μL | Sigma-Aldrich | CLS4071-1EA | https://www.sigmaaldrich.com/US/en/product/sigma/cls4071 |
| Corning Lambda plus single channel pipettor volume 100-1000 μL | Sigma-Aldrich | CLS4075-1EA | https://www.sigmaaldrich.com/US/en/product/sigma/cls4075 |
| Corning Lambda plus single channel pipettor volume 20-200 μL | Sigma-Aldrich | CLS4074-1EA | https://www.sigmaaldrich.com/US/en/product/sigma/cls4074 |
| FastDNA spin kit for soil | MP Biomedical | 116560200-CF | https://www.mpbio.com/us/116560000-fastdna-spin-kit-for-soil-samp-cf?srsltid=AfmBOoqOxxGilzY3IHNIZR ajegGTr9MoX1oMZUh 3dcbJqe0UvvukY128 |
| Gene copy calculator | Science Primer | https://scienceprimer.com/copy-number-calculator-for-realtime-pcr . | |
| High speed benchtop centrifuge | ThermoFisher Scientific | 75004241 | https://newlifescientific.com/products/thermo-scientific-sorvall-st16-high-speed-benchtop-centrifuge-75004241?gad_source=1&gclid=Cj0KCQiApN W6BhD5ARIsACmEbkVC_-cCIN9j 20TvYq8iDsBlUR5cPK_1_wN OBEcjMdv-CYVoGCfeOLYaAv enEALw_wcB |
| High speed microcentrifuge | VWR | 75838-336 | https://us.vwr.com/store/product/20546590/null |
| Lysing Matrix E tube | glass bead/ceramic sphere-containing tube | ||
| Microcentrifuge tube | ThermoFisher Scientific | 02-681-320 | https://www.fishersci.com/shop/products/fisherbrand-low-retention-microcentrifuge-tubes-8/02681320?gclid=Cj0KCQiAp NW6BhD5ARIsACm EbkWbG4_o3oUiGk HJPU-_31-CuexDwQ fmWPnfyhBOf2BHXsy K3fFW1toaAgJbEALw_ wcB&ef_id=Cj0KCQiAp NW6BhD5ARIsACmEb kWbG4_o3oUiGkHJPU- _31-CuexDwQfmWPnfy hBOf2BHXsyK3fFW1toa AgJbEALw_wcB:G:s&ppc _id=PLA_goog_21757693 617_171052169911_0268 1320__715434303113_10 349826094968484711&ev _chn=shop&s_kwcid=AL!4 428!3!715434303113!!!g!23 66517300713!&gad_source=1 |
| PCR Master mix | Promega | M7502 | https://www.promega.com/products/pcr/taq-polymerase/master-mix-pcr/?catNum=M7502 |
| Quantiflour ONE dsDNA system | Promega | E4871 | https://www.promega.com/products/rna-analysis/dna-and-rna-quantitation/quantifluor-one-dsdna-system/?gad_source=1&gbraid=0AAAAAD _rg189yJTY3cxeVqMdu8RPx10 Ma&gclid=CjwKCAjwxNW2BhAk EiwA24Cm9FUgViPNyWq7UfZL VeeoroLAZ5JIP6w07RGK_4D0w oZgAqf-G1XTmxoCxm8QAvD_B wE&catNum=E4871 |
| Quantus Fluorometer | Promega | E6150 | https://www.promega.com/products/microplate-readers-fluorometers-luminometers/fluorometers/quantus-fluorometer/?catNum=E6150 |
| YSI Pro 2030 | YSI a xylem brand | 603174 | https://www.ysi.com/product/id-p2030/pro2030-kits |
Références
- Xu, T., et al. Wetlands of international importance: Status, threats, and future protection. Int J Environ Res Public Health. 16 (10), 1818(2019).
- Corn, M. L. Deepwater Horizon oil spill: coastal wetland and wildlife impacts and response. , DIANE Publishing. (2010).
- Hendriks, I. E., Sintes, T., Bouma, T. J., Duarte, C. M. Experimental assessment and modeling evaluation of the effects of the seagrass Posidonia oceanica on flow and particle trapping. Marine Ecology Progress Series. 356, 163-173 (2008).
- Krause, S. J. E., Treude, T. Deciphering cryptic methane cycling: Coupling of methylotrophic methanogenesis and anaerobic oxidation of methane in hypersaline coastal wetland sediment. Geochimica et Cosmochimica Acta. 302, 160-174 (2021).
- Reddy, K. R., DeLaune, R. D., Inglett, P. W. Biogeochemistry of Wetlands: Science and Applications. , 2nd ed, CRC Press. (2022).
- La, W., et al. Sulfate concentrations affect sulfate reduction pathways and methane consumption in coastal wetlands. Water Research. 217, 118441(2022).
- Derwent, R. G. Global warming potential (GWP) for methane: Monte Carlo analysis of the uncertainties in Global Tropospheric Model predictions. Atmosphere. 11 (5), 486(2020).
- Potter, C., et al. Methane emissions from natural wetlands in the United States: Satellite-derived estimation based on ecosystem carbon cycling. Earth Interactions. 10 (22), 1-12 (2006).
- Understanding global warming potentials. , U.S. Environmental Protection Agency. https://www.epa.gov/ghgemissions/understanding-global-warming-potentials (2024).
- Wallenius, A. J., Dalcin Martins, P., Slomp, C. P., Jetten, M. S. M. Anthropogenic and environmental constraints on the microbial methane cycle in coastal sediments. Front Microbiol. 12, 631621(2021).
- Qu, Y., et al. Salinity causes differences in stratigraphic methane sources and sinks. Environmental Science and Ecotechnology. 19, 100334(2024).
- Vizza, C., West, W. E., Jones, S. E., Hart, J. A., Lamberti, G. A. Regulators of coastal wetland methane production and responses to simulated global change. Biogeosciences. 14 (2), 431-446 (2017).
- van Dijk, G., et al. Salinization lowers nutrient availability in formerly brackish freshwater wetlands; unexpected results from a long-term field experiment. Biogeochemistry. 143 (1), 67-83 (2019).
- Aronson, E., Allison, S., Helliker, B. R. Environmental impacts on the diversity of methane-cycling microbes and their resultant function. Front Microbiol. 4, 225(2013).
- Reeburgh, W. S. Oceanic methane biogeochemistry. Chem Rev. 107 (2), 486-513 (2007).
- Thauer, R. K. Biochemistry of methanogenesis: a tribute to Marjory Stephenson. Marjory Stephenson Prize Lecture. Microbiology (Reading). 144 (Pt 9), 2377-2406 (1998).
- Thauer, R. K. Anaerobic oxidation of methane with sulfate: on the reversibility of the reactions that are catalyzed by enzymes also involved in methanogenesis from CO2. Curr Opin Microbiol. 14 (3), 292-299 (2011).
- Friedrich, M. W. Methyl-coenzyme M reductase genes: Unique functional markers for methanogenic and anaerobic methane-oxidizing Archaea. Methods in Enzymology. 397, 428-442 (2005).
- Reeburgh, W. S. Oceanic methane biogeochemistry. Chem Rev. 107 (2), 486-513 (2007).
- Rasigraf, O., Schmitt, J., Jetten, M. S. M., Lüke, C. Metagenomic potential for and diversity of N-cycle driving microorganisms in the Bothnian Sea sediment. Microbiologyopen. 6 (4), e00475(2017).
- McDonald, I. R., Bodrossy, L., Chen, Y., Murrell, J. C. Molecular ecology techniques for the study of aerobic methanotrophs. Appl Environ Microbiol. 74 (5), 1305-1315 (2008).
- Tchawa Yimga, M., Dunfield, P. F., Ricke, P., Heyer, J., Liesack, W. Wide distribution of a novel pmoA-like gene copy among type II methanotrophs, and its expression in Methylocystis strain SC2. Appl Environ Microbiol. 69 (9), 5593-5602 (2003).
- Knief, C. Diversity and habitat preferences of cultivated and uncultivated aerobic methanotrophic bacteria evaluated based on pmoA as molecular marker. Front Microbiol. 6, 1346(2015).
- Huang, I. -S., et al. Preliminary assessment of microbial community structure of Wind-Tidal Flats in the Laguna Madre, Texas, USA. Biology. 9 (8), 183(2020).
- Wilding, T. K., Brown, E., Collier, K. J. Identifying dissolved oxygen variability and stress in tidal freshwater streams of northern New Zealand. Environ Monit Assess. 184 (10), 6045-6060 (2012).
- Roy Chowdhury, T., Mitsch, W. J., Dick, R. P. Seasonal methanotrophy across a hydrological gradient in a freshwater wetland. Ecological Engineering. 72, 116-124 (2014).
- Sun, Q. -Q., et al. Carbon dioxide and methane fluxes: Seasonal dynamics from inland riparian ecosystems, northeast China. Sci Total Environ. 465, 48-55 (2013).
- Lee, S., et al. Comparison and selection of conventional PCR primer sets for studies associated with nitrogen cycle microorganisms in surface soil. Appl Sci. 12 (20), 10314(2022).
- Bae, K. -S., et al. Development of diagnostic systems for wide range and highly sensitive detection of two waterborne hepatitis viruses from groundwater using the conventional reverse transcription nested PCR assay. J Virol Methods. 299, 114344(2022).
- Lecusay, D. Jr Assessment and Monitoring of Deltaic Wetlands and Fluvial Systems: Refining and Validating a Multimetric Index of Resaca Ecosystem Health. , The University of Texas Rio Grande Valley. (2021).
- FastDNA SPIN Kit for Soil (Cat No. 116560200). , MP Biomedicals. https://www.mpbio.com/media/productattachment/LS082019-EN-FastDNA-SPIN-Kit-for-Soil-116560200-Manual.pdf (2025).
- Luton, P. E., Wayne, J. M., Sharp, R. J., Riley, P. W. The mcrA gene as an alternative to 16S rRNA in the phylogenetic analysis of methanogen populations in landfill. Microbiology (Reading, England). 148 (Pt 11), 3521-3530 (2002).
- Changsoo, L., Jaai, K., Seung Gu, S., Seokhwan, H. Absolute and relative QPCR quantification of plasmid copy number in Escherichia coli. J Biotechnol. 123 (3), 273-280 (2006).
- Harms, G., et al. Real-time PCR quantification of nitrifying bacteria in a municipal wastewater treatment plant. Environ Sci Technol. 37 (2), 343-351 (2003).
- Holmes, A. J., Costello, A., Lidstrom, M. E., Murrell, J. C. Evidence that particulate methane monooxygenase and ammonia monooxygenase may be evolutionarily related. FEMS Microbiol Lett. 132 (3), 203-208 (1995).
- Fuse, H., et al. Oxidation of trichloroethylene and dimethyl sulfide by a marine Methylomicrobium strain containing soluble methane monooxygenase. Biosci Biotechnol Biochem. 62 (10), 1925-1931 (1998).
- Springer, E., Sachs, M. S., Woese, C. R., Boone, D. R. Partial gene sequences for the A subunit of methyl-coenzyme M reductase (mcrI) as a phylogenetic tool for the family Methanosarcinaceae. Int J Syst Bacteriol. 45 (3), 554-559 (1995).
- Costello, A. M., Lidstrom, M. E. Molecular characterization of functional and phylogenetic genes from natural populations of methanotrophs in lake sediments. Appl Environ Microbiol. 65 (11), 5066-5074 (1999).
- Flores, E. A. Effects of Nutrient Enrichment on Mangrove and Saltmarsh Habitats. , The University of texas Rio Grande Valley. (2022).
- Minjie, H., Jordi, S., Xianyu, Y., Josep, P., Chuan, T. Patterns and environmental drivers of greenhouse gas fluxes in the coastal wetlands of China: A systematic review and synthesis. Environ Res. 186, 109576(2020).
- Bridgham, S. D., Cadillo-Quiroz, H., Keller, J. K., Zhuang, Q. Methane emissions from wetlands: biogeochemical, microbial, and modeling perspectives from local to global scales. Glob Chang Biol. 19 (5), 1325-1346 (2013).
- Liu, D. Y., Ding, W. X., Jia, Z. J., Cai, Z. C. Relation between methanogenic archaea and methane production potential in selected natural wetland ecosystems across China. Biogeosciences. 8 (2), 329-338 (2011).
- Ke, Z., et al. Methane emissions and methanogenic community investigation from constructed wetlands in Chengdu City. Urban Climate. 39, 100956(2021).
- Chowdhury, T. R., Dick, R. P. Ecology of aerobic methanotrophs in controlling methane fluxes from wetlands. Applied Soil Ecology. 65, 8-22 (2013).
- Maja, S., et al. Humic substances cause fluorescence inhibition in real-time polymerase chain reaction. Anal Biochem. 487, 30-37 (2015).
- Sanches, T. M., Schreier, A. D. Optimizing an eDNA protocol for estuarine environments: Balancing sensitivity, cost and time. PLOS ONE. 15 (5), e0233522(2020).
- Xia, Z., et al. Conventional versus real-time quantitative PCR for rare species detection. Ecol Evol. 8 (23), 11799-11807 (2018).
- Bourne, D. G., McDonald, I. R., Murrell, J. C. Comparison of pmoA PCR primer sets as tools for investigating methanotroph diversity in three Danish soils. Appl Environ Microbiol. 67 (9), 3802-3809 (2001).
- Juottonen, H., Galand, P. E., Yrjala, K. Detection of methanogenic Archaea in peat: comparison of PCR primers targeting the mcrA gene. Res Microbiol. 157 (10), 914-921 (2006).
- Lueders, T., Friedrich, M. W. Evaluation of PCR amplification bias by terminal restriction fragment length polymorphism analysis of small-subunit rRNA and mcrA genes by using defined template mixtures of methanogenic pure cultures and soil DNA extracts. Appl Environ Microbiol. 69 (1), 320-326 (2003).
- Vaksmaa, A., Jetten, M. S., Ettwig, K. F., Luke, C. McrA primers for the detection and quantification of the anaerobic archaeal methanotroph 'Candidatus Methanoperedens nitroreducens'. Appl Microbiol Biotechnol. 101 (4), 1631-1641 (2017).
- Ren, G., et al. Electron acceptors for anaerobic oxidation of methane drive microbial community structure and diversity in mud volcanoes. Environ Microbiol. 20 (7), 2370-2385 (2018).
- Goldberg, C. S., et al. Critical considerations for the application of environmental DNA methods to detect aquatic species. Methods in Ecology and Evolution. 7 (11), 1299-1307 (2016).
- McKee, A. M., Spear, S. F., Pierson, T. W. The effect of dilution and the use of a post-extraction nucleic acid purification column on the accuracy, precision, and inhibition of environmental DNA samples. Biological Conservation. 183, 70-76 (2015).
- Gohl, D. M., et al. Systematic improvement of amplicon marker gene methods for increased accuracy in microbiome studies. Nat Biotechnol. 34 (9), 942-949 (2016).
- Ballarini, A., Segata, N., Huttenhower, C., Jousson, O. Simultaneous quantification of multiple bacteria by the BactoChip microarray designed to target species-specific marker genes. PLOS ONE. 8 (2), e55764(2013).
- Le Mer, J., Roger, P. Production, oxidation, emission and consumption of methane by soils: A review. Eur J Soil Biol. 37 (1), 25-50 (2001).
- Smith, C. J., Osborn, A. M. Advantages and limitations of quantitative PCR (Q-PCR)-based approaches in microbial ecology. FEMS Microbiol Ecol. 67 (1), 6-20 (2009).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon