Method Article
Visualización de la dinámica microbiana del ciclo del metano en los humedales costeros
En este artículo
Resumen
El protocolo detecta genes clave del ciclo del metano en los humedales costeros del sur de Texas y visualiza su distribución espacial para mejorar la comprensión de la regulación del metano y sus impactos ambientales en estos ecosistemas dinámicos.
Resumen
Los humedales costeros son la mayor fuente biótica de metano, donde los metanógenos convierten la materia orgánica en metano y los metanótrofos oxidan el metano, desempeñando así un papel fundamental en la regulación del ciclo del metano. Los humedales del sur de Texas, que están sujetos a frecuentes fenómenos meteorológicos, fluctuaciones en los niveles de salinidad y actividades antropogénicas debidas al cambio climático, influyen en el ciclo del metano. A pesar de la importancia ecológica de estos procesos, el ciclo del metano en los humedales costeros del sur de Texas sigue siendo insuficientemente explorado. Para abordar esta brecha, desarrollamos y optimizamos un método para detectar genes relacionados con metanógenos y metanótrofos, incluido el mcrA como biomarcador para los metanógenos y pmoA1, pmoA2 y mmoX como biomarcadores para los metanótrofos. Además, este estudio tuvo como objetivo visualizar los patrones de distribución espacial y temporal de la abundancia de metanógeno y metanótrofo utilizando el software del sistema de información geográfica (SIG) ArcGIS Pro. La integración de estas técnicas moleculares con la visualización geoespacial avanzada proporcionó información crítica sobre la distribución espacial y temporal de las comunidades de metanógeno y metanótrofo en los humedales del sur de Texas. Por lo tanto, la metodología establecida en este estudio ofrece un marco sólido para mapear la dinámica microbiana en los humedales, mejorando nuestra comprensión del ciclo del metano en condiciones ambientales variables y respaldando estudios más amplios sobre el cambio ecológico y ambiental.
Introducción
Los humedales costeros son ecosistemas vitales que contribuyen a la regulación del clima, la conservación de la biodiversidad y la gestión del agua a través de procesos como el secuestro de carbono, la evapotranspiración y las emisiones de metano (CH4)1. Estos ecosistemas, incluidos los humedales de agua dulce y salada2, son altamente productivos y actúan como zonas críticas para la absorción de dióxido de carbono (CO2) y la captura de materia orgánica de los ambientes terrestres y marinos 3,4. Las interacciones dinámicas dentro de estos humedales estimulan la producción y el consumo microbiano de CH4 5, posicionándolos como una de las mayores fuentes naturales de CH46. Como el segundo gas de efecto invernadero más importante, elCH4 tiene un potencial de calentamiento global aproximadamente 27-30 veces mayor que el del CO24,7,8,9, lo que hace que el estudio de las emisiones deCH4 de los humedales costeros sea esencial en la era del cambio climático. La emisión deCH4 está influenciada por diversos factores ambientales, en particular la salinidad, que desempeña un papel crucial en los procesos microbianos10. Los humedales de agua dulce contribuyen significativamente al metano atmosférico debido a sus niveles más bajos de sulfato, lo que facilita una mayor producción microbiana deCH4, mientras que los humedales de agua salada generalmente tienden a emitir menosCH4 debido a las mayores concentraciones de sulfato 11,12,13.
Las emisiones deCH4 de los humedales costeros generalmente están controladas por dos grupos de microorganismos, conocidos como metanógenos y metanótrofos14. Los metanógenos producen CH4 en sedimentos anóxicos al descomponer sustratos como formiato, acetato, hidrógeno o compuestos metilados a través de un proceso conocido como metanogénesis15. La enzima importante en esta vía es la metil-coenzima M reductasa (MCR), ya que cataliza el paso final y limitante de la tasa de metanogénesis 15,16,17. El gen mcrA, que codifica la subunidad alfa de MCR, es un marcador funcional que se puede encontrar en todas las arqueas metanogénicas18. Además, en los humedales costeros, la zona de transición sulfato-metano (ZMS) se forma por encima de la zona metanogénica, donde el metano que se difunde hacia arriba y el sulfato que se desplaza hacia abajo convergen y se agotan19. Dentro de esta zona, las arqueas metanotróficas anaeróbicas (ANME) oxidan el metano a dióxido de carbono utilizando la enzima MCR, mientras que las bacterias reductoras de sulfato (SRB) reducen el sulfato a sulfuro. Los SRB superan a los metanógenos por el hidrógeno y el acetato, limitando la producción de metano hasta que se agote el sulfato16,17.
Por el contrario, las bacterias metanotróficas aerobias oxidan el CH4 en ambientes aeróbicos20, utilizando diferentes formas de metano monooxigenasa (MMO). Estos incluyen la metano monooxigenasa particulada (pMMO), una enzima que contiene cobre incrustada en la membrana intracitoplasmática, y la metano monooxigenasa soluble (sMMO), una enzima que contiene hierro que se encuentra en el citoplasma. Sin embargo, para pMMO, hay tres operones de genes pmoCAB21; entre ellos, el gen pmoA es el más conservador para todos los metanótrofos. Hay dos genes biomarcadores diferentes para pmoA: pmoA1 y pmoA222. Además, para una comprensión completa de los metanótrofos, el gen mmoX se utiliza como herramienta en biología molecular para identificar metanótrofos que contienen sMMO23. Esta distinción en las rutas metabólicas y los requisitos ambientales de metanógenos y metanótrofos aeróbicos pone de relieve las complejas interacciones microbianas que regulan el ciclo del metano en los ecosistemas de humedales costeros.
El humedal de Boca Chica (BC), un ambiente productivo de agua salada en el sur de Texas, experimenta influencias de las mareas del Golfo de México (GOM), lo que lleva a niveles variables de salinidad superficial, especialmente debido a su proximidad a la hipersalina Laguna Madre24. Esta acción de las mareas, que alterna entre mareas altas y bajas, hace que los niveles de oxígeno fluctúen25 , lo que podría alterar la actividad del metanógeno y el metanótrofo en los sedimentos26. Por el contrario, los humedales costeros de agua dulce se consideran un importante punto caliente para los flujos de CH4 27. Los humedales costeros de agua dulce en el sur de Texas, incluyendo Resaca Del Rancho Viejo (RV) y Lozano Banco (LB), distantes de los efectos de las mareas del GOM, tienen un manejo hidrológico distinto. RV experimenta flujos de pulso complementados por el agua del río durante los niveles bajos de agua, mientras que LB funciona como un sistema de flujo fuera de línea sin dicha suplementación. Además, RV y LB mantienen niveles de salinidad más bajos debido a una alta descarga de agua dulce bombeada artificialmente y al ser un lago en meandro, respectivamente. Los diferentes factores ambientales pueden influir significativamente en el ciclo del metano a través de los humedales costeros del sur de Texas. Sin embargo, el ciclo del metano en los humedales costeros del sur de Texas sigue siendo un área que aún no se ha investigado a fondo.
La reacción en cadena de la polimerasa (PCR) y la PCR en tiempo real (también llamada PCR cuantitativa [qPCR]) representan técnicas fundamentales y ampliamente utilizadas para detectar y cuantificar la abundancia relativa de genes específicos en muestras ambientales. Estas técnicas amplifican específicamente regiones específicas del ADN para indicar la presencia y la cantidad relativa de genes relacionados con el ciclo de CH4, proporcionando indicadores de un posible ciclo del metano. Sin embargo, la disponibilidad y eficacia de los juegos de cebadores de PCR podría estar limitada por diversos factores inhibidores en el ADN ambiental extraído, siendo impactados por los tipos de ambientes28,29. Por lo tanto, este estudio estableció principalmente un método de PCR óptimo para detectar la presencia de genes relacionados con el ciclo de CH4 en los humedales costeros del sur de Texas (Figura 1) y luego visualizó su abundancia relativa cuantificada en estos ecosistemas. Los resultados de este estudio se pueden aplicar a otras regiones costeras para mejorar la comprensión del ciclo del CH4 y la dinámica microbiana en diversos ecosistemas costeros.
Protocolo
1. Recogida de muestras
- Recoja muestras de sedimentos con un muestreador de sedimentos o una pala.
NOTA: Se recolectaron muestras de dos estaciones de tres humedales costeros distintos durante las estaciones frescas (octubre-febrero, la temperatura promedio es de 20 °C) y cálidas (abril-junio, la temperatura promedio es de 27 °C) de 2023 y 2024. Se utilizó un muestreador de sedimentos cuando se recolectaron muestras de humedales costeros de agua dulce (Figura 2) y se usó una pala para los humedales costeros de agua salada influenciados por las mareas. - Baje primero el muestreador en el cuerpo de agua poco profundo y permita que se hunda hasta la superficie del sedimento (dentro de los 50 cm superiores) por su propio peso, minimizando la perturbación de la estructura del sedimento como se muestra en la Figura 2.
- Sáquelo de la columna de agua, donde la profundidad era típicamente de 60 cm a 215 cm30, transfiera las muestras de sedimento a bolsas con cierre hermético y guárdelas en una caja de hielo de inmediato.
NOTA: Limpie y lave el muestreador con agua desionizada (DI) antes de pasar a las siguientes estaciones. - Almacene todas las muestras a -20 °C inmediatamente en el laboratorio.
- En cada sitio de muestreo, mida los parámetros de calidad del agua superficial, como la salinidad y la temperatura, in situ utilizando un medidor de calidad del agua multiparamétrico.
NOTA: Las sondas se enjuagaron con agua desionizada (agua desionizada) después de su uso en cada estación.
2. Extracción de ADN genómico
- Descongele las muestras a temperatura ambiente antes de comenzar el procedimiento de extracción de ADN genómico.
- Transfiera aproximadamente 500 mg de muestras de sedimentos a un tubo de 15 ml y centrifugue a 4.250 × g durante 3 minutos para eliminar toda el agua.
- Extraiga el ADN genómico utilizando un kit de extracción de ADN para tierra siguiendo el protocolodel fabricante 31 con una pequeña modificación y almacene a -20 °C inmediatamente.
NOTA: Los cambios se realizaron para reducir la redundancia y la eficiencia del flujo de trabajo.- Agregue hasta 500 mg de muestra de suelo a un tubo que contenga perlas de vidrio/esfera de cerámica.
- Añada 978 μL de tampón de fosfato de sodio a la muestra en el tubo que contiene perlas/esferas.
- Añada 122 μL de solución tampón de lisis a la muestra en el tubo que contiene perlas/esferas para solubilizar los contaminantes externos.
- Homogeneice con un homogeneizador de molino de bolas a 5 veces el nivel de velocidad durante 20 s y repita 2 veces.
NOTA: En este estudio se utilizó un homogeneizador de molino de bolas, por lo que se ajustó la velocidad. - Centrifugar la mezcla durante 10 min a 14.000 × g.
- Transfiera el sobrenadante a un tubo de microcentrífuga limpio de 2,0 mL.
- Añadir 250 μL de solución de precipitación de proteínas (PPS) para separar los ácidos nucleicos solubilizados de los restos celulares y de la matriz de lisado. Mezclar invirtiendo el tubo 10x.
- Centrifugar a 14.000 × g durante 5 min para precipitar el pellet, eliminando los restos celulares y la matriz lisante.
- Transfiera el sobrenadante a un tubo de microcentrífuga limpio de 15 mL.
- Agregue 1.0 mL de la suspensión de la Matriz de Unión al sobrenadante en el tubo de 15 mL.
NOTA: Agite la suspensión de Binding Matrix para volver a suspenderla antes de agregarla. - Permita la unión del ADN a la matriz de unión colocando los tubos en un rotador durante 2 min.
- Coloque todos los tubos en una rejilla e incube durante 3 min para permitir la sedimentación de la matriz de unión.
- Después de 3 minutos, deseche 750 μL y mezcle suavemente el sobrenadante restante con el pellet con una pipeta.
- Transfiera 750 μL de la mezcla a un filtro SPIN y centrifugue a 14.000 × g durante 1 min. Vacíe el tubo de recogida y reutilícelo. Repita con la mezcla restante.
- Añada 500 μL de la solución de lavado preparada (con la cantidad adecuada de etanol añadida) para solubilizar aún más las impurezas. Vuelva a suspender suavemente el pellet utilizando la fuerza del líquido de la punta de la pipeta.
- Centrifugar a 14.000 × g durante 1 min para eliminar las impurezas. Vacíe el tubo de recogida y reutilícelo.
- Vuelva a centrifugar a 14.000 × g durante 2 min sin añadir nada.
- Reemplace el tubo de captura por un tubo de captura nuevo y limpio y seque al aire el filtro SPIN durante 5 minutos a temperatura ambiente.
- Añadir 50 μL de agua libre de ADNasa (DES) y centrifugar a 14.000 × g durante 1 min.
NOTA: En este protocolo, se utilizaron 50 μL de DES para la elución del ADN con el fin de garantizar una recuperación y estabilidad óptimas del ADN ambiental extraído (eDNA).
3. Cuantificación de ADN
- Añadir 1 μL de ADN extraído con 200 μL de colorante fluorescente en un tubo de 0,5 mL y mezclar bien mediante pipeteo.
- Envuelva el tubo inmediatamente con papel de aluminio para que la luz no pueda penetrar e incube a temperatura ambiente durante 5 minutos.
- Mida la concentración de ADN utilizando un fluorómetro en UN modo de concentración de ADN siguiendo el protocolo del fabricante.
4. Detección de ARNr 16S, pmoA1 , pmoA2 , mmoX y mcrA por PCR convencional
- Antes de realizar la PCR convencional (cPCR), descongele todas las muestras y reactivos en una cubeta de hielo.
- Diluir todas las muestras de eDNA extraídas a 10 ng/μL.
NOTA: En la Tabla 1 se da una lista de cebadores. - Prepare una mezcla de reacción de cPCR de 25 μL para cada muestra, que incluya 12,5 μL de mezcla maestra de PCR 2x, 0,5 μL de cebadores directos e inversos (10 μM) (consulte la Tabla 1 para ver la lista de cebadores), 1 μL de 10 ng/μL de eDNA y 10,5 μL de agua sin nucleasas.
NOTA: Prepare la mezcla maestra con suficiente volumen para una muestra adicional a fin de minimizar los errores de pipeteo. - Realizar la reacción de cPCR siguiendo el protocolo que consiste en una desnaturalización inicial a 95 °C durante 2 min, seguida de 40 ciclos de desnaturalización a 95 °C durante 45 s, extensión a 72 °C durante 30 s y extensión final a 72 °C durante 5 m, con temperatura de recocido variable para diferentes cebadores (consulte la Tabla 1 para conocer las temperaturas de recocido de diferentes cebadores utilizados para diferentes genes).
NOTA: Para el gen mcrA , los cebadores ML mostraron la banda deseada utilizando una velocidad de rampa lenta de 0,1 °C/s entre los pasos de recocido y extensión durante los primeros cinco ciclos32. - Visualice los productos de cPCR en un gel de agarosa preteñido con bromuro de etidio (EtBr).
NOTA: Use gel de agarosa al 2,5% para una escalera de 50 bp y gel al 0,9% para una escalera de 1 kb. Utilice 1x tampón TAE para un gel de agarosa al 2,5% y 0,5x tampón TAE para un gel de agarosa al 0,9%.
5. Detección de pmoA1 , pmoA2 , mmoX y mcrA por PCR cuantitativa en tiempo real
NOTA: Los genes dirigidos al metanógeno y al metanótrofo, como la abundancia de pmoA1, pmoA2, mmoX y mcrA , se observaron mediante qPCR utilizando un sistema de PCR en tiempo real.
- Prepare los patrones para cada gen por separado para obtener la curva estándar para cada gen.
- Amplifique el gen objetivo con cPCR, utilizando las muestras que produjeron la banda más brillante durante la electroforesis en gel de cada gen. Siga el método y las cartillas descritas en la sección 4.
- Purificar los productos amplificados con un kit de extracción en gel y medir la concentración de ADN siguiendo el método descrito en la sección 3 y almacenar a -20 °C.
NOTA: Alícuota de los patrones purificados en tubos separados para su uso posterior. - Calcule las copias de genes a partir de la concentración de ADN medida utilizando el sitio web de cálculo del número de copias para qPCR.
- Diluya el número de copias calculado del patrón con agua libre de nucleasas para preparar cada patrón, que oscile entre 108 y 102 copias/μL, antes de ejecutar la qPCR.
- Prepare la curva estándar utilizando tres réplicas de cada número de copia, incluido un control negativo (NTC, sin ADN plantilla).
NOTA: El valor de R2 de cada curva estándar fue mayor que 0,99.
- Prepare una mezcla de reacción de qPCR de 20 μL para todas las muestras, patrones y NTC. Realice el análisis de qPCR por triplicado para todas las muestras.
- Coloque todos los componentes de la reacción, incluida la mezcla maestra de qPCR, los cebadores, el agua sin nucleasas, los patrones y las muestras, en una rejilla de hielo antes de comenzar.
- Prepare una mezcla de reacción de qPCR de 20 μL para cada muestra, estándar y NTC, que contenga 10 μL de mezcla maestra SYBR Green, 0,5 μL de cada 10 μM de cebadores directos e inversos, 8 μL de agua libre de nucleasas y 1 μL de 10 ng/μL de ADN moltest, o agua estándar o desionizada, respectivamente.
NOTA: Utilice los juegos de cebadores que se muestran para obtener resultados óptimos para los genes pmoA1 y mcrA en PCR convencional para qPCR (consulte la Tabla 1). Para mejorar la precisión, ejecute cada muestra por triplicado. Los siguientes pasos proporcionan un método eficiente para preparar muestras por triplicado con un volumen combinado de 60 μL por muestra.- Prepare la mezcla de reacción para combinar la mezcla maestra, los cebadores directos e inversos para el gen específico y el agua libre de nucleasas en un tubo de 2 ml, excepto el ADN plantilla.
NOTA: Prepare el volumen de la mezcla de reacción teniendo en cuenta el error de pipeteo. Por ejemplo, si hay 24 muestras y 8 estándares, calcule el volumen total de 33 reacciones en lugar de 32 reacciones para minimizar los errores de pipeteo. En este caso, el volumen total requerido para las reacciones por triplicado sería el siguiente: 990 μL de mezcla maestra (33 muestras x 3 réplicas x 10 μL), 49,5 μL de cebadores directos (33 muestras x 3 réplicas x 0,5 μL), 49,5 μL de cebadores inversos (33 muestras x 3 réplicas x 0,5 μL) y 792 μL de agua libre de nucleasas (33 muestras x 3 réplicas x 8 μL). - Prepare los tubos de PCR de acuerdo con el número de muestras y los estándares.
- Dispense 57 μL de la mezcla de reacción preparada en cada tubo de PCR.
NOTA: Para realizar qPCR para cada muestra por triplicado, prepare un volumen total de mezcla de reacción de 57 μL por muestra (excluyendo el ADN molde, el patrón o el agua). Este volumen se dividirá en partes iguales en tres pocillos para una muestra, con 19 μL asignados a cada pocillo. - Agregue 3 μL de ADN molde, agua estándar o sin nucleasas a cada tubo y mezcle golpeando suavemente el fondo del tubo.
NOTA: El volumen total de la mezcla de reacción preparada para una muestra sería de 60 μL ahora en cada tubo.
- Prepare la mezcla de reacción para combinar la mezcla maestra, los cebadores directos e inversos para el gen específico y el agua libre de nucleasas en un tubo de 2 ml, excepto el ADN plantilla.
- Alícuota 20 μL de la mezcla de reacción preparada de cada tubo en los pocillos designados de una placa de qPCR de 96 pocillos. Selle la placa de PCR con una película adhesiva de sellado de PCR con un aplicador.
- Centrifugar la placa sellada a 1.000 × g durante 1 minuto para asegurar una mezcla adecuada y eliminar cualquier burbuja dentro de los pocillos.
- Coloque la placa PCR en el termociclador. Encienda la máquina de qPCR y, a continuación, abra el software relacionado para configurar el protocolo.
- Configure el protocolo de acuerdo con las directrices de mezcla maestra de qPCR. Utilice el siguiente protocolo: 95 °C durante 10 min, seguido de 95 °C durante 15 s, y un paso de extensión a 72 °C durante 30 s. Realice el paso de recocido a la temperatura de recocido especificada para los cebadores relevantes en la Tabla 1 durante 45 s. Realice todas las ejecuciones de qPCR durante 35 ciclos.
- Configure la placa qPCR de 96 pocillos con estándares y NTC en la misma configuración que la placa que contiene la muestra.
- Utilice el método de la curva estándar de cuantificación absoluta para cuantificar el producto amplificado y el número de copias del gen en cada muestra33.
6. Visualización de genes del ciclo del metano en el mapa de los humedales costeros del sur de Texas
- Abra el software del sistema de información geográfica (SIG) ArcGIS Pro y guarde el archivo de proyecto con el nombre Área de estudio en la carpeta especificada del equipo.
- Haga clic en Mapa en la parte superior izquierda | Mapa base y seleccione Terreno con etiquetas como mapa base.
- Haga clic en Localizar | Busque y cuando se abra la barra de búsqueda, ubique el área de estudio escribiendo el nombre del área; aparecerá el área.
- Dibuje el área específica utilizando la georreferenciación.
- Haga clic en Ver | Panel de catálogo de la capa superior.
- Haga doble clic en Carpeta del catálogo | Nombre del archivo.
- Haga clic con el botón derecho en el archivo de geodatabase (.gdb) y, a continuación, haga clic en Nuevo | Aparecerán Clase de entidad y Crear clase de entidad .
- Escriba el cuadro Nombre y alias y haga clic en Finalizar en la parte inferior.
- Haga clic en Ver | Contenidos. El nombre del alias aparecerá en el panel de contenido.
- Haga clic en Editar desde la capa superior | Crear. Se abrirá el panel Crear entidades . Haga doble clic en el panel Alias en Crear entidades y aparecerán las opciones de comentarios de la herramienta Configurar .
- Seleccione Líneas y, a continuación, dibuje líneas en el mapa para crear un límite exterior del área de estudio. Haga doble clic en el mapa cuando haya terminado.
- Minimice el software SIG y, a continuación, abra una hoja de cálculo. Escriba el nombre de la muestra en la primera columna, introduzca la latitud en la segunda columna y la longitud en la tercera columna. Utilice las cuatro columnas siguientes para los datos de qPCR de pmoA1, pmoA2, mmoX y mcrA.
- Guarde el archivo en formato CSV en cualquier carpeta específica de la computadora.
- Vuelva a abrir el software SIG y haga clic en Agregar datos | Datos de puntos XY.
- Seleccione el archivo CSV de la carpeta del equipo en el cuadro Tabla de entrada . Cambie el nombre del archivo en la Clase de entidad de salida y, a continuación, haga clic en Ejecutar para mostrar los puntos de muestreo en el mapa.
- Haga clic en el v barra de búsqueda en la parte superior y busque Kriging.
- Seleccione el archivo de la estación de muestreo y, a continuación, seleccione pmoA1.
- Haga clic en el botón Entorno | seleccione la estación de muestreo en capa y máscara | haga clic en Ejecutar.
- Siga los protocolos mencionados en los pasos 6.9, 6.10 y 6.11 para crear Kriging para pmoA2, mmoX y mcrA para toda el área de estudio.
- Cree un diseño del mapa.
- Haga clic en Insertar desde la capa superior | Nuevo diseño y seleccione ANSI - Horizontal.
- Haga clic en Marco de mapa, seleccione el mapa con el Kriging y coloque todos los mapas en el diseño dibujando un rectángulo. Esto hará que el mapa sea visible en el diseño.
- Seleccione la flecha norte y colóquela en el diseño para indicar la dirección norte.
- Seleccione Barra de escala para mostrar la escala del área en el mapa.
- Haga clic en Leyenda para mostrar las leyendas, luego colóquelo en el diseño.
- Haga clic en Cuadrícula y seleccione cualquiera de las opciones de retícula negra. Esto creará la cuadrícula con latitud y longitud y la mostrará en el Panel de contenido con la etiqueta Gravícula de etiqueta horizontal negra.
- Haga doble clic en Retícula de etiquetas horizontales negras y, a continuación, seleccione Componentes. Haga clic en las marcas 1 y cuadrícula y elimine estos componentes haciendo clic en el signo de cruz a su derecha.
- Haga clic en Compartir en la capa superior, luego haga clic en Exportar diseño. Seleccione el tipo de archivo como PDF, guarde el archivo en el equipo usando el cuadro Nombre, establezca la resolución vertical en 500 DPI y haga clic en Exportar para crear el archivo PDF del mapa.
Resultados
Para comprender la distribución y abundancia de los genes relacionados con el ciclo de CH4 (mcrA, pmoA1, pmoA2 y mmoX) en los humedales costeros del sur de Texas, se analizó el eDNA extraído de cada muestra mediante cPCR y qPCR. Se seleccionaron cebadores universales para cada biomarcador para realizar cPCR a partir de estudios previos (Tabla 1)22,34,35,36,37, y se realizaron modificaciones para optimizar las temperaturas y concentraciones de recocido en función de las características de la muestra y las condiciones ambientales. Por ejemplo, el ARNr 16S, pmoA1 y pmoA2 no se amplificaron utilizando la temperatura de recocido estándar porque la mayoría de ellos se amplificaron muy por debajo de la temperatura de recocido estándar (datos no mostrados). Además, durante el proceso de cPCR, una concentración de 10 ng/μL de ADN mostró una banda distinta para los genes pmoA1, pmoA2 y mmoX, mientras que el uso de 5 ng/μL de ADN molde mostró una banda más brillante para el gen mcrA (Archivo suplementario 1-Figura suplementaria S1).
El análisis de cPCR reveló variabilidad espacial en la detección de genes en tres tipos de humedales costeros del sur de Texas. Como se muestra en la Tabla 2, se detectaron genes mcrA en todas las muestras de LB y RV, que son humedales costeros de agua dulce. Sin embargo, la cPCR no pudo detectar ningún gen mcrA en el sedimento muestreado del humedal de agua salada, lo que sugiere un posible vínculo entre la salinidad y la distribución del metanógeno (Archivo Suplementario 1-Figura Suplementaria S2). Para la detección de mcrA, se utilizaron cuatro pares de cebadores en este estudio, incluidos MLF-MLR, MCRf-MCRr, ME1F-ME2R y McrA 159F-McrA 345R (Tabla 1); entre ellos, solo los pares de cebadores ML pudieron detectar la comunidad de metanógenos en los humedales del sur de Texas (Archivo Suplementario 1-Figura Suplementaria S2, Figura Suplementaria S3 y Figura Suplementaria S4). Por el contrario, para los metanótrofos aeróbicos, se detectó el gen pmoA1 en todas las muestras de RV, pero estuvo ausente en las muestras de LB y BC (Archivo suplementario 1-Figura suplementaria S5). Para identificar pmoA1, se utilizó A189 como cebador directo y A682 y mb661 como cebadores inversos, donde los pares de cebadores A189-A682 también pueden detectar amoA35,38. Sin embargo, este par de cebadores fue capaz de detectar la presencia de genes en la cPCR en este estudio, donde las imágenes en gel mostraron una banda muy tenue, lo que sugiere la baja abundancia del gen pmoA1. Otro cebador inverso, mb661, fue diseñado para identificar el gen pmoA1 específicamente con A189 como cebador directo38. Cuando se utilizó mb661 como cebador inverso, la cPCR mostró una banda más brillante (Archivo Suplementario 1-Figura Suplementaria S6), que posteriormente se utilizó en la qPCR. Curiosamente, el gen pmoA2 se detectó en dos muestras de BC y una muestra de LB, pero estaba ausente en una muestra de RV (Archivo Suplementario 1-Figura Suplementaria S7). Un estudio previo encontró que la temperatura de recocido era de 66 °C y 60 °C para pmoA2, donde a 60 °C se observaron dos amplicones de diferentes tamaños, 245 y 438 pb, mientras que a 66 °C, los cebadores 206f-703b pueden amplificar solo pmoA2 con un tamaño de producto22 de 438 pb. Sin embargo, en este estudio, la amplificación fue exitosa a una temperatura de recocido de 60 °C, produciendo una banda del tamaño esperado (438 pb) en sitios LB y BC (Archivo Suplementario 1-Figura Suplementaria S7). Además, el gen mmoX se detectó en una muestra de LB, pero estaba ausente en una muestra de RV (Archivo Suplementario 1-Figura Suplementaria S8) y estaba completamente ausente en las muestras de BC. Los pares de cebadores utilizados para mmoX también fueron adecuados para estudiar los humedales del sur de Texas en este estudio. Una temperatura de recocido de 55 °C36 produjo una banda brillante para mmoX en cPCR (Archivo Suplementario 1-Figura Suplementaria S8). Para verificar la integridad de la muestra, se utilizó el ARNr 16S como control positivo con una amplificación exitosa del ARNr 16S en todas las muestras durante la PCRc, lo que sugiere que las muestras no estaban contaminadas (Archivo Suplementario 1-Figura Suplementaria S9).
Para evaluar los cambios temporales y espaciales en la abundancia de genes, se realizó un análisis de qPCR en todos los genes objetivo utilizando conjuntos de cebadores optimizados identificados en cPCR. La qPCR de los genes pmoA2 y mmoX se llevó a cabo con los mismos cebadores utilizados en la cPCR. Sin embargo, para el gen pmoA1 se utilizó el par de cebadores A189-mb661, y para el gen mcrA se seleccionó el par de cebadores MLF-MLR, ya que ambos produjeron buenos resultados en la cPCR. También se consideraron las diferencias estacionales y la variación de la salinidad en los humedales del sur de Texas, ya que podrían influir en la dinámica de la comunidad microbiana. Como se muestra en la Figura 3 y la Figura 4, hubo una diferencia en la abundancia de genes de metanógenos y metanótrofos entre dos períodos de muestreo diferentes. Si bien no hubo variación estacional de la salinidad en los humedales costeros de agua dulce, se observó una diferencia estacional en la salinidad en los humedales costeros de agua salada39, lo que podría explicar la variación en la abundancia de metanógeno y metanótrofo. La Figura 3 muestra que durante la estación fría, el mmoX fue altamente abundante en todos estos humedales, con la mayor abundancia de 1.46 × 104 copias/μL. Había un pequeño porcentaje de pmoA2 en una estación de LB, RV y BC. Sin embargo, la qPCR no pudo detectar pmoA1 y mcrA debido a la baja abundancia durante la estación fría. En contraste, pmoA1 fue mayormente abundante en la estación cálida con la mayor abundancia de 2.44 × 103 copias/μL (Figura 4). El mmoX y el mcrA también se detectaron en las muestras de sedimento superficial de LB (dentro de los 50 cm superiores), mostrando la abundancia máxima de 1.92 × 102 copias/μL y 2.04 × 102 copias/μL, respectivamente, a pesar de su abundancia relativamente baja durante la estación cálida. Curiosamente, durante la estación cálida, no se detectó pmoA2 en absoluto, y ninguno de los genes objetivo se detectó en las muestras de BC, probablemente influenciado por factores ambientales únicos de este humedal de agua salada39 durante la estación cálida en el sur de Texas.
Este estudio demuestra distintas variaciones espaciales y estacionales en la distribución de los genes relacionados con el ciclo de CH4 en los humedales costeros. Específicamente, los genes mcrA se detectaron solo en la muestra de LB de agua dulce, lo que sugiere una preferencia de hábitat. Los genes mmoX y pmoA2 estuvieron presentes en todas las muestras en las estaciones frías, mientras que los pmoA1 se exhibieron en ambientes de agua dulce durante las estaciones cálidas. Estos resultados ponen de manifiesto la influencia de las condiciones ambientales y los cambios estacionales en las actividades microbianas relacionadas con el ciclo de CH4 . La visualización mejorada mediante interpolación de kriging, como se muestra en la Figura 3 y la Figura 4, avanzó nuestra comprensión de las actividades microbianas relacionadas con el ciclo del CH4 en los ecosistemas de humedales costeros.
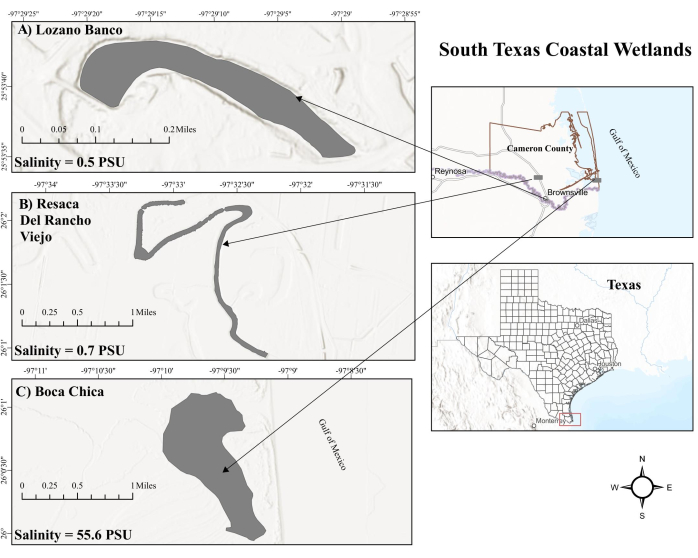
Figura 1: Mapa de los humedales costeros del sur de Texas. a) Lozano Banco. LB es un humedal costero de agua dulce. b) Resaca del Rancho Viejo. RV es también un humedal costero de agua dulce. (C) Humedal de Boca Chica. BC es un humedal costero de agua salada hipersalino influenciado por las mareas. Abreviaturas: LB = Lozano Banco; RV = Resaca Del Rancho Viejo; BC = Boca Chica; PSU = unidades prácticas de salinidad. Haga clic aquí para ver una versión más grande de esta figura.
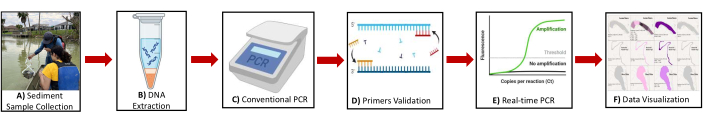
Figura 2: Diagrama esquemático que muestra la descripción general del protocolo. La ilustración de los pasos clave incluye: (A) Recolección de muestras de sedimentos, (B) Extracción de ADN, (C) PCR convencional, (D) Validación del cebador, (E) PCR en tiempo real, (F) Visualización de datos para identificar genes biomarcadores de metanógeno y metanótrofos. Haga clic aquí para ver una versión más grande de esta figura.
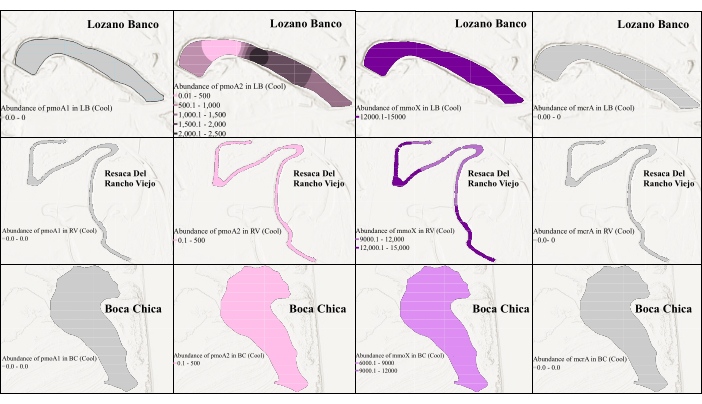
Figura 3: Distribución espacial de genes biomarcadores de metanógeno y metanótrofos en estación fría. La figura muestra la abundancia de mmoX durante la estación fría. Aquí, el color gris indica que la qPCR no pudo detectar el gen debido a la baja abundancia. Haga clic aquí para ver una versión más grande de esta figura.

Figura 4: Distribución espacial de los genes biomarcadores de metanógeno y metanótrofos en estación cálida. Aquí, el color gris representa que la qPCR no pudo detectar el gen debido a su baja abundancia. La figura muestra la abundancia de pmoA1 durante la estación cálida en Lozano Banco y Resaca Del Rancho Viejo, mientras que no se detectó abundancia en Boca Chica. Haga clic aquí para ver una versión más grande de esta figura.
| Nombre del cebador | Gen | Temperatura de recocido (°C) en este estudio | Longitud del producto (pb) | Secuencia (5'-3') | Referencia | |
| 1055F | ARNr 16S | 55 | 337 | ATGGCTGTCGTCAGCT | (Harms et al., 2003) | |
| 1392R | ACGGGCGGTGTGTAC | |||||
| A189F | pmoA1 | 55 | 525 | GGNGACTGGGACTTCTGG | (Holmes et al., 1995) | |
| A682R | GAASGCNGAGAAGAASGC | |||||
| A189F | 56 | 470 | GGNGACTGGGACTTCTGG | (Costello y Lidstrom, 1999) | ||
| mb661R | CCG GMG CAA CGT CYT TACC | |||||
| 206 y ss | pmoA2 | 60 | 438 | GGNGACTGGGACTTCTG GATCGACTTCAAGGATCG | (Tchawa Yimga et al., 2003) | |
| 703b | GAASGCNGAGAAGAASGC GGCGACCGGAACGACGT | |||||
| 536F | mmoX | 55 | 362 | CGCTGTGGAAGGGCATGAAGCG | (Fuse et al., 1998) | |
| 898R | GCTCGACCTTGAACTTGGAGCC | |||||
| MCRf | mcrA | 46 | ~490 | TAYGAYCARATHTGGYT | (Springer et al., 1995) | |
| MCRr | ACRTTCATNGCRTARTT | |||||
| ME1F | 50 | 763 | GCMATGCARATHGGWATGTC | (Hales et al., 1996) | ||
| ME2R | TCATKGCRTAGTTDGGRTAGT | |||||
| MLF | 55 | ~470 | GGTGGTGTMGGATTCA CACARTAYGCWACAGC | (Luton et al., 2002) | ||
| MLR | TTCATTGCRTAGTTWGGRTAGTT | |||||
| McrA 159F | 62 | 186 | AAAGTGCGGAGCAGCAATCACC | (Vaksmaa et al., 2017) | ||
| McrA 345R | TCGTCCCATTCCTGCTGCATTGC | |||||
Tabla 1: Conjuntos de cebadores utilizados en este estudio para detectar genes funcionales relacionados con el ciclo del metano. Todos estos pares de cebadores se utilizaron para la PCR convencional y, finalmente, los audaces se configuraron para detectar genes relacionados con el metanógeno y los metanótrofos en los humedales costeros del sur de Texas.
| Gen | Muestras | ||||||||
| Lozano Banco | Resaca Del Rancho Viejo | Boca Chica | |||||||
| LB-1 | RV-1 | RV-2 | M1 | M2 | B1 | B2 | MG1 | MG2 | |
| pmoA1 | - | + | + | - | - | - | - | - | - |
| pmoA2 | + | + | + | - | + | - | - | + | - |
| mmoX | + | - | + | - | - | - | - | - | - |
| mcrA | + | + | + | - | - | - | - | - | - |
Tabla 2: Detección de genes en tres tipos diferentes de cuerpos de agua en la estación fría mediante PCR convencional. '+', presente; '-', ausente. Abreviaturas: LB = Lozano Banco, RV = Resaca del Rancho Viejo, M = Barro, B = Batis, MG = Manglar.
Archivo complementario 1: Imágenes de electroforesis en gel para los genes mcrA, pmoA1, pmoA2, mmoX y 16S rRNA. Haga clic aquí para descargar este archivo.
Discusión
Se reconoce que los humedales costeros contribuyen significativamente al metano atmosférico, un importante gas de efecto invernadero40. Aunque se han realizado estudios sobre el flujo de metano y los metanógenos en los humedales41,42,43, se sabe poco sobre cómo operan los metanótrofos en diferentes entornos o bajo diversas prácticas de manejo, especialmente en humedales con niveles de agua fluctuantes44. Además, la presencia generalizada de inhibidores como los ácidos húmicos en el ADNe complica los procesos de extracción y amplificación45, lo que puede dar lugar a resultados falsos negativos46. Este desafío se complica aún más por la eficiencia variable de los cebadores que trabajan con eDNA, lo que sugiere la necesidad de una validación sistemática en este tipo de estudios47. Por lo tanto, esta investigación establece una metodología fundamental para comprender el ciclo del metano en los humedales costeros del sur de Texas, que no han sido suficientemente estudiados. Al centrarse en el desarrollo y la validación de técnicas de PCR, este estudio avanza en la detección de genes clave del ciclo del metano en estos ecosistemas dinámicos.
Uno de los aspectos críticos de esta investigación es la visualización de genes microbianos relacionados con el ciclo del metano, que desempeña un papel crucial en la observación de los patrones de distribución espacial de estos genes a lo largo de diferentes estaciones y gradientes de salinidad. Dicha visualización no es solo una herramienta complementaria; Es esencial para identificar áreas de presencia significativa de microbios metanogénicos y metanotróficos que indiquen que podría haber un posible ciclo del metano, mejorando así nuestra comprensión de las condiciones ambientales que influyen en estos procesos.
En este estudio, se especificó la metodología para la identificación de genes marcadores de metanógeno y metanótrofo como pmoA1, pmoA2, mmoX y mcrA mediante el uso de cPCR y qPCR. Se utilizaron cuatro pares de cebadores dirigidos al gen mcrA y dos pares dirigidos al gen pmoA1 , y solo un par de cada conjunto detectó eficazmente estos genes tanto en PCR convencional como en tiempo real. En un estudio anterior se utilizaron tres pares de cebadores para detectar pmoA1; entre ellos, los cebadores A189-A682 no pudieron detectar la diversidad de metanótrofos. En cambio, amplificaron principalmente el gen amoA y produjeron productos de PCR inespecíficos48. Ese estudio encontró que el par de cebadores utilizado fue efectivo para detectar el gen pmoA1 solo en entornos con una alta población de metanótrofos. Además, la mayor abundancia de metanótrofos se obtuvo utilizando los pares de cebadores A189-mb661 en ese estudio, lo que indica que estos cebadores son adecuados para los ecosistemas de agua dulce48. Por lo tanto, estos pares de cebadores se utilizaron para la qPCR como análisis adicional.
Entre los cuatro pares de cebadores utilizados para mcrA, los cebadores ML fueron capaces de amplificar las especies de metanógeno en todos los entornos49. Los cebadores ME tienen un rango de amplificación estrecho; No pueden detectar todo tipo de especies de metanógeno en diferentes tipos de entornos naturales. Los cebadores MCR tienen un alto nivel de degeneración, lo que los hace sensibles a la temperatura de recocido de PCR. Esta degeneración les permite unirse a una amplia gama de secuencias objetivo, a menudo acomodando múltiples desajustes. Sin embargo, esto también indica que un pequeño cambio en la temperatura de recocido puede influir significativamente en su eficiencia de unión y especificidad, lo que conduce a una amplificación insuficiente y, en consecuencia, a resultados falsos negativos para las especies de metanógeno49,50.
Por otro lado, en la comunidad de metanógenos de baja diversidad, durante las últimas etapas de la PCR, la alta acumulación de productos de PCR conduce al recocido de la plantilla e impide que los cebadores se unan, lo que tampoco detecta las bandas deseadas49. Curiosamente, los pares de cebadores McrA 159F-McrA 345R se diseñaron específicamente para identificar el metanotrófico anaeróbico dependiente de nitratos Candidatus M. nitroreducens archaea51, y este estudio no pudo detectar metanótrofos anaeróbicos utilizando estos pares de cebadores. Sin embargo, la oxidación anaeróbica del metano (AOM) puede acoplarse a la reducción de aceptores de electrones alternativos como el hierro (Fe), el manganeso (Mn) y el sulfato (SO42-)52, lo que podría explicar la incapacidad de detectar ANME utilizando este método. Además, podría deberse a las condiciones ambientales específicas del área de estudio. Por lo tanto, para la qPCR, se utilizaron cebadores ML para detectar la abundancia de metanógeno, ya que proporcionaron una amplificación confiable incluso en condiciones de baja diversidad comunitaria49.
Además, los inhibidores de la PCR, como los ácidos húmicos o el ADN de especies no objetivo, están ampliamente presentes en las muestras ambientales, lo que puede reducir la eficiencia de la PCR53. Dado que la alta concentración de ADN no pudo detectar ningún gen mediante cPCR, todas las muestras de ADN se diluyeron antes de ejecutar la cPCR para reducir el efecto de los inhibidores de la PCR54. El gen 16S rRNA, como marcador universal para bacterias y arqueas, proporcionó una línea de base sólida para validar nuestras condiciones de PCR y confirmar la integridad del ADN en todas las muestras 55,56. Sin embargo, las áreas de agua salada son reconocidas por tener mayores concentraciones de inhibidores de PCR46. Además, la salinidad influye indirectamente en las emisiones deCH4 en los humedales costeros, ya que está asociada a concentraciones de sulfato. Una mayor salinidad en los humedales de agua salada se correlaciona con niveles elevados de sulfato que suprimen la producción de metano debido a la competencia entre las bacterias reductoras de sulfato y los metanógenos, mientras que las concentraciones más bajas de sulfato en los humedales de agua dulce resultan en mayores emisiones de metano11,41. Además, las diferencias en las emisiones de CH4 entre los humedales de agua dulce y salada también están influenciadas por sus conexiones de marea con el océano57, lo que también podría explicar la detección infructuosa de genes relacionados con el ciclo del metano mediante cPCR en la Columbia Británica. En este estudio, la cPCR detectó con éxito todos los genes objetivo en las muestras de LB y RV, pero no pudo detectar la presencia de estos genes en las muestras de BC, siendo el gen pmoA2 la única excepción. Además, la qPCR fue capaz de detectar mmoX en todas las muestras, mientras que la cPCR no pudo detectarla en BC. Esto indica que la qPCR muestra una mayor resiliencia a los inhibidores de la PCR en comparación con la cPCR, atribuible a su mayor sensibilidad en los mecanismos de detección58. Otra razón es que la qPCR identifica los productos de PCR durante la fase exponencial del ciclo de PCR, mientras que la cPCR los detecta en la fase de meseta. Esta distinción permite que la qPCR exhiba una susceptibilidad reducida a la degradación del producto durante ciclos de reacción avanzados, donde los reactivos se agotan47.
Este estudio estableció una técnica de PCR para identificar genes relacionados con el ciclo del metano en los humedales costeros del sur de Texas para superar los desafíos asociados con los inhibidores de PCR y las eficiencias variables de los cebadores. Se utilizaron cuatro pares de cebadores dirigidos al gen mcrA y dos pares dirigidos al gen pmoA1 , y solo un par de cada conjunto detectó eficazmente estos genes tanto en PCR convencional como en tiempo real. Este estudio concluye el conjunto de pares de cebadores para los genes relacionados con el ciclo del metano, incluidos a189-mb6661 para pmoA1, 206f-703b para pmoA2, 536F-898R para mmoX y ML para mcrA. Esto sugiere la importancia de la selección adecuada de cebadores para detectar genes relacionados con el ciclo del metano en los humedales costeros, con los del sur de Texas como ejemplos. Además, el estudio mostró que el mmoX era más abundante durante la estación fría, mientras que el pmoA1 era abundante durante la estación cálida.
Por lo tanto, los resultados de este estudio proporcionan la base para la comprensión científica de los procesos microbianos involucrados en el ciclo del metano, destacando la influencia de los factores ambientales en las variaciones estacionales y espaciales en los humedales costeros del sur de Texas. Sobre la base de estos hallazgos, los estudios futuros apuntan a validar aún más este enfoque en otros ecosistemas, como los arrozales y los humedales de agua dulce, para aumentar la robustez y la generalización. Además, estos estudios también incluirán análisis geoquímicos, como las concentraciones de metano en los sedimentos y el sulfuro de agua de poro, y técnicas moleculares como la metagenómica y la transcriptómica, que se basarán en este trabajo preliminar para proporcionar una comprensión más completa de la dinámica del ciclo del metano.
Divulgaciones
Los autores no tienen conflictos de intereses que declarar.
Agradecimientos
Agradecemos a los miembros de C-REAL por su ayuda en la observación de campo y los análisis de laboratorio.
Materiales
| Name | Company | Catalog Number | Comments |
| 0.2 mL PCR tubes | ThermoFisher Scientific | AB0620 | https://www.thermofisher.com/order/catalog/product/AB0620?SID=srch-srp-AB0620 |
| 0.5 mL PCR Tubes | Promega | E4941 | https://www.promega.com/products/biochemicals-and-labware/tips-and-accessories/0_5ml-pcr-tubes/?catNum=E4941 |
| 10 μL tips | ThermoFisher Scientific | 05-408-187 | Fisherbrand SureGrip Pipet Tip Racked or Reload System Tips Natural; 10μL; | Fisher Scientific |
| 15 mL centrifuge tube | ThermoFisher Scientific | 14-959-53A | https://www.fishersci.com/shop/products/falcon-15ml-conical-centrifuge-tubes-5/p-193301 |
| 200 μL tips | ThermoFisher Scientific | 05-408-190 | Fisherbrand SureGrip Pipet Tip Racked or Reload System Tips Natural; 200μL; | Fisher Scientific |
| 1000 μL tips | ThermoFisher Scientific | 02-707-402 | https://www.fishersci.com/shop/products/sureone-micropoint-pipette-tips-specific-standard-fit/02707402?gclid=Cj0KCQiAp NW6BhD5ARIsACmEb kUsQ9Lu0YIq5i4vWege 17qPdtxIYZyvmJH1cDo ARuwereO1V4GLz9UaA lDREALw_wcB&ef_id=C j0KCQiApNW6BhD5ARI sACmEbkUsQ9Lu0YIq5i 4vWege17qPdtxIYZyvmJ H1cDoARuwereO1V4GLz 9UaAlDREALw_wcB:G:s &ppc_id=PLA_goog_2175 7693617_171052169911_02 707402__715434303113_1555 377385658230343&ev_chn=sh op&s_kwcid=AL!4428!3!71543430 3113!!!g!2366517300713!&gad_source=1 |
| Applied Biosystem Power SYBR Green Master Mix | ThermoFisher Scientific | 4368577 | https://www.thermofisher.com/order/catalog/product/4368577 |
| ArcGIS Pro | esri | https://www.esri.com/en-us/arcgis/products/arcgis-pro/overview?srsltid=AfmBOopatJ4 JvHJfscHRcAaDx0Jz5_Jrl8l5 vYkkBvfOqE-uNSsMghN1 | |
| CFX Duet Real-Time PCR system | Bio-Rad | 12016265 | https://www.bio-rad.com/en-us/product/cfx-duet-real-time-pcr-system?ID=97722926-9ed9-16a4-1d83-c92f587e427a |
| Corning Lambda plus single channel pipettor volume 0.5-10 μL | Sigma-Aldrich | CLS4071-1EA | https://www.sigmaaldrich.com/US/en/product/sigma/cls4071 |
| Corning Lambda plus single channel pipettor volume 100-1000 μL | Sigma-Aldrich | CLS4075-1EA | https://www.sigmaaldrich.com/US/en/product/sigma/cls4075 |
| Corning Lambda plus single channel pipettor volume 20-200 μL | Sigma-Aldrich | CLS4074-1EA | https://www.sigmaaldrich.com/US/en/product/sigma/cls4074 |
| FastDNA spin kit for soil | MP Biomedical | 116560200-CF | https://www.mpbio.com/us/116560000-fastdna-spin-kit-for-soil-samp-cf?srsltid=AfmBOoqOxxGilzY3IHNIZR ajegGTr9MoX1oMZUh 3dcbJqe0UvvukY128 |
| Gene copy calculator | Science Primer | https://scienceprimer.com/copy-number-calculator-for-realtime-pcr . | |
| High speed benchtop centrifuge | ThermoFisher Scientific | 75004241 | https://newlifescientific.com/products/thermo-scientific-sorvall-st16-high-speed-benchtop-centrifuge-75004241?gad_source=1&gclid=Cj0KCQiApN W6BhD5ARIsACmEbkVC_-cCIN9j 20TvYq8iDsBlUR5cPK_1_wN OBEcjMdv-CYVoGCfeOLYaAv enEALw_wcB |
| High speed microcentrifuge | VWR | 75838-336 | https://us.vwr.com/store/product/20546590/null |
| Lysing Matrix E tube | glass bead/ceramic sphere-containing tube | ||
| Microcentrifuge tube | ThermoFisher Scientific | 02-681-320 | https://www.fishersci.com/shop/products/fisherbrand-low-retention-microcentrifuge-tubes-8/02681320?gclid=Cj0KCQiAp NW6BhD5ARIsACm EbkWbG4_o3oUiGk HJPU-_31-CuexDwQ fmWPnfyhBOf2BHXsy K3fFW1toaAgJbEALw_ wcB&ef_id=Cj0KCQiAp NW6BhD5ARIsACmEb kWbG4_o3oUiGkHJPU- _31-CuexDwQfmWPnfy hBOf2BHXsyK3fFW1toa AgJbEALw_wcB:G:s&ppc _id=PLA_goog_21757693 617_171052169911_0268 1320__715434303113_10 349826094968484711&ev _chn=shop&s_kwcid=AL!4 428!3!715434303113!!!g!23 66517300713!&gad_source=1 |
| PCR Master mix | Promega | M7502 | https://www.promega.com/products/pcr/taq-polymerase/master-mix-pcr/?catNum=M7502 |
| Quantiflour ONE dsDNA system | Promega | E4871 | https://www.promega.com/products/rna-analysis/dna-and-rna-quantitation/quantifluor-one-dsdna-system/?gad_source=1&gbraid=0AAAAAD _rg189yJTY3cxeVqMdu8RPx10 Ma&gclid=CjwKCAjwxNW2BhAk EiwA24Cm9FUgViPNyWq7UfZL VeeoroLAZ5JIP6w07RGK_4D0w oZgAqf-G1XTmxoCxm8QAvD_B wE&catNum=E4871 |
| Quantus Fluorometer | Promega | E6150 | https://www.promega.com/products/microplate-readers-fluorometers-luminometers/fluorometers/quantus-fluorometer/?catNum=E6150 |
| YSI Pro 2030 | YSI a xylem brand | 603174 | https://www.ysi.com/product/id-p2030/pro2030-kits |
Referencias
- Xu, T., et al. Wetlands of international importance: Status, threats, and future protection. Int J Environ Res Public Health. 16 (10), 1818 (2019).
- Corn, M. L. . Deepwater Horizon oil spill: coastal wetland and wildlife impacts and response. , (2010).
- Hendriks, I. E., Sintes, T., Bouma, T. J., Duarte, C. M. Experimental assessment and modeling evaluation of the effects of the seagrass Posidonia oceanica on flow and particle trapping. Marine Ecology Progress Series. 356, 163-173 (2008).
- Krause, S. J. E., Treude, T. Deciphering cryptic methane cycling: Coupling of methylotrophic methanogenesis and anaerobic oxidation of methane in hypersaline coastal wetland sediment. Geochimica et Cosmochimica Acta. 302, 160-174 (2021).
- Reddy, K. R., DeLaune, R. D., Inglett, P. W. . Biogeochemistry of Wetlands: Science and Applications. , (2022).
- La, W., et al. Sulfate concentrations affect sulfate reduction pathways and methane consumption in coastal wetlands. Water Research. 217, 118441 (2022).
- Derwent, R. G. Global warming potential (GWP) for methane: Monte Carlo analysis of the uncertainties in Global Tropospheric Model predictions. Atmosphere. 11 (5), 486 (2020).
- Potter, C., et al. Methane emissions from natural wetlands in the United States: Satellite-derived estimation based on ecosystem carbon cycling. Earth Interactions. 10 (22), 1-12 (2006).
- . Understanding global warming potentials Available from: https://www.epa.gov/ghgemissions/understanding-global-warming-potentials (2024)
- Wallenius, A. J., Dalcin Martins, P., Slomp, C. P., Jetten, M. S. M. Anthropogenic and environmental constraints on the microbial methane cycle in coastal sediments. Front Microbiol. 12, 631621 (2021).
- Qu, Y., et al. Salinity causes differences in stratigraphic methane sources and sinks. Environmental Science and Ecotechnology. 19, 100334 (2024).
- Vizza, C., West, W. E., Jones, S. E., Hart, J. A., Lamberti, G. A. Regulators of coastal wetland methane production and responses to simulated global change. Biogeosciences. 14 (2), 431-446 (2017).
- van Dijk, G., et al. Salinization lowers nutrient availability in formerly brackish freshwater wetlands; unexpected results from a long-term field experiment. Biogeochemistry. 143 (1), 67-83 (2019).
- Aronson, E., Allison, S., Helliker, B. R. Environmental impacts on the diversity of methane-cycling microbes and their resultant function. Front Microbiol. 4, 225 (2013).
- Reeburgh, W. S. Oceanic methane biogeochemistry. Chem Rev. 107 (2), 486-513 (2007).
- Thauer, R. K. Biochemistry of methanogenesis: a tribute to Marjory Stephenson. Marjory Stephenson Prize Lecture. Microbiology (Reading). 144 (Pt 9), 2377-2406 (1998).
- Thauer, R. K. Anaerobic oxidation of methane with sulfate: on the reversibility of the reactions that are catalyzed by enzymes also involved in methanogenesis from CO2. Curr Opin Microbiol. 14 (3), 292-299 (2011).
- Friedrich, M. W. Methyl-coenzyme M reductase genes: Unique functional markers for methanogenic and anaerobic methane-oxidizing Archaea. Methods in Enzymology. 397, 428-442 (2005).
- Reeburgh, W. S. Oceanic methane biogeochemistry. Chem Rev. 107 (2), 486-513 (2007).
- Rasigraf, O., Schmitt, J., Jetten, M. S. M., Lüke, C. Metagenomic potential for and diversity of N-cycle driving microorganisms in the Bothnian Sea sediment. Microbiologyopen. 6 (4), e00475 (2017).
- McDonald, I. R., Bodrossy, L., Chen, Y., Murrell, J. C. Molecular ecology techniques for the study of aerobic methanotrophs. Appl Environ Microbiol. 74 (5), 1305-1315 (2008).
- Tchawa Yimga, M., Dunfield, P. F., Ricke, P., Heyer, J., Liesack, W. Wide distribution of a novel pmoA-like gene copy among type II methanotrophs, and its expression in Methylocystis strain SC2. Appl Environ Microbiol. 69 (9), 5593-5602 (2003).
- Knief, C. Diversity and habitat preferences of cultivated and uncultivated aerobic methanotrophic bacteria evaluated based on pmoA as molecular marker. Front Microbiol. 6, 1346 (2015).
- Huang, I. -. S., et al. Preliminary assessment of microbial community structure of Wind-Tidal Flats in the Laguna Madre, Texas, USA. Biology. 9 (8), 183 (2020).
- Wilding, T. K., Brown, E., Collier, K. J. Identifying dissolved oxygen variability and stress in tidal freshwater streams of northern New Zealand. Environ Monit Assess. 184 (10), 6045-6060 (2012).
- Roy Chowdhury, T., Mitsch, W. J., Dick, R. P. Seasonal methanotrophy across a hydrological gradient in a freshwater wetland. Ecological Engineering. 72, 116-124 (2014).
- Sun, Q. -. Q., et al. Carbon dioxide and methane fluxes: Seasonal dynamics from inland riparian ecosystems, northeast China. Sci Total Environ. 465, 48-55 (2013).
- Lee, S., et al. Comparison and selection of conventional PCR primer sets for studies associated with nitrogen cycle microorganisms in surface soil. Appl Sci. 12 (20), 10314 (2022).
- Bae, K. -. S., et al. Development of diagnostic systems for wide range and highly sensitive detection of two waterborne hepatitis viruses from groundwater using the conventional reverse transcription nested PCR assay. J Virol Methods. 299, 114344 (2022).
- Lecusay, D. . Assessment and Monitoring of Deltaic Wetlands and Fluvial Systems: Refining and Validating a Multimetric Index of Resaca Ecosystem Health. , (2021).
- . FastDNA SPIN Kit for Soil (Cat No. 116560200) Available from: https://www.mpbio.com/media/productattachment/LS082019-EN-FastDNA-SPIN-Kit-for-Soil-116560200-Manual.pdf (2025)
- Luton, P. E., Wayne, J. M., Sharp, R. J., Riley, P. W. The mcrA gene as an alternative to 16S rRNA in the phylogenetic analysis of methanogen populations in landfill. Microbiology (Reading, England). 148 (Pt 11), 3521-3530 (2002).
- Changsoo, L., Jaai, K., Seung Gu, S., Seokhwan, H. Absolute and relative QPCR quantification of plasmid copy number in Escherichia coli. J Biotechnol. 123 (3), 273-280 (2006).
- Harms, G., et al. Real-time PCR quantification of nitrifying bacteria in a municipal wastewater treatment plant. Environ Sci Technol. 37 (2), 343-351 (2003).
- Holmes, A. J., Costello, A., Lidstrom, M. E., Murrell, J. C. Evidence that particulate methane monooxygenase and ammonia monooxygenase may be evolutionarily related. FEMS Microbiol Lett. 132 (3), 203-208 (1995).
- Fuse, H., et al. Oxidation of trichloroethylene and dimethyl sulfide by a marine Methylomicrobium strain containing soluble methane monooxygenase. Biosci Biotechnol Biochem. 62 (10), 1925-1931 (1998).
- Springer, E., Sachs, M. S., Woese, C. R., Boone, D. R. Partial gene sequences for the A subunit of methyl-coenzyme M reductase (mcrI) as a phylogenetic tool for the family Methanosarcinaceae. Int J Syst Bacteriol. 45 (3), 554-559 (1995).
- Costello, A. M., Lidstrom, M. E. Molecular characterization of functional and phylogenetic genes from natural populations of methanotrophs in lake sediments. Appl Environ Microbiol. 65 (11), 5066-5074 (1999).
- Flores, E. A. . Effects of Nutrient Enrichment on Mangrove and Saltmarsh Habitats. , (2022).
- Minjie, H., Jordi, S., Xianyu, Y., Josep, P., Chuan, T. Patterns and environmental drivers of greenhouse gas fluxes in the coastal wetlands of China: A systematic review and synthesis. Environ Res. 186, 109576 (2020).
- Bridgham, S. D., Cadillo-Quiroz, H., Keller, J. K., Zhuang, Q. Methane emissions from wetlands: biogeochemical, microbial, and modeling perspectives from local to global scales. Glob Chang Biol. 19 (5), 1325-1346 (2013).
- Liu, D. Y., Ding, W. X., Jia, Z. J., Cai, Z. C. Relation between methanogenic archaea and methane production potential in selected natural wetland ecosystems across China. Biogeosciences. 8 (2), 329-338 (2011).
- Ke, Z., et al. Methane emissions and methanogenic community investigation from constructed wetlands in Chengdu City. Urban Climate. 39, 100956 (2021).
- Chowdhury, T. R., Dick, R. P. Ecology of aerobic methanotrophs in controlling methane fluxes from wetlands. Applied Soil Ecology. 65, 8-22 (2013).
- Maja, S., et al. Humic substances cause fluorescence inhibition in real-time polymerase chain reaction. Anal Biochem. 487, 30-37 (2015).
- Sanches, T. M., Schreier, A. D. Optimizing an eDNA protocol for estuarine environments: Balancing sensitivity, cost and time. PLOS ONE. 15 (5), e0233522 (2020).
- Xia, Z., et al. Conventional versus real-time quantitative PCR for rare species detection. Ecol Evol. 8 (23), 11799-11807 (2018).
- Bourne, D. G., McDonald, I. R., Murrell, J. C. Comparison of pmoA PCR primer sets as tools for investigating methanotroph diversity in three Danish soils. Appl Environ Microbiol. 67 (9), 3802-3809 (2001).
- Juottonen, H., Galand, P. E., Yrjala, K. Detection of methanogenic Archaea in peat: comparison of PCR primers targeting the mcrA gene. Res Microbiol. 157 (10), 914-921 (2006).
- Lueders, T., Friedrich, M. W. Evaluation of PCR amplification bias by terminal restriction fragment length polymorphism analysis of small-subunit rRNA and mcrA genes by using defined template mixtures of methanogenic pure cultures and soil DNA extracts. Appl Environ Microbiol. 69 (1), 320-326 (2003).
- Vaksmaa, A., Jetten, M. S., Ettwig, K. F., Luke, C. McrA primers for the detection and quantification of the anaerobic archaeal methanotroph 'Candidatus Methanoperedens nitroreducens'. Appl Microbiol Biotechnol. 101 (4), 1631-1641 (2017).
- Ren, G., et al. Electron acceptors for anaerobic oxidation of methane drive microbial community structure and diversity in mud volcanoes. Environ Microbiol. 20 (7), 2370-2385 (2018).
- Goldberg, C. S., et al. Critical considerations for the application of environmental DNA methods to detect aquatic species. Methods in Ecology and Evolution. 7 (11), 1299-1307 (2016).
- McKee, A. M., Spear, S. F., Pierson, T. W. The effect of dilution and the use of a post-extraction nucleic acid purification column on the accuracy, precision, and inhibition of environmental DNA samples. Biological Conservation. 183, 70-76 (2015).
- Gohl, D. M., et al. Systematic improvement of amplicon marker gene methods for increased accuracy in microbiome studies. Nat Biotechnol. 34 (9), 942-949 (2016).
- Ballarini, A., Segata, N., Huttenhower, C., Jousson, O. Simultaneous quantification of multiple bacteria by the BactoChip microarray designed to target species-specific marker genes. PLOS ONE. 8 (2), e55764 (2013).
- Le Mer, J., Roger, P. Production, oxidation, emission and consumption of methane by soils: A review. Eur J Soil Biol. 37 (1), 25-50 (2001).
- Smith, C. J., Osborn, A. M. Advantages and limitations of quantitative PCR (Q-PCR)-based approaches in microbial ecology. FEMS Microbiol Ecol. 67 (1), 6-20 (2009).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados