Method Article
可视化沿海湿地中的甲烷循环微生物动力学
摘要
该协议检测南德克萨斯州沿海湿地中的关键甲烷循环基因,并可视化它们的空间分布,以增强对甲烷调节及其在这些动态生态系统中的环境影响的理解。
摘要
沿海湿地是甲烷最大的生物来源,产甲烷菌将有机物转化为甲烷,而嗜甲烷菌氧化甲烷,从而在调节甲烷循环中起关键作用。由于气候变化,德克萨斯州南部的湿地经常受到天气事件、盐度水平波动和人为活动的影响,这些都会影响甲烷循环。尽管这些过程具有生态重要性,但南德克萨斯州沿海湿地的甲烷循环仍未得到充分探索。为了解决这一差距,我们开发并优化了一种检测与产甲烷菌和嗜甲烷菌相关的基因的方法,包括作为产甲烷菌生物标志物的 mcrA 和作为产甲烷菌生物标志物的 pmoA1、 pmoA2 和 mmoX 。此外,本研究旨在利用地理信息系统 (GIS) 软件 ArcGIS Pro 可视化产甲烷原和甲烷营养素丰度的时空分布模式。将这些分子技术与先进的地理空间可视化相结合,为了解南德克萨斯州湿地中产甲烷菌和嗜甲烷菌群落的时空分布提供了重要见解。因此,本研究中建立的方法为绘制湿地中的微生物动态图提供了一个强大的框架,增强了我们对不同环境条件下甲烷循环的理解,并支持更广泛的生态和环境变化研究。
引言
沿海湿地是重要的生态系统,通过碳封存、蒸发蒸腾和甲烷 (CH4) 排放等过程为气候调节、生物多样性保护和水资源管理做出贡献1。这些生态系统,包括淡水湿地和咸水湿地2,生产力很高,是吸收二氧化碳 (CO2) 和捕获陆地和海洋环境中有机物的关键区域 3,4。这些湿地内的动态相互作用刺激了微生物 CH4 的产生和消费5,使它们成为 CH46 的最大天然来源之一。作为第二重要的温室气体,CH4 的全球变暖潜能值约为 CO24、7、8、9 的 27-30 倍,因此在气候变化时代,研究沿海湿地的 CH4 排放量至关重要。CH4 的排放受各种环境因素的影响,尤其是盐度,在微生物过程中起着至关重要的作用10。淡水湿地的硫酸盐含量较低,因此对大气甲烷的贡献很大,这促进了微生物 CH4 的产生,而咸水湿地由于硫酸盐浓度较高,通常倾向于排放较少的 CH4 11,12,13。
沿海湿地的 CH4 排放通常由两组微生物控制,称为产甲烷菌和甲烷菌14。产甲烷菌通过称为甲烷生成15 的过程分解甲酸盐、乙酸盐、氢或甲基化化合物等底物,在缺氧沉积物中产生 CH4。该途径中的重要酶是甲基辅酶 M 还原酶 (MCR),因为它催化甲烷生成的最后和限速步骤 15,16,17。编码 MCR 的 α 亚基的 mcrA 基因是一种功能标记物,可以在所有产甲烷古细菌中找到18。此外,在沿海湿地中,硫酸盐-甲烷过渡区 (SMTZ) 在产甲烷区上方形成,甲烷向上扩散,硫酸盐向下移动汇聚并耗尽19。在该区域内,厌氧甲烷营养古细菌 (ANME) 使用 MCR 酶将甲烷氧化成二氧化碳,而硫酸盐还原细菌 (SRB) 将硫酸盐还原为硫化物。SRB 在氢和乙酸盐方面优于产甲烷菌,限制了甲烷的产生,直到硫酸盐耗尽16,17。
相比之下,需氧嗜甲烷菌在好氧环境中利用不同形式的甲烷单加氧酶 (MMO)20 氧化 CH4。这些包括颗粒甲烷单加氧酶 (pMMO),一种嵌入胞浆内膜的含铜酶,以及可溶性甲烷单加氧酶 (sMMO),一种在细胞质中发现的含铁酶。然而,对于 pMMO,有三个基因纵子 pmoCAB21;其中,pmoA 基因是所有嗜甲烷菌中最保守的。pmoA 有两种不同的生物标志物基因:pmoA1 和 pmoA222。此外,为了全面了解嗜甲烷菌,mmoX 基因被用作分子生物学中的工具,以识别含有 sMMO 的嗜甲烷菌23。产甲烷菌和好氧甲烷菌的代谢途径和环境要求的这种差异突出了沿海湿地生态系统中调节甲烷循环的复杂微生物相互作用。
博卡奇卡 (BC) 湿地是德克萨斯州南部一个多产的咸水环境,受到墨西哥湾 (GOM) 的潮汐影响,导致地表盐度水平变化,特别是由于靠近高盐度的马德雷湖24 号。这种潮汐作用在涨潮和退潮之间交替,导致氧气水平波动25 ,这可能会改变沉积物中的产甲烷菌和产甲烷菌活性26。相比之下,沿海淡水湿地被认为是 CH4 通量的重要热点27。德克萨斯州南部的沿海淡水湿地,包括 Resaca Del Rancho Viejo (RV) 和 Lozano Banco (LB),远离 GOM 的潮汐效应,具有独特的水文管理。RV 在低水位时经历由河水补充的脉冲流,而 LB 在没有这种补充的情况下作为离线流系统运行。此外,RV 和 LB 保持较低的盐度水平,这是由于人工抽取的淡水流量大,并且分别是一个牛轭湖。不同的环境因素会显着影响南德克萨斯州沿海湿地的甲烷循环。然而,南德克萨斯州沿海湿地的甲烷循环仍然是一个尚未彻底调查的领域。
聚合酶链反应 (PCR) 和实时 PCR(也称为定量 PCR [qPCR])是检测和定量环境样本中特定基因相对丰度的基本且广泛使用的技术。这些技术特异性扩增 DNA 的目标区域,以指示 CH4 循环相关基因的存在和相对数量,从而提供潜在甲烷循环的指标。然而,PCR 引物组的可用性和有效性可能受到提取的环境 DNA 中各种抑制因子的限制,并受到环境类型的影响28,29。因此,本研究主要建立了一种最佳的 PCR 方法,用于检测南德克萨斯州沿海湿地中 CH4 循环相关基因的存在(图 1),然后可视化它们在这些生态系统中的定量相对丰度。这项研究的结果可以应用于其他沿海地区,以增强对不同沿海生态系统中 CH4 循环和微生物动力学的理解。
研究方案
1. 样本采集
- 使用沉积物取样器或铲子收集沉积物样品。
注:样品是在 2023 年和 2024 年的凉爽季节(10 月至 2 月,平均气温为 20 °C)和温暖季节(4 月至 6 月,平均气温为 27 °C)从三个不同沿海湿地的两个站点收集的。从沿海淡水湿地收集样本时,使用沉积物抓取采样器(图 2),而受潮汐影响的沿海咸水湿地则使用铲子。 - 首先将采样器降低到浅水体中,让它在自重的作用下沉到沉积物表面(顶部 50 厘米以内),最大限度地减少对沉积物结构的干扰,如图 2 所示。
- 将其从深度通常为 60 厘米至 215 厘米30 的水柱中拉出,将沉积物样本转移到拉链袋中,并立即存放在冰柜中。
注:在进行下一个工作站之前,用去离子 (DI) 水清洁和清洗取样器。 - 将所有样品立即储存在实验室中-20 °C。
- 在每个采样点,使用多参数水质计原 位 测量地表水质量参数,例如盐度和温度。
注意:在每个工作站使用后,用去离子水(去离子水)冲洗探头。
2. 基因组 DNA 提取
- 在开始基因组 DNA 提取程序之前,在室温下解冻样品。
- 将大约 500 mg 沉淀物样品转移到 15 mL 试管中,并以 4,250 × g 离心 3 分钟以去除所有水分。
- 按照制造商的方案31 ,使用土壤DNA提取试剂盒提取基因组DNA,并立即储存在-20°C。
注意:进行更改是为了减少冗余和提高工作流程效率。- 将最多 500 毫克的土壤样品添加到含有玻璃珠/陶瓷球的试管中。
- 将 978 μL 磷酸钠缓冲液添加到含磁珠/球体的试管中的样品中。
- 在含磁珠/球体的试管中向样品中加入 122 μL 缓冲液裂解液,以溶解外部污染物。
- 使用珠磨机均质器以 5 倍的速度水平匀浆 20 秒,然后重复 2 倍。
注意:本研究中使用了珠磨机均质器,这就是调整速度的原因。 - 将混合物以 14,000 × g 离心 10 分钟 。
- 将上清液转移至干净的 2.0 mL 微量离心管中。
- 加入 250 μL 蛋白质沉淀溶液 (PPS),将溶解的核酸与细胞碎片和裂解基质分离。将试管倒置 10 次混合。
- 以 14,000 × g 离心 5 分钟以沉淀沉淀沉淀,去除细胞碎片和裂解基质。
- 将上清液转移至干净的 15 mL 微量离心管中。
- 将 1.0 mL 结合基质悬浮液添加到 15 mL 试管中的上清液中。
注:添加前摇动 Binding Matrix 悬浮液以重悬。 - 将试管放在旋转器上 2 分钟,让 DNA 与结合基质结合。
- 将所有试管放在架子上并孵育 3 分钟,以使结合基质沉淀。
- 3 分钟后,弃去 750 μL,并使用移液管将剩余的上清液与沉淀轻轻混合。
- 将 750 μL 混合物转移到 SPIN 过滤器中,并以 14,000 × g 离心 1 分钟。清空收集管并重复使用。对剩余的混合物重复此作。
- 加入 500 μL 制备好的洗涤液(加入适量的乙醇)以进一步溶解杂质。利用移液管吸头的液体力量轻轻重悬沉淀。
- 以 14,000 × g 离心 1 分钟以去除杂质。清空收集管并重复使用。
- 再次以 14,000 × g 离心 2 分钟,不添加任何东西。
- 用新的、干净的收集管更换收集管,并在室温下将 SPIN 过滤器风干 5 分钟。
- 加入 50 μL 游离 DNA 酶水 (DES),并以 14,000 × g 离心 1 分钟。
注:在该方案中,使用 50 μL DES 进行 DNA 洗脱,以确保提取的环境 DNA (eDNA) 的最佳回收率和稳定性。
3. DNA 定量
- 在 0.5 mL 试管中加入 1 μL 提取的 DNA 和 200 μL 荧光染料,并通过移液充分混合。
- 立即用铝箔包裹试管,使光线无法穿透并在室温下孵育 5 分钟。
- 按照制造商的方案,在 ONE DNA 浓度模式下使用荧光计测量 DNA 浓度。
4. 常规 PCR 检测 16S rRNA、pmoA1、pmoA2、mmoX 和 mcrA
- 在运行常规 PCR (cPCR) 之前,请在冰桶中解冻所有样品和试剂。
- 将所有提取的 eDNA 样品稀释至 10 ng/μL。
注意: 表 1 中给出了引物列表。 - 为每个样品制备 25 μL cPCR 反应混合物,包括 12.5 μL 2x PCR 预混液、0.5 μL 正向和反向引物 (10 μM)(引物列表见 表 1 )、1 μL 10 ng/μL eDNA 和 10.5 μL 无核酸酶水。
注:制备体积足以容纳一个额外样品的预混液,以最大限度减少移液错误。 - 按照方案进行 cPCR 反应,该方案包括在 95 °C 下初始变性 2 分钟,然后在 95 °C 下变性 45 秒,在 72 °C 下延伸 30 秒,最后在 72 °C 下延伸 5 m,不同引物的退火温度不同(参见表 1 用于不同基因的不同引物的退火温度)。
注:对于 mcrA 基因,ML 引物在前五个循环的退火和延伸步骤之间使用 0.1 °C/s 的缓慢升温速率显示所需的条带32。 - 在用溴化乙锭 (EtBr) 预染的琼脂糖凝胶中观察 cPCR 产物。
注:对于 50 bp 分子量标准,使用 2.5% 琼脂糖凝胶,对于 1 kb 分子量标准,使用 0.9% 凝胶。对于 2.5% 琼脂糖凝胶使用 1x TAE 缓冲液,对于 0.9% 琼脂糖凝胶使用 0.5x TAE 缓冲液。
5. 通过定量实时 PCR 检测 pmoA1 、 pmoA2 、 mmoX 和 mcrA
注:使用实时 PCR 系统通过 qPCR 观察到产甲烷菌和甲烷营养素靶向基因,如 pmoA1、 pmoA2、 mmoX 和 mcrA 丰度。
- 分别制备每个基因的标准品,以获得每个基因的标准曲线。
- 使用 cPCR 扩增目标基因,使用在每个基因的凝胶电泳过程中产生最亮条带的样品。遵循第 4 节中描述的方法和引物。
- 使用凝胶提取试剂盒纯化扩增的产物,并按照第 3 节中描述的方法测量 DNA 浓度,并储存在 -20 °C。
注:将纯化的标准品分装到单独的试管中以备进一步使用。 - 使用 qPCR 的拷贝数计算网站从测得的 DNA 浓度中计算基因拷贝数。
- 在运行 qPCR 之前,用不含核酸酶的水稀释计算出的标准品拷贝数,以制备 108 至 102 拷贝/μL 的每个标准品。
- 使用每个拷贝数的三个重复样本制备标准曲线,包括阴性对照(NTC,无模板 DNA)。
注:每条标准曲线的 R2 值大于 0.99。
- 为所有样品、标准品和 NTC 制备 20 μL qPCR 反应混合物。对所有样品一式三份进行 qPCR 分析。
- 在开始之前,将所有反应组分(包括 qPCR 预混液、引物、无核酸酶水、标准品和样品)放在冰架上。
- 为每个样品、标准品和 NTC 制备 20 μL qPCR 反应混合物,分别含有 10 μL SYBR Green 预混液、0.5 μL 每种 10 μM 正向和反向引物、8 μL 无核酸酶水和 1 μL 10 ng/μL 模板 DNA、标准品或 DI 水。
注:使用所示的引物对在常规 PCR 中产生 pmoA1 和 mcrA 基因的最佳结果进行 qPCR(参见 表 1)。为了提高准确性,请一式三份运行每个样品。以下步骤提供了一种制备一式三份样品的有效方法,每个样品的总体积为 60 μL。- 制备反应混合物,将预混液、特定基因的正向和反向引物以及无核酸酶的水混合在 2 mL 试管中,模板 DNA 除外。
注意:考虑到移液误差,准备反应混合物体积。例如,如果有 24 个样品和 8 个标准品,则计算 33 个反应的总体积,而不是 32 个反应,以最大程度地减少移液误差。在这种情况下,三次重复反应所需的总体积如下:990 μL 预混液(33 份样品 x 3 次重复 x 10 μL)、49.5 μL 正向引物(33 份样品 x 3 次重复 x 0.5 μL)、49.5 μL 反向引物(33 份样品 x 3 次重复 x 0.5 μL)和 792 μL 无核酸酶水(33 份样品 x 3 次重复 x 8 μL)。 - 根据样品数量和标准品准备 PCR 管。
- 将 57 μL 制备的反应混合物分配到每个 PCR 管中。
注:要对每个样品一式三份进行 qPCR,请为每个样品制备 57 μL 的总反应混合物体积(不包括模板 DNA、标准品或水)。对于一个样品,该体积将被平均分成三个孔,每个孔分配 19 μL。 - 向每个试管中加入 3 μL 模板 DNA、标准品或无核酸酶的水,轻轻敲击试管底部混合。
注:现在每个试管中为一个样品制备的反应混合物的总体积为 60 μL。
- 制备反应混合物,将预混液、特定基因的正向和反向引物以及无核酸酶的水混合在 2 mL 试管中,模板 DNA 除外。
- 将每管中 20 μL 制备的反应混合物分装到 96 孔 qPCR 板的指定孔中。使用涂抹器用粘性 PCR 密封膜密封 PCR 板。
- 将密封板以 1,000 × g 离心 1 分钟,以确保正确混合以消除孔内的任何气泡。
- 将 PCR 板放入热循环仪中。打开 qPCR 机器,然后打开相关软件设置协议。
- 根据 qPCR 预混液指南设置方案。使用以下方案:95 °C 10 分钟,然后 95 °C 15 秒,并在 72 °C 下延伸 30 秒。在 表 1 中为相关引物指定的退火温度下执行退火步骤 45 秒。进行 35 个循环的所有 qPCR 运行。
- 使用标准品和 NTC 设置与含样品板相同的配置中的 96 孔 qPCR 板。
- 使用绝对定量标准曲线法定量每个样品中的扩增产物和基因拷贝数33。
6. 在南德克萨斯州沿海湿地地图中可视化甲烷循环基因
- 打开地理信息系统 (GIS) 软件 ArcGIS Pro ,然后将名为 Study Area 的工程文件保存在计算机上的指定文件夹中。
- 点击左上角的地图 |Basemap 并选择 Terrain with Labels 作为底图。
- 单击 Locate |搜索 ,当搜索栏打开时,通过输入区域名称来定位研究区域;该区域将显示。
- 使用地理配准绘制特定区域。
- 单击 View |目录窗格 。
- 双击目录中的 Folder |文件名。
- 右键单击 地理数据库 (.gdb) 文件 ,然后单击 新建 |将显示 Feature Class 和 Create Feature Class 。
- 键入 名称 和 别名 框,然后单击 完成 在底部。
- 单击 View |目录。 别名 将显示在 内容窗格中。
- 单击顶部图层的 Edit |创建。Create Features 窗格将打开。双击 Alias in Create Features(创建要素中的 Alias)窗格,将出现 Configure Tool Feedback Options(配置工具反馈选项)。
- 选择 线,然后在地图上绘制线以创建研究区域的外部边界。完成后双击地图。
- 最小化 GIS 软件,然后打开电子表格。在第一列中键入 样本名称 ,在第二列中输入 纬度 ,在第三列中输入 经度 。将接下来的四列用于 pmoA1、 pmoA2、 mmoX 和 mcrA 的 qPCR 数据。
- 将文件以 CSV 格式 保存在计算机的任何特定文件夹中。
- 再次打开 GIS 软件,然后单击 添加数据 |XY 点数据。
- 从计算机上的文件夹中的 Input Table 框中选择 CSV 文件。重命名 Output Feature Class 中的文件名,然后单击 Run 以在地图上显示采样点。
- 单击顶部的 vsearch 栏并搜索 Kriging。
- 选择采样站文件,然后选择 pmoA1。
- 单击 “环境”| 在“图层”|“蒙 版”| ”单击 Run。
- 按照步骤 6.9、6.10 和 6.11 中提到的协议,为所有研究区域的 pmoA2、mmoX 和 mcrA 创建克里金法。
- 创建地图的布局。
- 单击顶层的 Insert |新建布局,然后选择 ANSI - 横向。
- 单击 地图框,选择 带有克里金法的地图,然后通过绘制矩形将所有地图放置在布局中。这将使地图在布局中可见。
- 选择 North Arrow 并将其放置在布局中以指示 North 方向。
- 选择 “比例尺 ”以在地图上显示该区域的比例。
- 单击 Legend 以显示图例,然后将其放置在布局中。
- 单击 格网 ,然后选择任意黑色经纬网选项。这将创建具有纬度和经度的格网,并将其显示在 内容窗格中 ,并带有标注 Black Horizontal Label Graticule。
- 双击 Black Horizontal Label Graticule,然后选择 Components 。单击 Ticks 1 和 Grid,然后单击它们右侧的叉号删除这些组件。
- 单击顶层的 Share ,然后单击 Export Layout。选择文件类型为 PDF,使用 名称框将文件保存在计算机上,将垂直分辨率设置为 500 DPI,然后单击 导出 以创建地图的 PDF 文件。
结果
为了解南德克萨斯州沿海湿地 CH4 循环相关基因 (mcrA 、 pmoA1 、 pmoA2 和 mmoX) 的分布和丰度,通过 cPCR 和 qPCR 分析每个样本中提取的 eDNA。从以前的研究中选择每个生物标志物的通用引物来运行 cPCR(表 1)22,34,35,36,37,并根据样品特性和环境条件进行修改以优化退火温度和浓度。例如,16S rRNA、pmoA1 和 pmoA2 在标准退火温度下未扩增,因为它们中的大多数扩增远低于标准退火温度(数据未显示)。此外,在 cPCR 过程中,浓度为 10 ng/μL 的 DNA 对 pmoA1、pmoA2 和 mmoX 基因显示出明显的条带,而使用 5 ng/μL 模板 DNA 对 mcrA 基因的条带更亮(补充文件 1-补充图 S1)。
cPCR 分析揭示了南德克萨斯州三种类型沿海湿地的基因检测空间变异性。如表 2 所示,在沿海淡水湿地 LB 和 RV 的所有样本中均检测到 mcrA 基因。然而,cPCR 无法在咸水湿地的采样沉积物中检测到任何 mcrA 基因,这表明盐度与产甲烷菌分布之间存在潜在联系(补充文件 1-补充图 S2)。对于 mcrA 检测,本研究使用了四个引物对,包括 MLF-MLR 、 MCRf-MCRr 、 ME1F-ME2R 和 McrA 159F-McrA 345R (表 1);其中,只有 ML 引物对能够检测到南德克萨斯州湿地中的甲烷原群落 (补充文件 1-补充图 S2 、补充图 S3 和补充图 S4)。相比之下,对于需氧嗜甲烷菌,在所有 RV 样品中检测到 pmoA1 基因,但在 LB 和 BC 样品中不存在(补充文件 1-补充图 S5)。为了鉴定 pmoA1,A189 用作正向引物,A682 和 mb661 作为反向引物,其中 A189-A682 引物对也可以检测 amoA35,38。然而,在这项研究中,这对引物能够检测到 cPCR 中基因的存在,其中凝胶图像显示非常微弱的条带,表明 pmoA1 基因的丰度较低。设计了另一种反向引物 mb661 来特异性识别 pmoA1 基因,以 A189 作为正向引物38。当 mb661 用作反向引物时,cPCR 显示出更亮的条带(补充文件 1-补充图 S6),后来用于 qPCR。有趣的是,在两个 BC 样本和一个 LB 样本中检测到 pmoA2 基因,但在一个 RV 样本中不存在(补充文件 1-补充图 S7)。先前的研究发现,pmoA2 的退火温度为 66 °C 和 60 °C,其中在 60 °C 时观察到两种不同大小的扩增子,245 和 438 bp,而在 66 °C 时,206f-703b 引物只能扩增 438 bp 产物大小为 22 的 pmoA2。然而,在本研究中,扩增在 60 °C 的退火温度下成功,在 LB 和 BC 位点产生了预期大小 (438 bp) 的条带(补充文件 1-补充图 S7)。此外,在 LB 样本中检测到 mmoX 基因,但在一个 RV 样本中不存在(补充文件 1-补充图 S8),并且在 BC 样本中完全不存在。在本研究中,用于 mmoX 的引物对也适用于研究南德克萨斯州湿地。55 °C36 的退火温度在 cPCR 中产生了 mmoX 的亮带(补充文件 1-补充图 S8)。为了验证样品的完整性,使用 16S rRNA 作为阳性对照,在 cPCR 过程中成功扩增了所有样品中的 16S rRNA,表明样品未被污染(补充文件 1-补充图 S9)。
为了评估基因丰度的时间和空间变化,使用 cPCR 中鉴定的优化引物对对所有靶基因进行 qPCR 分析。 pmoA2 和 mmoX 基因的 qPCR 使用 cPCR 中使用的相同引物进行。然而,对于 pmoA1 基因,使用 A189-mb661 引物对,对于 mcrA 基因,选择 MLF-MLR 引物对,因为两者都在 cPCR 中产生了良好的结果。南德克萨斯州湿地的季节性差异和盐度变化也被考虑在内,因为它们会影响微生物群落动态。如图 3 和 图 4 所示,产甲烷菌和嗜甲烷菌的基因丰度在两个不同的采样周期之间存在差异。虽然沿海淡水湿地的盐度没有季节性变化,但在沿海咸水湿地中观察到盐度的季节性差异39,这可能是产甲烷菌和嗜甲烷菌丰度变化的原因。 图 3 显示,在凉爽的季节,所有这些湿地中的 mmoX 都非常丰富,丰度最高,为 1.46 × 104 拷贝/μL。LB 、 RV 和 BC 的一个站点中有一小部分 pmoA2 。然而,由于凉爽季节丰度低,qPCR 无法检测 pmoA1 和 mcrA 。相比之下, pmoA1 在温暖季节最丰富,丰度最高,为 2.44 × 103 拷贝/μL(图 4)。在 LB 的表层沉积物样品中(顶部 50 cm 以内)中也检测到 mmoX 和 mcrA ,尽管它们在温暖季节的丰度相对较低,但最大丰度分别为 1.92 × 102 拷贝/μL 和 2.04 ×10 2 拷贝/μL。有趣的是,在温暖的季节,根本没有检测到 pmoA2 ,并且在 BC 样本中没有检测到任何靶基因,这可能是受到南德克萨斯州温暖季节这个咸水湿地特有的环境因素的影响39 。
本研究证明了 CH4 循环相关基因在沿海湿地中的分布存在明显的空间和季节性变化。具体来说,仅在淡水 LB 样本中检测到 mcrA 基因,表明栖息地偏好。 mmoX 和 pmoA2 基因在凉爽季节存在于所有样品中,而 pmoA1 在温暖季节出现在淡水环境中。这些结果突出了环境条件和季节变化对 CH4 循环相关微生物活性的影响。如图 3 和 图 4 所示,使用克里金插值法增强可视化,加深了我们对沿海湿地生态系统中 CH4 循环相关微生物活动的理解。
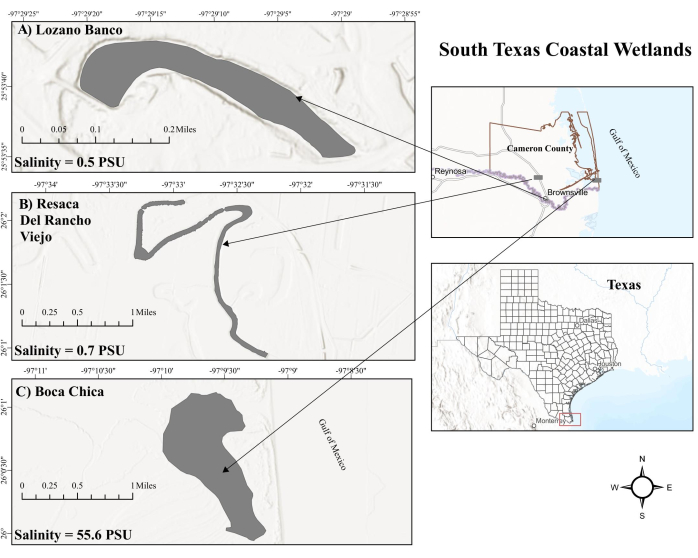
图 1:南德克萨斯州沿海湿地地图。 (A) Lozano Banco.LB 是一个沿海淡水湿地。(b) Resaca del Rancho Viejo。RV 也是一个沿海淡水湿地。(C) 博卡奇卡湿地。BC 是一个受潮汐影响的高盐沿海咸水湿地。缩写: LB = Lozano Banco;RV = Resaca Del Rancho Viejo;BC = 博卡奇卡;PSU = 实际盐度单位。 请单击此处查看此图的较大版本。
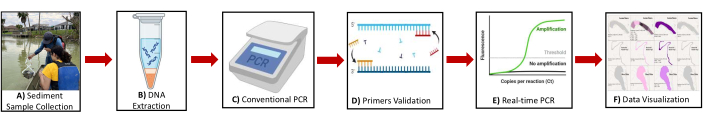
图 2:显示协议概述的示意图。 关键步骤的插图包括 (A) 沉积物样本采集 (B) DNA 提取 (C) 常规 PCR (D) 引物验证 (E) 实时 PCR (F) 用于识别产甲烷菌和嗜甲烷菌生物标志基因的数据可视化。 请单击此处查看此图的较大版本。
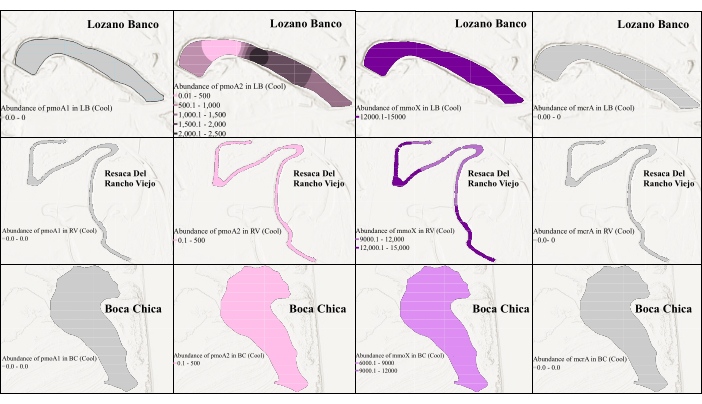
图 3:凉爽季节产甲烷菌和嗜甲烷菌生物标志基因的空间分布。 该图显示了凉爽季节 mmoX 的丰度。在这里,灰色表示 qPCR 由于丰度低而无法检测到该基因。 请单击此处查看此图的较大版本。

图 4:暖季产甲烷菌和嗜甲烷菌生物标志基因的空间分布。 在这里,灰色表示 qPCR 由于丰度低而无法检测到该基因。该图显示了 Lozano Banco 和 Resaca Del Rancho Viejo 在温暖季节的 pmoA1 丰度,而在 Boca Chica 没有检测到丰度。 请单击此处查看此图的较大版本。
| 引物名称 | 基因 | 本研究中的退火温度 (°C) | 产品长度 (bp) | 序列 (5'-3') | 参考 | |
| 1055F 系列 | 16S rRNA 抗体 | 55 | 337 | ATGGCTGTCGTCAGCT | (Harms 等人,2003 年) | |
| 1392R 系列 | ACGGGCGGTGTGT公司 | |||||
| A189F 系列 | pmoA1 | 55 | 525 | GGNGACTGGGACTTCTGG | (Holmes 等人,1995 年) | |
| A682R 系列 | GAASGCNGAGAAGAASGC | |||||
| A189F 系列 | 56 | 470 | GGNGACTGGGACTTCTGG | (Costello & Lidstrom,1999年) | ||
| mb661R 系列 | CCG GMG CAA CGT CYT TACC | |||||
| 206F | pmoA2 | 60 | 438 | GGNGACTGGGACTTCTG GATCGACTTCAAGGATCG | (Tchawa Yimga 等人,2003 年) | |
| 703b 系列 | GAASGCNGAGAAGAASGC GGCGACCGGAACGACGT | |||||
| 536F | mmoX | 55 | 362 | CGCTGTGGAAGGGCATGAAGCG | (Fuse 等人,1998 年) | |
| 898R 系列 | GCTCGACCTTGAACTTGGAGCC | |||||
| MCRf | 麦克拉 | 46 | ~490 | TAYGAYCARATHTGGYT | (Springer 等人,1995 年) | |
| MCRr | ACRTTCATNGCRTARTT | |||||
| ME1F | 50 | 763 | GCMATGCARATHGGWATGTC | (Hales 等人,1996 年) | ||
| ME2R 系列 | TCATKGCRTAGTTDGGRTAGT | |||||
| MLF 系列 | 55 | ~470 | GGTGGTGTMGGATTCA CACARTAYGCWACAGC | (Luton 等人,2002 年) | ||
| MLR | TTCATTGCRTAGTTWGGRTAGTT | |||||
| McrA 159F | 62 | 186 | AAAGTGCGGAGCAGCAATCACC | (Vaksmaa 等人,2017 年) | ||
| 麦克拉 345R | TCGTCCCATTCCTGCTGCATTGC | |||||
表 1:本研究中用于检测与甲烷循环相关的功能基因的引物组。 所有这些引物对都用于常规 PCR,最后,建立了大胆的引物对,用于检测南德克萨斯州沿海湿地的产甲烷菌和嗜甲烷菌相关基因。
| 基因 | 样品 | ||||||||
| Lozano Banco | Resaca Del Rancho Viejo | 博卡奇卡 | |||||||
| LB-1 型 | RV-1 型 | RV-2 | M1 系列 | M2 系列 | B1 层 | B2 层 | MG1 | MG2 | |
| pmoA1 | - | + | + | - | - | - | - | - | - |
| pmoA2 | + | + | + | - | + | - | - | + | - |
| mmoX | + | - | + | - | - | - | - | - | - |
| 麦克拉 | + | + | + | - | - | - | - | - | - |
表 2:使用常规 PCR 检测凉爽季节三种不同类型水体中的基因。 '+',现在;'-',不存在。缩写:LB = Lozano Banco,RV = Resaca del Rancho Viejo,M = 泥,B = Batis,MG = 红树林。
补充文件 1:mcrA、pmoA1、pmoA2、mmoX 和 16S rRNA 基因的凝胶电泳图像。 请点击此处下载此文件。
讨论
作者没有需要声明的利益冲突。沿海湿地被认为是大气中甲烷的重要贡献者,甲烷是一种重要的温室气体40。尽管已经对湿地中的甲烷通量和产甲烷菌进行了研究 41,42,43,但对嗜甲烷菌如何在不同环境或各种管理实践下运作知之甚少,尤其是在水位波动的湿地中 44。此外,eDNA 中腐殖酸等抑制剂的广泛存在使提取和扩增过程复杂化45,可能导致假阴性结果46。由于 eDNA 引物的效率可变,这一挑战进一步复杂化,表明需要在此类研究中进行系统验证47。因此,本研究为了解研究不足的南德克萨斯州沿海湿地的甲烷循环建立了一种基础方法。通过专注于 PCR 技术的开发和验证,本研究推进了这些动态生态系统中关键甲烷循环基因的检测。
这项研究的关键方面之一是微生物甲烷循环相关基因的可视化,这在观察这些基因在不同季节和盐度梯度的空间分布模式中起着至关重要的作用。这种可视化不仅是一种补充工具;它对于识别存在大量产甲烷和产甲烷微生物的区域至关重要,这些区域表明可能存在潜在的甲烷循环,从而增强我们对影响这些过程的环境条件的理解。
在本研究中,指定了使用 cPCR 和 qPCR 鉴定产甲烷菌和甲烷营养因子标记基因如 pmoA1 、 pmoA2 、 mmoX 和 mcrA 的方法。使用了 4 对靶向 mcrA 基因的引物对和 2 对靶向 pmoA1 基因的引物对,每组中只有一对在常规和实时 PCR 中有效检测这些基因。之前的一项研究使用三个引物对来检测 pmoA1;其中,A189-A682 引物无法检测嗜甲烷菌的多样性。相反,它们主要扩增了 amoA 基因并产生非特异性 PCR 产物48。该研究发现,所使用的引物对仅在嗜甲烷菌数量较多的环境中对检测 pmoA1 基因有效。此外,在该研究中,使用 A189-mb661 引物对获得了最高丰度的嗜甲烷菌,表明这些引物适用于淡水生态系统48。因此,这些引物对用于 qPCR 作为进一步分析。
在用于 mcrA 的四个引物对中,ML 引物能够在所有环境中扩增产甲烷菌49。ME 引物的扩增范围较窄;它们无法检测不同类型自然环境中的各种产甲烷菌种类。MCR 引物具有高水平的简并性,这使得它们对 PCR 退火温度敏感。这种简并性使它们能够与广泛的靶序列结合,通常适应多个错配。然而,这也表明退火温度的微小变化会显着影响它们的结合效率和特异性,导致扩增不足,从而导致产甲烷菌种类的假阴性结果49,50。
另一方面,在低多样性产甲烷菌群落中,在 PCR 的最后阶段,PCR 产物的高积累导致模板再退火并阻止引物结合,这也无法检测到所需的条带49。有趣的是,McrA 159F-McrA 345R 引物对是专门设计用于鉴定硝酸盐依赖性嗜甲烷念 珠菌 M. nitroreducens 古细菌51,本研究无法使用这些引物对检测厌氧嗜甲烷菌。然而,甲烷 (AOM) 的厌氧氧化可以与替代电子受体(如铁 (Fe)、锰 (Mn) 和硫酸盐 (SO42-)52)52 的还原耦合,这可能解释了无法使用这种方法检测 ANME 的原因。此外,这可能是由于研究区域的特定环境条件。因此,对于 qPCR,ML 引物用于检测甲烷原丰度,因为它即使在群落多样性低的条件下也能提供可靠的扩增49。
此外,腐植酸或非靶标物种 DNA 等 PCR 抑制剂广泛存在于环境样品中,可降低 PCR 效率53。由于高浓度的 DNA 无法通过 cPCR 检测任何基因,因此在运行 cPCR 之前稀释所有 DNA 样品以降低 PCR 抑制剂的影响54。16S rRNA 基因作为细菌和古细菌的通用标记物,为验证我们的 PCR 条件和确认所有样品的 DNA 完整性提供了强大的基线55,56。然而,咸水地区被认为具有较高浓度的 PCR 抑制剂46。此外,盐度间接影响沿海湿地的 CH4 排放,因为它与硫酸盐浓度有关。咸水湿地较高的盐度与硫酸盐水平升高相关,由于硫酸盐还原细菌和产甲烷菌之间的竞争,硫酸盐水平升高会抑制甲烷的产生,而淡水湿地中较低的硫酸盐浓度会导致甲烷排放量增加11,41。此外,咸水和淡水湿地之间的 CH4 排放差异也受到它们与海洋的潮汐联系的影响57,这也可能解释了在 BC 中通过 cPCR 未能成功检测到甲烷循环相关基因的原因。在这项研究中,cPCR 成功检测到 LB 和 RV 样本中的所有靶向基因,但未能检测到 BC 样本中这些基因的存在,pmoA2 基因是唯一的例外。此外,qPCR 能够在所有样品中检测到 mmoX,而 cPCR 在 BC 中无法检测到它。这表明与 cPCR 相比,qPCR 对 PCR 抑制剂表现出更大的抵抗力,这归因于其在检测机制中的敏感性增强58。另一个原因是 qPCR 在 PCR 循环的指数期识别 PCR 产物,而 cPCR 在平台期检测它们。这种区别使 qPCR 在试剂耗尽的晚期反应循环中表现出对产物降解的敏感性降低47。
本研究建立了一种 PCR 技术来鉴定南德克萨斯州沿海湿地中的甲烷循环相关基因,以克服与 PCR 抑制剂和可变引物效率相关的挑战。使用了 4 对靶向 mcrA 基因的引物对和 2 对靶向 pmoA1 基因的引物对,每组中只有一对在常规和实时 PCR 中有效检测这些基因。本研究总结了甲烷循环相关基因的引物对集,包括 pmoA1 的 a189-mb6661、 pmoA2 的 206f-703b、 mmoX 的 536F-898R 和 mcrA 的 ML。这表明适当的引物选择对于检测沿海湿地中甲烷循环相关基因的重要性,例如南德克萨斯州的那些。此外,研究表明, mmoX 在凉爽季节最丰富,而 pmoA1 在温暖季节丰富。
因此,本研究的结果为科学理解甲烷循环中涉及的微生物过程提供了基础,突出了环境因素对南德克萨斯州沿海湿地季节性和空间变化的影响。基于这些发现,未来的研究旨在在其他生态系统中进一步验证这种方法,例如稻田和淡水湿地,以提高稳健性和可推广性。此外,这些研究还将包括地球化学分析,如沉积物甲烷浓度和孔隙水硫化物,以及分子技术,如宏基因组学和转录组学,这些技术将基于此基础,提供更全面的甲烷循环动力学理解。
披露声明
The authors have no conflicts of interest to declare.
致谢
We are thankful to C-REAL members for their assistance in field observation and laboratory analyses.
材料
| Name | Company | Catalog Number | Comments |
| 0.2 mL PCR tubes | ThermoFisher Scientific | AB0620 | https://www.thermofisher.com/order/catalog/product/AB0620?SID=srch-srp-AB0620 |
| 0.5 mL PCR Tubes | Promega | E4941 | https://www.promega.com/products/biochemicals-and-labware/tips-and-accessories/0_5ml-pcr-tubes/?catNum=E4941 |
| 10 μL tips | ThermoFisher Scientific | 05-408-187 | Fisherbrand SureGrip Pipet Tip Racked or Reload System Tips Natural; 10μL; | Fisher Scientific |
| 15 mL centrifuge tube | ThermoFisher Scientific | 14-959-53A | https://www.fishersci.com/shop/products/falcon-15ml-conical-centrifuge-tubes-5/p-193301 |
| 200 μL tips | ThermoFisher Scientific | 05-408-190 | Fisherbrand SureGrip Pipet Tip Racked or Reload System Tips Natural; 200μL; | Fisher Scientific |
| 1000 μL tips | ThermoFisher Scientific | 02-707-402 | https://www.fishersci.com/shop/products/sureone-micropoint-pipette-tips-specific-standard-fit/02707402?gclid=Cj0KCQiAp NW6BhD5ARIsACmEb kUsQ9Lu0YIq5i4vWege 17qPdtxIYZyvmJH1cDo ARuwereO1V4GLz9UaA lDREALw_wcB&ef_id=C j0KCQiApNW6BhD5ARI sACmEbkUsQ9Lu0YIq5i 4vWege17qPdtxIYZyvmJ H1cDoARuwereO1V4GLz 9UaAlDREALw_wcB:G:s &ppc_id=PLA_goog_2175 7693617_171052169911_02 707402__715434303113_1555 377385658230343&ev_chn=sh op&s_kwcid=AL!4428!3!71543430 3113!!!g!2366517300713!&gad_source=1 |
| Applied Biosystem Power SYBR Green Master Mix | ThermoFisher Scientific | 4368577 | https://www.thermofisher.com/order/catalog/product/4368577 |
| ArcGIS Pro | esri | https://www.esri.com/en-us/arcgis/products/arcgis-pro/overview?srsltid=AfmBOopatJ4 JvHJfscHRcAaDx0Jz5_Jrl8l5 vYkkBvfOqE-uNSsMghN1 | |
| CFX Duet Real-Time PCR system | Bio-Rad | 12016265 | https://www.bio-rad.com/en-us/product/cfx-duet-real-time-pcr-system?ID=97722926-9ed9-16a4-1d83-c92f587e427a |
| Corning Lambda plus single channel pipettor volume 0.5-10 μL | Sigma-Aldrich | CLS4071-1EA | https://www.sigmaaldrich.com/US/en/product/sigma/cls4071 |
| Corning Lambda plus single channel pipettor volume 100-1000 μL | Sigma-Aldrich | CLS4075-1EA | https://www.sigmaaldrich.com/US/en/product/sigma/cls4075 |
| Corning Lambda plus single channel pipettor volume 20-200 μL | Sigma-Aldrich | CLS4074-1EA | https://www.sigmaaldrich.com/US/en/product/sigma/cls4074 |
| FastDNA spin kit for soil | MP Biomedical | 116560200-CF | https://www.mpbio.com/us/116560000-fastdna-spin-kit-for-soil-samp-cf?srsltid=AfmBOoqOxxGilzY3IHNIZR ajegGTr9MoX1oMZUh 3dcbJqe0UvvukY128 |
| Gene copy calculator | Science Primer | https://scienceprimer.com/copy-number-calculator-for-realtime-pcr . | |
| High speed benchtop centrifuge | ThermoFisher Scientific | 75004241 | https://newlifescientific.com/products/thermo-scientific-sorvall-st16-high-speed-benchtop-centrifuge-75004241?gad_source=1&gclid=Cj0KCQiApN W6BhD5ARIsACmEbkVC_-cCIN9j 20TvYq8iDsBlUR5cPK_1_wN OBEcjMdv-CYVoGCfeOLYaAv enEALw_wcB |
| High speed microcentrifuge | VWR | 75838-336 | https://us.vwr.com/store/product/20546590/null |
| Lysing Matrix E tube | glass bead/ceramic sphere-containing tube | ||
| Microcentrifuge tube | ThermoFisher Scientific | 02-681-320 | https://www.fishersci.com/shop/products/fisherbrand-low-retention-microcentrifuge-tubes-8/02681320?gclid=Cj0KCQiAp NW6BhD5ARIsACm EbkWbG4_o3oUiGk HJPU-_31-CuexDwQ fmWPnfyhBOf2BHXsy K3fFW1toaAgJbEALw_ wcB&ef_id=Cj0KCQiAp NW6BhD5ARIsACmEb kWbG4_o3oUiGkHJPU- _31-CuexDwQfmWPnfy hBOf2BHXsyK3fFW1toa AgJbEALw_wcB:G:s&ppc _id=PLA_goog_21757693 617_171052169911_0268 1320__715434303113_10 349826094968484711&ev _chn=shop&s_kwcid=AL!4 428!3!715434303113!!!g!23 66517300713!&gad_source=1 |
| PCR Master mix | Promega | M7502 | https://www.promega.com/products/pcr/taq-polymerase/master-mix-pcr/?catNum=M7502 |
| Quantiflour ONE dsDNA system | Promega | E4871 | https://www.promega.com/products/rna-analysis/dna-and-rna-quantitation/quantifluor-one-dsdna-system/?gad_source=1&gbraid=0AAAAAD _rg189yJTY3cxeVqMdu8RPx10 Ma&gclid=CjwKCAjwxNW2BhAk EiwA24Cm9FUgViPNyWq7UfZL VeeoroLAZ5JIP6w07RGK_4D0w oZgAqf-G1XTmxoCxm8QAvD_B wE&catNum=E4871 |
| Quantus Fluorometer | Promega | E6150 | https://www.promega.com/products/microplate-readers-fluorometers-luminometers/fluorometers/quantus-fluorometer/?catNum=E6150 |
| YSI Pro 2030 | YSI a xylem brand | 603174 | https://www.ysi.com/product/id-p2030/pro2030-kits |
参考文献
- Xu, T., et al. Wetlands of international importance: Status, threats, and future protection. Int J Environ Res Public Health. 16 (10), 1818 (2019).
- Corn, M. L. . Deepwater Horizon oil spill: coastal wetland and wildlife impacts and response. , (2010).
- Hendriks, I. E., Sintes, T., Bouma, T. J., Duarte, C. M. Experimental assessment and modeling evaluation of the effects of the seagrass Posidonia oceanica on flow and particle trapping. Marine Ecology Progress Series. 356, 163-173 (2008).
- Krause, S. J. E., Treude, T. Deciphering cryptic methane cycling: Coupling of methylotrophic methanogenesis and anaerobic oxidation of methane in hypersaline coastal wetland sediment. Geochimica et Cosmochimica Acta. 302, 160-174 (2021).
- Reddy, K. R., DeLaune, R. D., Inglett, P. W. . Biogeochemistry of Wetlands: Science and Applications. , (2022).
- La, W., et al. Sulfate concentrations affect sulfate reduction pathways and methane consumption in coastal wetlands. Water Research. 217, 118441 (2022).
- Derwent, R. G. Global warming potential (GWP) for methane: Monte Carlo analysis of the uncertainties in Global Tropospheric Model predictions. Atmosphere. 11 (5), 486 (2020).
- Potter, C., et al. Methane emissions from natural wetlands in the United States: Satellite-derived estimation based on ecosystem carbon cycling. Earth Interactions. 10 (22), 1-12 (2006).
- . Understanding global warming potentials Available from: https://www.epa.gov/ghgemissions/understanding-global-warming-potentials (2024)
- Wallenius, A. J., Dalcin Martins, P., Slomp, C. P., Jetten, M. S. M. Anthropogenic and environmental constraints on the microbial methane cycle in coastal sediments. Front Microbiol. 12, 631621 (2021).
- Qu, Y., et al. Salinity causes differences in stratigraphic methane sources and sinks. Environmental Science and Ecotechnology. 19, 100334 (2024).
- Vizza, C., West, W. E., Jones, S. E., Hart, J. A., Lamberti, G. A. Regulators of coastal wetland methane production and responses to simulated global change. Biogeosciences. 14 (2), 431-446 (2017).
- van Dijk, G., et al. Salinization lowers nutrient availability in formerly brackish freshwater wetlands; unexpected results from a long-term field experiment. Biogeochemistry. 143 (1), 67-83 (2019).
- Aronson, E., Allison, S., Helliker, B. R. Environmental impacts on the diversity of methane-cycling microbes and their resultant function. Front Microbiol. 4, 225 (2013).
- Reeburgh, W. S. Oceanic methane biogeochemistry. Chem Rev. 107 (2), 486-513 (2007).
- Thauer, R. K. Biochemistry of methanogenesis: a tribute to Marjory Stephenson. Marjory Stephenson Prize Lecture. Microbiology (Reading). 144 (Pt 9), 2377-2406 (1998).
- Thauer, R. K. Anaerobic oxidation of methane with sulfate: on the reversibility of the reactions that are catalyzed by enzymes also involved in methanogenesis from CO2. Curr Opin Microbiol. 14 (3), 292-299 (2011).
- Friedrich, M. W. Methyl-coenzyme M reductase genes: Unique functional markers for methanogenic and anaerobic methane-oxidizing Archaea. Methods in Enzymology. 397, 428-442 (2005).
- Reeburgh, W. S. Oceanic methane biogeochemistry. Chem Rev. 107 (2), 486-513 (2007).
- Rasigraf, O., Schmitt, J., Jetten, M. S. M., Lüke, C. Metagenomic potential for and diversity of N-cycle driving microorganisms in the Bothnian Sea sediment. Microbiologyopen. 6 (4), e00475 (2017).
- McDonald, I. R., Bodrossy, L., Chen, Y., Murrell, J. C. Molecular ecology techniques for the study of aerobic methanotrophs. Appl Environ Microbiol. 74 (5), 1305-1315 (2008).
- Tchawa Yimga, M., Dunfield, P. F., Ricke, P., Heyer, J., Liesack, W. Wide distribution of a novel pmoA-like gene copy among type II methanotrophs, and its expression in Methylocystis strain SC2. Appl Environ Microbiol. 69 (9), 5593-5602 (2003).
- Knief, C. Diversity and habitat preferences of cultivated and uncultivated aerobic methanotrophic bacteria evaluated based on pmoA as molecular marker. Front Microbiol. 6, 1346 (2015).
- Huang, I. -. S., et al. Preliminary assessment of microbial community structure of Wind-Tidal Flats in the Laguna Madre, Texas, USA. Biology. 9 (8), 183 (2020).
- Wilding, T. K., Brown, E., Collier, K. J. Identifying dissolved oxygen variability and stress in tidal freshwater streams of northern New Zealand. Environ Monit Assess. 184 (10), 6045-6060 (2012).
- Roy Chowdhury, T., Mitsch, W. J., Dick, R. P. Seasonal methanotrophy across a hydrological gradient in a freshwater wetland. Ecological Engineering. 72, 116-124 (2014).
- Sun, Q. -. Q., et al. Carbon dioxide and methane fluxes: Seasonal dynamics from inland riparian ecosystems, northeast China. Sci Total Environ. 465, 48-55 (2013).
- Lee, S., et al. Comparison and selection of conventional PCR primer sets for studies associated with nitrogen cycle microorganisms in surface soil. Appl Sci. 12 (20), 10314 (2022).
- Bae, K. -. S., et al. Development of diagnostic systems for wide range and highly sensitive detection of two waterborne hepatitis viruses from groundwater using the conventional reverse transcription nested PCR assay. J Virol Methods. 299, 114344 (2022).
- Lecusay, D. . Assessment and Monitoring of Deltaic Wetlands and Fluvial Systems: Refining and Validating a Multimetric Index of Resaca Ecosystem Health. , (2021).
- . FastDNA SPIN Kit for Soil (Cat No. 116560200) Available from: https://www.mpbio.com/media/productattachment/LS082019-EN-FastDNA-SPIN-Kit-for-Soil-116560200-Manual.pdf (2025)
- Luton, P. E., Wayne, J. M., Sharp, R. J., Riley, P. W. The mcrA gene as an alternative to 16S rRNA in the phylogenetic analysis of methanogen populations in landfill. Microbiology (Reading, England). 148 (Pt 11), 3521-3530 (2002).
- Changsoo, L., Jaai, K., Seung Gu, S., Seokhwan, H. Absolute and relative QPCR quantification of plasmid copy number in Escherichia coli. J Biotechnol. 123 (3), 273-280 (2006).
- Harms, G., et al. Real-time PCR quantification of nitrifying bacteria in a municipal wastewater treatment plant. Environ Sci Technol. 37 (2), 343-351 (2003).
- Holmes, A. J., Costello, A., Lidstrom, M. E., Murrell, J. C. Evidence that particulate methane monooxygenase and ammonia monooxygenase may be evolutionarily related. FEMS Microbiol Lett. 132 (3), 203-208 (1995).
- Fuse, H., et al. Oxidation of trichloroethylene and dimethyl sulfide by a marine Methylomicrobium strain containing soluble methane monooxygenase. Biosci Biotechnol Biochem. 62 (10), 1925-1931 (1998).
- Springer, E., Sachs, M. S., Woese, C. R., Boone, D. R. Partial gene sequences for the A subunit of methyl-coenzyme M reductase (mcrI) as a phylogenetic tool for the family Methanosarcinaceae. Int J Syst Bacteriol. 45 (3), 554-559 (1995).
- Costello, A. M., Lidstrom, M. E. Molecular characterization of functional and phylogenetic genes from natural populations of methanotrophs in lake sediments. Appl Environ Microbiol. 65 (11), 5066-5074 (1999).
- Flores, E. A. . Effects of Nutrient Enrichment on Mangrove and Saltmarsh Habitats. , (2022).
- Minjie, H., Jordi, S., Xianyu, Y., Josep, P., Chuan, T. Patterns and environmental drivers of greenhouse gas fluxes in the coastal wetlands of China: A systematic review and synthesis. Environ Res. 186, 109576 (2020).
- Bridgham, S. D., Cadillo-Quiroz, H., Keller, J. K., Zhuang, Q. Methane emissions from wetlands: biogeochemical, microbial, and modeling perspectives from local to global scales. Glob Chang Biol. 19 (5), 1325-1346 (2013).
- Liu, D. Y., Ding, W. X., Jia, Z. J., Cai, Z. C. Relation between methanogenic archaea and methane production potential in selected natural wetland ecosystems across China. Biogeosciences. 8 (2), 329-338 (2011).
- Ke, Z., et al. Methane emissions and methanogenic community investigation from constructed wetlands in Chengdu City. Urban Climate. 39, 100956 (2021).
- Chowdhury, T. R., Dick, R. P. Ecology of aerobic methanotrophs in controlling methane fluxes from wetlands. Applied Soil Ecology. 65, 8-22 (2013).
- Maja, S., et al. Humic substances cause fluorescence inhibition in real-time polymerase chain reaction. Anal Biochem. 487, 30-37 (2015).
- Sanches, T. M., Schreier, A. D. Optimizing an eDNA protocol for estuarine environments: Balancing sensitivity, cost and time. PLOS ONE. 15 (5), e0233522 (2020).
- Xia, Z., et al. Conventional versus real-time quantitative PCR for rare species detection. Ecol Evol. 8 (23), 11799-11807 (2018).
- Bourne, D. G., McDonald, I. R., Murrell, J. C. Comparison of pmoA PCR primer sets as tools for investigating methanotroph diversity in three Danish soils. Appl Environ Microbiol. 67 (9), 3802-3809 (2001).
- Juottonen, H., Galand, P. E., Yrjala, K. Detection of methanogenic Archaea in peat: comparison of PCR primers targeting the mcrA gene. Res Microbiol. 157 (10), 914-921 (2006).
- Lueders, T., Friedrich, M. W. Evaluation of PCR amplification bias by terminal restriction fragment length polymorphism analysis of small-subunit rRNA and mcrA genes by using defined template mixtures of methanogenic pure cultures and soil DNA extracts. Appl Environ Microbiol. 69 (1), 320-326 (2003).
- Vaksmaa, A., Jetten, M. S., Ettwig, K. F., Luke, C. McrA primers for the detection and quantification of the anaerobic archaeal methanotroph 'Candidatus Methanoperedens nitroreducens'. Appl Microbiol Biotechnol. 101 (4), 1631-1641 (2017).
- Ren, G., et al. Electron acceptors for anaerobic oxidation of methane drive microbial community structure and diversity in mud volcanoes. Environ Microbiol. 20 (7), 2370-2385 (2018).
- Goldberg, C. S., et al. Critical considerations for the application of environmental DNA methods to detect aquatic species. Methods in Ecology and Evolution. 7 (11), 1299-1307 (2016).
- McKee, A. M., Spear, S. F., Pierson, T. W. The effect of dilution and the use of a post-extraction nucleic acid purification column on the accuracy, precision, and inhibition of environmental DNA samples. Biological Conservation. 183, 70-76 (2015).
- Gohl, D. M., et al. Systematic improvement of amplicon marker gene methods for increased accuracy in microbiome studies. Nat Biotechnol. 34 (9), 942-949 (2016).
- Ballarini, A., Segata, N., Huttenhower, C., Jousson, O. Simultaneous quantification of multiple bacteria by the BactoChip microarray designed to target species-specific marker genes. PLOS ONE. 8 (2), e55764 (2013).
- Le Mer, J., Roger, P. Production, oxidation, emission and consumption of methane by soils: A review. Eur J Soil Biol. 37 (1), 25-50 (2001).
- Smith, C. J., Osborn, A. M. Advantages and limitations of quantitative PCR (Q-PCR)-based approaches in microbial ecology. FEMS Microbiol Ecol. 67 (1), 6-20 (2009).
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。