Method Article
단일 분자 수준에서 동질성을 라벨의 프로테옴 와이드 정량화
요약
여기, 선물이 단일 분자 수준에서 복잡 한 단백질 견본에서 각 단백질 종에 대 한 라벨 동질성을 평가 하는 프로토콜.
초록
셀 proteomes 전기 이동 법 분석 실험, 세포에 있는 단백질의 모든 종 형광 염료와 레이블이 아닌 구체적으로 하 고 그들의 분리에 따라 매칭에 의해 발견을 사용 하 여 특징. 단일 분자 형광 이미징 개별 형광 분자 시각화 능력 磁 단백질 검출을 제공할 수 있다. 그러나, 전기 이동 법 분석 실험에이 강력한 이미징 방법의 응용은 프로테옴 걸쳐 각 단백질의 형광 라벨의 동질성을 특성화 하는 방법의 부족에 의해 방해 된다. 여기, 우리는 단일 분자 형광 이미징 분석 결과에 따라 프로테옴 걸쳐 라벨 동질성을 평가 하는 방법을 개발 했다. 헬러 세포 샘플, 우리는 '라벨 인' 되 나 적어도 하나의 염료와 함께 표시 하는 단백질의 비율을 사용 하 여 측정에서 (LO), 결정 되었다 범위를 50%에서 90%, 단일 분자 이미징에 응용 프로그램의 높은 잠재력을 지 원하는 민감하고 정확한 프로테옴 분석입니다.
서문
프로테옴 분석, 셀에 표시 하는 단백질 분자의 전체 집합을 계량 하는 것을 목표로, 현재 생물의 약 연구에 귀중 한 접근 이다. 이 분석은 일반적으로 단백질 종 단백질 이온화1,2,3를 통해 생성 된 스펙트럼에 따라 식별 하는 질량 분석에 의존 합니다. 프로테옴 분석에 대 한 대체 방법 이며 전기 이동 법, polyacrylamide 젤 전기 이동 법 (페이지)를 포함 하 여, 모 세관 전기 이동 법 2 차원 (2D) 젤 전기 이동 법. 이 방법은 일반적인 전기 이동 별거 및 탐지 및 정량화 각 단백질의 분석 된 셀에서 모든 단백질 분자의 형광 라벨에 의존 합니다. 달성 하기 위해 필요한 일반적인 단백질 라벨, 하나의 전략 Coomassie Blue와 Sypro-루비4,5,6 등 정전기 및 소수 성 상호 작용을 통해 단백질을 바인딩할 수 있는 형광 염료를 사용 하는 . N-hydroxysuccinimide (NHS) 에스테 르 또는 maleimide, 1 차 아민 및 thiols와 같은 일반적인 잔류물을 통해 단백질에 묶는 covalently 수 있습니다, 각각7, 를 포함 하는 염료와 화학식 라벨을 사용 하는 대체 전략은 8.
한편, 형광 검출의 감도 낮은 풍부한 단백질 및 세포의 작은 숫자 분석에 이상적입니다. 단일 분자 형광 이미징 개별 형광 염료와 생체 외와 생체 조건9,10,,1112, 레이블이 지정 된 단백질의 검출을 허용 하는 가장 중요 한 방법 중 하나는 13,,1415. 응용 프로그램 이미징이 방법의 전기 기반 프로테옴 분석을 개별 붙일 표시 된 단백질을 계산 하 여 매우 민감하고 양적 분석을 사용 예정입니다. 그러나, 남아 있다 불분명 여부 형광 염료로 충분히 균질에 걸쳐 모든 단백질 분자 이며 어떻게이 동질성 다른 단백질 종 (그림 1)에 의해 영향을 받습니다. 간단한 대량 솔루션 측정 '커플링 효율'8 또는 '효율 라벨' 라는 단백질에 형광 염료의 어 금 니 비율을 얻기 위해 사용할 수 있지만이 속성 중 라벨의 동질성에 정보를 제공 하지 않습니다. 단백질 분자입니다.
여기, 우리는 라벨 동질성16셀 (그림 2) 모든 단백질 종에 대 한 조사 분석 결과 대 한 프로토콜을 설명 합니다. 이 분석 결과의 두 가지 주요 단계는 단백질 정화 및 이미징. 첫 번째 단계에서 셀에 모든 단백질은 붙일 표시 하 고 biotinylated, 다음 별도로 젤 전기 이동 법을 사용 하 여 압축을 푼 뒤에 electroelution. 두 번째 단계에서 추출 된 표본에서 개별 단백질 분자의 형광 속성 단일 분자 이미징에 따라 평가 됩니다. 우리가 라벨 인 (LO)16, 전화와 형광 염료의 평균 수는 단일 단백질 분자에 바인딩된는이 데이터를 매개 변수 계산 분석에 대 한 중요 한 단백질의 비율 이라고 적어도 한 같은 염색 (n̄ 염료), 특성화 될 수 있습니다. 프로토콜, NHS 에스테 르 기반 Cyanine 3 (Cy3) 염료와 HeLa 세포 프로테옴을 라벨에 대 한 최적화 절차를 예를 들어, 제시 하 고 원하는 연구 목표에 따라 다른 레이블 프로시저를 수정할 수 있습니다.
프로토콜
1. 세포 준비
- 37 ° C에서 10 cm 접시에 HeLa 세포 배양 미만 5% CO2 Dulbecco의 수정 10% 태아 둔감 한 혈 청을 포함 하는 글의 중간.
- 그들은 70 %confluency, ATCC 지침17다음에 도달 되 면 기 하 급수적으로 성장 세포를 수집 합니다.
- 버퍼링 하는 인산 염 (PBS) (pH 7.4) x 1의 5 mL로 세포를 씻어. PBS를 제거 합니다.
- 0.1% trypsin ethylenediaminetetraacetic 산 (EDTA)의 1 mL를 추가 합니다. 37 ° c.에서 5 분을 품 어
- 현미경으로 세포 분리의 진행을 모니터링 합니다.
- 일단 셀 라운드 되 고 접시에서 분리, 성장 매체의 4 mL를 추가 하 고 셀 덩어리를 pipetting으로 휴식.
- 셀 카운터를 사용 하 여 셀 수를 계산 합니다.
- 원심 분리기 튜브에서 10 분 150 x g 에서. 매체를 제거 하 고 resuspend 1 x PBS (pH 7.4)의 5 mL와 함께 셀 펠 릿.
- 1.2.6 단계를 반복 합니다. 106 셀/ml 단계 1.2.5에서에서 카운트 번호를 사용 하 여 최종 농도를 1 x PBS (pH 7.4)에 셀 resuspend
참고: 셀 정지 수 aliquoted-80 ° C에서 저장 몇 달 동안. 냉동/해 동 주기를 반복 피해 야 한다.
2. 세포 세포의 용 해 및 형광 라벨
- 게 lysing 버퍼의 10 mL (0.1 m M 붕 산 염, 1% 나트륨 라우릴 황산 염 (SDS)와 1% 트윈 20). PH 12 2 M NaOH를 사용 하 여 조정 됩니다.
주의: 집중된 NaOH 부식성입니다. 이 솔루션을 준비 하는 때 적합 한 장갑과 안경 착용.
참고: Lysing 버퍼는 실 온에서 1 주일까지 저장할 수 있습니다. - 1 M dithiothreitol (DTT)의 1 μ와 버퍼 및 4 lysing의 믹스 100 μ μ의 50 %3-[(3-cholamidopropyl) dimethylammonio]-1-propanesulfonate (챕).
- 2.2 단계에서 혼합된 lysing 버퍼와 세포 배양 (약 1, 000 셀)의 믹스 1 μ. 천천히 거품을 피하기 위해를 pipetting으로 솔루션을 균질. 부드러운 동요와 5 분 동안 실내 온도에 어둠 속에서 품 어. 다시 천천히 pipetting으로 솔루션을 균질.
- 2 μ g/mL Cy3 NHS 에스테 르 염료의 1 μ를 추가 합니다. 천천히 거품을 피하기 위해를 pipetting으로 솔루션을 균질. 부드러운 동요와 함께 실 온에서 어둠 속에서 5 분 동안 품 어.
참고:이 버퍼의 높은 산도 단백질, 높은 반응성 선도 리 진 잔류물의 중화를 발생 합니다. - 2 μ g/mL Cy3 NHS 에스테 르 염료의 1 μ의 추가 반복 합니다. 천천히 거품을 피하기 위해를 pipetting으로 솔루션을 균질. 부드러운 동요와 함께 실 온에서 어둠 속에서 10 분 동안 품 어.
참고:이 반복 NHS 에스테 르 염료의 추가 때문에 일부 Cy3 NHS 에스테 르 염료의 분해 반응 버퍼의 높은 pH에 의해 비활성화 라벨 효율을 증가 시킬 수 있습니다. - PH 7.2 0.8 M의 100 μ를 추가 하 여 4-(2-hydroxyethyl)-1-piperazineethanesulfonic 산 (HEPES) 조정-NaOH pH 7.2 반응 해소. 천천히 거품을 피하기 위해를 pipetting으로 솔루션을 균질.
- 추가 19 mg/mL biotin-(폴 리 에틸렌 글리콜 (PEG))2의 20 μ-아민 및 20 mg/mL 1의 5 μ-에틸-3-(3-dimethylaminopropyl) carbodiimide (EDC). 천천히 거품을 피하기 위해를 pipetting으로 솔루션을 균질. 부드러운 동요와 함께 실 온에서 어둠 속에서 1 시간에 품 어.
참고:이 biotinylation 단계 현미경 coverslip 고정 밀도 및 아무 단백질 종에 단백질 분자를 해결할 필요가 있다. - Unreacted 라벨 시 약을 제거 하 고 제조업체의 지침에 따라 10 kDa 커트 오프와 한 열을 사용 하 여 샘플을 집중.
참고: 프로토콜 수 수 일시 중지 여기. 샘플 몇 주 동안-80 ° C에 저장할 수 있습니다.
3. 프로테옴 분리 및 복구
- SDS 샘플 버퍼 (200 mM Tris HCl pH 6.8, 4 %SDS, 4% 글리세롤과 0.4 %bromophenol 블루) x 4를 추가 합니다.
참고: 염료 손상을 방지 하려면 단백질 샘플 수가 열 안 표준 SDS 페이지에서 다 고 실 온에서 보관 한다. - 단백질 견본 및 20% 아크릴 아 미드 젤을 사용 하 여 분자 사다리에 대 한 표준 SDS 페이지를 수행 합니다. 샘플을 마이그레이션하는 마이그레이션까지 일정 전압 (160 V)에서 젤 앞 그냥 젤 종료.
- 단백질 밴드 (그림 3)의 위치를 식별할 수 젤 이미지.
- 밴드 위치에 따라 날카로운 칼 날을 사용 하 여 관심사의 단백질 분수 포함 젤 부분을 잘라. 마찰 16, 23, 55 및 120 kDa 봉우리와 19-25, 25-32, 32-42, 42-54, 54-69, 69-97, 97-136, 136-226 kDa 영역은 그림 3에 나와 있습니다.
- 제조업체의 지침에 따라 6-8 kDa의 기 공 크기와 electroelution에 의해는 dialyzer를 사용 하 여 단백질을 추출 합니다.
참고: 추출 된 샘플 저장할 수 있습니다-80 ° C에서 몇 주 동안.
4. 현미경 coverslip 준비
- 각 샘플 avidin 버퍼 (5 mM HEPES NaOH pH 7.2, 10 ng/mL avidin, 2 mg/mL 소 혈 청 알 부 민 (BSA))의 200 μ를 준비 합니다.
- 22 m m x 22 m m, 0.15 m m 두께의 coverslips을 준비 합니다. 공기 플라즈마를 사용 하 여 플라즈마 1 분 동안 청소기 청소 하 고 그들의 표면 활성화 coverslips를 노출 합니다.
- 코트 1000 rpm에서 30 초 뒤 500 rpm에서 5 초 동안 스핀 coater를 사용 하는 avidin 버퍼의 스핀 코팅 200 μ에 의해 avidin와 coverslips. 드라이 그들을 실 온에서 15 분 동안 coverslips를 품 어.
- Coverslips 단백질 샘플 준비.
- 5 mM HEPES NaOH ph 7.2 (3.5)에서 단백질 샘플 100 배 희석.
- (4.3 단계)에서 avidin 코팅 coverslip의 중심에서 표면에 희석된 샘플의 장소 100 μ.
- 실 온에서 15 분 동안 어둠 속에서 품 어.
- 긍정적인 통제를 준비 합니다.
- 10 ng/mL의 장소 100 μ (단계 4.3)에서 5 mM HEPES NaOH pH 7.2는 avidin의 센터에서 코팅 coverslip 형광 biotin 정화.
- 실 온에서 15 분 동안 어둠 속에서 품 어.
참고:이 긍정적인 통제 coverslip 제공 한다 avidin 밀도 바인딩 관광 명소를, LO를 계산 하는 데 사용 되는.
- 부정적인 통제를 준비 합니다.
- 스핀-코트는 플라즈마 (avidin) 없이 5 mM HEPES NaOH 및 2 mg/mL BSA의 200 μ와 coverslip 청소.
- 건조 실 온에서 15 분 동안 품 어.
- 5 mM HEPES NaOH ph 7.2 10 ng/mL 정화 형광 biotin의 100 μ를 추가 합니다.
- 실 온에서 15 분 동안 어둠 속에서 품 어.
참고:이 부정적인 제어 coverslip avidin 없이 coverslip에 형광 biotin의 일반적인 바인딩 양을 제공합니다.
- 팁과 coverslip의 중간을 터치를 하지 coverslip의 가장자리에 pipetting으로 증류수 200 μ와 각 coverslip 린스. 이 세척 3 번 반복 합니다.
참고:는 coverslip에 발음, 후 물 즉시 두어야 한다 완전히 밖으로 건조에서 그것을 방지 하기 위해 coverslip에 다시. - 4.2 단계에서 coverslips의 동일한 수에 어 플라즈마에 노출.
- 건조를 피하기 위해 단계 4.7에서에서 샘플 바인딩된 coverslip 청소 coverslip을 놓습니다.
5입니다. 단일 분자 형광 현미경으로 관찰
- 넓은 필드 또는 사라져 분야 형광 현미경을 시작 합니다.
주의: 직접 또는 흩어져 레이저 방사선 눈 발생할 수 있습니다 또는 피부 손상. 적합 한 보호 고글을 착용 하 고 방패 레이저 빛 경로.
참고: 현미경 설치를 참조 하십시오 우리의 이전 게시16. 간단히, 고 전력 488 nm 레이저 구동, 높은 수 가늠 구멍 객 관 렌즈와 고감도 카메라 넓은 필드 또는 사라져 분야 형광 현미경이이 측정을 위해 사용할 수 있습니다. 또한, 자동 측정, 컴퓨터 제어 XY 변환 샘플 단, 레이저 업무가 대 한 기계적 셔터와 자동 초점 시스템 설치에 권장 됩니다. - 현미경에는 coverslip를 설정 하 고 포커스를 찾을.
- 적어도 100 이미지를 타일 스캔을 수행 합니다.
참고: 레이저 조명 photobleaching를 최소화 하기 위해 카메라와 이미지를 기록 하는 경우에 샘플에 노출 될 기계적 셔터와 제어 한다. 많은 샘플을 확인 하 고는, 자동된 측정 현미경 장치를 제어 하는 컴퓨터 프로그램을 사용 하 여 것이 좋습니다. 각 이미지 60 x 객관적 렌즈를 사용 하는 경우 일반적으로 100-500 단일 분자 관광 명소를 포함 합니다.
6. 이미지 분석 및 정보 추출
- 이미지 흘 레이저 조명 패턴 때문에, 그것에 게18를 해결 하기 위해 이미지 처리 실시 합니다.
- 균일 하 게 확산 염료 설정 단계 5.3 같은 카메라를 사용 하 여 구성 된 coverslip 샘플 측정 하 여 참조 이미지를 취득 합니다.
- 없는 레이저 여기 같은 카메라 설정을 사용 하 여 측정 하 여 오프셋된 이미지를 취득 합니다.
- 오프셋된 이미지의 가치와 모든 샘플 이미지와 참조 이미지에서 각 픽셀 값을 뺍니다.
- 뺀된 참조 이미지에 값 뺀된 샘플 이미지의 각 픽셀 값을 나눕니다.
- 제대로 획득된 이미지 수집 및 이미지 배경 제거.
- 큰 형광 집계를 수동으로 포함 된 이미지를 제거 합니다.
- ImageJ20를 사용 하 여 50 픽셀의 공 반경으로 회전 공 알고리즘19 를 적용 하 여 배경 이미지를 제거 합니다.
- ImageJ를 사용 하 여 흐린된 이미지를 빼서 이미지를 선명 하 게 (이미지 기능을 선명 하 게).
- 찾아서 이미지 관광 명소 단일 분자 분석.
- ImageJ를 사용 하 여 엔 임계값 알고리즘21 단일 분자 관광 명소를 식별 합니다.
- 제외할 카메라와 관광 명소의 어두운 소음 ImageJ (ROI 관리자)를 사용 하 여 단백질의 집계 제외 20 픽셀 미만 두 픽셀와 명소를 필터링 합니다.
- 각 이미지에 관광 명소의 수를 계산 합니다. 조명 수정된 이미지 (6.1.4 단계) 분석을 사용 하 여 모든 명소에 대 한 픽셀 내에서 농도 총 ImageJ를 사용 하 여.
- 다음 수식을 사용 하 여 라벨 인 (LO)를 계산.
LO = (샘플에서의 번호 / 긍정적인 컨트롤의 번호) x 100 - 단일 단백질 분자 (n̄염료)에 바인딩된 형광 염료의 평균 수를 계산 합니다.
- 모든 자리의 농도의 히스토그램을 분석 합니다.
참고: 히스토그램 일반적으로 여러 개의 봉우리, 그리고 모든 피크는 단백질에 바인딩된 염료 분자의 다른 수를 나타냅니다. 첫 번째, 두 번째 및 세 번째 피크 히스토그램에 각각 1, 2, 3 분자에 해당. - 히스토그램에서 평균 강도 계산 합니다. 또한 가우스 피팅을 적용 하 여 첫 번째 피크의 피크 강도 계산 합니다.
- N̄염료 피크 강도 평균 강도 나누어 계산 합니다.
- 모든 자리의 농도의 히스토그램을 분석 합니다.
결과
그림 4 는 긍정적이 고 부정적인 제어 뿐 아니라 HeLa 세포, lysate에서 단백질의 다른 분자량 분수에 대 한 원시 이미지 데이터를 나타냅니다. 단백질 견본 및 긍정적인 이미지 당 100-500 전시 명소를 제어 하는 동안 부정적인 컨트롤 없음 또는 프로토콜 충분히 coverslip 표면에 염료의 일반적인 바인딩을 억제를 보여주는 몇 가지 관광 명소를 표시 합니다. 자리 강도 히스토그램 단백질 샘플 및 긍정적인 통제에 대 한 여러 봉우리, 대표 하는 염료의 확률적 바인딩 단백질 및 avidin tetramer 구조22, 1 차 아민을 각각 제시. 모든 관광 명소 깜박이 stepwise photobleaching 연속 레이저 여기, (그림 5AB) 단일 분자 수준에서 관찰을 강조 했다.
LO는 긍정적인 통제에 그 모든 단백질 견본에 관광 명소의 수의 비율에서 계산 됩니다. LO는 HeLa 세포 lysate 샘플 작은 분자량 (16 kDa) 분수에 대 한 50%에서 90% 더 높은 분자량 (120 kDa) 분수에 대 한 원거리에서 측정 하 고 분리 (그림 6) 없이 전체 프로테옴 샘플에 대 한 72% 이었다. 대응 하 게, n̄염료 분자량을 증가 함께 증가 경향이 있었다. 이 증가 하는 경향을 증가 NHS 에스테 르로 이어지는 리 진 잔류물의 높은 숫자로 인해 발생으로 간주 됩니다 바인딩 염색. 예를 들어 23 kDa 분수 히스톤 단백질이 분수에 포함 된 리 진 잔류물의 많은 수 때문에 높은 LO 값을 갖습니다.
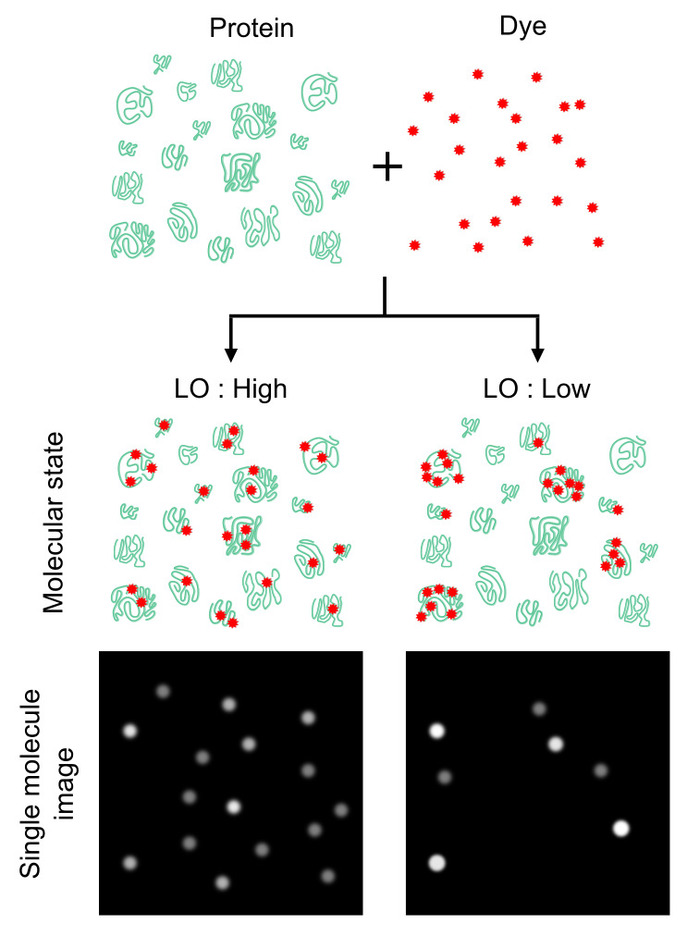
그림 1: 프로테옴 단백질 숫자 계산에 동질성을 라벨의 효과. 단백질 count 얼마나 강하게 높게에 지 이며 균질 염료는 단백질의 숫자 비율 보다 단백질 분자 표시 됩니다. 동질성 되 나 라벨 인 (LO), 총 단백질 분자에 대 한 레이블이 단백질 분자의 확률을 정의 하는 매개 변수를 사용 하 여 득점 될 수 있다. 이 값은 단백질 계산의 효율성을 제공 합니다 (즉, 더 높은 더 높은 LO 수익률 계산 숫자 (왼쪽)와 반대 (오른쪽)). LO 값이 100%는 이상적인, LO의 < 100% 절대 단백질 숫자 계산 한 감쇠 요소를 제공할 수 있습니다. 이 그림은 Leclerc 외16 저작권 2018 미국 화학 협회에서에서 수정 되었습니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭 하십시오.

그림 2: 분석 결과 워크플로. 첫째, coverslips (a)와 (b) avidin 고정된 밀도 달성 하기 위해 처리 됩니다. 두 번째, 붙일 레이블 샘플 단백질 biotinylated 고 avidin 비타민 b 복합체 상호 작용을 통해 coverslip (a)에 연결 된. 동시에, 거의 100% 붙일 레이블 순화 biotin coverslip (b)에 움직일 수는. 셋째,는 coverslips 형광 관광 명소의 수 및 밝기 분포를 단일 분자 형광 현미경 검사 법에 의해 몇 군데 있습니다. 마지막으로, 동질성 매개 변수, LO, 계산에 (한) 자리 숫자의 비율에서 그 (b)에서 합니다. 이 그림은 Leclerc 외16 저작권 2018 미국 화학 협회에서에서 수정 되었습니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭 하십시오.
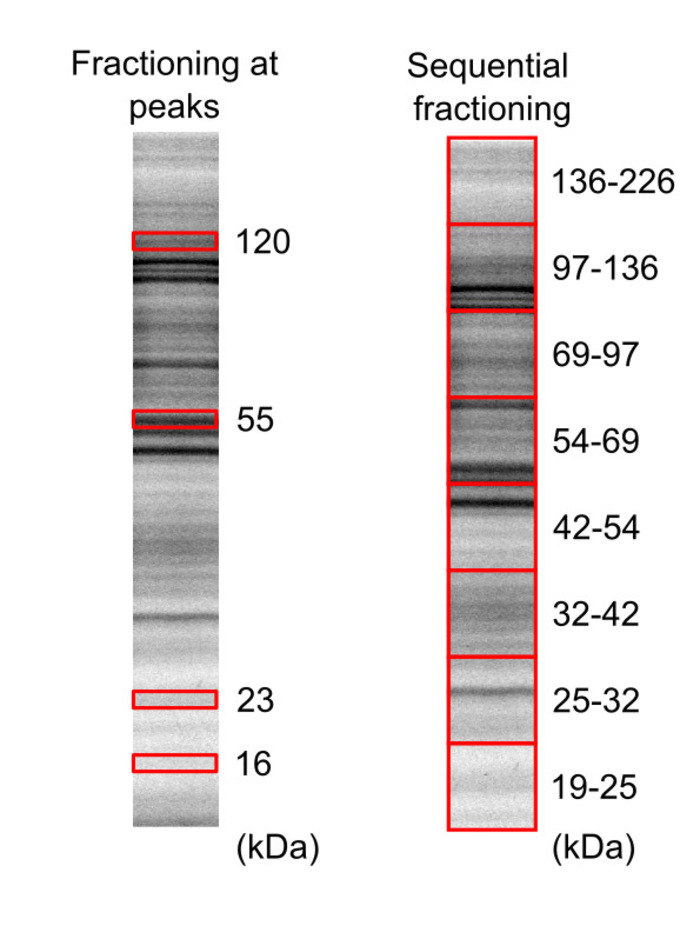
그림 3: SDS 페이지를 사용 하 여 헬러 세포 프로테옴 시료의 분리. 붙일 표시 및 biotinylated 세포 lysate에 두 개의 다른 젤 (20% 아크릴 아 미드), 마이그레이션된 고 단백질을 위한 5 분 및 10 분 노출 시간 젤 뷰어를 사용 하 여 시각 1 젤 젤 2, 각각. 빨간 상자를 잘라 하 고 추출 했다 젤 영역을 나타냅니다. 이 그림은 Leclerc 외16 저작권 2018 미국 화학 협회에서에서 수정 되었습니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭 하십시오.

그림 4: 프로테옴 샘플의 분자 이미지를 단일. 형광은 이미지 (왼쪽) 및 다른 분자량 프로테옴 분수에서 얻은 자리 농도 (오른쪽)의 히스토그램 자리. 눈금 막대는 10 μ m입니다. 긍정적인 통제에 화살표는 avidin의 다른 라벨 단계를 나타냅니다. 이 그림은 Leclerc 외16 저작권 2018 미국 화학 협회에서에서 수정 되었습니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭 하십시오.

그림 5: 형광 관광 명소의 Photobleaching. (A) 연속 레이저 여기에서 형광 명소의 시간 경과 이미지. (B) 시간 다른 장소에서 형광 강렬의 흔적. 추적 중 하나 또는 둘 스텝 photobleaching, 각각 수 염료의 정제 BSA 샘플의 자리에 단백질에 결합 되었다 나타내는 표시. 이 그림은 Leclerc 외16 저작권 2018 미국 화학 협회에서에서 수정 되었습니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭 하십시오.

그림 6: 프로테옴 샘플의 라벨. LO (파란색) 그리고 n̄염료 (짙은 빨간색) 다른 분자량 분수에서 얻은. 데이터는 의미 ± 남동 98, 46, 27, 77, 44, 39, 62, 101, 65, 62, 82, 61 및 70 이미지 표현 lysate, 전체 셀에 대 한 분석은 16, 23, 55, 120, 19-25, 25-32, 32-42, 42-54, 54-69, 69-97, 97-136, 136-226 kDa 분수 각각. 이 그림은 Leclerc 외16 저작권 2018 미국 화학 협회에서에서 수정 되었습니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭 하십시오.
토론
이 문서는 SDS 페이지 (3 단계)와 분리 후 셀에 각 레이블이 단백질 종의 라벨 동질성을 계량 하는 프로토콜을 설명 합니다. 분리 방법 분리 및 특수 장비23을 요구 하면서 높은 해상도, 세포질 단백질의 분류를 허용 하는 모 세관 전기 이동 법, 액체 크로마토그래피 등 다른 방법으로 교체하실 수 있습니다. 현재 프로토콜에 NHS 에스테 르를 사용 하 여 라벨 메서드 예 maleimide-에스테 르 또는 항 체를 사용 하 여 다른 방법으로 대체할 수 있습니다.
커플링 효율 분석에 가정 동질성 분석을 라벨에 대 한 요구 사항은 간단한 무작위 과정에서 일탈 얼마나 라벨에 따라 다릅니다. 우리의 최근 연구16 는 pH 및 세제 같은 반응 조건에 따라 임의의 프로세스에서 일탈 단백질의 NHS 에스테 르 레이블 표시. 우리는 몇 가지 조건 이종 단백질에 염료의 협력적인 바인딩 또는 불완전 한 가용 화 또는 염료에 대 한 선호도 낮춘된 비-반응성 단백질의 분수의 존재에 의해 유도 관찰. 대조적으로, 다른 조건 염료에 단백질 내의 반응 잔류물의 완전 한 바인딩에서 감소.
우리 고려는 pH 라벨 동질성에 가장 영향력 있는 요인 중 하나입니다. 높은 산도 단백질, NHS 에스테 르 염료와 더 높은 반응성 선도 리 진 잔류물의 중립화를 일으킬 수 있습니다. 또한, 높은 pH 세포 세포의 용 해를 일으킬 수 있는 고 단백질을 변성 수 있습니다. 그러나 높은 산도 단백질의 부정 하 게 있기 때문에 전자 전기 등 2D 전기 영동 실험에 적합 하지 않습니다,, pH 상태는 연구 목적에 따라 신중 하 게 고려 될 필요 합니다.
정확 하 게 측정 하려면 라벨 동질성 분석 결과에서 그것은 충분 한 biotinylated 단백질 견본을 사용 하는 것이 중요입니다. 이것은 분석 결과 단백질 채도 coverslip, 그리고 LO의 과소에 부족 한 단백질 농도 결과에 모든 avidin 분자에 바인딩할 가정 하기 때문에. 우리의 이전 연구16 일반적으로 단백질의 이상 10 세는 포화 바인딩을 위해 필요 하 고이 분류 후에 약 1000 HeLa 세포24에서 얻어질 수 있다 보이고 있다. Avidin 명소의 채도 확인 하려면 단백질 샘플의 희석 시리즈를 만들는 것이 좋습니다. 우리는 또한 참고 avidin 밀도 coverslip에 그렇게 하지 원인 통계 분석에 대 한 충분 한 자리 숫자를 주는 하는 동안 녹화 된 이미지에 명소 사이의 겹치는 최적화 되어야 할 수 있습니다.
이 분석 결과 가정 하나는 coverslip에 tetravalent avidin 분자22 한 단백질을 바인딩할 수 있습니다. 실제로, 우리의 이전 연구에서 긍정적인 컨트롤 데이터 표시 한 avidin 분자는 최대 4 개까지 붙일 레이블된 biotins, 하지만 과반수 (55%)에 바인딩할 수 있습니다. avidin 분자의 단 하나 또는 두 개의 biotin 분자를 바인딩하십시오. 단백질 비타민 b 복합체 보다 큰 있기 때문에, 그들의 입체 효과 적은 단백질 한 avidin에 바인딩된 될 한다. 우리의 가정은 더 이상 사실에 의해 지원 그 n̄염료 일정 하 게 유지의 혼합된 샘플 측정 표시 하 고 다른 비율16단백질 레이블 없음 때.
단백질 종류에 따라 불특정 바인딩 하는 coverslip 단백질의 BSA 코팅으로도 크게 발생할 수 있습니다. 이 경우 biotinylated 단백질 avidin, 단계 4.6.3 형광 biotin 대신 없이 coverslip에 코팅은 하나의 옵션 컨트롤 측정 하 수 있습니다. LO에 난다 바인딩의 효과 수학적으로 단백질 샘플에서이 컨트롤의 수를 빼서 제거할 수 있습니다.
이 의정서는 프로테옴 넓은 단일 분자 단백질 분석 계산에 대 한 귀중 한 것입니다. 단일 분자 감도 셀25에서 그들의 풍부에 관계 없이 모든 단백질 종에 대 한 단백질의 분석을 허용할 것 이다. 이 형광 명소 단백질 결합 수의 정량화 다음 프로테옴 분리에 의해 실현 될 수 있습니다. 또한, 높은 감도 또한 단일 세포 분석, 세포 인구에서이 검토 하는 연구를 활성화 하면 고 휴대의 클러스터링 할25,26상태.
공개
저자 선언 다음 경쟁 금융 interest(s): RIKEN SL 및 Y.T. 공동 발명가로 라는 이러한 결과에 특허 출원을 제기 했다.
감사의 말
저자 감사 Masae 오노와 카즈야 니시무라 실험적인 지원 및 조언. 이 작품은 프레스 토 (JPMJPR15F7), 일본 과학 및 기술 기관, 젊은 과학자 (A) (24687022), 도전 탐구 연구 (26650055), 및 혁신적인 지역 (23115005), 일본 학회에 과학 연구에 대 한 보조금에 의해 지원 되었다 승진의 과학, 그리고 다케다 과학 재단 및 모 기념 재단에서 교부 금에 의해 의료 및 제약 연구에 대 한. S.L. 인정 RIKEN 국제 프로그램 연결 (IPA) 프로그램에서 지원 합니다.
자료
| Name | Company | Catalog Number | Comments |
| 22x22x0.15 mm coverslip | VWR | 470019-004 | |
| 488 nm Argon laser | Coherent | Innova 70C | |
| 488 nm dichroic mirror | Semrock | FF495-Di03 | |
| 488 nm emission filter | Semrock | FF02-520/28 | |
| 560 nm dichroic filter | Semrock | Di02-R561 | |
| 560 nm emission filter | Semrock | FF02-617/73 | |
| 560 nm fiber laser | MBP Communications | F-04306-2 | |
| 60x oil immersion lens | Olympus | PLAPON 60x | |
| Avidin | Nacalai-tesque | 03553-64 | |
| Biotin-PEG-amine | Thermo Scientific | 21346 | |
| Biotinylated Alexa Fluor 488 | Nanocs | ||
| Borate | Nacalai-tesque | ||
| BSA | Sigma-Aldrich | A9547 | |
| CHAPS | Dojindo | C008-10 | |
| Cy3 NHS-ester dye | GE Healthcare | PA13101 | |
| Dialyzer - D-tube, 6-8 kDa | Merck Millipore | 71507-M | |
| DMEM | Sigma-Aldrich | ||
| DTT | Nacalai-tesque | 14112-94 | |
| EDC | Nacalai-tesque | ||
| EMCCD camera | Andor | IiXon 897 | |
| Epi fluorescence microscope | Olympus | IX81 | |
| Gel viewer | GE Healthcare | ImageQuant LAS 4000 | |
| Penicillin, streptomycine and Amphoterecin mix | Gibco | ||
| Plasma cleaner | Diener Electronic | ||
| SDS | Wako | NC0792960 | |
| Size purification column 10K | Merck Millipore | UFC5010 | |
| Tween 20 | Sigma-Aldrich | P9416 |
참고문헌
- Wilhelm, M., et al. Mass-spectrometry-based draft of the human proteome. Nature. 509 (7502), 582-587 (2014).
- Guo, A., et al. Immunoaffinity enrichment and mass spectrometry analysis of protein methylation. Molecular & Cellular Proteomics. 13 (1), 372-387 (2014).
- Tyanova, S., Temu, T., Cox, J. The MaxQuant computational platform for mass spectrometry-based shotgun proteomics. Nature Protocols. 11 (12), 2301-2319 (2016).
- Neuhoff, V., Arold, N., Taube, D., Ehrhardt, W. Improved staining of proteins in polyacrylamide gels including isoelectric focusing gels with clear background at nanogram sensitivity using Coomassie Brilliant Blue G-250 and R-250. Electrophoresis. 9 (6), 255-262 (1988).
- Berggren, K., et al. Background-free, high sensitivity staining of proteins in one- and two-dimensional sodium dodecyl sulfate-polyacrylamide gels using a luminescent ruthenium complex. Electrophoresis. 21 (12), 2509-2521 (2000).
- Butt, R. H., Coorssen, J. R. Coomassie blue as a near-infrared fluorescent stain: a systematic comparison with Sypro Ruby for in-gel protein detection. Molecular & cellular proteomics. 12 (12), 3834-3850 (2013).
- Nanda, J. S., Lorsch, J. R. Labeling a protein with fluorophores using NHS ester derivitization. Methods in Enzymology. , 87-94 (2014).
- Kim, Y., et al. Efficient site-specific labeling of proteins via cysteines. Bioconjugate chemistry. 19 (3), 786-791 (2008).
- Dickson, R. M., Cubitt, A. B., Tsien, R. Y., Moerner, W. E. On/off blinking and switching behaviour of single molecules of green fluorescent protein. Nature. 388 (6640), 355-358 (1997).
- Penna, A., et al. The CRAC channel consists of a tetramer formed by Stim-induced dimerization of Orai dimers. Nature. 456 (7218), 116-120 (2008).
- Ji, W., et al. Functional stoichiometry of the unitary calcium-release-activated calcium channel. Proceedings of the National Academy of Sciences of the United States of America. 105 (36), 13668-13673 (2008).
- Ulbrich, M. H., Isacoff, E. Y. Subunit counting in membrane-bound proteins. Nature Methods. 4 (4), 319-321 (2007).
- Sugiyama, Y., Kawabata, I., Sobue, K., Okabe, S. Determination of absolute protein numbers in single synapses by a GFP-based calibration technique. Nature Methods. 2 (9), 677-684 (2005).
- Puchner, E. M., Walter, J. M., Kasper, R., Huang, B., Lim, W. A. Counting molecules in single organelles with superresolution microscopy allows tracking of the endosome maturation trajectory. Proceedings of the National Academy of Sciences of the United States of America. 110 (40), 16015-16020 (2013).
- Grassart, A., et al. Actin and dynamin2 dynamics and interplay during clathrin-mediated endocytosis. The Journal of Cell Biology. 205 (5), 721-735 (2014).
- Leclerc, S., Arntz, Y., Taniguchi, Y. Extending Single Molecule Imaging to Proteome Analysis by Quantitation of Fluorescent Labeling Homogeneity in Complex Protein Samples. Bioconjugate chemistry. 29 (8), 2541-2549 (2018).
- Ian Freshney, R. Culture of Animal Cells: A Manual of Basic Technique and Specialized Applications. , John Wiley & Sons. (2015).
- Taniguchi, Y., et al. Quantifying E. coli proteome and transcriptome with single-molecule sensitivity in single cells. Science. 329 (5991), 533-538 (2010).
- Sternberg, Biomedical Image Processing. Computer. 16 (1), 22-34 (1983).
- Rueden, C. T., et al. ImageJ2: ImageJ for the next generation of scientific image data. BMC Bioinformatics. 18 (1), 529(2017).
- Yen, J. C., Chang, F. J., Chang, S. A new criterion for automatic multilevel thresholding. IEEE Transactions on Image Processing. 4 (3), 370-378 (1995).
- Livnah, O., Bayer, E. A., Wilchek, M., Sussman, J. L. Three-dimensional structures of avidin and the avidin-biotin complex. Proceedings of the National Academy of Sciences of the United States of America. 90 (11), 5076-5080 (1993).
- Mostovenko, E., Hassan, C., Rattke, J., Deelder, A. M., van Veelen, P. A., Palmblad, M. Comparison of peptide and protein fractionation methods in proteomics. EuPA Open Proteomics. 1, 30-37 (2013).
- Volpe, P., Eremenko-Volpe, T. Quantitative studies on cell proteins in suspension cultures. European Journal of Biochemistry. 12 (1), 195-200 (1970).
- Huang, B., et al. Counting low-copy number proteins in a single cell. Science. 315 (5808), 81-84 (2007).
- Johnson, A. C., Bowser, M. T. H. igh- High-Speed, Comprehensive, Two Dimensional Separations of Peptides and Small Molecule Biological Amines Using Capillary Electrophoresis Coupled with Micro Free Flow Electrophoresis. Analytical Chemistry. 89 (3), 1665-1673 (2017).
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기더 많은 기사 탐색
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유