Method Article
Échelle du protéome Quantification d’homogénéité à l’échelle de la molécule unique d’étiquetage
Dans cet article
Résumé
Nous présentons ici un protocole pour évaluer l’homogénéité étiquetage pour chaque espèce de protéine dans un échantillon complexe des protéines à l’échelle de la molécule.
Résumé
Cellule protéomes sont souvent caractérisées à l’aide de tests d’électrophorèse, où toutes les espèces de protéines dans les cellules sont non spécifiquement étiquetés avec un colorant fluorescent et sont repérés par un photodétecteur après leur séparation. Imagerie de fluorescence seule molécule peut fournir détection ultrasensible de protéines grâce à sa capacité de visualisation de molécules fluorescentes individuels. Toutefois, l’application de cette méthode d’imagerie puissante aux déterminations de l’électrophorèse est entravée par le manque de moyens pour caractériser l’homogénéité du marquage fluorescent de chaque espèce de protéines à travers le protéome. Ici, nous avons développé une méthode pour évaluer l’homogénéité d’étiquetage dans l’ensemble du protéome basé sur un test d’imagerie de fluorescence seule molécule. Dans notre mesure en utilisant un échantillon de cellules HeLa, la proportion de protéines marquées au moins un colorant, qui nous appelé « étiquetage occupation » (LO), était déterminé à gamme de 50 à 90 %, soutenant le potentiel élevé de l’application de l’imagerie de la seule molécule à analyse du protéome sensible et précis.
Introduction
Analyse du protéome, qui vise à quantifier l’ensemble des molécules de protéines exprimées dans la cellule, est une approche utile dans les études biologiques et pharmaceutiques actuelles. Cette analyse s’appuie généralement sur la spectrométrie de masse, qui identifie les espèces de protéines issus des spectres générées par l’intermédiaire de protéines ionisation1,2,3. Une autre méthode d’analyse du protéome est l’électrophorèse, y compris l’électrophorèse sur gel de polyacrylamide (PAGE), l’électrophorèse capillaire et l’électrophorèse sur gel en deux dimensions (2D). Cette méthode repose sur le marquage fluorescent non spécifique de toutes les molécules de protéine dans les cellules analysées, suivies de la séparation par électrophorèse et de détection et de quantification de chaque espèce de protéine. Pour atteindre l’étiquetage requis des protéines non spécifiques, une stratégie consiste à utiliser des colorants fluorescents qui peuvent se lier aux protéines via des interactions électrostatiques et hydrophobes, comme le bleu de Coomassie et Sypro Ruby4,5,6 . Une autre stratégie consiste à utiliser le marquage covalent avec colorants contenant de l’ester N-hydroxysuccinimide (NHS) ou maléimide, qui peut lier par liaison covalente à des protéines par le biais de résidus communs tels que les amines primaires et thiols, respectivement7, 8.
Pendant ce temps, la sensibilité de détection de fluorescence est idéale pour l’analyse des protéines de faible abondance et un petit nombre de cellules. Imagerie de fluorescence des molécules simples est une des méthodes plus sensibles qui permet la détection des colorants fluorescents individuels et des protéines marquées in vitro et in vivo9,10,11,12, 13,14,15. Application de cette méthode d’imagerie pour l’analyse protéomique axée sur l’électrophorèse est prévue pour permettre des essais hautement sensibles et quantitatives en comptant les différentes protéines fluorescent marquées. Cependant, on ne sait si l’étiquetage avec les colorants fluorescents est assez homogène dans l’ensemble de toutes les molécules de protéines, et comment cette homogénéité est affectée par des espèces de protéines différentes (Figure 1). Mesures de solution simple en vrac peuvent être utilisées pour obtenir un rapport molaire de colorants fluorescents aux protéines appelées « efficacité de couplage »8 « étiquetage efficacité », mais cette propriété ne fournit pas d’information sur l’homogénéité de l’étiquetage des molécules de protéines.
Nous décrivons ici le protocole pour un dosage d’enquêter sur l’homogénéité d’étiquetage pour toutes les espèces de protéines dans la cellule (Figure 2)16. Les deux principales étapes de ce test sont l’imagerie et purification des protéines. Dans la première étape, toutes les protéines dans les cellules sont étiquetés fluorescent et biotinylés, puis extrait séparément à l’aide de l’électrophorèse sur gel suivie par électroélution. Dans la deuxième étape, les propriétés de fluorescence des molécules de protéines individuelles dans les échantillons extraits sont évaluées basées sur l’imagerie seule molécule. Partir de ces données, paramètres importants pour l’analyse de l’inventaire, tels que le pourcentage de protéines marquées avec un moins un colorant, qui nous appelons étiquetage occupation (LO)16, et le nombre moyen de colorants fluorescents lié à une molécule de protéine unique (n̄ colorant), peuvent être caractérisés. Dans le protocole, une procédure optimisée pour l’étiquetage du protéome de cellules HeLa avec colorant de Cyanine 3 (Cy3) base d’esters NHS est présentée à titre d’exemple et peut être modifiée avec d’autres procédures d’étiquetage selon les objectifs de recherche désiré.
Protocole
1. préparation de la cellule
- Cultiver des cellules HeLa dans un plat de 10 cm à 37 ° C moins de 5 % de CO2 dans Dulbecco de l’aigle modifié contenant 10 % sérum de veau fœtal.
- Prélever des cellules exponentiellement croissants que lorsqu’ils ont atteint 70 % confluence, suivant les instructions de ATCC17.
- Rincer les cellules avec 5 mL de 1 x en solution saline tamponnée au phosphate (PBS) (pH 7,4). Supprimer les PBS.
- Ajouter 1 mL d’acide de 0,1 % trypsine-tétraacétique (EDTA). Incuber 5 min à 37 ° C.
- Surveiller la progression de la dissociation cellulaire au microscope.
- Une fois que les cellules deviennent ronds et sont détachent du plat, ajouter 4 mL de milieu de croissance et briser l’agglomération de cellule en pipettant également.
- Compter le nombre de cellules à l’aide d’un compteur de cellules.
- Centrifuger à 150 g pendant 10 min dans un tube à centrifuger. Retirez le support et resuspendre le culot cellulaire avec 5 mL de solution de 1 PBS x (pH 7,4).
- Répétez l’étape 1.2.6. Remettre en suspension les cellules dans du PBS 1 x (pH 7,4) à une concentration finale de 106 cellules/mL avec le numéro de compte à l’étape 1.2.5.
Remarque : La suspension cellulaire peut être aliquotés et congelés à-80 ° C pendant plusieurs mois. Répéter un cycle de gel/dégel devrait être évitée.
2. cellule lysis et marquage fluorescent
- Faire 10 mL de tampon de lyse (borate 0,1 M, de 1 % dodécylsulfate de sodium (SDS) et de 1 % Tween 20). Ajuster le pH 12 à l’aide de 2 M de NaOH.
Attention : NaOH concentré est corrosif. Porter des lunettes et des gants appropriés lors de la préparation de cette solution.
Remarque : Le tampon de lyse peut être stocké pendant une semaine à température ambiante. - Mélanger 100 μL de lyser un tampon avec 1 μL de 1 M le dithiothréitol (DTT) et 4 μL de 50 % (3-[(3-cholamidopropyl)) diméthylammonio] -1-propanesulfonate (CHAPS).
- Mélanger 1 μL de la culture cellulaire (environ 1 000 cellules) avec le tampon de lyse mixte de l’étape 2.2. Homogénéiser la solution en pipettant également lentement pour éviter les bulles. Incuber dans l’obscurité à température ambiante pendant 5 min avec agitation douce. Re-homogénéiser la solution en pipettant également lentement.
- Ajouter 1 μL de 2 μg/mL colorant Cy3 NHS-ester. Homogénéiser la solution en pipettant également lentement pour éviter les bulles. Incuber pendant 5 min dans l’obscurité à température ambiante avec agitation douce.
NOTE : Le pH élevé de ce tampon provoque la neutralisation des résidus de lysine dans les protéines, conduisant à une plus forte réactivité. - Répétez l’ajout de 1 μL de 2 μg/mL colorant Cy3 NHS-ester. Homogénéiser la solution en pipettant également lentement pour éviter les bulles. Incuber pendant 10 min dans l’obscurité à température ambiante avec agitation douce.
Remarque : Cette scène reproduit addition de colorant NHS-ester peut accroître l’efficacité étiquetage parce que certains de la teinture de Cy3 NHS-ester est hydrolysé et désactivé par le pH élevé de la mémoire tampon de réaction. - Ajuster le pH 7,2 en ajoutant 100 μL de 0,8 M 4-(2-hydroxyethyl)-1-piperazineethanesulfonic acide (HEPES)-NaOH pH 7,2 à étancher la réaction. Homogénéiser la solution en pipettant également lentement pour éviter les bulles.
- Ajouter 20 μL de 19 mg/mL biotine-(polyéthylène glycol (PEG))2-amine et 5 μL de 20 mg/mL 1-éthyl - 3-(3-dimethylaminopropyl) carbodiimide (EDC). Homogénéiser la solution en pipettant également lentement pour éviter les bulles. Incuber pendant 1 heure dans l’obscurité à température ambiante avec agitation douce.
Remarque : Cette étape biotinylation est nécessaire de fixer les molécules de protéines sur une lamelle de microscope à une densité fixe et avec aucun penchant pour les espèces de protéines. - Retirez les réactifs de marquage n’a pas réagis et concentrer l’échantillon en utilisant une colonne de l’ultrafiltration avec 10 kDa coupure, suivant les instructions du fabricant.
Remarque : Le protocole peut être suspendu ici. L’échantillon peut être stocké à-80 ° C pendant plusieurs semaines.
3. récupération et séparation de Proteome
- Ajouter 4 x tampon d’échantillon de SDS (200 mM Tris-HCl pH 6,8, SDS de 4 %, 4 % de glycérol et de 0,4 % de bleu de bromophénol).
Remarque : Pour éviter d’endommager de colorant, l’échantillon de protéines ne doit pas être chauffé comme fait par SDS-PAGE standard et doit être conservé à température ambiante. - Effectuer SDS-PAGE standard pour l’échantillon de protéine et une échelle moléculaire, à l’aide de 20 % gel d’acrylamide. Migrer de l’échantillon dans le gel à une tension constante (160 V) jusqu'à la migration avant sort juste le gel.
- Image du gel pour identifier l’emplacement des bandes protéiques (Figure 3).
- Découper les portions de gel, y compris les fractions de protéine d’intérêt à l’aide d’une lame tranchante, basée sur les endroits de la bande. Fractionnement à 16, 23, 55 et 120 kDa peaks et à 19 – 25, 25-32, 32 – 42, 42-54, 54-69, 69-97, 97-136 et 136 – 226 kDa régions sont affichées à la Figure 3.
- Extrait de protéines à l’aide d’un dialyseur par électroélution avec une taille de pore de 6 – 8 kDa, suivant les instructions du fabricant.
Remarque : L’échantillon extrait peut être stockée à-80 ° C pendant plusieurs semaines.
4. préparation de lamelle de microscope
- Préparez 200 μL de tampon de l’avidine (pH 7,2, 10 avidine ng/mL et 2 mg/mL d’albumine sérique bovine (BSA) de 5 mM HEPES-NaOH) pour chaque échantillon.
- Préparer les lamelles de 22 x 22 mm et 0,15 mm d’épaisseur. Exposer les lamelles couvre-objet pour plasma utilisant un plasma cleaner pendant 1 min pour nettoyer et activer leurs surfaces d’air.
- Enrober les lamelles couvre-objet avec avidine par enduction centrifuge 200 μL de tampon de l’avidine utilisant une coucheuse essorage pendant 5 secondes à 500 tr/min, suivi de 30 secondes à 1 000 tr/min. Incuber les lamelles pendant 15 min à température ambiante pour les laisser sécher.
- Préparer les lamelles couvre-objet pour l’échantillon de protéine.
- Diluer l’échantillon de protéines (à partir de 3.5) 100 fois dans 5 mM HEPES-NaOH de pH 7,2.
- Place 100 μl de l’échantillon dilué sur la surface au centre de la lamelle avidine-enduit (de l’étape 4.3).
- Incuber dans l’obscurité à température ambiante pendant 15 minutes.
- Préparer le contrôle positif.
- Place 100 μL de 10 ng/mL purifiée fluorescent biotine en lamelle pH 7,2 au centre de l’avidine enduit 5 mM HEPES-NaOH (de l’étape 4.3).
- Incuber dans l’obscurité à température ambiante pendant 15 minutes.
Remarque : Ce contrôle positif lamelle offre la densité de l’avidine points de liaison, qui sert à calculer le LO.
- Préparer le contrôle négatif.
- Spin-manteau le plasma nettoyer lamelle couvre-objet avec 200 μL de 5 mM BSA HEPES-NaOH et 2 mg/mL (sans avidine).
- Incuber pendant 15 min à température ambiante pour sécher.
- Ajouter 100 μL de 10 ng/mL purifiée fluorescent biotine en 5 mM HEPES-NaOH de pH 7,2.
- Incuber dans l’obscurité à température ambiante pendant 15 minutes.
Remarque : Cette lamelle de contrôle négatif fournit la quantité de liaisons non spécifiques de la biotine fluorescent à la lamelle sans avidine.
- Rincer chaque lamelle avec 200 μL d’eau distillée en pipettant également, sur les bords de la lamelle de manière à ne pas toucher au milieu de la lamelle avec la pointe. Répétez ce lavage trois fois.
Remarque : Après l’aspiration de l’eau sur la lamelle couvre-objet, l’eau doit être placé immédiatement sur la lamelle couvre-objet pour l’empêcher de se dessécher complètement. - Exposer le plasma d’air avec le même nombre de lamelles comme à l’étape 4.2.
- Placer la lamelle nettoyée sur la lamelle échantillon lié à l’étape 4.7 afin d’éviter le séchage.
5. l’observation en microscopie de fluorescence seule molécule
- Démarrage d’un microscope à fluorescence champ grand-angulaire ou évanescente.
Attention : Direct ou rayonnement laser épars peut entraîner des yeux ou des dommages de la peau. Porter des lunettes de protection appropriées et recouvrez le trajet de la lumière laser.
Remarque : Pour la configuration de microscope, veuillez vous référer à notre précédente publication16. En bref, un microscope à fluorescence champ grand-angulaire ou évanescente équipé d’une grande puissance 488 nm laser d’excitation, un objectif à grande ouverture numérique et une caméra de haute sensibilité peut être utilisé pour cette mesure. En outre, pour une mesure automatique, un stade d’échantillon translationnelle XY contrôlé par ordinateur, des volets mécaniques pour les excitations de laser et un système autofocus sont recommandés pour être installé. - La valeur de la lamelle sur le microscope et trouver le focus.
- Effectuez un scan de tuile pour obtenir au moins 100 images.
Remarque : L’illumination laser doit être contrôlée avec des volets mécaniques d’être exposés à l’échantillon uniquement lors de l’enregistrement des images avec l’appareil photo, afin de minimiser le Photoblanchiment. S’il y a beaucoup d’échantillons, mesure automatique en utilisant un programme informatique pour contrôler les périphériques du microscope est recommandé. Chaque image comprend généralement des taches seule molécule 100 – 500 lorsque vous utilisez un objectif de 60 x.
6. analyse et extraction d’informations de l’image
- Si l’image a inégalité en raison du modèle d’illumination laser, effectuer le traitement d’images pour le corriger18.
- Acquérir une image de référence en mesurant avec un échantillon de la lamelle couvre-objet composé des colorants uniformément répandue en utilisant une caméra définissant le même comme dans l’étape 5.3.
- Acquérir une image décalée en mesurant avec aucune excitation de laser en utilisant le même réglage de l’appareil.
- Soustraire à chaque valeur de pixel de chaque image de l’échantillon et l’image de référence avec la valeur de l’image décalée.
- Diviser chaque valeur des pixels dans les images d’exemple soustraite de la valeur à l’image de référence soustraites.
- Recueillir les images correctement reçues et supprimer l’arrière-plan de l’image.
- Supprimer les images contenant de gros agrégats fluorescents manuellement.
- Supprimer l’arrière-plan de l’image en appliquant l' algorithme de balles roulantes19 avec un rayon de la boule de 50 pixels à l’aide de ImageJ20.
- Netteté des images en soustrayant les images floues avec ImageJ (affiner en fonction de l’image).
- Trouver et analyser les taches de la molécule unique en images.
- Identifier les taches de la molécule unique avec le Yen seuil algorithme21 à l’aide de ImageJ.
- Filtrer les spots avec moins de deux pixels à exclure bruit sombre de la caméra et les taches plus de 20 pixels pour exclure les agrégations des protéines à l’aide de ImageJ (gestionnaire de retour sur investissement).
- Compter le nombre de places dans chaque image. À l’aide de l’éclairage, analyser les images corrigées (étape 6.1.4), total des intensités de pixels pour chaque tache à l’aide de ImageJ.
- Calculer le taux d’occupation étiquetage (LO) à l’aide de la formule suivante :
LO = (nombre de chefs d’accusation dans l’échantillon / nombre de chefs d’accusation dans le contrôle positif) x 100 - Calculer le nombre moyen de colorants fluorescents lié à une molécule de protéine unique (colorantden̄).
- Analyser un histogramme des intensités de chaque tache.
Remarque : L’histogramme a généralement plusieurs pics, et chaque sommet représente un nombre différent de molécules de colorant lié à une protéine. Le premier, deuxième et troisième pic dans l’histogramme correspond à des molécules 1, 2 et 3, respectivement. - Calculer l’intensité moyenne de l’histogramme. Aussi calculer l’intensité maximale du pic premier en appliquant un raccord gaussien.
- Calculer n̄colorant en divisant l’intensité moyenne avec le pic d’intensité.
- Analyser un histogramme des intensités de chaque tache.
Résultats
Figure 4 représente les données d’image raw pour les fractions de différents poids moléculaires des protéines des cellules HeLa lysat, ainsi que les témoins positifs et négatifs. Bien que l’échantillon de protéine et positif contrôlent pièce 100 – 500 places par image, contrôle négatif affiche aucun ou quelques taches, montrant que le protocole inhibe suffisamment non-spécifiques des colorants à la surface de la lamelle couvre-objet. Spot intensité histogrammes pour les échantillons de protéine et le contrôle positif présentent plusieurs pics, représentant une liaison stochastique des colorants d’amines primaires en protéines et en avidine tétramère structures22, respectivement. Tous les spots ont montré le Photoblanchiment clignotant et progressif avec excitation laser continu, mise en évidence d’observation à l’échelle de la molécule unique (Figure 5 aB).
La LO est calculée le rapport entre le nombre de places dans chaque échantillon de protéine à celle dans le contrôle positif. Le LO mesurée de l’échantillon de lysat de cellules HeLa varie de 50 % pour la plus petite fraction (16 kDa) de poids moléculaire à 90 % pour la fraction de (120 kDa) de poids moléculaire plus élevée et était de 72 % pour l’échantillon entier proteome sans séparation (Figure 6). Parallèlement, n̄colorant tend à augmenter avec l’augmentation de poids moléculaire. Ces tendances de croissantes sont censées se produire en raison d’un nombre plus élevé de résidus de lysine, conduisant à une augmentation NHS-ester teindre de liaison. Par exemple, la fraction de 23 kDa a une haute valeur LO à cause du grand nombre de résidus lysine histone protéiques contenues dans cette fraction.
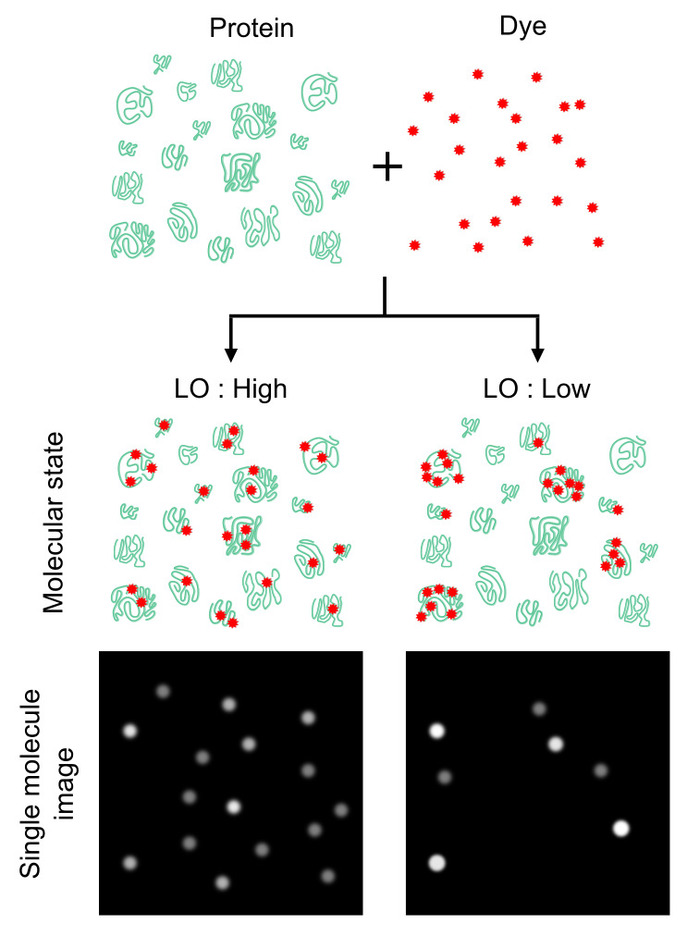
Figure 1 : effet du protéome étiquetage homogénéité sur le comptage nombre de protéine. Les nombre de protéines dépend très fortement et homogène des molécules protéiques sont étiquetés, plutôt que le ratio nombre de protéines aux colorants. L’homogénéité peut être marquée à l’aide d’un paramètre appelé étiquetage occupation (LO), qui définit la probabilité de molécules de protéines marquées contre les molécules de protéines totales. Cette valeur offre une efficacité de comptage des protéines (c.-à-d., LO rendements supérieurs comptent nombres (à gauche) et vice versa (à droite)). Alors qu’une valeur basse de 100 % est idéale, LO de < 100 % peut fournir un facteur d’atténuation pour estimer le nombre absolu de protéine. Ce chiffre a été modifié par Leclerc et coll.16 Copyright 2018 American Chemical Society. S’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.

Figure 2 : workflow Assay. Tout d’abord, lamelles couvre-objet (a) et (b) sont traités avec avidine pour atteindre une densité fixe. Deuxième échantillon fluorescent-étiqueté protéines sont biotinylé et attaché à la lamelle (un) par l’intermédiaire de l’interaction de l’avidine-biotine. En parallèle, presque 100 % fluorescent marquée purifiée biotine est immobilisé sur la lamelle couvre-objet (b). Troisièmement, les lamelles sont imagés par microscopie à fluorescence de molécules simples afin d’obtenir la distribution de nombre et de la luminosité des taches de fluorescence. Enfin, le paramètre homogénéité, LO, est calculé le rapport entre le nombre de spot dans (un) à celle de (b). Ce chiffre a été modifié par Leclerc et coll.16 Copyright 2018 American Chemical Society. S’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.
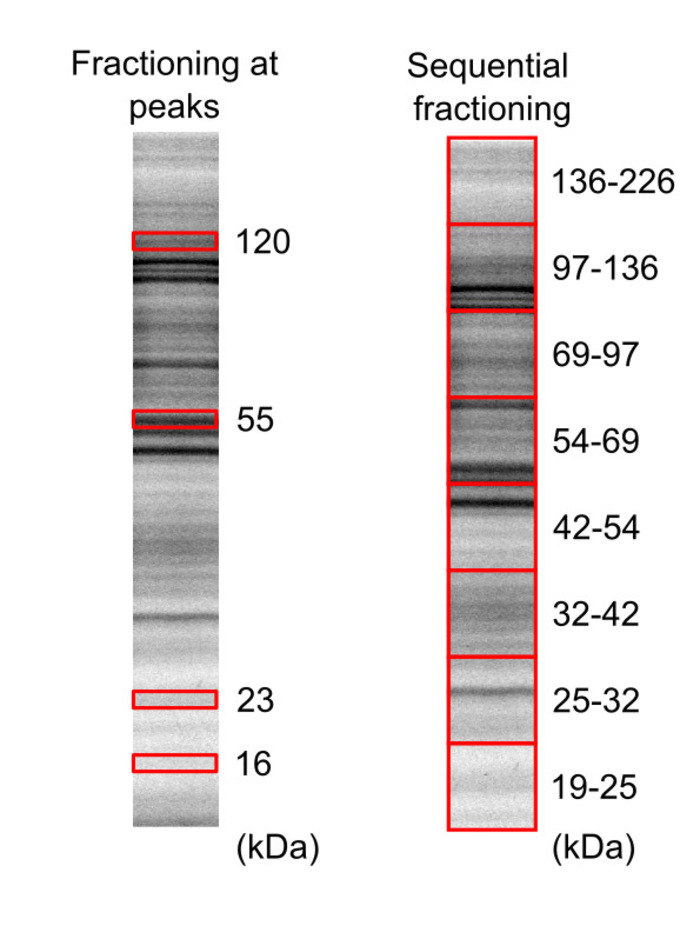
Figure 3 : séparation d’un échantillon de protéome de cellules HeLa par SDS-PAGE. Fluorescent étiqueté biotinylé lysat cellulaire ont été migrés sur deux gels différents (20 % d’acrylamide) et protéines visualisées à l’aide de la visionneuse de gel, avec un temps de pose 5 min et 10 min pour 1 de gel et gel 2, respectivement. Les cases rouges représentent les régions de gel qui ont été découpées et extraits. Ce chiffre a été modifié par Leclerc et coll.16 Copyright 2018 American Chemical Society. S’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.

Figure 4 : simples images de la molécule d’échantillons de proteome. Fluorescence spot images (à gauche) et histogrammes des intensités spots (à droite) provenant de différents poids moléculaires proteome fractions. La barre d’échelle est de 10 μm. Dans le contrôle positif, les flèches indiquent l’étape étiquetage différente de l’avidine. Ce chiffre a été modifié par Leclerc et coll.16 Copyright 2018 American Chemical Society. S’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.

Figure 5 : Photoblanchiment des taches fluorescentes. (A) des images time-lapse de taches fluorescentes sous excitation laser continu. (B) temps retrace des intensités de fluorescence à différents endroits. Les traces montrent soit Photoblanchiment one ou two - step, indiquant les numéros respectifs des colorants ont été couplés à la protéine à l’endroit de l’échantillon de BSA purifié. Ce chiffre a été modifié par Leclerc et coll.16 Copyright 2018 American Chemical Society. S’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.

Figure 6 : étiquetage d’homogénéité des échantillons de proteome. LO (bleu) et n̄colorant (rouge foncé) obtenu à partir des fractions de poids moléculaire différent. Données sont exprimées en moyenne ± images s.e. 98, 46, 27, 77, 44, 39, 62, 101, 65, 82, 61, 62 et 70 ont été analysés pour la cellule entière lysate, 16, 23, 55, 120, 19-25, 25-32, 32 – 42, 42 – 54, 54-69, 69-97, 97-136 et 136 – 226 kDa fractions , respectivement. Ce chiffre a été modifié par Leclerc et coll.16 Copyright 2018 American Chemical Society. S’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.
Discussion
Cet article décrit un protocole afin de quantifier l’homogénéité étiquetage de chaque espèce de protéines marquées dans les cellules après la séparation avec SDS-PAGE (étape 3). La méthode de séparation peut être substituée avec d’autres méthodes comme la chromatographie en phase liquide ou électrophorèse capillaire, permettant la séparation et le fractionnement des protéines cellulaires à haute résolution, tout en nécessitant un équipement spécial23. La méthode étiquetage du NHS-ester dans le protocole actuel peut être remplacée avec d’autres méthodes utilisant maléimide-ester ou des anticorps, par exemple.
L’exigence d’étiquetage analyse de l’homogénéité dépend de combien l’étiquetage s’écarte un processus aléatoire simple, comme supposé dans l’analyse de l’efficacité du couplage. Notre récente étude16 a indiqué que le NHS-ester marquage des protéines s’écarte un processus aléatoire en fonction des conditions de réaction comme le pH et les détergents. Nous avons observé que certaines conditions induites par l’hétérogénéité de liaison coopérative de colorants aux protéines ou l’existence d’une fraction de protéines non réactif en raison de la solubilisation incomplète ou abaissée d’affinité pour la teinture. En revanche, les autres conditions réduit hétérogénéité par la liaison complète des résidus réactives au sein d’une protéine à la teinture.
Nous considérons que le pH est un des facteurs plus influents sur l’homogénéité d’étiquetage. PH élevé peut causer la neutralisation des résidus de lysine dans les protéines, conduisant à une plus forte réactivité avec le colorant de NHS-ester. En outre, un pH élevé peut provoquer la lyse cellulaire et peut dénaturer les protéines. Toutefois, étant donné que le pH élevé rend la charge de protéines négatives et ne convient pas à certaines expériences comme l’électrophorèse isoélectrique ou électrophorèse 2D, la condition pH doit être soigneusement examinée selon le but de la recherche.
Pour mesurer avec précision l’étiquetage homogénéité dans le dosage, il est important d’utiliser assez échantillon de protéines biotinylées. C’est parce que l’analyse suppose que les protéines se lient avec une saturation de chaque molécule d’avidine sur la lamelle couvre-objet et les résultats concentration insuffisante de protéines sous-estimation de LO. Notre précédente étude16 a montré que plus de 10 pg de protéine est généralement nécessaire pour la liaison saturée, et cela peut provenir d’environ 1 000 de cellules HeLa24, même après fractionnement. Il est recommandé de créer une série de dilutions de l’échantillon de protéine pour confirmer la saturation des taches de l’avidine. Nous notons également que la densité de l’avidine sur la lamelle couvre-objet peut-être devoir être optimisé donc ne pas causer les chevauchements entre les taches dans les images enregistrées, tout en donnant les numéros de place suffisantes pour l’analyse statistique.
Ce test suppose que l'on avidine tétravalent molécule22 sur la lamelle peut se lier à une protéine. En effet, les données de contrôle positif dans notre précédente étude a indiqué qu’une molécule d’avidine peut lier à biotins fluorescent étiquetés jusqu'à quatre, mais la majorité (55 %) des molécules de l’avidine lier seulement une ou deux molécules de biotine. Parce que les protéines sont plus grandes que la biotine, leurs effets stériques devraient aboutir à des protéines moins liés à une avidine. Notre hypothèse est étayée par le fait que n̄colorant reste constante lorsque la mesure d’échantillons mixtes de marqués et non des protéines aux différents rapports16.
Selon l’espèce de protéine, liaison non-spécifique de protéines à la lamelle peut se produire sensiblement même avec un revêtement de BSA. Si c’est le cas, une seule option peut être pour mesurer un contrôle où les protéines biotinylées sont enduits sur la lamelle couvre-objet sans avidine, au lieu de biotine fluorescent à étape 4.6.3. L’effet de la liaison non-spécifique sur LO peut être mathématiquement éliminé en soustrayant le nombre de places de ce contrôle de celle de l’échantillon de protéine.
Ce protocole sera précieux pour proteome-échelle seule molécule protéique comptage des analyses. La sensibilité de la molécule unique permettra d’analyser des quantités de protéines pour chaque espèce de protéine indépendamment de leur abondance dans la cellule25. Cela pourrait être réalisé par séparation de proteome suivie de quantification du nombre de taches de fluorescence couplée avec des protéines. En outre, la forte sensibilité permettra aussi analyse cellulaire unique, permettant aux chercheurs d’étudier l’hétérogénéité entre les populations de cellules, et le pour faire la mise en cluster de systèmes cellulaires déclare25,26.
Déclarations de divulgation
Les auteurs déclarent les intérêts financiers concurrents suivants : RIKEN a déposé une demande de brevet sur ces résultats avec S.L. et Y.T. nommé comme des coïnventeurs.
Remerciements
Les auteurs remercient Masae Ohno et Kazuya Nishimura de conseils et d’assistance expérimentale. Ce travail a été soutenu par PRESTO (JPMJPR15F7), Japan Science and Technology Agency, subventions pour jeunes chercheurs (A) (24687022), contestant la recherche exploratoire (26650055) et la recherche scientifique sur les domaines innovants (23115005), la Japan Society for la Promotion de la Science et par des subventions de la Takeda Science Foundation et la fondation du mémorial Mochida à usage médical et la recherche pharmaceutique. S.L. reconnaît le soutien du programme RIKEN International programme Associate (IPA).
matériels
| Name | Company | Catalog Number | Comments |
| 22x22x0.15 mm coverslip | VWR | 470019-004 | |
| 488 nm Argon laser | Coherent | Innova 70C | |
| 488 nm dichroic mirror | Semrock | FF495-Di03 | |
| 488 nm emission filter | Semrock | FF02-520/28 | |
| 560 nm dichroic filter | Semrock | Di02-R561 | |
| 560 nm emission filter | Semrock | FF02-617/73 | |
| 560 nm fiber laser | MBP Communications | F-04306-2 | |
| 60x oil immersion lens | Olympus | PLAPON 60x | |
| Avidin | Nacalai-tesque | 03553-64 | |
| Biotin-PEG-amine | Thermo Scientific | 21346 | |
| Biotinylated Alexa Fluor 488 | Nanocs | ||
| Borate | Nacalai-tesque | ||
| BSA | Sigma-Aldrich | A9547 | |
| CHAPS | Dojindo | C008-10 | |
| Cy3 NHS-ester dye | GE Healthcare | PA13101 | |
| Dialyzer - D-tube, 6-8 kDa | Merck Millipore | 71507-M | |
| DMEM | Sigma-Aldrich | ||
| DTT | Nacalai-tesque | 14112-94 | |
| EDC | Nacalai-tesque | ||
| EMCCD camera | Andor | IiXon 897 | |
| Epi fluorescence microscope | Olympus | IX81 | |
| Gel viewer | GE Healthcare | ImageQuant LAS 4000 | |
| Penicillin, streptomycine and Amphoterecin mix | Gibco | ||
| Plasma cleaner | Diener Electronic | ||
| SDS | Wako | NC0792960 | |
| Size purification column 10K | Merck Millipore | UFC5010 | |
| Tween 20 | Sigma-Aldrich | P9416 |
Références
- Wilhelm, M., et al. Mass-spectrometry-based draft of the human proteome. Nature. 509 (7502), 582-587 (2014).
- Guo, A., et al. Immunoaffinity enrichment and mass spectrometry analysis of protein methylation. Molecular & Cellular Proteomics. 13 (1), 372-387 (2014).
- Tyanova, S., Temu, T., Cox, J. The MaxQuant computational platform for mass spectrometry-based shotgun proteomics. Nature Protocols. 11 (12), 2301-2319 (2016).
- Neuhoff, V., Arold, N., Taube, D., Ehrhardt, W. Improved staining of proteins in polyacrylamide gels including isoelectric focusing gels with clear background at nanogram sensitivity using Coomassie Brilliant Blue G-250 and R-250. Electrophoresis. 9 (6), 255-262 (1988).
- Berggren, K., et al. Background-free, high sensitivity staining of proteins in one- and two-dimensional sodium dodecyl sulfate-polyacrylamide gels using a luminescent ruthenium complex. Electrophoresis. 21 (12), 2509-2521 (2000).
- Butt, R. H., Coorssen, J. R. Coomassie blue as a near-infrared fluorescent stain: a systematic comparison with Sypro Ruby for in-gel protein detection. Molecular & cellular proteomics. 12 (12), 3834-3850 (2013).
- Nanda, J. S., Lorsch, J. R. Labeling a protein with fluorophores using NHS ester derivitization. Methods in Enzymology. , 87-94 (2014).
- Kim, Y., et al. Efficient site-specific labeling of proteins via cysteines. Bioconjugate chemistry. 19 (3), 786-791 (2008).
- Dickson, R. M., Cubitt, A. B., Tsien, R. Y., Moerner, W. E. On/off blinking and switching behaviour of single molecules of green fluorescent protein. Nature. 388 (6640), 355-358 (1997).
- Penna, A., et al. The CRAC channel consists of a tetramer formed by Stim-induced dimerization of Orai dimers. Nature. 456 (7218), 116-120 (2008).
- Ji, W., et al. Functional stoichiometry of the unitary calcium-release-activated calcium channel. Proceedings of the National Academy of Sciences of the United States of America. 105 (36), 13668-13673 (2008).
- Ulbrich, M. H., Isacoff, E. Y. Subunit counting in membrane-bound proteins. Nature Methods. 4 (4), 319-321 (2007).
- Sugiyama, Y., Kawabata, I., Sobue, K., Okabe, S. Determination of absolute protein numbers in single synapses by a GFP-based calibration technique. Nature Methods. 2 (9), 677-684 (2005).
- Puchner, E. M., Walter, J. M., Kasper, R., Huang, B., Lim, W. A. Counting molecules in single organelles with superresolution microscopy allows tracking of the endosome maturation trajectory. Proceedings of the National Academy of Sciences of the United States of America. 110 (40), 16015-16020 (2013).
- Grassart, A., et al. Actin and dynamin2 dynamics and interplay during clathrin-mediated endocytosis. The Journal of Cell Biology. 205 (5), 721-735 (2014).
- Leclerc, S., Arntz, Y., Taniguchi, Y. Extending Single Molecule Imaging to Proteome Analysis by Quantitation of Fluorescent Labeling Homogeneity in Complex Protein Samples. Bioconjugate chemistry. 29 (8), 2541-2549 (2018).
- Ian Freshney, R. Culture of Animal Cells: A Manual of Basic Technique and Specialized Applications. , John Wiley & Sons. (2015).
- Taniguchi, Y., et al. Quantifying E. coli proteome and transcriptome with single-molecule sensitivity in single cells. Science. 329 (5991), 533-538 (2010).
- Sternberg, Biomedical Image Processing. Computer. 16 (1), 22-34 (1983).
- Rueden, C. T., et al. ImageJ2: ImageJ for the next generation of scientific image data. BMC Bioinformatics. 18 (1), 529(2017).
- Yen, J. C., Chang, F. J., Chang, S. A new criterion for automatic multilevel thresholding. IEEE Transactions on Image Processing. 4 (3), 370-378 (1995).
- Livnah, O., Bayer, E. A., Wilchek, M., Sussman, J. L. Three-dimensional structures of avidin and the avidin-biotin complex. Proceedings of the National Academy of Sciences of the United States of America. 90 (11), 5076-5080 (1993).
- Mostovenko, E., Hassan, C., Rattke, J., Deelder, A. M., van Veelen, P. A., Palmblad, M. Comparison of peptide and protein fractionation methods in proteomics. EuPA Open Proteomics. 1, 30-37 (2013).
- Volpe, P., Eremenko-Volpe, T. Quantitative studies on cell proteins in suspension cultures. European Journal of Biochemistry. 12 (1), 195-200 (1970).
- Huang, B., et al. Counting low-copy number proteins in a single cell. Science. 315 (5808), 81-84 (2007).
- Johnson, A. C., Bowser, M. T. H. igh- High-Speed, Comprehensive, Two Dimensional Separations of Peptides and Small Molecule Biological Amines Using Capillary Electrophoresis Coupled with Micro Free Flow Electrophoresis. Analytical Chemistry. 89 (3), 1665-1673 (2017).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon